纳米孔测序提供长读取和快速提高准确性,有可能颠覆DNA测序市场。

克里斯托弗•梅森(Christopher Mason)有一个他喜欢在会议上施展的技巧。通过从志愿者手机上采集的棉签中提取DNA,他和他的同事可以在一小时内进行现场祖先分析,甚至可以讲述捐赠者当天的细节。梅森是纽约市威尔康奈尔医学院的计算生物学家,他说:“我们能够通过手机上的信息预测谁刚刚吃了一个橙子,谁吃了猪肉。”
梅森使用一种名为MinION的手持测序设备实现了这种快速分析,该设备由英国牛津纳米孔技术公司(ONT)开发。MinION通过将长DNA链穿过一个被称为纳米孔的小孔来读取序列信息,并检测由DNA的四个核苷酸组成的电流的微小变化。梅森的演示轻松地展示了该设备的功能,但早期用户也取得了一些引人注目的科学成就。MinION在监测2015年埃博拉病毒爆发方面发挥了重要作用,它已经航行到南极洲,甚至进入了轨道。
但是MinION——大约有一副扑克牌那么大——在世界测序输出中所占的比例相对较小,这一领域仍然由加州圣地亚哥的Illumina公司主导。Illumina领先了近10年,但ONT及其用户也在努力应对技术挑战,最显著的是较高的错误率。与此同时,竞争对手都希望通过这种概念简单但技术复杂的测序策略的创新来超越ONT。
一个艰难的开始
Illumina无处不在的DNA测序技术通过连续读取DNA复制反应中纳入样本的碱基,产生大量极其精确的“短读”。这些序列数据串横跨几百个核苷酸,然后可以通过计算组装成包含数百万个核苷酸的重叠“contigs”。
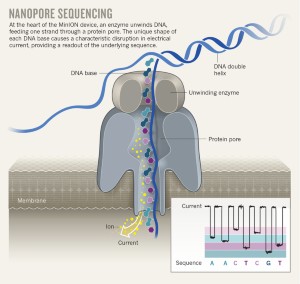
对于纳米孔测序,整个DNA片段直接通过物理穿线穿过纳米孔进行分析。
MinION的流式细胞每个成本为500-900美元,包含数百个纳米孔,因此它们可以并行分析许多分子。该系统在每个孔上施加电压,当一种酶稳定地将DNA通过时,核苷酸会阻止离子流动,并在电流中产生微小的变化,这些变化由专门的软件解释(参见“纳米孔测序”)。由此产生的“长读取”可以跨越数千个核苷酸,简化了组装。
在2014年发布时,基因组学社区被MinION的潜力所吸引,但早期用户遇到了许多挑战。英国伯明翰大学(University of Birmingham)微生物基因组学家、MinION最早的使用者之一尼古拉斯·洛曼(Nicholas Loman)说:“仅仅对单个细菌基因组进行测序需要很多努力,因为输出量很低,而且单次读取的准确性也很低。”Illumina对单个碱基的平均准确率通常达到99.9%,而第一代MinION则错误地识别了大约三分之一的碱基。还有其他问题。梅森说:“你会得到一个流动细胞,这很神奇,但你会得到一个只给你三个活跃毛孔的流动细胞,没有明确的原因,即使它们来自同一批次。”ONT不希望对本文发表公开评论。
这些限制将MinION主要局限于速度和简单性最重要的应用程序,例如病原体检测。位于马里兰州贝塞斯达的美国国家人类基因组研究所(US National Human genome Research Institute)基因组信息学主管亚当•菲利普(Adam Phillippy)表示:“如果你想问一封信中是否有炭疽病毒,你可以在MinION上很快地做出来,即使没有完美的读取精度。”
跨越式发展
如今,MinION已经成熟了,马萨诸塞州剑桥市药物研发公司Warp Drive Bio的首席科学家基思·罗宾逊(Keith robinson)说。更重要的是,ONT克服了上市前发布公告后的怀疑和轻蔑批评。罗宾逊说:“他们已经证明了所有这些人都错了,而且一再兑现了承诺。”
从细菌中提取的孔蛋白大肠杆菌,再加上流池化学技术的改进,在许多实验中,单个读数的错误率已降至2-5%。数据输出的巨大提升使研究人员能够通过并行测序更多分子来更好地识别错误,读取长度从最初的约7000个核苷酸跃升到今天的10万个核苷酸以上。洛曼的团队已经将单读长度提高到近100万个碱基。加拿大多伦多安大略癌症研究所的生物信息学家Jared Simpson说:“在我们早期的测试中,我们每次测试都能得到几百兆的序列。”“有了新的孔隙,我们很快就看到序列上升到千兆字节,现在可能每运行5或10千兆字节。”
该平台还得益于积极的软件开发。长读取比短读取更容易组装,因为有更多的重叠,但纳米孔读取也有更多的错误,操作较长的序列可能需要大量的计算。为了解决这个问题,Phillippy的团队设计了一种名为Canu的算法,在这种算法中,传统的短读组装过程被量身定制,以弥补长读数据的怪癖。另一个软件工具,ONT的Scrappie,可以解决当均聚物(包含多个相邻核苷酸实例的序列,如AAAAA)导致系统中断时可能出现的序列错误。
与微生物匹配
MinION在传染病研究人员中特别受欢迎。例如,洛曼与世界病毒学“热区”的同事合作,监测西非的埃博拉病毒和巴西的寨卡病毒的传播情况1,2.加州大学圣克鲁兹分校的生物物理学家马克·阿克森(Mark Akeson)说:“他们基本上能够在48小时内建立并运行一个测序实验室,这些实验室装在你可以带到飞机甲板下的行李里。”他在纳米孔测序方面进行了一些基础研究,也是ONT咨询委员会的成员。洛曼说,这种可移植性是一个巨大的福音,但他指出,大量的数据输出可能是压倒性的。“我们在巴西勉强做到了,但我的Mac过热了!”
一些研究小组正在探索临床微生物学应用。澳大利亚圣卢西亚昆士兰大学的生物信息学家Lachlan Coin开发了实时数据分析算法,用于检测血液样本中的耐药细菌。在使用培养的细菌和较老的流式细胞进行的早期试点测试中,科因的团队可以在10小时内识别出样本中的所有耐抗生素基因3..他说,目前的技术可以将这个时间缩短一半,但在现实世界的样本中,人类DNA超过了细菌DNA,这使得这个过程变得复杂。“我认为在一年左右的时间里,我们将能够在6小时内识别出患者样本中的抗生素抗性基因,”科因说。
其他研究人员正在研究宏基因组学,其目标是全面分析样本中的所有生物。原则上,流式细胞中的每个纳米孔都可以同时用于检测不同的基因组。“你可以得到那里物种的完整基因画像——细菌、病毒、人类DNA,”梅森说。他利用纳米孔测序技术对臭名昭著的肮脏的纽约地铁系统进行了宏基因组普查,并雄心勃勃地计划探索更不适宜居住的环境——包括火星。梅森与美国国家航空航天局的科学家合作,证明了MinION可以在国际空间站的零重力条件下表现强劲。他和他的同事希望有一天能将这项技术运送到这颗红色星球上,在那里这项技术可以帮助正在进行的对地外生命的搜索。
回到地球上,伯灵顿佛蒙特大学的遗传学家Scott Tighe在南极洲的麦克默多干谷运行MinION,在那里他的团队花了两个多小时对微生物样本进行测序。“停止跑步的原因是外面太冷了:电池最终耗尽了,”梅森解释道,他曾与Tighe合作过几个项目。
要么做大,要么回家
像Phillippy这样的纳米孔专家认为微生物基因组组装是“一个已解决的问题”。现在他们把目标对准了更大的目标:包含数十亿而不是数百万个核苷酸的哺乳动物基因组。今年,包括Phillippy、Loman和Simpson在内的一个多机构团队报告了仅使用MinION数据就组装了整个人类基因组,实现了高度的连续性和准确性4.Simpson说,平均contig大小达到了兆级,准确率高达99.44%。Illumina短读技术的补充使用使团队的准确度提高到99.96%,尽管这仍然落后于组装项目通常追求的99.99%的“黄金标准”准确度。
然而,在人类基因组分析的其他方面,纳米孔确实有优势。例如,人类基因组组装仍然不完整,因为高度重复的区域对短读分析有抵抗力。加州大学圣克鲁兹分校的基因组学研究员Karen Miga领导的团队表明,纳米孔可以帮助研究人员填补这些空白5.Miga的团队使用150千碱基对的读取来重建人类着丝粒——真核生物染色体紧腰处的超重复基因组延伸,这在以前代表着一个令人生畏的空白。与Miga合作的Akeson预测,真正完整的基因组序列可能只需要几年时间。
它能分辨两个氢原子大小的差别。
纳米孔分析也非常适合于绘制表观遗传标记——对单个核苷酸的微小化学修饰可以影响基因表达。大多数测序平台使用样品制备方法来消除这些标记,但纳米孔平台可以直接分析修饰过的DNA。马里兰州巴尔的摩市约翰·霍普金斯大学的辛普森和温斯顿·提姆普表示,他们可以训练软件区分甲基化胞嘧啶核苷酸和正常胞嘧啶的电子特征,准确率约为90%6.Akeson也取得了类似的成功7.阿克森说:“我们已经能够检测到我们试图看到的任何修改。”“它可以区分两个氢原子大小的差异。”
更多内容
尽管ONT在纳米孔市场上仍是孤军作战,但它也面临着一个成熟的长读取竞争对手。位于加州门洛帕克的太平洋生物科学公司(PacBio)以从测量数万个碱基的DNA片段中生成极其精确的数据而闻名。PacBio的平台比MinION更笨重,也更贵——它最小的系统Sequel大约有冰箱那么大,售价35万美元。它也不能完全达到ONT的读取长度极限。
但PacBio首席科学官乔纳斯·科拉赫(Jonas Korlach)指出,该系统可靠地提供了10-18千碱基的平均读数,最高可达100千碱基左右,PacBio在基因组学领域因其高质量的数据而受到信任。“你可以很容易地达到99.99%的准确率,”Phillippy说,他补充说,这项技术仍然是他第一次构建大型基因组组装时的首选。对于大型项目来说,它也更快:最近报道的仅用纳米孔的人类基因组组装4所用时间是PacBio机器的十倍,菲利普说。
一些用户发现纳米孔样品制备试剂盒可能令人不安地不可预测,一些DNA样品需要大量优化。罗宾逊说:“有些人做得非常好,取得了惊人的成绩,而另一些人只是在挣扎。”在2016年12月的一次演示中,ONT首席技术官Clive Brown宣布:“我们投入了大量精力……为人们提供特定样本类型的调试协议,这将帮助他们优化他们获得的产量。”
同样,该公司最大的资产之一——为提高业绩而进行的常规改造和改进——可能会让忠实用户手忙手乱。例如,ONT最新的孔隙技术给熟悉旧技术的客户带来了技术难题。洛曼说:“这种事情已经在我们身上发生过好几次了。”“这就是走在前沿的另一面——事情不会像你希望的那样持续很长时间。”
这些问题为竞争对手提供了机会。走得最远的是总部位于瑞士巴塞尔的罗氏(Roche),该公司于2014年收购了总部位于加州的纳米孔初创企业Genia Technologies。罗氏对其系统仍然保密,但Genia公司2016年的一份出版物描述了一种“合成纳米孔测序”策略,其中dna合成酶与蛋白质纳米孔耦合8.这种酶读取目标DNA,从化学标记的核苷酸中建立一个互补序列。当每个碱基被纳入生长链时,它的标签被释放出来,通过纳米孔产生独特的电信号。
罗氏测序解决方案主管尼尔•冈恩(Neil Gunn)表示,尽管这项研究早在罗氏被收购之前就开始了,但这项技术的核心原理基本上没有改变。“这非常符合产品的设计,”他说。“从那时起,我们就开始专注于提高准确性、读取速率和我们可以获得的测序长度。”Gunn指出,罗氏的平台将直接针对在体外诊断空间,目标是超越竞争对手的准确性和可重复性。罗宾逊认为罗氏的平台是一个潜在的强有力的竞争者,早期的出版物表明,对任何给定的核苷酸的准确性在78%到99%之间。“他们的设备可能会非常有趣,但魔鬼总是在细节中,”他说。“我们需要看到大规模的真实数据。”
但ONT并没有满足于现有的成绩:其最新的两款台式系统可以提供比以前型号大得多的数据量。今年3月发布的GridION基本上可以并行运行多个MinION设备。相比之下,PromethION使用了一种完全不同类型的流式细胞,是为人类基因组规模的项目而设计的。洛曼说:“很明显,他们的目标是在输出方面与Illumina平台竞争。”仪器已经交付给“早期访问”用户,但尚未公开发布任何数据。
无论这些发展如何成功,纳米孔测序技术无疑是占上风的。它为大众提供低成本、可靠的测序的承诺让研究人员兴奋不已。“作为一名计算机科学家,我总是渴望数据,”Phillippy说。“所有这些微生物实验室和所有这些大学教室都能够生成序列数据,这一想法很诱人。”
参考文献
快,j等人。自然530, 228-232(2016)。
法里亚,N. R.等人。自然546, 406-410(2017)。
曹,m.d.等。GigaSciencehttp://dx.doi.org/10.1186/s13742-016-0137-2(2016)。
Jain等人。在bioRxiv预印本http://dx.doi.org/10.1101/128835(2017)。
Jain等人。在bioRxiv预印本http://dx.doi.org/10.1101/170373(2017)。
辛普森,J. T.等。自然冰毒。14, 407-410(2017)。
兰德,a.c.等。自然冰毒。14, 411-413(2017)。
富勒,C. W.等。国家科学院学报美国113, 5233-5238(2016)。
权利和权限
关于本文
引用本文
这是DNA测序领域的王牌。自然550, 285-288(2017)。https://doi.org/10.1038/550285a
发表:
发行日期:
DOI:https://doi.org/10.1038/550285a
这篇文章被引用
Trycycler:细菌基因组的共识长读组件
基因组生物学(2021)
跨膜孔的计算设计
自然(2020)








