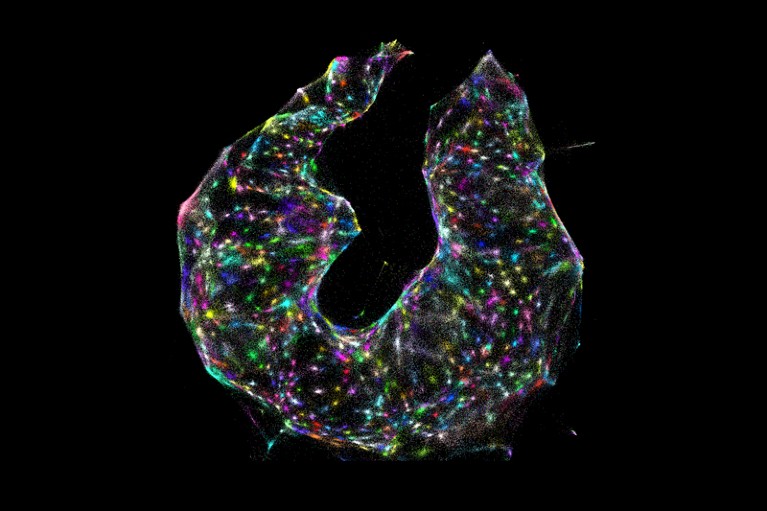
每个点都是一个单独的RNA分子,根据它与其他RNA的接近程度进行定位。这种成像方法被称为DNA显微镜,因为它使用DNA测序。资料来源:约书亚·韦恩斯坦/布罗德研究所
影迷们,空间果酱是1996年的一部喜剧电影,讲述了卡通人物兔八哥和篮球运动员迈克尔·乔丹对抗动画外星人的故事。对于神经科学家Ed Lein来说,这是一个以生物信息学为主题的聚会的名字——一种“黑客马拉松”。
今年4月,大约40名计算和转录生物学家出现在Lein工作的华盛顿州西雅图艾伦脑科学研究所。他们来这里是为了喝咖啡、写代码,还有一个共同的目标:找出日益增长的方法论工具集的优势、弱点和分析挑战原位(或空间)转录组学。
原位转录组学是一种技术的字母汤——方法包括MERFISH、seqFISH+、STARmap和FISSEQ——用于在其组织环境中绘制细胞的基因表达模式。有些依赖于杂交——短核酸探针在拥挤的细胞环境中找到补体的能力——而另一些则基于DNA测序。但所有产生概念上相似的数据-基因表达值匹配x而且y单元格坐标。
这样的数据可以揭示细胞间的关系,否则可能会被忽视,例如哪些细胞在与哪些细胞交谈,以及它们相对于结构特征和感兴趣的细胞的位置。正如计算和系统生物学家、麻省理工学院布罗德研究所和哈佛大学(马萨诸塞州剑桥市)人类细胞图谱(HCA)项目创始联合主席Aviv Regev所说:“告诉我谁是你的邻居,我就会告诉你你是谁。”
但这一领域的发展如此之快,以至于研究人员可能难以决定使用哪种方法。大量的数据分析算法、管道和文件格式使得分析和比较数据具有挑战性。莱因说:“该领域的现状是技术发展迅猛。”
在慈善组织陈-扎克伯格行动(CZI)的资助下,在HCA的赞助下,Lein和其他人于2017年组成了一个研究联盟,对不同的方法进行基准测试,称为SpaceTx——空间转录组学的缩写。与此同时,CZI的程序员开始建立一个统一的数据分析工具和文件格式,称为海星,以推进HCA的工作,并帮助更广泛的转录生物学社区。(加州雷德伍德市CZI计算生物学研究负责人杰里米·弗里曼解释说,这个名字“有点像个笑话”。许多空间方法依赖于FISH或荧光原位杂化。在编程中,星号或星号表示通配符。“笑话是,他们都是‘鱼一样的东西’。”)
海星是一款开源软件套件,可以读取图像文件,注册并去除图片中的噪声,找到斑点并识别它们在九种不同的实验策略中所代表的RNA分子,还有两种正在开发中。Lein说,这次“空间大Jam”活动是为了让开发人员和用户——空间转录组学专家自己——聚在一起讨论工作、排除故障并改进他们的方法。在这样做的过程中,研究小组暴露了一些微妙的差异,这些差异可能会让那些想要比较实验数据的人出错。但它也为如何驾驭一项快速增长的技术提供了一个模式。
原位转录组
研究基因表达的研究人员通常在大块水平上进行研究,从一块组织中提取RNA,然后对其进行整体分析。在过去的十年中,像Drop-seq这样的单细胞方法使得研究人员能够以牺牲空间细节为代价来探测细胞之间的差异。
这就是原位转录组学介入了。这些技术主要使用荧光显微镜和DNA测序来揭示组织本身细胞中RNA分子的存在和丰度。从那里,研究人员可以找出存在的细胞类型,它们的空间排列和彼此之间的关系。
雷格夫说,这就像一种水果甜点。她解释说:“如果所有的批量基因组学都是水果奶昔,那么单细胞基因组学就是水果沙拉,而空间基因组学就是水果馅饼。”“如果你从顶部看水果挞,所有的水果都被排列成这些非常漂亮的图案。”

如何构建人类细胞图谱
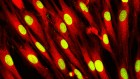
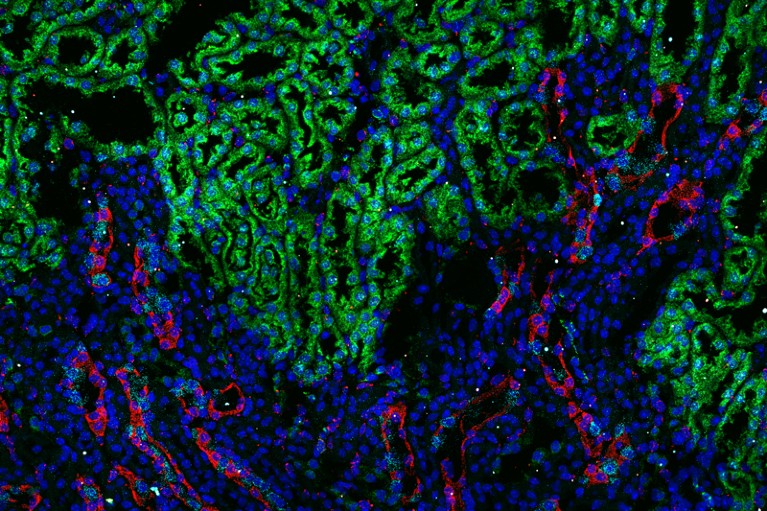
根据不同的方法,这些数据可以像漆黑天空中的星星,也可以像彩色的艺术品。例如,斯德哥尔摩卡罗林斯卡学院(Karolinska Institute) Sten Linnarsson实验室的生物图像信息学家Simone Codeluppi领导的一项研究,使用了一种单分子FISH的循环变体,称为osmFISH(读作“awesome FISH”),来绘制小鼠体感皮层的结构。结果是,这些细胞的图像根据它们的基因表达模式被着色,这张照片让人想起了彩色玻璃窗1。
但这些数据也可以揭示一些见解。在英国剑桥大学,神经生物学家和内科医生David Rowitch使用了一种叫做RNAscope的方法来研究小鼠大脑中星形胶质细胞的空间多样性和组织2。Rowitch发现,星形胶质细胞“在皮层中采用与神经元相似的层状模式,但与神经元不一致”。在帕萨迪纳市加州理工学院研究单细胞生物学的Long Cai和他的团队使用了一种名为seqFISH+的策略来识别相邻细胞表面编码相互作用蛋白质的转录本3.。
提供清晰的
seqFISH+和RNAscope都依赖于核酸杂交;他们利用荧光标记的短分子来点亮细胞中的目标序列。其他方法使用DNA测序甚至质谱法(参见“字母汤”)。
已经描述了十多种空间转录组学方法,包括2019年的六种3.- - - - - -8。它们在可检测的rna数量、空间分辨率和可探测的细胞数量上有所不同,但都能提供单细胞转录组学无法提供的空间定位细节。但雷格夫说,空间方法也有缺点。例如,显微技术速度慢(有时需要连续成像数周),价格昂贵,技术要求高。许多方法只能访问细胞转录组的预定义部分,实际的考虑可能会限制可以探测的细胞数量。
在试图为他们的工作选择正确的方法时,研究人员可能会不知所措。SpaceTx的目标是提供一些清晰度。
CZI发言人说,该项目是CZI过去两年半在HCA和辅助项目上花费的约1亿美元的一部分。每个研究小组——总共有19个——都将自己的方法应用于艾伦研究所制备的相同的人类和小鼠大脑样本。现在,Lein和他的同事们,以及更广泛的计算生物学社区,正在研究这些数据,看看这些方法之间的比较如何,以及哪种方法最适合特定的环境。
“这实际上很不寻常,”莱因说。通常情况下,研究人员致力于开发最佳方法,发表并继续研究。但对于SpaceTx,“我们试图把所有人聚集在一起,告诉他们,这些方法都很有用,但我们需要了解每种方法的用途,以及它们之间如何进行定量比较。”

NatureTech中心
但是这样做会带来计算问题,因为不同的方法产生不同的数据类型。例如,一些基于杂交的方法为每个转录本分配不同的颜色,而其他方法则使用多种颜色作为条形码。一些实验室通过跟踪每张图像中的荧光点,然后在成像轮之间监测其强度来识别RNA,而另一些实验室则测量每个像素的强度,将这些强度值与可能的条形码列表相关联,以确定是否存在RNA。这些图像在磁盘上的组织方式以及用于注释它们的“元数据”也会有所不同。
英国Hinxton欧洲生物信息学研究所的生物信息学家Matthew Green说,这种不兼容性会阻碍研究。即使他们不这样做,研究人员也常常很难安装他们同事的分析软件(由于这种软件需要复杂的计算要求和依赖性)。空间研究产生的大量数据可能令人生畏——Linnarsson的自动ossmfish设备每天大量生成2tb的图像,他说;他的团队为SpaceTx制作了大约25tb的数据。
空间果酱
由CZI的Deep Ganguli和Ambrose Carr领导的计算生物学家和软件工程师团队开始创建一个标准的文件格式和管道原位转录组分析——一种混合和匹配不同计算和湿实验室方法的方法,无论是在笔记本电脑上还是在云端。该团队甚至还去了实验室,并与生物信息学家交谈,以了解他们的工作流程。弗里曼说:“至少有一个实验室的研究生告诉Deep:‘这太棒了,因为以前从来没有人看过我的代码。’”
4月的黑客松让海星团队有机会让生物学家试用该软件,为研究人员和编码人员提供了面对面学习的机会,而不是通过代码共享平台GitHub上的错误报告。
“我们能够帮助所有这些人在海星中实现他们的数据处理管道,以帮助他们的科学努力,”CZI的海星开发负责人贾斯汀·基金斯(Justin Kiggins)说。“这让我们的团队对差距和挑战有了批判性的见解。”
加州大学圣地亚哥分校的生物工程研究生Matt Cai开发了一种叫做DARTFISH的方法,他说他在西雅图黑客马拉松有两个目标:与其他空间转录组组交换想法,并跟上海星的速度。他说:“我们有自己的内部分析方法,但它们的编写方式并不容易让人们使用。”“《海星》是为科学界写的。”
对格林来说,这次会议不同于他参加过的任何一次会议。尽管多年来他参加了多次会议,但他说:“我从来没有参加过第一次会议。”“实际上,每一次谈话都像是一次大规模的信息交换。这感觉非常令人兴奋。”
Lein说,每个团队都成功地将样本数据集转换为海星格式,数据生成仍在进行中。但软件本身仍在开发中。英国欣克斯顿威康桑格研究所(Wellcome Sanger Institute)的程序员亚历山德拉•塔科夫斯卡(Aleksandra Tarkowska)表示,由于“数据的复杂性”,她无法将自己的数据集转换成海星格式,也无法将不同的视野对齐成统一的图像。蔡龙实验室的软件工程师尼克·皮尔逊(Nico Pierson)报告了该软件的“斑点解码器”(将荧光模式与条形码匹配的算法)的问题,因为它无法处理seqFISH+数据的密度。“根据我们的数据,效率非常低,可能只有10%,”他说。
尽管如此,与会者还是称赞这次活动让程序员和生物学家们畅所欲言。程序员们带着一堆bug报告和功能请求离开,其中一些可以当场解决。研究人员带着新的、更好的数据分析思路回到实验室。例如,Codeluppi发现了一种“分割”策略,用于在他的图像数据中计算识别细胞边界,特别是对于小体积的细胞。
Matt Cai说,他的实验室现在经常运行海星和自己的计算管道来比较性能。但其他人可能不愿意放弃他们精心打造的内部管道。因此,海星可能会在那些试图实施其他人已经开发的方法的实验室中得到最大的采用。
纽约哥伦比亚大学朱克曼脑脑行为研究所的分子神经科学家阿巴斯·里兹维说:“在所有不同的方法中,有一种方法可以把它们以计算格式联系在一起,这是非常有价值的,我认为海星将允许这一情况发生。”Rizvi是HCA项目的成员之一,他正在部分使用空间方法构建人类脊髓图谱。
“这让我想起了单细胞转录组学的早期阶段,”他说。“让实验顺利进行已经很困难了,但研究数据并试图找到从中提取真正意义的方法也很令人兴奋。这就是我现在看到的领域。”

 如何构建人类细胞图谱
如何构建人类细胞图谱 人类细胞图谱:从视觉到现实
人类细胞图谱:从视觉到现实 NatureTech中心
NatureTech中心