和复杂多样的地形地貌的基因和调控序列,人类基因组通常被比作一个景观。但在许多地方,这个地形不太引人注目的vista和更多的沙漠公路:巨大的和重复性。
考虑一个染色体的着丝粒,链接两个gene-laden武器。着丝粒包含成千上万的几乎相同α-satellite序列- 171 base -对单位需要正确地组织确保染色体稳定性和细胞分裂。然而,二十年后人类基因组草案的公布,这些和其他具有挑战性的DNA特性仍顽固的差距在我们的染色体图谱。直到几年前,一些研究人员填的绝望。
贝思沙利文,着丝粒研究员达勒姆的杜克大学北卡罗莱纳回忆谈话Karen多边投资担保机构在2014年与基因组学研究员加州大学圣克鲁斯。“她告诉我,如果有什么不会发生大幅度的技术,我们要被困在这里很长一段时间的,”沙利文说。
但是确实发生了什么事情:测序技术的发展,可以阅读长段的DNA不间断。多边投资担保机构和她的同事在端粒,端粒(T2T)财团正准备完成开始的20年奥德赛与发布的第一份草稿序列。他们的目标是生产,对于每一个染色体,一个端到端的基因组地图,从一个端粒(染色体末端重复序列元素帽)。“这不仅仅是为了这样做,”多边投资担保机构说。“这是因为我觉得这里有一些很酷的生物学。基因组学”,但发现它,世界将需要许多这样的基因组序列,凿掉还不清楚这些基因变异的地区。
夹在中间
20年前的这个月出版1人类基因组的初稿是一个里程碑式的成就。但是也充满了漏洞。人类基因组计划生成大量的科学家们从染色体DNA短序列。他们和他们的邻居重叠的地方,这些都是组装成更大,连续延伸称为重叠群。理想情况下,每个染色体将是由一个叠连群,但初稿由1246个这样的片段。
里程碑在基因组测序
从那以后,科学家们作为参考基因组的一部分财团(GRC)充实大会上,手动检查和通过测序分析,识别出段错误和信息差距。人类基因组的最新版本,称为GRCh38,在2013年被释放。自那以后,一再“修补”。然而失踪5 - 10%的基因组,包括所有的着丝粒和其他具有挑战性的区域,如大量的基因编码的RNA序列形式具有细胞器称为核糖体。这些都是出现在许多很长一段时间里重复基因副本。“待结案差距的很大一部分,”亚当Phillippy说bioinformatician在美国国家人类基因组研究所的贝塞斯达,马里兰,T2T主持。基因组也穿插着hard-to-map绵延不尽相同的DNA称为节段重复——古老的染色体重组的产物。
这些具有挑战性的部分继续阻挠genome-assembly努力。这是因为大多数测序与短内容技术到目前为止已经完成,如广泛使用平台商业化生物科技公司Illumina公司在圣地亚哥,加利福尼亚。Illumina公司测序产生极其精确的数据,但通常在几百基地——太短跨度长重复和位置序列明确。说,“基因通常容易组装Kerstin豪,一个计算Hinxton Wellcome桑格研究所的生物学家,英国是集选区的一部分。“但一切,有很多重复的基因间的空间或基本上是没有可寻址的。”
到达对面的差距
两个读技术现在关闭这些空白。加州门罗公园的太平洋生物科学生物科技公司,使用一个成像系统直接读数十万甚至数百万并行DNA链,每个生成成千上万的基地。另一种方法,由英国牛津纳米孔技术商业化,线程DNA链通过微小的蛋白质,或纳米孔,阅读成千成千上万的基地通过测量电流发生细微变化作为核苷酸遍历通道。
当他们第一次推出(太平洋生物科学技术在2010年和2014年牛津纳米孔),这些技术更容易出错的Illumina公司,为个人提供了大于99%的准确率。“我们谈论15 - 20%的错误率PacBio初读、“Phillippy说。第一代纳米孔测序可能产生错误超过30%的基地。
但性能稳步提高,有了它,读长度。“在过去三、四年,我们现在可以读的长度超过100个碱基,“Phillippy说。“当我和凯伦推出这T2T财团。”
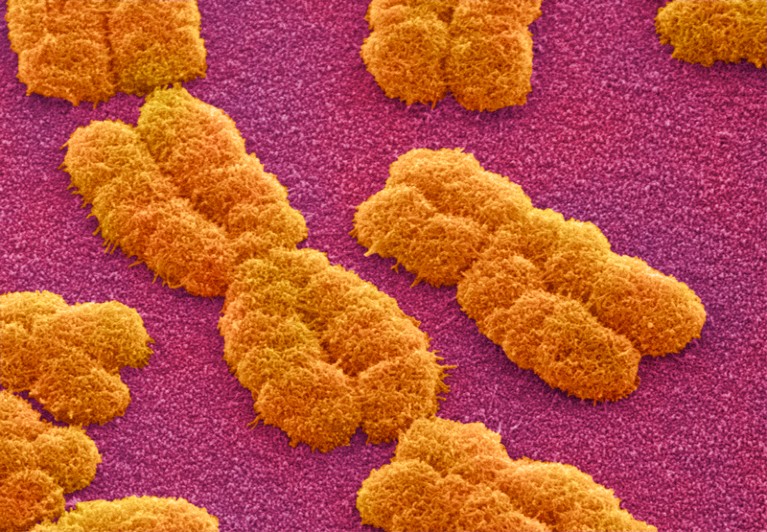
人类染色体由扫描电子显微镜成像。信贷:权力和锡/ SPL
成立于2019年初,该财团的目标是生产高质量,为每个人类染色体的端到端组件。100多个来自世界各地的测序和基因组学专家已经签署,其中许多人已经积极展示long-read-based分析的力量。
两篇论文发表在2018年强调他们的工作。在一个2计算生物学家马修·松诺丁汉大学的英国,和他的同事描述了第一个人类基因组组装完全从牛津纳米孔数据。以前读总成用Illumina公司数据正确的容易出错的纳米孔的输出。但松和他的同事们覆盖大约90%的GRCh38单独使用纳米孔数据有99.8%的准确度,同时关闭12个参考基因组的主要差距。
在第二个研究中3,多边投资担保机构和她的团队重组人类Y染色体的着丝粒,基因组最小的。它们产生大量的长阅读整个地区生成高质量的共识序列中随机误差可以很容易识别和消除。“我们可以遍历所有的方式在着丝粒,”多边投资担保机构说。“但它还是很手册在这一点上——只是观察模式和缝合在一起。”
第一个完成
这样的成功明确表示,T2T的目标是触手可及。为了简化工作,专注于CHM13财团,肿瘤提取细胞系基因组包含两套相同的染色体。这就消除了二倍体基因组的复杂性,不同的染色体来自父母。
2020年末,T2T科学家发表前两个完整的总成,为X染色体4和8(作为预印本)5。研究人员用牛津纳米孔技术序列的两条染色体长度通常超过70000基地,与一个读超过一百万个碱基。”这些,我们能够有一个骨干代表的染色体端粒和端粒,但以较低的准确性,“Phillippy说。然后补充这些数据与Illumina公司和太平洋生物科学读取波兰议会。

一个发现的财富建立在人类基因组计划——这些数字
Glennis Logsdon,实验室的一个博士后基因组科学家埃文为西雅图华盛顿大学和第一作者染色体8工作,说不同的测序技术有独特的怪癖。例如,T2T科学家已经发现太平洋生物科学化学可以斗争与G和高纯度的基因组区域基地,而纳米孔技术有时磕绊长重复相同的核苷酸。“如果一个数据集有一个缺陷,另一个没有,他们最终互补因为,“洛格斯登说。
完成和事实确认所需的程序集专业研究人员开发的软件工具,包括Phillippy和计算生物学家帕维尔Pevzner加州大学圣地亚哥。团队采取了谨慎的方法。“我们只会胶两个序列在一起如果他们基本上100%超过7000个碱基的长度相同,“Phillippy说。“一旦你引入一个错误的装配,很难解决这个问题。“但是通过这样的关心,他说,就可能产生组件在核苷酸水平与99.99%的准确率。
最初的工作4染色体X也受益于染色体的着丝点的先前知识,深入研究在结构层面。“我们使用各种大小的分子技术,以确保装配α-satellite数组的排序信息是正确的,”沙利文说。“总的来说,给我留下了深刻的印象的验证研究首次进入。”
研究人员还利用映射技术,如一个由Bionano基因组学,生物技术公司在圣迭戈,加利福尼亚,可以测量距离分离染色体DNA序列。
接近完成
虽然成功,但T2T方法染色体8和X是费力而艰苦的。但一个重要的进步在这段时间给团队的努力打了一针强心剂。太平洋生物科学仪器支持这一过程称为圆形共识测序(CCS),转化为个体的DNA链的封闭循环,可以反复阅读。通过比较这些重复读取,研究人员可以消除随机误差产生一个高度精确的结果。
早期版本的CCS超过了几千基地,限制其在基因组装配使用。但在2019年,该公司修改了这一过程6,现在产生的高保真的方法产生共识读取超过20000个碱基,准确率大于99%。“着丝粒,我们现在完全可以组装的高保真读——不需要额外的帮助,“Pevzner表示,尽管他补充说,校准算法,可以处理这些数据也是必需的。

破碎的承诺,破坏了人类基因组的研究
Pevzner比较着丝粒重建组装拼图的看似湛蓝的天空,所有作品最初出现无法区分。“有小,几乎看不见,云可以区分不同的难题,”他说。寻找那些云揭示了拼图的组织——与着丝粒和改进方法做了同样的事情,敏感地检测微妙的序列差异可以提供地标组装算法。
这种方法的组合与纳米孔的头衔就读取明显加速T2T的进步——洛格斯登报告hundred-thousand-base延伸现在是随处可见。“我们花了一年或以上的每个染色体X和8个项目,“Phillippy说,“但我们基本上能够完成所有剩下的染色体在两个月。“现在,终点就在眼前。“我们green-lit所有的着丝粒数组除了一个9号染色体上的“多边投资担保机构说。着丝粒,她说,是巨大的——跨越2700万基地,提出了特殊的挑战方面的验证。团队也仍然处于高度重复核糖体RNA基因。但该财团已经在GitHub上共享其数据,和多边投资担保机构预计,CHM13细胞系的完整基因组发布今年将到达。
数据已经产生的见解。洛格斯登和其他人使用纳米孔测序发现模式的DNA化学改性可以影响染色体的功能。“大多数的着丝粒是甲基化,但这是蘸甲基化似乎发现在所有的着丝粒,”她说。下降似乎标志着丝粒的位置,一个重要的着丝粒结构,管理相同分区的DNA在细胞分裂。洛格斯登希望利用这些发现工程师最小合成染色体着丝粒。
T2T的方法也取得了相对较短的庞大和复杂的基因阵列编码工作变量的区域表面的抗体和受体免疫系统的T细胞。“他们高度重复而且极难组装、“Pevzner说。“从今天开始,我们只有两个参考。“能够访问和描述这些具有挑战性的基因组片段可以指导努力理解感染和疫苗的免疫反应。
结束的开始
具有挑战性的,因为它已经建立一个端到端的基因组提供了研究人员有限的价值没有其他来自不同个体的基因组来比较。提高效用,在2020年末,T2T开始工作更紧密地与一个平行的努力,人类Pangenome参考财团(HPRC)。HPRC始于2019年,目标是取代GRCh38与参考基因组,更好地体现了人类的多样性的范围,基于全基因组的数据至少有350个人。“基因组医学成为常规越多,你越想要删除任何偏见,取决于一个人的祖先,“Tobias Marschall说,计算生物学家马克斯普朗克信息学在萨尔布吕肯,是来自德国的努力的一部分。
29岁铃木在实验室研究助理的计算生物学家Shinichi Morishita东京大学,利用太平洋生物科学测序研究36个人的着丝粒从日本和世界其他地区7。“只是日本人口中,我们看到不同的着丝粒几乎每个样本调查,”铃木说。“这是不够的只有一个引用,或者只是一个参考每个人口。”

NatureTech中心
Morishita计划分析数以百计的其他人类的着丝粒,他指出,几十个疾病有关的基因变异被映射到这些地区。“这表明有一些错在着丝粒重复,我们的印象是,他们的稳定可能会摧毁由于结构变异,”他说。Phillippy看到机会,更好地了解疾病相关的细胞蛋白的生产机械一旦核糖体RNA基因可以经常得到解决。
但首先,研究人员必须解决如何T2T过程应用于二倍体基因组。确定哪些序列位于染色体复制需要科学家来识别足够独特基因地标自信地为每个DNA链组装不同的重叠群,一个艰难的壮举ultra-repetitive地区如着丝点。在他们的染色体8预印本,洛格斯登,为和他们的同事们描述重建的可行性从黑猩猩和人类二倍体着丝粒区域,但只有当两条染色体高度遗传学上截然不同的。“我们需要更准确的或更长时间读取跨度为二倍体基因组完整的着丝粒区域,“Morishita说。
目前,大多数临床基因工作集中在已知基因——基因组分析快速和具有成本效益的方法。但先锋探索这种新的地形期望全面分析最终将成为一种标准,尽管可能更贵,夹具在医学和研究基因组学——特别是当研究人员开始例行公事地探索这些once-unmappable区域变化的临床影响。“如果我的孩子病了,我知道我可以得到100%的基因组与读,我想支付的区别,”多边投资担保机构说。


 一个发现的财富建立在人类基因组计划——这些数字
一个发现的财富建立在人类基因组计划——这些数字 破碎的承诺,破坏了人类基因组的研究
破碎的承诺,破坏了人类基因组的研究 未来20年的人类基因组必须更加公平,更加开放
未来20年的人类基因组必须更加公平,更加开放 非洲三百万基因组序列
非洲三百万基因组序列 突破人类的未知参考基因组
突破人类的未知参考基因组 罕见疾病的人类基因组转化研究如何
罕见疾病的人类基因组转化研究如何 从一个人类基因组复杂的祖先织锦
从一个人类基因组复杂的祖先织锦 里程碑在基因组测序
里程碑在基因组测序 DNA测序的杀手锏
DNA测序的杀手锏 NatureTech中心
NatureTech中心