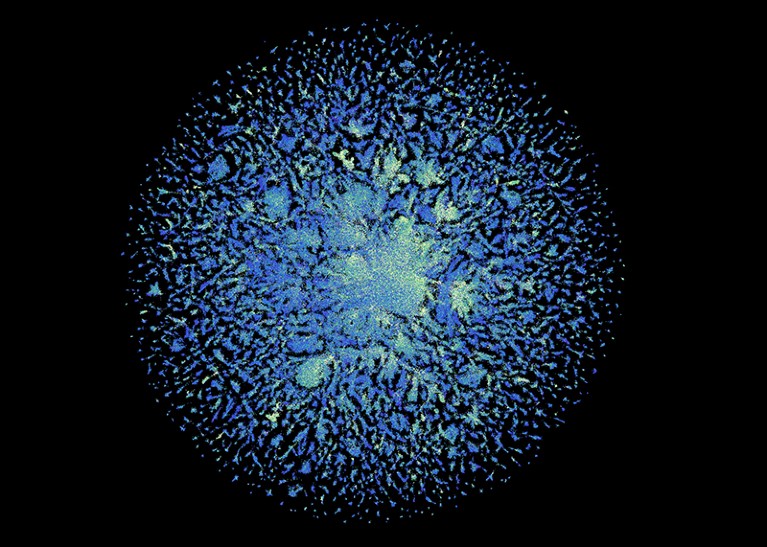
ESM宏基因组图谱包含6.17亿个蛋白质的结构预测。4.0信用:ESM宏基因组图谱(CC)
当伦敦的人工智能(AI)公司DeepMind公布今年早些时候预测蛋白质结构约2.2亿从已知的生物,宝库覆盖几乎所有的蛋白质在DNA数据库。现在,另一个科技巨头是填写蛋白质的“暗物质”的宇宙。
元研究员(以前Facebook,总部位于门洛帕克,加利福尼亚州)使用人工智能预测6亿蛋白质的结构从细菌、病毒和其他微生物,没有特点。
“它将改变一切”:DeepMind解决蛋白质结构的人工智能使巨大的飞跃
“这些都是我们最不了解的结构。这些都是非常神秘的蛋白质。我认为他们提供潜在的生物学的洞察力,”亚历山大说,元艾未未的蛋白质的研究领导团队。
生成的科学家描述的预测——11月1日预印1——用一个大的语言模型,可以预测的人工智能类型文本的几个字母或单词。
通常情况下,语言模型训练大量的文本。将它们应用于蛋白质,当和他的同事而不是美联储AI序列已知的蛋白质,可以写一系列信件,每个代表一个20的氨基酸。网络然后学会填写的蛋白质序列的氨基酸被遮住了。
蛋白质“自动完成”
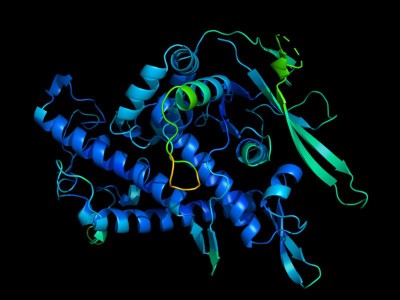
这种培训网络注入了一个直观的理解蛋白质的序列,其中包含的信息对他们的形状,当说。第二步——受DeepMind开创性protein-structure-predicting AI, AlphaFold——这样的洞察力与信息之间的关系已知蛋白质结构和序列,生成预测。
元的网络,称为ESMFold,不是像AlphaFold那么准确,当队今年早些时候报道2快,但这是约60倍在预测结构短序列,他说。“这意味着我们可以规模更大的数据库结构预测。”
作为测试,研究人员释放他们的模型的数据库bulk-sequenced元基因组的DNA从土壤等环境资源、海水和人类肠道和皮肤。绝大多数条目——潜在的蛋白质编码——来自单细胞生物体从未被隔离或培养,未知的科学。
总的来说,团队预计超过6.17亿种蛋白质的结构。努力了只有两周(相比之下,AlphaFold可以分钟生成一个单一的预测)。结构都是免费使用的,底层的代码模型,当说。

接下来是什么AlphaFold和AI蛋白质折叠革命
6.17亿年的预测,该模型认为高质量的三分之一以上,使得研究人员可以有信心,蛋白质的整体形状是正确的,在某些情况下,可以分辨原子水平的细节。数以百万计的这些结构与蛋白质结构数据库的完全是由实验决定的,或任何AlphaFold的预测从已知的生物。
大量AlphaFold数据库是由结构几乎完全相同,而宏基因组数据库”应包括很大一部分蛋白质前所未见的宇宙”,Martin Steinegger说,首尔国立大学计算生物学家。“有一个巨大的机遇现在解开更多的黑暗。”
谢尔盖Ovchinnikov,哈佛大学进化生物学家在剑桥,马萨诸塞州,奇迹的数亿预测ESMFold信心不足。有些人可能缺乏定义的结构,至少在隔离,而另一些可能非编码DNA误认为是编码蛋白质的物质。“似乎仍有超过一半的蛋白质空间我们一无所知,”他说。
更精简,更简单,更便宜
伯克哈德罗斯特,计算生物学家在德国慕尼黑工业大学,印象深刻的是,结合速度和准确度的元模型。但他质疑ESMFold是否真的提供了一个优势AlphaFold时精度预测蛋白质从宏基因组数据库。Language-model-based预测方法——包括一个由他的团队开发的3——更适合于快速确定突变改变蛋白质的结构,这与AlphaFold是不可能的。“我们将会看到结构预测变得更精简,更简单,更便宜,这将为新的东西开门,”他说。
DeepMind目前没有计划在其数据库包括宏基因组结构的预测,但不排除增加他们将来的版本中,根据公司的代表。但Steinegger和他的合作者已经使用的一个版本AlphaFold预测约3000万元基因组蛋白质的结构。他们希望找到新的类型的RNA病毒通过寻找未知形式的病毒的genome-copying酶。
Steinegger看到拖网生物学的暗物质等明显的下一步的工具。“我认为我们将很快有一个爆炸的分析这些宏基因组结构。”

 接下来是什么AlphaFold和AI蛋白质折叠革命
接下来是什么AlphaFold和AI蛋白质折叠革命 “整个宇宙蛋白质”:人工智能预测的形状几乎所有已知的蛋白质
“整个宇宙蛋白质”:人工智能预测的形状几乎所有已知的蛋白质 “它将改变一切”:DeepMind解决蛋白质结构的人工智能使巨大的飞跃
“它将改变一切”:DeepMind解决蛋白质结构的人工智能使巨大的飞跃