分类是一种机器学习的方法建立预测模型,可以分类样本正确分类。在微生物研究中,这些类别包括疾病和栖息地。在微生物生态学的问题是正确的层次的分析最好使用为了区分生物相关的样本。许多研究使用16 s rRNA基因分类标记,然后问如何有效分类概要文件从这个标记或集群不同的微生物群落进行分类根据样品类型。有趣的是,答案可能取决于被问到的问题。系统发育分析,不同级别的分辨率不同分组中成功在不同的分类任务。这些分类任务包括分离他们来自不同样本的人(这取决于细非常密切相关的菌株或物种之间的区别),和分离精益肥胖者(非常广泛组类群更有效)(骑士et al ., 2011 b)。
相关的争议是分类是否正确的分析级别:可能我们不期望函数而不是将更多的重要的生物意义的组分类样本比提供这些功能是谁?例如,一对草原是立即区分一双森林只要看看他们。这是真的,即使植物构成相关的草原和森林相关不密切相关的另一个系统。我们可能期望相同的微生物世界是真实的。因此,我们可能期望分类微生物群落的分子的成员函数将提供歧视性的力量远比看分类资料,特别是因为分类概要文件是非常变量在功能概要文件的情况下更稳定(恩伯et al ., 2008;人类微生物组工程协会,2012)。这可能是低噪音在解释功能配置;另一方面,功能配置文件包含更少的变化,所以也许有更少的共变临床或环境重要参数来解释。
最近,发展的方法来预测功能概要文件从相同的数据分类档案,PICRUSt (Langille et al ., 2013),使我们能够解决这个问题。PICRUSt是一个工具,用它可以预测基因标志基因的微生物群落调查的内容,使用现有的微生物基因组数据库。骑士et al。使用数据集出版,包括科斯特洛et al。身体的栖息地(CBH),科斯特洛et al。皮肤网站(CSS),科斯特洛et al。主题(CS)、菲勒et al。主题(FS)和菲勒et al。Subject-Hand (FSH),有效地问我们可以准确地分类样本身体不同的部位,不同的个人,不同的临床状态(骑士et al ., 2011 a)。使用相同的数据集,我们问是否所预测的功能配置PICRUSt提供更好或更差的能力根据生物学上有意义的类别分类样本的随机森林分类器。随机森林是一个符合一系列的系综分类方法决策树的次级样本数据集,然后将结果以提高分类精度。关键的输入特性(操作分类单元(辣子鸡)或基因在这种情况下)可以通过他们的贡献排名在区分不同类别的样本(补充数据S1和S2和补充表S1和S2)(Liaw维纳,2002;库恩,2008)。
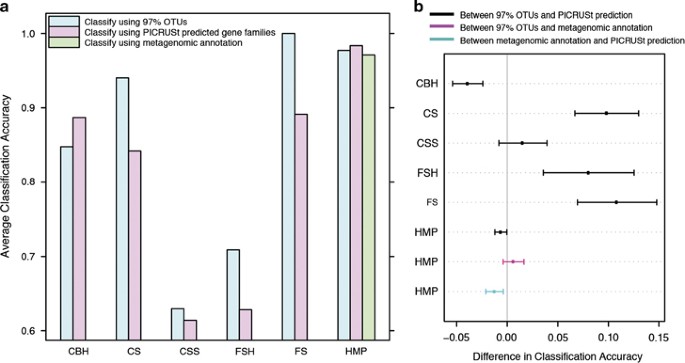
结果是有趣的(图1功能分类):在一个任务表现的更好,CBH(最简单的分类任务),更糟的是在三个,CS, FS和FSH,而不是在最后一个明显不同,CSS。明显的具有挑战性的分类任务差的精度,CSS和FSH、分类组成类之间的差异在哪里微妙,PICRUSt-predicted功能概要文件不提供任何改善在微生物组成数据。
监督分类的准确性。执行随机森林分类模型,使用插入符号(库恩,2008)R包,5重复10倍交叉验证CBH, CS, CSS, FSH, FS(从骑士et al ., 2011 a和遵守相同的命名)和HMP(从Langille et al ., 2013;其他数据集与配对猎枪和16 s序列在样本量太小分类)数据集。这里使用Kappa统计测量的精度评估预测分类和现实之间的协议。(一个)的平均精度使用辣子鸡集群作为输入序列相似度97%,预测功能概要文件从辣子鸡使用PICRUSt或注释猎枪宏基因组序列数据集(HMP)。(b)之间的两两比较使用这三个输入精度的预测功能。误差线表明95%置信区间。
添加功能信息,并不能提高分类精度是令人惊讶的。然而,一个可能的原因缺乏改善相对于分类,我们需要控制的功能预测可能质量不足。为了验证这个假设,我们使用人类微生物组计划(HMP)数据集从PICRUSt纸,成对的猎枪宏基因组注释和16 s rRNA可用同一样本配置文件数据。据推测,从宏基因组注释功能概要文件是比这更好的功能描述一个微生物群落与PICRUSt推断。这个数据集的分类基于PICRUSt-predicted函数实际上是略优于基于16 s概要文件和宏基因组注释,后者虽然没有显著区别两个(图1)。因此,我们可以得出结论,对环境有足够的参考基因组已经在数据库中,PICRUSt提供信息,有利于分类直接猎枪读取的功能任务,虽然在这两种情况下功能信息似乎并没有提高分类精度。
结果有几个复杂的微生物群落生态学研究的重要意义。首先,猎枪宏基因组和其他功能的研究仍远高于16 s rRNA剖析,但实际上可能提供更糟糕的结果,如果我们的目标是获得生物标志物为特定生理或生态状态。multi-omics研究其次,不同级别的功能可能需要检查经验理解提供最好的biomarkers-the研究高分子聚合物在执行数据,检查16 s rRNA和猎枪数据,应该被重复multi-omics水平获得,例如,在HMP2。最后,底层功能的数据库,它目前只提供相对粗粒度功能任务,可能需要大幅提高在我们能够使用功能基因环境分类的分类标记已经成功,特别是对环境弱势的数据库。当然,还有其他原因做猎枪宏基因组,从装配的小说的微生物基因组strain-level跟踪随着时间的推移,和功能分配的猎枪宏基因组或PICRUSt预测可以为获得非常有价值的功能了解给定的样本集。然而,改善我们的样本为生理上有意义的类别进行分类的能力显然不是在追求功能的原因,而不是分类,微生物群落的特征。不过,新技术和更好的生物信息学工具,如测序读长,更好的注释数据库工具更好地预测基因操纵子的结构,和工具,询问单核苷酸polymorphism-level数据将是至关重要的,提供更详细和准确的功能注释。这些注释将区分更加微妙的微生物样本之间的差异,并帮助我们理解微生物世界。
引用
人类微生物组项目财团。(2012)。结构、功能和人类健康的微生物多样性。自然486年:207 - 214。
骑士D,科斯特洛埃克,骑士R。(2011)。人类微生物群的监督分类。《牧师35:343 - 359。
骑士D, Parfrey LW, Zaneveld J, Lozupone C,骑士R。(2011 b)。Human-associated微生物签名:检查他们的预测价值。细胞宿主细菌10:292 - 296。
库恩M。(2008)。构建预测模型在R使用脱字符号包。J Stat Softw28:1-26。
Langille MGI Zaneveld J, Caporaso詹,麦当劳D,骑士D,雷耶斯农协等。(2013)。预测微生物群落的功能分析使用16 s rRNA标记基因序列。生物科技Nat》31日:814 - 821。
Liaw,维纳米。(2002)。由randomForest分类和回归。R新闻2:在18到22岁的。
恩伯PJ, Hamady M, Yatsunenko T, Cantarel提单,邓肯,雷再保险等。(2008)。肥胖和苗条的核心肠道微生物组双胞胎。自然457年:480 - 484。
确认
这项工作的部分支持由美国国立卫生研究院、美国国立正义,NSF智商生物学训练格兰特和霍华德休斯医学研究所。
作者信息
作者和联系
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
补充信息伴随着ISME日报网站上的这篇论文
补充信息
权利和权限
这项工作是基于知识共享署名3.0 Unported许可证。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在信贷额度;如果材料不包括在Creative Commons许可下,用户需要获得许可证持有人的许可复制的材料。查看本许可证的副本,访问http://creativecommons.org/licenses/by/3.0/
关于这篇文章
引用这篇文章
徐,Z。,Malmer, D., Langille, M.et al。微生物群落分类:哪个更重要或他们所能做的是谁?。ISME J8,2357 - 2359 (2014)。https://doi.org/10.1038/ismej.2014.157
发表:
发行日期:
DOI:https://doi.org/10.1038/ismej.2014.157
本文引用的
机器学习和深度学习在微生物研究的应用程序
ISME通信(2022)
人类urobiome
哺乳动物的基因组(2021)
bug和药物:系统生物学的方法来描述该药在马的粪便微生物的影响
动物微生物(2020)
Self-reinoculation粪便菌群的微生物群密度和成分的变化导致胆汁酸的改变在鼠标小肠
微生物组(2020)
叫人类皮肤微生物分析
微生物组(2019)