在这里,我们评论三个方面的最近的一份报告ISME J(相关的et al ., 2013),它认为:基因组的草案Smithella我1缺乏基因编码alkylsuccinate合成酶(屁股)子单元;因此它不表达这些基因在增长n -十六烷在混合文化产甲烷条件下;它可以使用另一种机制启动厌氧n十六烷退化。我们重新分析了草案的基因组Smithella我1和3个metatranscriptomes,达到的结论相反Embree et al。(2013)。
生物降解的n烷烃在有氧条件下易于理解和通路硝酸盐和硫酸盐还原条件下被阐明。相比之下,在产甲烷条件下完成n -烷烃降解需要财团的古菌和细菌;后者尚未分离,因此细菌代谢途径在很大程度上是未知的(艾特肯et al ., 2013;卡拉汉,2013)。理解产甲烷n烷烃降解是至关重要的优化生物修复在厌氧环境中,建模形成严重生物降解石油油藏和潜在转换未还原的残余碳氢化合物在石油储层为复苏甲烷作为宝贵的燃料。
厌氧的最广泛的报道机制n烷烃降解是由碳氢化合物延胡索酸酯。硝酸盐和硫酸盐还原条件下初始hydrocarbon-activating反应由氨基乙酰基自由基催化酶屁股,编码的assABC基因(卡拉汉,2013)。由于高的序列相似性和保护亚萨合基因编码的α-subunit屁股,它的存在还被用作诊断标记表明烷烃活化的基因功能补充延胡索酸酯(卡拉汉,2013)。几项研究已经涉及下产甲烷条件先天病种虫害和/或系统相关但基本上无教养的属Smithella作为主要n烷烃降能器由于其丰富浓缩文化(Zengler et al ., 1999;灰色et al ., 2011;程et al ., 2013)和环境影响石油碳氢化合物(灰色et al ., 2011)。alkane-activating机制(s),然而,保持神秘,因为签名alkylsuccinate代谢物并没有被发现原位产甲烷条件下或文化亚萨合基因没有被分配给属(艾特肯et al ., 2013;卡拉汉,2013)。因此,替代烷烃活化机制提出了(艾特肯et al ., 2013),但不证明。
Embree et al。(2013)最近出版的草案的基因组Smithellasp。我1 (DDBJ / EMBL /基因库加入号码AWGX00000000)获得一个n-hexadecane-degrading产甲烷的浓缩的文化。作者使用fluorescence-activated细胞排序分离6个细菌细胞相关Smithella从社区,使用全基因组总DNA多个位移放大,放大使用Illumina公司测序Hi-seq (Illumina公司、圣地亚哥、钙、美国),组装使用新创co-assembler和注释基因组使用拉斯特服务器(草案相关的et al ., 2013)。此外,他们从浓缩十六烷上的培养,获得metatranscriptomes丁酸或辛酸作为唯一的有机碳源。拉斯特未能检测和注释assABC基因在草案Smithella基因组;因此,映射的metatranscripts浓缩文化基因组草案并没有揭示的表达屁股在任何的三个文化基因。Embree et al。(2013)因此得出结论,Smithella是不能n烷烃活化,延胡索酸酯,即使他们观察到这些编码转录的基因同源氨基乙酰基彻底激活酶hexadecane-degradersβ-oxidation所需和脂肪酸利用基因的表达n烷烃之后激活。
这些结论与假设上面提到的暗示Smithella /先天病在烷烃降解,以及我们自己的结果,引发了进一步的调查Smithella基因组和metatranscriptomes草案。我们最近分析了metagenome产甲烷的短链alkane-degrading浓缩文化(SCADC;谭et al ., 2013)和恢复部分Smithella我们发现一个副本的基因组中亚萨合(KF824850;未发表)。我们使用这个序列加上一些注释亚萨合的基因(例如,卡拉汉et al ., 2012)tblastn筛选的Smithella我1基因组草案,其中发现七激进氨基乙酰基酶基因编码,包括一个几乎完整的假定的亚萨合基因重叠群5325(加入AWGX01000974数量;基因长度2584个基点)中没有注释Smithella我1基因(草案相关的et al ., 2013);一个假定的亚萨合基因片段重叠群9960 (AWGX01000099;基因长度235个基点);甲酸和五个公认的丙酮酸裂解酶(PFL)基因在6993年叠连群,13305年、13440年、7458年和4758年(AWGX01000042−380−095−531−777;基因长度1643 - 2543个基点)。截断的公认的亚萨合基因序列(099−)太短自信地分配函数,因此消除了进一步分析。
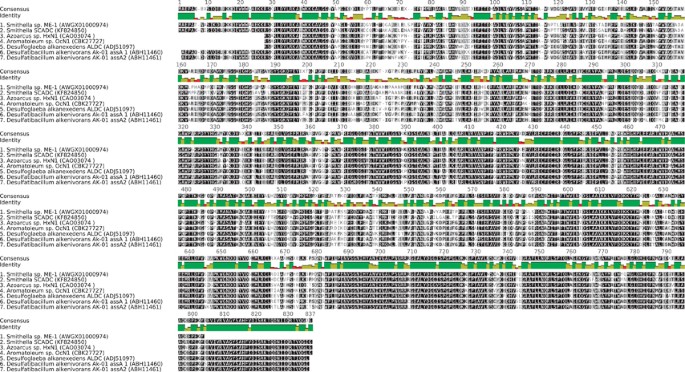
然后,我们翻译了全身的假定的附近亚萨合序列从Smithella我1,SmithellaSCADC和五个参考亚萨合序列。两两比较(补充表S1)表明,假定的亚萨合蛋白质Smithella我1 (AWGX01000974)和SmithellaSCADC (KF824850)高氨基酸彼此身份(87%)和已知的集团Azoarcus sp。HxN1 (CAO03074),Aromatoleumsp。OcN1 (CBK27727),Desulfoglaeba alkanexedensALDC (ADJ51097)和亚萨合册Desulfatibacillum alkenivoransAk-01 (ABH11460和ABH11461) -69%(61% > 800个氨基酸)。序列比对的假定的集团Smithella我1 (AWGX01000974)和SmithellaSCADC (KF824850)强劲的完整参考集团序列(图1)。此外,当翻译亚萨合序列从Smithella我1在NCBI nr-database BLASTP搜索,排名前100的点击了集团同系物中检测出细菌和浓缩的文化,其中许多是与产甲烷烷烃降解和所有的有效位分数和e-values;低冲击与琥珀酸苄酯合成酶α-subunit (BssA)而不是PFL(结果未显示)。
序列比对的公认的翻译亚萨合在Smithella种虫害,参照集团序列。3.3序列对齐使用肌肉Geneious R7,黑色的保守序列主题突出。成对百分比核苷酸排列所示的身份补充表S1。
因为注释基因序列序列比对本身是不够的,我们接受亚萨合基因Smithella我1系统发育分析(图2),显示它密切相关亚萨合基因在培养和细菌亚萨合基因注释SmithellaSCADC但其他氨基乙酰基激进的基因包括远亲bssA和pfl(图2)。很可能Embree et al。(2013)无意中未能发现亚萨合因为它不是自动注释的拉斯特服务器,可能由于叠连群长度短(< 3 kb);然而,亚萨合使用手动序列相似性搜索容易被探测到。因此,Embree et al。(2013)已经提供了第一个遗传证据直接链接Smithella延胡索酸酯激活的n烷烃,而不是记录。
最大似然树六翻译草案中甘氨酰基自由基酶基因Smithella我1基因(相关的et al ., 2013)恢复通过tblastn搜索(粗体所示,SCADC亚萨合序列);序列长度显示在括号中。密切相关的序列恢复从NCBI nr-database通过BLASTX搜索和使用MAFFT序列都是对齐的,其次是使用PhyML系谱树结构(Guindon et al ., 2010)和LG模型和100引导程序复制Geneious R7 (www.geneious.com)。引导支持⩾70%的表示。这棵树被中点加油的。的亚萨合序列的克隆(树)并不完整,范围从414 - 662个基点。树中使用的所有其他序列长度(> 2400个基点)。树的整体拓扑时获得只包括全长序列和删除空白(没有显示)。
我们也重新分析转录的新注解亚萨合基因在混合培养n十六烷,丁酸和辛酸相关的et al ., 2013)。我们绘制了metatranscripts (GSE49830)附近的六个完整的氨基乙酰基激进的基因同源染色体检测Smithella我1 (图2)使用CLC基因组工作台(CLC生物、奥尔胡斯、丹麦),有两个不匹配允许每读一致性。在这一过程中,我们观察到1822 metatranscriptome读取映射到亚萨合十六烷条件下基因(AWGX01000974)但是只有232读取映射下辛酸盐生长条件和2读取与增长丁酸(见补充图S1相对表达)。这个结果与报告Embree et al。(2013)没有表达AWGX01000974十六烷在增长,因为它不是由拉斯特注释。极低的表达AWGX01000974辛酸盐条件下(补充图S1)与低丰度的Smithella我1的辛酸盐文化与十六烷的压倒性优势文化(图4 b相关的et al ., 2013),支持我们的论点亚萨合表达式与增长Smithella我1在十六烷产甲烷混合文化。进一步支持我们的提议,Embree et al。(2013)检测到基因编码α-methylacyl-coA消旋酶和methyl-malonyl-coA提出参与差向异构化作用和碳骨架重排的代谢中间体,分别提出延胡索酸酯激活通路中的硝酸盐和硫酸盐还原条件下使用(卡拉汉et al ., 2012;首领et al ., 2012)。β-oxidation脂肪酸也高度基因转录中增长十六烷(相关的et al ., 2013),与利用一致n十六烷通过延胡索酸酯激活途径。假定的PFL基因的表达在叠连群AWGX01000042, 531年和777−−(补充图S1十六烷)增长可能反映了丙酮酸转化乙酰辅酶a和甲酸(陆et al ., 2012),一个在产甲烷过程常见基质退化。
根据我们的分析Smithella我1基因组和metatranscriptomes草案由人工管理使用tblastn而不是自动拉斯特延胡索酸酯添加基因的注释,我们得出结论相反的Embree et al。(2013)。相反,我们建议Smithella确实是基因能够激活和利用长链烷烃的呢n十六烷产甲烷条件下通过延胡索酸酯,由于拥有和表达屁股基因在产甲烷增长n十六烷。我们进一步注意到自动注释管道像拉斯特有时会导致misannotation延胡索酸酯添加基因的即使在良好的生物。例如,bssA基因序列核废料旁边daltonii FRC-32(NC_011979.1)注解为甲酸C-acetyltransferase (Geob_2448)。同样,在我们自己的工作SmithellaSCADC亚萨合基因被拉斯特misannotated PFL (EC 2.3.1.54)。因此,人工管理等准确识别的基因序列是必要的那些参与厌氧烃激活。
加入代码
引用
艾特肯厘米,琼斯DM,马奎尔MJ,灰色ND,雪莉,圆顶礼帽BFJ等。(2013)。证据表明,原油烷烃活化收益在硫酸盐还原和产甲烷条件下通过不同的机制。Geochim Cosmochim学报109年:162 - 174。
杰•麦克伦尼卡拉汉AV,莫里斯贝尔,佩雷拉IAC,乔丹,奥斯丁RN,树林JT等。(2012)。的基因组序列Desulfatibacillum alkenivoransAK-01:厌氧氧化烷烃的蓝图。环境Microbiol14:101 - 113。
卡拉汉AV。(2013)。酶参与的厌氧氧化n烷烃:从甲烷长链石蜡。前面Microbiol4:89。
程L,丁C,李问,他问,戴LR,张H。(2013)。DNA-SIP显示,Syntrophaceae发挥重要作用在产甲烷十六烷退化。《公共科学图书馆•综合》8:e66784。
相关的M,纳H,罪犯N, Chitsaz H, Zengler K。(2013)。单细胞基因组和metatranscriptome测序揭示代谢alkane-degrading产甲烷的互动社区。ISME J8:757 - 767。
灰色ND,雪莉,格兰特RJ,罗文AK,休伯特CRJ, Callbeck厘米等。(2011)。Syntrophaceae和syntrophic伙伴关系的定量意义的产甲烷降解原油烷烃。环境Microbiol13:2957 - 2975。
Guindon年代,Dufayard摩根富林明,Lefort V, Anisimova M,斯梯W, Gascuel O。(2010)。新算法和最大似然估计方法的发展史:评估PhyML 3.0的性能。系统医学杂志59:307 - 321。
首领R, Sadeghi M, Drozdowska M, Lahme年代,上W, Rabus R等。(2012)。立体化学的调查揭示了细菌激活的机制n烷烃没有氧气。Angew化学Int病51:1334 - 1338。
陆W, Du J,泽新泽西,Gerbig-Smentek E, Einsle O,安德拉德SLA。(2012)。甲酸通道恰城对妇女实施出口的产品混酸发酵。Nat美国国家科学109年:13254 - 13259。
谭B,董XL,靠CW Foght JM。(2013)。宏基因组分析的厌氧微生物文化:alkane-degrading社区成员的潜在hydrocarbon-activating通路和推断的角色。基因组56:599 - 611。
Zengler K, Richnow HH, Rossello-Mora R,米歇利斯W, Widdel F。(1999)。甲烷形成长链烷烃的厌氧微生物。自然401年:266 - 269。
确认
这项工作是由加拿大基因组和基因组通过烃宏基因组项目(阿尔伯塔省http://www.hydrocarbonmetagenomics.com/)。
作者信息
作者和联系
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
补充信息伴随着ISME日报网站上的这篇论文
权利和权限
关于这篇文章
引用这篇文章
棕褐色,B。,Nesbø, C. & Foght, J. Re-analysis of omics data indicatesSmithella产甲烷条件下可以通过补充延胡索酸酯降解烷烃。ISME J8,2353 - 2356 (2014)。https://doi.org/10.1038/ismej.2014.87
发表:
发行日期:
DOI:https://doi.org/10.1038/ismej.2014.87
本文引用的
回顾微生物多样性和遗传标记参与产甲烷碳氢化合物的降解:恢复受污染地区生物燃料的未来前景
环境科学与污染研究(2021)
用某种深度成层产热的碳氢化合物的生物降解不同微生物种群研究这个问题在Scotian盆地冷渗透
自然通讯(2020)