环境meta-omics正迅速扩大为测序能力提高,相关计算技术变得更容易,成本降低。的原位快照的海洋微生物提供这些数据提供了一个增长知识的功能角色社区生态系统的过程。宏蛋白质组学允许动态蛋白质组的描述一个复杂的微生物群落。它有可能揭示影响微生物代谢的生物地球化学运输、存储和骑自行车(例如,Hawley et al ., 2014),而另外澄清这分类群的执行这些角色。以前的工作照亮许多海洋微生物群落中的重要功能和交互(例如,莫里斯et al ., 2010海洋宏蛋白质组学),但回顾文献显示小标准化生物信息学管道检测肽和推断和注释的蛋白质。随着这些数据集患病率增长,有一个关键的需要开发标准化方法质谱(MS)蛋白质组谱识别和注释数据的科学价值得到最大化。在这里,我们表明,生物信息学决定整个肽识别过程一样重要的数据解释的选择采样协议和细菌社区操纵实验设计。我们的分析提供了一个最佳实践指南环境宏蛋白质组学。
MS-based宏蛋白质组学现在实际是由于工作周期和提高质量的进步精度前体和片段。这些改进允许超过10的检测4从单个视串联质谱采集女士混合微生物样品的分析。这些光谱必须从数以千计的蛋白质与多肽来自不同分类群。最常见的方法是数据库搜索:得分观察串联质谱对理论产生的肽谱在网上从蛋白质或肽数据库(Eng et al ., 1994)。然而,数据库选择的方法,或建筑,可能显著不同。在海洋宏蛋白质组学实验中,两个主要的方法创建一个蛋白质识别数据库(1)利用大量的公共序列数据或(2)序列和组装metagenome。进一步探索和装配时可能的公共数据库,广泛的使用数据库和序列选择方法。随着环境蛋白质组学领域的发展,宏蛋白质组学数据集的完整性和我们直接比较他们在时间和空间的能力取决于采用标准化程序肽识别和注释。在这里,我们显示如何高度影响力的蛋白质数据库选择宏蛋白质组学实验的生物学解释。
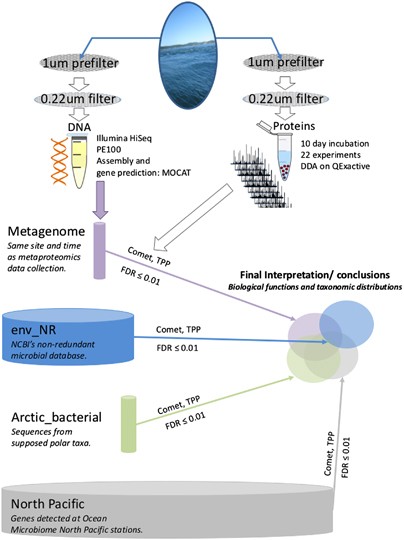
我们应用四个数据库选择技术为了执行肽检测、蛋白质推断,并从MS-based分类和功能分配,海洋微生物群落宏蛋白质组学(图1)。代表一个不同的问题和相对的metaproteome under-sequenced区域的海洋,太平洋北极。我们这项研究的结果提供了一个谨慎的前进道路以及生物的调查人员的结论来自宏蛋白质组学数据是高度特定的数据库。
原理图的工作流数据库搜索宏蛋白质组学样本。气缸的宽度描述每个数据库扩展到每个数据库的独特的胰蛋白酶的肽(补充信息4)。
目前我们的研究遵循传统的程序用于海洋宏蛋白质组学(细节补充信息1)。水样本收集和有选择地过滤所述,从白令海峡可能et al。(2016)和船上孵化10天(T0 = 0天,T10 = 10天)。细菌群落在Q-Exactive-HF孵化项目的蛋白质组分析(热费希尔科学、沃尔瑟姆,妈,美国)和结果数据搜索与四个不同的肽识别数据库(补充信息2):(1)网站/并发收集的详细时间metagenome孵化水;(2)NCBI env_NR数据库;(3)Arctic-bacterial NCBI数据库从已知极性分类群的蛋白质序列(补充信息3)北太平洋数据库来源于海洋微生物基因组测序项目的一个子集Sunagawa et al ., 2015;补充信息4)。肽是识别和蛋白质推断使用Comet v . 2015.01启2 (Eng et al ., 2012,2015年),其次是肽和蛋白质匹配得分(Pedrioli 2010;多伊奇et al ., 2015)的错误发现率阈值0.01 (补充信息5)。蛋白质数据库都使用BLASTp注释(Altschul et al ., 1990;卡马乔et al ., 2009)对UniProtKB TrEMBL数据库(下载2015年4月28日)的价值截止1平台以及(补充信息6)。转变社区在为期10天的孵化量化使用生物功能基因本体论(去)分析相关的肽谱匹配是去哪里。此外,数据库驱动的肽分数敏感数据库大小的函数被搜索网站/调查是有时限的使用越来越多的假肽metagenome数据库。
肽的数量产生光谱实验谱的匹配非常不同的数据库中。最多的自信取得了独特的肽和蛋白质推断是由于匹配搜索网站/详细时间metagenome数据库。这种肽匹配数量增加1.5倍通过搜索对未装配的读取相同的数据。这种“metapeptide”方法(可能et al ., 2016序列)避免损失和潜在的阅读汇编(例如引入的噪声Cantarel et al ., 2011)。四个装配发现的肽数据库重叠较少,表明不同的数据库涵盖的不同部分收购metaproteome (可能et al ., 2016)。直接比较的未装配的metagenome肽和env_NR metagenome包含更多的从metaproteome肽(可能et al ., 2016)。此外,数据库大小,尤其是在env_NR和北太平洋,对搜索灵敏度影响很大,使得统计自信检测肽困难(补充信息7;可能et al ., 2016)。与他人一致,我们发现大型数据库搜索遭受损失的统计力量来自多个假设检验的大量序列中没有代表表示metaproteome (Nesvizhiskii 2010;Jagtap et al ., 2013;Tanca et al ., 2013)。这个悖论的太多的序列导致太少的识别将变得越来越与更多的序列数据的可用性问题。我们的结果指向搜索metaproteome-specific数据库获得的成功,不包括非特异性序列,而平衡需要保留足够多的序列变异。
分类和功能解释造成的不同搜索相同metaproteome针对不同的数据库不同,建议,每个数据库,将产生不同的生物的结论。产生四个社区分类概要分化甚至在门级,这些差异被放大在细分类水平(图2)。metagenome也产生了更多种类的分类群在排名比类env_NR相比(更具体的可能et al ., 2016)。除了分类差异、功能反应10天孵化取决于所使用的数据库不同,差异已经被别人指出(Rooijers et al ., 2011;Tanca et al ., 2013)。在我们的北极微生物,几乎没有共识数据库搜索在十项改变最开始和结束之间的孵化实验(表1,补充信息8)。这些去条件将被视为最重要的贡献者社区功能变化在特定的实验中,并将导致非常不同的解释,这取决于选定的数据库。这些差异的重要性在功能分配搜索结果可以直接下游分析和解释。例如,他们推断时至关重要的社区和报告功能。我们的结果和其他人(例如,Rooijers et al ., 2011)强调宏蛋白质组学数据库选择功能任务的重要性和社区生物过程,尤其是在先前无特征的情况下,复杂的社区。
除了肽搜索结果的差异,发现真正的复杂性注释蛋白质被使用的标准方法只有顶部爆炸冲击的蛋白质注释定义的。爆炸算法返回一列可能的冲击与预期值相关联(e-values)当一个序列搜索;为了更好地理解这种方法的下游效应,我们包括500爆炸结果/蛋白质。平均每metagenome 403蛋白质匹配序列返回,通过价值截止1平台以及。分歧在功能和分类任务爆炸击中一个蛋白质是非常普遍的,即使结果都有很低的e-values (补充信息9)。这怀疑“顶级”爆炸击中了正确的注释感兴趣的蛋白质,尽管这是常见的做法在“组学”文学。通过爆炸不准确或缺乏精确的蛋白质注释方法将进一步模糊宏蛋白质组学数据的准确解释当加上一个无知的数据库的选择。
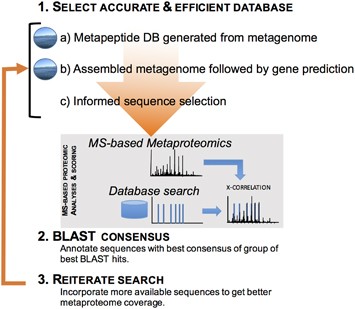
肽的蛋白质数据库的选择识别是其中一个最重要的生物信息学决定准确的生物和生态的解释原位社区功能。尽管更多的时间和金钱都必须完成一个网站/时间metagenome我们已经表明,这些投资更完整的metaproteome解释奠定了基础(Tanca et al ., 2013;可能et al ., 2016)。是否metagenome组装、数据解释必须谨慎行事。根据当前和以前的工作,我们提出一个通用的最佳实践指南(图3)来识别多肽和推断生物功能和分类分布的自然微生物组合:(1)以前但一个个社区,构建尽可能准确和高效的数据库(a)使用metapeptide方法(可能et al ., 2016,b)测序metagenome利用基因预测软件(例如,凯悦et al ., 2012)或(c)构建最准确的数据库可以避免损失的敏感性因大搜索空间当metagenome测序是不可能的;(2)当注释蛋白质,超越前爆炸冲击基础分类法和函数的注释上面达成协议在爆炸冲击一个特定价值阈值(补充信息9);(3)增加肽识别,利用公开可用的序列通过统计上更健壮的多步骤或迭代搜索(例如,Jagtap et al ., 2013;Kertesz-Farkas et al ., 2015)。当研究人员开始探索这些不同的搜索方法与各种宏蛋白质组学数据集,这种方法将提供最强大的搜索方法和最可靠的分类和功能推理环境宏蛋白质组学。
补充信息可在ISME杂志的网站。
女士1:metagenome序列的详细方法,宏蛋白质组学,数据库搜索和生物的解释数据。
2:最小、最大和平均蛋白质长度为每个标识数据库用于这项研究。
3:用于创建Arctic-bacterial数据库分类群。第一和第二列列表的组名和分类水平蛋白质序列从NCBI下载,紧随其后的是完整的分类树。对于每一个分类群,给出引用来自同行评议的文献被用来推断出这一群体的存在在我们的研究网站。第二个选项卡工作簿中列出完整的引用。
4:总结总独特的蛋白质和肽序列在每个数据库。三个不同的场景给肽序列生成构建一个数据库:(1)3错过了分裂和氧化蛋氨酸;(2)0错过了分裂,没有氧化;(3)3错过了分裂,没有氧化。
5:彗星参数文件用于运行所有数据库的搜索。
6:查询蛋白质,UniProt爆炸冲击,为所有人提供相应的价值高的蛋白质检测的信心。蛋白质列表中可以找到不同的数据库搜索的不同表Excel工作簿。
7:添加大量的随机诱饵肽1100万-肽metagenome-derived数据库沮丧肽检测灵敏度。水平轴是肽的数量在每个搜索数据库(1100万metagenome肽,越来越多的随机诱饵肽)。纵轴的数量metagenome肽检测到的错误发现率0.01由正反向数据库寻找五个不同的样例文件。错误发现率从反式计算蛋白质组学管道概率。
8:日志的方向2褶皱变化方面去发现PSM总数> 50 T0 vs T10, T0 vs T10, T0 vs T0”,和T10对T10”(' ' '代表一个技术复制)。一个日志2褶皱变化> 1是"正面",< 1是“消极”,1 - 1是“没有”,如果一个词并没有检测到高于50 PSM在数据库中有一个“X”。结果每个数据库(网站/时间metagenome env_NR, Arctic-bacterial和北太平洋)单独的列中列出每个比较。
9:热图代表类群的粒度从爆炸搜索返回(创造价值⩽1平台以及)作为标识阈值百分比的函数。每个颜色本代表蛋白质的数量在一个给定的最常见的分类单位水平500蛋白质。水平轴:最低百分比序列之间的身份查询蛋白质和爆炸冲击。纵轴:等级最低的常见分类单位代表所有爆炸击中高于阈值。颜色显示的自然对数的数量查询蛋白质,落入每一本,根据规模。“没有”表明击中被分配到多个超界。
引用
迈尔斯Altschul科幻,吉斯”W,米勒W,电子战,Lipman DJ。(1990)。基本的局部比对搜索工具。J杂志215年:403 - 410。
马卡马乔C, Coulouris G, Avagyan V, N,帕帕多普洛斯J, K比尔等。(2009)。爆炸+:体系结构和应用程序。BMC生物信息学10:421 - 430。
埃里克森AR, Cantarel提单VerBerkmoes数控,埃里克森BK,凯里PA,锅C等。(2011)。策略metagenomic-guided整个社区蛋白质组学复杂的微生物环境。《公共科学图书馆•综合》6:e27173。
多伊奇电子战,门多萨L, Shteynberg D, Slagel J,太阳Z,莫里茨RL。(2015)。Trans-Proteomic管道,标准化数据处理管道大规模可再生的蛋白质组学信息。蛋白质组学中国:9:745 - 754。
Eng JK, Hoopmann先生,贾汗助教,Egertson JD,高贵的WS, MacCoss乔丹。(2015)。深入了解一下彗星——实现和功能。J是Soc的质量范围26:1865 - 1874。
贾汗TA, Eng JK Hoopmann先生。(2012)。彗星:一个开放源码的串联质谱序列数据库搜索工具。蛋白质组学13:22 - 24。
Eng JK,麦考马克,耶茨JR。(1994)。肽的方法关联串联mass-spectral数据与氨基酸序列在蛋白质数据库中。J是Soc的质量范围5:976 - 989。
Hawley AK,布鲁尔嗯,Norbeck)广告,Pasa-Tolic L,哈勒姆SJ。(2014)。宏蛋白质组学揭示了微分模式之间的代谢耦合最小区域微生物无处不在的氧气。PNAS111年:11395 - 11400。
豪泽凯悦D, LoCascio PF, LJ, Uberbacher EC。(2012)。基因和翻译起始位点预测metagenome序列。生物信息学28:2223 - 2230。
Jagtap P, Goslinga J, Kooren是的,麦金尼T, Wroblewski女士,西摩SL等。(2013)。两步数据库搜索方法提高灵敏度的肽序列匹配proteogenomic metaproteomic研究。蛋白质组学13:1352 - 1357。
Kertesz-Farkas, Keich U,高贵的WS。(2015)。通过级联搜索串联质谱鉴定。J蛋白质组Res14:3027 - 3038。
可能DH, Timmins-Schiffman E,麦肯MP,哈维人力资源,伯伦斯坦E,南都提单,高贵的WS。(2016)。宏蛋白质组学鉴定微生物样本的翻译猎枪metagenomic读取。J蛋白质组Res15:2697 - 2705。
莫里斯RM,南都提单,Frazar C, Goodlett博士,Ting y, Rocap G。(2010)。宏蛋白质组学比较揭示ocean-scale微生物养分利用率和能量传导的变化。ISME J4:673 - 685。
Nesvizhskii AI。(2010)。计算方法的调查和错误率评估程序猎枪的肽和蛋白质识别蛋白质组学。J蛋白质组学73年:2092 - 2193。
Pedrioli PGA。(2010)。Trans-Proteomic管道:管道进行蛋白质组学分析。蛋白质组生物信息学604年:213 - 238。
Rooijers KK, Kolmeder C, C的字眼,多尔J·M, Boeren年代等。(2011)。迭代工作流挖掘人类肠道metaproteome。BMC基因组学12:6。
科埃略LP Sunagawa年代,Chaffron年代,Kultima JR,她们K,萨拉查G等。(2015)。全球海洋微生物的结构和功能。科学348年:1261359。
Tanca, Palomba, Deligios M, Cubeddu T, Fraumene C, Biosa G等。(2013)。评估不同的序列数据库对metaproteome的影响分析:从lab-assembled见解微生物混合。《公共科学图书馆•综合》8:e82981。
确认
这项工作是支持和由国家科学基金会的资助(NSF-OCE 1233014) ETS,左右,DHM MPM以及美国国立卫生研究院的培训对ETS (T32 HG00035)。DHM和网络支持的国家综合医学科学研究所的NIH奖号码P41 GM103533。微生物群落取样支持通过海洋能源管理局(BOEM-Hanna Shoal生态系统研究)卫生人力资源。这项工作是由华盛顿大学的支持部分蛋白质组学资源(UWPR95794)。我们感谢吉米Eng协助数据库搜索和生物信息学;杨晨赖特建议DNA提取;惠勒马科斯·佩雷斯和玛莎的援助metagenome测序;阿庄园与注释他的帮助;布莱恩·塞尔从NCBI编写代码下载序列;Jarrett Egertson和华盛顿大学基因组科学信息技术团队对他们的援助与数据分析; and Luis Pedro Coehlo for his advice and help with subsetting the Ocean Microbiome data set. BLN and ETS would like to thank TAN and IJE for their ongoing inspiration.
作者信息
作者和联系
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
补充信息伴随着ISME日报网站上的这篇论文
权利和权限
这项工作是在Creative Commons许可Attribution-NonCommercial-ShareAlike 4.0国际许可证。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在信贷额度;如果材料不包括在Creative Commons许可下,用户需要获得许可证持有人的许可复制的材料。查看本许可证的副本,访问http://creativecommons.org/licenses/by-nc-sa/4.0/
关于这篇文章
引用这篇文章
Timmins-Schiffman E。5月,D。,Mikan, M.et al。宏蛋白质组学的重要决定:实现高信心蛋白质注释的未知数。ISME J11,309 - 314 (2017)。https://doi.org/10.1038/ismej.2016.132
发表:
发行日期:
DOI:https://doi.org/10.1038/ismej.2016.132
本文引用的
复杂样品的蛋白质含量测定芳香族氨基酸分析,液体chromatography-UV吸光度比色法
分析和分析化学(2022)
使用丰富的蛋白质作为快速和有效的引导肽/蛋白从人类肠道metaproteomic数据识别
微生物组(2021)
耦合的微生物分析强调了相对细菌在双壳类孵化器的功能角色
环境微生物(2021)
原位矿物加工的机制和异质性海洋氮素固定者束毛藻属揭示了single-colony宏蛋白质组学
ISME通信(2021)
关键的评估MetaProteome调查(皮):multi-laboratory比较成熟的工作流
自然通讯(2021)