文摘gydF4y2Ba
只有一个非常小的一部分长非编码rna (lncRNAs)特征。lncRNAs的进化史可以提供洞察他们的功能,但缺乏lncRNA注释non-model生物妨碍了比较分析。在这里,我们提出一个大规模进化研究lncRNA体验和表达模式,在11个四足动物物种。我们确定了大约11000 primate-specific lncRNAs和2500高度保守的lncRNAs,包括大约400个基因可能起源于3亿多年前。我们发现lncRNAs,特别是古代的,一般积极监管和胚胎发育的主要功能。大多数lncRNAs发展迅速的序列和表达水平,但特异性组织往往是守恒的。我们比较表达的同源lncRNA和蛋白质编码的家庭模式在四足动物重建一个进化保存co-expression网络。这个网络表明潜在功能lncRNAs在精子发生和突触传递等基本流程,而且在更具体的胎盘等机制通过微rna的生产发展。gydF4y2Ba
这是一个预览的订阅内容,gydF4y2Ba通过访问你的机构gydF4y2Ba
相关的文章gydF4y2Ba
开放获取文章引用这篇文章。gydF4y2Ba
在哺乳动物精子形成的分子进化gydF4y2Ba
自然gydF4y2Ba开放获取gydF4y2Ba2022年12月21日gydF4y2Ba
全基因组鉴定和表征lncRNAs向日葵胚乳gydF4y2Ba
BMC植物生物学gydF4y2Ba开放获取gydF4y2Ba2022年10月22日gydF4y2Ba
ortho2align:一个敏感的方法寻找orthologues lncRNAs的小说gydF4y2Ba
BMC生物信息学gydF4y2Ba开放获取gydF4y2Ba2022年9月19日gydF4y2Ba
访问选项gydF4y2Ba
订阅本杂志gydF4y2Ba
收到51印刷问题和网络访问gydF4y2Ba
每年199.00美元gydF4y2Ba
只有3.90美元的问题gydF4y2Ba
本文租或购买gydF4y2Ba
本文得到只要你需要它gydF4y2Ba
39.95美元gydF4y2Ba
价格可能受当地税收计算在结帐gydF4y2Ba


加入代码gydF4y2Ba
登记入册gydF4y2Ba
基因表达综合gydF4y2Ba
顺序读取存档gydF4y2Ba
存款数据gydF4y2Ba
测序数据存入基因表达综合(加入gydF4y2BaGSE43520gydF4y2Ba)和SRA (gydF4y2BaPRJNA186438gydF4y2Ba和gydF4y2BaPRJNA202404gydF4y2Ba)。gydF4y2Ba
引用gydF4y2Ba
Kosiol, c . et al。在六个哺乳动物基因组的积极的选择模式。gydF4y2Ba公共科学图书馆麝猫。gydF4y2Ba4gydF4y2Bae1000144 (2008)gydF4y2Ba
Brawand, d . et al。哺乳动物器官的进化基因表达水平。gydF4y2Ba自然gydF4y2Ba478年gydF4y2Ba343 - 348 (2011)gydF4y2Ba
哈利勒,a . m . et al .许多人类大基因间的非编码rna与chromatin-modifying复合物,影响基因表达。gydF4y2BaProc。《科学。美国gydF4y2Ba106年gydF4y2Ba11667 - 11672 (2009)gydF4y2Ba
Cabili, m . n等。人类大型综合注释基因间的非编码rna显示全局属性和具体的子类。gydF4y2BaDev的基因。gydF4y2Ba25gydF4y2Ba1915 - 1927 (2011)gydF4y2Ba
Derrien, t . et al。人类长非编码rna的GENCODE v7目录:分析他们的基因结构、进化,和表达式。gydF4y2Ba基因组Res。gydF4y2Ba22gydF4y2Ba1775 - 1789 (2012)gydF4y2Ba
格特曼,m . et al .染色质签名显示超过一千个高度保守的非编码rna在哺乳动物中。gydF4y2Ba自然gydF4y2Ba458年gydF4y2Ba223 - 227 (2009)gydF4y2Ba
格特曼,m . et al。gydF4y2Ba从头开始gydF4y2Ba重建细胞类型特异性转录组的老鼠揭示了守恒multi-exonic lincRNAs结构。gydF4y2Ba生物科技自然》。gydF4y2Ba28gydF4y2Ba503 - 510 (2010)gydF4y2Ba
Carninci, p . et al .哺乳动物基因组的转录景观。gydF4y2Ba科学gydF4y2Ba309年gydF4y2Ba1559 - 1563 (2005)gydF4y2Ba
美世,t·R。,Dinger, M. E., Sunkin, S. M., Mehler, M. F. & Mattick, J. S. Specific expression of long noncoding RNAs in the mouse brain.Proc。《科学。美国gydF4y2Ba105年gydF4y2Ba716 - 721 (2008)gydF4y2Ba
年轻,r . s . et al . 1119个候选lincRNA位点的识别和属性gydF4y2Ba黑腹果蝇gydF4y2Ba基因组。gydF4y2Ba基因组医学杂志。另一个星球。gydF4y2Ba4gydF4y2Ba427 - 442 (2012)gydF4y2Ba
南,j . w . & Bartel d长非编码rnagydF4y2Ba秀丽隐杆线虫gydF4y2Ba。gydF4y2Ba基因组Res。gydF4y2Ba(2012)gydF4y2Ba
Ulitsky,我。,Shkumatava,。1月,c . H。,Sive, H. & Bartel, D. P. Conserved function of lincRNAs in vertebrate embryonic development despite rapid sequence evolution.细胞gydF4y2Ba147年gydF4y2Ba1537 - 1550 (2011)gydF4y2Ba
周润发,j . C。、日圆、Z。,Ziesche, S. M. & Brown, C. J. Silencing of the mammalian X chromosome.为基础。启基因组学的嗡嗡声。麝猫。gydF4y2Ba6gydF4y2Ba69 - 92 (2005)gydF4y2Ba
Sleutels F。,Zwart, R. & Barlow, D. P. The non-coding Air RNA is required for silencing autosomal imprinted genes.自然gydF4y2Ba415年gydF4y2Ba810 - 813 (2002)gydF4y2Ba
全垒打,m . e . et al .长非编码rna在小鼠胚胎干细胞多能性和分化。gydF4y2Ba基因组Res。gydF4y2Ba18gydF4y2Ba1433 - 1445 (2008)gydF4y2Ba
Rinn, j·l . et al .功能划分的活跃和沉默染色质域由非编码rna在人类HOX基因座。gydF4y2Ba细胞gydF4y2Ba129年gydF4y2Ba1311 - 1323 (2007)gydF4y2Ba
Ørom, et al。长非编码rna enhancer-like函数在人类细胞。gydF4y2Ba细胞gydF4y2Ba143年gydF4y2Ba46-58 (2010)gydF4y2Ba
Cesana, m . et al .长非编码RNA控制肌肉分化的功能作为一个竞争内源性RNA。gydF4y2Ba细胞gydF4y2Ba147年gydF4y2Ba358 - 369 (2011)gydF4y2Ba
Chodroff, r . a . et al。长非编码RNA基因:序列和脑保护表达不同的脊椎动物之一。gydF4y2Ba基因组医学杂志。gydF4y2Ba11gydF4y2BaR72 (2010)gydF4y2Ba
品牌,a c &桥,c . p .目录的哺乳动物长非编码rna:适度的保护和不完全性。gydF4y2Ba基因组医学杂志。gydF4y2Ba10gydF4y2BaR124 (2009)gydF4y2Ba
Cabili, m . n等。人类大型综合注释基因间的非编码rna显示全局属性和具体的子类。gydF4y2BaDev的基因。gydF4y2Ba25gydF4y2Ba1915 - 1927 (2011)gydF4y2Ba
库特,et al。快速的周转率长非编码rna和基因表达的进化。gydF4y2Ba公共科学图书馆麝猫。gydF4y2Ba8gydF4y2Bae1002841 (2012)gydF4y2Ba
篱笆,s B。,Dudley, J. & Kumar, S. TimeTree: a public knowledge-base of divergence times among organisms.生物信息学gydF4y2Ba22gydF4y2Ba2971 - 2972 (2006)gydF4y2Ba
林,m . f . et al .回顾蛋白质编码基因的目录gydF4y2Ba黑腹果蝇gydF4y2Ba使用12飞基因组。gydF4y2Ba基因组Res。gydF4y2Ba17gydF4y2Ba1823 - 1836 (2007)gydF4y2Ba
Siepel, a . et al .进化保守元素在脊椎动物,昆虫,蠕虫和酵母的基因组。gydF4y2Ba基因组Res。gydF4y2Ba15gydF4y2Ba1034 - 1050 (2005)gydF4y2Ba
1000人基因组计划联盟一个集成的地图从1092年人类基因组遗传变异。gydF4y2Ba自然gydF4y2Ba135年gydF4y2Ba56 - 65 (2012)gydF4y2Ba
病房,l d & > m .丰富的证据,净化人类选择最近收购了监管职能。gydF4y2Ba科学gydF4y2Ba337年gydF4y2Ba1675 - 1678 (2012)gydF4y2Ba
Galtier: & Duret l .适应或有偏见的基因转换?扩展分子进化的零假设。gydF4y2Ba趋势麝猫。gydF4y2Ba23gydF4y2Ba273 - 277 (2007)gydF4y2Ba
Soumillon, m . et al .细胞来源和哺乳动物睾丸的高转录组的复杂性机制。gydF4y2Ba细胞的代表。gydF4y2Ba3gydF4y2Ba2179 - 2190 (2013)gydF4y2Ba
Lindblad-Toh, k等。高分辨率的地图使用29哺乳动物人类进化的约束。gydF4y2Ba自然gydF4y2Ba478年gydF4y2Ba476 - 482 (2011)gydF4y2Ba
编码项目财团一个集成的百科全书在人类基因组中DNA的元素。gydF4y2Ba自然gydF4y2Ba489年gydF4y2Ba57 - 74 (2012)gydF4y2Ba
施密特,d . et al . Five-vertebrate ChIP-seq揭示转录因子的进化动态绑定。gydF4y2Ba科学gydF4y2Ba328年gydF4y2Ba1036 - 1040 (2010)gydF4y2Ba
沃克,E。,Manias, J. L., Chang, W. Y. & Stanford, W. L. PCL2 modulates gene regulatory networks controlling self-renewal and commitment in embryonic stem cells.细胞周期gydF4y2Ba10gydF4y2Ba45-51 (2011)gydF4y2Ba
室,即&汤姆林森,s . r .转录多能性的基础。gydF4y2Ba发展gydF4y2Ba136年gydF4y2Ba2311 - 2322 (2009)gydF4y2Ba
斯图尔特,j . M。西格尔,E。,Koller, D., Kim, S. K. & A Gene-coexpression network for global discovery of conserved genetic modules.科学gydF4y2Ba302年gydF4y2Ba249 - 255 (2003)gydF4y2Ba
Shkumatava,。鲜明的,。,Sive, H. & Bartel, D. P. Coherent but overlapping expression of microRNAs and their targets during vertebrate development.Dev的基因。gydF4y2Ba23gydF4y2Ba466 - 481 (2009)gydF4y2Ba
Franceschini, a . et al .字符串v9.1:蛋白质相互作用网络,增加覆盖率和集成。gydF4y2Ba核酸Res。gydF4y2Ba41gydF4y2BaD808-D815 (2013)gydF4y2Ba
Keniry, et al。gydF4y2Ba段H19gydF4y2BalincRNA发展水库的抑制增长和mir - 675gydF4y2BaIgf1rgydF4y2Ba。gydF4y2Ba自然细胞生物。gydF4y2Ba14gydF4y2Ba659 - 665 (2012)gydF4y2Ba
Van幅s & Abreu-Goodger c使用制程从网络中提取集群。gydF4y2Ba摩尔。生物方法。gydF4y2Ba804年gydF4y2Ba281 - 295 (2012)gydF4y2Ba
格兰特,j . et al。gydF4y2BaRsxgydF4y2Ba是一个metatherian RNAgydF4y2BaXistgydF4y2Ba——属性x染色体的失活。gydF4y2Ba自然gydF4y2Ba487年gydF4y2Ba254 - 258 (2012)gydF4y2Ba
杰尔C。,Pachter, L. & Salzberg, S. L. TopHat: discovering splice junctions with RNA-Seq.生物信息学gydF4y2Ba25gydF4y2Ba1105 - 1111 (2009)gydF4y2Ba
杰尔c . et al .成绩单组装和量化RNA-Seq揭示未经成绩单和同种型开关在细胞分化。gydF4y2Ba生物科技自然》。gydF4y2Ba28gydF4y2Ba511 - 515 (2010)gydF4y2Ba
UniProt。重组蛋白质空间在环球资源(UniProt)。gydF4y2Ba核酸Res。gydF4y2Ba40gydF4y2BaD71-D75 (2012)gydF4y2Ba
蓬,m . et al .蛋白质家庭包含了数据库。gydF4y2Ba核酸Res。gydF4y2Ba40gydF4y2BaD290-D301 (2012)gydF4y2Ba
Flicek, p . et al . 2012年运用。gydF4y2Ba核酸Res。gydF4y2Ba40gydF4y2BaD84-D90 (2012)gydF4y2Ba
柳井正,即et al。全基因组转录中档配置文件揭示人体组织规范中表达水平的关系。gydF4y2Ba生物信息学gydF4y2Ba21gydF4y2Ba650 - 659 (2005)gydF4y2Ba
斯穆特·m·E。小野,K。,Ruscheinski, J., Wang, P.-L. & Ideker, T. Cytoscape 2.8: new features for data integration and network visualization.生物信息学gydF4y2Ba27gydF4y2Ba431 - 432 (2011)gydF4y2Ba
R开发核心团队。gydF4y2Ba接待员:统计计算的语言和环境gydF4y2Bahttp://www.r-project.orggydF4y2Ba(R统计计算的基础,2011年)gydF4y2Ba
Langmead B。,杰尔C。,Pop, M. & Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome.基因组医学杂志。gydF4y2Ba10gydF4y2BaR25 (2009)gydF4y2Ba
Rhead, b . et al . UCSC基因组浏览器数据库:2010年更新。gydF4y2Ba核酸Res。gydF4y2Ba38gydF4y2BaD613-D619 (2010)gydF4y2Ba
>,M。,Patterson, N., Birren, B., Berger, B. & Lander, E. S. Methods in comparative genomics: genome correspondence, gene identification and regulatory motif discovery.j .第一版。医学杂志。gydF4y2Ba11gydF4y2Ba319 - 355 (2004)gydF4y2Ba
Altschul, s F。、《W。,Miller, W., Myers, E. & Lipman, D. Basic local alignment search tool.j·摩尔,杂志。gydF4y2Ba215年gydF4y2Ba403 - 410 (1990)gydF4y2Ba
Vilella, a . j . et al . EnsemblCompara GeneTrees:完成,duplication-aware脊椎动物系统发育树。gydF4y2Ba基因组Res。gydF4y2Ba19gydF4y2Ba327 - 335 (2009)gydF4y2Ba
布兰切特,m . et al .调整多个基因组序列与螺纹blockset对准器。gydF4y2Ba基因组Res。gydF4y2Ba14gydF4y2Ba708 - 715 (2004)gydF4y2Ba
香港,a . et al .精细复合率两性之间的差异,群体和个人。gydF4y2Ba自然gydF4y2Ba467年gydF4y2Ba1099 - 1103 (2010)gydF4y2Ba
格斯坦,m . b . et al .架构来源于人类的监管网络编码数据。gydF4y2Ba自然gydF4y2Ba489年gydF4y2Ba91 - 100 (2012)gydF4y2Ba
Carninci, p . et al .哺乳动物基因组分析启动子体系结构和演化。gydF4y2Ba自然麝猫。gydF4y2Ba38gydF4y2Ba626 - 635 (2006)gydF4y2Ba
Kozomara, a & Griffiths-Jones s miRBase:集成微注释和深度排序的数据。gydF4y2Ba核酸Res。gydF4y2Ba39gydF4y2BaD152-D157 (2011)gydF4y2Ba
确认gydF4y2Ba
我们感谢Froidevaux和d·科尔特斯帮助基因组测序,j·莫尼耶与初步microrna的分析,帮助英国洛桑Harshman表示和基因组学技术设施的高通量测序的支持,即Xenarios计算支持,伯格曼和z Kutalik co-expression分析建议。人类胚胎和胎儿MRC /威康信托基金会联合提供的材料(批准099175 / Z / 12 / Z)人类发育生物学资源(gydF4y2Bahttp://www.hdbr.orggydF4y2Ba)。通过它的计算进行(gydF4y2Bahttp://www.vital-it.chgydF4y2Ba高性能计算中心的SIB瑞士生物信息学研究所。这项研究支持由欧洲研究委员会(242597年开始独立研究员格兰特,SexGenTransEvolution)和瑞士国家科学基金会(31003 a_130287)授予香港A.N. 2月长期支持的博士后奖学金。gydF4y2Ba
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
A.N.构思和执行所有的生物分析和写的手稿,所有作者的输入。A.N.和M.W. RNA-seq处理数据。硕士和A.L. RNA-seq生成数据。杰和F.G.鸭嘴兽收集样本。U.Z.负鼠收集样本。J.C.B.提供鼠标胎盘样品和贡献gydF4y2BaH19XgydF4y2Ba分析。项目监督和最初由香港设计的gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有竞争的经济利益。gydF4y2Ba
扩展数据数据和表gydF4y2Ba
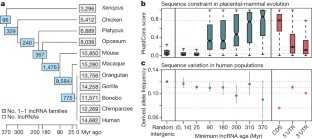
扩展数据图1 lncRNA进化年龄和序列保护模式。gydF4y2Ba
一个gydF4y2Ba,其实序列保护(意味着胎盘PhastCons分数),随机基因间区域,lncRNA最大进化时代类、编码和翻译蛋白编码基因的外显子。gydF4y2BabgydF4y2Ba,意味着DAF的常染色体non-CpG snp隔离在非洲人口(1000人基因工程gydF4y2Ba26gydF4y2Ba)。基因间的单核苷酸多态性被随机画地区匹配lncRNA重组率(方法)。gydF4y2BacgydF4y2Ba,意味着DAF的四类突变方向(W S (W→S)或在GC;S W (S→W)或GC在;W W (W→W),或者在;和年代年代(→年代),或为常染色体non-CpG GC, GC)单核苷酸多态性在primate-specific(25岁最高产量研究)lncRNA其实区域(蓝色)或基因间区域匹配的重组率(灰色)。W→S和S→W突变类已知受GC-biased基因转换。gydF4y2BadgydF4y2Ba一样,gydF4y2BacgydF4y2Ba但对于lncRNAs发现接近(左面板,最大距离10 kb)或远离(右面板,最小距离50 kb) Ensembl-annotated编码和非编码基因。gydF4y2BaegydF4y2Ba,意味着胎盘PhastCons分数(1 kb上游)启动子区域的lncRNA最低进化年龄类(米色)和蛋白质编码基因(蓝色)。gydF4y2BafgydF4y2Ba,意味着胎盘PhastCons分数(1 kb上游)启动子区域的lncRNA最大进化时代类(米色)和蛋白质编码基因(蓝色)。误差线,95%置信区间根据100年引导重采样复制。gydF4y2Ba
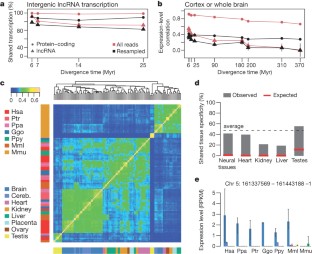
扩展数据图2 lncRNA表达模式在四个四足动物物种。gydF4y2Ba
一个gydF4y2Ba,与观察到的最大比例的基因表达在不同器官老鼠蛋白编码基因,老lncRNAs(至少两种)之间共享和年轻lncRNAs(特有)。gydF4y2BabgydF4y2Ba、Tissue-specificity指数为同一类的老鼠的基因。值接近1代表组织特异性高。gydF4y2BacgydF4y2Ba,分布的最大表达水平(日志gydF4y2Ba2gydF4y2Ba改变RPKM)。gydF4y2BadgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba一样,gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba但对于负鼠。gydF4y2BaggydF4y2Ba- - - - - -gydF4y2Ba我gydF4y2Ba一样,gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba但对于鸭嘴兽。gydF4y2BajgydF4y2Ba- - - - - -gydF4y2BalgydF4y2Ba一样,gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba但对于鸡。gydF4y2Ba
扩展数据图3在lncRNA启动子转录因子结合。gydF4y2Ba
一个gydF4y2Ba频率之间的比较gydF4y2Ba在网上gydF4y2Ba——转录因子(TF)绑定网站lncRNA推动者(2 kb上游)和随机的基因间区域。gydF4y2BabgydF4y2Ba频率之间的比较gydF4y2Ba在网上gydF4y2Ba——TF-binding网站lncRNA和蛋白质编码基因启动子上游(2 kb)。同源框TFs蓝色所示。gydF4y2BacgydF4y2Ba对比实验确定的频率(ChIP-seq编码)TF-binding网站lncRNA推动者(2 kb上游)和随机的基因间区域。gydF4y2BadgydF4y2Ba对比实验确定的频率(ChIP-seq编码)预测在lncRNA TF-binding网站和蛋白质编码基因启动子上游(2 kb)。gydF4y2BaegydF4y2Ba的绑定(ChIP-seq编码数据)、频率OCT4(也称为POU5F1)。gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2Ba,HNF4A比例——CEBPA-binding事件之间共享人类和小鼠,随机基因间区域,lncRNA (321 lncRNAs绑定事件和肝脏表达式,由笼数据)和蛋白质编码基因启动子上游(5 kb)。gydF4y2Ba
扩展数据图4 lncRNA表达模式的演变。gydF4y2Ba
一个gydF4y2Ba人力lncRNAs百分比(在蛋白编码基因的反义)发现在其他物种转录的证据,作为分歧时间的函数。转录评估证据的脑和睾丸strand-specific RNA-seq数据,2535年人类反义lncRNAs 1 - 1 orthologues在至少一个其他物种在人类(方法)和转录的证据。gydF4y2BabgydF4y2Ba斯皮尔曼相关的人类和小鼠表达水平,在不同的组织。箱线图表示100年观察到的变化引导复制。gydF4y2BacgydF4y2Ba人类瀑特异蛋白编码基因的比例(tissue-specificity指数> 0.9,RPKM > 0.1)在灵长类动物器官特异性的共享。红线,随机期望共同的器官特异性;水平黑线,平均守恒的特异性器官。gydF4y2BadgydF4y2Ba比例的人类瀑特异性lncRNAs(最低进化年龄> 90最高产量研究,tissue-specificity指数> 0.9,RPKM > 0.1)器官特异性的真兽类之间共享。红线,随机期望共同的器官特异性;水平黑线,平均守恒的特异性器官。gydF4y2BaegydF4y2Ba一样,gydF4y2BacgydF4y2Ba在真兽亚纲动物物种,保护。gydF4y2BafgydF4y2Ba主成分分析的真兽亚纲动物家属1 - 1 orthologues lncRNA表达水平。gydF4y2BaggydF4y2Ba主成分分析的蛋白质编码基因的表达水平真兽亚纲动物1 - 1 orthologues的家庭。gydF4y2Ba
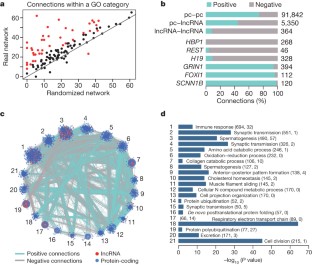
图5扩展数据特征的进化co-expression网络。gydF4y2Ba
一个gydF4y2Ba比例的激活/抑制关系注释字符串中的数据库,对积极和消极co-expression网络连接。gydF4y2BabgydF4y2Ba,基因表达水平(最大所有可用的样本为每个co-expression网络节点)和物种对不同网络连接类。gydF4y2BacgydF4y2Ba,基因表达水平(最大所有可用的样本和物种为每个co-expression网络节点)连接lncRNAs,转录因子(TFs)和non-TF蛋白质编码的基因。gydF4y2BadgydF4y2Ba,网络连接(节点度)lncRNAs(黑色),转录因子(中灰色)和non-transcription因素蛋白编码基因(浅灰色)。前,原始数据;底,纠正后表达水平的差异。gydF4y2BaegydF4y2Ba,观察和预期的比例关系的区别gydF4y2Bacis,gydF4y2BalncRNAs(红色),蛋白质编码基因(蓝色)和基因中发现gydF4y2BaHOXgydF4y2Ba集群(黑)。通过预期的比例随机化(方法)。gydF4y2Ba
扩展数据图6的表达模式和序列的进化gydF4y2BaH19XgydF4y2Ba相关的microrna。gydF4y2Ba
一个gydF4y2Ba,平均分布嵌入式microrna的密度(每kb microrna的发夹,基因下游身体或10 kb),积极的基因与每个网络节点。红色箭头,平均microrna基因积极与密度gydF4y2Ba段H19gydF4y2Ba。gydF4y2BabgydF4y2Ba,最大似然重建古代的发展史gydF4y2BaH19XgydF4y2Ba相关的microrna的家庭(成员代表gydF4y2Bamir - 503gydF4y2Ba,gydF4y2Bamir - 322gydF4y2Ba,gydF4y2Bamir - 424gydF4y2Ba,gydF4y2BamiR-15cgydF4y2Ba,gydF4y2BamiR-16cgydF4y2Ba)。microrna与gydF4y2BaH19XgydF4y2Ba显示在红色(亚科包含吗gydF4y2Bamir - 503gydF4y2Ba和gydF4y2BamiR-16cgydF4y2Ba)和蓝色(含亚科gydF4y2Bamir - 424gydF4y2Ba,gydF4y2Bamir - 322gydF4y2Ba和gydF4y2BamiR-15cgydF4y2Ba)。microrna的名字来源于miRBase可用,包括三个字母的缩写。保险公司,gydF4y2Ba智人gydF4y2Ba;Mdo,gydF4y2Ba把gydF4y2Ba(负鼠);Mml,gydF4y2Ba解剖gydF4y2Ba(猕猴);Mmu,gydF4y2Ba亩骶gydF4y2Ba(鼠标);赶紧走吧,gydF4y2Ba鸭嘴兽anatinusgydF4y2Ba(鸭嘴兽);Gga,gydF4y2Ba背带吊裤带gydF4y2Ba(鸡),额外的,gydF4y2Ba非洲爪蟾蜍tropicalisgydF4y2Ba。运用标识符给出了两个负鼠microrna。gydF4y2BacgydF4y2Ba,鼠标microrna的表达模式gydF4y2BammugydF4y2Ba- - - - - -gydF4y2Bamir - 322gydF4y2Ba相关的,gydF4y2BaH19XgydF4y2Ba。表达水平计算的数量每microrna的唯一映射读取,重采样后相同数量的读取/组织。gydF4y2BadgydF4y2Ba一样,gydF4y2BacgydF4y2Ba但对于鼠标micrornagydF4y2Bammu - mir - 351gydF4y2Ba。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
这个文件包含补充讨论,补充方法和额外的引用。(PDF 359 kb)gydF4y2Ba
补充表1gydF4y2Ba
这个补充表包含信息RNA-seq样品用于这项研究。(XLSX 76 kb)gydF4y2Ba
补充表2gydF4y2Ba
节点和边co-expression网络标识符。(XLSX 15528 kb)gydF4y2Ba
补充表3gydF4y2Ba
这个补充表包含蛋白质编码基因的列表,有过多的连接在独联体co-expression网络。(XLSX 181 kb)gydF4y2Ba
补充表4gydF4y2Ba
制程集群决定co-expression网络和浓缩每个集群的结果。(XLSX 263 kb)gydF4y2Ba
补充表5和6gydF4y2Ba
这个压缩文件包含补充表5和6。补充表5显示的结果去浓缩为每个co-expression lncRNA节点网络分析和补充表6包含每个物种的microrna与H19X列表。(邮政编码4765 kb)gydF4y2Ba
补充数据1gydF4y2Ba
这个补充数据集包含lncRNA注释用于这项研究。(邮政编码20104 kb)gydF4y2Ba
补充数据2gydF4y2Ba
这对同源lncRNA家庭补充数据集包含的信息。(邮政编码24012 kb)gydF4y2Ba
补充数据3gydF4y2Ba
这个补充数据集包含表达水平估计lncRNAs Ensembl-annotated蛋白编码基因。(邮政编码25470 kb)gydF4y2Ba
补充数据4gydF4y2Ba
这个补充数据集包含5种microrna的表达值。(邮政编码21311 kb)gydF4y2Ba
权利和权限gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
Necsulea,。,Soumillon, M., Warnefors, M.et al。gydF4y2Ba在四足动物lncRNA体验和表达模式的演变。gydF4y2Ba自然gydF4y2Ba505年gydF4y2Ba,635 - 640 (2014)。https://doi.org/10.1038/nature12943gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/nature12943gydF4y2Ba
本文引用的gydF4y2Ba
在哺乳动物精子形成的分子进化gydF4y2Ba
自然gydF4y2Ba(2023)gydF4y2Ba
ortho2align:一个敏感的方法寻找orthologues lncRNAs的小说gydF4y2Ba
BMC生物信息学gydF4y2Ba(2022)gydF4y2Ba
白肢野牛基因组驯化牛精液有气味的受体的扩张gydF4y2Ba
BMC基因组学gydF4y2Ba(2022)gydF4y2Ba
全基因组鉴定和表征lncRNAs向日葵胚乳gydF4y2Ba
BMC植物生物学gydF4y2Ba(2022)gydF4y2Ba
揭示了小说复杂的植物长非编码RNA strand-specific和全转录组测序的植物物种进化代表gydF4y2Ba
BMC基因组学gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论你同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条件或准则请国旗是不合适的。gydF4y2Ba
