摘要
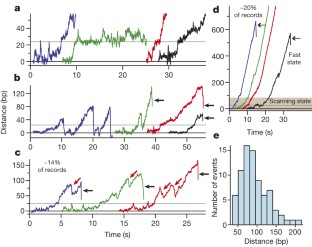
生物化学和结构研究表明,RNA聚合酶II转录的起始在以下几个阶段进行:聚合酶与一般转录因子和启动子DNA在一个“封闭的”起始前复合物(PIC)中组装。1,2;启动子DNA的大约15个碱基对展开,形成一个“开放”复合体3.,4;向下游扫描到转录起始位点;合成一个短的转录本,被认为大约有10个核苷酸长;启动子逃逸。在这里,我们组装了一个32个蛋白质,1.5百万加仑的PIC5来自酿酒酵母,并利用光镊实时观察后续起爆过程6.与预期相反,扫描是由转录因子IIH驱动的7,8,9,10,11,12包括一个扩展的转录泡的快速打开,平均85个碱基对,伴随着整个扩展泡长度的转录本的合成,随后是启动子逃逸。未能实现启动子逃逸的pic形成开放复合体和扩展气泡,气泡塌陷回闭合或开放复合体,导致重复徒劳扫描。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
TFIIH/ Rad4-Rad23-Rad33在核苷酸切除修复损伤DNA开口中的Cryo-EM结构
自然通讯开放获取2021年6月7日
在酿酒酵母转录启动过程中Pol II的通用启动子扫描
基因组生物学开放获取2020年6月2日
Sen1解旋酶体外转录终止的单分子表征
自然通讯开放获取4月4日
访问选项
订阅这本杂志
收到51个印刷问题和在线访问
199.00美元一年
每期仅需3.90美元
租或购买这篇文章
只要这篇文章,只要你需要它
39.95美元
价格可能受当地税收的影响,在结账时计算


参考文献
科纳威,R. C. &科纳威,J. W.一般启动因子的RNA聚合酶II。为基础。学生物化学启。62, 161-190 (1993)
真核生物转录的分子基础。国家科学院学报美国104, 12955-12961 (2007)
Holstege, F. C., Fiedler, U. & Timmers, H. T. RNA聚合酶II转录复合体起始过程中的三个转变。EMBO J。16, 7468-7480 (1997)
Pal, M. Ponticelli, A. S. & Luse, D. S.转录泡和TFIIB在RNA聚合酶II启动子清除中的作用。摩尔。细胞19, 101-110 (2005)
村上等。一个完整的31个蛋白质的RNA聚合酶II转录起始复合物的形成和命运。生物。化学。288, 6325-6332 (2013)
法扎尔,F. M.和Block, S. M.光镊研究张力下的生命。自然光子。5, 318-321 (2011)
Sawadogo, M. & Roeder, R. G.人类RNA聚合酶II系统特异性转录启动的能量需求。生物。化学。259, 5321-5326 (1984)
谢弗,L.等人。DNA修复解旋酶:BTF2 (TFIIH)基本转录因子的组成部分。科学260, 58-63 (1993)
Svejstrup, J. Q.等。不同形式的TFIIH用于转录和DNA修复:全TFIIH和核苷酸切除修复体。细胞80, 21-28 (1995)
Dvir, A.等人。ATP和TFIIH在转录起始前RNA聚合酶II预起始复合物激活中的作用。生物。化学。271, 7245-7248 (1996)
金,T. K, Ebright, R. H. & Reinberg, D.转录因子IIH熔化atp依赖启动子的机制。科学288, 1418-1421 (2000)
Grünberg, S., Warfield, L. & Hahn, S. RNA聚合酶II预起始复合物的结构和atp依赖启动子打开的机制。自然结构。摩尔。杂志。19, 788-796 (2012)
Galburt, E. A.等。回溯以与因子相关的方式确定RNAP II的力灵敏度。自然446, 820-823 (2007)
拉森,m.h.等。触发环动力学调节转录保真度和RNA聚合酶II的速度之间的平衡。国家科学院学报美国109, 655 - 6560 (2012)
Schweikhard, V.等人。转录因子TFIIF和TFIIS通过协同和独立的机制促进RNA聚合酶II的转录伸长。国家科学院学报美国111, 6642-6647 (2014)
西科尔斯基,t.w.等人。Sub1和RPA在转录的不同阶段与RNA聚合酶II结合。摩尔。细胞44, 397-409 (2011)
村上等。从起始点扫描解耦启动子开口。摩尔。细胞59, 133-138 (2015)
李志强,李志强,李志强,李志强。cdk活化激酶与CTD激酶TFIIH/TFIIK的关系。细胞79, 1103-1109 (1994)
罗伊,R.等。MO15细胞周期激酶与TFIIH转录dna修复因子相关。细胞79, 1093-1101 (1994)
Gnatt, a.l, Cramer, P., Fu, J., Bushnell, d.a . & Kornberg, r.d .转录的结构基础:3.3 Å分辨率的RNA聚合酶II延伸复合物。科学292, 1876-1882 (2001)
Giardina, C., Perez-Riba, M. & Lis, J. T.启动子熔化和TFIID配合物果蝇基因在活的有机体内.Dev的基因。6, 2190-2200 (1992)
Giardina, C. & Lis, J. T.酵母RNA聚合酶II启动子上的DNA熔化。科学261, 759-762 (1993)
Giardina, C. & Lis, J. T.酵母热休克促进剂的动态蛋白质- dna结构。摩尔。细胞。医学杂志。15, 2737-2744 (1995)
Dvir, A., Conaway, R. C. & Conaway, J. W. TFIIH在控制早期RNA聚合酶II延伸复合物活性中的作用。国家科学院学报美国94, 9006-9010 (1997)
Spangler, L., Wang, X., Conaway, J. W., Conaway, R. C. & Dvir, A. TFIIH在转录起始和启动子逃逸中的作用需要下游启动子DNA的不同区域。国家科学院学报美国98, 5544-5549 (2001)
Kapanidis, a.n.等人。RNA聚合酶的初始转录通过dna碾压机制进行。科学314, 1144-1147 (2006)
瑞维亚金,刘志刚,刘志刚,Ebright, R. H. & Strick, T. R. RNA聚合酶的流产起始和多产起始涉及DNA碾压。科学314, 1139-1143 (2006)
Miller, G. & Hahn, S.一种DNA系结的裂解探针揭示了酵母预起始复合物中启动子DNA的路径。自然结构。摩尔。杂志。13, 603-610 (2006)
村上等。RNA聚合酶II转录起始前复合物的结构。科学342, 1238724 (2013)
桑德林,A.等。哺乳动物RNA聚合酶II核心启动子:来自全基因组研究的见解。自然牧师热内。8, 424-436 (2007)
Laybourn, P. J. & Dahmus, M. E. RNA聚合酶IIA的磷酸化发生在与启动子相互作用之后和转录开始之前。生物。化学。265, 13165-13173 (1990)
李永华,李永华,李永华。CTD激酶与酵母RNA聚合酶II起始因子b的关系。细胞67, 1223-1230 (1991)
Koslover, d.j., Fazal, F. M., Mooney, R. A., Landick, R. & Block, S. M.在单分子水平上研究终止因子rho的结合和易位。J. Mol.生物学。423, 664-676 (2012)
村上等。Tfb6是一个以前未被识别的一般转录因子TFIIH的亚单位,在转录启动后促进Ssl2解旋酶的解离。国家科学院学报美国109, 4816-4821 (2012)
Ge, H. & Roeder, R. G.介导II类基因转录激活的人类辅激活因子PC4的纯化、克隆和表征。细胞78, 513-523 (1994)
Henry, N. L, Bushnell, D. A. & Kornberg, R. D.一种类似于人类PC4的酵母转录刺激蛋白。生物。化学。271, 21842-21847 (1996)
马利克,S, Guermah, M. & Roeder, R. G. RNA聚合酶II转录中PC4共激活子功能的动态模型。国家科学院学报美国95, 2192-2197 (1998)
Neuman, K. C., Abbondanzieri, E. A., Landick, R., Gelles, J. & Block, S. M.无处不在的转录暂停与RNA聚合酶回溯无关。细胞115, 437-447 (2003)
确认
我们感谢A. Chakraborty和B. Milic仔细阅读手稿P.-J。Mattei在蛋白质纯化方面的帮助,R. Landick和J. Gelles进行讨论。本研究由美国国立卫生研究院资助,GM36659和AI21144授予R.D.K., GM57035授予S.M.B, NSF研究生奖学金授予F.M.F.
作者信息
作者及隶属关系
贡献
F.M.F.、C.A.M.和K.M.在S.M.B.和R.D.K.的指导下设计实验,F.M.F.和C.A.M.收集单分子数据。K.M.对PIC组分进行了纯化和重组,并进行了批量实验。F.M.F.分析了单分子数据,C.A.M.和K.M.进行了建模。F.M.F C.A.M。,K.M S.M.B.和R.D.K.写道。
相应的作者
道德声明
相互竞争的利益
作者声明没有相互竞争的经济利益。
扩展的数据图形和表格
扩展数据图1集成了29个部件的PICSNR20*短启动子。
一个,在不含激酶结构域(TFIIK)的PIC上组装SNR20*短(邻近2.7 kb下游手柄序列),并沉积在甘油梯度上;采用SDS-PAGE对各组分进行分析。b,对分数12的结果进行了详细的注释,表明所有的PIC组分都被保留,证实了复合物完全由组分蛋白重组而成。Pol II的亚基标记为黑色,TFIIF标记为蓝色,TFIIE标记为洋红色,TFIIH标记为橙色,TFIIA标记为青色,TFIIB标记为红色,TBP标记为浅绿色,Sub1标记为深绿色。后来将TFIIK (3-subunits)添加到PIC中。
扩展数据图2哑铃组装示意图,横截面。
PICs通过生物素亲和素键(黄色)连接到一个珠子上。为了形成哑铃系带,一小部分PICs的另一端(4%)具有地高辛键,可以通过2.7 kb DNA手柄系到抗地高辛包被的珠(黑色和棕色)上。不参与系索形成的PIC有助于提高PIC组分的局部浓度。
扩展数据图3单分子实验条件下的径流转录。
一个,分离的PICs (0.1 pmol),形成于SNR20*短启动子片段融合到转录模板(覆盖区域- 62/+636),并附着在2.7 kb DNA柄上,与组装在PICs上的越来越多的PICs结合SNR20*短启动子,但无手柄,以下称为PIC(−62/+96)。这些成分用等体积的2倍NTP溶液(10 ml)孵育,其中含有1.6 mM ATP, 1.6 mM GTP, 1.6 mM CTP, 40 mM UTP和0.83 mM [α-]32P] UTP (2.5 μCi)。凝胶电泳分析得到的转录本。融合到DNA柄的PIC不能单独支持转录(通道1),但当反应中添加4倍(通道2)、8倍(通道3)、12倍(通道4)或15倍(通道5)的PIC(−62/+96)时,转录活性恢复(红色箭头)。在第6通道,反应中含有1.5 pmol PIC(−62/+96)。从PIC(−62/+96)流出的96 nt转录显示(黑色箭头)。25倍过量PIC(−62/+96)用于单分子测定(扩展图2).b将1.5 pmol等份的PIC(−62/+96)引入到不同体积的转录缓冲液中,使PIC的测定浓度在37 nM至4.5 nM之间变化。转录效率(流出带,黑色)随着PIC浓度的增加而降低∼18%到2-3%。单分子分析中使用的低浓度(<1 nM)不能直接使用凝胶进行分析,但我们预计转录效率也相应较低。
扩展数据图4 TFIIH扫描记录SNR20*长rntp或dATP。
一个,b如:SNR20*短(图3),较长的启动子显示TFIIH与两个rNTPs (一个)或只提供dATP (b),之后PIC解离(黑色箭头),或者气泡坍缩到闭合复合体(蓝色和绿色记录)或打开复合体(灰色线),TFIIH再次移动。虚线表示TSS(+1)的位置。
扩展数据图5 PIC上的核酸外切酶III足迹分析SNR20 *长。
在没有核苷酸的情况下在体外, PIC复合物结合到SNR20 *长启动子产生的障碍外切酶III消化定位∼TATA盒子下游50 bp处(距离TSS约- 40个核苷酸,黑色箭头)。这些障碍取决于TFIIH以及与TFIIH相互作用的TFIIE的存在。加入dATP后,势垒消失,暂停位置的波段在−30和+30之间增强(∼塔塔箱、支架下游60-120 bp)。
的转录起始途径SNR20*长(左)和SNR20*短(右)促销员。
左图为起始路径的模型SNR20 *漫长的启动子。从顶部开始的状态:带有gtf(蓝色)和Ssl2(橙色)的Pol II(米色)以“闭合”形式结合到DNA模板上TSS(箭头)上游的启动子元件(绿色和蓝色线)。标出了酶活性位点(白色圆圈打开)和TATA框(黑色方块关闭)的位置。TFIIH的unwind产生一个开放的复合物(OC),导致气泡的形成。在TFIIH的驱动下,扫描导致开放复合体到达TSS,形成一个延伸的气泡(虚线表示单链DNA的推测位置)。如果复合物不能识别TSS,它就会被TFIIH驱动,导致“快速状态”,不产生RNA,但以大约两倍于正常速度的速度前进(黑箱;看到文本)。当Pol II识别TSS时,它开始转录RNA(红线),对应于ITC。ITC的形成导致气泡破裂,随后是gtf的损失并过渡到伸长复合体(EC)。对,对应的起始路径模型SNR20 *短的启动子。类似的状态SNR20 *长。在这种情况下,开放复合体不需要扫描TSS,这是在其DNA足迹中发现的。因此,一旦活性位点识别了TSS, ITC就可以形成并开始RNA合成。因此,在向延伸复合物(EC)过渡之前,可以产生更长的RNA片段。
权利和权限
关于本文
引用本文
法扎尔,F.,孟,C.,村上,K.。et al。实时观察RNA聚合酶II转录的起始。自然525, 274-277(2015)。https://doi.org/10.1038/nature14882
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature14882
这篇文章被引用
TFIIH/ Rad4-Rad23-Rad33在核苷酸切除修复损伤DNA开口中的Cryo-EM结构
自然通讯(2021)
在酿酒酵母转录启动过程中Pol II的通用启动子扫描
基因组生物学(2020)
Sen1解旋酶体外转录终止的单分子表征
自然通讯(2019)
理解生物分子在溶液和活细胞中亚扩散行为的生物化学过程
生物物理评论(2019)
用光镊连接DNA手柄和功能选择蛋白质研究折叠和蛋白质配体相互作用的集成方法
科学报告(2017)
