摘要
人类细胞有23对染色体。然而,在癌症中,基因可以在染色体或环状染色体外DNA (ecDNA)中扩增,尽管ecDNA的频率和功能重要性尚不清楚1,2,3.,4.我们对17种不同类型的癌症进行了全基因组测序、结构建模和细胞遗传学分析,包括2572个分裂细胞的中期染色体结构和功能分析,并开发了一个名为ECdetect的软件包,可以进行无偏倚的、集成的ecDNA检测和分析。在这里,我们发现在近一半的人类癌症中发现了ecDNA;它的频率因肿瘤类型而异,但在正常细胞中几乎从未发现过。驱动癌基因在ecDNA中扩增最常见,从而增加转录水平。数学模型预测ecDNA扩增会比染色体扩增更有效地增加癌基因拷贝数和瘤内异质性。我们通过对癌症样本的定量分析验证了这些预测。这里提出的结果表明ecDNA有助于加速癌症的进化。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
TeCD:染色体外环状DNA的eccDNA收集数据库
BMC基因组学开放获取2023年1月27日
在控制和乳酸胁迫条件下,喂食批量培养的中国仓鼠卵巢细胞中eccDNA的微进化动态
科学报告开放获取2023年1月21日
结直肠癌染色体外环状DNA的分布及特征
分子生物医学开放获取12月2日
访问选项
订阅《自然》+
立即在线访问《自然》和其他55种《自然》杂志
29.99美元
每月
订阅期刊
获得1年的完整期刊访问权限
199.00美元
每期仅需3.90美元
所有价格均为净价格。
增值税稍后将在结帐时添加。
税务计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问。
32.00美元
所有价格均为净价格。
参考文献
Vogelstein等人。癌症基因组图景。科学339, 1546-1558 (2013)
Stark, g.r., Debatisse, M., Giulotto, E. & Wahl, g.m.理解哺乳动物DNA扩增机制的最新进展。细胞57, 901-908 (1989)
培养动物细胞的基因扩增。细胞37, 705-713 (1984)
范,Y.等。双分钟染色体频率与合并细胞遗传学异常及其特征。j:。麝猫。52, 53-59 (2011)
肿瘤细胞群的克隆进化。科学194, 23-28 (1976)
McGranahan, N. & Swanton, C.肿瘤内异质性在癌症进化中的生物学和治疗影响。癌症细胞27, 15-26 (2015)
Marusyk, A., Almendro, V. & Polyak, K.肿瘤内异质性:癌症的一面镜子?Nat. Rev. Cancer12, 323-334 (2012)
叶茨,L. R. &坎贝尔,P. J.癌症基因组的进化。Nat. Rev. Genet。13, 795-806 (2012)
格里夫斯,M. & Maley, C. C.肿瘤克隆进化。自然481, 306-313 (2012)
Andor, N.等人。肿瘤内异质性的程度和后果的泛癌分析。Nat,地中海。22, 105-113 (2016)
Gillies, R. J., Verduzco, D. & Gatenby, R. A.癌症发生的进化动力学和为什么靶向治疗不起作用。Nat. Rev. Cancer12, 487-493 (2012)
Von Hoff, d.d., Needham-VanDevanter, d.r., Yucel, J., Windle, b.e. & Wahl, g.m.扩增的人MYC癌基因定位于复制亚微观环状DNA分子。国家科学院学报美国85, 4804-4808 (1988)
加尔塞德,D. W.等。肿瘤新染色体的结构和进化。癌症细胞26, 653-667 (2014)
卡罗尔,s.m.等。双分钟染色体可以由染色体缺失的前体产生。摩尔。细胞。医学杂志。8, 1525-1533 (1988)
Windle, B., Draper, B. W., Yin, Y. X, O 'Gorman, S. & Wahl, g.m.染色体断裂在基因扩增、缺失形成和扩增子整合中的中心作用。Dev的基因。5, 160-174 (1991)
Kanda, T., Otter, M. & Wahl, g.m.病毒和细胞无中心染色体外分子的有丝分裂分离染色体系结。细胞科学。114, 49-58 (2001)
Mitelman, F. Johansson, B. & Mertens, F.。癌症染色体畸变和基因融合的Mitelman数据库.http://cgap.nci.nih.gov/Chromosomes/Mitelman(2016)
桑伯恩,J. Z.等。通过精确重建致癌扩增子,多形性胶质母细胞瘤中发现了双分钟染色体。癌症Res。73, 6036-6045 (2013)
Almendro, V.等人。通过计算建模和化疗过程中肿瘤演化的推断原位遗传和表型细胞多样性分析。细胞的报道6, 514-527 (2014)
扎克,t。i。等人。体细胞拷贝数改变的泛癌模式。Nat,麝猫。45, 1134-1140 (2013)
内桑森,d.a.等人。通过动态调节染色体外突变EGFR DNA介导的靶向治疗耐药。科学343, 72-76 (2014)
Storlazzi, c.t.等人。实体瘤中基因扩增为双分或均匀染色区域:起源和结构。基因组Res。20., 1198-1206 (2010)
Bozic, I.等人。肿瘤进展过程中驱动突变和乘客突变的积累。国家科学院学报美国107, 18545-18550 (2010)
李,X.等。体细胞染色体改变的时空演变:巴雷特食管的病例队列研究。癌症上一页。Res。7, 114-127 (2014)
米什拉,S. & Whetstine, J. R.拷贝数变化的不同方面:永久性,短暂性和适应性。摩尔。细胞。医学杂志。36, 1050-1063 (2016)
陈志强,陈志强,陈志强,陈志强。体外培养小鼠细胞耐药基因扩增的研究。科学202, 1051-1055 (1978)
尼古拉耶夫,S.等。胶质母细胞瘤和低级别胶质瘤的染色体外驱动突变。Commun Nat。5, 5690 (2014)
Biedler, J. L, Schrecker, A. W. & Hutchison, D. J.白血病L1210耐药亚系中二氢叶酸还原酶水平升高的染色体变异的选择。j .国家的。癌症本月。31, 575-601 (1963)
李,pm。贝叶斯统计:简介。第四版(约翰·威利父子,2012)
也颇,J。Bradley局部图像阈值.https://www.mathworks.com/matlabcentral/fileexchange/40854(2015)
布拉德利,D. &罗斯,G.自适应阈值使用积分图像。J.图形工具12, 13-21 (2007)
Lander, E. S.等。人类基因组的初步测序和分析。自然409, 860-921 (2001)
肯特,W. J.等。UCSC的人类基因组浏览器。基因组Res12, 996-1006 (2002)
李,H. & Durbin R.快速和准确的短读对齐与Burrows-Wheeler变换。生物信息学25, 1754-1760 (2009)
Miller, C. A., Hampton, O., Coarfa, C. & Milosavljevic, A. ReadDepth:用于检测短测序读取拷贝数变化的并行R包。《公共科学图书馆•综合》6, e16327 (2011)
巴甫洛娃,N. N. &汤普森,C. B.癌症代谢的新特征。细胞金属底座。23, 27-47 (2016)
确认
我们感谢R. Kolodner, W. Mischel, D. Geschwind, Mischel实验室的成员,A. Akbari, A. Iranmehr和A. Patel的有益评论。这项工作得到了路德维希癌症研究所(P.S.M., b.r., k.a., w.k.c., F.B.F.),国家脑肿瘤协会(P.S.M., F.B.F.)的战胜GBM计划,本和凯瑟琳·艾薇基金会(P.S.M.)的支持,Ziering家族基金会为纪念Sigi Ziering (P.S.M.)慷慨捐赠;Susan G. Komen基金会(SAC110036), Leona M. and Harry B. Helmsley慈善信托基金(2012-PG-MED002)和乳腺癌研究基金会(BCRF);这项工作还得到了以下NIH赠款的支持:NS73831 (P.S.M.), GM114362 (v.b., v.d., D.B.), NS80939 (F.B.F.), CA014195和CA159859 (G.M.W.)和CA151819 (D.A.N.)和T32CA121938 (K.M.T.)和NSF赠款:NSF- iis -1318386和NSF- dbi -1458557 (v.b., v.d., D.B.)。
作者信息
作者及隶属关系
贡献
K.M.T, v.d., d.b., V.B.和P.S.M.构思并设计了这项研究。K.M.T V.D。,D.B T.K。j。r、中一段和文学士进行实验。d.b., V.D.和V.B.开发了ECdetect软件,并进行了数学建模和模拟。V.D.和V.B.开发了AmpliconArchitect软件。K.A B.R。,D.A.N F.B.F, W.K.C P.N.R.和G.M.W.提供分析支持。H.i.k, m.d.t, s.k, r.w - r。S.R.V.提供了额外的临床样本和分析支持。K.M.T, v.d., d.b., V.B.和P.S.M.根据所有作者的反馈撰写了手稿。
相应的作者
道德声明
相互竞争的利益
V.B.是Digital Proteomics, LLC的联合创始人,拥有股权,并从该公司获得收入。该协议的条款已由加州大学圣地亚哥分校根据其利益冲突政策审查和批准。数字蛋白质组学没有参与这里提出的研究。
额外的信息
审核人信息:自然感谢C. Maley, A. Papenfuss和其他匿名审稿人对本工作的同行评审所作的贡献。
扩展的数据图形和表格
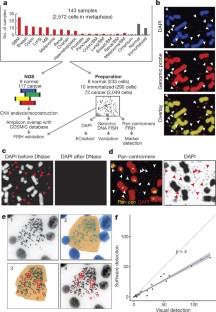
扩展数据图2根据不同的标准,按样品类型分层的ecDNA存在的替代分析。
在中期,平均每10个细胞具有最少数量ecDNA元件的样品x轴被分类为ecDNA阳性,其分数显示在y轴。垂直线为x= 4表明,平均每10个中期细胞中至少有4个ecDNA元件,0%的正常细胞,10%的永生化细胞,46%的肿瘤细胞系和89%的PDX样本被分类为ecDNA阳性。
图4肿瘤样本下一代测序的覆盖深度直方图。
我们测序了117个肿瘤样本,包括63个细胞系、19个神经球(PDX)和35个肿瘤组织,覆盖范围从0.6×到3.89×(不包括一个覆盖范围为0.06×的样本),中位覆盖范围为1.19×。
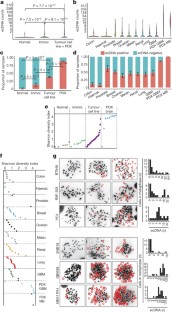
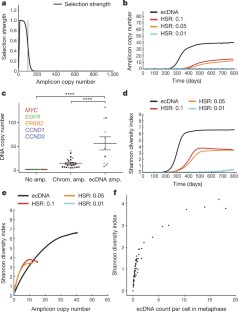
图7复制数扩增和ecDNA多样性。
为了测试复制数扩增和多样性有多少可以归因于ecDNA,我们选择了与我们样本集中最常扩增的四种致癌基因结合的FISH探针,表皮生长因子受体,MYC,CCND1或ERBB2,并从四种肿瘤细胞系(GBM39、MB411FH、SF295和PC3癌细胞)中量化了细胞间DNA拷贝数在中期扩散中的变异性。对于每个细胞系,已知只有标记为红色的靶癌基因在ecDNA上被扩增(表皮生长因子受体在GBM39;MYCMB411FH和PC3,以及CCND1在SF295)。其余3个基因位于染色体位点上。目标癌基因始终显示较高的拷贝数(上)和多样性(下)。
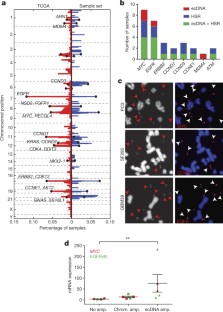
图8 GBM39细胞染色体外或染色体DNA中EGFRvIII扩增的精细结构分析。
一个, FISH图像显示表皮生长因子受体在GBM39细胞系的不同传代中,ecDNA(上)和HSRs(下)上的基因。对HSR FISH图像的分析表明,在不同的染色体上存在多个整合位点。b,对来自4个独立培养的GBM39的DNA进行下一代测序,以分析扩增的精细结构(补充资料4.3).在这些培养物的3个生物重复(第1-3行)中,EGFRvIII完全位于ecDNA上,而在后来的一个传代培养物(第4行)中,发现EGFRvIII完全位于HSRs上,没有检测到ecDNA。来自不同ecDNA培养物的DNA结构相同,但存在一定的异质性(P< 2.18 × 10−8对于所有对),表明有共同的起源。然而,来自hsr的DNA显示出与ecDNA结构相同的保守结构(P< 1.98 × 10−5,补充信息2.4),可能采用串联复制。c,正常基因组与EGFRvIII ecDNA的可能进展到癌症基因组,并扩增到约100个拷贝数。EGFRvIII ecDNA元件可能聚合成串联复制,并以HSRs的形式重新整合到多个染色体上,因此5-6个HSRs可以容纳大约100个EGFRvIII副本。
图9 EGFRvIII在未培养的GBM39细胞的染色体外或染色体DNA扩增以及厄洛替尼治疗和停药后的精细结构分析。
一个,未培养的GBM39细胞对厄洛替尼治疗(ERZ)和停药(ERZ被移除后)的FISH图像分别显示ecDNA扩增、HSR扩增和ecDNA扩增(从上到下)。b,对来自6个独立培养的GBM39进行了下一代DNA测序,以分析扩增的精细结构(补充资料4.3).测序分析确定的扩增间隔的平均拷贝数在初始样本中:110-150(生物重复在第1-3行),ERZ样本:5.4(第4行),停药后:100-105(生物重复在第5-6行)。这三个类别都表现出相似的精细结构,表明有共同的起源(方法)。与原始样本相比,药物戒断复制显示出额外的重排和异质性。c细胞遗传学和测序进展表明,药物应用后,未成熟细胞中的EGFRvIII ecDNA重新整合到HSRs中,HSRs中的副本再次从染色体中分离,形成与未成熟细胞相似的ecDNA。药物戒断样品在结构上也表现出额外的异质性。
相关的音频
补充信息
补充信息
该文件包含补充章节1-4,包括:章节1 ECDNA计数和存在统计;ECdetect -从DAPI染色中期图像中检测染色体外DNA的软件;第三节AmpliconArchitect:用于识别和重建焦点放大的序列分析;第4节染色体外和染色体内复制的理论模型及补充文献。(PDF 14810kb)
权利和权限
关于本文
引用本文
特纳,K.,德什潘德,V.,贝特,D.。et al。染色体外癌基因扩增驱动肿瘤进化和遗传异质性。自然543, 122-125(2017)。https://doi.org/10.1038/nature21356
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature21356
这篇文章被引用
TeCD:染色体外环状DNA的eccDNA收集数据库
BMC基因组学(2023)
在控制和乳酸胁迫条件下,喂食批量培养的中国仓鼠卵巢细胞中eccDNA的微进化动态
科学报告(2023)
水稻稻瘟病病原菌的染色体外环状dna包含多种LTR逆转录转座子、基因和效应子
BMC生物学(2022)
结直肠癌染色体外环状DNA的分布及特征
分子生物医学(2022)
ECCsplorer:从下一代测序数据中检测染色体外环状DNA (eccDNA)的管道
BMC生物信息学(2022)
