文摘
自然使用一个有限的,保守的氨基酸合成蛋白质。基因编码的能力扩展的构建块集和新的化学和物理性质转换研究中,操纵和蛋白质的进化,并使多样的应用程序,包括调查方法、图像和控制蛋白质功能,精确工程师疗法。支撑这一转变是工程师和重建的翻译策略。新兴战略目标对遗传密码进行了基因改造,使经典之中可以合成生物聚合物和演化,以及测试工程师的能力的限制平动机械和系统地重新编码的基因组。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
社会科学设计核糖体与有益的表型
自然通讯开放获取2023年2月21日
光诱导的β-fragmentation形成碳碳键的脂肪醇衍生物
自然通讯开放获取2022年12月02
蛋白质酪氨酸扰乱大规模自组织的卤化
自然通讯开放获取2022年8月17日
访问选项
访问其他自然组合期刊性质和54
得到自然+,请求书在线访问订阅
每月29.99美元
取消任何时候
订阅本杂志
收到51印刷问题和网络访问
每年199.00美元
只有3.90美元的问题
本文租或购买
本文得到只要你需要它
39.95美元
价格可能受当地税收计算在结帐

引用
Ambrogelly,。,Palioura, S. & Söll, D. Natural expansion of the genetic code.Nat,化学。医学杂志。329-35 (2007)
多尔蒂,d . a & Van Arnam e . B。在活的有机体内氨基酸的非规范使用化学氨酰化策略:一种广泛适用的机械工具。ChemBioChem151710 - 1720 (2014)
康沃尔,诉W。,Mendel, D. & Schultz, P. G. Probing protein structure and function with an expanded genetic code.Angew。化学。Int版英格兰。34621 - 633 (1995)
Bondalapati, S。,Jbara, M. & Brik, A. Expanding the chemical toolbox for the synthesis of large and uniquely modified proteins.Nat,化学。8407 - 418 (2016)
下巴,j . w .扩大和重新编程细胞和动物的遗传密码。为基础。学生物化学启。83年379 - 408 (2014)
刘,c . c &舒尔茨,p . g .添加新的化学反应的遗传代码。为基础。学生物化学启。79年413 - 444 (2010)
张,m . S。et al。生物合成和基因编码phosphothreonine通过并行选择和深度测序。Nat方法。14,729 - 736 (2017)。描述了一个可伸缩的方法来发现orthgonal合成酶通过平行biosynthesize的积极的选择和排序策略,编码一个关键翻译修饰。
安德森,j·c·舒尔茨& p·g .正交改编的热点leucyl-tRNA合成酶对四个基本,琥珀,蛋白石抑制。生物化学429598 - 9608 (2003)
诺伊曼,H。,Peak-Chew, S. Y. & Chin, J. W. Genetically encodingN(ε)-acetyllysine重组蛋白。Nat,化学。医学杂志。4232 - 234 (2008)
Ellefson, j . w . et al .定向进化遗传部分和电路的区分与复制。生物科技Nat。》。3297 - 101 (2014)
王,L。,Brock, A., Herberich, B. & Schultz, P. G. Expanding the genetic code of大肠杆菌。科学292年498 - 500 (2001)
年轻,d . d . et al。一种进化氨酰合成酶与非典型polysubstrate特异性。生物化学501894 - 1900 (2011)
厄尔·r·B。,Karplus, P. A. & Mehl, R. A. Gleaning unexpected fruits from hard-won synthetases: probing principles of permissivity in non-canonical amino acid-tRNA synthetases.ChemBioChem151810 - 1819 (2014)
小仲马,。,Lercher, L., Spicer, C. D. & Davis, B. G. Designing logical codon reassignment — Expanding the chemistry in biology.化学。科学。650 - 69 (2015)
澳网,s W。,Anderson, J. C., Lakshman, V. & Schultz, P. G. An archaebacteria-derived glutamyl-tRNA synthetase and tRNA pair for unnatural amino acid mutagenesis of proteins in大肠杆菌。核酸Res。31日6700 - 6709 (2003)
te¸ileanu, T。,Colwell, L. J. & Leibler, S. Protein sectors: statistical coupling analysis versus conservation.公共科学图书馆第一版。医学杂志。11e1004091 (2015)
Iraha, f . et al .内源性的功能替换tyrosyl-tRNA synthetase-tRNATyr一对由古酪氨酸大肠杆菌遗传密码的扩张。核酸Res。383682 - 3691 (2010)
休斯,r . a &艾灵顿,公元合理设计正交tryptophanyl胡说抑制tRNA。核酸Res。386813 - 6830 (2010)
意大利,j·S。et al。一个基因编码的使正交化平台在细菌和真核生物的扩张。Nat,化学。医学杂志。13,446 - 450 (2017)。进化的大肠杆菌TrpRS-tRNA在大肠杆菌和这双转移到哺乳动物细胞基因编码的扩张。
恩斯特,r . j . et al .遗传密码在老鼠大脑的扩张。Nat,化学。医学杂志。12776 - 778 (2016)
汉族,s . et al。扩大的遗传代码亩骶。Commun Nat。814568 (2017)
艾略特,t . s . et al .蛋白质组标签和蛋白质识别在特定组织和特定发展阶段的一种动物。生物科技Nat。》。32465 - 472 (2014)
艾略特,t·S。比安科,。,Townsley, F. M., Fried, S. D. & Chin, J. W. Tagging and enriching proteins enables cell-specific proteomics.细胞化学。医学杂志。23805 - 815 (2016)
石头,s E。,Glenn, W. S., Hamblin, G. D. & Tirrell, D. A. Cell-selective proteomics for biological discovery.咕咕叫。当今。化学。医学杂志。3650-57 (2017)
Schmied, w . H。,elsas, s . J。,Uttamapinant, C. & Chin, J. W. Efficient multisite unnatural amino acid incorporation in mammalian cells via optimized pyrrolysyl tRNA synthetase/tRNA expression and engineered eRF1.j。化学。Soc。136年15577 - 15583 (2014)
Amiram, m . et al。翻译机械记录细菌的进化使多站点的非标准氨基酸。生物科技Nat。》。331272 - 1279 (2015)
郑,Y。,Lewis, T. L., Jr, Igo, P., Polleux, F. & Chatterjee, A. Virus-enabled optimization and delivery of the genetic machinery for efficient unnatural amino acid mutagenesis in mammalian cells and tissues.ACS合成器。医学杂志。6十三至十八,(2017)
查。,太阳,s . B。,Furman, J. L., Xiao, H. & Schultz, P. G. A versatile platform for single- and multiple-unnatural amino acid mutagenesis in大肠杆菌。生物化学521828 - 1837 (2013)
王,K。,诺伊曼,H。,Peak-Chew, S. Y. & Chin, J. W. Evolved orthogonal ribosomes enhance the efficiency of synthetic genetic code expansion.生物科技Nat。》。25770 - 777 (2007)
诺伊曼,H。王,K。,Davis, L., Garcia-Alai, M. & Chin, J. W. Encoding multiple unnatural amino acids via evolution of a quadruplet-decoding ribosome.自然464年441 - 444 (2010)
王,k . et al .优化正交的非自然氨基酸翻译使自发蛋白质double-labelling和烦恼。Nat,化学。6393 - 403 (2014)
公园,h . s . et al。扩大的遗传代码大肠杆菌与磷酸丝氨酸。科学333年1151 - 1154 (2011)
粉丝,C。,Ip, K. & Söll, D. Expanding the genetic code of大肠杆菌phosphotyrosine。2月。590年3040 - 3047 (2016)
向井亚纪,t . et al .密码子的重新分配大肠杆菌遗传密码。核酸Res。388188 - 8195 (2010)
约翰逊,d . b . et al . RF1淘汰赛允许核糖体结合多个站点的非天然氨基酸。Nat,化学。医学杂志。7779 - 786 (2011)
Lajoie, m . j . et al .基因记录生物扩大生物功能。科学342年357 - 360 (2013)
吴,i . l . et al。多个site-selective不在经典里的氨基酸插入到sequence-repetitive多肽。ChemBioChem14968 - 978 (2013)
查。,Lajoie, M. J., Xiao, H., Church, G. M. & Schultz, P. G. A bacterial strain with a unique quadruplet codon specifying non-native amino acids.ChemBioChem151782 - 1786 (2014)
向井亚纪,t . et al .高度生殖大肠杆菌细胞与UAG密码子没有特定的任务。科学。代表。59699 (2015)
郑,y . et al .性能优化中的氨基酸突变系统没有释放因子1。摩尔。Biosyst。121746 - 1749 (2016)
罗杰森,d . t . et al .磷酸丝氨酸及其nonhydrolyzable模拟的有效遗传编码。Nat,化学。医学杂志。11496 - 503 (2015)
理查森,s m . et al .设计合成的酵母基因组。科学355年1040 - 1044 (2017)
戴维斯,l . &下巴,j . w .设计师蛋白质:遗传密码的扩张在细胞生物学中的应用。Nat。启摩尔。细胞杂志。13168 - 182 (2012)
Minnihan, e . C。,Seyedsayamdost, M. R., Uhlin, U. & Stubbe, J. Kinetics of radical intermediate formation and deoxynucleotide production in 3-aminotyrosine-substituted大肠杆菌核苷酸还原酶。j。化学。Soc。133年9430 - 9440 (2011)
杨,y . et al .基因编码的蛋白质photocrosslinker转移质量spectrometry-identifiable标签。Commun Nat。712299 (2016)
硬币,等。基因编码的化学探针在细胞显示的绑定路径urocortin-I GPCR CRF类B。细胞155年1258 - 1269 (2013)
朗,k &下巴,j . w .细胞的非自然氨基酸和bioorthogonal标签的蛋白质。化学。牧师。114年4764 - 4806 (2014)
查。郭,J。,Lee, H. S. & Schultz, P. G. A genetically encoded fluorescent probe in mammalian cells.j。化学。Soc。135年12540 - 12543 (2013)
罗,j . et al .基因编码optochemical光探测器为同步荧光报告和激活的蛋白质与双光子激发函数。j。化学。Soc。136年15551 - 15558 (2014)
朗,k &下巴,j . w . Bioorthogonal反应标记蛋白质。ACS化学。医学杂志。916 - 20 (2014)
Lukinavicˇ国际单位,g . et al。一个近红外荧光团live-cell超分辨率显微细胞蛋白质。Nat,化学。5132 - 139 (2013)
Uttamapinant, c . et al .遗传密码扩张使live-cell和超分辨率成像的网站标记细胞蛋白质。j。化学。Soc。137年4602 - 4605 (2015)
腌鱼、k等。应用中的氨基酸对蛋白质标签在一个基因上记录大肠杆菌。ACS合成器。医学杂志。6233 - 255 (2017)
彭,t &挂h . c .因地制宜bioorthogonal标记活细胞胞内蛋白的荧光成像技术。j。化学。Soc。138年14423 - 14433 (2016)
Baumdick, m . et al . EGF-dependent水泡回收交换机的路由自发磷酸化抑制表皮生长因子受体信号。eLife4e12223 (2015)
Sakin诉et al。万能工具live-cell成像和超分辨率nanoscopy研究hiv - 1 Env分布和流动。细胞。化学。医学杂志。24635 - 645 (2017)
天雪,L。,Prifti, E. & Johnsson, K. A general strategy for the semisynthesis of ratiometric fluorescent sensor proteins with increased dynamic range.j。化学。Soc。138年5258 - 5261 (2016)
Sachdeva,。王,K。,Elliott, T. & Chin, J. W. Concerted, rapid, quantitative, and site-specific dual labeling of proteins.j。化学。Soc。136年7785 - 7788 (2014)
肖,h . et al .基因整合多个非自然氨基酸蛋白在哺乳动物细胞。Angew。化学。Int版英格兰。5214080 - 14083 (2013)
李,j . et al . Palladium-triggered去化学活细胞的蛋白质激活。Nat,化学。6352 - 361 (2014)
李,J。,Jia, S. & Chen, P. R. Diels–Alder reaction-triggered bioorthogonal protein decaging in living cells.Nat,化学。医学杂志。101003 - 1005 (2014)
张,g . et al . Bioorthogonal化学活化激酶的生命系统。ACS分钱。科学。2325 - 331 (2016)
王,j . et al . Palladium-triggered化学救援的胞内蛋白通过基因编码allene-caged酪氨酸。j。化学。Soc。138年15118 - 15121 (2016)
贝克,a s & Deiters a .光学控制蛋白质功能通过非自然氨基酸突变和其他optogenetic方法。ACS化学。医学杂志。91398 - 1407 (2014)
Hemphill, J。,Borchardt, E. K., Brown, K., Asokan, A. & Deiters, A. Optical control of CRISPR/Cas9 gene editing.j。化学。Soc。137年5642 - 5645 (2015)
阮,d . p . et al . photocaged半胱氨酸的遗传编码允许光活化TEV蛋白酶在哺乳动物细胞。j。化学。Soc。136年2240 - 2243 (2014)
沃克,o . s . et al .光敏化突变异柠檬酸脱氢酶2显示快速癌症相关的代谢和表观遗传变异。j。化学。Soc。138年718 - 721 (2016)
玻色,M。,Groff, D., Xie, J., Brustad, E. & Schultz, P. G. The incorporation of a photoisomerizable amino acid into proteins in大肠杆菌。j。化学。Soc。128年388 - 389 (2006)
Hoppmann C。,Maslennikov, I., Choe, S. & Wang, L.原位形成偶氮桥蛋白可控的可见光。j。化学。Soc。137年11218 - 11221 (2015)
蔡,中州。,Essig年代。,James, J. R., Lang, K. & Chin, J. W. Selective, rapid and optically switchable regulation of protein function in live mammalian cells.Nat,化学。7554 - 561 (2015)
elsas, s . J。恩斯特,r . J。,Walker, O. S. & Chin, J. W. Genetic code expansion in stable cell lines enables encoded chromatin modification.Nat方法。13158 - 164 (2016)
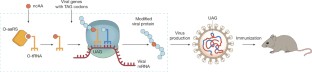
是的,L。et al。一代的A型流感病毒但replication-incompetent活病毒疫苗。科学354年,1170 - 1173 (2016)。描述了创建一个琥珀suppression-dependent甲型流感病毒和由此产生的使用减毒病毒免疫。
Wang z . A . et al .基因编码allysine与特定站点赖氨酸dimethylation蛋白质的合成。Angew。化学。Int版英格兰。56212 - 216 (2017)
王,z . A . et al .多才多艺的方法在蛋白质中的赖氨酸酰化。Angew。化学。Int版英格兰。561643 - 1647 (2017)
Hoppmann c . et al .因地制宜的phosphotyrosine使用扩大遗传密码。Nat,化学。医学杂志。13842 - 844 (2017)
罗,x et al .基因编码phosphotyrosine及其nonhydrolyzable模拟细菌。Nat,化学。医学杂志。13845 - 849 (2017)
赖特,t·h . et al .转译后的诱变:为探索蛋白质侧链化学策略的多样性。科学354年aag1465 (2016)
杨,A . et al .化学生物学途径特定站点真实蛋白质的修改。科学354年623 - 626 (2016)
肖,h &舒尔茨,p . g .界面的化学和生物合成:扩大遗传密码。冷泉哈布。教谕。医学杂志。8a023945 (2016)
田,f . et al .特有的抗体药物配合的一般方法。Proc。《科学。美国111年1766 - 1771 (2014)
VanBrunt, m . p . et al。含叠氮化基因编码的氨基酸在哺乳动物细胞使特定站点抗体药物配合使用点击化学环加成作用。Bioconjug。化学。262249 - 2260 (2015)
Kularatne, s . a . et al。招聘folate-receptor-positive癌细胞的细胞毒性T细胞。Angew。化学。Int版英格兰。5212101 - 12104 (2013)
妈,j . s . et al .多才多艺的战略控制设计的T细胞的特异性和活动。Proc。《科学。美国113年E450-E458 (2016)
王:et al .构建减毒活疫苗hiv - 1疫苗通过遗传密码扩张。Angew。化学。Int版英格兰。534867 - 4871 (2014)
林,s . et al .特定场地工程化学功能表面的D型肝炎病毒。Angew。化学。Int版英格兰。5213970 - 13974 (2013)
Loughran, g . et al .证据有效的终止密码子readthrough四哺乳动物的基因。核酸Res。428928 - 8938 (2014)
宣,w•舒尔茨& p·g·创造生物的策略依赖于不在经典里的氨基酸。Angew。化学。Int版英格兰。569170 - 9173 (2017)
Mandell, d . j . et al . Biocontainment转基因生物的合成蛋白质设计。自然518年,则高达55 - (2015)。使关键基因的功能依赖于琥珀抑制和身份中的氨基酸结合的琥珀色的抑制。
米尔斯,j . h . et al .计算设计的非自然氨基酸依赖金属蛋白原子水平精度。j。化学。Soc。135年13393 - 13399 (2013)
米尔斯,j . h . et al .计算设计homotrimeric金属蛋白trisbipyridyl核心。Proc。《科学。美国113年,15012 - 15017 (2016)。设计的金属离子扩大遗传密码。
皮尔森,公元et al。捕获一个过渡态计算设计蛋白质瓶。科学347年863 - 867 (2015)
肖,h . et al .探索扩大遗传密码的潜在影响蛋白质功能。Proc。《科学。美国112年6961 - 6966 (2015)
策略,d . s . et al .上瘾不同细菌中的氨基酸。Nat,化学。医学杂志。12138 - 140 (2016)
Hammerling坚持认为,m . j . et al .噬菌体使用一个扩大进化遗传密码路径更高的适应性。Nat,化学。医学杂志。10178 - 180 (2014)
洛佩兹、g和安德森,j . c .合成营养缺陷型与ligand-dependent重要基因BL21 (DE3)生物安全压力。ACS合成器。医学杂志。41279 - 1286 (2015)
福斯特,a . c . et al .编程peptidomimetic合成通过转换基因编码设计新创。Proc。《科学。美国One hundred.6353 - 6357 (2003)
Iwane, y . et al。扩大核糖体合成多肽的氨基酸曲目通过人工的密码子框。Nat,化学。8317 - 325 (2016)
安德森,j . c . et al .扩大遗传密码功能成套的密码子。Proc。《科学。美国101年7566 - 7571 (2004)
马里森,d . A . et al .半合成生物体与扩大遗传字母表。自然509年385 - 388 (2014)
张,Y。等半合成生物基因工程的稳定扩张字母表。Proc。《科学。美国114年,1317 - 1322 (2017)。正交碱基对的维护大肠杆菌。
非政府组织,d·a·j·t·&•蒂雷尔称有典中的氨基酸在细胞蛋白质合成的审讯。Acc。化学。Res。44677 - 685 (2011)
曾,Y。,Wang, W. & Liu, W. R. Towards reassigning the rare AGG codon in大肠杆菌。ChemBioChem151750 - 1754 (2014)
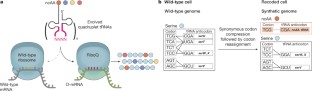
Ostrov, N。等。设计、合成和测试57-codon基因组。科学353年,819 - 822 (2016)。替换的50 kb部分大肠杆菌十独立菌株基因组同义密码子使用变量替换。
王,k . et al .定义同义密码子基因组重新编码的压缩方案。自然539年59 - 64 (2016)。描述了一个强大的基因替换策略大肠杆菌及其应用破译最好的同义替换为从基因组密码子目标。
纳波利塔诺,m . g . et al .紧急密码子的选择规则阐明通过编辑罕见的精氨酸的密码子大肠杆菌。Proc。《科学。美国113年E5588-E5597 (2016)
刘,黄懿慧et al。细菌基因组的大规模重新编码迭代recombineering DNA的合成。核酸Res。456971 - 6980 (2017)
诺伊曼,H。,Slusarczyk, A. L. & Chin, J. W.新创代的相互正交的氨酰合成酶/ tRNA对。j。化学。Soc。132年2142 - 2144 (2010)
查。肖,H。& Schultz, P. G. Evolution of multiple, mutually orthogonal prolyl-tRNA synthetase/tRNA pairs for unnatural amino acid mutagenesis in大肠杆菌。Proc。《科学。美国109年14841 - 14846 (2012)
郭,J。,Wang, J., Anderson, J. C. & Schultz, P. G. Addition of an alpha-hydroxy acid to the genetic code of bacteria.Angew。化学。Int版英格兰。47722 - 725 (2008)
小林,T。,Yanagisawa, T., Sakamoto, K. & Yokoyama, S. Recognition of non-alpha-amino substrates by pyrrolysyl-tRNA synthetase.j·摩尔,杂志。385年1352 - 1360 (2009)
棕褐色,Z。,Forster, A. C., Blacklow, S. C. & Cornish, V. W. Amino acid backbone specificity of the大肠杆菌翻译机器。j。化学。Soc。126年12752 - 12753 (2004)
Fujino, T。Goto, Y。,Suga, H. & Murakami, H. Reevaluation of thed在翻译中氨基酸兼容伸长的事件。j。化学。Soc。135年1830 - 1837 (2013)
Maini, r . et al .蛋白质合成与核糖体结合的选择β-amino酸。生物化学543694 - 3706 (2015)
Maini, r . et al . Ribosome-mediated合并二肽和二肽的类似物成蛋白质在体外。j。化学。Soc。137年11206 - 11209 (2015)
Dedkova, l . M。法赫米:E。,Golovine, S. Y. & Hecht, S. M. Construction of modified ribosomes for incorporation ofd胺基酸成蛋白质。生物化学4515541 - 15551 (2006)
梅洛Czekster C。,Robertson, W. E., Walker, A. S., Söll, D. & Schepartz, A.在活的有机体内生物合成的β-amino acid-containing蛋白质。j。化学。Soc。138年5194 - 5197 (2016)
Englander m . t . et al .核糖体能辨别的手性氨基酸在其peptidyl-transferase中心。Proc。《科学。美国112年6038 - 6043 (2015)
Terasaka, N。,Hayashi, G., Katoh, T. & Suga, H. An orthogonal ribosome-tRNA pair via engineering of the peptidyl transferase center.Nat,化学。医学杂志。10555 - 557 (2014)
炸,s D。,Schmied, w . H。,Uttamapinant, C. & Chin, J. W. Ribosome subunit stapling for orthogonal translation in大肠杆菌。Angew。化学。Int版英格兰。5412791 - 12794 (2015)
Orelle, c . et al .蛋白质合成核糖体拴在子单元。自然524年119 - 124 (2015)
确认
我感谢a . Chatterjee k . Wang和p·g·舒尔茨建议和编辑早期版本的手稿和诉贝拉尼克,和k .王寻求帮助。在我的实验室工作是医学研究委员会的支持下,英国(MC_U105181009和MC_UP_A024_1008)和一个伦理委员会先进格兰特(SGCR)。
作者信息
作者和联系
相应的作者
道德声明
相互竞争的利益
作者声明没有竞争的经济利益。
额外的信息
审核人信息自然由于a·c·福斯特a Schepartz,另一个匿名的评论家(s)为他们的贡献的同行评审工作。
出版商的注意:施普林格自然保持中立在发表关于司法主权地图和所属机构。
权利和权限
关于这篇文章
引用这篇文章
下巴,j .扩大和重组的遗传代码。自然550年53-60 (2017)。https://doi.org/10.1038/nature24031
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/nature24031
本文引用的
Virus-assisted定向增强哺乳动物细胞中抑制trna的进化
自然方法(2023)
社会科学设计核糖体与有益的表型
自然通讯(2023)
非天然的肽细胞解码
化学性质(2023)
短的tRNA反密码子茎和突变eRF1允许停止密码重置手术
自然(2023)
遗传密码显示UBE2W aminoacylated赖氨酸泛素化介导的扩张
《自然结构和分子生物》上(2023)
