摘要
飞行时间多路离子束成像(MIBI-TOF)是一种质谱成像形式,使用金属标记抗体和二次离子质谱同时成像同一组织切片中的数十种蛋白质。我们与美国国家癌症研究所(NCI)癌症免疫监测和分析中心(CIMAC)合作,进行了一项验证研究,评估了21个样本组织微阵列的12个系列切片的一致性,这些样本由MIBI-TOF或单一免疫组化(IHC)在12天内独立处理和成像。像素级特征在所有16个目标的染色强度(R2= 0.94±0.04),频率(R2= 0.95±0.04)。与相邻序列切片的数字化、单发免疫组化比较显示出相似的一致性(R2= 0.85±0.08)。最后,8个细胞群的自动分割和聚类发现,序列切片之间的细胞频率产生的平均相关性为R2= 0.94±0.05。综上所述,我们证明了MIBI-TOF,使用经过严格审查的试剂和自动化分析,可以在存档的人体组织中生成临床相关细胞状态的一致和定量注释,更广泛地说,为基准化多路免疫组合方法提供了一个可扩展的框架。
简介
免疫组化(IHC)通常用于临床诊断和基础研究,利用显色或荧光报告器在完整组织中可视化蛋白质1,2,3..免疫组化染色通常用于指导绝大多数实体组织恶性肿瘤的诊断和治疗选择4,5,6.虽然显色IHC仍然是解剖病理学中不可缺少的工具,但其固有的局限性阻碍了定量解释和常规的多重染色1,7,8,9,10.这些缺陷在癌症免疫治疗领域尤其受限,因为肿瘤免疫微环境的准确评估需要同时绘制数十种蛋白质的图谱11,12.具有多种分析解决方案的新兴空间组学领域正在努力取代传统的ihc驱动的决策11,13,14,15,16.虽然很明显,新技术和分析方法有可能产生新类型的数据,但尚不清楚它们是否能够可靠地复制当前“黄金标准”分析方法中的决策信息。
本实验室开发了飞行时间多路离子束成像技术(MIBI-TOF),利用二次质谱技术对同一组织切片上的数十种蛋白质进行成像,从而避免了光学成像的局限性17,18.MIBI-TOF取代显色或荧光报告剂,使用同位素富集的金属报告剂标记的一抗,可以使用飞行时间(TOF)质谱清晰地描绘和量化。组织切片同时用所有金属标记的一抗处理,使用一种简单的方案,不包括二抗体、酶扩增或循环染色。在MIBI-TOF分析过程中,组织被初级离子束以像素逐像素的方式溅射,从而将金属标签作为次级离子释放出来,随后由TOF量化。我们的实验室通常同时量化40个目标,目前正在努力将这一能力提高到60个或更多18,19,20.,21.值得注意的是,MIBI-TOF与福尔马林固定、石蜡包埋(FFPE)样品兼容,可以在跨越六个数量级的动态范围内检测低丰度和高丰度目标18.
像MIBI-TOF这样的技术要用于大型转化研究并最终用于常规临床诊断,就需要进行稳健性和可重复性研究,以及解释这些复杂数据的标准化工作流程。此外,重要的是要表明新技术与现有的、已建立的临床分析是一致的。与美国国家癌症研究所(NCI)合作,作为癌症免疫监测和分析中心-癌症免疫数据共享(CIMAC-CIDC)网络的一部分22,我们使用MIBI-TOF对多重抗体染色的重复性进行了基准测试。为了实现这一目标,我们比较了六个独立相邻组织微阵列(TMA)系列切片的MIBI-TOF成像数据。各系列切片的抗体染色和MIBI-TOF成像均独立进行,所有实验参数均随机化。此外,我们评估了MIBI-TOF与单发IHC显色染色的一致性。为了与CIMAC-CIDC的目标保持一致,为生物标志物的发现开发全面、标准化的免疫监测分析,我们表明MIBI-TOF是一种高度可重复的检测方法,并且与单一免疫组化一致。
材料与方法
组织微阵列构建与切片
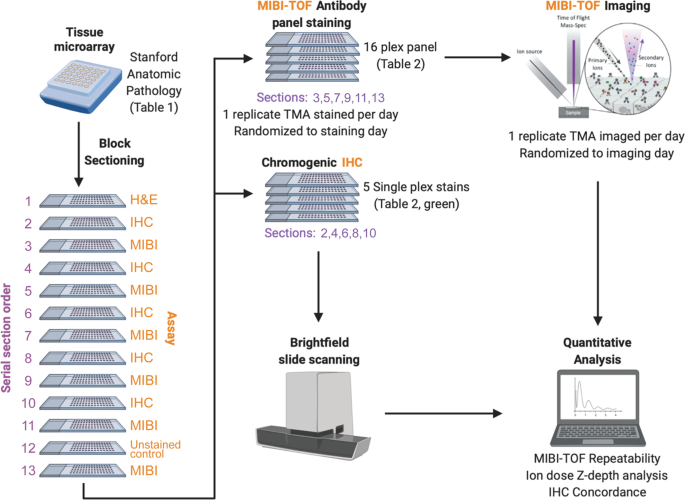
TMA是使用斯坦福病理学的人体FFPE块构建的。TMA包括无病对照以及多种类型的癌症、肉瘤和中枢神经系统病变。本研究中包含的核心的表可以在表中找到1(1mm芯)。对于每个TMA组织块,获取13个连续的连续切片(切片厚度为4 μm)。IHC和ibi - tof切除交替进行,以便IHC一致性分析,可以在相邻切片中比较MIBI-TOF和IHC。对每个切片进行的分析如下:
第一部分:H&E
第2、4、6、8、10节:显色IHC
第3、5、7、9、11、13节:MIBI-TOF全板染色
第12节:MIBI-TOF无染色对照
抗体制备
完整的MIBI-TOF面板包含16个抗体可以在表中找到2.冻干抗体板以预先配制的格式从商业供应商(Ionpath Inc.,“细胞分类”人体板,https://www.ionpath.com/antibody-panels/).抗体面板以冻干格式提供。预混合冻干抗体的设计是为了减少变异性和提高稳定性。每个抗体的滴度在冻干前由供应商进行了验证。每个小瓶都含有所有预先混合的抗体,一个小瓶足以染色一张载玻片。对于每个染色程序,新一瓶抗体面板悬浮在抗体稀释缓冲液(TBS-IHC补间,驴血清3%)中,并用0.1µm过滤器(Millipore)过滤。从Ionpath中也获得了单独的IHC金属标记抗体:CD3 (D7A6E)、CD8 (C8/144B)、CD68 (D4B9C)、Pax5 (D7H5X)、PanCK (AE1/AE3)。这些单独的抗体以溶液形式提供。供应商建议稀释系数为1:100。我们将此稀释因子用于除CD3外的所有标记物,我们确定1:60的稀释因子是合适的。
组织染色
6例MIBI-TOF再切割的组织染色分别在6天内进行,根据切片编号随机化(表2)1).详细的染色方案可以在这里找到:dx.doi.org/10.17504/protocols.io.byzrpx56, dx.doi.org/10.17504/protocols.io.bf6ajrae, dx.doi.org/10.17504/protocols.io.bhmej43e。我们使用Sequenza手动染色系统进行MIBI-TOF和IHC染色(dx.doi.org/10.17504/protocols.io.bmc6k2ze)。
简单地说,用徕卡ST4020线性染色器(徕卡生物系统)在70°C下烘烤过夜,然后脱蜡和再水化,连续清洗30秒,分别浸入二甲苯(三次清洗)、100%乙醇(两次清洗)、95%乙醇(两次清洗)、80%乙醇(一次清洗)、70%乙醇(一次清洗)和ddH2O(两次清洗)三次。接下来,组织进行抗原提取,方法是将载玻片浸泡在3-in-1目标提取液(pH值9,DAKO Agilent)中,并在97°C的Lab Vision PT模块(Thermo Fisher Scientific)中孵卵40分钟。冷却至室温后,将载玻片与盖板组装在Sequenza架上,并用PBS洗涤缓冲液清洗。用1× TBS IHC洗涤缓冲液,Tween 20, 3% (v/v)正常驴血清(Sigma-Aldrich)、0.1% (v/v)冷鱼皮凝胶(Sigma Aldrich)、0.1% (v/v) Triton X-100和0.05% (v/v)叠氮化钠在室温下封闭组织1 h。在Sequenza上腔中加入200 μL抗体稀释缓冲液,对阻塞缓冲液进行清洗。待稀释缓冲液流过后,将120 μL的抗体悬浮板加入载玻片。然后将Sequenza架放置在4°C过夜(16小时)。
过夜孵育后,用1ml PBS洗涤缓冲液清洗载玻片两次,并将载玻片固定在2%戊二醛(电子显微镜科学)的低钡PBS溶液中5分钟。将载玻片依次在PBS(一次清洗)、0.1 M Tris (pH 8.5)(三次清洗)、ddH2O(两次清洗)中清洗30 s,每次清洗三次,然后在70%乙醇(一次清洗)、80%乙醇(一次清洗)、95%乙醇(两次清洗)、100%乙醇(两次清洗)中洗涤脱水。切片在成像前在真空下干燥。
使用与MIBI-TOF染色组相同的抗体克隆,对5个靶点进行单发色免疫组化(表2)2).IHC组织切片的脱蜡、表位提取、阻断、杂交和洗涤与MIBI相同,但添加3% H阻断内源性过氧化物酶活性2O2(Sigma Aldrich)在ddH2O表位提取后。一夜一抗染色后,用1ml洗涤缓冲液清洗组织两次,用impress universal(抗小鼠/抗兔)试剂盒(Vector实验室)检测抗原:抗体复合物。显色液(DAB)孵育40 s,用PBS停止反应。切片用苏木精反染。
复查H&E组织切片,以确定缺失的组织芯、大面积坏死的组织芯、组织折叠和任何其他宏观缺陷。手工检查所有IHC和MIBI-TOF数据是否有过多的背景染色。
MIBI-TOF成像
每次重切的图像采集顺序根据切片编号随机化,并在6天内分别进行(表1).在每次重切的每次运行中,TMA核心的获取顺序也是随机的(表1).面积归一化Xe+一次离子剂量9na *hr*mm−2用于所有MIBI-TOF图像采集。在每次成像运行结束时,使用钼箔标准来量化灵敏度和质量分辨率(补充图。1).
图像处理
MIBI-TOF数据处理使用Ionpath的标准化管道进行。在数据提取过程中,为了只捕获与单原子同位素标签对应的单原子峰,并避免含有氢的多原子离子,我们使用质量窗口(X-0.3, X)集成了所有的峰,其中X是每个金属报告标签的标称质量电荷比(m/z)。
如前所述,使用预先定义的Ba、Ta和Au阈值去除裸露幻灯片产生的背景信号19,23.由双原子报告加合物和多原子碳氢化合物引起的等压干扰使用经验定义的补偿系数迭代去除(补充表1).最后,我们使用了几个密度指标的组合,包括可达性密度、连通性和Voronoi细分来估计和去除每个通道的噪声(补充表2).这些密度指标同时利用了空间和强度信息。去除背景和噪声后,任何非零值的像素都被认为是阳性的。
用NanoZoomer切片扫描仪在×40倍率下(Hamamatsu)对苏木精反染的免疫组化切片进行数字化。通过光谱解混从原始反染图像中提取DAB通道,对染色批中所有图像共同设置苏木素和各DAB通道的阈值。通过自动图像直方图阈值分割来设置初始阈值近似。然后由三名训练有素的用户在对照和额外的图像上确认阈值,并进行调整,直到达成共识(补充表3.).
标记定量
在所有MIBI-TOF图像中,对于每个标记,量化每个组织核心中阳性像素的频率和平均强度。这些图像的正像素百分比(PPP)和平均像素强度(MPI)的结果值,然后根据来自组织本身的内源性含碳质量峰值的信号强度进行归一化。因为单位面积的碳含量在组织样本之间是相对一致的,我们可以使用这个信号作为内部标准,在研究过程中对探测器灵敏度的变化进行归一化。在36、37和38 m/z处的峰分别对应3c、3C1H和3C2H对每幅图像进行积分(这里称为C我),并计算所有六个连续部分中所有图像的这些值的平均值(参考此处)Cavg).计算归一化系数(CN)对于给定的组织核心,C我因为组织核心被Cavg(CN=C我/Cavg).的中位数和标准差CN所有组织核的平均值为1.03±0.3。归一化前,归一化系数与PPP呈正相关。规范化后,这种关系不再明显(补充图。2).对于IHC图像,使用反卷积和阈值DAB图像(PPP)量化阳性像素的频率。
可重复性和一致性
为了评估连续切片的重现性,使用最小二乘线性回归将每个切片组织核心的PPP与所有六个连续切片的平均PPP进行比较。同样,使用最小二乘线性回归来比较每个切片中组织核心的MPI与所有六个连续切片的平均MPI。对于每个比较,我们计算斜率(米)和决定系数(R2).为了评估MIBI-TOF与单一免疫组化的一致性,我们使用最小二乘线性回归来比较MIBI-TOF和IHC在相邻序列组织切片中获得的PPP值。
细胞分割和表型
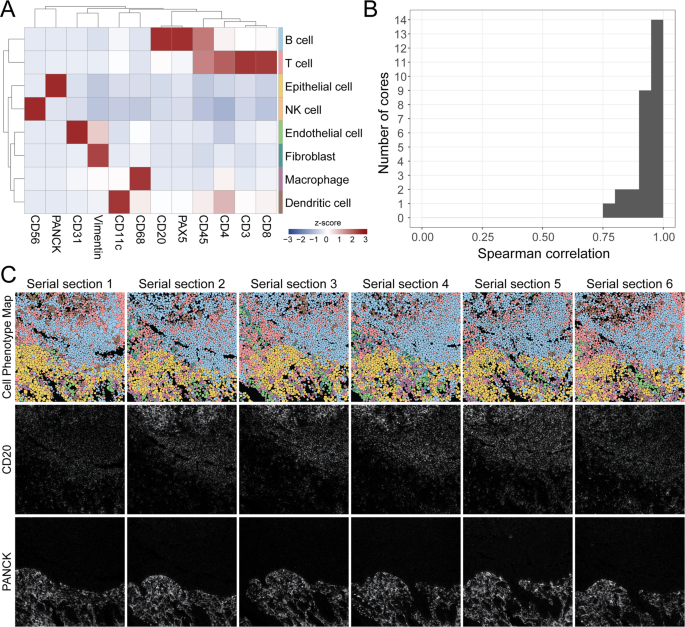
为了描述MIBI-TOF图像中单个细胞的位置,我们使用预先训练的Mesmer卷积神经网络架构进行细胞分割24.我们使用dsDNA作为核标记,HLA I类和na - k - atp酶作为膜标记作为网络输入。Mesmer的输出是图像中每个单元格的位置。
在细胞分割之后,分析管道的下一步是确定每个单个细胞的表型。我们使用了一种基于细胞平均标记表达的无监督聚类方法。这种方法已经在我们以前的出版物中使用过19,20.,21,25.对于每个细胞,确定每个标记物的总表达量,然后按细胞大小归一化。99.9%的归一化应用于每个标记。使用FlowSOM将细胞聚成100个簇26基于12个表型标记:CD3、CD4、CD8、CD11c、CD20、CD31、CD45、CD56、CD68、PanCK、Pax5和Vimentin的表达。找到了100个聚类中每个聚类的平均表达量z计算100个聚类中每个标记的-得分。所有z-分数被限制在3分,这样最大z- 3分。使用这些z-评分的表达式值,100个聚类采用分层聚类的方法进行元聚类,形成20个元聚类。然后,通过评估标记物的表达,手动注释每个细胞元簇的细胞表型,从而得到总共8种细胞类型:B细胞、T细胞、树突状细胞、巨噬细胞、NK细胞、成纤维细胞、内皮细胞和上皮细胞。为了评估同一组织核心的系列切片之间细胞表型的一致性,我们量化了每张图像中每种细胞类型的数量,然后计算了每个核心的系列切片图像之间这些频率的平均斯皮尔曼相关性。
结果
MIBI-TOF序列切片和IHC一致性的实验设计
为了评估MIBI的重复性以及相对于单发色IHC的基准,我们从本研究构建的TMA中取了13个系列切片(见方法),包括无病对照和多种类型的癌症、肉瘤和中枢神经系统病变(图2)。1).在这里,除H&E和未染色的组织对照外,每隔一页玻片都进行了染色和处理,用于MIBI-TOF分析或针对指示目标的单一显色IHC。为了控制由于组织染色和成像日产生的批次效应引起的混杂因素,每个TMA染色和成像的顺序按照连续切片顺序随机化。此外,对于每个TMA,每个组织核心的获取顺序也是随机的(表1).
MIBI-TOF串行分段一致性
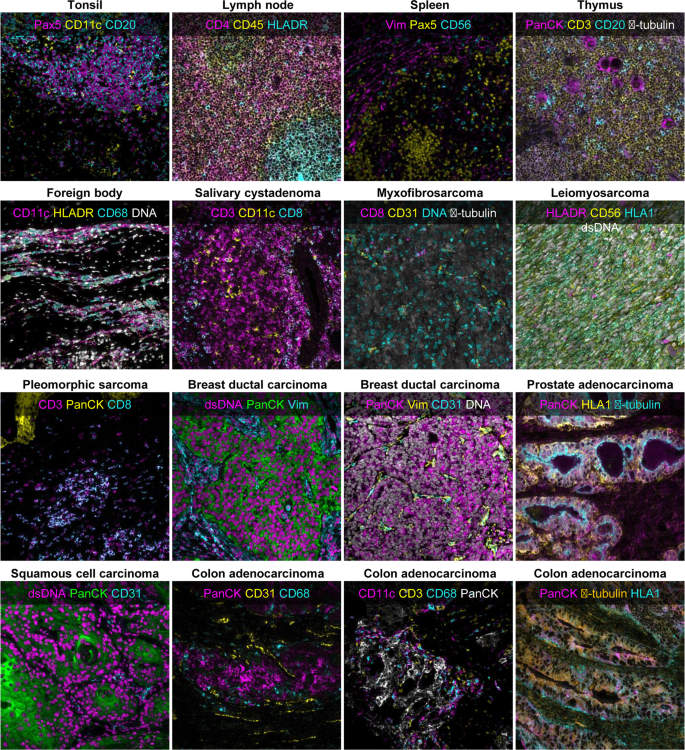
对于MIBI-TOF, 21个组织核各6个连续切片用16-plex抗体面板染色,并对总共126张MIBI-TOF图像进行分析2).在数据采集之前,使用钼箔标准对仪器稳定性进行了评估,我们知道离子计数的预期范围。一次离子电流和质量分辨率在整个研究过程中变化不显著(CV分别为1.8%和7%。1 a, B).离子检测器灵敏度的微小变化(CV = 22%)在历史规范范围内,未发现影响后续定量比较(补充图。1 c).与参考H&E系列切片的幻灯片扫描相比,每个组织核心的场是手动共同注册的,显示出高度一致的组织学特征(补充图)。3.).在所有6张TMA切片中,抗体染色板中的所有靶点都获得了高质量的MIBI-TOF成像数据。对每个标记物的手工评估显示,在正常和疾病组织核心中,对目标细胞都有稳健和特异性的染色。2).
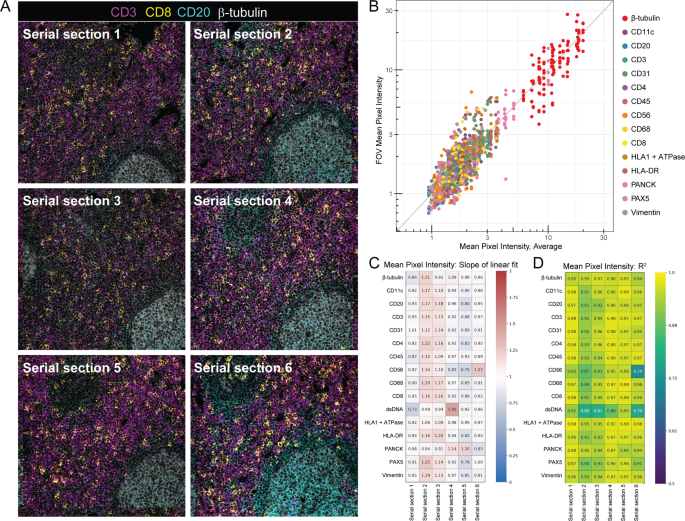
通过比较每个TMA系列切片中的标记物强度和丰度来评估分析的重复性。来自代表性组织核心的六个连续切片的共同注册视场(fov)的示例如图所示。3,说明了序列切片之间的高再现性。T细胞标记物CD3和B细胞标记物CD20的PPP值分别为22.6%±2.5和20.6%±8.1 (mean±SD)。CD20观察到的显著较高的标准偏差是由于从组织核心取切片时,两个生发中心(以B细胞标记物CD20的表达为标志)所占区域的切片到切片的变化。
对每个TMA系列切片中每个组织核心的标记物强度(MPI)和标记物频率(PPP)的归一化值进行线性回归(见方法),以确定与所有六个系列切片平均值的一致性程度。对于MPI(以正像素量化平均标记强度),合成斜率(米)和决定系数(R2)的值均接近1,且a米1.0±0.13和aR2的0.94±0.04 (mean±SD,图;3罪犯).因此,MPI捕获标记强度米而且R2接近1的值表示当存在标记时,该标记的亮度在串行部分中高度一致。类似地,对于PPP,它量化了给定标记的正数像素的总数,米而且R2在所有串行切片和标记也接近1,与a米1.0±0.17和aR2的0.95±0.04 (mean±SD,补充图4).购买力平价米而且R2接近1的值表示同一核心的串行部分的正像素数是一致的。除了整个TMA一致性外,我们还检查了核间变异性(补充图。5),表明核心内的序列切片是相对和谐的,并且在标记之间的变化是一致的。总的来说,MPI和PPP表明,在染色和成像日随机进行的同一组织核心的系列切片的MIBI-TOF图像高度一致,为MIBI-TOF的可重复性提供了证据。
MIBI-TOF细胞免疫表型分配的再现性
高维成像数据分析的一个重要步骤是通过图像中的免疫表型鉴定单细胞谱系。因此,我们接下来评估了每个组织核心的连续切片上单细胞注释的再现性。我们实验室最近开发了一种细胞分割算法,可以准确识别细胞在固体组织中的位置,而不需要手动微调或用户调整24.我们使用这种方法来描绘图像中单个细胞的位置(补充图。6),然后使用无监督聚类方法将单细胞分为8种细胞系:B细胞、T细胞、树突状细胞、巨噬细胞、NK细胞、成纤维细胞、内皮细胞和上皮细胞(图2)。4,补充图。7).在对每张图像中的细胞进行分类后,我们量化每张图像中每种细胞类型的数量,并通过计算每个组织核心的系列切片之间这些频率的平均斯皮尔曼相关性来评估序列切片之间的一致性(图2)。4 b).在比较MIBI-TOF系列切片图像时,我们观察到广泛的组织学特征是保守的,如生发中心或血管(图2)。3.一个,4摄氏度,补充图。7 a、B).平均Spearman相关性较高,为0.94±0.05 (mean±SD),说明在高维图像分析中,细胞表型分析这一必要步骤在相同组织核心的MIBI-TOF图像之间是可重复的。
我们预期这些Spearman相关值会有变化,因为同一组织核心的连续切片每个都是4 μm的切片(额外的连续切片用于IHC一致性分析)。每个组织共获得13个连续切片用于MIBI-TOF或IHC(见方法)。因此,当比较相距48 μm的连续切片时,我们会期望真正的生物学差异。为了量化这些差异,我们比较了每个组织核心的连续切片作为滑动距离的函数(补充图。8).对于具有多细胞组织学结构的组织,切片相似度与切片距离之间存在明显的线性关系(补充图)。8).例如,淋巴结生发中心的大小以一种可预测的方式变化,与椭球结构的连续切片一致。然而,对于缺乏周期结构的其他组织,如癌症,当我们切开组织核心时,我们看到细胞组成的差异非常小(补充图。8 b).综上所述,大多数差异被确定为主要归因于组织组成的变化。
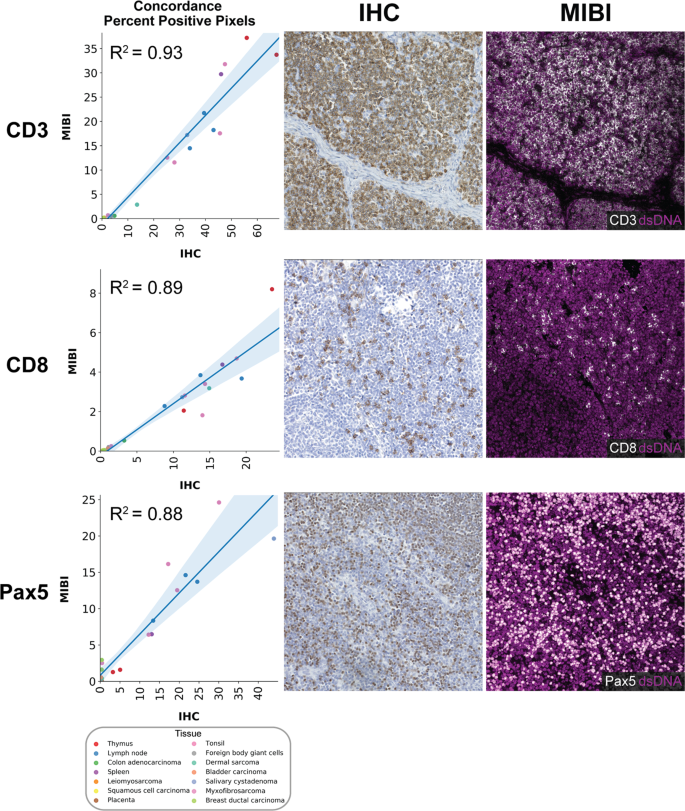
MIBI-TOF与单发显色IHC的一致性
显色性免疫组化是绝大多数实体肿瘤疾病预后和治疗选择的标准方式。因此,我们评估了一致性,以确定MIBI-TOF的多重成像是否可以定量概括单一免疫分析。与上述用于跨系列切片比较MIBI-TOF数据的分析相似,在MIBI-TOF图像中与单一显色IHC染色共同配准的标记阳性像素的频率在所有21个组织核上通过线性回归进行比较。因为我们交替重切MIBI-TOF和IHC,所以我们可以在相邻的连续切片上比较MIBI-TOF和IHC。获得CD3、CD8、Pax5、PanCK和CD68这5个靶点的单发色IHC图像。采用线性回归方法,将单发免疫组化检测相邻序列切片中CD3、CD8、Pax5、PanCK和CD68的标记物阳性值与MIBI-TOF检测的相应值进行比较(图2)。5,补充图。9).对于所有五个目标,MIBI-TOF和IHC的一致性都很高R2为0.85±0.08(均数±标准差,均R2> 0.7)。值得注意的是,比较IHC和MIBI-TOF的线性拟合斜率始终小于1(图2)。5,补充图。9).两种方法之间比例差异的原因可以归结为信号检测的差异。IHC采用二抗扩增,而MIBI-TOF仅直接检测标记的一抗。因此,在显色IHC中使用的非线性放大将导致每个细胞上更多的阳性像素27.
与单发色IHC染色共配的MIBI-TOF图像比较的代表性图像(CD3:扁桃体,CD8:淋巴结,Pax5:淋巴结)。每个数据点代表单个组织核的MIBI-TOF和IHC PPP。数据点的颜色表示组织类型。阴影区域表示95%置信区间。附加标记示于补充图。9.
讨论
在本研究中,我们评估了MIBI-TOF的重复性,方法是评估TMA的六个连续切片的检测一致性,包括无病扁桃体、淋巴结、胸腺和脾脏,以及多种类型的癌症、肉瘤和中枢神经系统病变。重要的是,每个系列切片分别在6天内用16标记染色板进行独立染色,然后在6天内进行分析。在每个序列切片中分析21个组织核,共126张MIBI-TOF图像。使用最小二乘线性回归将每个序列切片的正像素的染色强度和频率(分别为MPI和PPP)与所有运行的平均值进行比较。在所有的比较中,同一组织核心的连续切片之间的一致性很高米为1.0±0.13R2为0.94±0.04米为1.0±0.17R2(均值±标准差)为0.95±0.04。手工检查每个标记物的染色模式,以确认相关的适当亚细胞定位和组织学分布。例如,所有图像中扁桃体和淋巴结阳性区域的CD20染色均被证实为膜定位并富集于卵泡。此外,我们评估了连续切片中细胞表型的再现性,发现在连续切片中,使用我们的数据分析管道识别的细胞的位置和谱系高度一致,连续切片之间细胞类型频率的平均斯皮尔曼相关性为0.94±0.05(平均值±标准差)。重要的是,用于从MIBI-TOF图像到细胞表型的数据分析管道几乎完全自动化。细胞分割和聚类是完全自动化的,不需要用户输入,在用细胞注释标记细胞集群时只需要最小的用户干预,这表明MIBI-TOF图像的生物注释是鲁棒的,不需要大量的研究人员干预。总之,一致的染色、成像和特征提取的组合说明了MIBI-TOF的高度可重复性。此外,我们通过MIBI-TOF评估了5个标记物与单一免疫组化的PPP一致性,发现MIBI-TOF和单一免疫组化与平均值高度一致R20.85。
涉及连续组织切片的验证研究的一个局限性是,由于每个组织切片略有不同,且不是均质大样本,因此无法获得精确的生物学复制。在本研究中,由于组织切片各为4 μm,共获得13个连续切片,因此第一个组织切片和最后一个组织切片轴向间隔48 μm。此外,当平衡试验复制与与正交试验的一致性时,如这里的单发免疫组化,需要优先考虑使用物理相邻的玻片。在这种情况下,我们选择了物理上相邻的幻灯片来对单发IHC进行基准测试,因为任何一个标记的分布都严重依赖于细胞组成。在邻近切片中,大约10 μm的淋巴细胞保存最好。虽然在这个体积的组织中会有真正的生物学差异,但这项研究代表了次好的情况,其中我们比较了相邻的连续切片作为真实复制的代表。此外,这是一项单点研究,所有染色和成像都在我们的实验室进行。我们目前正在计划一项多地点研究,以评估仪器性能和组织染色的机构间差异。尽管如此,我们相信这里概述的基准测试的总体指标将足以评估这些未来的努力。
这项研究是癌症免疫监测和分析中心癌症免疫数据共享网络(CIMAC-CIDC)的一部分22这是一个NCI癌症登月计划,为免疫治疗临床试验提供技术和专业知识。多重组织成像是充分表征肿瘤免疫微环境的一个完整的分析方法,它需要同时分析多种肿瘤和免疫细胞类型。MIBI-TOF可以常规提供定量、多路成像数据,与FFPE组织档案和常规解剖病理工作流程兼容。试剂和仪器的技术创新将进一步提高MIBI-TOF的吞吐量和多路复用能力。在未来,我们设想MIBI-TOF不仅将用于可重复的基础科学研究,而且还可以在临床中采用定量空间特征,以实现更准确的诊断和治疗选择。与此同时,随着其他以ihc为中心的空间成像技术的发展,本研究中提出的分析框架和方法应作为评估的指南。
数据可用性
在当前研究期间生成和/或分析的数据集可在Zenodo上获得:https://doi.org/10.5281/zenodo.5945388.分析代码可以在Github上找到:https://github.com/angelolab/publications/tree/master/2022-Liu_Bosse_etal_CIMAC.
参考文献
Levenson, r.m., Borowsky, a.d. & Angelo, M.用于高度多重细胞分子成像的免疫组织化学和质谱。实验室。Investig。95, 397-405(2015)。
Matos, L. L., de, Trufelli, d.c., de Matos, M. G. L. & da Silva Pinhal, m.a.免疫组化作为生物标志物检测和临床实践的重要工具。Biomark。的见解5, 9-20(2010)。
Chlipala, e.a.,等人。一种定量测定免疫组化染色再现性的图像分析方法。达成。Immunohistochem。摩尔。Morphol。28, 428-36(2020)。
Thunnissen, E.等人。免疫组化的应用提高了小细胞肺癌的诊断及鉴别诊断。一组要求苛刻的案例的国际可重复性研究。J.胸廓肿瘤。12, 334-46(2017)。
Yatabe, Y.等。肺癌诊断免疫组织化学最佳实践建议。J.胸廓肿瘤。14, 377-407(2019)。
免疫组化在乳腺癌中的意义。世界J.克林。肿瘤防治杂志.5, 382-92(2014)。
McCabe, A., doled - filhart, M., Camp, R. L. & Rimm, D. L.原位蛋白表达、抗体浓度和预后的自动定量分析(AQUA)。国立肿瘤研究所。97, 1808-15(2005)。
陶布,J. M.等。癌症免疫治疗学会关于多重免疫组化(IHC)和免疫荧光(IF)染色和验证的最佳实践的声明。j . Immunother。癌症8, e000155(2020)。
Zimak, J., Schweller, R. M., Duose, D. Y., Hittelman, W. N. & Diehl, M. R.通过动态DNA复合物的可互换反应编程原位免疫荧光强度。Chembiochem13, 2722-8(2012)。
Neher, R. & Neher, E.优化荧光图像中多个标签分离的成像参数。j . Microsc。213, 46-62(2004)。
帕特尔SS,罗迪格SJ。组织成像方法概述。在癌症免疫治疗的生物标志物:方法和方案(eds。图林,M.,塞萨诺,A.和马林科拉,F.M.) 455-65(施普林格纽约,2020)。https://doi.org/10.1007/978-1-4939-9773-2_21.
Binnewies, M.等人。了解肿瘤免疫微环境(TIME)有助于有效治疗。Nat,地中海。24, 541-50(2018)。
用于发现和医疗保健应用的基于多表位的组织成像。细胞系统。2, 225-38(2016)。
Goltsev, Y.等人。CODEX多重成像对小鼠脾脏结构的深度分析。细胞174, 968年。e15(2018)。
舒尔茨,D等人。乳腺癌组织样本中mRNA和蛋白质亚细胞分辨率的同时多重成像细胞系统。6的技能。e5(2018)。
Giesen, C.等人。肿瘤组织的高多重成像与亚细胞分辨率的大规模细胞术。Nat方法。11, 417-22(2014)。
安吉洛,M.等人。人类乳腺肿瘤的多重离子束成像。Nat,地中海。20., 436-42(2014)。
Keren, L.等人。MIBI-TOF:一个涉及细胞表型和组织结构的多路成像平台。科学。睡觉。5, eaax5851(2019)。
Keren, L.等人。多重离子束成像显示三阴性乳腺癌中的结构化肿瘤免疫微环境。细胞174, 1373年。e19(2018)。
Risom, T.等。向浸润性乳腺癌的转变与肿瘤间质结构和组成的进行性变化有关。细胞185, 299 - 310。e18(2022)。
麦卡弗里,e.f.,等。人结核肉芽肿的免疫调节格局。Immunol Nat。https://doi.org/10.1038/s41590-021-01121-x(2022)。
陈海霞,等。癌症免疫治疗生物标志物免疫分析网络:癌症免疫监测和分析中心和癌症免疫数据共享(CIMAC-CIDC)。中国。癌症Res。27, 5038-48(2021)。
Baranski, A.等人。MAUI (MBI分析用户界面)-用于多路海量成像的图像处理管道。公共科学图书馆第一版。医学杂志。17, e1008887(2021)。
格林沃尔德,n.f.,等人。利用大规模数据注释和深度学习实现具有人类水平性能的组织图像全细胞分割。生物科技Nat。》。https://doi.org/10.1038/s41587-021-01094-0(2021)。
刘,C. C.,等。多路离子束成像:病理生物学的洞见。为基础。启分册。动力机械。说17, 403-23(2022)。
Van Gassen, S.等人。FlowSOM:使用自组织地图进行细胞术数据的可视化和解释。血细胞计数一次87, 636-45(2015)。
里姆,d。l。布朗不能为你做的。生物科技Nat。》。24, 914-6(2006)。
资金
这项工作得到了DOD EOH W81XWH2110143, 1-DP5-864 OD019822, 1R01AG056287, 1R01AG057915, R01AG068279, 1UH3CA246633, 1U24CA224309以及比尔和梅林达盖茨基金会的支持。
作者信息
作者及隶属关系
贡献
s。v。和m。r。制造了组织芯片。M.B.进行实验和成像。a.k., a.k., C.C.L和S.H.N.进行了数据分析。c.c.l和M.A.写的论文。s。c。b。r。k。s。m。h。审阅了论文。s.c.b和M.A.监督这项工作。
相应的作者
道德声明
相互竞争的利益
M.A.和S.C.B.是MIBI技术相关专利的发明人。M.A.和S.C.B.是Ionpath Inc.的顾问、董事会成员和股东。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
权利和权限
关于本文
引用本文
刘忠忠,波士,M,孔,A。et al。通过飞行时间(MIBI-TOF)的多重离子束成像,在档案人体组织中可重复的高维成像。实验室投资102, 762-770(2022)。https://doi.org/10.1038/s41374-022-00778-8
收到了:
修改后的:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41374-022-00778-8
这篇文章被引用
人类神经病理学的单细胞空间蛋白质组成像
神经病理学通讯学报(2022)
用单细胞技术推进T细胞癌症治疗
自然医学(2022)