摘要
目的:阐明本病不同临床病理谱的机制EWSR1 /付::分子易位相关肿瘤,我们对大量肿瘤类型的融合转录物变异、复发性基因改变(突变、拷贝数改变)、基因表达和甲基化谱进行了全面的基因组分析。的分布EWSR1/付家融合的合作伙伴,ATF1,CREB1,CREM而且外显子受累在不同肿瘤类型中有显著差异。我们的靶向测序显示,继发遗传事件与肿瘤类型相关,而不是融合类型。在39例进行定向NGS检测的病例中,18例(46%)有继发性OncoKB突变或拷贝数改变(共29例继发性遗传事件),其中15例(52%)复发。二次复发,但互斥,叔启动子和CDKN2A突变仅在透明细胞肉瘤(CCS)中被发现,并与较差的总生存率相关。CDKN2A / B纯合子缺失在血管瘤样纤维组织细胞瘤(AFH)中反复出现,局限于转移病例。mRNA的上调MITF,CDH19,PARVB,PFKP与AFH相比,在CCS中发现,并与低甲基化特征相关。相比之下,S100A4而且XAF1在AFH中有差异的上调和低甲基化,而在CCS中没有。甲基化谱的无监督聚类显示,CREB家族易位相关肿瘤形成相邻但紧密、独特的聚类。肉瘤甲基化分类器能够准确地将100%的CCS病例匹配到正确的甲基化类别;然而,当应用于其他组织时,它是次优的。综上所述,我们对EWSR1 /付::分子易位相关肿瘤在转录变异体、具有预后意义的继发性基因改变、基因表达和甲基化模式中主要发现组织型相关,而不是融合型相关。
简介
复发性基因融合EWSR1 /付与cAMP反应元件结合蛋白(CREB)家族成员(ATF1,CREB1而且CREM)在跨越广泛临床病理谱的多种肿瘤类型中共享。尽管有相似的基因融合,成员EWSR1:易位相关肿瘤的CREB家族表现出明显不同的临床病理特征。典型的例子是血管瘤样纤维组织细胞瘤(AFH)与透明细胞肉瘤(CCS) -两种形态不同的肿瘤,前者大多与良性行为相关,而后者是一种侵袭性肉瘤,具有高转移潜力和不良预后,如我们队列的生存分析所示。胃肠道透明细胞肉瘤样肿瘤(GICCS,又称胃肠道神经外胚层肿瘤)和唾液腺透明细胞癌(HCCC)相对于AFH和CCS的总生存率中等。
为了阐明其差异背后的分子机制,我们对融合转录本变异、继发性复发性基因改变(突变、拷贝数改变)、基因表达和甲基化特征进行了全面的基因组分析,这些特征涉及由遗传学家定义的大型肿瘤类型队列EWSR1 /付::分子基因融合。具体来说,本研究纳入的肿瘤包括AFH、CCS、GICCS、HCCC、透明细胞牙源性癌(CCOC)、恶性上皮样肿瘤,倾向于间皮瘤(ME)、间皮瘤(Meso)、黏液样间充质瘤(MMT)和原发性肺黏液样肉瘤(PPMS)。
材料与方法
病例选择和研究队列
经机构审查委员会批准后,根据肿瘤类型和/或肿瘤的存在,从纪念斯隆凯特琳癌症中心(MSKCC)手术病理档案或合作机构中确定病例EWSR1/付::ATF1/CREB1/CREM融合。所有137例病例的诊断包括两种融合伴的分子证实:37例采用荧光原位杂交,24例采用逆转录PCR (RT-PCR), 29例仅采用Memorial Sloan Kettering-Integrated Mutation Profiling of action Cancer Targets (MSK-IMPACT), 10例仅采用MSK-Fusion, 10例同时采用MSK-IMPACT和MSK-Fusion, 21例采用TruSight RNA Fusion Panel (Illumina, San Diego, CA),其余病例采用转诊机构进行的NGS检测。对已发表文献的荟萃分析基于对补充表中所有列出实体中报告的任何融合或基因重排的详尽文献检索1我们可以在PubMed上找到。
DNA序列和RNA序列
MSK-IMPACT工作流程和数据分析的详细描述,一种基于杂交捕获的实体肿瘤靶向DNA NGS检测,以及MSK-Fusion,一种基于扩增子的靶向RNA NGS检测,使用Archer™FusionPlex™标准协议,已经在前面描述过1,2.
850k甲基化阵列
甲基化阵列协议的细节已在前面描述3..简单地说,对于每个样本,250 ng输入DNA用于亚硫酸氢盐转化(EZ DNA甲基化试剂盒;Zymo研究;目录号D5002),然后使用Infinium HD FFPE DNA恢复试剂盒(Illumina;目录号WG-321-1002)。所有样品均在Infinium methylationEPIC 850k BeadChip阵列上处理,并使用Illumina iScan扫描。由Infinium数组查询的每个CpG站点都由一个唯一的cg标识符标识,格式为cg#,其中#是一个数字。每个CpG位点的甲基化水平使用β值(0到1之间的连续值)进行量化,计算为甲基化信号/总信号加偏置的比率。共对80个样本进行850k甲基化阵列分析,包括:7个AFH, 4个CCS, 8个GICCS,并与51个不同组织型的软组织肿瘤(4个血管肉瘤,27个胃肠道间质肿瘤,1个HCCC, 1个ME, 3个Meso, 11个副神经节瘤,4个小蓝色圆形细胞肿瘤)和10个正常组织(8个人外周血白细胞样本,2个正常肾上腺髓质)进行比较。日志的最小截断点2采用FC (fold change) >1.0和FDR < 0.01进行差异甲基化分析t测试,重点是比较7 AFH与所有其他样品和4 CCS与所有其他样品。采用t分布随机邻域嵌入(t-SNE)方法进行无监督分层聚类,使用上述样本和从海德堡肉瘤甲基化分类器参考队列下载的其他原始IDAT文件4(见补充图)3.有关样品和方法的详细信息)。
基于DNA甲基化谱的肉瘤分类
Koelsche等人描述了基于DNA甲基化的机器学习肉瘤分类算法的细节4.这种基于随机森林的机器学习算法是在德国海德堡的德国癌症研究中心(DKFZ)开发的。简单地说,该方法基于包含广泛肉瘤的1077个样本的参考队列定义了62种甲基化分类。分类器使用0到1之间的校准分数量化样本指定甲基化类的置信度。所有甲基化类别的所有校准评分之和为1.0。有信心的匹配通常被认为是>0.9,差匹配<0.54.采用DNA甲基化分类算法分析的22例病例中,80例进行甲基化分析的样本中AFH 6例,CCS 4例,GICCS 8例,ME 1例,Meso 3例。
Affymetrix芯片基因表达分析
微阵列协议的细节已在前面描述过5,6.使用RNAwiz RNA分离试剂(Ambion)分离RNA,并通过无rnase - DNase (Qiagen)色谱柱进行分离。然后将10微克标记和碎片化的cRNA杂交到人类基因组U133A表达阵列(Affymetrix, Santa Clara, CA)上。杂交后染色、洗涤和扫描按照制造商(Affymetrix)的说明进行。使用Affymetrix Microarray Analysis 5.0 (MAS 5.0)软件导出原始表达数据。数据使用标度目标强度500进行归一化,以考虑全球芯片强度的差异。表达式值使用以2为底的对数进行转换。Affymetrix U133A阵列基因表达分析在共58样本,包括3 AFH 4 CCS,相比1 GICCS, 44软组织肿瘤各种histotypes(3成人纤维肉瘤、5血管肉瘤、平滑肌肉瘤3 10胃肠道间质瘤,3黏液样脂肪肉瘤,6副神经节瘤,4个蓝色小圆细胞肿瘤,4单独纤维肿瘤,3滑膜肉瘤,3未分化的多形性肉瘤)和6正常组织(肾上腺、脑、肾、小肠,胃,睾丸)。对于差异基因表达分析,log的最小截断值2FC(折叠变化)>1.0和FDR调整p< 0.01用于t测试。我们将每种组织类型与所有其他肿瘤进行比较。例如:CCS(4例)与所有其他(54例)在一个分析,AFH(3例)与所有其他(55例)在另一个分析。使用pheatmap R包1.0.12版本进行无监督分层聚类,并使用Ward 's linkage和Euclidean distance进行聚类。
基因表达与甲基化分析的整合
首先,我们通过设置调整后的错误发现率(FDR)进行差异基因表达和差异甲基化分析p值为0.01和最小日志2FC(折叠变化)为1.0t每次比较一种组织类型与所有其他肿瘤(例如,CCS与所有其他肿瘤,AFH与所有其他肿瘤)。随后,为了整合转录组和甲基化数据,我们基于log对所有差异表达的基因进行匹配2根据log, FC >1.0和FDR < 0.01,甲基化差异2CCS和AFH比较,FC > 2.0和FDR < 0.01。在Affymetrix U133微阵列上的3个AFH、4个CCS和1个GICCS中,1个AFH和2个CCS与用于甲基化阵列的样品不重叠。
结果
临床病理的总结
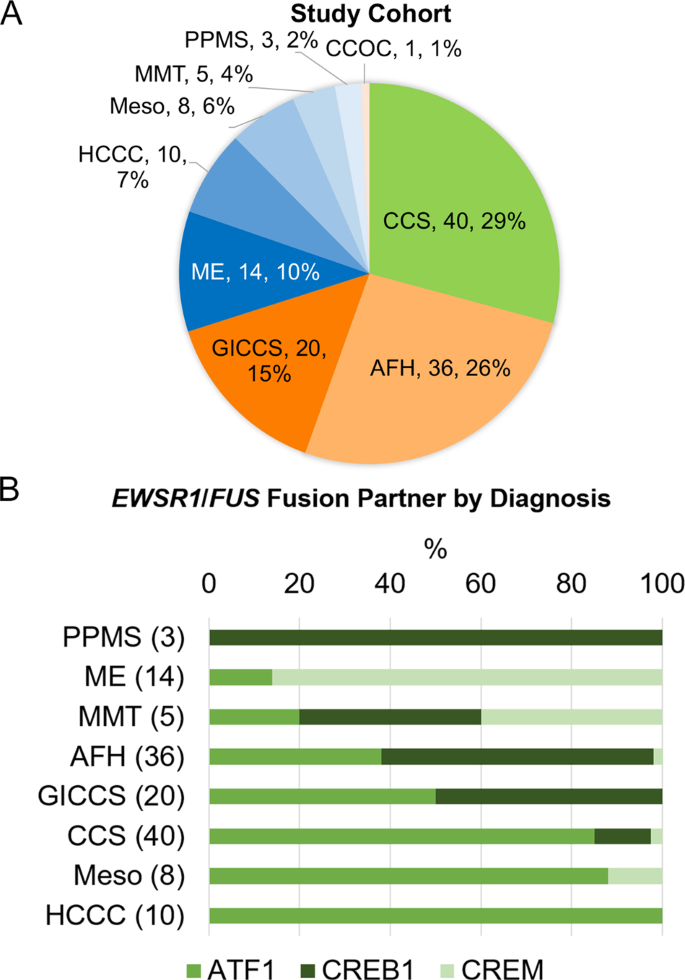
共鉴定出137例[女性76例,男性61例,平均年龄37岁(范围2-86)],包括:40例CCS(29%), 36例AFH(26%), 20例GICCS(15%), 14例ME(10%), 10例HCCC(7%), 8例Meso(6%), 5例MMT(4%), 3例PPMS(2%)和1例透明细胞牙源性癌(CCOC)(图)。1).HCCC、PPMS和CCOC的平均年龄高于AFH、CCS、GICCS、MMT和ME(表2)1).正如预期的那样,AFH和CCS的主要部位是软组织,GICCS的主要部位是胃肠道/骨盆,MMT的主要部位是大脑,Meso和ME的主要部位是胸廓或腹盆腔,PPMS的主要部位是肺,HCCC的主要部位是小唾液腺。
经诊断的融合类型和转录变异
的分布EWSR1/付家融合的合作伙伴,ATF1,CREB1,CREM,在不同肿瘤类型间差异显著(卡方P< 0.0001)(图1 b).具体地说,EWSR1: ATF1融合是HCCC中唯一的融合类型(100%),CCS和Meso中主要的融合类型(85%);EWSR1: CREB1融合是PPMS中唯一的融合类型(100%)和AFH中主要的融合类型(60%);EWSR1 /付::CREM融合是ME的主要融合类型(86%)。CREB1而且CREM融合在MMT中平均分布(40%)。ATF1而且CREB1融合在GICCS中均匀分布。单例CCOC有EWSR1: ATF1融合。在137例病例中,仅有5例(4%)为窝藏病例付家融合:4例付:CREMME的融合,一个是a付:ATF1内消旋的聚变。
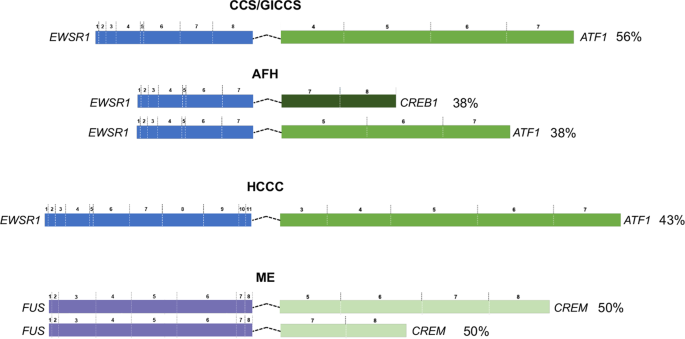
每种肿瘤类型的融合转录变体的外显子使用来自MSK-Fusion和/或MSK-IMPACT测试,在48例病例中可用(8例AFH, 18例CCS, 9例GICCS, 7例HCCC, 3例Meso, 1例PPMS, 2例ME)(表2)2).主要融合转录本变异为EWSR1ex8::ATF1CCS和GICCS中的ex4;EWSR1ex7::ATF1ex5和EWSR1ex7::CREB1AFH中的ex7;EWSR1ex11::ATF1HCCC中的ex3;付家ex8::CREMex5/7 for ME(图。2).补充表1总结了我们对已发表研究的荟萃分析得出的各种肿瘤类型的CREB家族融合变异,并将其与当前队列中检测到的肿瘤进行比较。
临床显著的复发性基因改变
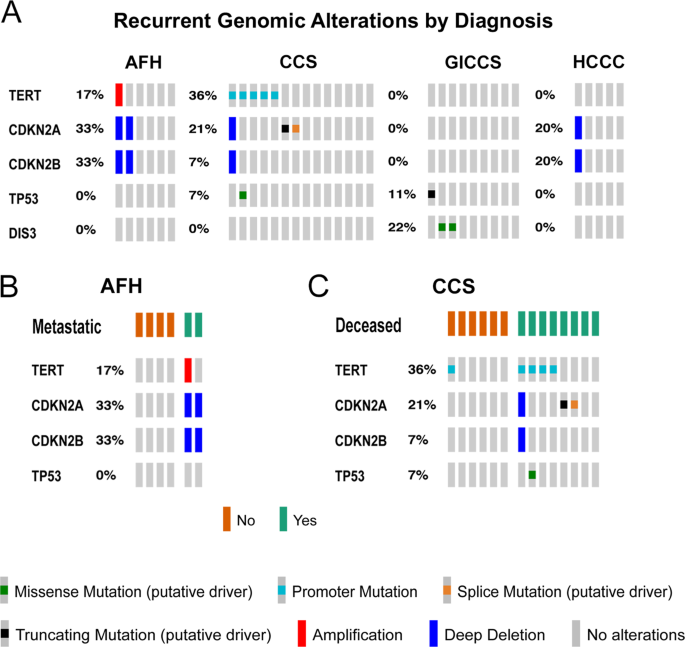
MSK-IMPACT分析39例[AFH 6例,CCS 14例,GICCS 9例,HCCC 5例,Meso 3例,PMMS 1例,透明细胞牙源性癌(CCOS) 1例]。只包括具有OncoKB注释的临床显著变异(Chakravarty 2017)(或已知复发热点)和继发性复发遗传改变(在我们队列中发生>1的事件)。意义未知的变异被排除。在39例进行定向NGS检测的病例中,18例(46%)有OncoKB突变或拷贝数改变(共29例继发性遗传事件),其中15例(52%)复发。具体地说,叔启动子热点突变(n5)和CDKN2AX51_splice和P81Lfs*30突变(n= 2)相互排斥,仅在CCS中识别。其他继发性复发性基因改变包括:TP53R248Q和T155Pfs*15个突变(n= 2, 1 CCS, 1 GICCS), 9p21.3 (CDKN2A/CDKN2B)拷贝数缺失(纯合缺失)(n= 4,2 AFH, 1 CCS, 1 HCCC),和DIS3D479G和D488N突变(n= 2,均为GICCS)(图;3).在3个Meso、1个PMMS或1个CCOC中均未发现继发性复发性基因改变。继发性复发性基因改变的类型与发病率无相关性EWSR1/付家融合伙伴类型(补充图)1).
有趣的是,AFH病例与CDKN2A / CDKN2B纯合缺失(n= 2,33%)仅在转移病例中发现,而其余的CDKN2A / CDKN2B未改变的AFH病例无转移(图;3 b).另一方面,CCS的情况叔启动子突变和CDKN2A功能丧失突变(移码和剪接位点突变)(n= 7,50%)与总生存率下降显著相关(Mantel Haenszel卡方P= 0.0196)(图3 c),中位生存期为5.13个月,而未发生改变的CCS病例为22.85个月(n= 7, 50%)。存在DIS3突变与GICCS的转移性和生存状态无关。
甲基化与基因表达的相关性
在Affymetrix U133A表达阵列上进行基因表达谱分析,将3例AFH、4例CCS和1例GICCS与44例不同组织类型的软组织肿瘤和6例正常组织样本进行比较。2及补充表2).将7个AFH, 4个CCS, 4个GICCS与51个不同组织型软组织肿瘤和10个正常组织样本进行850k甲基化阵列上的甲基化谱测试(补充表)3.).目的是确定差异基因表达(1.5 log2FC, FDR 0.01)和差异甲基化(4 log .2FC和FDR 0.01)EWSR1: ATF1-重新安排CCS和EWSR1: CREB1-重组AFH,分别与其他肿瘤类型相关。基因表达谱显示基因表达上调PMP22,MITF,SLC7A5,CDH19,WIPI1,菲英岛,PARVB,PFKP而非AFHSgk1, s100a4, xaf1而且LY96AFH表达,CCS不表达。来自Koelsche等人研究的额外甲基化数据。4(8例AFH, 7例CCS,以及不同组织型的软组织肿瘤和正常组织样本)被提取并纳入无监督t-SNE聚类分析。甲基化谱的无监督聚类显示,CREB家族易位肿瘤(AFH、CCS、GICCS、Meso)每个都形成紧密、不同的相互靠近的聚类(补充图)。3.及补充表3.).
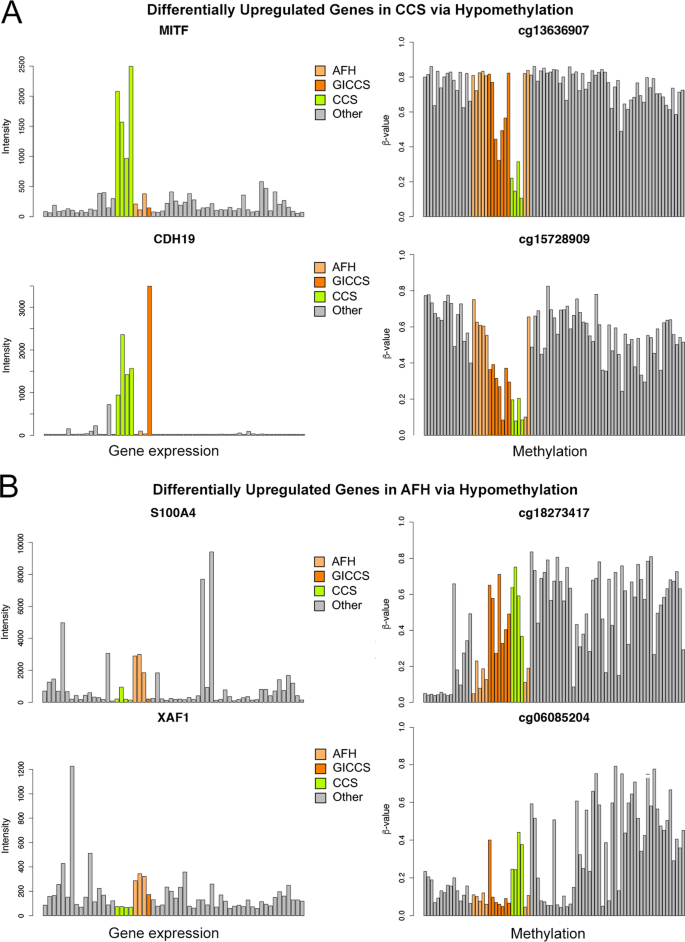
随后,根据染色体位置将差异表达基因与CpG位点进行匹配。我们根据对数匹配了所有表达差异的基因2根据log, FC > 1.0和FDR < 0.01,甲基化差异2FC > 2.0和FDR < 0.01。我们关注的是相应低甲基化的上调基因。我们的分析显示基因(MITF,CDH19,PARVB,PFKP), CCS中表达和低甲基化增加,而AFH中没有(图。4),以及基因(S100A4,XAF1), AFH中表达和低甲基化增加,而CCS中没有(图。4 b).MITF参与黑素生成并作为其核心基因特征的一部分在CCS中过表达5,7.CDH19而且PARVB参与细胞黏附并在原发性黑色素瘤中高度表达8.S100A4是否与细胞迁移和癌症转移有关9.XAF1促凋亡肿瘤抑制基因10.
一个CCS。BAFH。使用Affymetrix U133A对44个不同组织型的肿瘤和6个正常组织的3例AFH、4例CCS和1例GICCS进行比较2-fold变化阈值为1和P< 0.01。使用Infinium 850k甲基化阵列对29个不同组织型肿瘤和8个正常组织的7个AFH、4个CCS和4个GICCS进行比较2-fold变化阈值2和P< 0.01。差异表达的基因被匹配到CpG位点上,并以唯一的cg标识符(cg#)进行标识。在850k序列中,这4个基因的CpG位点数量如下所示S100A4, 14XAF127分MITF, 3个CDH19.其中,与基因表达呈负相关的CpG位点数如下S100A4, 6分XAF1, 3为MITF, 3个CDH19.图中显示了每个基因中最具代表性的CpG位点。简称:afh血管瘤样纤维组织细胞瘤,CCS透明细胞肉瘤,GICCS胃肠道透明细胞肉瘤样肿瘤。
利用肉瘤甲基化分类器预测肿瘤类型
Koelsche等人描述的基于dna甲基化的肉瘤分类算法。422例(AFH 6例,CCS 4例,GICCS 8例,ME 1例,Meso 3例)(表3.).该算法能够将4个CCS病例100%精确匹配到正确的甲基化类别(所有病例的校准评分= 0.99),但只能将33%(2 / 6)的AFH病例(校准评分分别为0.75和0.33)匹配到正确的甲基化类别。在原始分类器中,GICCS不是甲基化类。有趣的是,该算法匹配了1个GICCS与AFH(校准评分0.56)和2个GICCS与CCS(校准评分分别为0.65和0.96)。
生存分析
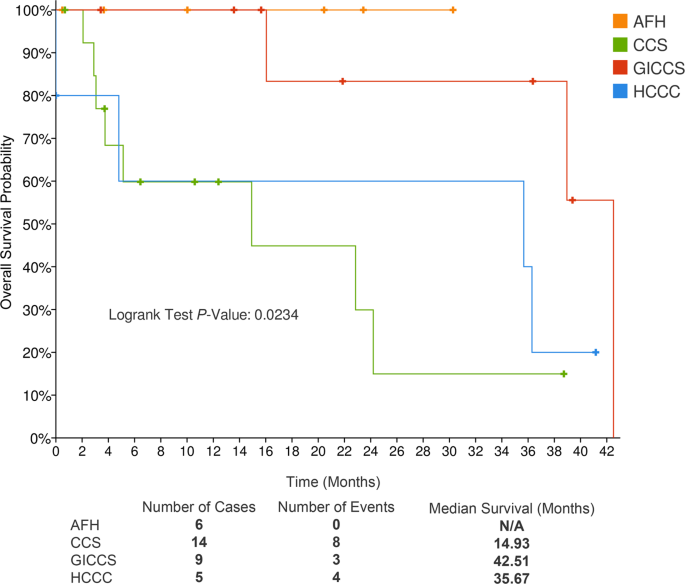
AFH、CCS、GICCS、HCCC的总体生存期有显著差异(log rank)P= 0.023), CCS与较差的生存相关(中位生存15个月),其次是HCCC(中位生存36个月),然后是GICCS(中位生存43个月)。在42个月的随访期间,所有AFH患者都存活了下来。5).
讨论
的EWSR1/FUS:易位相关肿瘤的CREB家族包含广泛和异质性的临床病理谱。为了了解将它们分开的发病机制,我们对融合变异、继发性复发性基因改变(突变、拷贝数改变)、基因表达和甲基化谱进行了全面的基因组分析EWSR1:易位相关肿瘤的CREB家族,重点是AFH、CCS、GICCS和HCCC。
尽管我们队列中融合转录本变异的分析在很大程度上与已发表的文献相似,但一些新的有趣的发现出现了。对于AFH,最常见的融合转录本变异是EWSR1: CREB1(ex7-ex7) (58%)7,11,12,13,14,15.然而,我们发现相当大比例(39%)的AFH病例与EWSR1: ATF1(主要ex7-ex5)。少数(3%)的AFH窝藏EWSR1: CREM融合。有趣的是,MMT16,17,18,19,20.,21一些作者仍对其与AFH的黏液样颅内变异有关存在争议22,23,24,25,26,27,含有大致相等比例的EWSR1: CREM而且EWSR1: CREB1融合,与少数港湾EWSR1: ATF1.对于CCS,主要的融合转录本为EWSR1ex8::ATF1ex415,28,29,30.,31,32,33,34.这种模式反映在Meso的一种亚型中,最初由我们的小组描述,发生在没有石棉接触史的年轻患者中,主要是由EWSR1: ATF1ex535,36,37.有趣的是,与之前的数据相比,GICCS显示了相似的比例EWSR1: ATF1(主要是ex8-ex4)和EWSR1: CREB1融合5,34,38.另一方面,最近描述的独特的肿瘤类型,所谓的“恶性上皮样肿瘤,倾向于间皮衬里腔”。39随后由Shibayama等人验证。40,最常见的是两者之间的融合EWSR1或付家7号外显子CREM.相比之下,PPMS几乎完全是由EWSR1: CREB1(主要是ex7-ex7)41,42,43,44,45,46,47,48除了一个罕见的案例EWSR1: ATF149.一些作者提出PPMS和AFH存在于形态和分子谱上43,49.最后,都是HCCC50,51,52,53,54,55和CCOC56,57,58,59,60港只EWSR1: ATF1支持HCCC和CCOC可能是相关肿瘤的观点。然而,从我们对已发表文献的荟萃分析和目前的研究中可以明显看出,在不同的CREB易位肿瘤中,融合转录本变异和外显子使用存在显著的瘤间重叠和瘤内异质性。在这里,肿瘤内异质性是指融合转录体变异,如:EWSR1: ATF1而且EWSR1: CREB1在GICCS中,以及外显子在同一组织类型中的使用,例如,付:CREM外显子5和付:CREMME中的外显子7。
这是第一个报道CCS、AFH和GICCS继发性复发基因组改变的研究。在CCS中,我们发现了复发的存在叔启动子和CDKN2A热点突变是相互排斥的,但结合在一起与较差的总生存率密切相关。叔启动子体细胞突变和扩增经常在多种肿瘤类型中被发现61,62.在软组织肿瘤中,叔启动子突变已在粘液样脂肪肉瘤中被发现63,非典型纤维黄色瘤/多形性皮肤肉瘤64,软骨肉瘤65和孤立性纤维性肿瘤66;据报道,后者的预后较差。我们的研究结果表明叔启动子和CDKN2A突变可以作为CCS患者生存率较差的预后生物标志物。
在AFH中,我们确定CDKN2A / CDKN2B纯合子缺失仅存在于转移病例中。多种肉瘤类型的基因组分析显示继发性复发CDKN2A改变67,68,其作用被认为是预后不良的生物标志物67.与其他CREB家族易位肿瘤相比,AFH是一种具有边缘性恶性潜能的软组织肿瘤,预后相对较好;转移率通常<2%。事实上,我们队列中所有AFH测序的患者在报告时仍然活着。我们发现CDKN2A / CDKN2B在两例经活检证实有转移的AFH病例中,但在未转移的AFH病例中没有,这增加了发生转移的可能性CDKN2A / CDKN2B缺失检测可作为转移潜力的生物标志物。虽然在这个队列中没有复发性异常,但其中一个转移性AFH病例显示同时存在BRAFV600E突变,经免疫组化证实为弥漫性强VE1表达。这是我们队列中唯一的一例BRAF突变检测。
基因表达谱显示AFH和CCS的差异基因表达,通过无监督分析,它们聚集在不同的基因组组中。与AFH相比,CCS中许多参与黑素细胞调节和细胞膜/迁移的基因上调,包括PMP22,MITF,SLC7A5,CDH19,WIPI1,菲英岛,PARVB,PFKP,而上调Sgk1, s100a4, xaf1而且LY96AFH中检测到mRNA的表达,而CCS中未检测到mRNA的表达。CCS细胞系的表达谱分析显示S100A11(编码S100蛋白),MITF(小眼相关转录因子),以及Pmel17(SILV)(免疫组化标记HMB45检测银鼠同源黑素体蛋白)69.此外,在体外CCS诱导的多能干细胞模型中,Komura等报道了几种雪旺氏细胞标记基因的表达,如我NTR,S100b,Mbp,Plp1,Pmp2270.他们提出表达s100的周围神经细胞可能是一种细胞起源EWS / ATF1 -诱导CCS。在最近的一项研究中,使用人类胚胎干细胞(hES)模型,hES细胞由EWSR1: CREB1而且EWSR1: ATF1融合分别概括了AFH的核心基因特征(SGK1而且MXRA5上调)及综合医疗保险(SGK1,MXRA5,SOX10,DUSP4upregulation)71.我们对患者样本的基因表达谱验证了这些临床前研究的一部分发现。
我们的甲基化分析和多维还原聚类分析显示,CREB家族易位相关肿瘤(AFH, CCS, GICCS, Meso)形成相邻但紧密的聚类,与其他软组织肿瘤类型和正常组织不同。当将差异表达基因与相应的甲基化探针/CpG位点进行匹配时,我们发现CCS中低甲基化的上调基因与AFH中低甲基化的上调基因之间存在显著相关性。这些基因包括MITF,CDH19,PARVB,PFKP在CCS。MITF参与黑素发生,并在CCS中作为其核心基因签名的一部分被发现过表达,但在AFH或GICCS中没有5,7.最近,是cre - loxp诱导的Ewsr1: Atf1驱动CCS模型证明了这一点Mitf而且Myc会不会导致肉瘤形成72.这两个CDH19而且PARVB参与细胞粘连并在原发性黑色素瘤中高度表达,与较差的生存率有关8.有趣的是,在我们的研究中发现它们在CCS中表达增加和低甲基化。另一方面,我们发现的上调和低甲基化S100A4而且XAF1在AFH,而不是CCS。S100A4蛋白是S100钙结合蛋白家族的一员,也被称为转移蛋白,并与细胞迁移和癌症转移有关9.XAF1促凋亡、干扰素刺激的肿瘤抑制基因是否抑制肿瘤发生10.而XAF1在大多数癌症中通常是高甲基化和下调的,但在具有适应性替莫唑胺耐药的胶质母细胞瘤中却发现了矛盾的低甲基化73.这些发现是一个概念证明的例子,说明整合基因表达和甲基化分析如何为共享相同驱动基因融合的肿瘤的不同发病机制提供有趣的生物学见解。整合DNA甲基化和基因表达研究已在尤因肉瘤中进行74小儿横纹肌肉瘤75、粘液样、去分化和多形性脂肪肉瘤76,发现了一组具有逆甲基化和基因表达关系的基因。在一项未分化肉瘤(USARC)的综合分子和基因组研究中,DNA甲基化分析未能识别不同的USARC亚群,且与基因表达无关,但显示MSH2而且叔启动子甲基化77.另一方面,DNA甲基化分析也揭示了同一肿瘤类型的表观遗传异质性,如尤文氏肉瘤78.遗憾的是,目前研究中用于甲基化分析的单个肿瘤类型的样本量不足以在同一肿瘤类型内进行差异甲基化分析,这可以在未来的研究中进行探索。
全基因组DNA甲基化分析已在广泛的间充质肿瘤中用于肿瘤分类,并取得了不同程度的成功。其中包括:良性和恶性神经鞘瘤79,骨肉瘤80未分化的小圆形蓝色细胞瘤81,中投公司-重排未分化肉瘤82.最近,海德堡的德国癌症研究中心(DKFZ)开发了一种随机森林机器学习肉瘤分类器,使用大型参考和验证队列对66种软组织和骨肿瘤进行了广泛的分类4.在我们的队列中,除了CCS外,海德堡甲基化分类器无法准确地对肿瘤实体进行分类,这说明了单独使用甲基化分析来区分软组织肿瘤的局限性。这种甲基化分类器对软组织肿瘤的适用性有几个主要缺点EWSR1/付家与CREB家族转录因子融合。首先,包括GICCS和Meso在内的几种肿瘤类型没有包括在用于开发分类器的参考队列中。第二,虽然参考队列包括8例AFH,但验证队列只使用了1例,被错误地归类为粘连性小圆细胞瘤4.在我们的研究中,分类器能够正确地分类三分之一的AFH病例。另一方面,在Koelsche等人的研究和我们的经验中,甲基化分类器在CCS的分类中表现非常好:正确分类了100%的病例。同样有趣的是,当我们将分类器应用于GICCS时,2例被归类为CCS, 1例被归类为AFH,说明了如上所述的甲基化图谱重叠。3例GICCS均位于胃肠道(1例胃,2例小肠),S100弥漫性强阳性,HMB45阴性。综合临床和免疫组化资料基本上排除了CCS和AFH。这些发现强调了甲基化分析在软组织肿瘤分类中的局限性,这可能需要进一步的算法改进以及更大的参考和验证队列83.
除了这些分子机制之外,融合的初始干细胞宿主的性质及其承诺/可塑性的程度也可能在最终的肿瘤类型中发挥重要作用(即,取决于全能性的位置/程度)。这些都是有趣的问题,超出了当前研究的范围。最近的研究使用Cre可诱导的小鼠和人类胚胎干细胞模型已经开始解决这些问题71,72.
每种技术的案例样本量缺乏一致性是我们论文的一个主要缺陷。进一步的研究聚焦于特定的分子分析技术,利用一些更罕见的组织类型的更大样本量进行更深入的基因组表征,这将有利于验证或扩展我们的发现。
综上所述,我们对EWSR1 /付::分子易位相关肿瘤发现融合转录本变异异质性,预后显著的继发性复发性基因改变,差异低甲基化和上调基因。这些发现强调了整合基因组方法在研究具有不同临床病理特征的易位相关肿瘤中的实用性,以及该家族中的一些实体是否可以在相同的形态/分子光谱下统一(例如,CCS和GICCS, AFH和PPMS)。
数据可用性
在这项研究中生成或分析的数据集包括在这篇发表的文章[及其补充信息文件)。原始Affymetrix和Illumina 850k甲基化阵列生成的数据由于缺乏无限期托管能力而无法公开,但可以通过通信作者的合理请求获得。
参考文献
泽希尔,A.等人。1万名患者的前瞻性临床测序揭示了转移性癌症的突变格局。Nat,地中海。23, 703-713(2017)。
Benayed, R.等人。RNA测序在肺腺癌中靶向激酶融合的高产量,DNA测序未检测到有丝分裂驱动改变,肿瘤突变负担低。中国。癌症Res。25, 4712-4722(2019)。
本哈米达,J. K.等。使用高通量全基因组甲基化阵列平台进行可靠的临床MLH1启动子高甲基化评估。J. Mol。22, 368-375(2020)。
Koelsche, C.等。基于DNA甲基化谱的肉瘤分类。Commun Nat。12, 498(2021)。
Antonescu, C. R., Nafa, K., Segal, N. H., Dal Cin, P. & Ladanyi, M. EWS-CREB1:透明细胞肉瘤中反复发生的变异融合,与胃肠道位置和缺乏黑素细胞分化有关。中国。癌症Res。12, 5356-5362(2006)。
黄,s.c .等。恶性间充质瘤中频繁的HRAS突变:胚胎横纹肌肉瘤的重叠遗传异常。点。J.外科。40, 876-885(2016)。
安东尼斯库,C. R.等。EWSR1-CREB1是血管瘤样纤维组织细胞瘤的主要融合基因。基因染色体癌症46, 1051-1060(2007)。
埃里克森,J.等人。原发性黑素瘤的基因表达分析表明,CTHRC1在黑素瘤的进展中起着重要作用。Oncotarget7, 15065-15092(2016)。
李志刚,李志刚,李志刚。转移相关蛋白S100A4在肿瘤进展和转移中的作用。Br。j .癌症92, 1955-1958(2005)。
里斯顿,P.等人。XAF1作为XIAP抗caspase活性拮抗剂的鉴定。细胞生物学。3., 128-133(2001)。
Waters, B. L., Panagopoulos, I. & Allen, E. F.血管瘤样纤维组织细胞瘤的遗传特征确定了涉及12q13和16p11条带的染色体易位诱导FUS和ATF-1基因融合。癌症麝猫。Cytogenet。121, 109-116(2000)。
Raddaoui, E., Donner, L. R. & Panagopoulos, I. FUS和ATF1基因在大的深层血管瘤样纤维组织细胞瘤中的融合。成岩作用。摩尔。病理学研究。11, 157-162(2002)。
Hallor, K. H.等。在血管瘤样纤维组织细胞瘤中融合EWSR1和ATF1基因而不表达MITF-M转录本。基因染色体癌症44, 97-102(2005)。
罗西,S.等。血管瘤样纤维组织细胞瘤中EWSR1-CREB1和EWSR1-ATF1融合基因的研究。中国。癌症Res。13, 7322-7328(2007)。
吉田等人。扩大EWSR1-CREM融合间充质肿瘤的表型谱。点。J.外科。43, 1622-1630(2019)。
高永昌等。EWSR1与CREB家族转录因子融合定义了一种倾向于颅内定位的新型黏液样间充质肿瘤。点。J.外科。41, 482-490(2017)。
Sciot, R.等人。颅内位置的原发性黏液样间充质瘤:一例EWSR1-ATF1融合报告。组织病理学72, 880-883(2018)。
小松,等。颅内黏液样间充质瘤伴EWSR1-CREB1基因融合1例报告并文献复习。脑瘤病理。37, 76-80(2020)。
巴莱斯特,L. Y.等。颅内黏液样间充质瘤伴EWSR1-ATF1融合。j . Neuropathol。经验,神经。79, 347-351(2020)。
Valente Aguiar, P., Pinheiro, J., Lima, J., Vaz, R. & Linhares, P.成年女性伴有EWSR1-CREB1基因融合的黏液样间充质脑室内肿瘤。菲尔绍拱门。478, 1019-1024(2021)。
王春萍,王春萍,刘建昌。成人颅内黏液样间充质瘤的研究。世界Neurosurg。143, 91-96(2020)。
贝尔,t.a.等。含有EWSR1-CREB家族基因融合的颅内黏液样间充质瘤:血管瘤样纤维组织细胞瘤的黏液样变异还是新形态?大脑病理学研究。28, 183-191(2018)。
斯隆,e.a.等人。颅内间充质瘤伴FET-CREB融合——颅内黏液样间充质瘤和血管瘤样纤维组织细胞瘤样肿瘤频谱的统一诊断。大脑病理学研究。31, 12918(2021)。
谭乃杰,等。具有“经典”组织学和EWSR1-CREM融合的颅内黏液样血管瘤样纤维组织细胞瘤为颅内黏液样间充质瘤的和解提供了见解。神经病理学41, 306-314(2021)。
Dunham, C., Hussong, J., Seiff, M., Pfeifer, J. & Perry, a .原发性脑血管瘤样纤维组织细胞瘤:一例t (12;22)(问题;q12)导致EWS和ATF-1基因的1型融合。点。J.外科。32, 478-484(2008)。
加莱顿等人。颅内黏液性血管瘤样纤维组织细胞瘤样肿瘤ESWR1-CREM融合1例报告及文献复习j . Neuropathol。经验,神经。77, 537-541(2018)。
康斯坦丁尼迪斯,A.等。颅内血管瘤样纤维组织细胞瘤伴EWSR1-CREB家族融合:2例小儿病例报告世界Neurosurg126, 113-119(2019)。
安东尼斯库,C. R.等。透明细胞肉瘤的分子诊断:12例透明细胞肉瘤EWS-ATF1和MITF-M转录本检测及组织病理和超微结构分析J. Mol。4, 44-52(2002)。
Panagopoulos等人。EWS/ATF1融合基因在肌腱和腱膜透明细胞肉瘤中的分子遗传学特征。Int。j .癌症99, 560-567(2002)。
Coindre, J. M.等。石蜡包埋组织实时逆转录-聚合酶链反应分析诊断透明细胞肉瘤:来自法国肉瘤组44例患者的临床病理和分子分析。癌症107, 1055-1064(2006)。
久冈,等人。软组织透明细胞肉瘤:33例临床病理、免疫组织化学和分子分析。点。J.外科。32, 452-460(2008)。
王文亮等。透明细胞肉瘤(软组织黑色素瘤)中EWSR1/ATF1和EWSR1/CREB1嵌合转录本的检测和鉴定国防部,病理学研究。22, 1201-1209(2009)。
Jakubauskas, A.等。在四种嵌合表达透明细胞肉瘤中发现两种新的EWSR1/ATF1转录本及其定量评价Exp、Mol、Pathol。90, 194-200(2011)。
Segawa, K.等。荧光原位杂交技术检测16例软组织透明细胞肉瘤和6例胃肠道透明细胞肉瘤样肿瘤特异性基因重排成岩作用。病理学研究。13, 73(2018)。
Desmeules, P.等。青年人恶性间皮瘤的一个子集与复发的EWSR1/FUS-ATF1融合有关。点。J.外科。41, 980-988(2017)。
Ajelero, O., Zhang P. L., Collingwood, R. & Fortuna, D.一例伴有EWSR-ATF1融合转录和异常免疫表型的恶性腹膜间皮瘤。嗡嗡声。病理学研究。代表。25, 200542(2021)。
Ren, H.等人。2例青少年男性恶性间皮瘤伴EWSR1-ATF1融合。Pediatr。Dev,病理学研究。12, 10935266211021222(2021)。
斯托克曼,D. L.等。胃肠道恶性神经外胚层肿瘤:16例胃肠道透明细胞肉瘤样肿瘤的临床病理、免疫组织化学、超微结构和分子分析点。J.外科。36, 857-868(2012)。
Argani, P.等人。EWSR1/FUS-CREB融合定义了一种独特的恶性上皮样肿瘤,倾向于间皮衬里腔。国防部,病理学研究。33, 2233-2243(2020)。
细胞角蛋白阳性腹部恶性肿瘤合并EWSR1/FUS-CREB融合:8例临床病理研究点。J.外科。46, 134-146(2022)。
Thway, K.等。原发性肺粘液样肉瘤合并EWSR1-CREB1融合:一种新的肿瘤实体点。J.外科。35, 1722-1732(2011)。
Matsukuma, S.等。原发性肺粘液样肉瘤合并EWSR1-CREB1融合,类似于骨外粘液样软骨肉瘤:一例报告并文献复习病理学研究。Int。62, 817-822(2012)。
史密斯,s.c.等。在原发性肺粘液样肉瘤和肺血管瘤样纤维组织细胞瘤的交叉点:来自三例新病例的观察。组织病理学65, 144-146(2014)。
Jeon, Y. K., Moon, K. C., Park, S. H.和Chung, D. H.原发性肺粘液样肉瘤伴EWSR1-CREB1易位可能源于原始支气管周围间充质细胞发生(肌)成纤维细胞分化。菲尔绍拱465, 453-461(2014)。
Yanagida, R., Balzer, B. L., Mckenna, R. J. & Fuller, C. B.原发性肺粘液样肉瘤,一种可能的转移性骨外粘液样软骨肉瘤。病理49, 792-794(2017)。
Prieto-Granada, C. N., Ganim, R. B., Zhang, L., Antonescu, C. & Mueller, J.原发性肺粘液样肉瘤:一个新描述的实体-一例病例报告及文献复习。Int。J.外科。25, 518-525(2017)。
陈震,杨艳,陈锐,吴昌松,石辉。原发性肺粘液样肉瘤合并EWSR1-CREB1融合:一例报告及文献复习。成岩作用。病理学研究。15, 15(2020)。
Koelsche, C.等。原发性肺粘液样肉瘤,在EWSR1的外显子7和CREB1的外显子5之间有不寻常的基因融合。菲尔绍拱476, 787-791(2020)。
桂红,苏斯曼,R. T., Jian, B., Brooks, J. S., Zhang, P. J. L.原发性肺黏液样肉瘤和黏液样血管瘤样纤维组织细胞瘤:具有共同和独特特征的统一连续体。点。J.外科。44, 1535-1540 (2020)
安东尼斯库,C. R.等。EWSR1-ATF1融合在唾液腺透明细胞癌中是一个新的和一致的发现。基因染色体癌症50, 559-570(2011)。
中野,T.等。含有EWSR1-ATF1融合基因的透明细胞癌:3例分子分析报告。菲尔绍拱466, 37-43(2015)。
查普曼,E.等。透明透明细胞癌的分子谱显示了一个肿瘤子集,具有新的EWSR1-CREM融合:3例报告。点。J.外科。42, 1182-1189(2018)。
广濑,K.等。腭小唾液腺透明细胞癌携带新的EWSR1-ATF1融合基因:1例报告并文献复习。头颈悲怆。15, 676-681(2021)。
野岛,等。口腔透明细胞癌伴三种新型EWSR1-ATF1易位:一例报告。头颈悲怆。(2021)
Heft Neal, m.e.等。整合测序发现atf1 motif富集的分子特征,可区分透明细胞癌与粘液表皮样癌。口腔肿瘤防治杂志。117, 105270(2021)。
比洛多,E. A.等。透明细胞牙源性癌显示EWSR1重排:一项新发现和与唾液透明细胞癌的生物学联系。点。J.外科。37, 1001-1005(2013)。
扬科斯基,A. E.等人。透明细胞牙源性癌中的EWSR1和ATF1重排:一例报告。口腔外科,口腔医学,口腔病理。口服广播118, 115-118(2014)。
沃格尔,R.等人。透明细胞牙源性癌:发生EWSR1-CREB1作为替代融合基因的EWSR1-ATF1。头颈悲怆。13, 225-230(2019)。
Santana, T., Andrade, F. L., Sousa Melo, m.c ., Rocha, G. B. L. & Trierveiler, M.含有EWSR1-ATF1融合基因的透明细胞牙源性癌:一例罕见病例报告。头颈悲怆。14, 847-851(2020)。
Breik O.等人。透明细胞牙源性癌:首次报道新型EWSR1-CREM融合基因在长期误诊情况下。头颈悲怆。(2021)。
贝尔,r.j.等。了解TERT启动子突变:通往永生的共同途径。Mol. Cancer Res。14, 315-323(2016)。
古普塔等人。临床基因组测序显示30773例肿瘤中TERT启动子突变和扩增的泛癌症研究J. Mol。23, 253-263(2021)。
Koelsche, C.等。TERT启动子热点突变在粘液样脂肪肉瘤中反复出现,但在其他软组织肉瘤中很少见。J.临床经验。癌症Res。33, 33(2014)。
格里旺克,K. G.等。TERT启动子突变在非典型纤维黄色瘤和多形性皮肤肉瘤中很常见。国防部,病理学研究。27, 502-508(2014)。
张勇,等。TERT启动子突变是软骨肉瘤疾病进展的客观临床标志。国防部,病理学研究。(2021)。
巴赫拉米,A.等人。TERT启动子突变与单发纤维性肿瘤的预后。国防部,病理学研究。29, 1511-1522(2016)。
Bui, n.q.等。软组织肉瘤患者的临床基因组分析显示CDKN2A缺失是预后不良的生物标志物。中国。肉瘤Res。9, 12(2019)。
Groisberg, R.等人。临床基因组分析,以确定不同肉瘤患者研究性治疗的可行改变。Oncotarget8, 39254-39267(2017)。
Schaefer, K. L.等。t (12;22)软组织细胞系透明细胞肉瘤阳性显示包括ERBB3在内的潜在新标记基因的特征性上调。癌症Res。64, 3395-3405(2004)。
高村,S.等。透明细胞肉瘤中EWS/ATF1融合基因的细胞类型依赖增强子结合Commun Nat。10, 3999(2019)。
Vanoli F.,等。生成人类胚胎干细胞模型,利用EWSR1-CREB融合混杂作为人类肿瘤转化的常见途径。致癌基因https://doi.org/10.1038/s41388-021-01843-0(2021)。
潘扎,E.等人。透明细胞肉瘤功能基因组图谱。j .中国。投资。131, 146301(2021)。
吴,Q.等。XAF1的反常表观遗传调控介导了mgmt -甲基化胶质母细胞瘤适应性耐药进化的可塑性。科学。代表。9, 14072(2019)。
Patel, N.等人。尤因肉瘤原发肿瘤的DNA甲基化和基因表达谱揭示了表观遗传失活的潜在靶点基因。肉瘤2012, 498472(2012)。
Mahoney, S. E.等人的甲基化研究表明,在小儿胚胎型和肺泡型横纹肌肉瘤中存在不同的DNA甲基化模式。表观遗传学7, 400-408(2012)。
雷纳等人。高度软组织肉瘤整合DNA甲基化和基因表达分析。基因组医学杂志。14, 137(2013)。
斯蒂尔,C. D.等人。未分化肉瘤通过不同的进化途径发展。癌症细胞35, 441-4568(2019)。
谢菲尔德,北卡罗来纳州等。DNA甲基化异质性定义了尤因肉瘤的病谱。Nat,地中海。23, 386-395(2017)。
Röhrich, M.等。基于甲基化的周围神经鞘肿瘤良恶性分类。Acta Neuropathol。131, 877-887(2016)。
吴,s.p.等。DNA甲基化分类器在骨肉瘤分子诊断中的应用。JCO大纲。肿瘤防治杂志。17, 00031(2017)。
Koelsche, C.等。DNA甲基化分析区分Ewing-like肉瘤合并EWSR1-NFATc2融合与Ewing肉瘤。J. Cancer Res. clins。肿瘤防治杂志。145, 1273-1281(2019)。
Miele, E.等人。DNA甲基化分析诊断伴Capicua转录受体改变的未分化肉瘤。Int。理学。21, 1818(2020)。
Lyskjaer, I.等。骨和软组织肿瘤的DNA甲基化分析:“DKFZ肉瘤分类器”的验证研究。j .分册。中国。Res。7, 350-360(2021)。
Chakravarty, D.等人。OncoKB:精准肿瘤学知识库。JCO大纲。肿瘤防治杂志。17, 00011(2017)。
高,J.等。使用cBioPortal对复杂癌症基因组学和临床资料进行综合分析。科学。信号6, 1(2013)。
确认
我们要感谢使用MSK-IMPACT数据的中心核心资助(P30 CA008748)、病学系分子诊断服务以及Marie-Josee和Henry R. Kravis分子肿瘤学中心。
资金
这项工作得到了P50 CA 140146-01 (SS, WT, CRA), P50 CA217694 (SS, WT, CRA), P30 CA008748 (SS, WT, CRA),生存周期(CRA, FV),克里斯汀·安·卡尔基金会(CRA)的支持。所有其他作者均报告没有与本研究相关的资金来源。
作者信息
作者及隶属关系
贡献
J.K.D.负责研究设计、数据采集、数据分析和解释、论文写作和修改。f.v., l.h., y.s., L.Z, s.s., w.t., r.b., t.b., j.b., B.D.对数据进行了采集、分析和/或解释,并对论文进行了审阅。c.r.a进行研究设计和构思,分析和解释数据,撰写,审查和修改论文。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
伦理批准并同意参与
这项研究得到了纪念斯隆凯特琳癌症研究所机构审查委员会的批准。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
权利和权限
关于本文
引用本文
德玛万,j.k., Vanoli, F., Herviou, L.。et al。综合基因组分析EWSR1 /付::分子易位相关肿瘤揭示了预后显著的复发性基因改变和甲基化-转录相关性。国防部分册35, 1055-1065(2022)。https://doi.org/10.1038/s41379-022-01023-9
收到了:
修改后的:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41379-022-01023-9