摘要
转录因子FOS和FOSB的重排最近被确定为骨样骨瘤和成骨细胞瘤的遗传驱动事件。尽管在特异性和敏感性方面存在局限性,但FOS和FOSB的核过表达已成为一种可靠的替代标记。事实上,骨肉瘤很少表现出FOS的核表达,一小部分成骨细胞瘤的发生似乎与FOS/FOSB重排无关。酸脱钙和组织保存是影响免疫组化检测的其他因素,使个别病例的诊断决策具有挑战性。特别具有侵袭性的成骨细胞瘤,也被称为上皮样成骨细胞瘤,和成骨细胞瘤样骨肉瘤可能很难区分,这强调了需要额外的标志物来支持诊断。甲基化和拷贝数分析,一种用于脑肿瘤分类的技术,可能会填补这一空白。在这里,我们开始通过免疫组织化学、荧光原位杂交以及拷贝数和甲基化分析全面描述77例成骨细胞瘤,并将我们的发现与组织学模拟进行比较。我们的研究结果表明,成骨细胞瘤具有一致的平坦拷贝数特征,这可以增加达到正确诊断的确定性。然而,到目前为止,由成骨细胞瘤形成的甲基化簇缺乏特异性,在个别病例中可能具有误导性。
简介
骨样骨瘤和成骨细胞瘤是形态相似的骨形成肿瘤。骨样骨瘤被认为是良性的,通常直径不超过2cm,成骨细胞瘤表现为局部侵袭性,大小可达> ~ 10cm。骨样骨瘤较为常见,占所有原发性骨肿瘤的10-12%,通常发生在长管和小管骨,优先累及皮质。成骨细胞瘤是罕见的(<1%的原发性骨肿瘤),主要发生在脊柱1.最常见的骨形成肿瘤是常规骨肉瘤,这是一种骨内高级别肉瘤,需要强烈的多模式治疗,在转移性和/或复发性疾病患者中仍与不良预后相关。虽然形态学通常有显著差异,但骨肉瘤和成骨细胞瘤之间的鉴别诊断可能具有挑战性,特别是在核心针活检中。罕见的成骨细胞瘤样骨肉瘤变体在组织学上与成骨细胞瘤相似。1).由于临床结果和治疗的关键差异,可靠的骨形成肿瘤分类对于提供足够的临床护理至关重要。
最近有研究表明,转录因子的重排”丛书在较小的程度上也是它的对话录FOSB,代表成骨细胞瘤和骨样骨瘤中高度复发的驱动突变2.这些易位导致基因融合,其中伴侣基因高度可变,但有助于FOS/FOSB蛋白的过表达2,3..因此,FOS/FOSB免疫染色与荧光原位杂交(FISH)一起成为常规诊断中有价值的替代标记和工具。基因融合已在绝大多数成骨细胞瘤中检测到,但约5-11%2,4缺乏这些畸变的证据。然而,免疫组化对潜在易位并不是特异性的,可以受到组织保存的影响,一小部分成骨细胞瘤具有纯合子的特征NF2缺失与缺失安全系数/ FOSB重组5,6.
识别FOS/FOSB过表达有助于常规诊断,但正如所述,并非所有病例都存在FOS/FOSB过表达,也可在一小部分骨肉瘤中识别。因此,一种替代分子标记的可用性将是非常有价值的。最近,DNA甲基化分析已被证明是脑肿瘤分类的另一种方法7.它与使用组织学和分子遗传学的传统肿瘤分型有良好的相关性,并已被证明可重复识别以前难以区分但对治疗和预后有不同反应的新肿瘤亚型。此外,使用Infinium Human Methylation450K BeadChip或Epic Array (850k)平台的甲基化分析提供了全基因组拷贝数变化,可以指向特定的基因扩增/重排、由变色引起的复杂畸变模式以及一般的非整倍体程度。
在方法上,甲基化分析是基于在整个基因组的CpG位点上检测5-甲基胞嘧啶,其中Infinium甲基化阵列检查每个样本450/850k。然后将单个肿瘤的特征与使用明确诊断病例(基本事实)建立的参考甲基化分类进行比较。这种比较是通过一种叫做随机森林的监督学习算法实现的8.这种分类器在随机选择的cpg系列上生成决策树。一旦森林被训练来识别参考样本,每棵树都为被测试的样本提供一个单独的预测,并选择多数投票。一旦建立了所有肿瘤亚型的甲基化分类,理论上任何给定的样本都应该根据其甲基组谱进行分类。这种方法已被证明为脑肿瘤提供了高度特异性和可重复性的分类7基于所谓的“海德堡肉瘤分类器”,初步研究对软组织和骨肿瘤也有很好的结果,8尽管一项验证研究显示,不同肿瘤类型的患者表现不同9.因此,甲基化分类器可能是一种有价值的诊断工具,只要其结果在适当的临床、形态学和免疫组织化学背景下得到解释。
在本研究中,我们对77例成骨细胞瘤进行了FOS/FOSB免疫组化、FOS荧光原位杂交、甲基化和拷贝数分析,并将我们的结果与纤维结构不良病例以及大量骨肉瘤(包括常规、骨旁、中央低级别、骨膜和成骨细胞瘤样亚型)进行了比较。
材料与方法
患者样本
在巴塞尔大学医院(CH)、卡罗林斯卡大学医院(SE)、Skåne大学医院(SE)和莱顿大学医学中心(NL)病理学部门的病理研究所的档案中,寻找成骨细胞瘤和骨样骨瘤,其中有足够数量保存完好的肿瘤组织,用于分子研究。共切除77例成骨细胞瘤和2例骨样骨瘤。所有可用组织学的肿瘤均由骨肿瘤病理学家(DB)进行复查,肿瘤含量低于40%的样本被排除在研究之外。临床病理资料见补充表S1.
荧光原位杂交(FISH)检测FOS
在77例成骨细胞瘤中,24例(来自莱顿大学医学中心)已经由FISH进行了调查10.另外28个样本缺乏足够数量的保存完好的组织切片。因此,如前所述,对25例成骨细胞瘤进行FISH分析10.BAC探针位于远端和近端两侧”丛书基因包括RP11-173A8与RP11-316E14和RP11-361H10与RP11-368K8。
FOS的免疫组化(IHC)
如前所述,对45例成骨细胞瘤和2例骨样骨瘤进行了免疫组化11.24例(来自莱顿大学医学中心)已经进行了免疫染色4另外8个肿瘤(来自隆德大学医院)缺乏足够数量的保存完好的组织切片。所有免疫反应均使用针对FOS n端区域的兔多克隆抗体(克隆,F7799, Sigma, St. Louis, Missouri, USA)。
DNA甲基化数据集
本研究纳入的77例成骨细胞瘤和2例骨样骨瘤均基于Infinium Human Epic Array (850k)平台(Illumina)进行DNA甲基化分析。24份福尔马林固定石蜡包埋(FFPE)和8份新鲜冷冻样品产生了可解释的结果,而41份样品未能通过质量控制,主要原因是组织保存有限。其余6例已经用850k阵列进行了评估,并用于生成“海德堡肉瘤分类器”的参考级成骨细胞瘤。此外,我们还研究了2例骨旁骨肉瘤、2例纤维结构不良增生病例、4例成骨细胞样骨肉瘤和10例中枢性低级别骨肉瘤的FFPE样本(补充表)S1).所有阵列都在一个完全自动化的平台(生命和大脑,波恩)进行杂交和外部扫描。然后将原始甲基化数据与已发表的几个外部数据集的数据一起处理8,9,12,以达到每种肿瘤类型至少7例(经验证明,这一阈值足以避免聚类伪影并实现充分的预测8).总的来说,DNA甲基化阵列数据集包括99例高级别骨肉瘤,48例成骨细胞瘤,22例骨旁骨肉瘤,20例纤维结构不良,15例中枢性低级别骨肉瘤,7例骨膜骨肉瘤和7例成骨细胞样骨肉瘤。所有甲基化数据已存入欧洲核苷酸档案,该研究的登录号为EGAD00010002279。
甲基化阵列处理
来自Methylation 450 K BeadChips或Methylation Epic (=850k) BeadChips的原始强度数据文件(IDATs)使用R-package minfi (https://bioconductor.org/packages/release/bioc/html/minfi.html).Epic阵列被转换为一个虚拟的450k阵列,用于联合规范化和处理来自两个平台的数据。与已知snp、非cpg和性染色体相关的探针未被考虑到评估中。此外,样品具有检测的均值p-value大于0.02被丢弃。在可用的不同归一化函数中,在生成降维可视化之前使用' preprocessIllimuna '函数,而在派生副本数概要之前首选' preprocessQuantile '函数。
副本编号配置文件
拷贝数变化由Infinium Human Methylation 450 K BeadChip或Epic Array平台使用R-package conumee推断(https://www.bioconductor.orgpackages/release/bioc/html/conumee.html),然后对上述数据进行预处理。在没有配对正常样本的情况下,一组20个对照样本作为参考(有反应性变化的组织n= 10和血n= 10来自Koelsche等人的研究,20208).拷贝数变化推断的设置如下:每个bin的最小探针数等于25;最小仓大小等于100,000 bp。任何背景噪声高于0.9的样本均被排除(6例成骨细胞瘤,2例成骨细胞瘤样骨肉瘤,1例纤维性发育不良病例和1例常规骨肉瘤,尽管有适当的甲基化组数据)。在图中,所有样品均用深灰色表示。3..复制数事件根据它们的背景噪声被调用。任何拷贝数变化低于个体背景噪声的三分之一被认为是不显著的,因此被过滤掉。最后,所有副本编号概要文件都已单独审查。然后计算出代表参与任何类型拷贝数变异的基因组百分比的分数(每个CNV的长度之和除以22个常染色体的长度之和,在排除着丝粒和端粒区域后)。
无监督聚类
使用R-package ChAMP对与不同阵列类型(450k/850k)以及不同组织条件(FFPE和新鲜冷冻)相关的批处理效果进行校正,以校正β值。然后,探针组被限制在15 ' 500个参考数据集中甲基化差异最大的前25 ' 000个(基于标准偏差),这些参考数据主要来自癌症基因组图谱(TCGA)和基因表达综合(GEO),如前所述13.本研究中使用的CpG id的完整列表已保存在上面提到的欧洲核苷酸档案中。均匀流形近似和投影(UMAP)是对主成分分析(20个PCs)的结果进行的,通过β甲基化矩阵的奇异值分解计算。用于生成图表的R-package可在以下网址(https://github.com/jlmelville/uwot).用于生成非线性回归模型的设置为:PCA = 20;邻居= 15。一旦模型建立,每个样本随后根据基因组重组的百分比(如上所述)根据蓝红梯度着色。拷贝数分析失败的样本被标记为“N/A”并涂成黑色。
结果
我们的研究包括77例成骨细胞瘤患者的样本。男性51例,女性26例(比例1.96:1),平均年龄22.2岁(范围2 ~ 57岁)。我们还纳入了2例骨样骨瘤(均为男性,13岁和20岁)。进一步的临床病理信息见补充表S1.
免疫组化和FISH
”丛书FOS蛋白免疫染色检测基因重排(n= 45个成骨细胞瘤,n= 2例骨样骨瘤)和FISH”丛书基因(n= 25)。85%的患者中FOS表达一致(n成骨细胞瘤/骨样骨瘤= 40),而15%为免疫组化阴性(n= 6)或不可评估(n= 1)。FISH分析显示6/6可评估病例杂交信号重排,所有病例均显示免疫染色(19例失败或信息不全)。因此,通过结合这两种方法,85%的成骨细胞瘤分析显示了证据”丛书重排。
Methylome分析
DNA甲基化分析在32个肿瘤中产生了可评估的结果,包括30个成骨细胞瘤和2个骨样骨瘤。28例为”丛书由于缺乏足够的保存完好的材料,其他4个样品没有进行原位分析(见补充表)S1).然后将原始甲基化数据提交给“DKFZ肉瘤分类器”进行评估。只有34%的病例诊断与组织学诊断一致(n= 11,包括两个骨样骨瘤),使用分类器的默认设置(预测评分> 0.9)。另有11例预测正确,但得分低于阈值。其余个案中(n= 10),分类器产生了其他预测得分较低(不显著)或根本没有预测的诊断。
拷贝数分析
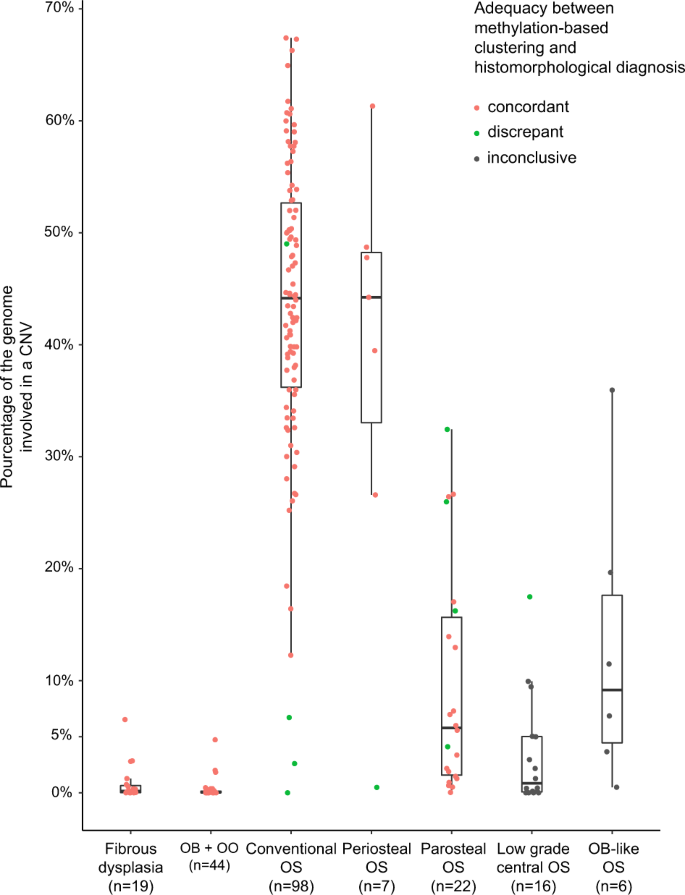
随后对DNA甲基化数据进行处理,以获得每个样本的拷贝数配置文件。为了将拷贝数变异(CNV)的程度总结为一个单一的客观评分,我们计算了每种情况下22个常染色体中重组碱基对的百分比。除了描述的成骨细胞瘤,这些分析还补充了最近发表的成骨细胞瘤以及其他骨形成肿瘤的数据(9,12,13)。数据集如图所示。2其中成骨细胞瘤42例,骨样骨瘤2例,常规骨肉瘤98例,中枢性低级别骨肉瘤15例,骨旁骨肉瘤22例,骨膜骨肉瘤7例,成骨细胞样骨肉瘤6例,纤维结构不良20例。平均0.28%(范围:0.00-4.71%;n= 42)的成骨细胞瘤基因组存在拷贝数变异,而重组基因组的比例达到43.42%(范围:0.00-67.41%;n= 98)。除1例外,所有中重度骨膜骨肉瘤的重组均与高级别骨肉瘤一样严重(38.37%,范围:0.47-61.31%;n= 7)。低级别中央骨肉瘤和骨旁骨肉瘤,两种亚型已知有扩增MDM2基因占25-30%,>占80%14分别表现出有限的CNV量:3.62%(范围:0.03 ~ 17.50%;n低级别中央骨肉瘤= 15),9.72% (0.03-32.45%;n骨旁骨肉瘤= 22)。纤维结构不良病例的拷贝数变化影响了约0.84%的常染色体(范围:0.00-6.54%;n= 20)。最后,成骨细胞瘤样骨肉瘤系列平均评分为13.0%(范围:0.49-36.0%;n= 6)他们的基因组与拷贝数变化有关。然而,肿瘤间的高变异性表明很难明确定义这种罕见的骨肉瘤亚型。
UMAP-based分类
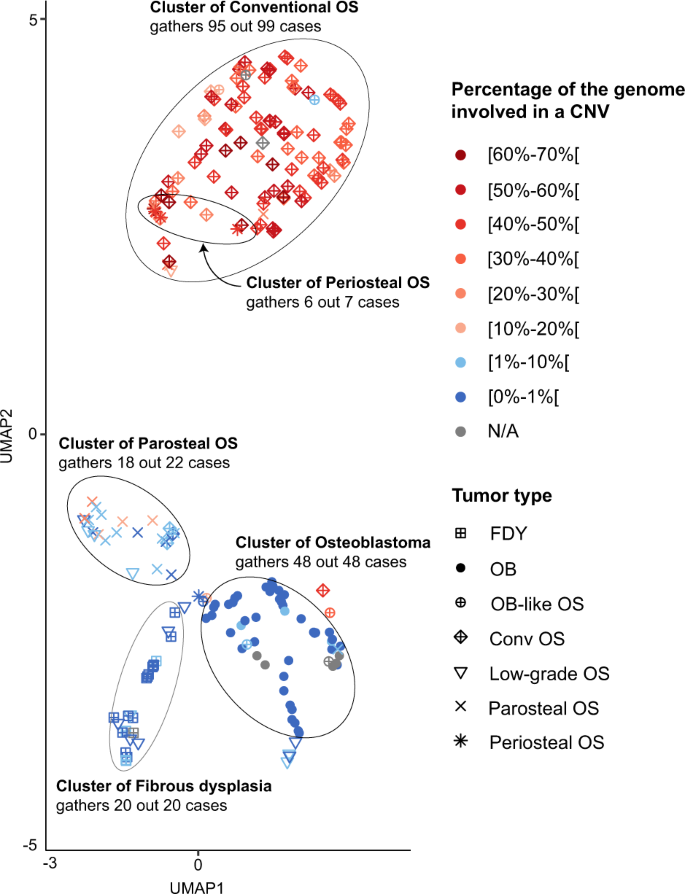
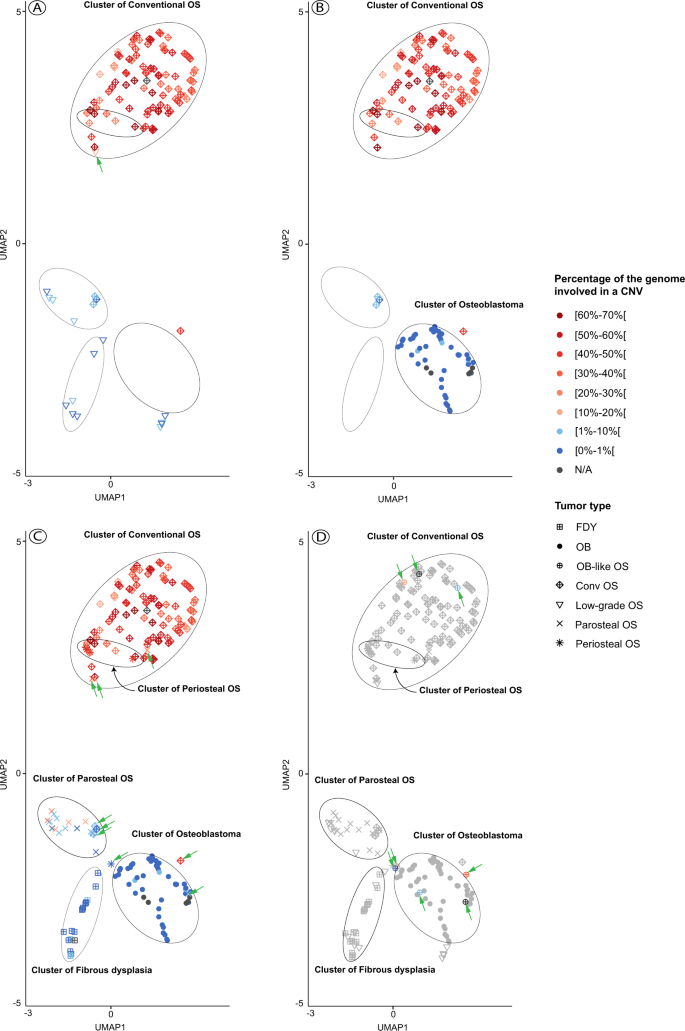
基于甲基化轮廓的骨肿瘤分类也使用统一流形近似和投影(UMAP)作为一种无监督聚类方法进行了研究15.我们研究中所有的肿瘤都被放在一张图表上(图2)。3.),其位置取决于周围甲基组体的相似性。在生成图时未考虑组织形态学诊断。然后根据拷贝数变化所涉及的碱基对的百分比,根据蓝-红梯度对每个病例进行着色。乍一看,我们确定了五个簇,其中绝大多数被分析的肿瘤聚集在一起。良性/低级别肿瘤和中/高级别肿瘤易于区分(图2)。3.).然而,低级别中心型和成骨细胞瘤样骨原性肉瘤分布不均匀,表明它们的甲基化谱只有有限的相似性。作为第二个观察,甲基化和拷贝数的分布似乎有很好的相关性。因此,它们各自聚类之外的大多数样本也是具有非典型性拷贝数分布的样本(图2)。3.).事实上,唯一出现基因组拷贝数改变百分比>10%的中央低级别骨肉瘤也是中/高级别骨肉瘤中唯一定位的标本(图2)。4).同样的观察结果也适用于骨膜骨肉瘤的唯一异常值,但具有反向相关。对于常规骨肉瘤,有4个肿瘤位于肿瘤簇外,其中3个具有特别平坦的拷贝数分布(图2)。4得了).有趣的是,位于OB簇外围的剩余第四个病变也被预测为成骨细胞瘤,“DKFZ肉瘤分类器”给出的置信度评分最高。然而,其拷贝数谱显示大量的CNV(49%)几乎与良性肿瘤不相容安全系数/ FOSBRNA测序检测基因融合。尽管具有典型的常规骨肉瘤组织学特征,但在分子水平上,该肿瘤似乎代表了ob样骨肉瘤。同样,3例CNV评分最高的骨旁骨肉瘤位于常规骨肉瘤簇中(图2)。2和无花果。4摄氏度).“DKFZ肉瘤分类器”尚未包括该肿瘤类型的已建立的甲基化分类,这解释了为什么13/15的肿瘤没有达到预测评分>0.9。2例预测为常规骨肉瘤(置信度评分最高),均属于所述的3个异常值。
总的来说,尽管几种肿瘤类型倾向于形成集群,但单个肿瘤类型有相当大的重叠。事实上,骨膜骨肉瘤和常规骨肉瘤不能仅根据其甲基化特征来区分,对于低级别的中心骨肉瘤和骨旁骨肉瘤也是如此(图2)。3.).此外,这些对的拷贝数谱在进一步的分层中似乎没有用处,因为低级别的中央和骨旁骨肉瘤都可以携带的扩增MDM2而骨膜肉瘤和常规骨肉瘤通常表现为高度重组1.存在或不存在MDM2在低级别骨肉瘤组中的扩增并不足以单独解释这类肿瘤的碎片化(补充图。S1).然而,我们发现大部分MDM2-扩增的低级别中央骨原性肉瘤靠近骨旁骨原性肉瘤,而在纤维性发育不良病例(可能是低级别骨原性肉瘤的组织学模拟)旁边没有,只有一个位于成骨细胞瘤簇的外围。
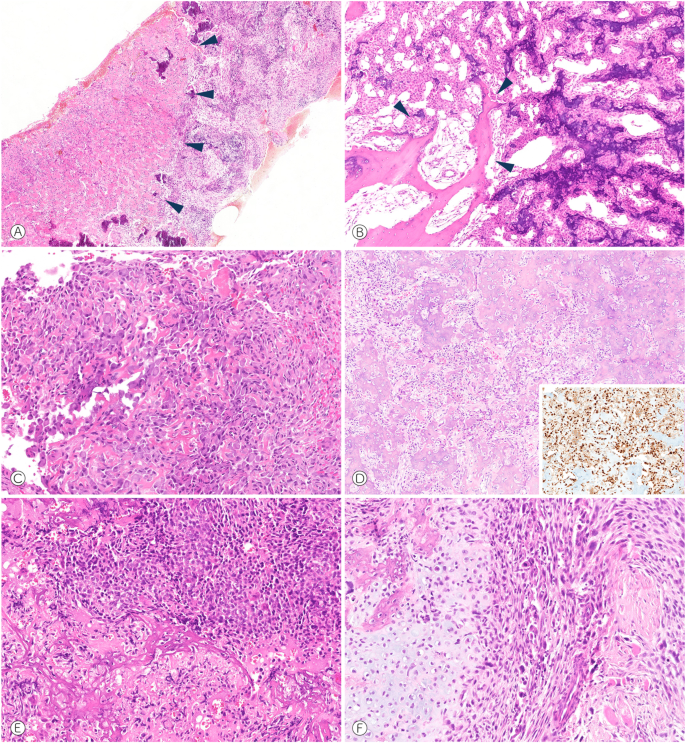
一个最初诊断为成骨细胞瘤的显著病例显示与骨巨细胞瘤(GCTB)的甲基化类高度相似。向“DKFZ肉瘤分类器”提交甲基化数据返回未分类(置信度评分低于阈值),尽管GCTB类别被认为是最接近的匹配。使用突变特异性H3F3A (G34W)抗体的免疫组化确实证实了GCTB的诊断,从而确定了基于甲基组的分类。组织学上肿瘤仅显示少量巨细胞和大量反应性新骨形成,这导致了形态学上的误解。
通过将可能性范围缩小到两种或三种单独甲基化分类的比较,可以做出重要的区分:成骨细胞瘤和传统的高级别骨肉瘤形成明显的独立簇,其中只有少数误定位病例(图2)。4 b).在除一个肿瘤外的所有肿瘤中,对相应拷贝数谱的独立评估都支持基于甲基化的分类,并强调了错误定位样本的罕见性。当甲基化和拷贝数分析同时使用时,96%的高级别骨肉瘤(95/99)和所有的成骨细胞瘤(48/48)可以被区分。然而,当仅使用拷贝数谱并应用基因组中受拷贝数改变影响的10%的任意截止分数时,在不考虑额外的甲基组数据的情况下,几乎可以识别出相同数量的肿瘤(96/99)。其余三例高级别骨肉瘤拷贝数异常平坦,通过甲基化聚类评估,均与骨旁骨肉瘤共定位。4 b)尽管组织学上有明确的高级特征。
值得注意的是,5例FOS/ fosb阴性成骨细胞瘤来自于Lyskjaer的研究9也在成骨细胞瘤簇内正确定位。其次,低级别中央骨肉瘤和高级别骨肉瘤也可以区分(图。4).虽然低级别骨肉瘤在图上分布不一致,但这些肿瘤仍然很容易与常规骨肉瘤区分开来。不幸的是,即使减少了评估肿瘤类型的数量,所描述的方法也不能在4/5的可评估病例中区分成骨细胞瘤样骨肉瘤与成骨细胞瘤(图5)。4 d5/6例未表达FOS,其余肿瘤仅局灶阳性。高级别肿瘤中拷贝数分布重排较多,低级别成骨细胞瘤样骨肉瘤中拷贝数变化较少(分别为6.85%和11.5%)。
讨论
基于甲基化的非脑肿瘤分类的作用是一个持续争论的问题7,16.在骨和软组织肿瘤中最有前途和先进的应用之一是“DKFZ肉瘤分类器”。尽管在高达83%的病例中有足够的预测,但已经发表了批评报告,警告这种方法的结果可能具有误导性,特别是如果非专业病理学家使用这种方法9.通过这项研究,我们建议DNA甲基化分析数据的替代和额外使用。
Illumina Epic Array可以很容易地获得全基因组拷贝数谱,并用于挑战基于甲基化谱的预测。在目前的研究中,我们使用分数来评估个体基因组中的拷贝数变异量。显然,并不是所有类型的重排都可以通过这种方法识别,例如,保持拷贝数配置文件不变的平衡易位。然而,这种简单的统计方法被证明是有价值的,以补充甲基化为基础的肿瘤分类。拷贝数谱分析在鉴别高、中恶性、低恶性/局部侵袭性/良性骨形成肿瘤方面非常有效。此外,人们可以想象实现特定cnv的自动检测(例如MDM2低级别骨肉瘤的扩增)进一步提高诊断的准确性。
当前版本的“DKFZ肉瘤分类器”(v12.2)旨在通过单独的甲基化分类来识别和区分54种不同的骨骼和软组织肿瘤类型8.由于用于生成甲基化分类的肿瘤样本数量不同,目前WHO分类区分了>170种肿瘤亚型,分类器的性能仍有可预测的局限性。甲基化类成骨细胞瘤仅包括7个肿瘤的甲基组数据,这可能不足以捕获这种良性和骨形成肿瘤的完整甲基组谱(www.molecularneuropathology.org/mnp).此外,据描述,与新鲜冷冻组织相比,使用FFPE样品的分类器灵敏度较低9.所有这些方面都可能导致我们研究中只有三分之一的病例被分类器正确预测为成骨细胞瘤(评分≥0.9)。然而,当同时(或仅)考虑拷贝数配置文件时,97%的病例可以确诊。基于甲基化的聚类似乎也是一种有趣的工具,可以使用非线性降维(UMAP)来可视化单个肿瘤之间甲基化轮廓的相似性。在这项研究中,我们发现成骨细胞瘤形成一个明显的簇,与传统的骨肉瘤截然不同。其他成骨性肿瘤类型也可区分,如低级别中央骨肉瘤与常规骨肉瘤或成骨母细胞瘤与骨膜骨肉瘤,但这些鉴别诊断通常易于在显微镜下和临床放射学背景下进行,不需要额外的遗传分析。所识别的聚类与单个肿瘤的预期拷贝数分布有很好的相关性。然而,UMAP图清楚地显示了局限性,一些相当相似的肿瘤不能区分,包括骨旁骨肉瘤与中枢性低级别骨肉瘤,骨膜骨肉瘤与常规骨肉瘤。我们研究中纳入的成骨细胞瘤样骨肉瘤在4/5的可评估病例中被错误分类。
除了甲基化和拷贝数分析外,FOS的免疫染色以及a”丛书FISH重排是可靠的诊断工具,85%的成骨细胞瘤在至少一项原位分析中呈阳性。值得注意的是,5例成骨细胞瘤样骨肉瘤的FOS免疫组化完全阴性,我们纳入的其余病例中有两种表现,一例为局灶性阳性,另一例为阴性。所有FOS/ fosb阴性成骨细胞瘤(n= 5,均由Lyskjaer等人先前发表。9)在本研究中通过甲基组分析评估的细胞被正确定位在成骨细胞瘤簇中。这表明,即使在没有可检测到的情况下,本文所示的方法确实有助于区分成骨细胞瘤与常规骨肉瘤”丛书/FOSB融合,代表一个有用的补充工具。尽管非常理想,但依赖于免疫组化表达FOS/FOSB的成骨细胞瘤分子表征在某些情况下可能会产生误导。事实上,在一小部分常规骨肉瘤(<14%)中有FOS的表达,尽管免疫反应通常是局部性的11.此外,先前的研究描述了富含上皮样细胞的成骨细胞瘤不携带”丛书/FOSB基因融合6,17.与这些观察结果相反,小系列的6个颌骨上皮样成骨细胞瘤均显示强烈的核FOS染色18.在我们的系列研究中,我们发现只有3个肿瘤表现出令人信服的上皮样形态,而在其他病例中,更大的上皮样细胞仅在常规成骨细胞瘤中发现。三例中有一例保存完好的DNA数量不足,因此甲基化阵列无法评估。然而,其他两例FOS免疫组化强阳性,并在甲基化聚类评估时与传统成骨细胞瘤共定位。参与拷贝数改变的基因组比例<2%。因此,上皮样成骨细胞瘤仍然是一种罕见且定义不明确的亚型,至少从这里非常有限的数据来看,它似乎显示出与常规成骨细胞瘤相似的甲基组和拷贝数分布。
总之,针对FOS/FOSB的免疫组织化学仍然是确认成骨细胞瘤诊断和排除(成骨细胞瘤样)骨肉瘤的最快、最具成本效益和合理的诊断附加。由于所有的骨形成肿瘤通常都需要脱钙,而在许多研究所仍然依赖于酸基方法,大量的活检将不适用于额外的分子分析,这也从我们研究的高辍学率中可以看出。然而,在形态学异常病例或FOS/FOSB分析不一致的情况下,甲基化和拷贝数分析是一种新兴的应用工具,可以提供有价值的额外信息,以准确分类单个病变。此外,当定义个体甲基化分类的肿瘤集显著扩大并且甲基化组分析的分辨率提高时,基于甲基化组的分类可能会变得更加准确。850k甲基化阵列仅查询基因组中存在的约3%的CpG位点,对于基于甲基化的聚类,目前仅使用和评估了10 - 25k的CpG位点。整个甲基组测序和更先进的算法来评估这些数据可能会导致更精确的预测,甚至是罕见的肿瘤亚型。在不久的将来,使用实时纳米孔测序的甲基化分析可以在2-3小时内进一步提供结果,这可能会改变手术病理的总体角度,因为组织切片和分子数据将同时可用19.
数据可用性
本研究期间生成的甲基化史诗(=850k)的所有原始强度数据文件(IDATs)已存入欧洲核苷酸档案,研究登录号为EGAD00010002279。
参考文献
世界卫生组织肿瘤分类:卷3.软组织与骨肿瘤.(世卫组织,2020年)。
Fittall, m.w.等。FOS和FOSB的反复重排定义成骨细胞瘤。Nat Commun9, 2150(2018)。
Panagopoulos等人。FOS-ANKH和FOS-RUNX2融合基因在成骨细胞瘤中的作用。癌症基因组学蛋白质组17, 161-168(2020)。
Lam, S. W.等。FOS作为骨样骨瘤和成骨细胞瘤诊断标志物的应用。菲尔绍拱476, 455-463(2020)。
诺德,K. H.等。成骨细胞瘤中复发的22号染色体缺失会影响Wnt/ -连环蛋白信号通路的抑制剂。《公共科学图书馆•综合》8, e80725(2013)。
Saba, K. H.等。NF2缺失定义了非fos重排成骨细胞瘤的遗传亚群。病理临床研究6, 231-237(2020)。
Capper, D.等人。基于DNA甲基化的中枢神经系统肿瘤分类。自然555, 469-474(2018)。
Koelsche, C.等。基于DNA甲基化谱的肉瘤分类。Nat Commun12, 498(2021)。
Lyskjaer, I.等。骨和软组织肿瘤的DNA甲基化分析:“DKFZ肉瘤分类器”的验证研究。病理临床研究7, 350-360(2021)。
van IJzendoorn, d.g.p.等。融合事件导致骨上皮样血管瘤中FOS的截断。基因染色体癌症54, 565-574(2015)。
玛丽,F.等人。FOS在骨样骨瘤和成骨细胞瘤中的表达:一种有价值的辅助诊断工具。Am J Surg Pathol43, 1661-1667(2019)。
最差的是b.c.等人。用于高危儿科癌症患者的下一代个性化药物——INFORM试点研究。欧元癌症65, 91-101(2016)。
Haefliger, S.等人。一例病例报告说明了一种新的有前途的诊断策略:通过单分子分析方法鉴别NUT中线癌及其差异。菲尔绍拱478, 1007-1012(2021)。
Duhamel, l.a.e.等人。小鼠双分2 (MDM2)和小鼠双分4 (MDM4)在骨旁肉瘤和常规骨肉瘤亚型中的扩增频率:对应。组织病理学60, 357-359(2012)。
McInnes, L., Healy, J. & Melville, J. UMAP:统一流形近似和投影降维。ArXivhttps://doi.org/10.48550/arXiv.1802.03426(2018)。
李文杰,李文杰,李文杰,李文杰。利用DNA甲基化分析进行分子肿瘤分类。哼Mol Genet29, r205-r213(2020)。
Panagopoulos等人。COL1A1和FYN基因在上皮样成骨细胞瘤中的融合。癌症基因组学蛋白质组16, 361-368(2019)。
Pereira, T.等人。6例颌骨上皮样成骨细胞瘤的临床病理研究及FOS、FOSB免疫表达分析。口腔外科,口腔医学,口腔病理,口腔无线电130, 191-199(2020)。
Euskirchen, P.等人。利用实时纳米孔测序对脑肿瘤进行当日基因组和表观基因组诊断。神经病理学报(Berl)134, 691-703(2017)。
资金
BA和DB由瑞士国家科学基金会、巴塞尔骨肿瘤参考中心基金会、Gertrude von Meissner基金会支持。BA, DB和SH由基金会für krebskranke Kinder, Regio Basiliensis赞助。FHF得到了瑞典癌症协会(资助号JCIA-190003)和斯德哥尔摩癌症协会(资助号284053)的支持。巴塞尔大学提供的开放获取资金。
作者信息
作者及隶属关系
贡献
DB, BA和SH构思并设计了该研究。BA、SH和JH获取、分析或解释数据。BA和DB起草了手稿。MN, KHN, FHF和JVMGB对重要的知识内容进行了批判性的修改。MN、KHN、FHF、JVMGB、AHK和JH提供行政、技术或物质支持。DB监督了这项研究。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
伦理批准
来自巴塞尔的案例由巴塞尔伦理委员会(Ethikkommission beider Basel)提供伦理批准(文献274/12),来自瑞典的案例由Regionala etikprövningsnämnden斯德哥尔摩(文献2013 1979-31),来自隆德的案例由隆德大学地区伦理委员会提供。来自莱顿的肿瘤是根据莱顿大学医学中心伦理委员会批准的“荷兰人体组织适当二次使用准则”中描述的道德准则进行编码(化名)处理的(B17.036和B19.007)。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
补充信息
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
阿米琳,B.,纳斯拉斯,M.,诺德,K.H.et al。甲基化和拷贝数分析:区分成骨细胞瘤与恶性模拟肿瘤的新兴工具?国防部分册35, 1204-1211(2022)。https://doi.org/10.1038/s41379-022-01071-1
收到了:
修改后的:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41379-022-01071-1