文摘gydF4y2Ba
CRISPR淘汰赛健身屏幕在癌症细胞系揭示许多基因的损失函数会导致细胞死亡或损失健身或更很少,相反的表现型更快的扩散。在这里我们展示一种系统化的方法来识别这些增殖抑制,肿瘤抑制基因的高纯度,并定义一个145个这样的基因网络在22个模块。一个模块包含几个元素glycerolipid生物合成途径和操作只在急性髓系白血病细胞系的子集。增殖抑制活性的基因参与的合成饱和脂肪酸,再加上更严重亏损健身减饱和通路中的基因表型,表明这些细胞操作的极限承载能力为饱和脂肪酸,我们确认生化反应。超表达的这个模块与青少年白血病的生存优势,建议临床相关的子类型。gydF4y2Ba
介绍gydF4y2Ba
基因遗传学家淘汰赛中是一个基本工具,和发现CRISPR-based基因组编辑gydF4y2Ba1克ydF4y2Ba及其适应基因敲除屏幕哺乳动物功能基因组学和癌症的目标发生了革命性变化gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba。数以百计的CRISPR / Cas9淘汰赛屏幕在癌症细胞系显示background-specific遗传缺陷gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,提供指导肿瘤特异性治疗和靶向制剂的发展。虽然血统和上下文相关的基因的突变状态强大的预测因子重要性,细胞生长介质和环境的变化也可以驱动不同的细胞状态,尤其是代谢基因gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,和有针对性的筛选可以揭示代谢途径的基因因素缓冲gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba。gydF4y2Ba
的存在和合成代谢和细胞的其他功能模块也可以推断通过综合分析大量的屏幕。相关基因敲除健身资料,测量在数以百计的屏幕,被用来推断基因功能和人类细胞的模块化的体系结构gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba。数据驱动的相关网络的分析,将揭示集群功能相关基因的紧急重要性在特定细胞背景往往是由底层血统或原因不明的突变gydF4y2Ba21gydF4y2Ba。有趣的是,在最近的一项研究的假字功能缓冲系统看不见的单基因CRISPR击倒屏幕呈现它们gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,结果表明,大部分的上下文相关的重要基因持续表达的细胞株gydF4y2Ba23gydF4y2Ba。集体这些观测结果表明,有很多无法解释的变异在遗传体系结构中,和紧急脆弱性,肿瘤细胞。gydF4y2Ba
构建人类功能交互网络相关基因敲除健身档案癌细胞类似于从相关基因交互配置文件生成功能交互网络gydF4y2Ba酿酒酵母gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba。两种方法之间的根本区别是,成对基因的酵母,大规模筛选淘汰赛中在一个单一的酵母菌株是为了衡量基因进行交互dual-knockout表型或多或少比预期严重的组合独立两个基因。在coessentiality网络,CRISPR-mediated单基因跨细胞小组淘汰赛中进行样本的多样性癌症基因型和血统。二基因型的扰动在人类细胞,更忠实的复制的酵母的方法,是有可能与Cas9及其变种,但图书馆建设,测序,位置偏差可以是有问题的gydF4y2Ba16gydF4y2Ba,gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29日gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31日gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba。最近,我们显示一个工程的变体的Cas12a核酸内切酶,enCas12agydF4y2Ba35gydF4y2Ba,可以有效地执行多重基因淘汰赛gydF4y2Ba34gydF4y2Ba,我们证明了其有效性分析合成目标假字之间的杀伤力gydF4y2Ba23gydF4y2Ba。这些原则的发展使研究人员可以测量出有生物网络不同背景,一个强大的方法来破解复杂的生物gydF4y2Ba24gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba。gydF4y2Ba
CRISPR干扰可能导致功能丧失的等位基因在人类细胞,增加以及体外增殖率下降;更快的扩散是一个极端罕见的酵母淘汰赛。这些fast-growers复杂化的预测基因的相互作用gydF4y2Ba29日gydF4y2Ba合用的药物抗性,并影响着屏幕gydF4y2Ba38gydF4y2Ba。然而,还没有受到广泛接受的方法识别这些基因从CRISPR屏幕。gydF4y2Ba
在这项工作中,我们描述一个方法来系统地分类的发展基因的敲除提供体外增殖优势。我们观察到的基因具有增殖优势通常是肿瘤抑制基因,他们显示相同的模块化和功能一致性上下文相关的重要基因。此外,我们发现一个模块,包括多个组件的glycerolipid生物合成途径,减缓了细胞增殖的一个子集急性髓系白血病(AML)细胞株。我们显示重新使用enCas12a多元筛选遗传相互作用网络,并找到强大的遗传相互作用通过临床生存数据证实。glycerolipid生物合成的一个假定的肿瘤抑制作用是值得注意的考虑这个过程被认为是需要为肿瘤细胞生长,生成生物质和抑制剂针对这个途径目前正在临床试验gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba。gydF4y2Ba
结果gydF4y2Ba
识别proliferation-suppressor签名gydF4y2Ba
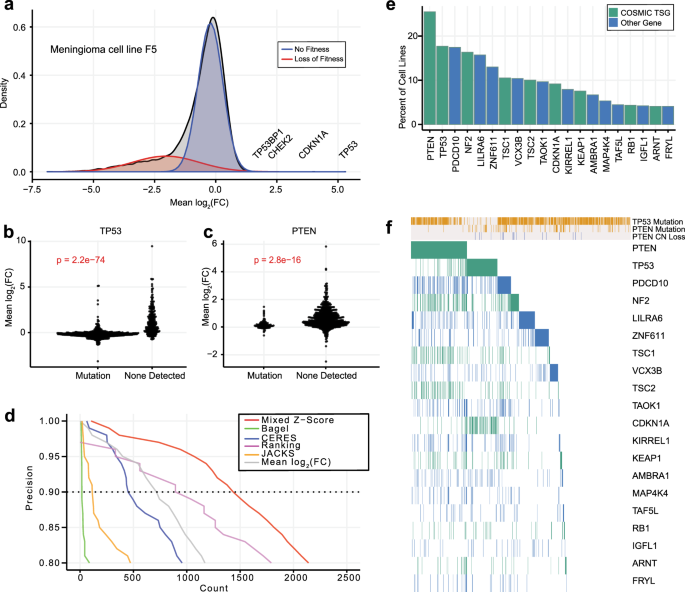
我们以前观察到的基因的敲除会导致群体在图书馆汇集淘汰赛屏幕。这些基因,我们学期proliferation-suppressor基因(PSG),表现出积极的选择健身屏幕,对面的表型的重要基因。正如预期的那样,许多PSG是已知的肿瘤抑制基因;例如,gydF4y2BaTP53gydF4y2Ba及相关通路的基因gydF4y2BaCDKN1AgydF4y2Ba,gydF4y2BaCHEK2gydF4y2Ba,gydF4y2BaTP53BP1gydF4y2Ba在选择细胞系(图显示积极的选择。gydF4y2Ba1gydF4y2Ba)。检测这些基因的异常值是健壮CRISPR分析方法的选择,当我们BAGEL2测试gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,谷神星gydF4y2Ba10gydF4y2Ba,杰克gydF4y2Ba43gydF4y2Ba,意味着log-fold改变(利物浦)gRNA针对每个基因(补充图。gydF4y2Ba1模拟gydF4y2Ba)。与core-essential基因,PSG非常特定于上下文的:gydF4y2BaTP53gydF4y2Ba淘汰赛只显示了积极的利物浦与野生型细胞系gydF4y2BaTP53gydF4y2Ba(无花果。gydF4y2Ba1 bgydF4y2Ba),gydF4y2BaPTENgydF4y2Ba淘汰赛显示了PS只表型gydF4y2BaPTENgydF4y2BawtgydF4y2Ba背景(图。gydF4y2Ba1 cgydF4y2Ba)。这些观察是一致的淘汰赛表型的肿瘤抑制基因(次数)细胞系:在野生型细胞,次数淘汰赛细胞培养中的增殖率增加,但当细胞系来源于次数已经丢失或非功能性肿瘤,基因敲除没有影响。次数是上下文相关的,但它并不一定与proliferation-suppressor表型的基因体外体内充当次数gydF4y2Ba;gydF4y2Ba增殖抑制充其量是假定的肿瘤抑制肿瘤没有确认的数据分析。gydF4y2Ba
一个gydF4y2Ba叠化分布的一个典型CRISPR淘汰赛屏幕有着悠久的左尾至关重要的基因,和少量的基因敲除增加健身(proliferation-suppressor基因,“PSG”)。双组份高斯混合模型(红、蓝)模型分布。gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba褶皱的变化在808细胞系(常见的肿瘤抑制gydF4y2BaPgydF4y2Ba值,两面Wilcoxon rank-sum测试)。gydF4y2BadgydF4y2Ba精度和召回的混合gydF4y2BaZgydF4y2Ba分数和其他CRISPR分析方法。罗斯福虚线,90%精度(10%)。gydF4y2BaegydF4y2Ba分数的细胞系(绿色)或其他已知的肿瘤抑制基因(蓝色,不是定义为宇宙次数)被归类为罗斯福PS基因在10%。gydF4y2BafgydF4y2Ba每个已知的次数在808细胞系,与细胞的遗传背景。黄金,突变;灰色,缺席。绿色或蓝色,配色方案后(gydF4y2BaegydF4y2Ba),被归为一种增殖抑制基因。gydF4y2Ba
虽然PSG的检测可以使用现有的信息管道,几个因素使一个健壮的检测这些基因。没有任何接受阈值算法我们认为检测PSG,因为所有被优化的关键基因进行分类。一个相关的第二个问题是细胞株屏幕显示广泛的方差在利物浦分布,使健壮的异常值检测具有挑战性(补充图。gydF4y2Ba1 e, fgydF4y2Ba)。第三,强烈background-dependent签名,证明了gydF4y2BaPTENgydF4y2Ba和gydF4y2BaTP53gydF4y2Ba。最后,没有一致的期望是否有多少假定的肿瘤抑制基因存在于给定的细胞系。gydF4y2Ba
为了解决这个差距,我们开发了一个方法来占可变性在屏幕之间的叠化分布。我们的方法使用高斯混合模型(K = 2)来估计每个屏幕的分布能够利物浦成绩(无花果。gydF4y2Ba1gydF4y2Ba)。混合分布模型曾被用于识别区分必要和不必要的健康基因的数量在CRISPR屏幕gydF4y2Ba44gydF4y2Ba。K = 2的混合模型,负分布(图。gydF4y2Ba1gydF4y2Ba,红色)通常是至关重要的基因,而高,窄峰值在零附近(无花果。gydF4y2Ba1gydF4y2Ba、蓝色)、模型淘汰赛中没有健身表型的庞大的人口。我们使用第二个模型来计算gydF4y2BaZ -gydF4y2Ba分数(以下称为“混合gydF4y2BaZ -gydF4y2Ba分数”)对所有能够意味着每个细胞株褶皱的变化。这种方法使方差(补充图规范化。gydF4y2Ba1 e, fgydF4y2Ba)利物浦分布在不同的细胞系,与消极的gydF4y2BaZgydF4y2Ba分数显示重要基因和积极的gydF4y2BaZgydF4y2Ba分数代表PSG表型。gydF4y2Ba
这种混合的有效性进行评估gydF4y2BaZgydF4y2Ba分数的方法,我们使用宇宙gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba肿瘤抑制基因作为一个真正的积极参考集,我们结合COSMIC-defined致癌基因(切除肿瘤抑制dual-annotated)与我们先前指定的不必要的基因作为一个真正的负面引用集gydF4y2Ba7gydF4y2Ba,gydF4y2Ba47gydF4y2Ba。由于没有期待的存在一组一致的PSG细胞系,我们分析每个808细胞系Avana独立2020年第四季度数据发布gydF4y2Ba10gydF4y2Ba,gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba计算在每个细胞株分别能够得分,然后合并到主列表的所有分数808×18 k = 1460万gene-cell行观察(补充数据gydF4y2Ba1克ydF4y2Ba)。此外,由于也没有期望,所有宇宙次数累计检测出所有的细胞系,我们认为传统召回指标(如参考集回收的百分比)是不合适的。因此,我们定义召回总数TSG-cell线的观察。使用该评价方案,喜忧参半gydF4y2BaZgydF4y2Ba得分方法显著优于同类方法的,超过722个PS-cell线实例分类错误发现率10%(罗斯福)(图。gydF4y2Ba1 dgydF4y2Ba)。这是公认的PSG ~ 50%比最近的选择,非参数rank-based的方法,在同一罗斯福。百吉饼gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba监督分类的重要基因,表现最差的次数,和原始意味着利物浦方法也表现不佳,强调在实验方差归一化的必要性。我们应用这10%罗斯福阈值为所有后续分析。gydF4y2Ba
常见的肿瘤抑制基因gydF4y2BaPTENgydF4y2Ba和gydF4y2BaTP53gydF4y2Ba观察在~ 25% ~ 18%的细胞系,分别(无花果。gydF4y2Ba1 egydF4y2Ba与其他著名的次数),出现的频率更低。309宇宙为我们健身次数概要(代表1.7%的18 k基因),我们发现116(37.5%)的这些基因发生增殖抑制至少一次(补充数据gydF4y2Ba2gydF4y2Ba),占总数的24.4% proliferation-suppressor调用(补充图。gydF4y2Ba2 a, bgydF4y2Ba),14倍浓缩。所有已知的次数达到808细胞系来自504(62.4%),其中proliferation-suppressor调用被确定(图。gydF4y2Ba1 fgydF4y2Ba),但我们没有观察到倾向于特定组织:在每一个血统,大多数细胞株进行至少一个PSG(补充图。gydF4y2Ba1克gydF4y2Ba)。gydF4y2Ba
进一步验证我们的方法,我们在PSG的次数相比其他分子分析数据。确认为增殖抑制时,53%的116次数展示更高意味着mRNA表达相对于背景相同的次数不是增殖抑制(补充数据gydF4y2Ba2gydF4y2Ba)。类似的,116年的次数的96.6%,分为增殖抑制时,演示nonsilent突变的频率较低相对于背景的次数不是(补充数据gydF4y2Ba2gydF4y2Ba)。然而,这些观察并不局限于宇宙的次数,因为这是所有PSG击中调用的基因对non-PSG打击调用(补充图。gydF4y2Ba2摄氏度,gydF4y2BadgydF4y2Ba)。人类基因组的比较并没有建议主要PSG和non-PSG调用(补充图之间的区别。gydF4y2Ba2 egydF4y2Ba),然而,却似乎有更多的PSG的变化观察,可能源于较小的分组样本大小。这些观察证实了我们的方法的可靠性检测基因的敲除导致细胞增殖快,而且,类似于重要的基因,这些基因必须表达和不能港口功能丧失的突变,以引起表型。gydF4y2Ba
我们试图证实发现使用第二CRISPR数据集342细胞株屏幕Behan et al。gydF4y2Ba13gydF4y2Ba,包括> 150屏幕在相同的细胞系Avana数据。然而,这些屏幕进行了在较短的时间内比Avana屏幕(14比21天),让更少的时间为积极的和消极的选择信号出现(见详细讨论的“方法”)。因此,当我们通过两组相比细胞株筛选,Avana数据产生了更多的次数(补充图。gydF4y2Ba3gydF4y2Ba)。虽然大多数的这些不符合我们的阈值桑格PSG的数据,在10%的罗斯福阈值在所有Avana屏幕强烈偏向积极的混合gydF4y2BaZgydF4y2Ba分数在桑格屏幕(补充图。gydF4y2Ba3 bgydF4y2Ba),与弱信号一致的积极的选择短化验的结果,而不是在屏幕上缺乏鲁棒性gydF4y2Ba49gydF4y2Ba。gydF4y2Ba
发现通路调节细胞生长与proliferation-suppressor共生网络gydF4y2Ba
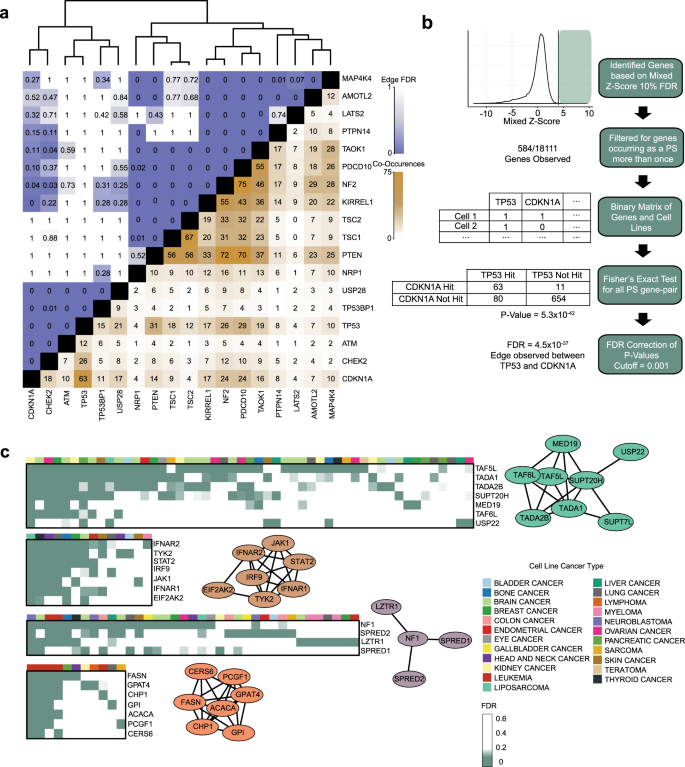
尽管已知的次数作为PSG只有一个子集的细胞系,我们观察到的同现模式功能相关基因之一。gydF4y2BaPTENgydF4y2Ba伴有mTOR监管机构gydF4y2BaNF2gydF4y2Ba50gydF4y2Ba(gydF4y2BaPgydF4y2Ba< 3×10gydF4y2Ba−11gydF4y2Ba,确切概率法)和gydF4y2BaTSC1gydF4y2Ba/gydF4y2BaTSC2gydF4y2Ba复杂(gydF4y2BaPgydF4y2Ba值< 7×10gydF4y2Ba−13gydF4y2Ba)gydF4y2Ba51gydF4y2Ba10、程序性细胞死亡(gydF4y2BaPDCD10gydF4y2Ba)gydF4y2Ba52gydF4y2Ba提出了肿瘤抑制gydF4y2Ba7gydF4y2Ba,gydF4y2Ba53gydF4y2Ba(无花果。gydF4y2Ba2gydF4y2Ba)。p53监管集群(gydF4y2BaTP53gydF4y2Ba,gydF4y2BaCDKN1AgydF4y2Ba,gydF4y2BaCHECK2gydF4y2Ba,gydF4y2BaTP53BP1gydF4y2Ba)也表现出强烈的同现模式是独立于mTOR监管集群(图。gydF4y2Ba2gydF4y2Ba)。mTORgydF4y2Ba54gydF4y2Ba基因和细胞周期检查点gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba已经严重导致癌症的发展,因为他们的角色在控制细胞生长和增殖,因此一直研究的重点描述病人基因组资料识别常见的途径改变吗gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba。gydF4y2Ba
mTOR监管机构的模块化和TP53监管机构表明pathway-level proliferation-suppressor活动。这反映了基因的概念与相关健身资料表明基因在相同的生化途径或生物过程gydF4y2Ba19gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba59gydF4y2Ba,gydF4y2Ba60gydF4y2Ba。然而,PSG的稀少,再加上小尺度效应,使得相关网络相对贫穷与proliferation-suppressor识别模块的基因活动。为了识别这类模块,我们开发了一个PSG网络(补充数据gydF4y2Ba3gydF4y2Ba)基于统计群体的共病的PSG(无花果。gydF4y2Ba2 bgydF4y2Ba);有关详细信息,请参阅“方法”。这种方法产生的145个基因网络断开连接的集群包含462边缘;只有8集群有3个或更多的基因(图。gydF4y2Ba2摄氏度gydF4y2Ba和补充图。gydF4y2Ba4摄氏度gydF4y2Ba)。这462的边缘,74(16.0%,实证gydF4y2BaPgydF4y2Ba< 10gydF4y2Ba−4gydF4y2BaHumanNet)存在gydF4y2Ba61年gydF4y2Ba功能交互网络(补充图。gydF4y2Ba4 a、bgydF4y2Ba),从随机抽样~ 8倍大于预期,表明高功能之间的相干性相关的基因。网络恢复PTEN和TP53模块以及河马通路,芳基碳氢化合物受体复杂(AHR / ARNT)的mTOR-repressing GATOR1复杂,STAGA染色质重构复杂,JAK-STAT信号,γ分泌酶复杂(图。gydF4y2Ba2摄氏度gydF4y2Ba和补充gydF4y2Ba4摄氏度gydF4y2Ba),所有这些都与肿瘤抑制相关活动。的功能一致性和生物相关性PSG共生网络进一步验证方法和建立这个数据集作为资源探索假定的肿瘤细胞系和肿瘤抑制基因活动。gydF4y2Ba
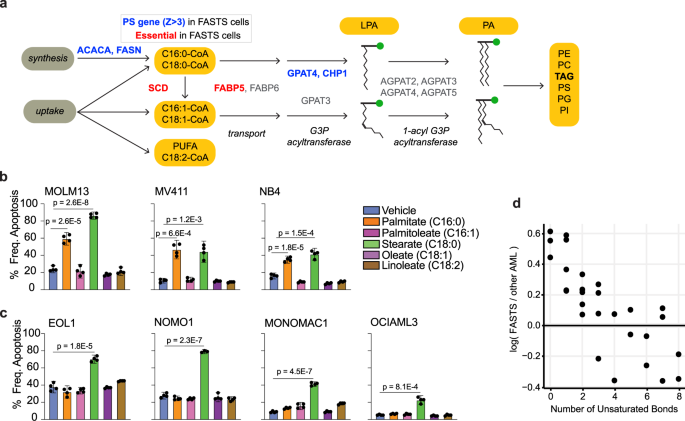
在AML细胞脂肪酸代谢的变化gydF4y2Ba
除了已知的肿瘤抑制,我们观察到一个大模块包含几种脂肪酸的元素(FA)和脂类的生物合成途径(图gydF4y2Ba2摄氏度gydF4y2Ba)。有趣的是,虽然似乎没有一个强有力的组织特异性签名对于大多数集群(图。gydF4y2Ba2摄氏度gydF4y2Ba),脂肪酸代谢集群显示了强大的浓缩AML细胞株(gydF4y2BaPgydF4y2Ba= 1.5×10gydF4y2Ba−4gydF4y2Ba)。AML,像大多数癌症一样,通常依靠增加葡萄糖消耗能源和转移所需的糖酵解生成的中间体生物细胞增殖。生物膜是由磷脂生物合成,利用脂肪酸作为构建块、与FA池补充一些甘油三酯分解代谢、transporter-mediated吸收,并通过的从头合成gydF4y2Baac / ACACA / FASNgydF4y2Ba棕榈酸酯生产使用柠檬酸前体转移从三羧酸循环途径。事实上,脂质代谢的作用在AML过程是由血清脂质含量的变化gydF4y2Ba62年gydF4y2Ba,特别是对长链饱和脂肪酸FAS管道的终端产品。抑制的合成是一个吸引人的化疗干预gydF4y2Ba63年gydF4y2Ba,gydF4y2Ba64年gydF4y2Ba和FASN抑制剂目前正在临床试验治疗实体肿瘤和代谢性疾病gydF4y2Ba40gydF4y2Ba。观察击出FAS通路基因的结果在一些AML细胞增殖快,和他们的签名作为假定的肿瘤抑制基因,因此非常意外。gydF4y2Ba
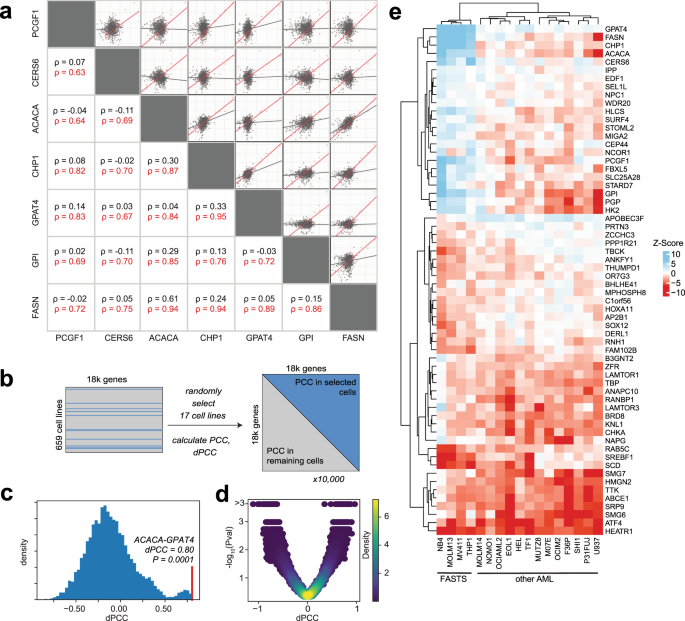
学习额外的元素是否与FAS集群相关的脂质代谢,我们检查了微分混合的相关性gydF4y2BaZgydF4y2Ba分数在AML细胞。我们和其他人已经表明,基因与相关基因敲除健身档案CRISPR屏幕可能会参与相同的生物通路或过程(“co-functional”)gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,类似于酵母相关基因交互配置文件gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba,gydF4y2Ba65年gydF4y2Ba。引人注目的是,完全连接集团内的所有基因对FAS集群(包含基因gydF4y2BaFASNgydF4y2Ba,gydF4y2BaACACAgydF4y2Ba,gydF4y2BaGPAT4gydF4y2Ba,gydF4y2BaCHP1gydF4y2Ba,gydF4y2BaGPI CERS6, PCGF1gydF4y2Ba,无花果。gydF4y2Ba2摄氏度gydF4y2Ba)平均0.76的皮尔森相关系数(PCC) 23 AML细胞株(范围0.63 - -0.95,无花果。gydF4y2Ba3gydF4y2Ba,红色),而相关的中位数0.05在785年剩下的细胞系(−0.11 - -0.62,最高的相关性gydF4y2BaFASNgydF4y2Ba和gydF4y2BaACACAgydF4y2Ba,毗邻线性棕榈酸酯合成途径酶;无花果。gydF4y2Ba3gydF4y2Ba、灰色)。这些高微分皮尔逊相关系数(dPCC)表明,脂质代谢的变化是明显的AML细胞gydF4y2Ba66年gydF4y2Ba。gydF4y2Ba
一个gydF4y2Ba基因中FAS模块,皮尔森相关系数的混合gydF4y2BaZ -gydF4y2Ba更高的分数概要文件在AML细胞比其他细胞(红色)(灰色)。gydF4y2BabgydF4y2Ba意义的测试微分PCC (dPCC)涉及质量的过滤Avana数据(gydF4y2BangydF4y2Ba= 659细胞株,其中17 AML细胞株),建立一个空分布随机选择17个细胞系,并计算所有基因对之间的PCC选定的细胞和剩余的细胞。gydF4y2BacgydF4y2Ba10000年之后重复,零分布为每一对生成,gydF4y2BaPgydF4y2Ba值(排列测试,gydF4y2BangydF4y2Ba= 10000)观察AML和其他dPCC计算。gydF4y2BadgydF4y2Ba火山的情节dPCC vs。gydF4y2BaPgydF4y2Ba值Co-PS集群中的所有基因。gydF4y2BaegydF4y2Ba热图的混合gydF4y2BaZ -gydF4y2Ba得分17 AML细胞株在选定的基因混合Z |和| |高dPCC |。集群的细胞系表示假定的脂肪酸合成/肿瘤抑制(禁食)亚型。gydF4y2Ba
我们试图探究这种差异是否在相关识别其他基因可能在AML给洞察代谢重新布线。我们第一次被嘈杂的数据为高质量的屏幕(科恩的过滤gydF4y2BaDgydF4y2Ba> 2.5,回忆> 60%gydF4y2Ba42gydF4y2Ba),留下659细胞株,其中包括17 AML细胞株。计算全球区别PCC的基因对17个AML和其余642细胞株产生了许多基因对其dPCC出现的随机抽样(补充图。gydF4y2Ba5 a、bgydF4y2Ba)。过滤这些,我们计算经验gydF4y2BaPgydF4y2Ba每个基因值对。我们从池中随机选择17个细胞系的屏幕,计算PCC中所有基因对选择和剩余的线,并计算dPCC从这些PCC值(无花果。gydF4y2Ba3 bgydF4y2Ba)。我们重复这个过程生成一个1000倍的零分布为每一对基因dPCC值,反对的gydF4y2BaPgydF4y2Ba值可以计算(无花果。gydF4y2Ba3 c, dgydF4y2Ba)。gydF4y2Ba
扩大将过滤列表的基因与基因相关FAS小团体显示显著改变AML细胞(gydF4y2BaPgydF4y2Ba< 0.001;参见“方法”)产生了共106个基因,包括小团体(图7基因。gydF4y2Ba3 egydF4y2Ba)+ holocarboxylase合成酶(gydF4y2BahlcgydF4y2Ba),biotinylates和激活acetyl-CoA-carboxylase蛋白质产品gydF4y2BaACACAgydF4y2Ba,以及糖酵解途径的基因gydF4y2BaPGPgydF4y2Ba和gydF4y2BaHK2gydF4y2Ba。有趣的是,大约有一半的基因显示显著增加anticorrelation FAS集群,表明基因优先重要作为增殖抑制FAS的基因(无花果。gydF4y2Ba3 egydF4y2Ba)。这些基因包括脂肪酸desaturase (gydF4y2Ba镜头分割gydF4y2Ba),直接下游gydF4y2BaFASN / ACACAgydF4y2Ba生成单一不饱和脂肪酸物种,sterol-regulatory元件结合转录因子1 (gydF4y2BaSREBF1gydF4y2Ba),主细胞脂质稳态的调节因子。gydF4y2Ba
集群AML细胞行根据这些high-dPCC基因揭示了两种截然不同的细胞亚群。FAS集群及其关联显示四个细胞系,强劲proliferation-suppressor表型NB4, MV411 MOLM13, THP1。其余13 AML细胞株显示弱基本可以忽略不计表型当这些基因被淘汰出局。anticorrelated基因,包括gydF4y2Ba镜头分割gydF4y2Ba和gydF4y2BaSREBF1gydF4y2Ba在这些相同的细胞系,显示高度重要性。基因敲除健身这些观察到的变化表明,该子集的AML细胞大量代谢重新布线。因为这些细胞之间共享基因签名脂肪酸合成途径与肿瘤抑制基因是一致的,我们称之为细胞系gydF4y2BaFgydF4y2BaattygydF4y2Ba一个gydF4y2BacidgydF4y2Ba年代gydF4y2Ba化合条件/gydF4y2BaTgydF4y2BaumorgydF4y2Ba年代gydF4y2Bauppressor(禁食)细胞(图。gydF4y2Ba3 egydF4y2Ba)。gydF4y2Ba
Cas12a-mediated基因交互屏幕确认重新在脂类代谢gydF4y2Ba
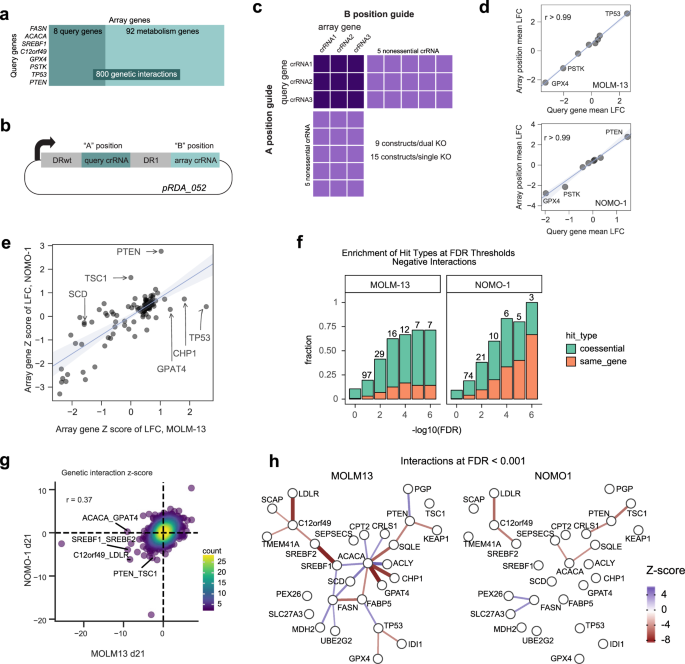
我们试图确定是否基因敲除授予改善细胞的健康,并收集一些深入了解为什么一些AML细胞显示绝食表型和其他人没有。遗传相互作用提供了一个强大的平台,了解细胞在生物模型重新布线,我们试图运用这种方法破译禁食表型。我们设计了一个CRISPR屏幕措施基因之间的相互作用8选择“查询基因”,~ 100其他基因(“阵列基因”)。查询基因包括gydF4y2BaFASNgydF4y2Ba和gydF4y2BaACACAgydF4y2Ba集群的proliferation-suppressor基因,以及脂质稳态转录因子gydF4y2BaSREBF1gydF4y2Ba集群,anticorrelated FAS的微分网络分析,和无特征基因gydF4y2Bac12orf49gydF4y2Ba,以前由coessentiality涉及脂质代谢gydF4y2Ba21gydF4y2Ba和最近的一个遗传相互作用研究gydF4y2Ba60gydF4y2Ba。附加查询基因包括控制肿瘤抑制基因gydF4y2BaTP53gydF4y2Ba和gydF4y2BaPTENgydF4y2Ba和控制上下文相关的重要基因gydF4y2BaGPX4gydF4y2Ba和gydF4y2BaPSTKgydF4y2Ba(无花果。gydF4y2Ba4gydF4y2Ba)。数组的基因包括两到三个基因来自多个代谢途径,包括各种分支的脂类的生物合成,糖酵解和glutaminolysis oxphos、过氧化物酶病和线粒体脂肪酸氧化。我们包括数组中的查询基因基因(图。gydF4y2Ba4gydF4y2Ba)为屏幕测试工件和进一步添加控制必要的和不必要的基因来衡量整体屏幕效果(补充数据gydF4y2Ba4 gydF4y2Ba和gydF4y2Ba5gydF4y2Ba)。gydF4y2Ba
一个gydF4y2Ba基因交互屏幕目标8查询基因,从禁食集群和dPCC分析,选择和100阵列基因采样脂质代谢通路,总共800成对淘汰赛。gydF4y2BabgydF4y2Ba图书馆设计使用一个dual-guide enCa12a查询目标基因的表达载体“a”位置和阵列基因“B”的位置。gydF4y2BacgydF4y2Ba图书馆整体设计包括三个crRNA /基因+控制crRNA针对不必要的基因。Single-knockout构造(目标基因与不必要的控制)允许Single-knockout健身的精确测量。gydF4y2BadgydF4y2Ba考虑single-knockout健身查询基因的“A”、“B”的位置crRNA表达载体没有位置效应在两个细胞系筛选(MOLM13 NOMO1)。利物浦log-fold变化。gydF4y2BaegydF4y2BaSingle-knockout健身(gydF4y2BaZ -gydF4y2Ba分数意味着利物浦)是MOLM13和NOMO1之间高度一致,但揭示了background-specific PS基因。gydF4y2BafgydF4y2Ba浓缩为同质的胃肠道和自动调节基因。Self-interactions中显示预计single-knockout健身表型的基因,反映GI观测的质量。gydF4y2BaggydF4y2Ba全球比较MOLM13 NOMO1遗传相互作用gydF4y2BaZ -gydF4y2Ba分数。gydF4y2BahgydF4y2Ba网络的相互作用在每一个背景显示重新布线MOLM13绝食细胞。gydF4y2Ba
我们使用了enCas12a CRISPR核酸内切酶系统进行多路基因淘汰赛gydF4y2Ba35gydF4y2Ba。我们使用dual-guide enCas12a设计,所述DeWeirdt et al。gydF4y2Ba34gydF4y2Ba,允许通过集中建设的具体指导对寡核苷酸合成(无花果。gydF4y2Ba4 bgydF4y2Ba)。图书馆有力措施single-knockout健身配对三Cas12a crRNA /目标基因每五crRNA针对不必要的基因gydF4y2Ba7gydF4y2Ba,gydF4y2Ba47gydF4y2Ba(gydF4y2BangydF4y2Ba= 15构造single-knockout健身),有效地分析double-knockout健身通过测量所有指南针对query-array基因双(gydF4y2BangydF4y2Ba=(图9)。gydF4y2Ba4摄氏度gydF4y2Ba和补充数据gydF4y2Ba5gydF4y2Ba)。使用这种高效的设计和enCas12a的内生多路复用功能,我们可以合成一个图书馆针对800个基因对一个12 k寡核苷酸阵列。gydF4y2Ba
我们筛选一个绝食子集AML细胞株,MOLM13,和第二个没有FAS表型,NOMO1,收集样本在14和21天后转导为期五天的嘌呤霉素选择(补充数据gydF4y2Ba6gydF4y2Ba和gydF4y2Ba7gydF4y2Ba)。重要的是,通过比较意味着log-fold改变查询基因的淘汰赛中在“A”的位置和相同的基因的“B”位置dual-knockout向量,我们找不到位置偏差的多路复用淘汰赛构造(图。gydF4y2Ba4 dgydF4y2Ba),与我们之前的结果一致gydF4y2Ba23gydF4y2Ba,gydF4y2Ba34gydF4y2Ba。Single-knockout健身测量有效地隔离已知基本来自不必要的基因,确认主屏幕(补充无花果的功效。gydF4y2Ba6gydF4y2Ba)。上下文相关的健身概要文件与细胞基因型一致,gydF4y2BaPTENgydF4y2Ba和gydF4y2BaTSC1gydF4y2Ba表现出积极的选择gydF4y2BaPTENgydF4y2BawtgydF4y2BaNOMO1细胞和gydF4y2BaTP53gydF4y2Ba作为一个强大的PS基因gydF4y2BaP53gydF4y2BawtgydF4y2BaMOLM13细胞。引人注目的是,gydF4y2BaCHP1gydF4y2Ba和gydF4y2BaGPAT4gydF4y2Ba在MOLM13未来两大支安打,确认proliferation-suppressor表型(无花果。gydF4y2Ba4 egydF4y2Ba),而不是显示NOMO1表型。一起这些观察验证enCas12a-mediated多元扰动平台,确认的能力CRISPR淘汰赛屏幕检测增殖抑制和确证background-specific fitness-enhancing FAS集群基因的影响。gydF4y2Ba
衡量遗传相互作用,我们每个指南之间的线性回归组合利物浦和single-guide利物浦,gydF4y2BaZgydF4y2Ba得分的残差这条线,结合所有指南针对同样的基因(补充图。gydF4y2Ba6gydF4y2Ba和补充数据gydF4y2Ba7gydF4y2Ba)。在这里,积极的基因相互作用gydF4y2BaZ -gydF4y2Ba分数反映比预期更大的健身和消极gydF4y2BaZ -gydF4y2Ba分数代表低于预期基于单基因独立淘汰赛,类似于遗传相互作用的方法应用于最近的一项调查在癌细胞中使用多路复用CRISPR干扰gydF4y2Ba33gydF4y2Ba(见“方法”)。基因self-interactions(当同样的基因在A和B的位置,无花果。gydF4y2Ba4 dgydF4y2Ba)应是负的增殖抑制和阳性必需品(无花果。gydF4y2Ba4 f, ggydF4y2Ba和补充图。gydF4y2Ba6gydF4y2Ba)。总的来说,基因相互作用gydF4y2BaZgydF4y2Ba分数在两个细胞系(图显示中度相关。gydF4y2Ba4 ggydF4y2Ba),之前报道合成之间的相互作用gydF4y2BaC12orf49gydF4y2Ba和低密度脂蛋白受体gydF4y2BaLDLRgydF4y2Ba17gydF4y2Ba和之间的gydF4y2BaSREBF1gydF4y2Ba和它的假字gydF4y2BaSREBF2gydF4y2Ba17gydF4y2Ba确定在两个细胞系(补充图。gydF4y2Ba6 f, ggydF4y2Ba)。gydF4y2Ba
与交互对比发现在这两种细胞系,background-specific基因交互反映细胞之间的基因型和表型差异。肿瘤抑制之间的负作用gydF4y2BaPTENgydF4y2Ba和mTOR抑制因子gydF4y2BaTSC1gydF4y2Ba在gydF4y2BaPTENgydF4y2BawtgydF4y2BaNOMO1细胞是一致的上位角色mTOR监管途径。两个基因显示积极的淘汰赛健身NOMO1(无花果。gydF4y2Ba4 egydF4y2Ba),但他们的双重淘汰赛不提供一个添加剂增长效应,导致抑制与负面的互动gydF4y2BaZgydF4y2Ba分数(无花果。gydF4y2Ba4 g hgydF4y2Ba)。同样,抑制基因之间的相互作用gydF4y2BaACACAgydF4y2Ba和下游proliferation-suppressor基因gydF4y2BaCHP1gydF4y2Ba和gydF4y2BaGPAT4gydF4y2Ba在MOLM13细胞明显,符合上位关系线性生化途径(图。gydF4y2Ba4 hgydF4y2Ba)。这些交互是不复制查询基因gydF4y2BaFASNgydF4y2Ba,但都gydF4y2BaFASNgydF4y2Ba和gydF4y2BaACACAgydF4y2Ba表现出负面的互动与脂肪酸运输基因gydF4y2BaFABP5gydF4y2Ba和积极的相互作用gydF4y2BaSREBF1gydF4y2Ba和gydF4y2Ba镜头分割gydF4y2Ba长链饱和脂肪酸的主要desaturase。所有这些相互作用在NOMO1缺席,展示了重新布线的脂质生物合成基因相互作用网络这两种细胞类型之间(无花果。gydF4y2Ba4 hgydF4y2Ba)。gydF4y2Ba
绝食签名预测敏感性饱和脂肪酸gydF4y2Ba
单的显著差异和double-knockout健身签名两个细胞系之间的建议在这些细胞脂质代谢的主要重新布线。gydF4y2BaCHP1gydF4y2Ba和gydF4y2BaGPAT4gydF4y2Ba是相互关联的Avana coessentiality网络(gydF4y2BargydF4y2Ba= 0.43,gydF4y2BaPgydF4y2Ba= 2.5×10gydF4y2Ba−34gydF4y2Ba),强烈co-functionality预测基因gydF4y2Ba21gydF4y2Ba。最近的两项研究lysophosphatidic酸酰基转移酶的作用特点gydF4y2BaGPAT4gydF4y2Ba在添加饱和酰基-磷酸甘油,半个生成lysophosphatidic酸(LPA)和磷脂酸(PA),细胞磷脂和甘油三酸酯的前体,进一步发现gydF4y2BaCHP1gydF4y2Ba作为一个关键调节因子gydF4y2BaGPAT4gydF4y2Ba活动gydF4y2Ba67年gydF4y2Ba,gydF4y2Ba68年gydF4y2Ba。在血液癌症细胞系,coessentiality网络重大改组,gydF4y2BaACACAgydF4y2Ba/gydF4y2BaFASNgydF4y2Ba模块与gydF4y2Ba镜头分割gydF4y2Ba在大多数背景(gydF4y2BargydF4y2Ba= 0.35,gydF4y2BaPgydF4y2Ba< 10gydF4y2Ba−18gydF4y2Ba),但强烈anticorrelated 36个血液癌症细胞系(gydF4y2BargydF4y2Ba=−0.52,gydF4y2BaPgydF4y2Ba< 10gydF4y2Ba−3gydF4y2Ba,无花果。gydF4y2Ba3 egydF4y2Ba)。这种变化的大小的相关性排名# 8 3100万个基因对(见“方法”)。相比之下,gydF4y2BaACACAgydF4y2Ba和gydF4y2BaFASNgydF4y2Ba是弱相关gydF4y2BaCHP1gydF4y2Ba在大多数组织但在AML强烈相关,与底层绝食细胞中的主要受到PS表型变异(无花果。gydF4y2Ba3 egydF4y2Ba)。这个途径逆转迹象证实在single-knockout健身中观察到我们的屏幕:gydF4y2Ba镜头分割gydF4y2Ba强烈的MOLM13但不是NOMO1(无花果。gydF4y2Ba4 egydF4y2Ba)。gydF4y2Ba
集体这些观察做出强烈预测特定脂质物种的代谢过程。快增殖相关基因敲除后饱和脂肪酸处理,加上依赖增加脂肪酸desaturase基因gydF4y2Ba镜头分割gydF4y2Ba(无花果。gydF4y2Ba5gydF4y2Ba),表明这些细胞是在或接近饱和脂肪酸的承载能力。为了测试这个预测,我们暴露出三个绝食细胞系和其他四个AML细胞株不同种类的饱和和不饱和脂肪酸。绝食细胞显示200年的存在显著增加细胞凋亡µM棕榈酸酯(无花果。gydF4y2Ba5 b, cgydF4y2Ba),而没有其他种类的饱和或不饱和脂肪酸显示类似的微分灵敏度。此外,分析癌症细胞系细胞代谢的百科全书gydF4y2Ba69年gydF4y2Ba,gydF4y2Ba70年gydF4y2Ba表明,饱和酰基链明显过多的三酰甘油(标签)禁食细胞(图。gydF4y2Ba5 dgydF4y2Ba),在与其他脂质物种测量(补充图。gydF4y2Ba7gydF4y2Ba)。Palmitate-induced lipotoxicity研究在许多环境和重要的是,所扮演的角色gydF4y2BaGPAT4gydF4y2Ba和gydF4y2BaCHP1gydF4y2Ba最近在调停lipotoxicity描述得很好gydF4y2Ba67年gydF4y2Ba,gydF4y2Ba68年gydF4y2Ba,但据我们所知,这是唯一的实例预测liposensitivity基因签名。gydF4y2Ba
一个gydF4y2Ba脂肪酸/ glycerolipid合成途径的示意图。蓝色,在绝食PSG细胞。红色,重要基因。路径分析表明,饱和脂肪酸是一个关键节点。gydF4y2BabgydF4y2Ba绝食细胞的凋亡在回应媒体补充200µM脂肪酸。所有三个细胞株显示标记对棕榈酸酯的敏感性。绘制酒吧(gydF4y2BabgydF4y2Ba)的均值和95%可信区间(CI)凋亡%。gydF4y2BaPgydF4y2Ba值(gydF4y2BabgydF4y2Ba)和(gydF4y2BacgydF4y2Ba)代表片面的孤gydF4y2BatgydF4y2Ba测试比较。每条情节展示了四个复制样品为测试的每个条件和细胞系。数据(gydF4y2BabgydF4y2Ba)和(gydF4y2BacgydF4y2Ba源数据文件中可以找到。gydF4y2BadgydF4y2Ba三酰甘油(标签)物种代谢物的差异。的gydF4y2BaxgydF4y2Ba轴代表log10归一化峰面积的平均差异代谢物的绝食AML细胞与其他细胞。y轴表示饱和债券的数量。每个点代表一个独特的代谢物。gydF4y2Ba
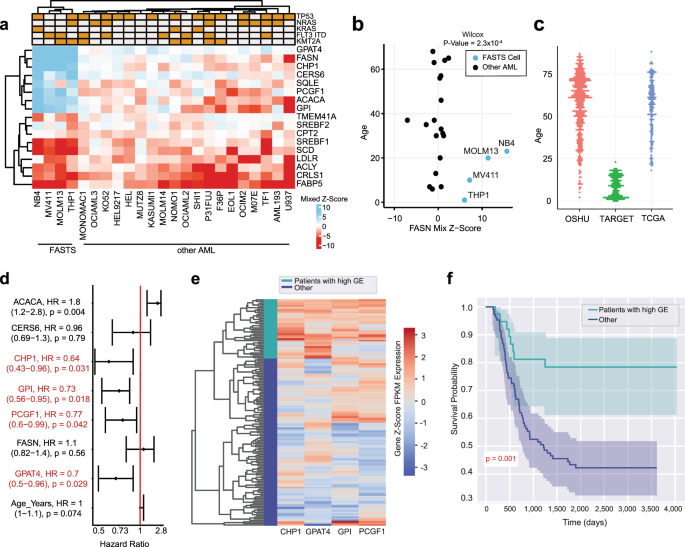
预后签名绝食基因gydF4y2Ba
探索禁食表型是否具有临床意义,我们将我们的结果与患者生存信息从公共数据库。利用基因从CCLE特征数据gydF4y2Ba69年gydF4y2Ba,我们没有发现任何损伤隔离绝食细胞从其他CD33 + AML细胞(无花果。gydF4y2Ba6gydF4y2Ba),所以没有提名驾驶禁食表型突变体内。相反,我们探索基因表达的变化是否与病人相关的结果。我们在绝食核心模块包括基因以及基因,并有很强的遗传相互作用gydF4y2BaACACA / FASNgydF4y2Ba在我们的屏幕(图。gydF4y2Ba6gydF4y2Ba)。选择一个适当的队列进行基因组分析,我们首先考虑患者年龄。尽管AML呈现在每一个十年的生活,患者从他绝食细胞系是派生的都是30岁以下(其他AML细胞来源的范围从6到68年;无花果。gydF4y2Ba6 bgydF4y2Ba)。记住这一点,我们从TARGET-AML探索数据gydF4y2Ba71年gydF4y2Ba项目,关注儿童癌症(无花果。gydF4y2Ba6摄氏度gydF4y2Ba)。使用目标数据,我们计算风险比率使用单变量Cox比例风险模型和连续感兴趣的基因的mRNA表达值作为独立的变量。我们观察到,4/7 FAS基因,gydF4y2BaGPAT4、CHP1 PCGF1gydF4y2Ba,gydF4y2Ba谷歌价格指数gydF4y2Ba,显示显著,负风险比率(人力资源)与肿瘤抑制签名(图一致。gydF4y2Ba6 dgydF4y2Ba),没有其他基因从我们组展示了一个消极的人力资源。实际上,当患者从目标群体的高表达gydF4y2BaGPAT4、CHP1 PCGF1gydF4y2Ba,gydF4y2Ba谷歌价格指数gydF4y2Ba(无花果。gydF4y2Ba6 egydF4y2Ba),我们观察到显著提高生存(gydF4y2BaPgydF4y2Ba值= 0.001,无花果。gydF4y2Ba6 fgydF4y2Ba)。这些发现并不复制gydF4y2BaGPAT4, CHP1gydF4y2Ba,gydF4y2Ba谷歌价格指数gydF4y2BaTCGA的gydF4y2Ba72年gydF4y2Ba或者也是OHSUgydF4y2Ba73年gydF4y2Ba肿瘤基因组学数据集,可能是因为他们旧样品同年龄组(Polycomb组单元gydF4y2BaPCGF1gydF4y2Ba观察到有一个人力资源< 1也是OHSU群组内补充图。gydF4y2Ba8gydF4y2Ba)。然而,年龄不是一般FAS集群中的相关基因的表达在肿瘤细胞系或样品(补充图。gydF4y2Ba8gydF4y2Ba)。gydF4y2Ba
一个gydF4y2Ba热图的混合gydF4y2BaZgydF4y2Ba分数的基因遗传相互作用网络。AML,常见的病变。gydF4y2BabgydF4y2Ba混合gydF4y2BaZgydF4y2Ba分数的FASN AML细胞株与年龄的病人细胞系是派生的。蓝色,绝食细胞。gydF4y2BacgydF4y2BaAML患者的年龄分布在三个公共肿瘤基因组学军团。红色表示OSHU,绿色表示目标,蓝色代表,TCGA。gydF4y2BadgydF4y2Ba风险比率(95%可信区间;单变量Cox比例风险测试)基因的表达(gydF4y2Ba一个gydF4y2Ba从目标),使用基因组学和生存数据。点代表人力资源价值显示在右边。95%可信区间范围为每个点显示在左边。基因显示红色代表有重大人力资源小于1的基因,用于分析(gydF4y2BaegydF4y2Ba)。gydF4y2BaegydF4y2Ba层次聚类的目标基因的表达情况,使用四种基因和消极的人力资源。绿色高表达集群。蓝色,其他。gydF4y2BafgydF4y2Bakaplan meier AML患者的生存分析目标,比较患者在高表达集群与他人。阴影区域表示曲线的95%置信区间。独立分析的患者的数量(gydF4y2BadgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba)= 145。gydF4y2Ba
讨论gydF4y2Ba
CRISPR屏幕癌症功能基因组学产生了深远的影响。而研究主要集中在重要的基因表型,仍有许多临床相关的生物,可以通过检查发现其他表型的遗传屏幕。我们建立一个方法,可以可靠地识别proliferation-suppressor从全基因组CRISPR敲除基因表型屏幕。在这里,我们提出一个系统研究的表型发表的1000多个屏幕gydF4y2Ba8gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba48gydF4y2Ba。gydF4y2Ba
proliferation-suppressor基因的活动本质上是上下文相关的,呈现全球分类困难。与上下文相关的重要基因,最强的信号是实现当比较淘汰赛表型与底层突变状态。例如,野生型和突变等位基因的肿瘤抑制的经典例子gydF4y2BaTP53gydF4y2Ba和gydF4y2BaPTENgydF4y2Ba存在于大量的细胞系,使相对容易在野生型PS行为背景的歧视,但大多数突变更罕见,减少统计力量。我们的基于模型的方法使PS表型的发现从null-phenotype淘汰赛中一个异类。使用这种方法,我们恢复COSMIC-annotated次数展示PS表型表达野生型等位基因时名义水平。gydF4y2Ba
同现的增殖抑制遵循模块化的生物学原理,与在同一通路作为增殖抑制基因在同一细胞系。我们观察PTEN通路background-specific假定的肿瘤抑制活性,P53规定,mTOR信号,染色质重塑等。同现网络还揭示了一个模块与glycerolipid生物合成相关,展品PS表型在AML细胞的一个子集。重新布线的分析在AML细胞脂质代谢coessentiality网络证实了这一发现,让我们定义脂肪酸合成/肿瘤抑制四AML细胞株(禁食)表型。遗传相互作用的调查,使用enCas12a多元淘汰赛平台,显示主要禁食和其他AML细胞之间的网络重新布线和显示强劲绝食细胞基因的相互作用gydF4y2BaGPAT4gydF4y2Ba,一个关键的酶处理的饱和脂肪酸,及其监管机构gydF4y2BaCHP1gydF4y2Ba。集体这些观测结果表明,在一些关键阈值附近的细胞为饱和脂肪酸承载能力,我们验证了用脂肪酸和生化反应的角度通过分析CCLE代谢组配置文件。gydF4y2Ba
确认临床体外表型的相关性是很困难的。没有明显的突变将绝食AML细胞与其他细胞,只有四个细胞系禁食表型,我们没有发现关联统计权力在一个公正的方式。然而,通过缩小我们的搜索微分的强烈打击网络分析,我们发现约的同龄群体的一个重要的生存优势gydF4y2BaGPAT4gydF4y2Ba和gydF4y2BaCHP1gydF4y2Ba超表达。这一发现指向一个肿瘤抑制签名PSG模块,尽管重要的进一步研究是必要的,以确定是否这一基因表达特征指出了一个类似的体内代谢重新布线和敏感的饱和脂肪。gydF4y2Ba
结合遗传、生化和临床支持的发现一个肿瘤抑制模块有几个含义。首先,它提供了一个临床的签名,需要进一步研究作为预后标记作为一个潜在的治疗目标。第二,它演示了微分网络分析的力量,特别是微分遗传相互作用网络,重新布线的解剖分子途径从模块化的表型。最后,它表明还有可能要学习从大型屏幕的数据驱动的分析数据,在低处的水果损伤/脆弱性关联。gydF4y2Ba
方法gydF4y2Ba
相关数据分析功能和包gydF4y2Ba
混合gydF4y2BaZgydF4y2Ba得分,分析使用评分指标,同现网络,进行生存分析4.0.4 R中的版本gydF4y2Ba74年gydF4y2Ba,gydF4y2Ba75年gydF4y2Ba。dPCC相关分析,包括实证计算,是在Python 3.8.2进行的gydF4y2Ba76年gydF4y2BaSciPy,使用包gydF4y2Ba77年gydF4y2Ba,NumPygydF4y2Ba78年gydF4y2Ba,MatplotlibgydF4y2Ba79年gydF4y2Ba和熊猫gydF4y2Ba80年gydF4y2Ba。gydF4y2Ba
R包tidyversegydF4y2Ba81年gydF4y2Ba,data.tablegydF4y2Ba82年gydF4y2Ba,knitrgydF4y2Ba83年gydF4y2Ba,gydF4y2Ba84年gydF4y2Ba,gydF4y2Ba85年gydF4y2Ba被用于图生成、数据操作和一般R函数;mixtoolsgydF4y2Ba86年gydF4y2Ba、交换gydF4y2Ba87年gydF4y2Ba,PRROCgydF4y2Ba88年gydF4y2Ba,gydF4y2Ba89年gydF4y2Ba被用于数据出现在模拟数据和评价;biomaRtgydF4y2Ba90年gydF4y2Ba,gydF4y2Ba91年gydF4y2Ba,org.Hs.eg.dbgydF4y2Ba92年gydF4y2Ba被用于集成数据类型;cowplotgydF4y2Ba93年gydF4y2Ba,ggbeeswarmgydF4y2Ba94年gydF4y2Ba,注释gydF4y2Ba95年gydF4y2Ba,RColorBrewergydF4y2Ba96年gydF4y2Ba,ComplexHeatmapgydF4y2Ba97年gydF4y2Ba,gplotsgydF4y2Ba98年gydF4y2Ba,ggpubrgydF4y2Ba99年gydF4y2Ba、网格gydF4y2Ba75年gydF4y2Ba,circlizegydF4y2BaOne hundred.gydF4y2Ba,ggthemesgydF4y2Ba101年gydF4y2Ba,ggExtragydF4y2Ba102年gydF4y2Ba,拼凑gydF4y2Ba103年gydF4y2Ba,ggplot2gydF4y2Ba104年gydF4y2Ba用于图美学和生成。R包生存gydF4y2Ba105年gydF4y2Ba,gydF4y2Ba106年gydF4y2Ba和survminergydF4y2Ba107年gydF4y2Ba被用于生存分析和图生成。分析相关kaplan meier和患者分层是在python版本3.8.5完成gydF4y2Ba108年gydF4y2Ba使用包熊猫gydF4y2Ba80年gydF4y2Ba,numpygydF4y2Ba78年gydF4y2Ba,scipygydF4y2Ba77年gydF4y2Ba统计函数和数据操作,seaborngydF4y2Ba109年gydF4y2Ba,情节gydF4y2Ba110年gydF4y2Ba,matplotlibgydF4y2Ba79年gydF4y2Ba图美学和代和生命线gydF4y2Ba111年gydF4y2Ba统计分析和图生成。gydF4y2Ba
enCas12a多元遗传分析屏幕是在R 4.0.0进行的gydF4y2Ba75年gydF4y2Ba和Python 3.8.3gydF4y2Ba112年gydF4y2Ba。可以在代码分析gydF4y2Bahttps://github.com/PeterDeWeirdt/FASTSgydF4y2Ba。R包tidyversegydF4y2Ba81年gydF4y2Ba和tidygraphgydF4y2Ba113年gydF4y2Ba用于数据操作和ggraph吗gydF4y2Ba114年gydF4y2Ba用于图形可视化。Python包SciPygydF4y2Ba77年gydF4y2Ba,NumPygydF4y2Ba78年gydF4y2Ba,MatplotlibgydF4y2Ba79年gydF4y2Ba、熊猫gydF4y2Ba80年gydF4y2Ba,statsmodelsgydF4y2Ba115年gydF4y2Ba,plotninegydF4y2Ba116年gydF4y2Ba被用于分析和可视化。定制包因gydF4y2Ba117年gydF4y2Ba是用来计算分数和gpplot基因交互作用gydF4y2Ba118年gydF4y2Ba用于生成点密度图。gydF4y2Ba
处理DepMap屏幕和CCLE基因组数据gydF4y2Ba
生读计数数据和指导rna从DepMap下载的地图数据库(gydF4y2Bawww.depmap.orggydF4y2Ba)gydF4y2Ba10gydF4y2Ba,gydF4y2Ba48gydF4y2Ba和项目评分数据库(gydF4y2Bahttps://depmap.sanger.ac.uk/gydF4y2Ba)gydF4y2Ba13gydF4y2Ba。2020年第四季度Avana数据版本gydF4y2Ba49gydF4y2Ba是用于分析。为了避免遗传交互作用,我们丢弃sgRNAs针对多个蛋白编码基因注解为公共或更新等待共识编码序列(ccd,释放22)gydF4y2Ba119年gydF4y2Ba。指导RNA的基因名称的地图Avana和项目得分是使用人类基因信息从ncbi ftp获得更新。然后,读为每个复制是通过CRISPRcleanR计数数据gydF4y2Ba120年gydF4y2Ba与位置信息的sgRNAs Avana基于GENCODE CRISPR库gydF4y2Ba121年gydF4y2Ba从而正确的损耗效应引起的放大。这个校正后,每个导游的日志gydF4y2Ba2gydF4y2Ba褶皱变化计算。对于项目评分数据,我们只用基因位置信息的肯塔基州图书馆v1.0 CRISPRcleanR建成的。规范化TPM RNA-seq数据,人类基因组数据和突变CCLE注释gydF4y2Ba69年gydF4y2Ba细胞也从DepMap下载。运用基因id RNA-seq数据转化为基因符号使用交叉引用从Emsembl下载BiomartgydF4y2Ba122年gydF4y2Ba。gydF4y2Ba
混合gydF4y2BaZ -gydF4y2Ba评分标准gydF4y2Ba
混合gydF4y2BaZ -gydF4y2Ba分数指标生成使用R版本4.0.4基础统计软件包gydF4y2Ba75年gydF4y2Ba和mixtoolsgydF4y2Ba86年gydF4y2BanormalmixEM函数。计算混合gydF4y2BaZ -gydF4y2Ba得分,个别指导日志gydF4y2Ba2gydF4y2Ba褶皱的变化对于每个细胞株是通过normalmixEM函数的默认设置以适应两个正态分布的区别。808细胞系通过这种分析,805细胞系能够收敛,1000次迭代后两个正态分布的区别。更高的计算平均值和标准偏差(积极)分布记录。随着未修正的原始基因日志gydF4y2Ba2gydF4y2Ba褶皱的变化,是用来计算相应的混合gydF4y2BaZgydF4y2Ba分数。原始和混合gydF4y2BaZ -gydF4y2Ba分方程如下:gydF4y2Ba
在哪里gydF4y2BaxgydF4y2Ba原始基因日志吗gydF4y2Ba2gydF4y2Ba褶皱的变化,gydF4y2BaμgydF4y2Ba高gydF4y2Ba更积极的拟合分布的平均值,然后呢gydF4y2BaσgydF4y2Ba高gydF4y2Ba是更积极的拟合分布的标准偏差。这个指标是计算DepMap 2020第四季度gydF4y2Ba49gydF4y2Ba屏幕设置,桑格DepMapgydF4y2Ba13gydF4y2Ba屏幕上设置补充图。gydF4y2Ba3gydF4y2Ba。可视化的混合gydF4y2BaZgydF4y2Ba分数为广泛的和桑格DepMap屏幕集可以看到泡菜gydF4y2Ba123年gydF4y2Ba数据库:gydF4y2Bahttps://pickles.hart-lab.org/。gydF4y2Ba
fitness-scoring指标的比较gydF4y2Ba
下面描述了检测算法的比较分析中补充图。gydF4y2Ba1克ydF4y2Ba。杰克gydF4y2Ba43gydF4y2Ba和百吉饼gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba124年gydF4y2Ba从相应的GitHub,软件下载官方站点分布:gydF4y2Bahttps://github.com/felicityallen/JACKSgydF4y2Ba,gydF4y2Bahttps://github.com/hart-lab/bagelgydF4y2Ba。我们跑千斤顶和面包圈DepMap 2020 q4版本的原始叠化数据gydF4y2Ba49gydF4y2Ba基因指导地图,复制信息。我们获得DepMap 2020第四季度从“dependency_score谷神星的分数。csv”从DepMap下载存托。排名是执行/屏幕和基于日志的意思gydF4y2Ba2gydF4y2Ba叠化每个基因值。gydF4y2Ba
我们使用了癌症基因从宇宙普查(公司治理文化)列表gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba比较健康的方法可以检测proliferation-suppressor活动。肿瘤抑制基因(次数)从公司治理文化表示一组基因的著名的增殖抑制。我们把公司治理文化基因在两个基因集,在癌症基因与肿瘤抑制作用代表真阳性proliferation-suppressor观察,与任何致癌基因和基因代表假阳性癌症的作用。此外,我们添加了引用不必要的基因gydF4y2Ba7gydF4y2Ba,gydF4y2Ba47gydF4y2Ba假阳性的列表,这些基因是不会展示任何表型。与这些编译列表,我们的健康评估每个指标的分数,看看哪些指标最好单独的真和假阳性基因列表。R包PRROC用于fitness-scoring评估gydF4y2Ba88年gydF4y2Ba,gydF4y2Ba89年gydF4y2Ba。gydF4y2Ba
直接proliferation-suppressor比较Avana和桑格屏幕数据集gydF4y2Ba
的CRISPRcleanRgydF4y2Ba120年gydF4y2Ba纠正叠化桑格屏幕设置gydF4y2Ba13gydF4y2Ba通过相同的管道被用于计算混合吗gydF4y2BaZ -gydF4y2Ba评分指标。混合的质量分析gydF4y2BaZgydF4y2Ba评分标准中描述的两个数据集被使用相同的基因集“健康评分指标的比较”一节。这种分析只局限于重叠的细胞系,186年,在这两个数据集。细胞系匹配使用护照的细胞模型数据库gydF4y2Ba125年gydF4y2Ba。gydF4y2Ba
健身引入增强PSG淘汰赛,相比相对较弱的严重缺陷的重要基因敲除,常常导致无法在更短的实验检测。在示例F5细胞系(无花果。gydF4y2Ba1gydF4y2Ba),2.5倍的变化在21天的时间对应于一个健身增加快速增长的细胞,只有~ 12%或倍增时间减少从24 - 21 h。在一个14天的实验,这增加增殖率会导致观察log-fold只改变~ 1.7,在预期的噪音从没有敲除基因表型。这是详细解释如下:gydF4y2Ba
理论叠化和增长率量化:评估假设的差异proliferation-suppressor fitness-scoring指标根据标准抽样的时候屏幕收集来自桑格和Avana数据库gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba48gydF4y2Ba,我们计算理论的细胞群野生型和报废proliferation-suppressor细胞株的差异。下面的情商。gydF4y2Ba2gydF4y2Ba)可以用来计算细胞群基于每天加倍率:gydF4y2Ba
在这个公式,gydF4y2BaXgydF4y2BafgydF4y2Ba是最后的人口数量的细胞,gydF4y2BaXgydF4y2Ba我gydF4y2Ba初始种群的细胞,gydF4y2BakgydF4y2Ba是细胞的倍增时间(天),然后呢gydF4y2BatgydF4y2Ba是在天。为了比较细胞,我们可以假设这些公式是一致的与野生型细胞和报废proliferation-suppressor细胞。,报废proliferation-suppressor细胞的假设是,这些细胞将增长更快,因此相对于野生型条件gydF4y2BakgydF4y2BapsgydF4y2Ba>gydF4y2BakgydF4y2BawtgydF4y2Ba,在那里gydF4y2BakgydF4y2BapsgydF4y2Ba的增长率proliferation-suppressor报废电池,gydF4y2BakgydF4y2BawtgydF4y2Ba是野生型细胞的生长速率。这两个独立的增长率相关:gydF4y2Ba
ΔgydF4y2BakgydF4y2Ba代表增长率的变化导致的基因敲除和被认为是积极的。野生型和proliferation-suppressor细胞的生长速率方程是:gydF4y2Ba
然后我们解决ΔgydF4y2BakgydF4y2Ba,日志gydF4y2Ba2gydF4y2Ba(XgydF4y2BapsgydF4y2Ba/ XgydF4y2BawtgydF4y2Ba)gydF4y2Ba作为日志gydF4y2Ba2gydF4y2Ba(FC),代表了叠化细胞种群在时间的区别gydF4y2BatgydF4y2Ba:gydF4y2Ba
一位代表日志gydF4y2Ba2gydF4y2Ba2.5 (FC),代表了一个相当大的涨幅从报废proliferation-suppressor来健身,和gydF4y2BatgydF4y2Ba= 21天,代表Avana屏幕的时间采样,我们计算ΔgydF4y2BakgydF4y2Ba:gydF4y2Ba
使用计算ΔgydF4y2BakgydF4y2Ba在0.12中,我们可以计算假设的日志gydF4y2Ba2gydF4y2Ba(FC),预计将在gydF4y2BatgydF4y2Ba代表时间= 14天,桑格屏幕取样:gydF4y2Ba
由此产生的理论证明Δ测量gydF4y2BakgydF4y2Ba两个样本之间可以是相同的,然而,时间的样本将会影响两者之间的比率测量细胞群。综上所述,这表明样品在较短的时间点将演示小野生型量化人口规模差异和proliferation-suppressor报废电池相比,在时间点采集的样本。gydF4y2Ba
Proliferation-suppressor共生网络gydF4y2Ba
基于FDR-corrected共生网络发达gydF4y2BaPgydF4y2Ba值从Fisher精确测试的所有gene-by-gene比较确定为一种增殖抑制不止一次(共584个基因)。并行处理,确切概率法,Benjamini &业务罗斯福gydF4y2BaPgydF4y2Ba使用R统计软件包基础值进行调整gydF4y2Ba75年gydF4y2Ba。图gydF4y2Ba2gydF4y2Ba创建的热图。2function from the R gplots98年gydF4y2Ba包,系统树图通过基地RgydF4y2Ba75年gydF4y2Ba欧几里得距离的函数,并完成凝聚方法聚类基因对之间的确切概率测试成绩。小的热图显示在无花果。gydF4y2Ba2摄氏度gydF4y2Ba是使用R ComplexHeatmap图书馆吗gydF4y2Ba97年gydF4y2Ba。网络可视化使用Cytoscape完工gydF4y2Ba126年gydF4y2Ba。gydF4y2Ba
创建网络遵循相应的步骤;(1)识别所有proliferation-suppressor观察罗斯福阈值(10%gydF4y2BaZgydF4y2Ba> = 3.83)。(2)筛选基因proliferation-suppressor观察发生的至少2次以上,选择总共584 18111个基因的全部可用的基因(3.2%);(3)创建一个二进制(1 =增殖抑制,0 =不扩散抑制器)矩阵所有584个基因的细胞系;(4)进行确切概率法的每一个可能的2×2列联表的584所选基因(gydF4y2BangydF4y2Ba= 170236测试);和(5)相应的调整gydF4y2BaPgydF4y2Ba值罗斯福值,使用截止0.001(0.1%罗斯福)定义边缘。通过评估基因边缘Fisher精确测试,我们观察到的基因联系是基于两个基因之间的共生的相对比例。gydF4y2Ba
Proliferation-suppressor网络浓缩gydF4y2Ba
测试网络浓缩观察到的边缘(补充图。gydF4y2Ba4gydF4y2Ba),我们花了10000的随机抽样462(总边数同现网络)基因对从170236年可用的所有基因对确切概率法集。我们将每个样本进行比较,看它们基因的频率对HumanNet内观察到有一些互动gydF4y2Ba61年gydF4y2Ba,不包括基因交互观察仅仅coessentiality网络组件gydF4y2Ba21gydF4y2Ba(从相同的数据生成的),以防止循环。此外,我们比较我们选择的混合gydF4y2BaZ -gydF4y2Ba分数对其他各种截止gydF4y2BaZgydF4y2Ba分数达标,确保我们观察到适当的边缘表示从HumanNet(补充图。gydF4y2Ba4 bgydF4y2Ba)。网络是使用相同的管道和确切概率法组被切断gydF4y2BaZgydF4y2Ba分数达标3至8 0.2增量。gydF4y2Ba
微分皮尔逊相关系数分析gydF4y2Ba
微分皮尔逊相关系数(dPCC)分析是识别基因进行健身区分AML细胞和其他细胞(图gydF4y2Ba3gydF4y2Ba)。最初的相关性(图。gydF4y2Ba3gydF4y2BaFAS集群基因的),gydF4y2BaPCGF1gydF4y2Ba,gydF4y2BaCERS6gydF4y2Ba,gydF4y2Ba谷歌价格指数gydF4y2Ba,gydF4y2BaFASNgydF4y2Ba,gydF4y2BaCHP1gydF4y2Ba,gydF4y2BaGPAT4gydF4y2Ba,gydF4y2BaACACAgydF4y2Ba计算R版本4.0.4基地统计包吗gydF4y2Ba75年gydF4y2Ba和策划ggplot2gydF4y2Ba104年gydF4y2Ba。gydF4y2Ba
这个观察后,后续dPCC分析进行绝食集群基因评估dPCC质量。细胞系与低质量(科恩的屏幕gydF4y2BaDgydF4y2Ba< 2.5或召回已知core-essential基因< 60%)被排除在外,留下659细胞系。这个过滤步骤后,两个gene-by-gene关联矩阵计算。第一个相关矩阵计算所有gene-by-gene对只有可用的AML细胞株(gydF4y2BangydF4y2Ba= 17)。第二个矩阵计算所有gene-by-gene对其余642细胞系。因此dPCC矩阵AML相关矩阵减去non-AML相关矩阵。gydF4y2Ba
每一对基因都有一个独特的混合的联合分布gydF4y2BaZgydF4y2Ba分数;因此,每个dPCC分数的意义必须单独计算。要做到这一点,我们生成零分布dPCC每一对基因。我们将随机选择没有更换17细胞系(匹配gydF4y2BangydF4y2BaAML细胞),计算所有gene-by-gene在其余两两相关性在这个选择和,和dPCC计算。我们重复这个采样和计算来生成唯一的零分布1000倍dPCC每一对基因和一个适当的计算gydF4y2BaPgydF4y2Ba值观察dPCC上面(right-tailed积极dPCC,离开尾随负dPCC)。gydF4y2Ba
显示明显的基因敲除基因表型(混合Z | | > 5)和(dPCC AML-specific变化相关gydF4y2BaPgydF4y2Ba< 0.001)与基因连接集团同现的集群(gydF4y2BaCHP1、GPAT4 ACACA、FASN GPI、CERS6 PCGF1gydF4y2Ba)被选为进一步分析(无花果。gydF4y2Ba3 egydF4y2Ba)。图gydF4y2Ba3 egydF4y2Ba使用R ComplexHeatmap图书馆吗gydF4y2Ba97年gydF4y2Ba。图gydF4y2Ba3 c, dgydF4y2Ba情节是Matplotlib使用Python包gydF4y2Ba79年gydF4y2Ba。gydF4y2Ba
细胞培养的遗传屏幕gydF4y2Ba
MOLM13与Cas12a-mediated NOMO1细胞筛选基因交互图书馆广泛研究所获得的癌症细胞系的百科全书。gydF4y2Ba
细胞系都经常进行支原体污染的检测和维护没有抗生素除了在屏幕,当媒体补充青霉素和链霉素为1%。细胞系都保存在一个37°C,湿度孵化器与5.0%二氧化碳和维护在对数生长期增长使每2−3天。下面的媒体环境和剂量的聚凝胺,嘌呤霉素,和杀稻瘟菌素,分别被使用:gydF4y2Ba
的边后卫MOLM13: RPMI + 10%;8μg毫升gydF4y2Ba−1gydF4y2Ba;4μg毫升gydF4y2Ba−1gydF4y2Ba;8μg毫升gydF4y2Ba−1gydF4y2Ba
的边后卫NOMO1: RPMI + 10%;8μg毫升gydF4y2Ba−1gydF4y2Ba;1μg毫升gydF4y2Ba−1gydF4y2Ba;8μg毫升gydF4y2Ba−1gydF4y2Ba
汇集屏幕gydF4y2Ba
稳定细胞系表达enCas12a (pRDA_174 Addgene 136476)被转导指南克隆到pRDA_052向量(Addgene 136474)在两个细胞培养复制低莫伊(~ 0.5)。与足够的细胞转导进行实现的表示至少有750细胞/指导构建/复制,考虑一个30 - 50%转导效率。整个屏幕,细胞分裂的密度保持表示至少1000细胞/指导构造,在每个通道和细胞计数被监控的增长。嘌呤霉素选择添加2天post-transduction并维持5天。转导后14天,21天,细胞被离心分离颗粒状,在PBS resuspended,立即冻结基因组DNA隔离。gydF4y2Ba
基因组DNA隔离和PCRgydF4y2Ba
基因组DNA (gDNA)孤立使用翠鸟Flex与Mag-Bind净化系统gydF4y2Ba®gydF4y2Ba血液和组织DNA HDQ工具包(ωBio-Tek # M6399-01)按照制造商的指示。gDNA浓度被量子位量化。PCR扩增,gDNA分为100μL反应,这样每个最多10μg gDNA。/ 96孔板,主结构由144μL 50×钛Taq DNA聚合酶(豆类),960μL 10 x钛Taq缓冲区,768μL核苷酸(股票为2.5毫米)提供的酶,48μL P5交错混合底漆(股票100μM浓度),480μL DMSO和1.44毫升的水。每个由50μL gDNA +水,40μL PCR大师混合,和10μL独特的条形码P7底漆(股票5μM浓度)。gydF4y2Ba
PCR循环条件:最初在95°C 1分钟;其次是30年代在94°C, 30年代在53°C, 30年代在72°C, 28周期;最后10分钟在72°C扩展。在集成PCR引物合成DNA技术(IDT)。PCR产物纯化了Agencourt AMPure XP撒珠子按照制造商的指示(贝克曼库尔特,A63880)。gydF4y2Ba
样本测序HiSeq2500快速跑flowcell (Illumina公司)和一个自定义序列的引物:5 ' -CTTGTGGAAAGGACGAAACACCGGTAATTTCTACTCTTGTAGAT。第一个核苷酸测序引物是第一个核苷酸的指导RNA,它将包含一个混合的四个核苷酸,因此交错引物不需要使用这种方法时保持多样性。读取被对齐到一个参考文件计算所有可能的指导rna出现在图书馆。阅读被分配给一个条件(比如,一个PCR板)的基础上8元索引包含在P7底漆。gydF4y2Ba
得分基因相互作用gydF4y2Ba
分数遗传交互我们使用一个自定义python包,因知识gydF4y2Ba117年gydF4y2Ba指数,可以在python包。我们使用log-fold变化(利物浦)作为输入得分管道。我们定义gydF4y2BaygydF4y2BaijgydF4y2Ba观察到利物浦的一双指南gydF4y2Ba我gydF4y2Ba,gydF4y2BajgydF4y2Ba,gydF4y2Ba\ (\ widehat {{y} _ {ij}} \)gydF4y2Ba这双的利物浦。然后我们计算残余gydF4y2Ba\ ({y} _ {ij} - \ widehat {{y} _ {ij}} \)gydF4y2Ba生成一个互动得分。定义预期的利物浦,gydF4y2Ba\ (\ widehat {{y} _ {ij}} \)gydF4y2Ba我们适合每个指南的线性回归,gydF4y2Ba我gydF4y2Ba,说gydF4y2Ba
在哪里gydF4y2BaxgydF4y2Ba每个指导单独的利物浦吗gydF4y2Ba米gydF4y2Ba我gydF4y2Ba和gydF4y2BabgydF4y2Ba我gydF4y2Ba斜率和截距适合指导吗gydF4y2Ba我gydF4y2Ba(补充图。gydF4y2Ba6 bgydF4y2Ba)。我们将gydF4y2Ba我gydF4y2Ba作为锚指南及其对目标指导。然后,我们gydF4y2BaZ -gydF4y2Ba分数残差在每个锚指南。这种方法类似于一个由Horlbeck et al。gydF4y2Ba33gydF4y2Ba。gydF4y2Ba
聚合互动得分在基因层面上,我们总和gydF4y2BaZgydF4y2Ba得到残差,gydF4y2BazgydF4y2BaijgydF4y2Ba,构造gydF4y2Ba我,我gydF4y2Ba针对该基因对gydF4y2Ba我,我gydF4y2Ba,修复gydF4y2Ba我gydF4y2Ba锚基因,除以根号构造目标的数量gydF4y2Ba我,我gydF4y2Ba。我们重复这个计算,修正gydF4y2BaJgydF4y2Ba锚的基因。我们为这两种取向和分数总和除以gydF4y2Ba\ \√{2}\)gydF4y2Ba能够到达gydF4y2BaZgydF4y2Ba分数。gydF4y2Ba
细胞培养对脂肪酸的反应gydF4y2Ba
人类癌症细胞系使用MD安德森获得如下:EOL1, MONOMAC1, NB4, OCIAML3 (DSMZ # acc - 386 # acc - 252 # acc - 207 # acc - 582);MOLM13和NOMO1(费舍尔,# NC0442994 # NC1515509);MV411(写明ATCC # crl - 9591)。身份确认收到,之前的实验STR打字(MDACC细胞系核心特征)。没有支原体确认每月(Invivogen # rep-pt1)。细胞系都生长在37°C公司5%gydF4y2Ba2gydF4y2Ba在低依恋烧瓶(格林尼)和维护在小于1毫升M细胞gydF4y2Ba−1gydF4y2Ba。但一行都培养与25毫米玫瑰(rpmi - 1640(σ# R5886)补充10%的边后卫(σ# F0926), 2毫米Glutamax (Gibco # 35050061), 1毫米丙酮酸钠(Gibco # 11360070), 10000毫升gydF4y2Ba−1gydF4y2Ba青霉素(Gibco # 15140122), 10毫克毫升gydF4y2Ba−1gydF4y2Ba链霉素(Gibco # 15140122),和100年µg毫升gydF4y2Ba−1gydF4y2BaNormocin (Invivogen # ANTNR2)。完全培养基与0.1毫米不必要的另外补充氨基酸对MONOMAC1 (Gibco # 11140050)。gydF4y2Ba
脂肪酸的解决方案gydF4y2Ba
脂肪酸的化学物质被购买的σ(圣路易斯,密苏里州)。根据罗等解决方案准备gydF4y2Ba。gydF4y2Ba127年gydF4y2Ba遵循最佳实践gydF4y2Ba128年gydF4y2Ba。脂肪酸股票的解决方案准备为硬脂酸在100%乙醇50毫米或200毫米。脂肪无酸的牛血清白蛋白(FAF-BSA)溶解在组织文化品位在1.5毫米(pyrogen-free)水10% (w / v),使用了0.1µm PES真空过滤单元(康宁)和整除存储−20°C。乙醇股票的解决方案被稀释到4毫米FAF-BSA(摩尔比2.7:1),在室温下混合轻轻2 h促进接合。车辆控制被稀释100%乙醇准备FAF-BSA匹配4毫米硬脂酸的乙醇浓度的解决方案。车辆或4毫米的解决方案被整除,储存在−80°C长达3个月。解冻后,整除与完全培养基稀释1:10 400µM,储存在4°C和1周内使用。gydF4y2Ba
细胞凋亡检测gydF4y2Ba
细胞被播种在500年之前24小时治疗µL完全培养基24-well低依恋板块(格林尼)250000个细胞gydF4y2Ba−1gydF4y2Ba。一式四份井收到500µL足总工作方案(400µM)或车辆(BSA + EtOH)。细胞治疗在200µM 48小时。治疗细胞转移到96孔板和介质离心后丢弃在500×gydF4y2BaggydF4y2Ba为5分钟。细胞被洗一次与1000年µL D-PBS(σ# D8537)。接下来,300年细胞resuspendedµL绑定缓冲区包含annexin-FITC (BD生物科学# BD556547)和propidium碘(英杰公司# P3566)根据制造商的协议(BD生物科学)和转移到浅96 - v形底板块(康宁)。染色后在黑暗中在室温下15分钟,细胞被洗一次在100年与300µL绑定缓冲最后resuspendedµL绑定缓冲。无污点的和single-stain控制准备线每一个细胞都在一个单独的盘子。盖茨进行调整,这样清白的汗衫低于每个阈值的99%。看到补充图。gydF4y2Ba9gydF4y2Ba完整的控制策略。流式细胞仪数据采集使用FACSCelesta分析仪配备一个autosampler (BD生物科学)和分析使用FlowJo 10.5.3。结果显示代表三个独立实验的顺序每个细胞株的段落。统计测试图所示。gydF4y2Ba5 b, cgydF4y2Ba是片面的孤gydF4y2BatgydF4y2Ba检测细胞凋亡的百分比和R统计计算使用基地gydF4y2Ba75年gydF4y2Ba功能。gydF4y2Ba
代谢组学分析gydF4y2Ba
本节描述图中使用的方法。gydF4y2Ba5 dgydF4y2Ba和补充图。gydF4y2Ba7gydF4y2Ba。代谢组学数据获得补充表gydF4y2Ba1克ydF4y2Ba李等人。gydF4y2Ba70年gydF4y2Ba。进行分析,归一化数据(“1-clean数据”)和变异系数为每个代谢物(“1-CV”)使用。规范化数据被过滤了,仅仅选择AML细胞出现在Avana 2020第四季度gydF4y2Ba49gydF4y2Ba屏幕设置。过滤后,中位数的物种现在的拍摄,按测量是否分组从一个绝食AML或其他AML细胞株。的差异值,代表日志比率,是每个代谢物。代谢物的差异中位数小于变异系数从故事情节被省略了。酰基组和不饱和键的数量获得直接从所提供的命名法。gydF4y2Ba
AML患者生存分析gydF4y2Ba
本节描述图中使用的方法。gydF4y2Ba6gydF4y2Ba和补充无花果。gydF4y2Ba8gydF4y2Ba和gydF4y2Ba10gydF4y2Ba。基因选择的分析都显示有一个互动gydF4y2BaACACAgydF4y2Ba在无花果。gydF4y2Ba4 hgydF4y2Ba和gydF4y2BaFASNgydF4y2Ba。基因注释在无花果。gydF4y2Ba6gydF4y2Ba热图包括任何nonsilent突变,人类损失gydF4y2BaTP53gydF4y2Ba和gydF4y2BaKMT2AgydF4y2Ba,人类获得的gydF4y2Ba喀斯特gydF4y2Ba,gydF4y2Ba国家管制当局方面gydF4y2Ba,gydF4y2BaFLT3gydF4y2Ba。gydF4y2Ba- itdgydF4y2Ba注释中gydF4y2BaFLT3gydF4y2Ba注释行酒吧。来自CCLE变异注释gydF4y2Ba69年gydF4y2Ba,人类来自cBioPortalgydF4y2Ba129年gydF4y2Ba,gydF4y2Ba130年gydF4y2Ba数据库,gydF4y2BaFLT-ITDgydF4y2Ba注释来自DSMZ目录gydF4y2Ba131年gydF4y2Ba。gydF4y2Ba
TARGET-AMLgydF4y2Ba71年gydF4y2Ba数据包括年龄、基因表达(HTseq FPKM UQ),时间事件,TCGA和生存事件结果和gydF4y2Ba72年gydF4y2Ba病人年龄和基因表达是直接从齐娜下载gydF4y2Ba132年gydF4y2Ba数据库。这也是OHSU BeatAMLgydF4y2Ba73年gydF4y2Ba年龄数据直接从Vizome下载数据库,和基因表达数据被从原来的出版。岁patient-derived细胞系获得Cellosaurus数据库gydF4y2Ba133年gydF4y2Ba。风险率计算从Cox比例风险模型使用R生存gydF4y2Ba105年gydF4y2Ba,gydF4y2Ba106年gydF4y2Ba包中。病人完成聚类分层聚类函数从scipy包gydF4y2Ba77年gydF4y2Ba,使用欧几里德集群和完整的链接设置。这个输出的热图TARGET-AML患者(无花果。gydF4y2Ba6 egydF4y2Ba从seaborn)创建使用功能gydF4y2Ba109年gydF4y2Ba包中。我们确定了包含最高的病人集群整体的表达gydF4y2BaCHP1gydF4y2Ba,gydF4y2BaGPAT4gydF4y2Ba,gydF4y2Ba谷歌价格指数gydF4y2Ba,gydF4y2BaPCGF1gydF4y2Ba从使用fcluster函数的热图scipygydF4y2Ba77年gydF4y2Ba。图gydF4y2Ba6 fgydF4y2Ba演示结果生存比较两组病人和创建函数的生命线gydF4y2Ba111年gydF4y2Ba包,具体来说,KaplanMeierFitter(α= 0.05,违约)函数kaplan meier曲线,和gydF4y2BaPgydF4y2Ba值反映了计算生存率较两条曲线。gydF4y2Ba
PgydF4y2Ba值与Schoenfeld测试计算由survminer内部包。目标数据分析,病人表达谱从原发肿瘤样本,选择过滤复发病人的样本(42例)。患者分层进行了基于分层患者团体到较低的基因表达(患者基因表达在第75个百分位,gydF4y2BangydF4y2Ba= 108个独立的患者),和更高的基因表达(75以上的患者,gydF4y2BangydF4y2Ba= 37独立病人)。计算风险比率对所有测试目标群体内基因通过Cox比例风险假设(补充图。gydF4y2Ba10gydF4y2Ba)没有拒绝Schoenfeld测试零假设。gydF4y2Ba
报告总结gydF4y2Ba
进一步研究信息设计是可用的gydF4y2Ba自然研究报告摘要gydF4y2Ba与这篇文章有关。gydF4y2Ba
数据可用性gydF4y2Ba
遗传相互作用(enCas12a)与图有关的数据。gydF4y2Ba4 gydF4y2Ba和补充图。gydF4y2Ba6gydF4y2Ba可以发现在gydF4y2Bahttps://github.com/PeterDeWeirdt/FASTSgydF4y2Ba。图gydF4y2Ba5 cgydF4y2Ba数据可以在源数据文件中找到。CytoscapegydF4y2Ba126年gydF4y2Ba网络文件PSG的网络(图。gydF4y2Ba2gydF4y2Ba和补充图。gydF4y2Ba4 gydF4y2Ba可以找到)gydF4y2Bahttps://doi.org/10.6084/m9.figshare.16746052.v1gydF4y2Ba。数据的相关数据,包括基因混合z分数评价,费舍尔边计算,dPCC评分指标,屏幕和其他指标的比较,可以发现gydF4y2Bahttps://doi.org/10.6084/m9.figshare.16746040.v1gydF4y2Ba。外部数据用于这项研究包括筛选来自Avana 2020年第四季度gydF4y2Ba10gydF4y2Ba,gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba释放,CCLEgydF4y2Ba69年gydF4y2Ba基因表达、突变和人类基因组数据,可以发现gydF4y2Bawww.depmap.orggydF4y2Ba;使用筛选数据从项目得分gydF4y2Ba13gydF4y2Ba可以发现gydF4y2Bahttps://depmap.sanger.ac.uk/gydF4y2Ba;细胞模型的护照gydF4y2Ba125年gydF4y2Ba数据被用于筛查数据对比,可以发现gydF4y2Bahttps://cellmodelpassports.sanger.ac.uk/gydF4y2Ba;癌症基因的人口普查gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba用于定义癌基因和肿瘤抑制,可以发现gydF4y2Bahttps://cancer.sanger.ac.uk/censusgydF4y2Ba;绝对的人类基因值从细胞系获得cBioPortal数据库gydF4y2Ba130年gydF4y2Ba在gydF4y2Bahttps://www.cbioportal.org/gydF4y2Ba;HumanNetgydF4y2Ba61年gydF4y2Ba数据用于网络比较可以发现gydF4y2Bahttps://www.inetbio.org/humannet/gydF4y2Ba;齐娜数据库gydF4y2Ba132年gydF4y2Ba用于获取特定的数据相关,TCGA LAML,目标AML, BeatAML数据集和可以找到吗gydF4y2Bahttps://xenabrowser.net/gydF4y2Ba;和额外的BeatAML分析直接取自Tyner et al。gydF4y2Ba73年gydF4y2Ba出版。结果发表在这里是部分基于数据生成的治疗研究适用于生成有效的治疗方法(目标)倡议,NCI phs000218、管理。可用的数据用于分析在研究dbGaP加入:gydF4y2Baphs000465.v19.p8gydF4y2Ba。目标可以找到的信息gydF4y2Bahttp://ocg.cancer.gov/programs/targetgydF4y2Ba。gydF4y2Ba源数据gydF4y2Ba本文提供的。gydF4y2Ba
代码的可用性gydF4y2Ba
遗传相互作用(enCas12a)代码笔记本相关图。gydF4y2Ba4 gydF4y2Ba和补充图。gydF4y2Ba6gydF4y2Ba可以发现在gydF4y2Bahttps://github.com/PeterDeWeirdt/FASTSgydF4y2Ba。代码相关的所有数据除了无花果。gydF4y2Ba4 gydF4y2Ba补充图。gydF4y2Ba6gydF4y2Ba,gydF4y2Ba9gydF4y2Ba可在:gydF4y2Bahttps://doi.org/10.6084/m9.figshare.16786063gydF4y2Ba。额外的分析代码(主要是共生网络,喜忧参半gydF4y2BaZgydF4y2Ba评分指标,dPCC相关性和临床分析)是可用的gydF4y2Bahttps://doi.org/10.6084/m9.figshare.16786078.v1gydF4y2Ba。gydF4y2Ba
引用gydF4y2Ba
Jinek, m . et al。可编程dual-RNA-guided DNA核酸内切酶在自适应细菌的免疫力。gydF4y2Ba科学gydF4y2Ba337年gydF4y2Ba,816 - 821 (2012)。gydF4y2Ba
马里、p等。通过Cas9 RNA-guided人类基因组工程。gydF4y2Ba科学gydF4y2Ba339年gydF4y2Ba,823 - 826 (2013)。gydF4y2Ba
马里、P。,Esvelt, K. M. & Church, G. M. Cas9 as a versatile tool for engineering biology.Nat方法。gydF4y2Ba10gydF4y2Ba,957 - 963 (2013)。gydF4y2Ba
琮、l . et al .多路复用基因组工程使用CRISPR / Cas系统。gydF4y2Ba科学gydF4y2Ba339年gydF4y2Ba,819 - 823 (2013)。gydF4y2Ba
城东O。,年代一个nj一个n一个,N. E. & Zhang, F. High-throughput functional genomics using CRISPR-Cas9.Nat,启麝猫。gydF4y2Ba16gydF4y2Ba,299 - 311 (2015)。gydF4y2Ba
王,T。,Wei, J. J., Sabatini, D. M. & Lander, E. S. Genetic screens in human cells using the CRISPR-Cas9 system.科学gydF4y2Ba343年gydF4y2Ba,80 - 84 (2014)。gydF4y2Ba
哈特,t . et al .高分辨率CRISPR屏幕显示健康基因和genotype-specific癌症负债。gydF4y2Ba细胞gydF4y2Ba163年gydF4y2Ba,1515 - 1526 (2015)。gydF4y2Ba
王,t . et al .识别和描述基本基因在人类基因组中。gydF4y2Ba科学gydF4y2Ba350年gydF4y2Ba,1096 - 1101 (2015)。gydF4y2Ba
Aguirre, a . j . et al .基因组拷贝数规定gene-independent CRISPR / Cas9瞄准细胞反应。gydF4y2Ba癌症。gydF4y2Ba6gydF4y2Ba,914 - 929 (2016)。gydF4y2Ba
Meyers, r . m . et al .拷贝数的计算校正效果提高特异性CRISPR-Cas9重要性屏幕癌细胞。gydF4y2BaNat,麝猫。gydF4y2Ba49gydF4y2Ba,1779 - 1784 (2017)。gydF4y2Ba
Tsherniak, a . et al .定义一个癌症地图的依赖。gydF4y2Ba细胞gydF4y2Ba170年gydF4y2Ba,564 - 576。e16(2017)。gydF4y2Ba
Tzelepis, k . et al . CRISPR辍学屏幕识别遗传脆弱性和急性髓系白血病的治疗目标。gydF4y2Ba细胞的代表。gydF4y2Ba17gydF4y2Ba,1193 - 1205 (2016)。gydF4y2Ba
Behan, f . m . et al .优先级使用CRISPR-Cas9屏幕癌症的治疗靶点。gydF4y2Ba自然gydF4y2Ba568年gydF4y2Ba,511 - 516 (2019)。gydF4y2Ba
Lagziel, S。,Lee, W. D. & Shlomi, T. Inferring cancer dependencies on metabolic genes from large-scale genetic screens.BMC医学杂志。gydF4y2Ba17gydF4y2Ba37 (2019)。gydF4y2Ba
Rossiter: j . et al . CRISPR屏幕生理中揭示基因在人类细胞有条件地重要。gydF4y2Ba细胞金属底座。gydF4y2Ba33gydF4y2Ba,1248 - 1263。e9 (2021)。gydF4y2Ba
赵,d . et al .组合CRISPR-Cas9代谢屏幕显示关键氧化还原控制点依赖KEAP1-NRF2监管轴。gydF4y2Ba摩尔。细胞gydF4y2Ba69年gydF4y2Ba,699 - 708。e7 (2018)。gydF4y2Ba
沙土荒漠,m . et al。系统的映射遗传交互新创脂肪酸合成标识C12orf49作为管理者的脂质代谢。gydF4y2BaNat,金属底座。gydF4y2Ba2gydF4y2Ba,499 - 513 (2020)。gydF4y2Ba
王,t . et al .基因本质分析揭示了与致癌Ras基因网络和合成致命的交互。gydF4y2Ba细胞gydF4y2Ba168年gydF4y2Ba,890 - 903。e15(2017)。gydF4y2Ba
博伊尔,大肠。,Pr我tch一个rd,J。K. & Greenleaf, W. J. High-resolution mapping of cancer cell networks using co-functional interactions.摩尔。系统。医学杂志。gydF4y2Ba14gydF4y2Bae8594 (2018)。gydF4y2Ba
拉舍尔,et al。一个集成的地图基因在肿瘤细胞的相互作用。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba14gydF4y2Bae7656 (2018)。gydF4y2Ba
金、大肠等。人类功能基因相互作用网络从淘汰赛健身屏幕癌细胞。gydF4y2Ba生命科学。联盟gydF4y2Ba2gydF4y2Ba,(2019)。gydF4y2Ba
凯格尔,b d &瑞安,c . j .假字缓冲有助于变量重要性在癌症细胞系的基因。gydF4y2Ba公共科学图书馆麝猫。gydF4y2Ba15gydF4y2Bae1008466 (2019)。gydF4y2Ba
黛德,M。,McLaughlin, M., Kim, E. & Hart, T. Multiplex enCas12a screens detect functional buffering among paralogs otherwise masked in monogenic Cas9 knockout screens.基因组医学杂志。gydF4y2Ba21gydF4y2Ba262 (2020)。gydF4y2Ba
Beltrao时,P。,Cagney, G. & Krogan, N. J. Quantitative genetic interactions reveal biological modularity.细胞gydF4y2Ba141年gydF4y2Ba,739 - 745 (2010)。gydF4y2Ba
使用,m . et al .细胞的遗传景观。gydF4y2Ba科学gydF4y2Ba327年gydF4y2Ba,425 - 431 (2010)。gydF4y2Ba
使用,m . et al。全球基因相互作用网络地图细胞功能的接线图。gydF4y2Ba科学gydF4y2Ba353年gydF4y2Baaaf1420 (2016)。gydF4y2Bahttps://doi.org/10.1126/science.aaf1420gydF4y2Ba。gydF4y2Ba
马丁,h . et al .微分遗传相互作用的酵母应激反应MAPK通路。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba11gydF4y2Ba800 (2015)。gydF4y2Ba
Wong a . s . l . et al .多路编码CRISPR-Cas9筛查CombiGEM启用。gydF4y2BaProc。《科学。美国gydF4y2Ba113年gydF4y2Ba,2544 - 2549 (2016)。gydF4y2Ba
沈,j . p . et al .组合CRISPR-Cas9屏幕新创的映射遗传相互作用。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba,573 - 576 (2017)。gydF4y2Ba
汉族,k . et al .协同药物组合对癌症中确定CRISPR屏幕成对基因的相互作用。gydF4y2Ba生物科技Nat。》。gydF4y2Ba35gydF4y2Ba,463 - 474 (2017)。gydF4y2Ba
Najm, f . j . et al .同源CRISPR-Cas9酶组合遗传屏幕。gydF4y2Ba生物科技Nat。》。gydF4y2Ba36gydF4y2Ba,179 - 189 (2018)。gydF4y2Ba
杜,d . et al .基因交互映射在哺乳动物细胞中使用CRISPR干扰。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba,577 - 580 (2017)。gydF4y2Ba
Horlbeck, m . a . et al .映射人类细胞的遗传景观。gydF4y2Ba细胞gydF4y2Ba174年gydF4y2Ba,953 - 967。e22(2018)。gydF4y2Ba
DeWeirdt, p . c . et al .优化组合的遗传屏幕AsCas12a的人类细胞。gydF4y2Ba生物科技自然》。gydF4y2Ba1 - 11;gydF4y2Bahttps://doi.org/10.1038/s41587 - 020 - 0600 - 6gydF4y2Ba(2020)。gydF4y2Ba
Kleinstiver, b . p . et al .工程CRISPR-Cas12a变异增加活动和提高基因的目标范围,表观遗传和基本编辑。gydF4y2Ba生物科技Nat。》。gydF4y2Ba37gydF4y2Ba,276 - 282 (2019)。gydF4y2Ba
Bandyopadhyay, s . et al .重组基因网络的响应DNA损伤。gydF4y2Ba科学gydF4y2Ba330年gydF4y2Ba,1385 - 1389 (2010)。gydF4y2Ba
Ideker t & Krogan: j .微分网络生物学。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba8gydF4y2Ba565 (2012)。gydF4y2Ba
绞痛,m . et al .识别chemogenetic交互与drugZ CRISPR屏幕。gydF4y2Ba基因组医学。gydF4y2Ba11gydF4y2Ba52 (2019)。gydF4y2Ba
•j . a . &卢普r .脂肪酸合成酶(FASN)在乳腺癌的治疗目标。gydF4y2Ba当今专家。其他。目标gydF4y2Ba21gydF4y2Ba,1001 - 1016 (2017)。gydF4y2Ba
搜索:FASN-List Results-ClinicalTrials.gov。gydF4y2Bahttps://clinicaltrials.gov/search?cond=FASNgydF4y2Ba(2020)。gydF4y2Ba
哈特,t &•莫法特j .百吉饼:计算框架,确定必要的基因池图书馆屏幕。gydF4y2BaBMC Bioinforma。gydF4y2Ba17gydF4y2Ba164 (2016)。gydF4y2Ba
金,大肠&哈特,t .改进分析CRISPR健身屏幕和减少非目标效应与BAGEL2基因重要性分类器。gydF4y2Ba基因组医学。gydF4y2Ba13gydF4y2Ba2 (2021)。gydF4y2Ba
艾伦,f . et al。杰克:联合分析CRISPR / Cas9淘汰赛屏幕。gydF4y2Ba基因组Res。gydF4y2Ba29日gydF4y2Ba,464 - 471 (2019)。gydF4y2Ba
戴利,t . p . et al . CRISPhieRmix:分层混合模型CRISPR汇集屏幕。gydF4y2Ba基因组医学杂志。gydF4y2Ba19gydF4y2Ba159 (2018)。gydF4y2Ba
Bamford, s . et al。宇宙(体细胞突变在癌症的目录)数据库和网站。gydF4y2BaBr。j .癌症gydF4y2Ba91年gydF4y2Ba,355 - 358 (2004)。gydF4y2Ba
Sondka, z . et al。宇宙癌症基因普查:描述在所有人类癌症基因功能障碍。gydF4y2BaNat。启癌症gydF4y2Ba18gydF4y2Ba,696 - 705 (2018)。gydF4y2Ba
哈特,T。,Brown, K. R., Sircoulomb, F., Rottapel, R. & Moffat, J. Measuring error rates in genomic perturbation screens: gold standards for human functional genomics.摩尔。系统。医学杂志。gydF4y2Ba10gydF4y2Ba733 (2014)。gydF4y2Ba
的法官,j . m . et al .提取生物见解从项目公司阿基里斯CRISPR屏幕癌症细胞系。预印在gydF4y2BabioRxivgydF4y2Bahttps://doi.org/10.1101/720243gydF4y2Ba(2019)。gydF4y2Ba
DepMap 20第四季度公开。gydF4y2Bahttps://doi.org/10.6084/m9.figshare.13237076.v4gydF4y2Ba(2020)。gydF4y2Ba
James m . f . et al . NF2 /梅林是一个小说-调节器mTOR复杂的1,和激活的mTORC1与脑膜瘤、神经鞘瘤增长相关联。gydF4y2Ba摩尔。细胞。医学杂志。gydF4y2Ba29日gydF4y2Ba,4250 - 4261 (2009)。gydF4y2Ba
黄,J。,D我bble, C. C., Matsuzaki, M. & Manning, B. D. The TSC1-TSC2 complex is required for proper activation of mTOR complex 2.摩尔。细胞。医学杂志。gydF4y2Ba28gydF4y2Ba,4104 - 4115 (2008)。gydF4y2Ba
马奇,s . et al .缺陷自噬脑海绵状畸形是一个关键的特性。gydF4y2BaEMBO摩尔。地中海。gydF4y2Ba7gydF4y2Ba,1403 - 1417 (2015)。gydF4y2Ba
朱、y . et al .内皮细胞程序性死亡损失10激活胶质母细胞瘤细胞,促进肿瘤的生长。gydF4y2BaNeuro-Oncol。gydF4y2Ba18gydF4y2Ba,538 - 548 (2016)。gydF4y2Ba
Populo, H。,Lopes, J. M. & Soares, P. The mTOR signalling pathway in human cancer.Int。j .摩尔。科学。gydF4y2Ba13gydF4y2Ba,1886 - 1918 (2012)。gydF4y2Ba
Massague, j . G1细胞循环控制和癌症。gydF4y2Ba自然gydF4y2Ba432年gydF4y2Ba,298 - 306 (2004)。gydF4y2Ba
埃文,g . i & Vousden k h .增殖、细胞周期和细胞凋亡在癌症。gydF4y2Ba自然gydF4y2Ba411年gydF4y2Ba,342 - 348 (2001)。gydF4y2Ba
Donehower、洛杉矶等。综合分析TP53基因和通路改变癌症基因组图谱。gydF4y2Ba细胞的代表。gydF4y2Ba28gydF4y2Ba,1370 - 1384。e5 (2019)。gydF4y2Ba
张,y . et al . Pan-Cancer Proteogenomic Atlas PI3K / AKT / mTOR通路的变化。gydF4y2Ba癌症细胞gydF4y2Ba31日gydF4y2Ba,820 - 832。e3 (2017)。gydF4y2Ba
锅,j . et al .审讯的哺乳动物蛋白质复杂的结构,功能,会员使用公司健身屏幕。gydF4y2Ba细胞系统。gydF4y2Ba6gydF4y2Ba,555 - 568。e7 (2018)。gydF4y2Ba
Bayraktar, e . c . et al .代谢coessentiality映射标识C12orf49的监管机构,如加工、胆固醇代谢。gydF4y2BaNat,金属底座。gydF4y2Ba2gydF4y2Ba,487 - 498 (2020)。gydF4y2Ba
黄,s . et al . HumanNet v2:人类疾病的基因网络研究。gydF4y2Ba核酸Res。gydF4y2Ba47gydF4y2BaD573-D580 (2019)。gydF4y2Ba
哈立德,。,年代我dd我qui, A. J., Huang, J.-H., Shamsi, T. & Musharraf, S. G. Alteration of serum free fatty acids are indicators for progression of pre-leukaemia diseases to leukaemia.科学。代表。gydF4y2Ba8gydF4y2Ba14883 (2018)。gydF4y2Ba
黄素,R。,Peluso, S., Nguyen, P. L. & Loda, M. Fatty acid synthase as a potential therapeutic target in cancer.未来的杂志。gydF4y2Ba6gydF4y2Ba,551 - 562 (2010)。gydF4y2Ba
Punekar s &曹特区小说疗法影响代谢途径。gydF4y2Ba点。Soc。中国。肿瘤防治杂志。建造。书gydF4y2Bae79-e87。gydF4y2Bahttps://doi.org/10.1200/EDBK_238499gydF4y2Ba(2019)。gydF4y2Ba
Roguev, a . et al。保护和重新布线功能模块显示的裂殖酵母的上位地图。gydF4y2Ba科学gydF4y2Ba322年gydF4y2Ba,405 - 410 (2008)。gydF4y2Ba
李,K.-C。全基因组coexpression动力学:理论和应用。gydF4y2BaProc。《科学。美国gydF4y2Ba99年gydF4y2Ba,16875 - 16880 (2002)。gydF4y2Ba
Piccolis, m . et al .探索全球lipotoxicity饱和脂肪酸引起的细胞反应。gydF4y2Ba摩尔。细胞gydF4y2Ba74年gydF4y2Ba,32-44。e8(2019)。gydF4y2Ba
朱、x g . et al . CHP1调节区分glycerolipid合成通过激活GPAT4。gydF4y2Ba摩尔。细胞gydF4y2Ba74年gydF4y2Ba,45-58。e7(2019)。gydF4y2Ba
甘地,m . et al .下一代描述癌症细胞系的百科全书。gydF4y2Ba自然gydF4y2Ba569年gydF4y2Ba,503 - 508 (2019)。gydF4y2Ba
李,h . et al。癌症细胞系代谢的景观。gydF4y2BaNat,地中海。gydF4y2Ba25gydF4y2Ba,850 - 860 (2019)。gydF4y2Ba
Meshinchi, s & Arceci r .目标:急性髓系白血病(AML),加入dbGaP研究:phs000465.v19.p8。gydF4y2Bahttps://www.ncbi.nlm.nih.gov/projects/gap/cgi-bin/study.cgi?study_id=phs000465.v19.p8gydF4y2Ba(2020)。gydF4y2Ba
癌症基因组图谱的研究网络。基因组和外遗传性景观成人新创的急性髓系白血病(aml)。gydF4y2Ba新英格兰。j .地中海。gydF4y2Ba368年gydF4y2Ba,2059 - 2074 (2013)。gydF4y2Ba
Tyner, j . w . et al .功能性基因组的急性髓系白血病。gydF4y2Ba自然gydF4y2Ba562年gydF4y2Ba,526 - 531 (2018)。gydF4y2Ba
Grolemund, g . &韦翰,H。gydF4y2BaR数据科学gydF4y2Ba(O ' reilly, 2020)。gydF4y2Ba
R核心团队。接待员:统计计算的语言和环境。R统计计算的基础,奥地利的维也纳。gydF4y2Bahttps://www.R-project.org/gydF4y2Ba(2016)。gydF4y2Ba
Python软件基金会。gydF4y2BaPython语言参考gydF4y2Ba,gydF4y2Ba版本3.8.2gydF4y2Ba。(2020)。gydF4y2Ba
维尔塔宁,p . et al . SciPy 1.0:在Python中科学计算的基本算法。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba,261 - 272 (2020)。gydF4y2Ba
哈里斯,c . r . et al .数组NumPy编程。gydF4y2Ba自然gydF4y2Ba585年gydF4y2Ba,357 - 362 (2020)。gydF4y2Ba
猎人,j . d . Matplotlib: 2 d图形环境。gydF4y2Ba第一版。科学。Eng。gydF4y2Ba9gydF4y2Ba,90 - 95 (2007)。gydF4y2Ba
麦金尼,w .数据结构在Python中统计计算。Stefan van der w . &杰罗德·m .编辑。gydF4y2Ba学报》第九Python在科学会议。gydF4y2Ba56 - 61 (2010)。gydF4y2Bahttps://doi.org/10.25080/majora - 92 - bf1922 - 00 -gydF4y2Ba。gydF4y2Ba
韦翰,h . et al。欢迎来到Tidyverse。gydF4y2Baj .开源Softw。gydF4y2Ba4 gydF4y2Ba1686 (2019)。gydF4y2Ba
Dowle, m . et al。gydF4y2Ba数据。t一个ble: Extension of ‘data.frame’。gydF4y2Bahttps://cran.r-project.org/package=data.tablegydF4y2Ba(2020)。gydF4y2Ba
谢,y . et al。gydF4y2Baknitr:动态报告生成的通用包RgydF4y2Ba。gydF4y2Bahttps://yihui.org/knitr/gydF4y2Ba(2020)。gydF4y2Ba
谢,y knitr:一个全面的工具,在R可再生的研究。gydF4y2Ba实现可再生的研究gydF4y2Ba3-31gydF4y2Bahttps://doi.org/10.1201/9781315373461-1gydF4y2Ba(2018)。gydF4y2Ba
谢,Y。gydF4y2Ba用R和knitr动态文档gydF4y2Ba(劳特利奇和CRC出版社,2015年)。gydF4y2Ba
Benaglia, T。,Chauveau, D., Hunter, D. & Young, D. mixtools: an R package for analyzing finite mixture models.Softw j . Stat。gydF4y2Ba32gydF4y2Ba1至29 (2009)。gydF4y2Ba
辛普森,g . L。,R Core Team, Bates, D. M. & Oksanen, J.交换:函数生成限制排列的数据gydF4y2Ba。gydF4y2Bahttps://cran.r-project.org/package=permutegydF4y2Ba(2019)。gydF4y2Ba
Keilwagen, J。,Grosse, I. & Grau, J. Area under precision-recall curves for weighted and unweighted data.《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Bae92209 (2014)。gydF4y2Ba
格劳,J。,Grosse, I. & Keilwagen, J. PRROC: computing and visualizing precision-recall and receiver operating characteristic curves in R.生物信息学gydF4y2Ba31日gydF4y2Ba,2595 - 2597 (2015)。gydF4y2Ba
Durinck, S。,年代pellman, P. T., Birney, E. & Huber, W. Mapping identifiers for the integration of genomic datasets with the R/Bioconductor package biomaRt.Protoc Nat。gydF4y2Ba4 gydF4y2Ba,1184 - 1191 (2009)。gydF4y2Ba
Durinck, s . et al . BioMart Bioconductor:一个强大的生物数据库和微阵列数据分析之间的联系。gydF4y2Ba生物信息学gydF4y2Ba21gydF4y2Ba,3439 - 3440 (2005)。gydF4y2Ba
卡尔森,m . org.Hs.eg.db。gydF4y2BaBioconductorgydF4y2Bahttp://bioconductor.org/packages/org.Hs.eg.db/gydF4y2Ba(2018)。gydF4y2Ba
Wilke, c .流线型的情节主题和情节注释为“ggplot2”。gydF4y2Bahttps://wilkelab.org/cowplot/gydF4y2Ba(2019)。gydF4y2Ba
克拉克,e . & Sherrill-Mix年代。gydF4y2Baggbeeswarm:直言散射(小提琴)情节gydF4y2Bahttps://cran.r-project.org/package=ggbeeswarmgydF4y2Ba(2017)。gydF4y2Ba
微阵列的绅士,r .注释:注释。(Bioconductor版本:版本(3.11),2020)。gydF4y2Bahttps://doi.org/10.18129/B9.bioc.annotategydF4y2Ba(2020)。gydF4y2Ba
Neuwirth E。gydF4y2BaRColorBrewer: ColorBrewer调色板gydF4y2Ba。gydF4y2Bahttps://cran.r-project.org/package=RColorBrewergydF4y2Ba(2014)。gydF4y2Ba
顾,Z。,Eils, R. & Schlesner, M. Complex heatmaps reveal patterns and correlations in multidimensional genomic data.生物信息学gydF4y2Ba32gydF4y2Ba,2847 - 2849 (2016)。gydF4y2Ba
警告,g . r . et al。gydF4y2Bagplots:绘图数据的各种R编程工具gydF4y2Ba。gydF4y2Bahttps://cran.r-project.org/package=gplotsgydF4y2Ba(2020)。gydF4y2Ba
Kassambara, a . ggplot2准备出版的情节。gydF4y2Bahttps://rpkgs.datanovia.com/ggpubr/gydF4y2Ba(2020)。gydF4y2Ba
顾,Z。,Gu, L., Eils, R., Schlesner, M. & Brors, B. circlize Implements and enhances circular visualization in R.生物信息学gydF4y2Ba30.gydF4y2Ba,2811 - 2812 (2014)。gydF4y2Ba
阿诺德,j . b . et al。gydF4y2Baggthemes:额外的主题,尺度和几何学ggplot2”gydF4y2Ba(2019)。gydF4y2Bahttps://cran.r-project.org/package=ggthemesgydF4y2Ba。gydF4y2Ba
Attali, d &贝克,C。gydF4y2BaggExtra:增加边际的ggplot2直方图ggplot2增强gydF4y2Ba(2019)。gydF4y2Bahttps://cran.r-project.org/package=ggExtragydF4y2Ba。gydF4y2Ba
彼得·t·L。gydF4y2Ba拼凑:情节的作曲家gydF4y2Ba(2020)。gydF4y2Bahttps://cran.r-project.org/package=patchworkgydF4y2Ba。gydF4y2Ba
韦翰,H。gydF4y2Baggplot2:优雅的图形进行数据分析gydF4y2Ba(Springer, 2016)。gydF4y2Ba
Therneau, t . M . & Grambsch p . M。gydF4y2Ba生存数据建模:延长Cox模型gydF4y2Ba(斯普林格出版社,2000年版)。gydF4y2Ba
Therneau, t . m .生存:生存分析。gydF4y2Bahttps://CRAN.R-project.org/package=survivalgydF4y2Ba(2020)。gydF4y2Ba
Kassambara,。,库辛斯M。,Biecek, P. & Fabian, S.survminer:绘制生存曲线使用“ggplot2”gydF4y2Ba。gydF4y2Bahttps://cran.r-project.org/package=survminergydF4y2Ba(2020)。gydF4y2Ba
Python软件基金会。gydF4y2BaPython语言参考,3.8.5版本gydF4y2Ba。(2020)。gydF4y2Ba
Waskom m . l . seaborn:统计数据可视化。gydF4y2Baj .开源Softw。gydF4y2Ba6gydF4y2Ba3021 (2021)。gydF4y2Ba
情节技术有限公司(协作数据科学,2015)。gydF4y2Ba
Davidson-Pilon, c . et al . CamDavidsonPilon /生命线:0.26.0。Zenodo。gydF4y2Bahttps://doi.org/10.5281/zenodo.4816284gydF4y2Ba(2021)。gydF4y2Ba
Python软件基金会。gydF4y2BaPython语言参考gydF4y2Ba,gydF4y2Ba版本3.8.3gydF4y2Ba(2020)。gydF4y2Ba
彼得·t·L。gydF4y2Ba为图形处理tidygraph:整洁的APIgydF4y2Ba。gydF4y2Bahttps://CRAN.R-project.org/package=tidygraphgydF4y2Ba(2020)。gydF4y2Ba
Pedersen t . l . & RStudio。gydF4y2Baggraph:语法的一个实现图形的图形和网络gydF4y2Ba。gydF4y2Bahttps://CRAN.R-project.org/package=ggraphgydF4y2Ba(2020)。gydF4y2Ba
Seabold, s & Perktold j . Statsmodels:计量经济学和统计建模与Python。在斯蒂芬·范德w . &杰罗德·M。gydF4y2Ba学报》第九Python在科学会议gydF4y2Ba,92 - 96 (2010)。gydF4y2Bahttps://doi.org/10.25080/majora - 92 bf1922 - 011gydF4y2Ba。gydF4y2Ba
哈桑,k等人has2k1 / plotnine: v0.7.1。ZenodogydF4y2Bahttps://doi.org/10.5281/zenodo.3973626gydF4y2Ba(2020)。gydF4y2Ba
DeWeirdt, p . C。gydF4y2Ba因:Python包装Caidentifying遗传相互作用组合筛选数据gydF4y2Ba。gydF4y2Bahttps://pypi.org/project/gnt/gydF4y2Ba(2020)。gydF4y2Ba
DeWeirdt, p . C。gydF4y2Bagpplot:绘图功能基因摄动平台的Broad研究所研发小组gydF4y2Ba。gydF4y2Bahttps://pypi.org/project/gpplot/gydF4y2Ba(2020)。gydF4y2Ba
Pujar, et al。共识编码序列(ccd)数据库:一套标准化的人类和小鼠蛋白质编码区域支持的管理专家。gydF4y2Ba核酸Res。gydF4y2Ba46gydF4y2BaD221-D228 (2018)。gydF4y2Ba
Iorio说道。f . et al . CRISPR-Cas9针对无监督纠正gene-independent细胞反应。gydF4y2BaBMC基因组学gydF4y2Ba19gydF4y2Ba604 (2018)。gydF4y2Ba
法兰克,a . et al . GENCODE参考人类和小鼠基因组的注释。gydF4y2Ba核酸Res。gydF4y2Ba47gydF4y2BaD766-D773 (2019)。gydF4y2Ba
Zerbino, d . r . et al . 2018年运用。gydF4y2Ba核酸Res。gydF4y2Ba46gydF4y2BaD754-D761 (2018)。gydF4y2Ba
Lenoir w·F。,Lim, T. L. & Hart, T. PICKLES: the database of pooled in-vitro CRISPR knockout library essentiality screens.核酸Res。gydF4y2Ba46gydF4y2BaD776-D780 (2018)。gydF4y2Ba
哈特,t . et al .评估和设计的全基因组CRISPR / SpCas9淘汰赛屏幕。gydF4y2BaG3(贝塞斯达)gydF4y2Ba7gydF4y2Ba,2719 - 2727 (2017)。gydF4y2Ba
范德梅尔先生,d . et al .细胞模型passports-a中心临床、临床癌症模型的基因和功能的数据集。gydF4y2Ba核酸Res。gydF4y2Ba47gydF4y2BaD923-D929 (2019)。gydF4y2Ba
香农,p . et al . Cytoscape:软件环境生物分子相互作用网络的集成模型。gydF4y2Ba基因组Res。gydF4y2Ba13gydF4y2Ba,2498 - 2504 (2003)。gydF4y2Ba
罗,Y。,Rana, P. & Will, Y. Palmitate increases the susceptibility of cells to drug-induced toxicity: an in vitro method to identify drugs with potential contraindications in patients with metabolic disease.Toxicol。科学。gydF4y2Ba129年gydF4y2Ba,346 - 362 (2012)。gydF4y2Ba
Alsabeeh, N。,Chausse, B., Kakimoto, P. A., Kowaltowski, A. J. & Shirihai, O. Cell culture models of fatty acid overload: problems and solutions.Biochim Biophys。《摩尔。细胞杂志。脂质gydF4y2Ba1863年gydF4y2Ba,143 - 151 (2018)。gydF4y2Ba
高,j . et al .综合分析复杂的使用cBioPortal癌症基因组学和临床资料。gydF4y2Ba科学。信号。gydF4y2Ba6gydF4y2Ba、pl1 (2013)。gydF4y2Ba
斯拉米、大肠等。cBio癌症基因组学门户:一个开放的平台,探索多维癌症基因组学数据。gydF4y2Ba癌症。gydF4y2Ba2gydF4y2Ba,401 - 404 (2012)。gydF4y2Ba
德国的微生物和细胞培养GmbH:欢迎来到莱布尼兹研究所DSMZ。gydF4y2Bahttps://www.dsmz.de/gydF4y2Ba(2020)。gydF4y2Ba
高盛,m . j . et al .可视化和解释癌症基因组学数据通过齐娜平台。gydF4y2Ba生物科技Nat。》。gydF4y2Ba38gydF4y2Ba,675 - 678 (2020)。gydF4y2Ba
Bairoch, a cellosaurus,细胞系知识资源。gydF4y2Baj . Biomol。技术。gydF4y2Ba29日gydF4y2Ba25-38 (2018)。gydF4y2Ba
确认gydF4y2Ba
本研究进行了部分实现的要求博士学位的德克萨斯大学MD安德森癌症中心UTHealth生物医学科学研究生院;德克萨斯大学MD安德森癌症中心,休斯顿,德克萨斯州77030。W.F.L.,M.Mc., M.Mo. and T.H. were supported by NIGMS grant R35GM130119. MC is supported by a Kopchick fellowship and Pauline Altman-Goldstein Foundation Discovery Fellowship. E.K. is supported by a grant from the Prostate Cancer Foundation. M.D. is supported by a Schissler Foundation fellowship. T.H. is a CPRIT Scholar in Cancer Research (RR160032), and is additionally supported by MD Anderson Cancer Center Support Grant P30 CA016672. W.F.L. is supported by the American Legion Auxiliary Fellowship in Cancer Research. This work was supported by the Andrew Sabin Family Foundation Fellowship (T.H.). Flow cytometry was performed at MDACC’s Advanced Cytometry & Sorting Facility supported by the NCI Cancer Center Support Grant P30CA16672.
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
W.F.L.所有PS发现进行分析。年报,一个。G. and A.S. performed genetic interaction screens; and P.D. and M.C. performed bioinformatic analysis. W.F.L., M.C., N.E.A., E.K. and M.D. performed all other bioinformatic analyses. M.Mo. and M.Mc. performed lipid profiling experiments. J.G.D. and T.H. supervised the research. W.F.L. and T.H. drafted the manuscript and all authors edited it.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
J.G.D.咨询贴水,迷宫疗法,微软研究院,辉瑞;J.G.D.咨询,股本的探戈治疗。W.F.L.在科隆诺斯生物公司股本。剩下的作者声明没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
同行审查的信息gydF4y2Ba自然通讯gydF4y2Ba感谢匿名审稿人(s)为他们的贡献的同行评审工作。gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然保持中立在发表关于司法主权地图和所属机构。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
Lenoir W.F.,Morgado, M., DeWeirdt, P.C.et al。gydF4y2Ba发现CRISPR屏幕显示的假定的肿瘤抑制中枢在急性髓系白血病细胞脂质代谢。gydF4y2BaNat CommungydF4y2Ba12gydF4y2Ba6506 (2021)。https://doi.org/10.1038/s41467 - 021 - 26867 - 8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41467 - 021 - 26867 - 8gydF4y2Ba
本文引用的gydF4y2Ba
染色质复杂的依赖关系揭示针对白血病的机会gydF4y2Ba
自然通讯gydF4y2Ba(2023)gydF4y2Ba
CRISPR / Cas9在癌症治疗中的应用:开拓基因组编辑工具gydF4y2Ba
细胞与分子生物学快报》上gydF4y2Ba(2022)gydF4y2Ba
生物活性的动态重组基因型和沿袭了上下文相关的功能交互gydF4y2Ba
基因组生物学gydF4y2Ba(2022)gydF4y2Ba
从图书馆功能交互网络的最优施工池CRISPR健身屏幕gydF4y2Ba
BMC生物信息学gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论你同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条件或准则请国旗是不合适的。gydF4y2Ba