摘要
基因组测序对于跟踪SARS-CoV-2的进化和传播,优化分子检测、治疗方法、疫苗,并指导公共卫生应对至关重要。为了调查全球SARS-CoV-2基因组监测,我们使用通过GISAID共享的序列来估计测序强度和周转时间对189个国家变异检测的影响。在大流行的头两年,78%的高收入国家对0.5%的COVID-19病例进行了测序,而42%的低收入和中等收入国家达到了这一水平。来自高收入国家的大约25%的基因组是在21天内提交的,而来自低收入和中等收入国家的5%的基因组也是如此。我们发现,对大约0.5%的病例进行测序,周转时间<21天,可以为SARS-CoV-2基因组监测提供基准。社会经济不平等破坏了全球大流行的防范,必须努力支持低收入和中等收入国家提高其地方测序能力。
简介
COVID-19大流行已过去两年多,许多国家继续面临SARS-CoV-2感染的大规模流行1这主要是由新型病毒变异的出现和传播引起的2以及获得疫苗的机会不平等,特别是在大流行早期3.,4,5,6.基因组监测对于研究许多快速进化的病原体至关重要7用于研究SARS-CoV-2的进化和传播,设计和优化诊断工具和疫苗,并快速识别和评估流行病学特征改变的病毒谱系,包括Alpha/B.1.1.7、Beta/B.1.351、Gamma/P等令人关注的变体。1, Delta/B.1.617.2和Omicron/B.1.1.529。这些世系由于其更强的传播性和潜在的免疫逃逸,对自然感染和/或疫苗诱导的中和抗体造成了更大的全球公共卫生风险8,9.感兴趣变异(VOIs)也需要持续监测传播性、疾病严重程度或抗原性的变化10.这种具有更高流行潜力的变异一直要求采取更具体的措施,与它们构成的风险成正比,为此,政策制定者需要知道“什么”病原体在当地存在,“在社区中在哪里”传播,“什么时候”这种变异可能到达,“为什么”它们代表更大的风险,以及“谁”面临的风险最大11.没有这些问题的答案,就无法实施有效的公共卫生政策,生命就会受到不必要的影响(高发病率:长冠,后遗症)或失去生命(高死亡率)。在此次大流行期间,基因组信息一直有助于低收入、中等收入和高收入国家制定措施,遏制变体的影响,这些国家实施了基于证据的政策,以应对挥发性有机化合物的出现和蔓延12,13,14,15,16,17,18,19,20.,21,22,23,24,25,26,27.为了帮助指导公共卫生应对不断演变的变种,必须近实时跟踪全球流行的SARS-CoV-2谱系的多样性8,28,29.世界各地的数据生成器一直在向可公开访问的数据库中提交数量空前的SARS-CoV-2基因组:截至2022年6月9日,通过GISAID数据科学计划托管的EpiCoV数据库共享了1130万个共识序列(FASTA)30..在国际核苷酸序列数据库合作组织的档案中还可以找到超过550万条序列31450万个原始读取序列(FASTQ)32.相比之下,自2008年以来,通过GISAID共享了1,614,498个流感序列33.尽管公平共享病原体基因组数据的模型有所改进34在美国,全球基因组监测的强度存在显著差异。在这里,我们研究了2019冠状病毒病大流行2年(2020年3月至2022年2月)全球公开获取的SARS-CoV-2基因组监测数据,以确定与测序强度和及时检测变异相关的关键方面,并调查监测差异的后果。
结果
SARS-CoV-2基因组监测的全球差异
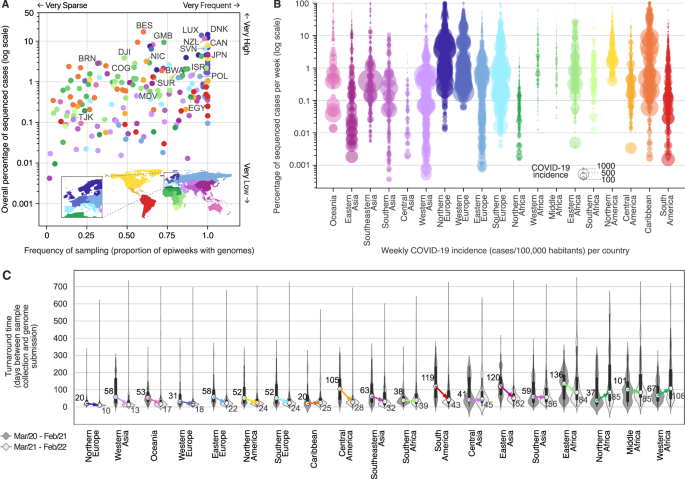
为了研究SARS-CoV-2基因组测序强度的时空异质性,我们探索了2020年3月至2022年2月每个国家每周测序的COVID-19病例百分比(图2)。1及补充资料1).有人建议对至少5%的SARS-CoV-2阳性样本进行测序,以检测流行率为0.1 - 1.0%的病毒谱系35但我们发现,在全球189个国家中,只有13个国家(6.8%)对其确诊病例总数进行了5%或以上的测序,而在189个国家中,有86个国家的确诊病例测序比例低于0.5%。1而且2而且S1).在大流行的头两年,只有7个国家或地区主要依赖其他国家的测序能力,其25%或更多的基因组测序来自国外(图2)。S2及补充资料2).截至2022年2月底,尽管高收入国家(HICs)和低收入/中等收入国家(LMICs)报告的病例总数相对相似(分别为23270万例和1.991亿例),但高收入国家提交的每例COVID-19病例序列多10倍(分别为3.53%和0.35%)(补充数据)3.).发病率大多为中等或较低(每10万人中<100例)的国家能够对较高比例的病例进行排序(图2)。1 b而且S3而且S4).例外的是,一些国家,如丹麦、日本和英国,尽管在大流行的头两年面临每周高COVID-19发病率(每10万人中有>例)的情况,但仍能够在大多数周内保持>5%的测序强度(图2)。1而且2 a, B而且S4).
根据2020年第10周(3月1日)至2022年第8周(2月26日)收集的基因组,每个国家、每个流行病学周(EW)报告的病例测序百分比,并将元数据提交给GISAID,截至2022年3月18日。序列提交的最新数字和测序病例的比例可在GISAID提交仪表板上获得,网址为“gisaid.org”。根据UNSD地理坐标,国家按区域分组,使用ISO 3161 -1命名法突出显示排序案例总体比例最高的国家:NZL新西兰、JPN日本、BRN文莱、MDV马尔代夫、TJK塔吉克斯坦、ISR以色列、DNK丹麦、LUX卢森堡、POL波兰、SVN斯洛文尼亚、EGY埃及、GMB冈比亚、COG刚果共和国、DJI吉布提、BWA博茨瓦纳、CAN加拿大、NIC尼加拉瓜、BES博奈尔和SUR苏里南。
一个每个国家测序病例的频率和总体百分比(颜色如图所示)。1).该图总结了图中所示的数据。1,在那里x-axis表示案例已排序的ew的百分比,y-axis显示病例的总体百分比(如图所示)。1作为最右边的一列)。使用ISO 3161 -1命名法强调了每个区域测序病例总体百分比最高的国家:NZL新西兰、JPN日本、BRN文莱、MDV马尔代夫、TJK塔吉克斯坦、ISR以色列、DNK丹麦、LUX卢森堡、POL波兰、SVN斯洛文尼亚、EGY埃及、GMB冈比亚、COG刚果共和国、DJI吉布提、BWA博茨瓦纳、CAN加拿大、NIC尼加拉瓜、BES博奈尔和SUR苏里南。B每个国家、每个地理区域的每一次电子战测序病例的百分比。每个圆代表一个EW,至少有一个序列的情况;圆直径代表发病率,这里定义为每个国家每EW每10万人报告的病例数。CCOVID-19大流行第一年(2020年3月至2021年2月)和第二年(2021年3月至2022年2月)不同地理区域收集的基因组周转时间分布,按提交年份分组(n= 8,947,455个基因组)。小提琴图中的元素表示中位数TATs(白色圆圈)、四分位范围(黑色矩形)以及数据集中的最小和最大数据点(黑色垂直线)。箭头突出显示大流行第一年和第二年之间tat中位数的变化。
尽管非洲和亚洲许多国家报告的COVID-19发病率在大多数周内较低(每10万人每周病例<10例,见图。S3而且S4),尚未达到与日本(4.6%)、冈比亚(9.3%)或新西兰(11.6%)类似的基因组监测水平,这些国家在大流行的头两年经历了类似的低COVID-19发病率(图2)。2 a, B而且S3而且S4).正如我们在下一节中所示,社会经济因素可以解释不同收入阶层国家之间的这些差异:58%的低收入国家(124个国家中有72个)和中上/下中等收入国家(UMCs和lmc)在大流行的头两年测序的病例不到0.5%,而在高收入国家中,只有21.5%的国家(65个国家中有14个)观察到如此低水平的监测(图)。1而且2及补充资料1而且5).然而,通过比较第一年和第二年,在HICs、UMCs和lmc中观察到测序强度的重要增加,每周测序病例的百分比分别增加了4.7倍、15倍和22.5倍。对于lic,没有观察到测序强度的重大改善(图。S5A).
基因组监测的另一个关键方面是及时性,我们通过观察周转时间(TAT;定义为19个地理区域SARS-CoV-2基因组测序从样本采集到向GISAID提交基因组之间的时间(图2)。2摄氏度;参见参考文献。36).我们观察到,在2020年底检测到更具传染性的变体(VOCs)后,几乎所有地理区域都降低了其TAT(图2)。2摄氏度看Fig。S6).北欧国家有最快的TAT(图。2摄氏度),在第二个大流行年将中位TAT从20天减少到10天。全球TAT的总体下降也与世卫组织和ECDC在检测到Alpha VOC后于2021年初发布的一系列SARS-CoV-2测序公告和指南相匹配37,38,39,40.在第二个大流行年,我们只观察到北非和西非的tat大幅增加(图2)。2摄氏度).当我们根据收入阶层比较各国的及时性时,除低收入国家外,所有阶层都观察到改善,低收入国家在第二个大流行年的TAT中位数较高(TAT中位数从71天变化为109天,见图。S5B).从定期收集的样本中快速生成和共享病原体序列数据对于最大限度地提高基因组数据对公共卫生的影响至关重要41,42.例如,在英国和玛瑙斯,VOCs Alpha和Gamma在2-3个月内分别达到了50%的频率43,44在美国,Omicron的疫情传播速度更快,不到一个月就在南非占据了主导地位45.这些例子表明,快速的tat对于早期识别和及时评估VOC的传播性至关重要41.高收入国家和中低收入国家对挥发性有机化合物和挥发性有机污染物的快速检测和表征,突出了快速基因组监测工作如何有助于地方和全球公共卫生应对的积极例子。基因组监测,特别是在低收入和中等收入国家,提供了关于四种新型挥发性有机化合物(Beta、Gamma、Delta和Omicron)早期传播和传播能力的关键信息,这一重要成就也为人兽共患疾病出现风险最高的地区的大流行防范奠定了基础。
在测序能力有限和/或TATs长的国家,更负担得起的基于pcr的检测,如基于目标失败(例如,“S基因目标失败”)来区分VOCs的RT-PCR检测,对于提供一些变体(如含有导致目标失败的特定缺失的VOCs Alpha和Omicron)传播的证据非常有价值46.然而,这些测试只有在对一个新谱系的足够多的基因组进行测序后才能部署,不仅要验证其公共卫生相关性,而且要确认允许差异RT-PCR检测的独特等位基因(缺失或广泛的遗传变化)的存在和高患病率。因此,如果没有快速测序和基因组表征,就像我们在2021年底为欧米克隆公司观察到的那样45,46在美国,基于pcr的低成本方法无法开发或部署。
快速变异检测的抽样策略
然后,我们研究了基因组测序强度和TAT对SARS-CoV-2谱系检测的影响。首先,我们发现全球观察到的世系数量与每个国家可用的SARS-CoV-2基因组数量相关r= 0.96,p值< 0.0001)和每个国家测序病例的总体比例(Pearson的r= 0.51,p值< 0.0001)(图S7),与英国的情况相似47.这表明,有限的基因组测序强度推迟了对具有改变流行病学和抗原特征的新病毒谱系的识别和反应。
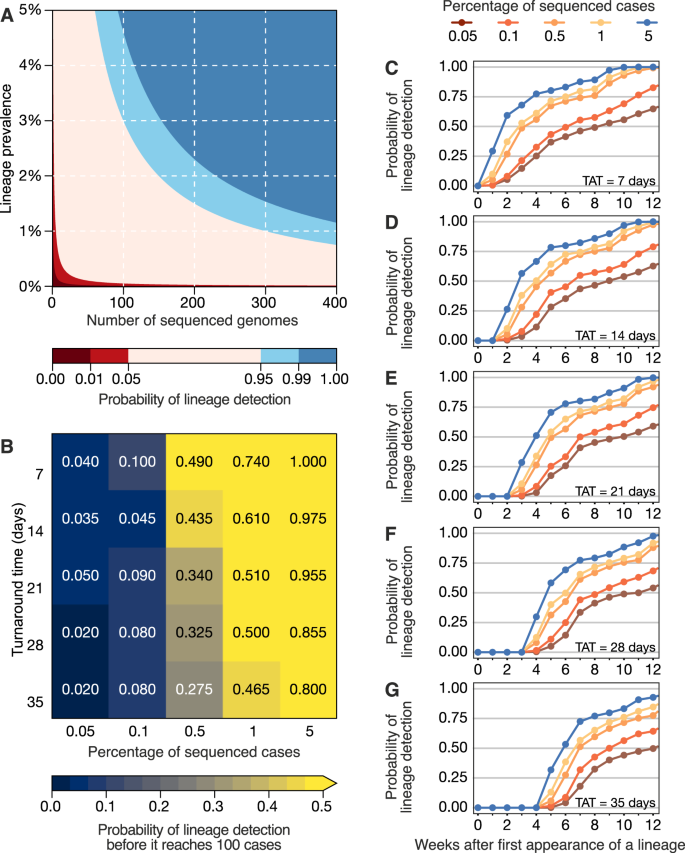
为了研究快速检测变异的策略,我们使用丹麦的元数据模拟了测序病例的百分比和TAT对可靠检测先前确定的SARS-CoV-2谱系的影响,丹麦拥有最全面的SARS-CoV-2基因组监测系统之一(见图“材料和方法”)。S8).在这里,我们假设了随机抽样的推荐场景,即病毒基因组测序的样本是独立于样本元数据(如年龄、性别或临床症状)选择的48.当计算在不同测序强度下检测到至少一个罕见谱系(0-5%流行率)基因组的概率时,我们发现每周至少测序300个基因组才能检测到95%概率为每周流行率为1%的人群中流行的谱系。对于每周5%的流行率,这个数字下降到每周75个基因组(图。3).这些数字与特定地点的疫情和人口规模无关,假设抽样具有代表性,只能说明是否存在某种世系,而不能说明其流行程度。通过模拟集中在一个国家人口最多的地区的非随机采样场景,我们观察到,检测谱系的能力下降,但当TAT低于21天,且测序强度至少占所有病例的0.5%时,仍具有一定的有用性(图2)。S9).对于其他国家来说,成功检测来自个别地区的国内血统还将取决于人口密度和人口流动性的分布,这些方面值得在未来的研究中进一步研究。平均而言,高收入国家的基因组监测规划在目前的tat和测序强度下,在随机采样的假设下,应该能够以最大概率检测到流行率为5%的流行病毒谱系(图2)。3 b和表1).然而,在随机抽样的情况下,低收入国家通常每周平均测序10个基因组,可能会错过流行率高达21.7%的SARS-CoV-2谱系(表2)1).这将极大地限制这些国家从基因组测序数据中获得的研究线索(表2)1).在这里考虑的排序病例的0.05-5%范围内,增加采样强度,并在较小程度上降低TAT,极大地提高了病毒谱系的快速检测(图5)。3 b).
一个在不同的测序制度下,检测到一个罕见谱系至少一个基因组的概率。B减少基因组测序周转时间(TAT)与增加测序百分比的相对重要性,以给定谱系(在模拟数据集中)在达到100例之前被检测到的概率来衡量(如图所示)。S8)n= 100次重采样。C- - - - - -G考虑到TATs为7天、14天、21天、28天和35天,检测到最常见的10个血统中的任何一个的概率n= 100次重采样。
接下来,我们模拟了25个场景,有100个重复,其中我们改变了采样频率(从0.05到5%)和TAT(从7天到35天),以计算在谱系达到100个病例的累积规模之前,至少检测到给定谱系的一个基因组的概率(图2)。3 b),使用来自良好特征设置的数据集作为“基本真相”(见“材料和方法”和图。S8).模拟场景显示,当在给定环境下实现每周5%的测序百分比和7天的tat时,总能在达到100例之前检测到病毒谱系。当每周测序病例的比例下降100倍至0.05%时,TAT为7天时,在达到100例之前及时检测到病毒谱系的概率下降到4%,当TAT为35天时,进一步下降到2.0%(图2)。3 b).然而,这些估计适用于随机抽样的情况。当抽样是非随机的时候,例如,当只关注一个国家人口最多的地区时,检测血统的能力就会下降;然而,即使在非随机抽样情况下,至少0.5% TAT <21天的报告病例的测序仍然是成功检测的重要因素(图2)。S9).
对于0.5%的测序病例(78%的高收入群体和40%的低收入群体)和21天的TAT(在高收入群体提交的25%的基因组中观察到,在低收入群体提交的5%的基因组中观察到)的乐观情况(补充数据)4),我们发现在100个病例之前发现谱系的概率为34%。在整个大流行期间,许多国家报告的每周发病率高达每10万居民100例(图2)。1 c而且S3而且S4).例如,在高发病率的情况下,对于巴西北部亚马逊州的一座拥有220万居民的城市玛瑙斯来说,0.5%的测序阈值相当于每周随机选择的11个基因组。通过21天的TAT,这将允许以34%的概率检测给定的谱系(图。3 b).对于São保罗市(1240万居民),这个数字增加到每周62个基因组。对于巴西(2.126亿居民)来说,在上述高发病率的情况下,这将对应于从随机样本人群中选择的1063个每周基因组。尽管在78%的高收入国家,每周近实时测序病例的0.5%比例是SARS-CoV-2基因组监测的合理基准(补充数据)4),这通常是诊断中心和资金充足、分散的基础设施密切协调的结果,以集成测序数据和样本相关的元数据(参见参考文献。49).
与基因组监测能力相关的因素
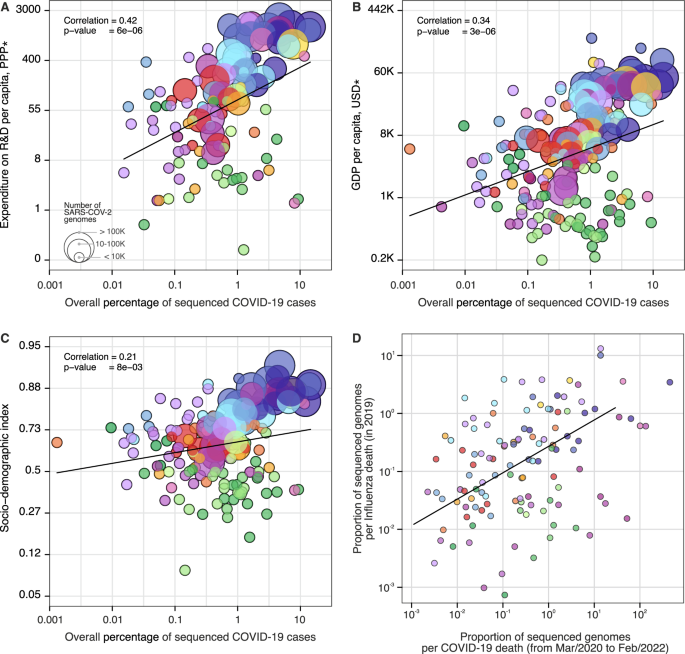
而许多hic能够依靠以前建立的网络和实验室基础设施来进行分子测试和测序50,51在美国,包括巴西、南非和印度在内的许多中低收入国家首次检测到四种VOCs43,52,53,54-在基因组监测的快速发展中面临着额外的挑战51,55,56.病原体基因组学与大流行应对的其他方面,例如监测和检测能力、医疗用品、实验室试剂、公共卫生和社会措施以及疫苗开发,相互补充,但往往争夺有限的资源57.为了调查社会经济因素如何影响全球SARS-CoV-2基因组监测反应,我们探索了每个国家COVID-19病例测序百分比与20个国家层面的社会经济和健康质量协变量之间的相关性(图2)。4及补充资料5).我们发现测序病例的百分比与人均研发(R&D)支出显著相关(r= 0.47,p值<0.0001)(图4)、人均本地生产总值(r= 0.37,p值<0.0001)(图4 b)、社会人口指数(r= 0.31,p值<0.001)(图;4摄氏度),并在COVID-19大流行之前建立了流感病毒基因组监测能力(r= 0.30,p值<0.001)(图;4 d及补充资料6).
与COVID-19测序病例总百分比相关性最高的协变量(在图中所示的期间)。1,地理区域颜色如图所示)。一个人均研发支出(斜率= 1.30,CI = (0.76, 1.84),t-value = 4.76)。B人均GDP(斜率= 0.75,CI = (0.44, 1.05),t-value = 4.83)。C社会人口学指数(斜率= 0.25,CI = (0.07, 0.44),t-value = 2.70)。D2019年每例流感死亡病例测序基因组的总比例(HA段)(斜率= 0.89,CI = (0.40, 1.37),t-value = 3.62)。协变量与周转时间的相关性见图。S10.配色方案与图中相同。1而且2.实线表示线性拟合;相关性是皮尔逊相关系数;p的值进行报告t-统计使用双侧假设,零假设是线性趋势的斜率为零。没有必要进行多次比较调整。*购买力平价,2005年美元。
2019年,向GISAID提交SARS-CoV-2基因组的国家中,共有74%(189个国家中有140个)也向同一数据库共享了流感病毒序列。当按收入阶层进行比较时,我们观察到目前对SARS-CoV-2进行测序的大多数umc(77%)和hic(78%)在2019年之前已经在公共数据库中报告了流感病毒序列。对于低收入国家而言,这一比例降至37.5%,这表明许多低收入国家在COVID-19大流行期间启动或加强了其基因组测序计划。在国家卫生、研究和发展方面的投资差异继续影响着各国扩大基因组测序强度的能力28,51,58在美国,许多中低收入国家最近在基因组监测方面取得了进展(图2)。S5)以及测序工作与已建立的基因组监测能力的联系,为未来的大流行防范计划描绘了一幅令人鼓舞的画面。
当我们探索与平均TAT(补充数据)的相关性时7),我们发现医疗服务可及性和质量指数(r=−0.56,p值<0.0001),全民健康覆盖(r=−0.56,p值<0.0001),卫生工作者密度(r=−0.56,p值<0.0001),以及人均卫生支出(r=−0.54,p值<0.0001)与平均TATs显著相关(图;S10及补充资料7).我们的结果只量化了社会经济协变量、测序强度和TAT之间的相关性,并不能被解释为因果关系。未来的研究应侧重于可能影响基因组监测的其他变量,特别是在中低收入国家,如培训实验室和生物信息学人员、元数据标准、与进口消耗品相关的成本,以及可能因边境关闭和旅行限制而加剧的装运延误28,55,56,58,59.与延迟报告VOCs相关的其他因素包括,在报告潜在VOCs时,社会和政治耻辱以及对旅行的负面影响,以及担心其他研究人员窃取和发表研究结果60.在病毒基因组学活动侧重于回顾性基因组学研究以调查SARS-CoV-2再感染的国家,预计tat时间也会更长61,疫苗突破感染62,以及过去的流行动态63,64.
讨论
加强病原体基因组监测工作,特别是在中低收入和中等收入国家,应成为改善全球大流行防范的优先事项60.我们的研究结果表明,全球SARS-CoV-2基因组监测工作目前高度不平衡,并取决于社会经济因素以及大流行前的实验室和监测能力。我们的结果表明,对总确诊病例中0.5%的TAT低于21天的病例进行测序,可以为针对SARS-CoV-2和未来新出现病毒的基因组监测研究提供基准。除了世卫组织和其他国际公共卫生当局提供的指导外(见37,38,40,65,66,67,68,69),正在进行的了解病毒基因组测序障碍和采样选择策略的调查将为未来的监测计划提供有价值的信息。实施宏基因组方法发现病毒,然后采用病毒基因组特异性测序方法,有助于克服分子和综合征监测策略的现有局限性70.采用具有代表性的基因组监测策略的标准化协议40,48根据公平的数据共享协议,建立数据和最低元数据标准,高效和便利地获取信息65以及学术界、公共卫生实验室、私人实验室和其他利益攸关方之间的合作,对于最大限度地提高基因组监测的成本效益和公共卫生影响至关重要。虽然随机采样策略可以为SARS-CoV-2变体的出现和频率估计提供准确的信息,但我们注意到,基因组采样策略应被视为病原体和问题特异性的48,65,66.例如,可能需要对按疾病严重程度分层的样本进行非随机选择,以确定与临床结果相关的基因或突变71.
世界各地正在开展几项旨在提高基因组测序能力的全球努力,包括非洲-非洲疾病控制中心、泛美卫生组织COVIGEN网络、世卫组织东南亚区域区域基因组监测联盟以及世卫组织全球风险监测框架ACT-A。全球必须作出努力,提高国内基因组监测能力,并提供可持续的研究资金,以加强测序能力和疫情分析,特别是在中低收入国家。还迫切需要改进人、动物和人-动物界面的病原体监测72.保留和扩大在SARS-CoV-2大流行期间取得的现有地方能力努力,对于遏制和应对下一个“X病”至关重要。72.
方法
基因组监测和流行病学数据
为了获得每个国家每周和累计测序病例的百分比,我们使用了与提交给GISAID的基因组“暴露国”相关的元数据30.截至2022年3月18日,收集自2020年EW(流行病学周)第10期(2020年3月1日)至2022年EW第8期(2022年2月26日)。我们从约翰霍普金斯大学系统科学与工程中心(http://github.com/CSSEGISandData/COVID-19),以及来自联合国经济和社会事务部(Department of Economic and Social Affairs)的各国人口数据73.按照世界银行现行的收入分类对国家进行了分组74.我们使用自定义管道“子采样器”(http://github.com/andersonbrito/subsampler)75.
基因组监测能力相关的协变量分析
与卫生系统相关的协变量可从卫生计量与评估研究所(IHME)获得。76的GDP数据也可从IHME获得77,人均研发支出数据可从联合国教科文组织获得78.对于IHME中的协变量76我们选择了2019年的值,2015年的GDP数据,以及2013年至2019年的研发支出,我们计算了国家层面的平均值。2019年收集的流感病毒基因组数据(HA段)来自GISAID30.2019年流感死亡估计数据下载自IHME 2019年全球疾病负担研究76.相关性和协变量细节在补充数据中提供5.为了计算相关性,排序病例的百分比为对数10改变了。应用于协变量的转换在补充数据中提供5,在“变换”栏。对于每个协变量,我们通过应用广义线性模型来估计线性拟合,回归协变量(可能是转换的,如补充数据所示)6)在日志上10-已排序案例的转换百分比;p与估算斜率对应的值见图。3.而且S10.
基因组采样场景模拟
如图所示。1丹麦在此次COVID-19大流行中拥有最全面的基因组监测计划之一,截至2022年2月26日,对约14.5%的报告病例进行了测序(2,733,807例病例和396,994个基因组,覆盖率为>70%;访问日期:2022年3月18日)79.为了模拟测序病例的百分比和TAT(样本收集和基因组提交之间的时间)在特定国家检测先前确定的SARS-CoV-2谱系中的影响,我们使用了丹麦COVID-19基因组联盟获得的基因组元数据,收集日期为2020年EW 10(3月1日)和2022年EW 8(2月26日)。79.
为了评估报告的样本收集日期和基因组提交日期之间的时间延迟对GISAID的影响,我们生成了提交日期调整后的基因组列表,以模拟TAT,表示样本收集和基因组提交之间的7至35天(5周)的延迟。考虑到丹麦每个EW测序病例的高百分比(通常超过20%),我们通过模拟每个EW测序病例的不同百分比(0.05、0.1、0.5、1和5%)的场景,生成了几个基因组数据集。在这样做的过程中,我们能够模拟25个场景(每个场景100个重复),使用不同的TAT组合和测序病例的百分比,以评估这两个参数如何影响我们检测循环谱系的能力(以概率表示)。具体来说,我们根据给定TAT后可获得的测序病例的目标百分比,对观察数据的每一列进行随机采样(将它们视为所有循环谱系的病例计数),忽略了从未达到100个样本基因组的罕见谱系。测序病例百分比和TAT的每种组合都产生了一个EWs中可用的基因组表。该过程重复100次以减轻随机抽样效应,结果用于生成每个循环谱系的检测概率。对100个重复进行汇总,得出每个流行病学周中每个谱系的检测概率。为了模拟测序病例的不均匀地理分布,我们还模拟了一个与上述情况类似的场景,但在模拟中只使用丹麦首都地区哥本哈根的测序强度,并将其与丹麦所有地区的实际谱系频率数据进行比较(图2)。S9).数字3表示不从泊松分布中抽取0的概率,其平均值是谱系流行率和排序案例的乘积。在无花果。3 b,我们展示了在给定的采样频率和延迟下,在模拟重复中计算出的检测概率,在完整数据集中达到100例的累积规模之前,能够至少对给定谱系进行一次检测,而没有延迟(“基本事实”,见图。S8).数字3 c g类似地,将其绘制出来,但要及时,询问使用“ground truth”数据集中的第一个谱系实例作为其出现,随着时间的推移,需要多长时间才能检测到给定的谱系。
报告总结
有关研究设计的进一步资料,请参阅自然研究报告摘要链接到这篇文章。
数据可用性
本研究的结果基于截至2022年3月18日GISAID上可用的8,949,097个序列的元数据https://doi.org/10.55876/gis8.220330me.全球报告病例的流行病学数据从约翰霍普金斯大学CSSE的GitHub帐户下载(https://github.com/CSSEGISandData/COVID-19).本研究中使用的所有相关数据均可作为本手稿中的补充文件,并在以下GitHub存储库中获得:https://github.com/andersonbrito/paper_2022_metasurveillance.
代码的可用性
用于计算每个国家排序病例百分比的管道可在以下GitHub存储库中获得:https://github.com/andersonbrito/subsampler75.
参考文献
谁。世卫组织COVID-19探索者。https://worldhealthorg.shinyapps.io/covid/(2021)。
劳林,A. S.和霍德克罗夫特,E. B. sars - cov -2的遗传变异——它们是什么意思?《美国医学会杂志》325, 529-531(2021)。
Rydland, H. T., Friedman, J., Stringhini, S., Link, B. G.和Eikemo, T. a . Covid-19疫苗接种的根本不平等:不平等根本原因的一个可预测但可避免的症状。Humanit。Soc。科学。Commun。9, 1-6(2022)。
是的,Y.等。公平获得COVID-19疫苗对所有国家都具有拯救生命的意义。Nat,哼。Behav。6, 207-216(2022)。
Mobarak, A. M.等。在低收入和中等收入国家消灭COVID-19。科学375, 1105-1110(2022)。
Yamey, G.等人。现在实现全球covid-19疫苗公平还为时不晚。BMJ376, e070650(2022)。
德拉蒙德,A. J., Pybus, O. G., Rambaut, A., Forsberg, R. & Rodrigo, A. G.可测量进化的种群。生态发展趋势。另一个星球。18, 481-488(2003)。
《柳叶刀》杂志上。流行病中的基因组测序。《柳叶刀》397, 445(2021)。
疾病预防控制中心。案例、数据和监控。https://www.cdc.gov/coronavirus/2019-ncov/cases-updates/variant-surveillance/variant-info.html(2021)。
谁。跟踪SARS-CoV-2变种。谁。https://www.who.int/en/activities/tracking-SARS-CoV-2-variants/(2021)。
Grubaugh, n.d.等人。追踪二十一世纪的病毒爆发。Nat。Microbiol4, 10-19(2019)。
非洲疾病预防控制中心。警报通知:新的SARS-CoV-2变种具有多个刺突蛋白突变。非洲疾病预防控制中心。https://africacdc.org/download/alert-notification-new-sars-cov-2-variant-with-multiple-spike-protein-mutations/(2020)。
ECDC。欧盟/欧洲经济区关注新型SARS-CoV-2变种传播的相关风险。https://www.ecdc.europa.eu/sites/default/files/documents/COVID-19-risk-related-to-spread-of-new-SARS-CoV-2-variants-EU-EEA.pdf(2020)。
格里菲斯,E.等。CanCOGeN关于命名、识别和报告SARS-CoV-2关注变种的临时建议。NCCID。https://nccid.ca/wp-content/uploads/sites/2/2021/02/CanCOGeN-Interim-Recommendations-for-Naming-Identifying-and-Reporting-SARS-CoV-2-Variants.pdf(2021)。
Fiocruz。Nota tÉcnica 2021/01-rede genÔmica fiocruz / ministÉrio da saÚde。https://portal.fiocruz.br/sites/portal.fiocruz.br/files/documentos/nota_tecnica_ms_2021-01-12.pdf(2020)。
谁。COVID-19流行病学每周更新世界卫生组织。https://www.who.int/docs/default-source/coronaviruse/situation-reports/weekly_epidemiological_update_23.pdf(2021)。
印度。INSACOG的基因组测序显示了令人关注的变种和印度的一种新变种。卫生和家庭福利部。https://pib.gov.in/PressReleaseIframePage.aspx?PRID=1707177(2021)。
威胁评估简介:印度出现SARS-CoV-2 B.1.617变种,以及欧盟/欧洲经济区的情况。ECDChttps://www.ecdc.europa.eu/en/publications-data/threat-assessment-emergence-sars-cov-2-b1617-variants(2021)。
叶,S.等。2021年4月,在印度-中国重庆市工作的归国中国雇员中发现了B.1.617血统的新变体。中国疾控中心3., 409-410(2021)。
谁。COVID-19流行病学每周更新世界卫生组织。https://www.who.int/docs/default-source/coronaviruse/situation-reports/20210427_weekly_epi_update_37.pdf(2021)。
乌干达。坎帕拉登记了印度Covid-19变种。乌干达病毒研究所。https://www.uvri.go.ug/news/kampala-registers-indian-covid-19-varriant(2021)。
镍镉。COVID-19更新:南非的Delta变种。镍镉。https://www.nicd.ac.za/covid-19-update-delta-variant-in-south-africa/(2021)。
非洲疾病预防控制中心。非洲疾病控制和预防中心关于新型SARS-COV-2病毒变种B.1.1.529的声明。非洲疾病预防控制中心。https://africacdc.org/news-item/africa-centres-for-disease-control-and-preventions-statement-regarding-the-new-sars-cov-2-virus-variant-b-1-1-529/(2021)。
疾病预防控制中心。科学简介:欧米克隆(B.1.1.529)变种。疾控中心COVID-19科学简报。https://www.ncbi.nlm.nih.gov/pubmed/34932278(2021)。
谁。欧米克隆的最新进展。世界卫生组织。https://www.who.int/news/item/28-11-2021-update-on-omicron(2021)。
ECDC。威胁评估简介:SARS-CoV-2出现和传播的影响EU/EEA的529变种关注(Omicron)。欧洲疾病预防控制中心。https://www.ecdc.europa.eu/en/publications-data/threat-assessment-brief-emergence-sars-cov-2-variant-b.1.1.529(2021)。
巴西。MCTI白果果RedeVírus MCTI变型副监测Ômicron。Ministério da Ciência, Tecnologia e Inovações。https://www.gov.br/mcti/pt-br/acompanhe-o-mcti/noticias/2021/11/mcti-aciona-pesquisadores-da-redevirus-mcti-para-monitorar-variante-omicron(2021)。
Inzaule, S. C, Tessema, S. K, Kebede, Y., Ogwell Ouma, A. E. & Nkengasong, J. N.非洲基因组信息病原体监测:机遇与挑战。柳叶刀感染。说.https://doi.org/10.1016/s1473 - 3099 (20) 30939 - 7(2021)。
Grubaugh, N. D., Hodcroft, E. B., Fauver, J. R., Phelan, A. L.和Cevik, M.控制新型SARS-CoV-2变种的公共卫生行动。细胞184, 1127-1132(2021)。
Shu, Y. & McCauley, J. GISAID:分享所有流感数据的全球倡议——从愿景到现实。欧元。Surveill.22, 30494(2017)。
Brister, J. R., Ako-Adjei, D., Bao, Y. & Blinkova, O. NCBI病毒基因组资源。核酸测定。43, d571-d577(2015)。
NCBI。Home-SRA-NCBI。https://www.ncbi.nlm.nih.gov/sra(2011)。
Elbe, S. & Buckland-Merrett, G.数据、疾病与外交:GISAID对全球卫生的创新贡献水珠。Chall。1, 33-46(2017)。
百万冠状病毒序列:流行的基因组网站达到巨大的里程碑。自然593, 21(2021)。
Vavrek, D.等。需要大规模的基因组监测,以便在早期时间点发现新出现的毒株。MedRxivhttps://doi.org/10.1101/2021.01.12.21249613(2021)。
Kalia, K., Saberwal, G. & Sharma, G. SARS-CoV-2基因组提交给GISAID的滞后。生物科技Nat。》。https://doi.org/10.1038/s41587-021-01040-0(2021)。
ECDC。sars - cov -2测序首次更新。欧洲疾病预防控制中心。https://www.ecdc.europa.eu/en/publications-data/sequencing-sars-cov-2(2021)。
ECDC。欧盟/欧洲经济区内SARS-CoV-2变体的检测和特征能力和能力。https://www.ecdc.europa.eu/en/publications-data/detection-and-characterisation-capability-and-capacity-sars-cov-2-variants(2021)。
ECDC。ECDC支持欧盟/欧洲经济区成员国快速检测SARS-CoV-2变种。欧洲疾病预防控制中心。https://www.ecdc.europa.eu/en/news-events/ecdc-supports-eueea-member-states-rapid-detection-sars-cov-2-variants(2021)。
谁。SARS-CoV-2基因组测序:对公共卫生产生最大影响的实施指南https://apps.who.int/iris/bitstream/handle/10665/338480/9789240018440-eng.pdf(2021)。
Frieden, T. R, Lee, C. T., Bochner, A. F, Buissonnière, M. & McClelland, A. 7-1-7:一个组织原则、目标和问责指标,使世界更安全,远离大流行。《柳叶刀》398, 638-640(2021)。
谁。世卫组织关于突发公共卫生事件中数据共享的政策声明。工作。论文。矩形。91, 237-240(2016)。
法里亚,N. R.等人。巴西玛瑙斯P.1 SARS-CoV-2谱系的基因组学和流行病学研究科学372, 815-821(2021)。
Volz, E.等人。评估英国SARS-CoV-2谱系B.1.1.7的传播性。自然593, 266-269(2021)。
维亚纳,R.等。SARS-CoV-2 Omicron变种病毒在南部非洲迅速蔓延。自然603, 679-686(2022)。
斯科特,L.等。用分子数据追踪欧米克隆的传播。科学374, 1454-1455(2021)。
杜立石,等。英国SARS-CoV-2流行的建立和谱系动态科学371, 708-712(2021)。
谁。SARS-CoV-2变种监测指南:临时指南,2021年8月9日。谁。https://www.who.int/publications/i/item/WHO_2019-nCoV_surveillance_variants(2021)。
尼科尔斯,s.m.等。CLIMB-COVID:支持SARS-CoV-2基因组监测的分散测序的持续整合。基因组医学杂志。22, 196(2021)。
令人担忧的新冠病毒变异显示了基因组监测的重要作用。自然589, 337-338(2021)。
盲点阻碍了全球冠状病毒追踪。科学372, 773-774(2021)。
拉格瓦尼,J.等人。印度古吉拉特邦早期SARS-CoV-2传播动态的基因组流行病学研究bioRxivhttps://doi.org/10.1101/2021.08.31.21262680(2021)。
Tegally, H.等人。在南非检测到令人关注的SARS-CoV-2变种。自然592, 438-443(2021)。
纳瓦卡,F. G.等。巴西亚马逊地区的COVID-19是由持续存在的地方病谱系和P.1的出现驱动的。Nat,地中海。27, 1230-1238(2021)。
Benítez, m.a.等。五个拉丁美洲国家对COVID-19的应对措施。卫生政策技术。9, 525-559(2020)。
Adebisi, Y. A.等。非洲SARS-CoV-2诊断检测:需求和挑战。潘误判率。地中海。J。35, 4(2020)。
Salyer, S. J.等人。非洲COVID-19大流行的第一波和第二波:一项横断面研究《柳叶刀》397, 1265-1275(2021)。
贝克尔,S. J.,泰勒,J. & Sharfstein, J. M.识别和跟踪SARS-CoV-2变种——挑战和机遇。心血管病。j .地中海。385, 389-391(2021)。
Maxmen, A.为什么美国冠状病毒追踪无法跟上相关变异。自然592, 336-337(2021)。
为什么一些研究人员反对无限制地共享冠状病毒数据。自然https://doi.org/10.1038/d41586-021-01194-6(2021)。
王j, Kaperak, C, Sato, T. & Sakuraba, a . COVID-19再感染:病例报告和病例系列的快速系统回顾。j . Investig。地中海。69, 1253-1255(2021)。
库斯汀,T.等人。bnt162b2 - mrna接种个体SARS-CoV-2变异突破率增加的证据值得关注。地中海Nat。.https://doi.org/10.1038/s41591-021-01413-7(2021)。
Alteri, C.等。新冠病毒基因组流行病学研究显示,意大利伦巴第地区新冠病毒感染存在多谱系和早期传播。Commun Nat。12, 434(2021)。
Oude Munnink, B. B.等。快速SARS-CoV-2全基因组测序和分析,用于荷兰知情的公共卫生决策。Nat,地中海。26, 1405-1410(2020)。
谁。2022-2032年具有大流行和流行潜力的病原体全球基因组监测战略。公牛。世界卫生组织。One hundred., 239-239a(2022)。
ECDC。代表性和靶向基因组SARS-CoV-2监测指南。(2021)。
谁。针对公共卫生目标的SARS-CoV-2基因组测序:临时指南,2021年1月8日。https://apps.who.int/iris/bitstream/handle/10665/338483/WHO-2019-nCoV-genomic_sequencing-2021.1-eng.pdf(2021)。
泛美卫生组织。SARS-CoV-2基因组特征和监测样本选择指南(2021)。
谁。SARS-CoV-2变种的检测和特征描述方法:首次更新,2021年12月20日。https://apps.who.int/iris/bitstream/handle/10665/351156/WHO-EURO-2021-2148-41903-62832-eng.pdf?sequence=1(2021)。
Gardy, J. L. & Loman, N. J.朝着基因组信息,实时,全球病原体监测系统。Nat. Rev. Genet。19, 9-20(2018)。
哈维,W. T.等。SARS-CoV-2变体、spike突变和免疫逃逸。Nat. Rev.微生物。19, 409-424(2021)。
Van Kerkhove, m.d., Ryan, m.j. & Ghebreyesus, t.a.为“X病”做准备。科学https://doi.org/10.1126/science.abm7796(2021)。
联合国。人口司经济和社会事务部(2019年)。《2019年世界人口展望》网络版。启1。联合国经济和社会事务部人口司(2019年)。《2019年世界人口展望》网络版。启1。(2019)。
世界银行。世界银行国家和贷款集团——世界银行数据服务台。世界银行https://datahelpdesk.worldbank.org/knowledgebase/articles/906519-world-bank-country-and-lending-groups(2021)。
布里托,A. F.亚采样器。https://doi.org/10.5281/zenodo.7065455(2022)。
健康指标。全球疾病负担合作网络。2019年全球疾病负担研究(GBD 2019) 1980-2019年协变量。卫生计量和评价研究所(IHME)。http://ghdx.healthdata.org/record/global-burden-disease-study-2019-gbd-2019-covariates-1980-2019(2020)。
健康指标。卫生计量和评价研究所(IHME)。1950-2015年各国国内生产总值(GDP)估算。卫生计量和评价研究所(IHME)。http://ghdx.healthdata.org/record/ihme-data/gross-domestic-product-gdp-estimates-country-1950-2015(2012)。
联合国教科文组织。教科文组织统计研究所。联合国教科文组织。http://data.uis.unesco.org/Index.aspx?DataSetCode=SCN_DS&lang=en(2021)。
迈克尔森,T. Y.等。B. SARS-CoV-2的介绍和传播丹麦有7个。medRxiv(2021)。
确认
我们感谢来自负责获取样本的原始实验室的作者,以及通过GISAID生成和共享基因组数据的提交实验室,这是本研究的基础。确认表可以在补充数据中找到8和在gisaid.org设置接入EPI_SET_20220330me。我们感谢James Nokes, Isabella Lynette Ochola和Sylvie Briand的宝贵意见。动力局感谢约书亚·巴特森(Joshua Batson),他在Twitter上分享的工作启发了Fig的创作。3.E.S.和S.F.确认EPSRC (EP/V002910/1)。gb承认来自KU Leuven内部基金的支持。C14/18/094)和研究基金会-弗兰德斯(“弗兰德基金会”,G0E1420N, G098321N)。G.W.H.感谢来自NIH F31 AI154824的支持。M.A.S.感谢NIH R01 AI153044和NIH U19 AI135995赠款的支持。M.U.G.K.感谢牛津大学马丁学院、EUH2020项目MOOD、Branco Weiss奖学金以及洛克菲勒基金会和Google.org的资助。n.d.g感谢乔治梅森大学莫卡特斯中心Emergent Ventures的快速拨款和CDC合同# 75D30120C09570的支持。O.G.P.感谢牛津大学马丁学院的支持。N.R.F.感谢维康基金会和皇家学会亨利·戴尔爵士奖学金(204311/Z/16/Z)的支持; acknowledges funding from the MRC Centre for Global Infectious Disease Analysis (reference MR/R015600/1), jointly funded by the UK Medical Research Council (MRC) and the UK Foreign, Commonwealth & Development Office (FCDO), under the MRC/FCDO Concordat agreement and is also part of the EDCTP2 programme supported by the European Union; and acknowledges funding by Community Jameel. N.R.F. and E.C.S. acknowledge support by the Medical Research Council-São Paulo Research Foundation (FAPESP) CADDE partnership award (MR/S0195/1 and FAPESP 18/14389-0) (http://caddecentre.org/)和比尔及梅琳达·盖茨基金会(INV-034540和INV-034652)。Rede Corona-ômica BR MCTI/FINEP隶属于RedeVírus/MCTI(奖项FINEP = 01.20.0029.000462/20, CNPq = 404096/2020-4)。B.P.H.和V.S.感谢澳大利亚传染病基因组网络成员对SARS-CoV-2基因组的贡献。cck感谢美国公共卫生服务局Ruth L. Kirschstein国家研究服务奖(5T35HL007649-35)的支持。R.S.A.承认CNPq资助:312688/2017-2和439119/2018-9;MEC /斗篷:14/2020 - 23072.211119/2020 - 10;FINEP: 0494/20 01.20.0026.00和UFMG-NB3 1139/20和FAPERJ: 202.922/2018。A.T.R.V.确认Corona-ômica-RJ (FAPERJ E-26/210.179/2020和E-26/211.107/2021)和CNPq(307145/2021-2和440931/2020-7)。i.a.、I.N.I.和I.P.感谢保加利亚教育和科学部的资助(合同:KП‐06‐H43/1-27.11.2020)。S.C.H.感谢惠康基金会亨利·惠康爵士奖学金(220414/Z/20/Z)的支持。
作者信息
作者及隶属关系
财团
贡献
概念:A.F.B, N.D.G, g.b., N.R.F.;数据采集:保加利亚SARS-CoV-2测序组、传染病基因组网络(澳大利亚和新西兰)、COVID-19影响项目、丹麦COVID-19基因组联盟、Fiocruz COVID-19基因组监测网络、GISAID核心管理团队、南非基因组监测网络(NGS-SA)、瑞士SARS-CoV-2测序联盟、g.g.、C.N.A、R.T.P.L、m.s.、P.C.R、c.v.f.c.、n.s.d.s.、S.M.S.;分析:A.F.B,大肠,G.D, G.W.H, C.C.K, J.H,克里,mit获得,顺丰速递,学士,G.B.;解释:A.F.B,大肠,G.D, G.W.H, M.U.G.K, J.H, H.T, G.G。C.N.A, T.d.O, R.T.P.L,克里,S.C.H, O.G.P,跨距,mit获得,顺丰速递,N.D.G, G.B。N.R.F.;起草:A.F.B。大肠,G.D, C.C.K, G.B。N.R.F.;修改:A.F.B。大肠,G.D, G.W.H, M.U.G.K, J.H, H.T, G.G。C.N.A, L.E.M,老温,b.p.h., vs, N.S.Z, O.M, H.M.B, T.d.O, R.T.P.L, m.m.,单链,A.T.R.V,联储,R.S.A。一、,I.N.I, I.P, C.V.F.C, N.S.D.S。是以,C.G。克里,D.N, K.v.E一下。,M.v.K, S.C.H,提到过,O.G.P,跨距,学士,N.D.G, G.B。N.R.F.;基金资助:N.D.G.和N.R.F.
相应的作者
道德声明
相互竞争的利益
n.d.g是Tempus实验室和国家篮球协会的传染病顾问。M.A.S.获得了美国国立卫生研究院、美国食品和药物管理局、美国退伍军人事务部和杨森研发公司的资助和合同。O.G.P.为阿斯利康开展了SARS-CoV-2分类和遗传谱系命名方面的工作。其余作者声明没有竞争利益。
同行评审
同行评审信息
自然通讯感谢匿名审稿人对本工作的同行评审所作的贡献。同行评审报告是可用的。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
布里托,a.f.,塞门诺娃,E,杜达斯,G。et al。SARS-CoV-2基因组监测的全球差异Nat Commun13, 7003(2022)。https://doi.org/10.1038/s41467-022-33713-y
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41467-022-33713-y
这篇文章被引用
跟踪SARS-CoV-2变种和资源
自然方法(2023)
下一个令人担忧的冠状病毒变种可能来自中国——它会被发现吗?
自然(2023)
SARS-CoV-2引发非消毒免疫并逃避疫苗诱导免疫:对未来疫苗接种策略的影响
欧洲流行病学杂志(2023)
德国基因组错误监测数据库Stärkung德国流行病和感染情况
Bundesgesundheitsblatt - Gesundheitsforschung - Gesundheitsschutz(2023)
Outbreak.info基因组报告:SARS-CoV-2变异和突变的可扩展和动态监测
自然方法(2023)