摘要gydF4y2Ba
针对HIV-1的gp160突刺蛋白的疫苗受到高病毒突变率和结构欺骗的阻碍。gp160的膜近端外区(膜近端外区)是自然产生的广泛中和抗体(bnAbs)的靶点,但基于MPER的疫苗不能产生bnAbs。在这里,通过冷冻电子显微镜和分子动力学模拟研究了纳米盘嵌入的刺突蛋白,揭示了自发的外畴倾斜,这造成了HIV-1的脆弱性。当每个MPER原聚体中心向三重轴辐射时,有助于形成一个膜相关的三脚架结构,该结构被垂直的尖刺遮挡,倾斜提供了对对面MPER的访问。结合4E10 bnAb fab的刺突蛋白的结构表明,抗体结合暴露的MPER,从而改变MPER动力学,改变平均外膜倾斜,并对病毒膜和刺突的跨膜段施加压力,导致膜融合的取消,并为未来的疫苗开发提供信息。gydF4y2Ba
简介gydF4y2Ba
人类免疫缺陷病毒-1 (HIV-1)作为一种病原体的起源gydF4y2Ba智人gydF4y2Ba被认为是在1920年左右发生在刚果民主共和国,由于逆转录病毒从黑猩猩传给人类,随后在20世纪70年代开始了目前的流行病gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba.过去35年汇编的数据表明,尽管接受了多剂抗病毒药物治疗,全球仍有约7800万人受到感染,约3500万人死亡,证明了这种人畜共患病的重要性gydF4y2Ba3.gydF4y2Ba.gydF4y2Ba
三聚体gp160 HIV-1包膜刺突蛋白(Env)是一种跨膜糖蛋白,由gp120和gp41三个原聚体组成,是病毒粒子上的单一病毒源蛋白。因此,它是患者自然产生的保护性抗体或通过接种疫苗在未感染个体中诱导产生的唯一靶点gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.也就是说,疫苗诱导的针对不同病毒株的具有必要宽度的抗体,即所谓的广泛中和抗体(bnAbs),未能产生抗体,既不能阻止最初的病毒与人CD4 T细胞的结合,也不能抑制后续的Env构象变化和伴随的融合事件,这些事件是结合后病毒进入宿主细胞所必需的(参见参考文献)。gydF4y2Ba7gydF4y2Ba).Env的免疫识别因其异常的序列可变性而受到阻碍gydF4y2Ba8gydF4y2Ba,密集的糖基化gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba关键位点和亚稳态的构象掩蔽gydF4y2Ba11gydF4y2Ba.尽管如此,一些慢性hiv -1感染患者在多年的感染后会产生bnAbsgydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.伴随的病毒多样化和广泛的抗体体细胞突变提高了结合亲和力,优化了partopes和表位之间的匹配gydF4y2Ba12gydF4y2Ba.BnAbs针对cd4结合位点以及gp120上的V1V2和V3糖基位点,gp41-gp120界面区域和gp41膜-近端外部区域(MPER)gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.连接Env外膜结构域和跨膜(TM)结构域的MPER是跨支菌株中最保守的HIV-1片段之一gydF4y2Ba13gydF4y2Ba.这种保守性和自然产生的针对MPER的bnAbs表现出最广泛的中和广度的观察,使MPER成为疫苗设计的关键靶点。gydF4y2Ba
通过光谱方法对MPER本身以及在膜模拟环境中TM结构域的结构进行了广泛的研究,建立了MPER多肽倾向于采用膜嵌入的扭结螺旋-铰链-螺旋构象,具有额外的侧翼灵活性gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba这表明bnab结合的MPER是从膜上部分提取的gydF4y2Ba15gydF4y2Ba.然而,MPER肽疫苗未能引发bnAbs, MPER作为整个膜嵌入Env三聚体的一部分的确切配置仍然存在争议。一项低温电子断层扫描(cryo-ET)研究报告称,MPERs形成了由三个分离的螺旋组成的三脚架状结构gydF4y2Ba16gydF4y2Ba,但其他低温et研究表明,三个MPER片段被组织成一个紧凑的柄gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba.因此,为了合理的疫苗设计目的,需要一个高分辨率的结构来阐明MPER在脂质双分子层和Env三聚体完整的糖基化外域中的实际分布。gydF4y2Ba
在这里,我们将哺乳动物细胞表达的HIV-1 Env蛋白重组为脂质纳米盘,并使用单颗粒冷冻电子显微镜(crio - em)来确定其单独结构以及与bnAb 4E10的多达三个fab的复合物结构。结合粗粒度分子动力学(CGMD)模拟,这些结构揭示了膜上Env外畴和MPER的动力学。此外,还确定了MPER的构象变化,以及随着这些4E10 fab结合数量增加而引起的邻近脂质和TM结构域的扰动。gydF4y2Ba
结果gydF4y2Ba
膜包埋的Env蛋白的MPER片段形成一个三脚架gydF4y2Ba
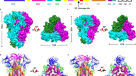
gp145 SOSIP构造gydF4y2Ba19gydF4y2Ba来自BG505分支A株的Env刺突蛋白,包括整个外膜结构域(残基1-662)、MPER和跨膜片段以及细胞质结构域的17个残基(图5)。gydF4y2Ba1gydF4y2Ba)在HEK293细胞中表达。由于与全长蛋白相比,c端截断的Env结构具有更好的表达,因此使用了它。使用PG9 Fab在十二烷基麦芽糖苷(DDM)中纯化后进行亲和层析,然后进行尺寸排除层析(SEC)(补充图)。gydF4y2Ba1 a, bgydF4y2Ba),用棕榈酰油基磷脂酰胆碱(POPC)、棕榈酰油基磷脂酰甘油(POPG)和脑极性脂质提取物的1.5:1:1.07混合物将重组gp145重组成纳米片。这种脂质混合物在组成上与HIV-1细胞膜相似gydF4y2Ba20.gydF4y2Ba除了缺少胆固醇。即使胆固醇是HIV-1脂质组中最丰富的成分,我们使用含有20%胆固醇的膜进行的CGMD模拟显示了外畴的动态行为,特别是倾斜角度,与我们的低温电镜图中看到的一致(见下文)。用SEC去除空的纳米盘(补充图。gydF4y2Ba1gydF4y2Ba).SDS-PAGE对SEC峰值部分的分析证实了gp145和膜支架蛋白MSP1D1dH5的存在,MSP1D1dH5用于纳米盘组装(补充图。gydF4y2Ba1 bgydF4y2Ba).DDM中gp145阴性的EM图像显示gp145的三重对称俯视图(补充图)。gydF4y2Ba1 cgydF4y2Ba),而纳米盘嵌入gp145的阴性染色EM图像显示侧视图,并显示了纳米盘(补充图。gydF4y2Ba1 dgydF4y2Ba).然后将纳米盘嵌入gp145与bnAb 4E10的Fab片段孵育,与BS3交联并在网格上玻璃化。利用RELION-3对所选颗粒进行图像处理,结果表明约80%的4E10 Fab颗粒没有明显的密度。对这些颗粒进行进一步处理,得到了总体分辨率为3.9的纳米盘嵌入gp145的密度图Å(图2)。gydF4y2Ba1 bgydF4y2Ba和补充图。gydF4y2Ba2gydF4y2Ba- - - - - -gydF4y2Ba4gydF4y2Ba).这张图使我们能够建立一个外畴残基31-662的原子模型和残基663-671的主干模型(详情请参阅“方法”)。该图谱没有解析跨膜螺旋,这在之前的纳米盘嵌入的全长Env蛋白图谱中也没有解析gydF4y2Ba21gydF4y2Ba,很可能是单跨TM结构域相对于纳米盘位置变化的结果。但是,额外的低分辨率密度(~5 Å;补充图。gydF4y2Ba4 bgydF4y2Ba)表示MPER- n段的开始,我们将MPER核磁共振结构的n端段放入其中(PDB: 2PV6)gydF4y2Ba14gydF4y2Ba(补充图。gydF4y2Ba4 dgydF4y2Ba),从而揭示了MPER在膜和三聚体环境外畴中的分布。每个原聚体的MPER-N片段形成三个独立的螺旋体,采用三脚架状构象(图2)。gydF4y2Ba1 b, cgydF4y2Ba),这与先前报道的mper收敛结构不同gydF4y2Ba16gydF4y2Ba或形成紧凑的茎gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba.gydF4y2Ba
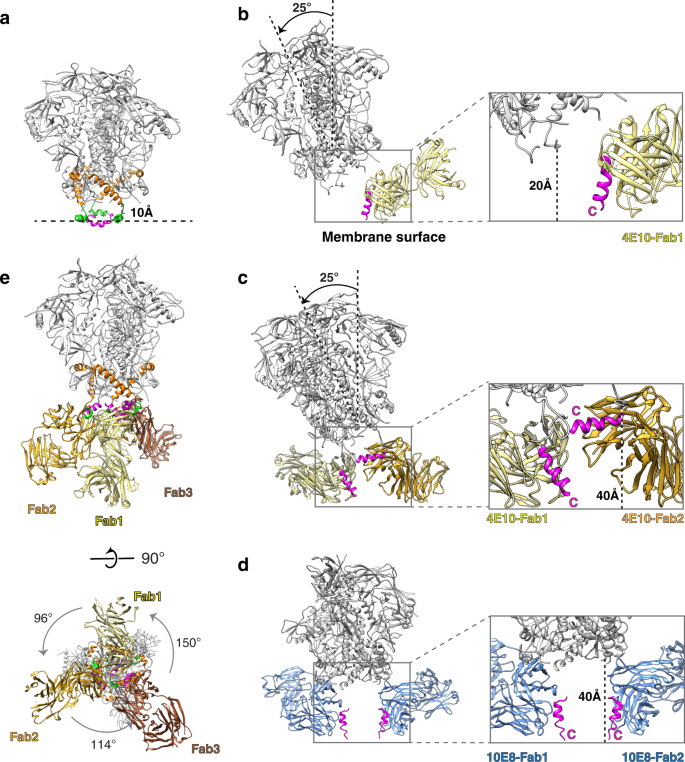
一个gydF4y2Bagp160的域架构。FP,融合肽;NHR, n端七键重复;CHR, c端七叉重复序列(橙色);MPER-N(绿色);MPER-C(红色);TM,跨膜结构域;CT,细胞质尾。上面的数字表示段的第一个残差,下面的数字表示最后一个残差。gydF4y2BabgydF4y2Ba左图:低等高线水平的纳米盘嵌入gp145的低温电镜图(0.011)。纳米圆盘的密度用浅黄色表示。右图:纳米盘嵌入gp145的高等高线(0.014)的Cryo-EM图,以半透明灰色显示,建模的gp145结构以带状表示,与图吻合。CHR区域为橙色,MPER-N段为绿色。外膜的其余部分用蓝色表示。gydF4y2BacgydF4y2Ba左:gp145的结构平行(上)和垂直于膜平面(下)。右:在将完整MPER (PDB: 2PV6)的NMR结构添加到我们的gp145模型(基于MPER- n段的叠加)后,与左侧面板中的视图相同。CHR区域为橙色,MPER-N段为绿色,MPER-C段为洋红色。gydF4y2BadgydF4y2Ba将MPER-TM的两个可用NMR结构放置到冷冻- em图中(基于MPER-N段的位置放置)。三脚架结构(PDB: 6DLN)很好地符合图(左),而柄泡结构(PDB: 6E8W)的MPER-N段与冷冻- em图对应不太好,因为跨膜螺旋远远突出于纳米盘密度(右)。gydF4y2BaegydF4y2Ba三张gp145的冷冻电镜图显示,gp145的外畴相对于膜平面采用了广泛的角度。地图显示为半透明的表面,外域的结构为彩色丝带。测量了从外畴末端(残基Ala662)到纳米盘中心(由虚线表示)的距离。gydF4y2Ba
我们的结构表明MPER-N片段部分埋藏在脂质双分子层中(图2)。gydF4y2Ba1 bgydF4y2Ba),与先前的核磁共振结构一致,表明MPER形成膜浸螺旋段gydF4y2Ba14gydF4y2Ba.由于一个铰链连接MPER-N和MPER-C螺旋gydF4y2Ba14gydF4y2Ba由于缺乏MPER-C段和TM区域的可靠密度,我们的地图没有提供MPER-C段的位置或TM螺旋的组织。也就是说,正如下面讨论的那样,每个MPER-C片段很可能向后延伸到Env三聚体的中心,并且每个原聚体的mper链接TM结构域近似于三重三聚体轴附近,要么是松散关联,要么是与MPER-TM的两个NMR结构中观察到的TM束相关gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba.值得注意的是,其他研究表明原生三聚体尖峰中的TM螺旋并不形成刚性三聚体gydF4y2Ba24gydF4y2Ba但可能在病毒融合过程中转变成这样的结构。其中一种核磁共振结构(PDB: 6E8W)gydF4y2Ba22gydF4y2Ba, MPER-C段与它们各自的TM结构域形成了某种连续的螺旋,尽管MPER-C螺旋扭曲并偏离了三重轴。MPER-C段和-N段之间的铰链区域形成强烈的扭结,导致MPER-N段近似于三重轴。我们将其称为柄泡构象。其他核磁共振结构(PDB: 6DLN)gydF4y2Ba23gydF4y2Ba,是TMD和MPER-C之间的铰链形成强烈的扭结,而MPER-C和-N段形成一个连续的螺旋,从三重螺旋延伸出去,形成三脚架构象。gydF4y2Ba
为了比较完整Env三聚体背景下MPER的构象,我们测量了MPER片段n端末端之间的距离,即残基Lys665的Cα原子之间的距离。在我们的cryo-EM结构中,距离为30 Å,而在NMR三脚架结构中,距离为53 Å,在NMR柄泡结构中,距离为16 Å (Supplementary Fig。gydF4y2Ba5gydF4y2Ba).测量结果表明,我们结构中的MPER段采用了介于两个核磁共振结构之间的构象。然而,来自三个MPER-N原聚体的Trp666残基在茎泡构象中聚集,形成疏水核心,这在低温em结构中不是这样的(补充图)。gydF4y2Ba5 bgydF4y2Ba).此外,将这两个核磁共振结构放入冷冻- em图中,使用MPER- n片段作为锚点,核磁共振三脚架结构的尺寸更好地符合图,建立了该结构在整个Env蛋白的背景下更接近MPER构象(图2)。gydF4y2Ba1 dgydF4y2Ba).核磁共振三脚架结构和我们的cryo-EM结构之间MPER构象的其余差异很可能是由于外畴对MPER施加的约束,这在cryo-EM研究中存在,但在NMR研究中不存在。gydF4y2Ba
Env蛋白经历自发的倾斜运动gydF4y2Ba
对gp145的数据处理表明,外畴相对于膜平面的角度范围很广。为了更好地表征外畴倾斜的范围,我们进行了迭代3D分类(补充图。gydF4y2Ba3.gydF4y2Ba),得到了20个具有明确的纳米盘密度和足够的外畴密度特征的映射,以明确地对接原子模型(示例如图所示)。gydF4y2Ba1 egydF4y2Ba).由对接模型生成的电影说明了Env三聚体相对于纳米圆盘膜可以经历的倾斜程度(补充电影gydF4y2Ba1gydF4y2Ba).图显示,外膜域的三折轴可以从膜法线倾斜至少30°(图2)。gydF4y2Ba1 egydF4y2Ba及辅助电影gydF4y2Ba2gydF4y2Ba).由于倾斜的结果,外膜域与膜表面的距离增加了多达20 Å(图2)。gydF4y2Ba1 egydF4y2Ba).gydF4y2Ba
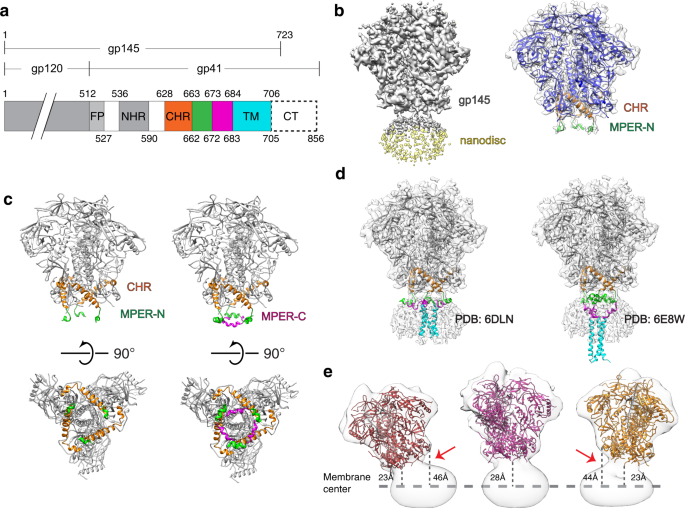
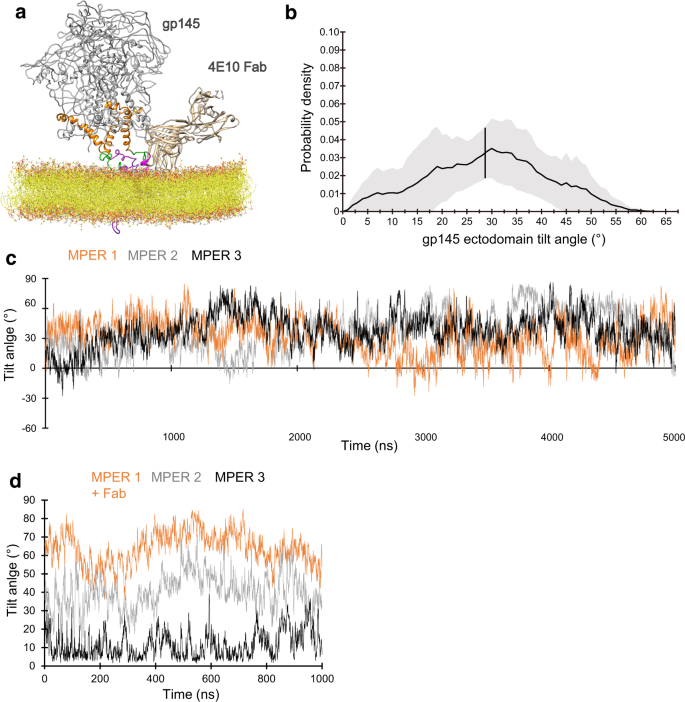
为了更好地理解膜上外畴的动力学,我们使用CGMD模拟(图2)。gydF4y2Ba2gydF4y2Ba).gp145被嵌入含有20%胆固醇的POPC双分子层,因为已知HIV-1膜富含胆固醇gydF4y2Ba20.gydF4y2Ba.模拟结果证实了低温电子显微镜的结果,因为外畴是高度移动的(补充电影gydF4y2Ba3.gydF4y2Ba),倾角可达~63°。从5个10 μs的模拟中,外畴平均倾斜17.6°±10.0°,但假设倾斜范围很广(图2)。gydF4y2Ba2 bgydF4y2Ba).CGMD模拟还表明,MPERs是高度动态的,并且偶尔会离开膜平面(补充电影gydF4y2Ba3.gydF4y2Ba),但没有迹象表明MPER运动与外畴倾斜有关。将CGMD快照转换为原子视图也表明,高外畴倾斜本身并不有利于从膜中提取MPER(图2)。gydF4y2Ba2摄氏度gydF4y2Ba).gydF4y2Ba
bnAb 4E10与倾斜的Env蛋白结合并对TM区施加力gydF4y2Ba
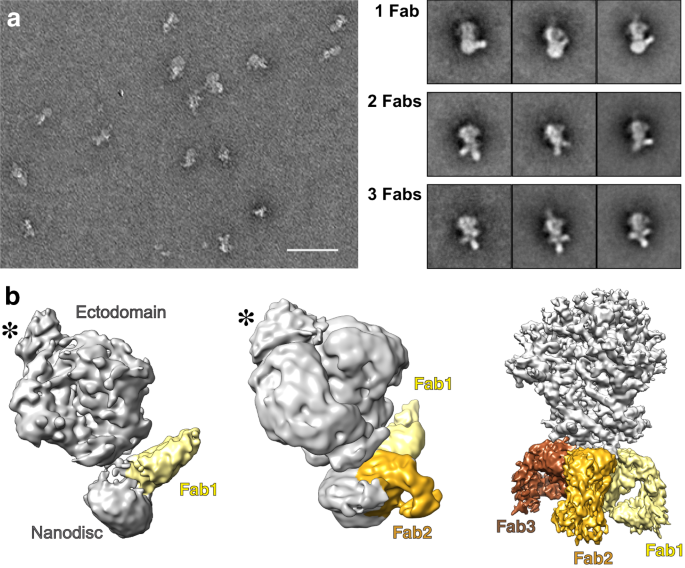
CGMD结果表明,MPER特异性bnAb与gp145的结合依赖于两个独立的动力学,(1)外畴的倾斜使MPER在空间上可接近Fab, (2) MPER运动使表位从膜上充分暴露,有利于Fab结合。我们使用结构良好的bnAb 4E10识别MPER铰链和c螺旋gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba让这个概念得以验证。将纳米盘嵌入gp145与超过100倍摩尔摩尔的4E10 Fab结合后,约20%的玻璃化外畴被至少一个Fab分子结合。通过对负染颗粒进行2D分类获得的投影平均值显示,一到三个fab可以与gp145结合(图1)。gydF4y2Ba3gydF4y2Ba).在对玻璃化晶圆结合颗粒进行大量处理后,我们能够在复合物中生成纳米盘嵌入gp145的密度图,其中一个4E10 Fab (gp145•1Fab)的分辨率为8.8-Å,两个4E10 Fab (gp145•1Fab)的分辨率为8.2-Å,三个4E10 Fab (gp145•1Fab)的分辨率为3.7-Å(图2)。gydF4y2Ba3 bgydF4y2Ba和补充图。gydF4y2Ba2gydF4y2Ba).尽管其中两张地图的分辨率有限,在fab结合区域只有~12 Å(补充图。gydF4y2Ba4 bgydF4y2Ba),附加密度的特征揭示了这些表示绑定晶圆,并允许对接4E10晶圆晶体结构(PDB: 1TZG)gydF4y2Ba25gydF4y2Ba(补充图。gydF4y2Ba6gydF4y2Ba).值得注意的是,这些图谱揭示了mper特异性4E10 Fab与gp145结合的两种不同模式。gp145•1Fab图说明了结合模式1,其中Fab从纳米盘延伸几乎平行于膜表面,但与膜平面在与外畴相同的方向略微远离膜平面,并且Fab平面平行于膜平面。在gp145•2Fab图中,一个4E10 Fab也表现出结合模式1,但第二个Fab以模式2结合,其中Fab沿与外畴相反的方向延伸,占据了通常属于脂质双分子层的空间。此外,Fab的平面旋转~90°,因此几乎垂直于膜平面。最后,在gp145•3Fab映射中,所有三个fab都以绑定模式2绑定到gp145。由于这三个晶圆厂现在都占据了通常被膜所占据的空间,这张图没有显示出代表纳米圆盘的密度。gydF4y2Ba
为了分析纳米盘嵌入gp145在一个和两个4E10 Fab复合物中的中分辨率图,我们将4E10 Fab与MPER-C肽复合物(PDB: 1TZG)对接。gydF4y2Ba25gydF4y2Ba(补充图。gydF4y2Ba6gydF4y2Ba).4E10的重链(CDRH3)上的长互补决定区3是高度疏水的,4E10利用其CDRH1和CDRH3环与脂质结合gydF4y2Ba26gydF4y2Ba这意味着这些cdr与脂质双分子层形成了相互作用表面。在我们的纳米盘嵌入gp145本身的结构中,外畴定向垂直于膜平面(图2)。gydF4y2Ba4gydF4y2Ba).然而,要使4E10 Fab能够与gp145结合,外畴必须倾斜~25°(图2)。gydF4y2Ba4 bgydF4y2Ba).最近在AMC011菌株的全长Env蛋白与VRC42.01的Fab复合物的低温电镜结构中也发现了类似的倾斜角度gydF4y2Ba21gydF4y2Ba.我们的gp145•2Fab结构的外畴显示出相似的倾斜角度~25°(图1)。gydF4y2Ba4摄氏度gydF4y2Ba).我们还测量了gp145外畴距膜表面的距离。对于gp145本身,Glu662之间的距离(c端七键重复;CHR)和Trp666(膜浸MPER的起始)为~10 Å。在gp145•1Fab结构中,这一距离增加到~20 Å。在gp145•2Fab结构中,第二个Fab的距离进一步增加到~40 Å。当我们将Env的原子模型和结合到其MPER表位(PDB: 4U6G)的10E8 Fab复合物对接到已发表的含有两个结合10E8 Fab的AMC011菌株的Env外膜结构域的冷冻- em图中时,我们发现了相似的~40 Å的距离。gydF4y2Ba4 dgydF4y2Ba)gydF4y2Ba21gydF4y2Ba.总的来说,这些发现表明,与mper靶向的bnAbs复合物中的Env外膜域倾斜并从膜上抬起。gydF4y2Ba
一个gydF4y2Bagp145本身的低温电镜模型(灰色为外畴,橙色为CHR,绿色为MPER-N)加上来自核磁共振结构(PDB: 2PV6)的MPER-C片段,表明MPERs是膜嵌入的,外畴(残基Ala662)距离膜表面的距离为~10 Å。gydF4y2BabgydF4y2Ba带有4E10 Fab结合(黄色)的gp145模型,显示外畴倾斜,增加其与膜的距离至~20 Å,并且MPER-C段(洋红色)必须暴露并近似垂直于膜平面(结合模式1)。gydF4y2BacgydF4y2Bagp145与两个4E10 Fab结合(黄色和金色)的模型,显示外畴仍然倾斜,两个Fab结合非常不同。而有一个Fab显示了与面板中相同的绑定模式1 (gydF4y2BabgydF4y2Ba),第二个Fab将MPER-C段从膜提升到~40 Å,在那里它与膜近似平行运行(结合模式2);这个Fab占据了通常由薄膜占据的空间。gydF4y2BadgydF4y2Ba将10E8 Fab - mper表位复合物(PDB: 4U6G)的晶体结构对接到AMC011菌株(EMDB: 21334) Env蛋白结合的10E8 Fab的低温电镜图谱中,生成带有两个bnAb 10E8结合Fab的gp160模型(蓝色)。该模型表明,外畴不倾斜,但距离膜表面约40 Å, 10E8晶圆与MPER-C的结合方式与4E10的结合方式1相似。gydF4y2BaegydF4y2Bagp145的Cryo-EM结构与三个4E10 Fabs结合(黄色、金色和棕色)平行(上)和垂直于膜平面(下)。虽然绑定是不对称的,但所有三个fab都显示绑定模式2。颜色编码与图中相同。gydF4y2Ba1gydF4y2Ba- - - - - -gydF4y2Ba3.gydF4y2Ba.gydF4y2Ba
由4E10 Fab结合的MPER-C段(残基674-683)的取向与未结合状态有本质上的不同。在我们的纳米盘包埋gp145结构中,MPER-C片段应该大部分平行于膜,部分浸入脂质双分子层(图2)。gydF4y2Ba4gydF4y2Ba).相比之下,gp145•1Fab复合物的模型表明,在结合模式1中由第一个Fab结合的MPER- c段从膜中提取出来,MPER的c端端仍然靠近膜表面,但MPER- c段现在相对于膜平面呈~80°角(图1)。gydF4y2Ba4 bgydF4y2Ba).在结合模式2中结合第二个Fab导致进一步去除MPER-C段,该段现在几乎平行于膜,但距离接近40 Å(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).MPER-C段的这个位置需要提取TM螺旋,因此不太可能在原位发生,但这确实意味着Fab结合对TM螺旋施加了巨大的拉力,可能导致跨膜区域的扰动,甚至可能将螺旋从膜中提取出来。值得注意的是,在结合模式2的第二个Fab的结合中看到的MPER-C段的剧烈重新定向,在两个结合10E8 Fab的Env外畴的模型结构中没有观察到(图2)。gydF4y2Ba4 dgydF4y2Ba).在该结构中,两个MPER-C段都采用了与gp145•1Fab和gp145•2Fab结构中以结合模式1结合的4E10晶圆所观察到的构象。4E10-和10e8结合结构的差异可能与4E10膜相互作用表面的疏水性和脂质结合有关gydF4y2Ba14gydF4y2Ba,gydF4y2Ba27gydF4y2Ba和/或gp145的截断c端结构域,用于与4E10形成复合物。gydF4y2Ba
gp145•3Fab映射的3.7-Å分辨率使我们能够为外畴以及其中一个子单元的整个MPER构建原子模型,并为其他两个子单元的MPER构建骨架模型,从而揭示MPER是如何与前者连接的(图1)。gydF4y2Ba4 egydF4y2Ba,补充图。gydF4y2Ba4 fgydF4y2Ba而且gydF4y2Ba7一个gydF4y2Ba).然而,我们既没有观察到纳米盘的密度,也没有观察到TM螺旋,这表明三个4E10 fab的结合导致三聚体从纳米盘中完全提取。之前通过等温滴定量热法直接测量4E10 Fab结合到HIV-1病毒模拟脂质体上的MPER肽,结果显示焓变为−25千卡/摩尔gydF4y2Ba14gydF4y2Ba.通过表面等离子体共振确定,放热过程与1.0 μM Kd的弱结合常数相关,表明存在显著的熵惩罚。虽然我们目前还没有直接的能量测量方法来测量4E10在纳米盘环境下提取gp41 TM,但单分子原子力显微镜方法显示,即使是多通道跨膜蛋白也可以在与粘附分子介导的力(pN)相同或更低的力(pN)下从膜中展开和提取gydF4y2Ba28gydF4y2Ba.尽管我们的gp145•3Fab图(在补充材料中进一步描述)清楚地代表了一种人为的情况,但它使我们得出两个结论。首先,在外结构域的背景下,4E10 bnAb识别的MPER表位保持与在与MPER肽复合物的4E10 Fab晶体结构中观察到的相同构象(补充图。gydF4y2Ba7 bgydF4y2Ba)gydF4y2Ba26gydF4y2Ba.其次,Env三聚体在3个4E10 fab结合后的完全膜提取说明了4E10结合对跨膜结构域施加的张力,这可能解释了gp145•1Fab图中纳米盘的扭曲外观(图1)。gydF4y2Ba3 bgydF4y2Ba).gydF4y2Ba
bnAb结合对环境动力学的影响gydF4y2Ba
为了了解bnAb结合对Env动力学的影响,我们分析了膜嵌入gp145的CGMD模拟,并将高度倾斜的外畴快照转换为原子模型。在许多模型中,MPER-C段的构象与4E10 Fab复合物晶体结构中的构象差异太大,无法实现明确的对接(补充图。gydF4y2Ba8gydF4y2Ba).这一发现表明4E10表位可能只是偶尔处于抗体结合的最佳构象。另外,可以想象抗体的初始相互作用可能只与其表位的一部分发生,如前所述gydF4y2Ba13gydF4y2Ba然后催化整个MPER-C段的折叠,使Fab与晶体结构中所见的整个表位完全接合gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba.此外,在几乎所有模型中,MPER-C段的定位方式是,对接4E10 Fab要么导致Fab部分嵌入膜中,要么导致Fab与外畴发生空间冲突(补充图)。gydF4y2Ba8 bgydF4y2Ba).然而,一个快照显示MPER-C段的方向与我们的gp145•1Fab结构相似,其构象允许与4E10 Fab对接。gydF4y2Ba5gydF4y2Ba).对膜嵌入gp145•1Fab复合物的CGMD模拟显示,与没有4E10 Fab时一样,外畴的角度范围也很广。gydF4y2Ba5 bgydF4y2Ba及辅助电影gydF4y2Ba4gydF4y2Ba),但其平均倾斜角度为28.7°±12.0°,与无配位外畴的平均倾斜角度(17.6°±10.0°)不同。此外,在未配位的gp145中,三个MPER-C片段的平均倾斜为29.0°±20.7°(补充图)。gydF4y2Ba9gydF4y2Ba),但似乎彼此独立移动(图;gydF4y2Ba5度gydF4y2Ba和补充图。gydF4y2Ba10gydF4y2Ba),以10E8 fab结合的gp145为例,MPER-C片段的角度更明显(图2)。gydF4y2Ba5 dgydF4y2Ba和补充图。gydF4y2Ba11gydF4y2Ba).fab结合的MPER-C稳定在64.9°±10.4°的高倾角,类似于从冷冻- em图中得到的~80°(图2)。gydF4y2Ba4 bgydF4y2Ba),而相邻的MPER采用了42.5°±15.3°的中间倾斜,这可能足以促进第二个Fab的结合,而最后一个MPER采用了20.6°±12.7°的低倾斜角度,与未配位gp145中的MPER- c段相似。gydF4y2Ba
一个gydF4y2Ba该膜嵌入gp145与结合4E10 Fab的系统用于粗粒度分子动力学(CGMD)模拟。gp145外畴为灰色,CHR为橙色,MPER-N为绿色,MPER-C为洋红色。结合的Fab以金色显示,脂质双分子层以黄色显示。gydF4y2BabgydF4y2Ba图中显示了4E10 fab - binding gp145外结构域所采用的倾斜角度分布,汇总了所有5个重复序列的数据。实线表示平均值,灰色带表示标准差。29°处的垂直线是外畴的总体平均倾斜。gydF4y2BacgydF4y2Ba在无配位gp145的CGMD模拟中,三个MPER-C片段相对于膜平面的角度随时间的变化,说明了三个片段高度动态和不相关的运动(见补充图)。gydF4y2Ba10gydF4y2Ba对于来自所有重复的数据)。gydF4y2BadgydF4y2Ba在4E10 Fab结合gp145的CGMD模拟中,三个MPER-C段随时间变化的角度,说明Fab结合使三个MPER-C段稳定在不同的平均角度(见补充图)。gydF4y2Ba11gydF4y2Ba对于来自所有重复的数据)。gydF4y2Ba
有效的抗mper bnab介导的病毒融合抑制的机制基础gydF4y2Ba
我们的纳米盘嵌入gp145本身以及与多达3个4E10 fab复合物的结构阐明了bnAb与MPER结合的前提条件和后果(图2)。gydF4y2Ba6gydF4y2Ba).大多数时候,高糖基化的Env外膜域会屏蔽bnAb表位在很大程度上浸在膜中的MPER。然而,我们的低温电镜和CGMD分析表明,相对于膜,外畴可以采用广泛的角度。在倾斜角度为~25°(4E10 Fab-结合gp145的冷冻电镜图中可见的角度)或更高时(24%的时间发生),MPER可以与Fab结合,这也独立地要求表位在膜上有足够的暴露,并采用至少允许与抗体初始结合相互作用的构像。在抗体与部分暴露的MPER接合后,完全结合将使MPER稳定在膜提取的结构中,从而破坏MPER原聚体的独立和可比较的运动,否则将促进下游的半融合和融合过程(见下文)。将其他靶向mper的bnAbs的表位结合Fab与各自表位引导的4E10 Fab重叠,表明这可能是这些bnAbs中和活性的一般原理(图2)。gydF4y2Ba6 bgydF4y2Ba).抗体结合也通过对跨膜结构域施加张力来干扰Env功能。虽然我们的地图没有揭示这种张力对TM结构域的影响,但之前的一项研究提出,抗体结合可能会破坏由TM结构域形成的假定的螺旋束gydF4y2Ba21gydF4y2Ba.此外,抗mper的bnAbs结合非配体以及受体结合的Env构型和发夹前中间状态的能力(补充图。gydF4y2Ba12gydF4y2Ba)提供较长的时间窗口来介导病毒中和。gydF4y2Ba
gp145外膜域为灰色,CHR为橙色,MPER-N为绿色,MPER-C为洋红色,跨膜域为浅蓝色。脂质双分子层为淡黄色/棕色,抗体为亮黄色。gydF4y2Ba一个gydF4y2Ba在Env蛋白的直立取向中,bnAb 4E10的MPER表位(*表示)被外膜遮挡,使其无法被bnAb接触,并且大多数时间不暴露在膜中,而是隐藏在膜中。外畴的短暂倾斜使相反的MPER可接近,如果这与表位的至少部分暴露同时发生,则bnAb可以进行第一次相互作用。完全抗体结合既改变了MPER在膜上的运动,又在跨膜结构域上产生了压力,从而防止了膜融合所需的进一步构象变化。gydF4y2BabgydF4y2Ba结合HIV-1 Env的mper靶向bnAbs fab的结构排列。4E10 (PDB: 4XC3)、DH511 (PDB: 5U3N)、10E8 (PDB: 4U6G)和PGZL1 (PDB: 6O3J)的fab基于共识MPER表位(品红)对齐,表明所有针对MPER- c的bnAbs以类似的方式与HIV-1 Env结合。注意4E10的脂质相互作用表面比其他bnAbs更疏水gydF4y2Ba26gydF4y2Ba.gydF4y2Ba
讨论gydF4y2Ba
实验观察到的HIV-1三聚体外畴的倾斜和扭转运动与MD模拟一致。虽然可以预测膜受体外结构域的运动,特别是那些具有单通道TM片段的膜受体,但我们不知道在其他系统中报告的类似经验发现。以前在膜模拟环境中对Env蛋白的冷冻电镜研究也观察到Fab结合Env的倾斜,但解释为观察到的倾斜主要是Fab结合的结果gydF4y2Ba21gydF4y2Ba.相比之下,在这项工作完成后发表的一项关于原生HIV-1病毒粒子的冷冻et研究也发现,Env蛋白相对于病毒膜采用不同的倾斜角度gydF4y2Ba29gydF4y2Ba,从而独立地证实了我们的单粒子低温em和CGMD结果,即在没有结合抗体的情况下,Env蛋白经历倾斜运动。在这方面,可以想象HIV-1受体CD4及其共受体(CXCR4或CCR5)与gp120的正交对接协调gydF4y2Ba30.gydF4y2Ba这是gp160介导的融合所需的初始构象变化gydF4y2Ba31gydF4y2Ba,分别构成挑战减轻外域手势。gydF4y2Ba
也就是说,值得强调的是,在MD模拟中,三聚体位于病毒膜附近,在大多数分析中阻塞了三脚架。不到10%的聚集单粒子用于没有4E10 Fabs结合的纳米盘嵌入三聚体的3.9-Å分辨率图。不出所料,其他的太不均匀,不允许可视化的MPER三脚架,这一事实强调的概念,MPER在膜上是灵活的。这种认为MPER大部分被膜浸没的观点也与gp160外畴靠近膜形成10-Å爬行空间的观点一致。gydF4y2Ba1gydF4y2Ba)和独立的低温et数据gydF4y2Ba29gydF4y2Ba.这两个发现与另一项对aldrithol -2灭活病毒颗粒上的HIV-1 BaL Env三聚体的冷冻et研究得出的结果形成了对比,其中MPER似乎在Env外结构域和病毒膜之间形成了茎状连接gydF4y2Ba32gydF4y2Ba.造成这种差异的原因目前尚不清楚。gydF4y2Ba
先前对病毒分支的核磁共振研究发现了一对结构保守的病毒脂浸N-和c -螺旋,由一个铰链隔开,串联接头被从20,000多个病毒HIV-1毒株的序列分析中确定的必要螺旋盖残基锁定。通过丙氨酸取代这些帽而破坏两个关节,消除了cd4依赖性和cd4非依赖性菌株介导的偏液和融合gydF4y2Ba13gydF4y2Ba.通过在病毒膜上同时覆盖这些可移动的疏水MPER片段,减少了病毒膜的脱水,使其在物理上接近溶剂化的脂质双分子层以介导融合,从而减少了所需的高动力学屏障gydF4y2Ba31gydF4y2Ba,gydF4y2Ba33gydF4y2Ba.此外,MPER-TM片段本身诱导膜弯曲和脂质迁移,也参与膜的半融合和融合gydF4y2Ba33gydF4y2Ba.MPER的这些融合促进特性值得注意,因为与包括甲型流感在内的多种其他病毒相比,每个HIV-1病毒粒子的峰值数量要低一个数量级gydF4y2Ba34gydF4y2Ba.因为单个Fab的结合不仅影响结合的MPER的膜表面运动,还影响其他两个原聚体的膜表面运动(图2)。gydF4y2Ba5 dgydF4y2Ba和补充图。gydF4y2Ba11gydF4y2Ba),结合一个抗体或其Fab片段足以使三聚体失活gydF4y2Ba35gydF4y2Ba, MPER的这种改变可能对中和有重要的贡献。gydF4y2Ba
考虑到MPER在HIV-1融合的所有阶段的重要作用,病毒保护其三脚架结构免受免疫抗体攻击的基本原理是显而易见的。虽然尖刺倾斜和可能的CD4启动,允许短暂访问抗体Fab臂,其他病毒外畴位点的免疫优势是一种有效的转移策略,以减轻MPER靶向gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba.我们之前的MPER免疫原性研究表明,膜表面排列的MPER在体内具有免疫原性gydF4y2Ba37gydF4y2Ba,大多数疫苗诱导抗体的接近角度与外膜域施加的空间阻滞不相容。未来的疫苗策略是限制现有bnAbs所采用的抗体接近角度(图2)。gydF4y2Ba6 bgydF4y2Ba)在倾斜过程中绑定本地MPER。疫苗对两种RNA病毒(即HIV-1)的有效性存在巨大差异gydF4y2Ba与gydF4y2BaCoV-2与前者的高突变率和聚糖屏蔽密度有关gydF4y2Ba与gydF4y2Ba后者gydF4y2Ba38gydF4y2Ba.鉴于本文所定义的基本原理,针对HIV-1最保守和最易接近的位点似乎对开发针对HIV-1的保护性疫苗至关重要。gydF4y2Ba
方法gydF4y2Ba
抗体表达及纯化gydF4y2Ba
PG9 HRV3C IgG在Freestyle中表达gydF4y2BaTMgydF4y2Ba293-F细胞(赛默飞世尔科学公司)使用FreeStyle悬浮培养gydF4y2BaTMgydF4y2Ba293表达介质。自由式的1-L文化gydF4y2BaTMgydF4y2Ba使用PEI ' Max ' (Polysciences, Inc)以2:1 (w/w)的HC: LC PG9 DNA混合物转染293-F细胞。6天后,将细胞制成颗粒,上清液装入蛋白a - sepharose柱上。IgG用0.5 M乙酸洗脱,溶液用3 M Tris-HCl中和,pH为9.0,用磷酸盐缓冲盐水(PBS;8mm NagydF4y2Ba2gydF4y2BaHPOgydF4y2Ba4gydF4y2Ba, 2 mM KHgydF4y2Ba2gydF4y2Ba阿宝gydF4y2Ba4gydF4y2Ba, pH 7.4, 137 mM NaCl, 2.7 mM KCl)。gydF4y2Ba
4E10 IgG在Expi中表达gydF4y2BaTMgydF4y2Ba293-F细胞(赛默飞世尔科学公司)使用Expi悬浮培养gydF4y2BaTMgydF4y2Ba293表达介质。1-L的Expi文化gydF4y2BaTMgydF4y2Ba使用PEI ' Max ' (Polysciences, Inc),用HC: LC 4E10 DNA 1:1 (w/w)的混合物转染293-F细胞。4天后,将细胞制成颗粒,上清液装入蛋白G-Sepharose柱上。IgG用500 mM乙酸洗脱,溶液用3 M Tris-HCl中和,pH 9.0, PBS透析过夜。将4E10 IgG (15 mg/mL)在100 mM二硫苏糖醇中还原1小时,在37℃下制备4E10 Fab,然后在2 mM碘乙酰胺中烷基化48小时,在4℃下制备。用内源性蛋白酶Lys-C (0.01 μg/μL;罗氏应用科学公司)在25 mM Tris-HCl, pH 8.5,和1 mM EDTA中浸泡4小时,在37°C。1 mM Nα-终止解理反应gydF4y2BapgydF4y2Ba-tosyl- lysine氯甲基酮(TLCK)和0.4 mM leupeptin,并将裂解产物通过Protein a - sepharose柱去除Fc和完整的IgG。gydF4y2Ba
Gp145蛋白表达gydF4y2Ba
将HIV-1株BG505的Env蛋白残基1-723序列,包含3个稳定突变(Ala501Cys、Thr605Cys、Ile559Pro)、1个引入糖基化位点(Thr332Asn)和1个改进的糠醛裂解位点(REKR to RRRRRR),分别克隆到pEG BacMam表达载体中。质粒被转化成gydF4y2Ba大肠杆菌gydF4y2BaDH10Bac细胞产生bacmid DNA。经三轮病毒扩增得到重组杆状病毒gydF4y2BaSpodoptera frugiperdagydF4y2BaSf9细胞在Sf-900III SFM培养基中培养,27°C。蛋白表达方面,分别含有gp145和furin DNA的杆状病毒以2:1 (v/v)的比例混合,用于感染FreestylegydF4y2BaTMgydF4y2Ba293-F细胞的比例为1:10 (v/v)。感染24小时后,向培养物中加入最终浓度为10 mM的丁酸钠,培养物从37°C移动到30°C,并让其再生长48小时。收集细胞并在添加了5 mM EDTA和0.1% (w/v)牛血清白蛋白(Sigma Aldrich)的PBS中洗涤。gydF4y2Ba
gp145净化gydF4y2Ba
gp145三聚体根据已发表的方案进行纯化gydF4y2Ba39gydF4y2Ba稍作改动。自由泳gydF4y2BaTMgydF4y2Ba表达gp145的293-F细胞用PG9 IgG孵育至少3小时,PBS洗涤,并用含有50 mM Tris-HCl、pH 7.5、150 mM NaCl、0.5% Triton X-100和蛋白酶抑制剂鸡尾酒的裂解缓冲液溶解(Roche)。细胞裂解液在38,400 ×离心后gydF4y2BaggydF4y2Ba4℃下1 h, 4℃下用Protein A树脂(GenScript)孵育上清过夜。依次用10柱容积(CV)的洗涤缓冲液1 (50 mM Tris-HCl, pH 7.5, 150 mM NaCl, 0.03 mg/mL脱氧胆酸钠,0.1% w/v CHAPS)、10 CV的洗涤缓冲液2 (50 mM Tris-HCl, pH 7.5, 500 mM NaCl, 0.03 mg/mL脱氧胆酸钠,0.1% (w/v) n-十二烷基β- d -麦乳醇(DDM))和10 CV的洗涤缓冲液3 (50 mM Tris-HCl, pH 7.5, 150 mM NaCl, 0.03 mg/mL脱氧胆酸钠,0.1% DDM, 2 mM EDTA)洗涤树脂。将与Protein A树脂结合的PG9 IgG用3c蛋白酶在添加80mm l -半胱氨酸(Sigma Aldrich)的洗涤缓冲液3中消化到Fabs中,在4°C下消化4小时。收集洗脱液,用SEC缓冲液(50 mM Tris-HCl, pH 7.4, 150 mM NaCl, 0.03 mg/mL脱氧胆酸钠,0.05% DDM)进行5 CV洗涤。使用Amicon Ultra 15 ml 100-kDa切断离心过滤器(Millipore Sigma)浓缩蛋白质溶液,并使用SEC缓冲液在Superose 6尺寸排除柱(GE Healthcare)上运行。gydF4y2Ba
gp145重组成纳米盘gydF4y2Ba
本研究使用的脂类为脑极脂提取物、棕榈酰油酰磷脂酰胆碱(POPC)和棕榈酰油酰磷脂酰甘油(POPG),均购自Avanti polar lipids。脂质用20 mM胆酸钠在30 mM Tris-HCl, pH 7.5, 150 mM NaCl中超声溶解,并以1.07:1.5:1的摩尔比混合。将在DDM中纯化的gp145、支架蛋白MSP1D1dH5和脂质溶液按1:20:300的摩尔比混合,重组成纳米片。在冰上孵卵30分钟后,Bio-Beads SM-2 (Bio-Rad)被添加到样品体积的30%以去除洗涤剂。3 h后更换Bio-Beads。在4°C恒定旋转孵卵过夜后,通过重力使Bio-Beads沉淀,收集含有纳米盘嵌入gp145的上清液。gydF4y2Ba
用4E10 Fab修饰纳米盘式gp145gydF4y2Ba
将纳米盘嵌入gp145与4E10 Fab以1:100的摩尔比孵育至少3小时。样品使用Amicon Ultra 15-mL 50- kda截止离心过滤器(Millipore Sigma)进行浓缩,并加载到Superose 6尺寸排阻柱上,与50 mM HEPES、pH 7.4和150 mM NaCl平衡。将含有4E10 Fab修饰的纳米盘嵌入gp145的峰值组分合并。gydF4y2Ba
EM阴性染色分析gydF4y2Ba
样品的均匀性首先用所述0.7% (w/v)甲酸铀酰进行负染色EM评估gydF4y2Ba40gydF4y2Ba.为了计算负染EM平均值,使用Philips CM10电子显微镜(Philips)上的XR16L-ActiveVu电荷耦合器件相机(AMT)在加速电压为100 kV的情况下为每个样品收集100张图像。校准的放大倍率为×41,513 (×52,000的标称放大倍率),在标本水平上产生2.65 Å的像素大小。离焦设置为-1.5µm。使用EMAN2软件包的e2boxer.py命令为每个样本手动选择了大约10,000个粒子gydF4y2Ba41gydF4y2Ba并窗口成180 × 180像素的图像。经过图像归一化和粒子定心后,利用在SPIDER中实现的K-means分类程序将粒子图像分为100组gydF4y2Ba42gydF4y2Ba.gydF4y2Ba
低温电镜样品制备和数据收集gydF4y2Ba
gp145-4E10 Fab配合物与BS3以1:60的摩尔比在冰上交联30 min。交联终止时加入50 mM Tris, pH为7.5。样品用50 mM Tris, pH 7.5, 150 mM NaCl对缓冲液进行透析,以去除交联剂。样品的均匀性通过阴性染色EM检查。对于低温EM,样品浓度用NanoDrop分光光度计(赛默飞世尔科学公司)测量,并调整为0.2 mg/mL。等分4 μL应用于轻度发光放电的氧化石墨烯(GO)网格(GO on Quantifoil R1.2/1.3, Cu, 400目,电子显微镜科学),使用Vitrobot Mark VI (Thermo Fisher Scientific)设置为4°C和100%湿度。20s后,网格以−2的印迹力进行1 s的印迹,并浸入液氮冷却的乙烷中。Cryo-EM数据集在300 kv Titan Krios电子显微镜(Thermo Fisher Scientific)上收集,配备K2 Summit电子探测器(Gatan),名义放大倍率为×29,000,超分辨率计数模式。经过超过2 × 2像素的装箱,在样本层面上校准的像素大小为1.03 Å。照射时间为10秒,剂量分成40帧(每帧0.25秒),剂量率为8 egydF4y2Ba- - - - - -gydF4y2Ba/像素/秒,导致总剂量为80 egydF4y2Ba- - - - - -gydF4y2Ba/一个gydF4y2Ba2gydF4y2Ba.数据收集使用SerialEMgydF4y2Ba43gydF4y2Ba在“超快模式”下,在将舞台移动到下一个位置之前,使用光束倾斜和图像移动来曝光3 × 3个孔gydF4y2Ba44gydF4y2Ba.离焦范围为−1.5 ~−2.5 μm。数据采集参数汇总见“补充表”gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
低温电子显微镜数据处理gydF4y2Ba
来自五个数据收集会话的30,404个电影堆栈被增益标准化,运动校正,剂量加权,并在MotionCorr2中以2 × 2像素为单位进行分组gydF4y2Ba45gydF4y2Ba.对比剂传递函数(CTF)参数由CTFFIND4(参考文献)估计。gydF4y2Ba46gydF4y2Ba)在RELION-3中实现gydF4y2Ba47gydF4y2Ba.粒子自动挑选没有模板与Gautomatch (gydF4y2Bahttp://www.mrc-lmb.cam.ac.uk/kzhang/Gautomatch/gydF4y2Ba),提取成单独的图像,在RELION-3中归一化并进行二维分类,分为100个类。具有良好2D平均值的类被组合在一起,产生3,039,896个粒子。gydF4y2Ba
gp145外畴密度的支配使4E10 Fabs结合粒子的鉴定变得复杂。因此,在cryoSPARC v2中使用从头算算法,只选择了2D类中显示清晰侧视图的粒子,并用于生成四个初始模型gydF4y2Ba48gydF4y2Ba.从头开始重建生成了一张图,显示了一个具有三个4E10晶圆的外畴的清晰密度,一张图显示了仅对外畴的强密度,以及两张“垃圾”图。前两个图和一个垃圾图被用作初始模型,在cryoSPARC v2中运行两轮异构细化,结果是362,646个粒子的堆栈显示了三个结合4E10晶圆的清晰密度,1,8878,941个粒子仅显示了外畴的清晰密度。gydF4y2Ba
在cryoSPARC v2中,362,646个颗粒显示出三个结合4E10晶圆的清晰密度,随后进行了均匀和不均匀的细化gydF4y2Ba49gydF4y2Ba,得到全局分辨率为3.66的最终地图Å(补充图。gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
18878,941个仅在外膜域显示清晰密度的颗粒首先在RELION-3中进行监督分类,使用Chimera中Segger工具生成的两个参考图,这些参考图来自菌株AMC011的纳米盘嵌入的全长HIV-1 Env蛋白的冷冻电镜图,其中结合了PGT151 Fab和10E8 Fab (EMDB 21332)。gydF4y2Ba21gydF4y2Ba.一个参考图只包含外畴和纳米盘的密度,而第二个参考图也包含10E8 Fab的密度。通过这种分类确定的具有Fab结合的224,730个颗粒被进一步进行3D分类。用于净化的PG9 Fab的密度总是被掩盖。在第一次3D分类为8个类别后,将四个显示出清晰Fab密度的类别组合在一起,并进行第二轮3D分类为4个类别。结果显示,其中两个类具有一个4E10 Fab结合的外畴。这两个类别被结合起来,并接受了进一步的两轮3D分类,分为四个类别。包含23,538个粒子的最终地图通过后处理进行锐化,以产生全局分辨率为8.8 Å (gp145•1Fab)的密度地图(补充图)。gydF4y2Ba4gydF4y2Ba).初步三维分类为四类的一类显示了一个外畴与两个4E10 fab结合。这个类包含21,454个粒子,通过后处理,地图被锐化,以产生一个全局分辨率为8.2的地图Å (gp145•2Fab)(补充图。gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
剩余的1,654,211个粒子从最初的监督分类中显示4E10 Fab没有密度,使用两张有和没有纳米盘密度的Env图作为参考,进行了进一步的监督分类。对1153,408个具有纳米盘密度的颗粒进行了5轮三维分类,在每一步分类中,选择外域和纳米盘密度均明确的类别进行进一步分类。最后,对MPER区域中密度最好的两类粒子47,616个进行组合和细化,然后进行CTF细化和贝叶斯抛光gydF4y2Ba50gydF4y2Ba.生成的密度图通过后处理进行锐化,以获得全局分辨率为3.9的最终地图Å(补充图。gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
电影一代gydF4y2Ba
说明外域可以在纳米圆盘表面上假设的方向范围的电影是由实验地图生成的。这些地图是从1,654,211个粒子的连续几轮3D分类中选择的,这些粒子没有4E10 Fab的密度。图的选择是基于具有1000个或更少的粒子数(以识别尽可能多的不同方向)以及具有明确定义的纳米盘密度(以能够确定相对于纳米盘的外畴方向)(补充图。gydF4y2Ba3.gydF4y2Ba).这些标准产生了20张地图,用于生成《奇美拉》中的电影gydF4y2Ba51gydF4y2Ba.gydF4y2Ba
模型构建和细化gydF4y2Ba
仅gp145, Env外结构域的晶体结构(残基31-662,PDB: 5I8H)gydF4y2Ba52gydF4y2BaMPER肽n段的核磁共振结构(残基663 ~ 671,PDB: 2PV6)gydF4y2Ba14gydF4y2Ba在Chimera中使用“fit in map”命令手动对接到gp145密度图。在合并链后,在Coot中手动调整模型gydF4y2Ba53gydF4y2Ba并使用phenix.real_space_refine对密度进行细化gydF4y2Ba54gydF4y2Ba始终保持几何约束和拉玛钱德兰约束。在去除密度不明确区域的侧链后,最终模型包含残基331-662的主链和侧链信息,而残基663-671只包含主链折叠。gydF4y2Ba
对于gp145•3Fab配合物,Env外畴的晶体结构(残基31-662,PDB: 5I8H)gydF4y2Ba52gydF4y2Ba, MPER肽n段的核磁共振结构(残基663-671,PDB: 2PV6)gydF4y2Ba14gydF4y2Ba以及与MPER-C肽配合物中的4E10 Fab(残基672-684,PDB: 4XC3)gydF4y2Ba26gydF4y2Ba在Chimera中使用“fit in map”命令手动对接到gp145•3Fab密度图。在合并链后,在白骨顶中手动调整模型,并使用凤凰对密度进行细化。real_space_refine始终保持几何和Ramachandran约束。去除密度不明确区域的侧链后,最终模型包含31-662残基的主链和侧链信息,663-684残基只包含主链折叠(除了B链上678残基和D链上664、671和674残基保留侧链外)。gydF4y2Ba
根据半映射1对模型进行细化,然后计算细化后的模型与半映射1(工作)、半映射2(自由)和组合映射(补充图)之间的FSC曲线。gydF4y2Ba4 c, egydF4y2Ba).gydF4y2Ba
粗粒度分子动力学模拟gydF4y2Ba
为了模拟非配位gp145,通过将外膜域(本研究)的低温em结构与mper跨膜域的核磁共振结构(包括细胞质域的前7个残基(PDB: 6DLN)连接起来建立了模型。将蛋白质以4:1的摩尔比置于由POPC和胆固醇组成的脂质双分子层中,并使用crazy .py脚本在150 mM NaCl中进行溶剂化gydF4y2Ba55gydF4y2Ba.gydF4y2Ba
粗粒度模拟使用Martinize2 (gydF4y2Bahttps://github.com/marrink-lab/vermouth-martinizegydF4y2Ba)和最新的MARTINI3力场模型gydF4y2Ba56gydF4y2Ba.将弹性网络应用于CG模型,力常数为500 kJ mol,保留了四阶和三级结构gydF4y2Ba−1gydF4y2Ba纳米gydF4y2Ba−2gydF4y2Ba,上下截止距离分别为0.9 nm和0.5 nmgydF4y2Ba57gydF4y2Ba.所有模拟都是在GROMACS 2020.6(参考文献。gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba)和蛙跳算法gydF4y2Ba60gydF4y2Ba是用来积分牛顿运动方程与20秒的时间步长。使用Verlet邻居搜索截断方案进行模拟,每20步更新一次gydF4y2Ba61gydF4y2Ba.静电相互作用采用反应场(Reaction-Field)计算,库仑截止距离为1.1 nmgydF4y2Ba62gydF4y2Ba.使用速度缩放恒温器将温度保持在310kgydF4y2Ba63gydF4y2Ba.使用Berendsen进行了平衡和生产模拟的压力耦合gydF4y2Ba64gydF4y2Ba以及Parrinello-Rahman气压计gydF4y2Ba65gydF4y2Ba,分别。从唯一的随机种子开始,以4.75 ns的平衡相和10 μs的生产运行进行了5次模拟。gydF4y2Ba
对于gp145在与4E10 Fab配合物中的模拟,从未配位的gp145模拟中获得快照,显示出与4E10 Fab结合的gp145的冷冻- em模型中相似的MPER-C构象。使用back .py脚本将这个粗粒度快照转换为全原子视图gydF4y2Ba66gydF4y2BaCHARMM-GUI全原子转换器gydF4y2Ba67gydF4y2Ba.由此得到的原子模型被用于对接4E10 Fab (PDB: 4XC3)的结构,由Fab结合的MPER-C肽引导,并进行了微小的调整,以避免与gp145外位域的空间冲突。将蛋白质以4:1的摩尔比置于由POPC和胆固醇组成的脂质双分子层中,并使用crazy .py脚本在150 mM NaCl中进行溶剂化。gydF4y2Ba
粗粒度的模拟被设置并运行为非配位的gp145。采用与上述参数相同的弹性网络。弹性网络相互作用也应用于4E10 Fab和MPER-C段之间,以保持结合相互作用。从唯一的随机种子开始,进行了5次模拟,平衡阶段为6.75 ns,每次生产运行1 μs。gydF4y2Ba
仿真轨迹采用gydF4y2Bagmx trjconv。gmx笨拙的移动gydF4y2Ba用于测量gp145外畴与MPER-C段的夹角。使用VMD v.1.9.4a12对轨迹进行可视化。gydF4y2Ba68gydF4y2Ba)和图表是用微软Excel制作的。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
支持本研究的数据可根据合理要求从通讯作者处获得。低温em图已按登录代码存入电子显微镜数据库(EMDB)gydF4y2Baemd - 25022gydF4y2Ba(gp145),gydF4y2Baemd - 25024gydF4y2Ba(gp145•1工厂),gydF4y2Baemd - 25025gydF4y2Ba(gp145•2工厂),gydF4y2Baemd - 25045gydF4y2Ba(gp145•3工厂)。原子坐标已根据登录代码存入蛋白质数据库(PDB)gydF4y2Ba7 sc5gydF4y2Ba(gp145)和gydF4y2BaPDB-7SD3gydF4y2Ba(gp145•3工厂)。以前报告的PDB接入代码如下:gydF4y2Ba1 tzggydF4y2Ba(4 e10工厂/ mp),gydF4y2Ba2 pv6gydF4y2Ba(MPER肽n段,NMR);gydF4y2Ba4 u6ggydF4y2Ba(10 e8工厂/ mp);gydF4y2Ba4 xc3gydF4y2Ba(4E10 Fab/MPER-C肽);gydF4y2Ba5 i8hgydF4y2Ba(Env ectodomain);gydF4y2Ba5 u3ngydF4y2Ba(DH511工厂/ mp);gydF4y2Ba6 dlngydF4y2Ba(mp三脚架);gydF4y2Ba6 e8wgydF4y2Ba(mp stalk-bubble);gydF4y2Ba6 o3jgydF4y2Ba(PGZL1工厂/ mp)。以前报告的EMDB接入代码如下:gydF4y2Baemdb - 21332gydF4y2Ba(AMC011与绑定PGT151 Fab和10E8 Fab)和gydF4y2Baemdb - 21334gydF4y2Ba(AMC011与绑定10E8 Fab)。gydF4y2Ba
参考文献gydF4y2Ba
曼恩,j.m.。gydF4y2Ba世界艾滋病gydF4y2Ba(哈佛大学出版社,1992)。gydF4y2Ba
夏普,P. M. & Hahn, B. H. HIV-1的进化和艾滋病的起源。gydF4y2Ba费罗斯。反式。r . Soc。Lond。医学杂志。科学。gydF4y2Ba365gydF4y2Ba, 2487-2494(2010)。gydF4y2Ba
2021年联合国艾滋病规划署全球艾滋病最新情况——对抗不平等——40年艾滋病防治工作的经验教训。gydF4y2Bahttps://www.unaids.org/en/resources/documents/2021/2021-global-aids-updategydF4y2Ba(2021)。gydF4y2Ba
Kwong, P. D. & Mascola, J. R.基于抗体鉴定、B细胞个体发育和表位结构的HIV-1疫苗。gydF4y2Ba免疫力gydF4y2Ba48gydF4y2Ba, 855-871(2018)。gydF4y2Ba
Mascola, J. R. & Haynes, B. F. HIV-1中和抗体:了解自然途径。gydF4y2BaImmunol。牧师。gydF4y2Ba254gydF4y2Ba, 225-244(2013)。gydF4y2Ba
Sok, D. & Burton, D. R.广泛中和HIV抗体的最新进展。gydF4y2BaImmunol Nat。gydF4y2Ba19gydF4y2Ba, 1179-1188(2018)。gydF4y2Ba
海恩斯,B. F.伯顿,D. R.和马斯科拉,J. R.艾滋病毒广泛中和抗体的多重作用。gydF4y2Ba科学。Transl。地中海。gydF4y2Ba11gydF4y2Ba, eaaz2686(2019)。gydF4y2Ba
斯塔奇,B. R.等。艾滋病逆转录病毒HTLV-III/LAV包膜基因保守和可变区域的鉴定和特征。gydF4y2Ba细胞gydF4y2Ba45gydF4y2Ba, 637-648(1986)。gydF4y2Ba
Berndsen, Z. T.等。HIV-1环境聚糖盾跨尺度的可视化。gydF4y2Ba国家科学院学报美国gydF4y2Ba117gydF4y2Ba, 28014-28025(2020)。gydF4y2Ba
魏,X.等。HIV-1的抗体中和和逃逸。gydF4y2Ba自然gydF4y2Ba422gydF4y2Ba, 307-312(2003)。gydF4y2Ba
邝普德等。HIV-1通过构象掩蔽受体结合位点来逃避抗体介导的中和作用。gydF4y2Ba自然gydF4y2Ba420gydF4y2Ba, 678-682(2002)。gydF4y2Ba
Bonsignori, M.等。从种系到CD4-mimic抗体的广泛HIV-1中和剂的成熟途径。gydF4y2Ba细胞gydF4y2Ba165gydF4y2Ba, 449-463(2016)。gydF4y2Ba
太阳,Z.-Y。J.等。螺旋盖残基671和674的破坏揭示了HIV-1进入gp41膜近端外部区域的一个特殊铰链段的作用。gydF4y2BaJ. Mol.生物学。gydF4y2Ba426gydF4y2Ba, 1095-1108(2014)。gydF4y2Ba
太阳,Z.-Y。J.等。HIV-1广泛中和抗体从病毒膜上弯曲的gp41外畴区域提取其表位。gydF4y2Ba免疫力gydF4y2Ba28gydF4y2Ba, 52-63(2008)。gydF4y2Ba
宋,L.等。广泛中和抗hiv -1抗体破坏gp41在膜界面的铰链相关功能。gydF4y2Ba国家科学院学报美国gydF4y2Ba106gydF4y2Ba, 9057-9062(2009)。gydF4y2Ba
Zhu, P., Winkler, H., Chertova, E., Taylor, K. A. & Roux, K. H. HIV-1包膜刺突的低温电子断层扫描:三脚架样腿的进一步证据。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba4gydF4y2Ba, e1000203(2008)。gydF4y2Ba
萨内蒂,G., Briggs, J. A. G., Grünewald, K., Sattentau, Q. J. & Fuller, S. D.原位免疫缺陷病毒包膜复合物的冷冻电子层析结构。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba2gydF4y2Ba, e83(2006)。gydF4y2Ba
怀特,t.a.等。完整病毒上三聚体SIV和HIV-1包膜糖蛋白的分子结构:菌株依赖的四阶结构变异。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba6gydF4y2Ba, e1001249(2010)。gydF4y2Ba
朱利安,j。et al。可溶性裂解HIV-1包膜三聚体的晶体结构。gydF4y2Ba科学gydF4y2Ba342gydF4y2Ba, 1477-1483(2013)。gydF4y2Ba
Brügger, B.等。HIV脂质组:一种不同寻常的组成。gydF4y2Ba国家科学院学报美国gydF4y2Ba103gydF4y2Ba, 2641-2646(2006)。gydF4y2Ba
Rantalainen, K.等。脂质组合中的HIV-1包膜和MPER抗体结构。gydF4y2Ba细胞的代表。gydF4y2Ba31gydF4y2Ba, 107583(2020)。gydF4y2Ba
傅,Q.等。HIV-1包膜糖蛋白的膜近端外区结构。gydF4y2Ba国家科学院学报美国gydF4y2Ba115gydF4y2Ba, e8892-e8899(2018)。gydF4y2Ba
Kwon B., Lee M., Waring, A. J. & Hong M. HIV gp41膜的寡聚结构和三维折叠-磷脂双分子层的近端外区域和跨膜域。gydF4y2Baj。化学。Soc。gydF4y2Ba140gydF4y2Ba, 8246-8259(2018)。gydF4y2Ba
王毅,等。与亚稳态HIV-1包膜预融合状态相关的脂质双分子层上gp41 MPER的拓扑分析。gydF4y2Ba国家科学院学报美国gydF4y2Ba116gydF4y2Ba, 22556-22566(2019)。gydF4y2Ba
卡多索,r.m.f.等。广泛中和抗hiv抗体4E10识别gp41中高度保守的融合相关基序的螺旋构象。gydF4y2Ba免疫力gydF4y2Ba22gydF4y2Ba, 163-173(2005)。gydF4y2Ba
Irimia, A., Sarkar, A., Stanfield, R. L. & Wilson, I. A.脂质作为HIV广泛中和抗体4E10表位的组成部分的晶体学鉴定。gydF4y2Ba免疫力gydF4y2Ba44gydF4y2Ba, 21-31(2016)。gydF4y2Ba
Irimia, A.等人。脂质相互作用和接近广泛中和抗体10E8的HIV-1病毒膜的角度:对疫苗和治疗设计的见解gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba13gydF4y2Ba, e1006212(2017)。gydF4y2Ba
孙瑞娥,闵东,王金玉,王金玉。单分子方法在膜蛋白折叠研究中的应用。gydF4y2BaJ. Mol.生物学。gydF4y2Ba430gydF4y2Ba, 424-437(2018)。gydF4y2Ba
Mangala Prasad, V.等。Env对完整HIV病毒粒子的Cryo-ET揭示了Gag晶格上的结构变化和位置。gydF4y2Ba细胞gydF4y2Ba185gydF4y2Ba, 641 - 653。e17(2022).
Shaik, m.m.等人。HIV-1包膜峰识别共受体的结构基础。gydF4y2Ba自然gydF4y2Ba565gydF4y2Ba, 318-323(2019)。gydF4y2Ba
哈里森,S. C.病毒膜融合。gydF4y2Ba病毒学gydF4y2Ba479 - 480gydF4y2Ba, 498-507(2015)。gydF4y2Ba
李,Z.等。aldrithol -2灭活病毒颗粒上HIV-1包膜三聚体的亚纳米结构gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba27gydF4y2Ba, 726-734(2020)。gydF4y2Ba
Sutherland, M. Kwon, B. & Hong, M. HIV gp41膜-近端外区域和跨膜域与磷脂膜的相互作用。gydF4y2BaBiochim。Biophys。生物化学学报。gydF4y2Ba1863gydF4y2Ba, 183723(2021)。gydF4y2Ba
朱,P.等。艾滋病病毒包膜刺突的分布和三维结构。gydF4y2Ba自然gydF4y2Ba441gydF4y2Ba, 847-852(2006)。gydF4y2Ba
杨晓霞,杨晓霞,李晓霞,李晓霞,杨晓霞。人免疫缺陷病毒1型抗体中和的化学计量学研究。gydF4y2Baj .性研究gydF4y2Ba.gydF4y2Bahttps://doi.org/10.1128/JVI.79.6.3500-3508.2005gydF4y2Ba(2005)。gydF4y2Ba
纳尔逊,J. D.等。一种针对人类免疫缺陷病毒1型gp41膜近端外部区域的亲和增强中和抗体识别2F5和4E10之间的表位。gydF4y2Baj .性研究。gydF4y2Ba81gydF4y2Ba, 4033-4043(2007)。gydF4y2Ba
Kim, M.等人。膜结合hiv - 1gp41膜-近端外区(MPER)片段的免疫原性受残基可达性支配,并受立体化学调控。gydF4y2Ba生物。化学。gydF4y2Ba288gydF4y2Ba, 31888-31901(2013)。gydF4y2Ba
费舍尔,W.等人。HIV-1和SARS-CoV-2:两种大流行病原体的进化模式gydF4y2Ba细胞宿主微生物gydF4y2Ba29gydF4y2Ba, 1093-1110(2021)。gydF4y2Ba
Lee, J. H., Ozorowski, G. & Ward, a . B.原生、完全糖基化、裂解的HIV-1包膜三聚体的Cryo-EM结构。gydF4y2Ba科学gydF4y2Ba351gydF4y2Ba, 1043-1048(2016)。gydF4y2Ba
李勇,程勇,李勇,郑勇和Walz, T.阴性染色和图像分类-现代电子显微镜的强大工具。gydF4y2Ba医学杂志。过程。在线gydF4y2Ba6gydF4y2Ba, 23-34(2004)。gydF4y2Ba
唐,G.等。EMAN2:用于电子显微镜的可扩展图像处理套件。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba157gydF4y2Ba, 38-46(2007)。gydF4y2Ba
弗兰克,J.等。蜘蛛和WEB:三维电子显微镜及相关领域的图像处理和可视化。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba116gydF4y2Ba, 190-199(1996)。gydF4y2Ba
自动电子显微镜断层扫描使用样本运动的稳健预测。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba152gydF4y2Ba, 36-51(2005)。gydF4y2Ba
Cheng, A.等。高分辨率单粒子冷冻电子显微镜使用光束图像移位。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba204gydF4y2Ba, 270-275(2018)。gydF4y2Ba
郑世强等。MotionCor2:改进冷冻电子显微镜光束诱导运动的各向异性校正。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 331-332(2017)。gydF4y2Ba
Rohou, A. & Grigorieff, N. CTFFIND4:快速准确的电子显微图像离焦估计。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba192gydF4y2Ba, 216-221(2015)。gydF4y2Ba
Zivanov, J.等人。在RELION-3中自动高分辨率冷冻- em结构测定的新工具。gydF4y2BaeLifegydF4y2Ba7gydF4y2Ba, e42166(2018)。gydF4y2Ba
Punjani, A., Rubinstein, J. L., Fleet, D. J. & Brubaker, M. A. cryoSPARC:快速无监督冷冻em结构确定的算法。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 290-296(2017)。gydF4y2Ba
彭贾尼,A.,张,H.和Fleet, D. J.非均匀细化:自适应正则化改进单粒子低温em重建。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 1214-1221(2020)。gydF4y2Ba
Zivanov, J., Nakane, T. & Scheres, S. H. W.在低温em单粒子分析中光束诱导运动校正的贝叶斯方法。gydF4y2BaIUCrJgydF4y2Ba6gydF4y2Ba, 5-17(2019)。gydF4y2Ba
彼得森,E. F.等人。UCSF chimera -用于探索性研究和分析的可视化系统。gydF4y2Baj .第一版。化学。gydF4y2Ba25gydF4y2Ba, 1605-1612(2004)。gydF4y2Ba
Kong, R.等人。HIV-1融合肽是中和抗体的易感位点。gydF4y2Ba科学gydF4y2Ba352gydF4y2Ba, 828-833(2016)。gydF4y2Ba
埃姆斯利,P. &考坦,K.库特:分子图形模型构建工具。gydF4y2BaActa Crystallogr。d杂志。Crystallogr。gydF4y2Ba60gydF4y2Ba, 2126-2132(2004)。gydF4y2Ba
亚当斯,P. D.等。PHENIX:一个全面的基于python的大分子结构解决方案系统。gydF4y2BaActa Crystallogr。d杂志。Crystallogr。gydF4y2Ba66gydF4y2Ba, 213-221(2010)。gydF4y2Ba
Wassenaar, T. A., Ingólfsson, H. I., Böckmann, R. A., Tieleman, D. P. & Marrink, S. J.计算脂质组学与疯狂:生成自定义膜分子模拟的多功能工具。gydF4y2Baj .化学。理论第一版。gydF4y2Ba11gydF4y2Ba, 2144-2155(2015)。gydF4y2Ba
Souza, p.c.t.等。Martini 3:粗粒度分子动力学的通用力场。gydF4y2BaNat方法。gydF4y2Ba18gydF4y2Ba, 382-388(2021)。gydF4y2Ba
佩里奥尔,X,卡沃利,M,马林克,s - j。& Ceruso, m.a.结合弹性网络与粗粒度分子力场:结构、动力学和分子间识别。gydF4y2Baj .化学。理论第一版。gydF4y2Ba5gydF4y2Ba, 2531-2543(2009)。gydF4y2Ba
Van Der Spoel, D.等。GROMACS:快速、灵活、免费。gydF4y2Baj .第一版。化学。gydF4y2Ba26gydF4y2Ba, 1701-1718(2005)。gydF4y2Ba
亚伯拉罕,m.j.等。GROMACS:通过从笔记本电脑到超级计算机的多级并行进行高性能分子模拟。gydF4y2BaSoftwareXgydF4y2Ba1 - 2gydF4y2Ba, 19-25(2015)。gydF4y2Ba
霍克尼,R. W.戈埃尔,S. P. &伊斯特伍德,J. W.等离子体的安静高分辨率计算机模型。gydF4y2Baj .第一版。理论物理。gydF4y2Ba14gydF4y2Ba, 148-158(1974)。gydF4y2Ba
经典流体的计算机“实验”。一、Lennard-Jones分子的热力学性质。gydF4y2Ba理论物理。牧师。gydF4y2Ba159gydF4y2Ba, 98-103(1967)。gydF4y2Ba
李国强,史培博,史培博,史培强,史培强。一种用于分子动力学模拟的广义反应场方法。gydF4y2Baj .化学。理论物理。gydF4y2Ba102gydF4y2Ba, 5451-5459(1995)。gydF4y2Ba
Bussi, G., Donadio, D. & Parrinello, M.通过速度重尺度的规范采样。gydF4y2Baj .化学。理论物理。gydF4y2Ba126gydF4y2Ba, 014101(2007)。gydF4y2Ba
李文杰,李文杰,李文杰,李文杰。分子动力学与外浴耦合的研究。gydF4y2Baj .化学。理论物理。gydF4y2Ba81gydF4y2Ba, 3684-3690(1984)。gydF4y2Ba
单晶的多态转变:一种新的分子动力学方法。gydF4y2Baj:。理论物理。gydF4y2Ba52gydF4y2Ba, 7182-7190(1981)。gydF4y2Ba
Wassenaar, t.a., Pluhackova, K, Böckmann, r.a., Marrink, s.j. & Tieleman, d.p.向后走:从粗粒度模型到原子模型的反向转换的灵活几何方法。gydF4y2Baj .化学。理论第一版。gydF4y2Ba10gydF4y2Ba, 676-690(2014)。gydF4y2Ba
Jo, S., Kim, T., Iyer, V. G. & Im, W. CHARMM- gui: CHARMM基于web的图形用户界面。gydF4y2Baj .第一版。化学。gydF4y2Ba29gydF4y2Ba, 1859-1865(2008)。gydF4y2Ba
汉弗莱,W., Dalke, A. & Schulten, K. VMD:视觉分子动力学。gydF4y2BaJ. Mol. Graph。gydF4y2Ba14gydF4y2Ba, 33-38(1996)。gydF4y2Ba
确认gydF4y2Ba
我们感谢洛克菲勒大学Evelyn Gruss Lipper Cryo-EM资源中心的M. Ebrahim, J. Sotiris和H. Ng在低温电镜数据收集方面的帮助。我们感谢P. Cesar Telles de Souza博士为我们提供了胆固醇的MARTINI3 beta拓扑文件。这项工作由美国国立卫生研究院资助P01 AI126901 (E.L.R, T.W.)。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
E.L.R.和T.W.构想了这项研究。S.Y, g.h., M.K, E.L.R和T.W.设计了实验。S.Y, Y.W, J.C.制备了抗体fab。S.Y.完成了所有的生物化学和结构生物学实验。G.H.进行了所有的分子动力学实验。j.h.w., m.k., E.L.R和T.W.监督实验。所有作者都对结果进行了分析。S.Y, E.L.R和T.W.写了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然通讯gydF4y2Ba感谢Tyler Reddy和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
杨,S, Hiotis, G,王,Y。gydF4y2Baet al。gydF4y2Ba动态的HIV-1尖刺运动使其膜结合的三脚架容易受到抗体攻击。gydF4y2BaNat CommungydF4y2Ba13gydF4y2Ba, 6393(2022)。https://doi.org/10.1038/s41467-022-34008-ygydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41467-022-34008-ygydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba