摘要
儿科室管膜瘤(EPN)常复发。我们对13年(5.8±3.8)年间30例患者匹配的重复复发(3.67±1.76次)进行了纵向综合分析,结果显示,在连续复发期间,稳定的分子亚型(RELA和PFA)和收敛的DNA甲基化重编程伴随着晚期复发中原位患者源性异种移植(PDX)的增加(13/27)。一组差异甲基化CpGs (DMCs)和DNA甲基化区域(DMRs)被发现持续存在于原发性和复发性肿瘤(潜在的驱动型DMCs)中,并且仅在复发中获得(潜在的促进型DMCs)。整合RNAseq揭示潜在驱动因子DMRs调控的差异表达基因(Cacna1h, slc12a7, rara在RELA和Hspb8, gmpr, itgb4)和潜在的促进剂DMRs (PLEKHG1在RELA和Notch, epha2, sufu, foxj1在PFA肿瘤中)。在原发肿瘤中也发现了DMCs复发的预测因子。本研究提供了一系列EPN复发的高分辨率表观遗传学路线图和13个原位PDX模型,以促进生物学和临床前研究。
简介
室管膜瘤(EPN)是儿童第三大常见恶性脑肿瘤,占儿童颅内肿瘤的12%。目前的治疗包括最大限度的手术切除和局灶放疗,5年总生存率(OS)和无进展生存率(PFS)分别为70%和57%1,2,3.,4,5.然而,近一半的患者会经历晚期复发1,3.,4.尽管反复治疗,大多数复发的肿瘤儿童最终还是死于这种疾病。10年OS和PFS分别下降到50±5%和29±5%1.化疗药物用于EPN已被广泛研究了几十年,然而,生存益处仍然存在争议6,7.需要深入了解EPN复发的生物学。
而肿瘤的位置在EPN生物学中是很重要的,这一点已经得到了证实8最近的研究表明,DNA甲基化介导的表观遗传改变在EPN肿瘤发生中起着重要作用9,10,11因为EPN中的基因突变比成人癌症中的基因突变要少得多9,10,11.事实上,非突变的表观遗传重编程最近已被纳入癌症的一个新标志12.和其他几种儿科癌症相似13,14,15, DNA甲基化分析已成功地将儿童epn细分为9个具有不同临床结果的分子亚群16,17.虽然原发性EPN的致癌驱动因素已被确定8,11,15,16,18,19,20.,21,22,23在美国,对复发性EPN生物学的理解仍处于起步阶段。其中一个挑战是难以获得复发的肿瘤组织。尽管对连续、连续复发的患者肿瘤样本进行纵向分析将能够从短暂的改变中分离出连续保守的潜在表观遗传驱动因素,但这仍然非常困难,需要由医生、神经病理学家和肿瘤生物学家组成的坚定合作团队。使用单个复发性EPN肿瘤伴或不伴患者匹配的原发肿瘤是一种常见的方法。第二个挑战在于可靠地检测细微的遗传/表观遗传变化。最近的一项研究表明,450k DNA甲基化阵列检测到的表观遗传变化不能充分解释复发性epn的形态变化24,结合基因表达模式的综合分析,提高了DNA甲基化位点的覆盖率9,10,25,26,27,28应该有助于发现基因相互作用和新的治疗机会。
研究EPN复发的第三个挑战是动物模型有限。在本课题组建立首个幕上EPN患者源性原位异种移植(PDOX,或orthotopic PDX)模型后29我们和其他人已经证明,将患者肿瘤直接植入小鼠大脑的匹配位置,促进了临床相关动物模型的建立,这些模型复制了原始患者的组织病理学特征、侵袭性/转移表型和遗传特征29,30.,31,32,33,34,35.虽然已经开发了几种基因工程动物模型22,23,36在肿瘤进展过程中,即每个患者从诊断到连续、连续复发,相关性EPN肿瘤的致瘤性,应作为确定关键致瘤驱动因素和确定EPN复发的可药靶点的体内功能测定。
在这里,我们报告了一项对30例患者匹配的原发性和复发EPN肿瘤的DNA甲基化情况进行深度纵向分析,并对这组匹配的原发性和复发EPN进行RNA-seq谱综合分析和PDOX致瘤性功能检查。我们的目标是应用这种综合分析和来自同一队列患者的临床相关患者匹配PDOX小鼠模型,以确定复发的潜在驱动因素(原发肿瘤中存在异常DNA甲基化,并在所有复发的epn中持续存在),复发的潜在促进因素(复发肿瘤中新获得的DNA甲基化,并随着肿瘤进展持续存在),以及原发肿瘤复发的潜在预测因素(原发肿瘤中的DNA甲基化预测未来/随后的复发)。
结果
EPN连续复发期间分子亚型的维持
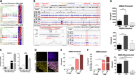
儿科EPN在诊断时的不同分子亚型会影响临床治疗13,14,16,37,38.虽然对单次复发的分析表明分子亚型的维持,但仍有待确定epn的分子亚型是否会在长时间的重复复发中发生变化。尽管50%的EPN患者会发生肿瘤复发1,3.,4在美国,复发的肿瘤样本,特别是复发的肿瘤样本很难获得。在我们的研究中,我们从110例(9.1%)儿科患者中收集了30例连续复发的EPN肿瘤样本,这些患者被随访了13年以上。已获得同意发布可能识别个人的临床信息。在患者接受化疗和/或放疗治疗后,获得了许多复发的肿瘤样本。1).从诊断到最后一次复发的时间为2.75年至13年,每例患者复发次数为1 ~ 7次(3.67±1.76次)。1、表1).
一个10例复发室管膜瘤的手术、放化疗治疗及致瘤性资料的时间过程总结。小脑内图像(银行独立委员会)和脑内(集成电路)原发原位异种移植瘤形成(p)或循环(r)插入PFA和RELA肿瘤(圆形括号内为模型ID),显示肿瘤(T)、脑积水(*)和CSF扩散。B原位移植复发性PFA epn小鼠模型。在大体(上图)的小鼠大脑(轮廓)上可以看到肿瘤形成。H&E染色(中)显示原发患者肿瘤与PDOX肿瘤的组织学比较。小脑内连续体内移植过程中动物生存时间的变化(下图)银行独立委员会) PDOX模型来自文章I (我)到V (V)以及两种PDOX模型中植入不同细胞数量(从每只小鼠1000个细胞到10万个细胞)对动物存活时间的影响(n= 10只/组)。比例尺= 50µM。C热图显示所有EPN肿瘤样本中最高变量20,000 CpGs的DNA甲基化比率。患者ID被垂直标记在热图的顶部。患者id下的黑色和灰色条用于区分同一患者的肿瘤。D在RELA(红圈)和PFA(蓝圈)肿瘤中,使用前6000个具有可变DNA甲基化比例的CpGs构建系统发育树。E以年龄匹配的儿童正常脑组织为参考,对EPN肿瘤样本RNA-seq数据进行主成分分析。F热图代表PFA(左))以及RELA(右)原发肿瘤特征基因在所有样本中的表达水平,包括多次复发。主特征基因从先前发表的数据库GSE64415中选择16,39,88,那些与我们持续减少(绿色)和增加(红色)的基因重叠的基因被突出显示。G与正常儿童小脑和脑组织相比,PFA(左)和RELA(下)原发性和复发性肿瘤中的CA甲基化比率。箱线图表示中位数,第一和第三四分位数(Q1和Q3),胡须延伸到最远的值;最上面一行和最下面一行分别表示最大值和最小值。在每个箱线图的顶部标记为分析的样本数量,包括小脑(n= 2);大脑(n= 3);PFA-P (n= 2);PFA-R1 (n= 5);PFA-R2 (n= 3);PFA-R3 (n= 1);PFA-R4 (n= 1);RELA-P (n= 3);RELA-R1 (n= 4);RELA-R2 (n= 2);RELA-R3 (n= 2);RELA-R4 (n= 2);RELA-lateR (n= 2)。
为了实现DNA甲基化的高分辨率分析,我们使用4个正常的儿童脑组织(2个小脑组织和2个脑组织)进行了全基因组DNA甲基化测序1)取自两名儿童作为对照的温尸检(<6小时)(图。1、表1).在所有样品中,我们鉴定出>2 × 106每个cpg至少被5个reads覆盖,平均DNA甲基化率为51%(补充数据1).EPN肿瘤表现出不同于正常组织的DNA甲基化特征(补充图。1 a, B).与原发肿瘤相似,复发性epn被细分为5个RELA和5个PFA肿瘤,使用前6000个变异CpGs在系统发育构建中显示出明显的分离(图2)。1 d, E和补充图。1 c, D)以及关键DNA甲基化特征的维持(图。1 c)和先前在原发性EPN肿瘤中发现的基因表达模式(GSE64415)39(无花果。1 f、补充数据2).这些数据表明,在多年的化疗和/或放疗期间,反复复发(≥2次)时,EPN分子亚型保持不变。
对CpG岛的进一步分析发现,与原发epn相比,复发肿瘤的DNA甲基化水平增加,其中CpG岛的水平高于正常脑组织(补充图。1 e).相比之下,在复发肿瘤的CpG岸边区域(距离CpG岛2 kb的侧翼)没有发现一致的DNA甲基化变化(补充图)。1 e).
非CpG甲基化最近被认为是一层组装在脊椎动物根部的表观遗传信息,并发挥着独立于CpG甲基化的祖先形式的新的调控作用40.它的模式通常是组织特异性的40,41,42,43.在本研究中,发现原发性和复发性肿瘤在RELA和PFA肿瘤中mCpA水平显著降低(图。1克)与正常组织相比。虽然RELA和PFA肿瘤之间的差异不显著,但这一发现表明mCpA的减少可能是EPN肿瘤的一个潜在的表观遗传特征,其功能作用值得进一步研究。
重复复发期间DNA甲基化景观的收敛
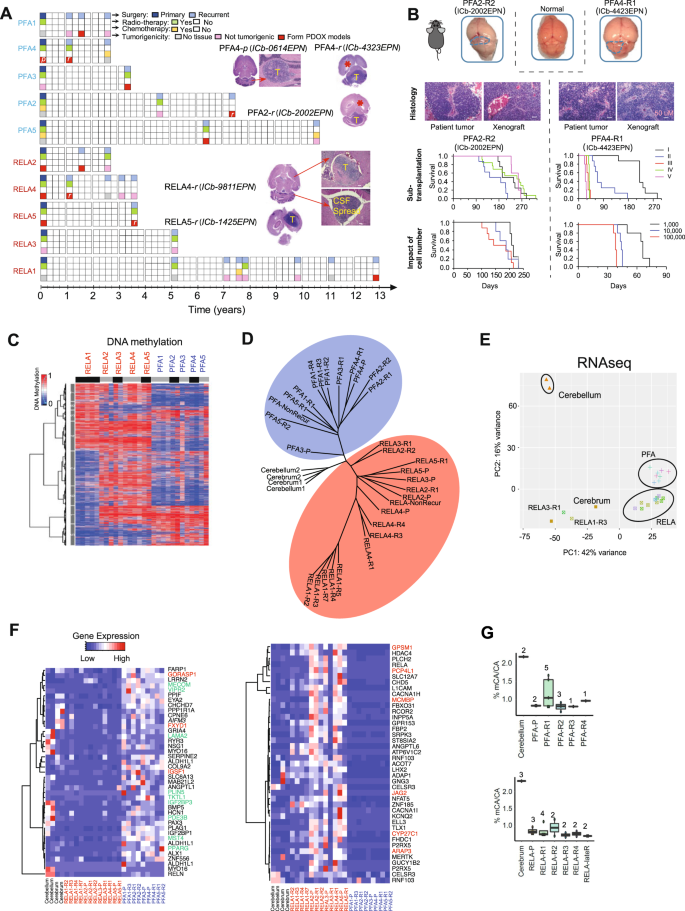
目前尚不清楚EPN的进展和反复复发如何影响细胞亚群。为了了解EPN长期连续复发期间选择性、适应性和进行性压力对表观遗传重编程的影响,我们计算了每个患者在肿瘤复发期间连续复发肿瘤之间DNA甲基化比率的Pearson相关系数。如果没有相邻的复发肿瘤,则使用先前可用的样本来计算相关性。不同于原发肿瘤与正常脑组织之间的低水平相关性(平均rRELA为0.24,PFA为0.23),两个连续纵向复发肿瘤之间的Pearson相关性显著增加,在RELA复发的8/11个成对比较中达到0.75,在6/7个PFA复发中达到0.83(图2)。2,补充图。2- - - - - -C),分别。例如,在RELA1中,相关系数(r)在第3次和第2次复发间增加到0.68,第4 /3次复发间增加到0.70,第7 /5次复发间增加到0.77,表明在重复复发(复发次数≥2次)时有收敛趋势。类似的模式也体现在PFA1(图。2 b).对转录数据的平行分析也发现了相似的相关性增加趋势(补充图。二维)。由于没有以前的样本/研究,我们的数据对EPN复发提供了更多的见解。这一发现也很重要,因为表观遗传同质肿瘤理论上可以比广泛异质肿瘤更有效地靶向。不像其他癌症在复发时表现出突变发散44这一结果表明,临床治疗(化疗和/或放射治疗)和生物进化在推动肿瘤复发过程中选择性富集了儿童epn的表观遗传病变。
一个RELA(上)和PFA(下)肿瘤重复复发期间相邻复发肿瘤DNA甲基化相关性变化的折线图。每个点代表一个肿瘤样本,并绘制到复发时间。B显示相关系数增加的代表性平滑密度散点图(rRELA1(上组)和PFA1(下组)患者相邻复发肿瘤之间的DNA甲基化谱。C染色体1q增益的FISH分析显示了FISH探针在1p(红色)和1q(绿色)(顶部)的位置,1q (G:绿色)相对于1p (R:红色)(中)配对的患者(Pt)和PDOX肿瘤。计数细胞的数量(n)在每个样品的列顶部标记(红色条表示1p计数,绿色条表示1q计数)。采用双侧Student进行统计分析t以及。**P< 0.01。P-values = 7.7186E-30 (Pt-2002), 5.32303E-34 (ICb-2002EPN), 1.17953E-13 (Pt-0614), 1.5575E-08 (ICb-0614EPN), 4.53091E-21 (Pt-4423), 5.4252E-14 (ICb-4423EPN)。数据以平均值±标准差表示。(放大倍数:×100)。D在匹配的PDOX模型中保存患者肿瘤的DNA甲基化谱P)或肿瘤复发(R1)直到7日(R7)复发。全局皮尔逊相关(r)匹配的患者肿瘤和PDOX肿瘤的DNA甲基化谱被标记在连接线上方。相关分析中使用的cpg数量为2,211,714 (RELA1-R7)、2,156,125 (RELA4-R1)、2,075,950 (RELA5-R1)、1,994,148 (PFA1-R3)、2,473,879 (PFA2-R2)、1,728,999 (PFA4-P)和1,200,936 (PFA4-R1)。大多数患者的DMRs维持在匹配的PDOX肿瘤中(下图)。用于患者和PDOX肿瘤分析的hyperDMRs总数为6919和6265 (RELA1-R7), 10,868和12,825 (RELA4-R1), 11,406和14,333 (RELA-R1)m 5815和6653 (PFA-R3), 13,541和15,036 (PFA2-R2), 4107和8309 (RFA4-P), 3155和8373 (RFA4-R1);而hypoDMRs的总数量分别为15147和27594 (RELA1-R7)、5797和8506 (RELA4-R1)、4361和7009 (RELA-R1)、12051和15519 (PFA-R3)、5824和5847 (PFA2-R2)、5373和10917 (RFA4-P)、8969和10050 (RFA4-R1)。E原发性/复发性肿瘤的无监督聚类(r1)直到7号(r7)复发以及在特定通道(通道)匹配的PDOX肿瘤。
晚期EPN复发表现出SCID小鼠大脑致瘤性增加
接下来,我们研究了复发肿瘤的进行性和收敛性表观遗传变化在促进致瘤能力增强方面是否具有重要的功能。尽管在小儿恶性脑肿瘤的PDOX模型开发方面取得了成功29,31,32,34,35,45,46,47,48,49通过我们的小组和其他人的研究,儿童EPN以低肿瘤发生率而闻名29,50,51.随着我们发现连续EPN复发过程中表观遗传变化的进行性和收敛性,我们假设这种表观遗传重编程使复发肿瘤获得致瘤能力。因此,我们系统地将27个EPN肿瘤(6个原发性,21个复发性)植入SCID小鼠大脑的匹配位置(相同位置,相同深度,年龄相似的两性动物)。29,30.,31,34,52(无花果。1).所有小鼠接受相同的数字(1 × 105)存活的肿瘤细胞。这些动物被密切监测长达15个月,如果它们出现神经功能缺陷或濒临死亡,就会被安乐死。13例(48.1%)肿瘤患者经大体和组织学检查证实存在脑内或小脑内异种移植。5例原发肿瘤患者(n= 1例患者:PFA3)或早期复发肿瘤(n= 4例:RELA1, PFA5, PFA2, PFA1)未形成异种移植,其晚期复发肿瘤形成PDOX肿瘤。在RELA1中,在第2、3、4、5次复发时肿瘤生长反复失败(第1、6次复发时未发现患者肿瘤组织),第7次复发时证实肿瘤形成。在PFA1和PFA2(图。1),第2次复发证实PDOX形成。尽管致瘤性会受到许多因素的影响,但我们独特的方法是检测来自同一患者的连续复发肿瘤组织,并使用标准化的肿瘤植入方案,提供了功能数据来支持肿瘤复发的恶性进展,并提示收敛的表观遗传重编程在促进EPN复发中的致瘤性方面的作用。
许多模型已经在小鼠大脑中进行了多达5个传代的体内亚移植,同时显示出植入的肿瘤细胞(1000-100,000个细胞/小鼠)与动物生存时间之间的反向相关性(图。1 b).这套模型将提供一个急需的平台,将生物学发现转化为复发性epn的功能和临床前测试。为了确定它们的分子保真度,我们比较了7对患者异种移植瘤的全球平均DNA甲基化比率和DMRs,并检测到高度的相似性(r> 0.9)在6/7模型(图;2 d, E,补充图。2- - - - - -D),甚至在亚移植期间(RELA5和PFA4),证明在其匹配的PDOX模型中忠实地再现了患者的表观遗传特征(图。2 e).
染色体1q的增加与PFA肿瘤的复发和不良预后有关1,50,53.为了检验第一季度的增益是否也可能与致瘤性相关,我们使用CNVkit使用我们的RRBS数据来推断CNV状态。由于技术的限制,即酶消化导致肿瘤中相同的reads和不均匀的覆盖,在PFA患者和PDOX肿瘤中没有明确的1q增益(补充图)。3.).为了补充这一方法,我们应用FISH并在三组PFA室管膜瘤患者和异种移植瘤的石蜡切片中检测1q增益(图。2摄氏度),表明患者肿瘤中的1q增益在PDOX肿瘤中也被保留。这些数据支持对其他PFA肿瘤(包括致瘤性和非致瘤性)的分析,以确定1q增益在PFA致瘤性中的作用。
发现了EPN复发的潜在DNA甲基化驱动因素
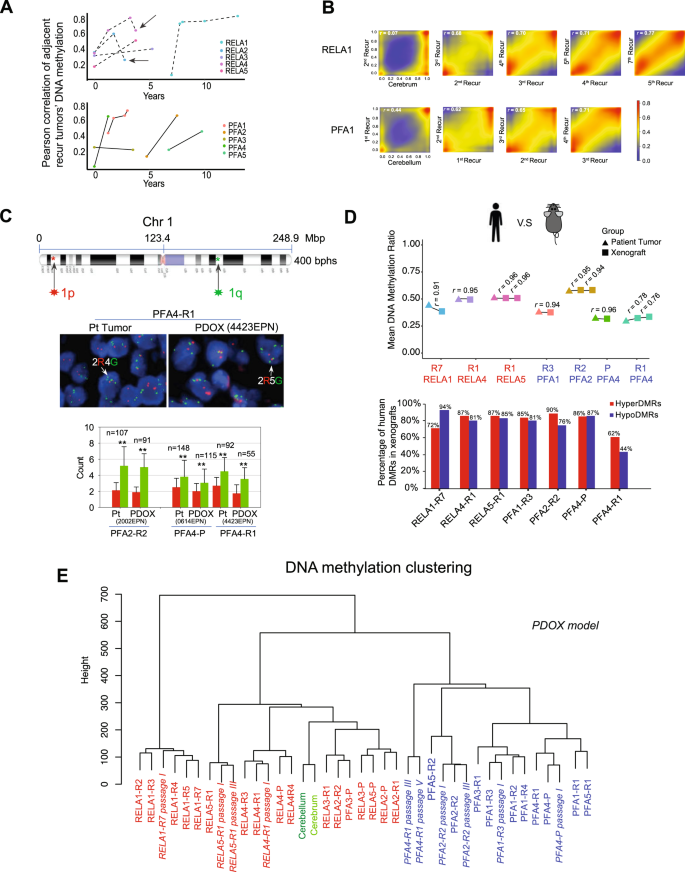
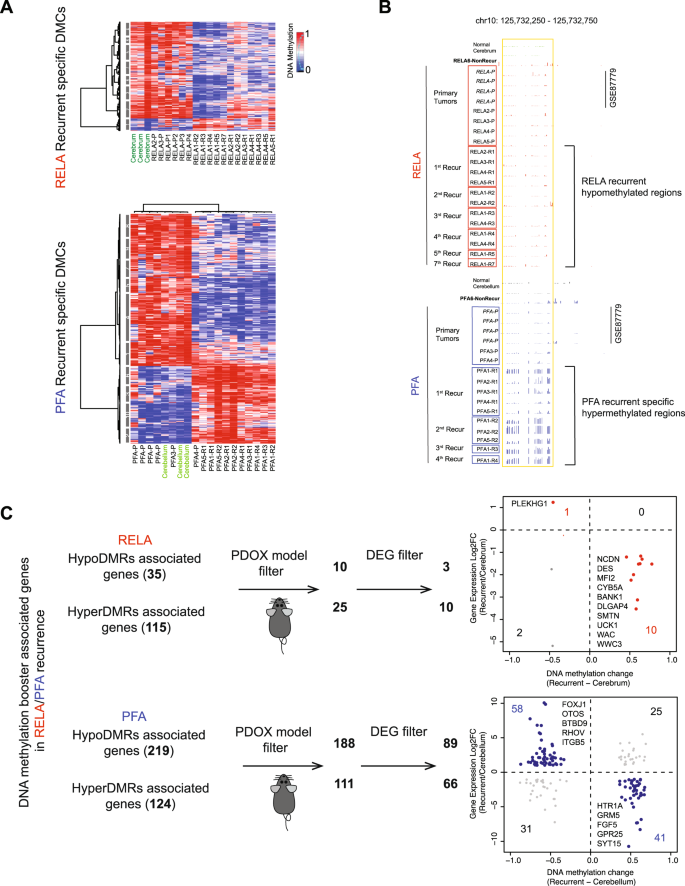
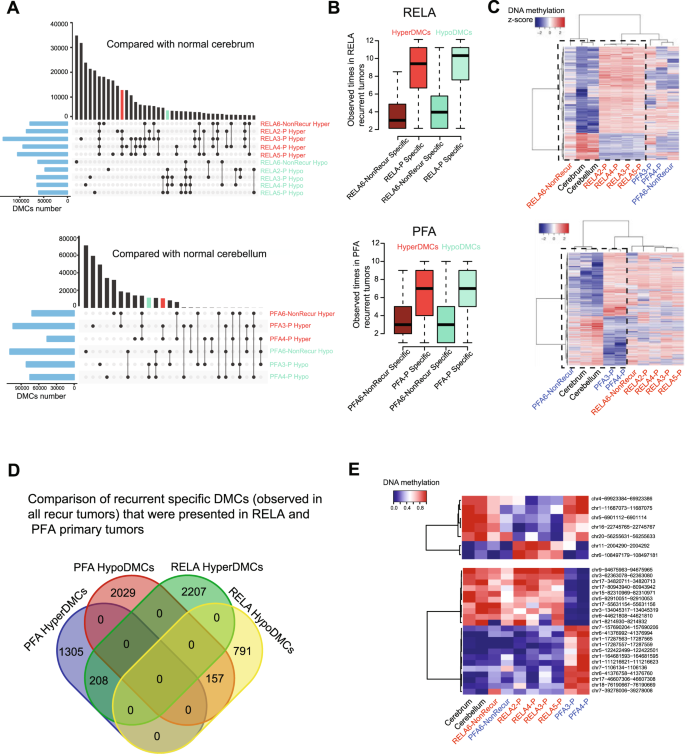
为了确定原发肿瘤中异常DNA甲基化的命运,并确定复发的潜在表观遗传驱动因素,我们对重复复发进行了深入的纵向分析,以从随机的DMCs转运中分离出连续复发期间一致存在的差异甲基化位点(DMCs)。3、补充数据3.).我们用冲积图显示了重复复发(≥2)时DNA甲基化的动态变化。大多数dmc发生了单向变化,而不到2%的dmc在hyperdmc和hypodmc之间切换(图2)。3 b,补充图。4).当单独分析肿瘤时,它们表现出广泛的变异范围,即在RELA和PFA复发中,HyperDMC为28,791 ~ 99,702(54380±24,495),HypoDMC为13,662 ~ 84,714(44672±21,560)。确定>2患者共享的dmc(图;3 c(垂直直方图)或在多次复发期间维持在每个患者(图。3 c(水平直直图),我们应用UpsetR绘制数字,识别出所有5名RELA患者共有的8155个HyperDMCs和2324个HypoDMCs共享和HypoDMC共享)(无花果。3 c);6845 HyperDMC共享6925 HypoDMC共享五名PFA患者共有。这些共享的dmc中有61.4 ~ 83.8%位于基因的近端调控区。为了进一步增加潜在DNA甲基化驱动因素发现的严格性,我们重点研究了肿瘤与正常小脑/大脑组织之间DNA甲基化比差异大于>0.3的dmc。来自DMC共享我们鉴定出57个consistent-HyperDMC共享51 consistent-HypoDMC共享在RELA肿瘤中;PFA肿瘤分别为148和118。3 d、补充数据4).这些dmc从原发肿瘤持续到晚期复发,从而构成了EPN复发的潜在DNA甲基化驱动信号。
一个时间过程DNA甲基化动态分析的示意图,以识别每个患者中从原发肿瘤到所有复发肿瘤的一致dmc,以及所有RELA或PFA患者中一致存在的共享dmc。B代表性的冲积层地块(左面板)显示RELA4(左上)和PFA1(左下)肿瘤在重复复发过程中DNA甲基化的动态变化。以正常脑组织为参照,确定CpG状态,即高甲基化、低甲基化和无变化。七种不同的模式(类别)和CpG变化的数量用不同的颜色列出。图(右图)显示了每个RELA(右上)和PFA患者(右下)7种不同类型CpG变化的百分比。C显示一致的Hyper-和hypodmc数量的打乱R图(y -RELA(左组)和PFA患者(右组)(点与线连接)共享的数据,以及每个患者一致的dmc数量(水平直方图)。所有RELA或PFA共享的一致dmc占一小部分,分别用红色突出显示。D热图显示RELA(左图)和PFA(右图)复发肿瘤的潜在DNA甲基化驱动因子(CpGs)的DNA高甲基化(红色)和低甲基化(蓝色)比率。E识别潜在DNA甲基化驱动因子调控的潜在驱动基因的数据分析步骤的示意图。从同一组肿瘤的RNA-seq中提取差异表达基因(DEGs)(肿瘤组织与正常组织之间的log2倍变化)。在患者肿瘤中发现但在匹配的PDOX肿瘤中不存在的deg被过滤出来,以识别有助于PDOX致瘤性的基因。FRELA(上)和PFA(下)的散点图显示了潜在基因的表达与DNA甲基化的相关性。在RELA中负相关的部分用红色标注(上面板),在PFA中以蓝色标注(下面板),并列在图的右侧。
潜在的DNA甲基化驱动因子调节了一组差异表达基因
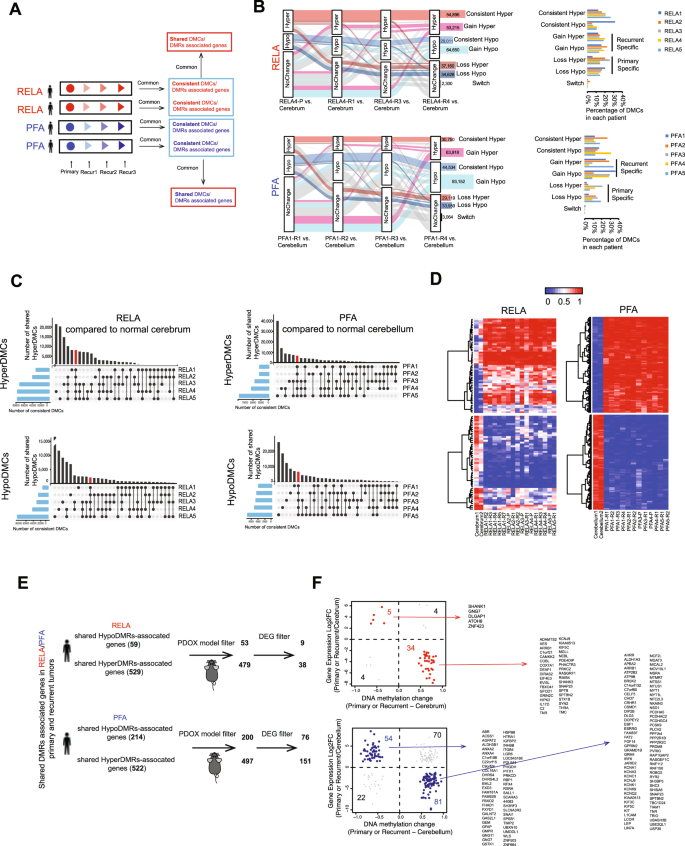
为了确定潜在驱动dmc的靶基因,我们检测了差异甲基化区域(DMRs),这是一组相邻的dmc,在调节基因表达方面比单个dmc发挥更重要的作用。在RELA原发性和复发性肿瘤中,我们平均检测到9730个HyperDMRs和8802个HypoDMRs,略高于PFA肿瘤中的7910个HyperDMRs和7977个HypoDMRs(补充数据)5)分布在不同的功能元件上(补充图;4 b).认识到位置对生物学的影响,我们比较了RELA和PFA epn之间的原发性和复发性肿瘤(补充图。5),并鉴定出2131例和976例仅在所有复发样本中出现的一致Hyper/ hypodma。使用公共数据集进行交叉检查(GSE65362)16揭示了在我们的DMRs中cpg的DNA甲基化比率的高度相似性(补充图。5 b).
启动子中的DNA甲基化可以抑制基因表达54.为了识别功能上重要的DMRs,即那些与基因相关的DMRs,我们将重点放在位于已知靶基因上/下游5 kb的基因转录起始位点(TSS)内的最显著的DMRs上(补充图。4 c, D).在RELA患者中,所有患者共有588个潜在驱动基因(529个与Hyper-DMRs相关,59个与hypod - dmrs相关)(补充数据)6);而在PFA患者中,有736个潜在的驱动基因(522个与Hyper-DMRs相关,214个与hypod - dmrs相关)(补充数据)7).为了进一步确定PDOX肿瘤形成所需的潜在DMR驱动因素,我们用PDOX DMRs过滤了潜在的患者DMR驱动因素。在RELA患者肿瘤中,90.5%(479/529)的hyperDMR相关潜在驱动基因和89.8%(53/59)的hypodmr相关潜在驱动基因在他们的PDOX模型中被保留,而在PFA肿瘤中,分别为95.4%(522/547)和93.8%(214/228)(图2)。3 e、补充数据8).这种对患者肿瘤及其匹配PDOX肿瘤的综合分析为发现潜在DMR驱动的功能重要靶基因提供了一种策略。
为了确定EPN复发中这些靶基因的表达水平是否确实受到潜在驱动因子DMRs的调控,我们分析了来自同一批样本的RNA-seq数据,以检测差异表达基因(DEGs)(补充图)。6),结果有显著不同(罗斯福< 0.05)从正常小脑或大脑中提取,然后将deg与潜在的DMR驱动因子进行匹配,显示小脑/大脑DMR比值与肿瘤>0.3的平均差异。对比PFA和RELA肿瘤显示PFA肿瘤共享更多度(调节= 1303,= 1290)和三组的肿瘤(从PFA-P PFA-R1 PFA-R20,显著高于RELA肿瘤(=三组复发的肿瘤从PFA-P PFA-R1和PFA-R2明显高于RELA肿瘤(调节= 208和表达下调= 281),选择性地共享RELA-P,只RELA1(补充图。6).RELA和PFA肿瘤细胞来源的差异20.,21,22,23,38,55可能导致了PFA复发时DEG面板的差异和相对保守的DEG。
在RELA肿瘤中,我们发现34/38(89.5%)下调靶基因(包括Syn2, phactr3, kcnj9, rims4, fbox41)共享, 5/9(55.6%)的靶基因上调(Cacna1h, slc12a7, cspg4, rara,而且ZNF423)的一致性- hypodmrs共享(无花果。3 f).这些基因在RELA肿瘤208个上调基因中占2.4%,在281个下调基因中占12.1%。同样,在PFA肿瘤中,81/151(53.6%)下调基因(包括Fat1, myt1l, grm4, kcnk9, pcdha5而且KCNA1)由consistent-hyperDMRs调控共享54/76(71.1%)基因上调(包括Fam92b, hspb8, gmpr, itgb4, fhad1,而且FXYD1)执行consistent-hypoDMRs命令共享(无花果。3 f、补充数据5).它们分别占PFA肿瘤中1303个上调基因的4.1%和1290个下调基因的6.3%(补充图)。6).尽管RELA和PFA肿瘤的细胞来源存在差异20.,21,22,23,38,55可能导致DEG小组的差异,共享的上调(n= 1303)和下调(n= 1290)在PFA复发过程中,从PFA- p到PFA- r1和PFA- r2的DEGs明显高于RELA肿瘤中的DEGs,在功能上,这些基因与神经元发育、神经元分化和神经发生相关(补充图)。6 b).DMRs与其靶基因之间存在较强的负相关(图;3 f)表明潜在DMR驱动因子在基因表达调控中具有潜在的调控作用。上述大多数DMR调控基因此前未被发现用于EPN复发。它们作为复发驱动基因的潜力被进一步增强了,因为其中许多基因,包括CACNA1H56, SLC12A757, CSPG458,59, RARA60在RELA,和HSPB861, ITGB462, FAT163,64在PFA肿瘤中,先前已与人类癌症或室管膜瘤肿瘤依赖基因(CACNA1H)11,65
尽管RELA和PFA肿瘤之间存在生物学差异,但两种类型的肿瘤共有7个下调基因(ARRB1, KCNJ9,Kiaa0513, kif5c, snap25, sptbn2,而且TNR),它们都受到DNA高甲基化的调控(补充数据6而且7).值得注意的是,我们还发现了过度表达WEE1其中多种抑制剂已进入临床试验阶段66,67在4/4 PFA和3/4 RELA肿瘤组中(补充图。5度,7 b).与RELA肿瘤相比,PFA肿瘤的潜在驱动基因增加了近3倍,这很可能是由于细胞来源的差异。总的来说,这种纵向DNA甲基化分析不仅确定了RELA和PFA肿瘤特异性或共同的复发潜在驱动因素(DMRs和基因),而且还揭示了一组与复发相关的潜在驱动基因表达改变的潜在机制。
在室管膜瘤的连续复发中发现了潜在的DNA甲基化促进因素
我们收集了来自同一患者的多个连续复发肿瘤,也为检查复发肿瘤中新获得的dmc提供了机会。在所有复发中持续存在的dmc可能持续(并促进)肿瘤复发,并导致了肿瘤发生性的增加。为了提高发现复发性特异性dmc的信心水平,我们从以前的研究中增加了4例PFA和4例RELA原发肿瘤(GSE87779)。16作为验证集(图;4 a、B,补充图。7 d),并在该公共数据库的复发肿瘤中鉴定了一组RELA和PFA复发特异性dmc(补充图。7 d).在我们对单个肿瘤进行分析后,我们提取了5组RELA或5组PFA复发共有的DMC(以下称为潜在DMC)升压),并鉴定出296个HypoDMC升压38个HyperDMC升压RELA;165年HypoDMC升压和323 HyperDMC升压在PFA递归(图。4).我们应用了如图所示的相同策略。3.鉴定DMC调控的基因推进器.在RELA肿瘤中,我们发现115例HyperDMRs升压相关基因和35个HypoDMRs升压相关基因);而在PFA肿瘤中124 HyperDMRs升压219个HypoDMRs升压相关基因(补充资料9).如果只检查一个复发性肿瘤,其中许多基因可能会被遗漏。
由于许多复发肿瘤直到晚期复发才具有致瘤性,我们推断一些潜在的促进因子相关基因有助于致瘤性升高。患者肿瘤与其PDOX肿瘤的直接比较显示,只有21.7%(25/115)的患者有hyperDMR升压hypoDMR 28.6% (10/35)升压RELA肿瘤中相关基因在PDOX肿瘤中维持,而PFA肿瘤中相关基因分别为89.5%(111/124)和89.8%(188/219)(补充数据)9),显著高于RELA肿瘤(P< 0.001)。这些数据表明DMR调控的基因数量推进器受到细胞来源差异的影响,并在PFA复发中发挥更重要的作用,PDOX肿瘤的形成尤其有助于识别致瘤性DMR升压RELA肿瘤的相关基因。
为了鉴定具有高严格性的潜在DMR助推器驱动基因,我们重点研究了在所有复发肿瘤中始终存在的deg。与潜在的dmc驱动相关基因不同,RELA和PFA肿瘤之间没有共享的潜在增强基因。在RELA复发的肿瘤中,10/10 (100%)hyperdmr调控基因的表达(Ncdn, des, mf12, cyb5a, bank1, dlgap4, smtn, uck1, wac,而且WWWC3)681/3(33.3%)的hypoDMR调控基因(PLEKHG1)与相应的DNA甲基化变化呈负相关(图;4摄氏度(上面板),分别占RELA肿瘤中下调和上调基因总数的3.4%和0.48%。更重要的是,所有这些基因在PDOX肿瘤中都得到了维持(补充数据9而且10),进一步支持了它们在致瘤性中的作用。在功能上,hyperDMR下调的一些基因与人类癌症有关,包括晚期癌症(SMTN)69和DES70, BANK171)、自噬相关死亡与胰腺癌预后(CYB5A)72,73神经胶质瘤和结直肠癌复发的侵袭性表型(MFI2)74,75.作为唯一上调的潜在促进基因,PLEKHG1似乎是一个有吸引力的治疗目标。虽然其生物学功能尚不完全清楚,但与PLEKHG1在低级别胶质瘤中,低生存率的表达已被注意到。8 a、B).在PFA肿瘤中,我们检测到41/66 (62.1%)hyperDMR下调基因(包括Htr1a, grm5, fgf5, gpr25而且嘘)和58/89(65.2%)的hypoDMR上调基因(包括Caps, aldh3a1, fam74a3, foxj1, ehf, itgb5, notch1, epha2,而且SUFU)(图。4摄氏度,下面板)(补充数据10),分别占PFA肿瘤中下调和上调基因总数的3.2%和4.5%。除了切口76,EPHA277而且SUFU78已知与EPN生物学有关的许多基因(FOXJ179,ALDH3A180,EHF81)与人类癌症或脑肿瘤有关(补充图。7).虽然少量的失调基因限制了我们详细的生物富集分析的能力,但我们发现它们在促进EPN复发中的潜在作用是令人兴奋的,并保证了未来的功能验证和药物开发。
DNA甲基化的复发预测因子可以在诊断时从原发肿瘤中确定
由于并非所有的epn都会复发,因此开发DNA甲基化标记物在肿瘤诊断时可以预测肿瘤复发是非常可取的。我们减去最终复发的epn的原发肿瘤dmc(即原发肿瘤)最终复发)从未复发的原发肿瘤(原发肿瘤Not-replapsed)超过10年的随访。使用UpsetR,我们绘制了原发肿瘤共享的dmc数量最终复发或原发肿瘤特有Not-replapsed(无花果。5(垂直直方图)以及各原发肿瘤的dmc(图;5,水平直方图)。我们进一步假设,在原发肿瘤中确定的活性预测因子也应该存在于随后的复发性肿瘤中。如图所示。5 b,原发肿瘤特异性dmc的频率最终复发是否明显高于原发肿瘤Not-relapsed.为了增加数据分析的严格性,我们重点研究了所有(12例)复发性RELA肿瘤和所有(10例)复发性PFA肿瘤中选择性存在的dmc(图2)。5 b, C),分别;并将候选预测因子dmc缩小到RELA肿瘤中2207个超、791个超、PFA肿瘤中1305个超、2029个超。5 d).从这个列表中,我们进一步对dmc进行了排名。5 e),并确定RELA肿瘤中7个DMCs和PFA肿瘤中22个DMCs (DNA甲基化比差异≥0.8)作为复发预测DMCs的首选候选(图。5 e).值得注意的是,一些置信度最高的标记,如chr17-80943940-80943942和chr5-92910051-92910053,显示出较大范围的DNA甲基化比例,提示样品存在变异的可能性。尽管样本量相对较小,但我们在原发肿瘤中发现的这组“复发绑定”的超、下dmcs非常令人鼓舞。它们为今后对这一重要主题的进一步研究提供了原理证明。新的复发诊断预测因子可能会导致儿童epn临床护理的范式转变。
一个显示RELA(上)和PFA(下)原发肿瘤中高甲基化和低甲基化CpGs位点(DMCs)的紊乱R图(上)- p)与正常的大脑或小脑组织相比。水平直方图表示原发肿瘤与正常脑组织每次比较中dmc的数量;垂直直方图表示由连接点标记的肿瘤共享的dmc数量。红色和绿色条突出了所有最终复发的肿瘤(原发肿瘤)共有的高、低dmc最终复发),但在正常脑组织和非复发性参考肿瘤中没有。B柱状图显示室管膜瘤原发肿瘤最终复发的高和低dmc的频率(-P-Specific)并且没有复发(-NonRecur特定)在RELA循环(n= 12)和PFA (n肿瘤复发。箱线图表示中位数,第一和第三四分位数(Q1和Q3),胡须延伸到最远的值;最上面一行和最下面一行分别表示最大值和最小值。在RELA肿瘤中,分析的hyperdmc数量为RELA6-非复发性(n= 10,309 CpGs), rela - p特异性(n= 357,141 cpg);而hypodmc的RELA6-NonRecurSpecific (n= 17,188 cpg)和rela - p特异性(n= 507,987 cpg)。在PFA肿瘤中,分析的HyperDMCs为pfa6 -非递归性(n= 24,296 CpGs)和pfa - p特异性(n= 349,746 cpg);和HypoDMCs PFA6NonRecurSpecific (n= 40,525 cpg);PFA-P-specific (n= 451,426 cpg)。C显示RELA(上)或PFA(下)原发肿瘤特异性DMCs DNA甲基化比例的热图最终复发存在于所有复发性肿瘤中。黑色虚线框显示,这些dmc在RELA-/ pfa - nonrecurur原发肿瘤、正常小脑和大脑中具有相似的DNA甲基化比例,但与原发肿瘤最终复发不同。D维恩图表示重叠和特定的超/ hypodmc的数量(从C)。E热图显示了最高CpGs的DNA甲基化比例,具有最高的置信度,可以潜在地预测RELA(上面板)和PFA(下面板)室管膜瘤原发肿瘤的复发。
讨论
在本研究中,我们通过收集诊断时与原发肿瘤相匹配的连续复发的EPN肿瘤,提高了对儿童EPN复发的认识。通过DNA甲基化和RNA-seq的高分辨率分析,我们发现分子亚型在长期连续的系列复发中保持不变,并发现它们的表观遗传特征在系列复发中逐渐收敛。来自同一组肿瘤样本的动物致瘤性系统分析显示,在一些晚期复发肿瘤中,PDOX致瘤性显著增加,这为支持EPN复发的进行性提供了功能数据。这些PDOX模型的并行分析也通过微调对EPN复发至关重要的潜在DMC驱动因素和促进因素,弥合了表观遗传重编程和增加的致瘤性之间的差距。
患者肿瘤是生物学研究最可靠的来源。了解肿瘤复发的主要挑战之一是复发肿瘤组织的可用性非常有限78.这需要一个强大的协作团队和长期的承诺。在目前的研究中,我们积极收集并仔细跟踪了110例EPN患者13年,并成功收集了10组(9%)复发肿瘤。然而,与儿童epn的50%复发率相比,我们只捕获了<20%的复发率。一个主要原因是EPN复发的治疗不包括手术或活检,因为原发肿瘤已经满足了诊断的需要,目前没有足够的理由进行常规的肿瘤病理“复查”。我们在复发性肿瘤中发现的表观遗传重编程为复发性epn的活检或手术切除提供了生物学证据,以更新分子诊断和复发性epn的临床治疗。
儿童epn反复复发时的表观遗传收敛表明复发肿瘤中DNA甲基化的细胞异质性降低,这与以往报道的其他类型癌症复发肿瘤中基因突变负荷增加或扩大的结果不同44.造成这种现象的一个可能的原因是临床治疗,特别是放射治疗,这仍然是儿科epn临床治疗的主流1,2,选择或调节存活的肿瘤细胞亚群复发。这一结果可能具有重要的临床意义,因为它提示了靶向一小部分复发的表观遗传驱动因素以显著提高疗效的可能性。
肿瘤复发是由一系列遗传和表观遗传事件推动的。我们对潜在的复发驱动因素(从原发肿瘤持续到所有复发肿瘤的dmc和基因)和促进因素(仅在复发肿瘤中出现的dmc和基因)的识别,破译了儿科EPN复发的详细长期路线图。然而,儿童时期的大脑往往处于不同的分化状态,例如,在我们的队列中,从2-10岁开始。当结合不同的复发时间框架(从1年到13年),一些潜在的DMC驱动因素可能归因于患者特定的大脑和小脑分化状态。除了发现一组潜在的EPN复发驱动和促进基因外,我们的发现令人鼓舞的一个方面是,许多基因已经参与了人类癌症生物学。例如,CACNA1H一种电压门控钙通道,已在乳腺癌中被检测到56,82;RARA视黄酸受体α在白血病和最近的成神经管细胞瘤和胶质瘤中发挥了重要作用83;而且HSPB8 (热休克蛋白β -8促进胶质瘤的生长和转移61,84.作为RELA复发中唯一潜在的促进基因,PLEKHG1(pleckstrin同源性和RhoGEF结构域含G1)在胃癌血浆中显著上调,并与总生存率差相关85.的识别切口,EPHA2而且SUFU作为PFA复发的潜在促进基因,不仅提示了这些基因在复发中的潜在作用,而且为难以治疗的PFA肿瘤提供了一组潜在的药物靶点76,86.
在诊断时识别可以预测肿瘤复发的标记物是非常可取的。与从复发性epn中获取肿瘤组织的困难类似,从诊断后至少>年无肿瘤的患者中定位手术样本也很困难。尽管我们进行了大量的努力,但我们只能获得一个RELA和PFA肿瘤样本,这些肿瘤没有复发5-10年。我们对DMC复发预测因子的识别可能有助于患者分层,以生物学为基础合理选择治疗策略。这组原理证明数据应该通过分析更大的循环和非循环样本集合来改进和验证预测因子列表,从而引发对该领域的广泛兴趣。尽管样本中候选预测因子存在差异,但我们非常希望目前或未来每种分子亚型的EPN复发预测因子数量减少或在临床上适用。
为了将生物学研究从实验室转化为临床,我们系统地将同一组肿瘤组织植入小鼠大脑的匹配位置。虽然需要近13年的持续努力,但我们通过发现晚期复发的致瘤性升高,过滤掉6-10%不直接参与PDOX形成的DMC,并从功能上验证了维持复发epn进展的潜在DMC驱动因素,证明了这一策略的力量。这些数据扩展了我们之前的发现,即染色体1q增加促进不良预后,并可能通过提供更广泛和更高分辨率的分子特征来推动PFA的致瘤性。这组临床相关的动物模型可以促进EPN复发的生物学和临床前研究。
我们的研究还存在一些局限性。由于儿科epn的罕见性,患者数量相对较少,我们希望我们的发现将有助于复发肿瘤的活检或手术,以更好地了解肿瘤生物学,并增加复发肿瘤的可用性。肿瘤的发生受多种因素的影响。虽然我们已经尝试将该方案标准化,但除了我们研究中涉及的遗传/表观遗传变化外,可能还有其他决定PDOX肿瘤形成的因素。单细胞分析可以揭示EPN复发的细胞异质性和驱动细胞。新兴数据表明,非cpg甲基化在大脑发育和癌症生物学中的潜在作用40,41,43,87.此外,需要更多的病例和更高的分辨率(例如,全基因组甲基化测序)来支持我们关于PFA和RELA EPN中mCpA降低的发现(无论复发与否)。
总之,我们定义了患者匹配的连续EPN复发的表观遗传进展路线图,发现了收敛的DNA甲基化特征作为连续复发的特征,并提出了一组潜在的DNA甲基化驱动因素和促进因素,维持和促进复发。我们还发现了与表观遗传进展并行的晚期复发的致瘤性增加,建立了一组PDOX模型,并证明了这些模型在微调EPN复发的潜在驱动因素和促进因素列表方面的力量。我们的发现可能会提高对EPN复发的潜在表观遗传机制的认识。
方法
儿童EPN患者的肿瘤组织
根据贝勒医学院机构审查委员会(IRB)批准的方案(H-4844),在采集样本之前,从患者或其法定监护人处获得签署的知情同意书。本研究从德克萨斯州儿童医院13年(5.8±3.8)年间接受原发性和复发性肿瘤(3.67±1.76/例)连续手术切除的10例患者中获得33例新切除的EPN肿瘤标本。4个正常脑组织样本(2个脑组织和2个小脑组织)取自儿童热尸检(<6 h),作为正常参考。患者的人口统计学和临床信息见表1.所有样本均按照WHO系统进行病理诊断和分级。肿瘤组织被分成两部分进行处理。其中一部分在液氮中快速冷冻,并保存在−80°C的冰箱中。第二部分新鲜肿瘤组织洗净,用细剪刀剪成小碎片。用35 μ细胞过滤器收集肿瘤细胞的单细胞和小块(每块3-5个细胞),重悬在DMEM生长培养基中,使最终浓度达到1 × 108每毫升活细胞,以台盼蓝染色评估,并转移到动物设施的冰上。
全球还原亚硫酸氢盐序列(RRBS)
采用Allprep DNA/RNA mini kit (Qiagen)提取基因组DNA。使用Qubit®dsDNA BR检测试剂盒(赛默飞世尔科学公司)测量浓度,随后使用片段分析仪进行DNA质量评估TM以及DNF-487标准灵敏度或DNF-488高灵敏度基因组DNA分析试剂盒(高级分析)。根据制造商的协议,使用优质还原亚硫酸氢盐测序(RRBS)试剂盒(Diagenode Cat# C02030033)制备RRBS库。每个样本使用100 ng基因组DNA开始文库制备。在文库准备完成后,将样本按8(小鼠污染高达10%)、7(小鼠污染高达25%)、5(小鼠污染高达40%)或4(小鼠污染高达55%)的比例组合在一起。总共准备了16个池子。最终文库扩增后,使用1.5×珠:样品比的agcourt®AMPure®XP (Beckman Coulter)进行PCR清理。通过使用Qubit®dsDNA HS Assay Kit (Thermo Fisher Scientific)测量池的DNA浓度来进行RRBS文库池质量控制,并使用2100 Bioanalyzer(安捷伦)的高灵敏度DNA芯片检查池的廓形。每个RRBS文库在HiSeq3000 (Illumina)上用50 bp单读测序(SR50)进行深测序。
RRBS数据生成和处理
使用BSMAP (v2.9) RRBS模式将原始RRBS FASTQ文件映射到NCBI Human Reference Genome Build GRCh37 (hg19)20..使用MOABS (v1.2.9)分析DNA甲基化率和差异甲基化胞嘧啶(DMCs/DMRs)21.覆盖5个或更多reads的CpG位点用于下游分析。亚硫酸氢盐转化率是根据噬菌体基因组spike-ins估计的。包括单碱基对DNA甲基化比的床图文件转换为bigwig文件格式,可以使用UCSC基因组浏览器进行可视化。采用R包绘制DNA甲基化热图https://www.rdocumentation.org/packages/heatmap3/versions/1.1.6/topics/heatmap3通过将所有样本之间的共享cpg作为输入,生成heatmap3。采用R包猿进行DNA甲基化系统发育分析22.为了比较多个组的DMC,我们合并所有两组比较中的所有DMC(联合DMC集)。的UpSetR23包用于在多个两组比较中可视化DMC集的并集。为了分析肿瘤复发过程中dmc的动态变化,我们首先将dmc分为3类(Hyper;海波;NoChange)基于相邻的两个复发期。然后,通过考虑四个相邻的复发期(P vs.小脑;R1 vs小脑;R2 vs.小脑和R3 vs.小脑),由于数量较少,我们过滤掉了在任何两个相邻阶段之间有高/低和低/高切换的dmc。我们最终将dmc分为7类:一致性Hyper;海波一致;获得超; Gain Hypo; Loss Hyper; Loss Hypo and switch (between hyper/hypo and NoChange). R alluvial package (https://www.rdocumentation.org/packages/alluvial/versions/0.1-2/topics/alluvial)观察DMCs随肿瘤复发的动态变化。DNA甲基化率的彩色密度散点图采用R包smoothScatter (https://www.rdocumentation.org/packages/graphics/versions/3.6.1/topics/smoothScatter).伟大的24用于预测DMRs的功能。分析代码可在https://github.com/lijiacd985/Mmint.
为了推断DNA拷贝数状态,特别是染色体1q,我们应用CNVkit (https://cnvkit.readthedocs.io/en/stable),利用PFA人类和PDX样本的RRBS数据推断CNV。CNVkits在bin级计算归一化覆盖率,然后使用循环二进制分割(CBS)来推断离散拷贝数区域作为段,从而去除系统偏差(如CG内容)。
RNA-seq分析
根据制造商的说明,使用TruSeq RNA样品制备试剂盒(Illumina)和安捷伦自动化NGS系统制备用于转录组分析的RNA-seq文库。样品准备从每个样品中提取1µg总RNA开始。用低聚dT磁珠从样品中纯化poly(A) RNA,并用二价阳离子对poly(A) RNA进行裂解。片段poly-A RNA通过逆转录转化为cDNA,并使用T4 DNA聚合酶、Klenow聚合酶和T4多核苷酸激酶进行修复。用Klenow聚合酶(exo-minus Klenow polymerase)进行3’a -tail,然后将Illumina配对端寡核苷酸适配器连接到cDNA片段上。PCR扩增15个循环,用AMPure XP珠纯化。PCR产物经AMPure XP珠纯化后,得到的质量和数量。
FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)用于对原始fastq文件进行质量检查。原始FASTQ文件使用HISAT2与NCBI人类参考基因组构建GRCh37 (hg19)比对16使用默认设置。唯一映射的读数用于下游分析。HTSeq17用于计算每个基因在外显子区域的reads计数。读取计数矩阵(行为基因;列作为样本)输入到DESeq218来鉴定差异表达基因(DEGs)。我们将≤FDR≤0.05、fold change≥2 fold的基因视为deg。用R包DESeq2进行DEGs的主成分分析。用GSEA富集DEGs的功能19.
患者源性原位异种移植(PDOX)小鼠模型的建立
SCID小鼠NOD.129S7(B6)-Ragltm1Mom/J (Jax实验室),小鼠被饲养在休斯顿德克萨斯儿童医院或芝加哥Lurie儿童医院的特定无病原体(SPF)动物设施中。所有实验均采用贝勒医学院或西北大学动物护理与使用委员会(IACUC)批准的协议进行。这两所机构的实验动物福利办公室都得到了国际实验动物护理评估和认可协会(AAALACI)的充分认可。养殖用水、温度和光照周期由中央计算机控制。该饲养场由全职兽医和支持人员组成,管理一个完整的兽医护理计划。使用徒手植入策略,将肿瘤细胞移植到小鼠大脑的肿瘤组织和手术移植29,30.,31.5-8周龄(模拟儿童大脑发育)的雄性和雌性小鼠均被吸入异氟醚和/或戊巴比妥(50 mg/kg)麻醉。i.p.注射)。在手术时,每只动物在颅内注射肿瘤细胞前都会被给予止痛药。肿瘤细胞(1 × 105)悬浮在2µl的培养基中,并通过10µl 26-gauge Hamilton Gastight 1701注射器针注射到右额/颞区(中线右侧1 mm,子宫内膜缝线前2.5 mm)或右小脑叶((中线右侧1 mm,子宫内膜缝线后1 mm) 3 mm深29,30.,31.注射肿瘤后,每天对动物进行3-4天的监测。在最初的每日观察期结束后,将监测所有荷瘤动物的不适或疼痛症状。如果动物无法进食或移动,或表现出蜷缩姿势或自残等行为,或任何感染迹象(如眼睛或耳朵),将会引起兽医干预。如果动物出现斜颈、不受控制的圈圈或肢体瘫痪等其他神经功能缺损迹象,或体重下降(>15%),或濒临死亡,将通过腹腔注射150 mg/Kg的乙硫醇(戊巴比妥钠和苯妥英钠)进行深度麻醉,对动物实施安乐死,然后将整个小鼠大脑取出进行组织病理学检查。12个月后,那些没有任何神经缺陷的小鼠被安乐死,并检查肿瘤的发展。为了进行连续次移植,供体小鼠的整个大脑被无菌移除,冠状切成两半,并转移回组织培养实验室。然后在显微镜下解剖肿瘤,机械分离成细胞悬液,计数并如上所述注射到受体SCID小鼠的大脑中30..
荧光原位杂交(FISH)分析
对5 μm石蜡包埋切片切片进行FISH分析48使用Vysis/Abbott Molecular (Des Plaines, IL)双色探针,针对染色体1p36.3/TP73和1q25.2/ANGPTL位点,其中1p36.3/TP73位点用光谱红色标记,1q25.2/ANGPTL位点用光谱绿色标记,按照临床细胞遗传学实验室的标准实验室程序,检测两个位点的拷贝数改变。共有100个不重叠的细胞由两名技术人员独立评估。计算1p36.3/TP73和1q25.2/ANGPTL的平均信号。1q/1p的信号比≥2.0,被解释为增益或放大1q25.2/ANGPTL。
报告总结
有关研究设计的进一步资料,请参阅自然组合报告摘要链接到这篇文章。
改变历史
12月22日
参考文献
里兹曼,t.a.等人。一项对复发性儿科室管膜瘤的回顾性分析显示,目前在中枢神经系统位置和分子亚群上的治疗极低的生存率和无效。Pediatr。血癌https://doi.org/10.1002/pbc.28426(2020)。
李,J.等。调强放疗治疗3岁以下间变性室管膜瘤疗效观察。Radiat。肿瘤防治杂志。J。38, 26-34(2020)。
古普塔等人。复发/进展性室管膜瘤再次切除的程度、挽救性再照射的顺序/时间和无病间隔对临床结果的影响。j . Neurooncol。147, 405-415(2020)。
佐藤,等人。调强放疗或质子束放疗治疗局限性室管膜瘤儿童的无进展生存。癌症123, 2570-2578(2017)。
Junger, s.t., Timmermann, B. & Pietsch, T.儿科室管膜瘤:一种复杂疾病的概述。蔡尔兹Nerv。系统.https://doi.org/10.1007/s00381-021-05207-7(2021)。
Rajagopal, R., Foo, J. C., Jawin, V., Qaddoumi, I. & Bouffet, E.婴儿和幼儿室管膜瘤的高剂量化疗与自体干细胞移植:Head Start II方案的10年经验。Pediatr。Transpl。23, e13421(2019)。
基兰,m.w.等人。AdV-tk基因介导细胞毒免疫疗法辅助小儿恶性胶质瘤和复发性室管膜瘤手术和放疗的I期研究神经肿瘤防治杂志。21, 537-546(2019)。
Ellison, D. W.等人。cIMPACT-NOW更新7:推进室管膜肿瘤的分子分类。大脑病理学研究。https://doi.org/10.1111/bpa.12866(2020)。
麦克,S. C.等。表观基因组改变定义致命的cmp阳性室管膜瘤的婴儿。自然506, 445-450(2014)。
Bayliss, J.等人。低H3K27me3和DNA低甲基化定义预后差的儿童后窝室管膜瘤。科学。Transl。地中海。8, 366ra161(2016)。
麦克,S. C.等。室管膜瘤的治疗靶向,由癌基因增强子分析得知。自然553, 101-105(2018)。
癌症的特征:新的维度。癌症。12, 31-46(2022)。
Capper, D.等人。DNA甲基化和基于拷贝数的中枢神经系统肿瘤诊断的实际实施:海德堡经验。Acta Neuropathol。136, 181-210(2018)。
Hovestadt, V.等人。利用DNA甲基化测序解码成神经管细胞瘤的调控格局。自然510, 537-541(2014)。
帕特勒,K. W.等。颅内室管膜瘤及其独特的分子变异的临床管理目前的共识。Acta Neuropathol。133, 5-12 (2017)
帕特勒,K. W.等。室管膜肿瘤在所有中枢神经区室、组织病理学分级和年龄组的分子分类。癌症细胞27, 728-743(2015)。
Zschernack, V.等人。儿童幕上室管膜瘤:不仅仅是RELA或YAP。Acta Neuropathol。141, 455-466(2021)。
约翰逊,R. A.等。跨物种基因组学匹配驱动突变和细胞室室模型室管膜瘤。自然466, 632-636(2010)。
库普,R.等人。zfta易位构成室管膜瘤染色质重塑和转录因子。癌症。https://doi.org/10.1158/2159-8290.CD-20-1052(2021)。
郑,T.等。跨物种基因组学揭示了ZFTA/C11orf95融合阳性幕上室管膜瘤的致癌依赖性。癌症。11, 2230-2247(2021)。
库普,R.等人。ZFTA易位构成室管膜瘤染色质重塑和转录因子。癌症。11, 2216-2229(2021)。
Arabzade, A.等。ZFTA-RELA指示致癌转录程序驱动侵袭性幕上室管膜瘤。癌症。11, 2200-2215(2021)。
小泽,T.等。一个C11orf95-RELA融合驱动的室管膜瘤新生小鼠模型识别了除了NF-kappaB之外的驱动程序功能。细胞的代表。23, 3787-3797(2018)。
Yang, D.等。室管膜瘤的复发伴随着相对稳定的表观基因组,但肿瘤形态严重改变。大脑病理学研究.https://doi.org/10.1111/bpa.12875(2020)。
Michealraj, K. A.等。表观基因组代谢调节驱动致死性婴儿室管膜瘤。细胞181, 1329-1345 e1324(2020)。
罗塞蒂,d.v.等。室管膜瘤儿童脑肿瘤蛋白质指纹的综合质谱平台:一个试点调查。癌症(巴塞尔)https://doi.org/10.3390/cancers12030674(2020)。
韩,Z.等。H3K27me3在儿童室管膜瘤中的预后价值。Pediatr。血癌67, e28121(2020)。
Jain, s.u.等。PFA室管膜瘤相关蛋白EZHIP通过H3 k27m样机制抑制PRC2活性。Commun Nat。10, 2146(2019)。
于,L.等。一种临床相关的室管膜瘤原位异种移植模型,保持原发肿瘤的基因组特征并在体内保存癌症干细胞。神经。肿瘤防治杂志。12, 580-594(2010)。
Kogiso, M.等。aurora a抑制剂MLN8237同时抑制儿童胶质母细胞瘤神经圈和单层细胞,预测PDOX模型的生存延长。中国。癌症Res.24, 2159-2170(2018)。
舒、q等人。新鲜手术标本直接原位移植保存临床相关髓母细胞瘤和胶质瘤小鼠模型中CD133+肿瘤细胞。干细胞26, 1414-1424(2008)。
刘,Z.等。肿瘤源性原位异种移植小鼠模型复制儿童幕上原始神经外胚层肿瘤第3组。神经。肿瘤防治杂志。16, 787-799(2014)。
格拉索,C. S.等。弥漫性固有脑桥神经胶质瘤功能明确的治疗靶点。Nat,地中海。21, 555-559(2015)。
林赛,H.等人。KIT基因型在一对新的患者来源的转移性小儿CNS生殖细胞瘤原位异种移植小鼠模型中的保存。j . Neurooncol.https://doi.org/10.1007/s11060-016-2098-9(2016)。
黄,L.等。基于系统生物学的药物重新定位确定地高辛为3组和4组成神经管细胞瘤的潜在治疗方法。科学。Transl。地中海。https://doi.org/10.1126/scitranslmed.aat0150(2018)。
埃德尔等人。YAP1/TAZ促进小鼠室管膜瘤样肿瘤的形成。Commun Nat。11, 2380(2020)。
Schwalbe, e.c.等。髓母细胞瘤的DNA甲基化分析允许使用福尔马林固定活检进行可靠的亚分类和改进的结果预测。Acta Neuropathol。125, 359-371(2013)。
帕克等人。C11orf95-RELA融合在室管膜瘤中驱动致癌NF-kappaB信号。自然506, 451-455(2014)。
Vladoiu, m.c.等人。儿童小脑肿瘤反映了保守的胎儿转录程序。自然572, 67-73(2019)。
德·门多萨等人。脊椎动物大脑非cpg甲基化系统的出现。Nat,生态。另一个星球。5, 369-378(2021)。
Ramasamy, D, Deva Magendhra Rao, A. K, Rajkumar, T. & Mani, S.非cpg甲基化-癌症中的关键表观遗传修饰。简短。功能。基因组学20., 304-311(2021)。
Ross, s.e., Hesselson, D. & Bogdanovic, O.斑马鱼大脑中基因体和转座子非cpg甲基化的发育积累。前面。细胞发育生物学。9, 643603(2021)。
Titcombe, P.等人。人类非cpg甲基化模式显示组织特异性和个体间的差异,提示潜在的功能。表观遗传学17, 653-664(2021)。
约翰逊,b.e.等人。突变分析揭示了复发性胶质瘤的起源和治疗驱动的进化。科学343, 189-193(2014)。
巴克斯特,p.a.等。沉默BMI1消除儿童胶质瘤CD133+细胞的肿瘤形成不是通过影响已知靶点,而是通过下调一组新的核心基因。Acta Neuropathol。Commun。2, 160(2014)。
Kogiso, M.等。小儿低级别胶质瘤的异种移植证实,在一种新的进展性多形性黄色星形细胞瘤原位移植小鼠模型中,BRAF V600E突变的丰富和CDKN2A缺失的保留。Oncotarget8, 87455-87471(2017)。
赵,X.等。全球基因表达谱证实了原发肿瘤为基础的原位异种髓母细胞瘤小鼠模型的分子保真度。神经。肿瘤防治杂志。14, 574-583(2012)。
赵,X.等。儿童恶性脑肿瘤成对神经球和传统单层培养的细胞遗传学格局。神经。肿瘤防治杂志。17, 965-977(2015)。
罗基塔,J. L.等。儿童肿瘤患者来源的异种移植模型的基因组分析,以实现合理的临床试验设计。细胞的代表。29, 1675-1689 e1679(2019)。
皮尔斯,a.m.等人。1q+后窝A组室管膜瘤患者源性原位异种移植模型的建立。神经肿瘤防治杂志。21, 1540-1551(2019)。
Pavon, L. F.等。小儿室管膜瘤原代细胞培养及颅内异种移植模型的建立:治疗发展及肿瘤生物学认识的展望。Oncotarget9, 21731-21743(2018)。
于,L.等。在原发肿瘤为基础的原位异种移植小鼠模型中,单次静脉注射溶瘤小rna病毒SVV-001可消除成神经管细胞瘤。神经。肿瘤防治杂志。13, 14-27(2010)。
卡沃利,F. M. G.等。PF-EPN-B室管膜瘤亚组的异质性。Acta Neuropathol。136, 227-237(2018)。
迪顿,a.m.和伯德,a.c pg岛和转录调控。Dev的基因。25, 1010-1022(2011)。
泰勒博士等人。放射状神经胶质细胞是室管膜瘤的候选干细胞。癌症细胞8, 323-335(2005)。
夏尔马,S.等。Ca(2+)和CACNA1H介导AM RF EMF靶向抑制乳腺癌脑转移。EBioMedicine44, 194-208(2019)。
布朗,t.c.等人。胰岛素样生长因子和SLC12A7失调:非功能性肾上腺皮质癌的新信号标志。j。科尔。杂志。229, 305-315(2019)。
Maggs, L., Cattaneo, G., Dal, A. E., Moghaddam, A. S. & Ferrone, S. CAR - T细胞免疫疗法治疗胶质母细胞瘤。前面。>。15, 662064(2021)。
斯文森,A.等。祖细胞标记物NG2/CSPG4的表达预示胶质母细胞瘤的生存率和对电离辐射的抵抗力较差。Acta Neuropathol。122, 495-510(2011)。
黄,G. L.等。视黄酸受体通过Akt和MMP2信号通路促进人类结直肠癌的进展。Onco瞄准Ther。12, 3087-3098(2019)。
沈娟,李敏,闵玲。HSPB8通过激活ERKCREB通路促进癌细胞生长,提示胃癌患者预后不良。肿瘤防治杂志。代表。39, 2978-2986(2018)。
宋,J. S.等。itgb4介导的癌症相关成纤维细胞代谢重编程。致癌基因39, 664-676(2020)。
帕斯图申科,等人。Fat1缺失促进混合EMT状态、肿瘤干性和转移。自然589, 448-455(2021)。
陈,M.等。FAT1通过结合β -连环蛋白抑制宫颈癌细胞的增殖和转移。J. clinin。实验病理学研究。12, 3807-3818(2019)。
朱俊杰,Jillette, N.,李晓楠,程安伟,刘志刚,幕上室管膜瘤C11orf95-RELA基因的三维表观基因组重编程。Acta Neuropathol.https://doi.org/10.1007/s00401-020-02225-8(2020)。
科尔布,E. A.等。AZD1775联合伊立替康治疗儿童实体瘤的临床前评估:儿科临床前测试联盟报告Pediatr。血癌67, e28098(2020)。
菲斯特,S. X.等。抑制WEE1通过dNTP饥饿选择性地杀死组蛋白h3k36me3缺陷的癌症。癌症细胞28, 557-568(2015)。
德格鲁特,J. F.等。aflibercept治疗复发性恶性胶质瘤的II期研究:一项北美脑肿瘤联盟研究。j .中国。肿瘤防治杂志。29, 2689-2695(2011)。
侯赛因,M. J.等。机器学习和基于网络的模型,以确定结直肠癌进展和生存的遗传危险因素。第一版杂志。地中海。135, 104539(2021)。
Shin, J., Wood, L. D., Hruban, R. H. & Hong, S. M. Desmin和CD31免疫标记用于检测胰胆道癌静脉浸润。《公共科学图书馆•综合》15, e0242571(2020)。
Ohshima, K.等。胃肠道间质瘤恶性进展的驱动基因改变和激活信号通路。癌症科学。110, 3821-3833(2019)。
CYB5A与自噬介导的胰腺癌细胞死亡。自噬10, 697-698(2014)。
乔凡内蒂,等人。CYB5A在胰腺癌预后及自噬调节中的作用国立肿瘤研究所。106, djt346(2014)。
刘凯,刘杰,波Q. F. MFI2-AS1通过正反馈回路调节MMP14调节胶质瘤侵袭性表型。欧元。Rev. Med. Pharm。科学。23, 5884-5895(2019)。
杨,W. J.等。II期结直肠癌复发风险的基于网络的预测基因表达特征。癌症医疗.9, 179-193(2020)。
de Almeida Magalhaes, T.等。室管膜瘤rela融合亚群中的Notch通路:上调和与癌症干细胞标记物表达的关联。癌症基因Ther。27, 509-512(2020)。
杨俊涛,汉密尔顿,R. L., Okada, H., Jakacki, R. I. & Pollack, I. F.儿科和成人室管膜瘤肿瘤相关抗原表达增加:疫苗治疗的意义。j . Neurooncol111, 103-111(2013)。
莫汉库马尔,K. M.等。在体内筛选确定室管膜瘤癌基因和肿瘤抑制基因。Nat,麝猫。47, 878-887(2015)。
雅凯,B. V.等。foxj1依赖的基因表达是产后大脑中放射状神经胶质细胞分化为室管膜细胞和星形胶质细胞亚群所必需的。发展136, 4021-4031(2009)。
Ucar, D.等人。乙醛脱氢酶活性作为肺癌的功能标志物。化学。医学杂志。交互。178, 48-55(2009)。
坂本,K.等。EHF通过抑制ets1介导的ZEB表达来抑制癌症进展。肿瘤形成10, 26(2021)。
潘,N. N.等人。电压门控钙通道:癌症治疗的新靶点。肿瘤防治杂志。列托人。14, 2059-2074(2017)。
Lopez-Valero等人。Midkine信号通路维持胶质瘤起始细胞的自我更新和致瘤能力。开展10, 5120-5136(2020)。
李霞,孙超,陈娟,马建峰,潘玉华。ERK-CREB通路参与hspb8介导的胶质瘤细胞生长和转移特性。Exp、Mol、Pathol。https://doi.org/10.1016/j.yexmp.2021.104653(2021)。
曹玮,周东,唐文华,安宏,张艳。血浆信使RNA作为胃癌的新型生物标志物的发现,通过生物信息学分析和临床验证。PeerJ7, e7025(2019)。
Herrera-Rios, D.等人。一种新型治疗性抗体的计算引导、功能验证提出Notch信号作为胶质瘤的临床相关和可药物靶点。科学。代表。10, 16218(2020)。
齐勒,m.j.等。人类细胞类型中非cpg甲基化的基因组分布和样本间变异。公共科学图书馆麝猫.7, e1002389(2011)。
帕特勒,K. W.等。后窝A组(PFA)室管膜瘤的分子异质性和CXorf67改变Acta Neuropathol。136, 211-226(2018)。
确认
本研究由NIH RO1 CA185402 (X.N.L.), St. Baldrick基金会(Grant 2532341503, J.M.S.), Golfers against Cancer (X.N.L.),儿童脑肿瘤基金会(X.N.L.),国家脑肿瘤基金会(X.N.L.),广州科学发展计划201707020001(Y.F.X.), CPRIT核心基金RP150578和RP200668 (C.S.和P.A.D.)资助。
作者信息
作者及隶属关系
贡献
概念和实验设计(S.Z Y.F.X。和X.L.N.),数据收集(S.Z H.Z。,L.Q Y.D。该调查,F.D.B, S.X,,12,学生论文,H.L。P.B。J.M.F,马丁,供货商,A.C.V,砂岩,G.B。S.R.D,清醒,王秋森,J.Y, P.J.D, X.L,司仪,D.W.P,和石油醚),组织病理学评价(大家),数据分析(论文,个终身制。,T.M S.Z, W.Y.T,,12,和X.N.L.),手稿准备(W.Y.T S.Z论文。,和X.N.L.)。
相应的作者
道德声明
相互竞争的利益
m.l., p.g., a.c v, s.s.和G.B.是比利时Diagenode公司Epigenetic Services的雇员,Liège。其余作者声明没有竞争利益。
同行评审
同行评审信息
自然通讯感谢Lukas Chavez和其他匿名审稿人对本工作的同行评审所作的贡献。同行评审报告是可用的。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
源数据
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
赵珊珊,李俊,张宏。et al。儿童室管膜瘤患者重复复发的表观遗传改变。Nat Commun13, 6689(2022)。https://doi.org/10.1038/s41467-022-34514-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41467-022-34514-z