摘要
残基-残基距离信息对于预测蛋白质单体的三级结构或蛋白质配合物的四级结构是有用的。目前已有许多深度学习方法可以准确预测单体的链内残馀距离,但很少有方法可以准确预测配合物的链间残馀距离。我们开发了一种基于二维注意驱动残差网络的深度学习方法CDPred(即复杂距离预测)来解决差距问题。在两个同型二聚体数据集上进行测试,CDPred对顶L/5链间接触(L:单体在同型二聚体中的长度)的预测精度分别为60.94%和42.93%,显著高于DeepHomo的37.40%和23.08%和GLINTER的48.09%和36.74%。在两个异源二聚体数据集上测试,CDPred的最高Ls/5链间接触预测精度(Ls:异源二聚体中较短单体的长度)分别为47.59%和22.87%,超过了GLINTER的23.24%和13.49%。此外,CDPred的预测与AlphaFold2-multimer的预测是互补的。
简介
蛋白质是生命的关键组成部分。蛋白质的功能很大程度上取决于它的三维结构1.有时单链蛋白质(单体)可以执行某些功能,而大多数单个蛋白质的结构相互作用形成多链复杂结构(多聚体)来执行其生物学功能2.因此,建立单体和蛋白质配合物的三维结构模型对于研究蛋白质功能至关重要。
自2012年以来,深度学习已被应用于推进单体三级结构的预测3..近十年来,人们开发了许多深度学习方法来预测链内残基-残基接触图或单体的距离图4,5,6,7,8,这些数据被CONFOLD等基于接触/距离的建模方法所使用9和罗塞塔10建造他们的三级结构。广泛的研究9,11,12,13已经表明,如果一个足够准确的链内距离图被预测,那么蛋白质的三级结构可以被准确地构建。最近的是AlphaFold214采用端到端深度学习方法预测单体的三级结构和残基-残基距离,取得了很高的平均精度(~90全球距离测试(GDT-TS)得分152020年第14届蛋白质结构预测技术关键评估(CASP14)。最近,AlphaFold2扩展为AlphaFold-multimer16和AF2Complex17改进对多聚体四元结构的预测。
随着深度学习在预测链内残馀距离和三级结构方面的革命,近年来开发了一些预测同型二聚体和/或异质二聚体链间残馀接触图的深度学习方法,如ComplexContact18, DeepHomo19, DRcon20.和GLINTER21预测同型二聚体和异型二聚体的接触映射,使用蛋白质单体结构的图形表示和MSA转换器从多个序列对准(MSA)生成的行注意映射作为输入22.由MSA转换器计算出的注意图是一种从MSA中提取的残差-残差协同进化特征。在无监督的预训练过程中,它已经在数百万个msa上进行了自动训练,以捕获许多不同蛋白质家族的共同进化信息。尽管取得了显著的进展,但链间接触预测的精度仍远低于链内接触/距离预测,这需要开发更多的方法来解决这一问题。
在这项工作中,我们开发了一种基于深度学习架构的蛋白质复合体距离预测方法(CDPred),该架构结合了深度残差网络的优势23一种通道级注意机制,以及一种空间级注意机制来预测同型二聚体和异质二聚体的链间距离图。与GLINTER一样,MSA转换器生成的MSA注意图被用作CDPred的一个输入。二聚体中单体的预测距离图被用作另一个输入特征。与现有的深度学习方法不同,CDPred预测的是链间距离,而不是目前DeepHomo和gllinter等方法预测的二元链间接触(接触或不接触)。我们在两个同二聚体测试数据集和两个异二聚体测试数据集上严格测试CDPred。对于这些数据集,CDPred的精度远远高于DeepHomo和glitter。
结果
同型二聚体链间接触预测的评价
我们比较CDPred和DNCON2_inter24, DeepComplex25在HomoTest1同型二聚体测试数据集上进行了、DeepHomo和glitter,结果如表所示1.这三种方法的输入三级结构都是与未结合单体结构相对应的预测结构。DNCON2_inter使用推荐的参数运行。DeepComplex web服务器用于获取其预测结果。DeepHomo的结果来自其出版物。测试了三个版本的CDPred。第一个版本(CDPred_BFD)使用BFD数据库生成的msa作为输入。第二个版本(CDPred_Uniclust)使用从Uniclust30数据库生成的msa作为输入。第三个版本(CDPred)使用CDPred_BFD和CDPred_Uniclust预测的距离映射的平均值作为预测。因为DeepHomo和GLINTER预测二进制链间接触的阈值是8 Å,而不是距离,所以我们将CDPred、CDPred_BFD和CDPred_Uniclust的链间距离预测转换为二进制接触预测进行比较。 The definition of inter-chain contact is the same as GLINTER and DeepHomo, i.e., a pair of inter-chain residues is considered to be in contact if the distance between their two closest heavy atoms is less than 8 Å. This definition is used to evaluate all the inter-chain contact predictions in this work.
CDPred的接触预测精度是所有方法中最高的。例如CDPred的top L/5接触预测精度为60.94%,比DNCON2_inter高50.34%,比DeepComplex高9.64%,比DeepHomo高23.54%,比GLINTER高12.85%。CDPred在准确率和AUC评分方面也优于DNCON2_inter、DeepComplex、DeepHomo和GLINTER,在准确率排序方面排名第二。从几乎所有的评价指标来看,CDPred的性能都优于CDPred_BFD和CDPred_Uniclust,这说明将两种msa的距离预测平均起来可以提高预测精度。
我们还比较了上述方法在HomoTest2同型二聚体测试数据集(表2).CDPred在所有评价指标方面表现最好。结合两种msa对CDPred的预测,提高了预测精度。
MSA深度对同型二聚体链间接触预测精度的影响
2.1节说明了两种不同的MSAs (BFD和Uniclust)对CDPred_BFD和CDPred_Uniclust的预测精度不同,CDPred对两种MSAs预测的两个接触映射的平均值得到了最好的结果。在这里,我们研究了MSAs的深度和两个MSAs的直接组合如何影响预测精度。补充表1,2报告序列数和有效序列数(Neff)26以及以BFD MSA和Uniclust MSA简单组合为输入的CDPred_BFD、CDPred_Uniclust、CDPred和CDPred_ComMSA的top L/2接触预测精度。Neff在MSA中相似序列的权重计算较少,被广泛用于MSA深度的测量。
CDPred_BFD和CDPred_Uniclust的Neff和接触预测精度因目标的不同而不同。在HomoTest1和HomoTest2上,CDPred_BFD和CDPred_Uniclust的Neff差值与top L/2精度差值之间的Pearson相关系数分别为0.31和0.67,说明MSA深度对接触预测精度有一定的正向影响。CDPred_ComMSA结合两个MSA生成一个更深层的MSA作为输入,在HomoTest1上的性能优于CDPred_BFD和CDPred_Uniclust,在HomoTest2上的性能优于CDPred_BFD,说明直接结合两个MSA是有益的。
在两个数据集的top L/2预测精度方面,CDPred仍然比CDPred_ComMSA表现略好(在HomoTest1上为55.19对55.13%,在HomoTest2上为38.14对36.14%),这表明平均两个msa预测的距离图比简单地将两个msa组合为输入更有效。
异源二聚体链间接触预测的评价
我们比较了CDPred和最先进的异二聚体接触预测器GLINTER在HeteroTest1和HeteroTest2异二聚体测试数据集上的差异(见表中的结果)3.,4分别)。用AlphaFold2预测了两种方法所使用的单体的输入三级结构。我们在每个异质二聚体中使用两种不同阶数的单体A和单体B (AB和BA)来生成CDPred进行预测的输入特征。两阶输出的平均值作为最终预测。对两个输出求平均的过程见补充图1.将BA预测图的链间部分取出,并将其转置为与AB预测图中链间部分相同的形状,然后求平均值。
在HeteroTest1数据集(表3.), CDPred在所有指标方面都比GLINTER好得多。它也大大优于DeepComplex的所有指标,但精度顺序。例如CDPred的顶级Ls/5接触预测精度为47.59%,是GLINTER 23.24%的两倍多,比DeepComplex高出40.19%个百分点。在HeteroTest2数据集(表4), CDPred在所有指标(接触精度、准确度顺序、准确度率和/或AUC)方面也大大优于DeepComplex和GLINTER。
补充表3.,4比较使用两种不同阶数的单体作为输入(CDPred(A_B)和CDPred(B_A))的性能,并分别在HeteroTest1和HeteroTest2数据集上平均两种不同阶数的输出(CDPred)。CDPred(A_B)和CDPred(B_A)的准确性因目标和数据集的不同而不同。有时两个阶数的精度有很大差异(见补充图)。2对HeteroTest1和HeteroTest2上的精度进行逐个目标的比较)。然而,双侧配对t检验表明,两者之间的平均差异不显著。即使以两种不同的顺序平均预测的接触图并不总是产生最好的精度,但它通过减少方差和平滑预测使性能更加稳定。例如,与CDPred(A_B)和CDPred(B_A)相比,CDPred通常提供最好或中等的预测精度。
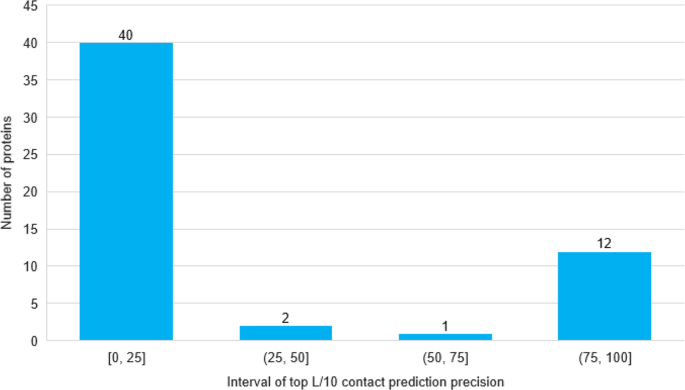
此外,我们将更具挑战性的HeteroTest2数据集中异二聚体的最高L/10接触预测精度划分为四个相等的区间,并绘制每个区间内异二聚体的数量(图2)。1).四个内部预测的精度是分岔的,主要集中在一个低精度区间[0-25%]和一个高精度区间[75-100%]。有40种异质二聚体的接触预测精度在0-25%范围内,仍有较大的改进空间。精度低的一个原因是40个异源二聚体中的大多数具有浅的msa。MSA的有效序列数对数(Neff)与最高L/10复接触精度之间的Pearson相关系数为0.46,表明两者之间存在一定的相关性。
还观察到,异质二聚体的链间接触预测精度平均低于同质二聚体。原因之一是同型二聚体的MSA生成只需要为同型二聚体中的一个单体生成MSA,通常比异型二聚体生成的MSA要深得多,后者需要将异型二聚体中两个不同单体的MSA中的相关序列配对。另一个原因是同型二聚体通常比异型二聚体具有更大的相互作用界面,这使得预测更容易。
统计优化方法与深度学习方法产生的协同进化特征的比较
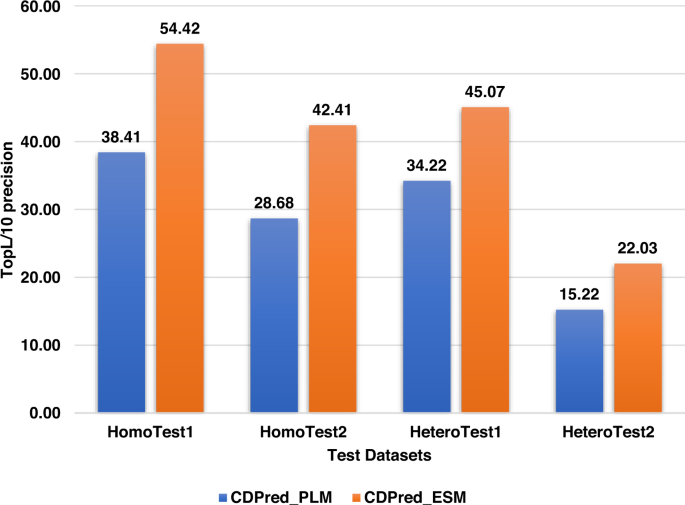
为了比较统计优化工具-CCMPred和深度学习工具- msa转换器生成的共同进化特征的性能,我们使用相同的神经网络架构对同一训练数据集的两种不同类型的共同进化特征训练了两种不同的模型。其中一个网络(CDPred_PLM)使用CCMPred生成的PLM共同进化特征进行训练。另一个模型CDPred_ESM是基于MSA转换器生成的行注意图特征进行训练的。两种模型在四种不同测试数据集上的top L/10接触预测精度如图所示。2.CDPred_ESM在所有4个测试数据集上的性能均优于CDPred_PLM,表明通过深度学习方法自动提取的协同进化特征比通过最大化直接协同进化信号的统计优化方法具有更大的信息量。然而,结合这两种共同进化的特征会产生更好的结果(见表中的结果)1,2,3.,4).补充图3.分别绘制两个同型二聚体测试数据集中同型二聚体和两个异型二聚体测试数据集中同型二聚体CDPred_ESM的顶级L/10精度与CDPred_PLM的顶级L/10精度的关系图。CDPred_ESM对51个同二聚体中的42个和64个异二聚体中的55个具有更高的精度。CDPred_ESM和CDPred_PLM在某些目标上都有较好的表现,表明两种方法所使用的协同进化特征具有一定的互补性。
预测单体三级结构质量对二聚体链间距离预测的影响
蛋白质复合体的四级结构取决于其单体单位的三级结构。由于Alphafold可以很好地预测单体的三级结构,我们研究了如何有效地将Alphafold预测的三级结构应用于预测蛋白质复合物的链间距离图。在四个数据集(HomoTest1, HomoTest2, heterotest1, HeteroTest2)上,每个二聚体的每个单体单元预测三级结构的tm -score和CDPred的接触预测精度见补充表5,6,7,8,分别。HomoTest1和HomoTest2预测的三级结构的tm -score平均值分别为0.95和0.90,HeteroTest1和HeteroTest2中异二聚体的A链的tm -score平均值分别为0.90和0.89,HeteroTest1和HeteroTest2中异二聚体的B链的tm -score平均值分别为0.95和0.88,表明alphafold预测的三级结构具有较高的质量。预测三级结构tm -评分与顶部L/2接触预测精度的Pearson相关系数为0.19。弱相关性的部分原因可能是预测的三级结构的质量足够高,以至于CDPred可以利用大多数三级结构信息来预测链间距离。
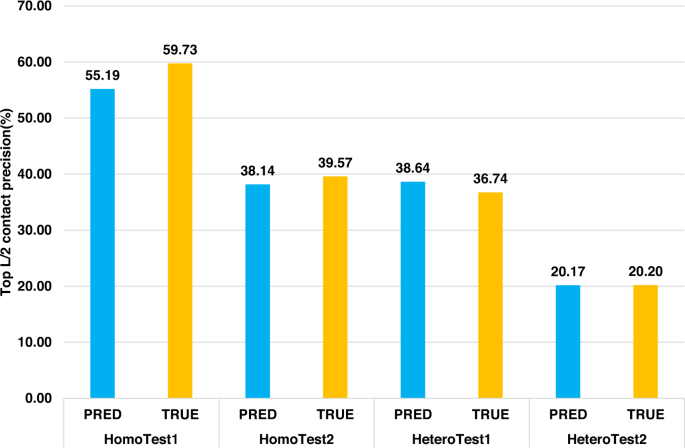
此外,我们在四个数据集上比较了以alphafold预测的单体三级结构为输入和以束缚态单体真三级结构为输入的top L/2链间接触预测精度(图2)。3.).在四个数据集中的三个(HomoTest1、HomoTest2和HeteroTest2)上,使用真正的三级结构产生的性能略好于使用alphafold预测结构,但在HeteroTest1上的性能略差。的p对的值t四个数据集的-test分别为0.6802,0.8892,0.9083,0.9963,说明差异不显著。结果表明,alphafold预测的三级结构对于CDPred进行链间距离预测是足够准确的,即使使用真正的三级结构作为输入可以略微提高整体预测精度。这与GLINTER不同,GLINTER使用真实的三级结构作为输入的准确性远远高于使用alphafold预测的三级结构作为输入21.
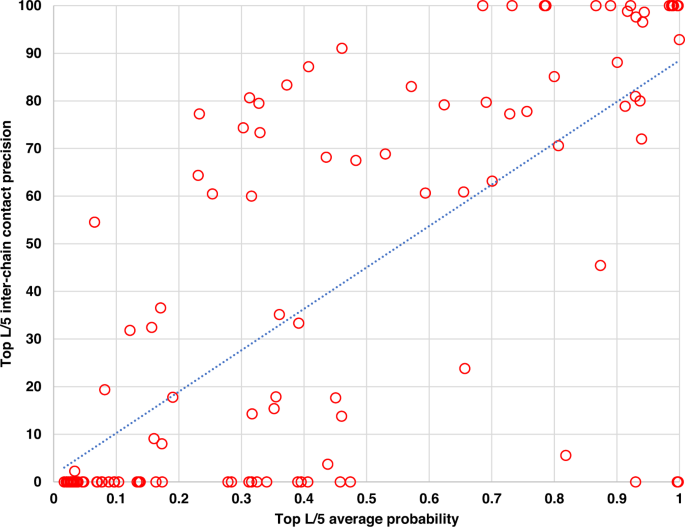
链间接触预测的精度与预测概率得分之间的高度相关
之前关于链内距离预测的工作27表明链内距离预测精度与预测概率得分具有较强的相关性,可用于选择预测链内距离图。在这里,我们研究了在链间距离预测中是否存在类似的相关性。数字4是4个测试数据集中每个目标的顶级L/5链间接触预测的精度及其概率得分的平均值。最高L/5链间接触精度与平均预测概率得分的相关系数为0.7345。高相关性表明CDPred预测的链间接触概率可以用来估计链间预测的置信度。
CDPred和AlphaFold2-multimer的比较
AlphaFold2-multimer是目前最先进的预测多聚体四元结构的方法。为了研究CDPred是否与AlphaFold2-multimer互补,我们比较了它们在四个数据集上的链间接触预测精度。这种比较并不完全公平,因为测试数据集和AlphaFold2-multimer训练数据集之间的冗余没有被去除。我们运行了最新版本(版本2)的AlphaFold2-multimer,没有模板来预测四个测试数据集中二聚体的四元结构。链间距离图从预测的第四系结构中提取。将图中的每个距离倒转,生成一个接触概率图,并与CDPred预测的链间接触图进行比较。补充图4给出了CDPred和AlphaFold2-multimer对四个测试数据集中每个目标的top L/2链间接触预测精度的逐目标比较。在大多数目标上,AlphaFold2-multimer比CDPred具有更高的顶部L/2精度。但是,对于AlphaFold2-multimer的top L/2精度小于10%的44个非常困难的目标,CDPred在15个目标上优于AlphaFold2-multimer,在25个目标上表现相同,在4个目标上表现较差。在两种方法性能不同的19个硬目标上,CDpred的平均精度为14.8%,远高于AlphaFold2-multimer的1.79%。的p差异的双侧两两t检验值为0.0068,说明差异显著。例如,对于7LB6目标,CDPred的top L/2精度为44.62%,远高于AlphaFold2-multimer的0%。目标的MSA的Neff为16.6。结果表明,CDPred与AlphaFold2-multimer是互补的,当目标非常困难且AlphFold2-multimer预测的置信度非常低时,CDPred尤其有用。CDPred的一个可能的应用是使用其预测的距离图来排序和选择由alphafold2 - multitimer预测的硬目标的不同的四元结构模型。
一个有趣的链间距离预测示例
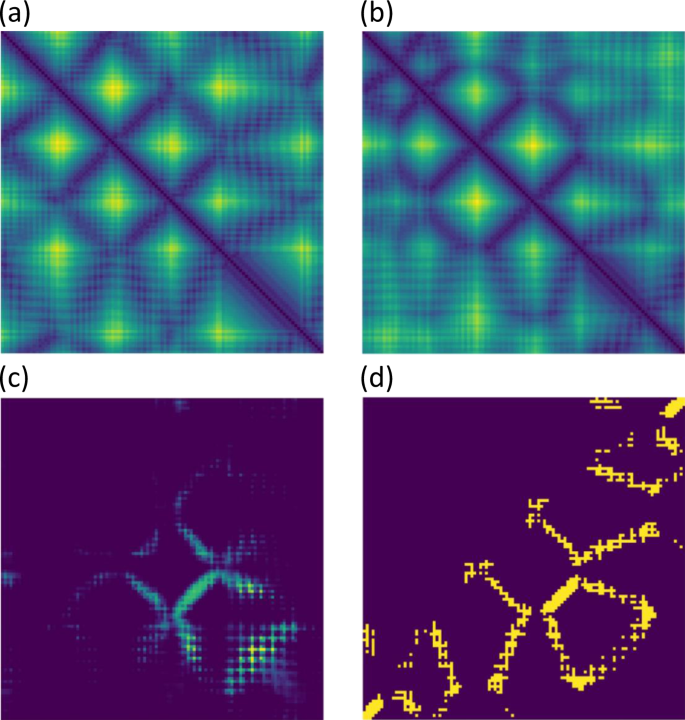
通常,当MSA较浅时,由于缺乏信息,链间距离预测的精度较低。然而,CDPred仍然可以准确预测一些MSAs较浅的目标的链间距离。数字5为CASP13同源二聚体靶T0991,距离图由matplotlib可视化28.它的MSA只有一个序列。的TM-score29AlphaFold2预测的T0991单体三级结构的褶数为0.3104,说明预测的三级结构褶数不正确。CDPred预测链间距离图得到的链间接触的顶部L/10、顶部L/5和顶部L/2的精度分别为72.73、68.18和56.36%,具有较高的精度。数据5一个,b如图所示为alpha fold预测的单体三级结构和单体真实三级结构的链内距离图。5度为CDPred预测的链间接触图,图。5 d真正的链间接触图。预测的链间接触图准确地回忆了大部分真实的链间接触。
方法
基于注意力的神经网络架构
数字6说明了基于通道型和空间型注意机制的CDPred的整体架构。CDPred以二聚体单体的三级结构为输入,提取单体序列和链内距离图。对于同型二聚体,由于一个同型二聚体的两个单体的序列是相同的,所以只用一个单体三级结构作为输入。利用单体序列搜索蛋白质序列数据库,生成二聚体的msa,生成残差-残差协同进化评分、行注意图和位置特异性评分矩阵(PSSM)作为输入特征(详见特征章节4.2)。CDPred的完整输入是所有输入特征的连接。
存储在多通道二维张量中的输入特征首先由二维卷积层转换,然后由Maxout层转换30.降低维数。Maxout层的输出被用作注意机制授权的一系列深层剩余网络块的输入。残差网络已广泛应用于计算机视觉、蛋白质链内距离和接触预测等领域5,7,31.在这里,我们将残差连接与其他有用的组件结合起来构造一个残差块,其中包括归一化块(称为RCIN),由行归一化层(RN)、列归一化层(IN)组成。32,实例规范化(IN)33用于规格化特征映射的通道注意挤压和激励(SE)块34用于捕捉不同特征通道的重要信息,并形成空间注意块35在通道注意块之后捕捉残基之间的信号。在残差块之后,使用带有softmax函数的二维卷积层将二聚体中来自两个单体的任意两个残差之间的距离划分为42个距离bin(即从2到22 Å的40个bin, bin大小为0.5 Å,加上0-2 Å bin和>22 Å bin)。同时预测了两种链间残基-残基距离:(1)该领域大多数现有工作中使用的离两个残基最近的两个重原子之间的距离(2)Cb- cb最近的一些作品中使用的两个残基之间的距离36,从而预测出两种距离图。
特性
CDPred的输入特征包含(1)单体链内距离图形式的三级结构信息,(2)成对共同进化特征,(3)序列氨基酸守恒特征,存储在L × L × N张量中(L为同型二聚体单体序列长度或异型二聚体两个单体长度之和(L1 + L2))。N为每对残基的特征通道数。
单体的三级结构信息
二聚体中单体的蛋白质三级结构信息表示为存储C和C之间距离的链内距离图b单体中两个残基的原子。对于同型二聚体,仅使用一个单体的三级结构计算的链内距离图(L × L × 1)。对于异质二聚体,由其三级结构计算出异质二聚体中两个单体的两个链内距离图(L1 × L1 × 1和L2 × L2 × 1),并分别作为(L1 + L2) × (L1 + L2) × 1维二聚体输入距离图的左上子矩阵和右下子矩阵相加。异质二聚体输入距离图的另一个区域的值设为0。在训练阶段,二聚体中单体的真三级结构被用来计算上面的链内距离图。在测试/预测阶段,使用AlphaFold预测的单体三级结构生成链内距离图作为输入。使用预测的三级结构作为输入更具有挑战性,但可以更客观地评估链间距离预测的性能,因为在大多数情况下,单体的真实三级结构是不知道的。预测的三级结构也对应于未结合的三级结构,这是蛋白质对接领域常用的术语。
进化论的特性
为同源二聚体或异源二聚体生成msa,作为计算其共同进化特征的输入。为了挑战深度学习方法,从噪声输入中有效地预测链间距离映射,在训练阶段,我们使用不太敏感的工具或较小的序列数据库来生成msa,但在测试阶段,我们使用最先进的工具和更大的数据库来生成所需的msa。具体来说,在训练阶段,对于同型二聚体,我们使用PSI-BLAST37比对Uniref90查找单体序列(2018-04)38为了生成msa,对于异质二聚体,我们遵循FoldDock中的步骤39使用HHblits40对Uniclust30(2017-10)进行搜索,分别生成两个单体的MSA,然后根据序列的有机体分类ID将两个MSA配对,生成异源二聚体的MSA。
在测试阶段,对于同型二聚体,我们使用HHblits根据大型奇妙数据库(BFD)搜索单体的序列。41和Uniclust30(2017-10),分别为同型二聚体的单链生成两个msa,分别用于生成输入特征,进行两次预测,平均后作为最终的预测距离图;对于异源二聚体,MSA的生成过程与EvComplex2相同42,利用手钻对Uniref90(2018-04)进行搜索,为两个单体各生成一个MSA,然后将两个MSA的序列配对,根据与每个物种中单体序列的最高序列同一性生成一个异源二聚体的MSA。同质二聚体或异质二聚体的MSA由统计优化工具CCMpred使用43生成残差-残差协同进化评分矩阵(L × L × 1)作为特征,并通过深度学习工具MSA transformer进行分析22生成残差-残差关系(注意)矩阵(L × L × 144)作为特征。L是MSA中的列数。
连续的特性
通过上述PSI-BLAST搜索生成的蛋白质序列剖面(即位置特定评分矩阵(PSSM))包含了残基守恒信息。将同型二聚体中单体的PSSM或异型二聚体中两个单体的PSSM垂直拼接成L × 20的形状平铺(即逐元素交叉拼接),得到L × L × 40维数的序列特征。
训练过程和超参数
深度神经网络使用上述输入特征来预测重原子距离图和Cb形状L × L × 42的距离图。42个通道存储了42个距离箱中两个残差之间距离的概率。将预测的链间距离图与真实的链间距离图进行比较,计算交叉熵损失,以调整训练过程中的权重。对于异质二聚体(L = L1 + L2), (L1 + L2) × (L1 + L2) × 42维的输出距离图既包含链间距离预测,也包含链内距离预测。仅使用链间距离预测计算交叉熵损失来训练网络,而忽略链内距离预测。CDPred的卷积层数设置为156,每个卷积层的过滤器数量设置为64。由于GPU内存限制,训练的批大小设置为1。我们使用学习率为1e-3的Adam优化器对模型进行前30个epoch的训练,以实现快速收敛,并使用开始学习率为1e-4的随机梯度下降,在剩余的50个epoch中每20个epoch进行10次缩减,以进一步减少训练损失。
数据集和评估指标
我们使用DeepHomo训练数据集19训练同二聚体链间距离预测器。整个数据集包括4132个C2对称的同二聚体蛋白。使用MMseq2去除与CASP/CAPRI实验目标盲测数据集(HomoTest1和HomoTest2)具有> =30%序列一致性的蛋白44, 4129个同型二聚体作为训练、验证和内部测试数据。和DeepHomo一样,我们选择其中的300个作为验证数据,300个作为内部测试数据,剩下的作为训练数据。DeepHomo使用的测试数据集包含从CASP10-13实验中收集的28个目标,被用作一个盲同二聚体测试数据集(HomoTest1).GLINTER使用的另一个测试数据集21,其中包括从CASP13和14中收集的23个同型二聚体目标,被用作另一个盲同型二聚体测试数据集(HomoTest2).两个盲测数据集有六个共同目标。
对于异二聚体,我们使用Apoc中的异二聚体45创建训练、验证和内部测试数据集。在40%序列一致性阈值处过滤出相似序列,去除与盲测数据集(HeteroTest1和HeteroTest2)序列一致性≥30%的序列后,还剩下3955个异源二聚体。我们随机选择其中的3576个作为训练数据,198个作为验证数据,181个作为内部测试数据。GLINTER使用的测试数据集包含来自CASP13和CASP14实验以及CAPRI实验的9个异源二聚体目标,被用作盲测试数据集(HeteroTest1).为了创建一个更大的盲测数据集,我们收集了PDB中09-2021年至11-2021年发布的异源二聚体。在以40%的序列身份阈值过滤出相似序列并排除>1000个残基目标的序列后,剩下55个异源二聚体创建另一个盲测试数据集(HeteroTest2).
由于GLINTER和DeepHomo两种外部方法预测的是链间接触而不是链间距离,为了更好地与CDPred进行比较,我们使用接触预测精度作为评价指标。具体来说,计算并比较了前5、10、L/10(或Ls/10)、L/5(或Ls/5)、L/2(或Ls/2)和L(或Ls)接触预测的精度(L:同型二聚体中单体的长度,Ls:异型二聚体中较短单体的长度)。类似的度量也广泛用于评估链内接触预测。因为DeepHomo和GLINTER预测链间接触的阈值为8 Å,所以我们使用相同的阈值将CDPred预测的距离图转换为二进制接触图。如果两个残基重原子之间的最小距离小于8 Å,则预测的链间接触是正确的。准确顺序,准确率46, AUC评分也用于评价CDPred的链间距离预测。精度顺序是第一个正确接触预测的秩除以二聚体的残留总数。AccRate是前10个链间接触预测中至少有一个是正确的二聚体的百分比。
报告总结
有关研究设计的进一步资料,请参阅自然组合报告摘要链接到这篇文章。
数据可用性
在本研究中产生的测试数据已保存在Zenodo数据库中,使用的是Creative Commons Attribution 4.0国际公共许可https://zenodo.org/record/6647564.本研究中使用的原始蛋白质二聚体数据可在CC0 1.0通用(CC0 1.0)公共领域奉献中获得https://www.rcsb.org/.本研究中生成的源数据在源数据文件中提供。本研究中使用的Uniclust30(2017-10)数据库可在以下地址获得:创作共用署名- sharealike 4.0国际许可https://wwwuser.gwdg.de/~compbiol/uniclust/2017_10/.本研究中使用的Uniref90(2018-10)数据库可在以下地址获得:CC BY 4.0国际创作共用属性许可证https://www.uniprot.org/help/uniref.本研究中使用的BFD (Big Fantastic Database)数据库请访问https://bfd.mmseqs.com/.源数据提供了这篇论文。
代码的可用性
CDPred的代码47可在以下网址索取:https://github.com/BioinfoMachineLearning/CDPred.
参考文献
结构-功能关系。食品蛋白质应用。80, 257(1997)。
斯派林,V.和米尔尼,L. A.分子网络中的蛋白质复合物和功能模块。国家科学院学报美国One hundred., 12123-12128(2003)。
Eickholt, J. & Cheng, J.预测蛋白质残基-残基接触使用深度网络和增强。生物信息学28, 3066-3072(2012)。
Adhikari, B., Hou, J. & Cheng, J. DNCON2:改进的蛋白质接触预测使用两级深度卷积神经网络。生物信息学34, 1466-1472(2018)。
王松,孙松,李志勇,张荣,徐娟。超深度学习模型对蛋白质接触图的精确从头预测。公共科学图书馆第一版。医学杂志。13, e1005324(2017)。
李勇,胡娟,张超,于东军。张宇。ResPRE:耦合精度矩阵与深度残差神经网络的高精度蛋白质接触预测。生物信息学35, 4647-4655(2019)。
吴涛,郭志军,侯俊杰,程杰。DeepDist:基于深度残差卷积网络的实值残差间距离预测。BMC生物信息学22, 1-17(2021)。
Senior, a.w.等。利用深度学习的潜力改进蛋白质结构预测。自然577, 706-710(2020)。
Adhikari, B. & Cheng, J. CONFOLD2:改进的接触驱动从头计算蛋白质结构建模。BMC Bioinforma。19, 22(2018)。
Rohl, c.a., Strauss, c.e., Misura, k.m., Baker, D。酶学方法(爱思唯尔,2004)。
Kandathil, s.m., Greener, j.g., Lau, a.m. & Jones, d.t.超快端到端蛋白质结构预测使未特征蛋白质的高通量探索成为可能。国家科学院学报美国119, e2113348119(2022)。
杨,J.等。利用预测残基间取向改进了蛋白质结构预测。国家科学院学报美国117, 1496-1503(2020)。
徐娟,王舒。CASP13中基于距离的蛋白质结构深度学习预测分析。蛋白质87, 1069-1081(2019)。
Jumper, J.等。高度准确的蛋白质结构预测AlphaFold。自然596, 583-589(2021)。
泽姆拉,a . LGA:一种寻找蛋白质结构三维相似性的方法。核酸测定。31, 3370-3374(2003)。
埃文斯等人。蛋白质复合物预测AlphaFold-Multimer。BioRxiv, 2021.2010。2004.463034(2022)。
高,M,中岛,安,D。,Parks, J. M. & Skolnick, J. AF2Complex predicts direct physical interactions in multimeric proteins with deep learning.Commun Nat。13, 1-13(2022)。
曾,H.等。ComplexContact:一个使用深度学习预测蛋白质间接触的网络服务器。核酸测定。46, w432-w437(2018)。
严昱&黄世永。均低聚蛋白复合物蛋白间残基-残基接触的准确预测。简短。Bioinforma。22, bbab038(2021)。
Roy R. S., Quadir, F., Soltanikazemi, E. & Cheng, J.预测蛋白质同型二聚体链间接触的深度扩张卷积残差网络。生物信息学381904-1910(2022)。
谢志,徐娟。蛋白质间接触的深度图学习。生物信息学38, 947-953(2022)。
Rao, R. M.等。在机器学习国际会议.8844-8856 (pmlr, 2021)。
何凯,张旭,任寿,孙俊仁IEEE计算机视觉与模式识别会议.770-778 (ieee, 2016)。
Quadir, F., Roy, R. S., Halfmann, R. & Cheng, J. DNCON2_Inter:利用单体多序列比对和深度学习预测同型二聚体和同型多聚体蛋白复合物的链间接触。科学。代表。11, 1-10(2021)。
Quadir, F., Roy, R. S., Soltanikazemi, E. & Cheng, J. DeepComplex:通过深度学习链间接触预测和基于距离的建模来预测蛋白质复合物结构的web服务器。前面。摩尔。Biosci。8, 716973(2021)。
Jones, D. T., Singh, T., Kosciolek, T. & Tetchner, S. MetaPSICOV:将联合进化方法用于精确预测蛋白质中的接触和长范围氢键。生物信息学31, 999-1006(2015)。
郭志刚,吴涛,刘娟,侯娟,程娟。基于深度学习的CASP14蛋白距离预测的改进。生物信息学37, 3190-3196(2021)。
亨特,J. D. Matplotlib:一个2D图形环境。第一版。科学。Eng。9, 90-95(2007)。
张勇,张志勇,张志勇。蛋白质结构模板质量自动评估的评分函数。蛋白质57, 702-710(2004)。
Goodfellow, I., ward - farley, D., Mirza, M., Courville, A. & Bengio, Y.在机器学习国际会议上。1319 - 1327 (PMLR)。
李勇,张超,贝尔,余东军,张勇。CASP13中多重原始共进化特征与深度残差神经网络的集成用于接触映射预测。蛋白质87, 1082-1091(2019)。
毛玮,丁玮,邢玉玉,龚海华。AmoebaContact和GDFold作为快速新生蛋白结构预测的管道。Nat,马赫。智能。2, 1-9(2019)。
Ulyanov, D., Vedaldi, A. & Lempitsky, V.实例规范化:快速程式化所缺少的成分。预印本在arXiv:1607.08022(2016)。
胡俊,沈良,孙国强。挤压-激励网络。在IEEE计算机视觉与模式识别会议.(ieee, 2018)。
吴邵,朴俊杰,李俊英。& Kweon, i.s. Cbam:卷积块注意力模块。在欧洲计算机视觉会议(ECCV)2018.3-19。
苏坦尼卡泽米,E., Quadir, F., Roy, R. S.,郭卓,程,J.基于距离的蛋白质四阶结构的链间接触重建。蛋白质90, 720-731(2022)。
巴格瓦特,M. &阿拉文,L.。比较基因组学(施普林格,2007)。
米尔迪塔,等人。聚类和深度注释的蛋白质序列和排列的Uniclust数据库。核酸测定。45, d170-d176(2017)。
Bryant, P., Pozzati, G. & Elofsson, A.使用AlphaFold2改进蛋白质-蛋白质相互作用的预测。Commun Nat。13, 1-11(2022)。
Remmert, M., Biegert, A., Hauser, A. & Söding, J. HHblits:通过HMM-HMM对齐进行闪电般的快速迭代蛋白质序列搜索。Nat方法。9, 173(2012)。
Steinegger, M., Mirdita, M. & Söding, J.蛋白质水平的组装增加了从宏基因组样本的蛋白质序列恢复数倍。Nat方法。16, 603-606(2019)。
格林,A. G.等。利用从基因组序列计算的协同进化,在残基分辨率上大规模发现蛋白质相互作用。Commun Nat。12, 1-12(2021)。
Seemayer, S., Gruber, M. & Söding, J. ccmpred -快速和精确预测相关突变的蛋白质残基-残基接触。生物信息学30., 3128-3130(2014)。
Steinegger, M. & Söding, J. MMseqs2使敏感蛋白质序列搜索用于大量数据集的分析。生物科技Nat。》。35, 1026-1028(2017)。
高,M. & Skolnick, J. APoc:相似蛋白口袋的大规模鉴定。生物信息学29, 597-604(2013)。
赵卓,龚霞。基于深度学习架构的蛋白质-蛋白质相互作用界面残留对预测。IEEE / ACM反式。第一版。医学杂志。Bioinforma。16, 1753-1759(2017)。
Guo, Z. & Cheng, J. BioinfoMachineLearning/CDPred:与描述CDPred的《自然通讯》论文的发表同时发布。Zenodohttps://doi.org/10.5281/zenodo.7218709(2022)。
确认
这项工作得到了美国能源部(授权#:DE-AR0001213 (J.C.), DE-SC0020400 (J.C.)和DE-SC0021303 (J.S.和J.C.)),国家科学基金会(授权#:DBI1759934 (J.C.)和IIS1763246 (J.C.))和美国国立卫生研究院(授权#:R01GM093123 (J.C.), R01GM146340 (J.C.)和R35GM118039 (J.S.))的部分支持。
作者信息
作者及隶属关系
贡献
J.C.构想了这个计划。Z.G.和J.C.设计了实验。z.g进行实验并收集数据。z.g., j.c., j.l.和J.S.分析了数据。J.L.和J.S.提供了一些数据集。Z.G.和J.C.撰写了手稿。z.g.、j.c.和J.S.编辑了手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
同行评审
同行评审信息
自然的交流S感谢匿名审稿人对这项工作的同行评议所作的贡献。同行评审报告是可用的。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
源数据
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
郭志刚,刘俊,郭志刚,郭志刚。et al。用二维基于注意力的深度神经网络预测蛋白质复合物的链间距离图。Nat Commun13, 6963(2022)。https://doi.org/10.1038/s41467-022-34600-2
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41467-022-34600-2