文摘
噬菌体和细菌之间的进化军备竞赛,发展抗噬菌体和细菌噬菌体resistance-countering突变的报复,是一个主要的创新驱动力的分子和遗传多样化。然而,试图在实验室中重现这种持续的报复动态挑战;实验室共同进化实验的噬菌体和细菌通常表现在混合环境中,往往会导致快速停滞遗传变异性。在这里,培养能动的大肠杆菌的裂解性噬菌体T7游泳板,我们观察复杂的时空动力学与多个基因多样化自适应周期。系统地定量超过10000 resistance-infectivity表型进化的细菌和噬菌体之间的隔离,我们观察到的多样化为多个共存生态型之间显示一个复杂的交互网络与宿主范围扩张和host-switch权衡。全基因组测序的进化噬菌体和细菌分离显示一组丰富的适应性基因突变在多个基因通路包括在未与phage-bacteria交互。综合改造这些新的突变,我们发现phage-general phage-specific抗性表型以及很强的协同作用与经典已知的噬菌体抗性突变。这些结果强调空间结构的重要性和迁移驾驶phage-bacteria共同进化,揭示新的分子机制提供一个具体的系统跨不同phage-bacterial系统。
介绍
自然微生物群落港内广泛的物种相互作用空间异构环境,如土壤、植物或人类的肠道。在这些复杂的社区中,物种多样性的关键因素是细菌和噬菌体寄生虫之间的敌对的关系1,2,3,4。当受到裂解性感染细菌时,选择优势phage-resistant细菌产生,和连续噬菌体能够克服这种进化的细菌耐药性5,6,7,8,9。这些选择压力,因此,引起持续的相互适应,导致新的阻力和counter-resistance途径的进化10,11,造成细菌和噬菌体的基因组的高时空的多样性。
形成鲜明对比,当实验室控制混合液体环境中培养phage-bacteria共同进化通常停滞后几自适应周期12,13,14,15,16,17,18,19,20.,21,很少表现出持续的共同进化动力学22,23,24,25,26。共同进化理论认为,长期的开始和多样化可能是通过空间结构27,28,29日,允许本地交互和亚种群的形成。事实上,空间结构引入bacteria-phage共同进化,通过培养他们作为殖民地,在生物膜,在unshaked或小连接液体环境中,或土壤中,证明了长期phage-bacteria共存和多样化30.,31日,32,33,34,35,36,37,38。然而,一个全面的长期时空跟踪自然迁移的细菌和噬菌体对空间结构的实验室环境中失踪。特别是,它是未知的结构化的环境是否会促进与更多的自适应的进化步骤和更高程度的基因多样化,有可能发现新的基因通路。
进化停滞和有限的多样性的挑战在混合环境中尤其体现在实验室里共同进化动力学大肠杆菌和溶解性噬菌体T7。先前的研究表明,共同进化的大肠杆菌和T7或噬菌体T3密切相关,在恒化器快速达到停滞,通常只有1.5周期后(bacteria-phage-bacteria突变)12,13,15,21。噬菌体和细菌显示有限的遗传多样性。在噬菌体中,观察到的遗传变异仅限于一个噬菌体tail-fiber基因密码子15。在细菌突变都集中在脂多糖(LPS)生物合成基因15,39也像噬菌体受体功能吗40,trxA基因15噬菌体编码的DNA聚合酶持续因素41或转录监管机构rcsB导致colanic酸生产过剩21形成一个屏障T7感染42。这个有限的进化多样性与丰富的遗传通路形成鲜明对比中发现综合遗传T7-resistance屏幕42,43,44,一大部分原因要归咎于全球交互和人工人口中固有的稀释混合环境和实施强有力的选择适当的突变体。
这里,开发一个时空长期bacterium-phage共培养试验,我们发现多步co-retaliating适应路径导致spatially-coexisting细菌和噬菌体表型和基因型的多样化。适应最近推出了swim-based设置,趋药性的细菌和裂解性噬菌体传播对大型半固体琼脂板上45,46,我们共同进化大肠杆菌和他们的T7噬菌体在两轮一周的进化,观察持续增长,移民和感染周期,导致进化的多样化和噬菌体和细菌的马赛克空间分布。高通量的映射之间的交互进化的细菌和噬菌体显示不同的阻力和传染性类,演示宿主范围扩大和主机切换动态。多元化进一步支持由多个基因变异,在已知和未知大肠杆菌t7的交互途径,包括一个新的阻力函数在两个细菌基因。这些发现强调空间组件的重要性在促进多元化和基因创新phage-bacteria共同进化。
结果
游泳板成像方法揭示了循环时空bacterial-phage联合进化动力学
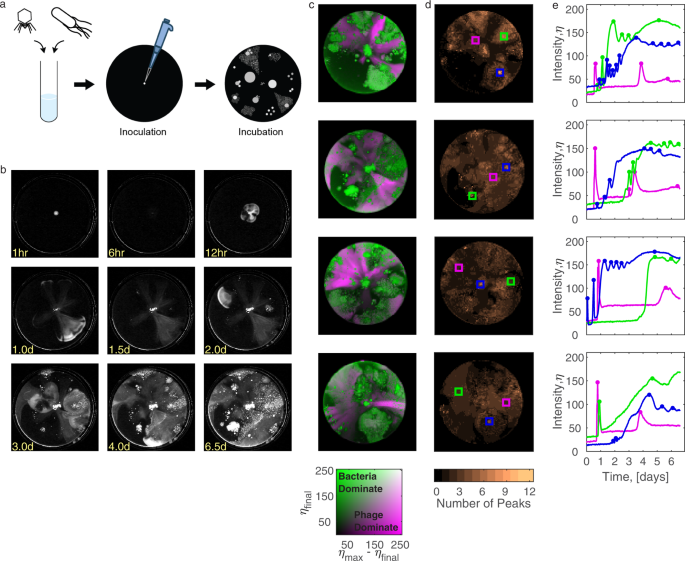
我们建立了一个空间结构共同进化平台,适应游泳板填充运动型长期潜伏的细菌和裂解性噬菌体。萍et al。45,我们接种细菌和噬菌体的混合物游泳板的中心和监控他们的增长动态成像方法。扩展这些早期的研究,我们允许共同进化四板复制总共15天;最初,细菌和噬菌体(图七天自发协同进化。1 a, b,补充电影1;方法:共同进化实验),那么当地的小样本第七天reinoculated在新鲜游泳板和共同进化持续了额外的8天(补充图。1 a, b;补充的电影2;方法:共同进化不断实验)。长期孵化一个主要挑战是水蒸发,从而增加了有效的琼脂浓度和掩盖了成像由于凝结在盖子上。为了减少蒸发,我们密封板的加热玻璃盖,防止蒸发和冷凝47。获取图像分辨率高,盘子都存储在定制的暗场照明箱(补充图。2),延时的图片每隔10分钟。,使高度准确的记录和分析。
一个实验设置长期演进:混合物的运动型MG1655细胞和T7病毒粒子植入的中心四个复制游泳板(直径14厘米,70毫升0.3%的琼脂磅)和孵化30°C为7天。b样品代表的暗场图像复制在不同的时间点。c最终的像素强度的复合的热图(η最后绿色)和最大的区别,最终强度(η马克斯- - - - - -η最后洋红色),分别代表细菌或噬菌体的主导地位在每个四个复制的最后时间点(2 d传说,底部)。d热图的观察growth-lysis周期,列举强度量化的山峰在每个位置(平均超过1.4毫米宽的区域)。e在特定位置(矩形强度随着时间的推移d),选择代表地区bacteria-dominance phage-dominance(绿色)或地区(红色)的端点和高度动态的区域与多个峰值强度(蓝色)。点代表检测峰值。源数据作为源数据文件提供。
获得的延时视频显示,紧随其后的是一波又一波的传播细菌噬菌体感染复发在所有复制(补充电影1)。不像在液体环境中,进化结束时并没有细菌的主导地位;分析像素强度在空间和时间的变化,我们观察到异构板殖民与生物共存的单独空间补丁端点,根据最终的像素强度高的地区,明显表示细菌生长,与高的区域差异最大和最终的强度值,代表噬菌体增殖(无花果。1 c分别,绿色和红色)。计算像素强度峰值的数量随着时间的推移,在小网站(1.4毫米宽广场)说明当地增长和感染周期。的平均数量在所有复制周期是2;然而,几个不同的位置显示更高growth-infection周期(12个周期;无花果。1 d, e补充图。3)。相比之下,控制的一组4 bacteria-only no-phage,游泳板没有显示多峰动态,确认噬菌体的作用在这些周期(方法:no-phage控制;补充的电影3;补充图。3)。虽然在最初的共同进化(图峰值振幅逐渐减小。1 e),接种在不断板块再次引发了强烈growth-infection周期和共存持续8天(补充额外的电影2;补充图。1汉英;补充图。3),这表明最初的共同进化的振幅末轮是由营养损耗而不是ecology-evolutionary停滞。综上所述,不同于典型的混合环境中观察12,13,21,大肠杆菌和T7正在进行游泳板体现共同进化与报复动力学15天,没有物种全球流行的在任何时间点。
交互的映射进化bacterium-phage隔离揭示表型多样性通过军备竞赛和主机开关动力学
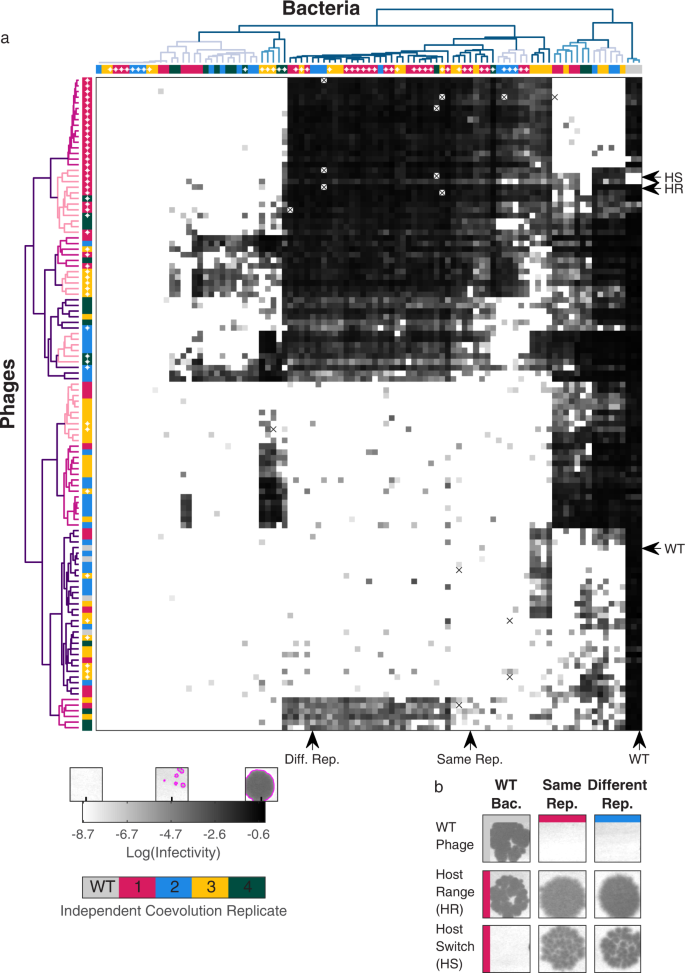
发现细菌和噬菌体在共同进化的进化表型多元化板块,我们取样多个站点从所有盘子终点的初始和持续的共同进化实验(51个不同的网站:29从最初从持续不断的实验和22)。在每个采样点,两个细菌克隆和四个噬菌体克隆分离(方法:单菌落分离,双色空斑实验;补充图。4)。噬菌体多样化样本,每个样本的噬菌体克隆站点被隔离在一个草坪上包含的混合物不同标记野生型细菌和细菌克隆分离出相同的网站(补充图。5)。整体图书馆94细菌分离和112噬菌体分离收集(补充数据1,2)。评估发展阻力和传染性表型的细菌和噬菌体分别隔离,我们测量所有成对bacteria-phage交互使用高通量空斑实验omni-tray琼脂板(共有11252 bacterium-phage交互包括3细菌野生型控制和4噬菌体野生型控制,方法:相互传染分析)。对于每个交互,我们自动量化一个“传染性分数”反映斑块的大小和浊度(0到1,方法:斑块图像自动处理;补充图。11)。传染性得分的可靠性测试通过测量三个野生型传染性细菌的相关性分数和所有四个噬菌体野生型传染性分数显示一个好的协议(R2= 0.73 - -0.81细菌野生型之间复制,R2= 0.99 - -1.00噬菌体野生型之间复制;补充图。6 c, d)。最后,细菌和噬菌体分离与分级聚类成集群综合交叉矩阵基于欧几里得距离成对的传染性分数(方法:系统建设;无花果。2)。
一个相互传染的结果分析的噬菌体分离(116行)在所有细菌隔离(97年,列)。方块矩阵表示的传染性分数,计算的区域显示细菌噬菌体裂解倍间隙在这个领域(斑块的方法:自动处理图像)。缺失的数据点是标有x。隔离下令分层聚类是基于表型欧几里得成对的距离,与每个表型类彩色系统树图中不同的颜色(左和分支机构)。每个隔离的颜色(系统树图的叶子)代表的四个独立进化复制和灰色表示祖先的应变。小白点系统树图的叶子说明隔离取样不断进化的盘子。源数据作为源数据文件提供。bphage-bacteria隔离感染检测的图像(箭头一个)展示宿主范围扩张(人力资源)和主机切换(HS)噬菌体。WT -野生型Bac Diff。不同。——细菌、众议员——复制。
系统的表型出现传染性细菌和噬菌体分离,我们确定了多个不同的适应性表型组成的一个复杂的交互网络的细菌和噬菌体。细菌和噬菌体多样化至少9和12个不同的表型类,分别(无花果。2系统树图分支的颜色;方法:系统树图施工)。值得注意的是,许多这些类在不同的进化复制独立进化,表明平行进化(无花果。2系统树图的叶子的颜色)。一些表型类,特别是最耐药细菌类和最广泛的范围噬菌体类,主要发现在连续板,支持进化军备竞赛。进一步分析了网络结构的演变我们利用BiMat表型48图书馆和发现,符合预期不同的和相互关联的环境49,50进化细菌扩大他们的电阻剖面和噬菌体扩展他们的宿主范围比他们的祖先进化而来(所有4共同进化板块显示重大nestedness, P < 10−4;只有两人也表现出显著的模块化,P= 0.01,P< 10−4方法:BiMat分析;补充图。7)。然而,一些噬菌体进行专门化增加对进化传染性细菌而显著减少对野生型传染性细菌(“主机开关”,无花果。2 b)。比较一个更直接的感染的传染性分数斑块测量浊度,相似的表型特征观察,包括host-switch现象,然而区分能力较弱的感染和较低的噪音(补充图。6 a、b;方法:斑块图像的自动处理)。在所有,我们系统的表型分析显示广泛的多样性的阻力和传染性类显示信号的并行进化军备竞赛51host-switch动力学。
一个回归模型识别自适应变异已知和未知phage-bacteria交互途径
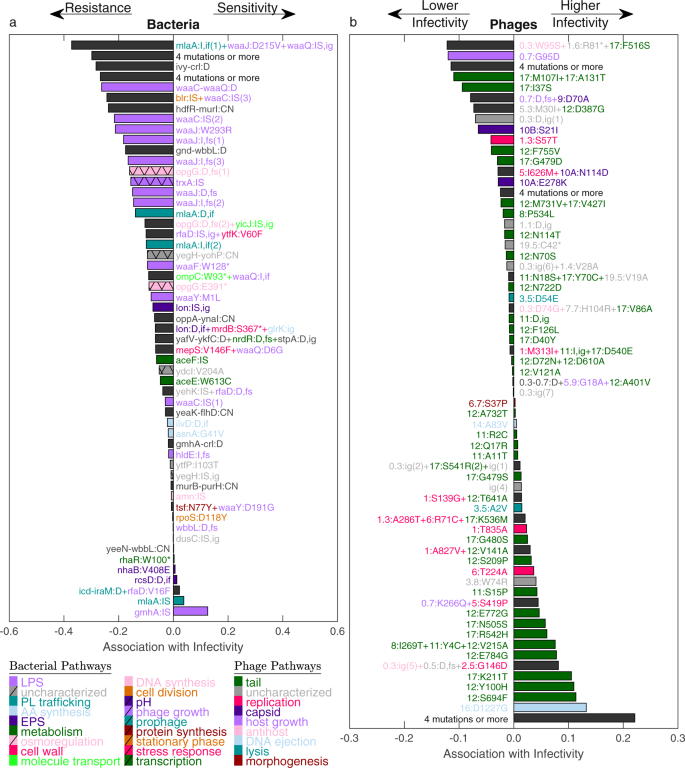
在基因型的层面,协会之间的突变和感染表型透露的重要基因和通路为适应细菌和噬菌体。全基因组测序的细菌和噬菌体隔离库显示40个snp, 49 indels和12个细菌隔离放大,和192个snp和18 indels噬菌体分离(补充数据3和4分别)。细菌和噬菌体显示产生同义突变比率高,表明积极的适应进化(细菌:5基因间32产生3同义,PdN / dS= 0.0078;噬菌体:15基因间,154产生的,23同义,PdN / dS< 10−4;方法:测序分析;补充图。8)。量化协会与感染表型变异,我们建立了一个线性回归模型的传染性得分每一对bacterium-phage(无花果。2)作为他们的联合基因型的函数。为了避免过度拟合和解决紧凑的突变,我们使用套索解决这个模型技术(至少绝对收缩和选择算子,方法)52。对传染性细菌突变强烈偏向负面联想,如预期的阻力突变(图。3)。符合之前的报道,这些细菌突变导致有限合伙人阻力包括突变生物合成,胞外生产(朗53),trxA丙酮酸脱氢酶编码美国节能54特别结合T7gp0.4与一个未知函数55。有趣的是,两个额外的基因,获得几个电阻突变(突变与传染性相关负面,无花果。3)之前没有联系大肠杆菌抵抗T7: osmoregulated周质的葡聚糖(功能)生物合成蛋白质opgG和膜间隙磷脂转运蛋白mlaA。专注于mlaA突变,而四分之三的突变被分配一个负系数(“mlaA:我,如果(1)”、“mlaA:我,如果(2)”、“mlaA: D,如果”,无花果。3补充数据3),一个突变收到积极的系数(“mlaA:”,无花果。3补充数据3)。这种插入序列突变造成的破坏mlaA基因,而其他三个短的插入或删除突变导致的在坐标系添加或删除2 40密码后密码子。有趣的是,隔离携带“mlaA:”也携带着一个短在坐标系indelsmlaA(Bac34 Bac33补充数据3),这表明增加的阻力在坐标系的突变造成的mlaA抵消了插入序列突变,破坏了基因。同样,积极系数最高的被分配到一个插入序列的突变gmhA(“gmhA:”,无花果。3),预计将导致短有限合伙人在野生型表型和增加T7抵抗背景42。这种突变出现在一个孤立的插入在坐标系mlaA(Bac71补充数据3),它显示了相比其他隔离携带类似的敏感性增加mlaA突变体和野生型gmhA(补充数据3和补充数据5),反映在积极与传染性。自gmhA对有限合伙人生物合成至关重要,它是可能的影响mlaA突变磷脂交通撤销了突变的有害影响gmhA有限合伙人生产。
基于联合协会的传染性和突变对所有phage-bacteria交互对回归模型,显示了所有的细菌(一个)和噬菌体(b拉索)突变与非零系数。酒吧的颜色代表每个突变基因的生物通路,与黑条表明突变影响了数个基因不同的生物学通路。AA -氨基酸,EPS -胞外,有限合伙人,脂多糖,PL -磷脂。突变是根据他们的类型:拷贝数变异(CN)、删除(D),插入(I),插入序列(是),基因间(ig), inframe(如果)和框移(fs)。指数(在括号中)添加到不同的突变与相同的标签。一开始每个噬菌体的突变数量名称(b)指噬菌体基因数量(“gp”省略了由于有限的空间)。例如,11:R2C代表改变第二个噬菌体尾管基因的密码子全科医生11。源数据作为源数据文件提供。
与细菌突变相比,噬菌体的突变包括更多的突变相关负面与传染性(无花果。3 b),可能是由于强烈的非线性等host-switch(无花果。2 b)和样本少的能力适应噬菌体基因型,在盘子里幸存下来。包括噬菌体的突变,如预期的那样,tail-fiber基因的突变gp17,还在尾管的基因gp11和gp12甚至包括非结构性基因gp16和gp1中、编码DNA弹射蛋白质和RNA聚合酶,分别。5相关突变高度传染性的组合,包括单核苷酸多态性gp1中(T23A),gp2(D12G),gp16(I197T)和gp11(E164A)以及一个在坐标系插入序列gp11被发现在一个噬菌体分离明显延长宿主范围(图。3 b与传染性,突变与最高协会;Phg29补充数据4和补充数据5)。最后,同样的关键基因,包括尾噬菌体基因,有限合伙人和EPS生物合成基因,以及新发现的mlaA和opgG,也确定了互补的平行进化分析基因突变超过预期偶然(方法、Multi-mutated基因),加强这些基因作为驾驶适应性进化(补充图。9)。
基因突变的重建mlaA和opgG表明这些基因在抗噬菌体的新角色
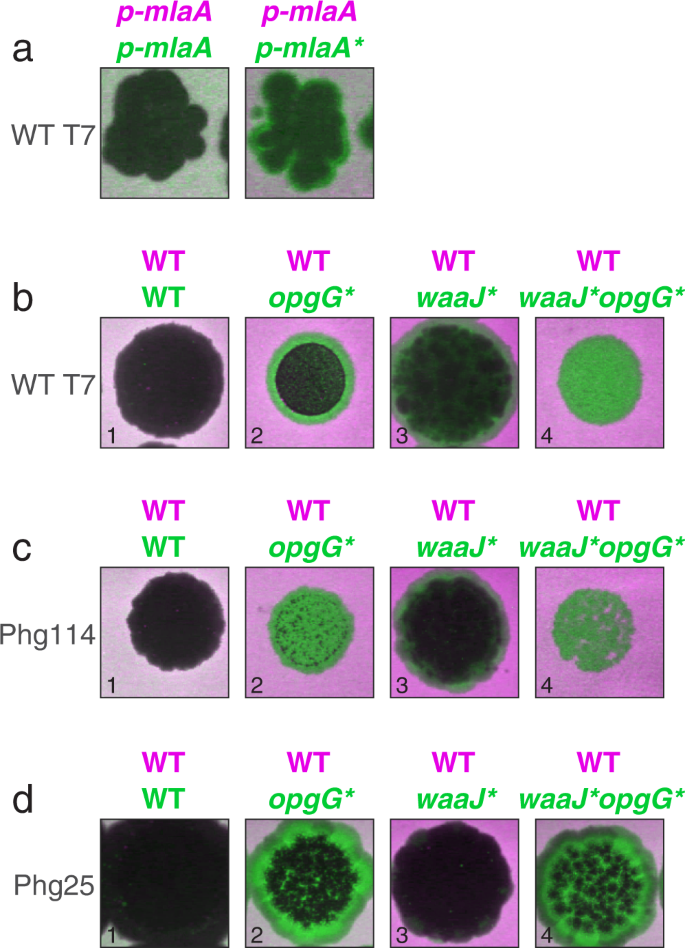
我们证实了新发现的噬菌体抗性作用mlaA和opgG通过合成重建和竞争表现型变异。专注于mlaA的祖先应变转换基因质粒表达特定在坐标系删除进化实验(图中观察到。3”,如果“mlaA: D),之前报道作为主导功能变异56,一个温和但健壮的优势野生型菌株时参加了野生型噬菌体的存在(无花果。4;染料在互换补充图。10;方法:重建mlaA和opgG突变体,双色空斑实验)。此外,实际的独立抗性表型分别衡量甚至可能比观察到的双色空斑实验实验由于支持噬菌体感染的敏感菌株的存在。专注于opgG基因,综合重建一个无义突变进化实验(图中观察到。3“opgG: E391 *”),我们最初观察到只有轻微的抵抗优势祖先背景野生型噬菌体(无花果。4 b在补充无花果,染料交换。10 b),与缺乏一致opgG在先前的遗传屏幕T7阻力42,43,44。然而,注意到所有的opgG突变隔离进行额外的有限合伙人葡糖基转移酶的突变waaJ基因,然后,我们构建了一个双突变体的opgG突变和waaJ突变(waaJW293R,最经常观察突变进化实验)。值得注意的是,虽然这waaJ单独突变也只引起轻微的表型,这两个突变显示强大的协同交互;的opgG突变表型有强劲阻力,当添加上waaJ突变背景(图。4 b在补充无花果,染料交换。10 bWT T7)。此外,我们还发现,当面对一些进化噬菌体,opgG明显的抗性表型的突变就足够(无花果。4 c, d补充图。10 c, d)。总的来说,这些结果显示一个新的角色opgG和mlaA在T7阻力。
一个与野生型的双色空斑实验T7噬菌体(WT T7)发现YFP和mCherry标记的野生型菌株携带质粒表达mlaA野生型(p -mlaA)或mlaA突变体(p -mlaA*)基因56。左:控制与表达野生型菌株mlaA;右:应变表达mlaA*和标记YFP(绿色)有一个小的选择性优势菌株表达野生型mlaA(红色标记mCherry),体现绿色斑块的光环在边境地带(方法:双色空斑实验)。b一个opgG突变体有一个轻微的T7阻力优势野生型(比较面板b2和野生型控制,面板b1)和强大的协同作用waaJ突变(在面板b4比较双突变体与单一突变体板b2, b3)。c,d与野生型T7阻力不同,当参加的存在两个选择进化T7噬菌体(Phg114,cPhg25,d在补充数据看到基因型4和传染性表型在补充数据5),opgG单独突变提供了强劲阻力(c2, d2)和结合waaJ突变(c4, d4),表明phage-specific利用这种突变。
讨论
使用长期共同进化空间结构化的环境中,我们显示的多元化大肠杆菌和T7成不同的阻力和传染性类通过多个步骤的互相适应。经常与有限的实验室共同进化中观察到混合设置,在空间结构化游泳板,我们观察到持续增长和感染周期导致显著的表型变异,包括初始步骤噬菌体形态通过主机开关,一般来说一个罕见的现象在实验室实验57,58,59,60只在人为和报道T7定制系统61年。在基因型的水平变化也很明显,一个Lasso-based phenotype-genotype协会分析显示广泛的耐药机制,包括两个潜在的小说T7通过突变耐药机制mlaA和opgG。的耐药表型opgG增加了相关的观察endogenous-lysis抵抗功能缺乏大肠杆菌细胞62年,T7-resistance transposon-mutagenizedopgH驻留在相同的操纵子基因opgG44。的抗性表型mlaA突变可能与最近显示hyper-vesiculation表型的突变56,63年。外膜囊泡分泌通常是增加抗噬菌体通过不可逆结合的噬菌体囊泡64年,65年针对T7,表明类似的抵抗机制。进化噬菌体显示高遗传变异性与50多个独特的尾巴纤维基因突变事件gp17和40多个独特的尾管基因突变事件gp12与增加相关,其中许多是传染性的套索模型。突变与传染性也发现non-tail RNA聚合酶相关基因如噬菌体编码gp1中和gp16基因参与DNA弹射。
我们的结果不同于共同进化结果混合设置,在研究,关注共同进化的大肠杆菌和T7或密切相关的T3噬菌体达到停滞后只有1.5周期(bacterial-phage-bacteria突变)12,13,15,21;平行进化显然也在基因型的层面,在一个有限合伙人与细菌第一电阻突变生物合成基因,噬菌体报复相同尾噬菌体基因密码子的突变,和细菌第二电阻突变在额外的有限合伙人生物合成基因,trxA15或胶囊的合成调控基因rcsBC21。我们的研究显示了突变路径和发现其他基因的突变和其他途径。空间结构很可能扮演了一个角色在允许较高的遗传多样性,尽管其他因素如人口规模和整体的进化时间可能也扮演了一定的角色。
我们的研究也有一些局限性。首先,取样做了只有在每个共同进化的终点。添加时间采样方法,虽然挑战由于污染的风险,将允许一个更详细的时间重建bacteria-phage co-phylogeny,揭示了按时间顺序的适应措施并确定bacteria-phage报复突变。第二,我们的研究依赖于之前文献相比,混合实验。我们注意到建立一个完全匹配的混合控制是很重要的,虽然在技术上和概念上的挑战。一个关键挑战是匹配的人口规模随着时间的增长将迅速分化的模式:在混合设置细菌成倍增长,同时,与增长大约是二次的游泳板,快速增长的细菌在前面和细菌生长缓慢。此外,游泳板不同于摊位和环境两个方面:一是空间结构,另一个是迁移(而不是固体,non-swim琼脂板)。这不是明显的如何正确设置液体实验来解决这两个方面。最后,在这项研究中我们没有参与遗传重建噬菌体的突变,这将有可能揭示新的噬菌体基因功能和细菌突变和噬菌体的突变之间的相互作用。
我们预期,扩大我们的方法更长的发展时间和更大的空间尺度上可以帮助揭示更多未知的阻力和counter-resistance机制大肠杆菌——T7和其他bacterial-phage系统,在小说的发现可能帮助噬菌体国防和anti-defense系统。应用于临床相关病原体及其噬菌体,这样的实验也可以帮助预测互相适应措施设计耐用噬菌体疗法66年,67年,68年,69年,70年。总的来说,这些结果提供一个一般的选择方案,以及分析方法,可以帮助揭示小说phage-bacteria相互作用的分子机制。
方法
品种和生长条件
所提供的实验与噬菌体T7请黛比Lindell (Technion——以色列理工学院),和一个能动的大肠杆菌MG1655应变携带IS1上游flhD请提供的阿迪Vaknin(耶路撒冷希伯来大学的)转化质粒表达YFP既定,CFP或mCherry和卡那霉素抗性基因71年。细胞培养在磅(10 g / L胰蛋白胨,5 g / L酵母提取物、5 g / L氯化钠在蒸馏水中,热压处理过的)补充了30µg /毫升卡那霉素在30°C用颤抖的除非另有注明。一个T7溶解产物是由传播100年μl最初的T7噬菌体在10毫升的指数增长的股票MG1655细胞磅(没有抗生素,OD600年≈0.1),直到达成文化间隙(75分钟。在30°C)。溶菌产物是消毒0.22µm Millex-GV过滤器,补充1%氯仿和储存在4°C的玻璃管。噬菌体浓度估计的电镀系列稀释溶解产物混合100μl MG1655细胞和4毫升暖0.7%琼脂LB-agar板块(30µg /毫升卡那霉素)和计算空斑形成单位(pfu),除非另有说明。
共同进化实验
游泳板是由注入70毫升的热压处理过的半固体培养基(0.3%的琼脂磅,高压灭菌后无菌蒸馏水添加弥补蒸发卷)补充了30µg /毫升卡那霉素每四个培养皿聚苯乙烯(⌀14厘米)。盘子被设置为1 h在室温和放在定制暗场成像盒配备LED灯垫(补充图。2)定位在一个单反相机(佳能EOS 100 d配备了佳能EF-S 18-55 mm f / 3.5 - -5.6二是单反镜头)在一个温度可控的房间(30°C,湿度70%)。长期以来琼脂板易受污染,细菌荧光标记(YFP, CFP)被用来允许视觉检测污染。MG1655-CFP和MG1655-YFP一夜之间文化被稀释1/100磅(25 ml, 30μg /毫升卡那霉素),孵化3小时(OD600年≈0.23),离心10分钟再悬浮于190年在2465 g和集中μl磅。集中文化被采样CFU计数和混合在一个1:1体积比。但是,回想起来,YFP标记已经主导了起初,因此标志的信息比没有利用和演化MG1655-CFP隔离被排除在后续的分析。接种前的盘子,30μl细胞混合30μl 10−6稀释T7溶解产物5μl混合文化(6∙106±1∙106MG1655-YFP细胞、8∙105±3∙105MG1655-CFP细胞,13±2 T7病毒粒子)接种每个板的中心。为了避免凝结在盖子上,盘子满是一个预热电热玻璃(85 cm X 65 cm, Seaclear工业LLC)连接到一个电源(OFI电子有限公司)设置为15 V, 1.48。玻璃的温度监测,范围从30 -°C在整个实验过程。捕获图像自动每10分钟(佳能EOS效用,暴露= 0.5 s,光圈F = 10, ISO = 100,白平衡=日光)在7天(共有954张图片)。
抽样
孵化后7天,5 - 8选择区域从每个板采样(补充图。4)由当地温和的混合软琼脂和移液5μl陷入245μl磷酸缓冲盐(pH值7.4,Sigma-Aldrich p - 5368,一袋是溶解在一个升蒸馏水和热压处理过的)。140μl每个样本和70μl甘油50%(最终浓度16.7%)和存储在−80°C。获得分离噬菌体样本,从每个样本被混入了11 100μlμl氯仿(~ 10%)并存储在小玻璃管在4°C。
共同进化不断的实验
四个新游泳板准备如上所述。四种选择从最初的实验中,样品从每个复制(补充图洋红色的样品。4),被解冻,接种(10μl甘油股票)板中心。孵化和成像继续如上所述共8天(1153张照片),然后从每个板2和12个区域之间采样如上所述(补充图。4)。
No-phage控制
四个新游泳板准备如上所述。MG1655-YFP一夜之间文化被稀释1/100磅(25 ml, 30μg /毫升卡那霉素),孵化3 h,离心10分钟再悬浮于190年在2465 g和集中μl磅。15μl集中文化的混合45μl磅(取代T7和MG1655-CFP文化卷在共同进化分析)和5μl接种的中心四个游泳板(准备如“共同进化实验”中描述的)。板被放置在暗场成像的设置(补充图。2),覆盖着一个加热玻璃(~ 32°C)。盘子被孵化(30°C,湿度70%)为33 h和成像每10分钟。如上所述。
图像分析
图像分析与定制MATLAB脚本。每个实验的第一形象(初始共同进化/持续共同进化/ no-phage控制)被从所有后续图像减少背景噪音,周围的图像裁剪板,只有红色通道显示最高的信噪比。时间流逝的视频共同进化轮都由LRTimelapse 5.6.0 (24 FPS和¼速度)从300年(初始实验)或340(持续实验)图像对数增加的时间差距。时间流逝no-phage控制的电影是由Adobe Premiere Pro 2022和包含所有200个图像与常数时间差距。计算生物优势,像素值是平均值的地区10 x 10 px2平均和峰值分析值进一步滑动时间窗的四个图片(30分钟)。峰值分析完成了MATLAB findpeaks函数(MinPeakDistance = 20, MinPeakProminence = 4)。
单菌落分离
共有51个采样区域选择:29从最初的共同进化的最后一轮和22从不断进化的最后一轮(补充图。4)。从每个区域孤立单一的殖民地,冷冻样本解冻和10μl每个样本的条纹在LB-agar(30μg /毫升卡那霉素)。经过2天的孵化,两个单菌落从每个板选择和传播在2毫升深井(400 ul磅30μg /毫升卡那霉素)。股票(16%甘油)保持在−80°C。共有94个殖民地独立进行进一步分析。虽然殖民地没有多尝试删除可能延滞,以下相互传染实验(见下文)旨在敏感地发现任何可能的结转:每个omni-tray覆盖的文化单一细菌分离和发现有96滴,大约25%的下降不含有噬菌体分离设计。噬菌体移行,噬菌体斑块预计将出现在一些空下降或周边地区。然而,分析的数据显示没有迹象表明延滞的隔离。
成空斑实验单斑块(隔离)
自从T7噬菌体可以持续好几天没有复制,我们怀疑噬菌体可以包含样品,除了进化噬菌体,野生型噬菌体从更早的时间点。为了隔离一组不同的进化噬菌体,我们建立了一个荧光技术空斑实验:所有细菌隔离共同进化轮(YFP)和野生型MG1655 (mCherry)一夜之间,生长和60的混合物μl每个隔离和野生型的4毫升琼脂(0.7%)在琼脂板覆盖。盘子被分为6段和10μl点1/10系列稀释相应噬菌体的样品(噬菌体和细菌从同一采样区)和野生型T7噬菌体被发现在内部和外部的部分每段,分别为(补充图的例子。5)。孵化后,盘子YFP成像和mCherry过滤器使用宏观(大规模的荧光成像设备72年)。噬菌体只能感染野生型(mCherry)应变形成绿色斑块(YFP孤立生存),噬菌体,可以感染菌株形成黑色的斑块,和噬菌体感染发展应变(YFP),但失去传染性与野生型(mCherry)形成紫红色斑块(host-switch,补充图。5)。而斑的颜色没有直接处理在我们的分析中,这一步骤给我们,除了斑块隔离,第一次目睹表型进化和演化周期的最小数量的测试的发展阻力野生型噬菌体的细菌分离和噬菌体的进化传染性隔离细菌隔离)。2斑块从每个盘子,最好是用不同的颜色和形态,是选择和接种成指数增长的细菌培养的野生型或进化压力相同的样本,根据斑块颜色,为了避免额外的适应周期在噬菌体传播;与野生型MG1655绿色斑块被传播,黑色或红色斑块被传播的细菌分离噬菌体示例相同。2 h后孵化的慢摇,噬菌体被转移到96孔样品准备盘子锥形0.7毫升玻璃插入(分析销售和服务),补充10%氯仿和储存在4°C。总共有112个斑块隔离进行进一步分析。
交叉分析
作为初步的一步,每一个噬菌体的空斑形成单位隔离和祖先T7衡量标准PFU试验在宽容的宿主菌株(野生型或进化),和所有隔离的标准化,以一个固定的效价~ 3.3·104空斑形成单位/毫升。接下来,所有的细菌分离株和野生型MG1655几乎在一夜之间长大。之后,120年7毫升每个孤立的μl涨跌互现琼脂(0.7%),覆盖在一口井LB-Agar盘子和接种~ 4μl斑点的噬菌体分离(100 - 150空斑形成单位)使用PLATEMASTER (Gilson)。盘子被孵化6.5人力资源增长30°C和宏观的感染和成像。
斑块图像的自动处理
交叉试验与MATLAB图像处理自定义脚本。短暂,每个板块分为96个平方地区集中在噬菌体的斑点(\ (L \乘以L = 181 \ 181 {{{{{rm \ p {}}}}}} {{{{{{rm \ {x}}}}}}} ^ {2} \)在每个板块,有96重叠广场)。每平方是规范化,使所有像素在90平方等于255百分位;然后,每个噬菌体的传染性隔离对每个细菌隔离广场内相应的计算如下:
在哪里\ ({{{{{rm \ {phageFraction}}}}}} \)噬菌体裂解的相对面积在一个给定的地区,
计算了一个预定义的plate-specific阈值(异常值与区域阈值),和间隙的定义是:
在哪里V \ ({} _ {{{{{{rm \{规范}}}}}}}^ {{{{{{rm \ {isPhage}}}}}}} \枚\压裂{V(我{{{{{rm \ {sPhage}}}}}})} {{V} _ {{{\ !\马克斯•}}}}\ \)都是像素值\ \ (V)在噬菌体区域在一个给定的地区,除以最大像素值\ ({V} _ {\ !\ \马克斯}= 255)。他们的意思是计算加法V \ ({} _ {{{{{{rm \{规范}}}}}}}^ {{{{{{rm \ {isPhage}}}}}}} \)价值观和除以噬菌体裂解区域。
感染的有效性测试得分,一个更直接的浊度得分计算通过像素值的总和,\ \ (V)在给定的噬菌体现货广场,规范化的盘子,和除以平方的面积L \ \ (L \倍)和最大像素值\ ({V} _{{{\马克斯}}}\):
浊度得分值将反向传染性分数:一块高传染性的分数应该接受一个低浊度得分,反之亦然。
系统树图施工
细菌和噬菌体分离集群根据他们的抵抗和传染性表型,根据他们的传染性得分向量。具体地说,所有细菌分离株之间的欧几里得距离成对与MATLAB pdist函数计算。这些距离集群与MATLAB连杆函数凝结的分层集群树(方法=“完成”)。最后,相互传染矩阵列被命令和系统树图与MATLAB可视化系统树图的功能。隔离被划分类ColorThreshold选项基于阈值的40%最大的链接。相同的过程是重复的噬菌体分离聚类和排序矩阵行相互传染。
BiMat分析
我们应用MATLAB BiMat包48计算每个复制感染的nestedness和模块化特性矩阵包括祖先。缺失的数据值(总共15)被分配的意思5×5 class-sorted矩阵(图周围的广场。2)和值都是关键。古普塔et al。73年、模块化和nestedness值计算而忽略空行和列的默认设置。统计显著性计算机会平等或更大的模块化或nestedness值在104和等概率的随机矩阵构造零模型。
DNA测序
细菌基因组DNA提取400年离心法μl新鲜的细菌培养和resuspending溶解的颗粒缓冲(20毫米三,2毫米EDTA, 1.5% Triton, pH值8.0)补充7毫克/毫升溶菌酶和孵化为40分钟37°C水浴。DNA分离的DNA、RNA和蛋白质纯化NucleoSpin®TriPrep工具包(MACHEREY-NAGEL)。用蛋白酶k治疗进行30分钟的56°C。噬菌体基因组DNA提取400年孵化μl新鲜溶解产物的蛋白酶k(75年μg /毫升决赛)和SDS(最后0.5%)为75分钟56°C,然后分离DNA的DNA, RNA,蛋白质纯化NucleoSpin®TriPrep工具包(MACHEREY-NAGEL)。细菌和噬菌体DNA浓度测量与高灵敏度Quant-iT™dsDNA分析工具包(表达载体)和标准化1.5 ng /分别μl和1 ng /μl。测序图书馆准备根据贝母等。74年。和测序Illumina公司HiSeq X机生产150基地paired-end读取(Admera健康)。原始测序文件存放到SRA数据库,见bioProjectPRJNA884167对噬菌体和bioProject测序数据PRJNA884682细菌的测序数据。
测序分析
Illumina公司读被过滤删除读污染Nextera适配器或劣质基地(> 2基地phr得分< 20)。SNP分析使用一个内部分析管道75年。短暂,读取对齐到一个参考基因组(基因库U00096.3细菌和噬菌体NC_001604)用领结1.2.1.1最多3不匹配/阅读。基本要求是使用SAMtools和BCFtools 0.1.19,和一个基因组位置确定SNP当超过一个等位基因被发现在所有隔离FQ <−质量阈值80。同时,插入和删除与0.32.0 Breseq版本识别76年使用相同的参考基因组。识别放大和额外的缺失,每个孤立的基因组覆盖率是归一化两个步骤:首先,每个孤立的报道值除以孤立中值覆盖,然后进一步规范化值除以相应的规范化碱基对覆盖祖先基因组的价值。手动高或低覆盖的地区被确定。自适应进化意义(PdN / dS)是由随机分布评估基因内的总数snp在所有orf X10,000时期,而占突变出现在不止一个复制和保存转换、颠换比率。
套索回归分析
我们结合所有细菌和噬菌体的基因型和表型数据隔离到一个回归模型来确定突变与对传染性最强的正面或负面的影响。具体来说,每一对phage-bacteria隔离的传染性的分数(无花果。2补充数据5)作为响应变量的回归模型,预测是联合噬菌体和细菌突变相应细菌分离和噬菌体的隔离。SNP和删除被分配一个二进制值,放大被分配一个整数根据他们估计拷贝数。长放大和删除事件重叠之间的隔离分段重叠,重叠区域。例如,删除从1000个基点至3000个基点的隔离和删除从2000个基点至4000个基点隔离B将截成3独特的事件:删除1000个基点和2000个基点之间的隔离,删除2000 bp - 3000 - bp在两个隔离,和删除3000个基点和4000个基点之间隔离B .突变,总是一起出现了,除了放大(由于分配的预测的差异值依赖的拷贝数),和同义snp都被省略了,导致204突变。只采取独特的基因型和表现型平均成对的相同的基因型,我们最终与5636年观测。套索正规化与MATLAB进行了内置套索函数使用X5交叉验证和马克斯λ= 200。选择变异系数在一个标准误差的最小均方误差。
Multiply-mutated基因
为了检测基因突变超过预期的机会,我们模拟一个随机突变分布如下:整个细菌或噬菌体的突变组拍摄的所有信息除了参考基因组的突变位置。为每个模拟,随机位置是来自每个突变的参考基因组和受影响的基因记录(长放大和删除突变可能影响多个基因)。如果一个突变出现在不止一个复制(平行进化),它的位置是复制的数量出现了。我们运行了仿真500倍对细菌突变和5000噬菌体的基因突变,并计算每个基因突变的数量的分布。multi-mutated基因阈值设定,只有5%的模拟达到了实验值。
重建mlaA和opgG突变体
OpgG突变体是由法师77年(多元自动化基因工程)。与pORTMAGE311B MG1655细胞转化(Addgene号码120418),和electrocompetent细胞准备Szili et al。78年。electrocompetent细胞(40μL)混合2μL法师的寡核苷酸(500μM) 1毫米试管和electroporated 1.8 kV脉冲,然后悬浮在5毫升prewarmed很棒的肉汤(酵母提取物24 g / L,胰蛋白胨12 g / L, K2HPO49.4 g / L, KH2阿宝4在水中2 g / L)和恢复为2.5 h在37°C下摇晃。最后,100细胞μl镀在磅卡那霉素的琼脂板上。waaJ突变体是通过两个T7阻力和MASC-PCR设计和验证79年(试剂盒多重PCR设备2μM 1:1混合引物)。然后,两个waaJ突变和野生型菌株被改造opgG突变与MASC-PCR殖民地进行验证。最后,所有菌株都改变了YFP / mCherry表达质粒携带氯霉素抗性基因。补充表中列出所有寡核苷酸引物1,法师寡核苷酸的设计使用温和的设计工具80年。mlaA突变体是由转换MG1655 pGS100-MlaA(野生型细胞mlaA)或pGS100-MlaA102 (mlaA删除的41和42码56)请提供的Paola Sperandeo(米兰大学),连同YFP / mCherry表达质粒通过热休克转换。一夜之间转化细胞的培养与氯霉素稀释1/100磅补充(12.5μg /毫升),卡那霉素(15μg /毫升)和IPTG(0.1毫米)和培养4 h在镀前30°C,它是根据双色omni-tray盘子空斑实验。
报告总结
进一步研究信息设计是可用的自然投资组合报告总结与这篇文章有关。
数据可用性
基因组测序数据存入公共SRA数据库,看到加入代码PRJNA884167加入噬菌体测序数据和代码PRJNA884682细菌测序数据。所有其他数据集生成和分析在当前研究存入Zenodo库https://doi.org/10.5281/zenodo.7347986(https://zenodo.org/record/7347986 .Y4aQbXbMLIU]。源数据本文提供的。
代码的可用性
代码进行数据分析和图生成可以在GitHub (https://github.com/Technion-Kishony-lab/Phage-bacteria-spatial-interactions.git)。
引用
沼泽,p &威灵顿,e . m . h . Phage-host土壤的相互作用。《。生态。15,99 - 107 (1994)。
Morella: M。戈麦斯,a . L。王,G。,Leung, M. S. & Koskella, B. The impact of bacteriophages on phyllosphere bacterial abundance and composition.摩尔。生态。27,2025 - 2038 (2018)。
Lim e . s . et al。早期生活动力的人类婴儿肠道virome和细菌微生物。Nat,地中海。21,1228 - 1234 (2015)。
Faruque, s m . et al .季节性流感流行的霍乱反向与环境的流行霍乱噬菌体。Proc。国家的。学会科学。美国102年,1702 - 1707 (2005)。
的种子,k·d . et al .进化后果intra-patient噬菌体在微生物种群捕食。Elife3e03497 (2014)。
Laanto E。,Hoikkala, V., Ravantti, J. & Sundberg, L.-R. Long-term genomic coevolution of host-parasite interaction in the natural environment.Commun Nat。8111 (2017)。
Avrani, S。,Wurtzel, O., Sharon, I., Sorek, R. & Lindell, D. Genomic island variability facilitates Prochlorococcus-virus coexistence.自然474年,604 - 608 (2011)。
nas,即b等。分析CRISPR-Cas系统的噬菌体在流行性霍乱弧菌菌株,活跃在孟加拉国。科学。代表。714880 (2017)。
Vos, M。,Birkett, P. J., Birch, E., Griffiths, R. I. & Buckling, A. Local adaptation of bacteriophages to their bacterial hosts in soil.科学325年833 (2009)。
斯特恩,a & 4 r . phage-host军备竞赛:形成微生物的进化。Bioessays3343-51 (2011)。
巴斯,z K。,Nguyen, M. H. & Seed, K. D. A chimeric nuclease substitutes a phage CRISPR-Cas system to provide sequence-specific immunity against subviral parasites.Elife10e68339 (2021)。
曹国伟,L。,Levin, B. R. & Stewart, F. M. A complex community in a simple habitat: An experimental study with bacteria and phage.生态58,369 - 378 (1977)。
Lenski认为,r . e . &莱文,b . r .限制细菌和烈性噬菌体的共同进化:一个模型,一些实验,预测自然社区。点。Nat。125年,585 - 602 (1985)。
魏、Y。,Ocampo, P. & Levin, B. R. An experimental study of the population and evolutionary dynamics of Vibrio cholerae O1 and the bacteriophage JSF4.Proc,杂志。科学。277年,3247 - 3254 (2010)。
佩里,e . B。,Barrick, J. E. & Bohannan, B. J. M. The molecular and genetic basis of repeatable coevolution between Escherichia coli and bacteriophage T3 in a laboratory microcosm.《公共科学图书馆•综合》10e0130639 (2015)。
魏、Y。,Kirby, A. & Levin, B. R. The population and evolutionary dynamics of Vibrio cholerae and its bacteriophage: conditions for maintaining phage-limited communities.点。Nat。178年,715 - 725 (2011)。
范Houte) s . et al . diversity-generating原核适应性免疫系统的好处。自然532年,385 - 388 (2016)。
常见的,J。,Morley, D., Westra, E. R. & van Houte, S. CRISPR-Cas immunity leads to a coevolutionary arms race between Streptococcus thermophilus and lytic phage.费罗斯。反式。r . Soc。Lond。B杂志。科学。374年,20180098 (2019)。
奥尔金市中心,a . v . et al .宿主抵抗,基因组学和种群动态肠炎沙门氏菌噬菌体系统。病毒11188 (2019)。
乔杜里,w . n . et al .漏水的阻力和裂解性噬菌体的存在的条件。公共科学图书馆杂志。16e2005971 (2018)。
乔杜里,w . et al . Mucoidy一般机制保持裂解性噬菌体在细菌的数量。《。生态。96年fiaa162 (2020)。
沟口健二,k . et al .共同进化的噬菌体PP01和大肠杆菌O157: H7在连续的文化。达成。环绕。Microbiol。69年,170 - 176 (2003)。
Kashiwagi, a & Yomo t持续的表型和基因变化实验共同进化的RNA噬菌体Qβ和大肠杆菌。公共科学图书馆麝猫。7e1002188 (2011)。
马斯顿,m . f . et al .共同进化海洋聚球藻属和病毒的快速多元化。Proc。《科学。美国109年,4544 - 4549 (2012)。
贝茨。,Kaltz, O. & Hochberg, M. E. Contrasted coevolutionary dynamics between a bacterial pathogen and its bacteriophages.Proc。《科学。美国的一个。111年,11109 - 11114 (2014)。
Paez-Espino, d . et al . CRISPR免疫驱动快速链球菌噬菌体基因组进化。MBio6e00262-15 (2015)。
Debarre F。狮子,S。,van Baalen, M. & Gandon, S. Evolution of host life-history traits in a spatially structured host-parasite system.点。Nat。179年52 - 63 (2012)。
一颗心去,j . o . & Sneppen k空间结构和拉马克的适应解释极端CRISPR位点的遗传多样性。MBio3e00126-12 (2012)。
束缚,p h &体细胞杂种,j。j gene-for-gene系统异质种群的进化:宿主和病原体传播的空间尺度的影响。植物病理学研究。51,169 - 184 (2002)。
施拉格,s . j . & Mittler j . e .开始共存:空间避难所的作用在稳定bacteria-phage交互。点。Nat。148年,348 - 377 (1996)。
Rainey屈曲,a & p . b .拮抗细菌和噬菌体之间的共同进化。Proc,杂志。科学。269年,931 - 936 (2002)。
Brockhurst, m·A。,Buckling, A. & Rainey, P. B. Spatial heterogeneity and the stability of host-parasite coexistence.j .另一个星球。医学杂志。19,374 - 379 (2006)。
斯坎兰,p D。大厅,a。R。,Lopez-Pascua, L. D. C. & Buckling, A. Genetic basis of infectivity evolution in a bacteriophage.摩尔。生态。20.,981 - 989 (2011)。
斯坎兰,p . d . et al .共同进化与噬菌体驱动全基因组进化和约束收购abiotic-beneficial突变。摩尔。杂志。另一个星球。32,1425 - 1435 (2015)。
克尔,B。,Neuhauser, C., Bohannan, B. J. M. & Dean, A. M. Local migration promotes competitive restraint in a host-pathogen ‘tragedy of the commons’.自然442年,75 - 78 (2006)。
戈麦斯,p &屈曲,a Bacteria-phage对立的土壤中共同进化。科学332年,106 - 109 (2011)。
西蒙斯,e . l . et al .促进共存phage-resistant和phage-susceptible细菌生物膜的结构。mSystems5e00877-19 (2020)。
Pyenson: c & Marraffini洛杉矶联合进化在结构化的细菌群落结果在多个扩张CRISPR位点和增强免疫力。Elife9e53078 (2020)。
福德,s E。,Thompson, J. N., Holt, R. D. & Bohannan, B. J. M. Coevolution drives temporal changes in fitness and diversity across environments in a bacteria-bacteriophage interaction.进化62年,1830 - 1839 (2008)。
加西亚诉a等。DNA构象变化导致T7交付与细菌受体交互。生物。化学。290年,10038 - 10044 (2015)。
马克·d·f·&理查森,c . c .大肠杆菌硫氧还蛋白:噬菌体T7 DNA聚合酶的亚基。Proc。《科学。美国73年,780 - 784 (1976)。
Qimron U。,Marintcheva, B., Tabor, S. & Richardson, C. C. Genomewide screens for Escherichia coli genes affecting growth of T7 bacteriophage.Proc。《科学。美国103年,19039 - 19044 (2006)。
Mutalik, v . k . et al .大规模大肠杆菌的噬菌体抗性格局的映射。bioRxiv2020.02.15.951020https://doi.org/10.1101/2020.02.15.951020(2020)。
Kortright k . E。,Chan, B. K. & Turner, P. E. High-throughput discovery of phage receptors using transposon insertion sequencing of bacteria.Proc。国家的。学会科学。美国。https://doi.org/10.1073/pnas.2001888117(2020)。
萍,d . et al .搭便车,崩溃,和应急噬菌体感染细菌种群迁移。ISME J。https://doi.org/10.1038/s41396 - 020 - 0664 - 9(2020)。
李X。,Gonzalez, F., Esteves, N., Scharf, B. E. & Chen, J. Formation of phage lysis patterns and implications on co-propagation of phages and motile host bacteria.公共科学图书馆第一版。医学杂志。16e1007236 (2020)。
贝母,m . et al .时空微生物进化在抗生素的风景。科学353年,1147 - 1151 (2016)。
弗洛雷斯,c . O。Poisot, T。,Valverde, S. & Weitz, J. S. BiMat: A MATLAB package to facilitate the analysis of bipartite networks.生态方法。另一个星球。7,127 - 132 (2016)。
桑特·G。,Woods, L. C., Barr, J. J. & McDonald, M. J. Host diversity slows bacteriophage adaptation by selecting generalists over specialists.Nat,生态。另一个星球。5,350 - 359 (2021)。
瓦尔韦德,s . et al .共存宿主-病原体感染nestedness和模块化的网络。Nat,生态。另一个星球。4,568 - 577 (2020)。
命运,m . a . et al .共同进化动力学形状bacteria-phage感染网络的结构。进化73年,1001 - 1011 (2019)。
Tibshirani, r .回归通过套索收缩和选择。j . r . Stat。Soc。爵士。B Methodol统计。58,267 - 288 (1996)。
Torres-Cabassa, a s &戈特斯曼,大肠杆菌k - 12 s胶囊的合成是由蛋白质水解。j . Bacteriol。169年,981 - 989 (1987)。
de角。,Hengeveld, A. F., Martin, A. & Westphal, A. H. The pyruvate dehydrogenase multi-enzyme complex from Gram-negative bacteria.Biochim。Biophys。学报1385年,353 - 366 (1998)。
Kiro, 0.4 r . et al .基因产物增加噬菌体T7竞争力通过抑制宿主细胞分裂。Proc。《科学。美国110年,19549 - 19554 (2013)。
Falchi, f . a . et al .突变抑制器的分析必不可少的脂多糖运输蛋白质LptA揭示策略来克服严重的大肠杆菌外膜通透性缺陷。j . Bacteriol。200年,(2018)。
达菲,S。,Turner, P. E. & Burch, C. L. Pleiotropic costs of niche expansion in the RNA bacteriophage phi 6.遗传学172年,751 - 757 (2006)。
迈耶,j . r . et al .生态噬菌体λ在异域性和同域分布的物种形成。科学354年,1301 - 1304 (2016)。
施瓦茨,d . a . & Lindell d基因障碍限制之间的军备竞赛原和球藻T7-like podoviruses感染他们。ISME J。11,1836 - 1851 (2017)。
De Sordi L。,Khanna, V. & Debarbieux, L. The gut microbiota facilitates drifts in the genetic diversity and infectivity of bacterial viruses.细胞宿主细菌22,801 - 808。e3 (2017)。
Holtzman, t . et al .连续进化系统感染噬菌体T7的宿主范围。科学。代表。10307 (2020)。
Holtje, j . V。菲德勒,W。,Rotering, H., Walderich, B. & van Duin, J. Lysis induction of Escherichia coli by the cloned lysis protein of the phage MS2 depends on the presence of osmoregulatory membrane-derived oligosaccharides.生物。化学。263年,3539 - 3541 (1988)。
Sutterlin, h·a . et al .中断革兰氏阴性细胞被膜的脂质稳态激活新细胞死亡通路。Proc。《科学。美国的一个。113年E1565-E1574 (2016)。
曼宁,a . j . & Kuehn m . j .贡献的细菌外膜囊泡的细菌防御。BMC Microbiol。11258 (2011)。
Reyes-Robles, t . et al .霍乱弧菌外膜囊泡抑制噬菌体感染。j . Bacteriol。200年e00792-17 (2018)。
Brockhurst, m·A。,Koskella, B. & Zhang, Q.-G. Bacteria-Phage Antagonistic Coevolution and the Implications for Phage Therapy. in噬菌体:生物学、技术治疗(eds。哈珀,D。,Abedon, S., Burrowes, B. & McConville, M.) 1–21 (Springer International Publishing, 2017).
牛,J·J。,Levin, B. R. & Molineux, I. J. Promises and Pitfalls of In Vivo Evolution to Improve Phage Therapy.病毒111083 (2019)。
Yehl - k . et al .工程噬菌体宿主范围和抑制细菌耐药性通过噬菌体尾巴纤维诱变。细胞179年,459 - 469。e9 (2019)。
李,C。,Shi, T., Sun, Y. & Zhang, Y. A novel method to create efficient phage cocktails via use of phage-resistant bacteria.达成。环绕。Microbiol。88年e02323-21 (2022)。
Castledine, m . et al .平行进化的铜绿假单胞菌噬菌体抗性和毒性损失对噬菌体治疗体内和体外。Elife11e73679 (2022)。
吉赫尔丹,Y。,Springer, M. & Kishony, R. A competitive trade-off limits the selective advantage of increased antibiotic production.Nat。Microbiol116175 (2016)。
印度历的1月,R。,Shrestha, S., Shah, A. K., Michel, J.-B. & Kishony, R. A differential drug screen for compounds that select against antibiotic resistance.《公共科学图书馆•综合》5e15179 (2010)。
古普塔,a . et al .超越动力学在phage-bacteria共同进化了联合分析相互传染表型和全基因组测序。bioRxiv2020.10.31.337758https://doi.org/10.1101/2020.10.31.337758(2021)。
贝母,m . et al。便宜的多路复用库准备megabase-sized基因组。《公共科学图书馆•综合》10e0128036 (2015)。
叶林,i . et al .基因组和流行病学的证据从益生菌胶囊细菌传播到ICU患者的血液。Nat,地中海。25,1728 - 1732 (2019)。
Deatherage, d . e . &巴里克,j . e .突变的识别使用breseq laboratory-evolved微生物从下一代测序数据。摩尔。生物方法。1151年,165 - 188 (2014)。
王,h . h . et al .编程细胞通过多路复用基因组工程和加速演化。自然460年,894 - 898 (2009)。
Szili, p . et al .快速进化的敏感性降低平衡双定位抗生素通过突变的垫脚石。Antimicrob。代理Chemother。63年e00207-19 (2019)。
王,h·h·&教堂,通用十八章——多路复用基因工程和基因分型方法:应用合成生物学和代谢工程。在方法酶学(ed。沃伊特c)卷。498 409 - 426(学术出版社,2011年)。
邦德,m . t . et al .谦虚:一个基于web的oligonucleotide-mediated基因组工程和recombineering设计工具。核酸Res。42W408-W415 (2014)。
确认
我们感谢d . Lindell Vaknin和p . Sperandeo提供菌株。我们感谢i叶林的支持与测序和实验。我们感谢Csorgő和。Nyerges建议法师协议和a·玛套索分析的建议。我们感谢d·拉斯,欧米尔曼诉Lazar, c·韦尔灵族和n Aframian彻底阅读手稿和宝贵的意见。部分本研究支持以色列科学基金(批准号455/19),以色列科学基金——BROAD研究所联合项目(批准号2790/19)和通过一个奖Kavli微生物思想的挑战,一个项目领导的美国微生物学会和美国化学学会、美国物理学会和支持的卡夫基金会(上面)。
作者信息
作者和联系
贡献
静电的T和上面设计的研究。E.S.T.进行实验和分析。E.S.T.,和R.K. interpreted the results and wrote the manuscript.
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
同行评审
同行审查的信息
自然通讯由于Dibyendu杜塔和其他匿名审稿人(s)为他们的贡献的同行评审工作。同行审查报告是可用的。
额外的信息
出版商的注意施普林格自然保持中立在发表关于司法主权地图和所属机构。
源数据
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
上海玛,E。,Kishony, R. Multistep diversification in spatiotemporal bacterial-phage coevolution.Nat Commun137971 (2022)。https://doi.org/10.1038/s41467 - 022 - 35351 - w
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41467 - 022 - 35351 - w
本文引用的
基因组合的多样化假单胞菌噬菌体提高效率对细菌的鸡尾酒
科学报告(2023)