摘要gydF4y2Ba
APOBEC3 (A3)蛋白是宿主编码的脱氧胞苷脱氨酶,为逆转录病毒感染提供先天免疫屏障,特别是针对HIV-1。低水平的脱氨被认为有助于HIV-1的遗传进化,而这些蛋白质的强烈催化活性可以诱导前病毒DNA的灾难性超突变,导致接近完全的HIV-1限制。到目前为止,关于A3胞嘧啶脱氨酶如何影响人类染色体DNA中的HIV-1前病毒DNA整合位点,我们知之甚少。使用深度测序方法,我们分析了催化活性和非活性APOBEC3F和APOBEC3G对HIV-1整合位点选择的影响。在这里,我们发现DNA编辑是在病毒的长末端重复区域的末端被检测到的。催化活性和非催化活性A3突变体都减少了对基因编码序列的插入,并增加了对sin元件、致癌基因和转录沉默非b DNA特征的整合位点。我们的数据表明A3是影响HIV-1整合位点选择的宿主因素,并促进了似乎更潜在的表达谱。gydF4y2Ba
简介gydF4y2Ba
人类A3家族由7个成员组成,其中5个已经证明了抗HIV-1的生物相关抗病毒活性:APOBEC3D (A3D), APOBEC3F (A3F), APOBEC3G (A3G),某些单倍型的APOBEC3H (A3H),和一个多态变体的APOBE3C (A3C)。gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.当HIV-1感染一个新的CD4+单核细胞或淋巴细胞时,A3蛋白与病毒蛋白和RNA结合,导致它们被包裹在新生的排出病毒粒子中gydF4y2Ba5gydF4y2Ba.病毒粒子包装的A3在逆转录过程中主要通过脱氨胞嘧啶(C)转化为尿嘧啶(U),即单链病毒DNA (vDNA)复制中间体,在靶细胞中发挥其抗逆转录病毒活性gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba.gydF4y2Ba
在感染早期观察到非常高水平的脱氨,称为高突变,可彻底灭活病毒gydF4y2Ba6gydF4y2Ba.然而,HIV-1可以通过增加病毒感染因子(Vif)的表达来克服A3蛋白的影响,Vif结合并诱导五种抗HIV-1 A3蛋白的多泛素化,从而通过蛋白酶体降解来协调它们的渐进性消耗gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba.因此,新生排出的病毒粒子包裹数量减少的A3蛋白,直到蛋白被Vif从细胞质中清除gydF4y2Ba12gydF4y2Ba.这些病毒缺乏A3,甚至限制因子蛋白水平高度降低,可以自由感染新的细胞,帮助快速传播感染。保持低比率的A3突变或低突变,被认为是HIV-1基因进化的重要贡献者gydF4y2Ba13gydF4y2Ba.gydF4y2Ba
A3蛋白还可以通过脱氨作用以外的机制限制HIV-1的复制(例如,与病毒RNA或病毒逆转录酶(RT)结合,减少vDNA合成)。gydF4y2Ba14gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba.先前研究表明,A3G和A3F可以与病毒整合酶(IN)和RT相互作用,但这种结合在病毒整合中的作用尚不清楚gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba.更重要的是,研究表明A3F和A3G蛋白可以通过修改或改变病毒长端重复序列(LTR)末端的充分处理来降低病毒的整合效率gydF4y2Ba25gydF4y2Ba,gydF4y2Ba26gydF4y2Ba.目前尚不清楚这可能如何影响HIV-1前病毒整合位点的选择。gydF4y2Ba
在合成前病毒DNA时,由病毒和宿主蛋白质(如A3)组成的预整合复合物(PIC)易位到细胞核,为整合做准备gydF4y2Ba27gydF4y2Ba,gydF4y2Ba28gydF4y2Ba.前病毒DNA整合到开放染色质涉及宿主晶状体上皮衍生生长因子(LEDGF/p75)结合到病毒IN和多聚腺苷酸特异性因子6 (CPSF6)在LTR末端(即肠道)。gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,gydF4y2Ba32gydF4y2Ba.这种HIV-1肠道有利于与组蛋白、活性转录单位、高G/C含量区域、高基因密度、高CpG岛密度、高频率短点缀核元件(SINEs)(如Alu重复序列)、表观遗传修饰和特定核区域(如靠近核孔复合体)相关的染色质的整合gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba.此外,非b DNA结构可能会影响HIV-1整合位点的靶向gydF4y2Ba36gydF4y2Ba.至少存在10种非b DNA构象,包括a相基序、倒置重复序列、直接重复序列、十字形DNA、鸟嘌呤四重体(G4) DNA、滑动DNA、镜像重复序列、短串联重复序列、三重体重复序列和Z-DNAgydF4y2Ba37gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba.gydF4y2Ba
A3蛋白主要在逆转录过程中通过脱氨依赖和独立机制来限制HIV-1, A3F和较小程度的A3G在PIC进入细胞核时仍然与PIC相关gydF4y2Ba41gydF4y2Ba.在本研究中,我们研究了A3蛋白对HIV-1整合位点选择的影响。我们发现A3F和A3G都对整合位点的选择有重要影响,A3脱氨酶依赖和-独立活动都有助于这种影响。gydF4y2Ba
结果gydF4y2Ba
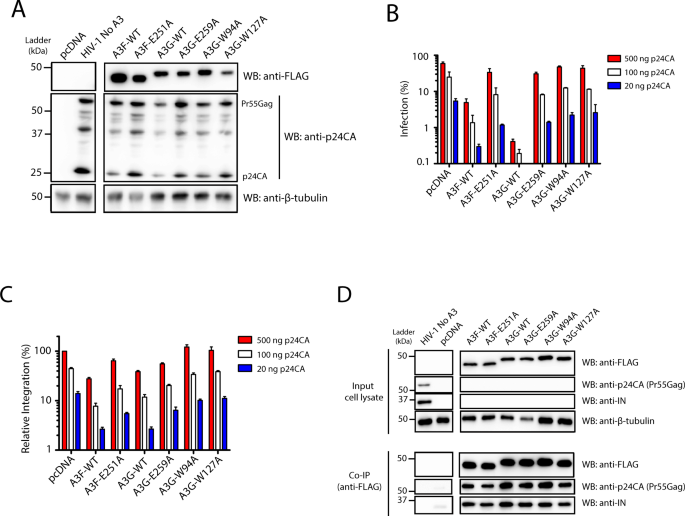
A3F和A3G以剂量依赖的方式强烈抑制HIV-1感染和整合gydF4y2Ba
通过转染293 T细胞,我们产生了HIV-1 (NL4-3)gydF4y2BaΔVif /ΔEnv-eGFPgydF4y2Ba)用水泡性口炎病毒包膜糖蛋白(VSV-G)进行假分型,同时存在野生型(wt)或脱氨缺陷突变型A3F [E251A]和A3G [E259A]。我们还包括A3G核酸结合缺陷突变体A3G [W94A]和A3G [W127A]。gydF4y2Ba14gydF4y2Ba.在非催化的A3G n端结构域内,W94是SgydF4y2BaWgydF4y2BaSPCxxC锌配合基序,而W127在ARLYYF内gydF4y2BaWgydF4y2Ba.这些色氨酸残基对于一般核酸结合能力、底物序列识别和蛋白质低聚化是重要的gydF4y2Ba14gydF4y2Ba,gydF4y2Ba21gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba.虽然W94A和W127A突变都减少了RNA结合,但W127A的取代是独特的,因为它阻止了A3G的同型二聚,从而降低了加工能力gydF4y2Ba14gydF4y2Ba.这突出了二聚化对ssDNA存在下A3G催化活性功能的重要性gydF4y2Ba45gydF4y2Ba.每个A3产生的等量病毒被用于感染许可人t4淋巴母细胞样细胞系CEM-SS。感染后48小时,通过流式细胞术检测CEM-SS细胞的生产性感染,采用病毒编码的eGFP报告基因表达(图2)。gydF4y2Ba1gydF4y2Ba,gydF4y2BaS1gydF4y2Ba).或者,收集感染细胞进行基因组DNA (gDNA)提取,以量化前病毒整合水平和整合位点的下游分析。除了在增加A3蛋白的情况下产生病毒外,还使用了三种不同数量的输入病毒进行感染(图2)。gydF4y2Ba1 bgydF4y2Ba,gydF4y2BaS1gydF4y2Ba).增加用于病毒生产的A3质粒的数量对HIV-1颗粒释放有显著影响(图2)。gydF4y2BaS2gydF4y2Ba).gydF4y2Ba
一个gydF4y2BaWestern blot分析病毒产生细胞。通过共转染NL4-3编码质粒-ΔEnv/ΔVif/eGFP、VSV-G和空pcDNA 3质粒(HIV-1 no A3)或每个A3表达质粒,在293 T细胞中产生HIV-1伪型病毒。控制通道是在没有病毒的情况下对pcDNA的转染。细胞裂解物SDS-PAGE和Western blot分析。gydF4y2BaBgydF4y2Ba用酶联免疫吸附法测定CEM-SS细胞归一化至衣壳(p24CA)蛋白后的病毒量。流式细胞术检测感染情况,以eGFP+细胞的百分比为指标。数据以平均值±标准差表示。gydF4y2BaCgydF4y2BaCEM-SS细胞中的整合前病毒gydF4y2BaBgydF4y2Ba采用铝基PCR结合巢式qPCR进行定量。数据以平均值±标准差表示。gydF4y2BaDgydF4y2Ba转染293个T细胞,分别表达A3蛋白、HIV-1(无A3)或pcDNA(转染pcDNA质粒的细胞)。病毒和细胞裂解物混合在一起,使用抗flag抗体进行免疫共沉淀,并通过western blotting进行分析。所示数据是三个独立实验的代表性数据。源数据作为源数据文件提供。gydF4y2Ba
在野生型(wt) A3F中观察到HIV-1的有效限制,在野生型A3G中更明显(图2)。gydF4y2Ba1 bgydF4y2Ba,gydF4y2BaS1gydF4y2Ba).催化无活性的A3F [E251A]和A3G [E259A]均表现出明显较小的限制。我们的小组之前已经确定A3G [W94A]和A3G [W127A]各自的限制能力减弱,但仍然能够进行病毒DNA编辑gydF4y2Ba14gydF4y2Ba.这些突变体对整体感染和整合效率的影响很小(图2)。gydF4y2Ba1 b, CgydF4y2Ba).此外,正如预期的那样,A3F和A3G的抑制水平取决于病毒生产过程中A3蛋白的表达量(图2)。gydF4y2BaS1gydF4y2Ba).gydF4y2Ba
鉴于A3具有多种编辑依赖和独立的限制机制,生产性感染与前病毒整合的总体水平相关。我们用Alu-PCR和ddPCR检测HIV-1前病毒DNA的相对整合拷贝数,在一个实验中以HIV-1 LTR为目标,在另一个实验中以报告基因eGFP作为对照,输入细胞DNA归一化为扩增的肌动蛋白DNA(图2)。gydF4y2Ba1 cgydF4y2Ba,gydF4y2BaS3gydF4y2Ba)gydF4y2Ba46gydF4y2Ba.整合水平与感染水平密切相关,除了wt A3G和A3F蛋白表现出比整合更明显的感染限制。这并不奇怪,因为eGFP报告基因的表达和荧光依赖于其编码序列的遗传完整性,而该编码序列经常因A3F和A3G超突变而失活gydF4y2Ba47gydF4y2Ba.gydF4y2Ba
A3F和A3G与病毒Gag和IN相互作用gydF4y2Ba
一些报道表明,A3F和A3G以依赖rna的方式与HIV-1 Gag和IN相互作用,它们都是PIC的组成部分gydF4y2Ba21gydF4y2Ba,gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba.然而,与失活脱氨酶和缺陷核酸结合特性的A3变异体的结合此前尚未平行评估。在本研究中,确定各种A3蛋白是否能直接或间接与IN相互作用至关重要,因为这种相互作用可能对PIC的形成和整合位点的选择至关重要。为了描述HIV-1 IN和Gag与各种A3蛋白的相互作用,用flag标记的A3变异体或HIV-1转染的细胞裂解液共孵育,然后使用anti-FLAG共免疫沉淀,并使用anti-IN和anti-p24CA进行Western blotting分析。如图所示。gydF4y2Ba1 dgydF4y2Ba,所有A3蛋白,包括脱氨酶非活性和核酸结合突变体与IN和Gag共免疫沉淀的效率相似,表明与A3直接或间接(即通过蛋白质复合物)相互作用。gydF4y2Ba
A3F和A3G改变HIV-1整合位点的基因组特征gydF4y2Ba
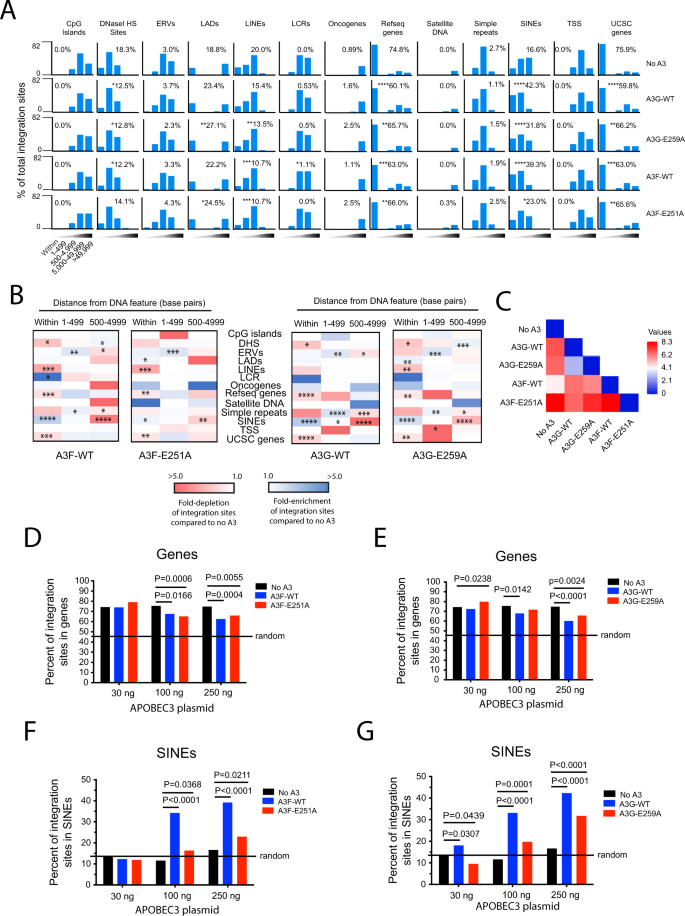
为了鉴定HIV-1整合位点,我们扩增了从感染HIV-1的细胞中分离出来的基因组DNA (gDNA)中的整合位点,这些细胞存在各种A3蛋白。如前所述,使用Barr实验室集成站点管道(BLISIP)生成集成站点概要gydF4y2Ba36gydF4y2Ba,gydF4y2Ba50gydF4y2Ba.BLISIP测量整合位点富集在和附近的基因组特征,如CpG岛,DNAseI超敏位点(DHS),内源性逆转录病毒,异色DNA区域(例如,椎板相关结构域(LADs)和卫星DNA), sin,长点缀核元件(LINEs),低复杂性重复序列(LCRs),癌基因,基因,简单重复序列和转录起始位点(TSS)。此外,BLISIP测量非b DNA中及其附近的富集特征,包括a相基序、十字基序、直接重复序列、G4重复序列、倒置重复序列、镜像重复序列、短串联重复序列、滑动基序、三联体基序和Z-DNA基序。gydF4y2Ba
与不含A3F或A3G的病毒相比,感染含有A3F或A3G的病毒的靶细胞在SINEs内和附近的整合显著增加(分别为39%和42%),而不含A3F或A3G的病毒为17%;gydF4y2BapgydF4y2Ba< 0.0001)(图gydF4y2Ba2 a, BgydF4y2Ba、补充数据文件gydF4y2Ba1gydF4y2Ba及补充数据文件gydF4y2Ba2gydF4y2Ba).与没有A3的对照相比,A3F和A3G的整合也显著富集了邻近的简单重复序列(1-500个核苷酸)(分别为26%和35%);gydF4y2BapgydF4y2Ba< 0.05)。此外,与无A3对照组相比,与A3F和A3G的整合在ERVs、LADs、癌基因和LCRs中及附近略有增加。值得注意的是,与没有A3对照相比,存在A3F和A3G的基因整合显著降低(分别为63%和60%),而不是75%;gydF4y2BapgydF4y2Ba< 0.001)。相比之下,46%的整合位点随机出现在基因中(补充数据文件gydF4y2Ba3.gydF4y2Ba).DHS和LINEs的整合程度也有所下降。gydF4y2Ba
一个gydF4y2Ba在感染HIV-1的CEM-SS T细胞中,存在A3F- wt、A3F [E251A]、A3G- wt或A3G [E259A]或不存在A3F或A3G(“没有A3”对照)时,在不同距离间隔内(1-499、500-4999、5000-49,999或>49,999 bp)距离各种常见基因组特征的整合位点的频率。插入数指的是直接落在特性内的全部集成站点的百分比。统计比较以No A3对照。gydF4y2BaBgydF4y2Ba与“no A3”对照病毒相比,热图描绘了在不同距离间隔内整合位点的折叠富集(蓝色阴影)和耗尽(红色阴影)。gydF4y2BaCgydF4y2Ba使用成对距离矩阵来确定感染A3对照病毒或A3F- wt、A3F [E251A]、A3G- wt或A3G [E259A]病毒的CEM-SS细胞的整合位点轮廓之间的总体相似性。比较中使用了每个共同DNA特征在每个距离仓中的折叠富集和耗尽值。热图显示了用欧几里得距离作为测量方法计算出的距离矩阵。较强的关系用深蓝色表示,较弱的关系用深红色表示。gydF4y2BaDgydF4y2Ba,gydF4y2BaEgydF4y2BaA3F- wt、A3F [E251A]、A3G- wt或A3G [E259A]病毒感染的CEM-SS细胞,其基因中总整合位点的百分比来自表达浓度不断增加的A3蛋白的细胞。gydF4y2BaFgydF4y2Ba,gydF4y2BaGgydF4y2Ba在被A3F- wt、A3F [E251A]、A3G- wt或A3G [E259A]病毒感染的CEM-SS细胞中,位于SINEs的总整合位点百分比来自表达A3蛋白浓度增加的细胞。阴影三角形表示不同距离的箱子,最暗的阴影表示离特征更远的距离。*gydF4y2BaPgydF4y2Ba< 0.05, **gydF4y2BaPgydF4y2Ba< 0.01, ***gydF4y2BaPgydF4y2Ba< 0.001, ****gydF4y2BaPgydF4y2Ba< 0.0001;费雪精确检验,双面检验。源数据作为源数据文件提供。gydF4y2Ba
使用缺乏脱氨酶活性的A3F和A3G突变体,SINEs的整合显著增加,而基因的整合显著减少,但与野生型突变体观察到的程度不同。与wt类似,与A3F [E251A]和A3G [E259A]的整合在ERVs、LADs和癌基因中或附近适度增加(图2)。gydF4y2Ba2 a, BgydF4y2Ba、补充数据文件gydF4y2Ba1gydF4y2Ba及补充数据文件gydF4y2Ba2gydF4y2Ba).为了比较不同A3结构之间整合位点概况的总体相似性,我们对整合位点概况进行了两两分析,基于“在”、“1-499 bp”和“500-4999 bp”每个bin内的整合位点富集或缺失,捕获每个基因组特征在5000 bp内的所有位点。如图所示。gydF4y2Ba2摄氏度gydF4y2Ba,不同含a3病毒的整合位点档案各不相同(gydF4y2BapgydF4y2Ba= 0.0398;双向方差分析,DF = 37),其中A3G和A3G [E259A]的相似度最高。gydF4y2Ba
鉴于整合位点选择的最大差异是在基因和SINEs中观察到的,我们询问这些偏好是否依赖于A3剂量。事实上,A3浓度的增加导致基因中整合位点百分比的降低(图。gydF4y2Ba2 d, EgydF4y2Ba及补充数据文件gydF4y2Ba3.gydF4y2Ba).相反,A3结构物浓度的增加导致SINEs中整合位点百分比的剂量依赖性增加(图。gydF4y2Ba2 f, GgydF4y2Ba及补充数据文件gydF4y2Ba3.gydF4y2Ba).总之,这些数据表明A3F和A3G影响HIV-1整合位点的靶向,A3F和A3G的脱氨活性在较小程度上影响这种靶向的大小。gydF4y2Ba
A3F和A3G的表达改变了非b DNA基序的整合位点靶向gydF4y2Ba
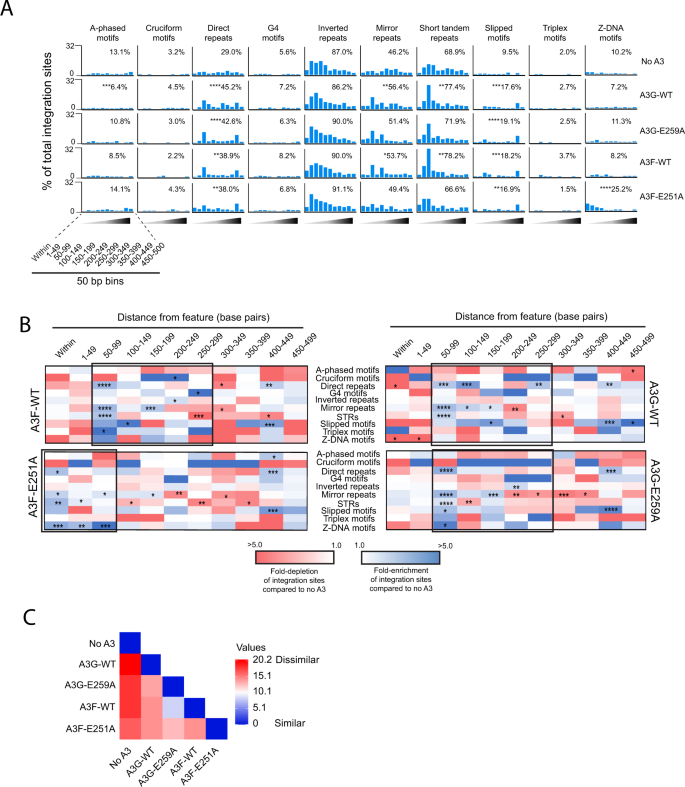
然后,我们确定了A3对靶向非b DNA基序进行整合的影响。在A3F或A3G存在下感染的细胞在大多数非b DNA特征的500 bp内表现出丰富的整合(图。gydF4y2Ba3gydF4y2Ba及补充数据文件gydF4y2Ba4gydF4y2Ba).与对照组相比,在A3F [E251A]或A3G [E259A]感染的细胞在大多数非b DNA附近表现出相似的整合水平,除了直接重复序列和滑动基序,在这些位点上观察到整合的显著增加。值得注意的是,与其他A3结构和对照组相比,A3F [E251A]在Z-DNA附近的整合显著增加(25%,对照组为10%;gydF4y2BapgydF4y2Ba< 0.0001)。gydF4y2Ba
为了确定靠近非b DNA特征的整合位点的分布是否存在差异,我们比较了距离每个非b DNA基序50 bp到500 bp的容器中整合位点的数量(图2)。gydF4y2Ba3 bgydF4y2Ba及补充数据文件gydF4y2Ba5gydF4y2Ba).由A3F、A3G和A3G [E259A]产生的病毒的整合偏好聚集在距离非b DNA 50 - 300bp的区域。A3F [E251A]的不同之处在于其整合集中在距离特征100 bp以内的区域。对整合位点剖面的两两分析(距离特征500 bp内)表明,虽然A3F和A3G [E259A]具有惊人的相似性,但所有其他整合剖面都是不同的(gydF4y2BapgydF4y2Ba< 0.0001;双向方差分析,DF = 109)(图gydF4y2Ba3 cgydF4y2Ba).总之,这些数据表明,A3F和A3G影响HIV-1非b DNA特征的整合位点靶向,它们在a相、镜像重复、str和Z-DNA特征的靶向上的脱氨基活性有很大贡献。gydF4y2Ba
一个gydF4y2Ba在感染HIV-1的CEM-SS T细胞中,存在A3F- wt、A3F [E251A]、A3G- wt或A3G [E259A],或不存在A3G或A3F(“没有A3”对照)时,在距离各种非b DNA特征不同的50 bp距离间隔内(1-500 bp)的整合位点频率产生。插入的数字是指位于特征500bp内的全部集成站点的百分比。统计比较是关于“无A3”的。gydF4y2BaBgydF4y2Ba与“no A3”对照病毒相比,热图描绘了在不同距离间隔内整合位点的折叠富集(蓝色阴影)和耗尽(红色阴影)。黑盒子突出了显著富集的区域。gydF4y2BaCgydF4y2Ba使用成对距离矩阵来确定不同集成站点轮廓之间的总体相似性。比较中使用了每个距离库中每个非b DNA特征的折叠富集和耗尽值。热图显示了用欧几里得距离作为测量方法计算出的距离矩阵。较强的关系用深蓝色表示,较弱的关系用深红色表示。阴影三角形表示不同距离的箱子,最暗的阴影表示离特征更远的距离。*gydF4y2BaPgydF4y2Ba< 0.05, **gydF4y2BaPgydF4y2Ba< 0.01, ***gydF4y2BaPgydF4y2Ba< 0.001, ****gydF4y2BaPgydF4y2Ba< 0.0001;费雪精确检验,双面检验。gydF4y2Ba
.gydF4y2Ba
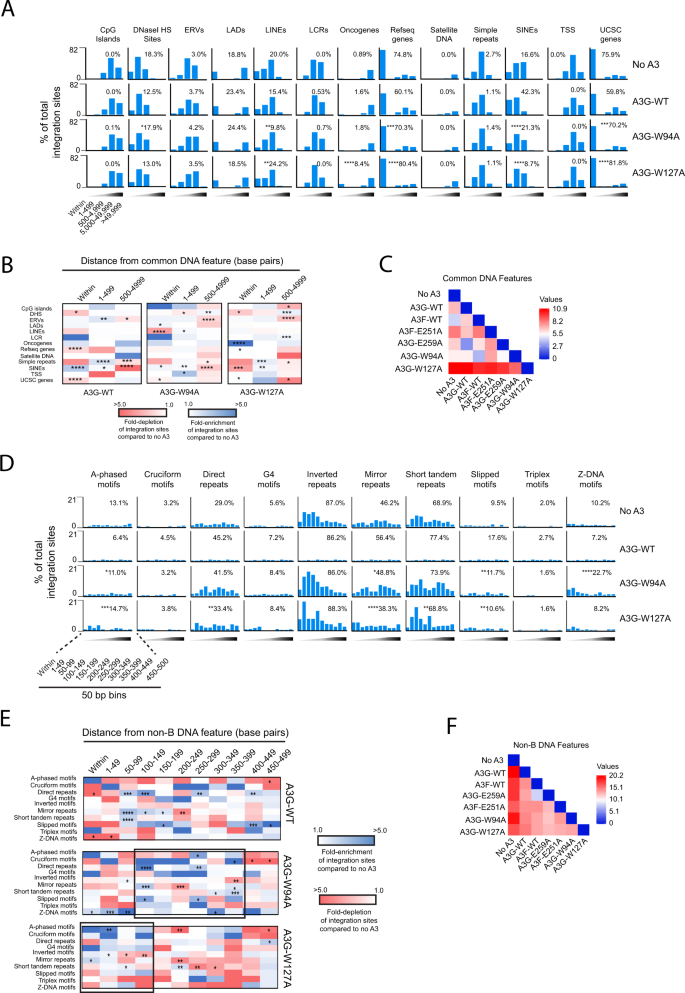
A3G残基W94和W127影响HIV-1整合位点的靶向gydF4y2Ba
我们分析了感染A3G [W94A]或A3G [W127A]突变体产生的病毒的细胞的整合位点谱,以确定这些残基是否影响wt A3G影响整合位点靶向的能力。与wt相比,A3G [W94A]和A3G [W127A]在基因整合方面显著增加,而在SINEs方面的整合则下降(图2)。gydF4y2Ba4 a、BgydF4y2Ba、补充数据文件gydF4y2Ba1gydF4y2Ba及补充数据文件gydF4y2Ba2gydF4y2Ba).此外,A3G [W127A]与wt A3G相比,癌基因整合事件显著增加。有趣的是,虽然A3G [W94A]表现出介于对照组和wt A3G之间的中间表型,但A3G [W127A]似乎加剧了HIV-1的整合位点偏好。对所有A3基因整合位点的配对分析(在各种特征的5000 bp范围内)表明,A3G [W94A]与wt A3F最相似,而A3G [W127]与所有测试的A3基因变体不同(gydF4y2BaPgydF4y2Ba= 0.014;双向方差分析,DF = 37)(图gydF4y2Ba4摄氏度gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba在感染HIV-1的CEM-SS T细胞中,在存在A3G- wt、A3G [W94A]、A3G [W127A]或不存在A3F或A3G(“没有A3”对照)的情况下,在各种常见基因组特征内或在不同距离间隔内的整合位点频率产生。插入的数字指的是直接位于该特性内的全部集成站点的百分比。统计比较是关于A3G-WT的。gydF4y2BaBgydF4y2Ba与“no A3”对照病毒相比,热图描绘了在不同距离间隔内整合位点的折叠富集(蓝色阴影)和耗尽(红色阴影)。gydF4y2BaCgydF4y2Ba使用成对距离矩阵来确定感染A3对照病毒或A3F- wt、A3F [E251A]、A3G- wt、A3G [E259A]、A3G [W94A]或A3G [W127A]病毒的CEM-SS细胞的整合位点轮廓之间的总体相似性。比较中使用了每个共同DNA特征在每个距离仓中的折叠富集和耗尽值。热图显示了用欧几里得距离作为测量方法计算出的距离矩阵。较强的关系用深蓝色表示,较弱的关系用深红色表示。gydF4y2BaDgydF4y2Ba在各种非b DNA特征内或不同距离间隔内的整合位点的频率。插入的数字是指位于特征500bp内的全部集成站点的百分比。统计比较是关于A3G-WT的。gydF4y2BaEgydF4y2Ba与“no A3”对照病毒相比,热图描绘了在不同距离间隔内整合位点的折叠富集(蓝色阴影)和耗尽(红色阴影)。黑盒子突出了显著富集的区域。gydF4y2BaFgydF4y2Ba利用两两距离矩阵来确定综合站点剖面之间的总体相似性。阴影三角形表示不同距离的箱子,最暗的阴影表示离特征更远的距离。*gydF4y2BaPgydF4y2Ba< 0.05, **gydF4y2BaPgydF4y2Ba< 0.01, ***gydF4y2BaPgydF4y2Ba< 0.001, ****gydF4y2BaPgydF4y2Ba< 0.0001;费雪精确检验,双面检验。源数据作为源数据文件提供。gydF4y2Ba
对于非b DNA特征,与小剂量A3G相比,感染A3G [W94A]-或A3G [W127A]-病毒的细胞在a相基序附近的整合显著增加,而在镜像重复和滑动基序附近的整合显著减少(图)。gydF4y2Ba4 dgydF4y2Ba及补充数据文件gydF4y2Ba4gydF4y2Ba).此外,A3G [W127A]在Z-DNA附近的整合显著增加。在距离非b DNA基序50 - 500 bp的区域内,A3G和A3G的整合位点分布相似[W94A],主要集中在距离特征100-400 bp的区域(图4)。gydF4y2Ba4 egydF4y2Ba及补充数据文件gydF4y2Ba5gydF4y2Ba).一个例外是Z-DNA,其位点高度富集在Z-DNA基序的100 bp内。相比之下,在A3G [W127A]存在下产生的细胞的整合位点倾向于聚集在非b DNA的150 bp内。对整合位点剖面(距离特征500 bp内)的两两分析表明,虽然A3F和A3G [E259A]具有相似性,但所有其他整合剖面都不同(gydF4y2BapgydF4y2Ba< 0.0001;双向方差分析,DF = 109)(图gydF4y2Ba4 fgydF4y2Ba).总之,这些数据表明,A3G残基W94和更大程度上的W127对A3G影响整合位点靶向的能力有不同的影响。gydF4y2Ba
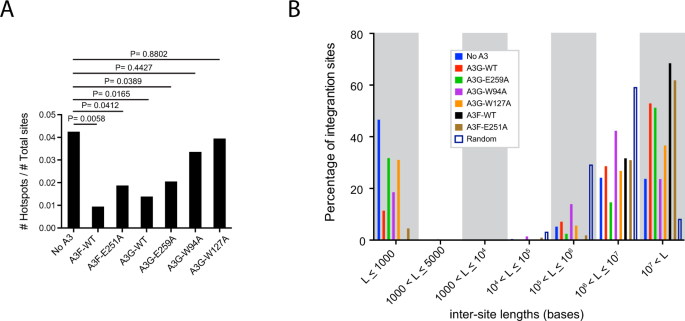
A3F和A3G减少了集成站点的热点和集群数量gydF4y2Ba
HIV-1整合“热点”的概念被引入,用于描述基因组中在没有任何选择过程的情况下,整合积累的数量偶然超过预期的区域gydF4y2Ba51gydF4y2Ba.鉴于我们发现A3F和A3G会影响整合站点的定位,我们询问它们是否也会影响整合热点的数量和站点的集群。我们将整合热点定义为包含四个或更多独特整合位点的1千碱基gDNA片段。与不表达A3的细胞相比,在A3F或A3G存在下产生的感染HIV-1的CEM-SS T细胞表现出大量减少的热点数量(gydF4y2BapgydF4y2Ba< 0.05, Fisher精确检验)(图gydF4y2Ba5gydF4y2Ba及补充数据文件gydF4y2Ba6gydF4y2Ba).A3F [E251A]和A3G [E259A]也表现出较少的热点数量,这表明A3脱氨活性对这种效应并不重要。相比之下,A3G [W94A]或A3G [W127A]的存在并没有显著减少集成站点热点的数量。gydF4y2Ba
一个gydF4y2Ba集成站点热点分析。热点被定义为基因组中包含4个或更多独特整合位点的1 kb窗口。整合热点显示为来自感染HIV-1的CEM-SS细胞在不存在A3F或A3G(“no A3”)对照(蓝色条)的情况下产生的总整合位点的比例,或来自表达A3F- wt(黑色条)、A3F [E251A](棕色条)、A3G- wt(红色条)、A3G [E259A](绿色条)、A3G [W94A](紫色条)或A3G [W127A](黄色条)的细胞。gydF4y2BaBgydF4y2Ba通过比较全基因组整合位点与相同数量的均匀分布(随机)位点之间的间隔来评估整合位点的聚类。站点之间的距离被收集在7个长度(L)“箱”中,站点之间的长度最短的在左边,最长的在右边。在计算机上生成一个匹配的随机控制数据集(详见方法)。费雪精确检验,双面检验。gydF4y2Ba
为了记录基因组区域内整合位点的聚类,我们比较了前病毒整合位点之间的距离(图2)。gydF4y2Ba5 bgydF4y2Ba).整合位点的对照群体包含比预期的偶然(即随机分布)更短的段间距离,表明了聚类。与对照细胞相比,用A3F或A3G产生的病毒表现出显著的聚类减少(gydF4y2BapgydF4y2Ba< 0.0001,费雪精确检验)。这种减少在含有A3G [E259A]和A3G [W127A]的病毒中丢失,在较小程度上A3G [W94A]和A3F [E251A]中丢失。这些数据表明,A3F和A3G的表达减少了整合热点的数量和整合位点的聚类。gydF4y2Ba
LTR中的G-to-A突变改变了体内和体外的整合位点靶向gydF4y2Ba
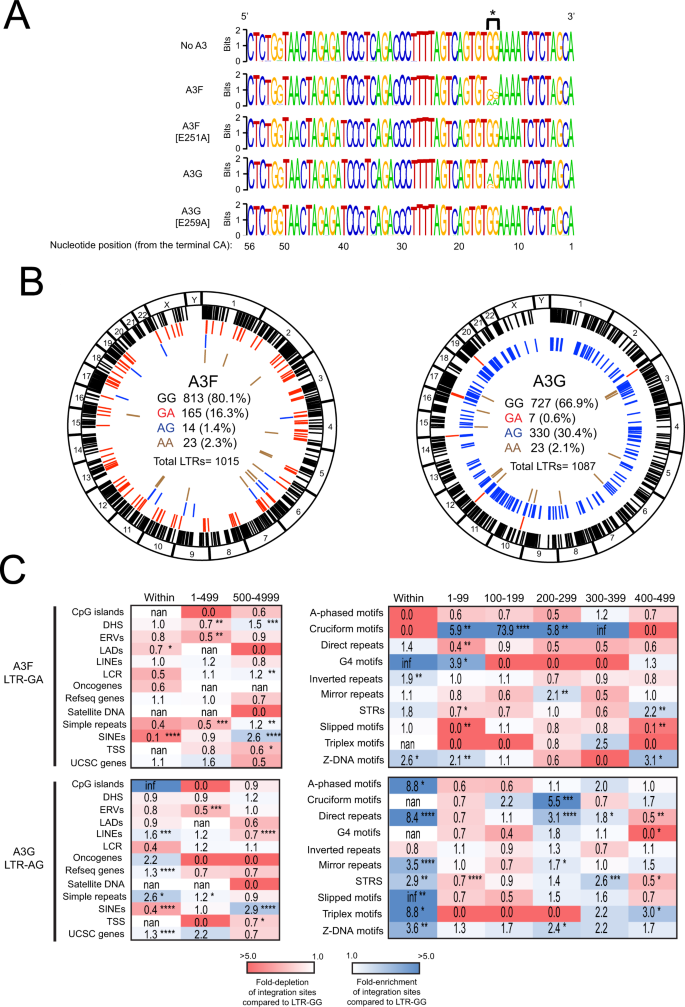
为了确定LTR末端脱氨是否影响整合位点靶向,我们对唯一的整合3’LTR核苷酸序列进行了对齐,并使用WebLogo将其图形化表示为序列标识gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba(无花果。gydF4y2Ba6gydF4y2Ba).正如预期的那样,对照的3 ' LTR端与脱氨缺陷A3突变体A3F [E251A]和A3G [E259A]高度相似。A3F和A3G的3 ' LTR末端除了位于LTR末端14和15位置的2个核苷酸外也高度相似。在A3F存在的情况下,80.0%的LTR在这些位置含有GG, 16.3%含有GA, 1.4%含有AG, 2.3%含有AA(图3)。gydF4y2Ba6 bgydF4y2Ba).在A3G存在的情况下,66.9%的lts含有GG, 0.6%的lts含有GA, 30.4%的lts含有AG, 2.1%的lts含有AA。gydF4y2Ba
一个gydF4y2Ba末端56个核苷酸的LOGO表示(A,绿色;C,蓝色;G,橙色;T,红色)在A3F、A3F [E251A]、A3G或A3G [E259A]存在的情况下产生的所有集成HIV-1前病毒的3 ' lts。gydF4y2BaBgydF4y2Ba显示感染CEM-SS细胞基因组中含有病毒的A3F-(左)或A3G-(右)整合位点的环状图。外圈代表不同的染色体。从LTR端开始的第14和15个核苷酸上,含有GG(黑色)、GA(红色)、AG(蓝色)和AA(棕色)的前病毒整合位点的染色体位置用彩色蜱表示。总场址的数量和百分比显示在环绕图内。gydF4y2BaCgydF4y2Ba热图描绘了与共同基因组特征(左)和非b DNA特征(右)在不同距离间隔的整合位点的折叠富集(蓝色阴影)和耗尽(红色阴影)。显示了来自A3F-LTR-GA(上)和A3G-LTR-AG(下)前病毒的整合位点。折叠变化分别为A3F-LTR-GG和A3G-LTR-GG。*gydF4y2BaPgydF4y2Ba< 0.05, **gydF4y2BaPgydF4y2Ba< 0.01, ***gydF4y2BaPgydF4y2Ba< 0.001, ****gydF4y2BaPgydF4y2Ba< 0.0001;费雪精确检验,双面检验。无限大(inf)表示当偶然期望0个积分时,观察到1个或多个积分。没有一个数字(nan)表示0个被观察到的积分和0个被期望的随机积分。gydF4y2Ba
为了确定3 ' LTR端第14和/或15位的G-to-A突变是否与整合位点谱的改变相关,我们将分别含有GA或AG的前病毒的A3F和A3G整合位点谱与含有3 ' LTR端第14和15位GG的前病毒的A3F和A3G整合位点谱进行了比较。含有GA二核苷酸的A3F lts (A3F- ltr -GA)在SINEs内的整合位点显著减少,而在SINEs远端(500-5000 bp)的整合位点增加(图。gydF4y2Ba6摄氏度gydF4y2Ba、补充数据文件gydF4y2Ba7gydF4y2Ba及补充数据文件gydF4y2Ba8gydF4y2Ba).在DHS、LCR和简单重复序列远端也观察到整合增加。引人注目的是,相对于非b DNA特征,整合位点的轮廓发生了巨大的变化。A3F-LTR-GA位点高度富集(高达74倍)1-400 bp的十字形基序(图。gydF4y2Ba6摄氏度gydF4y2Ba及补充数据文件gydF4y2Ba7gydF4y2Ba).G4 DNA、反向重复序列和Z-DNA位点富集。含有AG二核苷酸的A3G lts (A3G- ltr -AG)也表现出在SINEs中的整合减少,在SINEs更远端的整合增加;然而,与A3F-LTR-GA不同的是,基因和简单重复序列的整合也增加了(图3)。gydF4y2Ba6摄氏度gydF4y2Ba及补充数据文件gydF4y2Ba8gydF4y2Ba).与A3F-LTR-GA相似,A3G-LTR-AG相对于非b DNA在整合位点谱上有显著变化。整合富集在和/或接近大多数非b DNA特征。总之,这些数据表明,a3诱导的G-to-A突变位于3’LTR末端的14或15位,与一个显著改变的整合位点特征相关,并在转录沉默附近富集非b DNA特征。gydF4y2Ba
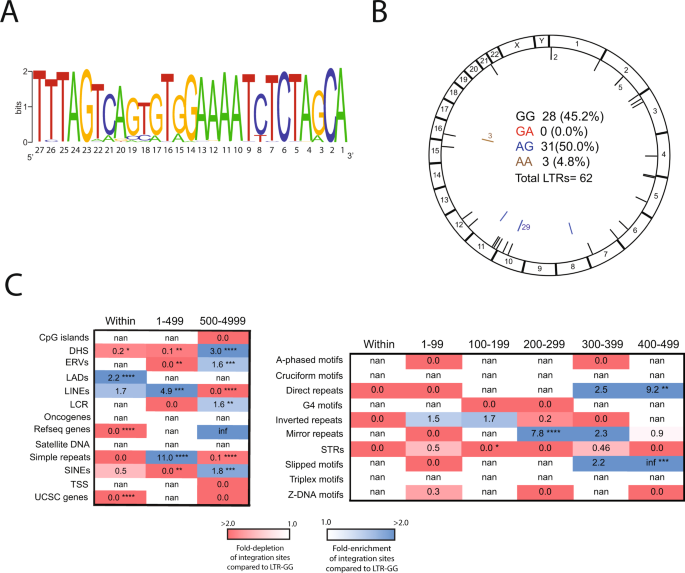
为了确定从HIV-1感染者的前病毒LTR序列末端的14和15位置是否发生类似的G-to-A突变,从93例患者队列的外周血单个核细胞(pmcs)中分离出基因组DNA,并用于对前病毒LTR进行测序并生成整合位点文库。我们将上述体外分析中所识别和过滤的独特集成3’LTR核苷酸序列进行比对,并使用WebLogo将其图形化表示为序列标识gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba(无花果。gydF4y2Ba7一个gydF4y2Ba).序列分析显示,从3 ' LTR端开始的14和15位G-to-A突变与A3G观察到的突变比例相似(图2)。gydF4y2Ba7一个gydF4y2Ba).45%的前病毒lts含有GG二核苷酸(LTR-GG), 50%含有AG (LTR-AG), 0%含有GA (LTR-GA), 5%含有AA (LTR-AA)(图)。gydF4y2Ba7 bgydF4y2Ba).尽管LTR-GG和LTR-AG前病毒的整合位点总数较低(共48个位点),可能是由于患者正在接受抗逆转录病毒治疗,但将包含LTR-AG的前病毒(25个位点)与LTR-GG(23个位点)的整合位点进行比较,结果显示基因内整合位点显著减少,而在板层相关结构域(LADs)的整合位点显著增加。与A3G-LTR-AG相似,来自含有LTR-AG的患者ltr的整合位点在一些非b DNA特征附近富集,特别是重复和滑动基序,这些基序已知会对基因表达产生负面影响(图3)。gydF4y2Ba7 cgydF4y2Ba及补充数据文件gydF4y2Ba9gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba末端27个核苷酸的LOGO表示(A,绿色;C,蓝色;G,橙色;T,红色)在HIV-1感染者中整合HIV-1前病毒的3 ' lts。gydF4y2BaBgydF4y2Ba显示感染个体基因组中前病毒整合位点的环状图。外圈代表不同的染色体。从LTR端开始的第14和15个核苷酸上,含有GG(黑色)、GA(红色)、AG(蓝色)和AA(棕色)的前病毒整合位点的染色体位置用彩色蜱表示。与蜱虫相邻的数字表示该地区无法被多个蜱虫区分的位点数量。总场址的数量和百分比显示在环绕图内。gydF4y2BaCgydF4y2Ba热图描绘了与共同基因组特征(左)和非b DNA特征(右)在不同距离间隔的整合位点的折叠富集(蓝色阴影)和耗尽(红色阴影)。显示了来自前病毒LTR-AG的整合位点,其折叠变化与前病毒LTR-GG整合位点有关。*gydF4y2BaPgydF4y2Ba< 0.05, **gydF4y2BaPgydF4y2Ba< 0.01, ***gydF4y2BaPgydF4y2Ba< 0.001, ****gydF4y2BaPgydF4y2Ba< 0.0001;费雪精确检验,双面检验。无限大(inf)表示当偶然期望0个积分时,观察到1个或多个积分。没有一个数字(nan)表示观察到0个积分,0个是偶然期望的。gydF4y2Ba
A3F和A3G促进体外潜伏感染gydF4y2Ba
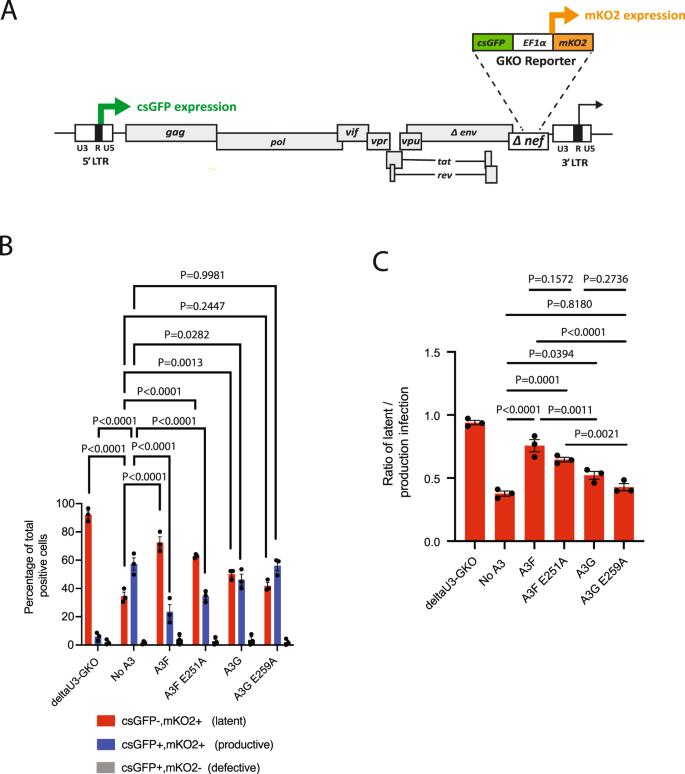
鉴于我们发现A3促进转录沉默基因组特征的整合,我们接下来确定A3的表达是否改变了潜伏性和生产性感染细胞的比例。在这些实验中,我们使用了一种双荧光HIV-1报告病毒(HIVgydF4y2Ba巨科gydF4y2Ba)设计用于流式细胞术定量潜伏感染细胞(图。gydF4y2Ba8gydF4y2Ba)gydF4y2Ba54gydF4y2Ba.艾滋病毒gydF4y2Ba巨科gydF4y2Ba该结构包含5 ' LTR启动子转录控制下的密码子开关增强绿色荧光蛋白(csGFP)和内部EF1α启动子控制下的不同的不相关荧光蛋白mKO2。HIV细胞的生产性感染gydF4y2Ba巨科gydF4y2Ba导致细胞同时表达csGFP和mKO2 (csGFP+, mKO2+),而细胞的潜伏感染只导致mKO2表达(csGFP−,mKO2+)。仅表现出csGFP表达的细胞(GFP+, mKO2−)被认为整合了缺陷原病毒。不表达任何一种标记(csGFP−,mKO2−)的细胞被认为是未感染的,和/或含有潜在的csGFP和mKO2表达的前病毒。这些(csGFP−,mKO2−)细胞被排除在分析之外。典型的流式细胞仪实验如图所示。gydF4y2BaS4gydF4y2Ba.gydF4y2Ba
一个gydF4y2BaHIV-1 GKO报告载体示意图,显示HIV-1 5 ' LTR启动子转录控制下的csGFP基因(绿框)和组成型EF1alpha启动子控制下的mKO2基因(橙框)。gydF4y2BaBgydF4y2BaCEM-SS细胞被HIV感染gydF4y2Ba巨科gydF4y2Ba或艾滋病毒gydF4y2Ba巨科——ΔU3LTRgydF4y2Ba在A3F、A3F[E251A]、A3G或A3G[E259A]存在或不存在48小时的情况下。使用流式细胞仪,用僵尸近红外对活细胞进行门控gydF4y2BaTMgydF4y2Ba双阳性细胞(csGFP+, mKO2+)(蓝色条)和单阳性细胞((csGFP−,mKO2+)(红色条)或(csGFP+, mKO2−)(灰色条)的百分比。采用Dunnett多重比较检验(自由度= 36)进行双因素方差分析。gydF4y2BaCgydF4y2Ba潜伏感染细胞(csGFP−,mKO2+)从面板的平均比例gydF4y2BaBgydF4y2Ba显示。采用单因素方差分析和Tukey多重比较检验(自由度= 12)进行统计分析。所示数据为三个独立实验的平均值(±S.E.M.)。源数据作为源数据文件提供。gydF4y2Ba
HIV感染CEM-SS细胞后gydF4y2Ba巨科gydF4y2Ba在A3缺失的情况下,我们观察到与潜在整合(csGFP−,mKO2+)相比,具有生产性整合(csGFP+, mKO2+)的细胞比例更高(图3)。gydF4y2Ba8 b, CgydF4y2Ba).相反,细胞感染了等量的艾滋病毒gydF4y2Ba巨科gydF4y2BaA3F或A3F[E251A]的存在导致潜在整合的比例显著高于生产性整合。艾滋病毒gydF4y2Ba巨科gydF4y2BaA3G的存在也导致潜在整合的显著增加,但程度低于A3F和A3F[E251A]。与无A3对照组相比,A3G[E259A]存在时未观察到潜在或有效整合的比例有显著变化。不出所料,阴性控制了HIVgydF4y2Ba巨科gydF4y2Ba缺少3 ' LTR的U3启动子区域的病毒(HIVgydF4y2BaΔU3-GKOgydF4y2Ba)导致整合病毒只表达mKO2 (csGFP−,mKO2+)。总之,这些数据表明,A3F和A3G显著增加了体外潜在整合的比例,而A3G和A3F的脱氨活性促成了这种增加。gydF4y2Ba
讨论gydF4y2Ba
尽管存在许多细胞宿主限制因子,共同抑制HIV-1感染的早期阶段,HIV-1基因组与宿主基因组的整合仍然可以发生。整合事件将有不同的结果取决于整合的基因组位点。这些可能涉及直接调节宿主基因网络,控制病毒转录水平,并在某些情况下影响活跃或潜伏感染的结果gydF4y2Ba55gydF4y2Ba,gydF4y2Ba56gydF4y2Ba.通常情况下,A3蛋白在阻碍HIV-1 RNA基因组的逆转录和突变复制中间体中起主要作用;然而,HIV-1 Vif通过降低生产性感染细胞中的A3蛋白水平来规避这一限制。此外,包括A3F和A3G在内的一些A3蛋白已被证明与PIC相互作用并被易位到细胞核中gydF4y2Ba41gydF4y2Ba.这种细胞核定位的后果及其对HIV-1整合的影响以前是未知的。在这里,我们已经证明A3F和A3G显著改变HIV-1整合位点的选择(图。gydF4y2BaS5gydF4y2Ba).gydF4y2Ba
HIV-1具有积极转录基因区域的整合位点偏好,特别是那些在感染期间激活的基因区域gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba.毫无疑问,选择过程在感染细胞的扩张和持久性中起着重要作用,这在接受cART治疗的患者中得到了证明gydF4y2Ba59gydF4y2Ba.已知宿主细胞蛋白在HIV-1整合位点选择中起着关键作用。例如,LEDGF/p75和CPSF6促进整合到居住在基因密集区域的积极转录基因中gydF4y2Ba29gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba58gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba,gydF4y2Ba63gydF4y2Ba,gydF4y2Ba64gydF4y2Ba.在这里,我们发现宿主细胞A3的表达以剂量依赖的方式将HIV-1整合位点的积累从基因转移到SINEs。此外,A3表达的增加减少了人类基因组中整合位点热点的数量,这表明A3增加了整合位点的多样性。考虑到A3与PIC内整合酶相互作用的能力,了解A3是否在立体上干扰整合位点靶向因子(如LEDGF和CPSF6)结合并靶向PIC到细胞核和基因组转录活性区域的能力将是很有趣的。类似地,A3在3 ' LTR上的脱氨活性(例如,位置14/15)可以改变PIC内或PIC与整合位点靶向因子之间的蛋白质-核酸相互作用。当然,在HIV-1复制的后期,A3水平被Vif降低,这可能是病毒促进整合到更活跃的基因组区域以帮助建立生产性感染的另一种机制。考虑到A3表达的时间梯度,它们对整合的影响可能发生在新细胞被感染后的早期时刻,此时它在HIV病毒粒子中释放出许可水平的A3。此外,正如本文和其他研究小组所示,有证据表明感染个体中确实存在传染性脱氨原病毒基因组,因此支持A3影响整合的机会gydF4y2Ba65gydF4y2Ba,gydF4y2Ba66gydF4y2Ba,gydF4y2Ba67gydF4y2Ba,gydF4y2Ba68gydF4y2Ba.虽然大多数脱氨原病毒基因组在某种程度上存在缺陷,但其中一些仍然可以产生HIV RNA和病毒蛋白,从而在没有感染颗粒排出的情况下促进慢性免疫激活gydF4y2Ba56gydF4y2Ba.此外,我们的实验数据支持A3蛋白可以促进潜在的整合。在这个实验系统中,我们将这些定义为综合前病毒,其中5'LTR启动子不能在其控制下表达GFP报告基因,但从内部启动子维持mKO2报告基因的表达。这在催化活性和非活性A3变异体中都被观察到,进一步支持脱氨酶与PIC的简单物理关联可能影响整合HIV前病毒DNA的表达结果。由于整合位点在hiv -1感染细胞的扩增和持久性中也起着关键作用,这些a3导向的整合位点可能在患者潜伏感染的持久性中发挥作用。最后,wt A3蛋白催化活性的失活突变也可能导致潜在表型。需要对来自HIV患者的人类样本进行更多的研究,以确定长期潜伏感染细胞是否主要包含我们在这里确定的LTR中第14和15位的突变标记。gydF4y2Ba
A3改变整合位点谱的能力部分依赖于A3F和A3G的脱氨酶活性,但更强烈地依赖于A3G的核酸结合能力。我们之前的研究表明,核酸结合突变体A3G [W94A]和A3G [W127A]被包裹成病毒颗粒,尽管包裹程度较低,但与wt A3G相比,它们表现出类似的脱氨酶活性gydF4y2Ba14gydF4y2Ba.此外,这些突变体并没有减少病毒的晚期逆转录或整合gydF4y2Ba14gydF4y2Ba.我们发现,与其他A3结构相比,这些相同的突变体对HIV-1 PICs的整合位点选择显示出独特的影响,这可能意味着脱氨酶独立活性是影响整合位点选择的重要因素,但不是唯一因素。A3G [W94A]和A3G [W127A]突变体之间的一个关键区别是,虽然A3G [W94A]可以形成同型二聚体,但A3G [W127A]突变体在这方面不太擅长gydF4y2Ba14gydF4y2Ba.因此,A3G和A3G [W94A]之间观察到的差异可能是由于核酸亲和力降低,而A3G [W94A]和A3G [W127A]之间观察到的差异可能是由于多聚化缺陷gydF4y2Ba14gydF4y2Ba.gydF4y2Ba
基因组位置效应已被证明可以影响HIV-1的表达和潜伏期逆转gydF4y2Ba54gydF4y2Ba,gydF4y2Ba69gydF4y2Ba,gydF4y2Ba70gydF4y2Ba.LADs代表与核外围紧密相关的抑制性染色质环境gydF4y2Ba54gydF4y2Ba,gydF4y2Ba71gydF4y2Ba.sin(例如,Alu重复序列)和其他转座序列由于其被抑制的染色质标记(组蛋白H3在Lys9位点甲基化)而被认为是基因表达的直接沉默者。gydF4y2Ba72gydF4y2Ba,gydF4y2Ba73gydF4y2Ba,gydF4y2Ba74gydF4y2Ba.此外,一些非b DNA结构,包括G4、十字形、三倍体和Z-DNA,已被证明可以有效地抑制邻近基因的表达gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba,gydF4y2Ba77gydF4y2Ba,gydF4y2Ba78gydF4y2Ba,gydF4y2Ba79gydF4y2Ba,gydF4y2Ba80gydF4y2Ba,gydF4y2Ba81gydF4y2Ba,gydF4y2Ba82gydF4y2Ba,gydF4y2Ba83gydF4y2Ba,gydF4y2Ba84gydF4y2Ba,gydF4y2Ba85gydF4y2Ba.我们在这里发现,A3F和A3G都增加了LADs、sine和几种非b DNA基序内或附近HIV-1整合的频率,并增加了体外潜伏感染细胞的比例,这可能意味着A3促进了基因组中转录沉默区域的整合。有趣的是,与非脱氨3 ' LTR末端的前病毒相比,脱氨3 ' LTR末端的前病毒在非b DNA基序中高度富集并接近基因沉默。然而,目前尚不清楚这些突变的病毒是否具有复制能力,因为LTR中的突变表明在病毒基因组的其他地方也可能发现突变。潜伏病毒颗粒的转录激活和特异性恢复是一项非常复杂的挑战,因为它们只占释放病毒总量的很小一部分。gydF4y2Ba
总之,我们首次证明A3酶可以通过脱氨酶依赖性和非依赖性机制调节HIV-1的整合位点谱。虽然这些A3蛋白最强的限制性特征被确定为它们的脱氨酶活性,但即使是非限制性突变体也保持了与整合酶相互作用和调节HIV-1整合位点选择的能力。目前,A3在影响HIV-1整合位点谱和疾病进展方面的总体影响尚不清楚。A3可能代表了最后的努力,将肠道从基因引导到更有可能转录沉默的基因组区域,以促进前病毒沉默。需要进一步的努力来解剖这一现象,并确定A3对前病毒沉默的影响。gydF4y2Ba
方法gydF4y2Ba
本研究符合所有相关的伦理规范。从JCRC和UHCMC/CWRU的irb获得伦理许可(EM-10-07和10-05-35)。gydF4y2Ba
细胞系和质粒gydF4y2Ba
细胞系保持在完全培养基(10%胎牛血清、100 U/mL青霉素和100µg/mL链霉素)中。HEK 293 T细胞(ATCC CRL-3216gydF4y2BaTMgydF4y2Ba)维持在高糖的完全DMEM中。完全RPMI维持CEM-SS细胞(NIH AIDS #776)。两种细胞储备均保存在37°C加5% CO的湿化培养箱中gydF4y2Ba2gydF4y2Ba.NL4-3- ΔVif/ΔEnv-eGFP是通过对NL4-3 ΔEnv-eGFP的定点诱变而开发的,它最初是从NIH艾滋病试剂计划(N.A.R.P.)(目录#11100)获得的。gydF4y2Ba14gydF4y2Ba.如前所述,用水泡性口炎病毒g蛋白(VSV-G) (pMDG)对NL4-3- ΔVif/ ΔEnv-eGFP进行假分型gydF4y2Ba21gydF4y2Ba,gydF4y2Ba86gydF4y2Ba.pcDNA 3.1 (Invitrogen)作为空载体对照进行转染,所有表达A3的质粒之前都有描述gydF4y2Ba21gydF4y2Ba,gydF4y2Ba86gydF4y2Ba.质粒pHIVgydF4y2Ba巨科gydF4y2Ba和pHIVgydF4y2Ba巨科——ΔU3LTRgydF4y2Ba由Eric Verdin博士(巴克研究所)提供。gydF4y2Ba
病毒产生和感染gydF4y2Ba
HEK 293 T细胞7.5 × 10接种gydF4y2Ba5gydF4y2Ba6孔板中每孔的细胞。播种24小时后,将携带NL4-3-ΔEnv/ΔVif/eGFP报告载体和pMDG的质粒与聚乙烯亚胺(PEI)指示的空载体或A3质粒一起共转染细胞。gydF4y2Ba87gydF4y2Ba.当NL4-3-ΔEnv/ΔVif/eGFP和pMDG质粒的比例保持不变(750 ng: 250 ng)时,共转染空载体或A3质粒的水平根据实验而变化(30、100和250 ng)。对于A3G和A3G- e259a,转染20 ng,以确保有足够的细胞数量用于流式细胞术分析。用空载体(pcDNA 3.1)保持DNA转染总量不变。细胞孵育72 h产生病毒。病毒生产经抗p24capsid (N.A.R.P. #1513)、抗flag(克隆M2;Sigma)和抗-β-微管蛋白(ab21058;Abcam)。收集病毒上清液,500 ×离心gydF4y2BaggydF4y2Ba用450nm注射器过滤5分钟,去除细胞碎片。此时,使用从杂交瘤31-90-25 (#HB-9725;和183-H12-5C (N.A.R.P. #1513)。收集病毒20小时后,将CEM-SS细胞以5 × 10的密度播种于12孔板中gydF4y2Ba5gydF4y2Ba每孔细胞,以标准衣壳水平(500,100,或20ng)感染,以900 × spinoculation 1 hgydF4y2BaggydF4y2Ba聚凝胺。培养48 h后收集细胞进行下游流式细胞仪分析和gDNA提取。采用BD FACSDiva (Software v8.0.1), BD FACSCelesta进行流式细胞仪分析。采集后分析使用FlowJo(软件v10.4.2)在一台单独的计算机上进行。使用Wizard gDNA纯化试剂盒(Promega)从CEM-SS细胞中分离纯化gDNA。准型艾滋病毒gydF4y2Ba巨科gydF4y2Ba和艾滋病毒gydF4y2Ba巨科——ΔU3LTRgydF4y2BaHEK293T细胞与pHIV共转染产生病毒gydF4y2Ba巨科gydF4y2Ba或pHIVgydF4y2Ba巨科——ΔU3LTRgydF4y2Ba和pMD。Gin the presence or absence of plasmids carrying A3 (200 ng) for 72 h. Sandwich-ELISA was performed to determine levels of capsid protein (p24CA) as described above. CEM-SS cells were infected with HIV巨科gydF4y2Ba或pHIVgydF4y2Ba巨科——ΔU3LTRgydF4y2Ba(相当于120 ng p24CA蛋白)通过900次spinoculationgydF4y2BaggydF4y2Ba在10µg/ml聚苯乙烯(Sigma-Aldrich, #H9268-5G)存在的室温下1小时。在消旋后,去除病毒,并将新鲜培养基添加到井中4天。然后使用流式细胞仪对感染细胞进行分析(BD FACSCelesta使用BD FACSDiva (Software v8.0.1))。采集后分析使用FlowJo(软件v10.4.2)在一台单独的计算机上进行。通过将(csGFP−,mKO2+)或(csGFP+, mKO2+)细胞的百分比分别除以阳性细胞的总百分比来计算潜在整合或有效整合的比例。gydF4y2Ba
铝基qPCR检测整合前病毒gydF4y2Ba
PowerUpgydF4y2BaTMgydF4y2Ba使用SYBR Master Mix (ThermoFisher)使用Viia量化细胞的相对水平gydF4y2BaTMgydF4y2Ba7 Real-Time PCR仪(Applied Biosystems), 50 ng gDNA模板,使用以下引物:Actin-FWD 5 ' -CAT GTA CGT TGC TAT CCA GGC-3 '和Actin-REV 5 ' -CTC CTT AAT GTC ACG CAC GAT-3 '。循环条件:95°C初始变性3分钟,然后95°C 15秒,60°C 1分钟,循环45次。数据分析采用QuantStudio (version 1.6.1)软件。接下来,与之前描述的方案类似,使用50 ng gDNA和PrimeStar GXL DNA聚合酶(Takara)在以下条件下进行Alu-PCR:在94°C初始变性1分钟,然后98°C变性10秒,55°C变性15秒,68°C变性10分钟,循环30次,最后以68°C延长10分钟结束gydF4y2Ba46gydF4y2Ba.所有针对HIV-1序列的引物都被设计为排除A3二核苷酸热点,以避免引起PCR偏差。为了量化整合的eGFP序列,使用了以下引物:Alu1 5 ' -TCC CAG CTA CTG GGG AGG CTG AGG-3 ', Alu2 5 ' -GCC TCC CAA AGT GCT GGG ATT ACA G-3 '和Lambda-eGFP-FWD 5 ' -ATG CCA CGT AAG CGA AAC TGT ACA ACT ACA ACA GCC ACA ACG TCT ATA TC-3 '。使用QX200系统(BioRad)在以下条件下对其稀释度进行ddPCR分析:在95°C初始变性10分钟,然后在94°C 30秒,60°C 30秒,72°C 30秒的45个循环。最后在98°C变性10分钟。使用了以下引物:lamb大- f2 5 ' -ATG CCA CGT AAG CGA AAC TGT ACA ACT AC-3 ', HIV eGFP REV 5 ' -TGA GGA TTG CTT AAA GAT TAT TGT TTT ATT ATT T-3 '。该探针使用:/5HEX/ CCC CGT GCT /ZEN/ GCT GCC CRA CAA CCA CTA CC /3IABkFQ。为了量化整合的5 ' LTR序列,使用了以下引物:Lambda-R-U5-REV1 5 ' -AGT TTC GCT TAC GTG GCA TCA GAC GGG CAC ACA CTA CTT TGA GCA C-3 ', Alu1 Comp 5 ' -CCT CAG CCT CCC CAG TAG CTG GGA-3 '和Alu2 Comp 5 ' -CTGT AAT CCC AGC ACT TTG GGA GGC-3 '。使用上述相同的条件和以下引物:LambdaR-REV2 5 ' -GTT TCG CTT ACG TGG CAT CAG ACG G-3 '和Late U3-FWD 5 ' -GCT ACA TAT AAG CAG CTG CTT TTT GCC TGT AC−3 ',用ddPCR对其进行稀释分析。使用探针:/5YAkYel/ CTT TAT TGA GGC T + T AAG + C + AG + T + G + GG T/3IABkFQ。 Nucleotides followed by a+ (N+) indicate an LNA base to improve the melting temperature of the probe. Results from the Alu-PCR that quantified integrated proviruses using the eGFP sequence or the 5’ LTR sequence primers were then averaged.
免疫沉淀反应gydF4y2Ba
用NL4-3-ΔEnv/ΔVif/eGFP和VSV-G转染HEK 293 T细胞,清除病毒上清中的细胞碎片。病毒上清在10万倍浓度下,用20%蔗糖垫超离心浓缩gydF4y2BaggydF4y2Ba使用70Ti型在4°C下加热3小时。病毒颗粒重悬于等渗1% triton - x100裂解缓冲液中。同时,使用500 mM NaCl软裂解缓冲液裂解病毒产生细胞,有效地击破细胞核,最大限度地释放整合酶。蛋白酶抑制剂(罗氏)一直用于防止蛋白质降解。盐活性核酸酶(Sigma)被用来去除gDNA根据制造商的协议。剩余的细胞碎片在4°C, 17000 ×离心去除gydF4y2BaggydF4y2Ba10分钟。细胞和上清液裂解物混合在一起,以最大限度地分离出病毒成分。使用无菌水将总盐水平恢复到等渗状态。上述A3或pcDNA 3.1质粒与NL4-3-ΔEnv/ΔVif/eGFP转染同时在各自的孔中分别转染。转染72小时后,收集细胞并用等渗的1% Triton-X 100裂解缓冲液裂解。对裂解物进行超声处理以提高蛋白质的溶解性,并在17000 ×下离心gydF4y2BaggydF4y2Ba在4°C下浸泡10分钟,以清除剩余的细胞碎片。在免疫沉淀之前收集每种裂解物的样本以评估输入水平。病毒裂解液在含有A3或对照的细胞裂解液中平均分配。然后将这些裂解物与30 μ L的抗flag共轭磁μ珠(Miltenyi)混合,并在管旋转器上在4°C下孵育3小时。然后,根据制造商的说明,使用µ柱将µ珠磁隔离。使用抗in (IN-2, Santa Cruiz BioTechnology, sc-69721)、抗p24衣壳(N.A.R.P. #1513)、抗flag(克隆M2;Sigma),抗-β-微管蛋白(ab21058;Abcam)。使用Image Quant LAS4000采集Western blots图像,并使用Image Quant TL 8.1版软件进行分析。gydF4y2Ba
集成站点库和计算分析gydF4y2Ba
利用Illumina MiSeq平台对基因组DNA进行整合位点分析和测序gydF4y2Ba36gydF4y2Ba,gydF4y2Ba50gydF4y2Ba.简单地说,用MseI和NarI酶切基因组DNA,用连接介导PCR扩增3 ' ltr -宿主基因组连接。PCR产物经凝胶纯化后,用Nextera XT DNA样品制备试剂盒对纯化的DNA样品进行处理。进行了有限环PCR反应来扩增插入DNA,然后在伦敦区域基因组学中心(加拿大西部大学Robarts研究所)使用Illumina MiSeq (2×150 bp化学)对其进行测序。Fastq测序reads经过高质量修剪,并使用我们内部的生物信息学管道确定了独特的整合位点gydF4y2Ba36gydF4y2Ba,它被称为Barr Lab集成站点标识管道(BLISIP版本2.9),包括以下更新:bedtools (v2.25.0), bioawk (awk版本20110810),bowtie2(版本2.3.4.1)和restrSiteUtils (v1.2.9)。如果3 ' LTR序列与人类基因组的任何区域(GRCh37/hg19)不匹配,则通过允许最多5个与参考NL4-3 ' LTR序列不匹配来识别和过滤HIV-1含有3 ' LTR的fastq序列。整合位点由3’LTR与人类基因组序列的序列交叉点确定。每个数据集中包含两个或多个位点(即相同位点)的所有基因组位点被分解为一个独特的位点供我们分析。使用我们内部的python程序BLISIPHA Heatmap (BLISIPHA v1.0)对位于各种常见基因组特征和非b DNA基基的位点进行量化,并生成热图。无法明确定位到基因组中单个区域的位点被排除在研究之外。所有非b DNA基序均根据先前建立的标准进行定义gydF4y2Ba88gydF4y2Ba.匹配的随机控制积分位点是通过将每个实验确定的位点与硅晶片上的10个随机位点相匹配来生成的,这些随机位点被构造成与实验位点距离限制位点的碱基数量相同gydF4y2Ba36gydF4y2Ba.独特的HIV 3 ' lts被BLISIP识别,与MUSCLE(10.1.7版本)对齐gydF4y2Ba89gydF4y2Ba与trimAl(版本1.2)的缺口剥离gydF4y2Ba90gydF4y2Ba.所有在40%以上的人群中存在间隙的柱都被间隙剥离。使用WebLogo(3.6版)生成唯一的LTR序列徽标gydF4y2Ba52gydF4y2Ba.gydF4y2Ba
病人样本的收集和准备gydF4y2Ba
样本采集自乌干达坎帕拉联合临床研究中心(JCRC)的WHO、CAP和nih - vqa认可的艾滋病研究中心(CFAR)实验室。JCRC是该国首批推出抗逆转录病毒疗法的艾滋病毒治疗中心之一,目前也是该国唯一获准提供inss的场所。根据各种纳入和排除标准,在2002年至2007年期间,在激素避孕药的HIV-1感染风险研究中,招募了艾滋病毒阴性的育龄妇女(18-35岁),并在咨询和签署同意书后自愿(无报酬)作为参与者。如果一名女性在上述父母研究期间被诊断为HIV-1,则会被邀请参加一项辅助研究,以确定疾病进展的标志物,同样是在征得同意并接受咨询的情况下。CFAR实验室的患者数据库仅用于访问HIV-1感染患者的样本ID号码。除了HIV-1感染状况外,作者对所有临床数据都不了解。从JCRC接受常规治疗的HIV-1感染者中,随机收集了93个先前冷冻和保存的PBMC样本,其中一些也来自激素避孕和HIV-1生殖器脱落和原发性HIV感染女性(GS)疾病进展研究gydF4y2Ba91gydF4y2Ba.从JCRC和UHCMC/CWRU的irb获得伦理许可(EM-10-07和10-05-35)。按照制造商的说明,使用DNeasy血液和组织试剂盒(Qiagen)提取基因组DNA,并将提取的DNA储存在−80°C。gydF4y2Ba
统计和再现性gydF4y2Ba
使用GraphPad Prism 9 9.4.1版本执行所有统计测试,如图图例所示。没有使用统计方法来预先确定样本量。分析中不排除任何数据。实验不是随机的。在实验和结果评估期间,研究人员并没有对分配盲目。研究人员对所有用于整合位点和LTR序列分析的患者样本进行盲法分析。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然组合报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
本研究生成的源数据在补充信息/源数据文件中提供。整合位点在人类基因组中的位置是从GRCh37/hg19数据库(gydF4y2Bahttps://hgdownload.soe.ucsc.edu/downloads.htmlgydF4y2Ba).本研究生成的整合位点测序数据已存入NCBI SRA数据库,登录码为SAMN31866157-SAMN31866258 [gydF4y2Bahttp://www.ncbi.nlm.nih.gov/bioproject/905178gydF4y2Ba]。gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
Bedtools (v2.25.0) (gydF4y2Bahttps://github.com/arq5x/bedtools2/releasesgydF4y2Ba)、生物awk (awk版本20110810)(gydF4y2Bahttps://github.com/lh3/bioawkgydF4y2Ba), bowtie2(版本2.3.4.1)(gydF4y2Bahttps://github.com/BenLangmead/bowtie2gydF4y2Ba),和ressiteutils (v1.2.9) (gydF4y2Bahttps://github.com/chasberry/integration-site-MRCsgydF4y2Ba)用于收集和分析基因组DNA序列和特征。非b DNA基基的整合位点是使用gquad R软件包(2.1-1版)识别的,该软件包可从综合R档案网络(gydF4y2Bahttps://cran.r-project.org/web/packages/gquad/index.htmlgydF4y2Ba).该包提供了预测非b DNA的功能。代码的关键特征和与测试数据集相关的细节可以在gquad文档中找到。gydF4y2Ba
参考文献gydF4y2Ba
OhAinle, M., Kerns, J. A., Li, M. M., Malik, H. S. & Emerman, M. APOBEC3H的抗逆转录元素活性在最近的人类进化中两次丢失。gydF4y2Ba细胞宿主微生物gydF4y2Ba4gydF4y2Ba, 249-259(2008)。gydF4y2Ba
王,等。人APOBEC3H单倍型与抗人类免疫缺陷病毒1型活性分析gydF4y2Baj .性研究。gydF4y2Ba85gydF4y2Ba, 3142-3152(2011)。gydF4y2Ba
维特科普,陈俊杰,吴丽丽,李丽娟,李丽娟,李丽娟。人APOBEC3C基因单核苷酸多态性增强慢病毒的限制性。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba12gydF4y2Ba, e1005865(2016)。gydF4y2Ba
哈里斯,R. S. & Dudley, J. P. APOBECs和病毒限制。gydF4y2Ba病毒学gydF4y2Bahttps://doi.org/10.1016/j.virol.2015.03.012gydF4y2Ba(2015)。gydF4y2Ba
Mariani, R.等人。Vif从HIV-1病毒粒子中排除APOBEC3G的物种特异性。gydF4y2Ba细胞gydF4y2Ba114gydF4y2Ba, 21-31(2003)。gydF4y2Ba
哈里斯,R. S.等。DNA脱氨介导逆转录病毒感染的先天免疫。gydF4y2Ba细胞gydF4y2Ba113gydF4y2Ba, 803-809(2003)。gydF4y2Ba
于,Q.等。APOBEC3G的单链特异性解释了HIV基因组的负链脱氨。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba11gydF4y2Ba, 435-442(2004)。gydF4y2Ba
Chelico, L., Pham, P. & Goodman, m.f. apobec3g催化脱氧胞苷在单链DNA上的过程脱氨机制。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba16gydF4y2Ba, 454-455(2009)。gydF4y2Ba
Sheehy, a.m., Gaddis, n.c., Choi, J. D.和Malim . M. H.一种抑制HIV-1感染的人类基因的分离,并被病毒Vif蛋白抑制。gydF4y2Ba自然gydF4y2Ba418gydF4y2Ba, 646-650(2002)。gydF4y2Ba
Stopak, K., de Noronha, C., Yonemoto, W. & Greene, W. C. HIV-1 Vif通过损害APOBEC3G的翻译和细胞内稳定性来阻断其抗病毒活性。gydF4y2Ba摩尔。细胞gydF4y2Ba12gydF4y2Ba, 591-601(2003)。gydF4y2Ba
Sheehy, a.m., Gaddis, n.c.和Malim, m.h.抗逆转录病毒酶APOBEC3G被蛋白酶体降解,以响应HIV-1 Vif。gydF4y2Ba地中海Nat。gydF4y2Ba.gydF4y2Ba9gydF4y2Ba, 1404-1407(2003)。gydF4y2Ba
冯勇,拜格,T. T., Love, R. P. & Chelico, L. Vif抑制apobec3介导的HIV-1限制。gydF4y2Ba前面。Microbiol。gydF4y2Ba5gydF4y2Ba, 450(2014)。gydF4y2Ba
奥尔森,M. E.哈里斯,R. S.和Harki, D. A. APOBEC酶作为病毒和癌症治疗的靶标。gydF4y2Ba细胞化学。医学杂志。gydF4y2Ba25gydF4y2Ba, 36-49(2018)。gydF4y2Ba
Belanger, K., Savoie, M., Rosales Gerpe, M. C., Couture, J. F. & Langlois, M. A.通过APOBEC3G结合RNA控制反转录病毒的脱氨独立限制。gydF4y2Ba核酸测定。gydF4y2Ba41gydF4y2Ba, 7438-7452(2013)。gydF4y2Ba
霍姆斯,R. K.马利姆,M. H.和Bishop, K. N. apobeck介导的病毒限制:不仅仅是编辑。gydF4y2Ba学生物化学的发展趋势。科学。gydF4y2Ba32gydF4y2Ba, 118-128(2007)。gydF4y2Ba
Pollpeter, D.等人。对HIV-1逆转录本的深度测序揭示了APOBEC3G的多方面抗病毒功能。gydF4y2BaMicrobiol Nat。gydF4y2Ba3.gydF4y2Ba, 220-233(2018)。gydF4y2Ba
纽曼,e.n.等人。APOBEC3G的抗病毒功能可与胞苷脱氨酶活性分离。gydF4y2Ba咕咕叫。医学杂志。gydF4y2Ba15gydF4y2Ba, 166-170(2005)。gydF4y2Ba
Iwatani, Y.等。APOBEC3G不依赖脱氨酶抑制HIV-1逆转录gydF4y2Ba核酸测定。gydF4y2Ba35gydF4y2Ba, 7096-7108(2007)。gydF4y2Ba
Bishop, K. N., Verma, M., Kim, E. Y., Wolinsky, S. M. & Malim . M. H. APOBEC3G抑制HIV-1逆转录本的延伸。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba4gydF4y2Ba, e1000231(2008)。gydF4y2Ba
Mangeat, B.等。人类APOBEC3G通过致命编辑新生逆转录本进行广泛的抗逆转录病毒防御。gydF4y2Ba自然gydF4y2Ba424gydF4y2Ba, 99-103(2003)。gydF4y2Ba
Belanger, K. & Langlois, m.a . APOBEC3G n端rna结合残基影响其DNA序列特异性和逆转录病毒限制效率。gydF4y2Ba病毒学gydF4y2Ba483gydF4y2Ba, 141-148(2015)。gydF4y2Ba
Luo, K.等。胞苷脱氨酶APOBEC3G和APOBEC3F与人类免疫缺陷病毒1型整合酶相互作用并抑制前病毒DNA的形成。gydF4y2Baj .性研究。gydF4y2Ba81gydF4y2Ba, 7238-7248(2007)。gydF4y2Ba
王,等。细胞抗病毒蛋白APOBEC3G与HIV-1逆转录酶相互作用,并在病毒复制过程中抑制其功能。gydF4y2Baj .性研究。gydF4y2Ba86gydF4y2Ba, 3777-3786(2012)。gydF4y2Ba
吴,等。人类免疫缺陷病毒1型整合酶蛋白通过与核蛋白逆转录复合物的特异性相互作用促进逆转录。gydF4y2Baj .性研究。gydF4y2Ba73gydF4y2Ba, 2126-2135(1999)。gydF4y2Ba
Mbisa, J. L., Bu, W. & Pathak, V. K. APOBEC3F和APOBEC3G通过不同机制抑制HIV-1 DNA整合。gydF4y2Baj .性研究。gydF4y2Ba84gydF4y2Ba, 5250-5259(2010)。gydF4y2Ba
Mbisa, J. L.等。在APOBEC3G存在下产生的人类免疫缺陷病毒1型cdna在正链DNA转移和整合方面表现出缺陷。gydF4y2Baj .性研究。gydF4y2Ba81gydF4y2Ba, 7099-7110(2007)。gydF4y2Ba
Raghavendra, n.k.等人。从受感染的CD4+细胞中分离出与HIV-1预整合复合物相关的宿主蛋白的鉴定。gydF4y2BaRetrovirologygydF4y2Ba7gydF4y2Ba, 66(2010)。gydF4y2Ba
Lusic, M. & Siliciano, R. F. HIV-1感染和整合的核景观。gydF4y2BaNat. Rev.微生物。gydF4y2Ba15gydF4y2Ba, 69-82(2017)。gydF4y2Ba
Maertens, G.等人。LEDGF/p75对于人类细胞中HIV-1整合酶的核靶向和染色体靶向至关重要。gydF4y2Ba生物。化学。gydF4y2Ba278gydF4y2Ba, 33528-33539(2003)。gydF4y2Ba
切列帕诺夫等人。HIV-1整合酶在人细胞中形成稳定的四聚体,并与LEDGF/p75蛋白结合。gydF4y2Ba生物。化学。gydF4y2Ba278gydF4y2Ba, 372-381(2003)。gydF4y2Ba
切列帕诺夫等人。LEDGF/p75中HIV-1整合酶结合域的溶液结构。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba12gydF4y2Ba, 526-532(2005)。gydF4y2Ba
Sowd, g.a.等。替代多聚腺苷酸因子CPSF6在靶向HIV-1整合转录活性染色质中的关键作用。gydF4y2Ba国家科学院学报美国gydF4y2Ba113gydF4y2Ba, e1054-e1063(2016)。gydF4y2Ba
Maskell, d.p.等人。逆转录病毒整合到核小体的结构基础。gydF4y2Ba自然gydF4y2Ba523gydF4y2Ba, 366-369(2015)。gydF4y2Ba
Kirk, P. D., Huvet, M., Melamed, A., Maertens, G. N. & Bangham, C. R.逆转录病毒整合成一个共享的非回文DNA基序。gydF4y2BaMicrobiol Nat。gydF4y2Ba2gydF4y2Ba, 16212(2016)。gydF4y2Ba
科恩,L. B.等。HIV-1在潜伏感染和活动性感染期间的整合情况。gydF4y2Ba细胞gydF4y2Ba160gydF4y2Ba, 420-432(2015)。gydF4y2Ba
McAllister, R. G.等。异染性脑白质营养不良模型室管膜细胞中的慢载体整合位点:非b DNA作为影响整合的新因素。gydF4y2Ba摩尔。其他。核酸gydF4y2Ba3.gydF4y2Ba, e187(2014)。gydF4y2Ba
Ho, p.s. d(CA/TG)n的非b - dna结构与Z-DNA的结构没有区别。gydF4y2Ba国家科学院学报美国gydF4y2Ba91gydF4y2Ba, 9549-9553(1994)。gydF4y2Ba
范霍德,K. & Zlatanova, J.不寻常的DNA结构,染色质和转录。gydF4y2BaBioessaysgydF4y2Ba16gydF4y2Ba, 59-68(1994)。gydF4y2Ba
Bacolla, A. & Wells, R. D.非b DNA构象,基因组重排与人类疾病。gydF4y2Ba生物。化学。gydF4y2Ba279gydF4y2Ba, 47411-47414(2004)。gydF4y2Ba
威尔斯,r.d.非b DNA构象,突变和疾病。gydF4y2Ba学生物化学的发展趋势。科学。gydF4y2Ba32gydF4y2Ba, 271-278(2007)。gydF4y2Ba
Burdick, R. C, Hu, W. S. & Pathak, V. K. apobec3f标记HIV-1预整合复合物的核导入。gydF4y2Ba国家科学院学报美国gydF4y2Ba110gydF4y2Ba, e4780-e4789(2013)。gydF4y2Ba
Huthoff, H., Autore, F., Gallois-Montbrun, S., Fraternali, F. & Malim . m.h. rna依赖的APOBEC3G寡聚是限制HIV-1所必需的。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba5gydF4y2Ba, e1000330(2009)。gydF4y2Ba
戈尔,S.等。单体全长APOBEC3G的计算模型及动力学研究。gydF4y2BaACS分科学gydF4y2Ba3.gydF4y2Ba, 1180-1188(2017)。gydF4y2Ba
拉文斯,D.等人。用MAPPIT突变分析定义Apobec3G和HIV-1 Vif的相互作用界面。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba38gydF4y2Ba, 1902-1912(2010)。gydF4y2Ba
Shlyakhtenko, L. S.等。原子力显微镜研究为HIV限制因子APOBEC3G的二聚化提供了直接证据。gydF4y2Ba生物。化学。gydF4y2Ba286gydF4y2Ba, 3387-3395(2011)。gydF4y2Ba
Vandergeeten, C.等人。跨支超灵敏pcr检测大队列研究中艾滋病毒持久性gydF4y2Baj .性研究。gydF4y2Ba88gydF4y2Ba, 12385-12396(2014)。gydF4y2Ba
Belanger, K. & Langlois, m.a .逆转录病毒限制因子APOBEC3F和APOBEC3G基因失活潜力的比较分析gydF4y2BaJ. Gen. virrolgydF4y2Ba.gydF4y2Bahttps://doi.org/10.1099/vir.0.000214gydF4y2Ba(2015)。gydF4y2Ba
Alce, T. M. & Popik, W. APOBEC3G通过与HIV-1 Gag核衣壳蛋白直接相互作用被合并到病毒样颗粒中。gydF4y2Ba生物。化学。gydF4y2Ba279gydF4y2Ba, 34083-34086(2004)。gydF4y2Ba
岑,S.等。HIV-1 Gag与APOBEC3G的相互作用。gydF4y2Ba生物。化学。gydF4y2Ba279gydF4y2Ba, 33177-33184(2004)。gydF4y2Ba
Ciuffi, A. & Barr, S. D.感染宿主基因组DNA中HIV整合位点的鉴定。gydF4y2Ba方法gydF4y2Ba53gydF4y2Ba, 39-46(2011)。gydF4y2Ba
Cattoglio, C.等。人CD34+造血细胞逆转录病毒整合的热点。gydF4y2Ba血gydF4y2Ba110gydF4y2Ba, 1770-1778(2007)。gydF4y2Ba
克鲁克斯,G. E, Hon, G., Chandonia, J. M. & Brenner, S. E. WebLogo:一个序列logo生成器。gydF4y2Ba基因组Res。gydF4y2Ba14gydF4y2Ba, 1188-1190(2004)。gydF4y2Ba
施耐德,T. D. &斯蒂芬斯,R. M.序列标识:显示一致序列的新方法。gydF4y2Ba核酸测定。gydF4y2Ba18gydF4y2Ba, 6097-6100(1990)。gydF4y2Ba
Battivelli, E.等人。在受感染的原代CD4(+) T细胞中,不同的染色质功能状态与HIV潜伏期再激活相关。gydF4y2BaElifegydF4y2Bahttps://doi.org/10.7554/eLife.34655gydF4y2Ba(2018)。gydF4y2Ba
Soto-Giron, M. J. & Garcia-Vallejo, F.人类免疫缺陷病毒1型(HIV-1)在巨噬细胞中整合基因表达网络拓扑结构的变化。gydF4y2Ba病毒Res。gydF4y2Ba163gydF4y2Ba, 91-97(2012)。gydF4y2Ba
西蒙斯,J.,卡梅伦,P. U. & Lewin, S. R. HIV整合位点及其对维持水库的影响。gydF4y2Ba咕咕叫。当今。艾滋病gydF4y2Ba13gydF4y2Ba, 152-159(2018)。gydF4y2Ba
Ciuffi, A. & Bushman, F. D.逆转录病毒DNA整合:HIV和LEDGF/p75的作用。gydF4y2Ba趋势麝猫。gydF4y2Ba22gydF4y2Ba, 388-395(2006)。gydF4y2Ba
施罗德,a.r.等。HIV-1在人类基因组中的整合有利于活跃基因和局部热点。gydF4y2Ba细胞gydF4y2Ba110gydF4y2Ba, 521-529(2002)。gydF4y2Ba
Maldarelli, F.等人。艾滋病毒延迟。特定的HIV整合位点与被感染细胞的克隆扩增和持久性有关。gydF4y2Ba科学gydF4y2Ba345gydF4y2Ba, 179-183(2014)。gydF4y2Ba
Achuthan, V.等人。衣壳- cpsf6相互作用许可核HIV-1贩运到病毒DNA整合位点。gydF4y2Ba细胞宿主微生物gydF4y2Ba24gydF4y2Ba, 392-404 e398(2018)。gydF4y2Ba
辛格,P. K.等。LEDGF/p75与mRNA剪接因子相互作用,并靶向HIV-1整合到高度剪接的基因。gydF4y2BaDev的基因。gydF4y2Ba29gydF4y2Ba, 2287-2297(2015)。gydF4y2Ba
马歇尔,h.m.等人。PSIP1/LEDGF/p75在慢病毒感染性和整合靶向中的作用gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba2gydF4y2Ba, e1340(2007)。gydF4y2Ba
Shun, m.c.等。LEDGF/p75在预整合复合物形成的下游发挥作用,影响基因特异性HIV-1整合。gydF4y2BaDev的基因。gydF4y2Ba21gydF4y2Ba, 1767-1778(2007)。gydF4y2Ba
Ciuffi, A.等。LEDGF/p75在靶向HIV DNA整合中的作用gydF4y2BaNat,地中海。gydF4y2Ba11gydF4y2Ba, 1287-1289(2005)。gydF4y2Ba
金,E. Y.等人。人类APOBEC3诱导的人类免疫缺陷病毒1型突变有助于自然感染的适应和进化。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba10gydF4y2Ba, e1004281(2014)。gydF4y2Ba
Cuevas, J. M., Geller, R., Garijo, R., Lopez-Aldeguer, J. & Sanjuan, R.体内HIV-1的高突变率。gydF4y2Ba公共科学图书馆杂志。gydF4y2Ba13gydF4y2Ba, e1002251(2015)。gydF4y2Ba
佐藤,K.等。APOBEC3D和APOBEC3F可有效促进HIV-1在人源小鼠模型中的多样化和进化。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba10gydF4y2Ba, e1004453(2014)。gydF4y2Ba
Sadler, H. A., Stenglein, m.d ., Harris, R. S. & Mansky, L. M. APOBEC3G通过亚致死诱变导致HIV-1变异。gydF4y2Baj .性研究。gydF4y2Ba84gydF4y2Ba, 7396-7404(2010)。gydF4y2Ba
陈,H. C.,马丁内斯,J. P., Zorita, E., Meyerhans, A. & Filion, G. J.位置效应影响HIV潜伏期逆转。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba24gydF4y2Ba, 47-54(2017)。gydF4y2Ba
Vranckx, L. S.等。ledin介导的整合酶- ledgf /p75相互作用抑制减少了残余潜伏HIV的再激活。gydF4y2BaEBioMedicinegydF4y2Ba8gydF4y2Ba, 248-264(2016)。gydF4y2Ba
Marini, B.等人。核结构决定HIV-1整合位点的选择。gydF4y2Ba自然gydF4y2Ba521gydF4y2Ba, 227-231(2015)。gydF4y2Ba
姜建昌,姜建昌,李丽娟。人转座子是乳腺癌细胞系中转录因子结合位点和启动子活性的丰富来源。gydF4y2Ba暴徒。DNAgydF4y2Ba10gydF4y2Ba, 16(2019)。gydF4y2Ba
Schmitz, J. SINEs作为基因组进化的驱动力。gydF4y2Ba基因组动力学。gydF4y2Ba7gydF4y2Ba, 92-107(2012)。gydF4y2Ba
近藤,Y. & Issa, J. P.组蛋白H3赖氨酸9甲基化在人细胞Alu重复序列的富集。gydF4y2Ba生物。化学。gydF4y2Ba278gydF4y2Ba, 27658-27662(2003)。gydF4y2Ba
鲍克曼,M. L., Paeschke, K. & Zakian, V. A. DNA二级结构:g -四重结构的稳定性和功能。gydF4y2BaNat. Rev. Genet。gydF4y2Ba13gydF4y2Ba, 770-780(2012)。gydF4y2Ba
Brazda, V., Laister, R. C., Jagelska, E. B. & Arrowsmith, C.十字形结构是一种常见的DNA特征,对调节生物过程很重要。gydF4y2BaBMC Mol. Biol。gydF4y2Ba12gydF4y2Ba, 33(2011)。gydF4y2Ba
Jain, A., Magistri, M., Napoli, S., Carbone, g.m. & Catapano, C. V.细胞中三重dna介导的转录起始抑制机制。gydF4y2BaBiochimiegydF4y2Ba92gydF4y2Ba, 317-320(2010)。gydF4y2Ba
Verma, A., Yadav, V. K., Basundra, R., Kumar, A. & Chowdhury, S.人类癌细胞中全基因组G4 dna介导基因表达的证据。gydF4y2Ba核酸测定。gydF4y2Ba37gydF4y2Ba, 4194-4204(2009)。gydF4y2Ba
Tornaletti, S., Park-Snyder, S. & Hanawalt, p.c.非转录DNA链中的g4形成序列对T7 RNA聚合酶和哺乳动物RNA聚合酶II构成阻断。gydF4y2Ba生物。化学。gydF4y2Ba283gydF4y2Ba, 12756-12762(2008)。gydF4y2Ba
Belotserkovskii, B. P.等。来自人类c-MYC启动子的三核苷酸序列干扰DNA转录。gydF4y2Ba生物。化学。gydF4y2Ba282gydF4y2Ba, 32433-32441(2007)。gydF4y2Ba
Siddiqui-Jain, A., Grand, C. L., Bearss, D. J. & Hurley, L. H.启动子区域g -四重结构的直接证据及其与小分子靶向抑制c-MYC转录。gydF4y2Ba国家科学院学报美国gydF4y2Ba99gydF4y2Ba, 11593-11598(2002)。gydF4y2Ba
Maher, L. J. 3rd, Dervan, P. B. & Wold, B.三螺旋DNA复合物在真核细胞无转录系统中启动子特异性抑制的分析。gydF4y2Ba生物化学gydF4y2Ba31gydF4y2Ba, 70-81(1992)。gydF4y2Ba
李志强,李志强,李志强,等。非b DNA序列对真核生物基因表达的影响。gydF4y2Ba物化学。Biophys。Commun >,gydF4y2Ba181gydF4y2Ba, 818-826(1991)。gydF4y2Ba
Waga, S., Mizuno, S. & Yoshida, M.染色体蛋白HMG1消除了超螺旋DNA中十字形引起的转录阻塞。gydF4y2Ba生物。化学。gydF4y2Ba265gydF4y2Ba, 19424-19428(1990)。gydF4y2Ba
Waga, S., Mizuno, S. & Yoshida, M.非组蛋白HMG1消除了超螺旋DNA中左旋z型段引起的转录阻滞。gydF4y2Ba物化学。Biophys。Commun >,gydF4y2Ba153gydF4y2Ba, 334-339(1988)。gydF4y2Ba
Renner, T. M., Belanger, K., Goodwin, L. R., Campbell, M. & Langlois, M. A.所有7种人类APOBEC3蛋白影响LINE-1限制的分子属性的表征。gydF4y2Ba病毒学gydF4y2Ba520gydF4y2Ba, 127-136(2018)。gydF4y2Ba
龙戈,P. A.,卡夫兰,J. M.,金,M. S.和Leahy, D. J.短暂哺乳动物细胞转染聚乙烯亚胺(PEI)。gydF4y2BaEnzymol方法。gydF4y2Ba529gydF4y2Ba, 227-240(2013)。gydF4y2Ba
Cer, R. Z.等。非b数据库v2.0:预测非b dna形成基序及其相关工具的数据库。gydF4y2Ba核酸测定。gydF4y2Ba41gydF4y2Ba, d94-d100(2013)。gydF4y2Ba
Edgar, r.c. MUSCLE:多序列比对,高精度,高通量。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba32gydF4y2Ba, 1792-1797(2004)。gydF4y2Ba
Capella-Gutierrez, S, Silla-Martinez, J. M. & Gabaldon, T. trimAl:大规模系统发育分析中自动校准修剪的工具。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 1972-1973(2009)。gydF4y2Ba
莫里森,C. S.等。乌干达和津巴布韦妇女在急性和早期HIV-1感染期间的血浆和宫颈病毒载量gydF4y2Ba艾滋病gydF4y2Ba24gydF4y2Ba, 573-582(2010)。gydF4y2Ba
确认gydF4y2Ba
女王伊丽莎白二世科学与技术研究生奖学金(QEII- GSST), T.M.R.加拿大卫生研究所(CIHR)运营拨款#159825和加拿大艾滋病毒治疗企业(CanCURE)拨款给m.a.l CIHR运营拨款FRN-150406给S.D.B.gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
m.a.l.和S.D.B.构思了这个项目并撰写了论文。t.m.r., k.b., m.g.和S.D.进行了实验。H.P.K.和M.D.C.进行了深度测序。E.N.和E.J.A.提供了资源。H.O.A, T.M.R, m.a.l.和S.D.B.进行数据分析。所有作者都编辑了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然通讯gydF4y2Ba感谢匿名审稿人对本工作的同行评议所作的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
阿约格,h.o.,雷纳,t.m., Bélanger, K。gydF4y2Baet al。gydF4y2Ba抗逆转录病毒APOBEC3胞苷脱氨酶改变HIV-1前病毒整合位点谱。gydF4y2BaNat CommungydF4y2Ba14gydF4y2Ba, 16(2023)。https://doi.org/10.1038/s41467-022-35379-ygydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41467-022-35379-ygydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba