摘要gydF4y2Ba
构建高质量的单倍型解析基因组组装已经大大提高了检测和表征遗传变异的能力。一种有针对性的方法可以方便地从单倍型解析的基因组集合中获取丰富的信息,这将吸引专注于特定基因组区域的基础研究人员和医学科学家群体。在这里,以4.5兆酶,众所周知难以组装的主要组织相容性复合体(MHC)区域为例,我们展示了一种基于crispr的富集构建目标基因组区域单倍型解析组装的方法。与单倍型解析基因组组装的结果相比,我们的目标方法实现了相当的完整性和准确性,降低了计算复杂性、测序成本以及起始材料的数量。此外,使用目标组合的个人MHC单倍型作为参考,既提高了测序数据的量化准确性,又使MHC区域的等位基因特异性功能基因组学分析成为可能。由于该方法对资源的高效利用,可以极大地促进目标区域的群体遗传研究,并可能为阐明疾病病因学的分子机制开辟新的途径。gydF4y2Ba
简介gydF4y2Ba
最近在构建高质量单倍型解析基因组组装方面的进展大大提高了检测和表征遗传变异的敏感性gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,并将极大地促进我们对人类基因组的理解。当亲子三胞胎信息可用时,三胞胎分箱方法允许为后代个体构建单倍型解析基因组组装gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba.或者,PacBio高保真(HiFi)长读测序,加上链特异性测序技术,提供了另一种选择来组装二倍体基因组,而不需要谱系信息gydF4y2Ba1gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba.然而,这两种实现高质量单倍型解析从头基因组组装的方法都需要大量的起始材料、广泛的计算资源和高水平的测序成本。有针对性的方法提供了从单倍型解析基因组集合中获取丰富信息的便捷途径,这将吸引基础研究人员和医学科学家群体,他们专注于特定基因组区域的功能相关性。gydF4y2Ba
最近,CRISPR/Cas9开始被用作富集目标基因组区域的工具gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba.与基于探针的DNA富集策略相比,该方法只需要知道目标基因组区域侧翼的DNA序列,使其成为对目标区域进行无偏富集的理想工具,即使具有高多态性。此外,它与长读测序技术兼容gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba单倍型解析程序集必需。我们设想,采用这种基于crispr的靶向富集方法可以大大减少实现单倍型解析组装所需的输入和资源。为了证明这种可能性,我们选择了4.5兆酶(Mb),这是出了名的难以组装的主要组织相容性复合体(MHC)区域,其中包含许多直接参与抗原免疫反应的基因,并与许多复杂的人类疾病有关gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba.MHC区域的高度多态性和强烈的连锁不平衡阻碍了对其的研究。实现MHC区域的单倍型解析组装对于无偏倚检测和MHC区域遗传变异的表征至关重要gydF4y2Ba18gydF4y2Ba用于精确的基因组推断gydF4y2Ba19gydF4y2Ba以期最终了解其在疾病病因学中的分子机制。gydF4y2Ba
在这项工作中,通过将基于crispr的富集与10x Genomics的链接读测序和PacBio HiFi长读平台相结合(图2)。gydF4y2Ba1gydF4y2Ba),我们能够从二倍体人类细胞中实现靶向单倍型分解的MHC区域组装。我们还将我们的靶向方法应用于两个具有挑战性的医学相关基因,并获得了单倍型解析的组装体。通过与来自同一细胞系的单倍型解析基因组组装进行比较,我们证明了我们获得单倍型解析组装的靶向方法具有相当的完整性和准确性,能够全面检测目标区域的遗传变异。有了目标组合的个人MHC单倍型作为参考,我们表明,与使用hg38作为参考的标准方法相比,MHC区域DNA甲基化和基因表达的量化要准确得多。最后,我们使用靶向组合的个人MHC单倍型来研究MHC区域的等位基因特异性转录调控。gydF4y2Ba
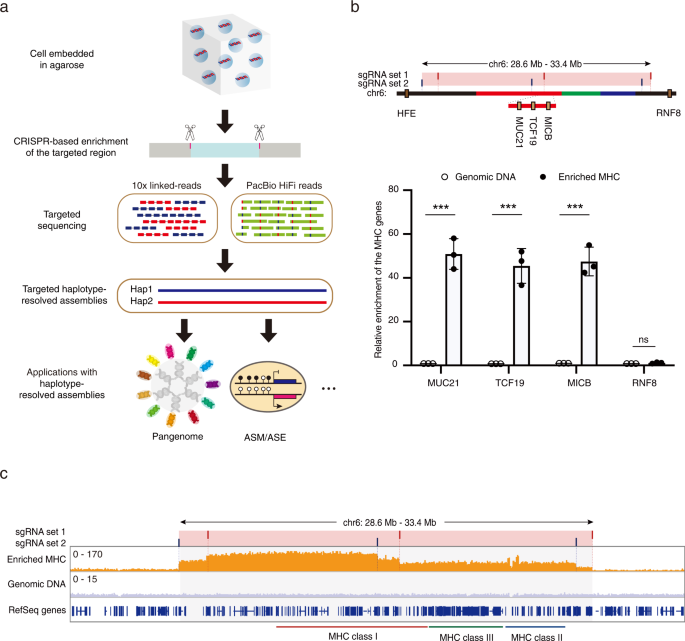
一个gydF4y2Ba基于crispr的靶向单倍型解析目标区域组装示意图。细胞被嵌入琼脂糖塞,然后通过结合基于crispr的凝胶内消化和脉冲场凝胶电泳富集目标区域。对富集的HMW DNA分子进行10x Genomics linked-read和PacBio HiFi long-read测序。一个高度可靠的相相杂合变异集被称为并用于将HiFi读集分离为两个单倍型分割的读集,从中构建目标区域的单倍型分辨组装。目标组合的个人单倍型可进一步用于下游分析。gydF4y2BabgydF4y2BaQPCR分析显示靶区MHC明显富集。顶部:两组针对MHC区域的sgrna的位置。粉色条表示目标MHC区域。红色、绿色和蓝色线分别表示HLA I类、III类和II类基因的区域。暗黄色方框表示用于qPCR分析的基因。基因是根据它们在hg38参考上的坐标排列的,但不是按比例排列的。下半部分:相对富集测定相对富集量gydF4y2BaHFEgydF4y2Ba在目标MHC区域之外的基因(如上图所示),然后归一化到没有经过sgRNA处理的细胞。目标MHC区域之外的另一个基因的相对富集(gydF4y2BaRNF8gydF4y2Ba)也作为阴性对照进行测试。数据用三个独立实验的均值±SEM表示,gydF4y2BaPgydF4y2Ba价值观:gydF4y2BaMUC21 PgydF4y2Ba= 0.000248,gydF4y2BaTCF19 PgydF4y2Ba= 0.000628,gydF4y2BaMICB PgydF4y2Ba= 0.000261,gydF4y2BaRNF8 PgydF4y2Ba= 0.7499 (*** .gydF4y2BaPgydF4y2Ba< 0.001, ns:不显著,学生的gydF4y2BatgydF4y2Ba以及,两面)。源数据作为源数据文件提供。gydF4y2BacgydF4y2Ba基于Illumina短读平台测序的目标MHC区域(灰色区域)的覆盖范围。两组sgrna的目标位点分别用红色和蓝色条表示。gydF4y2Ba
结果gydF4y2Ba
基于crispr的MHC区域靶向富集gydF4y2Ba
我们的研究使用了具有良好特征和广泛使用的人类二倍体细胞系GM12878(也称为HG001)。我们的方法的第一步是通过结合基于crispr的凝胶消化和脉冲场凝胶电泳,从琼脂糖嵌入的完整细胞中分离出靶向的百万酶大小的DNA区域(MHC区域)。gydF4y2Ba1gydF4y2Ba).四个琼脂糖塞,总共等于4 × 10gydF4y2Ba6gydF4y2Ba细胞被用来分离MHC区域。sgrna被设计用于靶向4.7 Mb区域两侧的非多态性DNA序列,已知包括整个MHC位点gydF4y2Ba20.gydF4y2Ba(无花果。gydF4y2Ba1 bgydF4y2Ba).所设计的sgrna的性能最初是通过从目标区域扩增的PCR产物进行体外裂解实验来评估的(补充图。gydF4y2Ba1 bgydF4y2Ba).大多数最终富集的DNA分子仍然是高分子量(HMW),长度大于50kb(补充图。gydF4y2Ba2gydF4y2Ba),因此适用于后续的长读测序。gydF4y2Ba
为了评估富集效率,我们对靶MHC区域的三个基因组位点(chr6: 28903952-33268517)进行了qPCR分析,结果显示,与无sgRNA对照相比,其富集程度超过40倍;注意,mhc侧翼区域未检测到富集(图。gydF4y2Ba1 bgydF4y2Ba).使用Illumina短读平台测序显示,整个靶向MHC区域成功富集(图2)。gydF4y2Ba1 cgydF4y2Ba).值得注意的是,整个2.3 Mb DNA片段的覆盖范围相对相同,但两个片段之间有很大差异(图2)。gydF4y2Ba1 cgydF4y2Ba),说明目标区域的富集依赖于相应sgRNAs的裂解效率。这些结果表明,通过CRISPR/Cas9裂解后的凝胶纯化,我们可以成功富集目标MHC区域。gydF4y2Ba
MHC区域的靶向单倍型解析组装gydF4y2Ba
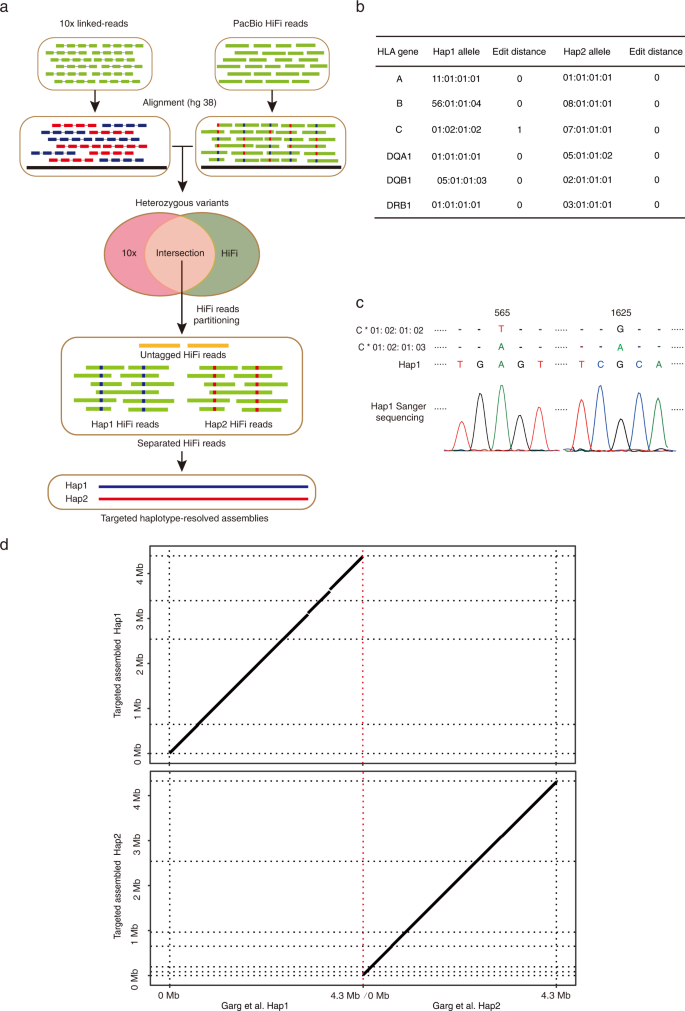
在富集MHC区域后,我们构建了10x基因组公司的HMW MHC分子链接阅读文库。目标MHC区域内的阶段性变异被称为(补充图。gydF4y2Ba3gydF4y2Ba),然后与GM12878细胞的两个基准数据集(GIAB (v.4.2.1))进行比较gydF4y2Ba21gydF4y2BaIllumina白金基因组gydF4y2Ba22gydF4y2Ba).大多数变异的假阴性率(FNRs)低于11%(表2)gydF4y2Ba1gydF4y2Ba).相控精度高,开关错误率和汉明错误率均低于1%(补充表gydF4y2Ba1gydF4y2Ba).我们用HLA- vbseq估计HLA类型gydF4y2Ba23gydF4y2Ba(补充图。gydF4y2Ba3gydF4y2Ba)gydF4y2BaHLAgydF4y2Ba基因,已被发现与许多人类疾病密切相关gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba.除了其中一个gydF4y2BaHLA-CgydF4y2Ba等位基因,所有其他等位基因达到了8位数的分辨率,这比最近基于牛津纳米孔数据生成的全基因组组装的HLA等位基因的预测更好gydF4y2Ba26gydF4y2Ba(补充表gydF4y2Ba2gydF4y2Ba).我们利用我们的10x基因组学链接读取目标MHC区域的数据进一步组装了两个单倍型,并为每个单倍型生成了18个contigs。对于这两种单倍型,NGA50支架尺寸一致超过0.513 Mb,最大contig长度超过1.93 Mb(补充表gydF4y2Ba3.gydF4y2Ba).gydF4y2Ba
尽管10x Genomics的linked-read测序在分阶段变异呼叫方面表现良好,但我们只能组装目标MHC区域的伪单倍型。因此,我们使用PacBio HiFi平台进行了额外的测序。虽然目前部署的PacBio HiFi平台用于基因组组装需要大量的起始材料(例如,来自二倍体人类细胞的基因组DNA超过25µg,以生成全基因组组装所需的至少80 Gb数据),但我们能够生成一个12 kb的HiFi文库,其靶向MHC区域的覆盖率至少为60倍(补充图。gydF4y2Ba3 bgydF4y2Ba)从20ng富集的HMW DNA中提取。在过滤了属于目标MHC区域的HiFi读取后,我们能够仅使用PacBio的HiFi数据生成一个主要的组合,并最终生成目标MHC区域的单倍型解析的组合(补充图)。gydF4y2Ba3 cgydF4y2Ba).该装配结果包含6个单倍型1的contigs和4个单倍型2的contigs,覆盖了这两种单倍型超过96%的靶MHC区域(补充表)gydF4y2Ba4 gydF4y2Ba).gydF4y2Ba
我们再次估计了六种主要的HLA类型gydF4y2BaHLAgydF4y2Ba基因。尽管他们都达到了8位数的分辨率,我们注意到三个HLA I类基因的相位信息是不正确的(补充表gydF4y2Ba5gydF4y2Ba).从一个单一测序平台生成的装配结果表明,当没有三重奏信息时,至少需要两个平台来实现高质量的单倍型解析装配,这与最近的其他发现是一致的gydF4y2Ba12gydF4y2Ba,gydF4y2Ba18gydF4y2Ba.gydF4y2Ba
通过将10x Genomics的链接读数据和HiFi读数据生成的变体交叉,我们能够获得一组高度可靠的相相杂合变体,将HiFi读数据分离为两个单倍型分割的读集。最终,两个MHC单倍型分别从每个单倍型分区的HiFi读集组装,以及未标记的HiFi读集(图2)。gydF4y2Ba2gydF4y2Ba).目标组合的MHC单倍型覆盖了hg38参考的大部分MHC区域(表2)gydF4y2Ba2gydF4y2Ba).我们在组装结果中观察到几个断点(补充表)gydF4y2Ba6gydF4y2Ba),它们似乎是由于缺乏HiFi读取(补充图。gydF4y2Ba4gydF4y2Ba,补充表gydF4y2Ba6gydF4y2Ba)来自偏置放大。gydF4y2Ba
一个gydF4y2BaMHC区域的目标单倍型解析组装原理图,具有10x链接读取数据和PacBio HiFi读取。gydF4y2BabgydF4y2BaHLA分型六大经典gydF4y2BaHLAgydF4y2Ba使用目标单倍型解析组装的基因。编辑距离:我们的组合与IMGT/HLA数据库中报告的HLA等位基因之间不同核苷酸的数量。gydF4y2BacgydF4y2Ba一本小说gydF4y2BaHLA-CgydF4y2BaSanger测序证实该等位基因为单倍型1。不同于最佳匹配的IMGT/HLA等位基因的核苷酸显示。不同核苷酸的位置gydF4y2BaHLA-CgydF4y2Ba表示基因。gydF4y2BadgydF4y2Ba我们的目标组装和之前报道的基因组组装的MHC区域之间的比较gydF4y2Ba8gydF4y2Ba对于每个单倍型。的gydF4y2BaYgydF4y2Ba-axis表示目标程序集的坐标,而gydF4y2BaXgydF4y2Ba-axis为Garg等人的坐标。gydF4y2Ba
我们打了六个主要的gydF4y2BaHLAgydF4y2Ba所有的基因都达到了8位数的分辨率(图2)。gydF4y2Ba2 bgydF4y2Ba).我们注意到1的序列gydF4y2BaHLA-CgydF4y2Ba我们鉴定的等位基因和所有可用的都不一样gydF4y2BaHLA-CgydF4y2BaIMGT/HLA数据库中报告的等位基因。通过Sanger测序,我们确认这是一个新发现gydF4y2BaHLA-CgydF4y2Ba与两个最匹配的IMGT/HLA等位基因有一个核苷酸差异的等位基因(gydF4y2BaC * 01:02:01:02gydF4y2Ba而且gydF4y2BaC * 01:02:01:03gydF4y2Ba)(图。gydF4y2Ba2摄氏度gydF4y2Ba).这些结果表明,利用我们针对MHC区域的单倍型分解组装,可以高精度地分型HLA等位基因。gydF4y2Ba
评估MHC区域的靶向单倍型分解组装gydF4y2Ba
为了评估我们的组装结果的准确性,通过将相阶段的contigs对准hg38参考,然后将其与GIAB (v.4.2.1)和Illumina Platinum基因组的基准进行比较,从而调用变体。我们在目标MHC区域内鉴定出30,100个FNRs低于2.8%的遗传变异(表gydF4y2Ba1gydF4y2Ba).使用来自高置信区域的床文件对变量进行评估,结果显示F1得分(代表精密度和召回率的调和平均值)均高于0.89(补充表)gydF4y2Ba7gydF4y2Ba).所有变体的开关错误率和汉明错误率均低于0.45%(补充表gydF4y2Ba1gydF4y2Ba),证实了相位的高质量。最近,一个染色体尺度的高质量单倍型解析组装GM12878gydF4y2Ba8gydF4y2Ba使用PacBio HiFi和Hi-C数据进行了报道。值得注意的是,我们针对MHC区域的单倍型分解组装实现了与Garg等人相当的一致准确性gydF4y2Ba1gydF4y2Ba、补充表格gydF4y2Ba1gydF4y2Ba而且gydF4y2Ba7gydF4y2Ba).gydF4y2Ba
相比之下,我们还评估了仅由PacBio HiFi数据生成的单倍型解析组装结果。与GIAB (v.4.2.1)和Illumina Platinum基因组的基准相比,共鉴定出29721个FNRs低于3%的遗传变异(表2)gydF4y2Ba1gydF4y2Ba).而开关错误率则低于0.5%(补充表gydF4y2Ba1gydF4y2Ba),有相当数量的杂合变异被称为相位信息不准确(表gydF4y2Ba1gydF4y2Ba),且汉明错误率高于45%(补充表gydF4y2Ba1gydF4y2Ba).这与三个HLA II类基因的相位信息不正确的结果是一致的(补充表gydF4y2Ba5gydF4y2Ba).事实上,这是在目标MHC区域中间的单一开关错误的结果(补充图。gydF4y2Ba3 dgydF4y2Ba).因此,这表明,即使我们仅通过使用PacBio HiFi数据就能够以相对较好的质量生成目标MHC区域的单倍型解析装配,但通过结合另一种链特异性测序技术(如10x Genomics的链读技术),仍然可以提高目标装配的完整性和相位精度。gydF4y2Ba
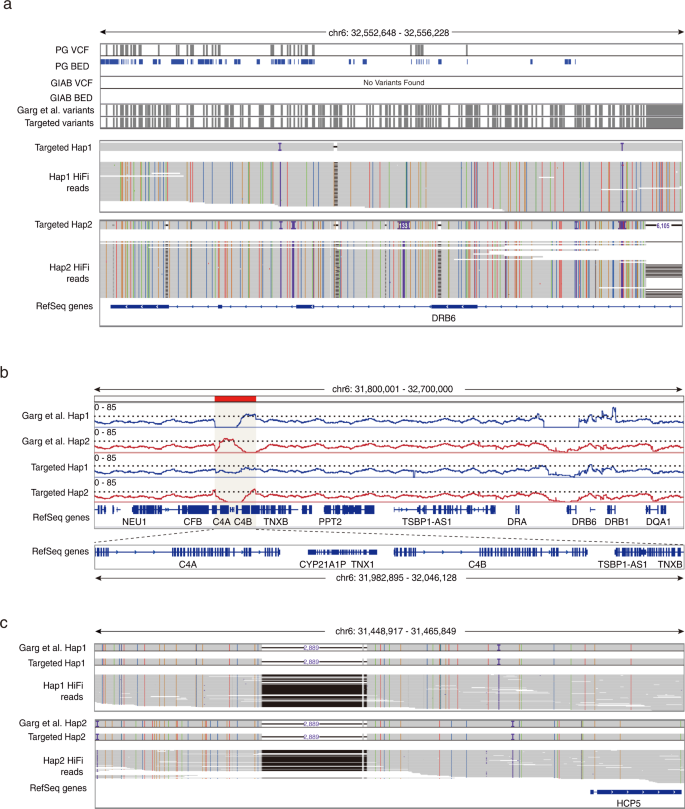
为了进一步评估我们的靶向方法与全基因组组装方法的性能,我们使用10x Genomics的链接读取数据和PacBio HiFi读取将我们的靶向单倍型解析组装与Garg等人进行了比较,并观察到整个目标区域的高度一致性(图2)。gydF4y2Ba二维gydF4y2Ba,补充图。gydF4y2Ba4 bgydF4y2Ba).与Garg等人类似,与GIAB (v.4.2.1)和Illumina Platinum基因组数据集相比,我们分别鉴定出19.3%和29.0%的新遗传变异(表2)gydF4y2Ba1gydF4y2Ba).通过对其中一些新鉴定的变异进行人工检测,这些变异大多位于高度多态或重复区域,并通过识别支持的高置信度HiFi reads来确认其准确性(图。gydF4y2Ba3gydF4y2Ba).为了评估传统上被折叠或排除的具有重复内容的复杂区域的装配精度,我们通过分析整个目标MHC区域的原始HiFi读覆盖来估计装配结果中折叠的碱基对的数量。与全基因组组装结果相比,我们的靶向方法对两种组装的单倍型产生了相似或更少的折叠重复(补充图。gydF4y2Ba4摄氏度gydF4y2Ba、补充数据gydF4y2Ba1gydF4y2Ba).重要基因片段复制区进一步评价gydF4y2BaC4A / BgydF4y2Ba,gydF4y2BaTNXA / B,gydF4y2Ba而且gydF4y2BaCYP21A2gydF4y2BaGarg等人在单倍型1的这一区域观察到20 kb的折叠重复,而我们的目标方法中没有折叠重复的证据(图2)。gydF4y2Ba3 bgydF4y2Ba、补充数据gydF4y2Ba1gydF4y2Ba).与此一致的是,当将两个基准的组装结果与该区域的床层文件进行比较时,F1得分高于我们的目标结果的0.83,而Garg等人的F1得分低于0.6gydF4y2Ba2gydF4y2Ba).所有这些结果都表明,我们的靶向方法可以达到与全基因组组装结果相当的高准确度。gydF4y2Ba
一个gydF4y2Ba一个新发现变异的地区的例子。具有新变体的高置信度HiFi读取如下所示。在单倍型1和HiFi读取的目标组装结果中定位的彩色线和点表明了四种不同于hg38参考的核苷酸。紫色字符“I”和圆点表示与hg38参考对照相比有小插入(<50 bp)。与hg38对照相比,黑点表示小的缺失(<50 bp)。加兰:Illumina白金基因组。gydF4y2BabgydF4y2Ba程序集中的折叠区域。蓝色和红色线表示从GIAB下载的GM12878细胞的PacBio HiFi读取的覆盖范围,该GIAB与不同组件的单倍型1或单倍型2对齐。虚线表示6号染色体预期覆盖的阈值(平均值+至少三个标准差),不包括坍塌区域的目标MHC区域。红色条表示补充数据中识别的折叠区域gydF4y2Ba1gydF4y2Ba.gydF4y2BacgydF4y2Ba上游2889 bp的缺失gydF4y2BaHCP5gydF4y2Ba两种单倍型的HiFi reads均显示并支持该基因。gydF4y2Ba
有了目标单倍型解析集,我们能够识别大的插入和删除,这是使用短读测序很难发现的。例如,我们确定了上游2889 bp的缺失gydF4y2BaHCP5gydF4y2Ba基因(图。gydF4y2Ba3 cgydF4y2Ba)和具有两个插入的区域(344和787 bp)(补充图。gydF4y2Ba4 dgydF4y2Ba),两者都得到了高保真长读的支持,并在Garg等人中观察到。据报道,基于组装的方法能够更准确和全面地描述遗传变异gydF4y2Ba1gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,并通过与单倍型解析的基因组组装进行比较,我们的结果表明,该靶向方法在目标区域遗传变异检测上具有相当的完整性和准确性。gydF4y2Ba
精确的功能基因组学分析与目标单倍型的决议汇编gydF4y2Ba
据报道,MHC位点的最多态部分位于三个HLA I类基因(gydF4y2Ba抗原gydF4y2Ba,gydF4y2BaHLA-BgydF4y2Ba,gydF4y2BaHLA-CgydF4y2Ba)和三个HLA II类基因(gydF4y2BaHLA-DRgydF4y2Ba,gydF4y2Bahla dqgydF4y2Ba,gydF4y2BaHLA-DPgydF4y2Ba)gydF4y2Ba24gydF4y2Ba,gydF4y2Ba27gydF4y2Ba.一致地,我们的目标单倍型解析组装表明,变异的主要峰值在这六个经典的基因组区域附近gydF4y2BaHLAgydF4y2Ba基因(补充图。gydF4y2Ba5gydF4y2Ba).而MHC区域内的高水平多态性对于面临各种病原体环境的人群是有益的gydF4y2Ba28gydF4y2Ba例如,携带许多多态性和结构变异的短测序reads将不能正确地与参考基因组对齐,导致结果模糊甚至不准确gydF4y2Ba19gydF4y2Ba,gydF4y2Ba29gydF4y2Ba.gydF4y2Ba
有人提出,使用个人基因组作为参考是一种解决方案,以处理与校准相关的伪影的基因表达gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2BaDNA甲基化分析gydF4y2Ba32gydF4y2Ba.据此,我们将hg38参考文献替换为个人基因组参考文献,其中两个目标组装的个人MHC单倍型分别与hg38的非MHC区域结合。然后将短读测序数据分别对齐到每个个人基因组参考,以限制遗传变异对序列对齐的影响(补充图。gydF4y2Ba5 bgydF4y2Ba).与之前的发现一致gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba,以hg38为参考,对GM12878细胞产生的RNA-Seq数据进行序列比对,测序reads的数量映射到含有许多变体的外显子上(补充图中灰色阴影突出的区域)。gydF4y2Ba5度gydF4y2Ba)的表达水平远低于同一基因的其他外显子gydF4y2BaHLA-BgydF4y2Ba基因。然而,当我们使用个人基因组作为参考时,不良排列的效果显著改善(补充图。gydF4y2Ba5度gydF4y2Ba).这突出了利用靶向单倍型解析组装对具有高多态性基因的基因表达进行精确量化的重要性。gydF4y2Ba
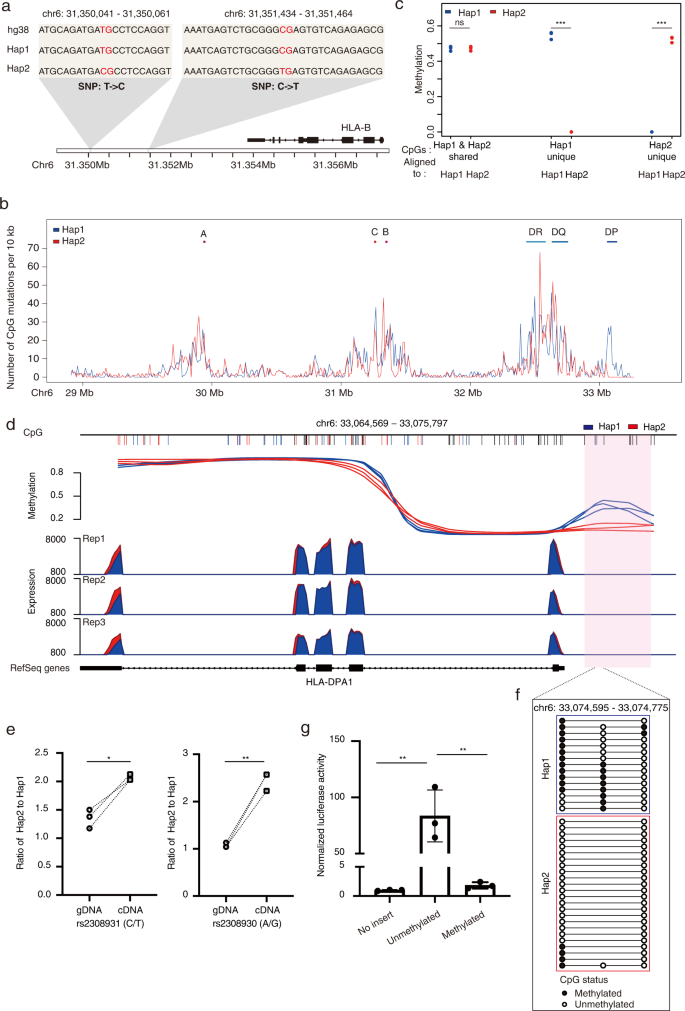
这在DNA甲基化的研究中变得更加复杂。众所周知,甲基化的胞嘧啶易于自发脱氨,导致最常见的二核苷酸变体(CGgydF4y2Ba→gydF4y2Ba在哺乳动物基因组中的TG)gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba.然而,量化亚硫酸氢盐转换后每个CpG位点DNA甲基化水平的金标准是计算用CG(表示亚硫酸氢盐受甲基保护)测序的reads数除以用TG(表示亚硫酸氢盐不含甲基转换)测序的reads数。在这种情况下,CpG位点上的C/T变异将不可避免地导致错误的甲基化量化。例如,在位于该基因下游的目标组装单倍型2中观察到一个特定的TG-to-CG变体gydF4y2BaHLA-BgydF4y2Ba基因(图中左边灰色方框。gydF4y2Ba4gydF4y2Ba).对于这个位置,如果测序读数与hg38参考值一致,则它们将从DNA甲基化评估中遗漏。另外,在单倍型2中观察到另一种CG-to-TG变异(图中右侧灰框)。gydF4y2Ba4gydF4y2Ba)将产生未甲基化的调用,而不会引起任何比对错配,并导致在单倍型2中实际上没有CpG位点的这个位置上有超过0%的甲基化“位点”。gydF4y2Ba
一个gydF4y2BaCpG站点上C/T变体的例子。gydF4y2BabgydF4y2Ba整个目标MHC区域的CpG变异密度图。三个MHC I类基因和三个MHC II类基因的位置显示在顶部。gydF4y2BacgydF4y2BaCpG变异对DNA甲基化分析的影响。数据来自三个独立的实验,gydF4y2BaPgydF4y2Ba价值观:共享gydF4y2BaPgydF4y2Ba= 0.851, Hap1唯一gydF4y2BaPgydF4y2Ba= 0.000496, Hap2唯一gydF4y2BaPgydF4y2Ba= 0.000318 (***gydF4y2BaPgydF4y2Ba< 0.001, ns:不显著,配对的学生gydF4y2BatgydF4y2Ba以及,两面)。gydF4y2BadgydF4y2Ba等位基因特异性转录调控gydF4y2BaHLA-DPA1gydF4y2Ba基因。上半部分:基因启动子区域的等位基因特异性甲基化gydF4y2BaHLA-DPA1gydF4y2Ba基因。两个单倍型之间的差异甲基化区域(DMR-DPA1)以粉红色条表示。两个单倍型之间共享的cpg以黑色短线表示,而单倍型特定的cpg则以蓝色或红色短线表示。的gydF4y2BaYgydF4y2Ba-axis分别表示每个单倍型的DNA甲基化水平。显示了三个重复。底部部分:等位基因特异性表达gydF4y2BaHLA-DPA1gydF4y2Ba基因。单倍型1和2的表达水平分别用蓝色和红色表示。显示了三个重复。gydF4y2BaegydF4y2Ba外显子内两个多态位置的焦磷酸测序gydF4y2BaHLA-DPA1gydF4y2BaGM12878细胞的基因。gydF4y2BaYgydF4y2Ba-axis为目标snp在两个单倍型之间的比例。数据来自三个独立的实验,gydF4y2BaPgydF4y2Ba值:rs2308931 (C/T)gydF4y2BaPgydF4y2Ba= 0.0177, rs2308930 (A/G)gydF4y2BaPgydF4y2Ba= 0.0056 (*0.01
我们评估了两个目标组合MHC单倍型和hg38参考中的CpG位点,发现2.38%(1355/57,010)的CpG在单倍型1中特异,2.14%(1191/55,659)的CpG在单倍型2中特异。然而,在hg38参考文献中,有3.91%(2291/58,570)的cpg在任何一种单倍型中都不存在。gydF4y2Ba6gydF4y2Ba).当描述这些CpG变体(相对于hg38的单倍型特异性的CpG增益和损失)在目标MHC区域的分布时,我们观察到主要的峰值出现在六个经典的基因组区域周围gydF4y2BaHLAgydF4y2Ba基因(图。gydF4y2Ba4 bgydF4y2Ba),与遗传变异产生的密度图相似(补充图。gydF4y2Ba5gydF4y2Ba).当我们分别对单倍型特异性或共享的CpG进行DNA甲基化分析时,由CpG变体引起的问题效应是明显的。正如预期的那样,当使用相应的目标组合个人MHC单倍型作为参考计算单倍型特异性CpGs的甲基化水平时,观察到了极端的差异,而共享CpGs没有观察到这种差异(图2)。gydF4y2Ba4摄氏度gydF4y2Ba).随着MHC区域的靶向单倍型分解组装的存在,通过将亚硫酸氢盐测序数据对准个人基因组作为参考,可以很容易地解决这种偏差(补充图。gydF4y2Ba5 bgydF4y2Ba),这与之前在分析两个高度不同的小鼠品系的甲基化数据时所显示的结果相似gydF4y2Ba32gydF4y2Ba.gydF4y2Ba
DNA甲基化阵列也观察到甲基化量化的偏倚,反映出跨越MHC区域的高水平多态性将影响snp相关探针的杂交行为gydF4y2Ba35gydF4y2Ba.我们观察到相关系数(gydF4y2BaRgydF4y2Ba2gydF4y2Ba对于阵列探针中没有snp的CpGs, Illumina甲基化EPIC串珠芯片和亚硫酸氢盐测序数据之间的DNA甲基化为0.96(补充图。gydF4y2Ba6 bgydF4y2Ba),而含有SNPs的探针的相关系数下降到0.9(补充图。gydF4y2Ba6摄氏度gydF4y2Ba).这些结果共同强调了使用目标组合的个人单倍型作为DNA甲基化和基因表达数据精确量化的参考的实用性,特别是对于高度多态区域,如MHC。gydF4y2Ba
等位基因特异性分析与目标单倍型解析汇编gydF4y2Ba
有了目标组合的MHC单倍型,我们进一步表征了二倍体GM12878细胞的等位基因特异性甲基化和表达。我们首先使用两个个人基因组作为参考分析了等位基因特异性表达(ASE),并确定了靶MHC区域内的7个基因表现出等位基因特异性表达(调整后)gydF4y2BaPgydF4y2Ba值≤0.05)(补充图gydF4y2Ba7一个gydF4y2Ba及补充资料gydF4y2Ba3.gydF4y2Ba).的表达式gydF4y2BaHLA-DPA1gydF4y2Ba基因,在两种单倍型之间有1.1倍的差异,我们观察到所有单倍型2外显子的高表达一致gydF4y2BaDPA1gydF4y2Ba(无花果。gydF4y2Ba4 dgydF4y2Ba).利用焦磷酸测序对ASE进行了验证。gydF4y2Ba4 egydF4y2Ba)、等位基因特异性qRT-PCR (gydF4y2BaPgydF4y2Bavalue = 0.0074,配对的学生gydF4y2BatgydF4y2Ba-test,双面)(补充图。gydF4y2Ba7 bgydF4y2Ba)及扩增PCR克隆的Sanger测序(gydF4y2BaPgydF4y2Bavalue = 0.0068, Fisher精确检验)(补充图。gydF4y2Ba7 cgydF4y2Ba的单倍型gydF4y2BaDPA1gydF4y2Ba基因在GM12878细胞中较单倍型1显著过表达。事实上,这个表达式gydF4y2BaDPA1gydF4y2Ba单倍型2基因(gydF4y2BaDPA1 * 01:03:01:02gydF4y2Ba)高于单倍型1 (gydF4y2BaDPA1 * 02:01:01:02gydF4y2Ba)在GM12878细胞中的表达与最近的一项发现相一致gydF4y2BaDPA1 * 01gydF4y2Ba等位基因显著高于等位基因gydF4y2BaDPA1 * 02gydF4y2Ba等位基因gydF4y2Ba36gydF4y2Ba.gydF4y2Ba
然后,我们分析了目标MHC区域的等位基因特异性甲基化(ASM),并在两种单倍型之间发现了211个差异甲基化区域(DMRs)gydF4y2Ba4 gydF4y2Ba).我们在甲基化分析中加入了单倍型特异性CpGs,因为这将增加差异分析的准确性和功率gydF4y2Ba32gydF4y2Ba(补充图。gydF4y2Ba7 dgydF4y2Ba).我们注意到,在启动子区域gydF4y2BaHLA-DRA1gydF4y2Ba基因中,存在一个差异甲基化区域(下文称为DMR-DPA1),其中单倍型1高甲基化(图1)。gydF4y2Ba4 dgydF4y2Ba).DMR-DPA1包含一个单倍型1特异性的CpG位点和几个共享的CpG位点,DMR-DPA1的等位基因特异性甲基化通过亚硫酸氢盐Sanger测序得到验证(图2)。gydF4y2Ba4 fgydF4y2Ba).gydF4y2Ba
研究DMR-DPA1的ASM是否对酶的ASE有调控作用gydF4y2BaDPA1gydF4y2Ba基因,我们对HEK293T细胞进行了基于细胞的双荧光素酶报告试验,并检测到当未甲基化的DMR-DPA1存在时,基因表达显著增加。相反,当荧光素酶报告基因上的DMR-DPA1在体外甲基化时,这种基因表达的上调被取消(图2)。gydF4y2Ba4 ggydF4y2Ba).这表明gydF4y2BaHLA-DPA1gydF4y2Ba基因可以通过ASM对基因的启动子区进行负调控gydF4y2BaHLA-DPA1gydF4y2Ba.这些结果表明,目标组合的个人单倍型能够在二倍体人类细胞中进行等位基因特异性的功能基因组学分析,并有助于我们理解目标基因组区域的等位基因特异性转录调控。gydF4y2Ba
具有挑战性的医学相关基因的靶向组装gydF4y2Ba
最近,GIAB联盟表明,他们从一个单倍型解析的全基因组组装中,解决并管理了395个具有挑战性的医学相关常染色体基因中的273个的变异基准gydF4y2Ba2gydF4y2Ba.为了扩展我们的靶向方法的应用,我们将我们的方法应用于一个giab分辨基因(gydF4y2BaRHCEgydF4y2Ba)和一个giab未解基因(gydF4y2BaCR1gydF4y2Ba).的基因gydF4y2BaRHCEgydF4y2Ba是Rh血型抗原的一部分gydF4y2Ba37gydF4y2BaSVs和复杂的变体。gydF4y2BaCR1gydF4y2Ba是一种与阿尔茨海默病有关的基因吗gydF4y2Ba38gydF4y2Ba在HG002细胞中含有18.5 kb与hg38相关的纯合缺失。通过CRISPR/Cas9裂解后凝胶纯化,从GM12878细胞中分离出这两个基因区域,或单独或一起(补充图)。gydF4y2Ba8gydF4y2Ba,gydF4y2BabgydF4y2Ba).qPCR分析(补充图;gydF4y2Ba8 c, dgydF4y2Ba)并用Illumina短读平台测序(补充图。gydF4y2Ba8 egydF4y2Ba)表明两个目标区域都成功富集。gydF4y2Ba
接下来,我们要么只使用PacBio HiFi reads,要么将其与10x Genomics的链接reads结合,以组装两个目标区域。除了只有hifi的组件gydF4y2BaCR1gydF4y2Ba基因,两种组装策略都产生了两个目标基因的单倍型解析组装,每个单倍型只有1个contig(补充表gydF4y2Ba8gydF4y2Ba).我们通过将其与两个基准进行比较,评估了来自整个目标区域组装的所谓变体的准确性,并观察到,对于这两个基因,两种组装策略都可以识别FNR低于7%的大多数遗传变体(gydF4y2BaRHCEgydF4y2Ba)及3% (gydF4y2BaCR1gydF4y2Ba)(补充资料gydF4y2Ba5gydF4y2Ba).因为挑战了医学相关领域gydF4y2BaRHCEgydF4y2Ba而且gydF4y2BaCR1gydF4y2Ba在两个基准测试中,基因都没有很好地解析,我们发现了许多新的变异,导致fdr水平很高(超过40%)(补充数据)gydF4y2Ba5gydF4y2Ba).比较来自两个基准的床文件也显示了被称为变体的高准确性(补充数据gydF4y2Ba6gydF4y2Ba).两种组件的开关误差和汉明误差都很低,只有hifi组件的开关误差和汉明误差较低gydF4y2BaCR1gydF4y2Ba基因,汉明误差较高(补充资料gydF4y2Ba5gydF4y2Ba).gydF4y2Ba
对于这两个gydF4y2BaRHCEgydF4y2Ba而且gydF4y2BaCR1gydF4y2Ba使用10x Genomics的linked-read数据和PacBio HiFi读取的组装结果被PacBio HiFi读取在目标区域的存在所支持(补充图。gydF4y2Ba9gydF4y2Ba),并在组件与hg38参考的对齐中出现一些中断(补充图。gydF4y2Ba9gydF4y2Ba).最近,发布了minimap2的新版本(v2.24),它改进了围绕长且排列不好的区域的对齐策略。为了区分这些断裂是由于组件断裂还是由于对准断裂,我们进一步比较了不同版本的minimap2的对准结果(补充图。gydF4y2Ba10gydF4y2Ba).而没有观察到变化gydF4y2BaRHCEgydF4y2Ba基因上,在单倍型1中发现了两个较大的缺失(17,095和18,555 bp)gydF4y2BaCR1gydF4y2Ba使用minimap2 (v2.24)的最新版本(补充图;gydF4y2Ba10gydF4y2Ba).所有这些结果都表明,与单倍型解析的全基因组组装相似,我们的靶向方法可以用于解析和表征其他基因组复杂区域的遗传变异。gydF4y2Ba
讨论gydF4y2Ba
最近在构建高质量单倍型解析基因组组装方面的进展使得在染色体尺度上全面发现遗传变异成为可能。然而,这种全基因组方法很难应用于大群体个体,甚至在不同人群中充分表征遗传变异,以揭示其潜在的功能相关性。在本研究中,我们以临床重要的MHC区域和两个具有挑战性的医学相关基因为例,将基于crispr的富集与10x Genomics的链读测序和PacBio HiFi平台相结合,成功组装了没有亲子三胞胎信息的目标单倍型。通过与单倍型解析基因组组装相比,我们获得单倍型解析组装的目标方法实现了相当的完整性和准确性,降低了计算复杂性、测序成本以及起始材料的数量(补充表)gydF4y2Ba9gydF4y2Ba而且gydF4y2Ba10gydF4y2Ba).gydF4y2Ba
尽管全基因组关联研究(GWAS)极大地促进了我们对人类基因组的理解,但对于具有高多态性和强连锁不平衡的遗传区域(例如MHC区域),确定涉及疾病病因学的因果变异仍然具有挑战性。有人认为,基于单倍型的方法可以克服这些挑战,实现更准确的基因组推断gydF4y2Ba19gydF4y2Ba.的单倍型就是一个例子gydF4y2BaHLA-DRB1-HLA-DQA1-HLA-DQB1gydF4y2Ba大大增加了患1型糖尿病的风险gydF4y2Ba39gydF4y2Ba.另一个例子是补体成分不同等位基因的鉴定4 (gydF4y2BaC4gydF4y2Ba) MHC区域内的基因与精神分裂症的发展有关gydF4y2Ba40gydF4y2Ba.尽管我们只使用GM12878细胞演示了我们的靶向方法,但已经表明,基于crispr的HMW DNA分子富集策略可以类似地应用于原发组织,包括外周血单个核细胞gydF4y2Ba11gydF4y2Ba还有乳腺肿瘤gydF4y2Ba13gydF4y2Ba.考虑到MHC区域是人类基因组中最难组装的区域之一gydF4y2Ba18gydF4y2Ba我们的目标方法可以(理论上)用于基于单倍型的群体遗传研究,以分析任何其他复杂的基因组区域,如MHC。gydF4y2Ba
一个可能使采用我们的靶向方法的研究复杂化的问题是,设计sgrna的富集策略依赖于对目标区域及其侧翼序列的理解。如果目标区域被复制、倒置或删除,这将改变富集DNA分子的分子量,使其难以从脉冲场凝胶中恢复。此外,即使使用Hifiasm从PacBio HiFi reads生成目标单倍型解析组装独立于参考基因组,为了过滤对准目标区域的HiFi reads,仍然需要参考序列。gydF4y2Ba
除了遗传变异,其他复杂基因组区域的功能分析也可以从我们构建目标区域的单倍型分辨组合的无偏倚方法中获益良多。对于具有高多态性的区域,如MHC,将短读测序数据映射到单个参考基因组(例如hg19或hg38)的传统策略已知会产生偏误的比对,导致不准确的量化gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba.这促使最近尝试用计算推断的个人基因型作为参考来取代单个参考基因组,所有这些都表明与标准方法相比,量化的准确性有所提高gydF4y2Ba36gydF4y2Ba,gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba.然而,这些计算策略高度依赖于我们对不同人类基因组的理解的完整性,即使对于被广泛研究的经典来说,这仍然远远没有饱和gydF4y2BaHLAgydF4y2Ba基因gydF4y2Ba47gydF4y2Ba.在这里,以靶向组装的个人MHC单倍型为例,我们不仅表明了MHC区域功能基因组学分析的结果更加准确,而且还揭示了MHC区域的等位基因特异性转录调控gydF4y2BaHLA-DPA1gydF4y2Ba基因通过荧光素酶测定。由于荧光素酶检测是在HEK293T细胞中进行的,因此需要进一步的实验来证实这种模式是生物学的,并且在其他类型的细胞中也适用。gydF4y2Ba
话虽如此,用短读测序数据准确识别ASE和ASM事件是相当具有挑战性的。尽管我们的方法可以限制由遗传变异引起的比对偏差的影响,但其他挑战,如测序读取与多个基因组位置或两个个人基因组参照对齐,仍有待解决。然而,可以有把握地假设,其他具有挑战性的区域(如MHC)的其他功能基因组学分析将受益于我们使用目标区域的单倍型解析组装作为个人参考的方法。这种遗传和表观遗传数据的综合分析可能为全面阐明疾病病因学的分子机制开辟新的途径。gydF4y2Ba
方法gydF4y2Ba
琼脂糖塞的制备gydF4y2Ba
从GM12878细胞中制备兆酶大小的MHC DNA分子或两个具有挑战性的医学相关常染色体基因基于前面描述的方法gydF4y2Ba48gydF4y2Ba与修改。简单地说,收获的GM12878细胞在冰冷的PBS中洗涤三次,重悬至最终浓度~2 × 10gydF4y2Ba7gydF4y2BaL缓冲液(0.1 M EDTA (pH 8.0);0.01 M Tris-Cl (pH 7.6);0.02 M NaCl), 42°C孵育5 min。将1.2%低熔点(LMP)琼脂糖在70°C熔化,然后在42°C孵育5分钟,并与细胞悬液1:1混合。将混合物立即分配到塞模中(1毫升细胞-琼脂糖混合物可填满模具中的10个孔),塞在4°C下孵育直到凝固。将固体琼脂糖塞转移到含有0.5 mg/ml蛋白酶K和1% (w/v) sarkozy的L缓冲液中,在50°C下孵育3小时。将原消化缓冲液替换为2卷新鲜消化缓冲液,在50°C下继续孵育12-16小时。在50体积TE缓冲液(10 mM Tris, pH 8, 1 mM EDTA)中冲洗三次,持续3小时,然后在室温下与2体积含有40 μg/ml PMSF的TE孵育1小时,然后在50℃下与相同的缓冲液孵育30分钟。接下来,用TE缓冲液在3小时内清洗塞3次,用于Cas9消化或储存在TE缓冲液中4°C以备将来使用。gydF4y2Ba
生成sgrnagydF4y2Ba
两组具有20 bp序列(20-mers)的sgRNAs,来自目标MHC区域两侧的非多态基因组区域(set1: chr6: 28.90-33.46 Mb;set2: chr6: 28.58-33.27 Mb)gydF4y2Ba1 bgydF4y2Ba),以及sgRNAs靶向gydF4y2BaRHCEgydF4y2Ba(chr1: 25.40-25.50 Mb)和gydF4y2BaCR1gydF4y2Ba(chr1: 207.47-207.79 Mb)基因,使用CRISPRdirect (gydF4y2Bahttps://crispr.dbcls.jpgydF4y2Ba)(补充资料gydF4y2Ba7gydF4y2Ba).使用EnGen sgRNA试剂盒(NEB)体外转录sgRNA,并使用RNA Clean & Concentrator™-25试剂盒(Zymo Research)进行纯化。纯化的sgrna通过NanoDrop(赛默飞世尔科学公司)进行定量。gydF4y2Ba
基于crispr的目标区域消化gydF4y2Ba
含GM12878细胞的琼脂糖塞体外消化gydF4y2Ba链球菌gydF4y2BaCas9核酸酶(NEB)基于前面描述的方法gydF4y2Ba49gydF4y2Ba与修改。简单地说,在消化之前,将4pmol Cas9酶与150ng sgRNAs、6 μl 10× Cas9缓冲液、1u /μl RNasin和无核酸酶水混合至最终体积为60 μl,在37℃孵育15 min,预组装sgRNAs和Cas9酶。每种预组装Cas9-sgRNA复合物的裂解效率在30 μl反应体积中进行评估,反应体积中包含30 nM的总sgRNA、30 nM的Cas9酶和3 nM的含有Cas9消化位点的PCR扩增DNA片段。琼脂糖塞在10mm Tris-HCl (pH 8.0)中洗涤三次,然后在1× Cas9缓冲液中培养2小时。4个琼脂糖栓用于MHC分离(~20 μg基因组DNA), 2个琼脂糖栓用于医学相关基因(gydF4y2BaRHCEgydF4y2Ba而且gydF4y2BaCR1gydF4y2Ba) (~10 μg基因组DNA)。将栓子分成两组,每组40 μl,用一组60 μl预组装的Cas9-sgRNA混合物在37℃消化2 h。将反应混合物替换为含有0.5 mg/ml蛋白酶K和1% (w/v) sarkozy的L缓冲液,然后在4°C下轻轻摇晃至少1小时,反应结束。gydF4y2Ba
脉冲场凝胶电泳(PFGE)靶向富集MHC区域gydF4y2Ba
经过Cas9消化,琼脂糖堵塞,一起gydF4y2Bah . wingeigydF4y2BaCHEF DNA大小标记(Bio-Rad),直接安装在凝胶梳的底边,并加入180毫升用1× TAE (Bio-Rad)制备的0.8%兆酶分辨率凝胶中。所有PFGE均在Bio-Rad CHEF-DR III系统上进行,角度为106°,电压为3 V/cm,持续48小时,固定切换时间为500秒。用3× Gel-Green染色法在0.1 M NaCl中在室温下染色30分钟。从凝胶中剪出约2.3 Mb对应的条带,放入4% LMP琼脂糖中1× TAE,再次用PFGE以106°角、3 V/cm分离17 h,固定开关时间为500 s。用3× Gel-Green染色法对LMP凝胶在0.1 M NaCl环境下室温下染色30分钟,从LMP凝胶中切割含HMW DNA的条带进行回收。gydF4y2Ba
目标富集gydF4y2BaRHCEgydF4y2Ba而且gydF4y2BaCR1gydF4y2BaPFGE区域gydF4y2Ba
Cas9消化后,琼脂糖栓和lambda DNA (NEB)作为标记直接安装在凝胶梳的底边,并加入0.5× TBE (Sangon)制备的180 ml 1%凝胶中。所有PFGE均在Bio-Rad CHEF-DR III系统上进行,分离距离为20 - 500kb。用3× Gel-Green染色法在0.1 M NaCl中在室温下染色30分钟。对应于~ 100kb的波段(gydF4y2BaRHCEgydF4y2Ba)及~ 300kb (gydF4y2BaCR1gydF4y2Ba)从凝胶中取出,放入4% LMP琼脂糖0.5× TBE中,再用PFGE在20-500 kb自动模式下分离4 h。用3× Gel-Green染色法对LMP凝胶在0.1 M NaCl环境下室温下染色30分钟,从LMP凝胶中切割含HMW DNA的条带进行回收。gydF4y2Ba
目标HMW DNA分子的恢复gydF4y2Ba
回收的LMP凝胶在70°C的热块中熔化10分钟,然后在42°C孵育5分钟。将4u的琼脂酶(赛默飞世尔科技公司)加入100mg (~100 μl) 4% LMP熔融琼脂糖中,轻轻混合,42°C孵育45分钟,使琼脂糖消化成HMW DNA溶液。同时,将10 ml TE缓冲液放入6 cm的培养皿中,将0.1 μm透析膜(Millipore)漂浮在TE缓冲液表面15 min,使膜水化。将高分子量DNA溶液单滴加入宽孔尖端透析膜中心,室温透析50 min。然后将透析后的DNA转移到宽口径的1.5 ml试管中,并通过量子比特DNA高灵敏度测定(赛默飞世尔科学公司)进行定量。采用定量实时聚合酶链反应(qPCR),以补充资料中所列引物序列确定目标区域是否在多个位置富集gydF4y2Ba7gydF4y2Ba.gydF4y2Ba
图书馆建设与排序gydF4y2Ba
采用DNeasy Blood & Tissue Kit (Qiagen)提取GM12878细胞基因组DNA。使用Ultra II DNA文库准备试剂盒(NEB)构建基因组短读或靶向富集DNA的下一代测序文库,并根据制造商的说明在Illumina Nova Novaseq 6000平台上测序。gydF4y2Ba
10x Genomics平台的测序文库是使用经过修改的Chromium Genome Library & Gel Bead Kit v2 (10x Genomics)构建的。简单地说,为了减少条形码碰撞,将目标富集的HMW DNA分子与lambda DNA (NEB) 1:1混合,总共200 pg的混合DNA用于液滴生成。取30 μl生成的液滴,按厂家说明书进行扩增和后续文库构建。根据制造商的说明,该文库在Illumina Hiseq X Ten平台上测序。gydF4y2Ba
根据超低DNA输入工作流(PacBio)进行了修改,构建了HiFi SMRTbell库。简单地说,用磁珠(ProNex, Promega, Madison, WI, USA)纯化20 ng靶向富集的HMW DNA,并用g-Tubes (Covaris)进行碎片化。使用BluePippin大小选择系统将构建的库大小选择为8-17 kb。最终的平均12 kb的文库在Sequel II System (PacBio)上测序。gydF4y2Ba
亚硫酸根测序文库是根据前面描述的方法制备的gydF4y2Ba50gydF4y2Ba使用EZ DNA甲基化金试剂盒(NEB)和NEBNext®Ultra™II DNA文库准备试剂盒(NEB)从10 ng靶向富集HMW MHC DNA分子中进行修饰,并在Illumina Hiseq X Ten平台上测序。Illumina甲基化EPIC珠片使用了500ng GM12878细胞的基因组DNA,实验由上海通用有限公司(中国上海)按照制造商的说明进行。gydF4y2Ba
为了构建链特异性RNA- seq文库,首先使用Trizol (Thermo Fisher Scientific)从GM12878细胞中提取总RNA,然后使用NEBNext Ultra Directional RNA library Prep Kit (NEB)提取1 μg RNA构建测序文库。根据制造商的说明,在Illumina Hiseq X Ten平台上对构建的文库进行测序。gydF4y2Ba
阶段性变异呼叫和HLA分型与10x基因组连接读取数据gydF4y2Ba
10x基因组学链读数据首先用proc10xG处理(process_10xReads.py v0.0.2, regen_10xReads.py v0.0.1, filter_10xReads.py v0.0.1) (gydF4y2Bahttps://github.com/CeciliaDeng/proc10xGgydF4y2Ba)来提取宝石条形码,然后使用Bwa mem (v0.7.15-r1140)与默认选项将没有条形码的链接读取与Lambda序列对齐。对齐后,用Seqkit (v.0.10.0)提取未映射的reads,并进一步过滤,得到:(1)每个条形码的reads数为>2,;(2)条形码序列中不存在N碱基。使用proc10xG将其余具有高条形码质量的未映射读取转换回10x Genomics链接读取格式,并使用Long Ranger (v.2.2.2)和gatk3.8将hg38引用对齐以调用分阶段变体。为了评估在目标MHC区域内的分阶段变体呼叫的性能,我们通过将结果与两个金标准(GIAB (v.4.2.1))进行比较,计算了切换错误率和汉明错误率。gydF4y2Ba21gydF4y2BaIllumina白金基因组gydF4y2Ba22gydF4y2Ba)为GM12878使用WhatsHap比较gydF4y2Ba51gydF4y2Ba.gydF4y2Ba
对于HLA分型,我们根据Long Ranger生成的分期信息将10x Genomics链接读取数据分割为两个单倍型分区读取集,并使用HLA- vbseq (v2)分别预测每个单倍型的HLA类型。gydF4y2Ba23gydF4y2BaIMGT/HLA数据库(v3.44)。gydF4y2Ba
针对MHC区域的伪单倍型组装与10倍基因组学链接读取数据gydF4y2Ba
如前所述,我们使用超新星汇编程序(v2.1.1)对MHC区域进行了10倍基因组学链接读取数据的靶向伪单倍型汇编gydF4y2Ba12gydF4y2Ba与修改。简单地说,首先将链接读取的数据与hg38引用对齐,并保留在目标MHC区域内至少有一个读取映射的条形码。对于每个条形码,计算每个1kb窗口内的目标映射读的数量,并记录每个映射读的基因组位置。为了去除条形码碰撞产生的伪影,相邻映射读取之间的距离为>40 kb的条形码被移除gydF4y2Ba52gydF4y2Ba.保留160kb窗口>10内的映射读数的条形码,以方便目标MHC区域的组装。每个160kb窗口内的链接读取数据被进一步分配到10kb的bins中,即0- 10,10 - 20kb等。对于每个箱子,带有映射读密度的条形码在gydF4y2BangydF4y2Ba去掉了第Th个百分位,使每个箱子的最终覆盖率略低于70×。每个160 kb窗口中的最终过滤条形码随后被合并并用于提取链接读取数据,以便使用带有mkoutput选项的Supernova汇编程序(v2.1.1)构建目标伪单倍型程序集。gydF4y2Ba
用PacBio HiFi对目标区域进行单倍型解析组装,只读gydF4y2Ba
首先,PacBio HiFi读取使用Pbmarkdup (v1.0.0) (gydF4y2Bahttps://github.com/PacificBiosciences/pbmarkdupgydF4y2Ba)以去除扩增中的重复reads,并使用Minimap2 (v2.17)招募对准hg38目标区域的reads。为了消除放大产生的偏差,进行如下所述的下采样。将HiFi读取分配到10kb的bin中,去除每个bin中读取长度最短的read,使最终覆盖达到60×。使用Hifiasm (v0.11-r302)从剩余的HiFi读取生成目标区域的主要组装gydF4y2Ba5gydF4y2Ba,并使用Racon (v1.4.20)进行了进一步的抛光gydF4y2Ba53gydF4y2Ba两轮来消除潜在的装配错误。然后,使用Minimap2招募与校正的主组装对齐的HiFi读取,并使用deepvariant从招募的HiFi读取调用变体(v0.10.0)gydF4y2Ba54gydF4y2Ba并逐步使用whatsapp (v0.17)gydF4y2Ba51gydF4y2Ba.相相杂合子变体,连同校正的主组装,使用whatsapp将HiFi reads分离为两个单倍型分区读集。最后,每个单倍型分区的HiFi读集,连同未标记的读集,使用Hifiasm组装目标区域的两个单倍型。对于目标MHC区域,我们进一步将所有HiFi reads对齐到两个组装的单倍型,以识别补充的HiFi reads(识别出79个HiFi reads),这些补充的HiFi reads与初始招募的HiFi reads相结合,生成目标MHC区域的最终单倍型解析集。gydF4y2Ba
目标区域的单倍型解析组装,具有10x Genomics的链接读取数据和PacBio HiFi读取gydF4y2Ba
首先,PacBio HiFi读取使用Pbmarkdup (gydF4y2Bahttps://github.com/PacificBiosciences/pbmarkdupgydF4y2Ba)以去除扩增中的重复reads,并使用Minimap2招募对准hg38目标区域的reads。然后,使用deepvariant从招募的HiFi reads中调用变体gydF4y2Ba54gydF4y2Ba,并利用whatsapp进行进一步的基因分型gydF4y2Ba51gydF4y2Ba.通过将deepvariant和WhatsHap生成的杂合子变体交叉,然后与从10x Genomics linked-read数据(Long Ranger)获得的靶MHC区域(chr6: 28903952-33268517)内的相相变体重叠,从HiFi reads中获得高可信度的杂合子变体集,以生成具有相位信息的高度可靠的杂合子变体集。最后,基于高度可靠的相相杂合变异集合,使用whatapp将HiFi reads与hg38参考文献分离为两个单倍型分割的读集。为了消除放大产生的偏差,对每个单倍型分区的读集进行下采样,如下所述。将HiFi读取分配到10kb的bin中,去除每个bin中读取长度最短的read,最终覆盖面积为30×。每个单倍型分区的HiFi读集,连同未标记的读集,被用来用Hifiasm组装目标区域的两个单倍型gydF4y2Ba5gydF4y2Ba.由于个体的MHC序列可能与hg38参考序列有很大差异,因此从高度多态区域读取的HiFi可能在读取招募的初始步骤中就被遗漏,从而导致组装结果中的空白。为了解决这个问题,我们将所有的HiFi reads对齐到两个组装的单倍型,以识别补充的HiFi reads(识别出68个HiFi reads),这些HiFi reads在最初对齐到hg38时被遗漏了。这些补充的HiFi reads与最初招募的HiFi reads相结合,生成目标MHC区域的最终单倍型解析组装。gydF4y2Ba
评估目标区域的单倍型分辨组装gydF4y2Ba
为了评估单倍型解析组装的完整性和准确性,将两个组装的单倍型排列到hg38的目标区域,或最近报道的GM12878细胞基因组组装的目标区域gydF4y2Ba8gydF4y2Ba使用Minimap2 (v2.17和v2.24)gydF4y2Ba55gydF4y2Ba.两种组合单倍型的完整性分别基于比对覆盖率和一致性进行评估。为了评估组装的准确性,通过使用Dipcall-1将相控contigs对准hg38参考来调用相控变体gydF4y2Ba56gydF4y2Ba(1) xasm5选项被移除,以包括高度发散的区域;(2) z - 200000、1000以提高对准的连续性;(3) -L 10000设置区域的最小长度为10 kb。切换错误率和汉明错误率是通过比较我们的分阶段变体和从GIAB (v.4.2.1)下载的GM12878的两阶段变体集合来计算的。gydF4y2Ba21gydF4y2BaIllumina白金基因组gydF4y2Ba22gydF4y2Ba使用whatsapp进行比较gydF4y2Ba51gydF4y2Ba.happy .py (v0.3.15) (gydF4y2Bahttps://github.com/Illumina/hap.pygydF4y2Ba)用于评估来自高置信区域的床文件的变体gydF4y2Ba57gydF4y2Ba.对于HLA分型,通过将两个组装的单倍型分别与IMGT/HLA数据库(v3.44)直接比对,预测了8位分辨率的HLA等位基因(gydF4y2Bahttps://www.ebi.ac.uk/ipd/imgt/hla/gydF4y2Ba)使用Minimap2。gydF4y2Ba
对于Sanger序列gydF4y2BaHLA-CgydF4y2Ba等位基因,我们首先扩增了含有不同于现有核苷酸的两个目标区域gydF4y2BaHLA-CgydF4y2Ba在IMGT/HLA数据库中使用嵌套PCR,引物序列包含EcoRI或BamHI限制性内切酶消化位点(补充数据)gydF4y2Ba7gydF4y2Ba).将扩增后的DNA片段克隆到pUC19载体中,从单独挑选的菌落中提取质粒,使用Sanger测序对其进行测序。gydF4y2Ba
倒塌分析gydF4y2Ba
折叠序列的计算如前所述gydF4y2Ba58gydF4y2Ba与修改。简单地说,我们首先生成了个人基因组参考,其中两个靶向组装的个人MHC单倍型分别与hg38的非MHC区域结合。然后,我们将下载的GM12878细胞的PacBio HiFi reads (15;20kb),使用Minimap2独立到每个引用。测序覆盖率使用samtools (v1.9)计算,我们将折叠碱基定义为参考文献中长度至少为10 kb的区域,且除目标MHC区域外的6号染色体的平均覆盖率大于预期覆盖率(平均值+至少三个标准差)。gydF4y2Ba
目标组合个人单倍型与hg38参考之间的坐标映射gydF4y2Ba
为了便于将两种目标组合的个人MHC单倍型与hg38的MHC区域进行基因组学分析的直接比较,我们根据hg38参考文献中的对应坐标生成了这两种单倍型的基因组位置。简单地说,两种单倍型分别使用MUMmer (v4.0.0beta2)与hg38的MHC区域进行比对。gydF4y2Ba59gydF4y2Ba.使用delta-filter命令(选项为“−1”)进一步处理结果,以保持一对一的对齐间隔,并使用show- aligned命令生成具有对齐格式的序列。使用定制的perl脚本将比对结果转换为基因组坐标,从而获得最终的映射坐标。两个单倍型之间的映射坐标也被类似地定义。gydF4y2Ba
目标MHC区域内遗传变异和CpG变异的数量gydF4y2Ba
对于每个单倍型,我们在每个10 kb窗口中计算相对于hg38参考基因的遗传变异总数,其中包括snp和InDels,然后沿着目标MHC区域绘制这些计数。同样,我们也计算了在每个10 kb窗口中相对于hg38的CpG增益和损失的总和,然后相应地绘制了图表。gydF4y2Ba
亚硫酸氢盐测序数据比对和证据提取gydF4y2Ba
亚硫酸氢盐原始测序reads进行质量控制,然后分别对准亚硫酸氢盐转换的三个参考文献:hg38的非MHC区域与目标组合个人MHC单倍型的单倍型1结合;2.hg38的非MHC区域具有目标组合个人MHC单倍型的单倍型2;和3。lambda噬菌体基因组序列),使用bowtie2 (v2.2.3)gydF4y2Ba60gydF4y2Ba使用选项-score-min L,0,0进行完美对齐,而读取则使用默认选项bowtie2映射到亚硫酸氢盐转换hg38参考。对齐后,bismark (v0.22.1)gydF4y2Ba61gydF4y2Ba用于证据提取,以获得每个CpG位点的甲基化和未甲基化胞嘧啶。gydF4y2Ba
CpG变异对DNA甲基化分析的影响gydF4y2Ba
单倍型特异性或共享cpg的平均DNA甲基化水平是通过将测序读数对准对应的目标组合个人MHC单倍型的每个个人基因组参考来估计的。gydF4y2Ba
等位基因特异性甲基化(ASM)分析gydF4y2Ba
进行等位基因特异性甲基化分析,以确定GM12878细胞靶MHC区域的两个单倍型之间的差异甲基化区域。对于ASM,我们进行了两种不同的分析:一种是包含单倍型特异性CpGs,另一种是去除单倍型特异性CpGs。对于没有单倍型特异性CpGs的分析,只有每个等位基因组至少出现两次的CpGs被纳入分析(对于包含单倍型特异性CpGs的分析,不进行此过滤步骤,因为单倍型特异性CpGs将不可避免地被排除)。在处理这些单倍型特异性CpGs后,下游分析相同,如下所述。我们使用bsseq (v1.24.4)平滑证据数据gydF4y2Ba62gydF4y2Ba选项:ns = 50,gydF4y2BahgydF4y2Ba= 800, maxGap = 10gydF4y2Ba8gydF4y2Ba.平滑后,gydF4y2BatgydF4y2Ba-statistic被计算,DMRs被识别使用平滑窗口包含50 cpg或800 bp宽度,两者取较大。接下来,满足以下条件的区域被认为是假定的DMRs:(1) agydF4y2BatgydF4y2Ba-statistic with an absolute value >3.6;(2)必须包含至少3个CpG站点;并且(3)必须有至少10%的甲基化差异。gydF4y2Ba
验证基因启动子区域的等位基因特异性甲基化gydF4y2BaHLA-DPA1gydF4y2Ba基因,我们进行亚硫酸氢盐Sanger测序。简单地说,使用EZ DNA甲基化金试剂盒(zyymo Research, Irvine, CA, USA)用亚硫酸氢钠转换GM12878细胞的500ng基因组DNA,并根据制造商的说明将样品在20 μl洗脱缓冲液中洗脱。DMR-DPA1中含有3个CpG位点的目标区域用嵌套PCR引物扩增(补充数据gydF4y2Ba7gydF4y2Ba)与EcoRI或BamHI酶切位点结合,随后克隆到pUC19载体中。质粒分别从采集的菌落中提取,并使用Sanger测序进行评估。gydF4y2Ba
荧光素酶报告试验gydF4y2Ba
荧光素酶报告基因检测如前所述gydF4y2Ba63gydF4y2Ba与修改。使用无cpg的萤火虫荧光素酶报告载体(pCpG-free basic vector, Invivogen)检测启动子的活性。在DMR-DPA1 (TTACGTTA)中合成含有8个重复片段的DNA,然后克隆到萤火虫荧光素酶基因上游pCpG-free基本载体的启动子区域。甲基化荧光素酶报告使用m.s sssi CpG甲基转移酶(NEB)进行。甲基化或非甲基化萤火虫荧光素酶报告基因与组成性表达Renilla荧光素酶的对照pRL-TK载体通过PEI共转染HEK293T细胞。转染48小时后,使用双荧光素酶报告检测系统(Promega)收集细胞,检测荧光素酶活性。启动子活性通过计算Firefly与Renilla的归一化来确定。所有试验均进行三次重复。gydF4y2Ba
Illumina Infinium EPIC甲基化阵列数据分析gydF4y2Ba
使用minfi包v1.28.0对原始EPIC idat文件进行预处理和规范化gydF4y2Ba64gydF4y2Ba使用默认选项。在0-1尺度上计算探针上每个CpG位点的甲基化β值(M/(M + U + 100),其中M和U分别代表甲基化和非甲基化信号强度)。对于该阵列上的CpG位点,我们将其基因组坐标从hg19转换为hg38,使用(gydF4y2Bahttps://genome.ucsc.edu/cgi-bin/hgLiftOvergydF4y2Ba),并将它们的位置与亚硫酸氢盐测序数据中的位置重叠,以确定两个平台之间共享的cpg。两组共享的CpG通过EPIC阵列探针上被测CpG位点5 bp内是否存在或探针上不存在遗传变异来确定。通过决定系数计算两组共享CpGs的EPIC和亚硫酸氢盐测序数据之间的甲基化相关性gydF4y2BaRgydF4y2Ba2gydF4y2Ba通过EPIC阵列对亚硫酸氢盐测序数据进行线性回归计算,与Pearson的平方相等gydF4y2BaRgydF4y2Ba.gydF4y2Ba
等位基因特异性表达分析与验证gydF4y2Ba
RNA-Seq数据来自GM12878细胞的3个重复。使用fastp (v0.21.0)对测序读取进行质量控制gydF4y2Ba65gydF4y2Ba,并使用Trimmomatic过滤以删除适配器序列、低质量碱基以及读取长度<50 bp的读取(v0.39)gydF4y2Ba66gydF4y2Ba.然后将过滤后的reads分别与两个个人基因组参考(hg38的非MHC区域与目标组合个人MHC单倍型的单倍型1结合;或hg38的非MHC区域,具有目标组合个人MHC单倍型2),使用HISAT2 (v2.1.0)gydF4y2Ba67gydF4y2Ba使用默认参数。WASP包的rmdup_pe.py命令(gydF4y2Bahttps://github.com/bmvdgeijn/WASPgydF4y2Ba)gydF4y2Ba68gydF4y2Ba用于去除PCR重复reads。对于每个对齐,只保留唯一对齐的读数以进行量化。通过比较NCBI下载的基因注释文件(gydF4y2Baftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/000/001/405/GCA_000001405.28_GRCh38.p13/GCA_000001405.28_GRCh38.p13_genomic.gff.gzgydF4y2Ba),并与hg38进行比对,得到两个组装单倍型的基因注释信息。gydF4y2Ba
使用bedtools multicov命令分别计算每个等位基因的表达水平。使用DESeq2包(v1.38.0)中的estimateSizeFactors命令对每个基因的读取进行标准化gydF4y2Ba69gydF4y2Ba.双侧瓦尔德检验gydF4y2BaPgydF4y2Ba对每个基因的两个等位基因进行DESeq2计算。等位基因特异性表达基因经校正后具有统计学意义gydF4y2BaPgydF4y2Ba-value < 0.05。gydF4y2Ba
等位基因特异性表达gydF4y2BaHLA-DPA1gydF4y2Ba通过焦磷酸测序、等位基因特异性qRT-PCR和扩增PCR克隆的Sanger测序进行验证。对于焦磷酸测序,外显子3区gydF4y2BaDPA1gydF4y2Ba从GM12878细胞的基因组DNA或cDNA中PCR扩增基因,并使用测序引物进行焦磷酸测序,计算扩增产物中两个等位基因的目标snp的比例(补充数据)gydF4y2Ba7gydF4y2Ba).对于等位基因特异性qRT-PCR,引物(补充数据gydF4y2Ba7gydF4y2Ba的每一个单倍型外显子4区域gydF4y2BaDPA1gydF4y2Ba用于扩增GM12878细胞的cDNA。的各个单倍型的相对表达gydF4y2BaDPA1gydF4y2Ba的水平归一化,计算基因gydF4y2BaGAPDHgydF4y2Ba.对于扩增PCR克隆的Sanger测序,编码区gydF4y2BaDPA1gydF4y2Ba用PCR引物从GM12878细胞的cDNA中扩增出两个不同单倍型之间包含多个snp的基因(补充数据gydF4y2Ba7gydF4y2Ba)与EcoRI或BamHI酶切位点结合,随后克隆到pUC19载体中。质粒分别从采集的菌落中提取,并使用Sanger测序进行评估。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然组合报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
所有相关数据均由GM12878单元生成。支持本研究关键发现的所有相关数据均可在文章及其中获得gydF4y2Ba补充信息gydF4y2Ba文件。目标组合单倍型和被称为变体可以从gydF4y2Bahttps://liulab.fudan.edu.cn/targeted_assemblies.htmlgydF4y2Ba.DNA测序数据与Illumina短读平台(登录代码no.;gydF4y2BaSRR17250933gydF4y2Ba全基因组测序;登记编码gydF4y2BaSRR17250934gydF4y2Ba目标富集MHC区域;登记编码gydF4y2BaSRR21079439gydF4y2Ba对于目标丰富的gydF4y2BaRHCEgydF4y2Ba而且gydF4y2BaCR1gydF4y2Ba区域),10x基因组连接读取(登录代码no.;gydF4y2BaSRR17250932gydF4y2Ba目标富集MHC区域;登记编码gydF4y2BaSRR21079437gydF4y2Ba对于目标丰富的gydF4y2BaRHCEgydF4y2Ba地区;登记编码gydF4y2BaSRR21079438gydF4y2Ba对于目标丰富的gydF4y2BaCR1gydF4y2Ba区域),PacBio HiFi长读(登录代码no。gydF4y2BaSRR17250931gydF4y2Ba目标富集MHC区域;登记编码gydF4y2BaSRR21079435gydF4y2Ba对于目标丰富的gydF4y2BaRHCEgydF4y2Ba地区;登记编码gydF4y2BaSRR21079436gydF4y2Ba对于目标丰富的gydF4y2BaCR1gydF4y2Ba区域)和RNA-Seq数据(登录码编号:;gydF4y2BaSRR17250928gydF4y2Ba- - - - - -gydF4y2BaSRR17250930gydF4y2Ba, (gydF4y2Bahttps://www.ncbi.nlm.nih.gov/sra/?term=SRR17250929gydF4y2Ba])存入序列读取档案(SRA)。来自目标富集MHC区域的亚硫酸氢盐测序数据和来自GM12878细胞基因组DNA的Illumina甲基化EPIC珠片数据可在基因表达综合(GEO)数据库中获得,登录号如下gydF4y2BaGSE192499gydF4y2Ba而且gydF4y2BaGSE192501gydF4y2Ba,分别。gydF4y2Ba
Garg等人的程序集和基于程序集的相阶段变体调用从[gydF4y2Baftp://ftp.dfci.harvardgydF4y2Ba.edu/pub/hli/whdenovo/]。折叠分析中使用的PacBio HiFi reads从GIAB Consortium获得:[gydF4y2Baftp://ftp-trace.ncbi.nlm.nih.gov/ReferenceSamples/giab/data/NA12878/HudsonAlpha_PacBio_CCS/PBmixSequel851_2_B01_PCCL_30hours_15kbV2PD_70pM_HumanHG001_CCS/m64109_200815_033514.fastq.gzgydF4y2Ba]、[gydF4y2Baftp://ftp-trace.ncbi.nlm.nih.gov/ReferenceSamples/giab/data/NA12878/HudsonAlpha_PacBio_CCS/PBmixSequel851_1_A01_PCCL_30hours_15kbV2PD_70pM_HumanHG001_CCS/m64109_200813_162416.fastq.gzgydF4y2Ba]、[gydF4y2Baftp://ftp-trace.ncbi.nlm.nih.gov/ReferenceSamples/giab/data/NA12878/HudsonAlpha_PacBio_CCS/PBmixSequel846_3_C01_PCDB_30hours_15kbV2PD_70pM_HumanHG001_CCS/m64109_200808_191025.fastq.gzgydF4y2Ba]、[gydF4y2Baftp://ftp-trace.ncbi.nlm.nih.gov/ReferenceSamples/giab/data/NA12878/HudsonAlpha_PacBio_CCS/PBmixSequel846_1_A01_PCCL_30hours_15kbV2PD_70pM_HumanHG001_CCS/m64109_200805_204709.fastq.gzgydF4y2Ba];[gydF4y2Baftp://ftp-trace.ncbi.nlm.nih.gov/ReferenceSamples/giab/data/NA12878/HudsonAlpha_PacBio_CCS/PBmixSequel846_4_D01_PBXM_30hours_21kbV2PD_70pM_HumanHG001_CCS/m64109_200810_062248.fastq.gzgydF4y2Ba]和[gydF4y2Baftp://ftp-trace.ncbi.nlm.nih.gov/ReferenceSamples/giab/data/NA12878/HudsonAlpha_PacBio_CCS/PBmixSequel846_2_B01_PCCM_30hours_21kbV2PD_70pM_HumanHG001_CCS/m64109_200807_075817.fastq.gzgydF4y2Ba].IMGT/HLA数据库(v3.44)来源于[gydF4y2Bahttps://www.ebi.ac.uk/ipd/imgt/hla/gydF4y2Ba].图1b、4e、4g和补充图7c、8c、866 8d底层的源数据作为源数据文件提供。gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
在此工作中使用的所有代码可在以下地址获得:gydF4y2Bahttps://liulab.fudan.edu.cn/targeted_assemblies_codes.htmlgydF4y2Ba
参考文献gydF4y2Ba
Ebert, P.等人。单倍型解析不同人类基因组和结构变异的综合分析。gydF4y2Ba科学gydF4y2Ba372gydF4y2Ba, eabf7117(2021)。gydF4y2Ba
瓦格纳等人。策展变异基准挑战医学相关的常染色体基因。gydF4y2Ba生物科技Nat。》。gydF4y2Ba40gydF4y2Ba, 672-680(2022)。gydF4y2Ba
柯伦,S.等人。单倍型测序基因组的重新组装。gydF4y2Ba生物科技Nat。》。gydF4y2Ba36gydF4y2Ba, 1174-1182(2018)。gydF4y2Ba
杨,C.等。来自绒猴二倍体基因组组装的进化和生物医学见解。gydF4y2Ba自然gydF4y2Ba594gydF4y2Ba, 227-233(2021)。gydF4y2Ba
程浩,李国涛,冯晓霞,张海华,李海华。基于相位装配图的单倍型解析从头装配。gydF4y2BaNat方法。gydF4y2Ba18gydF4y2Ba, 170-175(2021)。gydF4y2Ba
周,Q.等。杂合二倍体马铃薯单倍型解析基因组分析。gydF4y2BaNat,麝猫。gydF4y2Ba52gydF4y2Ba, 1018-1023(2020)。gydF4y2Ba
孙,x等。阶段性的二倍体基因组组装和泛基因组为了解苹果驯化的遗传历史提供了依据。gydF4y2BaNat,麝猫。gydF4y2Ba52gydF4y2Ba, 1423-1432(2020)。gydF4y2Ba
Garg, S.等人。染色体尺度,单倍型的人类基因组组装。gydF4y2Ba生物科技Nat。》。gydF4y2Ba39gydF4y2Ba, 309-312(2021)。gydF4y2Ba
张欣,等。单倍型解析基因组组装提供了对茶树进化史的深入了解gydF4y2Ba茶树gydF4y2Ba.gydF4y2BaNat,麝猫。gydF4y2Ba53gydF4y2Ba, 1250-1259(2021)。gydF4y2Ba
蒋,W.等。cas9辅助靶向染色体片段CATCH使大基因簇的一步靶向克隆。gydF4y2BaCommun Nat。gydF4y2Ba6gydF4y2Ba, 8101(2015)。gydF4y2Ba
Gabrieli, T.等人。通过cas9辅助靶向染色体片段(CATCH)对人BRCA1进行选择性纳米孔测序。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, e87(2018)。gydF4y2Ba
Shin, G.等人。靶向短读测序、重排和候选基因位点的组装提供了百万酶双倍型。gydF4y2Ba核酸测定。gydF4y2Ba47gydF4y2Ba, e115(2019)。gydF4y2Ba
吉尔帕特里克等人。cas9引导转接头结扎靶向纳米孔测序。gydF4y2Ba生物科技Nat。》。gydF4y2Ba38gydF4y2Ba, 433-438(2020)。gydF4y2Ba
Kovaka, S., Fan, Y., Ni, B., Timp, W. & Schatz, M. C.利用UNCALLED实时绘制原始电信号的靶向纳米孔测序。gydF4y2Ba生物科技Nat。》。gydF4y2Ba39gydF4y2Ba, 431-441(2021)。gydF4y2Ba
伦茨,T. L.,斯皮林,V.,乔丹,D. M.和桑尼亚耶夫,S. R. HLA基因周围有害突变的过剩揭示了平衡选择的进化成本。gydF4y2Ba摩尔。杂志。另一个星球。gydF4y2Ba33gydF4y2Ba, 2555-2564(2016)。gydF4y2Ba
Matzaraki, V, Kumar, V, Wijmenga, C. & Zhernakova, A. MHC位点和自身免疫和传染性疾病的遗传易感性。gydF4y2Ba基因组医学杂志。gydF4y2Ba18gydF4y2Ba, 76(2017)。gydF4y2Ba
丘玮,刘,杨。类风湿关节炎MHC区DNA甲基化:展望和挑战。gydF4y2Baj . Rheumatol。gydF4y2Ba47gydF4y2Ba, 1597-1599(2020)。gydF4y2Ba
Chin, C. S.等。主要组织相容性复合体变异的基于二倍体组装的基准。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 4794(2020)。gydF4y2Ba
人类MHC中最先进的基因组推断。gydF4y2BaInt。学生物化学j。细胞生物。gydF4y2Ba131gydF4y2Ba, 105882(2021)。gydF4y2Ba
人类主要组织相容性复合体的完整序列和基因图谱。MHC测序联盟。gydF4y2Ba自然gydF4y2Ba401gydF4y2Ba, 921-923(1999)。gydF4y2Ba
瓦格纳等人。对具有链接和长读取的小变量进行基准测试。gydF4y2Ba细胞染色体组。gydF4y2Ba2gydF4y2Ba, 100128(2022)。gydF4y2Ba
埃伯利,m.a.等人。通过对三代17人谱系进行测序,通过遗传遗传验证了540万阶段人类变异的参考数据集。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 157-164(2017)。gydF4y2Ba
王友友等。HLA- vbseq v2:改进HLA调用精度与全长日本class-I面板。gydF4y2Ba嗡嗡声。基因组Var。gydF4y2Ba6gydF4y2Ba, 29(2019)。gydF4y2Ba
崔氏,崔氏,崔氏。主要组织相容性复杂基因组学与人类疾病。gydF4y2Ba为基础。启染色体组。嗡嗡声。麝猫。gydF4y2Ba14gydF4y2Ba, 301-323(2013)。gydF4y2Ba
Dendrou, C. A., Petersen, J., Rossjohn, J. & Fugger, L. HLA变异与疾病。gydF4y2BaNat. Rev. Immunol。gydF4y2Ba18gydF4y2Ba, 325-339(2018)。gydF4y2Ba
Jain等人。纳米孔测序和超长读取人类基因组组装。gydF4y2Ba生物科技Nat。》。gydF4y2Ba36gydF4y2Ba, 338-345(2018)。gydF4y2Ba
诺曼,P. J.等。95个人类MHC单倍型序列揭示了除HLA I类和II类高度多态性外的基因的极端编码变异。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 813-823(2017)。gydF4y2Ba
Radwan, J., Babik, W., Kaufman, J., Lenz, T. L. & Winternitz, J. MHC多态性的进化理解进展。gydF4y2Ba趋势麝猫。gydF4y2Ba36gydF4y2Ba, 298-311(2020)。gydF4y2Ba
Dilthey, A., Cox, C., Iqbal, Z., Nelson, M. R. & McVean, G.使用种群参考图改进了MHC中的基因组推断。gydF4y2BaNat,麝猫。gydF4y2Ba47gydF4y2Ba, 682-688(2015)。gydF4y2Ba
罗佐斯基,J.等人。AlleleSeq:分析网络框架中特定等位基因的表达和结合。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba7gydF4y2Ba, 522(2011)。gydF4y2Ba
Saukkonen, A., Kilpinen, H. & Hodgkinson, A.对人群和疾病遗传学的等位基因表达的高度精确量化。gydF4y2Ba基因组Res。gydF4y2Ba32gydF4y2Ba, 1565-1572(2022)。gydF4y2Ba
Wulfridge, P., Langmead, B., Feinberg, A. P. & Hansen, K. D.分析高度不同基因型的全基因组亚硫酸氢盐测序数据。gydF4y2Ba核酸测定。gydF4y2Ba47gydF4y2Ba, e117(2019)。gydF4y2Ba
霍奇金森,A. &埃尔-沃克,A.在哺乳动物基因组突变率的变化。gydF4y2BaNat. Rev. Genet。gydF4y2Ba12gydF4y2Ba, 756-766(2011)。gydF4y2Ba
鸟类,a.p. DNA甲基化和动物DNA中CpG的频率。gydF4y2Ba核酸测定。gydF4y2Ba8gydF4y2Ba, 1499-1504(1980)。gydF4y2Ba
巴罗,t.m.和卞恩,h.m.。DNA甲基化微阵列上的单核苷酸多态性:预防混淆。gydF4y2Ba表观基因组学gydF4y2Ba6gydF4y2Ba, 577-579(2014)。gydF4y2Ba
Aguiar, V. R. C, Cesar, J., Delaneau, O., Dermitzakis, E. T. & Meyer, D. HLA基因的表达估计和eQTL定位与个性化管道。gydF4y2Ba公共科学图书馆麝猫。gydF4y2Ba15gydF4y2Ba, e1008091(2019)。gydF4y2Ba
带有选择的双位点基因转换模型及其在人类RHCE和RHD基因中的应用。gydF4y2Ba国家科学院学报美国gydF4y2BaOne hundred.gydF4y2Ba, 8793-8798(2003)。gydF4y2Ba
埃伯特,m.t.w.等人。对黑暗基因和伪装基因的系统分析揭示了隐藏在肉眼可见的疾病相关基因。gydF4y2Ba基因组医学杂志。gydF4y2Ba20.gydF4y2Ba, 97(2019)。gydF4y2Ba
胡,等。HLA-DQ和HLA-DR分子中三个氨基酸位置的加性和相互作用作用会导致1型糖尿病的风险。gydF4y2BaNat,麝猫。gydF4y2Ba47gydF4y2Ba, 898-905(2015)。gydF4y2Ba
Sekar, A.等人。补体成分复杂变异对精神分裂症的风险gydF4y2Ba自然gydF4y2Ba530gydF4y2Ba, 177-183(2016)。gydF4y2Ba
Gensterblum-Miller, E., Wu, W. & Sawalha, A. H.人类MHC区域的新转录活性和广泛的等位基因失衡。gydF4y2Baj . Immunol。gydF4y2Ba200gydF4y2Ba, 1496-1503(2018)。gydF4y2Ba
Johansson, T., Yohannes, D. A., Koskela, S., Partanen, J. & Saavalainen, P.用独特的分子标识符进行HLA RNA测序,揭示了mRNA表达的高等位基因特异性变异性。gydF4y2Ba前面。Immunol。gydF4y2Ba12gydF4y2Ba, 629059(2021)。gydF4y2Ba
Boegel, S.等人。从RNA-Seq序列中读取HLA分型。gydF4y2Ba基因组医学。gydF4y2Ba4 gydF4y2Ba, 102(2012)。gydF4y2Ba
Lee, W., Plant, K., Humburg, P. & Knight, J. C. AltHapAlignR:通过使用替代单倍型提高RNA-seq分析的准确性。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, 2401-2408(2018)。gydF4y2Ba
狄尔泰,a.t.等。利用群体参考图从全基因组测序数据进行HLA型的高精度推断。gydF4y2Ba公共科学图书馆第一版。医学杂志。gydF4y2Ba12gydF4y2Ba, e1005151(2016)。gydF4y2Ba
豪特曼,等人。健康个体和类风湿性关节炎患者MHC II类基因单倍型特异性表达分析gydF4y2Ba前面。Immunol。gydF4y2Ba12gydF4y2Ba, 707217(2021)。gydF4y2Ba
罗宾逊等人。IPD-IMGT / HLA数据库。gydF4y2Ba核酸测定。gydF4y2Ba48gydF4y2Ba, d948-d955(2020)。gydF4y2Ba
Sambrook, J. & Russell, D.W.用于脉冲场凝胶电泳的DNA制备:从哺乳动物细胞和组织中分离DNA。gydF4y2BaCSH Protoc。gydF4y2Ba2006gydF4y2Ba, pdb。prot3235(2006)。gydF4y2Ba
Bennett-Baker, P. E. & Mueller, J. L. crispr介导的基因组DNA特定兆酶片段的分离。gydF4y2Ba核酸测定。gydF4y2Ba45gydF4y2Ba, e165(2017)。gydF4y2Ba
刘,P.等。改变的DNA甲基化模式揭示了Hox基因在胸主动脉夹层中的表观遗传调控,并可作为疾病诊断的生物标志物。gydF4y2Ba中国。Epigenet。gydF4y2Ba13gydF4y2Ba, 124(2021)。gydF4y2Ba
帕特森等人。WhatsHap:用于未来一代测序读取的加权单倍型组装。gydF4y2Baj .第一版。医学杂志。gydF4y2Ba22gydF4y2Ba, 498-509(2015)。gydF4y2Ba
郑国祥等。单倍分型生殖系和癌症基因组高通量链接测序。gydF4y2Ba生物科技Nat。》。gydF4y2Ba34gydF4y2Ba, 303-311(2016)。gydF4y2Ba
Vaser, R., Sovic, I., Nagarajan, N. & Sikic, M.从长时间未校正的阅读中快速和准确的从头基因组组装。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 737-746(2017)。gydF4y2Ba
波普林,R.等人。使用深度神经网络的通用SNP和小indel变体调用器。gydF4y2Ba生物科技Nat。》。gydF4y2Ba36gydF4y2Ba, 983-987(2018)。gydF4y2Ba
Minimap2:核苷酸序列的成对比对。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, 3094-3100(2018)。gydF4y2Ba
李,H.等。一种用于精确评价变异调用的合成二倍体基准。gydF4y2BaNat方法。gydF4y2Ba15gydF4y2Ba, 595-597(2018)。gydF4y2Ba
Krusche, P.等。人类基因组中生殖系小变异呼叫基准测试的最佳实践。gydF4y2Ba生物科技Nat。》。gydF4y2Ba37gydF4y2Ba555-560(2019)。gydF4y2Ba
贾维斯,E.D.等人。高质量二倍体人类参考基因组的半自动组装。gydF4y2Ba自然gydF4y2Ba611gydF4y2Ba, 519-531(2022)。gydF4y2Ba
马凯伊斯,G.等。MUMmer4:一个快速和通用的基因组比对系统。gydF4y2Ba公共科学图书馆第一版。医学杂志。gydF4y2Ba14gydF4y2Ba, e1005944(2018)。gydF4y2Ba
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 357-359(2012)。gydF4y2Ba
Krueger, F. & Andrews, S. R. Bismark:用于亚硫酸酯- seq应用的灵活对准器和甲基化调用器。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 1571-1572(2011)。gydF4y2Ba
Hansen, K. D., Langmead, B. & Irizarry, R. A. B. smooth:从全基因组亚硫酸氢盐测序reads到差异甲基化区域。gydF4y2Ba基因组医学杂志。gydF4y2Ba13gydF4y2Ba, r83(2012)。gydF4y2Ba
孟,W.等。DLGAP2在酒精使用和依赖中的基因型依赖的表观遗传调控gydF4y2Ba精神病学摩尔。gydF4y2Ba26gydF4y2Ba, 4367-4382(2021)。gydF4y2Ba
阿尔耶,m.j.等人。Minfi:用于Infinium DNA甲基化微阵列分析的灵活而全面的Bioconductor包。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 1363-1369(2014)。gydF4y2Ba
陈淑娟,周勇,陈勇,顾杰。fastp:一种超快一体化FASTQ预处理器。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, i884-i890(2018)。gydF4y2Ba
Bolger, a.m., Lohse, M. & Usadel, B. Trimmomatic:用于Illumina序列数据的灵活修剪器。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 2114-2120(2014)。gydF4y2Ba
Kim, D, Paggi, J. M, Park, C., Bennett, C. & Salzberg, S. L.基于图的基因组比对和HISAT2和hisat基因型的基因分型。gydF4y2Ba生物科技Nat。》。gydF4y2Ba37gydF4y2Ba, 907-915(2019)。gydF4y2Ba
van de Geijn, B., McVicker, G., Gilad, Y. & Pritchard, J. K. WASP:用于稳健分子数量性状位点发现的等位基因特异性软件。gydF4y2BaNat方法。gydF4y2Ba12gydF4y2Ba, 1061-1063(2015)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
确认gydF4y2Ba
本工作得到国家自然科学基金项目(No. 82171837,刘云)、国家重点研发计划项目(No. 2021YFC2701001,刘云)、上海市学术技术带头人计划项目(No. 20XD1420400,刘云)、上海市科技重大专项(No. 2017SHZDZX01、2018SHZDZX01,刘云)、上海市科学技术基金项目(20ZR1404700,刘云)资助。我们感谢来自PacBio APAC的wangqianjie和Wilson Cheng的有益讨论、故障排除工作和分析建议。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
刘云想出了这个主意。tl.l在j.m.、w.m.、Z.J、h.y.、j.w.、s.d.、s.s.、w.y.、b.l.d.、d.d.、D.Z的协助下进行实验,林翼成在M.Z.、z.c的协助下进行分析,hl.l、Z.Z、Y.J和Z.X.提出实验设计。刘云和W.Q.监督了这项工作。t.l., W.Q.和刘云在所有作者的协助下完成了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
基于本文的研究成果,一项专利申请已经提交(Yun Liu et al。CN201911027231.0)。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然通讯gydF4y2Ba感谢Justin Zook和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
李,T.,杜,D.,张,D。gydF4y2Baet al。gydF4y2Ba基于crispr的靶向单倍型解析百万酶区域组装。gydF4y2BaNat CommungydF4y2Ba14gydF4y2Ba, 22(2023)。https://doi.org/10.1038/s41467-022-35389-wgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41467-022-35389-wgydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba