文摘
结构重组已被证明是重要的在当地适应和物种形成,但一直难以可靠地识别和描述non-model物种。我们结合长读,读和光学映射到柳树莺描述三个不同的染色体区域中国东北部柳莺,其中两个是与不同的移民和环境梯度。我们表明,反演(0.4 -13 Mb),每个地区之间的分歧时间倒和non-inverted单体型相似地区(~ 1.2最高产量研究),这是兼容了场景,反演两种分布区不重叠的人群,随后杂化。改进的基因组允许我们探测附加功能不同地区的差异,为移民提供候选基因和适应环境梯度。
介绍
潜在位点地方适应和物种形成是集中在反演在许多种类的动物和植物1。通过捕获co-adapted变异位点有关,表生的逆序对促进形成,复杂的表型遗传好像他们是由单个基因编码2。例如,反演多态性与鸟类不同的交配类型有关3,4、社会昆虫的多态性5并在鱼类迁徙的表型差异6,7。然而,对于non-model物种,更大的反演难以可靠地识别和描述,作为断点经常配合repeat-rich基因组区域很难装配,特别是短内容测序技术8。克服这些挑战将是重要的扩大我们的理解当地的适应和物种形成。
柳树莺中国东北部柳莺是由两个不同的迁移人口在欧洲吗9,10。南方迁徙表型(ssp。蜂鸟)发生在西欧和非洲西部迁移。北方迁徙表型(ssp。acredula)品种北部斯堪的纳维亚和东欧和非洲东部和南部的冬天。亚种是否则形态和生态相似11,12。
最全面的基因研究的柳树莺迄今为止,Lundberg et al。13草案基因组组装基于短内容数据和使用全基因组重测序和一个定制的4000个SNP数组来探索迁移表型之间的遗传差异。绝大多数的变体之间的高度分化迁移表型位于三个不同区域在染色体1,3,5。染色体变异在区域1和5与迁移表型紧密相关,而该地区3号染色体上显示的纬度和海拔。清晰地分隔高原高之间的遗传分化和重组的明显缺乏发散在这些地区南部和北部的单体型建议反演多态性的存在。但是,没有反转断点可以确定,大概是因为他们位于重复富裕地区。此外,染色体的两个migration-linked区域1和5,被分成两个,十个支架,分别,很难知道基因的顺序在这些地区不同于其他鸟类中发现的。
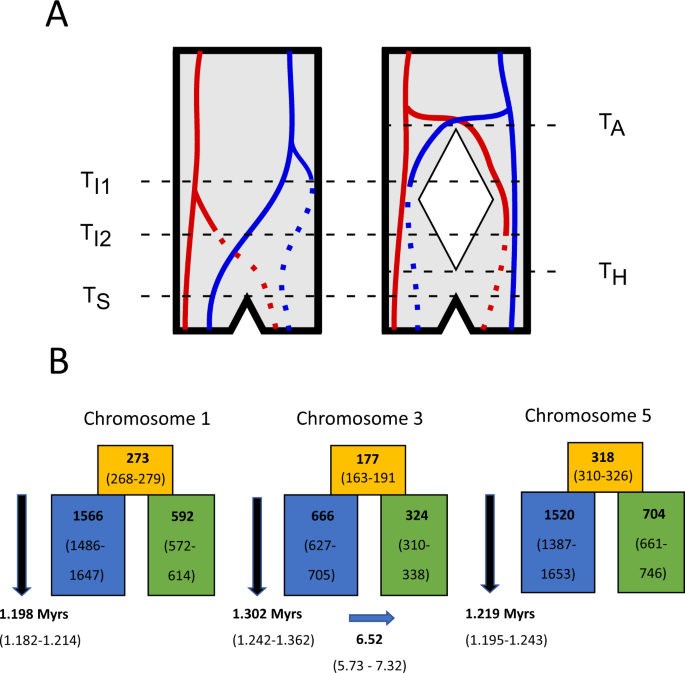
在这项研究中,我们使用读测序,linked-read测序,光学映射和RNAseq创建更完整、连续的和好的注释基因组装配的南部和北部柳树莺。新的基因组组件允许我们探索不同染色体的结构组织地区每一个亚种,检查如果额外高度分化迁移表型之间的地区可能驻留在基因组部分不包含在前面的短内容基因组组装,并评估功能高度分化变异的结果。我们也使用长阅读组装棕柳莺的基因组测序中国东北部collybita和比较这柳树莺组件来获得不同区域的进化历史的信息。最后,我们适合模型的人口差异使用块内的信息网站频谱(bsf)14。这种方法,改编自Lohse et al。15祖先的有效估计,人口规模以及迁移的速度,所以提供更准确的估计人口分歧时间比汇总统计,这些参数被假定或忽略。如果每个地区不同的单,目前与每个亚种关联与反演,独立在一个祖先的人口,我们就没有期望区域(图之间的分歧时间同步。1)。另一种情况是,不同的单是一个古老的杂交事件的结果,这曾被假设解释现存的柳树莺种群的多样性16。在这个场景中,北部和南部单是独特的杂交种群和互相结合,防止通过倒置,而其余的基因通过基因均相流。在这种情况下,我们期望散度次类似的跨区域(图。1),因为他们不代表实际的反转事件,而是古代人口划分的时间17。
一个人口替代方案有两个倒置(从固体到虚线行)出现在不同的位点(红色和蓝色)时间点TI1和TI2,分别。在左边的场景中,反演表现为多态性在单一祖先人口和最终会分为两个亚种(T年代)。散度次倒和non-inverted单,在这种情况下,反映了时间点的反转事件。在正确的情况下,人口祖先分裂成两个分区的数量(T一个)和反演出现在其中的一个。两种群后二次接触和合并(TH前),反演得到分为两个亚种(T年代)。在这个场景中,散度次倒和non-inverted两个位点的单将是相似的,因为他们并没有反映出时间反演的事件,但是祖先的时候人口分割(T一个)。B人口模型参数估计(最大复合可能性95 ci)三个不同区域的柳树莺。橙色,蓝色,和绿色框指祖先,北部和南部的有效种群大小,分别对数字以10为单位3。3号染色体,模拟确认更适合一个模型包括迁移(IM2),在这种情况下,蓝色箭头指示的方向迁移以10为单位给出的速度−7。这三个不同的地区也有类似的分离时间(~ 1.2最高产量研究),按预期从正确的场景(一个)。
结果
基因组装配
使用读的组合排序,linked-read测序和光学映射,我们获得高度连续新创总成南部和北部柳莺、含有547支架34 Mb的将军和496支架的将军17 Mb,分别为(补充表1)。这些邻近度量代表一个数量级改进以前的短内容相比,柳树莺基因组组装13和类似于或超过其他鸟类组件结合长读和光学映射数据18,19。使用音响读测序,我们生成棕柳莺的新创装配组成的517叠连群将军28 Mb(补充表1)。总成的质量也验证了很高比例的完整和较低比例的重复单副本鸟orthologues(94.1 - -95.2%和1.1 - -1.5% 4915年的目标基因,分别补充表1)。
柳树莺南部、北部柳树莺和棕柳莺大会包含21.0,14.8,9.7%和21.2%的带注释的重复,分别,而在前面的柳树莺组装(补充表2)。南部组装注释的22757个蛋白编码基因基于组合柳树莺RNAseq数据(补充表3)和蛋白质数据从其他鸟类。
不同亚种
我们研究了亚种间的遗传分化映射全基因组重测序数据的南部和北部11 11柳树莺(补充表4南部组装。加权平均F圣之间的北部和南部4500万年样本bi-allelic snp是0.006和平均体重平均F圣在10 kb windows也是0.006。只包括变异与未成年人等位基因频率(加)至少0.1(1200万),分别对应的值分别为0.01和0.007。高度差异化的变异的数量非常小,与先前识别几乎所有位于不同的染色体区域(补充表5)。例如,11855个变异的F圣≥0.7,只有140以外的三个地区被发现。绝大多数(84%)的140个变异被发现在9个支架(中等大小:3699632个基点,范围:511299 - 7950085个基点),不能自信地分配给特定染色体的鸡背带吊裤带,斑胸草雀Taeniopygia guttata或成卷的捕蝇草Ficedula albicollis基因组。这些支架含有高比例的重复序列(87 - 95的% ungapped长度),GC含量很高(50 - 51%)和主要嗅觉受体基因,虽然还不清楚在多大程度上这些基因的功能。此外,覆盖在这些支架是通常在北方比南方低重新测序样品(补充图。1)和类似的支架也发现在北部大会,但只能部分对齐支架在南部基因组。这表明,这些支架代表基因组区域显示不同亚种之间的高阶重复组织。
1号染色体区域
南部组装、1号染色体上的高度分化区域聚集成一个11.9 Mb免费(gap)支架(Scaffold19,无花果。2)。的开始和结束脚手架包含49和174 kb的数组,分别413个基点的串联重复序列。发散区域不能与其他染色体1,但预测相邻支架的两端,基于捕蝇草和斑胸草雀的基因组包含数组(67和70 kb)相同的串联重复序列(图。2),这可能解释了为什么这个地区是很难完全组装即使HiFi长读和光学映射数据。在北方大会,不同地区被组装成一个同样大小的脚手架(11.7 Mb),包含一个58 kb差距包围串联重复序列。南方支架缺乏长间隔的串联重复序列数组对应地区的差距,但一个270 kb的数组是出现在棕柳莺组装。棕柳莺,一端连接不同地区的部分染色体1预测从捕蝇草和斑胸草雀。在这个连接,有一个440 kb的串联重复序列数组。
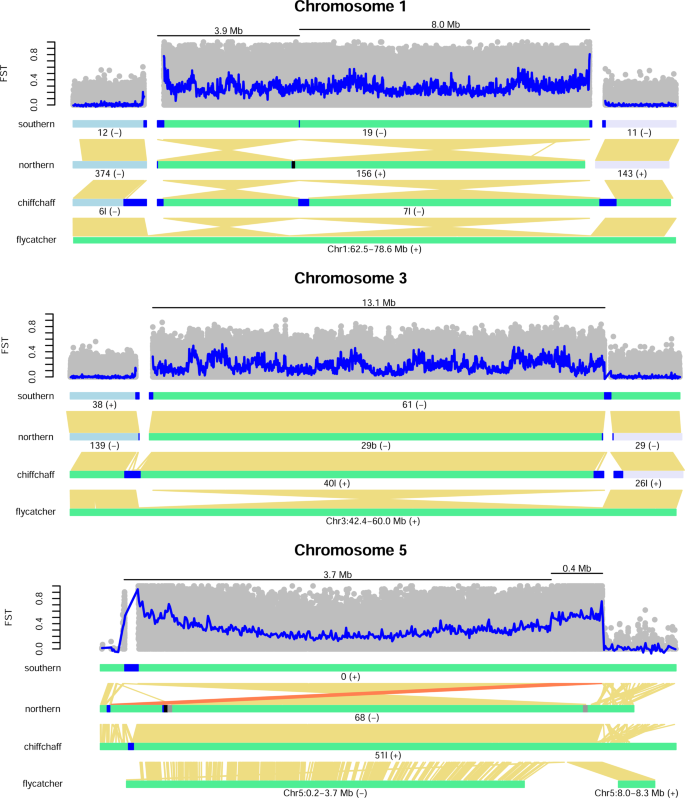
前面板显示遗传分化(F圣)11个亚种为每个变量的重新测序样本与未成年人等位基因频率(加)≥0.1,与蓝线代表加权平均加≥0.1 bi-allelic snp 10 kb重叠窗口。下面,支架或染色体(科)在每个装配匹配不同的地区显示为亮绿色矩形(ID和策划方向)和预测上游和下游支架浅蓝色和浅紫色的矩形,分别。蓝色部分支架显示大串联阵列的位置结束或断点区域,黑色部分代表差距和灰色部分的位置31 kb复制间隔在5号染色体区域北部组装。黄线支架上方代表1:1校准间隔(≥2 kb)支架在南部组装。染色体5,第二个反转时间间隔已用橙色突出提高能见度。斑胸草雀基因组同样显示了主要的结构差异的捕蝇草基因组和,因此,没有被包括在内。
南部新创组装不同于其他基因组的存在两个衔接着安排反演不同地区3.7 Mb和7.9 Mb(无花果。2)。共享断点区间伴随着更靠近串联重复序列数组在北部柳树莺和棕柳莺,但在南部组装,只有1.5 kb间隔相同的重复。断点在南部和北部之间的差异的样本也支持条形码读取(补充图有关的报道。2)以及光学地图,易位从开始到结束的发散式脚手架在北部南部组装检测样本。
11之间的遗传分化重新测序样品从每个亚种是整个地区的高(平均加权F圣在windows bi-allelic snp的乘加10 kb≥0.1: 0.28),但显示著名山峰开始时和结束时(无花果。2)。
3号染色体区域
南部组装,3号染色体上的高度分化区域(13.1 Mb)是位于69.3 Mb的脚手架(Scaffold61),展示了一个反方向相比,捕蝇草和斑胸草雀(无花果。2)。在不同地区之间和剩余的支架是一个184 kb的串联重复序列作为1号染色体上相同类型的数组。相同类型的串联重复序列数组还发现不同地区的另一端(两个间隔12和78 kb),最后预测相邻支架(94 kb, Scaffold38,无花果。2)。作为该地区1号染色体上,我们没有发现任何证据斑胸草雀的串联重复序列数组或捕蝇草。北部组装、发散区域是包含在13.2 Mb脚手架(Scaffold29b)不能可靠地搭建与其他未分化的部分染色体3。脚手架的开始包含8 kb串联重复序列数组和最终包含673个基点间隔相同的串联重复序列,这是紧随其后的是一个296个基点LTR / ERVL重复。棕柳莺组装,不同地区也发现了相同的(反向)取向在南部柳树莺,但与其他地区的染色体另一端(叠连群ptg000040l,无花果。2)。该连接与一个444 kb的串联重复序列数组,类似于柳树莺,串联重复序列数组也出现在另一端的发散区域间隔(279 kb),最终预测相邻重叠群(252 kb, ptg000026l)。
结束时,该地区的南部脚手架(Scaffold61,开始在无花果。2),有一个断点区别长阅读建议的柳树莺亚种的比对(补充图。3)。在这个区间,读取从北方柳树莺不能合理对齐以外的开始重复阵列和棕柳莺读取这种模式也被观察到。棕柳莺大会中的相应区域显示一个不同的结构配置与南方柳树莺相比,在一个额外的间隔9.7 kb为主的LTR / ERVL之间存在重复765和444 kb的串联重复序列数组。相比北方柳树莺,读取来自南部柳树莺不一致在这个区域(补充图。3)。这表明,北部柳树莺和棕柳莺分享更多的祖先配置在该地区,并且反演中观察到棕柳莺可能是一个独立事件的结果相比,南方的柳树莺。
高度分化变异出现在整个地区差异化(平均加权F圣在windows bi-allelic snp的乘加10 kb≥0.1: 0.19),但与该地区1号染色体上,我们没有看到任何明显的断点效应(无花果。2)。
5号染色体区域
南部组装,5号染色体上的高度分化区域(4.1 Mb)是一个67 Mb的一部分脚手架(Scaffold0),涵盖了大部分的染色体(无花果。2)。在不同的时间间隔,南方的柳树莺主要是共线的捕蝇器和斑胸草雀。两边的不同区间,有repeat-rich区域不能自信地对齐到其他物种。在北部组装、发散间隔是嵌入在一个4.6 Mb脚手架(Scaffold68,无花果。2)。柳树莺南部相比,北方柳树莺有3.7 Mb倒和0.4 Mb共线但进行间隔,分离的41个kb(图上的巨大差距。2)。这种差距是被重复串联的脚手架和形成一个116 kb的数组在南部地区大会的开始。此外,反向间隔在北部组装周围是31 kb节段重复显示94%彼此身份(无花果。2)。复制间隔富含重复和包含一个截断核仁的副本pre-ribosomal-associated蛋白1 (URB1)基因,它在南部发现装配的完整副本上游附近地区。
基于反向和共线的柳树莺亚种之间的间隔,可能的情况是,整个第一次倒在北方亚种分化间隔和第二个较小的反转事件恢复秩序。棕柳莺组装,发散区域嵌入在6.8 Mb叠连群(ptg000051l)和显示相同的取向在南部柳树莺(无花果。2)。这表明,北部柳树莺派生的取向。亚种之间的结构差异在这个区域被链接读取比对,进一步支持长期的wgs管道检测到断点的区别在北方~ 4.0 Mb脚手架(Scaffold68)和删除在北部样本与串联重复区域的区域在南部脚手架(Scaffold0)。此外,有关阅读条形码覆盖北方样本低于其他两个样品在断点地区南部脚手架(补充图。2),尽管周围的模式并不明确串联重复序列区开始时由于整体低覆盖率。最后,亚种之间的结构差异明显,组合光学北方样本映射到南部的组装样品(补充图。4)。
南部和北部之间的遗传分化重新测序鸟类高(平均加权F圣在windows bi-allelic snp的乘加10 kb≥0.1: 0.32)在整个地区,但高出平均在0.4 Mb内进行区间(图。2)。随着该地区的1号染色体上的遗传分化有断点的效果,特别高值区域的开始和结束。
不同的年龄和人口的地区
我们使用两个高覆盖率(注意寻找车号为24 - 44×)重新测序样品每个亚种的估计人口之间的分歧时间亚种的不同区域(补充表4)。首先,我们计算净散度(d一个)20.和发现了类似的值在三个区域(补充表6)。使用生殖系突变速率估计的成卷的捕蝇草21,假设一代时间1.7年11,这些值对应于散度乘以512,544和539可以对染色体区域1,3,5,分别。作为一个补充,但不是独立的散度测量,我们计算相对节点深度(RND)22使用一个昏暗的莺中国东北部fuscatus(补充表4)作为一个外围集团。研发估计是类似的跨区域(0.35 - -0.41,补充表6),表明分歧出现最近多的共同祖先柳树莺和昏暗的莺。
Divergence-based方法预计将导致低估了散度*如果有基因流动人口之间发生。此外,如果祖先人群的多样性远远大于或小于在当代人群,d一个分别将偏见向上或者向下。克服这些警告,并获得更健壮的估计人口差异,我们安装的人口模型涉及散度和两个种群间基因流为每一个不同的地区。我们使用的软件23,它利用信息块中的网站频谱(bsf)14比较支持(复合可能(CLs))对不同人口统计模型和参数。
不同地区的染色体1和5的最佳拟合模型(即。最高的CL)是我1模型。在这个模型中,人口分割是紧随其后的是一个常数的迁移(me来自南部人口)北部人口在时间维度。不同地区的3号染色体上的最大使用IM CL被发现2模型,IM迁移发生在相反的方向1。然而,IM模型预计总比严格隔离实现更大的CL (SI)模型,因为它们包括一个额外的参数,迁移的速率。此外,迁移率最大的复合(制程)估计可能性很小(4.45×10−7,6.53×10−7和3.46×10−7对于染色体1,3,5,分别),虽然符合有限内复合反演,表明IM模型可能不适合明显比斯。因此,测试是否在CL IM模型做了一个重大的进步,我们模拟复制下优化如果参数为每个不同的地区和记录CL IM和SI模型之间的差异。对染色体区域1和5的改善我们发现CL模型之间是完全符合严格的隔离(补充图的历史。5)。相比之下,改善CL观察到3号染色体区域大于我们希望如果没有迁移。以后我们提出并讨论参数估计的简单SI模型染色体1和5的区域和IM2模型3号染色体区域(图。1)。
制程参数估计,每个不同区域内的分歧时间大约是1.2最高产量研究,因此远比估计的d一个计算。有效种群大小都估计更高的北部柳树莺和祖先的数量小于在当代人群。类似的参数估计在三个区域,特别是发散时间(1.20 - -1.30最高产量研究),历史表明,他们有一个共同的人口和支持一个场景,逆序对发生在分区的人口。
一致认为,几乎所有的这些亚种之间的遗传分化仅限于只有三个地区,人口的制程评估分歧时间上面提到一个数量级大于类似估计超过其余的基因组(补充表7)。然而,分布的聚结时间之外的不同地区可能仍然包含这些亚种的人口历史的信息。例如,一段持续的人口结构将导致减少合并率(增加Ne),直到人群再次成为混。测试是否合并率随着时间的推移,遵循这样的模式,我们使用了连续马尔可夫链的合并(SMC) MSMC2中实现24。对于每一个样本,Ne估计是在更新世期间,最高达到~ 450 kya,和较低的在最近(< 200 kya)和遥远的过去(> 2米娅)(补充图。6)。虽然对这种模式有多种解释,但它至少符合一段分布区不重叠的可能性,1.2米娅开始和结束< 450 kya发散的原因,不同地区都有一个共同的人口历史。
我们也计算几个人口统计汇总数据来推断人口的影响,如瓶颈。不同地区,特别是在染色体1和5,柳树莺南部有一个整体的核苷酸多样性较低,较高的日本田岛的D和大量的高频衍生等位基因比北部高柳莺(补充无花果。7,8)。类似地,haplotype-based分析(XP-nsl)不同地区一般建议延长低多样性在南部地区样本相比,北方样品(补充图。9)。
功能不同地区的差异
改善组装和注释,我们检查潜在的功能性差异三个染色体区域的亚种。没有一个断点区间重叠或非常接近一个带注释的功能蛋白质编码基因(范围:1.5 - -71.2 kb,补充表8)。我们还探讨了单核苷酸多态性是否与高分化(F或短indels圣≥0.7)南部与北部的消费者之间,该预测有中度到高对蛋白编码基因的影响。在三个区域,我们发现73年产生的基因突变和一个在坐标系插入位于46(补充表9)。我们发现另外一个移码突变一般转录因子iii a (GTF3A),位于1号染色体上的不同区域。这种变化,代表北方亚种中删除,修改最后四个氨基酸,进一步扩展了蛋白质和三个氨基酸。尽管蛋白质编码的基因变化功能多样化,一些共享更具体的功能。特别是,产生的突变被发现在三个脂肪酸desaturase基因(FADS2,FADS1L1,FADS1L2),位于串联在第二分化间隔5号染色体上。其中两个基因(FADS2,FADS1L2)也标注为参与“氧化还原过程”与细胞色素b5还原酶2 (CYB5R2)和gamma-butyrobetaine羟化酶1 (BBOX1),5号染色体上,晶状体蛋白λ1 (CRYL11号染色体上。
我们也寻找高度分化的结构变异南部与北部的消费者之间,不同地区的样本。我们检测到31日删除(51 - 2934个基点),24插入(55 - 1511 bp)和两个重复(52 - 110个基点),F圣≥0.7南部与北部的消费者之间,比如在不同的地区。大部分的结构变体(35/57)以外的带注释的基因平均距离的47个kb和只有一个变体重叠蛋白质编码基因的外显子:插入207个基点的3 'utr Stomatin像3 (STOML3该地区)基因位于染色体上1。
最后,我们探讨是否有最近的迹象在基因在不同地区积极的选择。一个强烈的信号被发现在一个内含子的Spondin-1 (SPON1)基因,它位于5号染色体区域的开始(补充无花果。9,10)。在这里,我们找到了一个高比例的snp XP-nsl北部离群值,以及高Sweepfinder2 CLR值和北部的核苷酸多样性减少样本。
讨论
使用高度连续的基因组,我们已经表明,不同地区分离的两个柳树莺亚种与结构重组。我们也证实了她的结果等。13通过发现几乎所有亚种之间的高度分化SNPs和indels位于这些区域。然而,读测序和光学映射数据使我们能够识别额外的差异以前忽略repeat-rich支架可能代表更大规模的亚种之间的结构差异。由于其高重复内容,我们没能自信地分配这些支架特定染色体在其他鸟类,也不确定是否类似地区北部组装来自相同的基因组的一部分。然而,最近的一项研究25表明,这些重复的最大(12 Mb)支架在北部组装与小说转座因子的扩张,而不是与任何先前确定的三个不同的染色体区域。
通过拟合人口模型的频谱块网站,我们发现北部和南部单散度* ~ 1.2最高产量研究的三个区域(图1)。这些估计是远远低于柳树莺及其之间的分歧时间最近的亲戚,棕柳莺,大约在5最高产量研究26。因此,我们可以拒绝假设的存在不同的单在柳树莺从一个现存基因渗入的结果中国东北部物种。散度的估计是类似报道大常见的反演多态性在其他几个研究系统3,4,7,尽管最近的倒置会更难检测积累了更少的散度。
我们的分析提供支持之前的假说,现存的柳树莺是一个古老的结果两个不同种群之间的杂交事件16。类似的分歧时间跨区域兼容一个场景,一个祖先人口分成两个分区数量,随后相互杂化和均质基因组除了发散区域(图1)。在这个场景中,估计分歧时代将是一次分裂,人口和结构重组将会出现在一些时间人口分割和辅助触点之间的事件。倒单可以隔离在低频的分区数量和频率增加辅助触点的时候由于积极的选择17。在这种情况下,结构重组是选择,因为他们保护有利等位基因组合在不同的染色体区域,例如,那些与适应特定的在每个人口迁徙的路线,从分解由于基因流动和重组27。
随着时间的全基因组变化的有效种群大小决定从MSMC2分析(补充图。6)在很大程度上是符合该场景的分布区不重叠的人群。全基因组的有效种群大小可以增加当人口结构28在柳树莺我们观察增加在北部和南部的分歧时间估计单。人口规模的下降在400年开始kya可以反映合并分区的数量。然而,全基因组的改变Ne也可以由人口普查人口变化,尽管场景并不是相互排斥的。
不同地区对染色体1和5,这与迁移表型的差异,我们发现附近的重组或相互嵌套(无花果。2)。复杂的重组已经观察到在一个广泛的分类单元7,29日,30.并有可能进一步减少基因流动人口之间。比对棕柳莺大会和其他鸟组件使我们能够确定哪些亚种的派生或祖先的基因顺序在每个地区(图。2)。为该地区1号染色体上,南方亚种的派生基因顺序,而在该地区5号染色体上,北方柳树莺拥有派生的重组。意外,因为该地区3号染色体上,棕柳莺和南方柳树莺相比具有派生重排捕蝇草和斑胸草雀。柳树北部之间的共享结构配置的棕柳莺莺和地区开始表明,有两个独立的反转事件。跨物种基因不稳定区域与复发性反演曾被观察到在哺乳动物31日,32。
反向传播的单在人口的辅助触点事件可以预计到有选择性减少变异33。然而,我们没有观察到任何一致的减少Ne倒单,这表明任何多样性的清洁工发生在足够长的前积累和/或倒单已经隔离一段时间选择事件发生和产生柔和的清洁工。相反,南单都分配较低的有效种群大小在建模分析(无花果。1),总体上较低的核苷酸多样性,高日本田岛的D和大量的高频衍生等位基因(补充无花果。7- - - - - -9)。北单目前发现一个更大的地理范围比南方的单13,特别是对染色体1和5个地区,和,因此,合理的,甚至历史上保持更大的有效种群大小。
我们的分析只支持一个隔离与迁移模型对该地区3号染色体上,那里有从北方迁移到南方人口(无花果。1)。该地区3号染色体上明显不同于其他两个地区的北部和南部单体型的地理分布13,34。而不同的单染色体1和5只在狭窄的迁徙将在欧洲,不同的接触区单3号染色体上从斯堪的纳维亚半岛中部向东延伸至西伯利亚南部,这可能允许更多的基因流的机会。基因流增加在这一地区也可以促进更复杂的明显缺乏重组的其他两个地区(无花果。2)。虽然不支持的模拟,我们不能排除,至少一些北部和南部种群间基因流也在区域1和5号染色体上。双跨界车和基因转换,基因流的主要机制允许反向和共线单之间,和预计少接近断点35。与预测相一致的是,我们观察到最高的分化断点附近的染色体区域1和5(无花果。2)。
我们确定了相似的序列重复至少有一些不同的断点在每个不同的地区,有可能被直接参与形成的结构性变化36。有趣的是,相同类型的数组的串联重复序列相关的染色体断点区域1和3,和在这两个亚种在棕柳莺,但没有在相应的时间间隔在斑胸草雀或成卷的捕蝇草基因组。在柳树莺基因组内,高度相似的和完整的重复副本(至少50%的长度和90%的身份)被限制在两个地区和支架预测相邻。不同地区南部组装5号染色体上显示不同类型的串联重复序列数组开始,以及31 kb节段重复包含的截断和可能pseudogenized副本URB1基因在两个断点的北部组装。
识别选择性的目标在每个不同的地区是富有挑战性的,因为大量的基因(N= 47 - 197)和高连锁不平衡。断点自己可能面临选择,如果他们修改表达式或扰乱蛋白质编码基因的序列3,37。然而,没有一个断点区间重叠或非常接近注释功能基因(补充表8),尽管我们不能排除影响多个远程监管元素。
我们观察到明显减少高频衍生等位基因多样性和过多的北部的但不是在柳树莺内含子的南部SPON1基因,位于5号染色体区域(补充图。10)。该模式表明积极的选择发生在北部柳树莺和序列变化可能监管影响基因的表达。SPON1已被证明是重要的轴突指导38并在昼夜节律也被牵连39。这个基因的差异,因此,可以想象背后的一些亚种之间的迁徙行为观察到的差异。我们还发现了73高度分化snp或短indels 46个基因预测修改蛋白质编码序列(补充表9)。这些基因与多种生物过程相关联,其中一些缺乏任何功能注释。预测影响最大的变量是一个移码删除的GTF3A基因位于染色体1,它还包含了三个额外的高度差异化的snp。这个基因编码转录因子参与5 s核糖体rna基因的转录,在人类已经与身体质量指数相关40。高度分化,这个基因变体可能与生理适应不同亚种的迁徙路线。符合这一点,我们还发现高度分化产生的三个脂肪酸desaturase基因位于不同地区的串联5号染色体上。脂肪酸desaturase基因调节的未饱和脂肪酸和已被证明是人类饮食适应性的基础41,42。
该地区3号染色体上,另一方面,显示了很强的相关性与海拔和纬度在繁殖区域13,34,一个潜在的选择性单北部的好处可以增加耐寒性。在这种情况下,一个潜在的候选基因将低密度脂蛋白受体相关蛋白(11LRP11),这是注释的基因本体术语“应对寒冷”。
为了确定额外的假定的功能差异,我们也高度分化的结构变异筛查。虽然大多数的这些变异位于远离最近的基因,插入207个基点的3 '端非翻译区重叠STOML31号染色体上,可能会影响基因转录后调节,调节机械感受器的敏感性43。机械参与多种生理过程44和潜在的结构性变异的表型效应,因此,很难预测。然而,应该注意的是,我们能够准确的基因结构变异短内容的重新测序样本有限,尤其是在更多的重复间隔,未来的研究将从额外的读数据样本可能会发现一个更广泛的光谱相关的结构性差异。
总的来说,产生变化的功能注释和结构变异表明,区域影响不同的基因通路和可能广泛的表型效应。
总之,我们已经表明,结构性重组维护大量分化地区尽管广泛的杂交,和我们的结果添加到越来越多的证据表明,结构性重组往往是复杂和重复扩张。使用建模方法,我们获得更健壮的散度的估计时间和显示,不同地区的三个染色体是相似的。这个观察是兼容一个场景,反演了分区数量,后来为辅助触点和杂化。最后,我们改进的基因组和注释提供了一组新的候选基因与移民相关的适应性和环境梯度。
方法
本研究的研究是在协议执行许可M45-14出具马尔默/隆德伦理委员会动物研究,瑞典捕获和野生鸟类的血液抽样
样品
九柳树莺,确定为男性(基于机翼长度> 69 mm),传统上被抓着雾网在秋季迁徙的时间2016年9月在Krankesjon,隆德以东15公里,瑞典南部。虽然大多数个体表型相似的柳树莺育种在斯堪的纳维亚半岛南部,有的略大,有一个灰色的羽毛,这是常见的在斯堪的纳维亚半岛北部12。组样本从而可能含有柳树莺每个两大迁徙的表型。通过血液从鸟儿肱静脉穿刺和存储在两个帕子包含设置缓冲和70%的乙醇,分别。一个整除的血液被用于DNA提取phenol-chloroform协议。从提取的DNA,我们样品有两个位点的基因位于染色体1和5,分别为(NBEA和FADS2)45,46,bi-allelic 3号染色体上的不同区域内的标记(AFLP-ww1)47。基于纯合的基因分型结果我们选择两个样品北部或纯合子南部三个位点,分别。我们从一个棕柳莺还包括一个示例中国东北部collybita(女)为新创一个外围集团密切相关物种的基因组测序,以及额外的柳树莺(DD81063,男)确认断点差异与联系阅读顺序。这两种鸟类伺机抓在上面的网站一样在2019年秋天迁徙,和收集的血液其他鸟类都遵循着相同的方法。
光学地图
DNA的北部和南部柳树莺从血液中提取存储在乙醇使用塞溶解协议(v.30026D;美国CA Bionano基因组学)。血液是首先通过温和的离心分离乙醇和嵌入在熔融2%琼脂糖插头(DNA插入设备;美国CA Bio-Rad)。固化插头被淹没在裂解缓冲溶液(Bionano基因组学)和66.8µl每毫升缓冲Puregene蛋白酶K(试剂盒,医学博士,美国)2 h在50°C。的插头随后被洗1×洗缓冲区(Bio-Rad DNA塞工具包)TE缓冲紧随其后。在以下步骤中,插头是核糖核酸酶处理(试剂盒,20µl 1毫升TE缓冲)1 h在37°C,紧随其后的是另一个洗涤步骤使用相同的缓冲区和前面的步骤一样。接下来,2分钟的插头都融化在70°C和对待GELase(美国WI中心)为45分钟43°C。DNA使用下降然后从消化琼脂糖纯化透析对TE缓冲0.1µm透析膜(MF-Millipore、默克公司、德国)为2.5 h。
光学映射的两个样品是利用Bionano基因组的商业Irys系统48。BspQ1决心是最合适的轻伤酶后使用软件LabelDensityCalculator v.1.3.0和短裤v.1.5.5分析前一个短内容组装13。Bionano基因组的IrysPrep Labeling-NLRS协议(v.30024)是用于NLRS反应。在这一步中,DNA处理Nt.BspQ1(美国马内)来创建单链缺口molecule-specific模式。这些被贴上Bionano基因组(美国CA)标签组合(NLRS工具包),借助于Taq(内),并使用Bionano修复基因的修复组合(NLRS工具包),在Thermopol Rxn缓冲区,NAD +, Taq DNA连接酶(内)。最后,DNA骨干染色使用DNA染色Bionano基因组学的NLRS工具包。每个样本然后加载两个IrysChips (Bionano基因组学),和DNA染色BspQ1缺口是可视化使用Irys乐器,Bionano基因组Irys后用户指南(v.30047)。这导致了200年和182 Gb的北部和南部的数据样本,分别。
基因组地图在房子新创使用Bionano基因组的组装软件IrysView v.2.5.1,用噪声参数设置为“autonoise”和使用人工参数的xml文件。基因组地图然后进一步精炼的话语将所有数据,但使用第一个组装版本作为参考。总大小的最后程序集都是1.3 Gb,平均覆盖率92.3和96.4×0.93和0.95 Mb的将军,北部和南部的样本,分别。
阅读有关测序
样本和样本DD81063南部,DNA测序铬(10×基因组学、钙、美国)从血液中提取存储在缓冲区设置使用MagAttract高分子量DNAkit(试剂盒)Scilifelab,斯德哥尔摩,瑞典。北方bionano光学地图使用的示例提取。北部和南部的库样本都测序的一个单独的车道HiSeqX(美国CA Illumina公司)和DD81063样本测序在NovaSeq6000 (Illumina公司)。对所有样本测序进行使用2×150个基点的设置。
北部柳树莺新创组装
图书馆准备长阅读以前提取的DNA测序完成光学映射和太平洋生物科学(美国CA)后的10 - 20 kb的标准协议库。没有图书馆建设前进行剪切,但是库大小选择使用BluePippin脉冲磁场的大小选择系统(Sage科学、马、美国),大小截止> 25 kb。图书馆测序八SMRT细胞续集平台(太平洋生物科学)。测序产生了63.66英镑的数据由4690365 subreads平均长度为13573个基点(范围:50 - 170531个基点)。
Pacbio读取HGAP4组装新创49SMRT链接包的默认设置,除了指定一个预期的基因组大小的1.2英镑和抛光算法设置为“箭头”。我们跑猎鹰解压50在组装获得部分阶段性主要重叠群和完全阶段性haplotigs。在软件中,箭头用于波兰组装使用读取分配给每一个单体型。我们评估两个解压缩组件基于30×40×覆盖种子读取HGAP4预装配的一步。覆盖率较低阈值将导致再读入初始装配步骤,这可能会增加装配的连续性,但另一方面,限制阅读的数量,可以使用在逐步和抛光的步骤。虽然解压缩组件非常相似,40×版本被选为下游分析更连续,包含更多的单份鸟orthologues由车身3.0.2版本51。
大会进一步抛光Pilon 1.2252与Illumina公司铬读取相同的样本。Illumina公司读取映射到装配使用0.7.17-r1188 bwa版本53和重复读取标记使用picardtools 2.10.3 (http://broadinstitute.github.io/picard)。Pilon由只有纠正indels和总共1043827 275457插入和删除的软件,分别,其中绝大多数(94%)是单身完全改变。Illumina公司抛光有明显影响的单份鸟orthologues数量可以检测到主叠连群(补充表1)。
为进一步组装的步骤中,我们提取了Illumina-polished主Pacbio重叠群(N2.1 Mb = 2737,将军和1.29 Gb)的长度。这些叠连群表明意外高水平的重复单副本orthologues(7.4%),暗示部分或完全重叠部分重叠群。作为第一步,减少冗余,提高装配的连续性,我们杂化的主要叠连群光学映射相同的示例使用bionano解决版本3.2.2 (bionano基因组学)和默认设置除了指定咄咄逼人的脚手架参数。混合脚手架导致19削减bionano地图和259年削减Pacbio super-scaffolds叠连群,创造了363。大部分的重叠群间的缝隙super-scaffolds估计是负的(即。,一些重叠序列)。然而,在混合组装,序列两侧的差距没有倒塌,从而形成假节段重复。解决这个问题我们提取304套重叠叠连群(“supercontigs”)和GAP5用于2.0.0.b11 staden包54寻找潜在的重叠群结束之间的连接。使用这种方法,我们合并重叠群558(87%)的假定的重叠。指重叠排列长度是111 kb(范围:0.259 -661 kb)平均3.28%的序列差异(范围:0.31 - -15.55%)。最高的散度是由大型indels的存在。通过修剪掉一个或两端重叠群的差距(平均23 kb,范围:0.6 -60 kb),我们能够进一步接近23缺口。剩余的空白,GAP5未能找到潜在的重叠群之间的连接或末端应该加入被认为有过高的散度。新议会,包括supercontigs由2401叠连群6.5 Mb的将军,有一个低很多的重复单份基因(4.6% vs 7.4%)。
为了进一步减少冗余,我们使用了清洗haplotig管道55(下载2019-02-15)去除重叠群可以映射在大部分长度较大的重叠群和覆盖显示有限的二倍体。我们首先估计覆盖映射Pacbio subreads用于新创大会与minimap2 version2.13-r86056使用默认设置Pacbio读取(- x map-pb)。把损失减小到最低限度的重复序列,可以分离和搭建bionano光学地图,我们使用第一个bionano混合动力总成(363 superscaffolds和1500年削减和unscaffolded叠连群)映射作为参考。从映射的数据我们发现一个明确的单倍体和二倍体峰和设定一个阈值的二倍体覆盖率高于34 85××下面。任何脚手架,不到80%的头寸二倍体报道被认为是公认的haplotig并映射到软件内的其他使用minimap2支架。我们删除了1209个支架(平均大小:107655个基点,范围:598 - 495788个基点)的报道最好的达到至少70%(意思是:97.4%)。使用这种方法,我们特别排除叠连群superscaffolds不能合并。然而,我们也被三叠连群,每个完全由短superscaffolds可以唯一地分配给大型superscaffolds和高度的单倍体的报道。在这个阶段,我们也找到了五额外叠连群短于1000个基点,是切割组装的结果与bionano光学映射。这导致了一个装配1187叠连群,长度为1.1英镑和7.9 Mb的将军。过滤装置显示大幅减少单副本orthologue鸟基因(4.6% vs 1.3)。
提供一个中级的脚手架光学地图,我们绘制了10×铬读取相同的样品组装使用bwa和弧版本1.0.5使用571.8.6和链接版本58脚手架。弧是使用默认设置运行除了使缺口大小估计(——dist_est)和链接是通过设置支持链接的数量至少5 (- l = 5)和比值的最大链接两个最佳重叠群对0.3 (- a = 0.3)。脚手架导致739支架将军16.4 Mb,长度1.12 Gb。
最后搭建步骤,我们杂化10×chromium-Pacbio支架bionano光学映射使用相同的设置。混合搭建了23削减光学地图,122年削减支架,支架的将军曾经497年的16.8 Mb。两个叠连群代表1号染色体上的不同区域被弧搭建在一起但分离,而不是与其他序列re-scaffolded bionano混合组装。因为不匹配的光学映射是短,位于一个巨大的差距,和基因顺序是相同的在其他鸟类基因组,我们决定保持弧产生的脚手架。
这一轮的混合脚手架,有52个缺口,估计是负的。一样使用相同的方法在创建supercontigs时,我们能够接近10这些差距。我们另外使用PBJelly封闭缺口59从PBSuite 15.8.24与默认设置除了指定——spanOnly capturedOnly”。软件97缺口,12的一端差距,两端延伸18差距和满满的28差距(扩展的两端,但发现没有重叠,尽管扩展大于预测差距)。
我们进一步检查支架之间潜在的错接,源自不同的染色体。为此,我们使用的是SatsumaSynteny 2.060生产装配和鸡的基因组之间的全基因组比对(版本GRCg6a)和斑胸草雀(taeGut3.2.4版),从运用下载(www.ensembl.org)。使用这种方法,我们发现一个脚手架显示好的比对两个染色体10日和23日的其他物种。我们认为这个连接可能,决定将脚手架。
接下来,我们进行第二轮抛光的10×铬Illumina公司相同的样本数据。这一轮,因为我们有不到500支架,我们使用了长期2.1.14对齐管道61年地图读取barcode-aware。Pilon然后运行着相同的设置,导致417032年的修正indels,其中78.7%是single-basepair变化。第二轮抛光大大增加单副本orthologues鸟的数量可以确定组装(补充表1)。
线粒体基因组是没有发现在原始的Pacbio基因组组装。我们获得这个通过添加完整的线粒体基因组序列从先前的短内容组装13。然后使用bwa地图10×铬读取从北部样品组装和提取线粒体序列比对。接下来,freebayes是使用一个单倍体设置检测差异出现在读取保持一致。原始变量与vcftools文件过滤网站的质量小于30和两个间隔过度阅读报道(可能从未装配的读取NUMTs)。过滤后的文件包含11替换和三个变体indels,并使用bcftools 1.14版62年创建一个新的线粒体参考。
不同序列的提取和删除的组装步骤我们使用kentUtils 370 (https://github.com/ucscGenomeBrowser/kent)。摘要统计信息为每个组装(如将军)使用assemblathon_stats计算。pl脚本63年。
柳树莺南部和棕柳莺新创组件
南柳莺和棕柳莺每个测序两车道上续集II(太平洋生物科学)使用高保真(高保真)设置。测序库南柳莺从先前的提取制备用于光学地图(见上图),而棕柳莺,DNA从血液中提取使用Nanobind提取工具包(Circulomics,医学博士,美国)。2576876年柳树样本产生了南部HiFi读取平均长度为19303 bp和代表49.7英镑。棕柳莺样本产生2612165 HiFi读取19829个基点的平均长度和代表51.8英镑。
高保真读取新创组装使用0.15.5-r350 hifiasm版本64年默认设置和主要选择叠连群下游分析。的棕柳莺hifiasm大会,我们删除第一个6 Mb的一部分重叠群重叠与另一个重叠群和删除短间隔结束时,一个包含适配器序列重叠群。对于南方柳树莺,主重叠群(N= 540,补充表1)杂化光学映射相同的样品使用相同的管道的北部样本。虽然我们有铬相同的样本的数据,我们没有包括它执行一个中间搭建步骤(就像我们对北方柳树莺组装)因为读大会已经高度连续的。杂交步骤由39削减重叠群和20削减光学地图,导致组装superscaffolds 111和439 non-scaffolded重叠群。我们决定忽略一个光学map-supported叠连群,映射到单独的染色体融合在其他鸟类,这种融合是在大量重复区域。我们进一步排除45 bp序列产生的混合组装切割和蒙面四包含适配器很短的间隔序列。线粒体在南部的组装装配都遵循着相同的管道用于北方组装(见上图)。在这种情况下,10替换和两个indels被添加到线粒体序列前短内容组装基于对齐的读取来自南部样本有关。
重复注释
我们使用1.0.8 Repeatmodeler版本65年新创的识别重复的南部组装。的重复检测到repeatmodeler结合1023 bird-specific重复到自定义库。接下来,我们使用4.0.7 repeatmasker版本66年自定义库,通过使用一个更敏感的搜索(s标志)注释基因组中重复。Bedtools v2.29.267年一起带注释的重复,用来创建一个softmasked版本的南部组装、基因注释中使用步骤。同样的重复图书馆也用于注释重复新创组装的北部样本。棕柳莺大会我们使用相同的注释方法至于柳树莺南部,但包括与repeatmodeler种特异的库生成,还包括相关的串联重复序列与发散区域相关的染色体1和3的柳树莺图书馆。间隔串联重复序列在不同的地区也分析了串联重复序列4.0.9仪版本68年使用默认设置,除了指定2000个基点的最大周期大小。
复制间隔内不同支架被确定Minimap2和随后与浮雕担架6.6.0 (https://www.ebi.ac.uk/Tools/psa/emboss_stretcher/)。
RNA序列
我们使用总RNA提取整个大脑从六个样品用于早期研究量化微分表达式在迁徙和繁殖柳树莺69年(补充表3)。RNA的质量检查生物分析仪版2100(美国安捷伦,CA)。所有的痛苦有一个RNA完整性(RIN)的数量至少> 7.10。RNA库测序是准备使用TruSeq滞留信使RNA样本准备装备有96双索引(Illumina公司)根据制造商的指示除了自动化协议使用一个门店工作站(安捷伦)和使用纯化步骤中描述必et al70年。和Borgstrom等71年。原始RNA数据削减使用cutadapt 1.8版72年在削减大量的版本0.4.0 (https://github.com/FelixKrueger/TrimGalore用默认设置)。
我们使用Stringtie v1.3.373年从RNAseq创建记录数据。这些记录没有直接使用一代的基因模型,但用于人工管理一步作为潜在的替代文本。的软件,我们首先映射Hisat2版本2.1.0的读取74年使用默认设置为链序列库和下游转录分析。
基因注释
我们使用了奥古斯都版本3.2.375年创建基因模型使用从RNAseq数据提供提示和蛋白质数据从其他鸟类。RNAseq数据,我们将读取映射到装配使用明星2.7.9a版本76年。附件的脚本在奥古斯都包是用来过滤比对搭配和独特的映射读取和提取基因内区提示。我们另外生成覆盖率为每个链假发文件过滤使用软件stranded-coverage对齐文件(https://github.com/pmenzel/stranded-coverage)8月wig2hints和使用这些作为输入。pl生成exonpart提示。
同源性的证据,我们下载一组从NCBI鸟蛋白质(https://www.ncbi.nlm.nih.gov/)。从41214年鸡,这个数据集包括49673蛋白质蛋白质从斑胸草雀从大山雀和38619蛋白质。我们也从Uniprot(下载额外的数据集www.uniprot.org),3175年由手动审查鸟儿蛋白质和204年和12263年的蛋白质没有手动审查但由蛋白质或记录数据,分别。蛋白质数据映射到基因组使用开脱版本测试盒框77年。我们使用脚本align2hints。pl从制动器2.1.678年生成CDSpart基因内区,启动和停止从数据暗示。
奥古斯都和特有的运行参数(参见下面的训练奥古斯都)和默认设置除了指定“softmasking = true”,“——alternatives-from-evidence = true”,“——UTR =”,“——gff3 =”和”——allow_hinted_splicesites = atac”。在外部配置文件中,我们改变了内含子的马吕斯从0.34到0.001,这增加了预测的点球内含子所不支持的外部数据(RNAseq和蛋白质提示)。28491个基因和35389年预测结果记录。
Augustus-derived基因模型名称基于重叠与synteny-transferred斑胸草雀的基因。为了这个目的,我们使用与默认设置SatsumaSynteny获得全基因组比对我们的组装和斑胸草雀之间基因组bTaeGut1.4.pri版本79年。对齐的基础上,我们使用巨妖80年下载(2020-04-14)把斑胸草雀基因组注释(NCBI释放106)柳树莺组装。然后我们提取的奥古斯都的cd基因模型和北海巨妖bedtools相交量化使用基因和重叠。基因模型也被搜索的最长翻译每一个鸡,斑胸草雀和大山雀大山雀基因作为证据用于基因预测的步骤和使用blastp 2.5.0 + 86131 swissprot脊椎动物蛋白质81年的E值阈值1 E−5。基因通过同线性模型,没有注释指定一个基因名称基于爆炸的结果。基因的蛋白质域模型与interproscan v 5.30 - -69.0注释82年。减少假阳性的数量预测我们移除不支持5697个基因,通过同线性斑胸草雀基因,显示没有明显的相似之处脊椎动物蛋白质或不包含任何注释蛋白质域。
我们使用Webapollo 2.6.583年手动牧师基因模型在先前确定不同的染色体区域和其他区域差异。管理步骤,我们专门验证支持编码序列和UTR也删除基因可能是基于截断伪基因编码序列同源基因在其它脊椎动物相比,没有同线性的支持在其他鸟类和/或位于repeat-rich地区。
培训奥古斯都
我们使用之前repeat-masked短内容组装13和削减RNAseq数据用于这项研究获得奥古斯都特有的参数。RNAseq数据组装成记录使用2.0.2三一版本84年创建一个新创和genome-guided组装在一起组成的1929396记录。genome-guided成绩单大会是基于RNAseq映射到基因组使用GSNAP版本2016-07-1185年使用默认设置。我们使用2.0.2 PASA版本86年创建高质量的成绩单,导入Webapollo。评估记录的完整性,我们比他们synteny-transferred模型使用巨妖从鸡的基因组。我们选择1249年完整成绩单出现,不与其他基因重叠,显示不到80%的氨基酸相似性到另一个训练集,从这组基因,我们排除了21个基因给初始训练错误,给了我们一个训练集的1228个基因。这组基因随机分成1028个训练基因和200个基因用于测试。培训中,我们使用optimize_augustus。pl脚本with default settings except for the flag –UTR = on.
全基因组重测序和变体
从九个样品我们使用全基因组重测序数据的迁移表型中提供Lundberg et al13。和测序一个额外的两个高覆盖率样本每个迁移表型(补充表4)。测序新样本库准备与TruSeq DNA PCR-Free工具包(Illumina公司)与目标插入670个基点的大小或TruSeq DNA纳米(Illumina公司)与目标插入350个基点的大小。所有的新样本测序HiSeqX (Illumina公司)。0.36原始读取与trimmomatic修剪87年参数“ILLUMINACLIP: TruSeq3-PE-2。费尔南多-阿隆索:2:30:10领先:3落后:3 SLIDINGWINDOW: 15 MINLEN: 30”。
Quality-trimmed读取被映射到南部组装使用bwa mem和默认设置除了指定- m标志与下游重复删除步骤以确保兼容性和转换成二进制使用samtools对齐地图(bam)文件。样品测序跨多个通道,读取每个车道被独立映射和由此产生的bam文件与samtools合并。读副本被picardtools markduplicates工具提供。
一致的全基因组重测序的数据集,我们称为变体freebayes v1.1.0使用默认设置和并行分析使用GNU平行独立的支架88年。Vcflib版2017-04-0489年用于过滤器原始组变异对网站质量分数> 30和交替等位基因被至少一个支持阅读每个链(SAF > 0 & SAR > 0)和至少一个阅读平衡向左和向右(RPL > 0 & RPR > 0)。接下来,我们使用vcftools 0.1.1690年筛选基因型的覆盖率至少5 x和删除网站最多四个基因型失踪的人口。倒塌的变体也过滤重复通过删除网站平均中值两倍以上的报道意味着覆盖(30×)。我们接下来用vcflib分解单体型电话和复等位基因indels和snp和删除任何变体与注释重复重叠。这给了我们一个最后的5100万个变异4500万人bi-allelic snp。我们使用vcftools计算F圣91年为每一个变体和bi-allelic snp的重叠窗口10 kb。尽可能多的罕见变异在柳树莺种群隔离,这可能向下偏差微分估计92年与未成年人,我们专注于变异等位基因频率至少0.1。
为每个重新测序样本计算重叠覆盖1 kb windows正确使用bedtools,只包括配对阅读与映射至少1的质量。每个样本的原始报道值归一化的值覆盖所有窗户。
调用结构变体
我们使用del 0.9.1的组合93年和图像电传机第2.7.494年打电话给重新测序样品的结构变异。确定一组高信心变异,我们首先绘制了长读取从柳树莺北部到南部组装使用迷你地图2.22 -r110156Pacbio读取和使用默认设置的校准使用del称为变异。接下来,使用图像电传机del变异的基因型重新测序样品包含不同的染色体区域的支架。原始的变体被过滤集只包含网站“通过”国旗,对于每一个变体,聚合的基因型,即基因型模型的断点基因分型结果比对和覆盖率最高的质量,被选为下游分析。遗传分化(F圣)计算vcftools F和变异圣≥0.7之间,比如在每个不同的染色体区域提取和检查使用bedtools重叠的基因和基因特性。得到更可靠的微分估计,我们只包括地点至少80%的南部和北部该等位基因型。
反演重新测序样品的基因型
重新测序样本分配一个基因型的南部和北部单为每个不同的地区基于多维标度在invclust (MDS)的聚类95年Lundberg et al的SNP基因型数组。13。获得基因的单核苷酸多态性包含在数组重新测序样品,我们绘制了SNP阵列探测器序列的组装使用gmap北部和比对提取焦点SNP的位置。接下来,我们使用freebayes基因型的重新测序样品位置和叮铃声1.9版96年结合基因型和基因型的SNP数组。基因分型结果的步骤中,我们还包括映射10×铬北部和南部的库参考样本和额外的柳树莺样本。从合并后的数据集,我们提取的基因型为单核苷酸多态性位于每个不同区域和使用invclust分配每个样本的基因型倒和non-inverted单体型。倒和non-inverted单根据被记录为南方或北方单体型频率在每一个亚种。
断点分析
我们使用伶人4.0.0rc197年使南部和北部的基因组柳树莺、和南方柳树棕柳莺莺基因组的基因组,斑胸草雀(第3.2.4)和成卷的捕蝇草FicAlb (1.5)98年。
提供断点的进一步证据,我们绘制了10×铬读取每个样本的北部和南部组装和使用长期wgs管道称为结构变异。南部的基因组,我们选择了499大支架,连接到一个支架,使其兼容软件。我们还检查样本之间的差异与阅读分子覆盖。为此,每个样本的原始读取第一次处理长期基本质量削减和条形码处理。修剪读取映射到程序集使用bwa mem使用- c标记提取每个阅读和对齐的条形码信息转换成使用samtools bam文件。估计的条形码,我们首先从tigmint 1.1.2 tigmint-molecule脚本运行99年获取位置信息的条形码(分子)在每个不同的地区。软件运行与除了只使用默认设置读取映射至少1,只有质量报告分子估计至少10 kb。我们接下来用bedtools计算重叠的分子的数量1 kb的窗户。
我们探索光学地图使用runSV之间的区别。py脚本与南部bionano解决光学映射作为查询和北部大会目标和相互分析与北方光学地图查询和南部组装作为目标。我们也使用了bionano解决混合装配管道光地图可视化差异和基因组装配在断点的地区。
不同的功能注释
我们使用bedtools量化断点区间和注释的基因之间的距离。提供一个功能注释的SNPs和短indels,我们选择变异显示F圣≥0.7的南部与北部的消费者之间,该地区和使用这些作为输入Snpeff 5.0.0eOne hundred.与基因组注释和参考。我们使用Snpsift 5.0.0e101年选择变量,预测有中度到高影响的基因。基因本体术语提取基因的同源基因在其他鸟类基因组在运用(www.ensembl.org)或通过与interproscan域搜索的蛋白质。
年龄估计和人口统计分析不同地区
为了估计的时间反转事件,我们使用高覆盖率从南部两个重测序数据样本,两个样品和北部,作为外围集团,一个忧郁的莺中国东北部fuscatus(补充表4)。柳树莺样本选择,他们纯合子的南方或北方的三个不同的地区。昏暗的莺图书馆准备使用TruSeq纳米DNA库准备包Neoprep (Illumina公司)根据制造商的指示和测序HiSeq X (Illumina公司)。Quality-trimming修剪读取原始读取和映射的北方参考基因组都遵循着相同的方法用于柳树莺重测序样品(见上图)。
变体被称为使用freebayes和变异的原始设置过滤使用平衡台的预处理模块(v0.6.0)。Sample-specific可调用的网站被确定使用平衡台预处理和被定义为那些最小的报道最多8×0.75个标准差范围。基因和基因组的重复区域从调用删除网站为了限制下游分析基因间区域。
摘要遗传变异(π和统计dxy计算在不同地区使用。在这之后,净散度(d一个)之间的北部和南部样本计算d北,南−(π北+π南)/ 2。将净散度转化为年我们使用生殖系基因突变率(4.6×10−9)估计有领霸鹟科21。相对节点深度(RND)使用昏暗的莺(DW)作为外群计算d南北/ (dDW-north+ dDW-south)/ 2。为每个不同的地区,一个块网站频谱(bsf)生成平衡台使用块长度的64个基点。这个长度是指可调用的网站在一个块的数量,而物理块的长度被允许改变由于缺失的数据,但仅限于128个基点。下游分析依赖于生物沙子饮用水过滤系统使用的kmax 2,也就是说只有边际概率计算突变数> 2。复合的可能性(CL)的一个模型,给出不同的地区之一的生物沙子饮用水过滤系统,优化使用Nelder-Mead算法与最大迭代次数设置为1000。在软件中,我们评估了三种不同的人口模型。第一个模型是一个严格的隔离模型(SI),与参数的有效的人口规模,有效种群大小南部和北部柳树莺和分歧时间。第二个模型是一个隔离与迁移模型(IM1),还包括从北部到南部迁移率样本,第三个模型(IM2)而不是有一个迁移率从南部到北部柳树莺。
模拟是由msprime 0.7.4102年通过平衡台。这些模拟使用的复合率chromosome-specific估计高密度复合钻头定位捕蝇草的地图98年,2.04,1.95,和2.63厘米/ Mb染色体1,3,5,分别。总共100复制模拟如果每个地区的参数进行了优化。这些模拟生物沙子饮用水过滤系统优化的一个SI模型下以及最适合该地区的IM模型。CL这些模型之间的改善作为零分布测试改进CL观察到的实际数据是否大于预期的历史没有迁移。对于每个参数,我们计算95%置信区间为(制程)估计可能性最大复合±1.96 *标准差的模拟(补充表7)。因此,我们估计的不确定性是影响复合率,我们假设模拟。我们还利用模拟的结果量化制程的潜在偏差估计由于intra-block重组(补充表7)。然而,我们没有试图纠正这种偏见是相对较小(如制程分歧时间估计偏见向上7,24岁和10%)和我们的估计偏差本身在很大程度上是依赖于复合利率我们假设。
MSMC224被用来探索基因组N的变化e通过时间。作为软件的输入,我们使用了可调用的基因间的床上文件和过滤的vcf文件上面所提到的,进一步的过滤床文件只包含常染色体支架≥500 kb和排除不同的区域。MSMC2的输入文件,即,一个n unphased set of heterozygous sites for each sample, were generated using the generate_multihetsep.py script from msmc-tools. MSMC2 was run with a starting ρ/μ of 1 for 30 expectation-maximum iterations. For both the demographic modeling and MSMC2, we used the collared flycatcher germline mutation rate21和一代时间1.7年11将分歧时间转化为年。
来推断人口事件和选择的影响,我们还计算一些基因汇总统计。为此,我们首先估算缺失基因型和推断单过滤的变异使用小猎犬号版本5.4103年。全套的样品,我们选择了10个和7个样品纯合子的南方或北方三个不同的区域,分别确定MDS的分析(见上图),并提取bi-allelic snp。确定祖先和衍生等位基因,我们提取基因型的焦SNP的位置对齐棕柳莺和昏暗的莺读取使用bcftools 1.1462年mpileup命令。作为一个保守的方法,我们认为任何网站的参考和备选等位基因杂合的(不管他们的频率)和只包括报道地点至少三分之一的意思是覆盖在每个外围集团的所有网站的物种。我们下一个使用一个定制的脚本从最初的vcf文件,提取网站,如果有必要,开关和候补等位基因和交换基因型的引用。与极化基因型数据,我们使用PopGenome 2.7.5104年计算费和吴邦国委员长的H和vcftools衍生等位基因的数量。我们进一步用1.3.0 selscan版本105年计算XP-nsl106年南部和北部之间的样本,Sweepfinder2107年计算复合似然比(CLR)模型,选择扫描之间产生了影响基于全基因组等位基因的等位基因频率和模型频谱和使用vcftools计算核苷酸多样性,日本田岛的D和连锁不平衡(D)。
南部组装作为参考的使用可能会导致一个映射偏见从南部读取样本,特别是在地区高等亚种之间的分歧。反过来,这可能会影响基因的汇总统计和人口统计建模估计。探索参考偏见的影响,我们也因此重测序数据映射到北部组装、变体进行呼叫和计算核苷酸多样性和日本田岛的D 10 kb窗口。北部的组装,我们也使用相同的人口统计建模用于南方组装。对比平均遗传汇总统计和人口统计参数估计,我们发现微不足道的差异两个基因组装配(补充表10)。
报告总结
进一步研究信息设计是可用的自然投资组合报告总结与这篇文章有关。
数据可用性
原始序列数据、光学地图和新创组件生成的在这个研究是在NCBI bioproject之下PRJNA550489。使用全基因组重测序数据从先前的研究在NCBI bioproject之下PRJNA319295。图源数据和注释文件可用Figshare (https://doi.org/10.6084/m9.figshare.21821328.v1)。
代码的可用性
工作流和脚本可以在Github (https://github.com/maxlundberg/warbler_inversions)108年。
引用
Wellenreuther, m & Bernatchez l . Eco-evolutionary基因组染色体倒置。生态发展趋势。另一个星球。33,427 - 440 (2018)。
汤普森,m . j . & Jiggins c d表生的和他们在进化中的作用。遗传113年,1 - 8 (2014)。
Lamichhaney, et al。结构基因组变化背后另类生殖策略的飞边(Philomachus喜斗蜿)。Nat,麝猫。48,84 - 88 (2016)。
塔特尔,e . m . et al .散度和功能退化性染色体和表生的。咕咕叫。医学杂志。:CB26,344 - 350 (2016)。
王,j . et al . y染色体导致替代殖民地社会组织在火蚁。自然493年,664 - 668 (2013)。
Kirubakaran, t . g . et al。两个相邻逆序对维持基因组分化之间的迁徙和静止的生态型大西洋鳕鱼。摩尔。生态。https://doi.org/10.1111/mec.13592(2016)。
皮尔斯发起,d . e . et al . Sex-dependent优势维持迁移表生的虹鳟鱼。Nat,生态。另一个星球。https://doi.org/10.1038/s41559 - 019 - 1044 - 6(2019)。
洛格斯登,g。,Vollger, M. R. & Eichler, E. E. Long-read human genome sequencing and its applications.Nat,启麝猫。21,597 - 614 (2020)。
爬坡,a &佩特森,j .迁徙路线和越冬的柳树莺领域。Ornis Fennica64年,137 - 143 (1987)。
Bensch, S。,Bengtsson, G. & Åkesson, S. Patterns of stable isotope signatures in willow warbler Phylloscopus trochilus feathers collected in Africa.j .鸟类杂志。37,323 - 330 (2006)。
Bensch, S。,一个ndersson, T. & Åkesson, S. Morphological and molecular variation across a migratory divide in willow warblers Phylloscopus trochilus.另一个星球。Int。j . Org。另一个星球。53,1925 - 1935 (1999)。
Bensch, S。,Grahn, M., Müller, N., Gay, L. & Åkesson, S. Genetic, morphological, and feather isotope variation of migratory willow warblers show gradual divergence in a ring.摩尔。生态。18,3087 - 3096 (2009)。
Lundberg, m . et al。柳树莺迁移表型之间的遗传差异是几个大单体型和集群。另一个星球。列托人。1,155 - 168 (2017)。
Lohse, K。,Chmelik, M., Martin, S. H. & Barton, N. H. Efficient strategies for calculating blockwise likelihoods under the coalescent.遗传学202年,775 - 786 (2016)。
Lohse, K。,Clarke, M., Ritchie, M. G. & Etges, W. J. Genome-wide tests for introgression between cactophilic Drosophila implicate a role of inversions during speciation.另一个星球。Int。j . Org。另一个星球。69年,1178 - 1190 (2015)。
Bensch, S。,Irwin, D. E., Irwin, J. H., Kvist, L. & Akesson, S. Conflicting patterns of mitochondrial and nuclear DNA diversity in Phylloscopus warblers.摩尔。生态。15,161 - 171 (2006)。
菲德尔,j·L。Gejji, R。,Powell, T. H. & Nosil, P. Adaptive chromosomal divergence driven by mixed geographic mode of evolution.另一个星球。Int。j . Org。另一个星球。65年,2157 - 2170 (2011)。
Sigeman, h . et al .禽流感neo-sex染色体揭示复合抑制动力学和W变性。摩尔。杂志。另一个星球。38,5275 - 5291 (2021)。
Weissensteiner, m . h . et al .短内容、读和光学映射组件显示大规模串联重复序列数组与人口遗传影响。基因组Res。27,697 - 708 (2017)。
李Nei, m & w·h·数学模型研究遗传变异的限制内切酶。Proc。《科学。美国76年,5269 - 5273 (1979)。
Smeds, L。,Qvarnstrom, A. & Ellegren, H. Direct estimate of the rate of germline mutation in a bird.基因组Res。26,1211 - 1218 (2016)。
菲德尔,j . l . et al .娃提出警告,布什和Rhagoletis同域物种形成的复杂性。Proc。《科学。美国102年,6573 - 6580 (2005)。
Laetsch, d . r . et al .人口基因流动壁垒使用显式扫描。预印在bioRxivhttps://doi.org/10.1101/2022.10.27.514110(2022)。
Schiffels s & Wang K。统计人口基因组学(ed Dutheil j.y.) 147 - 166 (Springer, 2020)。
Caballero-Lopez, V。Lundberg, M。,年代okolovskis, K. & Bensch, S. Transposable elements mark a repeat-rich region associated with migratory phenotypes of willow warblers (Phylloscopus trochilus).摩尔。生态。31日,1128 - 1141 (2022)。
Alstrom、p . et al .完整了解发展史的柳莺(鸟类:Phylloscopidae)辐射。摩尔。Phylogenet。另一个星球。126年,141 - 152 (2018)。
柯克帕特里克,m &巴顿:染色体倒置,当地的适应和物种形成。遗传学173年,419 - 434 (2006)。
马瑟,N。,Traves, S. M. & Ho, S. Y. W. A practical introduction to sequentially Markovian coalescent methods for estimating demographic history from genomic data.生态。另一个星球。10,579 - 589 (2020)。
Joron, m . et al .染色体重组保持多态表生的控制蝴蝶模仿。自然477年,203 - 206 (2011)。
Calvete, O。,Gonzalez, J., Betran, E. & Ruiz, A. Segmental duplication, microinversion, and gene loss associated with a complex inversion breakpoint region in Drosophila.摩尔。杂志。另一个星球。29日,1875 - 1889 (2012)。
Porubsky, d . et al .复发性反转切换和类人猿进化基因组。Nat,麝猫。52,849 - 858 (2020)。
卡塞雷斯,m .美国国立卫生研究院的校内的测序中心比较排序,P。沙利文,r·t·托马斯& j . w .复发性反转的真兽亚纲动物X染色体。Proc。《科学。美国104年,18571 - 18576 (2007)。
格雷罗州,r F。,Rousset, F. & Kirkpatrick, M. Coalescent patterns for chromosomal inversions in divergent populations.费罗斯。反式。r . Soc。Lond。爵士。B、生物。科学。367年,430 - 438 (2012)。
拉森,k . w . et al .等位变异在柳树与气候渐变群莺基因组区域。《公共科学图书馆•综合》9e95252 (2014)。
纳瓦罗,。,Betran, E., Barbadilla, A. & Ruiz, A. Recombination and gene flux caused by gene conversion and crossing over in inversion heterokaryotypes.遗传学146年,695 - 709 (1997)。
卡瓦略,c . m & Lupski j . r .机制在基因组结构变异的形成障碍。Nat,启麝猫。17,224 - 238 (2016)。
普伊格,M。,Casillas, S., Villatoro, S. & Caceres, M. Human inversions and their functional consequences.简短。功能。染色体组。14,369 - 379 (2015)。
Burstyn-Cohen, t . et al . F-Spondin需要准确的寻路底板的合缝处的轴突。神经元23,233 - 246 (1999)。
Carrillo, g . L。苏,J。,Monavarfeshani, A. & Fox, M. A. F-spondin is essential for maintaining circadian rhythms.前面。神经回路1213 (2018)。
Speliotes, e . k . et al .协会249796个人的分析揭示18新位点与身体质量指数相关。Nat,麝猫。42,937 - 948 (2010)。
,去寻找更多a . et al .脂肪酸代谢的遗传适应:一个人类单体型越来越长链ω- 3和ω- 6脂肪酸的生物合成。点。j .的嗡嗡声。麝猫。90年,809 - 820 (2012)。
巴克利,m . t . et al。选择在欧洲脂肪酸desaturases与饮食相关的变化。摩尔。杂志。另一个星球。34,1307 - 1318 (2017)。
气,y . et al .膜加劲STOML3促进mechanosensation感觉神经元。Commun Nat。68512 (2015)。
没吃,s E。,Dubin, A. E. & Patapoutian, A. Piezos thrive under pressure: mechanically activated ion channels in health and disease.Nat。启摩尔。细胞杂志。18,771 - 783 (2017)。
Liedvogel, m . et al。没有证据表明选型交配在柳树莺迁徙的鸿沟。前面。黑旋风。1152 (2014)。
Lundberg, m . et al .转录组的描述找到序列差异两个不同迁移亚种的柳树莺中国蜂鸟。BMC染色体组。14330 (2013)。
Bensch, S。,Åkesson, S. & Irwin, D. E. The use of AFLP to find an informative SNP: genetic differences across a migratory divide in willow warblers.摩尔。生态。11,2359 - 2366 (2002)。
Lam e . t . et al .基因组映射在纳米通道阵列结构变异分析和装配序列。生物科技Nat。》。30.,771 - 776 (2012)。
下巴,c . s . et al . Nonhybrid完成微生物基因组从读SMRT测序数据组件。Nat方法。10563 (2013)。
下巴,c . s . et al .阶段性二倍体基因组组装单分子实时测序。Nat方法。13,1050 - 1054 (2016)。
西芒,f。,Waterhouse, R. M., Ioannidis, P., Kriventseva, E. V. & Zdobnov, E. M. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs.生物信息学31日,3210 - 3212 (2015)。
沃克,b . j . et al . Pilon:一个集成的全面的微生物基因组变异检测和装配工具的改进。《公共科学图书馆•综合》9e112963 (2014)。
李,h &杜宾,r .快速和准确的短阅读符合burrows - wheeler变换。生物信息学25,1754 - 1760 (2009)。
Bonfield j . k . & Whitwham a Gap5-editing十亿片段序列组装。生物信息学26,1699 - 1703 (2010)。
罗奇,m . J。,年代chmidt, S. A. & Borneman, A. R. Purge Haplotigs: allelic contig reassignment for third-gen diploid genome assemblies.BMC Bioinforma。19460 (2018)。
李,h . Minimap2:核苷酸序列两两对齐。生物信息学34,3094 - 3100 (2018)。
唷,S。,Coombe, L., Warren, R. L., Chu, J. & Birol, I. ARCS: scaffolding genome drafts with linked reads.生物信息学34,725 - 731 (2018)。
沃伦,r . l . et al .链接:可伸缩的,汇票alignment-free脚手架的基因组与读取。GigaScience435 (2015)。
英语,a . c . et al。心灵的差距:升级基因组与太平洋生物科学RS读测序技术。《公共科学图书馆•综合》7e47768 (2012)。
Grabherr, m . g . et al .全基因组通过高度敏感的同线性序列比对:无核小蜜橘。生物信息学26,1145 - 1151 (2010)。
标志、p . et al。解决的全部频谱使用Linked-Reads人类基因组变异。基因组Res。29日,635 - 645 (2019)。
李,h .统计框架SNP的召唤,突变的发现,协会从测序数据映射和人口遗传的参数估计。生物信息学27,2987 - 2993 (2011)。
Bradnam, k . r . et al . Assemblathon 2:评估新创三脊椎动物基因组组装的方法。GigaScience210 (2013)。
程,H。,Concepcion, G. T., Feng, X., Zhang, H. & Li, H. Haplotype-resolved de novo assembly using phased assembly graphs with hifiasm.Nat方法。18,170 - 175 (2021)。
Smit, a & Hubley r . RepeatModeler开放,http://www.repeatmasker.org(2015)。
Smit,。,Hubley, R. & Green, P. RepeatMasker Open-4.0,http://www.repeatmasker.org(2015)。
昆兰,a。r . &大厅,i m . BEDTools:一套灵活的工具来比较基因组的特性。生物信息学26,841 - 842 (2010)。
本森,g .串联重复序列仪:一个程序来分析DNA序列。核酸Res。27,573 - 580 (1999)。
老板,j . et al。大脑中的基因表达的迁徙songbird在繁殖和迁移。Mov。生态。41 - 11 (2016)。
Lundin年代。,年代tranneheim, H., Pettersson, E., Klevebring, D. & Lundeberg, J. Increased throughput by parallelization of library preparation for massive sequencing.《公共科学图书馆•综合》5e10029 (2010)。
Borgstrom E。,Lundin年代。&Lundeberg, J. Large scale library generation for high throughput sequencing.《公共科学图书馆•综合》6e19119 (2011)。
马丁,m . Cutadapt删除适配器序列高。吞吐量测序读。EMBnet J。173 (2011)。
Pertea, m . et al . StringTie使转录组的改善重建RNA-seq读取。生物科技Nat。》。33,290 - 295 (2015)。
金,D。,Paggi, J. M., Park, C., Bennett, C. & Salzberg, S. L. Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype.生物科技Nat。》。37,907 - 915 (2019)。
Stanke, M。,Diekhans, M., Baertsch, R. & Haussler, D. Using native and syntenically mapped cDNA alignments to improve de novo gene finding.生物信息学24,637 - 644 (2008)。
多布林,a . et al .明星:超速普遍RNA-seq对准器。生物信息学29日,15至21 (2013)。
斯莱特,g s &伯尼,大肠自动代启发式生物序列比较。BMC Bioinforma。631 (2005)。
米菲,T。,Hoff, K. J., Lomsadze, A., Stanke, M. & Borodovsky, M. BRAKER2: automatic eukaryotic genome annotation with GeneMark-EP+ and AUGUSTUS supported by a protein database.NAR染色体组。Bioinforma。3lqaa108 (2021)。
对完整、无误(a . et al .基因组的组装所有的脊椎动物。自然592年,737 - 746 (2021)。
Zamani: et al .通用基因协调翻译为比较基因组学。BMC Bioinforma。15227 (2014)。
卡马乔,c . et al .爆炸+:体系结构和应用程序。BMC Bioinformat。https://doi.org/10.1186/1471 - 2105 - 10 - 421(2009)。
琼斯公司p . et al . InterProScan 5:蛋白质功能分类。生物信息学30.,1236 - 1240 (2014)。
李,e . et al . Web阿波罗:一个基于Web的基因组注释编辑平台。基因组医学杂志。14R93 (2013)。
Grabherr, m . g . et al .全身没有参考基因组转录组装配从RNA-Seq数据。生物科技Nat。》。29日,644 - 652 (2011)。
吴,t D。,Reeder, J., Lawrence, M., Becker, G. & Brauer, M. J. GMAP and GSNAP for genomic sequence alignment: enhancements to speed, accuracy, and functionality.摩尔。生物方法。1418年,283 - 334 (2016)。
哈斯,b . j . et al .自动化真核基因结构注释使用EVidenceModeler和程序组装拼接对齐。基因组医学杂志。9R7 (2008)。
博尔格、a . M。,Lohse, M. & Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data.生物信息学30.,2114 - 2120 (2014)。
o . GNU平行Tange——命令行电动工具。登录:USENIX杂志。3642-47 (2011)。
驻军,E。,Kronenberg, Z. N., Dawson, E. T., Pedersen, B. S. & Prins, P. A spectrum of free software tools for processing the VCF variant call format: vcflib, bio-vcf, cyvcf2, hts-nim and slivar.公共科学图书馆第一版。医学杂志。18e1009123 (2022)。
Danecek, p . et al .变体调用格式和VCFtools。生物信息学27,2156 - 2158 (2011)。
堰,b s & Cockerham c . c .估算统计量分析人口结构。另一个星球。Int。j . Org。另一个星球。38,1358 - 1370 (1984)。
Roesti, M。,年代一个lzburger, W. & Berner, D. Uninformative polymorphisms bias genome scans for signatures of selection.BMC另一个星球。医学杂志。1294 (2012)。
Rausch t . et al . del:通过集成paired-end和split-read分析发现结构变体。生物信息学28I333-I339 (2012)。
Eggertsson,惠普et al . GraphTyper2使人口规模的基因结构变异使用pangenome图表。Commun Nat。105402 (2019)。
卡塞雷斯,a &冈萨雷斯,j . r .多态对SNP数据反演的足迹:从检测协会测试。核酸Res。43e53 (2015)。
珀塞尔,s . et al .叮铃声:全基因组协会的工具集,基于链接分析。点。j .的嗡嗡声。麝猫。81年,559 - 575 (2007)。
Marcais g . et al . MUMmer4:快速、通用的基因组比对系统。公共科学图书馆第一版。医学杂志。14e1005944 (2018)。
Kawakami (t . et al .高密度连锁图使第二代成卷的捕蝇草基因组组装和揭示了禽流感的模式重组率变异和染色体进化。摩尔。生态。23,4035 - 4058 (2014)。
杰克曼,s . d . et al . Tigmint:纠正使用链接读取从大分子组装错误。BMC Bioinforma。19393 (2018)。
Cingolani, p . et al .程序注释和预测单核苷酸多态性的影响,SnpEff:黑腹果蝇的基因组应变w1118单核苷酸多态性;iso-2;iso-3。飞6,80 - 92 (2012)。
Cingolani, p . et al。使用黑腹果蝇作为模式基因毒性化学突变研究一个新项目,SnpSift。前面。麝猫。335 (2012)。
凯莱赫,J。,Etheridge, A. M. & McVean, G. Efficient coalescent simulation and genealogical analysis for large sample sizes.公共科学图书馆第一版。医学杂志。12e1004842 (2016)。
布朗宁,b . L。田,X。,Zhou, Y. & Browning, S. R. Fast two-stage phasing of large-scale sequence data.点。j .的嗡嗡声。麝猫。108年,1880 - 1890 (2021)。
具有B。,Wittelsburger, U., Ramos-Onsins, S. E. & Lercher, M. J. PopGenome: an efficient Swiss army knife for population genomic analyses in R.摩尔。杂志。另一个星球。31日,1929 - 1936 (2014)。
Szpiech z, a &埃尔南德斯,r·d·selscan:一个高效的多线程程序执行EHH-based积极选择的扫描。摩尔。杂志。另一个星球。31日,2824 - 2827 (2014)。
Szpiech, z。诺瓦克,t·E。,B一个iley, N. P. & Stevison, L. S. Application of a novel haplotype-based scan for local adaptation to study high-altitude adaptation in rhesus macaques.另一个星球。列托人。5,408 - 421 (2021)。
DeGiorgio, M。,Huber, C. D., Hubisz, M. J., Hellmann, I. & Nielsen, R. SweepFinder2: increased sensitivity, robustness and flexibility.生物信息学32,1895 - 1897 (2016)。
Lundberg, M。,Mackintosh, A., Petri, A. & Bensch, S. Inversions maintain differences between migratory phenotypes of a songbird.https://github.com/maxlundberg/warbler_inversions,https://doi.org/10.5281/zenodo.7543037(2023)。
确认
我们要感谢康拉德Lohse和Lars Raberg评论手稿,莎拉Kingan讨论Pacbio组件的高度杂合的物种,以及Gertjan Bisschop和杜米尼克r . Laetsch平衡台分析的建议。这项研究是支持由皇家Physiograhic学会隆德马丁和由瑞典研究理事会(2017 - 03937)和Crafoord基金会(2017 - 0567)S.B.作者要感谢国家基因组的支持基础设施(进行下一代NGI)在乌普萨拉(乌普萨拉基因组中心/ SNP&SEQ技术平台)和在斯德哥尔摩。工作在进行下一代NGI由RFI / VR和生命的科学实验室,瑞典。计算和数据处理是通过计算资源提供的瑞典国家基础设施(SNIC) UPPMAX,部分由瑞典研究理事会通过赠款协议。2018 - 05973。
资金
隆德大学提供的开放获取资金。
作者信息
作者和联系
贡献
马丁和S.B.构思。马丁基因组组装和注释,执行断点分析,分析了重测序数据和写的手稿与其他作者的输入。点人口进行建模。美联社进行样本提取,生成和分析光学地图和基因组组装完成。
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
同行评审
同行审查的信息
自然通讯感谢匿名评论者对他们的贡献的同行评审工作。
额外的信息
出版商的注意施普林格自然保持中立在发表关于司法主权地图和所属机构。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。
关于这篇文章
引用这篇文章
Lundberg, M。,Mackintosh, A., Petri, A.et al。反演保持迁徙songbird的表型差异。Nat Commun14452 (2023)。https://doi.org/10.1038/s41467 - 023 - 36167 - y
收到了:
接受:
发表:
DOI:https://doi.org/10.1038/s41467 - 023 - 36167 - y