摘要gydF4y2Ba
在自然环境中检测内源性信号和精确控制遗传回路对于理解生物过程至关重要。然而,处理内生信息的工具是有限的。在这里,我们开发了一种可推广的内源性转录门控开关,在内源性启动子存在时释放单导向rna。当内源性转录门控开关与我们开发的高灵敏度crispr激活剂相关报告相结合时,我们可以可靠地检测内源性基因的活性,包括表达非常低的基因(相对于gydF4y2BaGapdhgydF4y2Ba;定量pcr分析)。值得注意的是,我们还可以使用这种方法监测活细胞中低水平表达的典型长非编码rna的转录活性。总之,我们的方法提供了一个强大的平台来感知潜在的细胞功能的内源性遗传元素的活动。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
内含子编码的顺激子转录本用于编码和非编码rna的微创监测gydF4y2Ba
自然细胞生物学gydF4y2Ba开放获取gydF4y2Ba11月7日gydF4y2Ba
通过合成CRISPRa电路精确的肿瘤免疫重布线,同时获得/失去转录因子gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2022年3月18日gydF4y2Ba
长链非编码RNA PAARH通过上调HOTTIP和激活HIF-1α/VEGF信号通路促进肝细胞癌进展和血管生成gydF4y2Ba
细胞死亡与疾病gydF4y2Ba开放获取gydF4y2Ba2022年2月2日gydF4y2Ba
访问选项gydF4y2Ba
订阅《自然》+gydF4y2Ba
立即在线访问《自然》和其他55种《自然》杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅期刊gydF4y2Ba
获得1年的完整期刊访问权限gydF4y2Ba
99.00美元gydF4y2Ba
每期只要8.25美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
增值税稍后将在结帐时添加。gydF4y2Ba
税务计算将在结账时完成。gydF4y2Ba
买条gydF4y2Ba
在ReadCube上获得时间限制或全文访问。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
数据可用性gydF4y2Ba
在此重新分析的先前发表的RNA测序数据可在登录代码下获得gydF4y2BaGSM2573084gydF4y2Ba.所有与数字相关的原始数据都列在源数据(统计源数据)中。所有western blot的原始图像都可以在Source数据中找到(未处理的western blot和凝胶)。所有向量的序列在补充图中提供。gydF4y2Ba1gydF4y2Ba.所有其他材料和数据均可按要求提供。gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
参考文献gydF4y2Ba
格林,A. A.,西尔弗,P. A.,柯林斯,J. J. &尹,P.立足点开关:基因表达的de-novo设计调节器。gydF4y2Ba细胞gydF4y2Ba159gydF4y2Ba, 925-939(2014)。gydF4y2Ba
刘,Y.等。通过CRISPR信号导体引导细胞信息流。gydF4y2BaNat方法。gydF4y2Ba13gydF4y2Ba, 938-944(2016)。gydF4y2Ba
格林,a.a.等。利用核计算设备进行复杂的细胞逻辑计算。gydF4y2Ba自然gydF4y2Ba548gydF4y2Ba, 117-121(2017)。gydF4y2Ba
Hirosawa, M.等。利用microrna响应性CRISPR-Cas9开关进行细胞类型特异性基因组编辑。gydF4y2Ba核酸测定。gydF4y2Ba45gydF4y2Ba, e118(2017)。gydF4y2Ba
Siu K. H. & Chen W.核调控的点控gRNA用于可编程CRISPR-Cas9功能。gydF4y2BaNat,化学。医学杂志。gydF4y2Ba15gydF4y2Ba, 217-220(2019)。gydF4y2Ba
王晓武等。microRNA诱导的CRISPR-Cas9平台作为microRNA传感器和细胞类型特异性基因组调控工具。gydF4y2Ba细胞生物学。gydF4y2Ba21gydF4y2Ba, 522-530(2019)。gydF4y2Ba
Miki, K.等。利用合成microRNA开关高效检测和纯化细胞群。gydF4y2Ba细胞干细胞gydF4y2Ba16gydF4y2Ba, 699-711(2015)。gydF4y2Ba
徐鹏东,李丽娟,张峰。CRISPR-Cas9基因工程的研究进展及应用。gydF4y2Ba细胞gydF4y2Ba157gydF4y2Ba, 1262-1278(2014)。gydF4y2Ba
姚,X.等。同源性介导的基于末端连接的CRISPR/Cas9靶向整合。gydF4y2Ba细胞Res。gydF4y2Ba27gydF4y2Ba, 801-814(2017)。gydF4y2Ba
Mikuni, T., Nishiyama, J., Sun, Y., Kamasawa, N.和Yasuda, R.通过体内基因组编辑在哺乳动物大脑中蛋白质定位的高通量、高分辨率定位。gydF4y2Ba细胞gydF4y2Ba165gydF4y2Ba, 1803-1817(2016)。gydF4y2Ba
西山,J., Mikuni, T. & Yasuda, R.病毒介导的基因组编辑通过同源定向修复在哺乳动物大脑有丝分裂和有丝分裂后细胞。gydF4y2Ba神经元gydF4y2Ba96gydF4y2Ba, 755-768(2017)。gydF4y2Ba
古特曼,M. &里恩,J. L.大型非编码rna的模块化调控原理。gydF4y2Ba自然gydF4y2Ba482gydF4y2Ba, 339-346(2012)。gydF4y2Ba
Kretz, M.等。长链非编码RNA TINCR对体细胞组织分化的控制。gydF4y2Ba自然gydF4y2Ba493gydF4y2Ba, 231-235(2013)。gydF4y2Ba
索瓦格等人。多个敲除小鼠模型揭示lincRNAs是生命和大脑发育所必需的。gydF4y2BaeLifegydF4y2Ba2gydF4y2Ba, e01749(2013)。gydF4y2Ba
贝斯特,a.c.等。一种整合的全基因组CRISPRa方法在耐药中功能化lncrna。gydF4y2Ba细胞gydF4y2Ba173gydF4y2Ba, 649-664(2018)。gydF4y2Ba
Cabili, M. N.等。人类lncrna在单细胞和单分子分辨率下的定位和丰度分析gydF4y2Ba基因组医学杂志。gydF4y2Ba16gydF4y2Ba, 20(2015)。gydF4y2Ba
陈,L.等。mrna与lncrna的组织表达差异。gydF4y2BaInt。理学。gydF4y2Bahttps://doi.org/10.3390/ijms19113416gydF4y2Ba(2018)。gydF4y2Ba
阿兹兰,A, Obeidat, s.m.,尤努斯,M. A. &阿扎姆,G.系统识别和表征gydF4y2Ba埃及伊蚊gydF4y2Ba长非编码rna (lncRNAs)。gydF4y2Ba科学。代表。gydF4y2Ba9gydF4y2Ba, 12147(2019)。gydF4y2Ba
周,H.等。使用crispr - dcas9激活剂转基因小鼠在体内同时转录激活大脑中的多个基因。gydF4y2BaNat。>。gydF4y2Ba21gydF4y2Ba, 440-446(2018)。gydF4y2Ba
王,J.等。通过RNA聚合酶II启动子表达CRISPR系统的sgRNA,产生细胞类型特异性基因突变。gydF4y2Ba蛋白细胞gydF4y2Ba6gydF4y2Ba, 689-692(2015)。gydF4y2Ba
布莱克,d.l.选择性前信使RNA剪接机制。gydF4y2Ba为基础。学生物化学启。gydF4y2Ba72gydF4y2Ba, 291-336(2003)。gydF4y2Ba
马特林,a . J.克拉克,F. &史密斯,C. W.理解可选剪接:朝向蜂窝代码。gydF4y2Ba细胞生物学。gydF4y2Ba6gydF4y2Ba, 386-398(2005)。gydF4y2Ba
谢凯,明肯伯格,杨旸。利用内源性trna处理系统提高CRISPR/Cas9多重编辑能力。gydF4y2Ba国家科学院学报美国gydF4y2Ba112gydF4y2Ba, 3570-3575(2015)。gydF4y2Ba
张,D.等。完美匹配的20个核苷酸引导RNA序列可以使用高保真SpCas9核酸酶进行稳健的基因组编辑。gydF4y2Ba基因组医学杂志。gydF4y2Ba18gydF4y2Ba, 191(2017)。gydF4y2Ba
Nissim, L., Perli, S. D., Fridkin, A., Perez-Pinera, P. & Lu, T. K.在人类细胞中使用集成的RNA和CRISPR/Cas工具包对基因网络进行多路和可编程调控。gydF4y2Ba摩尔。细胞gydF4y2Ba54gydF4y2Ba, 698-710(2014)。gydF4y2Ba
应Q. L., Stavridis, M., Griffiths, D., Li M. & Smith, A.胚胎干细胞在粘附单培养中转化为神经外胚层前体。gydF4y2Ba生物科技Nat。》。gydF4y2Ba21gydF4y2Ba, 183-186(2003)。gydF4y2Ba
张志华,陆玉云,岳俊。两个孔通道2对小鼠胚胎干细胞神经分化的差异调节。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba8gydF4y2Ba, e66077(2013)。gydF4y2Ba
王派布瓦塔纳,W. & Stavridis, M. P.小鼠胚胎干细胞在无血清单层培养中的神经分化。gydF4y2BaJ. Vis. Exp。gydF4y2Bahttps://doi.org/10.3791/52823gydF4y2Ba(2015)。gydF4y2Ba
Nair, G.,分支,E., Guedes, A. M., Henrique, D. & Raj, A.原生胚胎干细胞中异质谱系标记的表达主要是由于自发分化。gydF4y2Ba科学。代表。gydF4y2Ba5gydF4y2Ba, 13339(2015)。gydF4y2Ba
Neri, F.等人。在ESCs中TET1受多能性相关因子控制,在分化的细胞和组织中被PRC2下调。gydF4y2Ba核酸测定。gydF4y2Ba43gydF4y2Ba, 6814-6826(2015)。gydF4y2Ba
郭,F.等。小鼠早期胚胎和胚胎干细胞的单细胞多组学测序。gydF4y2Ba细胞Res。gydF4y2Ba27gydF4y2Ba, 967-988(2017)。gydF4y2Ba
拉莫斯,a.d.等人。整合全基因组方法在体内鉴定成体神经干细胞及其子代的lncrna。gydF4y2Ba细胞干细胞gydF4y2Ba12gydF4y2Ba, 616-628(2013)。gydF4y2Ba
刘,s.j.等。发育中的人类新皮层长链非编码rna的单细胞分析。gydF4y2Ba基因组医学杂志。gydF4y2Ba17gydF4y2Ba, 67(2016)。gydF4y2Ba
Salviano-Silva, A, Lobo-Alves, S. C, Almeida, R. C, Malheiros, D. & Petzl-Erler, M. L.除了病理学:细胞和组织稳态中的长链非编码RNA。gydF4y2Ba非编码RNAgydF4y2Bahttps://doi.org/10.3390/ncrna4010003gydF4y2Ba(2018)。gydF4y2Ba
孙,Z.等。长链非编码RNAgydF4y2BaLncenc1gydF4y2Ba通过促进糖酵解途径维持小鼠ESCs的幼稚状态。gydF4y2Ba干细胞代表gydF4y2Ba11gydF4y2Ba, 741-755(2018)。gydF4y2Ba
刘,Y.等。CRISPR激活屏幕系统地识别驱动神经元命运和重编程的因素。gydF4y2Ba细胞干细胞gydF4y2Ba23gydF4y2Ba, 758-771(2018)。gydF4y2Ba
查韦斯等人。高效cas9介导的转录编程。gydF4y2BaNat方法。gydF4y2Ba12gydF4y2Ba, 326-328(2015)。gydF4y2Ba
高宁,胡娟,周慧,杨华。一种生成SPH-OminiCMV-Ents mESCs的协议。gydF4y2BaProtoc。ExchgydF4y2Ba.gydF4y2Bahttps://doi.org/10.21203/rs.3.pex-1273/v1gydF4y2Ba(2020)。gydF4y2Ba
确认gydF4y2Ba
我们感谢ION的FACS设施的Quan L., Wu H.和Qian S.,以及光学成像设施的Wang Y., Zhang Y., Chen X., Xiang D.和Q. Hu。我们感谢钟n和王q的技术援助。本工作得到了中国科学院基础前沿科学研究计划从0到1原始创新项目(批准号:;ZDBS-LY-SM001),中国研发计划(批准号2017YFC1001300和2018YFC2000100),中科院战略重点研究计划(批准号:ZDBS-LY-SM001)。国家自然科学基金项目(批准号:31871502,31925016,91957122,31901047),上海市科技重大专项(批准号:XDB32060000);2018SHZDZX05)、上海市科委科技计划项目(批准号18411953700、18JC1410100、19XD1424400)和中国科学院国际合作计划项目(批准号:2018SHZDZX05);153 d31kysb20170059)。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
N.G.设计实验,构建载体,进行转染,生成细胞系并分析数据。j.h hu进行RNA FISH、免疫荧光染色,并协助构建载体。B.H., j.a huang, Yu Wei, J.P, Yinghui Wei, X.S.和L.S.协助细胞系的生成和分析。Z.J.构建载体,进行qPCR分析和细胞系基因分型。X.H.、Q.X.和H.L.在细胞培养物中进行瞬时转染和分析。N.G.和X.F.进行了western blot。Y.S.和Y.Z.设计了合成的sgRNA序列,并分析了公开的RNA测序数据。C.Z.协助构建了向量。H.Z.和H.Y.构想了这个项目,设计了实验,监督了这个项目,并写了论文。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
额外的信息gydF4y2Ba
同行评审信息gydF4y2Ba自然细胞生物学gydF4y2Ba感谢Rory Johnson, Ophir Shalem和其他匿名审稿人对本工作的同行评审所做的贡献。gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展数据gydF4y2Ba
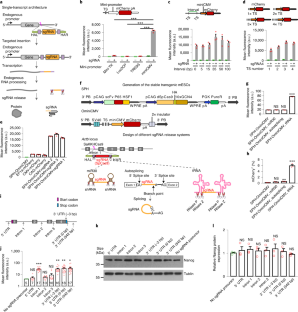
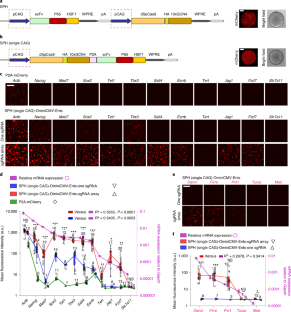
图1不同sgrna诱导的mCherry平均强度及miniCMV启动子的优化。gydF4y2Ba
一个gydF4y2Ba,不同sgrna诱导的mCherry荧光强度(gydF4y2BangydF4y2Ba= 2重复)。注意,在293T细胞中瞬时转染SPH、miniCMV和sgRNA 24小时后,用FACS定量了强度。gydF4y2BabgydF4y2Ba,不同微启动子诱导N2a细胞mCherry表达的代表性图像,瞬时转染24小时后,每个实验独立重复3次,结果相似。比例尺,200 μm。gydF4y2BacgydF4y2Ba,瞬时转染48小时后mCherry的平均荧光强度,gydF4y2BangydF4y2Ba=每组3次(miniCMV vs . Mini-TK,gydF4y2BapgydF4y2Ba< 0.0001;miniCMV vs . Luc2CPgydF4y2BapgydF4y2Ba< 0.0001;miniCMV vs . TRE3G,gydF4y2BapgydF4y2Ba< 0.0001;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试)。gydF4y2BadgydF4y2Ba,瞬时转染48小时后,不同TS间隔诱导的平均mCherry强度;gydF4y2BangydF4y2Ba每组重复3次。gydF4y2BaegydF4y2Ba,不同TS间隔下sgRNA拷贝数对mCherry表达的影响,柱状上方数字表示每组重复数。gydF4y2BafgydF4y2Ba,瞬时转染48小时后,SPH-OminiCMV和不同启动子诱导的mCherry平均荧光强度。gydF4y2BangydF4y2Ba每组重复3次。gydF4y2BaggydF4y2Ba代表性图像显示,SPH-OminiCMV诱导的mCherry水平高于常用的强启动子。tagBFP共转染以控制转染效率。每组独立重复实验2次,结果相似。比例尺:50 μm。所有实验均用质粒瞬时转染N2a细胞。所有数值均以均数±s.e.m.表示;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试;*gydF4y2BapgydF4y2Ba< 0.05, **gydF4y2BapgydF4y2Ba< 0.01, ***gydF4y2BapgydF4y2Ba< 0.001。统计源数据见源数据扩展数据图1。gydF4y2Ba
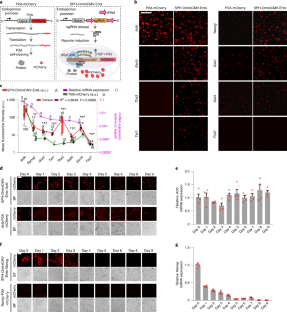
图2 SPH-OminiCMV诱导的基因表达水平高于sph介导的内源性激活和cmv介导的过表达。gydF4y2Ba
一个gydF4y2Ba, sph介导的内源性基因激活、CMV-和sph - ominicmv介导的外源性基因表达示意图。gydF4y2BabgydF4y2Ba,不同基因的相对mRNA表达量,gydF4y2BangydF4y2Ba每组重复2次。统计源数据见源数据扩展数据图2。gydF4y2Ba
图3 SPH-OminiCMV的特异性和SPH-OminiCMV转基因mESCs的生成。gydF4y2Ba
一个gydF4y2Ba,通过在N2a细胞中瞬时转染SPH、OminiCMV和sgRNA,改进的报告系统对错配sgRNA的耐受性。轴:没有。的sgRNA TS;轴:没有。sgRNA。标尺表示平均荧光强度(红色,高;白,低)。sgRNA信息见补充表gydF4y2Ba1gydF4y2Ba,gydF4y2BangydF4y2Ba每组重复2次。gydF4y2BabgydF4y2Ba,转基因示意图。gydF4y2BacgydF4y2Ba通过PCR对SPH-OminiCMV转基因mESCs进行基因分型,“+”表示阳性群体,“-”表示阴性群体,图像代表几个“+”群体。gydF4y2BadgydF4y2BaSPH-OminiCMV阳性菌群中瞬时表达sgRNA诱导mCherry表达的图像,具有3个实验的代表性。比例尺:200 μm。gydF4y2BaegydF4y2Ba, qPCR定量mCherry相对表达量;gydF4y2BangydF4y2Ba每组重复3次。所有数值均以均数±s.e.m表示。统计源数据和未处理凝胶见源数据扩展数据图3。gydF4y2Ba
图4将tRNA-sgRNA-tRNA靶向插入3’utr不影响靶基因正常的蛋白生成。gydF4y2Ba
一个gydF4y2Ba的3'UTR或内含子中插入了tRNA-sgRNA-tRNA的示意图gydF4y2BaActbgydF4y2Ba, mCherry的平均荧光强度。柱状图上方的数字表示每组的重复数(SPH-OminiCMV-Ents-Intron 1 vs .s。SPH-OminiCMV-Ents-3'UTR,gydF4y2BapgydF4y2Ba= 0.5689;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试)。gydF4y2BabgydF4y2Ba,西方墨迹,gydF4y2BangydF4y2Ba每组重复4次。gydF4y2BacgydF4y2Ba定量Western blots数据显示,tRNA-sgRNA-tRNA插入到的3'UTRgydF4y2BaActbgydF4y2Ba基因座不影响生产gydF4y2BaActbgydF4y2BaSPH-OminiCMV vs . SPH-OminiCMVgydF4y2BapgydF4y2Ba= 0.3456;SPH-OminiCMV-Ents vs . P2A-mCherrygydF4y2BapgydF4y2Ba= 0.6301;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试)。柱状上方的数字表示每组的重复次数。gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba,插入tRNA-sgRNA-tRNA靶向gydF4y2BaLacZgydF4y2Ba在SPH-OminiCMV中mESCs不诱导mCherry的表达,柱状图上方的数字表示每组的重复数。所有数值均以均数±s.e.m.表示;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试;*gydF4y2BapgydF4y2Ba< 0.05, **gydF4y2BapgydF4y2Ba< 0.01, ***gydF4y2BapgydF4y2Ba< 0.001。统计源数据和未处理的western blots见源数据扩展数据图4。gydF4y2Ba
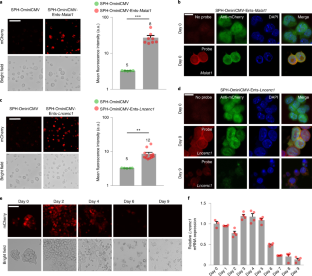
图5 SPH-OminiCMV-Ents能够在细胞分化过程中显示低丰度基因,将sgRNA阵列插入非表达基因中不会诱导mCherry表达。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba,分化定量时mCherry表达的代表性图像gydF4y2BaEsrrbgydF4y2BaqPCR检测SPH-OminiCMV-Ents-的mRNA水平gydF4y2BaEsrrbgydF4y2Ba分化过程中的mESCs。比例尺,50 μm;gydF4y2BangydF4y2Ba每组重复4次。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,显示mCherry表达和量化的代表性图像gydF4y2BaSox2gydF4y2BaqPCR检测SPH-OminiCMV-Ents-的mRNA水平gydF4y2BaSox2gydF4y2Ba分化过程中的mESCs。比例尺,50 μm;gydF4y2BangydF4y2Ba每组重复4次。gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba,显示mCherry表达和量化的代表性图像gydF4y2BaTet1gydF4y2BaqPCR检测SPH-OminiCMV-Ents-的mRNA水平gydF4y2BaTet1gydF4y2Ba分化过程中的mESCs。比例尺:50 μm,gydF4y2BangydF4y2Ba每组重复4次。gydF4y2BaggydF4y2Ba的3'UTR中插入一个sgRNA或sgRNA阵列的示意图gydF4y2BaSema3agydF4y2Ba轨迹。gydF4y2BahgydF4y2Ba,量化gydF4y2BaSema3agydF4y2BaqPCR在ESCs中的表达gydF4y2BangydF4y2Ba= 3重复。gydF4y2Ba我gydF4y2Ba,显示mCherry表达的图像,4个实验的图像具有代表性。比例尺,50 μm。gydF4y2BajgydF4y2Ba,平均mCherry荧光强度。柱状上方的数字表示每组的菌落数量(SPH-OminiCMV- ents -one sgRNA vs . SPH-OminiCMV,gydF4y2BapgydF4y2Ba= 0.5964;SPH-OminiCMV- ents - sgrna阵列vs . SPH-OminiCMV,gydF4y2BapgydF4y2Ba= 0.0761;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试)。所有数值均以均数±s.e.m.表示;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试;*gydF4y2BapgydF4y2Ba< 0.05, **gydF4y2BapgydF4y2Ba< 0.01, ***gydF4y2BapgydF4y2Ba< 0.001。统计源数据见源数据扩展数据图5。gydF4y2Ba
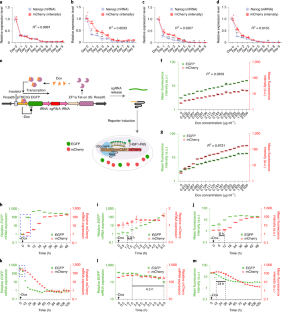
图6单一启动子下驱动dCas9和激活子mCherry的同质表达。gydF4y2Ba
一个gydF4y2Ba,矢量示意图。注意,dCas9和P65-HSF1分别由两个CAG启动子表达。gydF4y2BabgydF4y2Ba, SPH-OminiCMV-Ents-的FACS分析直方图gydF4y2BaActbgydF4y2Ba殖民地。gydF4y2BacgydF4y2Ba同一SPH-OminiCMV-Ents-的mCherry-high和mCherry-low细胞之间激活因子表达水平不同gydF4y2BaActbgydF4y2Ba殖民地,gydF4y2BangydF4y2Ba= 3次重复(高5% vs .低5%:dCas9,gydF4y2BapgydF4y2Ba= 0.0028;p65-HSF1,gydF4y2BapgydF4y2Ba= 0.9161;gydF4y2BaActbgydF4y2Ba,gydF4y2BapgydF4y2Ba= 0.0743;sgRNA,gydF4y2BapgydF4y2Ba= 0.0030;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试)。gydF4y2BadgydF4y2Ba, SPH(单个CAG)的示意图,注意dCas9和p65-HSF1的表达是由单个启动子驱动的。gydF4y2BaegydF4y2Ba, SPH(单CAG) - ominicmv - ents -的FACS分析直方图gydF4y2BaActbgydF4y2Ba殖民地。所有数值均以均数±s.e.m.表示;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试;*gydF4y2BapgydF4y2Ba< 0.05, **gydF4y2BapgydF4y2Ba< 0.01, ***gydF4y2BapgydF4y2Ba< 0.001。统计源数据见源数据扩展数据图6。gydF4y2Ba
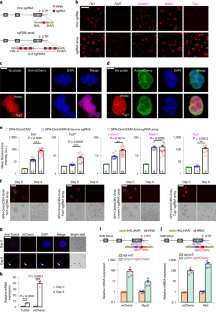
图7 mCherry表达的FACS分析。gydF4y2Ba
一个gydF4y2Ba分析流式细胞术数据,显示R1是如何被门控的(侧散射:SSC;前向散射:FSC)。gydF4y2BabgydF4y2Ba不同基因SPH(单CAG)- ominicmv - ents - 1 sgRNA、SPH(单CAG)-OminiCMV-Ents-sgRNA阵列和P2A-mCherry细胞的FACS分析代表直方图。每个实验都独立地重复了几次,得到了相似的结果。重复次数见图。gydF4y2Ba5 dgydF4y2Ba.gydF4y2BacgydF4y2Ba, SPH(单CAG)- ominicmv - ents - 1个sgRNA和SPH(单CAG)-OminiCMV-Ents-sgRNA阵列细胞对不同lncrna的代表性直方图。每个实验都独立地重复了几次,得到了相似的结果。重复次数见图。gydF4y2Ba5 fgydF4y2Ba.gydF4y2Ba
图8不同策略的并列比较以及分化过程中mCherry和Nanog蛋白水平的下调。gydF4y2Ba
一个gydF4y2Ba分析流式细胞术数据,显示R1是如何被门控的(侧散射:SSC;前向散射:FSC)。gydF4y2BabgydF4y2Ba不同基因SPH(单CAG)- ominicmv - ents - 1 sgRNA、SPH(单CAG)-OminiCMV-Ents-sgRNA阵列和P2A-mCherry细胞的FACS分析代表直方图。每个实验都独立地重复了几次,得到了相似的结果。重复次数见图。gydF4y2Ba5 dgydF4y2Ba.gydF4y2BacgydF4y2Ba, SPH(单CAG)- ominicmv - ents - 1个sgRNA和SPH(单CAG)-OminiCMV-Ents-sgRNA阵列细胞对不同lncrna的代表性直方图。每个实验都独立地重复了几次,得到了相似的结果。重复次数见图。gydF4y2Ba5 fgydF4y2Ba.gydF4y2Ba
图9 SPH(单CAG)-OminiCMV-Ents-sgRNA阵列诱导mCherry最高表达。gydF4y2Ba
一个gydF4y2BaSPH-OminiCMV-Ents-one sgRNA、SPH-OminiCMV-Ents-sgRNA阵列、SPH(单CAG)-OminiCMV-Ents-one sgRNA和SPH(单CAG)-OminiCMV-Ents-sgRNA阵列系统的荧光强度比较。栏中的数字表示每组菌落的数量。未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试。图中还显示了SPH(单CAG)- ominicmv - ents - 1个sgRNA和SPH(单CAG)-OminiCMV-Ents-sgRNA阵列数据。gydF4y2Ba5 dgydF4y2Ba;SPH-OminiCMV-Ents-one sgRNA和SPH-OminiCMV-Ents-sgRNA阵列数据如图所示。gydF4y2Ba2gydF4y2Bac,gydF4y2Ba4 egydF4y2Ba.gydF4y2BabgydF4y2Ba,胚胎干细胞LncRNA表达基质。请注意,数据是从公共GEO数据库(gydF4y2BaGSM2573084gydF4y2Ba)和lncrna按表达量由高到低排列。图中以红色和黑色标记的lncrna。gydF4y2Ba5 fgydF4y2Ba.共检测到FPKM值高于0的lncrna 14432个,FPKM值高于的lncrna 9640个gydF4y2BaPvt1gydF4y2Ba.所有数值均以均数±s.e.m.表示;未配对的两个gydF4y2Ba-gydF4y2Ba站在学生的gydF4y2BatgydF4y2Ba测试;*gydF4y2BapgydF4y2Ba< 0.05, **gydF4y2BapgydF4y2Ba< 0.01, ***gydF4y2BapgydF4y2Ba< 0.001。统计源数据和gydF4y2BapgydF4y2Ba值gydF4y2Ba一个gydF4y2Ba在源数据扩展数据图9中提供。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
互补序列。gydF4y2Ba
补充表gydF4y2Ba
表1:sgRNA序列。表2:内源性基因激活的sgRNA序列。补充表3:插入sgRNA前体的sgRNA序列补充表4:用于识别sgRNA插入的引物。补充表5:相对表达量。补充表6:qPCR引物。gydF4y2Ba
源数据gydF4y2Ba
图1 .来源数据gydF4y2Ba
统计源数据。gydF4y2Ba
图1 .来源数据gydF4y2Ba
未加工的西方墨迹。gydF4y2Ba
图2 .来源数据gydF4y2Ba
统计源数据。gydF4y2Ba
图3 .源数据gydF4y2Ba
统计源数据。gydF4y2Ba
图4 .来源数据gydF4y2Ba
统计源数据。gydF4y2Ba
图5 .来源数据gydF4y2Ba
统计源数据。gydF4y2Ba
图6 .来源数据gydF4y2Ba
统计源数据。gydF4y2Ba
图1 .源数据扩展数据gydF4y2Ba
统计源数据。gydF4y2Ba
图2 .扩展数据gydF4y2Ba
统计源数据。gydF4y2Ba
图3 .扩展数据gydF4y2Ba
统计源数据。gydF4y2Ba
图3 .扩展数据gydF4y2Ba
未加工的凝胶。gydF4y2Ba
图4 .扩展数据gydF4y2Ba
统计源数据。gydF4y2Ba
图4 .扩展数据gydF4y2Ba
未加工的西方墨迹。gydF4y2Ba
图5 .源数据扩展数据gydF4y2Ba
统计源数据。gydF4y2Ba
图6 .源数据扩展数据gydF4y2Ba
统计源数据。gydF4y2Ba
图9 .源数据扩展数据gydF4y2Ba
统计源数据。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
高楠,胡杰,何斌。gydF4y2Baet al。gydF4y2Ba内源性启动子驱动的sgRNA用于监测低丰度转录本和lncrna的表达。gydF4y2BaNat细胞生物学gydF4y2Ba23gydF4y2Ba, 99-108(2021)。https://doi.org/10.1038/s41556-020-00610-9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41556-020-00610-9gydF4y2Ba
这篇文章被引用gydF4y2Ba
通过合成CRISPRa电路精确的肿瘤免疫重布线,同时获得/失去转录因子gydF4y2Ba
自然通讯gydF4y2Ba(2022)gydF4y2Ba
内含子编码的顺激子转录本用于编码和非编码rna的微创监测gydF4y2Ba
自然细胞生物学gydF4y2Ba(2022)gydF4y2Ba
长链非编码RNA PAARH通过上调HOTTIP和激活HIF-1α/VEGF信号通路促进肝细胞癌进展和血管生成gydF4y2Ba
细胞死亡与疾病gydF4y2Ba(2022)gydF4y2Ba
通过在内含子中敲入带有tRNA的sgRNA,监测长链非编码rna的启动子活性和干细胞分化gydF4y2Ba
细胞的发现gydF4y2Ba(2021)gydF4y2Ba
