文摘
CRISPR-Cas持有庞大的承诺为发展中人类基因编辑核酸酶治疗应用1,2,3,4,5,6但是确定的非目标突变对临床翻译很重要7。一个有效的方法,它能够可靠地识别非标靶体内没有描述到目前为止,这意味着它目前还不清楚,这些突变的频率发生。这里我们描述体内非目标的验证(体内),强劲的高度敏感的战略可以识别的全基因组脱靶效应CRISPR-Cas核酸酶体内。我们使用活体和引导RNA故意设计成滥交表明CRISPR-Cas核酸酶可引起大量非目标突变老鼠体内肝脏。更重要的是,我们也使用活体证明设计合理引导rna能够直接有效的编辑在小鼠体内肝脏没有检测到非目标突变。体提供了一个通用的策略定义和量化的脱靶效应基因编辑核酸酶在整个生物,从而提供一个蓝图,促进使用体内基因治疗策略的发展编辑。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
CRISPR-GRANT:跨平台图形分析工具用于高通量基因组CRISPR-based编辑评估
BMC生物信息学开放获取2023年5月30日
改善体内CRISPR脱靶的敏感性检测DISCOVER-Seq +
自然方法开放获取2023年4月06
Extru-seq:全基因组预测方法的Cas9不相干的网站与细胞和体外方法的优势
基因组生物学开放获取2023年1月10
访问选项
访问其他自然组合期刊性质和54
得到自然+,请求书在线访问订阅
29.99美元/ 30天
取消任何时候
订阅本杂志
收到51印刷问题和网络访问
每年199.00美元
只有3.90美元的问题
本文租或购买
价格不同的文章类型
从1.95美元
来39.95美元
价格可能受当地税收计算在结帐


数据可用性
序列数据支持本研究的发现一直存放在SRA加入号码SRP151131。所有其他数据支持本研究的发现可以从相应的作者在合理的请求。
引用
Musunuru, k的希望和炒作CRISPR-Cas9基因组编辑:复习一下。JAMA心功能杂志。2,914 - 919 (2017)。
Fellmann C。,Gowen, B. G., Lin, P. C., Doudna, J. A. & Corn, J. E. Cornerstones of CRISPR–Cas in drug discovery and therapy.Nat。启药物。16,89 - 100 (2017)。
Komor, a . C。,Badran, A. H. & Liu, D. R. CRISPR-based technologies for the manipulation of eukaryotic genomes.细胞168年20-36 (2017)。
j·s·古,t & Kim的治疗应用CRISPR RNA-guided基因组编辑。简短。功能。基因组学1638-45 (2017)。
角,我t。,Mussolino, C. & Cathomen, T. Refining strategies to translate genome editing to the clinic.地中海Nat。。23,415 - 423 (2017)。
邓巴,c, e . et al。基因治疗的年龄。科学359年eaan4672 (2018)。
蔡,j·k·& Joung树群定义和改善CRISPR-Cas9全基因组特异性的核酸酶。Nat。启麝猫。17,300 - 312 (2016)。
蔡,s .问:et al . CIRCLE-seq:高度敏感在体外屏幕全基因组CRISPR-Cas9核酸酶的非标靶。Nat方法。14,607 - 614 (2017)。
许,p . d . et al。DNA靶向特异性RNA-guided Cas9核酸酶。Nat。。31日,827 - 832 (2013)。
蔡,s .问:et al . GUIDE-seq使全基因组分析的不相干的乳沟CRISPR-Cas核酸酶。Nat。。33,187 - 197 (2015)。
叮,问:et al。永久变更与体内CRISPR-Cas9 PCSK9基因编辑。中国保监会Res。。115年,488 - 492 (2014)。
阴,h . et al . Structure-guided病毒性指导RNA的化学改性使有效在活的有机体内基因组编辑。Nat。。35,1179 - 1187 (2017)。
阴,h . et al .治疗基因组编辑CRISPR结合病毒和病毒性交付的系统组件在活的有机体内。Nat。。34,328 - 333 (2016)。
高,x等。治疗常染色体显性的听力损失在活的有机体内交付基因组编辑代理。自然553年,217 - 221 (2018)。
Giannoukos, g . et al . UDiTaSTM一个基因组编辑检测方法indels和基因组重组。BMC基因组学19212 (2018)。
Bae, S。,P一个rk, J. & Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases.生物信息学30.,1473 - 1475 (2014)。
Kosicki, M。,Tomberg, K. & Bradley, A. Repair of double-strand breaks induced by CRISPR–Cas9 leads to large deletions and complex rearrangements.Nat。。36,765 - 771 (2018)。
霍吉金斯,a . et al . WGE: CRISPR数据库基因组工程。生物信息学31日,3078 - 3080 (2015)。
Rohland: &帝国,d .成本效益高通量DNA测序图书馆多元目标捕获。基因组Res。22,939 - 946 (2012)。
Pinello, l . et al。分析CRISPR用CRISPResso基因编辑实验。Nat。。34,695 - 697 (2016)。
李,h . et al .序列比对/地图格式和SAMtools。生物信息学25,2078 - 2079 (2009)。
确认
支持J.K.J.德斯蒙德和安Heathwood MGH研究学者奖。J.K.J.,马丁B. and J.A.G. were supported by a sponsored research agreement with AstraZeneca. L.P. is supported by a National Human Genome Research Institute (NHGRI) Career Development Award (R00HG008399). J.K.J., M.J.A. and J.M.-L. are supported by a National Institutes of Health Maximizing Investigators' Research Award (MIRA) (R35 GM118158). J.K.J., L.P. and K.C. are supported by the Defense Advanced Research Projects Agency (HR0011-17-2-0042). We thank M. Snowden, S. Platz and S. Rees for resource allocation from AstraZeneca Research Funds. We thank J. Y. Hsu for discussions and input.
审核人信息
自然由于f .莫斯科和其他匿名审稿人(s)为他们的贡献的同行评审工作。
作者信息
作者和联系
贡献
公共广播,M.J.P., T.B. and M.B. executed intravenous tail vein injections. P.A. and T.B. coordinated vena saphena blood sampling and performed plasma extractions. P.A., M.J.P., A.C., T.B., M.B., M.M. and R.N. performed in vivo terminations and organ collection. P.A. performed Surveyor assays for genomic DNAs of mouse livers. P.A. and T.B. performed ELISA for Pcsk9 protein detection in plasma samples. M.L.B, J.A.G., S.Q.T. and N.T.N. performed the CIRCLE-seq and targeted amplicon sequencing experiments. J.M.-L., K.C., S.P.G., L.P. and M.J.A. performed bioinformatic and computational analysis of the off-target experiments. M.A.F. generated AstraZeneca proprietary software for gRNA identification. P.A. designed gRNAs with help of M.A.F., and validated their functional activity. F.S. performed mouse phenotypic characterization. P.A., M.L.B., S.Q.T., M.M., M.B.-Y, A.C., R.N., M.D.F., L.M.M. and J.K.J. conceived of and designed the study. P.A., M.L.B., M.M. and J.K.J. organized and supervised experiments. P.A., M.L.B., J.A.G., M.M. and J.K.J. prepared the manuscript with input from all authors.
相应的作者
道德声明
相互竞争的利益
J.K.J.在梁疗法金融利益,眨眼疗法,Editas医学Endcadia,监测生物技术(原名信标基因组学),成对植物、波赛达疗法和Transposagen生物制药。在监测生物技术M.J.A.和S.Q.T.金融利益。J.K.J.和M.J.A.综述了利益和是由马萨诸塞州总医院和合作伙伴医疗依照他们的利益冲突的政策。S.Q.T.和J.K.J.共同发明人专利描述CIRCLE-seq方法。公共广播,M.D.F., M.J.P., M.A.F., F.S., M.B., R.N., M.B.Y. and M.M. are employees and shareholders of AstraZeneca. L.M.M. is an employee and shareholder of GE Healthcare and a shareholder of AstraZeneca.
额外的信息
出版商的注意:施普林格自然保持中立在发表关于司法主权地图和所属机构。
扩展数据数据和表
有效地扩展数据图1 gP-Cas9变异老鼠Pcsk9基因Pcsk9的蛋白含量,降低等离子体内。
一个全科医生是为了目标在鼠标的第1外显子序列Pcsk9有许多基因基因密切相关网站(也就是说,那些1 - 3相对于目标网站不匹配;扩展的数据表1)。蓝色酒吧指示老鼠基因外显子区域。b、测量分析和下一代DNA测序数据证明有效的老鼠体内修改目标Pcsk9由gP-Cas9基因在小鼠肝脏。化验进行管理后的第四天,星期3运载体编码gP-Cas9 (gP)或负控制GFP-Cas9 (GFP)。对于每一个时间点,从肝脏基因组DNA孤立使用的化验n= 3生物独立野生型C57BL / 6 n (WT)小鼠或C57BL / 6小鼠n-derived包含一个人类的副本PCSK9开放阅读框下白蛋白启动子,撞到Rosa26轨迹(KI)。星号表示裂解PCR产品预期与测量员核酸酶治疗后。百分比显示indel突变的频率取决于目标是使用新一代测序的扩增子测序;这些是相同的值显示为图的目标网站。1 b。行划分车道从不同的位置在同一凝胶。对源数据测量分析和有针对性的扩增子测序,看到补充图。1和补充表2,分别。c、鼠标Pcsk9等离子体中的蛋白质含量的测量n= 3生理上独立的野生型和敲入小鼠和人类PCSK9等离子体中的蛋白质含量的测量n= 3生物独立敲入小鼠,在核酸酶治疗。蛋白质含量是评估4天,7周后3 gP或控制GFP运载体和归一化到基线水平。实验和对照组之间的显著差异是决定使用双向方差分析和Sidak双边调整多个对比测试;* *P< 0.01,* * *P< 0.001,* * * *P< 0.0001。看到图扩展数据的源数据。4 b为精确调整P值。所有值都呈现组的含义,以及误差表示平均数标准误差。在等离子体增强减少Pcsk9的水平相对于观察到的频率Pcsk9基因改变是一致的与先前发表的研究(补充讨论)。
扩展数据图2生物分布的研究adenovirus-serotype 5在老鼠身上。
一个记者综合构造的示意图R26R老鼠用于评估交付Cre重组酶使用腺病毒血清型5向量。Cre-mediated切除的液态氧P-flanked转录终止信号的上游lacZ基因导致β-galactosidase酶的表达。β-Galactosidase表达式可以被染色的组织量化与半乳糖苷化合物,蓝色当这种酶裂解。b、量化β-galactosidase表达各种解剖的部分器官n= 2生物独立R26R小鼠静脉注射注射腺病毒血清型向量编码Cre 5。匹配器官部分从R26R小鼠静脉注射腺病毒血清型5向量编码绿色荧光蛋白是用来确定背景染色水平和作为一个消极的控制。匹配器官部分从Z /如老鼠持续表达lacZ(β-galactosidase)和静脉注射PBS(而非腺病毒)被用来提供积极的染色控制。所有小鼠评估腺病毒后一周或PBS注入。实验被执行一次。
扩展数据图3包含人类生成实验小鼠的繁殖策略PCSK9开放阅读框撞到了Rosa26轨迹。
C57BL / 6 n-derived鼠标线包含一个人类的副本PCSK9开放阅读框撞到了Rosa26轨迹(C57BL / 6 n hPCSK9KI+ /−)用于与C57BL / 6 n小鼠繁殖。后代产生C57BL / 6 n hPCSK9KI实验动物+ /−(称为敲入)和C57BL / 6 n hPCSK9KI−−/男性(称为野生型)。
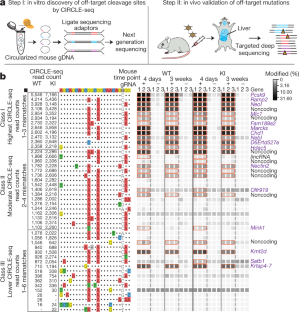
扩展数据图4 CIRCLE-seq读计数的散点图网站认同gP-Cas9基因组DNAn= 1野生型和n= 1敲入小鼠。
读计数显示在对数刻度和颜色显示不匹配的数量在每个非目标站点相对于目标站点。网站显示为三角形选择有针对性的扩增子测序。的相关性r2价值获得使用散点图中所有的值显示在左上角,并使用线性回归获得。
扩展数据图5的比较密切匹配网站中确定硅片和非目标被CIRCLE-seq乳沟网站。
维恩图比较不相干的乳沟网站在小鼠基因组DNA被CIRCLE-seq实验密切匹配网站(六不匹配相对于目标网站)在小鼠基因组中确定硅片由Cas-OFFinder显示Cas9 gRNAs gP,通用和gMH。
扩展数据图6的遗传和表型变化引起的交付gM-Cas9 gMH-Cas9体内。
一个、顺序和位置的Cas9通用的(老鼠)和gMH(老鼠和人类)目标站点内生鼠标Pcsk9基因和人类PCSK9转基因插入在鼠标Rosa26轨迹。单一的基地位置不同gMH目标站点的老鼠Pcsk9基因和人类PCSK9转基因是用红色突出显示。蓝色酒吧指示老鼠基因外显子区域和紫色酒吧代表外显子的人类基因组位点;网站的PAM序列是粗体下划线间隔序列。b、测量分析和下一代DNA测序数据证明有效的内生老鼠体内修改目标Pcsk9网站和人类PCSK9转基因小鼠的肝脏。进行了化验后第四天,星期3运载体编码通用和Cas9(通用),gMH和Cas9 (gMH)或GFP和Cas9 (GFP)使用从肝脏基因组DNA分离n= 3生理上独立的野生型和敲入小鼠。星号表示裂解PCR产品预期与测量员核酸酶治疗后。百分比显示indel突变的频率取决于目标是使用新一代测序的扩增子测序;这些是相同的值显示为图的目标网站。3和扩展数据图。7。行划分车道从不同的位置在同一凝胶。源数据测量分析,看到补充图。1。对源数据有针对性的扩增子测序,看到补充表6和7分别对通用汽车和gMH。b、鼠标Pcsk9蛋白质含量测量等离子体n= 3生物独立野生型和敲入小鼠和人类PCSK9蛋白质含量测量等离子体n= 3生物独立敲入小鼠后CRISPR-Cas核酸酶治疗。等离子体中的蛋白质含量进行评估在第四天,7和星期3通用管理后,gMH或控制GFP运载体和规范化与基线水平在每个时间点。组间显著差异确定使用双向方差分析和Dunnett双边调整多个对比测试;*P< 0.05,* *P< 0.01,* * *P< 0.001,* * * *P< 0.0001。看到源数据精确调整P值。值作为集团意味着误差表示的标准错误的意思。
扩展数据图7的评估体内脱靶indel gMH-Cas9引起的突变。
Indel突变频率取决于目标扩增子测序(使用高通量测序)提出的热图gMH-Cas9目标网站(黑色广场)和63非目标网站从CIRCLE-seq实验确定。每个轨迹化验n= 3生物独立老鼠(标注1、2和3)使用基因组DNA分离野生型和敲入小鼠肝脏的处理实验adenoviral向量编码gMH-Cas9 (gRNA +)或控制adenoviral向量GFP-Cas9 (gRNA−)。对于每一个网站,不匹配相对于目标网站显示彩色盒子和基地间隔序列和编号从1(最近端PAM) 20(大多数PAM)的远端。阅读的数量计数发现的每个站点CIRCLE-seq实验对野生型和敲入小鼠基因组DNA所示左列(从最高到最低排名基于计数的野生型基因组DNA CIRCLE-seq实验)。每个热图中的方框代表一个测序实验。网站之间明显不同的实验(gRNA +)和控制(gRNA−)样品与盒子桔子轮廓突出显示。额外的紧密匹配的网站在小鼠基因组中从CIRCLE-seq实验(不确定),检查indel突变是盒装红色底部的图。看到补充表7源数据和P值(负二项)。
补充信息
补充信息
这个文件包含一个补充讨论。额外的讨论提出了包括查阅以往研究脱靶突变体内,可能的解释为相对更大的减少血浆Pcsk9的效率相比Pcsk9基因突变观察,描述如何使用体内CRISPR-Cas变异和orthologues,其他基因编辑平台/交付方法,非哺乳类生物。
补充数据
这个文件包含uncropped凝胶为扩展数据数据。
补充表
这个文件包含补充表1:非标靶乳沟网站被CIRCLE-seq gP / SpCas9核酸酶。染色体提供了坐标,其次是CIRCLE-seq读计数为非目标网站中发现WT老鼠基因组DNA,非标靶序列,不匹配,膨胀不匹配序列,分数与凸起的数量不匹配,和CIRCLE-seq读计数的非目标识别KI老鼠基因组DNA。
补充表
这个文件包含补充表2:潜在的非目标网站gP / SpCas9分析有针对性的扩增子深测序WT和KI小鼠肝脏的运载体。染色体提供了坐标,其次是MiSeq运行使用ID和FASTQ文件,样本信息,包括组织类型、时间点、治疗和复制。第二列详细读计数为每个样本和统计测试结果在治疗组之间,其次是底漆使用。注:负二项测试。
补充表
这个文件包含补充表3:非标靶乳沟网站被CIRCLE-seq SpCas9和通用gRNA。染色体坐标提供后跟CIRCLE-seq读计数非目标网站中发现WT老鼠基因组DNA,非标靶序列,不匹配,膨胀不匹配序列,分数与凸起的数量不匹配,和CIRCLE-seq读计数的非目标识别KI老鼠基因组DNA。
补充表
这个文件包含补充表4:非标靶的乳沟网站被CIRCLE-seq SpCas9和gMH gRNA。染色体坐标提供后跟CIRCLE-seq读计数非目标网站中发现WT老鼠基因组DNA,非标靶序列,不匹配,膨胀不匹配序列,分数与凸起的数量不匹配,和CIRCLE-seq读计数的非目标识别KI老鼠基因组DNA。
补充表
这个文件包含补充表5:潜在的非目标网站通用/ SpCas9被CIRCLE-seq没有读取WT或KI未经处理的小鼠进行有针对性的扩增子深测序来评估潜在的单核苷酸多态性。数字和百分比的顺序读取显示,不同于预期的非目标网站。“WT vs KI百分比差异”列显示了这些读取的差别的绝对值。差异发现最高为7.5%,表明snp不解释微分读计数观察与CIRCLE-seq相比。
补充表
这个文件包含补充表6:潜在的非目标网站通用/ SpCas9分析有针对性的扩增子深测序WT和KI小鼠肝脏的运载体。染色体提供了坐标,其次是MiSeq运行使用ID和FASTQ文件,样本信息,包括组织类型、时间点、治疗和复制。第二列详细读计数为每个样本和统计测试结果在治疗组之间,其次是底漆使用。注:负二项测试。
补充表
这个文件包含补充表7:潜在的非目标网站gMH / SpCas9分析有针对性的扩增子深测序WT和KI小鼠肝脏的运载体。染色体提供了坐标,其次是MiSeq运行使用ID和FASTQ文件,样本信息,包括组织类型、时间点、治疗和复制。第二列详细读计数为每个样本和统计测试结果在治疗组之间,其次是底漆使用。注:负二项。
权利和权限
关于这篇文章
引用这篇文章
Akcakaya, P。,Bobbin, M.L., Guo, J.A.et al。体内CRISPR编辑没有可检测全基因组的非目标突变。自然561年,416 - 419 (2018)。https://doi.org/10.1038/s41586 - 018 - 0500 - 9
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586 - 018 - 0500 - 9
关键字
- 基因编辑核酸酶
- CRISPR-Cas9核酸酶
- 有针对性的扩增子测序
- 补充讨论
- PCSK9基因
本文引用的
Extru-seq:全基因组预测方法的Cas9不相干的网站与细胞和体外方法的优势
基因组生物学(2023)
CRISPR-GRANT:跨平台图形分析工具用于高通量基因组CRISPR-based编辑评估
BMC生物信息学(2023)
全基因组分析的主要编辑器使用PE-tag非目标网站在体外和体内
自然方法(2023)
发现CRISPR-Cas不相干的优惠
自然方法(2023)
CRISPR工程在瀑样基因修复和疾病模型
自然评论生物工程(2023)
