摘要gydF4y2Ba
减弱同源重组(HR)介导的修复的突变促进肿瘤发生,并使细胞对导致复制叉折的化疗药物敏感,这种表型被称为“BRCAness”gydF4y2Ba1gydF4y2Ba.乳腺癌是由22个基因的功能缺失突变引起的gydF4y2Ba1gydF4y2Ba.在这些基因中,除了一个(gydF4y2BaCDK12gydF4y2Ba)直接在HR修复通路中起作用gydF4y2Ba1gydF4y2Ba.CDK12磷酸化RNA聚合酶II c端结构域七肽重复的丝氨酸2gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,一种调节转录延伸、剪接、裂解和多聚腺苷酸的修饰gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba.全基因组表达研究表明,CDK12的缺失相对特异性地取消了几个HR基因的表达,从而削弱了HR修复gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba.这一观察表明,突变状态gydF4y2BaCDK12gydF4y2Ba可能预测针对BRCAness的靶向治疗的敏感性,如PARP1抑制剂,CDK12抑制剂可能诱导HR-competent肿瘤对这些治疗的敏化gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba.尽管临床兴趣日益浓厚,但CDK12调控HR基因的机制仍不清楚。在这里,我们发现CDK12在小鼠胚胎干细胞中全面抑制内含子多聚腺苷酸事件,使全长基因产物的产生成为可能。许多HR基因比其他表达基因含有更多的内含子多聚腺苷酸位点,这些位点对CDK12的缺失特别敏感。这些位点的累积效应解释了HR基因表达对CDK12缺失的增强敏感性,我们发现这种机制在包含功能缺失的人类肿瘤中是保守的gydF4y2BaCDK12gydF4y2Ba突变。这项工作阐明了CDK12的功能,并强调了其作为化疗靶点和肿瘤生物标志物的潜力。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
PNUTS-PP1复合物是疱疹病毒KSHV基因表达和复制的内在屏障gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba12月2日gydF4y2Ba
在braf突变的黑素瘤中,CDK12被过度激活并成为合成致死靶点gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2022年10月29日gydF4y2Ba
PROTACs:学术界和工业界的巨大机遇(2020年至2021年更新)gydF4y2Ba
信号转导与靶向治疗gydF4y2Ba开放获取gydF4y2Ba2022年6月9日gydF4y2Ba
访问选项gydF4y2Ba
访问《自然》和其他54种《自然》杂志gydF4y2Ba
获取Nature+,我们最超值的在线订阅gydF4y2Ba
每月29.99美元gydF4y2Ba
随时取消gydF4y2Ba
订阅这本杂志gydF4y2Ba
收到51个印刷问题和在线访问gydF4y2Ba
199.00美元一年gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
租或购买这篇文章gydF4y2Ba
只要这篇文章,只要你需要它gydF4y2Ba
39.95美元gydF4y2Ba
价格可能受当地税收的影响,在结账时计算gydF4y2Ba
数据可用性gydF4y2Ba
测序数据已存入基因表达集合,登录号为gydF4y2BaGSE116017gydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
Lord, C. J. & Ashworth, A. BRCAness再版。gydF4y2BaNat. Rev. CancergydF4y2Ba16gydF4y2Ba, 110-120(2016)。gydF4y2Ba
巴特科维亚克,B.等人。CDK12是一种转录伸长相关的CTD激酶,是酵母Ctk1的后生同源物。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba24gydF4y2Ba, 2303-2316(2010)。gydF4y2Ba
Blazek, D.等人。细胞周期蛋白K/CDK12复合物通过调控DNA损伤反应基因的表达来维持基因组的稳定性。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba25gydF4y2Ba, 2158-2172(2011)。gydF4y2Ba
张,T.等。共价靶向远端半胱氨酸残基开发CDK12和CDK13抑制剂。gydF4y2BaNat,化学。医学杂志gydF4y2Ba.gydF4y2Ba12gydF4y2Ba, 876-884(2016)。gydF4y2Ba
Ekumi, K. M.等。卵巢癌CDK12突变通过CDK12/CycK复合物的形成和功能缺陷来调控DNA修复基因的表达。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba43gydF4y2Ba, 2575-2589(2015)。gydF4y2Ba
约翰逊,s.f.等人。在BRCA野生型和突变型三阴性乳腺癌模型中,CDK12抑制逆转从头和获得性PARP抑制剂耐药。gydF4y2Ba细胞的报道gydF4y2Ba17gydF4y2Ba, 2367-2381(2016)。gydF4y2Ba
Iniguez, A. B.等人。EWS/FLI在尤文氏肉瘤中赋予CDK12抑制的肿瘤细胞合成致死率。gydF4y2Ba癌症细胞gydF4y2Ba33gydF4y2Ba, 202 - 216。e6(2018)。gydF4y2Ba
辛建平,马利等。RNA聚合酶II CTD协调转录和RNA加工。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba26gydF4y2Ba, 2119-2137(2012)。gydF4y2Ba
Davidson, L., Muniz, L. & West, S.在人类细胞中,pre-mRNA的3 '端形成和RNA聚合酶II CTD上Ser2的磷酸化是相互耦合的。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba28gydF4y2Ba, 342-356(2014)。gydF4y2Ba
Bajrami等人。基因合成致死率的全基因组分析确定CDK12是PARP1/2抑制剂敏感性的新决定因素。gydF4y2Ba癌症ResgydF4y2Ba.gydF4y2Ba74gydF4y2Ba, 287-297(2014)。gydF4y2Ba
Joshi, P. M., Sutor, S. L., Huntoon, C. J. & Karnitz, L. M.卵巢癌相关突变使CDK12的催化活性丧失,CDK12是一种促进同源重组修复和对顺铂和多聚(adp -核糖)聚合酶抑制剂耐药性的激酶。gydF4y2Ba生物。化学gydF4y2Ba.gydF4y2Ba289gydF4y2Ba, 9247-9253(2014)。gydF4y2Ba
Hong Y. & Stambrook, P. J.电离辐射后胚胎干细胞G1阻滞的恢复和凋亡保护。gydF4y2Ba国家科学院学报美国gydF4y2Ba101gydF4y2Ba, 14443-14448(2004)。gydF4y2Ba
Aladjem, m.i.等人。ES细胞不激活p53依赖性应激反应,并在DNA损伤时发生p53依赖性凋亡。gydF4y2Ba咕咕叫。医学杂志gydF4y2Ba.gydF4y2Ba8gydF4y2Ba, 145-155(1998)。gydF4y2Ba
Tichy, e.d.等人。小鼠胚胎干细胞,而不是体细胞,主要使用同源重组来修复双链DNA断裂。gydF4y2Ba干细胞开发gydF4y2Ba.gydF4y2Ba19gydF4y2Ba, 1699-1711(2010)。gydF4y2Ba
泽曼,M. K. & Cimprich, K. A.复制压力的原因和后果。gydF4y2Ba细胞生物学gydF4y2Ba.gydF4y2Ba16gydF4y2Ba, 2-9(2014)。gydF4y2Ba
Lin, T. et al. p53通过抑制Nanog表达诱导小鼠胚胎干细胞分化。gydF4y2Ba细胞生物学gydF4y2Ba.gydF4y2Ba7gydF4y2Ba, 165-171(2005)。gydF4y2Ba
刘,j.c .等。高线粒体启动使hESCs对dna损伤诱导的凋亡敏感。gydF4y2Ba细胞干细胞gydF4y2Ba13gydF4y2Ba, 483-491(2013)。gydF4y2Ba
van der Laan, S, Tsanov, N, Crozet, C. & Maiorano, D.小鼠ESCs中Dub3的高表达将G1/S检查点与多能性结合。gydF4y2Ba摩尔。细胞gydF4y2Ba52gydF4y2Ba, 366-379(2013)。gydF4y2Ba
Shieh, s.y ., Ikeda, M., Taya, Y. & Prives, C. DNA损伤诱导的p53磷酸化缓解MDM2的抑制作用。gydF4y2Ba细胞gydF4y2Ba91gydF4y2Ba, 325-334(1997)。gydF4y2Ba
李,K.-H。et al。一项全基因组研究发现Wnt信号通路是小鼠胚胎干细胞中p53的主要靶点。gydF4y2Ba国家科学院学报美国gydF4y2Ba107gydF4y2Ba, 69-74(2010)。gydF4y2Ba
Ku, M.等。对PRC1和PRC2占用的全基因组分析确定了两类二价结构域。gydF4y2Ba公共科学图书馆麝猫gydF4y2Ba.gydF4y2Ba4gydF4y2Ba, e1000242(2008)。gydF4y2Ba
Tien, J. F.等。CDK12调控替代最后外显子mRNA剪接并促进乳腺癌细胞侵袭。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba45gydF4y2Ba, 6698-6716(2017)。gydF4y2Ba
田波,李志强,李志强。mRNA前体的选择性聚腺苷酸化。gydF4y2Ba细胞生物学gydF4y2Ba.gydF4y2Ba18gydF4y2Ba, 18-30(2017)。gydF4y2Ba
田斌,潘卓,李建勇。内含子中广泛存在的mRNA多聚腺苷酸事件表明多聚腺苷酸与剪接之间的动态相互作用。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba17gydF4y2Ba, 156-165(2007)。gydF4y2Ba
Almada, A. E, Wu, X., Kriz, A. J, Burge, C. B. & Sharp, P. A.启动子定向受U1 snRNP和多聚腺苷酸信号控制。gydF4y2Ba自然gydF4y2Ba499gydF4y2Ba, 360-363(2013)。gydF4y2Ba
韦伯,C. M., Ramachandran, S. & Henikoff, S.核小体是环境特异性的,H2A。z -调制的RNA聚合酶屏障。gydF4y2Ba摩尔。细胞gydF4y2Ba53gydF4y2Ba, 819-830(2014)。gydF4y2Ba
癌症基因组图谱研究网络。原发性前列腺癌的分子分类学。gydF4y2Ba细胞gydF4y2Ba163gydF4y2Ba, 1011-1025(2015)。gydF4y2Ba
吴彦明,等。CDK12失活描述了一种不同的免疫原性晚期前列腺癌。gydF4y2Ba细胞gydF4y2Ba173gydF4y2Ba, 1770 - 1782。e14灯头(2018)。gydF4y2Ba
癌症基因组图谱研究网络。卵巢癌的综合基因组分析。gydF4y2Ba自然gydF4y2Ba474gydF4y2Ba, 609-615(2011)。gydF4y2Ba
孟喜,F.等。串联复制表型是一种由不同基因突变驱动的普遍的全基因组癌症配置。gydF4y2Ba癌症细胞gydF4y2Ba34gydF4y2Ba, 197 - 210。e5(2018)。gydF4y2Ba
Ran, F. A.等。利用CRISPR-Cas9系统进行基因组工程。gydF4y2BaProtoc Nat。gydF4y2Ba8gydF4y2Ba, 2281-2308(2013)。gydF4y2Ba
Cong, L.等。利用CRISPR/Cas系统进行多重基因组工程。gydF4y2Ba科学gydF4y2Ba339gydF4y2Ba, 819-823(2013)。gydF4y2Ba
Gurtan, A. M., Lu, V., Bhutkar, A. & Sharp, P. A.人体Dicer的体内结构-功能分析揭示了前体miRNAs的定向加工。gydF4y2Ba核糖核酸gydF4y2Ba18gydF4y2Ba, 1116-1122(2012)。gydF4y2Ba
Gyori, b.m., Venkatachalam, G., Thiagarajan, p.s., Hsu, D. & Clement, m.v。OpenComet:用于彗星分析图像的自动化工具。gydF4y2Ba氧化还原生物gydF4y2Ba.gydF4y2Ba2gydF4y2Ba, 457-465(2014)。gydF4y2Ba
Skene, P. J. & Henikoff, S.生成全基因组蛋白质结合高分辨率地图的简单方法。gydF4y2BaeLifegydF4y2Ba4gydF4y2Ba, e09225(2015)。gydF4y2Ba
Skene, P. J., Hernandez, A. E, Groudine, M. & Henikoff, S. RNA聚合酶II对启动子逃逸的核小体屏障被染色质重塑体Chd1所克服。gydF4y2BaeLifegydF4y2Ba3.gydF4y2Ba, e02042(2014)。gydF4y2Ba
特拉普内尔,C.等。TopHat和Cufflinks RNA-seq实验的差异基因和转录本表达分析。gydF4y2BaProtoc Nat。gydF4y2Ba7gydF4y2Ba, 562-578(2012)。gydF4y2Ba
Dobin, A.等人。STAR:超快通用RNA-seq对准器。gydF4y2Ba生物信息学gydF4y2Ba29gydF4y2Ba, 15-21(2013)。gydF4y2Ba
Li, B. & Dewey, C. N. RSEM:有或没有参考基因组的RNA-Seq数据的精确转录物定量。gydF4y2BaBMC生物信息学gydF4y2Ba12gydF4y2Ba, 323(2011)。gydF4y2Ba
冷,N.等。EBSeq: RNA-seq实验推理的经验贝叶斯层次模型。gydF4y2Ba生物信息学gydF4y2Ba29gydF4y2Ba, 1035-1043(2013)。gydF4y2Ba
安德斯,S. & Huber, W.序列计数数据的差分表达式分析。gydF4y2Ba基因组医学杂志gydF4y2Ba.gydF4y2Ba11gydF4y2Ba, r106(2010)。gydF4y2Ba
Katz, Y.,王,E. T., Airoldi, E. M. & Burge, C. B.用于识别异构体调控的RNA测序实验的分析和设计。gydF4y2BaNat方法。gydF4y2Ba7gydF4y2Ba, 1009-1015(2010)。gydF4y2Ba
Voong, L. N.等。通过化学制图深入了解小鼠胚胎干细胞的核小体组织。gydF4y2Ba细胞gydF4y2Ba167gydF4y2Ba, 1555 - 1570。e15(2016)。gydF4y2Ba
Vainshtein, Y., Rippe, K. & Teif, V. B. NucTools:从高通量测序数据分析染色质特征占用谱。gydF4y2BaBMC基因组学gydF4y2Ba18gydF4y2Ba, 158(2017)。gydF4y2Ba
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 841-842(2010)。gydF4y2Ba
高,J.等。使用cBioPortal对复杂癌症基因组学和临床资料进行综合分析。gydF4y2Ba科学。信号gydF4y2Ba.gydF4y2Ba6gydF4y2Ba, pl1(2013)。gydF4y2Ba
Cerami, E.等。cBio癌症基因组学门户:探索多维癌症基因组学数据的开放平台。gydF4y2Ba癌症越是加大gydF4y2Ba.gydF4y2Ba2gydF4y2Ba, 401-404(2012)。gydF4y2Ba
Cui, Y. & Denis, C. L.体内证据表明,转录延伸因子RPB2, TFIIS和SPT5的缺陷增强了上游聚(A)位点的利用。gydF4y2Ba摩尔。细胞。医学杂志gydF4y2Ba.gydF4y2Ba23gydF4y2Ba, 7887-7901(2003)。gydF4y2Ba
杨,Y.等。PAF复合物在选择性裂解和多聚腺苷酸化中起着新的亚基特异性作用。gydF4y2Ba公共科学图书馆麝猫gydF4y2Ba.gydF4y2Ba12gydF4y2Ba, e1005794(2016)。gydF4y2Ba
刘,等。转录延伸率有组织特异性的影响选择性切割和多聚腺苷酸gydF4y2Ba黑腹果蝇gydF4y2Ba.gydF4y2Ba核糖核酸gydF4y2Ba23gydF4y2Ba, 1807-1816(2017)。gydF4y2Ba
在时间和空间上耦合mRNA加工与转录。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba15gydF4y2Ba, 163-175(2014)。gydF4y2Ba
确认gydF4y2Ba
我们感谢夏普实验室、J. Arribere、F. Solomon和L. Cote的讨论和阅读手稿。pAC4、PBNeoTetO-Dest、OVCAR4细胞、THZ531分别来自A. Cheng、S. Correa Echavarria和N. Gray。我们感谢H. Suzuki提供的第一个稳定核小体坐标,以及F. Lam在彗星实验中的协助。这里显示的结果部分基于TCGA研究网络生成的数据:gydF4y2Bahttp://cancergenome.nih.gov/gydF4y2Ba.我们感谢科赫研究所的Robert A. Swanson(1969)麻省理工学院生物技术中心的技术支持,特别是G. Paradis的流式细胞仪核心和S. Levine的麻省理工学院生物显微中心。这里描述的研究得到了来自NCI (P.A.S.)的计划项目拨款P01-CA042063,美国公共卫生服务拨款R01-GM034277和R01-CA133404,以及来自NCI的Koch研究所支持(核心)拨款P30-CA14051的支持。S.J.D.还获得了David H. Koch奖学金和NIH博士预科培训补助金T32-GM007287(麻省理工学院生物系)的支持。gydF4y2Ba
审核人信息gydF4y2Ba
自然gydF4y2Ba感谢R. Fisher, B. Tian和其他匿名审稿人对本工作的同行评议所作的贡献。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
s.j.d., P.L.B.和P.A.S.构想并设计了实验和分析。S.J.D.进行了实验。P.L.B.进行了计算分析。s.j.d., P.L.B.和P.A.S.分析了数据并撰写了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意:gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
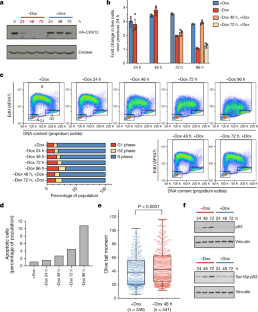
扩展数据图1gydF4y2BaCdk12gydF4y2BamES细胞的基因敲除和表型数据来自第二,独立推导gydF4y2BaCdk12gydF4y2BaΔ克隆。gydF4y2Ba
一个gydF4y2Ba,原理图gydF4y2BaCdk12gydF4y2BaΔ细胞系生成、LoxP位点(红色三角形)、sgRNA切割位点(*)、内源性启动子(黑色箭头)和强力霉素诱导启动子(橙色箭头)。gydF4y2BabgydF4y2Ba, PCR产物横跨gydF4y2BaCdk12gydF4y2Ba基因位点侧面外显子4(引物显示为橙色箭头)的野生型mES细胞和gydF4y2BaCdk12gydF4y2Ba∆克隆。在本研究中使用的克隆28和36用红色表示。gydF4y2BacgydF4y2Ba- - - - - -gydF4y2BahgydF4y2Ba,表型数据来自两个独立推导的第二个gydF4y2BaCdk12gydF4y2Ba如图所示结果所示的Δ克隆。gydF4y2Ba1 fgydF4y2Ba为了另一个克隆体。gydF4y2BacgydF4y2Ba,停用强力霉素(Dox)后CDK12转基因(HA表位)表达的代表性免疫印迹。gydF4y2BadgydF4y2Ba,流式细胞仪测定活细胞24小时内的折叠变化;柱状表示平均折叠变化(±s.e.m.)gydF4y2BangydF4y2Ba= 3个生物重复。细胞在Dox中持续生长(蓝色条),在时间0时从Dox中提取并保持在Dox外(红色条),或在时间0时从Dox中提取,在48小时(橙色条)或72小时(黄色条)后重新引入Dox,直到剩余时间。gydF4y2BaegydF4y2Ba,在相同的条件下,一个具有代表性的复制的基于facs的细胞周期分析gydF4y2BadgydF4y2Ba(上)和量化(下)。gydF4y2BafgydF4y2Ba,基于流式细胞术的caspase 3阳性(凋亡)细胞定量分析。一个有代表性的生物复制。gydF4y2BaggydF4y2Ba中性彗星法量化未修复DNA双链断裂程度gydF4y2BaCdk12gydF4y2Ba停用强力霉素48小时后,Δ细胞。箱线图:第25和第75个四分位数的中值;胡须显示最小值到最大值。gydF4y2BaPgydF4y2Ba基于单边曼-惠特尼的价值gydF4y2BaUgydF4y2Ba测试。gydF4y2BahgydF4y2Ba,免疫印迹检测CDK12缺失时总Ser15 (P-Ser15) p53和磷酸化的p53。HSP90作为负载控制。gydF4y2Ba
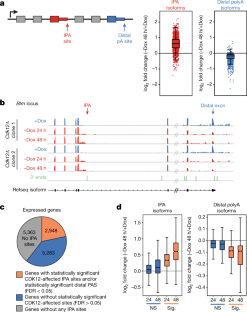
图2 cdk12缺失mES细胞的基因表达变化主要由IPA使用增加引起。gydF4y2Ba
一个gydF4y2Ba,停药24小时(左)或48小时(右)后总基因水平显著基因表达事件的火山图。gydF4y2BaygydF4y2Ba设在:FDR-adjustedgydF4y2BaPgydF4y2Ba由R中的DESeq包确定的值;彩色点:PPDE > 0.95(由EBSeq包在R中确定)。gydF4y2BabgydF4y2Ba,饼图显示了停药24或48 h后总基因表达量下降(左)或增加(右)的基因。可能的次要影响表明:p53抑制基因(红色),p53增强基因(蓝色),二价启动子基因(黄色),p53抑制和二价启动子基因(橙色),p53增强和二价启动子基因(绿色)。不属于上述任何一类的基因用灰色表示。gydF4y2BacgydF4y2Ba,总结了24和48小时后观察到的重要的替代剪接事件gydF4y2BaCdk12gydF4y2BaΔ细胞。gydF4y2BadgydF4y2Ba- - - - - -gydF4y2BaggydF4y2Ba,亚型特异性RT-qPCR证实了RNA测序数据中观察到的差异亚型使用。蓝柱(+Dox)和红柱(-Dox 48 h)代表平均值(±s.e.m.),gydF4y2BangydF4y2Ba= 4个生物重复。来自五个基因的七个IPA异构体在两个独立的实验中得到验证gydF4y2BaCdk12gydF4y2BaΔ克隆在gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba并在这五个基因中验证了相应的远端多聚腺苷酸异构体gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2Ba.gydF4y2BadgydF4y2Ba,gydF4y2BafgydF4y2Ba而且gydF4y2BaegydF4y2Ba,gydF4y2BaggydF4y2Ba表示两个独立推导出的相应数据gydF4y2BaCdk12gydF4y2Ba在整个研究过程中使用了Δ克隆。gydF4y2BahgydF4y2Ba,左,IPA站点显示有统计学意义(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05, FDR调整gydF4y2BaPgydF4y2BaR中DEXSeq包测定的值在Dox消耗24或48 h后,在表达基因中改变(橙色)或不改变(蓝色)。右,表达的末端多聚腺苷酸位点显著改变(橙色)或与其余转录本归一化后无统计学意义(蓝色)的基因。gydF4y2Ba我gydF4y2Ba,散点图显示日志gydF4y2Ba2gydF4y2Ba远端外显子CDK12缺失后的折叠变化(gydF4y2BaygydF4y2Ba-轴)与IPA位点(gydF4y2BaxgydF4y2Ba-轴)的基因具有统计学意义上显著的(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05) IPA位点及远端多聚腺苷酸改变有统计学意义;gydF4y2BangydF4y2Ba=每个条件4个生物重复。gydF4y2Ba
扩展数据图3识别相同靶蛋白的ChIP抗体表现出强烈重叠的元基因模式。gydF4y2Ba
用单个抗体分解的元基因谱。蓝线:中特定ChIP抗体的归一化读密度gydF4y2BangydF4y2Ba= 2个生物重复。橙色线:阴性对照(结合全细胞提取物和所有抗体阴性对照,gydF4y2BangydF4y2Ba= 4个生物重复)。黑色虚线:折叠富集(特定ChIP/阴性对照)。阴影区域:−loggydF4y2Ba10gydF4y2Ba(bin-wisegydF4y2BaPgydF4y2Ba值,Kolmogorov-Smirnov单侧检验)的读深差异,蓝色阴影表示正CDK12信号明显更大。−日志gydF4y2Ba10gydF4y2Ba的gydF4y2BaPgydF4y2Ba值显示在右边的轴上,水平虚线为gydF4y2BaPgydF4y2Ba= 0.05。TSS,转录起始位点。远端聚a,远端聚腺苷酸位点。gydF4y2Ba
图4 ChIP实验与数据分析示意图。gydF4y2Ba
一个gydF4y2Ba、生物复制和抗体复制实验设计示意图。每个芯片组(RNAPII和Ser2p,在CDK12gydF4y2Ba+gydF4y2Ba或CDK12gydF4y2Ba−gydF4y2Ba细胞)由两个生物重复组成,每个芯片上都有两种识别相同蛋白质的不同抗体。然后将这4个重复组合用于ChIP宏基因分析。gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba,用于确定ChIP测定的平均读密度的步骤示意图,以及用于确定依赖于CDK12表达的读密度的显著差异的统计检验。gydF4y2Ba
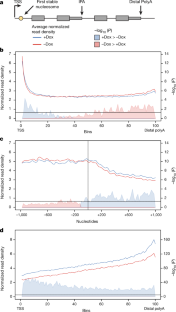
图5 RNAPII元基因模式受基因长度和表达的影响。gydF4y2Ba
一个gydF4y2Ba、出现显著变化的基因长度和表达量四分位数(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05, FDR调整gydF4y2BaPgydF4y2Ba在IPA或远端异构体中由DEXSeq包在R中确定的值)。箱线图:中位数,第25和第75四分位数;胡须显示1.5 ×四分位范围。gydF4y2BangydF4y2Ba=每个条件4个生物重复。顶部面板:大小分布(对数gydF4y2Ba10gydF4y2Ba每个长度四分位数(左)和基因表达分布(loggydF4y2Ba10gydF4y2Ba每个表达四分位数(右)与所有表达基因的各自分布进行比较。底部面板:每个长度四分位数的表达式分布(左)和每个表达式四分位数的长度分布(右)。请注意,基因长度通常与表达水平呈负相关,但显著变化的IPA/远端亚型的所有四分位的中位表达均高于所有表达基因的中位表达。此外,显著变化的IPA/远端异构体的所有表达四分位数的中位数长度都长于所有表达基因的中位数。因此,组成显著变化的IPA/远端亚型集的基因比更广泛的基因群的长度更长,表达水平更高。gydF4y2BabgydF4y2Ba,在具有统计学意义的cdk12敏感IPA或末端位点的基因中,RNAPII密度的宏基因谱分为基于长度的四分位数。在最短的四分位数中,CDK12缺失的细胞在3 '端呈密度增加的趋势,但在聚合酶达到比CDK12活性细胞更高的密度之前,最短的基因终止。相反,最长的基因表达在较低的水平(见gydF4y2Ba一个gydF4y2Ba),导致RNAPII ChIP讯号降低。由于这些原因,最短和最长的四分位数被排除在图中。gydF4y2Ba3 b、dgydF4y2Ba.gydF4y2BacgydF4y2Ba,在具有统计学意义的cdk12敏感IPA或末端位点的基因中,RNAPII密度的宏基因谱分为基于表达的四分位数。gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba,实线表示归一化读密度(蓝色,gydF4y2BangydF4y2Ba= 4个独立芯片)或没有(红色,gydF4y2BangydF4y2Ba= 4个独立芯片)CDK12;阴影区域表示−loggydF4y2Ba10gydF4y2Ba(bin-wisegydF4y2BaPgydF4y2Ba值,Kolmogorov-Smirnov单侧检验)的读密度差异(蓝色表示CDK12 .gydF4y2Ba+gydF4y2Ba信号较大,粉红色表示CDK12gydF4y2Ba−gydF4y2Ba信号更大)。水平虚线:gydF4y2BaPgydF4y2Ba= 0.05。DPA,远端多聚腺苷酸位点。gydF4y2Ba
扩展数据图6 RNAPII ChIP模式不特异性于cdk12敏感IPAs基因。gydF4y2Ba
一个gydF4y2Ba,一组对照基因中RNAPII密度的Metagene谱与IPA或远端异构体显著变化的基因组长度匹配。顶部,所有控制基因。底部,去掉最短和最长的四分位数(如图所示)。gydF4y2Ba3 bgydF4y2Ba).gydF4y2BabgydF4y2Ba,一组对照基因中RNAPII密度的Metagene谱与IPA或远端异构体显著变化的基因组长度匹配,分为长度四分位数。gydF4y2BacgydF4y2Ba,一组对照基因表达中RNAPII密度的Metagene谱,与IPA或远端异构体显著变化的基因组相匹配。gydF4y2BadgydF4y2Ba,一组对照基因表达RNAPII密度的Metagene谱,与IPA或远端异构体显著变化的基因组相匹配,分为表达四分位数。gydF4y2Ba模拟gydF4y2Ba,实线表示归一化读密度(蓝色,gydF4y2BangydF4y2Ba= 4个独立芯片)或没有(红色,gydF4y2BangydF4y2Ba= 4个独立芯片)CDK12;阴影区域表示−loggydF4y2Ba10gydF4y2Ba(bin-wisegydF4y2BaPgydF4y2Ba值,Kolmogorov-Smirnov单侧检验)的读密度差异(蓝色表示CDK12 .gydF4y2Ba+gydF4y2Ba信号较大,粉红色表示CDK12gydF4y2Ba−gydF4y2Ba信号更大)。水平虚线:gydF4y2BaPgydF4y2Ba= 0.05。gydF4y2Ba
扩展数据图7在所有基因表达四分位数中,第一稳定核小体上游RNAPII增加,下游RNAPII减少。gydF4y2Ba
对于IPA或远端亚型发生显著变化的基因,第一个稳定核小体上游和下游1 kb的RNAPII总宏基因密度,分为基因表达四分位。如Fig。gydF4y2Ba3 cgydF4y2Ba,实线表示有(蓝色)或没有(红色)CDK12的标准化读深度,阴影区域表示−loggydF4y2Ba10gydF4y2Ba(bin-wisegydF4y2BaPgydF4y2Ba(Kolmogorov-Smirnov单侧检验),蓝色阴影表示正CDK12信号明显更大,粉色阴影表示负CDK12信号明显更大。水平虚线为gydF4y2BaPgydF4y2Ba= 0.05。垂直虚线表示第一个稳定核小体二分体的位置。gydF4y2BangydF4y2Ba=每个条件4个生物重复。gydF4y2Ba
扩展数据图8 Ser2p因CDK12缺失而耗尽,宏基因模式受基因表达和长度的影响。gydF4y2Ba
左为Ser2p RNAPII基因密度的宏基因谱,具有统计学意义(gydF4y2BaPgydF4y2Ba邻接的gydF4y2Ba< 0.05, FDR调整gydF4y2BaPgydF4y2Ba由DEXSeq包在R中确定的值)cdk12敏感IPA或末端位点分为基于长度的四分位数。如Fig。gydF4y2Ba3 dgydF4y2Ba,蓝色实线表示CDK12的平均归一化读密度gydF4y2Ba+gydF4y2Ba红色实线为cdk12缺失样本的平均归一化读密度。浅蓝色阴影表示正CDK12信号明显更大。gydF4y2BangydF4y2Ba=每个条件4个生物重复。右,在具有统计学意义的cdk12敏感IPA或末端位点的基因中,Ser2p RNAPII密度的宏基因谱被划分为基于表达的四分位数。gydF4y2BangydF4y2Ba=每个条件4个生物重复。gydF4y2Ba
扩展数据图9 cdk12依赖的基因表达效应模型。gydF4y2Ba
顶部,由于RNAPII通过包含IPA位点(红色八边形)的基因区域转录(蓝色为外显子区域,5 '和3 '剪接位点(SS),灰色为内含子),cdk12依赖的RNAPII- ctd Ser2磷酸化抑制IPA位点的使用。底部,在CDK12缺失的情况下,RNAPII-CTD Ser2磷酸化降低。IPA位点使用增加,导致截断亚型增加,远端亚型减少。通过下游外显子转录的RNAPII向基因的3 '端聚集,密度不断增加。IPA的使用与其内含子的剪接存在竞争。降低剪接效率或提高裂解和聚腺苷酸活性均可增加IPA的使用量。另外,转录延伸效率的降低可能会改变动力学平衡,有利于IPA的使用。事实上,先前的研究表明,由于聚合酶突变或转录延伸因子的改变,RNAPII较慢的延伸率增加了IPA的使用gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba.所有这三种可能性都与RNAPII Ser2p有关,但尚不清楚cdk12依赖的Ser2p磷酸化如何与这些非互斥的可能性相关。gydF4y2Ba
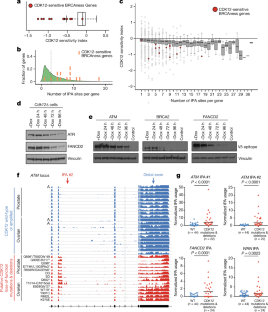
扩展数据图10上调IPA在人类肿瘤中的使用是特定于gydF4y2BaCDK12gydF4y2BaLOF突变,其他BRCAness基因无突变;用THZ531表型复制治疗人类卵巢癌和前列腺癌细胞系,观察到IPA位点使用增加gydF4y2BaCDK12gydF4y2Ba遗传的损失。gydF4y2Ba
一个gydF4y2Ba在cdk12敏感的IPA位点有突变状态的人前列腺癌或卵巢囊腺癌患者的TCGA肿瘤的RNA-seq读取密度gydF4y2Ba自动取款机gydF4y2Ba轨迹(gydF4y2Ba自动取款机gydF4y2Ba异丙醇# 2)。蓝色显示的肿瘤是野生型gydF4y2BaCDK12gydF4y2Ba和二倍体,除非标记为扩增(A)。红色所示的肿瘤携带错误意义的假定驱动突变,截断突变,或浅(SD)或深(DD)基因缺失gydF4y2BaCDK12gydF4y2Ba轨迹。值得注意的是,所有卵巢囊腺癌所携带的肿瘤gydF4y2BaCDK12gydF4y2Ba点突变也有一个浅的缺失gydF4y2BaCDK12gydF4y2Ba除了肿瘤gydF4y2BaCDK12gydF4y2Ba(R882L)错义突变,在基因座上为二倍体。橙色的23个肿瘤包含其他BRCAness基因的假定驱动突变(gydF4y2Ba自动取款机gydF4y2Ba,gydF4y2Ba乳腺癌易感基因1gydF4y2Ba,gydF4y2BaBRCA2gydF4y2Ba,gydF4y2BaFANCAgydF4y2Ba,或gydF4y2BaCHEK2gydF4y2Ba)。gydF4y2BabgydF4y2Ba两种不同IPA位点在人体内使用的定量研究gydF4y2Ba自动取款机gydF4y2Ba在IPA网站gydF4y2BaFANCD2gydF4y2Ba而且gydF4y2BaWRNgydF4y2Ba在人类前列腺和卵巢肿瘤中使用TCGA数据(在本分析中结合)。野生型或放大型肿瘤gydF4y2BaCDK12gydF4y2Ba为蓝色(WT),其中gydF4y2BaCDK12gydF4y2Ba红色(Mut)基因缺失、错义突变或截断突变,以及5个BRCAness基因中假定的驱动突变(gydF4y2Ba自动取款机gydF4y2Ba,gydF4y2Ba乳腺癌易感基因1gydF4y2Ba,gydF4y2BaBRCA2gydF4y2Ba,gydF4y2BaFANCAgydF4y2Ba,gydF4y2BaCHEK2gydF4y2Ba)。中位数由水平黑条表示,样本量如下所示。gydF4y2BaPgydF4y2Ba数值由单边曼-惠特尼法确定gydF4y2BaUgydF4y2Ba测试。gydF4y2BacgydF4y2Ba免疫印迹显示,THZ531治疗4小时对两种前列腺癌细胞系(22RV1和PC-3)和一种高级别浆液性卵巢癌细胞系(OVCAR4)中RNAPII pSer2 (3E10抗体)的影响。总RNAPII (8WG16抗体),HSP90和Vinculin显示为加载对照。gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba, isoforms -specific RT-qPCR用于检测两种前列腺癌细胞株(22RV1和PC-3)和一种高级别浆液性卵巢癌细胞株(OVCAR4)经400 nM THZ531处理4小时后IPA和远端多聚腺苷酸异构体的表达。蓝色柱状(DMSO)和红色柱状(THZ531)代表的平均值(±s.e.m.)gydF4y2BangydF4y2Ba= 3个生物重复。gydF4y2BadgydF4y2Ba,测定了4个IPA位点。在人类卵巢和前列腺肿瘤的TCGA数据中发现了三个IPA位点(gydF4y2Ba自动取款机gydF4y2Ba异丙醇# 1,gydF4y2BaFANCD2gydF4y2Ba异丙醇,gydF4y2BaWRNgydF4y2Ba异丙醇;无花果。gydF4y2Ba4 f, ggydF4y2Ba和扩展数据图。gydF4y2Ba10 a, bgydF4y2Ba).在我们的mES细胞中,一个IPA位点对应着一个显著变化的IPA位点gydF4y2BaCdk12gydF4y2BaΔ克隆(APAF1)。gydF4y2BaegydF4y2Ba中基因远端多聚腺苷酸异构体gydF4y2BadgydF4y2Ba.gydF4y2Ba
补充信息gydF4y2Ba
补充数据gydF4y2Ba
该文件包含补充图1-2。补充图1包括图1b、1c、1d和扩展数据图1d、1e、1f中使用的FACS门控策略示例。补充图2包含了图1a, 1f, 4d, 4e和扩展数据图1c, 1h, 10c未裁剪的Western blots。gydF4y2Ba
补充表1gydF4y2Ba
Cdk12缺失24小时后总基因表达量的变化;Cdk12缺失48小时后总基因表达变化;基因分类为p53转录靶基因或二价启动子基因。gydF4y2Ba
补充表2gydF4y2Ba
全基因组内含子多聚腺苷酸(IPA)切割位点。gydF4y2Ba
补充表3gydF4y2Ba
内含子多聚腺苷酸位点(IPA)在Cdk12耗尽24或48小时后发生显著变化。gydF4y2Ba
补充表4gydF4y2Ba
Cdk12耗尽24或48小时后远端多聚腺苷酸位点亚型发生显著变化。gydF4y2Ba
补充表5gydF4y2Ba
sgRNA和用于制造Cdk12-Flox和Cdk12Δ v5标记的Atm、Brca2和Fancd2细胞系的修复结构。gydF4y2Ba
补充表6gydF4y2Ba
qPCR引物序列列表。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Dubbury, s.j., Boutz, P.L. & Sharp, P.A. CDK12通过抑制内含子多聚腺苷酸调节DNA修复基因。gydF4y2Ba自然gydF4y2Ba564gydF4y2Ba, 141-145(2018)。https://doi.org/10.1038/s41586-018-0758-ygydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-018-0758-ygydF4y2Ba
关键字gydF4y2Ba
- Intronic聚腺苷酸化gydF4y2Ba
- 到损耗gydF4y2Ba
- 远端聚腺苷酸位点gydF4y2Ba
- 流式细胞仪门控策略gydF4y2Ba
- 部分外显子gydF4y2Ba
这篇文章被引用gydF4y2Ba
rna介导的免疫治疗调节肿瘤免疫微环境:下一波癌症治疗gydF4y2Ba
分子癌症gydF4y2Ba(2022)gydF4y2Ba
在braf突变的黑素瘤中,CDK12被过度激活并成为合成致死靶点gydF4y2Ba
自然通讯gydF4y2Ba(2022)gydF4y2Ba
转录相关的周期蛋白依赖激酶作为前列腺癌的治疗靶点gydF4y2Ba
致癌基因gydF4y2Ba(2022)gydF4y2Ba
CDK12促进肿瘤发生,但在乳腺癌中诱导对抑制叶酸一碳代谢的疗法的脆弱性gydF4y2Ba
自然通讯gydF4y2Ba(2022)gydF4y2Ba
增强子调节3 '端加工活性以控制替代3 ' utr异构体的表达gydF4y2Ba
自然通讯gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
