摘要gydF4y2Ba
遗传稳健性,或有机体在有害突变存在时保持适应性的能力,可以通过蛋白质反馈回路来实现。先前的研究表明,生物体也可能通过转录适应对突变做出反应,转录适应是一个相关基因独立于蛋白质反馈回路上调的过程。然而,转录适应的普遍性及其潜在的分子机制尚不清楚。在这里,通过分析斑马鱼和小鼠的几种转录适应模型,我们揭示了突变mRNA降解的需求。不能转录突变基因的等位基因不表现出转录适应,这些等位基因比显示突变mRNA衰变的等位基因产生更严重的表型。在显示突变mRNA衰变的等位基因的转录组分析显示,与突变基因的mRNA序列相似的基因中有相当一部分上调,这表明了一种序列依赖机制。这些发现有助于我们理解致病突变,并有助于设计具有最小转录适应衍生补偿的突变等位基因。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
crispr - cas9介导的F0筛选识别斑马鱼视网膜色素上皮中的促再生基因gydF4y2Ba
科学报告gydF4y2Ba开放获取gydF4y2Ba2023年2月23日gydF4y2Ba
房室瓣膜的形成需要多个pkd和piezo基因家族成员gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2023年1月13日gydF4y2Ba
使用RagTag的自动化组装脚手架提升了一个新的番茄系统,用于高通量基因组编辑gydF4y2Ba
基因组生物学gydF4y2Ba开放获取gydF4y2Ba2022年12月15日gydF4y2Ba
访问选项gydF4y2Ba
订阅《自然》+gydF4y2Ba
立即在线访问《自然》和其他55种《自然》杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅期刊gydF4y2Ba
获得1年的完整期刊访问权限gydF4y2Ba
199.00美元gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
增值税稍后将在结帐时添加。gydF4y2Ba
税务计算将在结账时完成。gydF4y2Ba
买条gydF4y2Ba
在ReadCube上获得时间限制或全文访问。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
数据可用性gydF4y2Ba
ATAC-seq和RNA-seq数据根据登录代码存入基因表达Omnibus (GEO)gydF4y2BaGSE107075gydF4y2Ba而且gydF4y2BaGSE114212gydF4y2Ba.gydF4y2Ba
改变历史gydF4y2Ba
4月17日gydF4y2Ba
在本文中,补充表和源数据文件最初是按照错误的顺序上传的。gydF4y2Ba
参考文献gydF4y2Ba
冗余、发展和信息流。gydF4y2BaBioEssaysgydF4y2Ba14gydF4y2Ba, 263-266(1992)。gydF4y2Ba
Barabási, A. L. & oltai, Z. N.网络生物学:了解细胞的功能组织。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba5gydF4y2Ba, 101-113(2004)。gydF4y2Ba
滕,X.等。删除任何单个基因的全基因组后果。gydF4y2Ba摩尔。细胞gydF4y2Ba52gydF4y2Ba, 485-494(2013)。gydF4y2Ba
罗西等人。有害突变引起的遗传补偿,而不是基因敲除。gydF4y2Ba自然gydF4y2Ba524gydF4y2Ba, 230-233(2015)。gydF4y2Ba
遗传补偿:一种寻找机制的现象。gydF4y2Ba公共科学图书馆麝猫gydF4y2Ba.gydF4y2Ba13gydF4y2Ba, e1006780(2017)。gydF4y2Ba
Isken, O. & Maquat, L. E.真核生物mRNA的质量控制:保护细胞免受异常mRNA功能的影响。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba21gydF4y2Ba, 1833-1856(2007)。gydF4y2Ba
慕克吉,C.等人。鉴定细胞质帽靶揭示了帽内稳态在翻译和mRNA稳定性中的作用。gydF4y2Ba细胞的报道gydF4y2Ba2gydF4y2Ba, 674-684(2012)。gydF4y2Ba
Boehm, V., Gerbracht, J. V., Marx, M. C. & Gehring, N. H.用xrn1抗性序列询问不稳定mrna的降解途径。gydF4y2BaNat。CommungydF4y2Ba.gydF4y2Ba7gydF4y2Ba, 13691(2016)。gydF4y2Ba
Doi, t.s.等人。缺乏肿瘤坏死因子可使rela缺陷小鼠免于胚胎死亡。gydF4y2Ba国家科学院学报美国gydF4y2Ba96gydF4y2Ba, 2994-2999(1999)。gydF4y2Ba
Hao, S. & Baltimore, D. mRNA的稳定性影响编码炎症分子的基因诱导的时间顺序。gydF4y2BaNat。ImmunolgydF4y2Ba.gydF4y2Ba10gydF4y2Ba, 281-288(2009)。gydF4y2Ba
Elkon, R., Zlotorynski, E., Zeller, K. I. & Agami, R. mRNA稳定性在塑造基因诱导动力学中的主要作用。gydF4y2BaBMC基因组学gydF4y2Ba11gydF4y2Ba, 259(2010)。gydF4y2Ba
Rabani, M.等。RNA的代谢标记揭示了哺乳动物细胞中RNA产生和降解动态的原理。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba29gydF4y2Ba, 436-442(2011)。gydF4y2Ba
孙,等。比较动态转录组分析(cDTA)揭示了mRNA合成和降解之间的相互反馈。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba22gydF4y2Ba, 1350-1359(2012)。gydF4y2Ba
柯林斯,S. R.等。使用遗传相互作用图对酵母染色体生物学中涉及的蛋白质复合物进行功能解剖。gydF4y2Ba自然gydF4y2Ba446gydF4y2Ba, 806-810(2007)。gydF4y2Ba
Berretta, J., Pinskaya, M. & Morillon, A.一个神秘的不稳定转录本介导转录gydF4y2Ba反式gydF4y2Ba-沉默的Ty1逆转录转座子gydF4y2Ba酿酒酵母gydF4y2Ba.gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba22gydF4y2Ba, 615-626(2008)。gydF4y2Ba
Pinskaya, M., Gourvennec, S. & Morillon, A. H3赖氨酸4二甲基化和三甲基化通过隐转录沉积减弱启动子激活。gydF4y2BaEMBO JgydF4y2Ba.gydF4y2Ba28gydF4y2Ba, 1697-1707(2009)。gydF4y2Ba
海莫维奇,G.等人。基因表达是圆形的:信使rna降解的因素也促进信使rna合成。gydF4y2Ba细胞gydF4y2Ba153gydF4y2Ba, 1000-1011(2013)。gydF4y2Ba
Ghanbarian, H.等。小rna引导的胚胎干细胞心脏分化的表观遗传编程。gydF4y2Ba科学。代表gydF4y2Ba.gydF4y2Ba7gydF4y2Ba, 41799(2017)。gydF4y2Ba
Modarresi, F.等人。自然反义转录的抑制gydF4y2Ba在活的有机体内gydF4y2Ba结果基因特异性转录上调。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba30.gydF4y2Ba, 453-459(2012)。gydF4y2Ba
艾森史密斯,R. C. & Woo, S. L.苯丙酮尿症和相关高苯丙氨酸血症的分子基础:人类苯丙氨酸羟化酶基因的突变和多态性。gydF4y2Ba嗡嗡声。MutatgydF4y2Ba.gydF4y2Ba1gydF4y2Ba, 13-23(1992)。gydF4y2Ba
海克斯A基因的致病突变和中性多态性。gydF4y2Ba嗡嗡声。MutatgydF4y2Ba.gydF4y2Ba9gydF4y2Ba, 195-208(1997)。gydF4y2Ba
Genschel, J. & Schmidt, H. H.编码层膜A/C的LMNA基因突变。gydF4y2Ba嗡嗡声。MutatgydF4y2Ba.gydF4y2Ba16gydF4y2Ba, 451-459(2000)。gydF4y2Ba
Chuzhanova, N. A., Anassis, E. J., Ball, E. V., Krawczak, M. & Cooper, D. N. indels引起人类遗传疾病的元分析:突变机制和局部DNA序列复杂性的作用。gydF4y2Ba嗡嗡声。MutatgydF4y2Ba.gydF4y2Ba21gydF4y2Ba, 28-44(2003)。gydF4y2Ba
Ferec, C. & Cutting, G. R.评估CFTR突变的疾病风险。gydF4y2Ba冷泉港。教谕。地中海gydF4y2Ba.gydF4y2Ba2gydF4y2Ba, a009480(2012)。gydF4y2Ba
周,Q.等。ADA2基因突变与早发性卒中和血管病变相关。gydF4y2Ba心血管病。j .地中海gydF4y2Ba.gydF4y2Ba370gydF4y2Ba, 911-920(2014)。gydF4y2Ba
Dietz, H. C.等。四种新的FBN1突变:突变转录本水平和egf样结构域钙结合在马凡综合征发病机制中的意义gydF4y2Ba基因组学gydF4y2Ba17gydF4y2Ba, 468-475(1993)。gydF4y2Ba
霍尔,G. W. & Thein, S. β-珠蛋白基因末端外显子的无意义密码子突变与β-mRNA积累的减少无关:显性β-地中海贫血表型的机制。gydF4y2Ba血gydF4y2Ba83gydF4y2Ba, 2031-2037(1994)。gydF4y2Ba
Sulem, P.等人。鉴定出一大批罕见的完整的人类敲除基因。gydF4y2BaNat麝猫。gydF4y2Ba.gydF4y2Ba47gydF4y2Ba, 448-452(2015)。gydF4y2Ba
Lek, M.等人。60706人蛋白质编码遗传变异分析。gydF4y2Ba自然gydF4y2Ba536gydF4y2Ba, 285-291(2016)。gydF4y2Ba
Gerri, C.等人。Hif-1α在斑马鱼血管发育过程中调节巨噬细胞-内皮细胞相互作用。gydF4y2BaNat。CommungydF4y2Ba.gydF4y2Ba8gydF4y2Ba, 15492(2017)。gydF4y2Ba
罗西等人。天然和合成配体对Vegf信号通路的调控。gydF4y2Ba血gydF4y2Ba128gydF4y2Ba, 2359-2366(2016)。gydF4y2Ba
劳森,n.d.和温斯坦,b.m.。gydF4y2Ba在活的有机体内gydF4y2Ba利用转基因斑马鱼胚胎血管发育成像。gydF4y2BaDev医学杂志gydF4y2Ba.gydF4y2Ba248gydF4y2Ba, 307-318(2002)。gydF4y2Ba
普洛克斯,陆,a, Sumanas, S.斑马鱼形式的脑血管通过血管母细胞簇衍生的血管生成。gydF4y2BaDev医学杂志gydF4y2Ba.gydF4y2Ba348gydF4y2Ba, 34-46(2010)。gydF4y2Ba
Labun, K., Montague, T. G., Gagnon, J. A., Thyme, S. B. & Valen, E. CHOPCHOP v2:下一代CRISPR基因组工程的网络工具。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba44gydF4y2Ba, w272-w276(2016)。gydF4y2Ba
瑟马克,T.等人。高效设计和组装定制TALEN和其他基于TAL效应的DNA靶向结构。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba39gydF4y2Ba, e82(2011)。gydF4y2Ba
道尔,e.l.等人。通过替换隐重复序列−1中的色氨酸,DNA靶碱基0的TAL效应子特异性以一种复杂的、效应子和分析依赖的方式改变。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba8gydF4y2Ba, e82120(2013)。gydF4y2Ba
加农,J. A.等。Cas9蛋白介导的寡核苷酸插入有效诱变和单导向rna的大规模评估。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba9gydF4y2Ba, e98186(2014)。gydF4y2Ba
Vejnar, c.e., Moreno-Mateos, m.a., Cifuentes, D., Bazzini, a.a. & Giraldez, a.j.优化的CRISPR-Cas9系统用于斑马鱼基因组编辑。冷泉港。Protoc。gydF4y2Bahttps://doi.org/10.1101/pdb.prot086850gydF4y2Ba(2016)。gydF4y2Ba
狄奥多西等人。Kindlin-2与talin协同激活整合素,通过直接结合paxillin诱导细胞扩散。gydF4y2BaeLifegydF4y2Ba5gydF4y2Ba, e10130(2016)。gydF4y2Ba
Gapuzan, m.e., Yufit, P. V. & Gilmore, T. D.永不朽的胚胎小鼠成纤维细胞缺乏转录因子NF-κB的RelA亚基,具有恶性转化的表型。gydF4y2Ba致癌基因gydF4y2Ba21gydF4y2Ba, 2484-2492(2002)。gydF4y2Ba
Ran, F. A.等。利用CRISPR-Cas9系统进行基因组工程。gydF4y2BaProtoc Nat。gydF4y2Ba8gydF4y2Ba, 2281-2308(2013)。gydF4y2Ba
Thakore, p.i.等人。通过CRISPR-Cas9阻遏物对远端调控元件进行高度特异性的表观基因组编辑。gydF4y2BaNat方法。gydF4y2Ba12gydF4y2Ba, 1143-1149(2015)。gydF4y2Ba
马丁,L.等。鉴定和鉴定抑制无意义RNA衰变和抑制无意义p53突变的小分子。gydF4y2Ba癌症ResgydF4y2Ba.gydF4y2Ba74gydF4y2Ba, 3104-3113(2014)。gydF4y2Ba
Rädle, B.等。新转录RNA的代谢标记,用于细胞培养中RNA合成、加工和衰变的高分辨率基因表达谱。gydF4y2BaJ. Vis. ExpgydF4y2Ba.gydF4y2Ba78gydF4y2Ba, e50195(2013)。gydF4y2Ba
Sun, W. & Chen, W.新合成RNA的代谢标记与4sU并行评估RNA的转录和衰变。gydF4y2Ba方法分子生物学gydF4y2Ba.gydF4y2Ba1720gydF4y2Ba, 25-34(2018)。gydF4y2Ba
Vanhollebeke等人。在大脑血管生成过程中,尖端细胞对非典型Gpr124和reck依赖的Wnt/β-catenin通路的特异性要求。gydF4y2BaeLifegydF4y2Ba4gydF4y2Ba, e06489(2015)。gydF4y2Ba
权,H. B.等。Apelin受体信号在体内对内皮细胞极化的调节。gydF4y2BaNat。CommungydF4y2Ba.gydF4y2Ba7gydF4y2Ba, 11805(2016)。gydF4y2Ba
Blecher-Gonen, R.等人。高通量染色质免疫沉淀用于体内蛋白质- dna相互作用和表观基因组状态的全基因组映射。gydF4y2BaProtoc Nat。gydF4y2Ba8gydF4y2Ba, 539-554(2013)。gydF4y2Ba
Buenrostro, J. D., Giresi, P. G., Zaba, L. C., Chang, H. Y. & Greenleaf, W. J.天然染色质转座用于开放染色质,dna结合蛋白和核小体位置的快速和敏感表观基因组分析。gydF4y2BaNat方法。gydF4y2Ba10gydF4y2Ba, 1213-1218(2013)。gydF4y2Ba
Bolger, a.m., Lohse, M. & Usadel, B. Trimmomatic:用于Illumina序列数据的灵活修剪器。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 2114-2120(2014)。gydF4y2Ba
Dobin, A.等人。STAR:超快通用RNA-seq对准器。gydF4y2Ba生物信息学gydF4y2Ba29gydF4y2Ba, 15-21(2013)。gydF4y2Ba
罗宾逊,J. T.等。整合基因组学查看器。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba29gydF4y2Ba, 24-26(2011)。gydF4y2Ba
Ramírez, F., Dündar, F., Diehl, S., Grüning, B. a. & Manke, T. deepTools:用于探索深度测序数据的灵活平台。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba42gydF4y2Ba, w187-w191(2014)。gydF4y2Ba
安德斯,S. & Huber, W.序列计数数据的差分表达式分析。gydF4y2Ba基因组医学杂志gydF4y2Ba.gydF4y2Ba11gydF4y2Ba, r106(2010)。gydF4y2Ba
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 841-842(2010)。gydF4y2Ba
Davis, M. P., van Dongen, S., abreuu - goodger, C., Bartonicek, N. & Enright, a. J. Kraken:一套高通量序列数据质量控制和分析工具。gydF4y2Ba方法gydF4y2Ba63gydF4y2Ba, 41-49(2013)。gydF4y2Ba
Liao, Y., Smyth, G. K. & Shi, W. featuremets:一种高效的通用程序,用于分配序列读取基因组特征。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 923-930(2014)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志gydF4y2Ba.gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
阿特舒,S. F.,吉什,W.,米勒,W.,迈尔斯,E. W. &李普曼,D. J.基本的局部对齐搜索工具。gydF4y2BaJ. Mol.生物学gydF4y2Ba.gydF4y2Ba215gydF4y2Ba, 403-410(1990)。gydF4y2Ba
温特辛格,J. A.和Wasmuth, J. D. Kablammo:一个交互式的,基于web的BLAST结果可视化工具。gydF4y2Ba生物信息学gydF4y2Ba31gydF4y2Ba, 1305-1306(2015)。gydF4y2Ba
MUSCLE:一种减少时间和空间复杂度的多序列比对方法。gydF4y2BaBMC生物信息学gydF4y2Ba5gydF4y2Ba, 113(2004)。gydF4y2Ba
谢,C.等。KOBAS 2.0:用于注释和识别丰富途径和疾病的web服务器。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba39gydF4y2Ba, w316-w322(2011)。gydF4y2Ba
确认gydF4y2Ba
我们感谢V. Serobyan、F. Mueller、Z. Jiang、A. Beisaw和F. Gunawan对手稿的讨论和评论;J. Pestel的gydF4y2BaalcamagydF4y2Ba突变体;A. Atzberger支持单元排序;N. Gehring和V. Böhm提供抗xrn1序列质粒。M.A.E.-B。得到勃林格殷格翰基金会博士奖学金的支持。D.Y.R.S.实验室的研究得到了马克斯·普朗克学会、欧盟、DFG和Leducq基金会的支持。gydF4y2Ba
审核人信息gydF4y2Ba
自然gydF4y2Ba感谢Miles Wilkinson和其他匿名审稿人对本工作的同行评审所作的贡献。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
M.A.E.-B。设计并执行大部分实验,分析数据并撰写手稿;Z.K.和A.R.设计并进行了mESC实验和部分成像,并编辑了手稿;C.K.进行生物信息学分析;S.G.进行ATAC-seq和RNA-seq;N.F.生成了一些斑马鱼突变体,并进行了一些qPCR实验;K.K.进行了一些qPCR实验;G.L.M.B.进行了一些成像;ct产生了gydF4y2Baupf1gydF4y2Ba突变体,在A.J.G.的监督下;s.l.l.、R.F.和C.G.提供了未发表的突变体;D.Y.R.S.帮助设计实验、分析数据、监督工作并撰写手稿。所有作者都对手稿作了评论。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意:gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
扩展数据图1本研究中产生的突变等位基因示意图。gydF4y2Ba
本研究中产生的不同突变等位基因的部分DNA序列,以及凝胶图像提供了缺失rna等位基因的证据。红色表示突变(星号,缺失;大写字母,替换;小写字母,插入);绿色表示具有PTC的等位基因的终止密码子;箭头表示基因分型引物。gydF4y2Ba
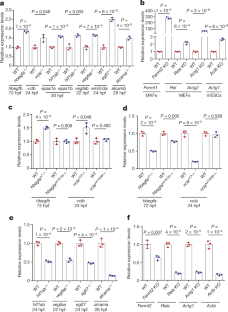
转录适应与蛋白质功能的丧失无关。gydF4y2Ba
一个gydF4y2Ba, qPCR分析gydF4y2Baepas1agydF4y2Ba,gydF4y2Baepas1bgydF4y2Ba,gydF4y2BavegfabgydF4y2Ba,gydF4y2Baemilin3agydF4y2Ba而且gydF4y2BaalcambgydF4y2Ba野生型和野生型的mRNA表达水平gydF4y2Bahif1abgydF4y2Ba,gydF4y2BavegfaagydF4y2Ba,gydF4y2Baegfl7gydF4y2Ba而且gydF4y2BaalcamagydF4y2Ba注入(注射)突变胚胎gydF4y2BaeGFPgydF4y2Ba信使rna(控制;Ctrl)或野生类型gydF4y2Bahif1abgydF4y2Ba,gydF4y2BavegfaagydF4y2Ba,gydF4y2Baegfl7gydF4y2Ba或gydF4y2BaalcamagydF4y2Ba信使rna。gydF4y2BabgydF4y2Ba, qPCR分析gydF4y2BavclbgydF4y2Ba,gydF4y2Baepas1agydF4y2Ba,gydF4y2Baepas1bgydF4y2Ba而且gydF4y2Baemilin3agydF4y2BamRNA表达水平gydF4y2BavclagydF4y2Ba,gydF4y2Bahif1abgydF4y2Ba而且gydF4y2Baegfl7gydF4y2Ba野生型,杂合和突变斑马鱼。gydF4y2BacgydF4y2Ba, qPCR分析gydF4y2BahbegfagydF4y2Ba,gydF4y2Bahif1abgydF4y2Ba,gydF4y2BavegfaagydF4y2Ba而且gydF4y2BaalcamagydF4y2BamRNA表达水平gydF4y2BahbegfagydF4y2Ba,gydF4y2Bahif1abgydF4y2Ba,gydF4y2BavegfaagydF4y2Ba而且gydF4y2BaalcamagydF4y2Ba野生型和杂合斑马鱼,使用野生型等位基因特异性引物。gydF4y2BadgydF4y2Ba, qPCR分析gydF4y2BaFermt1gydF4y2Ba而且gydF4y2BaRelgydF4y2Ba野生型和野生型的mRNA表达水平gydF4y2BaFermt2gydF4y2Ba而且gydF4y2BaRelagydF4y2Ba敲除细胞转染空载体(对照)或编码野生型FERMT2或RELA的质粒。gydF4y2BaegydF4y2Ba, Western blot分析小鼠FERMT2和ACTB水平gydF4y2BaFermt2gydF4y2Ba敲除细胞转染空载体(对照)或编码野生型FERMT2的质粒。gydF4y2BafgydF4y2BaWestern blot检测血清中RELA和ACTB水平gydF4y2BaRelagydF4y2Ba敲除细胞转染空载体(对照)或编码野生型RELA的质粒。gydF4y2BaggydF4y2Ba, qPCR分析gydF4y2BaActg1gydF4y2Ba野生型和杂合型mRNA表达水平gydF4y2BaActbgydF4y2Ba制。gydF4y2BangydF4y2Ba= 3个生物独立的样本。每个试验的野生型或对照型表达水平设置为1。数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值(gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba,gydF4y2BaggydF4y2Ba).美国的实验gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba只执行了一次。western blots的源数据见补充图。gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
转录适应包括转录的增强,并且与DNA损伤本身无关。gydF4y2Ba
一个gydF4y2Ba, qPCR分析gydF4y2BahbegfbgydF4y2Ba而且gydF4y2Baemilin3agydF4y2BamRNA和pre-mRNA表达水平gydF4y2BahbegfagydF4y2Ba而且gydF4y2Baegfl7gydF4y2Ba野生型和突变斑马鱼。gydF4y2BabgydF4y2Ba, qPCR分析gydF4y2BaFermt1gydF4y2Ba而且gydF4y2BaRelgydF4y2BamRNA和pre-mRNA表达水平gydF4y2BaFermt2gydF4y2Ba而且gydF4y2BaRelagydF4y2Ba野生型和敲除细胞。gydF4y2BacgydF4y2Ba, IGV的轨迹gydF4y2BaFermt1gydF4y2Ba显示ATAC-seq信号的基因座gydF4y2BaFermt2gydF4y2Ba淘汰赛细胞。gydF4y2BadgydF4y2Ba, qPCR分析gydF4y2BahbegfagydF4y2Ba,gydF4y2BahbegfbgydF4y2Ba,gydF4y2Baegfl7gydF4y2Ba而且gydF4y2Baemilin3agydF4y2BamRNA表达水平gydF4y2BahbegfagydF4y2Ba而且gydF4y2Baegfl7gydF4y2Ba野生型和Δ3突变斑马鱼。gydF4y2BaegydF4y2Ba, qPCR分析gydF4y2BavegfaagydF4y2Ba,gydF4y2BavegfabgydF4y2Ba,gydF4y2Baegfl7gydF4y2Ba而且gydF4y2Baemilin3agydF4y2BamRNA表达水平gydF4y2BavegfaagydF4y2Ba而且gydF4y2Baegfl7gydF4y2Ba野生型和5'UTR突变斑马鱼。gydF4y2BafgydF4y2Ba, qPCR分析gydF4y2BavclagydF4y2Ba而且gydF4y2BavclbgydF4y2BamRNA表达水平gydF4y2BavclagydF4y2Ba野生型和最后的外显子(外显子22)突变斑马鱼。gydF4y2BangydF4y2Ba= 3个生物独立的样本。每个试验的野生型表达水平设置为1。数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值。gydF4y2Ba
图4突变体转录水平的降低是由mRNA衰变引起的。gydF4y2Ba
一个gydF4y2Ba, qPCR分析gydF4y2BahbegfagydF4y2Ba,gydF4y2Baegfl7gydF4y2Ba而且gydF4y2BaalcamagydF4y2BamRNA和pre-mRNA表达水平gydF4y2BahbegfagydF4y2Ba,gydF4y2Baegfl7gydF4y2Ba而且gydF4y2BaalcamagydF4y2Ba野生型和突变斑马鱼。gydF4y2BabgydF4y2Ba, qPCR分析gydF4y2BaFermt2gydF4y2Ba而且gydF4y2BaRelagydF4y2BamRNA和pre-mRNA表达水平gydF4y2BaFermt2gydF4y2Ba而且gydF4y2BaRelagydF4y2Ba野生型和敲除细胞。gydF4y2BacgydF4y2Ba, qPCR分析4su标记gydF4y2BaFermt2gydF4y2Ba,gydF4y2BaRelagydF4y2Ba而且gydF4y2BaActg1gydF4y2BamRNA和pre-mRNA表达水平gydF4y2BaFermt2gydF4y2Ba,gydF4y2BaRelagydF4y2Ba而且gydF4y2BaActg1gydF4y2Ba野生型和敲除细胞。gydF4y2BadgydF4y2Ba的拟合指数衰减曲线gydF4y2BaFermt2gydF4y2Ba野生型和野生型的mRNA表达水平gydF4y2BaFermt2gydF4y2Ba淘汰赛细胞。gydF4y2BatgydF4y2Ba1/2gydF4y2Ba,半衰期。gydF4y2BaegydF4y2Ba的拟合指数衰减曲线gydF4y2BaRelagydF4y2Ba野生型和野生型的mRNA表达水平gydF4y2BaRelagydF4y2Ba淘汰赛细胞。gydF4y2BafgydF4y2Ba的拟合指数衰减曲线gydF4y2BaActg1gydF4y2Ba野生型和野生型的mRNA表达水平gydF4y2BaActg1gydF4y2Ba淘汰赛细胞。gydF4y2BangydF4y2Ba= 3 (gydF4y2Ba一个gydF4y2Ba,gydF4y2BabgydF4y2Ba,gydF4y2BadgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba)或gydF4y2BangydF4y2Ba= 2 (gydF4y2BacgydF4y2Ba)生物独立样本。每个试验的野生型表达水平设置为1 (a-c)。数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值。gydF4y2Ba
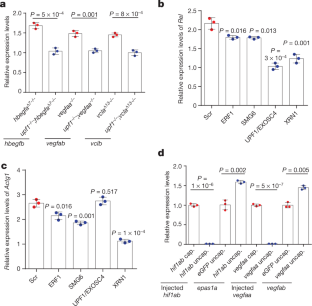
图5 RNA衰变诱导转录适应。gydF4y2Ba
一个gydF4y2Ba, qPCR分析gydF4y2BahbegfagydF4y2Ba,gydF4y2BavegfaagydF4y2Ba而且gydF4y2BavclagydF4y2BamRNA表达水平gydF4y2Baupf1; hbegfagydF4y2Ba,gydF4y2Baupf1; vegfaagydF4y2Ba而且gydF4y2Baupf1gydF4y2Ba;gydF4y2BavclagydF4y2Ba双突变斑马鱼。gydF4y2BabgydF4y2Ba, qPCR分析gydF4y2BaRelagydF4y2BamRNA表达水平在sirna介导的敲低指示蛋白gydF4y2BaRelagydF4y2Ba淘汰赛细胞。gydF4y2BacgydF4y2Ba, qPCR分析gydF4y2BaActbgydF4y2BamRNA表达水平在sirna介导的敲低指示蛋白gydF4y2BaActbgydF4y2Ba淘汰赛细胞。gydF4y2BadgydF4y2Ba, qPCR分析gydF4y2BahbegfagydF4y2Ba6 dpf mRNA表达水平gydF4y2BahbegfagydF4y2Ba用NMD抑制剂(NMDi)处理突变体。gydF4y2BaegydF4y2Ba, qPCR分析gydF4y2BahbegfbgydF4y2Ba6 dpf mRNA表达水平gydF4y2BahbegfagydF4y2Ba用NMDi处理的突变体。gydF4y2BafgydF4y2Ba, qPCR分析gydF4y2BaRelagydF4y2BamRNA表达水平gydF4y2BaRelagydF4y2Ba用环己亚胺(CHX)处理敲除细胞。gydF4y2BaggydF4y2Ba, qPCR分析gydF4y2BaRelgydF4y2BamRNA表达水平gydF4y2BaRelagydF4y2Ba用CHX处理的敲除细胞。gydF4y2BaHgydF4y2Ba, qPCR分析内源性gydF4y2Bahif1abgydF4y2Ba而且gydF4y2BavegfaagydF4y2Ba注射uncapped的6个hpf野生型胚胎mRNA表达水平gydF4y2Bahif1abgydF4y2Ba或gydF4y2BavegfaagydF4y2BaRNA。gydF4y2Ba我gydF4y2Ba, qPCR分析gydF4y2BaActg1gydF4y2Ba转染uncapped的mESCs中mRNA的表达水平gydF4y2BaActbgydF4y2Ba转染后不同时间的RNA。gydF4y2BajgydF4y2Ba, qPCR分析注射gydF4y2Bahif1abgydF4y2Ba,gydF4y2Baepas1agydF4y2Ba,注射gydF4y2BavegfaagydF4y2Ba而且gydF4y2BavegfabgydF4y2Ba6 hpf野生型胚胎中RNA的表达水平gydF4y2Bahif1abgydF4y2Ba或gydF4y2BavegfaagydF4y2Ba5 '抗xrn1 (xr)序列的转录本。gydF4y2BakgydF4y2Ba, qPCR分析gydF4y2Baepas1agydF4y2Ba而且gydF4y2BavegfabgydF4y2Ba6个hpf野生型斑马鱼胚胎mRNA表达水平的研究gydF4y2Bahif1abgydF4y2Ba或gydF4y2BavegfaagydF4y2BaRNA;相同的gydF4y2BaeGFPgydF4y2Ba脱帽致意。对照样品用于gydF4y2Baepas1agydF4y2Ba实验。每个试验的野生型或对照型表达水平设置为1 (gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba,gydF4y2BafgydF4y2Ba,gydF4y2BahgydF4y2Ba- - - - - -gydF4y2BakgydF4y2Ba).gydF4y2BangydF4y2Ba= 3个生物独立的样本。数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值。gydF4y2Ba
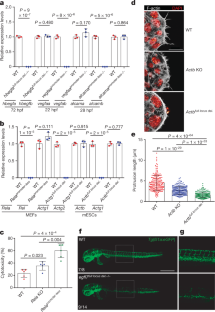
扩展数据图6突变mRNA衰变有助于遗传稳健性。gydF4y2Ba
一个gydF4y2Ba, qPCR分析gydF4y2BaFermt2gydF4y2Ba而且gydF4y2BaFermt1gydF4y2BaCRISPR干扰介导的基因敲除后的mRNA表达水平gydF4y2BaFermt2gydF4y2Ba转录的gydF4y2BaFermt2gydF4y2Ba淘汰赛细胞。gydF4y2BabgydF4y2Ba, qPCR分析gydF4y2Baemilin3agydF4y2Ba,gydF4y2Baemilin3bgydF4y2Ba而且gydF4y2Baemilin2agydF4y2Ba20个hpf野生型的mRNA表达水平gydF4y2Baegfl7gydF4y2BaΔgydF4y2Ba4gydF4y2Ba突变体,gydF4y2Baegfl7gydF4y2Ba全轨迹gydF4y2Ba.gydF4y2Ba突变体。gydF4y2BacgydF4y2Ba, 58 hpf内连接基底动脉(BA)的中央动脉(cta)数量gydF4y2BavegfaagydF4y2BaΔgydF4y2Ba10gydF4y2Ba而且gydF4y2BavegfaagydF4y2Bapromoter-lessgydF4y2Ba突变体。gydF4y2BadgydF4y2Ba, 78 hpf野生型血流速;gydF4y2BahbegfagydF4y2BaΔgydF4y2Ba7gydF4y2Ba突变体,gydF4y2BahbegfagydF4y2Ba全轨迹gydF4y2Ba.gydF4y2Ba突变体。gydF4y2BaegydF4y2Ba100 hpf野生型心室长度的定量分析gydF4y2BaalcamagydF4y2BaΔgydF4y2Ba8gydF4y2Ba突变体,gydF4y2BaalcamagydF4y2Bapromoter-lessgydF4y2Ba突变体。每个试验的野生型或对照型表达水平设置为1 (gydF4y2Ba一个gydF4y2Ba,gydF4y2BabgydF4y2Ba).gydF4y2BangydF4y2Ba= 3 (gydF4y2Ba一个gydF4y2Ba,gydF4y2BabgydF4y2Ba);gydF4y2BangydF4y2Ba= 13 (gydF4y2BavegfaagydF4y2BaΔgydF4y2Ba10gydF4y2Ba−−/gydF4y2Ba)及19 (gydF4y2BavegfaagydF4y2Bapromoter-lessgydF4y2Ba−−/gydF4y2Ba) (gydF4y2BacgydF4y2Ba);gydF4y2BangydF4y2Ba= 25 (gydF4y2BadgydF4y2Ba);而且gydF4y2BangydF4y2Ba= 18(野生型的兄弟姐妹gydF4y2BaalcamagydF4y2BaΔgydF4y2Ba8gydF4y2Ba−−/gydF4y2Ba), 7 (gydF4y2BaalcamagydF4y2BaΔgydF4y2Ba8gydF4y2Ba−−/gydF4y2Ba), 22(野生型的兄弟姐妹gydF4y2BaalcamagydF4y2Bapromoter-lessgydF4y2Ba−−/gydF4y2Ba)及15 (gydF4y2BaalcamagydF4y2Bapromoter-lessgydF4y2Ba−−/gydF4y2Ba) (gydF4y2BaegydF4y2Ba)动物。数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值。gydF4y2Ba
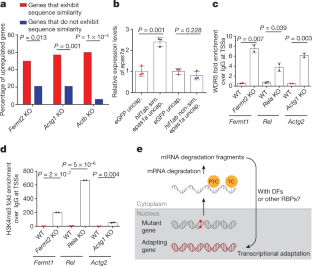
图7转录适应模型中序列相似参数的分析。gydF4y2Ba
一个gydF4y2Ba,不同敲除细胞系模型中差异表达基因的数量;gydF4y2BaPgydF4y2Ba≤0.05;这些基因分布在整个基因组中(数据未显示)。gydF4y2BabgydF4y2Ba,在3种不同的细胞系模型中,L2F敲除>野生型和基因上调的维恩图gydF4y2BaPgydF4y2Ba≤0.05。gydF4y2BacgydF4y2Ba中常见上调基因的KEGG通路富集分析gydF4y2BaFermt2gydF4y2Ba,gydF4y2BaActg1gydF4y2Ba而且gydF4y2BaActbgydF4y2Ba与野生型细胞相比的敲除。十大途径基于gydF4y2BaPgydF4y2Ba的值。虚线表示agydF4y2BaPgydF4y2Ba值为0.05。圆的大小提供了规模的估计;外层的灰色圆圈代表通路中基因的总数;中间的彩色圆圈表示通路中通常上调的基因数量。gydF4y2BadgydF4y2Ba,三种不同BLASTn对准质量参数(对准长度,位分和gydF4y2BaEgydF4y2Ba值)的意义上观察到的上调和序列相似性之间的相关性,因此识别或预测假定的适应基因。的gydF4y2BaEgydF4y2BaValue描述由偶然性产生的匹配概率(较低的值对应较低的概率),位分数评估对齐质量和长度的组合(较高的值对应较好的对齐)。的gydF4y2BaygydF4y2Ba每个图的轴表示负对数gydF4y2Ba10gydF4y2Ba的gydF4y2BaPgydF4y2Ba价值和gydF4y2BaxgydF4y2BaAxis显示了各自的参数值。一个gydF4y2BaPgydF4y2Ba数值0.05用黑色水平线标记。的gydF4y2BaEgydF4y2Ba我们分析中使用的值阈值用圆圈突出显示。行将初步结束表示该点之后没有任何剩余的对齐。第一行图探讨了阈值的巨大变化,试图确定总的范围,而第二行则关注所调查的三个基因的最相关窗口。根据所分析基因的不同,最佳阈值有很大差异。gydF4y2BangydF4y2Ba= 2个生物独立的样本。gydF4y2BaPgydF4y2Ba值通过自举随机子样本计算(参见方法的“序列相似性和子样本分析”部分)。gydF4y2BaPgydF4y2Ba多次测试的值没有校正。gydF4y2Ba
图8不同小鼠细胞系模型中序列相似基因的表达水平。gydF4y2Ba
一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba,与序列相似的基因的RNA-seq分析gydF4y2BaFermt2gydF4y2Ba(gydF4y2Ba一)gydF4y2Ba,gydF4y2BaActg1gydF4y2Ba(gydF4y2Bab)gydF4y2Ba或gydF4y2BaActbgydF4y2Ba(gydF4y2BacgydF4y2Ba)与野生型细胞相比。粗体,相对于野生型细胞敲除显著上调;红色,L2F>0,蓝色,L2F<0;绿色,gydF4y2BaPgydF4y2Ba价值或调整gydF4y2BaPgydF4y2Ba值≤0.05;紫色;在启动子区显示与突变基因mRNA序列相似的基因;黄色为3'UTR区与突变基因mRNA序列相似的基因。其他非彩色基因在其外显子或内含子中与突变基因的mRNA序列相似。盒状,基因敲除上调但没有rna缺失细胞;没有gydF4y2BaFermt2gydF4y2Ba分析RNA-less等位基因。gydF4y2BadgydF4y2Ba, qPCR分析gydF4y2BaUbaplgydF4y2Ba,gydF4y2BaFmnl2gydF4y2Ba,gydF4y2BaCdk12gydF4y2Ba而且gydF4y2BaActr1agydF4y2Ba中pre-mRNA表达水平gydF4y2BaActg1gydF4y2Ba相对于野生型细胞的敲除。gydF4y2BaegydF4y2Ba, qPCR分析gydF4y2Baactb1gydF4y2Ba无帽小鼠注射6 hpf野生型斑马鱼mRNA表达水平gydF4y2BaActbgydF4y2BaRNA。gydF4y2BafgydF4y2Ba,序列之间相似性区域的示意图表示gydF4y2Bahif1abgydF4y2BamRNA和gydF4y2Baepas1agydF4y2Ba轨迹。灰色阴影三角形表示对齐;强度代表对准质量;底部的宽度表示相似区域的长度。gydF4y2BaggydF4y2Ba, qPCR分析gydF4y2Baepas1agydF4y2Ba在6个hpf野生型斑马鱼胚胎中,mRNA的表达水平被注射了完全由dna组成的无帽RNAgydF4y2Bahif1abgydF4y2Ba与gydF4y2Baepas1agydF4y2Ba启动子、外显子、内含子或3'UTR;相同的gydF4y2BaeGFPgydF4y2Ba脱帽致意。所有比较均采用对照样本。gydF4y2BangydF4y2Ba= 2 (gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba)或gydF4y2BangydF4y2Ba= 3 (gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba,gydF4y2BaggydF4y2Ba)生物独立样本。采用DESeq2检验检验负二项广义线性模型系数的显著性,并采用Wald检验(gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba).gydF4y2BaPgydF4y2Ba多次测试的值没有校正。每个试验的野生型或对照型表达水平设置为1 (gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba,gydF4y2BaggydF4y2Ba).数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值。gydF4y2Ba
转录适应涉及依赖于衰变因子活性的染色质重塑。gydF4y2Ba
一个gydF4y2Ba, qPCR分析gydF4y2BaRelgydF4y2BamRNA表达水平在sirna介导的敲低指示蛋白gydF4y2BaRelagydF4y2Ba淘汰赛细胞。gydF4y2BabgydF4y2Ba, ChIP-qPCR分析H3K4me3在非启动子区域的占用情况(作为对照)gydF4y2BaFermt1gydF4y2Ba,gydF4y2BaRelgydF4y2Ba而且gydF4y2BaActg2gydF4y2Ba在gydF4y2BaFermtgydF4y2Ba2,gydF4y2BaRelagydF4y2Ba而且gydF4y2BaActg1gydF4y2Ba敲除细胞,分别与野生型细胞进行比较。gydF4y2BacgydF4y2Ba, ChIP-qPCR分析H3K4me3附近的占用率gydF4y2BaRelgydF4y2BaTSS和非启动子区(作为对照)在sirna介导的敲除指示蛋白后gydF4y2BaRelagydF4y2Ba淘汰赛细胞。gydF4y2BadgydF4y2Ba目前转录适应突变的扩展模型。RNA衰变片段可能作为中间产物,使衰变因子和染色质重塑因子适应基因位点,从而引发基因表达增加。另外,RNA衰变片段可能通过抑制适应基因位点上的反义RNA发挥作用,从而增加意义mRNA的表达。然而,转录适应可能还涉及其他机制,而且可能是以基因依赖的方式。gydF4y2BangydF4y2Ba= 3 (gydF4y2Ba一个gydF4y2Ba)或gydF4y2BangydF4y2Ba= 2 (gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba)生物独立样本。数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值。gydF4y2Ba
图10反义转录本在转录适应反应中的潜在作用。gydF4y2Ba
一个gydF4y2Ba, qPCR分析gydF4y2BaCdk9gydF4y2Ba而且gydF4y2BaSox9gydF4y2Ba转染uncapped基因的细胞mRNA表达水平gydF4y2BaCdk9gydF4y2Ba或gydF4y2BaSox9gydF4y2BaRNA。gydF4y2BabgydF4y2Ba, qPCR分析gydF4y2Ba脑源性神经营养因子gydF4y2Ba而且gydF4y2BaBDNF-ASgydF4y2Bauncapped转染HEK293T细胞的mRNA表达水平gydF4y2Ba脑源性神经营养因子gydF4y2BaRNA。gydF4y2BacgydF4y2Ba,集成基因组查看器跟踪gydF4y2BavclbgydF4y2Ba而且gydF4y2BahbegfbgydF4y2Ba位点,显示注释反义转录本的位置。在105和147 bp之间观察到两个对齐gydF4y2BavclagydF4y2Ba信使rna和gydF4y2BavclbgydF4y2Ba反义rna,两者之间的比对为39 bpgydF4y2BahbegfagydF4y2Ba信使rna和gydF4y2BahbegfbgydF4y2Ba反义RNA。显示的反义转录本来自GSE32898的数据集。gydF4y2BadgydF4y2Ba, qPCR分析gydF4y2BavclbgydF4y2Ba而且gydF4y2BahbegfbgydF4y2Ba反义(AS) RNA表达水平gydF4y2BavclagydF4y2Ba而且gydF4y2BahbegfagydF4y2Ba野生型和突变斑马鱼分别在24和72 hpf。每个试验的对照表达量设置为1。gydF4y2BangydF4y2Ba= 3个生物独立的样本。数据为均值±标准差,双尾学生gydF4y2BatgydF4y2Ba-test用于计算gydF4y2BaPgydF4y2Ba值。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
该文件包含补充讨论;补充数据和补充数字。合并后的补充信息PDF包括:补充讨论,2。补充数据:包括(1)斑马鱼的肌肉排列gydF4y2Baactb1gydF4y2Ba和鼠标gydF4y2BaActbgydF4y2Ba编码序列,(2)肌肉对齐合成gydF4y2Bahif1abgydF4y2Ba由类似的序列组成的转录本gydF4y2Baepas1agydF4y2Ba,和完整的gydF4y2Bahif1abgydF4y2Ba(3) BLASTn对齐gydF4y2Bahif1abgydF4y2Ba记录和gydF4y2Baepas1agydF4y2Ba(4)由序列组成的合成转录本序列gydF4y2Bahif1abgydF4y2Ba未表现出相似的mRNAgydF4y2Baepas1agydF4y2Ba基因组位点,3。补充图1:未裁剪的western blots图像。gydF4y2Ba
补充表gydF4y2Ba
此文件包含补充表1:常见上调基因gydF4y2BaFermt2gydF4y2Ba,gydF4y2BaActg1gydF4y2Ba而且gydF4y2BaActbgydF4y2Ba快速出拳细胞。gydF4y2Ba
补充表gydF4y2Ba
该文件包含补充表2:研究中使用的siRNAs。gydF4y2Ba
补充表gydF4y2Ba
此文件包含补充表3:基因分型引物。gydF4y2Ba
补充表gydF4y2Ba
此文件包含补充表4:qPCR引物。gydF4y2Ba
补充表gydF4y2Ba
该文件包含补充表5:研究中使用的grna。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
El-Brolosy, M.A, Kontarakis, Z, Rossi, A。gydF4y2Baet al。gydF4y2Ba突变体mRNA降解引发的遗传补偿。gydF4y2Ba自然gydF4y2Ba568gydF4y2Ba, 193-197(2019)。https://doi.org/10.1038/s41586-019-1064-zgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-019-1064-zgydF4y2Ba
这篇文章被引用gydF4y2Ba
房室瓣膜的形成需要多个pkd和piezo基因家族成员gydF4y2Ba
自然通讯gydF4y2Ba(2023)gydF4y2Ba
crispr - cas9介导的F0筛选识别斑马鱼视网膜色素上皮中的促再生基因gydF4y2Ba
科学报告gydF4y2Ba(2023)gydF4y2Ba
GOT1抑制TH17细胞分化,促进iTreg细胞分化gydF4y2Ba
自然gydF4y2Ba(2023)gydF4y2Ba
神经元极性的分子机制研究进展gydF4y2Ba
分子神经生物学gydF4y2Ba(2023)gydF4y2Ba
grin2B(一种asd相关基因)的破坏会在斑马鱼中产生社会缺陷gydF4y2Ba
分子自闭症gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
