摘要gydF4y2Ba
CRISPR-Cas碱基编辑器技术可以实现靶向核苷酸改变,并越来越多地用于研究和潜在的治疗应用gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba.最广泛使用的胞嘧啶碱基编辑器(CBEs)使用大鼠APOBEC1酶诱导DNA胞嘧啶脱氨,该酶被连接的Cas蛋白引导RNA复合物靶向gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.先前对CBEs特异性的研究已经确定了哺乳动物细胞中的脱靶DNA编辑gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.在这里,我们发现带有大鼠APOBEC1的CBE可导致人类细胞中广泛的转录组范围内的RNA胞嘧啶脱氨,在38-58%的表达基因中诱导成千上万次c - u编辑,频率从0.07%到100%不等。cbe诱导的RNA编辑发生在蛋白质编码和非蛋白质编码序列中,并产生错义、无义、剪接位点以及5 '和3 '非翻译区突变。我们在大鼠APOBEC1中设计了两种CBE变体,它们大大减少了人类细胞中RNA编辑的数量(减少了390多倍和3800多倍)。这些变体也显示出比野生型CBE更精确的对靶DNA编辑,对于大多数测试的指导rna,编辑效率没有显著降低。最后,我们展示了一个腺嘌呤碱基编辑器gydF4y2Ba7gydF4y2Ba也能诱导转录组范围内的RNA编辑。这些结果对碱基编辑器在研究和临床环境中的使用都有影响,说明了工程改进变体的可行性,降低了RNA编辑活性,并表明需要在碱基编辑器平台上更全面地定义和描述脱氨酶的RNA脱靶效应。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
基于crispr的基因组编辑技术及其在心血管研究中的应用gydF4y2Ba
军事医学研究gydF4y2Ba开放获取gydF4y2Ba2023年3月10日gydF4y2Ba
TadA orthologs可以同时编辑碱基编辑器的胞嘧啶和腺嘌呤gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2023年1月26日gydF4y2Ba
TadA重编程生成强大的微型碱基编辑器与高精度gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2023年1月26日gydF4y2Ba
访问选项gydF4y2Ba
访问《自然》和其他54种《自然》杂志gydF4y2Ba
获取Nature+,我们最超值的在线订阅gydF4y2Ba
每月29.99美元gydF4y2Ba
随时取消gydF4y2Ba
订阅这本杂志gydF4y2Ba
收到51个印刷问题和在线访问gydF4y2Ba
199.00美元一年gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
租或购买这篇文章gydF4y2Ba
只要这篇文章,只要你需要它gydF4y2Ba
39.95美元gydF4y2Ba
价格可能受当地税收的影响,在结账时计算gydF4y2Ba

数据可用性gydF4y2Ba
编码本研究中显示的最相关结构的质粒,包括两种SECURE BE3变体,已被沉积到Addgene (gydF4y2Bahttp://www.addgene.org/browse/article/28197497/gydF4y2Ba;添加基因id 123611-123616)。gydF4y2Ba
本研究中使用的所有RNA-seq数据都保存在基因表达综合(GEO)存储库(国家生物技术信息中心)中。这些文件可通过GEO系列登录号访问gydF4y2BaGSE121668gydF4y2Ba.所有WES和靶向扩增子测序数据已存放在SRA的生物项目号仓库中gydF4y2BaPRJNA497753gydF4y2Ba.所有其他相关数据可根据要求从通讯作者处获得。gydF4y2Ba
代码的可用性gydF4y2Ba
作者将在合理的要求下提供本工作中使用的所有以前未报告的定制计算机代码。gydF4y2Ba
参考文献gydF4y2Ba
Rees, H. A. & Liu, D. R.碱基编辑:活细胞基因组和转录组的精确化学。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba19gydF4y2Ba, 770-788(2018)。gydF4y2Ba
Seo H. & Kim J. S.对治疗碱基编辑。gydF4y2Ba地中海Nat。gydF4y2Ba.gydF4y2Ba24gydF4y2Ba, 1493-1495(2018)。gydF4y2Ba
Komor, a.c, Kim, Y. B., Packer, M. S., Zuris, J. a . & Liu, D. R.基因组DNA中目标碱基的可编程编辑,没有双链DNA切割。gydF4y2Ba自然gydF4y2Ba533gydF4y2Ba, 420-424(2016)。gydF4y2Ba
科莫尔,a.c.等。改进了碱基切除修复抑制和噬菌体Mu Gam蛋白,得到了C:G-to-T:A碱基编辑器,具有更高的效率和产品纯度。gydF4y2Ba科学。阿德gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, eaao4774(2017)。gydF4y2Ba
金,D.等人。CRISPR rna引导的可编程脱氨酶的全基因组靶向特异性。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba35gydF4y2Ba, 475-480(2017)。gydF4y2Ba
左,E.等。胞嘧啶碱基编辑器在小鼠胚胎中产生大量脱靶单核苷酸变异。gydF4y2Ba科学gydF4y2Ba364gydF4y2Ba, 289-292(2019)。gydF4y2Ba
高德利,n.m.等人。基因组DNA A·T到G·C的可编程碱基编辑,无需DNA切割。gydF4y2Ba自然gydF4y2Ba551gydF4y2Ba, 464-471(2017)。gydF4y2Ba
索尔特,J. D.,班尼特,R. P.和史密斯,H. C. APOBEC蛋白家族:结构统一,功能各异。gydF4y2Ba学生物化学的发展趋势。ScigydF4y2Ba.gydF4y2Ba41gydF4y2Ba, 578-594(2016)。gydF4y2Ba
哈里斯,R. S.彼得森-马特,S. K. & Neuberger, M. S. RNA编辑酶APOBEC1及其一些同源物可以作为DNA突变体。gydF4y2Ba摩尔。细胞gydF4y2Ba10gydF4y2Ba, 1247-1253(2002)。gydF4y2Ba
刘鹏鹏,陈淑华,王俊昌,陈丽丽,40千吨大鼠肝脏核蛋白与载脂蛋白B mRNA在RNA编辑位点周围特异性结合。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba18gydF4y2Ba, 5817-5821(1990)。gydF4y2Ba
Boström, K.等。载脂蛋白B mRNA编辑。直接测定编辑的碱基和发生在非载脂蛋白b产生细胞系。gydF4y2Ba生物。化学gydF4y2Ba.gydF4y2Ba265gydF4y2Ba, 22446-22452(1990)。gydF4y2Ba
Skuse, G. R., Cappione, A. J., Sowden, M., Metheny, L. J. & Smith, H. C.神经纤维瘤病I型信使RNA经历碱基修饰RNA编辑。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba24gydF4y2Ba, 478-485(1996)。gydF4y2Ba
Rosenberg, B. R., Hamilton, C. E., Mwangi, M. M., Dewell, S. & Papavasiliou, F. N.转录组范围测序揭示了转录本3 ' utr中大量APOBEC1 mrna编辑靶点。gydF4y2BaNat。结构。摩尔。杂志gydF4y2Ba.gydF4y2Ba18gydF4y2Ba, 230-236(2011)。gydF4y2Ba
Sowden, M., Hamm, J. K. & Smith, H. C. apobec1过表达导致系泊序列依赖的混杂RNA编辑。gydF4y2Ba生物。化学gydF4y2Ba.gydF4y2Ba271gydF4y2Ba, 3011-3017(1996)。gydF4y2Ba
Yamanaka, S., Poksay, K. S., Driscoll, D. M. & Innerarity, T. L. apobeck -1对载脂蛋白B mRNA的多胞苷超编辑需要辅助蛋白,而不需要系泊序列基序。gydF4y2Ba生物。化学gydF4y2Ba.gydF4y2Ba271gydF4y2Ba, 11506-11510(1996)。gydF4y2Ba
鲍威尔,l.m.等。一种新型的组织特异性RNA处理在肠道中产生载脂蛋白b48。gydF4y2Ba细胞gydF4y2Ba50gydF4y2Ba, 831-840(1987)。gydF4y2Ba
陈,s.h.等。载脂蛋白B-48是一种信使RNA的产物,具有器官特异性的框架内终止密码子。gydF4y2Ba科学gydF4y2Ba238gydF4y2Ba, 363-366(1987)。gydF4y2Ba
杨晓燕,曾国强,王晓燕,王晓燕,王晓燕。兔ApoB mRNA编辑蛋白的克隆与突变。锌基序对于催化活性至关重要,而编辑配合物的非催化辅助因子(s)广泛分布。gydF4y2Ba生物。化学gydF4y2Ba.gydF4y2Ba269gydF4y2Ba, 21725-21734(1994)。gydF4y2Ba
Navaratnam, N.等人。apoB mRNA编辑的进化起源:由胞苷脱氨酶催化,该酶在其活性位点获得了一种新的rna结合基序。gydF4y2Ba细胞gydF4y2Ba81gydF4y2Ba, 187-195(1995)。gydF4y2Ba
滕,B. B.等。载脂蛋白B mRNA编辑酶(APOBEC1)突变分析。RNA编辑与二聚化的结构-功能关系。gydF4y2BaJ.脂质测定gydF4y2Ba.gydF4y2Ba40gydF4y2Ba, 623-635(1999)。gydF4y2Ba
陈,Z.等。apobec1过表达引起的超突变可以消除。gydF4y2Ba核糖核酸gydF4y2Ba16gydF4y2Ba, 1040-1052(2010)。gydF4y2Ba
MacGinnitie, A. J., Anant, S. & Davidson, N. O. apobec1(哺乳动物载脂蛋白B mRNA编辑酶的催化亚基)的突变揭示了介导胞核苷脱氨酶、RNA结合和RNA编辑活性的不同结构域。gydF4y2Ba生物。化学gydF4y2Ba.gydF4y2Ba270gydF4y2Ba, 14768-14775(1995)。gydF4y2Ba
Wolf, J., Gerber, A. P. & Keller, W. tadA,一种必需的trna特异性腺苷脱氨酶gydF4y2Ba大肠杆菌gydF4y2Ba.gydF4y2BaEMBO JgydF4y2Ba.gydF4y2Ba21gydF4y2Ba, 3841-3851(2002)。gydF4y2Ba
金,J.等人。的结构和动力学特征gydF4y2Ba大肠杆菌gydF4y2BaTadA,摆动特异性tRNA脱氨酶。gydF4y2Ba生物化学gydF4y2Ba45gydF4y2Ba, 6407-6416(2006)。gydF4y2Ba
Koblan, L. W.等。通过表达优化和祖先重建改进胞苷和腺嘌呤碱基编辑器。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba36gydF4y2Ba, 843-846(2018)。gydF4y2Ba
吉布森,d.g.等。DNA分子的酶组装可达几百个碱基。gydF4y2BaNat方法。gydF4y2Ba6gydF4y2Ba, 343-345(2009)。gydF4y2Ba
莱尔德,P. W.等。简化的哺乳动物DNA分离程序。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba19gydF4y2Ba, 4293(1991)。gydF4y2Ba
Rohland, N. & Reich, D.用于多目标捕获的高成本效益,高通量DNA测序文库。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba22gydF4y2Ba, 939-946(2012)。gydF4y2Ba
麦肯纳等人。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba20.gydF4y2Ba, 1297-1303(2010)。gydF4y2Ba
DePristo, m.a.等人。利用下一代DNA测序数据进行变异发现和基因分型的框架。gydF4y2BaNat麝猫。gydF4y2Ba.gydF4y2Ba43gydF4y2Ba, 491-498(2011)。gydF4y2Ba
Dobin, A.等人。STAR:超快通用RNA-seq对准器。gydF4y2Ba生物信息学gydF4y2Ba29gydF4y2Ba, 15-21(2013)。gydF4y2Ba
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 357-359(2012)。gydF4y2Ba
Olarerin-George, a . O. & Hogenesch, J. B.通过NCBI的RNA-seq档案调查评估细胞培养中支原体污染的流行程度。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba43gydF4y2Ba, 2535-2542(2015)。gydF4y2Ba
McLaren, W.等人。综变效应预测器。gydF4y2Ba基因组医学杂志gydF4y2Ba.gydF4y2Ba17gydF4y2Ba, 122(2016)。gydF4y2Ba
克莱门特等人。CRISPResso2提供准确、快速的基因组编辑序列分析。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba37gydF4y2Ba, 224-226(2019)。gydF4y2Ba
克鲁克斯,G. E, Hon, G., Chandonia, J. M. & Brenner, S. E. WebLogo:一个序列logo生成器。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba14gydF4y2Ba, 1188-1190(2004)。gydF4y2Ba
Kelley, L. A., Mezulis, S., Yates, C. M., Wass, M. N. & Sternberg, M. J. Phyre2蛋白质建模、预测和分析门户网站。gydF4y2BaNat协议。gydF4y2Ba10gydF4y2Ba, 845-858(2015)。gydF4y2Ba
确认gydF4y2Ba
j.k.j., J.G.和R.Z.由美国国防高级研究计划局(hr0011-17,2 -0042)支持。美国国立卫生研究院也提供了支持(RM1 HG009490给J.K.J.和J.G., R35 GM118158给J.K.J.和M.J.A.)。J.K.J.还获得了Desmond and Ann Heathwood MGH研究学者奖的支持。我们感谢M. M. Kaminski, B. P. kleinstver和K. Petri的讨论;V. Pattanayak对手稿的输入;Y. E. Tak, G. Boulay, M. K. Clement, A. A. Sousa, R. T. Walton, M. L. Bobbin, M. V. Maus和A. Schmidts提供技术建议;p·k·卡贝茜拉斯和o·r·塞万提斯提供技术援助。J.K.J.谨以本文纪念c.j. Park。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
j。g。和r。z。一起做了所有的湿实验。s.p.g.、s.i.、C.A.L.和M.J.A.对数据进行了所有生物信息学和计算分析。J.G.和J.K.J.构想并设计了这项研究。j.g., M.J.A.和J.K.J.组织并监督工作。J.G.和J.K.J.撰写了手稿的初稿,所有作者都参与了最终手稿的撰写。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
J.K.J.在Beam Therapeutics、Editas Medicine、Endcadia、Pairwise Plants、Poseida Therapeutics和Transposagen biopharmacticals拥有财务权益。这些利益由马萨诸塞州总医院和合作伙伴医疗保健公司根据其利益冲突政策进行审查和管理。j。k。j。和m。j。a。还持有Excelsior Genomics的股权。j.k.j是美国基因和细胞治疗学会的董事会成员。j.g., R.Z.和J.K.J.是Partners Healthcare/Massachusetts General Hospital关于减少RNA编辑活动的工程碱基编辑器架构的专利申请的共同发明人。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意:gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
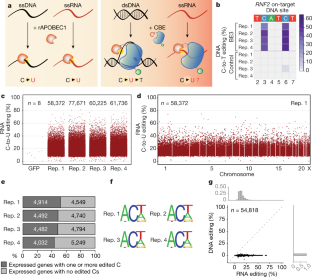
图1 BE3诱导HepG2细胞转录组范围脱靶C-to-U RNA编辑的附加数据和分析。gydF4y2Ba
一个gydF4y2Ba,的点图gydF4y2BaRNF2gydF4y2Ba靶上DNA编辑数据如图所示。gydF4y2Ba1 bgydF4y2Ba,描述了跨越间隔序列的所有胞嘧啶的编辑频率。gydF4y2BabgydF4y2Ba,热图显示RNA和DNA编辑效率与BE3和对人胞嘧啶的控制gydF4y2Ba飞机观测gydF4y2Ba.编号表示核苷酸的位置gydF4y2Ba飞机观测gydF4y2Ba成绩单;星号表示之前被APOBEC1修改过的基因。gydF4y2BacgydF4y2Ba,显示已识别的rna编辑胞嘧啶数量的直方图(gydF4y2BaygydF4y2Ba-轴)与RNA c - u编辑频率(gydF4y2BaxgydF4y2Ba-轴),如图4个重复所示。gydF4y2Ba1 cgydF4y2Ba.虚线红线,中位数;红线实线,平均值。gydF4y2BadgydF4y2Ba,图中重复2、3和4的曼哈顿数据图。gydF4y2Ba1 cgydF4y2Ba显示在转录组中鉴定的修饰胞嘧啶的分布。gydF4y2BangydF4y2Ba,鉴定出的修饰胞嘧啶总数。gydF4y2BaegydF4y2Ba,在每个RNA-seq复制中鉴定的不同预测效应和编辑胞嘧啶位置的百分比。gydF4y2BafgydF4y2Ba, BE3表达修饰的胞嘧啶抖动图gydF4y2BaRNF2gydF4y2Ba在HepG2细胞中进行的4、3、2或1个复制RNA-seq实验中,根据gRNA的存在情况进行分类(gydF4y2BangydF4y2Ba= 4个生物独立样本,如图。gydF4y2Ba1 cgydF4y2Ba).Box跨越四分位数范围(IQR)(第一到第三四分位数);水平线为中位数(第二四分位数);须延伸至±1.5 × IQR。gydF4y2BangydF4y2Ba,每一类中修饰胞嘧啶的总数。每个类别中所有修饰胞嘧啶的百分比也显示出来。gydF4y2Ba
在HEK293T细胞中,用两种不同的gRNAs表达BE3诱导转录组范围的脱靶RNA编辑。gydF4y2Ba
一个gydF4y2Ba, HEK293T细胞中BE3和nCas9-UGI-NLS(对照)靶DNA碱基编辑效率的热图(均为GFP分选)gydF4y2BaRNF2gydF4y2Ba或gydF4y2BaEMX1gydF4y2BagRNA。所显示的碱基位于目标间隔序列的编辑窗口内(编号位于底部,1是最pam -远端间隔位置)。gydF4y2BabgydF4y2Ba的点图gydF4y2BaRNF2gydF4y2Ba而且gydF4y2BaEMX1gydF4y2Ba靶向DNA编辑数据见gydF4y2Ba一个gydF4y2Ba,描述了跨越间隔序列的所有胞嘧啶的编辑频率。gydF4y2BacgydF4y2Ba,来自RNA-seq实验的抖动图显示RNA胞嘧啶被BE3表达修饰gydF4y2BaRNF2gydF4y2Ba或gydF4y2BaEMX1gydF4y2BagRNA。gydF4y2BangydF4y2Ba,每个重复中鉴定的修饰胞嘧啶总数。gydF4y2BadgydF4y2Ba,显示已识别的rna编辑胞嘧啶数量的直方图(gydF4y2BaygydF4y2Ba-轴)与RNA c - u编辑频率(gydF4y2BaxgydF4y2Ba-轴)的实验gydF4y2BacgydF4y2Ba.虚线红线,中位数;红线实线,平均值。gydF4y2BaegydF4y2Ba中显示的曼哈顿数据图gydF4y2BacgydF4y2Ba描述修饰胞嘧啶在转录组中的分布。gydF4y2BangydF4y2Ba,鉴定出的修饰胞嘧啶总数。gydF4y2Ba
附加数据分析显示HEK293T细胞中含有BE3和两种不同gRNAs的转录组范围脱靶RNA编辑。gydF4y2Ba
一个gydF4y2Ba,每个RNA-seq复制中不同预测效应和编辑胞嘧啶位置的百分比。gydF4y2Ba2摄氏度gydF4y2Ba.gydF4y2BabgydF4y2Ba、百分比(gydF4y2BaxgydF4y2Ba-轴)和每个RNA-seq复制中表达基因的数量(用竖条表示),这些数据来自扩展数据图中的数据。gydF4y2Ba2摄氏度gydF4y2Ba显示至少有一个编辑过的胞嘧啶。gydF4y2BacgydF4y2Ba, BE3表达修饰的胞嘧啶抖动图gydF4y2BaRNF2gydF4y2Ba或gydF4y2BaEMX1gydF4y2Ba在HEK293T细胞中进行的3,2或1个复制RNA-seq实验中,根据它们的存在进行分类的gRNA (gydF4y2BangydF4y2Ba= 3个生物独立样本,如扩展数据图所示。gydF4y2Ba2摄氏度gydF4y2Ba).盒子、胡须和gydF4y2BangydF4y2Ba如扩展数据图所定义。gydF4y2Ba1 fgydF4y2Ba.还显示了在每个类别中识别的所有修饰胞嘧啶的百分比。gydF4y2BadgydF4y2Ba,从每个RNA-seq复制中识别的编辑胞嘧啶中获得的序列标识。使用cDNA生成的RNA-seq数据进行分析;每一个T都应该被认为是RNA中的U。gydF4y2BaegydF4y2Ba,维恩图显示的胞嘧啶的数量与编辑gydF4y2BaRNF2gydF4y2Ba而且gydF4y2BaEMX1gydF4y2BagRNAs。对于每个gRNA,胞核嘧啶的数量代表三个重复中识别的胞核嘧啶的并集。gydF4y2Ba
图4在HEK293T细胞中,BE3表达的增加导致转录组范围内RNA胞嘧啶编辑的数量和频率增加。gydF4y2Ba
一个gydF4y2Ba, HEK293T细胞中BE3和nCas9-UGI-NLS(对照)靶DNA碱基编辑效率热图(前5% GFP排序)gydF4y2BaRNF2gydF4y2Ba或gydF4y2BaEMX1gydF4y2BagRNA。所显示的碱基位于目标间隔序列的编辑窗口内(编号位于底部,1是最pam -远端间隔位置)。gydF4y2BabgydF4y2Ba中所示的数据点图gydF4y2Ba一个gydF4y2Ba,描述了跨越间隔序列的所有胞嘧啶的编辑频率。gydF4y2BacgydF4y2Ba,来自重复RNA-seq实验的抖动图显示RNA胞嘧啶被BE3表达修饰gydF4y2BaRNF2gydF4y2Ba,gydF4y2BaEMX1gydF4y2Ba或非靶向gRNA。gydF4y2BangydF4y2Ba,每个重复中鉴定的修饰胞嘧啶总数。gydF4y2BadgydF4y2Ba,显示经RNA编辑的胞嘧啶数量的直方图(gydF4y2BaygydF4y2Ba-轴)与RNA c - u编辑频率(gydF4y2BaxgydF4y2Ba-轴)的实验gydF4y2BacgydF4y2Ba.虚线红线,中位数;红线实线,平均值。gydF4y2BaegydF4y2Ba,曼哈顿的数据图gydF4y2BaRNF2gydF4y2Ba,gydF4y2BaEMX1gydF4y2Ba的非靶向gRNAsgydF4y2BacgydF4y2Ba显示经过修饰的胞嘧啶在转录组中的分布。gydF4y2BangydF4y2Ba,鉴定出的修饰胞嘧啶总数。gydF4y2Ba
额外的数据和分析表明,BE3表达的增加导致HEK293T细胞转录组范围内RNA胞嘧啶编辑的数量和频率增加。gydF4y2Ba
一个gydF4y2Ba,在扩展数据图中,在每个RNA-seq复制中识别的不同预测效应和编辑胞嘧啶位置的百分比。gydF4y2Ba4摄氏度gydF4y2Ba.gydF4y2BabgydF4y2Ba、百分比(gydF4y2BaxgydF4y2Ba-轴)和每个RNA-seq复制中至少有一个编辑胞嘧啶的表达基因的数量(在条内显示)。gydF4y2BacgydF4y2Ba,从每个RNA-seq重复实验中识别的编辑胞嘧啶中获得的序列标识,来自扩展数据图。gydF4y2Ba4摄氏度gydF4y2Ba为gydF4y2BaRNF2gydF4y2Ba,gydF4y2BaEMX1gydF4y2Ba和非靶向grna。使用cDNA生成的RNA-seq数据进行分析;每一个T都应该被认为是RNA中的U。gydF4y2BadgydF4y2Ba,维恩图显示的编辑胞嘧啶的数量与鉴定gydF4y2BaRNF2gydF4y2Ba,gydF4y2BaEMX1gydF4y2Ba和非靶向grna。对于每个gRNA,圆圈包含两个重复中识别的胞嘧啶的并集(数据来自扩展数据图所示的实验)。gydF4y2Ba4摄氏度gydF4y2Ba).gydF4y2BaegydF4y2Ba,维恩图显示了所有可能的成对比较的编辑胞嘧啶在重复实验中确定用gydF4y2BaRNF2gydF4y2Ba,gydF4y2BaEMX1gydF4y2Ba和非靶向grna(数据来源于扩展数据图所示的实验)。gydF4y2Ba4摄氏度gydF4y2Ba).gydF4y2BafgydF4y2Ba, RNA编辑频率相关的散点图(gydF4y2BaxgydF4y2Ba-轴)之前显示的154,264个胞嘧啶被DNA编辑频率的RNA-seq编辑(gydF4y2BaygydF4y2Ba-轴),由WES测定,取自相同实验的DNA (gydF4y2BangydF4y2Ba= 3个生物独立样本,合并数据)。叠加直方图(上和右)描述了显示RNA上不同编辑速率的胞嘧啶的百分比(上)gydF4y2BaxgydF4y2Ba-轴)或DNA(对gydF4y2BaygydF4y2Ba设在)。gydF4y2Ba
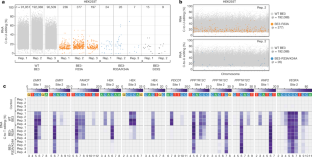
附加数据显示,SECURE BE3变体诱导的RNA编辑数量大幅减少,但在HEK293T细胞中具有相当和更精确的DNA编辑活性。gydF4y2Ba
一个gydF4y2Ba,包含各种rAPOBEC1突变并在HEK293T细胞(GFP信号最高5%的分选细胞)中高水平表达的6个BE3变体转录组范围内RNA编辑活性的初步筛选。单重复RNA-seq实验的抖动图显示RNA胞嘧啶被野生型BE3、BE3- e63q (rAPOBEC1催化位点突变体)、BE3- p29f、BE3- p29t、BE3- l182a、BE3- r33a、BE3-K34A和BE3- r33a /K34A变体的表达修饰。gydF4y2BangydF4y2Ba,每个样品中鉴定出的修饰胞嘧啶总数。gydF4y2BabgydF4y2Ba, HEK293T细胞中nCas9-UGI-NLS(对照)、野生型BE3、BE3- r33a和BE3- r33a /K34A靶DNA碱基编辑效率热图gydF4y2BaRNF2gydF4y2BagRNA(图中实验细胞)gydF4y2Ba2gydF4y2Ba).目标间隔序列的编辑窗口内的碱基按照前面描述的方式编号。请注意C12的包含,在这些示例中,野生型BE3不能有效地编辑C12,但SECURE BE3变体不能编辑C12,即使具有更高的表达。gydF4y2BacgydF4y2Ba中HEK293T对靶数据的点图gydF4y2BabgydF4y2Ba,扩展到跨越间隔序列的所有胞嘧啶。gydF4y2Ba
扩展数据图7 BE3和SECURE BE3变体的靶上DNA和脱靶RNA活性的附加数据和分析。gydF4y2Ba
一个gydF4y2Ba,在HEK293T细胞的12个基因组位点上,点阵图显示了nCas9-UGI-NLS(对照)、野生型BE3、BE3- r33a和BE3- r33a /K34A的靶向DNA编辑效率。这些数据与图中所示数据相同。gydF4y2Ba2摄氏度gydF4y2Ba,扩展到跨越间隔序列的所有胞嘧啶。gydF4y2BabgydF4y2Ba来自HepG2细胞RNA-seq实验的抖动图显示RNA胞嘧啶被野生型BE3、BE3- r33a和BE3- r33a /K34A修饰。野生型BE3的数据来自图中所示的实验。gydF4y2Ba1 cgydF4y2Ba(复制2 - 4)。gydF4y2BangydF4y2Ba,鉴定出的修饰胞嘧啶总数。gydF4y2BacgydF4y2Ba显示BE3-R33A或BE3-R33A/K34A表达诱导的修饰胞嘧啶分布的数据图gydF4y2BabgydF4y2Ba覆盖在野生型BE3表达诱导的修饰胞嘧啶上(野生型BE3数据在顶部和底部图中相同)。gydF4y2BangydF4y2Ba,鉴定出的修饰胞嘧啶总数。gydF4y2BadgydF4y2Ba, nCas9-UGI-NLS(对照)、野生型BE3、BE3- r33a和BE3- r33a /K34A在HepG2细胞中靶DNA碱基编辑效率的热图gydF4y2BaRNF2gydF4y2BagRNA(来自相同实验的细胞,见gydF4y2BabgydF4y2Ba).野生型BE3和nCas9-UGI-NLS的重复1、2和3显示了与野生型BE3和nCas9-UGI-NLS的重复2、3和4相同的数据。gydF4y2Ba1 bgydF4y2Ba.目标间隔序列的编辑窗口内的碱基按照前面描述的方式编号。再次注意位置C12的包含。gydF4y2BaegydF4y2Ba, HepG2对靶数据的点图,如图所示gydF4y2BadgydF4y2Ba,扩展到跨越间隔序列的所有胞嘧啶。gydF4y2BafgydF4y2Ba,野生型BE3、BE3- r33a和BE3- r33a /K34A基于实验数据的编辑窗口(彩色框)示意图。gydF4y2Ba2摄氏度gydF4y2Ba和扩展数据图。gydF4y2Ba7一个gydF4y2Ba.颜色较深和较半透明的框分别表示通常显示较高和较低的C-to-T编辑效率的位置。BE3-R33A/K34A对5t的严密性也有所增加。突出显示了DNA骨架中的PAM (NGG)和缺口位点。图纸经参考表1的许可进行改编。gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
图8 HEK293T细胞中ABEmax表达诱导的转录组范围脱靶A-to-I RNA编辑的其他数据和分析。gydF4y2Ba
一个gydF4y2Ba, HEK位点2上靶DNA编辑数据点图如图所示。gydF4y2Ba3gydF4y2Ba,描述了跨越间隔序列的所有腺嘌呤的编辑频率。gydF4y2BabgydF4y2Ba,显示已识别的rna编辑腺嘌呤数量的直方图(gydF4y2BaygydF4y2Ba-轴)与RNA a - i编辑频率(gydF4y2BaxgydF4y2Ba-轴),如图3所示。gydF4y2Ba3 bgydF4y2Ba.虚线红线,中位数;红线实线,平均值。gydF4y2BacgydF4y2Ba,图中重复1和2的曼哈顿数据图。gydF4y2Ba3 bgydF4y2Ba显示在转录组中鉴定的修饰腺嘌呤的分布。gydF4y2BangydF4y2Ba,鉴定出的修饰腺嘌呤总数。gydF4y2BadgydF4y2Ba,图中所示的每个RNA-seq复制中编辑腺嘌呤的不同预测效应和位置的百分比。gydF4y2Ba3 bgydF4y2Ba.gydF4y2BaegydF4y2Ba、百分比(gydF4y2BaxgydF4y2Ba-轴)和每个RNA-seq复制中表达基因的数量(条内)显示至少一个编辑过的腺嘌呤。gydF4y2BafgydF4y2Ba,如图3、2或1个复制RNA-seq实验所示,ABEmax表达修饰的腺嘌呤抖动图,HEK位点2 gRNA根据它们的存在进行分类。gydF4y2Ba3 bgydF4y2Ba(gydF4y2BangydF4y2Ba= 3个生物独立的样本)。盒和须的定义见扩展数据图。gydF4y2Ba1 fgydF4y2Ba.gydF4y2BangydF4y2Ba,每一类中修饰腺嘌呤的总数。还显示了在每个类别中发现的所有修饰腺嘌呤的百分比。gydF4y2BaggydF4y2Ba, RNA编辑频率相关的散点图(gydF4y2BaxgydF4y2Ba-轴)的52,462个腺嘌呤,先前显示RNA编辑与DNA编辑频率(gydF4y2BaygydF4y2Ba-轴)由WES测定(gydF4y2BangydF4y2Ba= 3个生物独立样本,合并数据)。叠加直方图(上和右)描述了RNA上编辑腺嘌呤的百分比(上)gydF4y2BaxgydF4y2Ba-轴)或DNA(对gydF4y2BaygydF4y2Ba设在)。gydF4y2Ba
图9 BE3和SECURE BE3变体对细胞活力、rAPOBEC1结构模型和脱靶RNA编辑位点扩展序列标识的影响。gydF4y2Ba
一个gydF4y2Ba,比较转染nCas9-UGI-NLS、野生型(WT) BE3、BE3- r33a、BE3- r33a /K34A或BE3- e63q质粒的HEK293T细胞活力测定(gydF4y2BangydF4y2Ba=每个条件下3个生物独立样本)。每个点代表一个生物复制(并且是三个技术复制的平均值)。所有数据点归一化为nCas9-UGI-NLS对照组的平均发光(设置为100%,灰色虚线),用于每次生物重复实验。在电镀后的第1、2、3和4天(对所有gfp阳性细胞进行分选后)进行测定。数据显示为平均值±s.e.m. RLU,相对光单位;n.s,与匹配的nCas9-UGI-NLS对照相比,无显著降低;*gydF4y2BaPgydF4y2Ba< 0.05, ***gydF4y2BaPgydF4y2Ba< 0.001,与匹配的nCas9-UGI-NLS对照相比显著降低。统计学意义的确定,如gydF4y2Ba补充的方法gydF4y2Ba.gydF4y2BabgydF4y2BarAPOBEC1的结构模型,包括催化残基的位置以及在SECURE变体中被改变的R33和K34位置。预测的rAPOBEC1结构是由蛋白质同源/类比识别引擎v 2.0 (Phyre2)生成的。gydF4y2Ba37gydF4y2Ba并在PyMOL (v 1.8.2.1)中可视化。在SECURE变体中突变的R33和K34残基分别以橙色和蓝色显示。催化位点残基(H61, E63, C93和C96)先前已被描述过gydF4y2Ba19gydF4y2Ba用绿色表示。gydF4y2BacgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba, BE3-和abemax诱导的RNA编辑位点的扩展序列标识。显示了由BE3 (ACW)或ABEmax (UA)在RNA中编辑的基序上游和下游的核苷酸100个碱基对派生的序列标识。标记来源于HepG2细胞中BE3表达的数据(gydF4y2BacgydF4y2Ba;无花果。gydF4y2Ba1 cgydF4y2Ba), BE3在HEK293T细胞中的表达(gydF4y2BadgydF4y2Ba;所有gfp分选细胞;扩展数据图。gydF4y2Ba2摄氏度gydF4y2Ba), HEK293T细胞中BE3表达升高(gydF4y2BaegydF4y2Ba;前5% gfp分选细胞;扩展数据图。gydF4y2Ba4摄氏度gydF4y2Ba)和HEK293T实验中ABEmax的表达(gydF4y2BafgydF4y2Ba;前5% gfp分选细胞;无花果。gydF4y2Ba3 bgydF4y2Ba).使用cDNA生成的RNA-seq数据进行分析;每一个T都应该被认为是RNA中的U。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
该文件包含补充方法、补充讨论、补充参考文献和补充说明,其中包括不同实验条件下的FACS原始数据和门控示例。gydF4y2Ba
补充表gydF4y2Ba
该文件包含补充表1-36和补充表指南。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Grünewald,周,R.,加西亚,S.P.gydF4y2Baet al。gydF4y2Ba由crispr引导的DNA碱基编辑器诱导的转录组范围脱靶RNA编辑。gydF4y2Ba自然gydF4y2Ba569gydF4y2Ba, 433-437(2019)。https://doi.org/10.1038/s41586-019-1161-zgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-019-1161-zgydF4y2Ba
这篇文章被引用gydF4y2Ba
利用ITER平台系统优化小麦和玉米Cas12a碱基编辑器gydF4y2Ba
基因组生物学gydF4y2Ba(2023)gydF4y2Ba
基于crispr的基因组编辑技术及其在心血管研究中的应用gydF4y2Ba
军事医学研究gydF4y2Ba(2023)gydF4y2Ba
TadA orthologs可以同时编辑碱基编辑器的胞嘧啶和腺嘌呤gydF4y2Ba
自然通讯gydF4y2Ba(2023)gydF4y2Ba
评估和提高CRISPR-Cas工具的安全性:从DNA到RNA编辑gydF4y2Ba
自然通讯gydF4y2Ba(2023)gydF4y2Ba
酶辅助腺苷脱氨法检测n6 -甲基腺苷的转录组全谱分析和定量gydF4y2Ba
自然生物技术gydF4y2Ba(2023)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
