摘要gydF4y2Ba
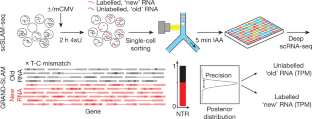
单细胞RNA测序(scRNA-seq)强调了细胞间异质性在健康和疾病表型变异中的重要作用gydF4y2Ba1gydF4y2Ba.然而,目前的scRNA-seq方法仅提供基因表达的快照,并传达很少关于转录的真实时间动态和随机性质的信息。scRNA-seq分析的另一个关键限制是每个单个细胞的RNA谱只能分析一次。在这里,我们介绍了单细胞巯基(SH)链接的RNA烷基化代谢标记测序(scSLAM-seq),它集成了代谢RNA标记gydF4y2Ba2gydF4y2Ba,生化核苷转化gydF4y2Ba3.gydF4y2Ba和scRNA-seq,通过区分每个细胞中数千个基因的新RNA和旧RNA来直接记录转录活性。我们使用scSLAM-seq来研究单个小鼠成纤维细胞中溶解性巨细胞病毒感染的发生。从旧RNA推导出的细胞周期状态和感染剂量使基于新RNA的剂量反应分析成为可能。因此,scSLAM-seq既可以可视化,也可以解释单细胞水平上转录活性的差异。此外,它描述了宿主基因表达中的“开关”开关和转录爆发动力学,具有广泛的基因特异性差异,与启动子固有特征相关(TBP-TATA-box相互作用和DNA甲基化)。因此,基因特异性而非细胞特异性的特征解释了个体细胞之间转录组的异质性和对扰动的转录反应。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
利用重编程tracrnas记录单个细菌细胞中的RNAgydF4y2Ba
自然生物技术gydF4y2Ba开放获取gydF4y2Ba2023年1月5日gydF4y2Ba
哺乳动物mRNA降解率的遗传和生化决定因素gydF4y2Ba
基因组生物学gydF4y2Ba开放获取gydF4y2Ba2022年11月23日gydF4y2Ba
单细胞RNA-seq方法询问病毒-宿主相互作用gydF4y2Ba
免疫病理学研讨会gydF4y2Ba开放获取gydF4y2Ba2022年11月21日gydF4y2Ba
访问选项gydF4y2Ba
访问《自然》和其他54种《自然》杂志gydF4y2Ba
获取Nature+,我们最超值的在线订阅gydF4y2Ba
每月29.99美元gydF4y2Ba
随时取消gydF4y2Ba
订阅这本杂志gydF4y2Ba
收到51个印刷问题和在线访问gydF4y2Ba
199.00美元一年gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
租或购买这篇文章gydF4y2Ba
只要这篇文章,只要你需要它gydF4y2Ba
39.95美元gydF4y2Ba
价格可能受当地税收的影响,在结账时计算gydF4y2Ba
数据可用性gydF4y2Ba
测序数据和基因表可从基因表达Omnibus (GEO)获得,并有登录号gydF4y2BaGSE115612gydF4y2Ba.脚本文件可以在zenodo (doi: 10.5281/zenodo.1299119)上获得。GRAND-SLAM可用于非商业用途gydF4y2Bahttp://software.erhard-lab.degydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
Wagner, A., Regev, A. & Yosef, N.用单细胞基因组学揭示细胞身份的载体。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba34gydF4y2Ba, 1145-1160(2016)。gydF4y2Ba
Dölken, L.等。高分辨率基因表达谱同时动力学参数分析的RNA合成和衰变。gydF4y2Ba核糖核酸gydF4y2Ba14gydF4y2Ba, 1959-1972(2008)。gydF4y2Ba
赫尔佐格,v.a.等人。巯基链烷基化RNA评估表达动力学。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 1198-1204(2017)。gydF4y2Ba
Jürges, C., Dölken, L. & Erhard, F.使用GRAND-SLAM解剖新转录和旧RNA。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, i218-i226(2018)。gydF4y2Ba
拉曼诺,G.等。单细胞的RNA速度。gydF4y2Ba自然gydF4y2Ba560gydF4y2Ba, 494-498(2018)。gydF4y2Ba
Terhune, s.s., Schröer, J. & Shenk, T. rna按照细胞内浓度的比例被包装成人类巨细胞病毒病毒粒子。gydF4y2Baj .性研究gydF4y2Ba.gydF4y2Ba78gydF4y2Ba, 10390-10398(2004)。gydF4y2Ba
Butler, A, Hoffman, P, Smibert, P, Papalexi, E. & Satija, R.整合不同条件、技术和物种的单细胞转录组数据。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba36gydF4y2Ba, 411-420(2018)。gydF4y2Ba
吴志刚,张勇,吴红,吴海涛,张志刚,张志刚。单细胞rna序列的两相差异表达分析。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, 3340-3348(2018)。gydF4y2Ba
沙雷克,A. K.等。单细胞转录组学揭示了免疫细胞表达和剪接的双态性。gydF4y2Ba自然gydF4y2Ba498gydF4y2Ba, 236-240(2013)。gydF4y2Ba
Marcinowski, L.等人。在溶细胞巨细胞病毒感染期间细胞和病毒基因表达的实时转录分析。gydF4y2Ba公共科学图书馆PathoggydF4y2Ba.gydF4y2Ba8gydF4y2Ba, e1002908(2012)。gydF4y2Ba
Krause, E., de Graaf, M., Fliss, P. M., Dölken, L. & Brune, W.小鼠巨细胞病毒病毒粒子相关蛋白M45在感染后介导NF-κB快速激活。gydF4y2Baj .性研究gydF4y2Ba.gydF4y2Ba88gydF4y2Ba, 9963-9975(2014)。gydF4y2Ba
范,J.等。通过途径和基因集过度分散分析来表征转录异质性。gydF4y2BaNat方法。gydF4y2Ba13gydF4y2Ba, 241-244(2016)。gydF4y2Ba
Pachkov, M., Balwierz, P. J., Arnold, P., Ozonov, E. & van Nimwegen, E. SwissRegulon,调控位点全基因组注释数据库:最新更新。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba41gydF4y2Ba, d214-d220(2013)。gydF4y2Ba
利奥,C.-W。cGAS-STING信号通路调控巨细胞病毒感染的初始先天控制。gydF4y2Baj .性研究gydF4y2Ba.gydF4y2Ba90gydF4y2Ba, 7789-7797(2016)。gydF4y2Ba
Rand, U.等。多层随机性和旁分泌信号传播形成了i型干扰素反应。gydF4y2Ba摩尔。系统。医学杂志gydF4y2Ba.gydF4y2Ba8gydF4y2Ba, 584(2012)。gydF4y2Ba
Hinata, K., Gervin, A. M., Jennifer Zhang, Y. & Khavari, P. A. NF-κ B在人皮肤上皮细胞和间充质细胞中的分化基因调控和生长效应。gydF4y2Ba致癌基因gydF4y2Ba22gydF4y2Ba, 1955-1964(2003)。gydF4y2Ba
Hodges, C., Bintu, L., Lubkowska, L., Kashlev, M. & Bustamante, C.核小体波动控制RNA聚合酶II的转录动力学。gydF4y2Ba科学gydF4y2Ba325gydF4y2Ba, 626-628(2009)。gydF4y2Ba
Tantale, K.等。单分子转录的观点揭示了RNA聚合酶的运输和多尺度的爆发。gydF4y2BaNat。CommungydF4y2Ba.gydF4y2Ba7gydF4y2Ba, 12248(2016)。gydF4y2Ba
皮切利等人。Smart-seq2用于单细胞敏感的全长转录组分析。gydF4y2BaNat方法。gydF4y2Ba10gydF4y2Ba, 1096-1098(2013)。gydF4y2Ba
Zoller, B. Nicolas, D., Molina, N. & Naef, F.哺乳动物基因沉默转录间隔的结构和噪声特征。gydF4y2Ba摩尔。系统。医学杂志gydF4y2Ba.gydF4y2Ba11gydF4y2Ba, 823(2015)。gydF4y2Ba
科赫等人。癌症中DNA甲基化的分析:重新定位。gydF4y2BaNat. Rev. clinin。肿瘤防治杂志gydF4y2Ba.gydF4y2Ba15gydF4y2Ba, 459-466(2018)。gydF4y2Ba
玛扎,我,等人。iPSC重编程因子在体细胞转分化过程中瞬时获得多能性。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba33gydF4y2Ba, 769-774(2015)。gydF4y2Ba
Reinius, B. & Sandberg, R.常染色体基因的随机单等位基因表达:随机转录和等位基因水平调控。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba16gydF4y2Ba, 653-664(2015)。gydF4y2Ba
基弗,L.,斯科菲尔德,J. A.和西蒙,m.d.扩展核苷编码工具包:揭示6-硫鸟苷的RNA种群动态。gydF4y2Baj。化学。SocgydF4y2Ba.gydF4y2Ba140gydF4y2Ba, 14567-14570(2018)。gydF4y2Ba
Brennecke, P.等。考虑单细胞RNA-seq实验中的技术噪声。gydF4y2BaNat方法。gydF4y2Ba10gydF4y2Ba, 1093-1095(2013)。gydF4y2Ba
Schwanhäusser, B.等。哺乳动物基因表达控制的全球定量研究。gydF4y2Ba自然gydF4y2Ba473gydF4y2Ba, 337-342(2011)。gydF4y2Ba
刘,Z.等。利用单细胞转录组数据重建细胞周期伪时间序列。gydF4y2BaNat。通讯gydF4y2Ba.gydF4y2Ba8gydF4y2Ba, 22(2011)。gydF4y2Ba
确认gydF4y2Ba
我们感谢J. Vogel和S. Gorski对手稿的评论和T. Walzthoeni(计算生物学研究所生物信息学核心设施,Helmholtz Zentrum München)的生物信息学支持。这项工作由欧洲研究委员会(ERC-2016-CoG 721016-疱疹和erc -2018- pocc - dl3 832409 T-GRAND-SLAM)资助,并由感染- era资助eDEVILLI (031L0005B)给L.D.。Helmholtz rna感染研究所(HIRI)通过巴伐利亚经济事务和媒体、能源和技术部的资金支持这项工作。0703/68674/5/2017和0703/89374/3/2017)。F.J.T.和M.L.感谢研究生院QBM (GSC 1006)的财政支持;F.J.T.得到了德国联邦政府资助01IS18036A和01IS18053A,德国研究基金会(DFG) 1243合作研究中心的子项目A17, Helmholtz协会(孵化器资助sparse2big, ZT-I-0007)和陈-扎克伯格倡议DAF(硅谷社区基金会的建议基金,182835)的支持。M.L.和P.A.分别感谢Joachim Herz Stiftung和IZKF的财政支持。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
概念化:f.e., a.e.s.和L.D.;计算方法学:m.l., C.S.J., F.E.;调查:例如,M.A.P.B T.K, T.H。马丁,年利,F.J.T. A.-E.S。;感染实验:M.A.P.B.、T.H.;建立scSLAM-seq: M.A.P.B, t.k., p.a., a.e.s.和L.D;写作:f.e., a.e.s.和L.D.;融资收购:a.e.s.、F.E.和L.D.;监督:F.J.T, a.e.s, F.E.和L.D.gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
已经申请了一项关于GRAND-SLAM方法的专利(EP 18 17 9371),该方法用于分析基于U-to-C转换的转录活性的相对贡献。作者声明没有其他竞争利益。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意:gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
扩展数据图1 scSLAM-seq质量控制。gydF4y2Ba
一个gydF4y2Ba,在所有四种实验条件下(未感染和cmv感染细胞;两个生物重复)与单个细胞的总读取计数。水平线表示一个阈值,低于该阈值的细胞被排除在分析之外。gydF4y2BabgydF4y2Ba在所有单个细胞中,用于宿主(细胞)、病毒、尖刺控制(外部RNA控制联盟(ERCC))和线粒体基因(Mitoch)的reads分区。gydF4y2BacgydF4y2Ba核苷酸取代率表明,与4sU幼稚细胞(no4sU)相比,4sU处理的单细胞(4sU)具有有效的转化率。这适用于源于cDNA链(正义和反义)以及配对末端测序的重叠部分(重叠)。gydF4y2BadgydF4y2Ba,如gydF4y2BacgydF4y2Ba,放大到范围(gydF4y2BaygydF4y2Ba轴)0到0.004。gydF4y2BaegydF4y2Ba,与检测基因和可靠检测基因(TPM >10)相比,每个细胞的NTR可以被高精度量化(90%可信区间(CI) < 0.2)的基因数。gydF4y2BafgydF4y2Ba,大块RNA-seq的表达水平与总RNA、新RNA和旧RNA的聚合scRNA-seq数据之间的相关性。基因是根据RNA的半衰期来着色的。皮尔逊相关系数(gydF4y2BaRgydF4y2Ba)和使用的基因数目(gydF4y2BangydF4y2Ba)。gydF4y2BaggydF4y2Ba,使用ERCC spike-ins对应用于总RNA的技术噪声进行建模,鉴定高变量基因(洋红色)(1%的错误发现率)。变异系数的平方(CVgydF4y2Ba2gydF4y2Ba)与所有通过质量控制过滤器的单元格的平均规范化读计数相对应。粉红色实线表示ERCC峰值的平均值(蓝点)。gydF4y2Ba25gydF4y2Ba.虚线表示生物变异系数为50%的基因的预期位置。gydF4y2BahgydF4y2Ba高度可变基因(包括病毒转录本(感染:gydF4y2BangydF4y2Ba= 2个重复,49个细胞;感染:gydF4y2BangydF4y2Ba= 2个重复,45个细胞)。gydF4y2Ba我gydF4y2Ba,在前两个主成分中病毒读取百分比与到未感染细胞的距离的相关性,由PCA的逻辑回归测量gydF4y2BafgydF4y2Ba(gydF4y2BangydF4y2Ba= 2个重复,49个细胞)。gydF4y2BajgydF4y2Ba,如gydF4y2Ba我gydF4y2Ba为图中的主成分分析。gydF4y2Ba2gydF4y2Ba(gydF4y2BangydF4y2Ba= 2个重复,49个细胞)。gydF4y2Ba
图2调控基因的半衰期估计和主成分分析。gydF4y2Ba
一个gydF4y2Ba, bulk SLAM-seq估计RNA半衰期的相关性(gydF4y2BangydF4y2Ba= 2个重复)。gydF4y2BabgydF4y2Ba,折叠变化(FC), loggydF4y2Ba2gydF4y2Ba(MCMV/Mock),从bulk SLAM-seq中获得的总RNA的分布分布在日志上gydF4y2Ba2gydF4y2Ba从批量测序中获得的新RNA的-转化折叠变化,根据不同的RNA半衰期(平均为gydF4y2BangydF4y2Ba= 2个重复)。gydF4y2BacgydF4y2Ba在大量实验中,对新RNA中差异表达的基因进行PCA分析(绝对对数gydF4y2Ba2gydF4y2Ba-变换折叠变化> 0.5)。上图为RNA半衰期短(小于2小时)基因的PCA。下图为RNA半衰期较长的基因(超过4小时)的主成分分析。从左到右,主成分分析使用总RNA、旧RNA或新RNA,或NTR(感染的、gydF4y2BangydF4y2Ba= 2个重复,49个细胞;未受感染的,gydF4y2BangydF4y2Ba= 2个重复,45个细胞)。ARI,调整后的兰特指数。gydF4y2BadgydF4y2Ba, PCA的相关分析gydF4y2BacgydF4y2Ba病毒式阅读的百分比。Pearson相关系数和gydF4y2BaPgydF4y2Ba由gydF4y2BatgydF4y2BaPearson相关系数的-test(见扩展数据图)。gydF4y2Ba1 j) (gydF4y2BangydF4y2Ba= 2个重复,49个细胞)。gydF4y2Ba
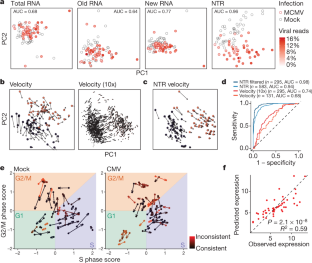
图3 RNA和NTR速度。gydF4y2Ba
一个gydF4y2Ba, PCA是根据速度值、使用速度预测未来1小时的表达值以及使用如图所示的同一组基因的scSLAM-seq数据的内含子/外显子计数比计算的。gydF4y2Ba2gydF4y2Ba(未受感染的,gydF4y2BangydF4y2Ba= 2个重复,43个细胞;感染,gydF4y2BangydF4y2Ba= 2个重复,44个细胞)。gydF4y2BabgydF4y2Ba,对同一组基因的10倍数据进行主成分分析,如图所示。gydF4y2Ba2gydF4y2Ba在成熟成绩单的基础上(外传只读;左)未感染细胞与受感染细胞的边缘分离。相比之下,根据内含子/外显子reads的比例(右),感染细胞与未感染细胞几乎完全分离(右;未受感染的,gydF4y2BangydF4y2Ba= 2个重复,793个细胞;感染,gydF4y2BangydF4y2Ba= 2个重复,353个细胞)。gydF4y2BacgydF4y2Ba,如Fig。gydF4y2Ba二维gydF4y2Ba,但速度仅根据未感染细胞的降解率计算;即不违反稳态假设,但提供类标签(违反预测盲检验)。gydF4y2BadgydF4y2Ba,将NTRs与来自下调和上调基因的10x scRNA-seq数据的速度进行比较的散点图。gydF4y2Ba
扩展数据图4 scSLAM-seq区分传入的病毒粒子相关RNA与从头转录的病毒RNA。gydF4y2Ba
热图显示旧(左)和新(右)RNA水平相对于每个cmv感染细胞的每个病毒基因(行)的最大总水平(列)。细胞是根据病毒读取在细胞中所有读取的百分比进行排序的。指出了动力学类。E,早;IE,即刻早;L,迟了;ND,没有定义。新RNA与旧RNA的比值(loggydF4y2Ba2gydF4y2Ba(新的/旧的))和每个病毒基因从池细胞的总表达被描绘。gydF4y2Ba
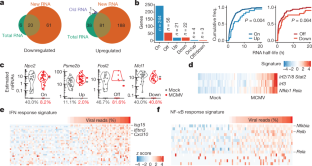
图5病毒基因表达与感染剂量、细胞周期的相关性。gydF4y2Ba
一个gydF4y2BaCMV基因的病毒源RNA(病毒粒子相关RNA)和旧RNA水平的比较。从四个独立的病毒储存瓶获得的平均水平和cmv感染细胞的平均表达水平(gydF4y2BangydF4y2Ba= 2个重复,49个细胞)。颜色表示不同动力学类的病毒基因。TL,真晚。皮尔森相关系数和gydF4y2BaPgydF4y2Ba由gydF4y2BatgydF4y2Ba给出了Pearson相关系数的检验方法。gydF4y2BabgydF4y2Ba,如gydF4y2Ba一个gydF4y2Ba但cmv感染细胞中各自基因的总表达水平归一化。皮尔森相关系数和gydF4y2BaPgydF4y2Ba值是指定的。gydF4y2BacgydF4y2Ba,比较单个细胞病毒基因表达预测程度的散点图(gydF4y2BangydF4y2Ba= 2个重复,49个细胞),以观察到的表达为感染剂量。gydF4y2BaPgydF4y2Ba由似然比检验确定的值。gydF4y2BadgydF4y2Ba, G1细胞的病毒读数分布(gydF4y2BangydF4y2Ba= 9单元格),S (gydF4y2BangydF4y2Ba= 20个细胞)或G2/M (gydF4y2BangydF4y2Ba= 20个细胞)感染初期的阶段。gydF4y2BaPgydF4y2Ba值由双面Wilcoxon检验确定。gydF4y2BaegydF4y2Ba,基于未感染的旧RNA和总RNA的细胞周期预测的细胞周期破坏程度(mock,gydF4y2BangydF4y2Ba= 2个重复,45个细胞)和cmv感染(gydF4y2BangydF4y2Ba= 2个重复,49个细胞)用于G1、S和G2/M期。单个单元格显示为圆点。gydF4y2BaPgydF4y2Ba由双面Wilcoxon检验确定的值。gydF4y2BafgydF4y2Ba、无偏路径和基因集过分散分析(PAGODA)gydF4y2Ba12gydF4y2Ba揭示了与模拟细胞和cmv感染细胞相关的基因本体(GO)术语。表示每个签名被发现的分数(总数、新或旧)。gydF4y2BaggydF4y2Ba,gydF4y2BahgydF4y2Ba, NF-κB (gydF4y2BaggydF4y2Ba)和干扰素(gydF4y2BahgydF4y2Ba)每个单元格的响应特征分数(gydF4y2BangydF4y2Ba= 2个重复,49个细胞)绘制其病毒RNA含量。线性回归拟合(直线),95%可信区间(阴影),斯皮尔曼gydF4y2BaρgydF4y2Ba价值观和gydF4y2BaPgydF4y2Ba由gydF4y2BatgydF4y2Ba-测试斯皮尔曼的gydF4y2BaρgydF4y2Ba是表示。gydF4y2Ba我gydF4y2Ba,gydF4y2BajgydF4y2Ba, NF-κB的分布范围(gydF4y2Ba我gydF4y2Ba)和干扰素(gydF4y2BaggydF4y2Ba)对G1期细胞的反应(gydF4y2BangydF4y2Ba= 9单元格),S (gydF4y2BangydF4y2Ba= 20个细胞)或G2/M (gydF4y2BangydF4y2Ba= 20个细胞)阶段。gydF4y2BaPgydF4y2Ba由双面Wilcoxon检验确定的值。所有箱形图如图所示。gydF4y2Ba4摄氏度gydF4y2Ba.圆点代表异常值。gydF4y2Ba
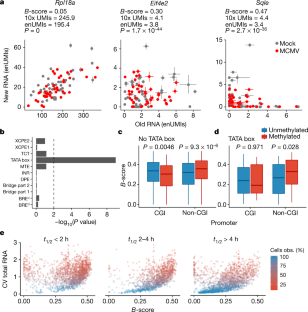
扩展数据图6gydF4y2BaBgydF4y2Ba-分数,nUMIs和转录爆发。gydF4y2Ba
一个gydF4y2Ba,相关性gydF4y2BaBgydF4y2Ba分数(gydF4y2BangydF4y2Ba= 2个重复)。皮尔森相关系数和gydF4y2BaPgydF4y2Ba由gydF4y2BatgydF4y2Ba给出了Pearson相关系数的检验方法。gydF4y2BabgydF4y2Ba,旧分子和新分子的数量(由RNA spike-ins回归分析估计)显示gydF4y2BaHif1agydF4y2Ba而且gydF4y2BaAtg12gydF4y2Ba.两者都表现出极端的NTR方差,这是由非常少的mRNA分子样本造成的。点表示未感染的最大后验估计(模拟,gydF4y2BangydF4y2Ba= 2个重复,45个细胞)和cmv感染(gydF4y2BangydF4y2Ba= 2个重复,49个细胞)细胞。误差条表示由GRAND-SLAM提供的90%可信区间。gydF4y2BacgydF4y2Ba,从ref获得的平均mRNA拷贝数。gydF4y2Ba26gydF4y2Ba与RNA spike-ins回归分析估计的平均拷贝数(gydF4y2BaPgydF4y2Ba< 2 × 10gydF4y2Ba−16gydF4y2Ba,双面gydF4y2BatgydF4y2Ba- Pearson相关系数检验;看到gydF4y2Ba补充的方法gydF4y2Ba).gydF4y2BadgydF4y2Ba在6个未标记4sU的样本中,显示了所有可检测reads的细胞中nUMIs > 0基因的比例。gydF4y2BaegydF4y2Ba,显示了在10x和scSLAM-seq数据中至少检测一次的所有基因的基因检出率分布(gydF4y2BangydF4y2Ba= 12784)。在10x或scSLAM-seq数据中,一个基因分别被称为至少有一个UMI或读取。gydF4y2BafgydF4y2Ba,在scSLAM-seq或10x实验中,分别显示了未感染和感染细胞的nUMI(左)或UMI(右)计数的分布。gydF4y2BaggydF4y2Ba,从ref中获得的平均拷贝数。gydF4y2Ba26gydF4y2Ba与10倍未感染的umi和numi的平均数量(gydF4y2BangydF4y2Ba= 2个重复,353个细胞)和scSLAM-seq (gydF4y2BangydF4y2Ba= 2个重复,45个细胞)试验。线表示捕获率的中值。gydF4y2BahgydF4y2Ba,如gydF4y2BafgydF4y2Ba,但使用枚举。gydF4y2Ba
扩展数据图7示例基因的nUMIs可视化基因组浏览器截图。gydF4y2Ba
一个gydF4y2Ba,示例基因组查看器截图的两个单细胞为gydF4y2BaAtg12gydF4y2Ba显示个体读数的基因。灰色和黑色表示单序列部分和双序列部分。基因组序列用颜色编码(A,红色;C,绿色;G,蓝色;T,橙色)。不匹配是在读取相同颜色代码时表示的。在双序列部分,顶部和底部三角形分别表示第一次和第二次读取的对应不匹配。在84单元格中,有两个特征的4sU错配,在所有读取中都观察到。在单元格29中,有6个不匹配,这与在单元格84中观察到的不匹配不同。 In both cells, only a single new transcript with stochastic 4sU incorporation thus gave rise to the respective reads.bgydF4y2Ba,示例基因组浏览器截图为gydF4y2BaSqlegydF4y2Ba基因。在这里,至少有4种(nUMI = 4) mrna产生了观察到的reads。然而,由于并非所有读取都重叠,这可能是对克隆转录本实际数量的低估。gydF4y2Ba
扩展数据图8gydF4y2BaBgydF4y2Ba-分数反映单细胞的随机转录活性。gydF4y2Ba
一个gydF4y2Ba,比较gydF4y2BaBgydF4y2Ba分数(从gydF4y2BangydF4y2Ba= 2个重复,45个细胞),按RNA半衰期分层的体RNA表达水平。的gydF4y2BargydF4y2Ba2gydF4y2Ba普通线性回归值如图所示(用线表示)。特别是对于短时间转录本的基因,相关性并不高gydF4y2BaBgydF4y2Ba-分数不是由于低效的RNA捕获。gydF4y2BabgydF4y2Ba,细胞对中最易变的前10%基因的NTR值的Pearson相关系数(gydF4y2BangydF4y2Ba= 2个重复,45个细胞),要么处于相同的细胞周期阶段(紫色),要么处于细胞周期的不同阶段(绿色)。gydF4y2BacgydF4y2Ba,细胞(gydF4y2BangydF4y2Ba= 2个重复,45个细胞)使用reCAT根据细胞周期(恢复周期随时间)进行排序gydF4y2Ba27gydF4y2Ba(gydF4y2BaxgydF4y2Ba轴)。图中显示了顺序中每个单元格与下一个单元格的相关性,或顺序中最远的单元格(相反)。Pearson的相关系数是计算前10%最易变基因的NTR值。gydF4y2BadgydF4y2Ba,显示细胞周期S期和G2/M期标记基因NTR值的热图。灰色区域表示未检测到的基因。细胞(列,gydF4y2BangydF4y2Ba= 2个重复,45个细胞)根据日志概率(on-off vs S-G2 /M)进行排序。日志的概率和关联gydF4y2BaPgydF4y2Ba值(双面费雪精确检验,由Benjamini-Hochberg校正)。根据NTR值与对数比值顺序的相关性对基因(行)进行排序。gydF4y2BaegydF4y2Ba,前10%变异最大基因的Spearman相关系数分布(gydF4y2BaBgydF4y2Ba-按细胞周期对数赔率顺序评分;看到gydF4y2BadgydF4y2Ba).为了便于比较,将细胞随机排列100次,并表示相关系数的分布情况。gydF4y2BafgydF4y2Ba, RNA半衰期与细胞上的分数的散点图(gydF4y2BangydF4y2Ba= 2个重复,45个细胞)。所有箱形图如图所示。gydF4y2Ba4摄氏度gydF4y2Ba.点表示异常值。gydF4y2Ba
扩展数据图9二分式基因启动子区序列标志过多。gydF4y2Ba
结合位点较低的基因中显著富集的转录因子序列标识gydF4y2Ba真沸点gydF4y2Ba)或高(gydF4y2BaPatz1gydF4y2Ba,gydF4y2BaPmlgydF4y2Ba,gydF4y2BaChd1gydF4y2Ba,gydF4y2BaSin3agydF4y2Ba而且gydF4y2BaZbtb14gydF4y2Ba)gydF4y2BaBgydF4y2Ba-从SwissRegulon数据库获得的分数gydF4y2Ba13gydF4y2Ba.gydF4y2Ba
扩展数据图10基因子集重复的爆发动力学分析。gydF4y2Ba
一个gydF4y2Ba,分析如图所示。gydF4y2Ba4 a egydF4y2Ba根据异质性检验(基于nUMIs和enUMIs),仅对1718个显著调控基因重复gydF4y2Ba补充的方法gydF4y2Ba).gydF4y2BabgydF4y2Ba,分析如图所示。gydF4y2Ba4 a egydF4y2Ba重复前50%的基因(gydF4y2BangydF4y2Ba= 2770)的表达基因。所有箱形图如图所示。gydF4y2Ba4摄氏度gydF4y2Ba.gydF4y2BaPgydF4y2Ba数值采用双侧Wilcoxon检验。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
该文件包含补充方法、补充参考和补充表格指南。补充方法包括关于病毒原液生产和滴定、病毒原液衍生RNA测序、病毒感染和代谢RNA标记、文库制备和测序(包括使用10x Genomics的液滴测序)以及数据处理和分析的详细信息。gydF4y2Ba
补充表gydF4y2Ba
该文件包含补充表1-8 -参见补充信息文件完整的表说明。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
艾哈德,F.,巴普蒂斯塔,m.a.p.,克莱默,T.。gydF4y2Baet al。gydF4y2BascSLAM-seq揭示了单细胞转录动力学的核心特征。gydF4y2Ba自然gydF4y2Ba571gydF4y2Ba, 419-423(2019)。https://doi.org/10.1038/s41586-019-1369-ygydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-019-1369-ygydF4y2Ba
这篇文章被引用gydF4y2Ba
人类巨细胞病毒感染的单细胞转录组学分子特征gydF4y2Ba
微生物学性质gydF4y2Ba(2023)gydF4y2Ba
利用重编程tracrnas记录单个细菌细胞中的RNAgydF4y2Ba
自然生物技术gydF4y2Ba(2023)gydF4y2Ba
单细胞RNA-seq方法询问病毒-宿主相互作用gydF4y2Ba
免疫病理学研讨会gydF4y2Ba(2023)gydF4y2Ba
哺乳动物mRNA降解率的遗传和生化决定因素gydF4y2Ba
基因组生物学gydF4y2Ba(2022)gydF4y2Ba
使用代谢RNA标记的时间分辨单细胞RNA序列gydF4y2Ba
自然评论方法引物gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
