摘要gydF4y2Ba
许多基因组加工反应,包括转录、复制和修复,都会产生DNA旋转。直接测量DNA旋转的方法,如转子头跟踪gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba、角光捕获gydF4y2Ba4gydF4y2Ba磁性镊子gydF4y2Ba5gydF4y2Ba,已经帮助揭示了一系列基因组加工酶的作用机制,包括RNA聚合酶(RNAP)。gydF4y2Ba6gydF4y2Ba,促旋酶gydF4y2Ba2gydF4y2Ba一种病毒DNA包装马达gydF4y2Ba7gydF4y2Ba和DNA重组酶gydF4y2Ba8gydF4y2Ba。尽管旋转测量有可能改变我们对基因组加工反应的理解,但测量DNA旋转仍然是一项艰巨的任务。现有方法的时间分辨率不足以跟踪生理条件下许多酶引起的旋转,而且测量通量通常较低。在这里,我们介绍了基于折纸转子的成像和跟踪(ORBIT),一种使用荧光标记的DNA折纸转子在单分子水平上以毫秒级的时间分辨率跟踪DNA旋转的方法。我们使用ORBIT来跟踪由RecBCD复合体(一种参与DNA修复的解旋酶)解绕引起的DNA旋转gydF4y2Ba9gydF4y2Ba,以及RNAP的转录。我们描述了在recbcd诱导的DNA解绕过程中发生的一系列事件,包括起始、过程易位、暂停和回溯,并揭示了一种起始机制,该机制涉及可逆的atp非依赖性DNA解绕和RecB马达的参与。在RNAP转录过程中,我们直接观察到与单碱基对解绕相对应的旋转步骤。我们设想ORBIT将使研究蛋白质和DNA之间广泛的相互作用成为可能。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用这篇文章的开放获取文章。gydF4y2Ba
数字纳米反应器控制绝对化学计量和时空行为的DNA受体内脂质双层gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2023年3月20日gydF4y2Ba
DNA折纸旋转棘轮马达gydF4y2Ba
自然gydF4y2Ba开放获取gydF4y2Ba2022年7月20日gydF4y2Ba
MINFLUX的替代方案,可在共聚焦显微镜中实现纳米分辨率gydF4y2Ba
光:科学与应用gydF4y2Ba开放获取gydF4y2Ba2022年6月30日gydF4y2Ba
访问选项gydF4y2Ba
访问Nature和其他54种Nature组合期刊gydF4y2Ba
获取自然+,我们最具价值的在线订阅gydF4y2Ba
每月29.99美元gydF4y2Ba
随时取消gydF4y2Ba
订阅本刊gydF4y2Ba
接收51个印刷问题和在线访问gydF4y2Ba
每年$199.00gydF4y2Ba
每期只要3.90美元gydF4y2Ba
租用或购买这篇文章gydF4y2Ba
只要你需要,就可以得到这篇文章gydF4y2Ba
39.95美元gydF4y2Ba
价格可能会在结账时计算当地税收gydF4y2Ba
数据可用性gydF4y2Ba
支持本研究结果的数据可根据通讯作者的合理要求提供。gydF4y2Ba
代码的可用性gydF4y2Ba
使用定制Python和Igor Pro代码分析单分子数据,可在gydF4y2Bahttps://github.com/altheimerb/python-sma/gydF4y2Ba。gydF4y2Ba
参考文献gydF4y2Ba
布莱恩特,Z.等。DNA上扭矩测量的结构转变和弹性。gydF4y2Ba自然gydF4y2Ba424gydF4y2Ba; 338-341(2003)。gydF4y2Ba
戈尔等人。利用转子磁珠跟踪对DNA回转酶进行力学化学分析。gydF4y2Ba自然gydF4y2Ba439gydF4y2Ba, 100-104(2006)。gydF4y2Ba
Lebel, P, Basu, A, Oberstrass, F. C, Tretter, E. m和Bryant, Z. Gold转子头跟踪用于DNA扭曲,扭矩和延伸的高速测量。gydF4y2BaNat方法。gydF4y2Ba11gydF4y2Ba, 456-462(2014)。gydF4y2Ba
Deufel, C., Forth, S., Simmons, C. R., Dejgosha, S.和Wang, M. D.用于角捕获的纳米石英圆柱体:DNA超卷曲扭矩检测。gydF4y2BaNat方法。gydF4y2Ba4gydF4y2Ba, 223-225(2007)。gydF4y2Ba
Lipfert, J., van Oene, M. M., Lee, M., Pedaci, F. & Dekker, N. H.用于生物系统旋转运动研究的扭矩光谱。gydF4y2Ba化学。牧师gydF4y2Ba。gydF4y2Ba115gydF4y2Ba, 1449-1474(2015)。gydF4y2Ba
原田,Y.等。直接观察转录过程中的DNA旋转gydF4y2Ba大肠杆菌gydF4y2BaRNA聚合酶。gydF4y2Ba自然gydF4y2Ba409gydF4y2Ba, 113-115(2001)。gydF4y2Ba
Liu, S.等。病毒包装马达改变其DNA旋转和步长,以在衣壳填充时保持亚基协调。gydF4y2Ba细胞gydF4y2Ba157gydF4y2Ba, 702-713(2014)。gydF4y2Ba
Lipfert, J., Wiggin, M., Kerssemakers, J. W. J., Pedaci, F. & Dekker, N. H.自由轨道磁镊子直接监测核酸扭曲的变化。gydF4y2BaNat。CommungydF4y2Ba。gydF4y2Ba2gydF4y2Ba, 439(2011)。gydF4y2Ba
rebcd酶与双链DNA断裂的修复。gydF4y2BaMicrobiol。摩尔。杂志。牧师gydF4y2Ba。gydF4y2Ba72gydF4y2Ba, 642-671(2008)。gydF4y2Ba
折叠DNA以创造纳米级的形状和图案。gydF4y2Ba自然gydF4y2Ba440gydF4y2Ba, 297-302(2006)。gydF4y2Ba
道格拉斯,s.m.等。DNA自组装成纳米级三维形状。gydF4y2Ba自然gydF4y2Ba459gydF4y2Ba, 414-418(2009)。gydF4y2Ba
Nomidis, s.k., Kriegel, F., Vanderlinden, W., Lipfert, J. & Carlon, E.双链DNA的扭-弯耦合和扭转响应。gydF4y2Ba理论物理。(1gydF4y2Ba。gydF4y2Ba118gydF4y2Ba中文信息学报,217801(2017)。gydF4y2Ba
罗曼,L. J.和科瓦尔奇科夫斯基,S. C.解旋酶活性的表征gydF4y2Ba大肠杆菌gydF4y2BaRecBCD酶使用一种新的解旋酶测定。gydF4y2Ba生物化学gydF4y2Ba28gydF4y2Ba, 2863-2873(1989)。gydF4y2Ba
Bianco, p.r.等人。单个RecBCD酶分子的遗传易位和DNA解绕。gydF4y2Ba自然gydF4y2Ba409gydF4y2Ba, 374-378(2001)。gydF4y2Ba
李晓东,李晓东,李晓东,等。DNA序列识别和DNA易位的研究进展。gydF4y2Ba自然gydF4y2Ba409gydF4y2Ba, 370-374(2001)。gydF4y2Ba
珀金斯,t.t.,李,h - w。,Dalal, R. V., Gelles, J. & Block, S. M. Forward and reverse motion of single RecBCD molecules on DNA.Biophys。JgydF4y2Ba。gydF4y2Ba86gydF4y2Ba, 1640-1648(2004)。gydF4y2Ba
刘,B., Baskin, R. J., Kowalczykowski, S. C. DNA解绕的非均匀性研究。gydF4y2Ba自然gydF4y2Ba500gydF4y2Ba, 482-485(2013)。gydF4y2Ba
Saikrishnan, K, Griffiths, S. P, Cook, N., Court, R. & Wigley, D. B. DNA与RecD的结合:1B结构域在SF1B解旋酶活性中的作用。gydF4y2BaEMBO JgydF4y2Ba。gydF4y2Ba27gydF4y2Ba, 2222-2229(2008)。gydF4y2Ba
Farah, J. A. & Smith, G. R. DNA解绕的RecBCD酶起始复合物:酶定位和DNA打开。gydF4y2BaJ.摩尔生物学gydF4y2Ba。gydF4y2Ba272gydF4y2Ba, 699-715(1997)。gydF4y2Ba
冯·希佩尔,p.h.,约翰逊,n.p.和马库斯,a.h.五十年的DNA“呼吸”:对新旧方法的反思。gydF4y2Ba生物聚合物gydF4y2Ba99gydF4y2Ba科学通报,923-954(2013)。gydF4y2Ba
Wu, C. G.和Lohman, T. M. DNA末端结构对DNA解绕起始机制的影响gydF4y2Ba大肠杆菌gydF4y2BaRecBCD和RecBC解旋酶。gydF4y2BaJ.摩尔生物学gydF4y2Ba。gydF4y2Ba382gydF4y2Ba中文信息学报,312-326(2008)。gydF4y2Ba
卡特,a.r.等人。单个RecBCD-DNA复合物的序列依赖的纳米级构象动力学。gydF4y2Ba核酸类gydF4y2Ba。gydF4y2Ba44gydF4y2Ba, 5849-5860(2016)。gydF4y2Ba
Dillingham, M. S, Webb, M. R. & Kowalczykowski, S. C.双极DNA易位有助于RecBCD酶的高进程DNA解绕。gydF4y2Ba生物。化学gydF4y2Ba。gydF4y2Ba280gydF4y2Ba, 37069-37077(2005)。gydF4y2Ba
Forde, N. R., Izhaky, D., Woodcock, G. R., Wuite, G. J. L.和Bustamante, C.使用机械力来探测在连续拉伸过程中暂停和停止的机制gydF4y2Ba大肠杆菌gydF4y2BaRNA聚合酶。gydF4y2Ba自然科学进展。美国gydF4y2Ba99gydF4y2Ba[j], 2002。gydF4y2Ba
阿德尔曼等人。RNA聚合酶延伸的单分子分析揭示了均匀的动力学行为。gydF4y2Ba自然科学进展。美国gydF4y2Ba99gydF4y2Ba, 13538-13543(2002)。gydF4y2Ba
Neuman, K. C, Abbondanzieri, E. A, Landick, R., Gelles, J. & Block, S. M.无所不在的转录暂停与RNA聚合酶回溯无关。gydF4y2Ba细胞gydF4y2Ba115gydF4y2Ba, 437-447(2003)。gydF4y2Ba
Abbondanzieri, E. A, Greenleaf, W. J, Shaevitz, J. W, Landick, R. and Block, S. M.直接观察RNA聚合酶的碱基对步进。gydF4y2Ba自然gydF4y2Ba438gydF4y2Ba, 460-465(2005)。gydF4y2Ba
Righini, M.等。RNA聚合酶在单碱基对分辨率下的完整分子轨迹。gydF4y2Ba自然科学进展。美国gydF4y2Ba115gydF4y2Ba, 1286-1291(2018)。gydF4y2Ba
卡布希,W.,桑德,C.和特里福诺夫,E. N. B-DNA的十个螺旋扭曲角。gydF4y2Ba核酸类gydF4y2Ba。gydF4y2Ba10gydF4y2Ba, 1097-1104(1982)。gydF4y2Ba
Kopperger, E.等。由电场控制的自组装纳米级机械臂。gydF4y2Ba科学gydF4y2Ba359gydF4y2Ba, 296-301(2018)。gydF4y2Ba
致谢gydF4y2Ba
这项工作得到了美国国立卫生研究院的部分支持。b.d.a获得了哈佛大学生物物理学研究生项目国家卫生研究院培训基金和国家科学基金会研究生研究奖学金的支持。医学博士由霍华德休斯医学研究所国际学生研究奖学金支持。X.Z.是霍华德休斯医学研究所的研究员。gydF4y2Ba
作者信息gydF4y2Ba
作者及单位gydF4y2Ba
贡献gydF4y2Ba
p.k., B.D.A.和X.Z.构思了这个项目并设计了实验。P.K.和b.d.a进行了ORBIT实验并分析了数据。m.d., P.K.和P.Y.设计了折纸结构。B.D.A, M.D.和P.K.准备了折纸结构。P.K.和M.D.进行了原子力显微镜和透射电子显微镜成像。p.k., B.D.A.和X.Z.在医学博士和P.Y.的帮助下撰写了这份手稿gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益冲突。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意:gydF4y2Bab施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。gydF4y2Ba
扩展数据图和表gydF4y2Ba
图1折纸转子和锚的设计。gydF4y2Ba
一个gydF4y2Ba,两条160nm臂组成的折纸转子走线图(补充表)gydF4y2Ba1gydF4y2Ba).完整的臂(六螺旋束)穿过正交臂(两个半长六螺旋束)的断裂。附加的螺旋稳定结。其中两个螺旋包含从转子中心延伸的短纤维(黑色)(延伸未显示;看到gydF4y2BacgydF4y2Ba).在完整臂末端14 nm内的6个订针(浅绿色)在其3 '端标记Cy3。gydF4y2BabgydF4y2Ba、转子设计的三维效果图。gydF4y2BacgydF4y2Ba放大图显示两条短链(红色)从转子中心延伸,形成一个14bp的dsDNA片段和一个12nt的ssDNA悬垂用于结扎。gydF4y2BadgydF4y2Ba突出部分连接到较长的dsDNA片段,后者作为dna相互作用酶的底物。gydF4y2BaegydF4y2Ba原子力显微镜图像与大视野的折纸转子。代表了十多个独立的生物重复。标尺:1 μm。gydF4y2BafgydF4y2Ba,由三个20nm翅膀组成的折纸锚的路线图,每个翅膀由一个短的6螺旋束基序组成(补充表)gydF4y2Ba2gydF4y2Ba).一些短纤维链被延长了与生物素标记的次级低聚物的结合位点,用于表面附着。从结构的中心,三股(黑色)被用来制作适配器,以便连接额外的DNA。在最后的链交叉之后,接头由26 bp的双链dna和一个12 nt的双链dna悬垂组成。gydF4y2BaggydF4y2Ba,折纸锚的三维效果图。gydF4y2BahgydF4y2Ba,用于表征布朗动力学的折纸结构。将折纸转子、锚和dsDNA片段(根据需要)连接在一起。折纸锚通过多个生物素标签附着在显微镜表面。gydF4y2Ba我gydF4y2Ba,折纸锚的代表性透射电镜图像。gydF4y2Ba
图2折纸转子的角度和径向位置不确定性表征。gydF4y2Ba
一个gydF4y2Ba,功率谱显示了52bp dsDNA附着在锚点上的转子角位置的布朗波动。3个独立生物重复的代表性。红线表示修改后的洛伦兹拟合,如gydF4y2Ba补充讨论gydF4y2Ba,补充式(1)。这种拟合得到的扭转刚度为gydF4y2BaκgydF4y2Ba)和水动力阻力(gydF4y2BaγgydF4y2Ba).gydF4y2BabgydF4y2Ba的逆的依赖关系gydF4y2BaκgydF4y2Ba在转子和折纸之间的dsDNA片段的长度。线性拟合的斜率在没有施加力的情况下产生dsDNA单位长度的扭转刚度(补充讨论,补充方程(3))。gydF4y2BaCgydF4y2Ba= 200±10 pN nmgydF4y2Ba2gydF4y2BaradgydF4y2Ba−1gydF4y2Ba,这与之前在零力下的测量结果一致gydF4y2Ba12gydF4y2Ba。的倒数gydF4y2BaygydF4y2Ba-从这个匹配中抵消,gydF4y2BaκgydF4y2Ba其他gydF4y2Ba= 30±8 pN nm radgydF4y2Ba−1gydF4y2Ba(补充讨论,补充方程(3)),表示结构剩余部分的扭转刚度,相当于约20bp的dsDNA。gydF4y2BacgydF4y2Ba,弛豫时间的依赖性,gydF4y2BaτgydF4y2Ba=gydF4y2BaγgydF4y2Ba/gydF4y2BaκgydF4y2Ba,对转子和锚之间的dsDNA段的长度进行计算gydF4y2BaκgydF4y2Ba和gydF4y2BaγgydF4y2Ba由功率谱拟合(gydF4y2Ba补充讨论,补充方程(1)gydF4y2Ba).数据gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba平均值±S.E.M. (gydF4y2BangydF4y2Ba= 203, 195和133(从左至右),来自3个独立的生物重复。gydF4y2BadgydF4y2Ba的依赖性gydF4y2BaγgydF4y2Ba折纸转子对缓冲液粘度的影响。折纸转子通过一个92-bp的dsDNA片段连接到锚点。使用0%、10%和25%的甘油可获得不同的粘度。数据为平均值±s.e.m。(gydF4y2BangydF4y2Ba= 195, 210和150(从左到右),来自3个独立的生物重复。gydF4y2BaegydF4y2Ba,转子角位置的标准差作为积分时间的函数。黑线显示了在降采样到指定的积分时间后,从连接到锚的单个转子测量到的sd,该转子具有以3 kHz跟踪的52 bp dsDNA片段。3个独立生物重复的代表性。红色和蓝色曲线分别表示考虑了定位不确定性(gydF4y2Ba补充讨论,补充方程(2)gydF4y2Ba).gydF4y2BaκgydF4y2Ba= 7.8 pN nm radgydF4y2Ba−1gydF4y2Ba,gydF4y2BaγgydF4y2Ba= 5.0 fN nm s,每帧定位不确定度gydF4y2Ba\({\σ}_ {L} ^ {2} \)gydF4y2Ba= 0.038 radgydF4y2Ba2gydF4y2Ba。gydF4y2BaκgydF4y2Ba和gydF4y2BaγgydF4y2Ba是由多个(gydF4y2BangydF4y2Ba= 203)转子,52-bp dsDNA片段将转子连接到锚点。gydF4y2Ba\({\σ}_ {L} ^ {2} \)gydF4y2Ba利用径向位置的测量不确定度进行估计,并利用圆形轨迹的半径转换为角度值。顶部和底部虚线与sd与积分时间曲线的交叉点分别给出了检测单碱基对旋转(34.6°)所需的积分时间,信噪比分别为1和3。gydF4y2BafgydF4y2Ba径向定位不确定性(s.d)与单个转子平均荧光信号强度(平均值)的函数关系。3个独立生物重复的代表性。我们采用16 nm(0.1像素)的定位不确定性阈值,如图所示为红色,以选择具有相对较高定位精度的轨迹。所有的测量都用0%甘油进行,除了gydF4y2BadgydF4y2Ba。gydF4y2Ba
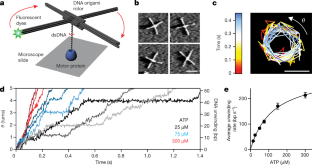
图3放卷速率分布。gydF4y2Ba
一个gydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba,不同ATP浓度(溶液pH 8)下单个分子平均解绕速率直方图:15 μM ATP (gydF4y2Ba一个gydF4y2Ba), 25 μm atp (gydF4y2BabgydF4y2Ba), 50 μm atp (gydF4y2BacgydF4y2Ba), 75 μm atp (gydF4y2BadgydF4y2Ba), 150 μm atp (gydF4y2BaegydF4y2Ba)和300 μM ATP (gydF4y2BafgydF4y2Ba).gydF4y2Ba
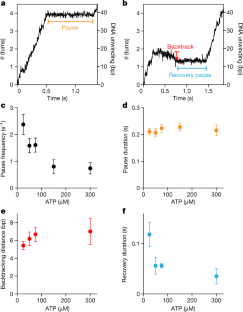
图4溶液pH值为6时RecBCD放卷速率和暂停特性gydF4y2Ba
一个gydF4y2Ba平均解绕速率作为ATP浓度的函数符合Michaelis-Menten依赖性gydF4y2BavgydF4y2Ba马克斯gydF4y2Ba= 290±10 bp sgydF4y2Ba−1gydF4y2Ba,gydF4y2BaKgydF4y2Ba米gydF4y2Ba= 130±10 μm。数据为平均值±s.e.m。(gydF4y2BangydF4y2Ba= 47、94、80、37和110(从左到右),每种条件下至少有3个独立的生物复制轨迹)。gydF4y2BabgydF4y2Ba, ATP浓度对暂停频率的依赖性。暂停频率确定为每个单分子轨迹每秒暂停的平均次数。数据为平均值±s.e.m。(gydF4y2BangydF4y2Ba= 47、94、80、37和110(从左到右)轨迹,每种条件下至少有3个独立的生物重复)。gydF4y2BacgydF4y2Ba,在不同浓度的ATP下,正向解绕过程中停顿的中位数持续时间。误差条为重采样(gydF4y2BangydF4y2Ba= 76, 62, 27和18(从左到右)事件,每种条件下至少有3个独立的生物重复)。gydF4y2BadgydF4y2Ba,不同ATP浓度下的平均回溯距离。数据为平均值±s.e.m。(gydF4y2BangydF4y2Ba= 20、22、16和10(从左到右)事件,每种条件下至少有3个独立的生物重复)。gydF4y2BaegydF4y2Ba,在不同浓度的ATP下,回溯事件后的中位数恢复暂停时间。误差条是重采样得到的中位数的标准差。的gydF4y2BaPgydF4y2Ba25 μ m - atp数据点与50、75和300 μ m - atp数据点的差值分别为0.05、0.004和0.09,由暂停持续时间分布的双侧Kolmogorov-Smirnov检验得出(gydF4y2BangydF4y2Ba= 20、22、16和10,分别对应25、50、75和300 μM数据点)。注意,并非所有轨迹都包含回溯事件。数据在10%甘油,溶液pH值6500 Hz下获得。gydF4y2BafgydF4y2BaRecBCD诱导DNA解绕动力学模型示意图。在DNA解绕过程中,暂停频繁发生,一些暂停导致酶回溯;酶可以退出回溯状态,并通过恢复暂停中间体恢复DNA解绕,这与初始暂停状态不同。gydF4y2Ba
扩展数据图5使用整体停流法测量RecBCD展开。gydF4y2Ba
一个gydF4y2Ba,停流实验DNA底物的设计gydF4y2Ba21gydF4y2Ba。A链上的Cy3最初被b链上的Cy5猝灭,RecBCD活性导致A链解离,导致荧光增加。左侧的发夹(链C)确保活动只能从右侧开始。gydF4y2BabgydF4y2Ba、双混停流实验设计。在与ATP和肝素混合之前,将RecBCD和DNA在延迟环路中混合200 ms,以防止单次翻转后RecBCD - DNA的额外结合。gydF4y2BacgydF4y2Ba在ATP为50 μM,溶液pH为8,10%甘油条件下,A链为26 bp(黑色)或52 bp(绿色)的钝端底物上的停流荧光测量。具有至少3个独立生物重复的代表性。两个样品的A链长度之差与测量到的两个样品的半上升时间之差之比可以估计出解绕速率约为100 bp sgydF4y2Ba−1gydF4y2Ba假设这两种底物的起始动力学没有不同(因为它们在双链断裂处具有相同的几何形状和序列)。这个解绕速率相当于85 bp / sgydF4y2Ba−1gydF4y2Ba我们的单分子ORBIT测量在相同浓度的ATP下确定的值。gydF4y2Ba
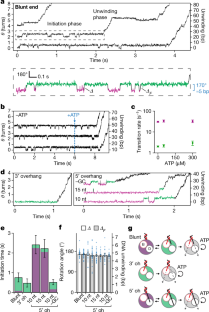
图6在ATP为50 μM,溶液pH为8的条件下,不同类型底物上的RecBCD起始相持续时间分布。gydF4y2Ba
图的复制gydF4y2Ba3 egydF4y2Ba,但将起始期持续时间的单个数据点叠加为点图。柱状图统计如图所示。gydF4y2Ba3 egydF4y2Ba。注意,由于动力学转变的随机性,起爆期持续时间的分布预期为长尾指数型分布。gydF4y2Ba
图7 300 μM ATP下,溶液pH为8时,RecBCD的起始动力学。gydF4y2Ba
一个gydF4y2Ba例如,钝端DNA底物和6-nt 3 '或10-nt 5 '悬垂DNA底物的起始轨迹。具有至少3个独立生物重复的代表性。绿色和品红的颜色编码如图所示。gydF4y2Ba3gydF4y2Ba。gydF4y2BabgydF4y2Ba,钝端底物和6-nt 3 '或10-nt 5 '悬垂底物起始阶段的平均持续时间(从底物结合到过程解绕的总时间)。绿色和品红部分分别表示绿色和品红状态下的平均累积停留时间。误差条为s.e.m。(gydF4y2BangydF4y2Ba= 80, 44和85(从左到右)至少3个独立生物复制的轨迹)。起始期持续时间的单个数据点被叠加为点图。在300 μM ATP,溶液pH 8, 10%甘油,500 Hz条件下采集数据。gydF4y2Ba
图8采用整体停流法测量的RecBCD解绕动力学。gydF4y2Ba
一个gydF4y2BaDNA底物的设计为停止流动实验,如扩展数据图所示。gydF4y2Ba5 a、bgydF4y2Ba,除了添加3 '或5 ' ssDNA悬垂(虚线),以制备具有悬垂的相应底物。gydF4y2BabgydF4y2Ba对钝端底物和6-nt 3 '或10-nt 5 '悬垂底物的停流荧光测量显示,具有5 '悬垂的底物动力学较慢。gydF4y2BacgydF4y2Ba对10-nt 5′悬垂底物或10-nt 5′悬垂底物的停流荧光测量,初始5个碱基对中G-C对转化为A-T。gydF4y2BadgydF4y2Ba基于单分子数据(在溶液pH值为8时收集)得出的起始和解卷速率预测的集合时间过程(红线)与在几种pH值下钝端底物上测量的停止流动时间过程(彩色符号)的比较。为了模拟没有拟合参数的集合时间过程,我们使用我们从单分子数据中测量的起始速率将起始过程建模为单个指数过程,并将unwind建模为一系列1 bp的unwind步骤,其中模拟中的每个分子的unwind速率来自我们从单分子数据中实验测量的unwind速率分布。正如预期的那样,由于二氧化硅复盖表面带电(导致局部pH值漂移约2个单位;看到gydF4y2Ba补充讨论gydF4y2Ba),在溶液pH为8(表面pH为6)时,单分子实验测量的起始和解绕速率预测曲线与pH为6时测量的停止流动数据相匹配。gydF4y2BaegydF4y2BapH值为6时钝端底物的停止流动数据(红色符号)与从溶液pH值为8(表面pH值为6)的单分子数据中获得的起始和解绕速率(红色)或仅从解绕速率(黑线)预测的时间过程的比较。因此,需要在模拟中包含起始相以匹配实验结果。gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2BapH值为6时的停流数据(彩色符号)的比较见gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba利用从溶液pH为8(表面pH为6)的单分子数据得出的起始和解绕速率来预测集合时间过程(颜色匹配的线),如下所述gydF4y2BadgydF4y2Ba。所有停流实验均在50 μM ATP、10%甘油、pH 6条件下进行gydF4y2BadgydF4y2Ba,其中pH值在图例中表示),并且每种条件下至少代表3个单独的实验。相同颜色的图是相同数据集的副本,放置在不同的面板中,用于比较。gydF4y2Ba
图9不同pH值下的RecBCD起始动力学。gydF4y2Ba
一个gydF4y2Ba在50 μM ATP,溶液pH 6, 10%甘油,500 Hz下记录的单分子轨迹显示RecBCD在钝端底物上引发。任意垂直偏移应用于不同的轨迹显示目的。虚线框中的区域放大如下,其中缠绕和未缠绕的角度状态分别用品红和绿色标记。gydF4y2BabgydF4y2Ba在相同条件下,单分子底物在6-nt 3 '或10-nt 5 '悬垂的底物上显示RecBCD起始。虚线框划定的区域在插图中被放大。数据gydF4y2Ba一个gydF4y2Ba,gydF4y2BabgydF4y2Ba至少代表3个独立的生物重复。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,溶液pH为6时,钝端底物和3′或5′悬垂底物的起始期平均持续时间(从底物结合到过程解绕的总时间)(gydF4y2BacgydF4y2Ba)及溶液pH值7 (gydF4y2BadgydF4y2Ba), 50 μM ATP, 10%甘油。绿色和品红部分分别表示在绿色和品红状态下的累积停留时间。误差条为s.e.m。(gydF4y2BangydF4y2Ba= 40, 27和44(从左到右)的轨迹gydF4y2BacgydF4y2Ba;gydF4y2BangydF4y2Ba= 149, 154和108(从左到右)的轨迹gydF4y2BadgydF4y2Ba(每种条件下至少3个独立的生物重复)。起始期持续时间的单个数据点被叠加为点图。gydF4y2Ba
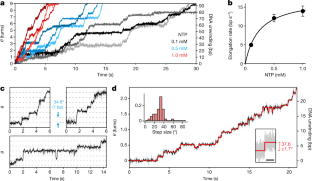
图10 RNAP碱基对步进的附加分析。gydF4y2Ba
从rnap诱导DNA旋转的单分子轨迹的隐马尔可夫模型分析得到前向步长(>5°)的全概率分布。在这里,使用单个单分子轨迹中检测到的所有步长概率来构建直方图(而不是使用每个轨迹的最可能步长,如图所示)。gydF4y2Ba4 dgydF4y2Ba).gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
补充讨论的PDF文件,包含对技术要点的额外讨论,补充方法,包含与本文相关的所有方法细节,补充参考文献,补充表3和4,以及所有补充表的标题。gydF4y2Ba
补充表1gydF4y2Ba
一个.xlsx文件与补充表1 | DNA低聚物用于折纸转子。该表包含用于折叠DNA折纸转子的DNA寡聚物序列的列表。gydF4y2Ba
补充表2gydF4y2Ba
一个.xlsx文件与补充表2 | DNA低聚物用于折纸基础。此表包含用于折叠折纸碱基的DNA低聚物序列的列表。gydF4y2Ba
补充视频1gydF4y2Ba
recbcd诱导的DNA解绕过程的单分子ORBIT轨迹。recbcd诱导的DNA过程解绕的单分子ORBIT轨迹。折纸- dsdna复合物从溶液中扩散并与RecBCD结合。经过相对短暂的起始期后,由于在50µM ATP条件下,RecBCD对dsDNA底物进行解绕,折纸进行了持续旋转。在折纸转子的尖端染料的定位显示。轨迹至少代表了三个独立的实验。gydF4y2Ba
补充视频2gydF4y2Ba
recbcd诱导的具有长起始期的DNA解绕过程的单分子ORBIT轨迹。recbcd诱导的具有长起始期的DNA解绕过程的单分子ORBIT轨迹。折纸双链DNA复合体从溶液中扩散并与RecBCD结合。经过起始阶段,在两个角位置之间显示出几个可逆的转变~170gydF4y2Ba°gydF4y2Ba由于可逆的、与ATP无关的前5bp的解绕转变,折纸开始旋转,这是由于DNA底物在50µM ATP下被RecBCD解绕。在折纸转子的尖端染料的定位显示。轨迹至少代表了三个独立的实验。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Kosuri, P., Altheimer, b.d., Dai, M.。gydF4y2Baet al。gydF4y2Ba利用DNA折纸转子进行基因组加工酶的旋转跟踪。gydF4y2Ba自然gydF4y2Ba572gydF4y2Ba, 136-140(2019)。https://doi.org/10.1038/s41586-019-1397-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-019-1397-7gydF4y2Ba
这篇文章是由gydF4y2Ba
利用分子扭转弹簧在DNA纳米机器人中储存机械能gydF4y2Ba
自然物理gydF4y2Ba(2023)gydF4y2Ba
DNA双螺旋结构,微型电动机gydF4y2Ba
自然纳米技术gydF4y2Ba(2023)gydF4y2Ba
数字纳米反应器控制绝对化学计量和时空行为的DNA受体内脂质双层gydF4y2Ba
自然通讯gydF4y2Ba(2023)gydF4y2Ba
功能化DNA折纸研究和与生物系统的相互作用gydF4y2Ba
《自然评论》gydF4y2Ba(2022)gydF4y2Ba
MINFLUX的替代方案,可在共聚焦显微镜中实现纳米分辨率gydF4y2Ba
光:科学与应用gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
