摘要gydF4y2Ba
确定染色体结构、核体、染色质状态和基因表达之间的关系是核组织研究的首要目标gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.因为单个细胞在所有这些水平上都是高度可变的gydF4y2Ba5gydF4y2Ba,在同一细胞中绘制不同的模式是至关重要的。在这里,我们报道了使用DNA seqFISH+在单个小鼠胚胎干(ES)细胞中成像3660个染色体位点,以及17个染色质标记和序列免疫荧光亚核结构,以及70个rna的表达谱。在单个小鼠胚胎干细胞中,许多基因座总是与免疫荧光标记相关。这些位点在单细胞的核组织中形成“固定点”,经常出现在核体表面和由组合染色质标记定义的区域上。此外,高表达基因似乎预先定位于活跃的核区,独立于单细胞的爆发动力学。我们的分析还发现了几种具有特征组合染色质状态的不同小鼠ES细胞亚群。通过克隆分析,我们发现一些染色质标记的整体水平,如赖氨酸27的H3三甲基化(H3K27me3)和macroH2A1 (mH2A1),至少可以遗传3-4代,而其他标记则在更快的时间尺度上波动。这种基于seqFISH+的空间多模态方法可用于探索不同生物系统中的核组织和细胞状态。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
哺乳动物组织的空间表观基因组-转录组联合分析gydF4y2Ba
自然gydF4y2Ba开放获取gydF4y2Ba2023年3月15日gydF4y2Ba
高效、鲁棒的π-FISH彩虹,用于多种生物分子的多重原位检测gydF4y2Ba
自然通讯gydF4y2Ba开放获取gydF4y2Ba2023年1月27日gydF4y2Ba
用于从多重DNA FISH中解析染色质结构的空间基因组校准器gydF4y2Ba
自然生物技术gydF4y2Ba开放获取gydF4y2Ba2023年1月2日gydF4y2Ba
访问选项gydF4y2Ba
访问《自然》和其他54种《自然》杂志gydF4y2Ba
获取Nature+,我们最超值的在线订阅gydF4y2Ba
每月29.99美元gydF4y2Ba
随时取消gydF4y2Ba
订阅这本杂志gydF4y2Ba
收到51个印刷问题和在线访问gydF4y2Ba
199.00美元一年gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
租或购买这篇文章gydF4y2Ba
只要这篇文章,只要你需要它gydF4y2Ba
39.95美元gydF4y2Ba
价格可能受当地税收的影响,在结账时计算gydF4y2Ba
数据可用性gydF4y2Ba
本研究的原始数据和处理数据可在Zenodo (gydF4y2Bahttps://zenodo.org/record/3735329gydF4y2Ba).本研究期间获得的其他原始显微数据可根据合理要求从通讯作者处获得。本研究中使用的公开数据集(GSE96107, 4DNESOJRTZZR, GSE17051, GSE102076, GSE48895, ENCSR000CFN, ENCSR000CGP, ENCSR000CGQ)在方法中有详细说明。gydF4y2Ba
代码的可用性gydF4y2Ba
本研究中使用的自定义脚本可从以下网站获得gydF4y2Bahttps://github.com/CaiGroup/dna-seqfish-plusgydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
戴克尔,J.等人。4D核体计划。gydF4y2Ba自然gydF4y2Ba549gydF4y2Ba, 219-226(2017)。gydF4y2Ba
Kelsey, G., Stegle, O. & Reik, W.单细胞表观基因组学:记录过去和预测未来。gydF4y2Ba科学gydF4y2Ba358gydF4y2Ba, 69-75(2017)。gydF4y2Ba
Kempfer, R. & Pombo, A.绘制3D染色体结构的方法。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba21gydF4y2Ba, 207-226(2020)。gydF4y2Ba
朱春,普里斯尔,任斌。单细胞多模态组学:多人的力量。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 11-14(2020)。gydF4y2Ba
冯志伟,陈志伟,陈志伟。空间基因组组织变异的分子基础和生物学功能。gydF4y2Ba科学gydF4y2Ba365gydF4y2Ba, eaaw9498(2019)。gydF4y2Ba
Lieberman-Aiden, E.等人。对远距离相互作用的全面测绘揭示了人类基因组的折叠原理。gydF4y2Ba科学gydF4y2Ba326gydF4y2Ba, 289-293(2009)。gydF4y2Ba
藜麦酮,S. A.等。高阶染色体间轮毂在细胞核中形成三维基因组组织。gydF4y2Ba细胞gydF4y2Ba174gydF4y2Ba, 744-757(2018)。gydF4y2Ba
王,S.等。单染色体染色质结构域和区室的空间组织。gydF4y2Ba科学gydF4y2Ba353gydF4y2Ba, 598-602(2016)。gydF4y2Ba
Bintu, B.等。超分辨率染色质示踪揭示单细胞结构域和协同作用。gydF4y2Ba科学gydF4y2Ba362gydF4y2Ba, eaau1783(2018)。gydF4y2Ba
Nir, G.等。通过超分辨率成像、接触图和综合建模沿着染色体行走。gydF4y2Ba公共科学图书馆麝猫gydF4y2Ba.gydF4y2Ba14gydF4y2Ba, e1007872(2018)。gydF4y2Ba
卡多佐·吉齐,等人。基于显微镜的染色体构象捕获能够同时可视化完整生物体的基因组组织和转录。gydF4y2Ba摩尔。细胞gydF4y2Ba74gydF4y2Ba, 212-222(2019)。gydF4y2Ba
芬恩,e.h.等人。空间基因组组织的广泛异质性和内在变异。gydF4y2Ba细胞gydF4y2Ba176gydF4y2Ba, 1502-1515(2019)。gydF4y2Ba
马特奥,L. J.等。在单细胞分辨率下可视化胚胎中的DNA折叠和RNA。gydF4y2Ba自然gydF4y2Ba568gydF4y2Ba, 49-54(2019)。gydF4y2Ba
Nguyen, H. Q.等人。三维测绘和加速超分辨率成像的人类基因组使用原位测序。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 822-832(2020)。gydF4y2Ba
苏,黄永发。,Zheng,P.,Kinrot, S. S., Bintu, B. & Zhuang, X. Genome-scale imaging of the 3d organization and transcriptional activity of chromatin.细胞gydF4y2Ba182gydF4y2Ba, 1641-1659(2020)。gydF4y2Ba
贝利沃,B. J.等。使用Oligopaint FISH探针可视化基因组的多功能设计和合成平台。gydF4y2Ba国家科学院学报美国gydF4y2Ba109gydF4y2Ba, 21301-21306(2012)。gydF4y2Ba
Lubeck, E., Coskun, A. F., Zhiyentayev, T., Ahmad, M. & Cai, L.单细胞原位RNA序列杂交分析。gydF4y2BaNat方法。gydF4y2Ba11gydF4y2Ba, 360-361(2014)。gydF4y2Ba
Shah, S., Lubeck, E., Zhou, W. & Cai, L.单细胞原位转录剖析揭示了小鼠海马细胞的空间组织。gydF4y2Ba神经元gydF4y2Ba92gydF4y2Ba, 342-357(2016)。gydF4y2Ba
Takei, Y., Shah, S., Harvey, S., Qi, L. S. & Cai, L.结合CRISPR成像和DNA序列FISH的基因组位点的多重动态成像。gydF4y2BaBiophys。JgydF4y2Ba.gydF4y2Ba112gydF4y2Ba, 1773-1776(2017)。gydF4y2Ba
Shah, S.等人。内含子seqFISH的新生转录组动力学和空间基因组学研究。gydF4y2Ba细胞gydF4y2Ba174gydF4y2Ba, 363-376(2018)。gydF4y2Ba
英格,C. L.等。RNA seqFISH+在组织中的转录组尺度超分辨成像。gydF4y2Ba自然gydF4y2Ba568gydF4y2Ba, 235-239(2019)。gydF4y2Ba
陈凯宏,陈建荣,陈建荣,王淑娟,庄晓霞,RNA成像。空间分辨率,单细胞中高度多路RNA分析。gydF4y2Ba科学gydF4y2Ba348gydF4y2Ba, aaa6090(2015)。gydF4y2Ba
Bonev, B.等人。小鼠神经发育过程中的多尺度三维基因组重布线。gydF4y2Ba细胞gydF4y2Ba171gydF4y2Ba, 557 - 572。e24(2017).
沈,Y.等。小鼠基因组中的顺式调控序列图。gydF4y2Ba自然gydF4y2Ba488gydF4y2Ba, 116-120(2012)。gydF4y2Ba
伯蒂格,a.n.等人。超分辨率成像揭示了不同表观遗传状态下不同的染色质折叠。gydF4y2Ba自然gydF4y2Ba529gydF4y2Ba, 418-422(2016)。gydF4y2Ba
van Steensel, B. & Belmont, A. S. lamina相关结构域:与染色体结构、异染色质和基因抑制有关。gydF4y2Ba细胞gydF4y2Ba169gydF4y2Ba, 780-791(2017)。gydF4y2Ba
斯派特,d.l.和拉蒙德,a.i.核斑点。gydF4y2Ba冷泉港。教谕。医学杂志gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, a000646(2011)。gydF4y2Ba
Pederson, T.核仁。gydF4y2Ba冷泉港。教谕。医学杂志gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, a000638(2011)。gydF4y2Ba
路德维希,C. H. &宾图,L.在单细胞水平上映射染色质修饰。gydF4y2Ba发展gydF4y2Ba146gydF4y2Ba, dev170217(2019)。gydF4y2Ba
Söderberg, O.等。近距离结扎法原位直接观察个体内源性蛋白复合物。gydF4y2BaNat方法。gydF4y2Ba3.gydF4y2Ba, 995-1000(2006)。gydF4y2Ba
阿加斯蒂,s.s.等人。用于高复用Exchange-PAINT成像的dna条形码标记探针。gydF4y2Ba化学。ScigydF4y2Ba.gydF4y2Ba8gydF4y2Ba, 3080-3091(2017)。gydF4y2Ba
Guenatri, M., Bailly, D., Maison, C. & Almouzni, G.小鼠中心和中心周围卫星重复序列形成不同的功能异染色质。gydF4y2BaJ.细胞生物学gydF4y2Ba.gydF4y2Ba166gydF4y2Ba, 493-505(2004)。gydF4y2Ba
Solovei, I.等。哺乳动物进化中视杆感光细胞的核结构适应视觉。gydF4y2Ba细胞gydF4y2Ba137gydF4y2Ba, 356-368(2009)。gydF4y2Ba
毛玉生,张波,张德龙。核体的生物成因与功能。gydF4y2Ba趋势麝猫gydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 295-306(2011)。gydF4y2Ba
佩里-哈普克斯,D.等人。分化过程中基因组-核层相互作用重组的分子图谱。gydF4y2Ba摩尔。细胞gydF4y2Ba38gydF4y2Ba, 603-613(2010)。gydF4y2Ba
Kind, J.等。单个人类细胞中核层相互作用的全基因组图谱。gydF4y2Ba细胞gydF4y2Ba163gydF4y2Ba, 134-147(2015)。gydF4y2Ba
陈,Y.等。使用TSA-seq作为细胞学标尺绘制相对于核隔室的3D基因组组织。gydF4y2BaJ.细胞生物学gydF4y2Ba.gydF4y2Ba217gydF4y2Ba, 4025-4048(2018)。gydF4y2Ba
Gut, G, Herrmann, m.d. & Pelkmans, L.多路蛋白图链接亚细胞组织到细胞状态。gydF4y2Ba科学gydF4y2Ba361gydF4y2Ba, eaar7042(2018)。gydF4y2Ba
McSwiggen, D. T., Mir, M., Darzacq, X. & Tjian, R.评估活细胞中的相分离:诊断、警告和功能后果。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba33gydF4y2Ba, 1619-1634(2019)。gydF4y2Ba
马克斯,H.等人。基态多能性的转录和表观基因组基础。gydF4y2Ba细胞gydF4y2Ba149gydF4y2Ba, 590-604(2012)。gydF4y2Ba
辛格,Z. S.等。胚胎干细胞的动态异质性和DNA甲基化。gydF4y2Ba摩尔。细胞gydF4y2Ba55gydF4y2Ba, 319-331(2014)。gydF4y2Ba
Kolodziejczyk, a.a.等人。多能状态的单细胞rna测序解锁模块化转录变异。gydF4y2Ba细胞干细胞gydF4y2Ba17gydF4y2Ba, 471-485(2015)。gydF4y2Ba
托索里尼等人。不同类型小鼠多能干细胞异染色质表观遗传状态的对比。gydF4y2Ba科学。代表gydF4y2Ba.gydF4y2Ba8gydF4y2Ba, 5776(2018)。gydF4y2Ba
van Mierlo, G.等人。整合蛋白质组分析显示prc2依赖的表观遗传串扰保持基态多能性。gydF4y2Ba细胞干细胞gydF4y2Ba24gydF4y2Ba, 123 - 137。e8(2019)。gydF4y2Ba
Gerlich, D.等人。在哺乳动物细胞中,全局染色体位置通过有丝分裂传递。gydF4y2Ba细胞gydF4y2Ba112gydF4y2Ba, 751-764(2003)。gydF4y2Ba
Walter, J., Schermelleh, L., Cremer, M., Tashiro, S. & Cremer, T. HeLa细胞的染色体顺序在有丝分裂和G1早期发生变化,但在随后的间期阶段稳定维持。gydF4y2BaJ.细胞生物学gydF4y2Ba.gydF4y2Ba160gydF4y2Ba, 685-697(2003)。gydF4y2Ba
Thomson, I., Gilchrist, S., Bickmore, W. A. & Chubb, J. R.染色质的径向定位不是通过有丝分裂遗传的,而是在G1早期重新建立的。gydF4y2Ba咕咕叫。医学杂志gydF4y2Ba.gydF4y2Ba14gydF4y2Ba, 166-172(2004)。gydF4y2Ba
Essers, J.等人。细胞周期内染色体相对位置的动态变化。gydF4y2Ba摩尔。杂志。细胞gydF4y2Ba16gydF4y2Ba, 769-775(2005)。gydF4y2Ba
Hormoz, S.等人从谱系树和端点单细胞测量推断细胞状态转换动态。gydF4y2Ba细胞系统gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, 419-433(2016)。gydF4y2Ba
坎宁安等人。运用2019年。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba47gydF4y2Ba, d745-d751(2019)。gydF4y2Ba
克莱因等人。用于单细胞转录组学的液滴条形码应用于胚胎干细胞。gydF4y2Ba细胞gydF4y2Ba161gydF4y2Ba, 1187-1201(2015)。gydF4y2Ba
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 357-359(2012)。gydF4y2Ba
卡马乔等。BLAST+:架构和应用程序。gydF4y2BaBMC生物信息学gydF4y2Ba10gydF4y2Ba, 421(2009)。gydF4y2Ba
鲍,W., Kojima, K. K. & Kohany, O. Repbase Update,一个真核生物基因组重复元素数据库。gydF4y2Ba暴徒。DNAgydF4y2Ba6gydF4y2Ba, 11(2015)。gydF4y2Ba
Eng, C. L., Shah, S., Thomassie, J. & Cai, L.用RNA斑点分析转录组。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 1153-1155(2017)。gydF4y2Ba
Cremer, C.等在gydF4y2Ba计算机视觉与应用手册gydF4y2BaVol. 3 (ed. Jahne, B. et al) 839-857(学术,1999)。gydF4y2Ba
克罗夫特,J. A.等。人类细胞核中染色体位置和形态的差异。gydF4y2BaJ.细胞生物学gydF4y2Ba.gydF4y2Ba145gydF4y2Ba, 1119-1131(1999)。gydF4y2Ba
Esa, A.等。三色DNA标记后染色质纳米结构的三维光谱精确距离显微镜:22号染色体和费城染色体上BCR区域的研究。gydF4y2Baj . MicroscgydF4y2Ba.gydF4y2Ba199gydF4y2Ba, 96-105(2000)。gydF4y2Ba
Cremer, M.等人。多色三维荧光原位杂交成像间期染色体。gydF4y2Ba方法分子生物学gydF4y2Ba.gydF4y2Ba463gydF4y2Ba, 205-239(2008)。gydF4y2Ba
张震,雷亚金,格林,J. B. Lavis, L. D. & Tjian, R.亚秒RNA检测的单分子转录周期跟踪。gydF4y2BaeLifegydF4y2Ba3.gydF4y2Ba, e01775(2014)。gydF4y2Ba
陈,B.等。通过优化的CRISPR/Cas系统对活人类细胞中的基因组位点进行动态成像。gydF4y2Ba细胞gydF4y2Ba155gydF4y2Ba, 1479-1491(2013)。gydF4y2Ba
尼尔森,M.等。挂锁探针:用于局部DNA检测的环状寡核苷酸。gydF4y2Ba科学gydF4y2Ba265gydF4y2Ba, 2085-2088(1994)。gydF4y2Ba
鲁哈尼法德,s.h.等人。ClampFISH利用点击化学放大技术检测单个核酸分子。gydF4y2BaNat。gydF4y2Ba.(2018)。gydF4y2Bahttps://doi.org/10.1038/nbt.4286gydF4y2Ba
Edelstein, A., Amodaj, N., Hoover, K., Vale, R. & Stuurman, N.使用μManager的显微镜计算机控制。gydF4y2Ba咕咕叫。Protoc。摩尔。杂志gydF4y2Ba.gydF4y2BaCh。14gydF4y2Ba, Unit14.20(2010)。gydF4y2Ba
刘,S.-L。et al。快速高精度定位三维单粒子跟踪。gydF4y2Ba科学。代表gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, 2462(2013)。gydF4y2Ba
通过计算径向对称中心,快速,准确的粒子跟踪。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 724-726(2012)。gydF4y2Ba
杜兰德,n.c.等人。Juicer提供了一键式系统,用于分析循环分辨率Hi-C实验。gydF4y2Ba细胞系统gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, 95-98(2016)。gydF4y2Ba
奈特,P. A. &鲁伊斯,D.矩阵平衡的快速算法。gydF4y2BaIMA J.数字。分析的gydF4y2Ba.gydF4y2Ba33gydF4y2Ba, 1029-1047(2013)。gydF4y2Ba
斯坦尼特,R.等人。细胞周期进程中姐妹染色单体分解的动态。gydF4y2BaJ.细胞生物学gydF4y2Ba.gydF4y2Ba217gydF4y2Ba, 1985-2004(2018)。gydF4y2Ba
McInnes, L., Healy, J., Saul, N. & Großberger, L. UMAP:均匀流形近似和投影。gydF4y2BaJ.开源软件gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, 861(2018)。gydF4y2Ba
Nmezi, B.等人。A-型和b型层蛋白的同心结构预示着它们在核层结构的空间组织和稳定性中所起的不同作用。gydF4y2Ba国家科学院学报美国gydF4y2Ba116gydF4y2Ba, 4307-4315(2019)。gydF4y2Ba
Suzuki, H., Kurihara, Y., Kanehisa, T. & Moriwaki, K.野生小鼠染色体上银染色核仁组织区分布的变化,gydF4y2Ba亩骶gydF4y2Ba.gydF4y2Ba摩尔。杂志。另一个星球gydF4y2Ba.gydF4y2Ba7gydF4y2Ba, 271-282(1990)。gydF4y2Ba
栗原,Y., Suh, D. S., Suzuki, H. & Moriwaki, K.实验室小鼠品系Ag-NORs的染色体位置和核糖体DNA簇。gydF4y2BaMamm。基因组gydF4y2Ba5gydF4y2Ba, 225-228(1994)。gydF4y2Ba
强金,D. E., Groudine, M. & Politz, J. C. R.核仁系带介导IgH和Myc位点的配对。gydF4y2Ba核gydF4y2Ba5gydF4y2Ba, 474-481(2014)。gydF4y2Ba
Jonkers, I., Kwak, H. & Lis, J. T. Pol II伸长的全基因组动力学及其与启动子近端暂停、染色质和外显子的相互作用。gydF4y2BaeLifegydF4y2Ba3.gydF4y2Ba, e02407(2014)。gydF4y2Ba
Dileep, V. & Gilbert, D. M.测量哺乳动物复制时间随机变异的单细胞复制分析。gydF4y2BaNat。CommungydF4y2Ba.gydF4y2Ba9gydF4y2Ba, 427(2018)。gydF4y2Ba
Bacher, R.等人。SCnorm:单细胞RNA-seq数据的稳健归一化。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 584-586(2017)。gydF4y2Ba
Vallejos, C. A., Risso, D., Scialdone, A., Dudoit, S. & Marioni, J. C.规范化单细胞RNA测序数据:挑战与机遇。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 565-571(2017)。gydF4y2Ba
Hafemeister, C. & Satija, R.使用正则化负二项回归的单细胞RNA-seq数据归一化和方差稳定。gydF4y2Ba基因组医学杂志gydF4y2Ba.gydF4y2Ba20.gydF4y2Ba, 296(2019)。gydF4y2Ba
特拉格,V. A.沃特曼,L. &范埃克,N. J.从鲁汶到莱顿:保证良好连接的社区。gydF4y2Ba科学。代表gydF4y2Ba.gydF4y2Ba9gydF4y2Ba, 5233(2019)。gydF4y2Ba
斯图尔特,T.等人。单细胞数据的全面集成。gydF4y2Ba细胞gydF4y2Ba177gydF4y2Ba, 1888-1902(2019)。gydF4y2Ba
Serrano, L.等。肿瘤抑制因子SirT2通过调节H4K20甲基化的有丝分裂沉积来调节细胞周期进程和基因组稳定性。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 639-653(2013)。gydF4y2Ba
哈斯蒂,T. & Stuetzle, W.主曲线。gydF4y2Baj。统计协会。gydF4y2Ba.gydF4y2Ba84gydF4y2Ba, 502-516(1989)。gydF4y2Ba
Haghverdi, L., Büttner, M., Wolf, F. A., Buettner, F. & Theis, F. J.扩散伪时间稳健重建谱系分支。gydF4y2BaNat方法。gydF4y2Ba13gydF4y2Ba, 845-848(2016)。gydF4y2Ba
van Dijk, D.等。利用数据扩散从单细胞数据中恢复基因相互作用。gydF4y2Ba细胞gydF4y2Ba174gydF4y2Ba, 716-729(2018)。gydF4y2Ba
命运:R中大规模单细胞数据的扩散图。gydF4y2Ba生物信息学gydF4y2Ba32gydF4y2Ba, 1241-1243(2016)。gydF4y2Ba
香农等人。细胞景观:生物分子相互作用网络集成模型的软件环境。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba13gydF4y2Ba, 2498-2504(2003)。gydF4y2Ba
x染色体失活:重复假设。gydF4y2BaCytogenet。细胞麝猫gydF4y2Ba.gydF4y2Ba80gydF4y2Ba, 133-137(1998)。gydF4y2Ba
Meuleman, W.等人。本构核层-基因组相互作用高度保守,与富含A/ t的序列相关。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba23gydF4y2Ba, 270-280(2013)。gydF4y2Ba
确认gydF4y2Ba
我们感谢I. Strazhnik在数字方面的帮助;A. Anderson请他帮忙写手稿;C. Karp定制流池;h - j。Ahn为抗体偶联的早期阶段;B. Bonev的Hi-C数据。本项目由NIH 4DN DA047732和补充,以及保罗·g·艾伦前沿基金会发现中心资助。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
Y.T.和L.C.构思了这个想法并设计了实验。Y.T.在J.T.和c.h.l.e的帮助下设计了探针。Y.T.和J.Y.准备并验证了所有的实验材料。Y.T.在j.y.t.的帮助下完成了所有的实验,N.P.在J.W.和S. Shah的帮助下进行了图像分析。y.t., S.Z.和L.C.与N.O.和s.o Suo分析了数据。l.c., M.G.和g.c.y监督了分析过程。Y.T.和L.C.根据c.h.l.e.的意见撰写了手稿,g.c.y L.C.监督了项目的所有方面。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
L.C.是Spatial Genomics Inc.的联合创始人。gydF4y2Ba
额外的信息gydF4y2Ba
同行评审信息gydF4y2Ba自然gydF4y2Ba感谢Peter Fraser和其他匿名审稿人对这项工作的同行评审所做的贡献。同行评审报告是可用的。gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
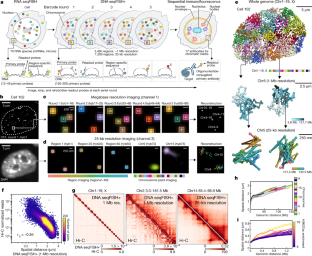
扩展数据图1包含DNA seqFISH+、RNA和内含子seqFISH和多重免疫荧光的集成空间基因组学方法的详细示意图。gydF4y2Ba
一个gydF4y2Ba,实验过程流程图。样品用多聚甲醛固定,然后用寡核苷酸偶联一抗孵育,用多聚甲醛和BS(PEG)5和RNA seqFISH固定。然后制备DNA seqFISH+样品。这种优化的方案确保了DNA seqFISH+数据与RNA seqFISH数据和在体素水平上的多重免疫荧光数据之间的良好对齐(扩展数据图。gydF4y2Ba2gydF4y2Ba).右下方的漫画显示了RNA FISH和DNA seqFISH+的成像程序,使用初级探针和寡核苷酸偶联一抗的顺序免疫荧光。gydF4y2BabgydF4y2Ba, 1mb分辨率数据集的DNA seqFISH+示意图。5轮条形码允许在每个荧光通道中检测2048个条形码,并进行2轮掉线误差校正。两个荧光通道用于覆盖总共2460个位点,在基因组中间隔约1mb。在每一轮条形码中,进行16轮杂交,生成16个伪色。在每个伪色通道中检测到的DNA点被三维拟合以确定它们的超分辨质心位置,并在所有16种伪色上编译以生成超分辨定位图像。通过5轮条形码(共80轮序列杂交),解码出所有DNA位点的身份。每个DNA位点应该在每个条形码轮中以单一伪色出现一次。条码表(gydF4y2Ba补充表2gydF4y2Ba)显示在右侧。DNA seqFISH+探针包含全部5轮条形码读出序列。对于给定的条形码轮,每个序列都有16个序列可供选择,对应于其中一种伪颜色。对于每个基因,80个杂交中有5个将导致杂交事件和荧光读出探针结合在主DNA杂交探针上。为了在所有80轮杂交中保存染色体上的DNA主探针,主探针被锁住gydF4y2Ba62gydF4y2Ba,gydF4y2Ba63gydF4y2Ba在初始杂交后的引物结合位点通过T4 DNA连接酶转移到染色体上(gydF4y2Ba方法gydF4y2Ba).gydF4y2BacgydF4y2Ba,条形码方案为25kb分辨率DNA seqFISH+。在60轮杂交中依次读出60个相邻的25kb区域并成像。这是在20条染色体上并行进行的。换句话说,每一轮杂交在不同的染色体上成像20个不同的位点。进行额外的20轮杂交,一次标记每一条染色体,以便在前60轮分别为每个成像位点分配染色体身份。1mb分辨率数据采集于643 nm(通道1)和561 nm(通道2)通道gydF4y2BabgydF4y2Ba25kb分辨率数据采集于488 nm通道(通道3)gydF4y2BacgydF4y2Ba.gydF4y2Ba
图2 DNA seqFISH+的优化和验证。gydF4y2Ba
一个gydF4y2Ba主探针的连接和后固定可以防止它们在读出探针剥离步骤中解离,端粒DNA FISH验证了这一点。将55%甲酰胺洗涤缓冲液(WB)在37°C添加到细胞中,分别锁住和不锁住主探针16小时gydF4y2Ba62gydF4y2Ba,gydF4y2Ba63gydF4y2Ba到染色体DNA上探针保留在结扎的样本中,而不保留在未结扎的样本中。请注意,在seqFISH例程中,55%的WB在室温下的每个剥离步骤中使用2分钟,该条件不如本文使用的条件严格。gydF4y2BabgydF4y2Ba,量化苛刻洗入后的信号保留gydF4y2Ba一个gydF4y2Ba,在多种条件下使用端粒DNA FISH。从单个核到单个核的总强度gydF4y2BazgydF4y2Ba-切片进行了对比。DNA seqFISH+实验采用结扎后固定的条件。来自两次独立测量的细胞数被写在图中。对于盒形图gydF4y2BabgydF4y2Ba而且gydF4y2BaggydF4y2Ba,方框中线标记中值,方框的上下限标记四分位范围,胡须延伸到1.5倍四分位范围内最远的数据点,灰点标记异常值。gydF4y2BacgydF4y2Ba在DNA seqFISH+实验中,Primary探针在经过81轮以上的杂交后仍然被结合,特异性信号恢复。DNA seqFISH+的初始hyb0用hyb80读出探针进行比较。使用单一主探针靶向基因组重复区域的基准标记物也被初步成像,并包括在所有80个成像轮中进行校准。gydF4y2BadgydF4y2Ba, DNA seqFISH+实验中80轮杂交的基准标记强度的量化,相对于hyb0基准标记的强度。基准标记(gydF4y2BangydF4y2BaDNA seqFISH+实验中446个细胞中= 506- 1117个点/杂交轮)用于定量。阴影区域代表平均值(中心)与s.d。gydF4y2BaegydF4y2Ba, DNA seqFISH+实验中基准标记在hyb 1到80之间的定位误差,gydF4y2BangydF4y2Ba= 71,981个对齐点gydF4y2BaxgydF4y2Ba而且gydF4y2BaygydF4y2Ba,gydF4y2BangydF4y2Ba= 87,879个对齐点gydF4y2BazgydF4y2BaDNA seqFISH+实验中的446个细胞。为gydF4y2BaxgydF4y2Ba而且gydF4y2BaygydF4y2Ba对线,我们过滤掉在每次杂交时距离平均位移超过2× s.d.的对齐点,并计算新的对线。gydF4y2BafgydF4y2Ba,通过双固定程序保存核结构。加热前后核散斑(SF3A66)共定位良好(右图黄色)。gydF4y2BaggydF4y2Ba,核内SF3A66免疫荧光信号保留的定量(左)和定位精度(右)通过核内像素强度与单个像素的Pearson相关性测量gydF4y2BazgydF4y2Ba- hyb0 (pre-DNA seqFISH+ steps)图像和hyb40 (pre-DNA seqFISH+ steps)或hyb130 (post-DNA seqFISH+ steps)之间的section。gydF4y2BangydF4y2Ba两个DNA seqFISH+生物复制的中心视野= 326个细胞gydF4y2BaggydF4y2Ba- - - - - -gydF4y2BakgydF4y2Ba.gydF4y2BahgydF4y2Ba,每个单元在通道1和通道2中对靶和脱靶条形码的频率。平均每个细胞检测到3,636.0±1,052.6个(中位数±s.d)靶上条形码和14.0±7.4个脱靶条形码(gydF4y2BangydF4y2Ba=两个生物重复的中心视野中的326个细胞)。gydF4y2Ba我gydF4y2Ba、个别准靶及脱靶条码的平均频率(gydF4y2BangydF4y2Ba= 4096个条形码在通道1和2),证明了DNA seqFISH+的准确性。gydF4y2BajgydF4y2Ba,单个细胞中每个荧光通道中检测到的点总数。通道1和2包含1mb的数据,通道3包含25kb的数据。gydF4y2BakgydF4y2Ba在所有20条染色体中,每个细胞的每个位点检测到的点的平均数量。注意,每个细胞2点并不是100%的检测效率,因为一些细胞处于细胞周期的G2阶段(总共4个等位基因)。与其他常染色体(1.57±0.27)相比,X染色体每个位点检测到的点数(0.84±0.21(中位数±s.d))减少了一半,因为E14小鼠ES细胞是雄性二倍体细胞系(gydF4y2Ba方法gydF4y2Ba).gydF4y2BalgydF4y2Ba,在DNA seqFISH+实验的两个生物重复之间,搜索半径为500 nm (1 mb数据)和150 nm (25 kb数据)的位点对的概率Pearson相关。计算1-Mb的所有唯一染色体内位点对(gydF4y2BangydF4y2Ba= 2,460个位点)及25kb数据(gydF4y2BangydF4y2Ba= 1200个位点)gydF4y2BangydF4y2Ba分别为201、245个细胞,用于生物重复1和2。gydF4y2Ba米gydF4y2Ba,与中使用的500纳米搜索半径(1 mb数据)和150纳米搜索半径(25 kb数据)相比,位点对之间的接近概率作为搜索半径的函数的PCCgydF4y2BalgydF4y2Ba.gydF4y2BangydF4y2Ba=来自两个DNA seqFISH+生物重复的446个细胞。gydF4y2Ba
扩展数据图3 DNA seqFISH+的附加验证。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba, DNA seqFISH+在100 nm-2 μm搜索半径内对位点的概率与Hi-C频率之间的Spearman相关性gydF4y2Ba23gydF4y2Ba在小鼠胚胎干细胞中使用一定的容器大小。计算1-Mb的所有唯一染色体内位点对(gydF4y2BangydF4y2Ba= 2340个常染色体位点)和25kb数据(gydF4y2BangydF4y2Ba=每染色体60个位点),并在bin内重叠区域gydF4y2Ba一个gydF4y2Ba均被排除在此分析之外。在1.5 Mb的染色体区域,分辨率为25 kbgydF4y2BabgydF4y2Ba,中位数Hi-C读数因目标1.5 Mb区域而异,范围为0.9至203.2。我们使用5个Hi-C读数大于40 / 25kb bin的常染色体区域进行比较。gydF4y2BacgydF4y2Ba, DNA seqFISH+ (1 mb分辨率数据)中常染色体内染色体位点对在500 nm搜索半径内的概率与Hi-C频率的比较gydF4y2Ba23gydF4y2Ba小鼠胚胎干细胞的数据。斯皮尔曼相关系数为0.89,由gydF4y2BangydF4y2Ba= 84,707个独特的染色体内成对组合。Hi-C数据与1mb数据进行分箱,1mb以内的重叠区域排除。gydF4y2BadgydF4y2Ba, DNA seqFISH+ (1 mb分辨率数据)和SPRITE频率在500 nm搜索半径内对常染色体染色体内位点对的概率比较gydF4y2Ba7gydF4y2Ba在小鼠胚胎干细胞中。Spearman相关系数为0.83。在Hi-C分析中采用了相同的分类和滤波方法gydF4y2BacgydF4y2Ba.gydF4y2BaegydF4y2Ba,通过DNA seqFISH+ (25 kb分辨率数据)和Hi-C对所选常染色体中位点对在150 nm搜索半径内的概率进行比较gydF4y2Ba23gydF4y2Ba在小鼠胚胎干细胞中。斯皮尔曼相关系数从0.82到0.94计算gydF4y2BangydF4y2Ba= 948-1,776个唯一的成对组合,使用相同的选择和过滤条件gydF4y2BabgydF4y2Ba.gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2Ba,中1mb分辨率数据对位点空间距离中值的关系gydF4y2BafgydF4y2Ba和25kb分辨率的数据gydF4y2BaggydF4y2BaDNA seqFISH+和Hi-C频率。红线是带有拟合参数的幂律拟合gydF4y2Ba年代gydF4y2Ba用斯皮尔曼相关系数R表示。gydF4y2BahgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,热图显示搜索半径为500nm in内对位点的概率gydF4y2BahgydF4y2Ba150纳米gydF4y2Ba我gydF4y2Ba(右上三角形),以及每条染色体中1 Mb分辨率数据的位点对(左下三角形)的中位数空间距离gydF4y2BahgydF4y2Ba和25kb分辨率的数据gydF4y2Ba我gydF4y2BaDNA seqFISH+。gydF4y2BangydF4y2Ba= 446个细胞,来自两个生物重复的DNA seqFISH+数据gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2Ba我gydF4y2Ba.gydF4y2Ba
图4 DNA seqFISH+对染色体的单细胞组织和物理缩放。gydF4y2Ba
一个gydF4y2Ba,小鼠胚胎干细胞的DAPI染色图像(上)和对应的带有不同颜色标记的单个染色体的细胞核的3D图像(下)。gydF4y2BabgydF4y2Ba,单个染色体的3D图像,根据染色体坐标着色(浅到深颜色)。染色体来自于细胞gydF4y2Ba一个gydF4y2Ba.这些图像具有代表性gydF4y2BangydF4y2Ba= 446个细胞的DNA seqFISH+。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba中位数空间距离作为基因组距离的函数的缩放,用于20条染色体,分辨率为1 mbgydF4y2BacgydF4y2Ba和25kb分辨率的数据gydF4y2BadgydF4y2Ba.灰色点表示给定位点对的中位数距离。蓝色虚线为各基因组距离仓的空间距离中值,红线为幂律函数与图中拟合参数的拟合。gydF4y2BangydF4y2Ba= 446个细胞。gydF4y2BaegydF4y2Ba, 1 mb DNA seqFISH+数据中所有位点之间的完整空间接近图,搜索半径为1 μm(左下三角形面板)。放大后的chr6和chr7染色体图谱(右上)显示,来自不同染色体的非重复区域靠近熵点周围的重复区域更可能在空间上彼此接近。色条以对数刻度表示。gydF4y2BafgydF4y2Ba, 20条染色体的平均空间接近图,只考虑每条染色体的前5 Mb非重复区域,搜索半径为1 μm。gydF4y2BaggydF4y2Ba,中染色体对间空间接近性变异系数(CV)的分布gydF4y2BafgydF4y2Ba.gydF4y2BahgydF4y2Ba,空间接近地图的单细胞版本gydF4y2BafgydF4y2Ba在染色体近端5 Mb非重复区域之间的空间接近性上显示出异质性。gydF4y2Ba我gydF4y2Ba单核图像显示,在单个核中,只有一个染色体子集的近端5 Mb非重复区域出现在富含dapi的近核周围异染色质区域附近。这些图像具有代表性gydF4y2BangydF4y2Ba= 446个细胞,从2个生物重复中定量分析gydF4y2BaegydF4y2Ba- - - - - -gydF4y2BahgydF4y2Ba.gydF4y2Ba
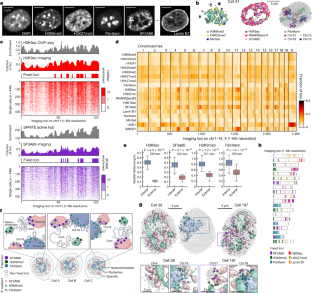
扩展数据图5序列免疫荧光和重复元件DNA FISH的可视化和验证。gydF4y2Ba
一个gydF4y2Ba与DAPI同时成像17个抗体和4个重复元件,包括基因贫乏的长穿插核元件(LINE1)、基因丰富的短穿插核元件(sinineb1)、着丝粒的MinSat、端粒。单个细胞具有不同的免疫荧光染色模式。注意,单元之间的DAPI模式并不相同。同样地,与富含dapi的周围熵点异染色质区域共定位的标记在细胞之间是不同的,甚至在单个细胞中不同的周围熵点区域之间也是不同的。gydF4y2BabgydF4y2Ba,代表性的H3K9ac图像和检测H3K9ac小球外部体素的边缘变换图像(gydF4y2Ba方法gydF4y2Ba).gydF4y2BacgydF4y2Ba,代表H3K9ac图像从单个gydF4y2BazgydF4y2Ba-截面或最大强度gydF4y2BazgydF4y2Ba-投影强度gydF4y2BaZgydF4y2Ba-得分阈值大于2。对具有强度的像素进行三维可视化(右)gydF4y2BaZgydF4y2Ba-得分在2分以上(gydF4y2Ba方法gydF4y2Ba).gydF4y2BadgydF4y2Ba,为所述像素点附加免疫荧光标记的单细胞3D图像gydF4y2BaZgydF4y2Ba-得分2分以上。异染色质成分(H3K9me3、DAPI和MinSat)聚在一起,RNAPIISer5-P、活性标记(H3K9ac、H3K27ac)、sinineb1和核斑点(SF3A66)物理位置近端。DNA FISH中LINE1的高强度像素主要定位于富含LINE1的X染色体gydF4y2Ba88gydF4y2Ba.gydF4y2BaegydF4y2Ba,从不同免疫荧光标记的内部和外部体素的距离产生的1 Mb分辨率的所有2460个位点的染色质谱的相关性(gydF4y2BangydF4y2Ba= 446个细胞)。gydF4y2BafgydF4y2Ba,在1 mb分辨率下,每个位点到内部体素与外部体素的距离的散点图,比2460个位点的平均值高出2倍s.d. (gydF4y2BangydF4y2Ba= 446个细胞)。PCCs显示。gydF4y2BaggydF4y2Ba,通过DNA seqFISH+在25kb分辨率下显示300 nm内免疫荧光标记和重复元件的部分位点的热图(gydF4y2BangydF4y2Ba= 1200个基因座和446个细胞)。gydF4y2Ba
扩展数据图6固定位点和染色质谱的额外可视化和验证。gydF4y2Ba
一个gydF4y2Ba, DNA seqFISH+和免疫荧光与其他方法比较染色质谱的相关矩阵gydF4y2Ba7gydF4y2Ba,gydF4y2Ba24gydF4y2Ba,gydF4y2Ba35gydF4y2Ba.使用1 mb DNA seqFISH+数据,参考数据与1 mb数据进行分组。染色质谱计算为2460个位点在距离免疫荧光标记外部300 nm范围内的位点比例(gydF4y2BangydF4y2Ba= 446个细胞)。gydF4y2BabgydF4y2Ba,各标记比较的二维密度图,如图所示gydF4y2Ba一个gydF4y2Ba.gydF4y2BangydF4y2Ba= 2460个位点。gydF4y2BacgydF4y2Ba,不同阈值下距离lamin B1外部300 nm范围内位点比例的比较(gydF4y2BaZgydF4y2Ba-得分大于2或3),或由核像素的凸包计算出的核外围(gydF4y2Ba方法gydF4y2Ba),显示在不同量化标准下的剖面具有良好的一致性(gydF4y2BangydF4y2Ba= 2460个位点来自446个细胞)。gydF4y2BadgydF4y2Ba,通过先前DamID研究中指定的细胞型不变本构层相关结构域(cLADs)、细胞型依赖兼性LADs (fLADs)和本构间LADs (ciLADs)等位点对层蛋白B1富集的验证gydF4y2Ba35gydF4y2Ba,gydF4y2Ba89gydF4y2Ba.与来自cLADs的基因座相比,被归类为cLADs和fLADs的基因座显示了与lamin B1邻近位点的富集,这代表了我们的测量结果的良好一致性(gydF4y2BangydF4y2Ba在ddamid研究中,cLADs、fLADs、ciLADs分别为351、405和1023个位点,类别平均为446个细胞)。N是基因座的数目。对于盒形图gydF4y2BadgydF4y2Ba而且gydF4y2BaggydF4y2Ba,方框中线标记中值,方框的上下限标记四分位范围,胡须延伸到1.5倍四分位范围内最远的数据点,灰点标记异常值。gydF4y2BaegydF4y2Ba中不同标准的Lamin B1染色质谱的额外可视化gydF4y2BacgydF4y2Ba(gydF4y2BangydF4y2Ba= 446个细胞)gydF4y2Ba35gydF4y2Ba.为了只考虑核外周的Lamin B1染色,我们计算了细胞核凸包附近的DNA位点与Lamin B1信号之间的距离以及不同强度阈值。gydF4y2BafgydF4y2Ba,与ChIP-seq比较的单细胞染色质谱的附加示例gydF4y2Ba24gydF4y2BaH3K27me3(上)和雪碧gydF4y2Ba7gydF4y2Ba.计算并以与图相同的方式显示轮廓。gydF4y2Ba2摄氏度gydF4y2Ba.gydF4y2BangydF4y2Ba= 446个细胞。gydF4y2BaggydF4y2Ba,单细胞中与基于染色质谱定义的固定位点的免疫荧光标记的外部相关的位点的比例(gydF4y2BangydF4y2Ba= 446个细胞)。注意,不同的免疫荧光标记对固定位点的调用有不同的阈值。因此,一些免疫荧光标记的固定位点与单细胞中的免疫荧光标记更一致。gydF4y2BahgydF4y2Ba,免疫荧光标记及其相关固定位点的附加3D图像。在每个细胞中,6个免疫荧光标记(每面板2个)显示为视觉清晰度。gydF4y2Ba我gydF4y2Ba中所示的3个细胞中突出显示了5条染色体gydF4y2BahgydF4y2Ba.在相应的图像可视化中为每个染色体显示一对免疫荧光标记的固定位点。固定的位点用彩色点表示,染色体上剩余的位点用灰点表示。使用相同的颜色代码gydF4y2BahgydF4y2Ba.gydF4y2Ba
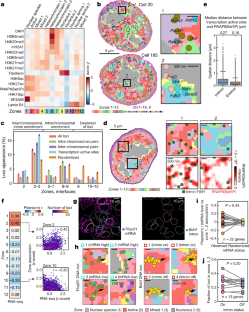
图7与染色质标记相关的群体水平和单细胞水平染色体组织的比较。gydF4y2Ba
一个gydF4y2Ba,个体位点的整体平均免疫荧光空间接近谱的聚类。gydF4y2BangydF4y2Ba= 2,460个位点(gydF4y2BangydF4y2Ba每个聚类分别为805、278、877,500个位点)。gydF4y2BabgydF4y2Ba在单个细胞中,与每个簇相关的位点被映射到它们的空间位置上。请注意,DNA位点的聚类定义是从人群平均数据中获得的,这些聚类分配的位点分布可能不一定反映单细胞中的免疫荧光标记物定位。gydF4y2BacgydF4y2Ba,每个聚类中基因座的免疫荧光标记箱状图。簇1富含H3K9me3、mH2A1、DAPI等抑制性标记。簇2富含与纤原蛋白的相互作用。簇3富集了RNAPII(Ser5P)、H3K27ac和SF3A66(核散斑标记)等活性标记。簇4富集于层蛋白B1。对于盒形图gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,gydF4y2BahgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,方框中线标记中值,方框的上下限标记四分位范围,胡须延伸到1.5倍四分位范围内最远的数据点,灰点标记异常值。gydF4y2BadgydF4y2Ba,特定簇对在单个细胞内1 μm搜索半径内的位点概率。中的集群定义遵循gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba.随机数据是通过打乱细胞中单个位点的簇身份,同时保持细胞内每个簇内的位点总数相同来生成的。每个单元观察数据和随机数据的概率显示为箱形图。gydF4y2BaegydF4y2Ba,在给定的搜索半径内找到具有聚类分配的位点对的概率,作为搜索半径的函数。误差条表示20次引导试验的标准误差。gydF4y2BafgydF4y2Ba, A/B隔间定义的映射gydF4y2Ba23gydF4y2Ba到gydF4y2BatgydF4y2Ba-基于整体平均位点-免疫荧光标记空间接近图的sne图。请注意,没有被分配到某个区室的区域被排除在分析之外。(gydF4y2BangydF4y2BaA区和B区分别为1188个和960个位点)。gydF4y2BaggydF4y2Ba,单个细胞的重建,将A或B室的位点映射到它们的空间位置上。观察与随机数据的2个细胞gydF4y2BabgydF4y2Ba.gydF4y2BahgydF4y2Ba, A或B区室位点的免疫荧光标记箱形图。gydF4y2Ba我gydF4y2Ba, A/B区室中位点在单个细胞中1 μm搜索半径内的概率与gydF4y2BadgydF4y2Ba.gydF4y2BajgydF4y2Ba,在给定搜索半径内找到具有A/B分配的位点对的概率,作为空间接近性搜索半径的函数,类似于gydF4y2BaegydF4y2Ba.gydF4y2BangydF4y2Ba= 446个细胞,来自两个生物重复gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BajgydF4y2Ba.gydF4y2Ba
扩展数据图8核带和界面的进一步表征。gydF4y2Ba
一个gydF4y2Ba,基于像素的组合染色质分析工作流。具有15个染色质标记的个体体素通过分层聚类进行聚类,并通过非线性降维技术(UMAP)进行可视化表示。gydF4y2Ba70gydF4y2Ba.来自单个集群(区域)的体素被映射回单个核,并覆盖DNA seqFISH+点。gydF4y2BabgydF4y2Ba, UMAP表示为取自201个单元的44,000像素,标记为12个区域。UMAP投影用于视觉清晰度。gydF4y2BacgydF4y2Ba,基于与区域和界面关联的DNA位点的Pearson相关矩阵,如图所示gydF4y2BafgydF4y2Ba(gydF4y2BangydF4y2Ba= 2460个位点)。出现在区域1的位点也更有可能出现在区域2以及界面1/2。gydF4y2BadgydF4y2Ba,比较DNA seqFISH+处理和未处理的区域外观,测量结果总体一致。20个引导试验的平均值显示在与标准误差相对应的误差条上。gydF4y2BaegydF4y2Ba,作为免疫荧光标记下采样函数的区域分配。在每个下样本量中选择20个免疫荧光标记的随机子集。曲线的中心反映平均值,宽度反映在每个下样本量下正确区域分配的标准偏差(gydF4y2Ba方法gydF4y2Ba).gydF4y2BafgydF4y2Ba,重建区域和DNA位点在额外的细胞。gydF4y2BaggydF4y2Ba,细胞31中不同区域的重建gydF4y2BazgydF4y2Ba飞机。gydF4y2BahgydF4y2Ba、条带重建、1000个基因内含子点、RNAPIISer5-P染色(去除背景)和RNAPIISer5-P染色边缘。gydF4y2Ba我gydF4y2Ba, 1 mb数据的DNA位点、核区和界面之间关联概率的热图。区域和界面根据与DNA位点关联的总体概率进行排序。右边的面板显示周围的基因座gydF4y2BaPou5f1gydF4y2Ba如图所示。gydF4y2Ba3 bgydF4y2Ba(图1).单个细胞中的每个位点被分配到一个区域或界面。热图中显示的分布反映了每个位点区域关联的单细胞变异性。例如,gydF4y2BaEhmt2gydF4y2Ba而且gydF4y2BaPou5f1gydF4y2Ba位点主要与活性区2和接口1/2和2/3有关gydF4y2BaOpn5gydF4y2Ba而且gydF4y2BaDazlgydF4y2Ba基因座在许多区域分布更为均匀。gydF4y2BajgydF4y2Ba, 25 kb数据的DNA位点、核区和界面之间关联概率的热图。相同Mb区域内的位点具有相似的核区和界面关联概率。gydF4y2BakgydF4y2Ba,单细胞中DNA位点与区或界面的结合频率,计算所有位点、染色体内和染色体间对的位点、内含子FISH测量的TAS以及随机位点(随机对照)。20个引导试验的平均值显示在与标准误差相对应的误差条上。gydF4y2BalgydF4y2Ba,条带关联与基因表达水平的相关性(RNA-seq)gydF4y2Ba49gydF4y2Ba,位点上RNA聚合酶的密度(GRO-seq)gydF4y2Ba75gydF4y2Ba早期复制域(Repli-seq)gydF4y2Ba76gydF4y2Ba所有1mb分辨率的基因座(gydF4y2BangydF4y2Ba= 2460个位点)。gydF4y2Ba米gydF4y2Ba,各免疫荧光标记物固定位点的表达水平gydF4y2BangydF4y2Ba= 446个细胞。群体水平表达来自大量RNA-seq研究gydF4y2Ba49gydF4y2Ba并集成为1mb的区域。对于盒状图,盒中的中线标记中位数,盒的上下限标记四分位范围,胡须延伸到1.5倍四分位范围内最远的数据点,灰点标记异常值。gydF4y2BangydF4y2Ba,在单个细胞中处于单个mRNA活性区域的给定位点300 nm内的mRNA水平和体素分数的相关性。20个自举试验的平均值显示与每个mRNA的标准误差对应的误差条。随机样本对应于每个细胞的mRNA和区域分配值的打乱。gydF4y2BaogydF4y2Ba,比较DNA基因座300 nm内处于活跃区(1区和2区)的体素比例,对于有活跃内含子信号的基因座(ON)与单个内含子没有内含子信号的基因座(OFF)。20个引导试验的平均值显示在与标准误差相对应的误差条上。每个内含子RNA。gydF4y2BangydF4y2Ba= 201和172细胞的DNA seqFISH+和内含子FISH测量gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BalgydF4y2Ba,gydF4y2BangydF4y2Ba,gydF4y2BaogydF4y2Ba,分别。gydF4y2Ba
图9单细胞中转录和染色质状态的异质性及其相互关系。gydF4y2Ba
一个gydF4y2Ba, RNA seqFISH和bulk RNA-seq的平均mRNA计数Pearson相关性。RNA seqFISH的误差条表示两次测量的平均值的标准误差(gydF4y2BangydF4y2Ba= 151和175单元格从中心视野)。gydF4y2BabgydF4y2Ba,基于scRNA-seq识别的两种不同细胞簇中的单个细胞的UMAP表示gydF4y2Ba42gydF4y2Ba并映射到RNA seqFISH数据(集群a表示具有更多多能状态的细胞,集群b表示分化路径上的细胞)(左),以及在不同的数据集中(右)(gydF4y2BangydF4y2BaRNA seqFISH和scRNA-seq = 326和250个细胞gydF4y2Ba42gydF4y2Ba数据集,分别)。gydF4y2BacgydF4y2Ba,箱形图显示scRNA-seq和seqFISH数据集中差异表达基因的良好一致性。gydF4y2BaPgydF4y2Ba值来自双侧Wilcoxon 's秩和检验,在群集a和b中的细胞(gydF4y2BangydF4y2Ba= 298和209细胞在集群a和gydF4y2BangydF4y2Bab簇中RNA seqFISH和scRNA-seq分别为28和41个细胞gydF4y2Ba42gydF4y2Ba数据集,分别)。对于盒状图,盒中的中线标记中位数,盒的上下限标记四分位范围,胡须延伸到1.5倍四分位范围内最远的数据点,灰点标记异常值。gydF4y2BadgydF4y2Ba, UMAP表示由免疫荧光强度谱定义的细胞簇。gydF4y2BaegydF4y2Ba,具有不同免疫荧光谱的细胞簇热图,显示与细胞周期相关的免疫荧光标记和所有mRNA标记,如图所示。gydF4y2Ba4 bgydF4y2Ba.gydF4y2BafgydF4y2Ba在细胞周期进程的伪时间过程分析中,细胞周期标记物(H4K16ac, H4K20me1和H3pSer10)在细胞周期伪时间过程中表现出明显的富集,而其他标记物在细胞周期伪时间过程中不表现出特定的富集,这表明所描述的大多数免疫荧光标记物主要不受细胞周期阶段的影响。gydF4y2BaggydF4y2Ba,基于mRNA表达水平的小鼠ES细胞多能性状态的伪时间过程分析,显示与幼稚多能性相关的标记的富集,如gydF4y2BaTfcp2l1gydF4y2Ba而且gydF4y2BaNanoggydF4y2Ba与启动多能性相关的标记,如gydF4y2BaDnmt3agydF4y2Ba,gydF4y2Ba基因Lin28bgydF4y2Ba而且gydF4y2BaOtx2gydF4y2Ba以及在多能性伪时间过程中某些染色质标记的富集。gydF4y2BahgydF4y2Ba,沿细胞的多能伪时间顺序缩放标记基因表达(顶部面板)或强度(底部面板)。原始数据gydF4y2BaggydF4y2Ba与拟合曲线(gydF4y2Ba方法gydF4y2Ba).gydF4y2Ba我gydF4y2BamRNA和免疫荧光标记的网络分析表明,标记之间存在正相关和负相关。gydF4y2BajgydF4y2Ba,基于单细胞中规模化表达或强度谱的mRNA与免疫荧光标记之间的联合Pearson相关矩阵(gydF4y2BangydF4y2Ba= 41个mRNA和25个免疫荧光标记)。gydF4y2BangydF4y2Ba= 326个细胞在中心视野为RNA seqFISH和免疫荧光数据gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BajgydF4y2Ba.gydF4y2Ba
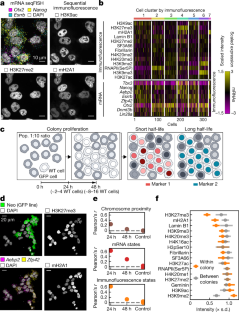
扩展数据图10菌落水平细胞状态异质性的附加分析。gydF4y2Ba
一个gydF4y2Ba在48小时克隆示踪实验中获得菌落中的mRNA和免疫荧光图像。在集落的WT细胞(GFP/Neo阴性)中H3K27me3和mH2A1总体强度相似。gydF4y2BabgydF4y2Ba,集落内(红色)和集落间(灰色)归一化mRNA水平的标准差。错误条显示了20个引导试验的标准错误。gydF4y2BaTbx3gydF4y2Ba而且gydF4y2BaNanoggydF4y2Ba在菌落内是否更加同质,与先前通过单细胞活体成像实验发现的这些基因在几代人之间的长寿命转录状态一致gydF4y2Ba41gydF4y2Ba,gydF4y2Ba49gydF4y2Ba.gydF4y2BangydF4y2Ba=来自48小时数据集的菌落内117个未标记细胞。gydF4y2BacgydF4y2Ba,菌落内(红色)和菌落间(灰色)的细胞间染色体到染色体接近图的细胞间相关性直方图。具有相似染色体结构的细胞(高相关值的红点)很可能是姐妹细胞。的gydF4y2BaygydF4y2Ba-轴为PCC,由对细胞的20 × 20染色体接近矩阵计算。gydF4y2BaPgydF4y2Ba值来自双面Wilcoxon 's秩和检验,对细胞为180、1198、966和5820(从左到右)。gydF4y2BadgydF4y2Ba, 48小时克隆示踪实验中菌落细胞间染色体邻近性的相关性。在假定的姐妹细胞之间可以看到很强的相关性,这表明粗染色体的接近性被保留了一代。彩色条表示计算的PCCgydF4y2BacgydF4y2Ba.gydF4y2BaegydF4y2Ba,来自24小时菌落的未标记细胞的染色体图像显示两组相邻细胞之间的相似性(最大gydF4y2BazgydF4y2Ba投影)。单细胞中的染色体组织在物理上接近的细胞对(可能是姐妹细胞)之间高度相关,并且大多数与菌落中的其他细胞不相关。为了清晰可见,我们展示了6条染色体。gydF4y2BargydF4y2Ba中计算的PCCgydF4y2BacgydF4y2Ba.gydF4y2Ba
补充信息gydF4y2Ba
补充表gydF4y2Ba
补充表1:DNA seqFISH+中3660个DNA位点的基因组坐标列表。gydF4y2Ba
补充表gydF4y2Ba
补充表2:三个荧光通道中3660个DNA位点的编码本。通道1和通道2中每个位点的Base 16伪色编码方案,包括用于脱靶评估的控制区域。区域和染色体为通道3中的每个位点绘制成像方案。gydF4y2Ba
补充表gydF4y2Ba
补充表3:在单细胞中同时在给定染色体上找到一对IF标记固定点的染色体配置概率。在单个细胞中,每个IF标记的内外体素300 nm范围内相关的基因座比例。从3,660个DNA位点到个体免疫荧光标记的内部和外部体素的中位数空间距离。gydF4y2Ba
补充表gydF4y2Ba
补充表4:3660个DNA位点上单个区域和界面的归一化关联频率。gydF4y2Ba
补充表gydF4y2Ba
补充表5:单细胞中与区域/界面相关的DNA位点比例列表,包括所有位点、染色体内和染色体间对位点、内含子FISH检测的转录活性位点以及随机定位的位点。用双侧Wilcoxon秩和检验从20个自举试验中计算类别对的p值。gydF4y2Ba
补充表gydF4y2Ba
补充表6:E14复制和克隆实验中的目标rna列表。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
武井,杨,云,J,郑,S。gydF4y2Baet al。gydF4y2Ba整合空间基因组学揭示单核的整体结构。gydF4y2Ba自然gydF4y2Ba590gydF4y2Ba, 344-350(2021)。https://doi.org/10.1038/s41586-020-03126-2gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-020-03126-2gydF4y2Ba
这篇文章被引用gydF4y2Ba
高效、鲁棒的π-FISH彩虹,用于多种生物分子的多重原位检测gydF4y2Ba
自然通讯gydF4y2Ba(2023)gydF4y2Ba
用于从多重DNA FISH中解析染色质结构的空间基因组校准器gydF4y2Ba
自然生物技术gydF4y2Ba(2023)gydF4y2Ba
用Micro Capture-C测定染色质结构gydF4y2Ba
自然的协议gydF4y2Ba(2023)gydF4y2Ba
哺乳动物组织的空间表观基因组-转录组联合分析gydF4y2Ba
自然gydF4y2Ba(2023)gydF4y2Ba
转录控制的空间组织gydF4y2Ba
自然评论遗传学gydF4y2Ba(2023)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
