摘要gydF4y2Ba
分子噪声是所有生物系统所固有的一种自然现象gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba.随机过程如何产生支持组织稳态的稳健结果仍不清楚。在这里,我们使用单分子RNA荧光原位杂交(smFISH)对来源于造血组织的小鼠干细胞进行测量,以测量编码转录因子的三个关键基因的转录动态:gydF4y2BaPU.1gydF4y2Ba(亦称gydF4y2BaSpi1gydF4y2Ba),gydF4y2BaGata1gydF4y2Ba而且gydF4y2BaGata2gydF4y2Ba.我们发现,在大多数造血干细胞和祖细胞中,罕见的随机转录爆发导致这些拮抗转录因子的共同表达。此外,通过将smFISH与延时显微镜和谱系分析相结合,我们发现尽管单个干细胞克隆产生的后代处于转录相关状态(类似于转录启动现象),但状态之间的潜在转变动态最好由随机和可逆模型捕获。因此,随机过程可以产生细胞行为,这些行为可能被错误地推断为由确定性动力学产生。我们提出了一个模型,其中基因表达的内在随机性促进,而不是阻碍,伴随的转录可塑性和干细胞稳健性的维持。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
RS-FISH:精确、交互式、快速、可扩展的FISH斑点检测gydF4y2Ba
自然方法gydF4y2Ba开放获取gydF4y2Ba11月17日gydF4y2Ba
多参数时序成像分析揭示巨核红细胞祖细胞克隆扩增和分化动力学gydF4y2Ba
科学报告gydF4y2Ba开放获取gydF4y2Ba2022年9月28日gydF4y2Ba
通过嵌入双稳定子系统设计多稳定系统的鲁棒方法gydF4y2Ba
系统生物学与应用gydF4y2Ba开放获取gydF4y2Ba2022年3月25日gydF4y2Ba
访问选项gydF4y2Ba
访问《自然》和其他54种《自然》杂志gydF4y2Ba
获取Nature+,我们最超值的在线订阅gydF4y2Ba
每月29.99美元gydF4y2Ba
随时取消gydF4y2Ba
订阅这本杂志gydF4y2Ba
收到51个印刷问题和在线访问gydF4y2Ba
199.00美元一年gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
租或购买这篇文章gydF4y2Ba
只要这篇文章,只要你需要它gydF4y2Ba
39.95美元gydF4y2Ba
价格可能受当地税收的影响,在结账时计算gydF4y2Ba
数据可用性gydF4y2Ba
用于生成图形的所有源数据都可在手稿文件或GitHub存储库(gydF4y2Bahttps://github.com/justincwheat/Single-Molecule-Imaging-of-Transcription-Dynamics-in-Somatic-Stem-CellsgydF4y2Ba)与这份手稿有关。进一步的信息和对资源、试剂和数据的合理要求应直接联系通讯作者。用于生成与亲缘关系分析或模拟相关的数字的数据(图gydF4y2Ba2gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,扩展数据图。gydF4y2Ba8gydF4y2Ba而且gydF4y2Ba9gydF4y2Ba),已提供单独的.mat文件作为补充数据gydF4y2Ba1gydF4y2Ba并上传到上面列出的GitHub存储库,或者在运行相关脚本时生成。所有数据均可根据合理要求从通讯作者处获得。gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
为参数估计和随机模拟编写的软件在补充数据中提供gydF4y2Ba2gydF4y2Ba(FSP。米,getKLD。米,GSSA.m)。Figs相关软件。gydF4y2Ba3.gydF4y2Ba而且gydF4y2Ba4gydF4y2Ba也可以在补充数据中找到gydF4y2Ba2gydF4y2Ba: KCA (KCA.m)的代码,生成3单元频率矩阵(ThreePtFreqs.m),测试不同的分子截止(KCA_thresholdtesting.mlx),并计算在每个状态下花费的时间(GenerateAllTrees.m)。还提供了每个殖民地的数据结构(colony [#].mat)。所有脚本和数据文件也已发布在一个公开可用的存储库中gydF4y2Bahttps://github.com/justincwheat/Single-Molecule-Imaging-of-Transcription-Dynamics-in-Somatic-Stem-CellsgydF4y2Ba.本研究中使用的其他组生成的所有软件均列在补充表中gydF4y2Ba7gydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
列夫斯基,J. M.辛格,R. H.基因表达和平均细胞的神话。gydF4y2Ba细胞生物学趋势gydF4y2Ba.gydF4y2Ba13gydF4y2Ba, 4-6(2003)。gydF4y2Ba
埃洛维茨,M. B.,莱文,a . J.,西吉亚,E. D. &斯温,P. S.单细胞中的随机基因表达。gydF4y2Ba科学gydF4y2Ba297gydF4y2Ba, 1183-1186(2002)。gydF4y2Ba
雷泽,J. M. & O 'Shea . E. K.真核生物基因表达随机性的控制。gydF4y2Ba科学gydF4y2Ba304gydF4y2Ba, 1811-1814(2004)。gydF4y2Ba
Bar-Even, A.等。蛋白质表达中的噪声与天然蛋白质丰度成比例。gydF4y2BaNat麝猫。gydF4y2Ba.gydF4y2Ba38gydF4y2Ba, 636-643(2006)。gydF4y2Ba
甘地,S. J.,曾克鲁森,D., Lionnet, T. & Singer, R. H.功能相关的组成基因的转录是不协调的。gydF4y2BaNat。结构。摩尔。杂志gydF4y2Ba.gydF4y2Ba18gydF4y2Ba, 27-34(2011)。gydF4y2Ba
哈,D. & Paulsson, J.细胞分裂时分子的随机分配。gydF4y2Ba国家科学院学报美国gydF4y2Ba108gydF4y2Ba, 15004-15009(2011)。gydF4y2Ba
Lestas, I., Vinnicombe, G. & Paulsson, J.分子波动抑制的基本限制。gydF4y2Ba自然gydF4y2Ba467gydF4y2Ba, 174-178(2010)。gydF4y2Ba
奥尔森,A.等。单细胞混合谱系状态分析导致二元细胞命运选择。gydF4y2Ba自然gydF4y2Ba537gydF4y2Ba, 698-702(2016)。gydF4y2Ba
杜思,B. K.等。种群快照预测早期造血和红细胞等级。gydF4y2Ba自然gydF4y2Ba555gydF4y2Ba, 54-60(2018)。gydF4y2Ba
Femino, A. M., Fay, F. S., Fogarty, K. & Singer, R. H.原位单个RNA转录本的可视化。gydF4y2Ba科学gydF4y2Ba280gydF4y2Ba, 585-590(1998)。gydF4y2Ba
托瑞等人。通过单分子RNA FISH指导下的单细胞RNA测序检测罕见细胞。gydF4y2Ba细胞系统gydF4y2Ba.gydF4y2Ba6gydF4y2Ba, 171 - 179。e5(2018)。gydF4y2Ba
陈凯辉,陈建荣,陈建荣,王淑娟,庄晓霞。单细胞中空间分辨的高多重RNA谱分析。gydF4y2Ba科学gydF4y2Ba348gydF4y2Ba, aaa6090(2015)。gydF4y2Ba
Tsanov, N.等人smiFISH和FISH-quant -一种具有超分辨率能力的灵活的单RNA检测方法。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba44gydF4y2Ba, e165(2016)。gydF4y2Ba
陈红梅,Pahl, H. L., Scheibe, R. J,张德娥,Tenen, d.g . Sp1转录因子在骨髓细胞中特异性结合CD11b启动子,并对骨髓特异性启动子活性至关重要。gydF4y2Ba生物。化学gydF4y2Ba.gydF4y2Ba268gydF4y2Ba, 8230-8239(1993)。gydF4y2Ba
Koschmieder, S., Rosenbauer, F., Steidl, U., Owens, B. M. & Tenen, d.g.转录因子C/EBPα和PU.1在正常造血和白血病中的作用。gydF4y2BaInt。j .内科杂志gydF4y2Ba.gydF4y2Ba81gydF4y2Ba, 368-377(2005)。gydF4y2Ba
Rekhtman, N., Radparvar, F., Evans, T. & Skoultchi, a.i .造血转录因子pu1和GATA-1的直接相互作用:红细胞功能拮抗。gydF4y2Ba基因开发gydF4y2Ba.gydF4y2Ba13gydF4y2Ba, 1398-1411(1999)。gydF4y2Ba
张,P.等。PU.1通过阻断GATA-1 DNA结合抑制GATA-1功能和红系分化。gydF4y2Ba血gydF4y2Ba96gydF4y2Ba, 2641-2648(2000)。gydF4y2Ba
卢森鲍尔,F.等人。急性髓系白血病是由一种谱系特异性转录因子PU.1的逐渐减少引起的。gydF4y2BaNat麝猫。gydF4y2Ba.gydF4y2Ba36gydF4y2Ba, 624-630(2004)。gydF4y2Ba
Steidl, U.等人。Jun家族转录因子在PU.1敲除诱导的白血病干细胞中的重要作用。gydF4y2BaNat麝猫。gydF4y2Ba.gydF4y2Ba38gydF4y2Ba, 1269-1277(2006)。gydF4y2Ba
威尔,B.等人。最小的PU.1降低诱导白血病前状态,促进急性髓系白血病的发展。gydF4y2Ba地中海Nat。gydF4y2Ba.gydF4y2Ba21gydF4y2Ba, 1172-1181(2015)。gydF4y2Ba
斯金纳,s.o.等人。跨细胞周期的单细胞转录动力学分析。gydF4y2BaeLifegydF4y2Ba5gydF4y2Ba, e12175(2016)。gydF4y2Ba
Giladi, A.等人。造血祖细胞的单细胞特征及其在稳态和紊乱造血中的运动轨迹。gydF4y2Ba细胞生物学gydF4y2Ba.gydF4y2Ba20.gydF4y2Ba, 836-846(2018)。gydF4y2Ba
保罗,F.等。髓系祖细胞的转录异质性和谱系承诺。gydF4y2Ba细胞gydF4y2Ba163gydF4y2Ba, 1663-1677(2015)。gydF4y2Ba
Nestorowa, S.等人。小鼠造血干细胞和祖细胞分化的单细胞分辨率图。gydF4y2Ba血gydF4y2Ba128gydF4y2Ba, e20-e31(2016)。gydF4y2Ba
Tabula Muris财团。20个小鼠器官的单细胞转录组学创造了Tabula Muris。gydF4y2Ba自然gydF4y2Ba562gydF4y2Ba, 367-372(2018)。gydF4y2Ba
周,S. T.等。分级抑制gydF4y2BaPU.1gydF4y2Ba/gydF4y2BaSfpi1gydF4y2BaGATA因子基因转录调控造血细胞命运。gydF4y2Ba血gydF4y2Ba114gydF4y2Ba, 983-994(2009)。gydF4y2Ba
Doré, L. C., Chlon, T. M., Brown, C. D., White, K. P. & Crispino, J. D.染色质占用分析揭示了造血过程中全基因组GATA因子切换。gydF4y2Ba血gydF4y2Ba119gydF4y2Ba, 3724-3733(2012)。gydF4y2Ba
格拉斯,J. A.等。gata -1依赖的转录抑制GATA-2通过破坏积极的自我调节和全域染色质重塑。gydF4y2Ba国家科学院学报美国gydF4y2BaOne hundred.gydF4y2Ba, 8811-8816(2003)。gydF4y2Ba
辛格,Z. S.等。胚胎干细胞的动态异质性和DNA甲基化。gydF4y2Ba摩尔。细胞gydF4y2Ba55gydF4y2Ba, 319-331(2014)。gydF4y2Ba
数值模拟耦合化学反应随机时间演化的一般方法。gydF4y2Baj .第一版。理论物理gydF4y2Ba.gydF4y2Ba22gydF4y2Ba, 403-434(1976)。gydF4y2Ba
Haghverdi, L., Büttner, M., Wolf, F. A., Buettner, F. & Theis, F. J.扩散伪时间稳健重建谱系分支。gydF4y2BaNat方法。gydF4y2Ba13gydF4y2Ba, 845-848(2016)。gydF4y2Ba
霍普,p.s.等人。早期骨髓系的选择并不是由pu1和GATA1蛋白的随机比例决定的。gydF4y2Ba自然gydF4y2Ba535gydF4y2Ba, 299-302(2016)。gydF4y2Ba
Buggenthin, F.等。基于深度学习的造血谱系选择的前瞻性识别。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 403-406(2017)。gydF4y2Ba
斯特拉瑟,M. K.等。造血谱系中的谱系标记同步性驳斥了PU.1/GATA1拨动开关范式。gydF4y2BaNat。CommungydF4y2Ba.gydF4y2Ba9gydF4y2Ba, 2697(2018)。gydF4y2Ba
Arinobu, Y.等人。GATA-1和PU.1的相互激活标志着造血干细胞进入髓系和髓淋巴系的初始规范。gydF4y2Ba细胞干细胞gydF4y2Ba1gydF4y2Ba, 416-427(2007)。gydF4y2Ba
Laslo, P.等。多谱系转录启动和交替造血细胞命运的确定。gydF4y2Ba细胞gydF4y2Ba126gydF4y2Ba, 755-766(2006)。gydF4y2Ba
Hormoz, S.等。从谱系树和端点单细胞测量推断细胞状态转换动态。gydF4y2Ba细胞系统gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, 419 - 433。e8(2016)。gydF4y2Ba
吕弗勒等人。通过CD43-或cd44 -抗体涂层在液体培养中固定小鼠和人HSPC。gydF4y2Ba血gydF4y2Ba131gydF4y2Ba, 1425-1429(2018)。gydF4y2Ba
拉曼诺,G.等。单细胞的RNA速度。gydF4y2Ba自然gydF4y2Ba560gydF4y2Ba, 494-498(2018)。gydF4y2Ba
希尔森贝克,O.等。用于单细胞跟踪和定量细胞和分子特性的软件工具。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba34gydF4y2Ba, 703-706(2016)。gydF4y2Ba
确认gydF4y2Ba
我们感谢D. Shechter、K. Gritsman、R. Coleman、J. Biswas、E. Tutucci、M. V. Ugalde和R. Pisczcatowski的讨论;F. Mueller为FISH-QUANT提供帮助;M. Elowitz和S. Hormoz为KCA所用的脚本;M. Lopez-Jones协助探头设计;D. Loeffler和T. Schroeder对HSC延时成像的输入;D. Sun协助流式细胞仪和细胞分选。R.H.S.是霍华德休斯医学研究所的高级研究员。a.b.是圣菲研究所的外聘教授。本研究由Ruth L. Kirschstein国家研究服务奖F30GM122308-03和MSTP培训补助金T32GM007288-43资助给J.C.W, U01DA047729资助给R.H.S., R01CA217092资助给美国。美国是白血病和淋巴瘤协会的研究学者,是阿尔伯特·爱因斯坦医学院的黛安娜和亚瑟·贝尔弗癌症研究学院学者。这项工作得到了阿尔伯特·爱因斯坦癌症中心核心支持基金(P30CA013330),以及Ruth L.和David S. Gottesman干细胞研究和再生医学研究所的干细胞分离和异种移植核心设施(NYSTEM基金#C029154)的支持。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
J.C.W, U.S.和R.H.S.对研究进行了概念化并设计了实验。j.c.w., A.B.和Y.S.概念化了数学模型。J.C.W.完成了所有的实验并生成了手稿中的所有数据。J.C.W.进行了mRNA分析、转录参数拟合、随机模拟、scRNA-seq分析和亲缘关系分析。M.W.提供了scRNA-seq分析的基本脚本。Y.S.和A.B.在谱系结构的条件下发展了与状态转变历史相关的分析。J.C.W.写了手稿,并生成了所有的图形和数据可视化。j.c.w., u.s., r.h.s., a.i.s., y.s., A.B.和M.W.审阅并编辑了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
额外的信息gydF4y2Ba
同行评审信息gydF4y2Ba自然gydF4y2Ba感谢Thomas Gregor, Ellen Rothenberg和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
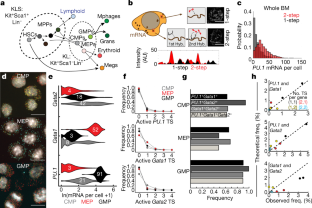
图1有条件的基因的转录动力学gydF4y2BaPU.1gydF4y2Ba状态。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba,光斑强度的累积分布函数(CDF)gydF4y2Ba一个gydF4y2Ba)和光点强度与局部背景强度的信噪比(SNR)直方图(gydF4y2BabgydF4y2Ba)对所有通过强度的点进行显示,三维点扩散函数(PSF)拟合FISH-QUANT中的阈值。gydF4y2BacgydF4y2Ba, Cy3-、Alexa Fluor 594-和cy5标记读出探针在HPC-7细胞中荧光(对应于mRNA分子)的概率密度。Insets是gydF4y2BaXYgydF4y2Ba而且gydF4y2BaXZgydF4y2Ba每个荧光团的平均psf。重叠线是高斯分布的拟合。每个荧光团获得超过10,000个斑点。gydF4y2BadgydF4y2Ba,代表图像三色smFISH为gydF4y2BaPU.1gydF4y2Ba(Cy5、红色)gydF4y2BaGata2gydF4y2Ba(Cy3,白色)和gydF4y2BaGata1gydF4y2Ba(AF594,绿色)在HPC-7细胞中。比例尺,5 μm。gydF4y2BaegydF4y2Ba的双变量分布gydF4y2BaGata1gydF4y2Ba而且gydF4y2BaGata2gydF4y2Ba(左),gydF4y2BaGata2gydF4y2Ba而且gydF4y2BaPU.1gydF4y2Ba(中间)和gydF4y2BaPU.1gydF4y2Ba而且gydF4y2BaGata1gydF4y2Ba(右)在两个独立的实验(gydF4y2BangydF4y2Ba> 400个细胞/实验)与HPC-7细胞。gydF4y2BafgydF4y2Ba,代表图像的多路smFISH之间gydF4y2BaPU.1gydF4y2Ba以及Kit中其他8个造血基因gydF4y2Ba+gydF4y2Ba林gydF4y2Ba−gydF4y2Ba野生型小鼠骨髓(gydF4y2BangydF4y2Ba=每个基因对应258 - 2488个细胞,来源于单个实验;比例尺,5 μm)。gydF4y2BaggydF4y2Ba,的概率分布gydF4y2BaPU.1gydF4y2Ba野生型小鼠骨髓KL细胞中每个细胞的mRNA。重叠的是拟合数据的双分量负二项分布的高分量(红色)和低分量(蓝色)。gydF4y2BahgydF4y2Ba, PU.1高、低状态破裂动力学比较。左,来自smFISH的代表图像gydF4y2BaPU.1gydF4y2Ba在细胞核中有一个大的转录位点。中间,频率的细胞与指示的活跃数量gydF4y2BaPU.1gydF4y2Ba转录的网站。右,每个细胞的新生mRNA总和的频率分布gydF4y2BaPU.1gydF4y2Ba状态。gydF4y2Ba我gydF4y2Ba,展示假设的转录相位图的示意图。gydF4y2BajgydF4y2Ba,每个基因的相位图基于gydF4y2BaPU.1gydF4y2Ba单元格的状态。gydF4y2Ba
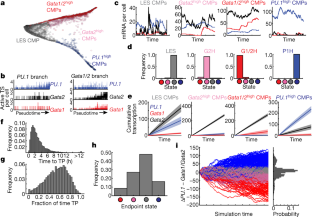
图2 smFISH与scRNA-seq比较分析gydF4y2Ba
一个gydF4y2Ba, 5个scRNA-seq数据集和smFISH的每个细胞mRNA的CDF图。数据被归一化为每个数据集中每个基因的最大计数。gydF4y2BabgydF4y2Ba计算每个scRNA-seq数据集(从白色到黑色)和smFISH数据集(红色)中7种转录因子mrna的基尼指数。gydF4y2BacgydF4y2Ba, 5个scRNA-seq数据集基尼系数的CDF图(见补充表)gydF4y2Ba2gydF4y2Ba基因列表)。gydF4y2BadgydF4y2Ba,分层聚类原理图,然后随机森林分类,以识别重要变量进行聚类分配。gydF4y2BaegydF4y2Ba,四个scRNA-seq数据集的变量重要性与基尼指数的关系。底部和右侧面板分别显示了基尼指数和变量重要性的边际分布。gydF4y2BafgydF4y2Ba,平均互信息(上)或皮尔逊相关系数的平均绝对值(下)与归一化丰度的关系图gydF4y2BangydF4y2Ba= 200个随机选择的基因与数据集中的所有其他基因。的gydF4y2BargydF4y2Ba所列值为相关系数。看到gydF4y2Ba补充讨论gydF4y2Ba有关所执行分析的进一步细节。gydF4y2Ba
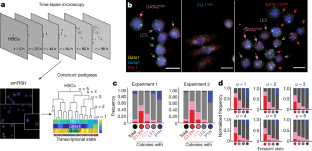
扩展数据图3原发性KL mRNA拷贝数的汇总统计。gydF4y2Ba
一个gydF4y2Ba,经smFISH染色的cmp、gmp和mep的代表图像gydF4y2BaPU.1gydF4y2Ba,gydF4y2BaGata1gydF4y2Ba而且gydF4y2BaGata2gydF4y2Ba.比例尺,5 μm。箭头指向共同表达所有三种mrna的cmp。gydF4y2BabgydF4y2Ba,每个细胞mRNA计数的箱线图,覆盖单细胞mRNA值(点)。粉色框为95%置信区间,红线为均值表达式,灰框为±s.e.m。gydF4y2BacgydF4y2Ba,各基因统计汇总表。数据gydF4y2Ba一个gydF4y2Ba- - - - - -gydF4y2BacgydF4y2Ba来源于两个实验(cmp和mep)或一个实验(gmp)。样本容量列于gydF4y2BacgydF4y2Ba.gydF4y2Ba
图4 FISH-QUANT斑点检测与T淋巴细胞斑点召唤。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba,比较原始(gydF4y2Ba一个gydF4y2Ba)和过滤(gydF4y2BabgydF4y2Ba来自cmp的smFISH图像(代表cmp中的2个以上实验;斑点质量与本手稿中所有报道的实验一致)。插图显示了线条强度图;细胞上的白线表示图是从哪里获得的。比例尺,10 μm。gydF4y2BacgydF4y2Ba,平均PSFgydF4y2BaXYgydF4y2Ba(左栏)和gydF4y2BaXZgydF4y2Ba(右列)为来自CMPs数据集中所有检测点的每个基因。gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba,经验(左)相对于理论(中)PSF和残差(右)gydF4y2BaXYgydF4y2Ba(gydF4y2BadgydF4y2Ba),gydF4y2BaXZgydF4y2Ba(gydF4y2BaegydF4y2Ba)飞机。gydF4y2BafgydF4y2Ba中所有通过滤波强度(上一行)、残差平方(第二行)和宽度的初始强度阈值的点的cfsgydF4y2BaXgydF4y2Ba,gydF4y2BaYgydF4y2Ba,gydF4y2BaZgydF4y2Ba以纳米为单位(分别为第三至第五行)。斑点是根据每个细胞的mRNA拷贝数超过5份,每个细胞的mRNA拷贝数在2到5份之间,以及每个细胞的mRNA拷贝数为1份而分离出来的。3D拟合失败的废弃点以橙色显示。gydF4y2BaggydF4y2Ba, mRNA在初级CD4细胞中的检测gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba胸腺细胞(gydF4y2BangydF4y2Ba= 136gydF4y2BaGata1gydF4y2Ba,gydF4y2BangydF4y2Ba= 154gydF4y2BaPU.1gydF4y2Ba).gydF4y2Ba
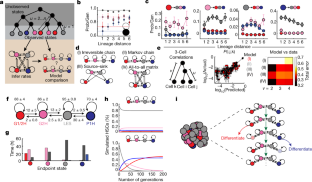
扩展数据图5为状态分配CMP的门控策略。gydF4y2Ba
一个gydF4y2Ba,不同州中医的代表图像。比例尺,10 μm。gydF4y2BabgydF4y2Ba,将cmp分配到转录状态的门控方案。看到gydF4y2Ba补充讨论gydF4y2Ba有关门控策略的详细信息。t-SNE图显示了状态之间以及免疫表型gmp和mep之间的邻近性。从图中报道的实验数据集中得到的图像和分析。gydF4y2Ba1gydF4y2Ba和扩展数据图。gydF4y2Ba3.gydF4y2Ba.gydF4y2BacgydF4y2Ba,各转录状态下各基因转录爆发的频率分布。的gydF4y2BaxgydF4y2Ba轴是活动等位基因的数量。gydF4y2BadgydF4y2Ba,顶部,“状态”的示意图,是LES状态的简单转录噪声(右)与需要转换事件的真正独立的转录状态(右)的结果(箭头)。底部,在噪声(灰色)或状态转换系统(红色)中模拟细胞的时间依赖行为,显示为的二元图gydF4y2BaGata1 + Gata2gydF4y2Ba编号按gydF4y2BaPU.1gydF4y2Ba拷贝数。T表示经过的模拟时间是最终时间的一部分。gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba,吉莱斯皮模拟的状态转变,调制半衰期单独。如果仅通过噪声发生向另一种状态的转变,则细胞仅改变定义该状态的mRNA的半衰期。gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba,模拟中达到的端点状态(gydF4y2BangydF4y2Ba= 10,000) (gydF4y2BaegydF4y2Ba)和1,000个代表性模拟轨迹(gydF4y2BafgydF4y2Ba),以颜色标记最终端点状态。每个面板是mRNA半衰期的不同因素变化,最左边的面板作为参考(即图中使用的半衰期)。gydF4y2Ba2gydF4y2Ba),其余分别为2×(左二)、3×(右二)、4×(右二)。gydF4y2Ba
扩展数据图6造血干细胞72小时后代。gydF4y2Ba
一个gydF4y2Ba, HSC子代的代表性图像。gydF4y2BaPU.1gydF4y2Ba,红色;gydF4y2BaGata2gydF4y2Ba青色;gydF4y2BaGata1gydF4y2Ba,黄色。转录位点用方框划分。全箭头表示三阳性细胞,箭头表示巨核细胞。来自两个独立实验的代表性图像。gydF4y2BabgydF4y2Ba,每个HSC子代的mRNA计数的CDFs。所示为每个细胞大于或等于1个mRNA的细胞数。两个独立的实验,用gydF4y2BangydF4y2Ba图中所示的值。gydF4y2BacgydF4y2Ba的双变量分布gydF4y2BaPU.1gydF4y2Ba与gydF4y2BaGata1gydF4y2Ba(左)和gydF4y2BaPU.1gydF4y2Ba与gydF4y2BaGata2gydF4y2Ba(右)。gydF4y2Ba
扩展数据图7 HSC子代的状态分配。gydF4y2Ba
一个gydF4y2Ba,门控策略。左,首先去除巨核细胞。对,细胞有超过10个拷贝gydF4y2BaGata1gydF4y2Ba被分配到G1/2H,而有超过150个gydF4y2BaPU.1gydF4y2Ba被分配给巨噬细胞。gydF4y2BabgydF4y2Ba的概率密度分布gydF4y2BaPU.1gydF4y2Ba(左)和gydF4y2BaGata2gydF4y2Ba(右)在去除巨核细胞、G1/2H和巨噬细胞后,用重叠拟合得到细胞间的双组分负二项分布。gydF4y2BacgydF4y2Ba,相同细胞的二元分布。与cmmp的情况相反,的种群gydF4y2BaGata2gydF4y2Ba高gydF4y2BaPU.1gydF4y2Ba高gydF4y2BaHSC子代都具有与GMP数据集中的巨噬细胞样细胞相似的形态特征,而GMP数据集中的巨噬细胞也是如此gydF4y2BaGata2gydF4y2Ba高gydF4y2BaPU.1gydF4y2Ba高gydF4y2Ba(见扩展数据图。gydF4y2Ba3.gydF4y2Ba).因此,所有的细胞gydF4y2BaPU.1gydF4y2Ba> 75和< 150归为P1H。gydF4y2BadgydF4y2Ba,的概率分布gydF4y2BaGata2gydF4y2Ba在剩余的单元格中,拟合一个双组分负二项式。gydF4y2BaegydF4y2Ba中那样的分布gydF4y2BadgydF4y2Ba由于分布重叠,不能明确分为高分量和低分量;因此,在KCA期间,细胞被概率地分配到G2H或LES状态,以纠正分配中的不确定性引起的错误转换。看到gydF4y2Ba补充讨论gydF4y2Ba有关概率门控的原理和实现的更多细节。gydF4y2Ba
扩展数据图8 HSC群体数据。gydF4y2Ba
一个gydF4y2Ba端点细胞是每个谱系上的叶子。请注意,边缘长度没有在分裂之间的时间上缩放,并且所有端点细胞从实验开始到96小时。单元格是根据整个手稿使用的配色方案进行颜色编码的。巨核细胞用橙色标记。在端点上游观察到的节点(细胞)(即没有转录数据)被标记为黑色。gydF4y2BabgydF4y2Ba,单个HSC后代数量的直方图。gydF4y2BacgydF4y2Ba- - - - - -gydF4y2BaegydF4y2Ba,基于终点状态识别的细胞增殖表型(P1H,gydF4y2BangydF4y2Ba= 137;莱斯,gydF4y2BangydF4y2Ba= 1571;G1/2H,gydF4y2BangydF4y2Ba= 81;G2H,gydF4y2BangydF4y2Ba= 166)。细胞寿命gydF4y2BaegydF4y2Ba是细胞诞生(最后一次分裂)与下一次细胞分裂或细胞死亡之间的时间间隔。小提琴图归一化为面积,中心盒须图显示平均值(线)、标准差(盒)和95%置信区间(须)。在gydF4y2BaegydF4y2Ba,单点代表第99百分位的异常值。gydF4y2Ba
图9推断的转换矩阵对mRNA阈值的鲁棒性。gydF4y2Ba
一个gydF4y2Ba,每个阈值的推断转换矩阵的归一化偏差(gydF4y2BangydF4y2Ba= 200次自举迭代)的gydF4y2BaGata1gydF4y2Ba相对于本文报道的参考基质,每个细胞的mRNA(截断值=每个细胞的10 mRNA)。参考矩阵是盒装的。对于任何给定的转换(即矩阵项),初始状态是列,最终状态是行。颜色代码与手稿中其他地方使用的颜色相同。gydF4y2BabgydF4y2Ba,如gydF4y2Ba一个gydF4y2Ba为gydF4y2BaPU.1gydF4y2Ba(手稿中截断值=每个细胞75个mRNA)。gydF4y2BacgydF4y2Ba, Frobenius距离gydF4y2Ba\ \√{\ _ {ij}和{({T} _{我{j} _ {{\ rm {ref}}}} - {T} _{我{j} _ {{\ rm{测试}}}})}^ {2}}\)gydF4y2Ba在每个矩阵与参考转换矩阵之间。黑色实线表示参考转换矩阵中统计不确定性导出的背景Frobenius距离,通过分析自举得到gydF4y2BangydF4y2Ba= 1000次,并从转移矩阵的推断均值和标准差定义的高斯分布中随机选择转移率。这条线以上的Frobenius距离值与手稿中报道的矩阵有显著差异。gydF4y2Ba
图10 mRNA分割误差分析。gydF4y2Ba
一个gydF4y2Ba, CMP后期的代表图像。gydF4y2BabgydF4y2Ba, cmmp中每个姐妹细胞的mRNA拷贝数(gydF4y2BangydF4y2Ba= 52)及造血干细胞(gydF4y2BangydF4y2Ba= 46)。gydF4y2BargydF4y2Ba为姐妹细胞mRNA拷贝数的Pearson相关系数;红色虚线是gydF4y2BaygydF4y2Ba=gydF4y2BaxgydF4y2Ba.gydF4y2BacgydF4y2Ba,最近1小时内分裂的造血干细胞之间mRNA水平的相关性(gydF4y2BangydF4y2Ba= 171)。皮尔逊相关系数(gydF4y2BargydF4y2Ba)。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
该文件包括补充方法章节1-6,补充讨论章节1-3,补充图1-2和补充参考。gydF4y2Ba
补充表1gydF4y2Ba
smFISH探针的寡核苷酸序列。对于用两步smFISH检测的基因,列出了适当的读出探针。gydF4y2Ba
补充表2gydF4y2Ba
用于scRNAseq分析的基因列表。每个scRNAseq数据集中分析的所有基因集的基因名称。gydF4y2Ba
补充表3gydF4y2Ba
GO项(按富集分数排序)和基因VI的前十分位数进行scRNAseq分析。从分析的每个scRNAseq数据集中按变量重要性排序的前十分位基因。相关的GO术语,按富集分数排序,为那些前十分位基因。gydF4y2Ba
补充表4gydF4y2Ba
CMP数据的推断转录参数。Kon:基因开启概率;koff: ON基因关闭的概率;kini:开启时,RP2引发事件的概率;kd: mRNA的衰减率。gydF4y2Ba
补充表5gydF4y2Ba
本研究中使用的抗体列表。gydF4y2Ba
补充表6gydF4y2Ba
本研究实验中使用的试剂。gydF4y2Ba
补充表7gydF4y2Ba
软件列表和软件下载的相关url。gydF4y2Ba
补充数据1gydF4y2Ba
这个压缩文件包括:gydF4y2BaColonyIDs.matgydF4y2Ba- KCA的所有殖民地名称;gydF4y2BaBdry.matgydF4y2Ba-每个菌落的细胞数目;gydF4y2BaColony_ 1:117 .matgydF4y2Ba- 117个数据帧,包含执行KCA所需的所有数据;gydF4y2BaKLdatamatrix.matgydF4y2Ba- KL祖先数据;gydF4y2BaKCA_datamatrix.matgydF4y2Ba- KCA中使用的所有单元的数据矩阵。gydF4y2Ba
补充数据2gydF4y2Ba
这个压缩文件包括:gydF4y2BaFSP.mgydF4y2Ba-使用该软件进行参数推断,基于最适合的爆发频率和mRNA计数/细胞;gydF4y2BagetKLD.mgydF4y2Ba- FSP.m使用;gydF4y2BamarkovBackTrace.mgydF4y2Ba-给定一个单元格在时间t时处于状态i,可用于确定对状态j的访问次数;gydF4y2BaGSSA.mgydF4y2Ba-随机模拟;gydF4y2BatreeBackTrace2.mgydF4y2Ba-由GenerateAllTrees.m使用;gydF4y2BaGenerateAllTrees.mgydF4y2Ba-生成所有殖民地的谱系地图,并计算在每个州花费的时间;gydF4y2BaKCA.mgydF4y2Ba-根据殖民地中存在的状态产生状态频率,并运行转换概率推断;gydF4y2BaThreePtFreqs.mgydF4y2Ba- 3单元状态频率测试;gydF4y2BaKCA_thresholdtesting.mlxgydF4y2Ba-用于测试KCA的不同mRNA截断值,并与参考矩阵进行比较。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
麦特,j.c.,塞拉,Y.,威尔考克森,M。gydF4y2Baet al。gydF4y2Ba体细胞干细胞转录动力学的单分子成像。gydF4y2Ba自然gydF4y2Ba583gydF4y2Ba, 431-436(2020)。https://doi.org/10.1038/s41586-020-2432-4gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-020-2432-4gydF4y2Ba
这篇文章被引用gydF4y2Ba
利用贝叶斯非参数从快照RNA数据推断基因表达模型gydF4y2Ba
自然计算科学gydF4y2Ba(2023)gydF4y2Ba
造血分化的特点是在单细胞水平上有一个短暂的熵峰gydF4y2Ba
BMC生物学gydF4y2Ba(2022)gydF4y2Ba
RS-FISH:精确、交互式、快速、可扩展的FISH斑点检测gydF4y2Ba
自然方法gydF4y2Ba(2022)gydF4y2Ba
通过嵌入双稳定子系统设计多稳定系统的鲁棒方法gydF4y2Ba
系统生物学与应用gydF4y2Ba(2022)gydF4y2Ba
多参数时序成像分析揭示巨核红细胞祖细胞克隆扩增和分化动力学gydF4y2Ba
科学报告gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
