摘要gydF4y2Ba
经过20年的改进,目前的人类参考基因组(GRCh38)是迄今为止最准确、最完整的脊椎动物基因组。然而,没有一个染色体被首尾相连,数百个未解决的空白仍然存在gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba.在这里,我们展示了一个超越GRCh38连续性的人类基因组组装gydF4y2Ba2gydF4y2Ba,以及人类染色体端粒到端粒的无间隙组装。这是通过对葡萄胎CHM13全基因组进行高覆盖、超长读取的纳米孔测序,并结合用于质量改进和验证的补充技术实现的。我们把精力集中在人类X染色体上gydF4y2Ba3.gydF4y2Ba,我们重建了中心粒卫星DNA阵列(约3.1 Mb),并填补了当前参考文献中剩余的29个空白,包括来自人类假常染色体区域和癌症睾丸扩增基因家族(CT-X和GAGE)的新序列。这些序列将被整合到未来发布的人类参考基因组中。此外,完整的X染色体与超长纳米孔数据相结合,使我们能够在复杂的串联重复序列和卫星阵列中绘制甲基化模式。我们的研究结果表明,完成整个人类基因组现在触手可及,这里提供的数据将促进正在进行的完成其他人类染色体的努力。gydF4y2Ba
主要gydF4y2Ba
完整的端粒到端粒参考基因组组装是必要的,以确保所有的基因组变体被发现和研究。目前,人类基因组中尚未解决的区域是由位于染色体周围区域的多兆酶卫星阵列和位于染色体末端短臂上的核糖体DNA阵列,以及长度大于数百千碱基的片段重复丰富的区域来定义的,这些区域在对位序列之间显示出超过98%的序列一致性。由于缺乏参考文献,这些富含重复序列经常被排除在遗传学和基因组学研究之外,这限制了关联和功能分析的范围gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba.未解决的重复序列也会导致意想不到的后果;例如,副同源序列变异被错误地称为等位变异gydF4y2Ba6gydF4y2Ba以及细菌基因数据库的污染gydF4y2Ba7gydF4y2Ba.完整的人类基因组有望有助于我们对染色体功能的理解gydF4y2Ba8gydF4y2Ba,人类疾病gydF4y2Ba9gydF4y2Ba以及基因组变异,这将改善生物医学中使用短读映射到参考基因组的技术(例如RNA测序(RNA-seq))gydF4y2Ba10gydF4y2Ba,染色质免疫沉淀,测序(ChIP-seq)gydF4y2Ba11gydF4y2Ba利用测序(ATAC-seq)测定转座酶可及染色质gydF4y2Ba12gydF4y2Ba).gydF4y2Ba
从许多相对较短的测序读数(称为基因组组装)重建基因组的根本挑战是将重复序列彼此区分开来gydF4y2Ba13gydF4y2Ba.解决这种重复依赖于测序读取足够长,以跨越整个重复或准确到区分每个重复副本的基础上的唯一变体gydF4y2Ba14gydF4y2Ba.2001年首次公开的人类基因组在20年后仍未完成,这突显了组装问题的困难和过去技术的局限性gydF4y2Ba15gydF4y2Ba.美国国家生物技术信息中心(NCBI Build 28)发布的第一个人类参考基因组高度碎片化,一半的基因组包含在500kb或更多的连续序列(contigs)中(NG50)。努力完成基因组gydF4y2Ba16gydF4y2Ba,以及基因组参考联盟(GRC)的管理工作gydF4y2Ba2gydF4y2Ba这大大增加了最近发布的grch38中NG50 contig长度为56 Mb的引用的连续性,但基因组中最重复的区域仍然没有解决,没有染色体是完全代表端粒到端粒的。超长(大于100 kb)纳米孔读数的从头组装在最困难的区域显示出有希望的组装连续性gydF4y2Ba1gydF4y2Ba但是这个概念验证项目对基因组的测序只有5倍的覆盖深度,并且未能组装最大的人类基因组重复序列。先前基于人类基因组中大重复序列的大小和分布进行的建模预测,30×超长reads的组装将接近人类参考序列的连续性gydF4y2Ba1gydF4y2Ba.因此,我们假设高覆盖率超长读取纳米孔测序将使人类染色体的第一次完整组装成为可能。gydF4y2Ba
为了避免组装二倍体基因组的两种单倍型的复杂性,我们选择了有效的单倍体CHM13hTERT细胞系进行测序(以下简称CHM13)。gydF4y2Ba17gydF4y2Ba.该细胞系来源于一个完整的葡萄胎(CHM),核型为46,XX。这种子宫痣的基因组来源于一个经历了减数分裂后染色体复制的单一精子;因此,对于一组等位基因来说,这些基因组是均匀纯合的。CHM13以前曾被用于修补人类参考中的空白gydF4y2Ba2gydF4y2Ba、基准基因组组装器和二倍体变体调用器gydF4y2Ba18gydF4y2Ba,并研究人类片段复制gydF4y2Ba19gydF4y2Ba.CHM13系的核型分析证实为稳定的46,XX核型,没有可观察到的染色体异常。gydF4y2Ba1gydF4y2Ba,补充说明gydF4y2Ba1gydF4y2Ba).最大似然混合分析gydF4y2Ba20.gydF4y2Ba有信心地将大多数单倍型确定为欧洲起源,有可能是一些亚洲或美洲印第安人的混合物(扩展数据图。gydF4y2Ba2gydF4y2Ba,补充说明gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
高度连续的全基因组组装gydF4y2Ba
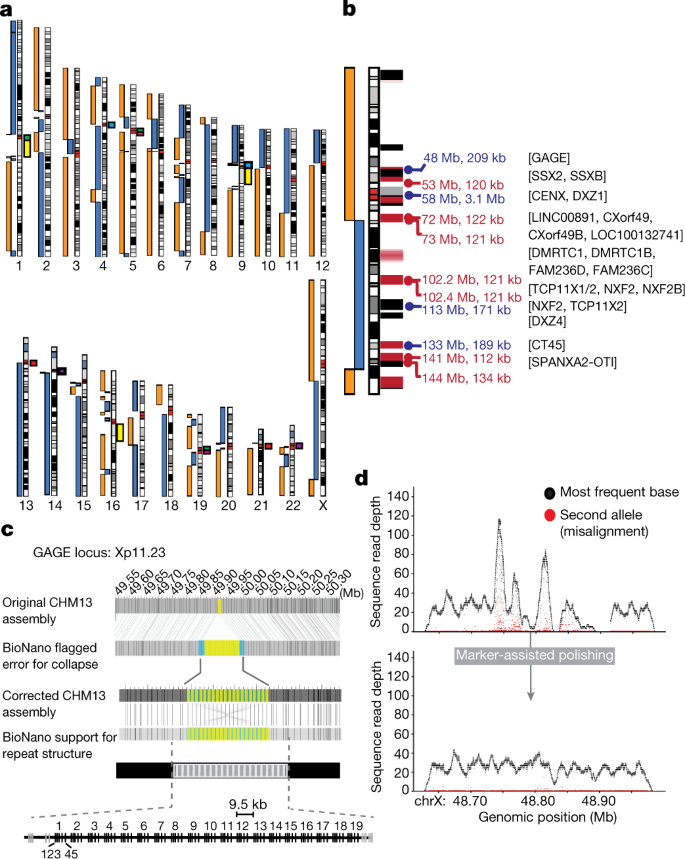
从CHM13细胞中提取高分子量DNA,并使用先前描述的超长读取方案制备纳米孔测序gydF4y2Ba1gydF4y2Ba.我们总共测序了98个MinION流单元,总容量为155 Gb (50×覆盖率,每个流单元1.6 Gb,补充说明gydF4y2Ba3.gydF4y2Ba).所有测序的碱基中有一半包含在70 kb或更长的reads中(78 Gb, 25×基因组覆盖),最长的验证reads为1.04 Mb。一旦我们收集到足够的从头组装测序覆盖,我们将39×覆盖的超长reads与70×覆盖的先前生成的PacBio数据结合起来,并使用Canu组装CHM13基因组gydF4y2Ba21gydF4y2Ba.Canu选择了最长的30×-coverage ultra-long和7×-coverage PacBio reads进行校正和组装。这个初始组装的总长度为2.90 Gb,其中一半的基因组包含在长度为75 Mb或更大的连续序列(contigs)中(NG50),这超过了GRCh38参考基因组的连续性(NG50为75 Mb, NG50为56 Mb)。然后,通过一系列测序技术(nanoore、PacBio和链接读取Illumina)对该组装进行迭代优化。共识精度从初始装配的99.46%提高到经过Nanopore抛光的99.67%,经过PacBio抛光的99.99%。Illumina数据仅用于纠正基因组中唯一可映射区域的小插入和删除错误,这对平均精度有边际影响,但减少了帧移基因的数量。通过分析未用于初始装配的Illumina链接读取条形码(10X Genomics)和光学映射(Bionano Genomics)数据,确定了假定的误装配。最初的contigs在低映射覆盖区域被破坏,然后使用光学映射对校正后的contigs进行排序和相对定向。超过90%的6条染色体在2个contigs中表示,10条染色体由2个支架表示。gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba,无间隙contigs在染色体表意图旁以蓝色和橙色条表示(突出显示contig断裂)。有几条染色体只在着丝粒区断裂。contigs之间的大间隙(例如,chr1的中间)表明存在较大的异色块(黄色为人类卫星2和3的阵列)或没有GRCh38序列的核糖体DNA阵列。预计非同源染色体之间序列相似的着丝粒卫星阵列显示:chr1、chr5和chr19(绿色);Chr4和chr9(浅蓝色);Chr5和chr19(粉红色);Chr13和chr21(红色);还有chr14和chr22(紫色)gydF4y2BabgydF4y2BaX染色体被选择进行人工组装,最初在三个位置被破坏:着丝粒(在组装中人工坍塌)、一个大的片段重复(DMRTC1B, 120 kb)和第2号染色体上的第二个片段重复(134 kb)。GRCh38参考基因(黑色)和已知片段重复(红色;与Y平行,粉色)都有注释。大于100 kb的重复序列以预期大小(kb)命名(蓝色,串联重复序列;红色,部分重复)。gydF4y2BacgydF4y2Ba,由光学图(上)识别的GAGE位点的误组装,以及修正后的版本(下)显示19个(9.5 kb)全长重复单元和两个部分重复单元的最终组装。gydF4y2BadgydF4y2Ba,使用独特的(单拷贝)标记放置长读,进行抛光前后GAGE位点的质量。点表示从映射的PacBio HiFi读取中恢复的主(黑色)和次(红色)等位基因的覆盖深度(每个碱基重叠的映射测序读的数量)gydF4y2Ba4gydF4y2Ba).由于CHM13基因组是有效的单倍体,低覆盖区域或增加的次级等位基因频率表明低质量区域或潜在的重复崩溃。标记辅助抛光显著改善了整个GAGE位点的等位基因均匀性。gydF4y2Ba
最终组装由448个contigs中的2.94 Gb组成,其中contig NG50为70 Mb。共有98个支架(173个contigs)被明确分配到一条参考染色体上,代表了组装碱基的98%。基于之前完成的两个BAC序列,我们估计该全基因组组装的一致准确性中位数至少为99.99%gydF4y2Ba22gydF4y2Ba和映射的Illumina链接读取(补充说明gydF4y2Ba4gydF4y2Ba).虽然与GRCh38未加壳长度(2.95 Gb)相似,但我们的组装大小比估计的人类基因组大小3.2 Gb短。我们使用段复制汇编器(SDA)方法估计大约170 Mb的崩溃基gydF4y2Ba19gydF4y2Ba.与最近的其他组装相比,我们解析了更大比例的341个CHM13细菌人工染色体(BAC)序列,这些序列之前已经从基因组的分段复制和其他难以组装的区域中分离和完成gydF4y2Ba19gydF4y2Ba(表gydF4y2Ba1gydF4y2Ba,补充说明gydF4y2Ba4gydF4y2Ba).我们的全基因组组装的比较注释也表明,与以前的组装相比,映射转录本的一致性更高,与GRCh38相比,潜在的框架转移率仅略有增加gydF4y2Ba23gydF4y2Ba.在CHM13 de novo组合中注释的19,618个蛋白质编码基因中,只有170个(0.86%)包含预测的移码,或者,如果通过转录本测量,83,332个转录本中只有334个(0.40%)包含预测的移码(补充表)gydF4y2Ba1gydF4y2Ba).当被用作调用其他基因组中的结构变异的参考序列时,CHM13报告了插入和删除调用的平衡(扩展数据图。gydF4y2Ba3.gydF4y2Ba,补充说明gydF4y2Ba5gydF4y2Ba),正如预期的那样,而GRCh38显示出删除偏倚,如先前报道的那样gydF4y2Ba24gydF4y2Ba.与其他长读程序集相比,GRCh38调用的反转次数是CHM13的两倍(平均每个基因组26次对13次反转),这表明一些定向错误的序列仍然存在于当前的人类参考中(补充说明)gydF4y2Ba5gydF4y2Ba).在这些倒置中,有19个是GRCh38特异性的,而在最近组装的5个长读人类基因组中没有发现(补充表)gydF4y2Ba5gydF4y2Ba).我们在组装和读取中识别了端粒序列(扩展数据图。gydF4y2Ba4gydF4y2Ba,补充说明gydF4y2Ba4gydF4y2Ba),它们在端粒大小上高度一致,并且我们的组装在contig末端包括46个预期端粒中的41个。因此,在连续性、完整性和正确性方面,我们的CHM13组装体通过一些质量指标超过了所有以前的人类从头组装体——包括当前的人类参考基因组(补充表)gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
一个完整的人类X染色体gydF4y2Ba
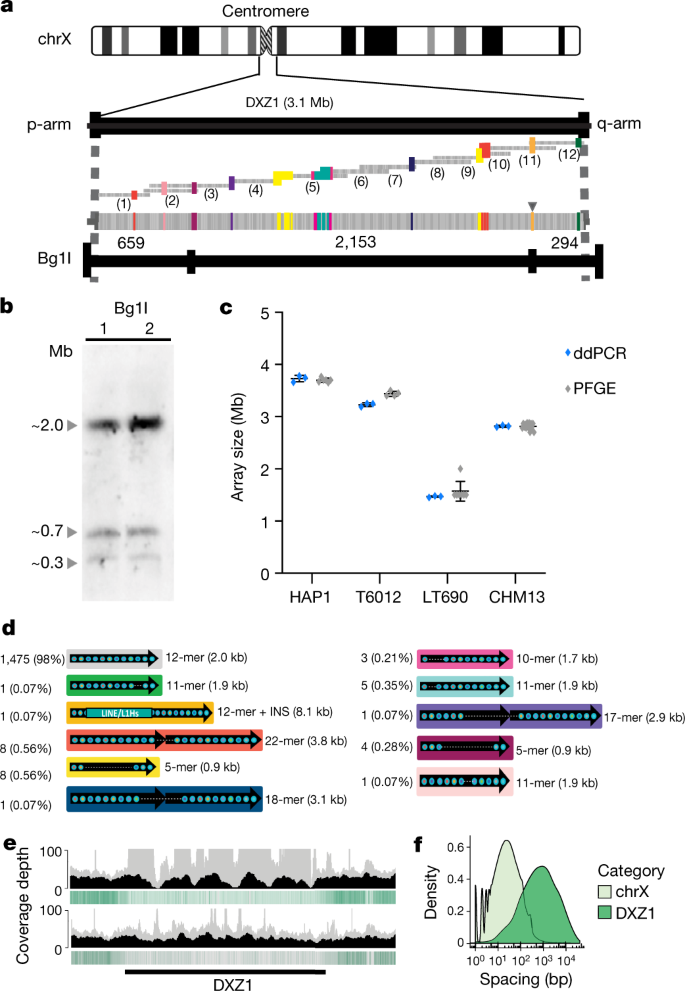
以这种全基因组组装为基础,我们选择了X染色体进行人工整理和验证,因为它在初始组装时具有较高的连续性;独特且特征良好的中心点阿尔法卫星阵列gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba25gydF4y2Ba;开发过程中的独特行为gydF4y2Ba26gydF4y2Ba;以及与孟德尔病的比例失调gydF4y2Ba3.gydF4y2Ba.X染色体的从头组装在三个地方被破坏:着丝粒和两个几乎相同的大于100 kb的片段重复(图2)。gydF4y2Ba1 bgydF4y2Ba).通过识别完全跨越重复的超长读取,并在两侧唯一锚定,手动解决了破坏组件的两个片段重复,从而允许在组件中有一个自信的位置。通过映射从CHM13生成的PacBio高保真(HiFi)长读取的正交集来评估这些困难区域的组装质量的改进gydF4y2Ba22gydF4y2Ba以及评估信息性单核苷酸变异差异的阅读深度(方法)。此外,采用液滴数字PCR (ddPCR)的实验验证证实,现在完整的组装正确地代表了CHM13基因组的串联重复序列,包括7个CT47基因(7.02±0.34 (mean±s.d.)), 6个CT45基因(6.11±0.38),19个完整和2个部分GAGE基因(19.9±0.745),55个DXZ4重复序列(55.4±2.09)和3.1 mb的中心体DXZ1阵列(1408±40.69 2,057 bp重复序列)(补充注)gydF4y2Ba6gydF4y2Ba).gydF4y2Ba
先前对X染色体上单倍体着丝粒卫星阵列(DXZ1)的高分辨率研究为我们目前的人类着丝粒组织基因组模型提供了信息gydF4y2Ba8gydF4y2Ba.与所有正常的人类着丝粒一样,X着丝粒在序列水平上由α卫星dna定义——一个富含at(约171 bp)的串联重复,或“单体”。gydF4y2Ba27gydF4y2Ba.DXZ1阵列的标准重复是由12个分散的单体定义的,这些单体被有序地形成一个约2 kb的更大的重复单元,这被称为“高阶重复”(HOR)。gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba.HORs被串联排列成一个大型的、多兆字节大小的卫星阵列(即2.2-3.7 Mb;平均3,010 KB (s.d = 429,gydF4y2BangydF4y2Ba= 49))gydF4y2Ba25gydF4y2Ba重复副本之间的核苷酸差异有限gydF4y2Ba8gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba.这些之前的评估被用来指导我们对DXZ1组件的评估,并提供了既定的实验方法来评估DXZ1阵列的结构gydF4y2Ba25gydF4y2Ba,gydF4y2Ba32gydF4y2Ba(扩展数据图。gydF4y2Ba5gydF4y2Ba).为了组装X着丝粒,我们在经典DXZ1重复单元内构建了一个结构和单核苷酸变体目录(约2 kb)gydF4y2Ba28gydF4y2Ba,gydF4y2Ba33gydF4y2Ba并使用这些变体作为路标gydF4y2Ba8gydF4y2Ba在整个中心点卫星阵列(DXZ1)上唯一地平铺超长读取(扩展数据图。gydF4y2Ba5中gydF4y2Ba),就像之前对Y着丝粒所做的那样gydF4y2Ba34gydF4y2Ba.通过脉冲场凝胶电泳(PFGE) Southern blotting估计DXZ1阵列在大约2.8-3.1 Mb的范围内(图。gydF4y2Ba2 bgydF4y2Ba扩展数据图gydF4y2Ba6gydF4y2Ba),得到的约束轮廓与预测的阵列组件结构一致(图2)。gydF4y2Ba2 a, bgydF4y2Ba).ddPCR对DXZ1重复序列的拷贝数估计与PFGE Southern blotting之前大小的阵列面板进行了比较,并进一步支持了约2.8 Mb(1408±81.38)个2057 kb标准重复序列的拷贝数。gydF4y2Ba2摄氏度gydF4y2Ba,补充表gydF4y2Ba3.gydF4y2Ba,补充说明gydF4y2Ba7gydF4y2Ba).此外,DXZ1结构变异频率与PacBio HiFi数据的直接比较高度一致gydF4y2Ba22gydF4y2Ba(无花果。gydF4y2Ba二维gydF4y2Ba扩展数据图gydF4y2Ba5度gydF4y2Ba).gydF4y2Ba
一个gydF4y2Ba, Top,数组,大约有2-kb的重复单元,由垂直带标记(灰色为规范单位;彩色的是结构变体)。数组中的单个LINE/L1Hs插入用箭头标记。底部,酶BglI的预测限制图,虚线表示DXZ1阵列之外的区域。为了说明目的,重构了最小平铺路径,这不是初始装配的机制(扩展数据图)。gydF4y2Ba5 bgydF4y2Ba).gydF4y2BabgydF4y2Ba,用于BglI摘要副本的实验性PFGE Southern印迹(用三角形表示条带大小;BglI, 2.87 Mb±0.16),符合硅内预测的波段模式(gydF4y2Ba一个gydF4y2Ba)用于CHM13阵列(实验重复了六次,结果相似)。gydF4y2BacgydF4y2Ba,使用ddPCR估计阵列大小(重复3次;mean±s.d.)针对PFGE Southern blots (HAP1,gydF4y2BangydF4y2Ba= 6;T6012,gydF4y2BangydF4y2Ba= 4;LT690,gydF4y2BangydF4y2Ba= 7;CHM13,gydF4y2BangydF4y2Ba= 13)。gydF4y2BadgydF4y2Ba,相对于2057 bp标准重复单元(灰色)识别的33个DXZ1结构变体的目录,以及观察到的实例数量、阵列中的频率、α卫星单体的数量和大小。INS,插入(即8.1 kb插入的LINE/L1Hs)。gydF4y2BaegydF4y2Ba,映射(灰色)和唯一锚定(黑色)纳米孔的覆盖深度读取到DXZ1阵列。标记辅助抛光(底部)与未抛光(顶部)组件相比提高了覆盖均匀性。单个副本的唯一标记显示为垂直的绿色带,整个数组的密度减小但非零。gydF4y2BafgydF4y2Ba,分布表示染色体X和DXZ1上相邻唯一标记之间的间隔。在X染色体上,平均每66个碱基就有一个唯一的标记,而在DXZ1中,每2.3 kb就有一个唯一的标记,任意两个相邻标记之间的最长间隔为42 kb。gydF4y2Ba
当前的长读取程序集需要严格的共识抛光,以实现最大的基本调用精度gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba.给定每个读取点在组件中的位置,这些抛光工具对底层信号数据进行统计建模,以对每个测序碱基进行准确预测。这个过程的关键是每次阅读的正确位置,这将有助于抛光。由于模糊的读映射,我们最初的抛光尝试降低了最大的x染色体重复中的组装质量(扩展数据图。gydF4y2Ba7 a、bgydF4y2Ba).为了克服这一问题,我们分析了Illumina测序数据,编目了CHM13 X染色体上的短(21 bp)、唯一(单拷贝)序列(扩展数据图)。gydF4y2Ba8gydF4y2Ba).即使在最大的重复序列阵列中,如DXZ1,重复拷贝之间也有足够的变化,以半规则的间隔诱导独特的21-mer标记(图2)。gydF4y2Ba2 e, fgydF4y2Ba扩展数据图gydF4y2Ba8 cgydF4y2Ba).这些标记被用来告知长x染色体在组装中的正确位置(方法)。每种技术进行两轮迭代抛光;首先是Oxford Nanopore,然后是PacBio,最后是Illumina链接阅读gydF4y2Ba37gydF4y2Ba,并且在每一轮之后,共识的准确性都有所提高。Illumina数据太短,无法使用唯一的标记来确定,只能用于抛光映射明确的唯一区域。这种精心的抛光过程被证明是精确完成超过纳米孔和PacBio读取长度的x染色体重复的关键。gydF4y2Ba
我们手工完成的x染色体组装是完整的,无间隙的,根据x特异性BACs估计准确率为99.991%,根据Illumina数据映射准确率为99.995%。有明确的支持99.9%的装配基地(补充说明gydF4y2Ba4gydF4y2Ba),以符合已完成基因组序列的原始百慕大标准gydF4y2Ba38gydF4y2Ba.预测最大重复序列(如DXZ1卫星阵列)的准确度略低(中位数识别率99.3%),但由于缺乏来自这些区域的BAC克隆,这很难测量。映射的长读和光学映射数据显示完整的X染色体覆盖均匀,在可映射区域没有结构错误的证据(图2)。gydF4y2Ba2 egydF4y2Ba扩展数据图gydF4y2Ba8 b, cgydF4y2Ba,补充说明gydF4y2Ba4gydF4y2Ba),以及Strand-seq数据证实没有任何反转错误gydF4y2Ba39gydF4y2Ba,gydF4y2Ba40gydF4y2Ba(扩展数据图。gydF4y2Ba8 d, egydF4y2Ba).通过长读映射的单核苷酸变体调用显示,在大量串联重复的GAGE和CT47基因家族中,初始组装质量较低,但这些问题通过优化和超长读映射和光学映射得到了解决(图2)。gydF4y2Ba1 c, dgydF4y2Ba扩展数据图gydF4y2Ba7 c-jgydF4y2Ba,补充表gydF4y2Ba4gydF4y2Ba).通过TandemQUAST测量,DXZ1阵列映射的长读覆盖显示了均匀的覆盖深度和高精度gydF4y2Ba41gydF4y2Ba(无花果。gydF4y2Ba2 e fgydF4y2Ba,扩展数据图。gydF4y2Ba7gydF4y2Baj,gydF4y2Ba8 cgydF4y2Ba).我们识别了所有与DXZ1重复序列匹配的高保真读数。所有的读数——除了一个大的,可能是错误的均聚物——都被我们的重建解释了,证实了DXZ1阵列的完整性。整个X染色体的覆盖范围是均匀的,只有一小部分碱基的覆盖范围比平均值高出三个标准差(0.44% Nanopore, 0.77% PacBio continuous long reads (CLR), 2.4% HiFi)。低覆盖的HiFi区域因其较低的唯一标记密度而富集,这使得它们由于长度相对较短而难以分配(补充说明)gydF4y2Ba4gydF4y2Ba).此外,变量调用没有从HiFi或CLR数据中识别出高频变量,只从超长读取数据中识别出低复杂度变量,这些变量可能代表超长读取数据中的错误,而不是真正的装配错误。我们完整的端粒到端粒版本的X染色体完全解决了29个参考空白gydF4y2Ba3.gydF4y2Ba,总计1,147,861 bp之前的模糊碱基(n -碱基)。gydF4y2Ba
染色体范围的DNA甲基化图gydF4y2Ba
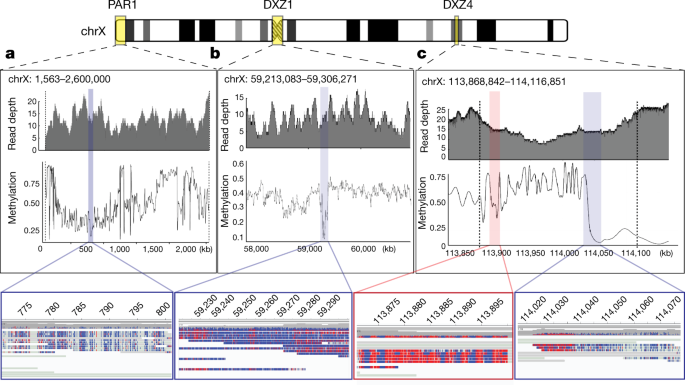
纳米孔测序对甲基化碱基敏感,正如原始电信号的调制所揭示的那样gydF4y2Ba42gydF4y2Ba.精确锚定的超长reads提供了一种新的方法来分析重复区域的甲基化模式,这通常很难用短读测序来检测。X染色体具有许多人类基因组中独一无二的表观基因组特征。X染色体失活,其中一个女性X染色体在发育早期沉默,并在体细胞组织中保持不活跃,预计将提供一个独特的甲基化染色体范围。与之前的研究一致gydF4y2Ba43gydF4y2Ba,我们观察到位于x染色体臂两端尖端的大多数假常染色体区域(PAR1和PAR2)甲基化下降(图2)。gydF4y2Ba3gydF4y2Ba).失活的X染色体也采用了不寻常的空间构象和,与先前的研究一致gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2BaCHM13染色体构象捕获(Hi-C)数据支持在宏卫星重复序列DXZ4上划分的两个大的超域(扩展数据图)。gydF4y2Ba9gydF4y2Ba).在对DXZ4阵列的进一步分析中,我们发现了明显的甲基化带(图2)。gydF4y2Ba3 cgydF4y2Ba),在远端边缘观察到低甲基化,这通常与先前描述的染色质结构一致gydF4y2Ba46gydF4y2Ba.值得注意的是,我们还在DXZ1着丝粒阵列中发现了一个甲基化降低的区域(约60 kb, chrX: 59,217,708-59,279,205)(图2)。gydF4y2Ba3 bgydF4y2Ba).为了测试这一发现是否只存在于X阵列,或者也存在于其他着丝粒卫星上,我们在8号染色体(D8Z2)上手工组装了一个约2.02 Mb的着丝粒阵列。gydF4y2Ba47gydF4y2Ba,gydF4y2Ba48gydF4y2Ba并使用相同的唯一标记映射策略在数组中自信地锚定长读取(G.A.L.等人,手稿正在准备中)。在此过程中,我们在D8Z2阵列中发现了另一个低甲基化区域,与我们在DXZ1阵列上的观察相似(扩展数据图。gydF4y2Ba10gydF4y2Ba),这进一步证明了我们的超长读取映射策略能够提供基本水平的染色体范围DNA甲基化图。还需要进一步的研究来验证这一发现,并评估这些甲基化模式的潜在重要性(如果有的话)。gydF4y2Ba
甲基化估计通过平滑甲基化频率数据计算,窗口大小为500个核苷酸。PAR1、DXZ1和DXZ4的覆盖深度和高质量甲基化调用(|log-likelihood| > 2.5)显示为插图。只有具有可靠的唯一锚映射且至少存在一个高质量甲基化调用的读取才会被考虑。gydF4y2Ba一个gydF4y2Ba纳米孔覆盖和甲基化需要X染色体的伪常染色体区域1 (PAR1)(1,563-2,600,000)。底部集成基因组学查看器(IGV)插图显示PAR1(770,545-801,293)内的低甲基化区域,蓝色为未甲基化碱基,红色为甲基化碱基。gydF4y2BabgydF4y2Ba, DXZ1序列中的甲基化,底部IGV插图显示X染色体着丝粒附近约93 kb的低甲基化区域(59,213,083-59,306,271)。gydF4y2BacgydF4y2Ba,黑色垂直虚线表示DXZ4数组的开始坐标和结束坐标。左IGV插图显示X染色体DXZ4的甲基化区域(113,870,751-113,901,499);右侧IGV插图显示DXZ4从甲基化区域到非甲基化区域的转变(114,015,971-114,077,699)。gydF4y2Ba
一条完成人类基因组的道路gydF4y2Ba
人类染色体端粒到端粒的完整组装表明,现在有可能使用现有技术完成整个人类基因组。虽然我们在这里的重点是完成X染色体,但我们的全基因组组装已经重建了其他几条染色体,只剩下一些空白,并且可以作为完成其他染色体的基础。然而,仍有许多挑战需要克服。例如,将这些方法应用于二倍体样本将需要对潜在的单倍型进行分期,以避免复杂结构变异的混合区域。我们对其他染色体的初步分析表明,比X染色体大的重复区域和着丝粒卫星将需要开发更多的方法gydF4y2Ba49gydF4y2Ba.这尤其适用于人类的末端染色体,其大量的卫星阵列和片段复制尚未在序列水平上得到解决。此外,图。gydF4y2Ba1gydF4y2Ba突出显示了预计非同源染色体之间序列相似的着丝粒卫星阵列。像这样的阵列需要在染色体之间和染色体内部进行相控。gydF4y2Ba
完成人类基因组将随着这些剩余挑战的解决而继续进行,从相对容易组装的染色体(例如,3、6、8、10、11、12、17、18和20)开始,最终以包含大块经典人类卫星染色体(1、9和16)和末端染色体(13、14、15、21和22)的染色体结束。在短期内,CHM13基因组中闭合的参考缺口将使用GRC现有的“补丁”基础设施整合到GRCh38中。一旦所有CHM13染色体完成,我们计划将其提供给GRC,作为一个新的、完全无间隙的参考基因组发布的基础,这可能是当前参考序列与CHM13序列在最困难区域的马赛克。最终完成GRC人类参考基因组的努力将有助于推进必要的技术,实现我们的最终目标,即所有人类基因组的完整、端粒到端粒的二倍体组装。gydF4y2Ba
方法gydF4y2Ba
数据报告gydF4y2Ba
没有使用统计方法来预先确定样本量。实验不是随机的,研究人员在实验和结果评估期间没有对分配盲目。gydF4y2Ba
细胞培养gydF4y2Ba
完整葡萄胎CHM13的细胞最初是从马吉妇女医院(匹兹堡)的一例葡萄胎中培养出来的,这是21世纪初进行的一项研究的一部分(IRB mh -20-054)。同时,培养CHM13细胞,使用Q带核型,随后使用人端粒酶逆转录酶(hTERT)进行永生化。在本研究中,冷冻保存的CHM13细胞在完整的AmnioMax C-100基础培养基(Thermo Fisher Scientific)中解冻培养,并添加1%青霉素-链霉素(Thermo Fisher Scientific),并在37°C的湿度控制环境中生长,95% OgydF4y2Ba2gydF4y2Ba5% COgydF4y2Ba2gydF4y2Ba.每三天更换一次新鲜培养基,用于本研究的所有细胞均未超过10代。细胞已鉴定,支原体污染检测呈阴性。gydF4y2Ba
核型分析gydF4y2Ba
中期载玻片制剂取自人葡萄胎细胞系CHM13,并采用前面描述的标准风干技术制备gydF4y2Ba51gydF4y2Ba.采用DAPI条带技术根据ISCN鉴定核型中的结构和数量染色体畸变gydF4y2Ba52gydF4y2Ba.核型分析使用蔡司M2荧光显微镜和应用光谱成像软件(补充注gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
DNA提取,文库制备和测序gydF4y2Ba
从5 × 10中提取高分子量DNAgydF4y2Ba7gydF4y2BaCHM13细胞使用改良的Sambrook和Russell协议gydF4y2Ba1gydF4y2Ba,gydF4y2Ba53gydF4y2Ba.文库使用Oxford Nanopore Technologies公司的快速测序试剂盒(SQK-RAD004)和15 μg DNA构建。初始反应通常分为三部分进行加载,并加入FRA缓冲液(104 mM Tris pH 8.0, 233 mM NaCl),使体积达到21 ul。这些反应在4°C下孵育48小时,以使缓冲液在加载前达到平衡。大多数测序在nanoore GridION上使用FLO-MIN106或FLO-MIN106D R9流池进行,只有一个Flongle流池用于测试。在初始组装中使用的测序读数是测序仪上的第一个碱基。在收集了所有数据后,读取数据再次使用最新的Guppy算法(v.2.3.1中启用了' flip-flop '模型)进行基调用。gydF4y2Ba
根据制造商的协议,使用10X Genomics Chromium设备和Chromium Reagent Kit v.2,从1ng高分子量基因组DNA制备了10X Genomics链接阅读基因组文库。该文库在Illumina NovaSeq 6000 DNA测序仪上对S4流式细胞进行测序,产生5.86亿对端151碱基reads。采用RTA 3.3.3和bwa 0.7.12对原始数据进行处理gydF4y2Ba54gydF4y2Ba.由此产生的分子大小被计算为来自超新星的130.6 kbgydF4y2Ba55gydF4y2Ba组装。gydF4y2Ba
使用“Bionano Prep细胞培养DNA分离方案”制备DNA。收集细胞后,在植入琼脂糖之前,将细胞进行多次清洗。进行蛋白酶K消化,随后进行额外的洗涤和琼脂糖消化。使用Qubit dsDNA BR Assay试剂盒和CHEF凝胶对DNA的数量和质量进行评估。按照Bionano Prep直接标记和染色(DLS)方案标记和染色750 ng的DNA。染色后,使用Qubit dsDNA HS Assay试剂盒对DNA进行量化,并在Saphyr芯片上运行。gydF4y2Ba
Hi-C文库由Arima Genomics使用四种限制性内切酶复制生成。经过修饰的染色质消化后,标记消化的末端,近端结扎,然后纯化近端结扎的DNA。在Arima-HiC方案后,通过首先剪切然后使用SPRI珠选择大小的DNA片段制备illumina兼容的测序文库。使用Arima-HiC试剂盒中提供的Enrichment Beads对含有连接连接的大小选择片段进行富集,并使用Swift Accel-NGS 2S Plus试剂盒(P/N: 21024)试剂将其转化为illumina兼容的测序文库。连接接头后,用pcr扩增DNA,用SPRI珠纯化。纯化的DNA经过标准质量控制(qPCR和Bioanalyzer),并按照制造商的方案在HiSeq X上测序。gydF4y2Ba
纳米孔和PacBio全基因组组装gydF4y2Ba
Canu v.1.7.1gydF4y2Ba21gydF4y2Ba使用2018年11月7日或之前生成的所有rel1 Oxford Nanopore数据(on-instrument basecaller, rel1)和2014年和2015年生成的PacBio序列(Sequence Read Archive (SRA): PRJNA269593)(总覆盖率为70倍)运行。gydF4y2Ba2gydF4y2Ba,gydF4y2Ba56gydF4y2Ba.装配中的一些染色体仅在着丝粒区被破坏(例如,chr10, chr12, chr18等)。gydF4y2Ba1gydF4y2Ba).尽管几个着丝粒之间存在明显的连续性(例如chr8、chr11和chrX),但组装者报告的重复拷贝数量远低于预期。gydF4y2Ba
手动封缝gydF4y2Ba
X染色体上的缺口是通过将所有读对装配进行映射,并手动识别不包括在自动Canu装配中的连接contigs的读来关闭的。这产生了一个初始的候选染色体集合,除了着丝粒。候选装配的四个区域被发现与Bionano光学图在结构上不一致,并通过手动从这些区域中选择读取值并使用Canu进行局部重组来纠正gydF4y2Ba21gydF4y2Ba和Flye v.2.4gydF4y2Ba57gydF4y2Ba.低覆盖率的长读数自信地横跨整个重复区域,用于指导和评估可用的最终装配。使用hmm (v.3)评估重组版本和跨读之间的拷贝数和重复组织gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba在特定的串联重复单元上进行训练,并手动比较所报告的结构。Minimap2的默认参数gydF4y2Ba60gydF4y2Ba导致不均匀的覆盖和抛光精度在串联重复序列。通过将Minimap2 -r参数从500增加到10,000,并将报告的次要对准的最大数量(-N)从5增加到50,成功地解决了这个问题。通过PacBio数据集(CLR和HiFi)的映射来确定重复基本水平质量的最终评估(扩展数据图)。gydF4y2Ba7gydF4y2Ba,补充说明gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
X着丝粒中的α卫星阵列,由于其在男性基因组中作为单倍体阵列的可用性,是基因组水平上研究最好的着丝粒区域之一,具有明确的2-kb重复单位gydF4y2Ba28gydF4y2Ba物理和基因图谱gydF4y2Ba8gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba以及数组长度的预期范围gydF4y2Ba25gydF4y2Ba.我们最初生成了一个包含超长reads的alpha卫星数据库,通过用至少一个完整的一致序列标记这些readsgydF4y2Ba33gydF4y2Ba在两个方向都有171bp的标准重复,如前所述gydF4y2Ba61gydF4y2Ba.反向补全含有alpha的Reads,并使用2057 bp DXZ1重复单元用HMMER (v.3)筛选。然后,我们使用运行长度编码,其中2057 bp标准重复(定义为最小值:1957 bp,最大值:2157 bp范围内的任何重复)的运行被存储为单个数据值和计数,而不是原始运行。这允许我们将所有的读取重新定义为一系列的变体或重复,它们在大小或结构上与预期的规范重复单元不同,并在两者之间定义一个间隔。在超长读取数据中鉴定出的CHM13 DXZ1结构变异与先前发表的PacBio (CLRgydF4y2Ba50gydF4y2Ba和音响系统gydF4y2Ba22gydF4y2Ba)使用Alpha-CENTAURI,如所述gydF4y2Ba61gydF4y2Ba.结构变量的输出注释和每个读取的规范DXZ1间距被手动聚类以生成六个初始contig,其中两个已知锚定在相邻的Xp或Xq中。为了定义contigs之间的顺序和重叠,我们在从CRISPR-Cas9 Duplex-seq (CRISPR-DS)靶向重测序中获得的高质量DXZ1阵列数据中确定了所有精确匹配的21个mersgydF4y2Ba62gydF4y2Ba(补充注gydF4y2Ba8gydF4y2Ba).两个或多个21-mer之间的重叠以相等的间距指导了组装的组织。附加超长读覆盖支持contig间距(和contig结构)的正交验证,为除三个区域外的所有区域提供了高置信的重复单元计数。gydF4y2Ba
X染色体长读抛光gydF4y2Ba
我们使用了一种新颖的映射管道,使用唯一的标记在重复中放置读取。长度gydF4y2BakgydF4y2Ba子(gydF4y2BakgydF4y2Ba-mers)是从Illumina的链接reads中收集的,在修剪条形码(成对的第一个reads的前23个碱基)后。读取数据被放置在与读取数据具有最独特标记的程序集的位置。进一步筛选以排除短的和低身份的队列。这一过程在每一轮抛光后重复,在每一轮抛光后重新计算新的唯一标记和对齐。抛光过程先进行一轮Racon,然后是两轮Nanopolish和两轮Arrow。抛光后,所有先前标记的低质量基因座都显示出显著改善,除了139-140.3的覆盖率仍有下降,并被Canu使用PacBio HiFi数据生成的替代补丁组件所取代。gydF4y2Ba
全基因组长读抛光gydF4y2Ba
整个基因组的其余部分被打磨得与X染色体类似,但没有使用uniquegydF4y2BakgydF4y2Ba- m锚定。相反,使用上述参数运行了两轮Nanopolish,然后是两轮Arrow,这些参数依赖于映射质量和长度以及身份阈值来确定长读取的最佳位置。由于没有一致的努力来正确地在X染色体以外的染色体上组装大型卫星阵列,这种默认的抛光方法被认为对基因组的其余部分足够了。然而,未来的努力,以完成这些剩余的染色体,预计将受益于独特的gydF4y2BakgydF4y2Ba-mer锚定映射方法。gydF4y2Ba
全基因组短读抛光gydF4y2Ba
Illumina链接读取被用于整个组装的最终抛光,包括X染色体,但只使用明确的映射和纠正小的插入和删除错误(补充说明gydF4y2Ba4gydF4y2Ba).gydF4y2Ba
甲基化分析gydF4y2Ba
为了测量纳米孔数据中的CpG甲基化,我们使用了NanopolishgydF4y2Ba63gydF4y2Ba.Nanopolish在纳米孔电流信号上使用隐马尔可夫模型来区分5-甲基胞嘧啶和未甲基胞嘧啶。甲基化调用程序生成一个对数似然值,表示特定位置上甲基化cg与非甲基化cg的概率之比gydF4y2BakgydF4y2Ba- m。接下来,我们使用nanopore_methylation_utilities工具(gydF4y2Bahttps://github.com/isaclee/nanopore-methylation-utilitiesgydF4y2Ba),它使用2.5的对数似然比作为调用甲基化的阈值gydF4y2Ba64gydF4y2Ba.对数似然比大于2.5(甲基化)或小于- 2.5(非甲基化)的CpG位点被认为是高质量的,并被纳入分析。没有任何高质量CpG位点的Reads被排除在后续的甲基化分析中。数字gydF4y2Ba3.gydF4y2Ba显示了至少一个高质量CpG站点的读取覆盖率。Nanopore_methylation_utilities将甲基化信息集成到比对BAM文件中,以便在IGV的亚硫酸氢盐模式下查看gydF4y2Ba65gydF4y2Ba并创建俾斯麦风格的文件,然后我们用R Bioconductor包BSseq (v.1.20.0)进行分析gydF4y2Ba66gydF4y2Ba.我们使用BSmooth算法gydF4y2Ba66gydF4y2Ba在BSseq包中平滑数据,以估计特定感兴趣区域的甲基化水平。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
斯托尔斯医学研究所产生的原始数据是本手稿的基础,可以从斯托尔斯原始数据存储库访问gydF4y2Bahttp://www.stowers.org/research/publications/libpb-1453gydF4y2Ba.基因组组装和测序数据包括原始信号文件(FAST5)、事件级数据(FAST5)、基调用(FASTQ)和比对(BAM/CRAM)作为亚马逊网络服务开放数据集提供。访问数据的说明,以及对原始数据和程序集的未来更新,可从gydF4y2Bahttps://github.com/nanopore-wgs-consortium/chm13gydF4y2Ba.所有数据也存档,并可在NCBI生物项目接入下使用gydF4y2BaPRJNA559484gydF4y2Ba,包括全基因组组装(GCA_009914755.1)和完整的X染色体(CM020874.1)。gydF4y2Ba
代码的可用性gydF4y2Ba
本手稿中的分析没有使用自定义代码。所有使用的软件都是免费的:Canu,gydF4y2Bahttps://github.com/marbl/canugydF4y2Ba;BWA,gydF4y2Bahttps://github.com/lh3/bwagydF4y2Ba;Minimap2,gydF4y2Bahttps://github.com/lh3/minimap2gydF4y2Ba;箭头,gydF4y2Bahttps://github.com/PacificBiosciences/GenomicConsensusgydF4y2Ba;Nanopolish,gydF4y2Bahttps://github.com/jts/nanopolishgydF4y2Ba;HMMER,gydF4y2Bahttp://hmmer.orggydF4y2Ba;超新星,gydF4y2Bahttps://support.10xgenomics.comgydF4y2Ba;管理员,gydF4y2Bahttps://support.10xgenomics.comgydF4y2Ba;榨汁机和果汁盒,gydF4y2Bahttps://github.com/aidenlab/JuiceboxgydF4y2Ba;Flye,gydF4y2Bahttps://github.com/fenderglass/FlyegydF4y2Ba;Bioconductor,gydF4y2Bahttps://github.com/BioconductorgydF4y2Ba;Samtools,gydF4y2Bahttp://samtools.github.iogydF4y2Ba;Freebayes,gydF4y2Bahttps://github.com/ekg/freebayesgydF4y2Ba;哑剧演员,gydF4y2Bahttp://mummer.sourceforge.netgydF4y2Ba;CRISPR-DS,gydF4y2Bahttps://github.com/risqueslabgydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
Jain等人。纳米孔测序和超长读取人类基因组组装。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba36gydF4y2Ba, 338-345(2018)。gydF4y2Ba
施耐德,v.a.等。对GRCh38和从头单倍体基因组组合的评估表明了参考组合的持久质量。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 849-864(2017)。gydF4y2Ba
罗斯,m.t.等人。人类X染色体的DNA序列。gydF4y2Ba自然gydF4y2Ba434gydF4y2Ba, 325-337(2005)。gydF4y2Ba
Mefford, H. C. & Eichler, E. E.复制热点,罕见的基因组疾病和常见疾病。gydF4y2Ba咕咕叫。当今。麝猫。DevgydF4y2Ba.gydF4y2Ba19gydF4y2Ba, 196-204(2009)。gydF4y2Ba
Langley, s.a., Miga, K. H., Karpen, G. H. & Langley, C. H.单倍型跨越着丝粒区域揭示了大块古代DNA的持久性。gydF4y2BaeLifegydF4y2Ba8gydF4y2Ba, e42989(2019)。gydF4y2Ba
《伪装重复:人类基因组的谬误陷阱》。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba8gydF4y2Ba, 758-762(1998)。gydF4y2Ba
Breitwieser, F. P., Pertea, M., Zimin, A. V. & Salzberg, S. L.人类对细菌基因组的污染已经产生了成千上万的假蛋白质。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba29gydF4y2Ba954-960(2019)。gydF4y2Ba
Schueler, m.g., Higgins, a.w., Rudd, m.k, Gustashaw, K. & Willard, h.f.功能性人类着丝粒的基因组和遗传学定义。gydF4y2Ba科学gydF4y2Ba294gydF4y2Ba, 109-115(2001)。gydF4y2Ba
艾希勒,E. E.等。遗传性缺失和寻找复杂疾病潜在原因的策略。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba11gydF4y2Ba, 446-450(2010)。gydF4y2Ba
Mortazavi, A., Williams, B., McCue, K., Schaeffer, L. & Wold, B.通过RNA-seq绘制和量化哺乳动物转录组。gydF4y2BaNat方法。gydF4y2Ba5gydF4y2Ba, 621-628(2008)。gydF4y2Ba
Park, P. J. ChIP-seq:成熟技术的优势和挑战。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba10gydF4y2Ba, 669-680(2009)。gydF4y2Ba
张海燕,吴斌,张海燕,张文杰。ATAC-seq:一种检测染色质可达性的方法。gydF4y2Ba咕咕叫。Protoc。摩尔。杂志gydF4y2Ba.gydF4y2Ba109gydF4y2Ba, 21.29.1-21.29.9(2015)。gydF4y2Ba
一种利用计算机程序进行DNA测序的策略。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba6gydF4y2Ba, 2601-2610(1979)。gydF4y2Ba
Nagarajan, N. & Pop, M.序列组装去神秘化。gydF4y2BaNat. Rev. GenetgydF4y2Ba.gydF4y2Ba14gydF4y2Ba, 157-167(2013)。gydF4y2Ba
国际人类基因组测序联盟。人类基因组的初步测序和分析。gydF4y2Ba自然gydF4y2Ba409gydF4y2Ba, 860-921(2001)。gydF4y2Ba
国际人类基因组测序联盟。完成人类基因组的纯色序列。gydF4y2Ba自然gydF4y2Ba431gydF4y2Ba, 931-945(2004)。gydF4y2Ba
斯坦伯格,k.m.等。葡萄胎人类基因组的单倍型组装。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba24gydF4y2Ba, 2066-2076(2014)。gydF4y2Ba
李,H.等。一种用于精确评价变异调用的合成二倍体基准。gydF4y2BaNat方法。gydF4y2Ba15gydF4y2Ba, 595-597(2018)。gydF4y2Ba
Vollger, m.r.等人。长读序列和片段重复的组装。gydF4y2BaNat方法。gydF4y2Ba16gydF4y2Ba, 88-94(2019)。gydF4y2Ba
Alexander, D. H., Novembre, J. & Lange, K.基于模型的不相关个体祖先估计。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba19gydF4y2Ba, 1655-1664(2009)。gydF4y2Ba
柯伦,S.等人。Canu:通过自适应可伸缩和精确的长读汇编gydF4y2BakgydF4y2Ba-mer加权并重复分离。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 722-736(2017)。gydF4y2Ba
Vollger, m.r.等人。改进的组装和变异检测单倍体人类基因组使用单分子,高保真长读取。gydF4y2Ba安。嗡嗡声。麝猫gydF4y2Ba.gydF4y2Ba84gydF4y2Ba, 125-140(2020)。gydF4y2Ba
Fiddes, i.t.等人。比较注释工具箱(CAT) -同时注释分支和个人基因组。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba28gydF4y2Ba, 1029-1038(2018)。gydF4y2Ba
柴森,m.j.p.等。利用单分子测序解决人类基因组的复杂性。gydF4y2Ba自然gydF4y2Ba517gydF4y2Ba, 608-611(2015)。gydF4y2Ba
Mahtani, m.m. & Willard, h.f.人类X染色体着丝粒α -卫星DNA的脉冲场凝胶分析:高频多态性和阵列大小估计。gydF4y2Ba基因组学gydF4y2Ba7gydF4y2Ba, 607-613(1990)。gydF4y2Ba
米金,B. R. &肯尼迪,J. F.人类女性发育早期X染色体失活的证据。gydF4y2Ba点。j .的嗡嗡声。麝猫gydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 233-239(1975)。gydF4y2Ba
吴俊杰,李志刚,李志刚。人猿重复DNA的同源性研究。gydF4y2Ba自然gydF4y2Ba276gydF4y2Ba, 92-94(1978)。gydF4y2Ba
韦拉德,H. F.史密斯,K. D. & Sutherland, J.从人类X染色体一个主要串联重复家族的分离和特征。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba11gydF4y2Ba, 2017-2034(1983)。gydF4y2Ba
威拉德,H. F.韦,J. S.染色体特异性人类阿尔法卫星DNA的等级顺序。gydF4y2Ba趋势麝猫gydF4y2Ba.gydF4y2Ba3.gydF4y2Ba, 192-198(1987)。gydF4y2Ba
米加,K. H.等。人类染色体X和Y卫星阵列的着丝粒参考模型。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba24gydF4y2Ba, 697-707(2014)。gydF4y2Ba
杜菲,S. J. &威拉德,H. F.人类X染色体α卫星阵列内和阵列间序列变异的模式:串联重复DNA序列短距离均质化的证据。gydF4y2Ba基因组学gydF4y2Ba5gydF4y2Ba, 810-821(1989)。gydF4y2Ba
Wevrick, R. & Willard, H. F.人类染色体着丝粒α卫星DNA串联阵列的远程组织:高频阵列长度多态性和减数分裂稳定性。gydF4y2Ba国家科学院学报美国gydF4y2Ba86gydF4y2Ba, 9394-9398(1989)。gydF4y2Ba
韦,J. S.和威拉德,H. F. α卫星重复DNA的核苷酸序列异质性:来自不同人类染色体的alphoid序列的调查。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba15gydF4y2Ba, 7549-7569(1987)。gydF4y2Ba
Jain等人。人类着丝粒在Y染色体上的线性组合。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba36gydF4y2Ba, 321-323(2018)。gydF4y2Ba
洛曼,N. J.,奎克,J. &辛普森,J. T.一个完整的细菌基因组组装从头仅使用纳米孔测序数据。gydF4y2BaNat方法。gydF4y2Ba12gydF4y2Ba, 733-735(2015)。gydF4y2Ba
Koren, S, Phillippy, a.m., Simpson, J. T., Loman, N. J. & Loose, M.对“长读集合中的错误可以严重影响蛋白质预测”的回复。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba37gydF4y2Ba, 127-128(2019)。gydF4y2Ba
Garrison, E. & Marth, G.基于单倍型的短读测序变异检测。预印在gydF4y2Bahttps://arxiv.org/abs/1207.3907gydF4y2Ba(2012)。gydF4y2Ba
Schmutz, J.等。人类基因组序列的质量评估。gydF4y2Ba自然gydF4y2Ba429gydF4y2Ba, 365-368(2004)。gydF4y2Ba
Falconer, E.等人。单细胞DNA模板链测序在高分辨率绘制基因组重排。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 1107-1112(2012)。gydF4y2Ba
桑德斯,a.d.等人。通过单细胞测序来表征人类基因组的多态反转。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba26gydF4y2Ba, 1575-1587(2016)。gydF4y2Ba
Mikheenko, A., Bzikadze, A. V., Gurevich, A., Miga, K. H. & Pevzner, P. A.串联工具:在超长串联重复中映射长读取和评估/提高装配质量。gydF4y2Ba生物信息学gydF4y2Ba36gydF4y2Ba, i75-i83(2020)。gydF4y2Ba
兰德,a.c.等。利用高通量纳米孔测序绘制DNA甲基化图谱。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 411-413(2017)。gydF4y2Ba
卡雷尔,L.,卡托尔,A. A.,戈林,K. C.和威拉德,H. F.人类X染色体的第一代X失活剖面。gydF4y2Ba国家科学院学报美国gydF4y2Ba96gydF4y2Ba, 14440-14444(1999)。gydF4y2Ba
乔尔杰蒂,L.等。小鼠失活X染色体的结构组织。gydF4y2Ba自然gydF4y2Ba535gydF4y2Ba, 575-579(2016)。gydF4y2Ba
达罗,e.m.等人。删除gydF4y2BaDXZ4gydF4y2Ba在人类不活跃的X染色体上改变了更高阶的基因组结构。gydF4y2Ba国家科学院学报美国gydF4y2Ba113gydF4y2Ba, e4504-e4512(2016)。gydF4y2Ba
Chadwick, B. P. DXZ4染色质采用了与周围染色体相反的构象,并获得了一种新的非活性x特异性作用,涉及CTCF和反义转录本。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba18gydF4y2Ba, 1259-1269(2008)。gydF4y2Ba
唐伦,T. A.,布伦斯,G. A.,拉特,S. A.,穆赫兰,J. & Wyman, A. R.一个8号染色体富集的alphoid重复。Cytogen。gydF4y2Ba细胞创gydF4y2Ba.gydF4y2Ba46gydF4y2Ba, 607(1987)。gydF4y2Ba
葛,Y.,瓦格纳,M. J., Siciliano, M. & Wells, D. E.序列,高阶重复结构,和人类8号染色体特定的α卫星DNA的远程组织。gydF4y2Ba基因组学gydF4y2Ba13gydF4y2Ba, 585-593(1992)。gydF4y2Ba
Bzikadze, A. V. & Pevzner, P. A.从超长易出错的reads中自动组装着丝粒。gydF4y2Ba生物科技本质》gydF4y2Ba.gydF4y2Bahttps://doi.org/10.1038/s41587-020-0582-4gydF4y2Ba(2020)。gydF4y2Ba
Kronenberg, Z. N.等人。类人猿基因组的高分辨率比较分析。gydF4y2Ba科学gydF4y2Ba360gydF4y2Ba, eaar6343(2018)。gydF4y2Ba
杜特拉,A. S., Mignot, E. & Puck, J. M. FISH在狗中的基因定位和同步定位。gydF4y2BaCytogenet。细胞麝猫gydF4y2Ba.gydF4y2Ba74gydF4y2Ba, 113-117(1996)。gydF4y2Ba
Willatt, L., Morgan, S. M., Shaffer, L. G., Slovak, M. L. & Campbell, L. J. ISCN 2009人类细胞遗传学命名的国际系统。gydF4y2Ba嗡嗡声。麝猫gydF4y2Ba.gydF4y2Ba126gydF4y2Ba, 603(2009)。gydF4y2Ba
快,J. RAD004 V.3超长读取测序协议。gydF4y2Baprotocols.iogydF4y2Bahttps://doi.org/10.17504/protocols.io.mrxc57ngydF4y2Ba(2018)。gydF4y2Ba
李,H. & Durbin R.快速和准确的短读对齐与Burrows-Wheeler变换。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 1754-1760(2009)。gydF4y2Ba
Weisenfeld, n.i, Kumar, V., Shah, P., Church, d.m. & Jaffe, d.b.二倍体基因组序列的直接测定。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 757-767(2017)。gydF4y2Ba
Huddleston, J.等人。长读单倍体基因组序列数据结构变异的发现和基因分型。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 677-685(2017)。gydF4y2Ba
Kolmogorov M., Yuan J., Lin Y. & Pevzner, P. A.使用重复图的长且易出错的读取的组装。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba37gydF4y2Ba, 540-546(2019)。gydF4y2Ba
贝特曼等人。Pfam 3.1: 1313多重比对和轮廓hmm匹配大多数蛋白质。gydF4y2Ba核酸测定gydF4y2Ba.gydF4y2Ba27gydF4y2Ba, 260-262(1999)。gydF4y2Ba
基于概率推断的新一代同源搜索工具。gydF4y2Ba基因组的通知gydF4y2Ba.gydF4y2Ba23gydF4y2Ba, 205-211(2009)。gydF4y2Ba
Minimap2:核苷酸序列的成对比对。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, 3094-3100(2018)。gydF4y2Ba
Sevim, V., Bashir, A., Chin, c - s。& Miga, K. H. Alpha-CENTAURI:用长读测序评估新的着丝粒重复序列变异。gydF4y2Ba生物信息学gydF4y2Ba32gydF4y2Ba, 1921-1924(2016)。gydF4y2Ba
Nachmanson, D.等人。使用CRISPR/Cas9靶向基因组碎片可以快速高效地富集小基因组区域,并以低DNA输入(CRISPR- ds)进行超精确测序。gydF4y2Ba基因组ResgydF4y2Ba.gydF4y2Ba28gydF4y2Ba, 1589-1599(2018)。gydF4y2Ba
辛普森,J. T.等。利用纳米孔测序检测DNA胞嘧啶甲基化。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 407-410(2017)。gydF4y2Ba
李,我等人。用纳米孔测序同时分析人类细胞系上染色质可及性和甲基化。在bioRxiv预印本gydF4y2Bahttps://doi.org/10.1101/504993gydF4y2Ba(2019)。gydF4y2Ba
罗宾逊,J. T.等。整合基因组学查看器。gydF4y2BaNat。gydF4y2Ba.gydF4y2Ba29gydF4y2Ba, 24-26(2011)。gydF4y2Ba
Hansen, K. D., Langmead, B. & Irizarry, R. A. B. smooth:从全基因组亚硫酸氢盐测序reads到差异甲基化区域。gydF4y2Ba基因组医学杂志gydF4y2Ba.gydF4y2Ba13gydF4y2Ba, r83(2012)。gydF4y2Ba
Sullivan, L. L., Boivin, C. D., Mravinac, B., Song, I. Y. & Sullivan, B. A. CENP-A结构域的基因组大小与人类着丝粒上alpha卫星阵列的总大小成正比,并在癌细胞中扩展。gydF4y2Ba染色体ResgydF4y2Ba.gydF4y2Ba19gydF4y2Ba, 457-470(2011)。gydF4y2Ba
确认gydF4y2Ba
我们承认与I. Lee关于甲基化分析的对话和H. F. Willard对手稿的审查。基金资助:NIH/NHGRI R21 1R21HG010548-01和NIH/NHGRI U01 1U01HG010971 (K.H.M.);美国国立卫生研究院国家人类基因组研究所内部研究计划(s.k., a.r., v.m., a.d., g.g.b., a.m.c., n.f.h., a.y., J.C.M.和A.M.P.);通过韩国健康产业发展研究院的韩国健康技术研发项目HI17C2098 (A.R.);国家卫生研究院国家医学图书馆校内研究方案(V.A.S.和f.t.n);国立卫生研究院主任办公室共同基金(V.M.);斯托尔斯医学研究所(e.h.、T.P.和J.L.G.);Nih r01 gm124041 (b.a.s.);NIH HG002385和HG010169 (E.E.E.);E.E.E.是霍华德休斯医学研究所的研究员; National Library of Medicine Big Data Training Grant for Genomics and Neuroscience 5T32LM012419-04 (M.R.V.); NIH 1F32GM134558-01 (G.A.L.); NIH/NHGRI U54 1U54HG007990, W. M. Keck Foundation DT06172015, NIH/NHLBI U01 1U01HL137183 and NIH/NHGRI/EMBL 2U41HG007234 (B.P.); NIH/NHGRI R01 HG009190 and NIGMS T32 GM007445 (W.T. and A.G.); NIH R01CA181308 (R.R.); NIH/NHGRI 2R44HG008118 (A.D.S. and S.S.); Wellcome Trust (212965/Z/18/Z) (N.H., N.J.L. and M.L.); and National Institute for Health Research (NIHR) Surgical Reconstruction and Microbiology Research Centre (SRMRC) (J.Q.). The views expressed are those of the author(s) and not necessarily those of the NIHR or the Department of Health and Social Care. This work used the computational resources of the NIH HPC Biowulf cluster (https://hpc.nih.govgydF4y2Ba).gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
s.b.、g.a.l.、k.t.、v.m.、g.g.b.、m.y.d.、d.c.s.、r.s.、g.k.、n.h.、m.l.、a.y.、J.C.M.和E.E.E.进行了CHM13纳米孔测序、细胞系制备和原始数据分析。A.Y.和J.C.M.进行了10倍的全基因组测序和组装。B.A.S.进行PFGE Southern blotting阵列尺寸分析。m.k., c.m., r.f., T.A.G.L.和I.H.生成Bionano数据并进行数据分析。J.F.和R.R.进行CRISPE-DS分析。e.h.、T.P.和J.L.G.进行ddPCR和SKY分析。e.p., a.d., e.h., T.P.和J.L.G.进行CMH13细胞系核型分析。A.B.W.和E.E.E.进行了外加剂分析。K.H.M.进行了重复表征和卫星DNA组装。k.h.m., s.k., M.R.V, A.M.C.和A.M.P.进行了自动和手动装配。 K.H.M., S.K., A.R., M.R.V., G.A.L., D.P., J.W., W.C., K.H., E.E.E. and A.M.P. performed assembly curation and validation. S.K., A.R. and A.M.P. performed marker-based assembly polishing. A.G. and W.T. performed methylation analysis. A.B. and P.A.P. generated automated satellite DNA assemblies. A.D.S., J.-M.B. and S.S. performed Hi-C CHM13 sequencing. A.R. performed Hi-C analysis. N.F.H. performed structural variant analysis. J.A. and B.P. performed annotation analysis. V.A.S. and F.T.-N. performed alignment versus RefSeq, repeat characterization and frameshift analysis. U.S. provided access to critical resources. J.Q. developed the initial ultra-long-read protocol and updated to current chemistry. N.J.L. provided an Amazon Web Services (AWS) account and coordinated data sharing. K.H.M., S.K., A.R., M.R.V. and A.M.P. developed figures. K.H.M. and A.M.P. coordinated the project. K.H.M., S.K. and A.M.P. drafted the manuscript. All authors read and approved the final manuscript.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
E.E.E.是DNAnexus的科学顾问委员会成员。k.h.m., S.K.和W.T.收到了旅费在牛津纳米孔组织的研讨会上发言。W.T.拥有两项专利授权给牛津纳米孔(美国专利8,748,091项和美国专利8,394,584项)。a。d。s。j。m。b。s。s。是Arima基因公司的员工。R.R.拥有NanoString Technologies的股权,并且是与TwinStrand Biosciences签订的NIH SBIR转包研究协议的主要研究员。所有其他作者都没有竞争利益需要申报。gydF4y2Ba
额外的信息gydF4y2Ba
同行评审信息gydF4y2Ba自然gydF4y2Ba感谢Tomi Pastinen, Steven Salzberg和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
扩展数据图1 CHM13的光谱核型分析证实为正常的46,XX核型。gydF4y2Ba
一个gydF4y2Ba, CHM13细胞系第10传代染色体和核型。用秋水胺处理的细胞制备有丝分裂中期扩散,并按照方法中的详细处理。光谱核型分析显示正常。46, XX染色体组型。有代表性的核型显示从一个10个传播分析,所有10个报告有类似的结果。比例尺,10 μm。gydF4y2BabgydF4y2Ba, CHM13 g带核型。共20个CHM13中期扩增被独立鉴定,均表现出相似的正常46,XX女性核型,如图所示。gydF4y2Ba
扩展数据图2 CHM13的推断祖先。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba,每个聚类解释的祖先比例,由使用admix估计gydF4y2BaKgydF4y2Ba= 6 (gydF4y2Ba一个gydF4y2Ba)或gydF4y2BaKgydF4y2Ba= 9 (gydF4y2BabgydF4y2Ba),从每个群体和CHM13中随机抽取10个个体。分析基于1964个来自1KG和SGDP的无关联个体。CHM13以红色字体和黑色边框突出显示。gydF4y2Ba
扩展数据图3以CHM13为参考描述结构变异的结果。gydF4y2Ba
对于CHM13(暗红色或红色)和GRCh38(黑色或灰色),汇编的大型插入和删除需要四个长读程序集。使用CHM13作为参考产生了插入和删除的平衡计数,而使用GRCh38时观察到过多的插入调用,这表明GRCh38可能存在删除偏差。SVs,结构变体。gydF4y2Ba
扩展数据图4 reads和assembly中的端粒长度。gydF4y2Ba
组装端粒大小与reads中观察到的较大尺寸一致。读取端粒长度较短的峰值可能是过早读取末端的人工产物,而不是真正的端粒末端。ONT,牛津纳米孔技术;PB,太平洋生物科学公司。gydF4y2Ba
扩展数据图5 x心粒卫星阵列(DXZ1)组件的结构评估。gydF4y2Ba
一个gydF4y2BaX染色体上的卫星阵列(DXZ1)在序列水平上被定义为一个数兆酶大小的α卫星DNA阵列。DXZ1阵列的标准重复序列由12个不同的单体定义,这些单体被有序地形成一个更大的约2-kb的重复单元,称为“高阶重复”(HOR)(灰色显示,HOR为黑色,圆圈代表12个约171-bp的单体)。这些HORs被串联排列成一个大型的、数兆酶大小的卫星阵列(先前发表的PFGE-Southern估计平均为3 Mb),在HOR重复结构中有有限数量的重排(如黄色所示的5个mer变体的缺失)和重复拷贝之间的核苷酸差异。我们的组装策略最初识别和注释了所有不间断的“规范”重复序列的从头到尾串联阵列,以及在我们的DXZ1文库中读取的每个纳米孔中的结构变异位点(gydF4y2Ba方法gydF4y2Ba).规范重复序列与侧翼结构变异的间隔决定了读取之间的精确对齐。contig是通过对这些独特位置的超长读取的共识来生成的。gydF4y2BabgydF4y2BaT2T-X CHM13序列最初被分割成7个结构变异确定的contigs。contig之间的排序和重叠使用Duplex-seq DXZ1 kmers的共享位置和低覆盖(即1-2个读取)支持超长数据,自信地跨越contig排序。三个区域(用星号标记)仅由单核苷酸变体重叠确定。我们通过实施一种正交方法centroFlye改进了这些重叠的预测,该方法研究DXZ1纳米孔reads中单个不同的位置,以指导接合物之间重叠的最终定位(并确认最靠近p臂的区域存在重叠)。gydF4y2BacgydF4y2Ba与DXZ1高阶重复变频在纳米孔超长读数据中HiFi长读PacBio数据高度一致。使用Alpha-CENTAURI在HiFi数据集中预测DXZ1重复单元变体gydF4y2Ba61gydF4y2Ba.DXZ1重复单元如箭头所示,由12个较小的约171-bp重复单元(箭头内的小圆圈表示)组成。我们总共鉴定出7316个含有dxz1的HiFi reads。我们描述了一个包含38184个(98.2%)全长DXZ1典型12位重复序列和691个具有不同重复结构的HORs(1.8%)的数据库。从标准重复单元的变化用虚线表示,每个结构变体标记一种颜色,其在阵列组件中的位置在上面表示(有序p臂到qhifi臂)。大多数读数被确定为纯DXZ1-alpha卫星(7,305/7,316,或99.85%)。在剩余的读取中,有10个读取提供了从DXZ1过渡到我们程序集中的单个L1Hs插入的证据。由于存在902 bp的均聚物([G]n),我们只鉴定了一个无法分配到我们的组装中的读数,这可能是一个测序假象。gydF4y2BadgydF4y2Ba,为了便于说明,重构了最小平铺路径(如图2所示)。gydF4y2Ba2gydF4y2Ba),并不是最初组装的机制。gydF4y2BaegydF4y2Ba, DXZ1读重叠组件采用结构变型重叠和定位。读取id和长度从Xp到Xq:(1) ab9c12a7-08db-4524-8332-373129eaa4fb, 442,119 bp。(2) 063fca09-81fc-4c2d-81ad-16fb2bfee76f, 364,710 bp。(3) 3d0fa869-028f-45be-be41-b2487897bb25, 380,361 bp。(4) a5cf4e19-8eff-4035-8238-ae81963b854f, 362052 bp。(5) c6f29ca1-d84d-4881-9042-dfb37bc9f111, 482907 bp。(6) 1ccd919f-5726-4d79-8cfe-fe2b344070a1, 275,718 bp。(7) e39308c6-0c73-45d5-9b8d-7f764af858be, 351045 bp。(8) 86ac29ba-5a93-4c08-aa18-c07829a5b696, 393,007 bp。(9) 64d464d1-f317-4dff-a259-de6097a5cd4c, 221,510 bp。 (10) 08e000a1-69dd-40fb-9fd1-942f159ec6b7, 262,585 bp. (11) 1ef64f71-9477-4a5b-bf7e-a356785cc656, 421,096 bp. (12) a1e01c13-7ca1-4dc5-85b1-6b69ec2124f9, 371,129 bp.
扩展数据图6 DXZ1阵列PFGE Southern blotting评价。gydF4y2Ba
采用PFGE和Southern blotting方法估计Alpha卫星阵列大小gydF4y2Ba25gydF4y2Ba,gydF4y2Ba67gydF4y2Ba.在大约3.1 Mb的硅片摘要中,DXZ1阵列预计将产生三个具有完整BglI摘要的波段:约659 kb,约2153 kb和约294 kb,这与BglI的重复PFGE Southern实验一致(约2.1 Mb,约0.7 Mb和约0.3 Mb)。在BstEII的硅晶摘要中提供了6个波段的证据,其中3个波段小于约200 kb且低于检测范围(用灰色波段标记)。其余三个条带再次与观察到的BstEII PFGE-Southern重复序列一致(约1.8 Mb,约0.7 Mb和约0.3 Mb)。内部控制包括HAP1和DLD1。这个实验重复了七次,得到了相似的结果。gydF4y2Ba
扩展数据图7初始抛光降低了最大重复次数内的装配质量。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba, GAGE位点的初始Canu组装(gydF4y2Ba一个gydF4y2Ba)由于标准的长读抛光(箭头,纳米抛光)而进一步损坏(gydF4y2BabgydF4y2Ba).黑点是主等位基因的覆盖率,红点是次等位基因的覆盖率(PacBio CLR数据)。CHM13基因组是有效的单倍体,因此一个等位基因是预期的。低覆盖率区域或增加的次级等位基因频率表明低质量区域或潜在的重复崩溃。由于在抛光过程中读取的错误映射,等位基因覆盖变得不均匀。采用改良抛光工艺,使用独特gydF4y2BakgydF4y2Ba-mer策略,纠正这种效果。gydF4y2BacgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba,左侧图为打磨前的装配图。右边的图显示了unique后的相同区域gydF4y2BakgydF4y2Ba-mer辅助抛光(racon, 2轮纳米抛光,2轮箭头,2轮10X)。区域为GAGE位点(48.6 ~ 49 Mb) (gydF4y2BacgydF4y2Ba), 70.8-71.3 Mb (gydF4y2BadgydF4y2Ba), 138.6-139.7 Mb (gydF4y2BaegydF4y2Ba)及cenX (57-61 Mb) (gydF4y2BafgydF4y2Ba).gydF4y2BaggydF4y2Ba- - - - - -gydF4y2BajgydF4y2Ba,相同的位点gydF4y2BacgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba但使用PacBio HiFi而不是CLR映射。gydF4y2Ba
扩展数据图8使用存在于CHM13 X染色体上的独特(单拷贝)序列的标记辅助映射提高了抛光效果。gydF4y2Ba
一个gydF4y2Ba来自10X Genomics的21个mer分布读到。用Meryl收集21个mers,用GenomeScope1.0生成图,以可视化和确认CHM13的单倍体性质和基因组大小(len)。计数在5 ~ 58(含)之间的k-mers被用作打磨X染色体时的唯一标记。gydF4y2BabgydF4y2BaPacBio CLR(黑色)、HiFi(蓝色)和超长(绿色)的覆盖直方图横跨整个X染色体。读取的过滤使用与抛光相同的基于唯一标记的过滤。gydF4y2BacgydF4y2Ba,绘制的纳米孔读数显示整个X染色体的均匀覆盖。读取的过滤使用与抛光相同的基于唯一标记的过滤。标记密度显示在读取对齐的下方。gydF4y2BadgydF4y2Ba,染色体X组装的Strand-seq验证。每条同源染色体上只有一条模板链。源于这种单链DNA的测序读取具有方向性,这一特征可用于评估个体同源体的长范围相邻性。在单链DNA遗传的基础上,我们区分了三种可能的链态:WW -同系物都继承了Watson模板链,CC -同系物都继承了Watson模板链,WC -同系物一个继承了Watson模板链,另一个继承了Crick模板链。通过跟踪每条染色体上链态的变化,我们能够精确定位表明基因组误组装的反复链态变化的位置。我们总共分析了57个strand -seq库,并映射了28个局部链状态变化。这些链状态的变化是沿着染色体X组装随机分布的,因此表明在DNA复制过程中发生了双链断裂,而不是真正的基因组误组装。这种断裂通常由可用的姐妹染色单体修复,因此经常导致链方向的改变。黑色星号表示小的局部链状态变化。这样的事件要么是由Strand-seq库准备所固有的噪声读取引起的,要么是发生在彼此非常接近的两个双链中断。gydF4y2BaegydF4y2Ba由于双链断裂不太可能在多个单细胞中完全相同的位置发生,因此在strand- seq数据中可以看到,在给定的contig或支架的相同位置上,链状态的周期性变化是真正的基因组误组装。在CHM13染色体X组合中没有观察到这些特征。gydF4y2Ba
扩展数据图9 X染色体的Hi-C读映射。gydF4y2Ba
整个X显示在左边,右边在DXZ4轨迹上放大。热图显示DXZ4周围有清晰的边界,表明DXZ4分隔了两个大的超域。gydF4y2Ba
图10 8号染色体(D8Z2)上中心粒卫星阵列装配的甲基化估计(chr8: 43,281,085-45,333,062)。gydF4y2Ba
甲基化值通过平滑频率数据计算,窗口大小为500个核苷酸。所显示的读覆盖依赖于我们唯一的锚映射,以及在读|日志上至少存在一个高质量的甲基化调用——可能性为| > 2.5。与我们之前对X染色体着丝粒卫星阵列(DXZ1)的甲基化分析类似,我们在8号染色体的着丝粒(chr8: 44,830,000-44,900,000)中观察到一个未甲基化区域(约75 kb)。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
该文件包括补充注释1-8,详细分析了主要文本,补充表1提供了基因组注释结果,补充表2提供了反转调用,补充表3提供了NCBI中所有人类基因组组件的描述,contig NG50 >25 Mb或源自CHM13;补充表4提供了DXZ1阵列估计,补充表5列出了BioNano光学地图识别的结构变体,以及其他参考文献(详情见内容)。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
米加,k.h.,柯伦,S,瑞,A。gydF4y2Baet al。gydF4y2Ba一个完整的人类X染色体端粒到端粒的组装。gydF4y2Ba自然gydF4y2Ba585gydF4y2Ba, 79-84(2020)。https://doi.org/10.1038/s41586-020-2547-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-020-2547-7gydF4y2Ba
这篇文章被引用gydF4y2Ba
使用肿瘤-正常配对参考样本进行精确的癌症体细胞突变发现的个性化基因组组装gydF4y2Ba
基因组生物学gydF4y2Ba(2022)gydF4y2Ba
INSERT-seq能够使用纳米孔测序对基因组整合DNA进行高分辨率测绘gydF4y2Ba
基因组生物学gydF4y2Ba(2022)gydF4y2Ba
端粒到端粒组装时代的多基因组对齐gydF4y2Ba
基因组生物学gydF4y2Ba(2022)gydF4y2Ba
识别和纠正端粒纳米孔测序中的重复调用错误gydF4y2Ba
基因组生物学gydF4y2Ba(2022)gydF4y2Ba
斑马鱼基因组的长读测序重组了基因组结构gydF4y2Ba
BMC基因组学gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba