摘要gydF4y2Ba
同源重组是精确修复双链DNA断裂(dsb)的关键。gydF4y2Ba1克ydF4y2Ba.最初,RecBCD复合体gydF4y2Ba2gydF4y2Ba将DSB的末端切成3 '单链DNA, RecA细丝在其上组装gydF4y2Ba3 gydF4y2Ba.接下来,灯丝将同源修复模板定位在姐妹染色体上gydF4y2Ba4gydF4y2Ba.在这里,我们使用高通量微流体和荧光显微镜直接可视化单细胞中dsb的修复。我们发现gydF4y2Ba大肠杆菌gydF4y2Ba,分离姐妹位点间dsb的修复在15±5min(平均±s.d)内完成,适应度损失最小。我们进一步表明,搜索需要不到9±3分钟(平均±s.d),并由一个薄的,高度动态的RecA灯丝贯穿整个细胞介导。我们提出RecA灯丝的结构有效地降低了搜索维数。该模型预测的搜索时间与我们的测量结果一致,并且通过观察到搜索时间不依赖于细胞的长度或DNA的数量来证实。考虑到RecA同系物的丰富gydF4y2Ba5gydF4y2Ba在美国,我们相信这种模式在生物体中是广泛保守的。gydF4y2Ba
主要gydF4y2Ba
为了研究活细菌中的同源重组机制,我们创建了一个诱导DSB系统,包括(1)一个诱导Cas9核酸酶,在特定的染色体位置(“切割位点”)产生DSB,(2)一个荧光体gydF4y2Ba帕尔斯gydF4y2Ba/ mCherry-ParB系统(以下简称ParB)来可视化染色体断裂位置,以及(3)sos反应gydF4y2Ba6克ydF4y2Ba报告者选择进行DSB修复的细胞(图;gydF4y2Ba1gydF4y2Ba、扩展数据图gydF4y2Ba1gydF4y2Ba)。我们使用了微流体母机的变种gydF4y2Ba7gydF4y2Ba这允许短暂的Cas9核酸酶诱导(图。gydF4y2Ba1gydF4y2Ba扩展数据图gydF4y2Ba1 bgydF4y2Ba)。gydF4y2Ba
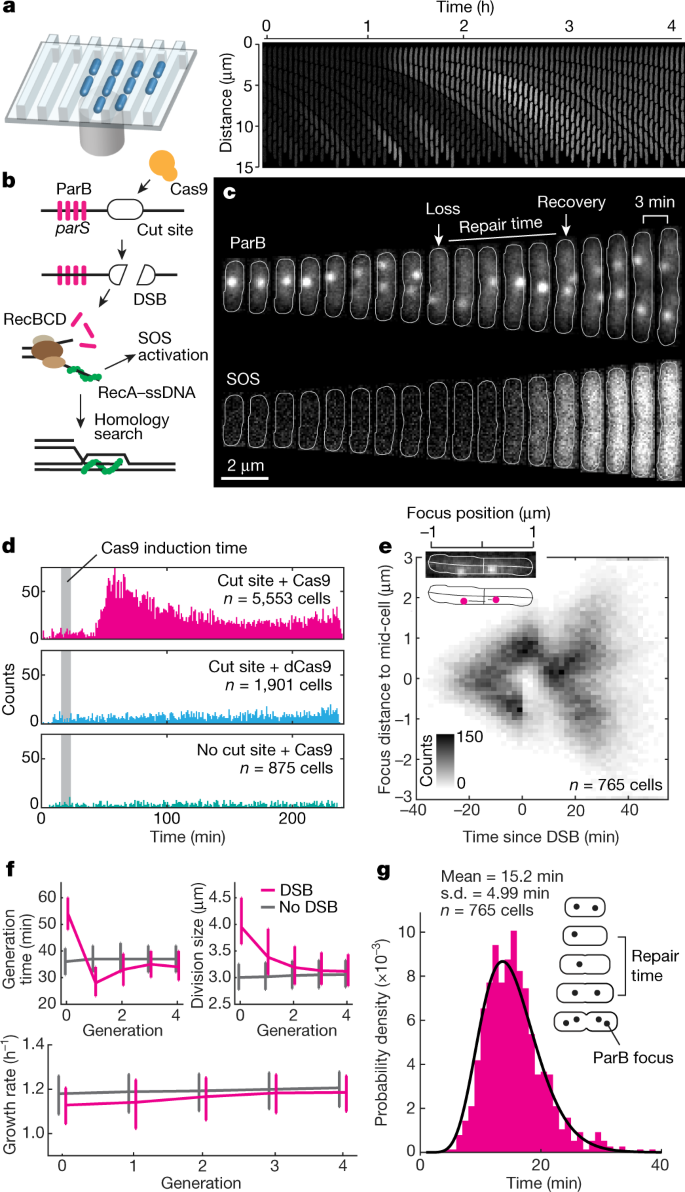
一个gydF4y2Ba左图是用来培养细胞的母机装置。右,单个生长通道的蒙太奇显示在dsb诱导后SOS激活。gydF4y2BabgydF4y2Ba,卡通显示DSB的形成、加工和修复。Cas9结合到切割部位并创建一个DSB。接下来,RecBCD与DNA结合,在DSB位点结束,并开始末端处理,弹出ParB蛋白并生成3 ' ssDNA尾巴,RecA与此结合并诱导SOS反应。RecA-ssDNA丝寻找同源性,在同源性被定位后,DSB被修复。gydF4y2BacgydF4y2Ba,进行DSB修复的细胞。ParB焦点的丢失和恢复被注释。单元格轮廓用白线显示。gydF4y2BadgydF4y2Ba,单个细胞中SOS诱导的时间,定义为Cas9诱导开始到SOS报告器发出的CFP信号达到最大信号一半的时间。只有CFP信号增加4倍以上的细胞被绘制出来。数据来自四个(上)或三个(中,下)实验。gydF4y2BaegydF4y2Ba, DSB修复过程中ParB病灶沿细胞长度的定位。细胞定向,使剩余的ParB焦点位于顶部。时间由ParB焦点损失对齐,如注释中所示gydF4y2BacgydF4y2Ba.插入图显示单个细胞的ParB通道与轮廓和主干(上)叠加,以及焦点沿细胞主干的位置映射(下),gydF4y2BangydF4y2Ba= 717个细胞,来自4个实验。gydF4y2BafgydF4y2Ba,单个DSB及其后代细胞修复时的生成时间(左上)、分裂时的大小(右上)和生长速率(下)。参考(无DSB)样本由在最大DSB诱导时间窗内出生的无DSB的细胞组成。这条线表示中位数,误差条表示第一和第三个四分位数,gydF4y2BangydF4y2Ba= 60405个细胞,来自4个实验。gydF4y2BaggydF4y2Ba, DSB修复时间,gamma分布拟合数据,gydF4y2BangydF4y2Ba= 765个细胞,4个实验。gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
DSB修复快速准确gydF4y2Ba
DSB形成后,RecBCD进行末端处理,去除断口附近的ParB标记gydF4y2Ba8gydF4y2Ba,并通过激活sos响应报告器(图。gydF4y2Ba1 bgydF4y2Ba)。Cas9脉冲在染色体切割位点的细胞中诱导特定的dsb,在诱导后不久,伴随着sos激活细胞比例的增加(图。gydF4y2Ba1 c, dgydF4y2Ba扩展数据图gydF4y2Ba2gydF4y2Ba)。在没有切割位点的细胞中表达Cas9或表达催化死亡的Cas9 (dCas9)不会诱导SOS反应(图2)。gydF4y2Ba1 dgydF4y2Ba扩展数据图gydF4y2Ba2 bgydF4y2Ba)。SOS反应的激活依赖于同源重组,在缺失的细胞中不存在gydF4y2BarecAgydF4y2Ba或gydF4y2BarecBgydF4y2Ba基因(扩展数据图。gydF4y2Ba2 bgydF4y2Ba)。由于dsb而丢失的ParB焦点在野生型细胞中被恢复和分离,但在缺乏关键重组成分的细胞中却没有(扩展数据图)。gydF4y2Ba2 c fgydF4y2Ba)。这些结果表明,诱导的dsb可以通过同源重组得到修复。修复是非常稳健的:95.5%(468个中有447个)保留了未裂解模板的细胞修复了DSB,随后分裂。在没有修复模板的细胞中,修复功能受损,因为在4小时的实验中,没有一个细胞在切割部位的所有副本上分裂(gydF4y2BangydF4y2Ba= 27个细胞,3次实验)。gydF4y2Ba
接下来,我们重点研究了修复过程中切口位点的动态变化。通常,在DSB后,未切割的位点首先易位到细胞中部,然后分裂成两个病灶,随后分离(图2)。gydF4y2Ba1 cgydF4y2Ba)。当我们沿着细胞长度绘制ParB焦点的位置与相对于DSB事件的时间的关系时,这种模式是可见的。gydF4y2Ba1 egydF4y2Ba)。然后,我们测量了单个细胞的修复时间,即ParB焦点丢失和再现之间的时间(图2)。gydF4y2Ba1 cgydF4y2Ba)。我们将分析限制在DSB之前有两个分离的ParB病灶的细胞。我们发现DSB在15.2±5.0 min内修复。gydF4y2Ba1克gydF4y2Ba)。这些结果在重复之间是一致的(扩展数据图。gydF4y2Ba二维gydF4y2Ba),特别是当I-SceI代替Cas9诱导断裂时(扩展数据图。gydF4y2Ba2 egydF4y2Ba),或将ParB标记替换为gydF4y2Ba不全gydF4y2Ba由MalI - venus蛋白结合的阵列(称为“MalI”)(扩展数据图。gydF4y2Ba3gydF4y2Ba)。当DSB的一侧是ParB,另一侧是MalI时,两端同时处理,并遵循相同的动力学(扩展数据图。gydF4y2Ba3 bgydF4y2Ba)。考虑到修复时间只是生成时间的一小部分(这里是35±10分钟),我们测试了它是否对适应度有负面影响。单次DSB将分裂延迟至55±10 min;然而,这种延迟在接下来的几代中被更快的分裂所补偿。gydF4y2Ba1 fgydF4y2Ba)。在接受DSB修复的细胞中,生长速率暂时降低了4%。gydF4y2Ba1 fgydF4y2Ba)。gydF4y2Ba
遥远同源间的配对gydF4y2Ba
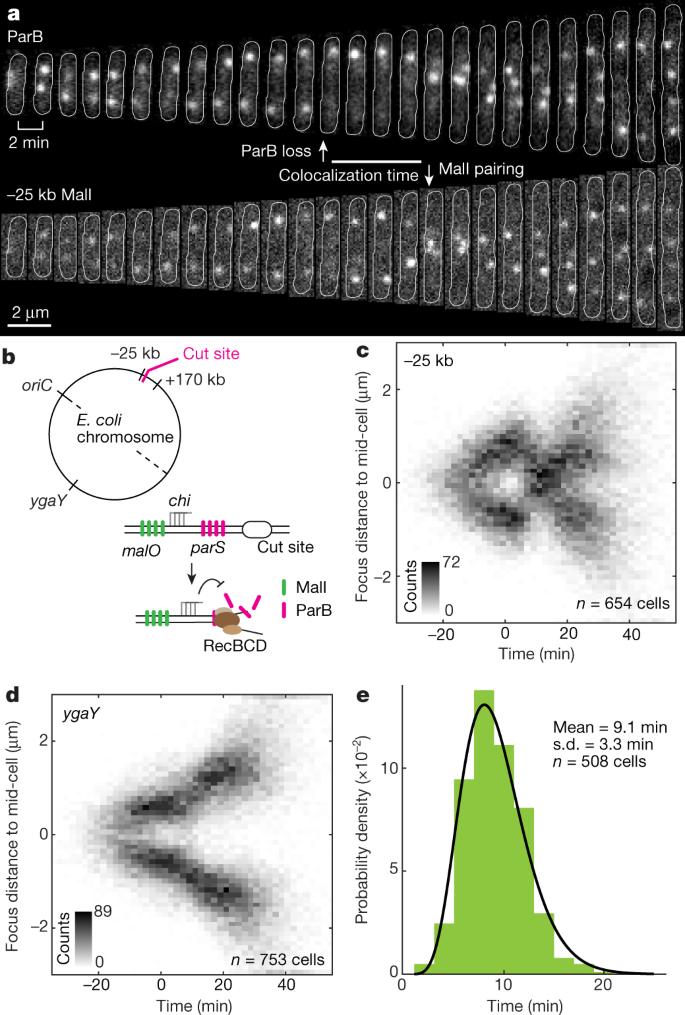
ParB病灶的丢失阻碍了对切割位点位点的观察。因此,为了可视化断裂点的动态,我们使用了一组在RecBCD未处理的距离上集成的MalI标记gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba(距离被切割的位置为- 25kb或+ 170kb)gydF4y2Ba2 bgydF4y2Ba)。两个MalI标记中的任何一个成像显示,在DSB后,两个姐妹位点都移动到细胞中部,在那里它们共定位(图2)。gydF4y2Ba2, cgydF4y2Ba扩展数据图gydF4y2Ba4 a, cgydF4y2Ba)。姐妹MalI标记的运动是对称的,不像先前报道的动态,在DSB后,分裂的姐妹位点移动得更远gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba.为了测试姐妹位点的共定位是特异性的还是由染色体的整体排列引起的,我们使用了一个整合在染色体另一侧的远端MalI标记(gydF4y2BaygaYgydF4y2Ba)。遥远的gydF4y2BaygaYgydF4y2Ba标记在修复过程中保持其典型位置(图;gydF4y2Ba二维gydF4y2Ba扩展数据图gydF4y2Ba4 bgydF4y2Ba),排除了同源重组修复诱导整个染色体配对的模型。相反,它似乎共定位是特定的裂切部位及其同源性。由于姐妹位点在通过搜索定位之前与其他染色体位点没有区别,我们得出结论,姐妹位点的共定位意味着完成同源性搜索(扩展数据图)。gydF4y2Ba4 egydF4y2Ba)。−25 kb标记的共定位时间为9.1±3.3 min。gydF4y2Ba2 egydF4y2Ba),因此同源搜索速度比此更快。gydF4y2Ba
一个gydF4y2Ba,具有−25 kb MalI标记的细胞正在进行DSB修复。对ParB焦点丢失和MalI焦点共定位进行了标注。单元格轮廓显示为一条白线。gydF4y2BabgydF4y2Ba,顶部,显示圆形的卡通gydF4y2Ba大肠杆菌gydF4y2Ba染色体与插入切位点的位置和gydF4y2Ba不全gydF4y2Ba/马里标记。下图,卡通显示了DSB在加工过程中的存在性gydF4y2Ba不全gydF4y2Ba受保护的数组gydF4y2Ba气gydF4y2Ba网站。的gydF4y2Ba气gydF4y2Ba位点阻止RecBCD在到达之前降解DNAgydF4y2Ba不全gydF4y2Ba网站。gydF4y2BacgydF4y2Ba,在进行DSB的细胞中定位−25 kb的MalI病灶。对于每个细胞,时间以ParB焦点损失对齐。gydF4y2BangydF4y2Ba= 654个细胞,来自3个实验。gydF4y2BadgydF4y2Ba,因为gydF4y2BacgydF4y2Ba,但对于gydF4y2BaygaYgydF4y2BaMalI标记,在与切割部位相对的染色体臂上。gydF4y2BangydF4y2Ba= 753个细胞,2个实验。gydF4y2BaegydF4y2Ba,在DSB修复过程中,−25 kb MalI标记的MalI焦点共定位时间分布。实线表示gamma与数据的拟合。gydF4y2BangydF4y2Ba= 508个细胞从3个实验。gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
RecA细丝薄且动态gydF4y2Ba
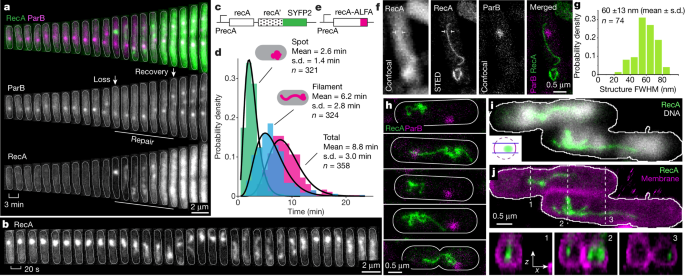
同源性搜索由组装在单链DNA (ssDNA)上的RecA细丝介导。gydF4y2Ba3 gydF4y2Ba荧光RecA融合形成的结构之前已经成像gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba,gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba.我们使用与野生型结合的RecA黄色荧光蛋白(YFP)融合来观察DSB修复过程中的RecA活性gydF4y2BarecAgydF4y2Ba,一个结构被发现是完全活跃的(图。gydF4y2Ba3 cgydF4y2Ba扩展数据图gydF4y2Ba5 b, ggydF4y2Ba)。一个类似的结构之前已经被证明具有完全的功能gydF4y2Ba14gydF4y2Ba但还没有被用来描述遥远的修复事件。gydF4y2Ba
一个gydF4y2Ba在DSB修复期间,RecA-YFP(绿色)和ParB(洋红色)。单元格轮廓用白线显示。gydF4y2BabgydF4y2Ba, DSB修复过程中RecA-YFP的快速延时。gydF4y2BacgydF4y2Ba, RecA-RecA-YFP串联结构插入gydF4y2BarecAgydF4y2Ba轨迹。gydF4y2BadgydF4y2Ba, RecA结构的寿命。实线表示gamma与数据的拟合。gydF4y2BangydF4y2Ba= 3次实验,所分析的细胞数量显示。gydF4y2BaegydF4y2Ba, RecA-ALFA构造插入gydF4y2BarecAgydF4y2Ba轨迹。gydF4y2BafgydF4y2Ba,在共聚焦和STED显微镜下成像的具有扩展RecA-ALFA结构的细胞。白色标记之间的强度分布见扩展数据图。gydF4y2Ba6 egydF4y2Ba.gydF4y2BaggydF4y2Ba,所测RecA-ssDNA丝的厚度分布如图扩展数据图所示。gydF4y2Ba6 egydF4y2Ba.gydF4y2BangydF4y2Ba2次实验= 74。gydF4y2BahgydF4y2Ba,具有RecA-ALFA结构的细胞的STED图像。白线表示单元格轮廓。gydF4y2Ba我gydF4y2Ba, RecA-ALFA三维STED图像叠加Pico488染色DNA共焦图像。只显示了中间的400纳米部分(插图)。gydF4y2BajgydF4y2Ba,上,覆盖尼罗红细胞膜的RecA-ALFA 3D STED图像。底部,高亮位置的横截面。gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
诱导dsb导致gydF4y2BarecBgydF4y2Ba在DSB位点依赖于RecA结构的形成(图;gydF4y2Ba3gydF4y2Ba扩展数据图gydF4y2Ba5 fgydF4y2Ba)。这些结构出现时间为35±98 s(扩展数据图;gydF4y2Ba5 hgydF4y2Ba),并在6.6±5.2 min后拆解(扩展数据图;gydF4y2Ba5我gydF4y2Ba)在修复完成之前,由ParB病灶分离定义。SOS响应由组装在ssDNA上的RecA灯丝激活gydF4y2Ba3 gydF4y2Ba.我们预测,如果结构是RecA-ssDNA细丝,它们的寿命将与SOS响应的强度相关。事实确实如此(皮尔逊的gydF4y2BargydF4y2Ba= 0.36,gydF4y2BaPgydF4y2Ba= 3 × 10gydF4y2Ba−12gydF4y2Ba扩展数据图gydF4y2Ba5 d, egydF4y2Ba),我们确信该结构为RecA-ssDNA丝状结构。gydF4y2Ba
RecA结构在几十秒的时间尺度上是高度动态的(图。gydF4y2Ba3 bgydF4y2Ba)。平均寿命为8.8±3.0 min。gydF4y2Ba3 dgydF4y2Ba),在这段时间内,它们表现出两种形式:在DSB处的初始点(存在2.6±1.4 min),强度增加(扩展数据图。gydF4y2Ba5gydF4y2Ba),提示在ssDNA上加载RecA;以及从初始点挤出并延伸到整个电池的灯丝(存在6.2±2.8分钟)。gydF4y2Ba
据报道,在细菌DSB修复过程中,RecA形成束状结构gydF4y2Ba10gydF4y2Ba.为了测试我们是否可以观察到这种结构,我们使用激发发射耗竭超分辨率显微镜(STED)对标记有ALFA表位的RecA进行成像gydF4y2Ba15gydF4y2Ba.单独的RecA-ALFA在DSB修复中完全起作用(扩展数据图。gydF4y2Ba5 b, ggydF4y2Ba)。进行DSB修复的细胞免疫染色显示,RecA形成薄的丝状结构(图。gydF4y2Ba3 f、hgydF4y2Ba扩展数据图gydF4y2Ba6gydF4y2Ba),它们与细胞膜无关,相反,它们出现在细胞的中心区域(图。gydF4y2Ba3 i, jgydF4y2Ba扩展数据图gydF4y2Ba6罪犯gydF4y2Ba,补充视频gydF4y2Ba1克ydF4y2Ba)。用成像系统的点扩散函数(PFS)对观测到的60±13 nm半最大全宽(FWHM)进行反褶积,得到丝宽为37.5±23.5 nm(图5)。gydF4y2Ba3 ggydF4y2Ba扩展数据图gydF4y2Ba6 e, fgydF4y2Ba)。正如预期的那样,鉴于RecA-YFP结构在活细胞中的高流动性,固定细胞表现出各种各样的构象,包括复杂的、纠结的线或横跨细胞长度的单一的、缠绕的细丝(图2)。gydF4y2Ba3 hgydF4y2Ba)。值得注意的是,当免疫染色RecA-YFP的串联结构时,我们观察到相同类型的结构。gydF4y2Ba6克gydF4y2Ba),或RecA-ALFA, RecA-YFP串联结构(扩展数据图。gydF4y2Ba6 h-kgydF4y2Ba)。这些结果表明,同源性搜索是通过薄而灵活的RecA细丝进行的。gydF4y2Ba
RecA降低了搜索的维数gydF4y2Ba
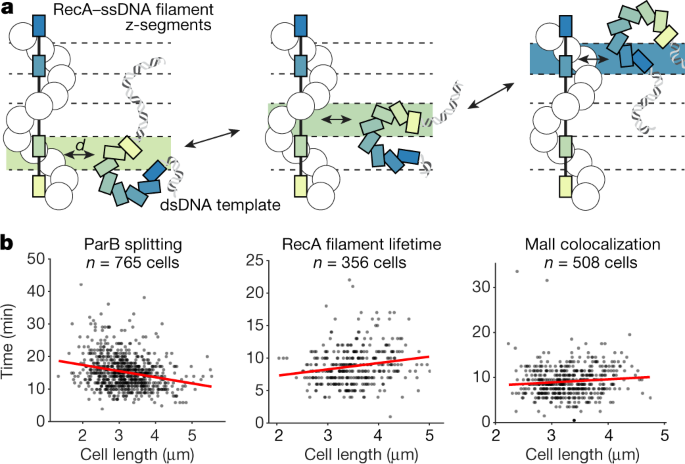
与其他依赖同源定向搜索的系统相比,RecA的目标搜索速度更快gydF4y2Ba16gydF4y2Ba需要一个不同的定量模型。由于大的RecA-ssDNA复合体和染色体位点的扩散速度都很慢gydF4y2Ba17gydF4y2Ba在三维空间中,无法用双分子反应-扩散来解释gydF4y2Ba18gydF4y2Ba.我们提出,RecA同源搜索由“降维”机制促进,该机制以两种方式加速了该过程。首先,ATP水解使RecA-ssDNA丝在不到一分钟的时间内穿过细胞,迅速覆盖断端和搜索目标之间的大部分距离,如图所示。gydF4y2Ba3gydF4y2Ba.第二,延长的纤维与许多不同的序列并行相互作用。这种同时探测在单分子实验的基础上已经被提出过gydF4y2Ba19gydF4y2Ba和冷冻电子显微镜结构gydF4y2Ba20.gydF4y2Ba.我们对模型的补充是认识到在任何gydF4y2BazgydF4y2Ba坐标(即沿着细胞的长轴),始终有至少一段拉伸的RecA-ssDNA丝同时与双链DNA (dsDNA)片段同源gydF4y2BazgydF4y2Ba位置(图。gydF4y2Ba4gydF4y2Ba)。这使得搜索问题独立于gydF4y2BazgydF4y2Ba协调并将复杂性从三维降低到二维。也就是说,同源配对的时间等于染色体dsDNA片段径向扩散到RecA-ssDNA丝的时间,而不是两个片段在整个细胞中通过3D扩散找到彼此所需的时间。在这种情况下,2D搜索大约比3D搜索快100倍gydF4y2Ba21gydF4y2Ba(有关详细描述,请参见gydF4y2Ba方法gydF4y2Ba)。gydF4y2Ba
一个gydF4y2Ba, RecA-ssDNA丝和修复模板各有一个同源片段(彩色条)gydF4y2BazgydF4y2Ba沿着单元格的长度坐标。当修复模板沿着单元移动时,只有当前的段gydF4y2BazgydF4y2Ba坐标是相关的。因此,相关段的搜索在2D中进行。gydF4y2BabgydF4y2Ba,修复时间是中断时单元长度的函数。时间是根据ParB焦点分裂(左),RecA细丝寿命(中)和MalI共定位(右)来测量的。红线显示与数据的线性拟合。显示被分析细胞的数量,gydF4y2BangydF4y2Ba= 4次(ParB)或3次(RecA, MalI)实验。gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
在补充信息中,我们推导了同源序列相遇的期望时间的表达式。我们的模型根据目标DNA在类核半径长度尺度上的扩散速率预测搜索将在5分钟内完成gydF4y2Ba17gydF4y2Ba.值得注意的是,该模型还预测搜索时间应该与单元长度不变,因为只有径向距离是相关的。这将我们的模型与以前的模型区分开来,例如参考文献中提出的模型。gydF4y2Ba22gydF4y2Ba.实验数据证实,细胞的长度对ParB焦点分裂、RecA结构寿命或马里焦点在细胞中部共定位估计的搜索时间有微小的影响(图2)。gydF4y2Ba4 bgydF4y2Ba),尽管更大的细胞有更多的DNA需要探索(扩展数据图。gydF4y2Ba8gydF4y2Ba)。gydF4y2Ba
讨论gydF4y2Ba
我们提出,拉伸的RecA-ssDNA丝——以一种简单而优雅的方式——将至少一个ssDNA片段定位在其同源姐妹的附近,这样同源dsDNA片段就可以使用快速、近距离搜索找到ssDNA片段。RecA是链交换蛋白家族的原型成员,它存在于所有形式的生命中,并具有共同的机制gydF4y2Ba3 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba.降维机制可能是这些蛋白质的保守性质。拉伸纤维的优势在拉长细胞中是明显的,长RecA结构确实在gydF4y2Ba茎菌属crescentusgydF4y2Ba11gydF4y2Ba而且gydF4y2Ba枯草芽孢杆菌gydF4y2Ba13gydF4y2Ba.当DSB修复发生在姐妹模板附近的复制分叉处时,荧光RecA仅在断裂部位形成短暂焦点gydF4y2Ba14gydF4y2Ba这表明,在RecA灯丝完全延伸之前,研究可能就会结束。先前的研究表明,I-SceI诱导的dsb修复是通过“束”进行的,“束”是由RecA形成的复杂结构gydF4y2Ba10gydF4y2Ba.这些束的特点是中心体厚,流动性低,寿命只有几十分钟,位于类核和内膜之间。我们发现参与DSB修复的RecA细丝与这些束明显不同:它们很薄,动态,仅持续几分钟,并且存在于类核内(图2)。gydF4y2Ba3 h,我gydF4y2Ba)。值得注意的是,根据降维模型,搜索时间不受DNA量增加的影响,只要灯丝的长度与DNA量成比例。因此,这种机制可以在基因组比细菌大的生物体中进行搜索。gydF4y2Ba
方法gydF4y2Ba
紧张施工gydF4y2Ba
在这项工作中使用的菌株是衍生物gydF4y2Ba大肠杆菌gydF4y2BaTB28gydF4y2Ba23gydF4y2Ba其中我们恢复了gydF4y2Barph-1gydF4y2Ba突变为野生型并删除gydF4y2Ba马里gydF4y2Ba基因和gydF4y2Ba不全gydF4y2Ba操作符。遗传整合是用lambda红整合完成的gydF4y2Ba24gydF4y2Ba,用pCP20质粒去除抗性盒gydF4y2Ba24gydF4y2Ba,标记物通过P1噬菌体转导进行组合。gydF4y2Ba
标签mCherry-ParBMt1gydF4y2Ba25gydF4y2Ba和MalI-Venus由整合到基因中的本构启动子表达gydF4y2BagtrAgydF4y2Ba轨迹。的gydF4y2Ba不全gydF4y2Ba/MalI标记由12个麦芽糖操作符组成,这些操作符是MalI- venus的结合位点。gydF4y2Ba
DSB盒由两侧的I-SceI识别位点组成gydF4y2Ba虫胶gydF4y2Ba运营商,gydF4y2BaparSMt1gydF4y2Ba站点和三gydF4y2Ba气gydF4y2Ba位于parSMt1和I-SceI识别序列之外的位点(扩展数据图。gydF4y2Ba1克ydF4y2Ba)。Cas9靶点选在距离细胞约100bp的地方gydF4y2BaparSMt1gydF4y2Ba网站。值得注意的是,该结构的设计方式没有gydF4y2Ba气gydF4y2Ba站点之间的DSB站点和gydF4y2BaparSMt1gydF4y2Ba网站。将DSB卡带集成成gydF4y2BacodAgydF4y2Ba轨迹。gydF4y2Ba
RecA-YFP融合基因在内源性基因下游直接表达gydF4y2BarecAgydF4y2Ba通过ref将构造中的mCherry替换为SYFP2。gydF4y2Ba14gydF4y2Ba.RecA-ALFA融合体通过引入ALFA c端来实现gydF4y2BarecAgydF4y2Ba无论是野生型还是野生型gydF4y2BarecAgydF4y2Ba轨迹或进入上面提到的串联结构。gydF4y2Ba
本研究中使用的菌株列表可在扩展数据表中找到gydF4y2Ba1克ydF4y2Ba.gydF4y2Ba
质粒构建gydF4y2Ba
p15a-SceIdeg-amp采用HiFi DNA Assembly (NEB)融合两个PCR片段进行克隆:(1)pSC101SceI_deg_ampgydF4y2Ba8gydF4y2Ba(2) p15aSceI_deg_Kan片段gydF4y2Ba8gydF4y2BaJwu037和Jwu038扩增片段。从p15a-SceIdeg-amp模板扩增出的2个PCR片段(1)Jwu088和Jwu090, (2) Jwu085和Jwu091,经Gibson组装克隆得到p15a-dSceIdeg-amp。p15a-Cas9deg-amp由两个片段(1)Jwu273和Jwu274扩增的p15a-SceIdeg-amp片段和(2)Jwu263和Jwu264扩增的pCRED片段(来自Daniel Camsund)组成。采用Gibson组装法克隆了质粒p15a-SceIdeg-amp- sos,分别采用引物Jwu330和Jwu331扩增p15a-SceIdeg-amp片段gydF4y2Ba大肠杆菌gydF4y2Ba引物Jwu332和Jwu333。采用Gibson组装法克隆p15a-Cas9deg-amp-SOS质粒,引物Jwu263和Jwu272扩增p15a-wtCas9deg-amp片段,引物Jwu273和Jwu274扩增p15a-SceIdeg-smp-SOS片段。p15a-dCas9deg-amp-SOS采用Gibson组装协议克隆(1)p15a-SceIdeg-amp-SOS扩增片段,引物为Jwu273和Jwu274gydF4y2Ba大肠杆菌gydF4y2Ba利用引物Jwu264和Jwu272对菌株EL1605进行测序。pKD13-recA:: alfa - cash - syfp2 -frt-kan-frt采用金门协议克隆(1)片段扩增gydF4y2Ba大肠杆菌gydF4y2Ba引物Jwu485和Jwu486,(2)从菌株EL2515中扩增出片段gydF4y2Ba大肠杆菌gydF4y2Ba(3)引物Jwu489和Jwu490从pKD13-P58-SYFP2-frt-kan-frt中扩增的片段。gydF4y2Ba
psgRNA-CS1被克隆gydF4y2Ba大肠杆菌gydF4y2Ba将Jwu267、Jwu184和psgRNA PCR生成的片段钝端结扎获得Top10gydF4y2Ba26gydF4y2Ba质粒作为模板(psgRNA是D. Bikard的礼物;添加基因质粒号。114005),引导RNA (gRNA)序列为ACTGGCTAATGCACCCAGTA。gydF4y2Ba
本研究中使用的引物列表可在扩展数据表中找到gydF4y2Ba2gydF4y2Ba.gydF4y2Ba
高通量DSB成像gydF4y2Ba
生长条件gydF4y2Ba
在微流控实验中,细胞在添加0.4%葡萄糖、0.08% RPMI 1640氨基酸(Sigma-Aldrich R7131)、表面活性剂Pluronic F-109 (Sigma-Aldrich 542342, 21µg ml的M9培养基中37°C生长gydF4y2Ba−1gydF4y2Ba),当相关卡本西林(40µg mlgydF4y2Ba−1gydF4y2Ba)或卡那霉素(20µg mlgydF4y2Ba−1gydF4y2Ba)。从- 80°C冷冻库中取出的细胞接种添加了足够抗生素的LB培养基,并在37°C下培养过夜。第二天,细胞在M9 0.4%葡萄糖0.08% RPMI培养基中稀释1/250,培养2 h后加载到微流控芯片上。在实验开始前,细胞在微流控芯片中生长至少2小时。Cas9和dCas9的诱导时间为5分钟(扩展数据图中的点计数实验为6分钟)。gydF4y2Ba2摄氏度gydF4y2Ba) aTc脉冲(20 pg mlgydF4y2Ba−1gydF4y2Ba)。gydF4y2Ba
显微镜gydF4y2Ba
显微镜实验采用Ti2-E(尼康)倒置显微镜,配备CFI Plan Apochromat DM Lambda 100×物镜(尼康),Sona 4.2B-11 sCMOS相机(Andor), Spectra III (Lumencor)荧光光源。显微镜由Micro-Manager控制gydF4y2Ba27gydF4y2Ba运行内部构建插件。荧光光源由TTL连接的相机触发。使用定制荧光立方体:CFP激发滤波器:BrightLine FF02-438/24 (Semrock),发射滤波器:BrightLine FF01-494/41 (Semrock),二向色镜:Di02-R442 (Semrock);YFP激励滤波器:FF01-514/3-25 (Semrock),发射滤波器:ET550/50M 200362 (Chroma),二向色镜:Di02-R514 (Semrock);mCherry励磁滤波器:FF01-559/34 (Semrock),发射滤波器:T590LP 262848 (Chroma),二向色镜:T585lpxr (Chroma)。用1.5倍中倍镜成像。在插入CFP立方体的情况下拍摄相位对比图像。通常情况下,每分钟采集一张相位对比图像,曝光80 ms, CFP通道每3分钟采集一张,荧光强度设置为5%,曝光40 ms, mCherry通道每分钟采集一张,荧光强度设置为20%,曝光80 ms, YFP通道每分钟采集一张(用于RecA成像),或每2分钟采集一张(用于MalI实验),荧光强度设置为40%,曝光100 ms。用于扩展数据图中的点计数实验。gydF4y2Ba2摄氏度gydF4y2BamCherry通道每3分钟成像一次gydF4y2BazgydF4y2Ba在每个时间点取间隔300 nm的切片,采用最大强度投影法重建荧光图像。gydF4y2Ba
微流控实验gydF4y2Ba
微流控实验采用前期研制的PDMS母机微流控芯片进行gydF4y2Ba7gydF4y2Ba.这种芯片设计允许加载两种不同的应变和自动切换介质。使用OB1 MK3+微流体流量控制器(Elveflow)控制介质压力。在3小时实验开始时脉冲aTc,或在图像采集开始后20分钟脉冲aTc, 4小时实验。gydF4y2Ba
染色体DAPI染色gydF4y2Ba
菌株EL2504和EL1743(与gydF4y2BadnaC2gydF4y2Ba28gydF4y2Ba)在LB培养基中过夜生长。第二天,将细胞培养物稀释在5 ml M9中,加入0.4%葡萄糖和0.08% RPMI 1640氨基酸(Sigma-Aldrich R7131),并在37°C下培养(菌株EL2504)。EL1734菌株在30°C下培养2小时,然后在42°C下培养一个培养基以诱导复制停止。DAPI加入最终浓度为1µg mlgydF4y2Ba−1gydF4y2Ba含DAPI的细胞在生长温度下孵育30 min。然后将1 ml细胞在4℃、7000转/分(5424 R, Eppendorf)下离心3分钟,再悬于50 μl添加10 mM MgCl的ITDE (Integrated DNA Technologies)冷缓冲液中gydF4y2Ba2gydF4y2Ba.将2 μl浓缩细胞置于琼脂糖垫上成像。成像用功率为12 mW cm的445 nm激光完成gydF4y2Ba−2gydF4y2Ba曝光时间为220毫秒。采用Nested-Unet神经网络对相位对比图像进行分割gydF4y2Ba29gydF4y2Ba他在公司内部接受培训。DAPI图像通过减去细胞外区域的平均像素强度来校正背景。gydF4y2Ba
图像分析gydF4y2Ba
数据分析是用MATLAB (Mathworks)完成的,除了单元格分割是用Python完成的。显微镜数据使用之前开发的自动化分析管道进行处理gydF4y2Ba30.gydF4y2Ba,经过了几处修改。首先,利用Nested-Unet神经网络对相位对比图像进行分割gydF4y2Ba29gydF4y2Ba内部培训,专门为我们的显微镜设置。神经网络使用Pytorch 1.7.1。我们训练了两个网络,一个用于分割细胞,另一个用于在相位对比图像上检测微流体生长通道(扩展数据图)。gydF4y2Ba1 cgydF4y2Ba)。测量不同滤波立方体获取的图像之间的转换矩阵,并对荧光图像进行转换,以校正荧光图像和相位对比度图像之间的像素移位。格拉姆gydF4y2Ba31gydF4y2Ba工具箱在Matlab中生成部分图。gydF4y2Ba
DSB细胞的选择和分析gydF4y2Ba
接受DSB修复的细胞是在质粒携带的SOS报告细胞CFP信号增加至少4倍的基础上选择的。首先,通过从原始CFP图像中减去一个核大小为20像素的高斯滤波器滤波的图像(使用Matlab函数imgaussfilt),对CFP图像进行背景校正。我们将分析局限于在分割中没有重大错误、存活至少9分钟并在实验中分裂的细胞。手动修复动力学注释仅在DSB之前包含两个ParB病灶的细胞上进行,并在修复后进行分裂。有>2 ParB病灶的细胞,或诱导DSB不止一次的细胞,被排除在分析之外。在扩展数据图的实验中。gydF4y2Ba2摄氏度gydF4y2Ba采用基于径向对称的方法自动检测ParB病灶。使用自定义编写的Matlab代码将斑点位置映射到单元长度上。所有显示样例细胞的图像都来自至少重复两次的实验,除了RecA-YFP和RecA-ALFA的costaining只做了一次(扩展数据图。gydF4y2Ba6 h-kgydF4y2Ba)。gydF4y2Ba
超分辨率STED显微镜gydF4y2Ba
样品制备gydF4y2Ba
表达RecA-ALFA, RecA-YFP或两者的细胞在37℃的M9培养基中培养3小时,培养基中含有葡萄糖(0.4%),RPMI 1640氨基酸(0.08%),卡本西林(20µg ml)gydF4y2Ba−1gydF4y2Ba)和卡那霉素(10µg mlgydF4y2Ba−1gydF4y2Ba)。用aTc (0.8 pg ml)诱导Cas9 40 mingydF4y2Ba−1gydF4y2Ba),然后用甲醛(3.5%)固定细胞10分钟。用100 mM甘氨酸淬灭固定,细胞在PBS中洗涤,然后在70%乙醇中渗透1小时。染色时,细胞在含牛血清白蛋白(1%)的PBS中封闭30分钟,然后用1:200稀释的抗体孵育至少1小时。我们使用偶联Star635P或Star580的骆驼单结构域抗体。用于RecA-ALFA flutag - x2抗alfa (N1502-Ab635P)和RecA-YFP flutag - x4抗gfp (N0304-Ab635P和N0304-Ab580, NanoTag生物技术公司)。在没有表位的情况下,没有检测到抗体结合(扩展数据图。gydF4y2Ba7gydF4y2Ba)。为了显示类核和膜,分别用Pico488(1:400稀释,Lumiprobe)和尼罗红(5µM)对细胞进行染色。在PBS中清洗三次后,细胞被安装在琼脂糖垫上进行显微镜检查。gydF4y2Ba
2D和3D STED显微镜gydF4y2Ba
使用定制的STED装置进行超分辨率成像gydF4y2Ba32gydF4y2Ba.用脉冲二极管激光器对染料进行激发;561 nm (PDL561, Abberior Instruments), 640 nm (LDH-D-C-640, PicoQuant)和510 nm (LDF-D-C-510, PicoQuant)。使用775 nm激光(KATANA 08 HP, OneFive)作为耗尽光束,利用空间光调制器(LCOS-SLM X10468-02, Hamamatsu Photonics)将其分裂成两束正交偏振光束,分别在焦平面上形成甜甜圈和礼帽形状,实现2D和3D-STED成像。激光束用HC PL APO 100×/1.40 Oil STED White物镜(15506378,Leica Microsystems)聚焦在样品上,并采集荧光信号。在物镜的第一共轭后焦平面测量,561 nm激发激光功率为8-20 μ W, 640 nm激发激光功率为4-10 μ W, 775 nm耗尽激光功率为128 mW,完成成像。gydF4y2Ba
RecA-YFP与RecA-ALFA以逐行扫描方式进行双色STED成像,平均超过4或8行;ParB和RecA-ALFA逐帧记录,第一通道为共焦,第二通道为STED。所有2D图像的像素大小设置为20 nm,像素停留时间为50µs。gydF4y2Ba
逐行扫描方式记录RecA-ALFA和尼罗红的体积双色3D-STED成像,随后在细菌细胞中间记录Pico488的单共焦帧。的体素大小gydF4y2BaxyzgydF4y2Ba体积设置为25 × 25 × 80 nmgydF4y2Ba3 gydF4y2Ba.像素停留时间设置为30或50µs。gydF4y2Ba
使用ImSpector软件(Max-Planck Innovation)和ImageJ对原始图像进行处理和可视化gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba.亮度和对比度对整个图像进行线性调整。计算垂直于结构方向的RecA细丝和RecA束的尺寸,并在原始图像上的两个像素上进行平均。然后用OriginPro2020软件对数据进行高斯函数拟合,从中提取全宽半最大值。为gydF4y2BaxzgydF4y2Ba在ImSpector中实现的Richardson Lucy反褶积算法对图像进行反褶积。在Imaris 9.1 (Bitplane)中使用惠更斯反褶积完成三维体绘制。gydF4y2Ba
显微镜的分辨率是在校准样品上测量的,校准样品由附着在玻璃上的稀疏抗体制成,再加上Star635P染料。提取直线轮廓,并用洛伦兹函数进行拟合gydF4y2Ba35gydF4y2Ba,其中宽度(gydF4y2BaWgydF4y2Ba)提取为网点大小。gydF4y2Ba
我们估计丝的直径为37.5±23.5 nm,作为观测到的60±13 nm FWHM宽度的反褶积(图2)。gydF4y2Ba3 ggydF4y2Ba)与成像系统的35±11 nm (FWHM) Lorentzian PFS(扩展数据图;gydF4y2Ba6 fgydF4y2Ba)假设灯丝是一个表面有3纳米荧光团层的圆柱体。gydF4y2Ba
I-SceI实验gydF4y2Ba
对于I-SceI超切酶的实验,将含有p15a-I-SceI质粒的细胞培养在M9最小培养基中,加载在微流控芯片中,然后在37°C的M9培养基中与葡萄糖(0.4%)、RPMI 1640氨基酸补充物(Sigma-Aldrich R7131, 0.05%)、卡本西林(20µg ml)孵育gydF4y2Ba−1gydF4y2Ba)和Pluronic F-108(21µg mlgydF4y2Ba−1gydF4y2Ba)。通过切换到同样含有aTc (20 ng ml)的培养基3分钟诱导dsbgydF4y2Ba−1gydF4y2Ba)和IPTG (1 mM),然后3分钟至中仅IPTG。然后对细胞进行成像,同时在初始培养基中修复和恢复。gydF4y2Ba
连续稀释镀法gydF4y2Ba
细菌培养物从冷冻库中取出,在洛杉矶的盘子上划线,在37°C下培养一夜。第二天,用隔夜培养皿中的单个菌落接种5 ml LB培养基,在37℃下培养6 h。接着,在LB中连续稀释10倍,每稀释4 μl分别镀于LA板或含1 μg ml的LA板上gydF4y2Ba−1gydF4y2Ba萘啶酸。在37°C下孵育过夜。gydF4y2Ba
用扩展的RecA细丝搜索模型gydF4y2Ba
同源搜索将被视为一个扩散限制反应,具有同源序列到达反应半径的运输时间和序列相遇后的探测时间。与获取同源序列的时间相比,正确序列的探测时间可以忽略不计,但需要探测的大量不正确句子会减慢整体反应。gydF4y2Ba
为了量化这些情况,我们从很少的近似开始。假设RecA-ssDNA细丝是圆柱形核体中心的细杆,从一个极点延伸到另一个极点gydF4y2BazgydF4y2Ba而同源dsDNA序列在类核的随机位置上盘绕。同源序列找到彼此的相关时间是盘绕起来的dsDNA片段径向扩散到细胞中心的棒的时间。在我们的模型中,核心的认识是,在哪个位置并不重要gydF4y2BazgydF4y2Ba-坐标它到达杆子。这将搜索问题从3D转换为2D,因为我们可以从dsDNA片段的角度来描述搜索过程,该dsDNA片段与恰好位于的ssDNA序列同源gydF4y2BazgydF4y2Ba-杆最先到达的位置。gydF4y2Ba
考虑这种情况的等效方法是考虑许多独立搜索器(即染色体dsDNA片段)的第一个绑定事件,每个可以绑定到许多目标中的一个(即reca结合的ssDNA片段),与一个可以绑定所有目标的搜索器具有相同的速率。总结合速率为gydF4y2Ba\ (r ={\总和}_{我}{r} _{我}\)gydF4y2Ba,其中gydF4y2BargydF4y2Ba我gydF4y2Ba是模板段收费吗gydF4y2Ba我\ \ ()gydF4y2Ba找到它的同源ssDNA片段。如果我们写出at的依赖关系gydF4y2BazgydF4y2Ba协调,gydF4y2Ba\ ({z} _ {j} \)gydF4y2Ba时,灯丝到达时,总速率可表示为gydF4y2Ba\ (r ={\总和}_ {j}{\总和}_{我}{r} _{我}({z} _ {j}) p ({z} _ {j}) \)gydF4y2Ba,其中gydF4y2Baz \ (p ({} _ {j}) \)gydF4y2Ba到达灯丝位置的概率是多少gydF4y2Ba\ ({z} _ {j} \)gydF4y2Ba而且gydF4y2Bar \ ({} _ {} ({z} _ {j}) \)gydF4y2Ba是分段的条件费率吗gydF4y2Ba我\ \ ()gydF4y2Ba考虑到灯丝达到gydF4y2Ba\ ({z} _ {j} \)gydF4y2Ba.在这里,除非模板片段与特定的ssDNA匹配,否则结合率为零gydF4y2BazgydF4y2Ba-position,即,gydF4y2Bar \ ({} _ {} ({z} _ {\ ne我})= 0 \)gydF4y2Ba这意味着gydF4y2Ba\ (r ={\总和}_{我}{r} _{我}({z} _{我})\)gydF4y2Baz \ (p({} _{我})\)gydF4y2Ba,即总结合率与单个dsDNA片段的结合率相同,该片段可以在灯丝的任何位置结合,与gydF4y2BazgydF4y2Ba位置,并且每个位置都是同源的。gydF4y2Ba
搜索时间预测gydF4y2Ba大肠杆菌gydF4y2Ba
对于染色体dsDNA片段从类核中的随机径向位置扩散到其中心的纤丝所需要的时间的一阶近似,我们可以使用扩散限制速率来达到圆柱体中心的棒gydF4y2Ba21gydF4y2Ba长度为2gydF4y2BalgydF4y2Ba,也就是说,gydF4y2BakgydF4y2Ba= 2π(2gydF4y2BalgydF4y2Ba)gydF4y2BaDgydF4y2Ba/ ln (gydF4y2BaR / RgydF4y2Ba),其中gydF4y2BargydF4y2Ba反应棒的反应半径,假设在核苷酸的量级(1nm),和gydF4y2BaRgydF4y2Ba是类核半径。搜索dsDNA片段的浓度为gydF4y2BacgydF4y2Ba= 1 /gydF4y2BaVgydF4y2Ba= 1 / (2gydF4y2BalgydF4y2BaπgydF4y2BaRgydF4y2Ba2gydF4y2Ba),其中gydF4y2BaVgydF4y2Ba是类核体积。因此到达杆的平均时间为gydF4y2BaTgydF4y2Ba=gydF4y2BaVgydF4y2Ba/gydF4y2BakgydF4y2Ba=gydF4y2BaRgydF4y2Ba2gydF4y2Baln (gydF4y2BaRgydF4y2Ba/gydF4y2BargydF4y2Ba) / 2gydF4y2BaDgydF4y2Ba.核半径gydF4y2BaRgydF4y2Ba≈200 nm,反应半径约为gydF4y2BargydF4y2Ba≈1nm,虽然确切的值是无关紧要的,因为只有它的对数进入时间。复杂的参数是扩散速率常数gydF4y2BaDgydF4y2Ba,因为DNA位点的运动是次扩散的gydF4y2BaDgydF4y2Ba因此在长长度尺度上比短长度尺度上要低。然而,该过程将受到相应的长距离运动的限制gydF4y2BaRgydF4y2Ba.在长度尺度上gydF4y2Ba17gydF4y2Ba的gydF4y2BaRgydF4y2Ba= 200nm,gydF4y2BaDgydF4y2BaRgydF4y2Ba≈0.0007 μmgydF4y2Ba2gydF4y2Ba年代gydF4y2Ba−1gydF4y2Ba.搜索过程的关联步骤是这样的gydF4y2BaTgydF4y2Ba≈(0.2 μm)gydF4y2Ba2gydF4y2Ba× ln(200 nm/1 nm)/0.0007 μmgydF4y2Ba2gydF4y2Ba年代gydF4y2Ba−1gydF4y2Ba= 300秒= 5分钟。如果我们考虑RecA灯丝也在分钟的时间尺度上移动,这只会进一步加快搜索速度。gydF4y2Ba
探测时间gydF4y2Ba
然而,由于RecA细丝还需要探测所有其他的dsDNA片段,因此它不能一直被用于结合。如果同源序列只需要测量到的搜索时间的一半(约10分钟),那么对其他序列的探测还有5分钟的时间。这足以探测所有序列吗?如果dsDNA被探测gydF4y2BangydF4y2Ba-bp长的片段,在RecA纤维中2 kb长的ssDNA的每一个等长片段平均每2000 /一次gydF4y2BangydF4y2Ba染色体的片段。细胞中dsDNA有9.6 Mb(每个基因组4.8 Mb ×每个细胞约2个基因组),对应9.6 × 10gydF4y2Ba6克ydF4y2Ba/gydF4y2BangydF4y2Ba探测部分。反过来,这意味着RecA灯丝中的每个ssDNA片段都需要测试(9.6 × 10gydF4y2Ba6克ydF4y2Ba/gydF4y2BangydF4y2Ba) / (2000 /gydF4y2BangydF4y2Ba) = 4,800个dsDNA片段。每次测试的平均时间不能超过300 s/ 4800≈63 ms,考虑到Cas9-sgRNA执行类似任务平均需要30 ms,这应该是足够的时间gydF4y2Ba16gydF4y2Ba.gydF4y2Ba
ATP结合和水解gydF4y2Ba
ATP-RecA对ssDNA具有较高的亲和力gydF4y2Ba15gydF4y2Ba并在结合后的结果是一个拉伸和刚性的灯丝与持久的长度gydF4y2Ba16gydF4y2Ba大约900纳米。ATP水解降低了RecA对ssDNA的亲和力,是快速丢弃不匹配序列所必需的gydF4y2Ba17gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba.因此,需要ATP周转来拉伸细胞内环境中的纤维,否则它将陷入部分同源。gydF4y2Ba
替代模型gydF4y2Ba
如何获得足够接近的序列以探测同源性的替代模型可以有许多其他形式。gydF4y2Ba
最简单的比较是考虑扩散速率对应dsDNA片段的粒子和反应半径对应的非扩散片段的扩散限制双分子反应gydF4y2BargydF4y2Ba(约1nm)在核碘的任何位置。这里,扩散限制反应的速率是gydF4y2BakgydF4y2Ba= 4πgydF4y2Ba理查德·道金斯gydF4y2BalgydF4y2Ba,其中gydF4y2BaDgydF4y2BalgydF4y2Ba就是扩散gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba细胞长度尺度上的速率,而段的浓度与上面相同(gydF4y2BacgydF4y2Ba= 1 /gydF4y2BaVgydF4y2Ba= 1 / (2gydF4y2BalgydF4y2BaπgydF4y2BaRgydF4y2Ba2gydF4y2Ba))。结果是gydF4y2BaTgydF4y2Ba=gydF4y2BaVgydF4y2Ba/gydF4y2BakgydF4y2Ba=gydF4y2BaLRgydF4y2Ba2gydF4y2Ba/ 2gydF4y2Ba理查德·道金斯gydF4y2BalgydF4y2Ba.这应该与gydF4y2BaRgydF4y2Ba2gydF4y2Baln (gydF4y2BaRgydF4y2Ba/gydF4y2BargydF4y2Ba) / 2gydF4y2BaDgydF4y2BaRgydF4y2Ba在二维情况下。重要的是,在类核半径长度尺度上的扩散速率,gydF4y2BaDgydF4y2BaRgydF4y2Ba,是一个数量级gydF4y2Ba17gydF4y2Ba快于;gydF4y2BalgydF4y2Ba.比率为(gydF4y2BalgydF4y2Ba/gydF4y2Ba理查德·道金斯gydF4y2BalgydF4y2Ba) / (ln (gydF4y2BaRgydF4y2Ba/gydF4y2BargydF4y2Ba)/gydF4y2BaDgydF4y2BaRgydF4y2Ba)≈1750,考虑gydF4y2BalgydF4y2Ba= 1µm,gydF4y2BaRgydF4y2Ba= 300 nm,gydF4y2BargydF4y2Ba= 1 nm和gydF4y2BaDgydF4y2BaRgydF4y2Ba/gydF4y2BaDgydF4y2BalgydF4y2Ba≈10。的实际值gydF4y2BargydF4y2Ba在这种情况下更重要,因为重新绑定事件的数量比在2D情况下更重要。gydF4y2Ba
建立二维模型的中间步骤是将原始模型并行化,并将同源dsDNA分成片段,这些片段可以并行和独立地在RecA细丝中搜索它们各自的同源性。例如,如果我们将1750个碱基长的灯丝分成10段,速度将增加175倍,与2D模型相比,剩余的差异为(gydF4y2BaDgydF4y2BaRgydF4y2Ba/gydF4y2BaDgydF4y2BalgydF4y2Ba)−倍的短期和远程扩散的差异。gydF4y2Ba
最后一个模型是认为同源DNA是静态的,只有RecA细丝的运动类似于针织针在纱线球中的运动。这种情况不是直接量化的,因为我们不知道在细胞中不同长度尺度上的RecA细丝有多刚性。它似乎在100纳米的长度尺度上是灵活的,但探测相互作用必须发生在1纳米尺度上,因此我们不知道灯丝探索基因组的速度有多快。它可以清楚地同时探测多个DNA片段gydF4y2Ba19gydF4y2Ba但是,当细丝移动到基因组的其他部分时,用细丝的一部分探测序列会造成复杂的限制。可能需要详细的模拟来预测这类模型的预期搜索时间。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然研究报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
原始显微数据可在gydF4y2Bahttps://doi.org/10.17044/scilifelab.14815802gydF4y2Ba.gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
用于分析和绘图的计算代码可在gydF4y2Bahttps://doi.org/10.17044/scilifelab.14815802gydF4y2Ba.gydF4y2Ba
改变历史gydF4y2Ba
2021年11月12日gydF4y2Ba
对本文的更正已发表:gydF4y2Bahttps://doi.org/10.1038/s41586-021-04154-2gydF4y2Ba
参考文献gydF4y2Ba
重组DNA修复的分子机制概述。gydF4y2Ba冷泉港。教谕。医学杂志。gydF4y2Ba7gydF4y2Ba, a016410(2015)。gydF4y2Ba
dicingham, m.s. & Kowalczykowski, s.c. RecBCD酶和双链DNA断裂的修复。gydF4y2BaMicrobiol。摩尔。杂志。牧师。gydF4y2Ba72gydF4y2Ba, 642-671(2008)。gydF4y2Ba
贝尔,J. C.和科瓦尔奇科夫斯基,S. C. RecA:分子搜索引擎的调节和机制。gydF4y2Ba学生物化学的发展趋势。ScigydF4y2Ba41gydF4y2Ba, 491-507(2016)。gydF4y2Ba
Renkawitz, J., Lademann, C. A. & Jentsch, S.重组中同源搜索的机制和原则。gydF4y2Ba细胞生物学。gydF4y2Ba15gydF4y2Ba, 369-383(2014)。gydF4y2Ba
Bianco, P. R., Tracy, R. B. & Kowalczykowski, S. C. DNA链交换蛋白:生化和物理比较。gydF4y2Ba前面。Biosci。gydF4y2Ba3 gydF4y2Ba, d570-d603(1998)。gydF4y2Ba
SOS修复假说:伴随突变的可诱导DNA修复的现象学。gydF4y2Ba基础生命科学。gydF4y2Ba5gydF4y2Ba, 355-367(1975)。gydF4y2Ba
Baltekin, O。,Boucharin, A., Tano, E., Andersson, D. I. & Elf, J. Antibiotic susceptibility testing in less than 30 min using direct single-cell imaging.国家科学院学报美国gydF4y2Ba114gydF4y2Ba, 9170-9175(2017)。gydF4y2Ba
Wiktor, J., van der Does, M, Büller, L., Sherratt, D. J. & Dekker, C. RecBCD在体内双链DNA断裂修复过程中末端切除的直接观察。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, 1821-1833(2018)。gydF4y2Ba
Cockram, C. A., Filatenkova, M., Danos, V., El Karoui, M. & Leach, D. R. F. DNA双链断裂修复过程中RecA蛋白结合的定量基因组分析揭示了RecBCD在体内的作用。gydF4y2Ba国家科学院学报美国gydF4y2Ba112gydF4y2Ba, e4735-e4742(2015)。gydF4y2Ba
Lesterlin, C., Ball, G., Schermelleh, L. & Sherratt, D. J. RecA束在DNA断裂修复过程中介导远亲姐妹之间的同源配对。gydF4y2Ba自然gydF4y2Ba506gydF4y2Ba, 249-253(2014)。gydF4y2Ba
Badrinarayanan, A., Le, T. B. K. & Laub . M. T.远距离同源位点的快速配对和再分离使细菌的双链断裂修复成为可能。gydF4y2BaJ.细胞生物学。gydF4y2Ba210gydF4y2Ba, 385-400(2015)。gydF4y2Ba
Renzette, N。gydF4y2Ba等gydF4y2Ba.RecA的本地化gydF4y2Ba大肠杆菌gydF4y2BaK-12使用RecA-GFP。gydF4y2Ba摩尔。Microbiol。gydF4y2Ba57gydF4y2Ba, 1074-1085(2005)。gydF4y2Ba
Kidane, D. & Graumann, P. L.活细胞DNA双链断裂修复中心RecA细丝的动态形成。gydF4y2BaJ.细胞生物学。gydF4y2Ba170gydF4y2Ba, 357-366(2005)。gydF4y2Ba
Amarh, V. White, M. A. & Leach, D. R. F. reca介导的复制依赖性DNA断裂修复的动力学。gydF4y2BaJ.细胞生物学。gydF4y2Ba217gydF4y2Ba, 2299-2307(2018)。gydF4y2Ba
Gotzke, H。gydF4y2Ba等gydF4y2Ba.alfa标签是基于纳米体的生物科学应用的一种高度通用的工具。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 4403(2019)。gydF4y2Ba
琼斯,D. L。gydF4y2Ba等gydF4y2Ba.dCas9靶搜索动力学gydF4y2Ba大肠杆菌gydF4y2Ba.gydF4y2Ba科学gydF4y2Ba357gydF4y2Ba, 1420-1424(2017)。gydF4y2Ba
韦伯,S. C., Spakowitz, A. J. & Theriot, J. A.非热atp依赖性波动有助于染色体位点的体内运动。gydF4y2Ba国家科学院学报美国gydF4y2Ba109gydF4y2Ba, 7338-7343(2012)。gydF4y2Ba
柯林斯,F. C.和金博尔,G. E.扩散控制反应速率。gydF4y2Ba胶体科学。gydF4y2Ba4gydF4y2Ba, 425-437(1949)。gydF4y2Ba
Forget, a . L. & Kowalczykowski, S. C. RecA DNA配对的单分子成像揭示了三维同源性搜索。gydF4y2Ba自然gydF4y2Ba482gydF4y2Ba, 423-427(2012)。gydF4y2Ba
杨海华,周春华,周春华,杨海华。RecA-DNA突触和D-loop结构的链交换机制。gydF4y2Ba自然gydF4y2Ba586gydF4y2Ba, 801-806(2020)。gydF4y2Ba
伯格,O. G. &布隆伯格,C.耦合扩散流的关联动力学。gydF4y2BaBiophys。化学。gydF4y2Ba4gydF4y2Ba, 367-381(1976)。gydF4y2Ba
高楚佳娃,史维茨,A. A. & Kolomeisky, A. B. RecA蛋白丝同源性搜索机制的研究。gydF4y2BaBiophys。J。gydF4y2Ba112gydF4y2Ba, 859-867(2017)。gydF4y2Ba
伯恩哈特,t.g.,德波尔,p.a.j.gydF4y2Ba大肠杆菌gydF4y2Ba酰胺酶是一种通过双精氨酸转运途径输出的质周间隔环成分。gydF4y2Ba摩尔。Microbiol。gydF4y2Ba48gydF4y2Ba, 1171-1182(2003)。gydF4y2Ba
李国强,李国强,李国强,等。小鼠染色体基因一步失活研究gydF4y2Ba大肠杆菌gydF4y2BaK-12使用PCR产物。gydF4y2Ba国家科学院学报美国gydF4y2Ba97gydF4y2Ba, 6640-6645(2000)。gydF4y2Ba
尼尔森,H. J.,奥特森,J. R.,杨仁,B.,奥斯汀,S. J. &汉森,F. G. ThegydF4y2Ba大肠杆菌gydF4y2Ba染色体由左右染色体臂组成,分别位于不同的细胞半部分。gydF4y2Ba摩尔。Microbiol。gydF4y2Ba62gydF4y2Ba, 331-338(2006)。gydF4y2Ba
崔,L。gydF4y2Ba等gydF4y2Ba.CRISPRi屏幕gydF4y2Ba大肠杆菌gydF4y2Ba揭示了dCas9序列特异性毒性。gydF4y2BaCommun Nat。gydF4y2Ba9gydF4y2Ba, 1912(2018)。gydF4y2Ba
埃德尔斯坦gydF4y2Ba等gydF4y2Ba.采用μManager软件控制显微镜的先进方法。gydF4y2Ba生物。方法gydF4y2BaCh.14, Unit14.20(2010)。gydF4y2Ba
威瑟斯。H. L.伯纳德gydF4y2BadnaC2gydF4y2Ba而且gydF4y2BadnaC28gydF4y2Ba流式细胞仪检测突变体。gydF4y2Baj . Bacteriol。gydF4y2Ba180gydF4y2Ba, 1624-1631(1998)。gydF4y2Ba
周z ., Siddiquee, m.m.r, Tajbakhsh, N.和Liang, J. UNet++:重新设计跳跃连接以利用图像分割中的多尺度特征。gydF4y2BaIEEE反式。医疗成像。gydF4y2Ba39gydF4y2Ba, 1856-1867(2020)。gydF4y2Ba
Camsund D。gydF4y2Ba等gydF4y2Ba.基于时间分辨成像的CRISPRi筛选。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 86-92(2020)。gydF4y2Ba
Pierre, M. Gramm: Matlab中的图形绘图语法。gydF4y2BaJ.开源软件gydF4y2Ba3 gydF4y2Ba, 568(2018)。gydF4y2Ba
张国强,张国强,张国强,等。扩展样品区域的稳定受激发射耗竭成像。gydF4y2Ba期刊。DgydF4y2Ba53gydF4y2Ba, 024001(2019)。gydF4y2Ba
Schindelin, J。gydF4y2Ba等gydF4y2Ba.斐济:生物图像分析的开源平台。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 676-682(2012)。gydF4y2Ba
鲁登,c.t.。gydF4y2Ba等gydF4y2Ba.ImageJ2:用于下一代科学图像数据的ImageJ。gydF4y2BaBMC Bioinf。gydF4y2Ba18gydF4y2Ba, 529(2017)。gydF4y2Ba
Vicidomini G。gydF4y2Ba等gydF4y2Ba.带时间门控检测的STED纳米镜:理论和实验方面。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba8gydF4y2Ba, e54421(2013)。gydF4y2Ba
李,J. Y, Qi, Z. & Greene, E. C. ATP水解促进RecA突触前复合物的双链DNA释放。gydF4y2Ba生物。化学。gydF4y2Ba291gydF4y2Ba, 22218-22230(2016)。gydF4y2Ba
Danilowicz, C., Hermans, L., Coljee, V., Prévost, C. & Prentiss, M. ATP水解提供了促进长重复序列不同副本之间配对排斥的功能。gydF4y2Ba核酸测定。gydF4y2Ba45gydF4y2Ba, 8448-8462(2017)。gydF4y2Ba
关于扩散控制的解离。gydF4y2Ba化学。理论物理。gydF4y2Ba31gydF4y2Ba, 47-57(1978)。gydF4y2Ba
爸爸,T.等人。建设gydF4y2Ba大肠杆菌gydF4y2BaK-12帧内,单基因敲除突变体:Keio集合。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba2gydF4y2Ba, 2006.0008(2006)。gydF4y2Ba
确认gydF4y2Ba
我们感谢D. Fange, I. Barkefors和D. Jones对手稿的帮助,感谢D. Camsund对pCRED质粒的帮助,感谢S. Zikrin和P. Karempudi对图像分析的帮助。J.W.是EMBO非津贴研究员(EMBO ALTF 1029-2018)。这项工作由H2020玛丽·居里个人奖学金(RecPAIR:842047)资助给J.W,欧洲研究理事会(BIGGER:885360),瑞典研究理事会(2016.06213和2018.03958),克努特和爱丽丝·瓦伦堡基金会(2016.0077,2017.0291和2019.0439)和eSSENCE e-science计划资助给J.E, SSF (FFL15-0031)资助给I.T.gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
j.e., J.W.和A.H.G.构想了这项研究并解释了结果。a.h.g., J.W.和J.L.进行了实验。J.W.和J.L.进行cas9诱导断裂成像;A.H.G.进行scei诱导断裂成像和STED样品制备。J.W.和A.H.G.编写了分析代码并分析了数据,A.H.G.、J.W.和P.L.构建了菌株,P.L.制作并测试了新的RecA融合,J.E.推导了方程,G.C.和I.T.贡献了STED成像。j.e., A.H.G.和J.W.在所有作者的输入下撰写了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
额外的信息gydF4y2Ba
同行评审信息gydF4y2Ba自然gydF4y2Ba感谢Anatoly Kolomeisky, David Leach和Stephan Uphoff对这项工作的同行评审所做的贡献。同行评审报告是可用的。gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
扩展数据图1 DSB感应系统及图像处理。gydF4y2Ba
一个gydF4y2Ba, DSB报告系统中使用的遗传结构。Cas9和SOS-reporter在一个质粒上编码,sgRNA在一个单独的质粒上编码。圆形地图gydF4y2Ba大肠杆菌gydF4y2Ba显示了染色体与被切部位整合的位置。底部卡通显示了一个放大的观点,削减现场卡带集成到gydF4y2BacodAgydF4y2Ba染色体上的位点。直方图显示了以ParB病灶数量为测量值的细胞中切割位点拷贝数的分布。gydF4y2BangydF4y2Ba= 9263个细胞,来自3个实验。gydF4y2BabgydF4y2Ba,上,显示微流控芯片简化示意图的漫画。下图:在实验的生长(左)和诱导(右)阶段,显示介质输入结点和介质流动的动画。最右边的卡通显示了芯片上生长通道的排列。通常,用活性Cas9和dCas9转化的应变被加载在芯片的不同一侧,以作为感应强度的控制。gydF4y2BacgydF4y2Ba,左:图像分析管道中的步骤。正确的:gydF4y2Ba前gydF4y2Ba图像显示了微流控芯片单个位置的全视场(FOV)。在一个实验中,通常会对16个位置进行成像。gydF4y2Ba中间gydF4y2Ba图像显示了FOV的放大部分,并覆盖了分割通道的掩模。gydF4y2Ba底gydF4y2Ba图像显示了与中间相同的FOV部分,但重叠的是分段细胞gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
扩展数据图2 DSB诱导特异性gydF4y2Ba
一个gydF4y2Ba,上:在DSB实验中显示SOS报告通道的细胞,激活SOS响应的细胞被概述并显示在底部。箭头指向下一帧丢失的ParB焦点。比例尺为2µm。gydF4y2BabgydF4y2Ba,在不同菌株和活跃或死亡的Cas9变体中激活SOS反应的细胞片段。切位点菌株缺乏染色体gydF4y2Bacut-sitegydF4y2Ba.柱状图代表平均值,点代表每个生物重复实验的平均值。gydF4y2BacgydF4y2Ba, Cas9或dCas9诱导后平均每个细胞ParB病灶数(诱导时间为t = 20min)。实线表示平均值,浅色区域表示自举法测量的95%置信区间。Wt: n = 3次实验,gydF4y2Ba∆recAgydF4y2Ba: n = 2次实验,gydF4y2Ba∆recBgydF4y2Ba;N = 2次实验。gydF4y2BadgydF4y2Ba,在每4个实验重复中,通过ParB焦点损失到恢复来测量修复时间。中线为中位数,方框边界为25gydF4y2BathgydF4y2Ba和75年gydF4y2BathgydF4y2Ba如果最极端的值超出了截断值,则晶须显示最极端的值,或上下四分位范围(IQR)的1.5倍,实点表示超出截断值的数据点。N(每个实验):1 - 250细胞,2 - 150细胞,3 - 174细胞,4 - 171细胞。gydF4y2BaegydF4y2Ba,由Cas9引起的ParB焦点损失到dsb恢复所测量的修复时间(Cas9数据与图中相同)。gydF4y2Ba1克gydF4y2Ba)或I-gydF4y2Ba南加州爱迪生公司gydF4y2Ba我的酶。箱形图如d. I-gydF4y2Ba南加州爱迪生公司gydF4y2BaI: n = 73个细胞,8次实验;Cas9: n = 765个细胞,4次实验。gydF4y2BafgydF4y2Ba,gydF4y2Ba∆ruvC∆放射心电图gydF4y2BaDSB后的细胞。比例尺为2µmgydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
扩展数据图3不同标记存在时DSB修复动态。gydF4y2Ba
一个gydF4y2Ba两个正在进行DSB修复的示例细胞。注释MalI焦点丢失和恢复事件,以及修复时间。单元格轮廓显示为纯灰色线。比例尺为2µm。漫画显示了一个切割工地建设与gydF4y2Ba不全gydF4y2Ba/MalI标记,表示标记与切割点之间的距离。箱形图显示了带有MalI标记的应变与带有ParB标记的应变的修复时间测量结果(ParB数据在图中相同)。gydF4y2Ba1克gydF4y2Ba)。中线为中位数,方框边界为25gydF4y2BathgydF4y2Ba和75年gydF4y2BathgydF4y2Ba如果最极端的值超出了截断值,则晶须显示最极端的值,或上下四分位范围(IQR)的1.5倍,实点表示超出截断值的数据点。ParB: 4个实验n = 765个细胞,MalI: 2个实验n = 221个细胞。gydF4y2BabgydF4y2Ba两个正在进行DSB修复的示例细胞。修复时间用实线标注。单元格轮廓显示为灰色实线。比例尺为2µm。漫画展示了一个被切割的地方gydF4y2Ba不全gydF4y2Ba/马里和gydF4y2Ba帕尔斯gydF4y2Ba/ParB标记,标记与切割点之间的距离gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
扩展数据图4远同源的配对。gydF4y2Ba
一个gydF4y2Ba,带有+170kb MalI标记的细胞进行DSB修复。ParB焦点损失和恢复事件显示。gydF4y2BabgydF4y2Ba,如gydF4y2Ba一个gydF4y2Ba,但对于一个细胞用gydF4y2BaygaYgydF4y2Ba马里标记。显示MalI标记配对事件。gydF4y2BacgydF4y2Ba,在DSB细胞中+170kb MalI病灶在时间上的空间定位。对于每个细胞,时间根据ParB焦点损失时间进行对齐。彩色条显示计数的密度,n = 337个细胞,来自2个实验。gydF4y2BadgydF4y2Ba, -25 kb MalI焦点配对后ParB焦点分裂时间,n = 507个细胞,来自3个实验。gydF4y2BaegydF4y2Ba,漫画显示了导致马里病灶在细胞中心共定位的事件。1.Cas9诱导DSB。2.由于末端切除,DSB部位的ParB焦点丢失。3.修复模板被识别,同源位点移动并在细胞中心共定位。4.DSB修复,ParB焦点恢复,同源位点分离gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
扩展数据图5 RecA结构是特定的。gydF4y2Ba
一个gydF4y2Ba带有ParB和RecA标签的细胞进行DSB修复。gydF4y2BabgydF4y2Ba,不同RecA变异株在没有或存在萘啶酸诱导的DNA损伤的情况下生长。gydF4y2BacgydF4y2Ba,质粒SOS报告基因荧光检测SOS反应激活时间与RecA表达的相关性。通过荧光信号达到最大值一半的时间来测量SOS激活。分析仅限于激活SOS响应的细胞。红线表示与数据的线性拟合(黑色),虚线表示95%置信区间。右图显示单个细胞的SOS(蓝色)和RecA(绿色)强度轨迹。显示每个通道半最大信号的时间。相关性是Pearson的r, n = 255个细胞,来自一个实验。gydF4y2BaPgydF4y2Ba值来自f检验。gydF4y2BadgydF4y2Ba, DSB修复细胞中RecA结构寿命与SOS信号强度增加的相关性。红色实线表示线性拟合,红色虚线表示置信界限,相关性为Pearson的r, n = 358个细胞,来自3个实验。gydF4y2BaPgydF4y2Ba值来自f检验。gydF4y2BaegydF4y2Ba和在gydF4y2BadgydF4y2Ba而对于RecA结构寿命与RecA- yfp表达增加之间的相关性,相关性为Pearson的r,来自3个实验的n = 358个细胞。gydF4y2BaPgydF4y2Ba值来自f检验。gydF4y2BafgydF4y2Ba, DSB诱导后具有RecA结构的细胞比例(时间=0),n = 2次实验。gydF4y2BaggydF4y2Ba,对于不同的RecA变体,由ParB焦点损失到dsb恢复测量的修复时间(野生型数据与图中相同)。gydF4y2Ba1克gydF4y2Ba)。中线为中位数,方框边界为25gydF4y2BathgydF4y2Ba和75年gydF4y2BathgydF4y2Ba如果最极端值超出该截断值,则须显示最极端值,或上下四分位范围(IQR)的1.5倍,实点表示超出截断值的数据点,RecA-YFP: n = 371个细胞来自4个实验,wt: n = 765个细胞来自4个实验,RecA-ALFA: 361个细胞来自2个实验。gydF4y2BahgydF4y2Ba, RecA结构出现与ParB焦点丢失之间的时间,3次实验n = 358个细胞。gydF4y2Ba我gydF4y2Ba, RecA结构拆卸到ParB焦点分裂的时间,3次实验n = 358个细胞gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
图6 RecA细丝的STED镜检。gydF4y2Ba
一个gydF4y2Bacas9诱导的dsb细胞,表达RecA-ALFA,并用抗alfa - star635p抗体标记。Star635P在STED和ParB用共聚焦显微镜成像。单元格轮廓由虚线表示。所示单元如图所示。gydF4y2Ba3 fgydF4y2Ba.gydF4y2BabgydF4y2Ba,细胞的体积三维视图(也显示在图中。gydF4y2Ba3我gydF4y2Ba),采用扩展的RecA-ALFA结构,3D STED成像。gydF4y2BacgydF4y2Ba,上:细胞经RecA-ALFA和尼罗红膜染色后的3D STED切面,如图所示。gydF4y2Ba3 jgydF4y2Ba.底部:如上所示的RecA和膜强度的线剖面。指出了RecA和膜之间的峰到峰的距离。gydF4y2BadgydF4y2Ba(左)膜染料和RecA在RecA结构扩展的细胞段横截面上的平均分布。右:左侧横截面上膜和RecA的径向密度,2次实验中n = 17个细胞。gydF4y2BaegydF4y2Ba,图中标记点之间的线剖面图。gydF4y2Ba3 fgydF4y2Ba共聚焦显微镜和STED显微镜。实线表示STED剖面的高斯拟合。长丝宽度的测量是高斯拟合的FWHM。gydF4y2BafgydF4y2Ba,使用star635p偶联抗体测量STED分辨率。左图:插入图中所示单抗体的剖面,以及该剖面的洛伦兹拟合。右图:抗体洛伦兹拟合线轮廓的FWHM分布,代表STED显微镜的分辨率,一次实验n = 50。gydF4y2BaggydF4y2Ba,具有扩展结构的细胞和RecA-RecA-YFP结构的STED图像,抗gfp抗体标记。gydF4y2BahgydF4y2Ba,同时表达RecA-ALFA和RecA-YFP的基因构建物,插入原代gydF4y2BarecAgydF4y2Ba轨迹。gydF4y2Ba我gydF4y2Ba经抗gfp - star580和抗alfa - star635p抗体染色,经RecA纤维延长的细胞的STED图像。gydF4y2BajgydF4y2Ba中所示的荧光强度分布图gydF4y2Ba我gydF4y2Ba,显示了两种抗体之间的重叠。gydF4y2BakgydF4y2Ba,有免疫标记细胞的进一步STED图像,如图gydF4y2Ba我gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
扩展数据图7抗体验证。gydF4y2Ba
细胞内抗体标记的特异性,通过结合star635p偶联抗体特异于ALFA和gfp衍生物,在有或没有表位的菌株中,有或没有DNA损伤诱导。gydF4y2Ba
扩展数据图8 DNA数量随细胞大小而变化。gydF4y2Ba
显示DNA含量(DAPI)作为细胞大小的函数的散点图,n = 9065个细胞来自一个实验gydF4y2Ba
源数据gydF4y2Ba.gydF4y2Ba
补充信息gydF4y2Ba
RecA细丝的亚细胞定位gydF4y2Ba
用cas9诱导的dsb三维STED重建细胞,表达RecA-ALFA,并用抗alfa Star635P抗体标记(绿色),细胞膜用尼罗红(洋红色)染色。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba.gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
维克多,Gynnå, a.h.,勒罗伊,P。gydF4y2Baet al。gydF4y2BaRecA通过降维搜索找到同源DNA。gydF4y2Ba自然gydF4y2Ba597gydF4y2Ba, 426-429(2021)。https://doi.org/10.1038/s41586-021-03877-6gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-021-03877-6gydF4y2Ba
这篇文章被引用gydF4y2Ba
分手和和好:DNA搜索和修复gydF4y2Ba
自然微生物学评论gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba