摘要gydF4y2Ba
染色体外DNA (ecDNA)在人类癌症中普遍存在,并通过基因扩增和改变基因调控介导癌基因的高表达gydF4y2Ba1gydF4y2Ba.基因诱导通常包括gydF4y2Ba独联体gydF4y2Ba-与同一染色体上的基因接触并激活的调控元件gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba.在这里,我们发现ecDNA枢纽(细胞核内约10-100个ecDNA簇)能够使分子间增强子-基因相互作用促进癌基因过表达。编码多种不同癌基因的ecdna在不同的癌细胞类型和原发肿瘤中形成枢纽。当每个ecDNA与其他ecDNA在空间上聚集时,更有可能转录癌基因。ecDNA中心由溴域和外域(BET)蛋白BRD4连接gydF4y2BaMYCgydF4y2Ba-扩增结直肠癌细胞系。BET抑制剂JQ1分散ecDNA中心,优先抑制ecDNA衍生的癌基因转录。的BRD4-boundgydF4y2BaPVT1gydF4y2Ba启动子异向融合gydF4y2BaMYCgydF4y2Ba并在ecDNA中复制,接收混杂的增强子输入以驱动基因的有效表达gydF4y2BaMYCgydF4y2Ba.此外,gydF4y2BaPVT1gydF4y2Ba外源片段上的启动子足以介导基因激活gydF4y2Ba反式gydF4y2Ba通过ecDNA中心以jq1敏感的方式。通过CRISPR干扰系统沉默ecDNA增强子,揭示了在不同ecDNA上扩增的多个癌基因位点之间的分子间增强子-基因激活。因此,蛋白质系结的ecDNA中心能够进行分子间转录调节,并可能作为癌基因功能和协同进化的单位,并作为癌症治疗的潜在靶点。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
染色体外环状DNA:生物发生、结构、功能与疾病gydF4y2Ba
信号转导与靶向治疗gydF4y2Ba开放获取gydF4y2Ba10月2日gydF4y2Ba
染色体外环状DNA (eccDNA):癌症领域的新星gydF4y2Ba
生物标志物的研究gydF4y2Ba开放获取gydF4y2Ba2022年7月26日gydF4y2Ba
癌症中的三维染色质结构和转录调控gydF4y2Ba
血液学与肿瘤学杂志gydF4y2Ba开放获取gydF4y2Ba5月4日gydF4y2Ba
访问选项gydF4y2Ba
访问《自然》和其他54种《自然》杂志gydF4y2Ba
获取Nature+,我们最超值的在线订阅gydF4y2Ba
每月29.99美元gydF4y2Ba
随时取消gydF4y2Ba
订阅这本杂志gydF4y2Ba
收到51个印刷问题和在线访问gydF4y2Ba
199.00美元一年gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
租或购买这篇文章gydF4y2Ba
只要这篇文章,只要你需要它gydF4y2Ba
39.95美元gydF4y2Ba
价格可能受当地税收的影响,在结账时计算gydF4y2Ba
数据可用性gydF4y2Ba
本研究生成的ChIP-seq、HiChIP、Hi-C、RNA-seq和单细胞多组学(10x Chromium Single Cell Multiome ATAC + Gene Expression)数据已存入GEO,可在登录号下查询gydF4y2BaGSE159986gydF4y2Ba.本研究生成的CRISPR-Cas9酶切后的纳米孔测序数据、WGS测序数据、sgRNA测序数据、靶向ecDNA测序数据以及PFGE测序数据均已存入SRA,登录号可查gydF4y2BaPRJNA670737gydF4y2Ba.本研究生成的光学作图数据已使用BioProject代码存入GenBankgydF4y2BaPRJNA731303gydF4y2Ba.本研究还使用了以下公开数据:TR14 H3K27ac ChIP-seqgydF4y2Ba93gydF4y2Ba(GEO:gydF4y2BaGSE90683gydF4y2Ba);col320 - dm, col320 - hsr和PC3 WGSgydF4y2Ba1gydF4y2Ba(SRA:gydF4y2BaPRJNA506071gydF4y2Ba);SNU16 WGSgydF4y2Ba60gydF4y2Ba(SRA:gydF4y2BaPRJNA523380gydF4y2Ba);及HK359 WGSgydF4y2Ba6gydF4y2Ba(SRA:gydF4y2BaPRJNA338012gydF4y2Ba).显微镜图像文件可在figsharegydF4y2Bahttps://doi.org/10.6084/m9.figshare.c.5624713gydF4y2Ba.gydF4y2Ba
代码的可用性gydF4y2Ba
本研究中使用的自定义代码可在gydF4y2Bahttps://github.com/ChangLab/ecDNA-hub-code-2021gydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
吴,S.等。环状ecDNA促进可接近染色质和高癌基因表达。gydF4y2Ba自然gydF4y2Ba575gydF4y2Ba, 699-703(2019)。gydF4y2Ba
高金,梁德华,任斌。三维基因组在转录调控和多能性中的作用。gydF4y2Ba细胞干细胞gydF4y2Ba14gydF4y2Ba, 762-775(2014)。gydF4y2Ba
郑浩,谢伟。三维基因组组织在细胞发育和分化中的作用。gydF4y2Ba细胞生物学。gydF4y2Ba20.gydF4y2Ba535-550(2019)。gydF4y2Ba
Bailey, C., Shoura, M. J., Mischel, P. S. & Swanton, C.染色体外dna缓解遗传限制,加速肿瘤进化。gydF4y2Ba安。肿瘤防治杂志。gydF4y2Ba31gydF4y2Ba, 884-893(2020)。gydF4y2Ba
Kim, H.等人。染色体外DNA与多种癌症的癌基因扩增和不良预后相关。gydF4y2BaNat,麝猫。gydF4y2Ba52gydF4y2Ba, 891-897(2020)。gydF4y2Ba
特纳,K. M.等。染色体外癌基因扩增驱动肿瘤进化和遗传异质性。gydF4y2Ba自然gydF4y2Ba543gydF4y2Ba, 122-125(2017)。gydF4y2Ba
李志刚,李志刚,李志刚。染色体外癌基因扩增在肿瘤发病和进化中的作用。gydF4y2BaNat. Rev. CancergydF4y2Ba19gydF4y2Ba, 283-288(2019)。gydF4y2Ba
考克斯,D.,尤肯,C. &斯普里格斯,a.i .儿童恶性肿瘤中的微小染色质体。gydF4y2Ba《柳叶刀》gydF4y2Ba286gydF4y2Ba, 55-58(1965)。gydF4y2Ba
van der Bliek, a.m., Lincke, C. R. & Borst, P. 3T6R50双分钟染色体的环状DNA。gydF4y2Ba核酸测定。gydF4y2Ba16gydF4y2Ba, 4841-4851(1988)。gydF4y2Ba
Hamkalo, B. A., Farnham, P. J., Johnston, R. & Schimke, R. T.甲氨蝶呤耐药小鼠3T3细胞系中微小染色体的超微结构特征。gydF4y2Ba国家科学院学报gydF4y2Ba82gydF4y2Ba, 1126-1130(1985)。gydF4y2Ba
毛瑞杰,赖文杰,李文杰,胡德,李文杰,等。人类细胞亚显微外染色体元件的研究进展。gydF4y2Ba自然gydF4y2Ba327gydF4y2Ba, 434-437(1987)。gydF4y2Ba
VanDevanter, d.r., Piaskowski, v.d., Casper, j.t., Douglass, e.c.和Von Hoff, d.d.在人体内成神经细胞瘤中环状染色体外DNA分子携带扩增MYCN原癌基因的能力。gydF4y2Ba中华泌尿外科肿瘤研究所。gydF4y2Ba82gydF4y2Ba, 1815-1821(1990)。gydF4y2Ba
内桑森,d.a.等人。通过动态调节染色体外突变EGFR DNA介导的靶向治疗耐药。gydF4y2Ba科学gydF4y2Ba343gydF4y2Ba, 72-76(2014)。gydF4y2Ba
Ståhl, F., Wettergren, Y. & Levan, G.多药耐药小鼠细胞中的扩增子结构:基因组DNA中对应于大环状DNA的非重排区域。gydF4y2Ba摩尔。细胞。医学杂志。gydF4y2Ba12gydF4y2Ba, 1179-1187(1992)。gydF4y2Ba
Vicario, R.等人。HER2基因扩增模式和抗HER2治疗的反应。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba10gydF4y2Ba, e0129876(2015)。gydF4y2Ba
卡罗尔,s.m.等。双分钟染色体可以由染色体缺失的前体产生。gydF4y2Ba摩尔。细胞。医学杂志。gydF4y2Ba8gydF4y2Ba, 1525-1533(1988)。gydF4y2Ba
Kitajima, K., Haque, M., Nakamura, H., Hirano, T. & Utiyama, H.二甲基亚甲亚胺诱导HL-60亚系中粒细胞分化不可逆性的丧失。gydF4y2Ba物化学。Biophys。Commun >,gydF4y2Ba288gydF4y2Ba, 1182-1187(2001)。gydF4y2Ba
Quinn, L. A., Moore, G. E., Morgan, R. T. & Woods, L. K.具有不寻常细胞产物的人结肠癌细胞系,双分,染色区域均匀。gydF4y2Ba癌症Res。gydF4y2Ba39gydF4y2Ba, 4914-4924(1979)。gydF4y2Ba
Storlazzi, c.t.等人。实体瘤中基因扩增为双分或均匀染色区域:起源和结构。gydF4y2Ba基因组Res。gydF4y2Ba20.gydF4y2Ba, 1198-1206(2010)。gydF4y2Ba
环状DNA在哺乳动物基因扩增中的重要性。gydF4y2Ba癌症Res。gydF4y2Ba49gydF4y2Ba, 1333-1340(1989)。gydF4y2Ba
库马尔,P.等人。ATAC-seq在癌症和细胞系中鉴定了数千个染色体外环状DNA。gydF4y2Ba科学。睡觉。gydF4y2Ba6gydF4y2Ba, eaba2489(2020)。gydF4y2Ba
莫顿,a.r.等人。功能增强子形成染色体外癌基因扩增。gydF4y2Ba细胞gydF4y2Ba179gydF4y2Ba, 1330-1341(2019)。gydF4y2Ba
赫尔姆绍尔等人。增强子劫持决定染色体外环状gydF4y2BaMYCNgydF4y2Ba成神经细胞瘤中的扩增子结构。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 5823(2020)。gydF4y2Ba
Itoh, N. & Shimizu, N.双分钟染色质的DNA复制依赖性核内重定位。gydF4y2Ba细胞科学。gydF4y2Ba111gydF4y2Ba, 3275-3285(1998)。gydF4y2Ba
Kanda, T, Sullivan, K. F. & Wahl, g.m.组蛋白- gfp融合蛋白使活哺乳动物细胞染色体动态的敏感分析。gydF4y2Ba咕咕叫。医学杂志。gydF4y2Ba8gydF4y2Ba, 377-385(1998)。gydF4y2Ba
Oobatake, Y. & Shimizu, N.染色体外双分粒中的双链断裂触发它们在细胞核中的聚集、微核和形态转化。gydF4y2Ba基因染色体癌症gydF4y2Ba59gydF4y2Ba, 133-143(2020)。gydF4y2Ba
贝利沃,B. J.等。使用Oligopaint FISH探针可视化基因组的多功能设计和合成平台。gydF4y2Ba国家科学院学报美国gydF4y2Ba109gydF4y2Ba, 21301-21306(2012)。gydF4y2Ba
Koche, R. P.等。染色体外环状DNA驱动成神经细胞瘤的致癌基因组重塑。gydF4y2BaNat。遗传学gydF4y2Ba52gydF4y2Ba, 29-34(2019)。gydF4y2Ba
帕克,s.c.j等。染色质拉伸增强子状态驱动细胞特异性基因调节和港湾人类疾病风险变异。gydF4y2Ba国家科学院学报美国gydF4y2Ba110gydF4y2Ba, 17921-17926(2013)。gydF4y2Ba
怀特,w.a.等人。主转录因子和中介在关键细胞识别基因上建立超级增强子。gydF4y2Ba细胞gydF4y2Ba153gydF4y2Ba, 307-319(2013)。gydF4y2Ba
Lovén, J.等。通过破坏超增强子选择性抑制肿瘤癌基因。gydF4y2Ba细胞gydF4y2Ba153gydF4y2Ba, 320-334(2013)。gydF4y2Ba
Filippakopoulos, P.等人。选择性抑制BET溴域。gydF4y2Ba自然gydF4y2Ba468gydF4y2Ba, 1067-1073(2010)。gydF4y2Ba
萨巴里,B. R.等。在超增强子处的共活化剂凝聚连接相分离和基因控制。gydF4y2Ba科学gydF4y2Ba361gydF4y2Ba, eaar3958(2018)。gydF4y2Ba
Ren, C.等。空间约束的串联溴域抑制支持持续抑制BRD4转录活性的TNBC细胞生长。gydF4y2Ba国家科学院学报美国gydF4y2Ba115gydF4y2Ba, 7949-7954(2018)。gydF4y2Ba
Deshpande, V.等人。使用AmpliconArchitect探索癌灶放大的景观。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 392(2019)。gydF4y2Ba
Luebeck, J.等人。AmpliconReconstructor集成了NGS和光学映射来解决焦距放大的复杂结构。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 4374(2020)。gydF4y2Ba
施瓦布,M., Klempnauer, K. H., Alitalo, K., Varmus, H. & Bishop, M.放大c-的5 '端重排gydF4y2BamycgydF4y2Ba在人类COLO 320细胞中与异常转录有关。gydF4y2Ba摩尔。细胞。医学杂志。gydF4y2Ba6gydF4y2Ba, 2752-2755(1986)。gydF4y2Ba
L 'Abbate, A.等。基因组组织和进化的双分/均匀染色区域与gydF4y2BaMYCgydF4y2Ba人类癌症中的扩增。gydF4y2Ba核酸测定。gydF4y2Ba42gydF4y2Ba, 9131-9145(2014)。gydF4y2Ba
Hann, s.r., King, m.w., Bentley, d.l., Anderson, c.w.和Eisenman, r.n. c-myc外显子1的非aug翻译启动产生n端不同的蛋白,其合成在Burkitt淋巴瘤中被中断。gydF4y2Ba细胞gydF4y2Ba52gydF4y2Ba, 185-195(1988)。gydF4y2Ba
Carramusa, L.等。PVT-1癌基因是Myc蛋白靶蛋白,在转化细胞中过表达。gydF4y2Baj .细胞。杂志。gydF4y2Ba213gydF4y2Ba, 511-518(2007)。gydF4y2Ba
赵,S. W.等。lncRNA基因启动子gydF4y2BaPVT1gydF4y2Ba是一种肿瘤抑制DNA边界元件。gydF4y2Ba细胞gydF4y2Ba173gydF4y2Ba, 1398-1412(2018)。gydF4y2Ba
Tolomeo, D., Agostini, A., Visci, G., Traversa, D. & Storlazzi, C. T.。gydF4y2BaPVT1gydF4y2Ba:一种长链非编码RNA,经常参与肿瘤相关融合转录本。gydF4y2Ba基因gydF4y2Ba779gydF4y2Ba, 145497(2021)。gydF4y2Ba
Mumbach, m.r.等人。HiChIP:高效、灵敏的蛋白质定向基因组结构分析。gydF4y2BaNat方法。gydF4y2Ba13gydF4y2Ba, 919(2016)。gydF4y2Ba
Fulco, c.p.等。来自数千个CRISPR扰动的增强子-启动子调控的接触活性模型。gydF4y2BaNat,麝猫。gydF4y2Ba51gydF4y2Ba, 1664-1669(2019)。gydF4y2Ba
Park, J.等。CD44和FGFR2之间通过c-myc相互调节电路控制胃癌细胞的生长。gydF4y2BaOncotargetgydF4y2Ba7gydF4y2Ba, 28670-28683(2016)。gydF4y2Ba
弗隆,E. E. M.莱文,M.发育增强子和染色体拓扑结构。gydF4y2Ba科学gydF4y2Ba361gydF4y2Ba, 1341-1345(2018)。gydF4y2Ba
朱毅,等。致癌染色体外DNA作为移动增强子,在全球范围内放大染色体转录。gydF4y2Ba癌症细胞gydF4y2Ba39gydF4y2Ba, 694-707(2021)。gydF4y2Ba
薛,K. S., Hooper, K. A., Ollodart, A. R., Dingens, A. S. & Bloom, J. D.不同病毒变体之间的合作促进细胞培养中H3N2流感病毒的生长。gydF4y2BaElifegydF4y2Ba5gydF4y2Ba, e13974(2016)。gydF4y2Ba
Vignuzzi, M., Stone, J. K., Arnold, J. J., Cameron, C. E. & Andino, R.准种多样性通过病毒种群中的合作相互作用决定致病机制。gydF4y2Ba自然gydF4y2Ba439gydF4y2Ba, 344-348(2006)。gydF4y2Ba
Henssen, A.等。通过BET-bromodomain抑制mycn驱动的转录。gydF4y2Ba中国。癌症Res。gydF4y2Ba22gydF4y2Ba, 2470-2481(2016)。gydF4y2Ba
谢,等。3D ATAC-PALM:可访问基因组的超分辨率成像。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 430 - 436(2020)。gydF4y2Ba
安布罗斯,P. F.等人。成神经细胞瘤分子诊断的国际共识:来自国际成神经细胞瘤风险组(INRG)生物学委员会的报告。gydF4y2BaBr。j .癌症gydF4y2BaOne hundred.gydF4y2Ba, 1471-1482(2009)。gydF4y2Ba
Balaban-Malenbaum, G. & Gilbert, F.人成神经细胞瘤细胞系染色体中的双分钟染色体和均匀染色区域。gydF4y2Ba科学gydF4y2Ba198gydF4y2Ba, 739-741(1977)。gydF4y2Ba
Marrano, P. Irwin, m.s. & Thorner, P. S.异质性gydF4y2BaMYCNgydF4y2Ba神经母细胞瘤在诊断、治疗、复发和转移中的扩增。gydF4y2Ba基因染色体癌症gydF4y2Ba56gydF4y2Ba, 28-41(2017)。gydF4y2Ba
Villamón, E.等。神经母细胞瘤的遗传不稳定性和瘤内异质性gydF4y2BaMYCNgydF4y2Ba扩增加11q缺失。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba8gydF4y2Ba, e53740(2013)。gydF4y2Ba
Schindelin, J.等。斐济:生物图像分析的开源平台。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 676-682(2012)。gydF4y2Ba
Rajkumar, U.等。EcSeg:包含染色体外DNA的中期图像的语义分割。gydF4y2BaIsciencegydF4y2Ba21gydF4y2Ba, 428-435(2019)。gydF4y2Ba
韦奇,S. L.等。相关函数量化超分辨率图像和估计由于计数过多的明显聚类。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba7gydF4y2Ba, e31457(2012)。gydF4y2Ba
Bolger, a.m., Lohse, M. & Usadel, B. Trimmomatic:用于Illumina序列数据的灵活修剪器。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 2114(2014)。gydF4y2Ba
甘地等人。肿瘤细胞系百科全书的下一代特性。gydF4y2Ba自然gydF4y2Ba569gydF4y2Ba, 503-508(2019)。gydF4y2Ba
诺曼诺,D.等。利用TetR作为模型搜索器,探索哺乳动物细胞中dna结合蛋白的目标搜索。gydF4y2BaCommun Nat。gydF4y2Ba6gydF4y2Ba, 7357(2015)。gydF4y2Ba
杨晓燕,张晓燕,张晓燕,等。蛋白质介导的重复序列染色体配对研究。gydF4y2BaJ. Mol.生物学。gydF4y2Ba426gydF4y2Ba, 550-557(2014)。gydF4y2Ba
格林,J. B.等人。红移罗丹明染料优化与功能化的一般方法。gydF4y2BaNat方法。gydF4y2Ba17gydF4y2Ba, 815-821(2020)。gydF4y2Ba
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 357-359(2012)。gydF4y2Ba
张杨,等。基于模型的ChIP-seq (MACS)分析。gydF4y2Ba基因组医学杂志。gydF4y2Ba9gydF4y2Ba, r137(2008)。gydF4y2Ba
Ramírez, F. et al. deepTools2:用于深度测序数据分析的下一代web服务器。gydF4y2Ba核酸测定。gydF4y2Ba44gydF4y2Ba, w160-w165(2016)。gydF4y2Ba
Sedlazeck, F. J.等。利用单分子测序精确检测复杂结构变异。gydF4y2BaNat方法。gydF4y2Ba15gydF4y2Ba, 461-468(2018)。gydF4y2Ba
Overhauser, J. ingydF4y2Ba脉冲场凝胶电泳,分子生物学中的方法gydF4y2Ba第12卷(编。Burmeister, M. & Ulanovsky, L.) 129-134 (Humana出版社,1992)。gydF4y2Ba
皮切利等人。用于大规模测序项目的Tn5转座酶和标记程序。gydF4y2Ba基因组Res。gydF4y2Ba24gydF4y2Ba, 2033-2040(2014)。gydF4y2Ba
Corces, m.r.等人。一种改进的ATAC-seq协议减少了背景,并能够对冷冻组织进行询问。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 959-962(2017)。gydF4y2Ba
Talevich, E., Shain, A. H., Botton, T. & Bastian, B. C. CNVkit:来自靶向DNA测序的全基因组拷贝数检测和可视化。gydF4y2Ba公共科学图书馆第一版。医学杂志。gydF4y2Ba12gydF4y2Ba, e1004873(2016)。gydF4y2Ba
Raeisi Dehkordi, S., Luebeck, J. & Bafna, V. FaNDOM:快速嵌套的基于距离的光学地图播种。gydF4y2Ba模式gydF4y2Ba2gydF4y2Ba, 100248(2021)。gydF4y2Ba
哈斯,B. J.等。基于读映射和从头融合转录本组装方法的融合转录本检测准确性评估。gydF4y2Ba基因组医学杂志。gydF4y2Ba20.gydF4y2Ba, 213(2019)。gydF4y2Ba
Hahne, F. & Ivanek, R. ingydF4y2Ba统计基因组学,分子生物学中的方法gydF4y2Ba第1418卷(编)Mathé, E. & Davis, S.) 335-351 (Humana出版社,2016)。gydF4y2Ba
Butler, A, Hoffman, P, Smibert, P, Papalexi, E. & Satija, R.整合不同条件、技术和物种的单细胞转录组数据。gydF4y2Ba生物科技Nat。》。gydF4y2Ba36gydF4y2Ba, 411-420(2018)。gydF4y2Ba
Granja, J. M.等。ArchR是一个可扩展的软件包,用于整合单细胞染色质可达性分析。gydF4y2BaNat,麝猫。gydF4y2Ba53gydF4y2Ba, 403-411(2021)。gydF4y2Ba
情感,a.t.等。人类免疫细胞发育和瘤内T细胞衰竭的大规模平行单细胞染色质景观。gydF4y2Ba生物科技Nat。》。gydF4y2Ba37gydF4y2Ba, 925-936(2019)。gydF4y2Ba
Mumbach, m.r.等人。原代人类细胞中的增强子连接组可识别疾病相关DNA元件的靶基因。gydF4y2BaNat,麝猫。gydF4y2Ba49gydF4y2Ba1602-1612(2017)。gydF4y2Ba
Mumbach, m.r.等人。HiChIRP揭示rna相关染色体构象。gydF4y2BaNat方法。gydF4y2Ba16gydF4y2Ba, 489-492(2019)。gydF4y2Ba
仆人,N.等。HiC-Pro:用于Hi-C数据处理的优化灵活管道。gydF4y2Ba基因组医学杂志。gydF4y2Ba16gydF4y2Ba, 259(2015)。gydF4y2Ba
Bhattacharyya, S., Chandra, V., Vijayanand, P. & Ay, F.通过FitHiChIP从HiChIP数据中识别显著染色质接触。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 4221(2019)。gydF4y2Ba
饶,s.s.p.等。千碱基分辨率的人类基因组3D图揭示了染色质环的原理。gydF4y2Ba细胞gydF4y2Ba159gydF4y2Ba, 1665-1680(2014)。gydF4y2Ba
维达尔等人。OneD:增加异常核型Hi-C样本的重现性。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, e49(2018)。gydF4y2Ba
弗林,R. A.等。SARS-CoV-2 rna -宿主蛋白相互作用的发现和功能探究。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 2394-2411(2021)。gydF4y2Ba
李,W.等。MAGeCK能够从基因组规模的CRISPR/Cas9敲除筛选中强大地识别必要基因。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 554(2014)。gydF4y2Ba
李,H. & Durbin R.快速和准确的长读对齐与Burrows-Wheeler变换。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 589-595(2010)。gydF4y2Ba
谢宁,等人。通过浅全基因组测序分析新鲜和福尔马林固定标本的DNA拷贝数,识别和排除基因组组装中的问题区域。gydF4y2Ba基因组Res。gydF4y2Ba24gydF4y2Ba, 2022-2032(2014)。gydF4y2Ba
哈迪,K.等。在数千个癌症基因组图谱中发现了不同类别的复杂结构变异。gydF4y2Ba细胞gydF4y2Ba183gydF4y2Ba, 197-210(2020)。gydF4y2Ba
Blumrich, A.等人。的gydF4y2BaFRA2CgydF4y2Ba常见的易碎站点地图的边界gydF4y2BaMYCNgydF4y2Ba成神经细胞瘤中的扩增子,并与不同癌症的总体染色体重排有关。gydF4y2Ba嗡嗡声。摩尔,麝猫。gydF4y2Ba20.gydF4y2Ba, 1488-1501(2011)。gydF4y2Ba
戈戈林等人。CDK4抑制恢复GgydF4y2Ba1gydF4y2Ba-S逮捕在gydF4y2BaMYCNgydF4y2Ba-放大的成神经细胞瘤细胞在阿霉素诱导的DNA损伤的背景下。gydF4y2Ba细胞周期gydF4y2Ba12gydF4y2Ba, 1091-1104(2013)。gydF4y2Ba
杜兰德,n.c.等人。Juicer提供了一键式系统,用于分析循环分辨率Hi-C实验。gydF4y2Ba细胞系统。gydF4y2Ba3.gydF4y2Ba, 95-98(2016)。gydF4y2Ba
奈特,P. A. &鲁伊斯,D.矩阵平衡的快速算法。gydF4y2BaIMA J.数字。分析的gydF4y2Ba33gydF4y2Ba, 1029-1047(2013)。gydF4y2Ba
Boeva, V.等人。由转录回路定义的成神经细胞瘤细胞身份的异质性。gydF4y2BaNat,麝猫。gydF4y2Ba49gydF4y2Ba, 1408-1413(2017)。gydF4y2Ba
确认gydF4y2Ba
我们感谢Chang、Liu、Mischel和Bafna实验室成员的讨论;R. Zermeno, M. Weglarz和L. Nichols在斯坦福共享FACS设施协助进行细胞分类实验;X. Ji, D. Wagh和J. Coller在斯坦福功能基因组学设施协助高通量测序;以及Bionano Genomics的A. Pang在光学测绘方面的帮助。H.Y.C.得到NIH R35-CA209919和RM1-HG007735的支持;K.L.H.获得了斯坦福大学研究生奖学金的支持;K.E.Y.获得了美国国家科学基金会研究生研究奖学金计划(NSF DGE-1656518)、斯坦福大学研究生奖学金和NCI博士预科到博士后过渡奖(NIH F99CA253729)的支持。这个项目的细胞分类是在斯坦福共享FACS设施的仪器上完成的。测序由斯坦福功能基因组学设施进行(由NIH拨款S10OD018220和1S10OD021763支持)。在UCSD显微镜核心(由NINDS NS047101支持)中的仪器上进行显微镜检查。 A.G.H. is supported by the Deutsche Forschungsgemeinschaft (DFG; German Research Foundation) (398299703) and the European Research Council (ERC) under the European Union’s Horizon 2020 research and innovation programme (grant agreement no. 949172). Z.L. is a Janelia Group Leader, and H.Y.C. and R.T. are Investigators of the Howard Hughes Medical Institute.
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
K.L.H、k.e.y和h.y.c构想了这个项目。K.L.H.进行并分析了CRISPRi和体外ecDNA消化和PFGE实验,并分析了单细胞多组学、RNA-seq和ATAC-seq实验。k.e.y进行并分析了中期DNA FISH成像、ChIP-seq、HiChIP、WGS、COLO320-DM纳米孔测序和JQ1微扰实验。L.X.进行并分析了间期DNA和RNA FISH成像、TetO-eGFP细胞系生成和活细胞成像以及PVT1p-nLuc成像实验。Q.S.执行并分析了除PVT1p-nLuc RNA和DNA FISH外的所有荧光素酶报告实验,并帮助进行了CRISPRi实验。K.H.和R.S.分析了TR14 Hi-C数据和扩增子重建。J.L.和S.R.D.分析了COLO320-DM WGS、纳米孔测序、光学作图数据和扩增子重建。J.T.L, S.W, C.C.和J.T.进行并分析了DNA FISH成像。rc.g生成TR14的Hi-C、DNA FISH、WGS和纳米孔测序数据。N.E.W.对MS645处理后的小分子抑制剂实验和DNA FISH成像进行了分析。 M.E.V. performed Hi-C experiments and data analysis for TR14. I.T.-L.W. performed metaphase DNA FISH imaging. C.V.D. performed and analysed ChIP–seq experiments. K.K. performed HiChIP experiments. J.A.B. helped with CRISPRi experimental design and cloning of the sgRNA pool. R.L. performed RNA-seq experiments. U.R. analysed metaphase DNA FISH data. J.F. generated COLO320-DM WGS data. M.R.C. and J.M.G. wrote the HiChIP data processing pipeline. M.R.C., J.C.R., A.B., A.T.S., R.T., S.M., V.B., A.G.H., P.S.M., Z.L. and H.Y.C. guided data analysis and provided feedback on experimental design. K.L.H., K.E.Y. and H.Y.C. wrote the manuscript with input from all authors.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
他是Accent Therapeutics、Boundless Bio和Cartography Biosciences的联合创始人,也是10x Genomics、Arsenal Biosciences和Spring Discovery的顾问。P.S.M.是无边生物公司的联合创始人。他拥有股权,并担任科学顾问委员会主席,为此他获得了报酬。V.B.是无边生物的联合创始人和顾问。A.T.S.是Immunai和Cartography Biosciences的创始人。K.E.Y.是生物制图学的顾问。gydF4y2Ba
额外的信息gydF4y2Ba
同行评审信息gydF4y2Ba自然gydF4y2Ba感谢Charles Lin和其他匿名审稿人对本工作的同行评审所作的贡献。gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
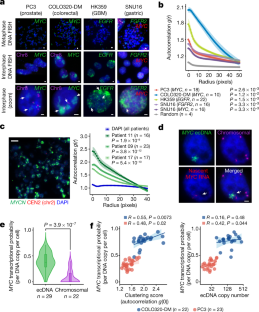
扩展数据图1 ecDNA FISH策略和拷贝数估计。gydF4y2Ba
一个gydF4y2Ba, WGS跟踪DNA FISH探针位置。对于COLO320-DM和PC3, 1.5 MbgydF4y2BaMYCgydF4y2BaFISH探针(图。gydF4y2Ba1 a, bgydF4y2Ba),大小为100kbgydF4y2BaMYCgydF4y2BaFISH探针(图。gydF4y2Ba1 d-fgydF4y2Ba),或使用1.5 Mb 8号染色体FISH探针。商业探针用于SNU16和HK359细胞。gydF4y2BabgydF4y2Ba,具有代表性的DNA FISH图像,使用染色体和1.5 MbgydF4y2BaMYCgydF4y2Ba非ecdna扩增的HCC1569探针显示染色体位点预期的成对信号。gydF4y2BacgydF4y2Ba,个体col320 - dm细胞ecDNA自相关聚类gydF4y2BaggydF4y2Ba(gydF4y2BargydF4y2Ba).gydF4y2BadgydF4y2Ba,代表性的FISH图像显示ecDNA在原发性成神经细胞瘤肿瘤中聚集(患者11和17)。gydF4y2BaegydF4y2Ba,利用自相关技术对所有3例患者的单个原发肿瘤细胞进行ecDNA聚类gydF4y2BaggydF4y2Ba(gydF4y2BargydF4y2Ba).gydF4y2BafgydF4y2Ba,比较gydF4y2BaMYCgydF4y2Ba根据WGS (n=7个基因组箱与DNA FISH探针重叠)、中期FISH (n=82个细胞)和间期FISH (n=47个细胞)计算COLO320-DM中的拷贝数。用双面Wilcoxon检验确定p值。gydF4y2BaggydF4y2Ba,新生的代表形象gydF4y2BaMYCgydF4y2BaPC3细胞中新生RNA(内含子)和总RNA(外显子)FISH探针重叠的RNA FISH(独立重复两次)。gydF4y2BahgydF4y2Ba,结合DNA FISH的代表图像gydF4y2BaMYCgydF4y2BaecDNA (100kb探针)和带有新生的染色体DNAgydF4y2BaMYCgydF4y2Bacol320 - dm细胞中的RNA FISH(独立重复4次)。gydF4y2Ba我gydF4y2Ba,gydF4y2BaMYCgydF4y2Ba通过新生RNA FISH归一化到DNA拷贝数来测量转录概率,通过FISH将单例ecdna与col320 - dm枢纽中发现的ecdna进行比较(方框中线,中位数;方框限制,上下四分位数;盒须,1.5倍四分位范围)。为了控制少量ecdna转录概率中的噪声,我们根据hub大小分组随机重新采样RNA FISH数据并计算转录概率。小提琴图表示每个ecDNA枢纽基于枢纽大小匹配采样的转录概率。用双面Wilcoxon检验确定p值。gydF4y2Ba
图2用于活细胞ecDNA成像的tetra - gfp COLO320-DM细胞的生成。gydF4y2Ba
一个gydF4y2Ba,基于TetO阵列敲入和tetra - egfp标记的ecDNA成像(左)。TetO-eGFP col320 - dm细胞中TetO-eGFP信号在时间过程中指定时间点的代表性图像(右;独立重复两次)。gydF4y2BabgydF4y2Ba, ecDNA-TetO COLO320-DM细胞中的GFP信号。在活细胞中tetra -eGFP和单体tetra -eGFP(A206K)标记的ecDNA中心似乎比固定细胞的DNA FISH研究中更小,这可能是因为TetO阵列没有集成在所有ecDNA分子中,并且在DNA FISH和eGFP二聚过程中存在变性引起的潜在差异。gydF4y2BacgydF4y2Ba, ecDNA中心直径,单位为微米(方框中线,中位数;方框限制,上下四分位数;盒须,1.5倍四分位范围)。tet -eGFP标记的中心比单体tet -eGFP(A206K)标记的中心略小,这可能是由于eGFP二聚化效应(方法)。用双面Wilcoxon检验确定p值。gydF4y2BadgydF4y2Ba,每个细胞的ecDNA中心数。直线表示中位数。用双面Wilcoxon检验确定p值。gydF4y2BaegydF4y2Ba在chr8-chromosomal-TetO (chr8:116,860,000-118,680,000,左)和ecDNA-TetO (TetO-eGFP COLO320-DM,右)col320 - dm细胞中的TetO-eGFP信号。gydF4y2BafgydF4y2Ba, chr8- chromosome - teto和ecDNA-TetO病灶的荧光强度。gydF4y2BaggydF4y2Ba,gydF4y2BahgydF4y2Ba,每个病灶推测的ecDNA拷贝数(g;N =病灶数/细胞),每个细胞(h;n =细胞数)用于ecDNA-TetO标记细胞,基于相对于chr8-染色体- teto焦点的总和荧光强度。直线表示中位数。gydF4y2Ba我gydF4y2Ba,未集成TetO阵列的亲本COLO320-DM中tetra - gfp信号的代表图像,显示最小的tetra - gfp焦点。gydF4y2BajgydF4y2Ba, ecDNA (TetO-eGFP)和BRD4 (HaloTag)焦点的平均荧光强度横跨最大ecDNA (TetO-eGFP)信号中心的一条线。数据为n=5个ecDNA病灶的平均值±标准差。gydF4y2BakgydF4y2Ba,无TetO阵列集成覆盖BRD4-HaloTag信号的COLO320-DM细胞tetra - egfp信号代表图像。虚线为核边界。我们在没有TetO阵列整合的COLO320-DM细胞亚群中注意到细胞质tetra - egfp信号,但它没有与BRD4-HaloTag共定位。gydF4y2BalgydF4y2Ba,gydF4y2BaMYCgydF4y2Ba用RT-qPCR检测亲本COLO320-DM细胞和BRD4-HaloTag COLO320-DM细胞经DMSO或500 nM JQ1处理6小时的RNA,显示相似水平gydF4y2BaMYCgydF4y2BaBRD4表位标记后对JQ1抑制的转录和敏感性。3个生物重复之间的数据为平均值±标准差。gydF4y2BaPgydF4y2Ba由双面学生决定的值gydF4y2BatgydF4y2Ba以及。gydF4y2Ba
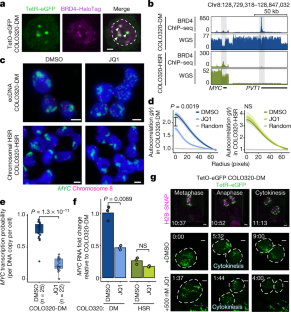
扩展数据图3 BET抑制导致ecDNA hub扩散。gydF4y2Ba
一个gydF4y2Ba,具有代表性的中期FISH图像和示意图显示了COLO320-DM中的ecDNA和COLO320-HSR中的染色体HSRs(在COLO320-DM中独立重复两次,在COLO320-HSR中不重复)。gydF4y2BabgydF4y2Ba, BRD4级ChIP-seq信号。ecDNA或HSR扩增的峰值被高亮显示,并用最近的基因标记。gydF4y2BacgydF4y2Ba、ATAC-seq、BRD4 ChIP-seq、H3K27ac ChIP-seq和扩增MYC位点的WGS。gydF4y2BadgydF4y2Ba,经DMSO或500 nM JQ1处理6小时后,个体COLO320-DM细胞间期FISH成像的ecDNA位置数量(包括>1 ecDNA的ecDNA中心和单个ecDNA)。N =每个条件量化的细胞数。用双面Wilcoxon检验确定p值。gydF4y2BaegydF4y2Ba,在DMSO或500 nM JQ1处理6小时后,col320 - dm的间期FISH成像中每个ecDNA位置都有ecDNA拷贝(框中线,中位;方框限制,上下四分位数;盒须,1.5倍四分位范围)。N =每种条件下定量的ecDNA位点数。用双面Wilcoxon检验确定p值。gydF4y2BafgydF4y2Ba,在指定时间点用DMSO或500 nM JQ1处理后tetra - egfp标记的ecDNA的代表性实时图像(上;独立重复两次)和ecDNA中心放大图(底部)。gydF4y2BaggydF4y2Ba,经DMSO、500 nM JQ1或1% 1,6-己二醇处理6小时的COLO320-DM细胞中DNA/RNA组合FISH的代表图像。gydF4y2BahgydF4y2Ba,gydF4y2BaMYCgydF4y2Ba用DMSO、1% 1,6-己二醇或100µg/mL α -amanitin处理6 h后,用双DNA/RNA FISH测量转录概率(方框中线,中位数;方框限制,上下四分位数;箱形须,1.5倍四分位范围;N =单元格数)。用双面Wilcoxon检验确定p值。gydF4y2Ba我gydF4y2Ba,用1% 1,6-己二醇或100µg/mL α -amanitin处理6 h, col320 - dm间期MYC ecDNA的代表性DNA FISH图像。gydF4y2BajgydF4y2Ba, ecDNA在间期细胞中的自相关聚类gydF4y2BaggydF4y2Ba(gydF4y2BargydF4y2Ba),用DMSO、1% 1,6-己二醇或100µg/mL α -amanitin处理6小时。数据为均值±标准差(每个条件定量n = 10个细胞)。gydF4y2BakgydF4y2Ba, DMSO或500 nM JQ1处理细胞6 h后,所有BRD4峰上的平均BRD4 ChIP-seq信号和热图。gydF4y2BalgydF4y2Ba,用不同浓度的JQ1处理48小时后,通过ATP水平(CellTiterGlo)测定细胞活力。3个生物重复之间的数据为平均值±标准差。gydF4y2BaPgydF4y2Ba由双面学生决定的值gydF4y2BatgydF4y2Ba以及。gydF4y2Ba米gydF4y2Ba,不同JQ1浓度处理72 h后细胞增殖。3个生物重复之间的数据为平均值±标准差。gydF4y2BangydF4y2Ba,不同JQ1浓度在72小时内处理后的细胞倍增时间(上)或正常化至dmso处理后的细胞(下)。3个生物重复之间的数据为平均值±标准差。gydF4y2BaPgydF4y2Ba由双面学生决定的值gydF4y2BatgydF4y2Ba以及。gydF4y2BaogydF4y2Ba,gydF4y2BaMYCgydF4y2Ba用指示抑制剂处理6小时后,RT-qPCR检测RNA(上;每个点代表一个生物复制,DMSO和JQ1治疗n=6,所有其他药物治疗n=3)。数据为平均值±标准差。gydF4y2BaPgydF4y2Ba由双面学生决定的值gydF4y2BatgydF4y2Ba以及。抑制剂组,蛋白靶点,作用意义gydF4y2BaMYCgydF4y2Ba转录,以及对ecDNA和HSR转录影响的比较(下)。gydF4y2BapgydF4y2Ba,gydF4y2Ba问gydF4y2Ba,代表性DNA FISH图像(gydF4y2BapgydF4y2Ba)和自相关聚类gydF4y2BaggydF4y2Ba(gydF4y2BargydF4y2Ba) (gydF4y2Ba问gydF4y2Ba)gydF4y2BaMYCgydF4y2Ba用DMSO或500 nM MS645处理col320 - dm中的ecdna 6小时。数据为均值±SEM。半径= 0处双面Wilcoxon检验确定的p值。gydF4y2Ba
图4 col320 - dm ecDNA扩增子结构重建。gydF4y2Ba
一个gydF4y2Ba,结构变体(SV)视图的AmpliconArchitect (AA)重建的gydF4y2BaMYCgydF4y2Ba结肠320- dm细胞中的扩增子。gydF4y2BabgydF4y2Ba, col320 - dm细胞的纳米孔测序(左)和读取长度的分布。gydF4y2BacgydF4y2Ba通过WGS和纳米孔测序检测到col320 - dm的连接。gydF4y2BadgydF4y2Ba,用于光学映射和统计的分子长度。gydF4y2BaegydF4y2Ba,整合WGS、光学作图和体外ecDNA消化后重建COLO320-DM ecDNA。标记起始染色体和对应坐标(hg19)。三条内环形轨道(浅棕色、石板色和棕色);引导A, B和C)表示使用三种不同的sgrna及其预期大小作为Cas9裂解结果的预期片段。引导序列见补充表gydF4y2Ba2gydF4y2Ba(PFGE_guide_A-C)。gydF4y2BafgydF4y2Ba,体外Cas9酶切col320 - dm ecDNA,然后用PFGE酶切(左)。碎片大小的确定基于gydF4y2Bah . wingeigydF4y2Ba而且gydF4y2Ba酿酒酵母gydF4y2Ba梯子。未裁剪的凝胶图像见补充图。gydF4y2Ba1gydF4y2Ba.中间面板显示的短读测序gydF4y2BaMYCgydF4y2Ba所有分离片段的ecDNA扩增子,按片段大小排序。右图显示了通过光学映射重建的预期片段大小的一致性,以及通过体外Cas9消化观察到的片段大小(不一致的片段圈出)。每次sgRNA消化在一个独立的实验中进行。gydF4y2BaggydF4y2Ba,显示共定位的中期FISH图像gydF4y2BaMYCgydF4y2Ba,gydF4y2BaPCAT1gydF4y2Ba而且gydF4y2Ba确切地gydF4y2Ba正如光学绘图和体外消化预测的那样。N = 20个细胞,1270个ecdna定量gydF4y2BaMYCgydF4y2Ba/gydF4y2BaPCAT1gydF4y2BaDNA FISH和n = 15个细胞和678个ecdnagydF4y2BaMYCgydF4y2Ba/gydF4y2Ba确切地gydF4y2BaDNA FISH来自一个实验。gydF4y2BahgydF4y2Ba,通过RT-qPCR检测col320 - dm细胞中稳定表达dCas9-KRAB和指示sgRNAs的指示转录本的RNA表达(n=2个生物重复)。规范gydF4y2BaMYCgydF4y2Ba引物MYC_exon1_fw和MYC_exon2_rv扩增;融合gydF4y2BaPVT1-MYCgydF4y2BaPVT1_exon1_fw和MYC_exon2_rv扩增;用total_MYC_exon2_fw和total_MYC_exon2_rv扩增total MYC。引物序列见补充表gydF4y2Ba1gydF4y2Ba导序见补充表gydF4y2Ba2gydF4y2Ba.gydF4y2Ba我gydF4y2Ba,连接处的对齐读数gydF4y2BaPVT1-MYCgydF4y2Ba断点。gydF4y2Ba
图5单细胞多组分析揭示了与之相关的组合和异质ecDNA调控元件活性gydF4y2BaMYCgydF4y2Ba表达式。gydF4y2Ba
一个gydF4y2Ba联合单细胞RNA和ATAC-seq同时检测基因表达和染色质可及性,并识别与之相关的调控元件gydF4y2BaMYCgydF4y2Ba表达式。gydF4y2BabgydF4y2Ba,独特的ATAC-seq片段和RNA特征的细胞通过过滤器(都是log2转换)。gydF4y2BacgydF4y2Ba,之间的相关性gydF4y2BaMYCgydF4y2Ba可及性评分和归一化RNA表达。gydF4y2BadgydF4y2Ba,来自RNA或ATAC-seq数据的UMAP(左)。对数标准化和缩放gydF4y2BaMYCgydF4y2BaRNA表达(右上)和gydF4y2BaMYCgydF4y2Ba可达性评分(右下)在ATAC-seq UMAP上可视化,显示细胞水平的异质性gydF4y2BaMYCgydF4y2Ba含ecdna的COLO320-DM中的RNA-seq和ATAC-seq信号。gydF4y2BaegydF4y2Ba的基因表达评分(用Seurat在R中计算)gydF4y2BaMYCgydF4y2Ba-上调基因(基因集M6506,分子特征库;MSigDB)gydF4y2BaMYCgydF4y2BaRNA分位数箱。水平线标记中间值。图中显示了所有单个细胞的总体方差(上图)。由双侧f检验确定p值。gydF4y2BafgydF4y2Ba,gydF4y2BaMYCgydF4y2Ba顶部和底部箱子的表达水平(左)。标准化ATAC-seq覆盖显示(右)。gydF4y2BaggydF4y2Ba,与col320 - hsr中的染色体HSRs相比,col320 - dm ecdna上识别的可变元素数量(左)。ecDNA上有45个唯一的可变元件。ecDNA上的所有可变元素显示在右侧(y轴显示-log10(FDR),圆点大小表示log2倍变化。的相对位置突出显示并命名了五个最重要的变量元素gydF4y2BaMYCgydF4y2BaTSS(负,5 ';积极的,3 ')。gydF4y2BahgydF4y2Ba,估计之间的相关性gydF4y2BaMYCgydF4y2Ba复制数字和规范化的log2转换gydF4y2BaMYCgydF4y2Ba所有个体细胞的表达均表现出与表达增加相关的高水平拷贝数变异性,特别是COLO320-DM。gydF4y2Ba我gydF4y2Ba估计,gydF4y2BaMYCgydF4y2Ba所有细胞箱的扩增子拷贝数gydF4y2BaMYCgydF4y2BaRNA表达。gydF4y2BajgydF4y2Ba中确定的五个最显著变量元素的ATAC-seq覆盖率的放大图gydF4y2BaggydF4y2Ba(由虚线框标记)。gydF4y2BakgydF4y2Ba,高单元和低单元箱中TSS富集的相似分布,表明变量元素可达性的差异并不是数据质量差异的人为因素。gydF4y2BalgydF4y2Ba,平均拷贝数回归,对数归一化,缩放ATAC-seq覆盖差异峰值相对均值gydF4y2BaMYCgydF4y2Ba橙色为每个细胞箱的RNA(对数归一化,平均中心,缩放)。来自相同扩增子区间的相同数量的随机非微分峰,以灰色显示。误差带显示线性模型的95%置信区间。gydF4y2Ba米gydF4y2Ba,的累积概率gydF4y2BaMYCgydF4y2Ba单细胞ATAC-seq数据和DNA FISH数据的扩增子拷贝数分布(以平均为中心,按比例分布),表明单细胞ATAC-seq数据的拷贝数估计反映了DNA FISH测量的ecDNA拷贝数的异质性。由Kolmogorov-Smirnov检验确定的p值(1000次自举模拟)。gydF4y2Ba
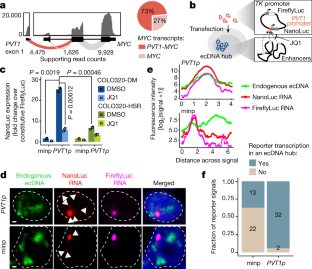
图6 col320 - dm内源性增强子连接组gydF4y2BaMYCgydF4y2BaecDNA扩增子和启动子序列效应,gydF4y2Ba独联体gydF4y2Ba增强子,BET抑制episomal报告激活。gydF4y2Ba
一个gydF4y2Ba,从上到下:COLO320-DM H3K27ac HiChIP接触图(kr归一化读计数,10kb分辨率),重构的COLO320-DM扩增子,H3K27ac ChIP-seq信号,BRD4 ChIP-seq信号,WGS覆盖,交互剖面gydF4y2BaPVT1gydF4y2Ba(上,深粉色)和gydF4y2BaMYCgydF4y2Ba(底部,淡粉色)10 kb分辨率的启动子,FitHiChIP循环如下所示,调整后的p值着色。通过scATAC识别的活性元件和重叠的H3K27ac HiChIP触点,根据到MYC起始位点的基因组距离命名:−1132E,−1087E,−679E,−655E,−401E,−328E,−85E。gydF4y2BabgydF4y2Ba10 kb分辨率下COLO320-DM H3K27ac HiChIP矩阵归一化方法的比较。HiChIP信号对不同的归一化方法具有鲁棒性。gydF4y2BacgydF4y2Ba,质粒NanoLuc荧光素酶信号的定量gydF4y2BaPVT1pgydF4y2Ba-,gydF4y2BaminpgydF4y2Ba,或gydF4y2BaMYCpgydF4y2Ba驱动NanoLuc报告表达式。通过将NanoLuc读数归一化到Firefly读数来计算荧光素酶信号。条形图显示平均值±SEM。gydF4y2BaPgydF4y2Ba使用双面Student 's计算gydF4y2BatgydF4y2Ba-test (n=3个生物重复)。gydF4y2BadgydF4y2Ba,在pvt1p -报告者和minp-报告者转染的细胞中,小提琴图显示NanoLuc报告RNA的平均荧光强度和信号大小。p值采用双面Wilcoxon检验计算。gydF4y2BaegydF4y2Ba,原理图gydF4y2BaPVT1gydF4y2Ba启动子驱动的荧光素酶报告质粒gydF4y2Ba独联体gydF4y2Ba增强剂。的细节gydF4y2Ba独联体gydF4y2Ba-enhancer在Methods中。gydF4y2BafgydF4y2Ba,柱状图显示PVT1p、MYCp或本构TKp驱动的荧光素酶信号,无论有无顺式增强子(均值±SEM)。所有的值都归一化为相应的仅启动子结构,不含顺式增强子。gydF4y2BaPgydF4y2Ba使用双面Student 's计算gydF4y2BatgydF4y2Ba-test (n=3个生物重复)。gydF4y2BaggydF4y2Ba,点阵图显示jq1处理的荧光素酶信号(萤火虫标准化NanoLuc信号)在dmso处理的COLO320-DM和COLO320-HSR细胞中,在转染PVT1p或带有或不带有顺式增强子的MYCp质粒后的倍数变化。gydF4y2BaPgydF4y2Ba使用双面Student 's计算gydF4y2BatgydF4y2Ba-test (n=3个生物重复)。gydF4y2Ba
图7减少ecDNA融合的单克隆SNU16-dCas9-KRAB的生成。gydF4y2Ba
一个gydF4y2Ba,具有代表性的DNA FISH图像显示染色体外单阳性gydF4y2BaMYCgydF4y2Ba而且gydF4y2BaFGFR2gydF4y2Ba放大(左上和中上)和双阳性gydF4y2BaMYCgydF4y2Ba而且gydF4y2BaFGFR2gydF4y2Ba在亲本SNU16细胞(右上)的中期扩散放大(右上)。N = 42个细胞和8222个ecdna。具有代表性的DNA FISH图像显示明显的染色体外gydF4y2BaMYCgydF4y2Ba而且gydF4y2BaFGFR2gydF4y2Basn16 - dcas9 - krab细胞中期扩散的扩增(下)。N = 29个细胞和3893个ecdna。gydF4y2BabgydF4y2Ba,在AmpliconArchitect中显示支持每个断点的连接读取数量的排序图。断点的颜色是基于它们是否跨越来自同一扩增子的区域(gydF4y2BaMYCgydF4y2Ba/gydF4y2BaFGFR2gydF4y2Ba)或来自两个不同扩增子的区域。gydF4y2BacgydF4y2Ba亲本SNU16细胞系(左)和SNU16- dcas9 - krab细胞系(右)在10kb分辨率下经KR归一化的HiChIP接触矩阵。亲代细胞的接触基质含有增加的区域gydF4y2Ba顺式gydF4y2Bachr8和chr10之间的接触频率如所示,而相比之下,SNU16-dCas9-KRAB细胞的chr8和chr10之间的接触频率大大降低。中描述的chr8和chr10之间的低频结构重排重叠的区域灶相互作用增加gydF4y2BabgydF4y2Ba用方框表示。gydF4y2Ba
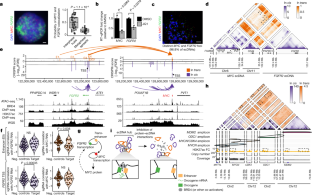
扩展数据图8 CRISPRi对ecDNA增强子的扰动揭示了功能的分子间增强子-基因相互作用。gydF4y2Ba
一个gydF4y2Ba, CRISPRi实验在SNU16-dCas9-KRAB细胞中扰动候选增强子。单导rna (sgRNAs)被设计用于靶向候选增强子gydF4y2BaFGFR2gydF4y2Ba而且gydF4y2BaMYCgydF4y2Ba基于染色质可及性的ecdna。gydF4y2BabgydF4y2Ba,聚合CRISPRi抑制假定增强子的实验工作流程。通过单细胞克隆获得稳定的SNU16-dCas9-KRAB细胞。用抗生素筛选的慢病毒sgRNAs库转导细胞,并用flowFISH评估癌基因RNA。通过基于癌基因表达的荧光激活细胞分选(FACS)将细胞分为6个箱。对每个箱子中的细胞进行sgrna定量。gydF4y2BacgydF4y2Ba, FACS门控策略。gydF4y2BadgydF4y2Ba,与针对任何一种增强子的CRISPRi库的未排序细胞相比,每个候选增强子元素的sgRNAs的变化为Log2倍gydF4y2BaMYCgydF4y2Ba或gydF4y2BaFGFR2gydF4y2Baecdna,其次是基于表达水平的细胞排序gydF4y2BaMYCgydF4y2Ba或gydF4y2BaFGFR2gydF4y2Ba.每个点代表针对一个候选元素的20个sgrna的平均log2倍变化。与相同池中阴性对照sgRNA分布相比,与癌基因表达负相关的元素用红色标记。gydF4y2BaegydF4y2Ba, Bar图显示了CRISPRi抑制候选增强子元素的重要性,如图所示。gydF4y2Ba4 egydF4y2Ba(上)。重要的在gydF4y2Ba反式gydF4y2Ba而在gydF4y2Ba独联体gydF4y2Ba增强子的颜色如所示。SNU16-dCas9-KRAB H3K27ac HiChIP 1D信号轨迹和相互作用配置文件gydF4y2BaFGFR2gydF4y2Ba而且gydF4y2BaMYCgydF4y2Ba启动子在10kb分辨率gydF4y2Ba独联体gydF4y2BaFitHiChIP循环如下所示。中的交互概要文件gydF4y2Ba独联体gydF4y2Ba用紫色和in表示gydF4y2Ba反式gydF4y2Ba用橙色表示。gydF4y2BafgydF4y2Ba,针对MYC TSS的单个sgrna的Spearman相关性gydF4y2BaMYCgydF4y2Ba而且gydF4y2BaFGFR2gydF4y2Ba表达式。使用低尾t检验比较目标sgrna与阴性对照sgrna (negcontrols)的P值显示。每个点代表一个独立的sgRNA。gydF4y2Ba
扩展数据图9分子间增强子和gydF4y2BaMYCgydF4y2Ba对于绝大多数ecdna来说,它们位于不同的分子上。gydF4y2Ba
一个gydF4y2Ba,上:中期扩散的双色DNA FISH,用于定量共定位的频率gydF4y2BaMYCgydF4y2Ba基因和分子间增强子如图所示。gydF4y2Ba4 egydF4y2Ba.以上随机共定位将表明融合事件。下图:代表性DNA FISH图像。DNA FISH探针靶向以下hg19基因组坐标:E1, chr10:122,635,712-122,782,544 (RP11-95I16;N = 11单元格);E2, chr10:122,973,293-123,129,601 (RP11-57H2;N = 12 cell);E3/E4/E5, chr10:123,300,005-123,474,433 (RP11-1024G22;N = 10个cell)。gydF4y2BabgydF4y2Ba,上:不同和共定位的FISH信号数。为了估计随机共定位,使用匹配数量的信号生成100个模拟图像,并将平均模拟频率与观测到的共定位进行比较。通过双侧t检验(Bonferroni-adjusted)确定P值。底部:明显高于随机概率的共定位信号数量。超过模拟随机分布的共定位是所有FISH图像中超过随机均值的共定位分子之和,其中总共定位高于随机均值加上95%置信区间(每张FISH图像100个模拟图像)。gydF4y2BacgydF4y2BaCas9体外消化gydF4y2BaMYCgydF4y2Ba在SNU16-dCas9-KRAB中含有- ecDNA,然后是PFGE(一个独立的实验)。碎片大小的确定基于gydF4y2Bah . wingeigydF4y2Ba而且gydF4y2Ba酿酒酵母gydF4y2Ba梯子。未裁剪的凝胶图像见补充图。gydF4y2Ba1gydF4y2Ba.gydF4y2BaMYCgydF4y2BaCDS指南对应《补充表》中的指南BgydF4y2Ba2gydF4y2Ba.gydF4y2BadgydF4y2Ba,分离增强子DNA序列的富集gydF4y2BaMYCgydF4y2Baecdna带gydF4y2BacgydF4y2Ba基于5kb窗口中标准化读取的背景(从未消化的基因组DNA在相应大小范围内的单独PFGE通道中分离的DNA)。每个点代表来自不同凝胶带的DNA。红色表示折叠变化大于4。gydF4y2BaegydF4y2Ba,测序轨迹为凝胶纯化gydF4y2BaMYCgydF4y2BaecDNA显示富集gydF4y2BaMYCgydF4y2Ba扩增子和耗竭gydF4y2BaFGFR2gydF4y2Ba含有增强子E1-E5的扩增子。gydF4y2Ba
图10 TR14成神经细胞瘤细胞系中四个不同扩增子的重建以及与H3K27ac标记相关的分子间扩增子相互作用模式。gydF4y2Ba
一个gydF4y2Ba,从上到下:四种不同扩增子的长读重建;基因组图谱,以长reads为基础的结构变异体>为10kb大小,>为20支持reads,红色边表示;短读全基因组测序的拷贝数变异和覆盖范围,所选基因的位置。gydF4y2BabgydF4y2Ba的代表性DNA FISH图像gydF4y2BaMYCNgydF4y2Ba间期TR14细胞中的ecDNA(上)和ecDNA聚类与相同细胞中DAPI对照的自相关评估gydF4y2BaggydF4y2Ba(gydF4y2BargydF4y2Ba)(底部)。数据为平均值±SEM (n = 14个单元)。gydF4y2BacgydF4y2Ba,自定义重构TR14扩增子Hi-C图。的gydF4y2BaMYCNgydF4y2Ba/gydF4y2Ba到gydF4y2Ba扩增子和gydF4y2BaMYCNgydF4y2BaecDNA共享序列,这阻止了在这些区域进行明确的短读映射,并显示为白色区域。gydF4y2Ba反式gydF4y2Ba之间的相互作用出现局部升高gydF4y2BaMYCNgydF4y2BaecDNA和gydF4y2BaODC1gydF4y2Ba扩增子(由箭头指示)。gydF4y2Ba独联体gydF4y2Ba- - -gydF4y2Ba反式gydF4y2Ba-接触频率为所示颜色。gydF4y2BadgydF4y2Ba,对连接扩增子的长读测序识别的结构变异的读取支持。不同扩增子之间只有一个结构变异(gydF4y2BaMYCNgydF4y2Ba而且gydF4y2BaMDM2gydF4y2BaAmplicons)被鉴定为3个支持reads。gydF4y2BaegydF4y2Ba,连接扩增子结构变异的变异等位基因频率。gydF4y2BafgydF4y2Ba,gydF4y2Ba反式gydF4y2Ba增强子之间的交互模式gydF4y2BaMYCNgydF4y2Ba扩增子片段(垂直)和一个gydF4y2BaODC1gydF4y2Ba扩增子片段(水平)。短读WGS覆盖(灰色),H3K27ac ChIP-seq轨迹显示输入的平均折叠变化(黄色),Hi-C接触图显示(kr归一化计数在5kb箱)。gydF4y2BaggydF4y2Ba,从上到下:三个扩增子重建,富增强子的虚拟4C相互作用谱gydF4y2BaHPCAL1gydF4y2Ba轨迹在gydF4y2BaODC1gydF4y2Ba带有其他扩增子位点的扩增子(红色),以及H3K27ac ChIP-seq(输入的折叠变化;黄色)。gydF4y2BahgydF4y2Ba,gydF4y2Ba反式gydF4y2Ba不同扩增子之间的相互作用(kr归一化计数为5kb bin)取决于相互作用位点的H3K27ac信号(左;盒子中心线,中间;方框限制,上下四分位数;盒须,1.5倍四分位范围)。gydF4y2Ba反式gydF4y2Ba相互作用(kr归一化计数在5kb箱)由扩增子对(右)分开。H3K27ac高与低表示在5kb的箱子中至少比输入的平均富集3倍。N = 114,636对H3K27ac Low + Low, N = 11,990对H3K27ac High + Low, N = 296对H3K27ac High + High。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
此文件包含补充表1和2以及补充表1 - 3的图例。gydF4y2Ba
补充图1gydF4y2Ba
琼脂糖凝胶的原始图像。与扩展数据图4f, 9c相关。gydF4y2Ba
补充表3gydF4y2Ba
参见补充表3图例中的补充信息。gydF4y2Ba
补充视频1gydF4y2Ba
用未处理的TetO-eGFP col320 - dm细胞进行活细胞成像。未处理细胞的快照显示在30分钟的过程中。GFP标记TetO-knock-ingydF4y2BaMYCgydF4y2BaecDNAs。gydF4y2Ba
补充视频2gydF4y2Ba
dmso处理的TetO-eGFP col320 - dm细胞的活细胞成像。用DMSO处理的对照细胞在1小时内被跟踪。GFP标记TetO-knock-ingydF4y2BaMYCgydF4y2BaecDNAs。gydF4y2Ba
补充视频3gydF4y2Ba
JQ1处理后TetO-GFP col320 - dm细胞活细胞成像。用500 nM JQ1处理的细胞在1小时内被跟踪。GFP标记TetO-knock-ingydF4y2BaMYCgydF4y2BaecDNAs。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
洪国良,约斯特,谢国良,谢国良。gydF4y2Baet al。gydF4y2BaecDNA中心驱动协同分子间癌基因表达。gydF4y2Ba自然gydF4y2Ba600gydF4y2Ba, 731-736(2021)。https://doi.org/10.1038/s41586-021-04116-8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-021-04116-8gydF4y2Ba
这篇文章被引用gydF4y2Ba
染色体外环状DNA (eccDNA):癌症领域的新星gydF4y2Ba
生物标志物的研究gydF4y2Ba(2022)gydF4y2Ba
癌症中的三维染色质结构和转录调控gydF4y2Ba
血液学与肿瘤学杂志gydF4y2Ba(2022)gydF4y2Ba
染色体外环状DNA:生物发生、结构、功能与疾病gydF4y2Ba
信号转导与靶向治疗gydF4y2Ba(2022)gydF4y2Ba
染色体外DNA的基因组和空间流动性及其对癌症治疗的意义gydF4y2Ba
自然遗传学gydF4y2Ba(2022)gydF4y2Ba
转录活动的中心gydF4y2Ba
自然评论癌症gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
