摘要
10-23 DNAzyme是最显著的催化活性DNA序列之一1,2.它具有高选择性地切割广泛的RNA靶标的能力,具有巨大的治疗和生物技术潜力2.然而,很高的期望尚未得到满足,这一事实与缺乏关于其行动方式的高分辨率和时间分辨的资料是一致的3..在这里,我们提供了10-23 DNAzyme原型的所有表观状态的高分辨率核磁共振表征,并对其催化功能的动力学和动力学进行了全面的调查。预催化DNAzyme-RNA复合物的确定结构和确定的金属离子结合位点揭示了dna介导的催化的基础是三个因素之间的相互作用:一个意想不到的,但令人兴奋的分子结构;明显的构象可塑性;以及金属离子的动态调制。通过实时核磁共振测量,我们进一步识别了dna介导的催化过程中先前隐藏的限制速率的瞬态中间态。使用合理选择的单原子替代,我们可以大大提高DNAzyme的性能,证明获得的分子结构知识,其可塑性和长寿命中间态的出现构成了合理设计下一代DNAzyme的宝贵起点。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
在膜系统动力学和生物催化下的原子
BIOspektrum开放获取2月13日
访问选项
订阅《自然》+
立即在线访问《自然》和其他55种《自然》杂志
29.99美元
每月
订阅期刊
获得1年的完整期刊访问权限
199.00美元
每期仅需3.90美元
所有价格均为净价格。
增值税稍后将在结帐时添加。
税务计算将在结账时完成。
买条
在ReadCube上获得时间限制或全文访问。
32.00美元
所有价格均为净价格。

代码的可用性
结构计算、MD模拟和实时核磁共振数据模拟的代码可根据合理要求从相应作者处获得。
参考文献
桑托罗,S. W.和乔伊斯,g.f.一种通用的rna切割DNA酶。国家科学院学报美国94, 4262-4266(1997)。
催化DNA:脱氧核酶的范围、应用和生物化学。学生物化学的发展趋势。Sci41, 595-609(2016)。
罗森巴赫,H.等人。影响10-23 DNAzyme活性的分子特征和金属离子。分子25, 3100(2020)。
蔡国强,李志强,李志强。一种具有DNA连接酶活性的DNA金属酶。自然375, 611-614(1995)。
普拉迪库马尔,p.i., Höbartner, C.,鲍姆,d.a.和西尔弗曼,s.k. dna催化形成的核苷酸连接。Angew。化学Int。艾德。47, 1753-1757(2008)。
沈,Z.等。一种由特定的细菌病原体菌株激活的催化DNA。Angew。化学Int。艾德。55, 2431-2434(2016)。
Torabi S.-F。et al。钠特异性DNAzyme的体外筛选及其在细胞内传感中的应用。国家科学院学报美国112, 5903-5908(2015)。
Ponce-Salvatierra, A., Wawrzyniak-Turek, K., Steuerwald, U., Höbartner, C. & Pena, V. DNA催化剂的晶体结构。自然529, 231-234(2016)。
刘,H.等。rna裂解DNAzyme的晶体结构。Commun Nat。8, 2006(2017)。
Victor, J., Steger, G. & Riesner, D. DNAzymes不能在体内切割RNA是由于有限的Mg2 +细胞的浓度。欧元。Biophys。J。47, 333-343(2018)。
Young, D. D. D., Lively, M. O. & de, A. DNAzyme的激活和失活以及反义功能在哺乳动物细胞中基因表达的光化学调节。j。化学。Soc。132, 6183-6193(2010)。
王,杨,阮,K, Spitale, R. C. & Chaput, J. C.一种生物稳定的DNAzyme,能有效地沉默细胞中的基因表达。Nat,化学。13, 319-326(2021)。
刘,C.等。自组装铜- dnazyme纳米杂化物用于双催化肿瘤治疗。Angew。化学Int。艾德。60, 14324-14328(2021)。
刘恩恩,王玉玉,林昌华,裴林2 +-独立的DNAzyme,通过多重催化翻转有效地切割RNA。化学。科学。9, 1813-1821(2018)。
Nowakowski, J., Shim, P. J., Prasad, G. S., Stout, C. D. & Joyce, G. F.由10-23 DNA酶形成的82-核苷酸RNA-DNA复合体的晶体结构。Nat。结构。医学杂志。6, 151-156(1999)。
Zaborowska, Z., Fürste, J. P., Erdmann, V. A. & Kurreck, J.“10-23”DNA酶催化核心的序列要求。生物。化学。277, 40617-40622(2002)。
Vögeli, B., Olsson, S., Güntert, P. & Riek, R.精确的NOE作为集成结构确定的替代方法。Biophys。J。110, 113-126(2016)。
Elstner, M., Hobza, P., Frauenheim, T., Suhai, S. & Kaxiras, E.核酸碱基对的氢键和堆叠相互作用:基于密度功能理论的治疗。j .化学。理论物理。114, 5149-5155(2001)。
Kisseleva, N., Khvorova, A., Westhof, E. & Schiemann, O.通过电子顺磁共振波谱研究锰(II)与三级稳定锤头核酶的结合。核糖核酸11, 1-6(2005)。
罗森巴赫,H.等人。单价金属离子对10-23 DNAzyme金属结合及催化活性的影响。医学杂志。化学。402, 99-111(2020)。
巴尔巴赫,J.等人。在二维核磁共振实验中,在单个残基上监测蛋白质折叠。科学274, 1161-1163(1996)。
Etzkorn, M., Böckmann, A. & Baldus, M.通过实时2D固态NMR光谱监测蛋白质聚集的动力学分析。j . Biomol。核磁共振49, 121-129(2011)。
纳罗特,B.等人。脱氧核酶10-23催化核心中功能磷酸基团的图谱。2月J。274, 1062-1072(2007)。
霍顿,T. E.,克拉迪,D. R. &德罗斯,V. J.电子顺磁共振波谱测量锰2 +与锤头核酶的结合亲和性及与裂解活性的相关性。生物化学37, 18094-18101(1998)。
Hunsicker-Wang, L., Vogt, M. & Derose, V. J.研究RNA中特定金属离子结合位点的EPR方法。Enzymol方法。468, 335-367(2009)。
Rinnenthal, J., Klinkert, B., Narberhaus, F. & Schwalbe, H.调节的稳定性沙门氏菌4u型RNA温度计。核酸测定。39, 8258-8270(2011)。
Delaglio, F.等人。NMRPipe:基于UNIX管道的多维光谱处理系统。j . Biomol。核磁共振6, 277-293(1995)。
凯勒,r.l.j。计算机辅助共振作业教程(酒吧,2004)。
Lee, W., Tonelli, M. & Markley, J. L. NMRFAM-SPARKY:生物分子核磁共振光谱的增强软件。生物信息学31, 1325-1327(2015)。
舒伯特,S. RNA裂解' 10-23 ' DNAzymes具有增强的稳定性和活性。核酸测定。31, 5982-5992(2003)。
Thakur, C. S., Luo, Y., Chen, B., Eldho, N. V.和Dayie, T. K.利用野生型和转酮醇酶大肠杆菌突变体标记RNA进行高分辨率NMR的生物质生产。j . Biomol。核磁共振52, 103-114(2012)。
Orts, J., Vögeli, B. & Riek, R.自旋扩散弛豫矩阵分析用于eNOEs核磁共振结构计算。j .化学。理论第一版。8, 3483-3492(2012)。
Schwieters, C. D., Kuszewski, J. J., Tjandra, N. & Marius Clore, G. Xplor-NIH NMR分子结构测定包。j .增效。的原因。160, 65-73(2003)。
Valafar, H. & Prestegard, J. H. REDCAT:残差偶极耦合分析工具。j .增效。的原因。167, 228-241(2004)。
黄,K.等。MEF2A-DNA复合物的溶液结构:MADS-box转录因子调节DNA弯曲和特异性的结构基础。EMBO J。19, 2615-2628(2000)。
Pimenta, J.等人。NMR溶液结构和SRP54M预测了绵羊Doppel蛋白n端序列(1-30)的相互作用。肽49, 32-40(2013)。
Case, d.a.等。2020年琥珀(加州大学,2020)。
王杰,Cieplak, P. & Kollman, P. a .约束静电势(RESP)模型在计算有机和生物分子构象能中的表现如何?j .第一版。化学。21, 1049-1074(2000)。
巴斯,D. C.罗杰斯,D. M.和詹森,J. H.非常快速的预测和合理化的pKa值的蛋白质配体复合物。蛋白质73, 765-783(2008)。
乔根森,钱德拉塞卡,J.,马杜拉,J. D., Impey, R. W. & Klein, M. L.模拟液态水的简单势函数的比较。j .化学。理论物理。79, 926-935(1983)。
jung, i.s. & Cheatham, t.e.碱和卤化物单价离子参数的测定用于明确溶剂化的生物分子模拟。期刊。化学。B112, 9020-9041(2008)。
Schott-Verdugo, S. & Gohlke, H. packmole - memgen:一种简单易用的膜-蛋白质-脂-双层体系构建的通用工作流。j .化学。正无穷。模型。59, 2522-2528(2019)。
李,罗伯茨,B. P.,查克拉沃蒂,D. K. & Merz, K. M.显式溶剂中+2金属阳离子粒子网格Ewald兼容Lennard-Jones参数的合理设计。j .化学。理论第一版。9, 2733-2748(2013)。
达顿,T.,约克,D. & Pedersen, L.粒子网格Ewald:一个N⋅大系统中Ewald和的log(N)方法。j .化学。理论物理。98, 10089-10092(1993)。
Ryckaert, j。,Ciccotti, G. & Berendsen, H. J. C. Numerical integration of the cartesian equations of motion of a system with constraints: molecular dynamics of n-alkanes.j .第一版。理论物理。23, 327-341(1977)。
汉克,c.a. &高尔克,H. In理解核糖开关的计算方法第553卷(编)陈,S.-J。伯克-阿圭罗,D. H. B.) 163-191(学术出版社,2015)。
Roe, d.r. & Cheatham, t.e. PTRAJ和CPPTRAJ:用于处理和分析分子动力学轨迹数据的软件。j .化学。理论第一版。9, 3084-3095(2013)。
里克特,C.等。用溶液核磁共振波谱分析偶极子-偶极子相互关联弛豫法测定大分子量RNA寡核苷酸中的糖构象。j . Biomol。核磁共振15, 241-250 (1999)
双链核酸的热变性:梯度凝胶电泳和聚合酶链反应临界温度的预测。核酸测定。22, 2760-2768(1994)。
杉本,N.等。预测RNA/DNA杂化双工稳定性的热力学参数。生物化学34, 11211-11216(1995)。
具有长程相关性的特定序列大分子的概率分布的递归关系生成。生物聚合物13, 1859-1871(1974)。
确认
我们承认访问Jülich-Düsseldorf生物分子核磁共振中心。我们感谢H. Arthanari, A. Boeszoermenyi和T. Viennet对我们的宝贵支持19F-NMR实验;R. Biehl进行有益的讨论;R. Kümmerle用于1.1 GHz的数据采集。H.G.非常感谢海因里希·海涅大学(Heinrich Heine University)的“ZIM (für information -und Medientechnologie)”(ZIM)和约翰·冯·诺伊曼计算研究所(NIC)(用户ID: HKF7, VSK33)提供的计算支持和基础设施。这项工作得到了德国研究基金会(DFG) (ET 103/2-1, ET 103/2-2, ET 103/4-1, ET 103/5-1)对M.E的支持,化学工业基金(Li 196/05给I.S., Hoe 700080给H.R.),德国学术奖学金基金会(给H.R.),拜耳公司(Grants4Ag项目给I.S.),以及欧盟的地平线2020研究和创新计划,根据Marie Sklodowska-Curie资助协议号660258 A.V.结构研究中心由DFG资助(资助号417919780)。
作者信息
作者及隶属关系
贡献
j.b., j.v., h.r., a.v., c.g.w.g., c.w., m.g., H.K.和M.E.进行了实验和/或模拟,并与g.s., o.s., H.G.和i.s.一起分析了数据。核磁共振数据采集和处理由j.b.、a.v.、M.G.和M.E.进行。活性测定由j.v.、h.r.、H.K.和M.E.进行。MD模拟由C.W.进行。EPR谱记录由C.W.进行。j.b.、j.v.、h.r.、d.r.、H.G.和M.E.撰写。所有作者都设计了实验并对手稿进行了评论。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
额外的信息
同行评审信息自然感谢Harald Schwalbe, Scott Silverman和其他匿名审稿人对这项工作的同行评审所做的贡献。同行评审报告是可用的。
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
扩展的数据图形和表格
扩展数据图1使用结构的属性和应用修改的验证。
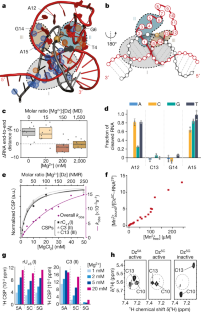
a)使用结构表。b)所示Dz变体底物裂解的凝胶电泳分析(上,使用荧光素标记的RNA)和2ʹF稳定的影响(下,使用GelRed染色)。c)[中2ʹF RNA修饰(左)及受影响质子示意图]1H,1H]-TOCSY指纹谱(Dz5度核糖核酸2ʹF,蓝色和Dz5度rna,黑色)。2ʹF在rG的修改0仅在rU的H5和H6位置直接诱导CSP-1.而没有13RNA底物中C的富集阻碍了准确测定2ʹF修饰对裂解位点周围假旋转相的潜在影响48,分析2ʹF修饰诱导的CSP模式完全解决1H在配合物中的位置(d)证实了底物的稳定不会改变预催化配合物的整体结构。e)存在和不存在1 mM Mg时核苷酸特异性峰值强度比2 +对Dz5核糖核酸2ʹF(红色)和Dz5度核糖核酸2ʹF(蓝色)。为了在不同核苷酸之间进行可靠的比较,我们给出了每个Dz核苷酸H1ʹ与H6/H8之间相关性的交叉峰强度的变化。在环区消失的峰值与核磁共振中间交换体系中发生的交换过程有关,而其他观察到的CSPs揭示了核磁共振快速交换体系中的交换过程。可以得出Mg2 +在5A变异的催化环内停留时间更长,可能促进裂解。f)验证用于PRE自旋标记的点击化学方法的标记效率和裂解能力。使用tempo -叠氮化物(2)和5-乙基-2 ' -脱氧尿苷(EdU) (1) (f,上)的铜催化click反应的示意图。EdU被用来取代一个选定的胸腺嘧啶核苷酸在Dz序列。为了测试标记效率和裂解能力,使用了fam -叠氮化物,通过SDS-PAGE直接检测(f,底部)。为了评估标记效率,在指示车道上装载了等量的商用fam标记的Dz。g) Dz活性的验证6 xf.Dz随时间变化的底物裂解变性SDS-PAGE结果5度不带和带六2ʹ-19F替换(Dz6 xf).结果表明,氟原子不影响Dz的解理活性,也不影响Mg的解理活性2 +的依赖。
图2 Dz的核磁共振赋值5度核糖核酸2ʹF.
a) a [1H,1H]-NOESY谱显示Dz序列dG的顺序相关性+ 2对dT+ 9如b). c) [1H,13C]-HSQC谱显示1个ʹ-CH基团的分配(见方法有关详情)。
图3预催化状态的分子特性。
a)氨基峰的存在作为稳定氢键形成的指标(例如,沃森-克里克碱基配对)。2D NOESY光谱提取(右)包含胞嘧啶H5和氨基之间的交叉相关性(图中高亮显示(左))。数据显示,结合臂中所有碱基配对的胞嘧啶都有明确的信号,而Dz环中的5个胞嘧啶中没有检测到任何信号,这表明各自的氨基缺乏稳定的氢键。(b,c,d)任一单链Dz中所示核苷酸的温度依赖性TOCSY NMR峰值强度5度(b)或预催化区5度核糖核酸2ʹF复杂的(c, d)。偏离连续强度增加表明局部稳定结构发生变性(详见补充讨论)。T4表现出与其邻居不同的行为(d)。e)[的比较1H,1H]-TOCSY DNA指纹谱c·RNA(青色)和DNA5度·RNA2ʹF(黑色)。大多数受影响核苷酸的配位被指出,并表明在没有催化环的情况下,裂解位点周围的各自峰转变为规则双工结构的区域特征,就像在其余的结合臂中发现的那样。f)催化环的存在引起的化学位移扰动(CSPs)的分布。Dz之间束缚臂区所有指定质子的CSPs5度-RNF2ʹF和DNAc-RNA显示为等高线图(左)或映射在预催化复合物的确定结构上(右)。后者表明,具有强CSPs的区域与预催化络合物中与经典a型螺旋不同的区域非常一致。g)总结基于nmr的预催化配合物结构测定的示意图。指示距离和同源限制相结合,在一个从头开始结构计算,然后使用rdc进行聚类分析和构象滤波,得到与所有实验数据最相似的结构集合(簇I)(参见方法参阅详细资料及扩展数据图。4代表性实验数据)。
图4基于nmr的Dz结构确定5度核糖核酸2ʹF.
a,b) PRE实验的实验数据和结果分析。) (1H,1H]-NOESY光谱提取的样品,在T8位置有一个TEMPO标签,通过添加抗坏血酸(黑色)降低氮氧化物自旋标签之前(红色)和之后(黑色)。b)三个样品中每个分解质子的两个光谱之间的强度比。标记尺寸(in I/I0方向)表示从分析各自质子的所有解析交叉峰的值得到的误差幅度(SD)。只有具有至少两个不同的交叉峰的质子才被考虑。c)用于eNOE方法的noe -累积曲线子集。归一化NOE强度记录在混合时间在40到800毫秒之间(填充圆)和由eNORA确定的累积曲线(线)32.[1H,1H]-NOESY交叉峰出现在对角线以上(红色)或对角线以下(绿色)被认为是大多数NOE接触。值得注意的是,即使是短的和几何上固定的距离,它们的累积行为也有相当大的差异,这表明有必要考虑交叉松弛效应。计算的交叉弛豫率(σ)和提取的eNOE距离范围(在20%的误差范围内)(蓝色)。d - i)19f基核磁共振实验。d) 1 d19Dz的F-NMR谱5度核糖核酸2ʹF(上)和Dz5度变体包含6个2ʹ19F在G位置的变化−6G−5、G2、C7、A11、G14 (Dz6 xf(下)与RNA复合体2ʹF.虽然[19F,19F] Dz的-NOESY谱6 xf核糖核酸2ʹF未显示任何可检测到的峰值(数据未显示),1H-detected (e) or19f检测(f) [1H,19F]-HOESY光谱显示了有限数量的明显交叉相关,用于顺序共振分配以及远程距离限制。g)各自氟化核糖部分的H1ʹ和H2ʹ质子可以在一个[1H,1H]-NOESY谱经强峰分裂诱导J跳频耦合。h+i)为了克服二维HOESY相关性的灵敏度限制,19F-saturated和1使用h -检测的STD NMR。h)产生的19Dz的F-STD谱5度核糖核酸2ʹF.我)19Dz的F-STD谱6 xf核糖核酸2ʹF使用指示的19F饱和频率(颜色代码参考面板d中的分配)。j-m)剩余偶极耦合(rdc)的表征。j) rdc诱导的预催化配合物频移示例。图中为a[]中回路位置C13的H5-C5交叉峰。1H,13C]-HSQC谱记录在20°C时Pf1噬菌体不存在(黑色)和存在(红色)13C同位素标记的Dz5度与未标记RNA的复合物2ʹF.k)交叉峰的一维横截面,如j)所示,表示由于线宽、峰重叠和信噪比效应造成的实验限制。l,m)簇III (l)的非匹配结构的观测和反计算RDC常数的相关图,以及簇I (m)结构的改进相关性。尽管实验限制在确定值中引入了更大的误差范围(如RMSD值所示),RDC效应仍然明显(如相关性显著增加所示)。
图5 nmr衍生的构造集成特征。
a)后的聚类分析结果从头开始结构计算不使用rdc。20个能量最低的结构(在计算的200个结构中)根据它们在Dz环的相对方向进行排序:围绕RNA(簇I)完全缠绕,定位于解理位点的下游(簇II)或上游(簇III),或其他(簇IV)。结构下面的数字表示各自折叠中的结构数量。b)程序与a)相同,但包括RDC数据。虽然集群I在没有RDC限制的情况下已经是人口最多的,但在包括RDC之后,它将成为目前为止的主要部分。一般来说,rdc可被视为最可靠的基于nmr的相对分子取向探针之一。因此,尽管光谱质量和精度的剩余限制(扩展数据图。4 j, k),对聚类选择的影响显著。c)在更严格的结构优化和能量最小化后,覆盖每个集群(集群I-III)的四个最低能量结构(以100个为单位)。
图6预催化配合物的构象塑性。
a) Dz的均方根波动(RMSF)5度-RNA在MD模拟。所示为每个核苷酸在所有副本上的平均RMSF±SEM(黑色区域)。橙色区域突出了催化环的核苷酸,蓝色区域代表裂解位点周围的核苷酸。b) MD模拟的平均结构(卡通)与MD轨迹生成的结构集合叠加(半透明带,每10 ns取一次结构)。c)所有7个最低能量Dz的叠加5度核糖核酸2ʹF在从头算结构计算中采用松散约束得到的属于簇I的结构。与扩展数据图中所示的集合相反。5,此时,将环路区域排除在结构对准之外。可以看出,包括结合臂和解理位点方向在内的核心区域定义合理,全原子RMSD为2.6 Å。相反,催化环区域在不同结构中的位置显示出明显的变化,提供了允许构象空间的估计。值得注意的是,与定义较好的区域直接比较,环路区域在检测到的时间范围内并没有显示出大量增加的动态(图。1 f),它仍然显示了核苷酸与其邻居的堆叠相互作用,表明局部定义的构象(扩展数据图。9 d及补充数字1).根据md衍生图,因此可以假设Dz在环区相比于结合臂,特别是解理位点区域表现出更高的构象塑性。
图7金属离子与预催化配合物的结合。
一)毫克2 +结合引起Dz指纹谱的化学位移扰动(CSPs)5度核糖核酸2ʹF表明在快速兑换制度中发生的约束性和结构性的重新安排。b)获得的核苷酸特异性图KD值(黑色,拟合值±标准误差)以及Mg的大小2 +-诱导的最大CSP(红色)。注意,CSPs报告直接相互作用和/或继发构象变化。因此,得到的KD值只能作为Mg的直接或间接效应的报告2 +绑定。c)毫克2 +依赖于卵裂活性,如FRET所示。d)原子特异性Mg图2 +-诱导的CSPs沿着Dz-RNA序列。e) Mn2 +-和Mg2 +Dz的诱导活性5度.f)锰2 +在过量镁的存在下结合2 +不诱导CSPs,但明确的PRE效应,允许区分结合和结构变化。g)由于与Mn的距离不同,所选核苷酸的PRE速率不同2 +绑定的网站。h)原子特异性Mn图2 +-诱导的PRE速率沿Dz-RNA序列。i) Mg的核苷酸结合的可能性2 +在多个微秒长的MD模拟中,预催化复合体。j)直接NOE交叉峰[Co(NH3.)6]3 +和Dz5度核糖核酸2ʹF.虚线表示猎取质子的频率。根据观察到的低亲和力和快速交换速率,这些NOEs指向二价金属离子的水合壳与核苷酸的相互作用。观察到的相互作用明显涉及金属离子结合区I的核苷酸,也可能涉及金属离子结合区III的核苷酸,证实了预催化Dz-RNA复合体的中心金属离子结合位点采用了能够容纳二价金属离子水化壳的构象。k) Dz的PAGE分析5度活性:在不同浓度的金属离子存在时的活性有趣的是,Co2 +离子能充分激活Dz,促进底物裂解,[Co(NH3.)6]3 +不介导Dz裂解反应,但抑制Mg2 +介导催化。这些抑制作用与观察到的相同结合边的竞争相一致。然而,数据也表明,水合壳模拟不保持催化活性状态。l)与i)相同,但不是聚类1(如i中所使用),而是使用聚类2-4的结构作为MD模拟的起点。可以看出,使用聚类1 (i)的MD数据与实验数据(h)的一致性明显好于其他聚类(l),验证了基于RDC数据的聚类选择。
扩展数据图8预催化配合物结构与功能之间的实验联系。
a)脚手架的结构特征包括两个结合臂之间的紧密接触,+1位置的非碱基配对,i区域的金属离子结合。b) NOE图中明显没有各自的峰,证实了dG的翻转+1.不像所有核苷酸预期的寄存器内堆叠,dG没有交叉相关+1对dG+ 2检测到的(应该沿蓝色虚线出现)。dA的堆叠方式+ 6- - - - - - dT+ 9显示为阳性对照(绿色箭头)。c) +1和−1位置突变对Dz的影响5度与位置−1不同,确认位置+1的活动不会形成基本的沃森-克里克碱基对。d)核磁共振数据的例子(NOESY光谱的提取)证实dG在空间上接近−5和dG+ 2在毫克2 +绑定。e)从Mn中获得的原子特异性PRE速率2 +DNA滴定法c-RNA(上)与Dz中各核苷酸的比较5度核糖核酸2ʹF(底部)。数据表明,催化环的存在使M2 +-臂的结合行为,从一个相当分散的模式(上)到定义的结合区域,形成脚手架台阶的基础(下)。f)预催化结构俯视图,重点关注裂解位点周围环境。g) noe生成速率强烈表明syn- dA的构象−1.上图所示的示意图模型可视化了所考虑的质子间相互作用与固定距离(H1ʹ-H2ʹ,紫色,和H1ʹ-H2″,红色)或距离强烈依赖于χ-角(H1ʹ到指示的基底质子,蓝色)。h) NOE模式指示Mg2 +-诱导T4翻转。值得注意的是,T4的所有10个核苷酸间相关性都观察到相同的行为。i)催化环5ʹ端(金属离子结合位点II)突变对解理活性的影响。5位突变作为参考;所有其他突变都是Dz的变体5度.数据以三次重复实验的平均值+/- SD表示。j)变异体Dz解理活性比较5(黑色)、Dz5度(蓝色)和Dz5克(红色)存在3mm Mg2 +(数据为Dz5和Dz5度与图中各条件相同。1 b).k)简化的示意图模型,突出三维结构的中心特征。l-n) Mn的cw EPR表征2 +绑定。l) Mn的双积分EPR信号2 +在不存在(蓝色)和存在40 μM Dz时5度·RNA2ʹF配合物(黑色)。全范围和缩小到较低的浓度显示。m,n)实验确定的结合行为(黑色数据点)对任何一个满Mn的拟合2 +浓度范围(左图)或只有高亲和力结合位点(右图)。数据拟合到不同的绑定模型(红色曲线,见方法和补充讨论,以了解更多应用数据拟合的细节)。n)假设有三个协同结合位点,分别有高亲和性和低亲和性结合位点,总体上与得到的数据最拟合(左)。在右侧面板中,假设只有一个合作绑定位点的模型,并且只考虑显示的数据范围。所显示的拟合以最小数量的参数捕获绑定行为的关键特征。
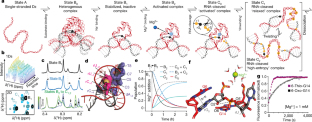
图9催化循环中中心Dz态的性质。
a) Dz指示态的核磁共振指纹区5度和催化循环中的底物。如图所示的椭圆。1 d作为每个频谱的参考。光谱允许跟随复杂的形成,钠的影响+和毫克2 +, RNA裂解和RNA解离。光谱下面的示意图模型说明了各自的(简化的)条件和结构特征。b)加入RNA前(红色)和加入RNA后(黑色)的Dz指纹区对比2ʹF.c-d)在自由(红色)和络合Dz(黑色)的NOESY光谱中发生的顺序接触分析。c)分析的顺序接触示意图。d)代表核苷酸碱基质子(H6/H8)(顺序)交叉相关性的谱区提取。数据表明,催化环的中心区域已经采用了一种(最低结构)构象,类似于与底物复杂形成后在同一区域发现的特征(更多细节见补充讨论)。e-k)单价(Na+)金属离子。在缺乏(单价和二价)金属离子的情况下,在NMR指纹图谱中发现了额外的复杂构象(顶部e中的青色箭头)。加入100 mM Na后,这些构象消失+(中)或1mm Mg2 +(e,下),不代表催化相关的状态(f,从扩展数据图的数据外推可见。7一个,灰色箭头)。人们很容易推测,这种非通路状态可能代表Dz-RNA复合物的非活性构象,其特征是杂交结合臂缺乏围绕底物的额外循环。然而,其他构象也是可能的。除了不同的Dz - RNA复合物构象外,在不含金属离子的情况下,光谱还显示了自由RNA和Dz的存在。g)来自自由RNA(灰色)、自由Dz(紫色)和附加复杂构象(青色)的信号的峰值体积分析。数据是在没有NaCl和存在指示浓度的MgCl的情况下获得的2.发现初始(低)Mg对络合物的形成(总体和构象)有相当强的影响2 +浓度。在100 mM NaCl和不含MgCl的情况下,光谱可见2(e,中),两种特征均可被NaCl饱和。换句话说,在与生理离子强度条件更相关的条件下(例如在100 mM NaCl下),络合物的形成似乎不是一个限制步骤。然而,这些数据有力地表明,在低离子强度条件下,添加少量金属离子可以诱导对复合物形成和构象的强烈影响(后者可能与催化无关)。这一观察可能有助于澄清以前的发现,涉及从低离子强度条件的转变。h) MgCl结果2Dz指纹区核磁共振滴定法5度核糖核酸2ʹF在没有(上)或存在(下)100 mM NaCl时(后者与扩展数据图中的数据相同)。7一个).i)放大两个指定核苷酸的行为。在不添加和存在100 mM NaCl时,滴定终点非常相似,但在不添加NaCl时,最大CSPs (j)增加,趋势延长KDMg值2 +在NaCl存在的情况下观察到结合。k)在不存在和存在100 mM NaCl时,各滴定步骤的总CSPs(所有分解CSPs的总和)的相关性。对于MgCl2浓度大于1mm时,NaCl的存在对Mg的影响不大2 +全身的csp。相比之下,1mm以下的MgCl2时,相关图的最小值偏离对角线(白色箭头)。数据表明,100 mM NaCl对Dz的影响相当5度核糖核酸2ʹF复杂结构近似。0.7 mM MgCl2.l)二维比较[1H -1H] TOCSY光谱提取记录之前(黑色)和之后完全裂解(橙色)。m)具有(橙色)或不具有(绿色)的核苷酸在裂解后检测到多个构象,并在预催化配合物的结构上突出显示(虚线表示具有剩余不确定性的核苷酸)。n)随着温度的升高,检测到的信号强度和所示核苷酸的催化后复合体中发生的不同状态的相对数量。o)最佳分辨核苷酸的两种状态的核磁共振峰强度之比。解理后的数值(橙色,谱图数据为l)以及解理反应过程中的实时NMR数据(蓝色,谱图数据为扩展数据图数据)。10 d).数据表明,随着时间的推移,各自状态的相对总体是稳定的,包括它们在裂解反应中的初始外观。所观察到的行为可以用缓慢交换状态的稳定平衡来最好地解释(详情见补充讨论)。p)在NMR条件下显示RNA底物裂解的原生PAGE(这里显示了实时NMR实验后的样品)。q)在37℃和变性温度下的理论变性度ΘT米与分裂RNA的Dz复合物(用程序POLAND计算49使用RNA:DNA杂交参数50,循环参数为σ= 1e−351,解离常数β= 1e−3,和给定的复合物浓度。
扩展数据图10催化反应的时间分辨原子级洞察。
a) 1D系列1加入Mg后记录的H谱2 +到预成型的非稳定Dz5度-RNA复合物显示明显的峰值强度随时间变化。b) Dz上记录的1D光谱提取5度-RNA之前(黑色),直接之后(蓝色),并在指定的时间点(直到约2小时)Mg2 +加法。在稳定的Dz上记录的频谱5度核糖核酸2ʹF,与Mg预孵育2 +作为Mg的参考2 +-平衡预催化配合物(红色)。c)解理反应后时间分辨1D NMR数据等高线图。如图(A)所示的数据的放大显示了两个峰,分别代表排出(右)和产物(左)状态。在反应的初始阶段,排泄态的最大峰值明显移位,而生成物态则不存在(虚线作为参考)。观测到的频率变化方向遵循Mg诱导的CSP2 +结合(见(b)),因此与有效Mg的增加相一致2 +: Dz比率。这些数据有力地表明,催化后配合物对Mg具有较低的亲和力2 +离子比预催化配合物产生有效Mg2 +在产品发布之前发布。d,e)二维级数[1H -1H]加入1mm MgCl前(黑色)和加入1mm MgCl后依次记录的TOCSY光谱2.各光谱采集时间均为3 h。在第一光谱(d,青色)和第二光谱(e,棕色)之间存在清晰的峰位置、峰形状和/或强度变化。在第二光谱和第三光谱之间只有非常微弱的强度变化(e,橙色)。所示放大倍数比较Mg前(黑色)和Mg时的峰值位置2 +-诱导转变(d,青色/蓝色表示正/负轮廓水平)和完全裂解后(e,橙色)。f) rU信号放大+ 6如图(d)所示2 +Dz滴定法5度核糖核酸2ʹF在指定Mg时2 +浓度用紫色虚线表示。在实时光谱中,代表排泄峰和产物峰的区域以棕色和橙色突出显示。Mg的区域2 +自由状态用灰色突出显示。数据表明,观察到的转变不是从Mg开始的2 +而是从一个与Mg的频率相匹配的状态2 +Mg时的平衡态2 +一个交叉峰(T8-H6/H7;正/负轮廓分别用蓝色/青色表示)与最佳拟合模拟光谱(红色/黄色表示正/负轮廓)。给出了初始态和最终态的特征。仿真结果能较好地再现实验数据。请注意,面板a、b、d和g中显示的数据显示了图3中各自数据的放大版本。h)实验和模拟实时核磁共振数据之间的差异作为模拟中应用的不同速率常数的函数。每个分解原子的结果单独显示。最小值代表最佳拟合条件。原子核可以分为两组,一组显示速率比匹配条件下确定的FRET速率慢(棕色虚线),用蓝色表示,另一组显示速率更快(红色表示)。各自的核苷酸被映射在图中的结构上。3 d解理反应时记录的二维光谱间接维数得到T8(H6-H7)的1D切片(d,青色)。实验数据(黑色,表示状态C1)与用2态转换模型(蓝色)或3态转换模型(红色)模拟的峰值形状进行比较。两个模拟光谱都代表了各自模型的最佳拟合。请注意,负肩用3状态模型(虚线箭头)更好地表示。j)所示核苷酸的实验获得的数据与模拟数据之间的差异,作为模拟中使用的速率常数的函数。While for dC+ 5(灰色)整体拟合接近实验数据(可见的差异的相当低的整体值),峰值是不敏感的速率常数的变化(可见的低变化超过应用速率常数的范围)。因此,各自的峰并不是一个很好的转变动力学传感器,因此被排除在数据解释之外。相反,C3的数据显示出一个明确的最小值,并在速率常数的变化方面显示出良好的敏感性。相应的峰值因此被纳入进一步的数据分析。k) G14或六水合Mg相互作用的可能性2 +rG的O2ʹ原子0或rU的O5ʹ原子-1在RNA裂解位点,当处于裂解位点的任何构象时(左),以及当裂解位点处于直列攻击构象时(角O2ʹ-P-O5ʹ:130-180°,右)。如果是G14或Mg的杂原子,则认为存在相互作用2 +距离目标原子5 Å以内。l) Mg的作用2 +在20 mM Mg的MD模拟过程中,与金属离子结合位点II的结合对RNA裂解反应结构特征的频率有影响2 +.m) Mg存在时基于fret的活性测定2 +(红色数据点,1mm)或Mn2 +(蓝色数据点,0.5 mM)5度在G14(左)或G6(右)进行6-硫代修饰。而6-硫代g6的测量行为与以前的报道很一致23, 6-硫代- g14的不寻常行为将与图k所示的酸碱机制一致(右,X1= G14)。
权利和权限
关于本文
引用本文
Borggräfe, J.,维克多,J.,罗森巴赫,H.。et al。rna裂解DNA催化剂的时间分辨结构分析。自然601, 144-149(2022)。https://doi.org/10.1038/s41586-021-04225-4
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586-021-04225-4
这篇文章被引用
在膜系统动力学和生物催化下的原子
BIOspektrum(2022)
