摘要gydF4y2Ba
透明质酸是一种酸性杂多糖gydF4y2BaNgydF4y2Ba-乙酰葡萄糖胺和葡萄糖醛酸糖,在脊椎动物细胞外基质中普遍表达gydF4y2Ba1gydF4y2Ba.高分子质量聚合物调节健康和疾病的基本生理过程,包括细胞分化、组织稳态和血管生成gydF4y2Ba2gydF4y2Ba.透明质酸是由一种膜嵌入的过程糖基转移酶,透明质酸合成酶(HAS)合成的,它催化透明质酸的合成和膜转运从尿苷二磷酸激活的前体gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.在这里,我们描述了在底物结合和聚合物合成起始阶段,病毒HAS同源物在不同状态下的五种冷冻电子显微镜结构。结合生物化学分析和分子动力学模拟,我们的数据揭示了HAS如何选择其底物,水解第一个底物以启动合成反应,打开一个透明质酸传导跨膜通道,确保交替底物聚合并协调透明质酸在其跨膜孔内。我们的研究为酸性细胞外杂多糖的形成提供了一个详细的模型,并为人体中最丰富和最基本的糖胺聚糖之一的生物合成提供了见解。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
几丁质合酶的结构、机制及其抗真菌药物尼可霉素的抑制作用gydF4y2Ba
细胞的发现gydF4y2Ba开放获取gydF4y2Ba12月6日gydF4y2Ba
定向甲壳素生物合成的结构基础gydF4y2Ba
自然gydF4y2Ba开放获取gydF4y2Ba2022年9月21日gydF4y2Ba
访问选项gydF4y2Ba
订阅《自然》+gydF4y2Ba
立即在线访问《自然》和其他55种《自然》杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅期刊gydF4y2Ba
获得1年的完整期刊访问权限gydF4y2Ba
199.00美元gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
增值税稍后将在结帐时添加。gydF4y2Ba
税务计算将在结账时完成。gydF4y2Ba
买条gydF4y2Ba
在ReadCube上获得时间限制或全文访问。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
数据可用性gydF4y2Ba
原始EM视频和地图已按登录代码存入蛋白质数据库和电子显微镜数据库gydF4y2Ba7 sp7gydF4y2Ba而且gydF4y2Baemd - 25367gydF4y2Ba;gydF4y2Ba7 sp6gydF4y2Ba而且gydF4y2Baemd - 25366gydF4y2Ba;gydF4y2Ba7 sp8gydF4y2Ba而且gydF4y2Baemd - 25368gydF4y2Ba;gydF4y2Ba7 sp9gydF4y2Ba而且gydF4y2Baemd - 25369gydF4y2Ba;而且gydF4y2Ba7水疗gydF4y2Ba/gydF4y2Baemd - 25370gydF4y2Ba分别为udp结合,D302N apo, udp - glcacc结合,引物(关闭)和引物(打开)状态。gydF4y2Ba
参考文献gydF4y2Ba
魔术胶透明质酸和它的橡皮透明质酸酶:生物学概述。gydF4y2Ba生命科学。gydF4y2Ba80gydF4y2Ba, 1921-1943(2007)。gydF4y2Ba
Cyphert, J. M., Trempus, C. S. & Garantziotis .尺寸问题:透明质酸在细胞生物学中的作用的分子量特异性。gydF4y2BaInt。J.细胞生物学。gydF4y2Ba2015gydF4y2Ba, 563818(2015)。gydF4y2Ba
Vigetti, D.等人。透明质酸:生物合成和信号。gydF4y2BaBiochim。Biophys。学报gydF4y2Ba1840gydF4y2Ba, 2452-2459(2014)。gydF4y2Ba
透明质酸合成酶:来自脊椎动物、细菌病原体和藻类病毒的糖基转移酶。gydF4y2Ba细胞。生命科学gydF4y2Ba56gydF4y2Ba, 670-682(1999)。gydF4y2Ba
西罗宁等人。透明质酸在人类恶性肿瘤中的作用。gydF4y2Ba细胞实验。。Res。gydF4y2Ba317gydF4y2Ba, 383-391(2011)。gydF4y2Ba
考曼,M. K.李,H.-G。,Schwertfeger, K. L., McCarthy, J. B. & Turley, E. A. The content and size of hyaluronan in biological fluids and tissues.前面。Immunol。gydF4y2Ba6gydF4y2Ba, 261(2015)。gydF4y2Ba
Hubbard, C., McNamara, J., Azumaya, C., Patel, M. & Zimmer, J.透明质酸合成酶催化透明质酸的合成和膜转运。gydF4y2BaJ. Mol.生物学。gydF4y2Ba418gydF4y2Ba, 21-31(2012)。gydF4y2Ba
Weigel, P. H. & Deangelis, P. L.透明质酸合成酶:十多年的新糖基转移酶。gydF4y2Ba生物。化学。gydF4y2Ba282gydF4y2Ba, 36777-36781(2007)。gydF4y2Ba
李丽娟,李志强,李志强,李志强。一种透明质酸合成酶的克隆与鉴定gydF4y2Ba巴斯德菌multocidagydF4y2Ba.gydF4y2Ba生物。化学。gydF4y2Ba273gydF4y2Ba, 8454-8458(1998)。gydF4y2Ba
Camenisch, t.d.等人。透明质酸合成酶-2的破坏取消了正常的心脏形态发生和透明质酸介导的上皮细胞向间质的转化。gydF4y2Baj .中国。投资。gydF4y2Ba106gydF4y2Ba, 349-360(2000)。gydF4y2Ba
Itano, N.等人。哺乳动物透明质酸合成酶的三种异构体具有不同的酶性质。gydF4y2Ba生物。化学。gydF4y2Ba274gydF4y2Ba, 25085-25092(1999)。gydF4y2Ba
李志刚,李志刚,李志刚,李志刚。小球藻病毒PBCV-1透明质酸合成酶的研究进展。gydF4y2Ba科学gydF4y2Ba278gydF4y2Ba, 1800-1803(1997)。gydF4y2Ba
布莱克本,m.r.等。透明质酸在不同生命王国中生物合成的不同反应机制。gydF4y2Ba糖生物学gydF4y2Ba28gydF4y2Ba, 108-121(2018)。gydF4y2Ba
对不起,e等人。结构生物学纳米体生成的一般方案。gydF4y2BaProtoc Nat。gydF4y2Ba9gydF4y2Ba, 674-693(2014)。gydF4y2Ba
特拉帕克-西蒙斯,V. L., Baron, C. A. & Weigel, P. H.从纯化的透明质酸合成酶的表征gydF4y2Ba链球菌equisimilisgydF4y2Ba.gydF4y2Ba生物化学gydF4y2Ba43gydF4y2Ba, 9234-9242(2004)。gydF4y2Ba
Punjani, A., Rubinstein, J. L., Fleet, D. J. & Brubaker, M. A. cryoSPARC:快速无监督冷冻em结构确定的算法。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 290-296(2017)。gydF4y2Ba
Lairson, L. L., Henrissat, B., Davies, G. J. & Withers, S. G.糖基转移酶:结构、功能和机制。gydF4y2Ba为基础。学生物化学启。gydF4y2Ba77gydF4y2Ba, 521-555(2008)。gydF4y2Ba
Cantarel, B., Coutinho, P., Rancurel, C. & Bernard, T.碳水化合物活性酶数据库(CAZy):糖基因组学的专家资源。gydF4y2Ba核酸测定。gydF4y2Ba37gydF4y2Ba, d233-d238(2009)。gydF4y2Ba
吴,Q.等。利用宏基因组序列数据和残余神经网络预测蛋白质接触。gydF4y2Ba生物信息学gydF4y2Ba36gydF4y2Ba, 41-48(2020)。gydF4y2Ba
唐雅苏vunakool, K. Adler, J., Wu, Z. Jumper, J. & Hassabis, D.人类蛋白质组的高精度蛋白质结构预测。gydF4y2Ba自然gydF4y2Ba596gydF4y2Ba, 590-596(2021)。gydF4y2Ba
Baek, M.等。使用三轨道神经网络准确预测蛋白质结构和相互作用。gydF4y2Ba科学gydF4y2Ba373gydF4y2Ba, 871-876(2021)。gydF4y2Ba
李志强,李志强,李志强,等。核苷酸-二磷酸-糖转移酶的结构研究gydF4y2Ba枯草芽孢杆菌gydF4y2Ba,以天然和核苷酸复合物的形式存在。gydF4y2Ba生物化学gydF4y2Ba38gydF4y2Ba, 6380-6385(1999)。gydF4y2Ba
Morgan, J., Strumillo, J. & Zimmer, J.纤维素合成和膜易位的晶体学快照。gydF4y2Ba自然gydF4y2Ba493gydF4y2Ba, 181-186(2013)。gydF4y2Ba
何丽娟,何丽娟,张志刚,等。一种具有催化活性的同种三聚体植物纤维素合成酶的结构。gydF4y2Ba科学gydF4y2Ba369gydF4y2Ba, 1089-1094(2020)。gydF4y2Ba
陈建平,陈建平,陈建平,等。细菌纤维素合成酶活性的研究进展。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba21gydF4y2Ba, 489-496(2014)。gydF4y2Ba
McManus, J., Yang, H., Wilson, L., Kubicki, J. & Tien, M.细菌纤维素合成的起始,延伸和终止。gydF4y2BaACSωgydF4y2Ba3.gydF4y2Ba, 2690-2698(2018)。gydF4y2Ba
Orlean, P. & Funai, D.几丁质链的启动和延伸:对几丁质合酶机制的影响。gydF4y2Ba细胞冲浪。gydF4y2Ba5gydF4y2Ba, 100017(2019)。gydF4y2Ba
杨,J.等。透明质酸合酶羧基端在透明质酸聚合物的加工合成和尺寸控制中的关键作用。gydF4y2Ba《生物高分子gydF4y2Ba18gydF4y2Ba, 1064-1073(2017)。gydF4y2Ba
摩根,j.l.等。晶体中纤维素生物合成和膜转运的观察。gydF4y2Ba自然gydF4y2Ba531gydF4y2Ba, 329-334(2016)。gydF4y2Ba
PyMol: PyMol分子图形系统v.2.5.0 (Schrödinger, 2021)。gydF4y2Ba
Ho, B. & Gruswitz, F. HOLLOW:在分子结构中生成通道和内部表面的准确表示。gydF4y2BaBMC结构。医学杂志。gydF4y2Ba8gydF4y2Ba, 49(2008)。gydF4y2Ba
研究人员,F.在高密度震动培养中通过自动感应产生蛋白质。gydF4y2Ba蛋白质Expr。Purif。gydF4y2Ba41gydF4y2Ba, 207-234(2005)。gydF4y2Ba
凯利,泰勒,K. B.李,S. & Jedrzejas, M. JgydF4y2Ba链球菌引起的肺炎gydF4y2Ba透明质酸盐裂合酶。gydF4y2Ba糖生物学gydF4y2Ba11gydF4y2Ba, 297-304(2001)。gydF4y2Ba
Rohou, A. & Grigorieff, N. CTFFIND4:快速准确的电子显微图像离焦估计。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba192gydF4y2Ba, 216-221(2015)。gydF4y2Ba
埃姆斯利,P. &考坦,K.库特:分子图形模型构建工具。gydF4y2BaActa Crystallogr。DgydF4y2Ba60gydF4y2Ba, 2126-2132(2004)。gydF4y2Ba
亚当斯等人。PHENIX:一个全面的基于python的大分子结构解决方案系统。gydF4y2BaActa Crystallogr。DgydF4y2Ba66gydF4y2Ba, 213-221(2010)。gydF4y2Ba
彼得森,E. F.等人。UCSF chimera -用于探索性研究和分析的可视化系统。gydF4y2Baj .第一版。化学。gydF4y2Ba25gydF4y2Ba, 1605-1612(2004)。gydF4y2Ba
黄,J.等。CHARMM36m:用于折叠和内在无序蛋白质的改进力场。gydF4y2Ba自然gydF4y2Ba14gydF4y2Ba, 71-73(2016)。gydF4y2Ba
Jo, S., Kim, T., Iyer, V. G. & Im, W. CHARMM- gui: CHARMM基于web的图形用户界面。gydF4y2Baj .第一版。化学。gydF4y2Ba29gydF4y2Ba, 1859-1865(2008)。gydF4y2Ba
李,J.等。CHARMM- gui输入发生器NAMD, GROMACS, AMBER, OpenMM,和CHARMM/OpenMM模拟使用CHARMM36加性力场。gydF4y2Baj .化学。西奥。第一版。gydF4y2Ba12gydF4y2Ba, 405-413(2016)。gydF4y2Ba
Sondergaard, c.r, Olsson, m.h.m, Rostkowski, M. & Jensen, j.h.改进的配体处理和耦合效应在经验计算和合理化的pgydF4y2BaKgydF4y2Ba一个gydF4y2Ba值。gydF4y2Baj .化学。理论第一版。gydF4y2Ba7gydF4y2Ba, 2284-2295(2011)。gydF4y2Ba
奥尔森,M. H. M., Sondergaard, C. R., Rostkowski, M. & Jensen, J. H. PROPKA3:经验p中内部和表面残留的一致处理gydF4y2BaKgydF4y2Ba一个gydF4y2Ba预测。gydF4y2Baj .化学。理论第一版。gydF4y2Ba7gydF4y2Ba, 525-537(2011)。gydF4y2Ba
Bussi, G., Donadio, D. & Parrinello, M.通过速度重尺度的规范采样。gydF4y2Baj .化学。理论物理。gydF4y2Ba126gydF4y2Ba, 014101(2007)。gydF4y2Ba
单晶的多态转变:一种新的分子动力学方法。gydF4y2Baj:。理论物理。gydF4y2Ba52gydF4y2Ba, 7182-7190(1981)。gydF4y2Ba
Berendsen, H. J. C., van der Spoel, D. & van Drunen, R. GROMACS:消息传递并行分子动力学实现。gydF4y2Ba第一版。理论物理。Commun。gydF4y2Ba91gydF4y2Ba, 43-56(1994)。gydF4y2Ba
汉弗莱,W., Dalke, A. & Schulten, K. VMD:视觉分子动力学。gydF4y2BaJ. Mol. Graph。gydF4y2Ba14gydF4y2Ba, 33-38(1996)。gydF4y2Ba
michael - agrawal, N., Denning, E. J., Woolf, T. B. & Beckstein, O. MDAnalysis:用于分子动力学模拟分析的工具包。gydF4y2Baj .第一版。化学。gydF4y2Ba32gydF4y2Ba, 2319-2327(2011)。gydF4y2Ba
哈里斯,C. R.等。使用NumPy进行数组编程。gydF4y2Ba自然gydF4y2Ba585gydF4y2Ba, 357-362(2020)。gydF4y2Ba
亨特,J. D. Matplotlib:一个2D图形环境。gydF4y2Ba第一版。科学。Eng。gydF4y2Ba9gydF4y2Ba, 90-95(2007)。gydF4y2Ba
Bernsel, A., Viklund, H., Hennerdal, A. & Elofsson, A. TOPCONS:膜蛋白拓扑结构的共识预测。gydF4y2Ba核酸测定。gydF4y2Ba37gydF4y2Ba, w465-w468(2009)。gydF4y2Ba
确认gydF4y2Ba
我们感谢弗吉尼亚大学MEMC的K. Dryden和M. Purdy,以及A. Wier和NCI-NCEF的支持人员。E.P.和J.S.感谢Instruct-ERIC(欧洲研究基础设施战略论坛(ESFRI)的一部分)和研究基金会-弗兰德斯(FWO)对纳米体发现的支持,并感谢E. Beke的技术援助。J.Z.和F.P.M.由NIH拨款R21AI148853资助。R.H.得到了NIH拨款R01GM101001(授予J.Z.)的支持。R.A.C.和P.J.S.由惠康(208361/Z/17/Z)资助。p.j.s.的实验室得到了BBSRC (BB/P01948X/1, BB/R002517/1和BB/S003339/1)和MRC (MR/S009213/1)的奖励。P.J.S.承认华威大学科学计算研究技术平台的计算访问。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
yb克隆了Cv-HAS并制造了用于纳米体生产的材料。E.P.和J.S.制造了纳米体。yb和L.M.对纳米体进行了表征。L.M.进行了热稳定性测试。F.P.M.纯化了cv - has -纳米体复合物,并制备了纳米圆盘。R.H.收集EM数据,F.P.M.和J.K.处理数据。F.P.M.测定了载脂蛋白和udp结合的Cv-HAS结构。jk确定了底物结合和引物的Cv-HAS结构。R.A.C.完成了所有的MD模拟。J.Z.写了第一份手稿。 F.P.M., J.K., R.A.C., P.J.S. and J.Z. edited the draft. All of the authors commented on the manuscript.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢匿名审稿人对本工作的同行评议所作的贡献。同行评审报告是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
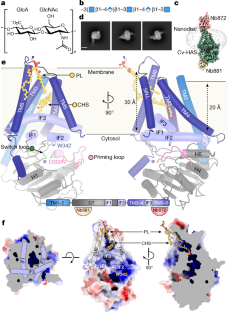
扩展数据图1 HAS正交基序列比对。gydF4y2Ba
中HAS主序列的比较gydF4y2Ba小球藻病毒gydF4y2Ba(简历)gydF4y2Ba智人gydF4y2Ba(Hs)和gydF4y2Ba链球菌equisimilisgydF4y2Ba(Se)。拓扑预测使用TopCons执行gydF4y2Ba50gydF4y2Ba.圆柱体表示在Cv-HAS中观察到的二级结构元素。gydF4y2Ba
扩展数据图2与两个纳米体和UDP绑定的Cv-HAS的识别、数据收集和处理。gydF4y2Ba
(gydF4y2Ba一个gydF4y2BaNb872存在时Cv-HAS的熔化温度升高。蛋白质融化是基于实时量化UDP释放检测的酶活性来测量的。(gydF4y2BabgydF4y2Ba) HA生物合成在指示的纳米体的存在,并基于定量gydF4y2Ba3.gydF4y2Ba闪烁计数h标记HA。在没有纳米体的情况下,数据相对于产品收率进行归一化。误差条表示n = 3个独立实验时与均值的偏差。(gydF4y2BacgydF4y2Ba有代表性的放射自显影gydF4y2Ba14gydF4y2Bac标记的HA在指定的纳米体存在下产生。这个实验至少重复了4次,结果基本相同。NC:无UDP-GlcNAc底物(面板b)或UDP-GlcA底物(面板c)阴性对照。PC:无纳米体阳性对照。裂解酶:SDS-PAGE前的透明质酸裂解酶处理。(gydF4y2BadgydF4y2Ba这个工作流产生了udp绑定的Cv-HAS结构。gydF4y2Ba
扩展数据图3 Cv-HAS D302N在衬底存在下的Cryo-EM数据采集与处理。gydF4y2Ba
该工作流程生成了载脂蛋白、底物结合、引物和开通道Cv-HAS结构引物。gydF4y2Ba
图4 udp绑定Cv-HAS的地图质量和模型构建。gydF4y2Ba
(gydF4y2Ba模拟gydF4y2Ba)地图概览,基于FSC的估计分辨率,粒子方向分布。(gydF4y2BaegydF4y2BaCv-HAS的二级结构元素和拓扑结构。(gydF4y2Baf jgydF4y2BaCv-HAS的TM螺旋为2 ~ 6。(gydF4y2BakgydF4y2Ba) tmh2 -4细胞外环。(gydF4y2BalgydF4y2Ba)细胞外tmh6 -6环。(gydF4y2Ba米gydF4y2Ba) QxxRW主题。(gydF4y2BangydF4y2Ba) c端细胞质螺旋。(gydF4y2BaogydF4y2Ba)未解决的TMH5-IF3循环。所有地图的等高线都是7.0σ。gydF4y2Ba
图5 TMH1的预测位置。gydF4y2Ba
(gydF4y2Ba一个gydF4y2BaCv-HAS ' TM和GT区域内进化耦合残基的关系,基于65,535个序列在MapPred中生成。TMH1在其预测位置显示为紫色圆柱体。(gydF4y2BabgydF4y2Ba) RoseTTAfold全长度Cv-HAS模型。Cv-HAS显示为曲面,其tmh2显示为蓝色圆柱体。TMH1在其预测位置以卡通形式显示。(gydF4y2BacgydF4y2Ba) AlphaFold2预测的人类HAS2结构(从N-到c端被涂成蓝色到红色)与Cv-HAS结构叠加,显示为灰色卡通和半透明表面。(gydF4y2BadgydF4y2Ba)当两个细胞质纳米体用于低温电镜分析时,TMH1仍然是无序的。(gydF4y2BaegydF4y2BaTMH1截断Cv-HAS的催化活性。左:用于imv的Western blotgydF4y2Ba在体外gydF4y2Ba活动测量。右图:相对于野生型酶表达的指示Cv-HAS突变体的催化活性。该方法量化gydF4y2Ba3.gydF4y2Ba闪烁计数h标记HA。对照反应在缺乏UDP-GlcA作为背景,并被减去。误差条表示n = 3个独立实验时与均值的偏差。gydF4y2Ba
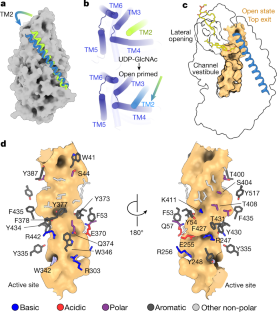
扩展数据图6脂质堵塞侧通道开口。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)建模脂质轮廓在7.0σ的代表性地图区域(来自UDP-GlcNAc边界集)。(gydF4y2BabgydF4y2Ba)来自POPE双分子层内Cv-HAS(黑色区域)MD模拟的2D切片。水和脂密度分别用蓝色和绿色表示。右图:与选定通道残基的脂质接触时间。(gydF4y2BacgydF4y2BaCv-HAS(从N端到c端呈彩虹色)和RsBcsA(灰色,4P00)的比较。与BcsA相关的纤维素呈黑色棒状。除了BcsA的n端两个ths以线圈的形式显示外,螺旋显示为圆柱体。gydF4y2Ba
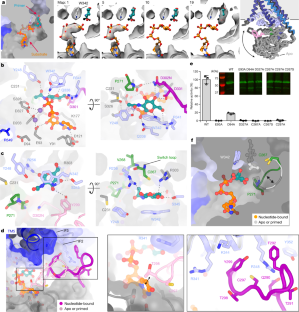
扩展数据图7底物结合和引物诱导构象变化的细节。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba而且gydF4y2BabgydF4y2Ba) UDP- glcnac, UDP和Mn的映射质量gydF4y2Ba2+gydF4y2Ba配体。(gydF4y2BacgydF4y2Ba) UDP和UDP- glcnac位置的比较。(gydF4y2BadgydF4y2Ba)核苷酸结合态启动环的图谱。(gydF4y2BaegydF4y2BaGlcNAc引物的代表图谱。(gydF4y2BafgydF4y2Ba)在启动状态下的启动循环映射。(gydF4y2BaggydF4y2BaTMH2与处于引物状态的IF1的接触点(蓝色打开,灰色关闭)。(gydF4y2BahgydF4y2Ba而且gydF4y2Ba我gydF4y2BaTMH2在闭合位置(UDP-GlcNAc结合)和开放位置的质量图。所有地图的等高线都是7.0σ。gydF4y2Ba
图8 HA生物合成的GlcNAc启动。gydF4y2Ba
所示为SDS-PAGE后14c标记HA的自显像图。这个实验至少重复了3次,结果基本相同。gydF4y2Ba
图9单糖对底物水解的影响。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba而且gydF4y2BabgydF4y2Ba) UDP-GlcA和UDP-GlcNAc水解反应方案。(gydF4y2BacgydF4y2Ba而且gydF4y2BadgydF4y2Ba)原始吸光度测量。(gydF4y2BaegydF4y2Ba)在单糖浓度增加的情况下,水解速率的定量。蓝色和红色分别为UDP-GlcNAc和UDP-GlcA的水解。浅色和深色代表不含酶的对照反应。右图:背景减去水解速率。误差条表示n = 3个独立实验时与均值的偏差。gydF4y2Ba
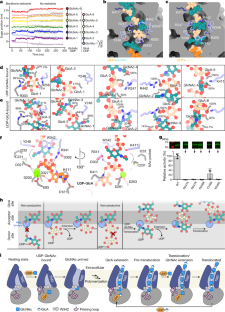
图10底物交替聚合的可能机理及与纤维素合酶的比较。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)底物结合和启动的Cv-HAS结构的叠加。引物与供体糖之间的距离用灰色条表示。(gydF4y2BabgydF4y2Ba)中系统C231与GlcNAc的接触可能性gydF4y2Ba一个gydF4y2Ba在每个模拟的最后125纳秒。在GlcNAc同时处于供体和受体位置的情况下,两个GlcNAc单元都不太可能结合C231,并且在结合位姿方面表现出非常高的方差。特别值得注意的是,两个GlcNAc同时出现在C231口袋中的几率非常低(约1.5±0.8%)。(gydF4y2BacgydF4y2BaCv-HAS与thegydF4y2BaRhodobacter sphaeroidesgydF4y2Ba(Rs)基于二级结构匹配的BcsA-B配合物(PDB: 4P00)。Rs-BcsA-B为灰色,Cv-HAS为TM和GT域的蓝色和绿色。与Rs-BcsA-B相结合的纤维素聚合物显示为黑色棒状。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
补充讨论1-3,补充表1-3和补充参考文献。gydF4y2Ba
补充视频1gydF4y2Ba
连续的体积级数,表示活性部位的密度差。图2a所示的简单三维变异性显示作业的密度叠加。Trp342以棒状表示,UDP-GlcNAc和GlcNAc引物分别以碳原子为灰色和青色的球状和棒状表示。gydF4y2Ba
补充视频2gydF4y2Ba
启动回路的运动。从一个简单的3D变异性显示输出叠加一个Chimera变形,由启动开放和udp - glcacc结合状态的模型创建。视频从启动的开放状态振荡到udp - glcacc结合的状态,然后回到启动的开放状态。gydF4y2Ba
补充视频3gydF4y2Ba
底物诱导Cv-HAS的整体构象变化。从一个简单的3D变异性显示输出叠加一个Chimera变形,由启动开放和UDP-GlcNAc结合状态的模型创建。从启动开放状态振荡到udp - glcacc结合状态,再回到启动开放状态。gydF4y2Ba
补充视频4gydF4y2Ba
TMH2的倾斜和假定HA通道的打开。从一个简单的3D变异性显示输出叠加一个奇美拉变形从启动打开和启动关闭状态的模型创建。蓝色,TMH2;橙色,TMH4;绿色TMH6。从导导闭合状态振荡到导导开启状态再回到导导闭合状态。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
马洛尼,f.p.,库克勒维茨,J.,科里,R.A.gydF4y2Baet al。gydF4y2Ba透明质酸合酶的结构、底物识别及启动。gydF4y2Ba自然gydF4y2Ba604gydF4y2Ba, 195-201(2022)。https://doi.org/10.1038/s41586-022-04534-2gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-04534-2gydF4y2Ba
这篇文章被引用gydF4y2Ba
硫酸肝素共聚合酶作用的结构基础EXT1-2配合物gydF4y2Ba
自然化学生物学gydF4y2Ba(2023)gydF4y2Ba
几丁质合酶的结构、机制及其抗真菌药物尼可霉素的抑制作用gydF4y2Ba
细胞的发现gydF4y2Ba(2022)gydF4y2Ba
定向甲壳素生物合成的结构基础gydF4y2Ba
自然gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
