摘要
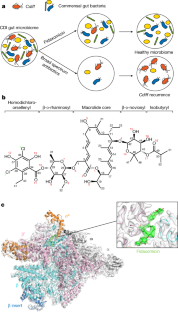
非达霉素(Fdx)被广泛用于治疗Clostridioides固执的(Cdiff但其在人类肠道微生物组中的窄谱活性的分子基础仍然未知。Cdiff感染是医院死亡的主要原因1.抑制RNA聚合酶的非达霉素是靶标Cdiff对肠道共生菌影响最小,减少复发Cdiff感染2,3..在这里,我们提出了冷冻电子显微镜结构CdiffRNA聚合酶与非达霉素的复合物,并鉴定出一个关键的非达霉素结合行列式Cdiff大多数肠道菌群如变形菌门和拟杆菌门中不存在的RNA聚合酶。通过结合结构,生化,遗传和生物信息学分析,我们确定了一个单一的残基CdiffRNA聚合酶是非达霉素窄谱活性的增敏元件。我们的研究结果为针对一种重要的人类病原体的靶向药物设计提供了蓝图。
这是订阅内容的预览,通过你所在的机构访问
相关的文章
引用本文的开放获取文章。
从信号转导到蛋白质毒素——艰难梭菌毒素研究路线里程碑的叙事回顾
Naunyn-Schmiedeberg的药理学档案开放获取10月7日
访问选项
访问《自然》和其他54种《自然》杂志
获取Nature+,我们最超值的在线订阅
每月29.99美元
随时取消
订阅这本杂志
收到51个印刷问题和在线访问
199.00美元一年
每期仅需3.90美元
租或购买这篇文章
只要这篇文章,只要你需要它
39.95美元
价格可能受当地税收的影响,在结账时计算
参考文献
疾病预防控制中心。2019年美国抗生素耐药性威胁(美国卫生与公众服务部,疾控中心2019年);https://www.cdc.gov/drugresistance/pdf/threats-report/2019-ar-threats-report-508.pdf
路易,t.j.等人。非达霉素和万古霉素艰难梭状芽胞杆菌感染。心血管病。j .地中海。364, 422-431(2011)。
Crawford, T., Huesgen, E. & Danziger, L. Fidaxomicin:一种新型大环抗生素治疗艰难梭状芽胞杆菌感染。点。J.健康系统。制药。69, 933-943(2012)。
Louie, t.j, Emery, J, Krulicki, W, Byrne, B. & Mah, M. OPT-80消除艰难梭状芽胞杆菌并且在处理过程中保留了拟杆菌的种类梭状芽孢杆菌感染。Antimicrob。代理Chemother。53, 261-263(2009)。
李,Y. J.等。肠道微生物组中的保护因子艰难梭状芽胞杆菌异基因造血干细胞移植受者的感染。j .感染。说。215, 1117-1123(2017)。
Vincent, C. & Manges, a.r.抗菌药物的使用,人类肠道菌群和艰难梭状芽胞杆菌定植和感染。抗生素4, 230-253(2015)。
波雅齐等人。Fidaxomicin堵塞结核分枝杆菌通过RbpA接触启动所需的RNA聚合酶运动。eLife7, e34823(2018)。
林,W.等。非达霉素(脂霉素A3)转录抑制的结构基础。摩尔。细胞70, 60 - 71。e15(2018)。
Morichaud, Z., Chaloin, L. & Brodolin, K. RNA聚合酶σ亚基的1.2和3.2区域促进DNA融化并减弱抗生素脂霉素的作用。J. Mol.生物学。428, 463-476(2016)。
Tupin, A., Gualtieri, M., Leonetti, J. P. & Brodolin, K.转录抑制剂脂霉素阻断DNA进入RNA聚合酶催化位点。EMBO J。29, 2527-2537(2010)。
boaci, H., Chen, J., Jansen, R., Darst, S. A. & Campbell, E. A. RNA聚合酶启动子熔化中间体的结构阐明DNA解绕。自然565, 382-385(2019)。
陈俊杰,柏亚奇,H. & Campbell, E. A.细菌转录起始机制的多样性和统一。Nat. Rev.微生物。19, 95-109(2021)。
Lane, W. J. & Darst . S. A.多亚基RNA聚合酶的分子进化:序列分析。J. Mol.生物学。395, 671-685(2010)。
马尼,N., Dupuy, B. & Sonenshein, A. L. RNA聚合酶的分离艰难梭状芽胞杆菌谷氨酸脱氢酶和rRNA基因启动子的体外和体内特性。j . Bacteriol。188, 96-102(2006)。
Cardone, G., Heymann, J. B. & Steven, A. C.一个数字并不适用于所有情况:在低温em重建中映射分辨率的局部变化。j . Struct。医学杂志。184, 226-236(2013)。
Bae, B.等人。噬菌体T7 Gp2的抑制作用大肠杆菌RNA聚合酶涉及σ的挪用70域1.1。国家科学院学报美国110, 1972 - 19777(2013)。
裴,H. H.等。δ亚基和NTPase HelD建立了RNA聚合酶循环的双管齐下机制。Commun Nat。11, 6418(2020)。
方,C.等。细菌多药耐药调节因子BmrR扭曲启动子DNA以激活转录。Commun Nat。11, 6284(2020)。
坎贝尔,e.a.等人。细菌RNA聚合酶启动子特异性σ亚基的结构。摩尔。细胞9, 527-539(2002)。
胡斌,E. A.等。分枝杆菌转录起始复合物的结构和功能与必要的调节RbpA。eLife6, e22520(2017)。
Chen, J.等。6S RNA模仿b型DNA调节大肠杆菌RNA聚合酶。摩尔。细胞68, 388 - 397。e386(2017)。
费克里斯托夫,A.等人。启动子熔化过程中RNA聚合酶的运动。科学356, 863-866(2017)。
Babakhani, F., Seddon, J. & Sears, P.转录抑制剂非达霉素和利福霉素的比较微生物学研究艰难梭状芽胞杆菌.Antimicrob。代理Chemother。58, 2934-2937(2014)。
Kuehne, S. A.等。rpoB突变对小鼠体外和体内竞争适应度影响的表征艰难梭状芽胞杆菌以及对非达霉素的易感性。j . Antimicrob。Chemother。73, 973-980(2018)。
Goldstein, E. J., Babakhani, F. & Citron, D. M. fidaxomicin的抗菌活性。中国。感染。说。55, s143-s148(2012)。
Kurabachew等人。脂霉素以RNA聚合酶为靶点,对多药耐药菌株有较好的抑制作用结核分枝杆菌.j . Antimicrob。Chemother。62, 713-719(2008)。
斯里瓦斯塔瓦,A.等。抑制细菌RNA聚合酶的新靶点:“开关区”。咕咕叫。当今。Microbiol。14, 532-543(2011)。
福斯特,s.c.等。用于改进宏基因组分析的人类肠道细菌基因组和培养集。生物科技Nat。》。37, 186-192(2019)。
秦,等。宏基因组测序建立的人体肠道微生物基因目录。自然464, 59-65(2010)。
韦克斯勒,a . G. &古德曼,a . L.内部人士的观点:拟杆菌作为微生物组的窗口。Nat。Microbiol2, 17026(2017)。
金,C. H.等人。健康人肠道菌群的基线概况和标准报告模板。《公共科学图书馆•综合》14, e0206484(2019)。
Corbett, D.等人。一种新型阳离子肽增强抗生素活性:SPR741的效力和活性谱。Antimicrob。代理Chemother。61, e00200-17(2017)。
瓦拉,等人。一种新的多粘菌素衍生物缺乏脂肪酸尾巴,只携带三个正电荷,与完整的外膜所排除的制剂具有很强的协同作用。Antimicrob。代理Chemother。54, 3341-3346(2010)。
韦尔奇等人。设计控制合成基因表达的参数大肠杆菌.《公共科学图书馆•综合》4, e7002(2009)。
Salis, H. M. Mirsky, E. A. & Voigt, C. A.合成核糖体结合位点控制蛋白表达的自动化设计。生物科技Nat。》。27, 946-950(2009)。
Czyz, A., Mooney, R. A., Iaconi, A. & Landick, R.分枝杆菌RNA聚合酶在固有终止位点上需要u束,在次优终止位点上由NusG辅助。Mbio5, e00931(2014)。
杨晓林,杨晓林,李建平。重组蛋白的生产过剩与纯化枯草芽孢杆菌RNA聚合酶。蛋白质Expr。Purif。59, 86-93(2008)。
Davis, E, Chen, J, Leon, K, Darst, S. A. & Campbell, E. A.分枝杆菌RNA聚合酶形成不稳定的开放启动子复合物,由CarD稳定。核酸测定。43, 433-445(2015)。
莫林,A.等。协作可以最大限度地利用软件。eLife2, e01456(2013)。
Suloway, C.等人。自动化分子显微镜:新的Leginon系统。j . Struct。医学杂志。151, 41-60(2005)。
郑世强等。MotionCor2:改进冷冻电子显微镜光束诱导运动的各向异性校正。Nat方法。14, 331-332(2017)。
Punjani, A., Rubinstein, J. L., Fleet, D. J. & Brubaker, M. A. cryoSPARC:快速无监督冷冻em结构确定的算法。Nat方法。14, 290-296(2017)。
Zivanov, J.等人。在RELION-3中自动高分辨率冷冻- em结构测定的新工具。eLife7, e42166(2018)。
彭贾尼,A.,张,H.和Fleet, D. J.非均匀细化:自适应正则化改进单粒子低温em重建。Nat方法。17, 1214-1221(2020)。
海曼,j.b.和贝尔纳普,d.m.b软:图像处理和分子建模的电子显微镜。j . Struct。医学杂志。157, 3-18 (2007)
沃特豪斯等人。瑞士模型:蛋白质结构和复合物的同源模型。核酸测定。46, w296-w303(2018)。
郭,X.等。NusA稳定转录暂停的结构基础。摩尔。细胞69, 816 - 827。e814(2018)。
彼得森,E. F.等人。UCSF chimera -用于探索性研究和分析的可视化系统。j .第一版。化学。25, 1605-1612(2004)。
Afonine, P. V.等。用于冷冻电镜和结晶学的PHENIX实空间细化。Acta Crystallogr。D74, 531-544(2018)。
Emsley, P., Lohkamp, B., Scott, W. G. & Cowtan, K.白骨顶的特征与发育。Acta Crystallogr。D66, 486-501(2010)。
Jumper, J.等。高度准确的蛋白质结构预测AlphaFold。自然596, 583-589(2021)。
Afonine, P. V.等。用于分析和验证低温电磁图和原子模型的新工具。Acta Crystallogr。D74, 814-840(2018)。
图洛霍诺夫,张杰,帕朗加,M.和Landick, R. RNA聚合酶触发环在转录暂停期间活性位点重排中的核心作用。摩尔。细胞27, 406-419(2007)。
Weilbaecher, R., Hebron, C., Feng, G. & Landick, R. β '亚基的末端改变氨基酸取代大肠杆菌RNA聚合酶识别RNA链延伸的区域。Dev的基因。8, 2913-2927(1994)。
Burby, p.e. & Simmons, l.a . CRISPR/Cas9编辑枯草芽孢杆菌基因组。生物。Protoc。7, e2272(2017)。
Hockett, K. L. & Baltrus, D. A.使用软琼脂覆盖技术筛选细菌产生的抑制化合物。J. Vis. Exp。119, 55064(2017)。
华莱士,a . C.拉斯科夫斯基,R. a .和桑顿,J. M. LIGPLOT:生成蛋白质配体相互作用示意图的程序。蛋白质中。8, 127-134(1995)。
RAxML-VI-HPC:基于最大似然的系统发育分析,涉及数千个类群和混合模型。生物信息学22, 2688-2690(2006)。
Letunic, I. & Bork, P.交互式生命树(iTOL) v4:最新更新和新发展。诊断。酸Res。47(w1), w256-w259(2019)。
确认
我们感谢S. Darst在本研究期间的有益讨论;R. Mooney提供了σ70用于转录分析的蛋白质;Wang J.提供质粒pJW557;请M. Young和J. Yang指导枯草芽孢杆菌遗传学;洛克菲勒大学Evelyn Gruss Lipper冷冻电子显微镜资源中心的M. Ebrahim, J. Sotiris和Honkit Ng;E. Eng和K. Maruthi负责收集低温电磁数据。其中一些工作是在西蒙斯电子显微镜中心和位于纽约结构生物学中心的国家自动化分子显微镜资源进行的,由西蒙斯基金会(SF349247)、NYSTAR、Agouron研究所(F00316)和美国国立卫生研究院(GM103310, OD019994)资助。这项研究得到了美国国立卫生研究院(NIH)对R.L. (GM38660)和E.A.C. (GM114450)的资助,以及Revson基金会对H.B. (CEN5650030)的资助。
作者信息
作者及隶属关系
贡献
e。a。c。r。l监督这项工作。X.C.和H.B.进行了生化和功能检测。h.b., E.A.C.和J.C.确定了低温电镜结构并建立了结构模型。X.C.进行了生物信息学分析。Y.B.协助进行蛋白质纯化。x.c., h.b., E.A.C.和R.L.根据所有作者的意见撰写了这份手稿。
相应的作者
道德声明
相互竞争的利益
作者声明没有利益竞争。
同行评审
同行评审信息
自然感谢Robert Britton和其他匿名审稿人对这项工作的同行评审所做的贡献。可以获得同行评审报告。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
扩展的数据图形和表格
图1基因的过表达和纯化CdiffRNAP。
一个, pXC026为过表达质粒CdiffrpoA, rpoZ, rpoB和rpoC基因(编码α, ω, β和β′亚基)Cdiff分别为RNAP)。β和β’亚基与亚基间10-氨基酸(aa)连接子(LARHVGGSGA)和c端鼻病毒3C蛋白酶可裂解His10标记融合。b,(上)为组装的排除尺寸色谱剖面CdiffRNAP Eσ一个.(下)各主峰组分的考马斯染色SDS-PAGE。RNAP亚基标记在凝胶的右侧。收益率为CdiffRNAP Eσ一个从汇集的馏分的第二个峰是足够从单一纯化和用于生物化学和结构生物学实验。c,流产转录测定Cdiff核和Eσ一个使用CdiffrrnC启动子作为DNA模板。的转录活性CdiffEσ一个随Fdx浓度的增加而被抑制。转录实验独立重复三次,结果相似(n = 3)。最后给出了一个具有代表性的实验结果。最后给出了一个具有代表性的实验结果。巷1,CdiffRNAP核心;2,CdiffEσ一个;巷3,CdiffEσ一个添加0.2µM Fdx;巷4,CdiffEσ一个加入2µM Fdx。
扩展数据图2冷冻电镜处理管道。
的冷冻电子显微镜数据的图像处理管道流程图CdiffEσ一个/Fdx复合物,从配备K3 Summit直接电子探测器(Gatan)的300kev Titan Krios (FEI)上收集的6,930个剂量分级电影开始。电影帧对齐并使用MotionCor2进行汇总41.使用cryoSPARC2计算每张显微照片的CTF估计42.MotionCor2处理后的代表性显微照片41.用cryoSPARC2从每张显微照片中自动挑选颗粒42然后利用cryoSPARC2对Blob Picker进行二维分类,以评估质量。显示了从2D分类中选择的类。通过二维分类进行拾取和清洗后,数据集包含2415902个粒子。粒子的一个子集被用来生成一个从头开始使用cryoSPARC2对这些模板进行三维异构细化42.出现了一个主要的高分辨率类,使用RELION对其进行了优化43并用另外两个3D异质细化进一步清洗。最终的182,390个颗粒使用cryoSPARC非均匀细化44.
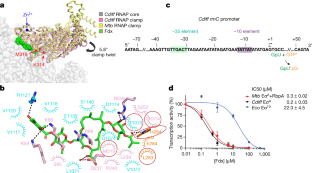
图3低温电镜分析。
一个,左上为3.26 Å-resolution冷冻- em密度图CdiffEσ一个/不会。右上,结构的横截面,显示Fdx。底部,与上面相同的视图,但由局部分辨率着色,框内区域被放大并显示为插图。Fdx的密度用红色标出15.b,金标FSC地块CdiffEσ一个/Fdx配合物来自cryoSPARC42.虚线表示黄金标准0.143 FSC截止值,表示标称分辨率为3.26 Å。c,在cryoSPARC中计算的角分布CdiffEσ一个/Fdx粒子投影。热图显示了每个视角的粒子数量(少=蓝色,多=红色)42.d,在精化结构之间计算映射到模型拟合的交叉验证FSC图CdiffEσ一个/Fdx和用于细化的半映射(工作,红色)、另一个半映射(自由,蓝色)和完整映射(黑色)。黑点线表示为完整地图确定的0.5 FSC界限52.
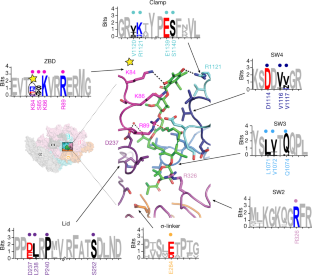
扩展数据图5 σ的差异一个-Fdx联系人之间Cdiff而且快艇和σ一个序列比对。
一个的保守地区Cdiffσ一个相比快艇σ一个而且大肠杆菌σ70.快艇σ一个有一个更短的σ一个NCR比大肠杆菌σ70,不过是残在短快艇与RbpA接触的NCR也不存在Cdiff或大肠杆菌20..快艇RbpA接触Fdx,而Cdiffσ一个与Fdx的联系要多于快艇σ一个.黑色箭头表示RpbA- σ一个接触,而彩色箭头表示Fdx接触σ一个和RpbA,其中包括之间的一个共享接触快艇而且Cdiffσ一个(红色箭头)。b, σ的氨基酸序列比对一个细菌种类的不同代表。相同的残基用黄色突出显示。缺口由虚线表示。保守的σ区域标记在对齐的下方。彩色方框表示Fdx的联系人:蓝色,唯一的Cdiff;红色,由Cdiff而且快艇.三个字母的物种代码如下:Clostridioides固执的;Bsu,枯草芽孢杆菌;包子,拟杆菌均匀化;生态,大肠杆菌;快艇,结核分枝杆菌.
扩展数据图6 Fdx结合残基快艇RbpA-Eσ一个而且CdiffEσ一个.
一个, Ligplot57用来确定Fdx和快艇RbpA-Eσ一个(左)和CdiffEσ一个(右)。青色球体,H2O;绿色虚线,氢键或盐桥;红弧,范德华相互作用;红色虚线表示阳离子-π相互作用。注意在的ligplot中CdiffEσ一个V1143(在文中被讨论为突变导致Fdx抗性的残基之一)没有达到距离截止值(4.5 Å),因为它距离Fdx 4.7 Å。RNAP β, β'和σ一个残留物分别为青色、粉色和橙色。这两个快艇与Fdx相互作用的RbpA残基(E17, R10)用紫色表示,并在文本中标出。fdx相互作用残基之间没有相应的相互作用Cdiff而且快艇用红圈突出显示。b,残基与Fdx相互作用的低温电镜密度图。残基的着色与图中RNAP亚基的着色一致。3.,棒状模型和低温电磁密度用颜色编码如下:粉红色:β亚基,青色:β′亚基,橙色:σ一个.水分子显示为红色球体。与Fdx形成氢键的残基(黑色虚线)被标记。
扩展数据图7在体外用于测定Fdx IC50的流产转录试验Cdiff而且快艇Eσ一个而且生态Eσ70与无花果有关。二维.
流产32P-RNA产物(GpUpG)Cdiff rrnC启动子在增加Fdx浓度的存在下定量。对于每个Eσ一个(或Eσ70),在同一凝胶上进行了三个独立的实验并进行了分析。
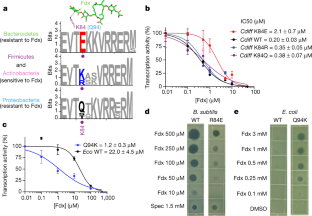
图8耐Fdx菌与敏感Fdx菌之间与Fdx相互作用的rnap关键结构成分的比较序列比对。
Fdx相互作用区域被标记在序列比对的顶部。两者中接触Fdx的残留物位置Cdiff而且快艇由序列下方的三角形标记。对于对Fdx敏感的革兰氏阳性细菌,在Cdiffβ ' K84是K或R,在粉红色背景中突出显示。对于对Fdx耐药的革兰氏阴性菌,β ' K84位点的残留物为中性Q、L或阴性E,即蓝色背景中突出显示的部分。保守残馀以红色背景上的白色字母表示,相似残馀则以蓝色框中的红色字母表示。它强调,Clostridioides固执的;快艇,结核分枝杆菌;Bsu,枯草芽孢杆菌;公元前,蜡样芽胞杆菌、Sab、金黄色葡萄球菌;Lcb,干酪乳杆菌;体,Peptococcus麦格纳;成立分公司,肠球菌都有效;生态,大肠杆菌;Hpy,幽门螺杆菌;Pae,铜绿假单胞菌;包子,拟杆菌均匀化;美国银行,拟杆菌ovatus;Pdi,Parabacteroides distasonis;Stm,沙门氏菌Choleraesuis;Nmc,脑膜炎奈瑟氏菌.
扩展数据图10 WT和突变体Fdx的抑制Cdiff而且生态RNAPs。
一个,转录测定Cdiffβ ' K94E和β ' K84Q Eσ一个s与Fig有关。4 b.的Cdiff rrnC启动子(无花果。2摄氏度)被用作模板。b,转录测定生态WT和β ' Q94K Eσ70它与无花果有关。4摄氏度.相同的Cdiff rrnC使用了促进剂。对于每个RNAP,在同一凝胶上进行了三个独立的实验并进行了分析。c,在活的有机体内琼脂板测定大肠杆菌WT和Q94K突变株。用编码号为pRL662的对照质粒对温度敏感菌株RL602进行转化rpoC、WTrpoC和变异rpoC -Q94K。菌株在40°C下生长一夜。含有表达质粒的细菌rpoCWT和Q94K生长良好,而空质粒不支持细胞生长。d.抗生素抑制试验使用大肠杆菌rpoC将3 μ L DMSO (Fdx, SPR741和利福平(Rif))或水(卡那霉素(Kan))中的抗生素移液到含有细菌的覆盖软琼脂上(见方法).SPR741没有抑制细胞生长,但增加了Rif和Fdx的效力,这表明它增加了抗生素向细胞的扩散。Rif是一种靶向与Fdx(±SPR741)不同的RNAP区域的抗生素,Kan是一种靶向核糖体且不受SPR741影响的抗生素,它们同样抑制了WT和突变Q94K菌株。相比之下,Fdx仅有效抑制突变菌株,这表明Q94E突变对Fdx具有特异性敏感性。
源数据
权利和权限
关于本文
引用本文
曹秀文,波雅契,H.,陈俊杰。et al。非达霉素窄谱活性的基础Clostridioides固执的.自然604, 541-545(2022)。https://doi.org/10.1038/s41586-022-04545-z
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586-022-04545-z
这篇文章被引用
从信号转导到蛋白质毒素——艰难梭菌毒素研究路线里程碑的叙事回顾
Naunyn-Schmiedeberg的药理学档案(2023)
