摘要gydF4y2Ba
只有一小部分癌症患者对基于免疫检查点封锁(ICB)的单一疗法表现出持久的反应。rna编辑酶ADAR1是对ICB治疗的耐药性的一个新兴决定因素,并通过抑制免疫原性双链rna (dsRNAs)来防止ICB的反应,例如那些由内源性逆转录病毒元件(EREs)表达失调引起的rna。gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.这些dsRNA通过激活a型dsRNA (A-RNA)传感蛋白如MDA-5和PKR来触发干扰素依赖的抗肿瘤反应gydF4y2Ba5克ydF4y2Ba.在这里,我们发现ADAR1还可以阻止内源性z型dsRNA元件(z - rna)的产生,这些z - rna富集在干扰素刺激的mrna的3′非翻译区。ADAR1的缺失或突变导致Z-RNA的积累和Z-RNA传感器ZBP1的激活,最终导致ripk3介导的坏死。由于目前没有临床可行的ADAR1抑制剂,我们寻找一种可以覆盖ADAR1抑制需求并直接激活ZBP1的化合物。我们发现了一个小分子,curaxin CBL0137,它通过触发细胞中Z-DNA的形成来有效地激活ZBP1。CBL0137在癌症相关成纤维细胞中诱导zbp1依赖性坏死,并逆转了黑色素瘤小鼠模型中的ICB无反应性。总的来说,这些结果表明ADAR1抑制内源性z - rna,并确定zbp1介导的坏死是ADAR1掩盖的肿瘤免疫原性的新决定因素。zbp1诱导的坏死的治疗激活为重新点燃icb耐药人类癌症的免疫反应提供了一个容易翻译的途径。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
相关的文章gydF4y2Ba
引用本文的开放获取文章。gydF4y2Ba
针对细胞死亡途径的癌症治疗:坏死、焦亡、铁亡和铜腐病研究的最新进展gydF4y2Ba
血液学与肿瘤学杂志gydF4y2Ba开放获取gydF4y2Ba2022年12月8日gydF4y2Ba
访问选项gydF4y2Ba
订阅《自然》+gydF4y2Ba
立即在线访问《自然》和其他55种《自然》杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅期刊gydF4y2Ba
获得1年的完整期刊访问权限gydF4y2Ba
199.00美元gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
增值税稍后将在结帐时添加。gydF4y2Ba
税务计算将在结账时完成。gydF4y2Ba
买条gydF4y2Ba
在ReadCube上获得时间限制或全文访问。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
数据可用性gydF4y2Ba
RNA-seq, ChIP-seq和RIP-seq实验数据可从基因表达Omnibus数据库(gydF4y2Bahttps://www.ncbi.nlm.nih.gov/geogydF4y2Ba)gydF4y2BaGSE184966gydF4y2Ba.DeepZ特征分析中使用的基因组注释来自ChIP-Atlas公共资源(gydF4y2Bahttps://chip-atlas.orggydF4y2Ba).gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
详细的命令和脚本可以在GitHub上在线获得:gydF4y2Bahttps://github.com/alnfedorov/MEF-CBL0137gydF4y2Ba.DeepZ模型的数据预处理管道可以在gydF4y2Bahttps://github.com/Nazar1997/DeepZ_data_creationgydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
刘,H.等。肿瘤来源的IFN触发慢性通路激动和对ADAR丢失的敏感性。gydF4y2BaNat,地中海。gydF4y2Ba25gydF4y2Ba, 95-102(2019)。gydF4y2Ba
伊什祖卡,J. J.等。肿瘤中ADAR1的缺失克服了对免疫检查点封锁的抵抗。gydF4y2Ba自然gydF4y2Ba565gydF4y2Ba, 43-48(2019)。gydF4y2Ba
Mehdipour, P.等人。表观遗传治疗诱导逆转录sin和ADAR1依赖。gydF4y2Ba自然gydF4y2Ba588gydF4y2Ba, 169-173(2020)。gydF4y2Ba
甘农,H. S.等人。肿瘤细胞亚群中ADAR1腺苷脱氨酶依赖性的鉴定。gydF4y2BaCommun Nat。gydF4y2Ba9gydF4y2Ba, 5450(2018)。gydF4y2Ba
陈,R., Ishak, C. A. & De Carvalho, D. D.内源性逆转录因子和癌症治疗和细胞稳态中的病毒模拟反应。gydF4y2Ba癌症。gydF4y2Ba11gydF4y2Ba, 2707-2725(2021)。gydF4y2Ba
Loo Yau, H., Ettayebi, I. & De Carvalho, D.癌症表观基因组:利用其弱点进行免疫治疗。gydF4y2Ba细胞生物学趋势。gydF4y2Ba29gydF4y2Ba, 31-43(2019)。gydF4y2Ba
herord - farlow, j.e, Chalk, a.m.和Walkley, c.r.通过血液学定义腺苷-肌苷RNA编辑的功能。gydF4y2Ba咕咕叫。当今。内科杂志。gydF4y2Ba26gydF4y2Ba, 241-248(2019)。gydF4y2Ba
Eisenberg, E. & Levanon, E. Y. A-to-I RNA编辑-免疫保护和转录组多样化。gydF4y2BaNat. Rev. Genet。gydF4y2Ba19gydF4y2Ba, 473-490(2018)。gydF4y2Ba
Samuel, C. E.腺苷脱氨酶作用于RNA (ADAR1),双链RNA触发先天免疫反应的抑制因子。gydF4y2Ba生物。化学。gydF4y2Ba294gydF4y2Ba, 1710-1720(2019)。gydF4y2Ba
ALU非b - dna构象,翻转,二进制代码和进化。gydF4y2Bar . Soc。开放的科学。gydF4y2Ba7gydF4y2Ba, 200222(2020)。gydF4y2Ba
钟,H.等。人类ADAR1阻止内源性RNA触发翻译关闭。gydF4y2Ba细胞gydF4y2Ba172gydF4y2Ba, 811 - 824。e14(2018).
George, C. X., Ramaswami, G., Li, J. B.和Samuel, C. E.用腺苷脱氨酶ADAR1编辑细胞自我rna抑制先天免疫应激反应。gydF4y2Ba生物。化学。gydF4y2Ba291gydF4y2Ba, 6158-6168(2016)。gydF4y2Ba
Liddicoat, B. J.等。ADAR1编辑RNA可阻止MDA5感知内源性dsRNA为非己。gydF4y2Ba科学gydF4y2Ba349gydF4y2Ba, 1115-1120(2015)。gydF4y2Ba
赫伯特,A.等。Z-DNA结合域存在于人类编辑酶,双链RNA腺苷脱氨酶。gydF4y2Ba国家科学院学报美国gydF4y2Ba94gydF4y2Ba, 8421-8426(1997)。gydF4y2Ba
由双链RNA编辑酶ADAR的Zα域影响Z-DNA和Z-RNA识别的变异引起的孟德尔病。gydF4y2Ba欧元。j .的嗡嗡声。麝猫。gydF4y2Ba28gydF4y2Ba, 114-117(2020)。gydF4y2Ba
张,T.等。流感病毒z - rna诱导zbp1介导的坏死。gydF4y2Ba细胞gydF4y2Ba180gydF4y2Ba, 1115 - 1129。e13(2020)。gydF4y2Ba
哈丁,C. C.等。化学溴化稳定Z-RNA及其被抗z - dna抗体识别。gydF4y2Ba生物化学gydF4y2Ba26gydF4y2Ba, 5191-5199(1987)。gydF4y2Ba
Schade, M., Turner, C. J., Lowenhaupt, K., Rich, A. & Herbert, A. dsRNA腺苷脱氨酶I型的z - dna结合域Zα的结构-功能分析揭示了与(α + β)螺旋-转弯-螺旋蛋白家族的相似性。gydF4y2BaEMBO J。gydF4y2Ba18gydF4y2Ba, 470-479(1999)。gydF4y2Ba
巴扎克,L., Levanon, E. Y. & Eisenberg, E. Alu可编辑性的全基因组分析。gydF4y2Ba核酸测定。gydF4y2Ba42gydF4y2Ba, 6876-6884(2014)。gydF4y2Ba
尼科尔斯,P. J.等。ADAR1的Z-RNA结合域识别Alu和核糖体rna中的非cpg重复序列可诱导A-Z连接。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 793(2021)。gydF4y2Ba
鲍海林,徐勇。2 ' -gydF4y2BaOgydF4y2Ba-甲基-8-甲基鸟苷作为Z-RNA结构和功能研究的稳定剂。gydF4y2Ba分子gydF4y2Ba23gydF4y2Ba, 2572-2579(2018)。gydF4y2Ba
Brown, B. A. 2, Lowenhaupt, K., Wilbert, C. M., Hanlon, E. B.和Rich, A.编辑酶dsRNA腺苷脱氨酶的Zα结构域结合左旋Z-RNA和Z-DNA。gydF4y2Ba国家科学院学报美国gydF4y2Ba97gydF4y2Ba, 13532-13536(2000)。gydF4y2Ba
金,K.等。人类dna依赖性ifn调控因子激活剂Zβ域的溶液结构及其与B-和z - dna的结合方式gydF4y2Ba国家科学院学报美国gydF4y2Ba108gydF4y2Ba, 6921-6926(2011)。gydF4y2Ba
Peck, L. J., Nordheim, A., Rich, A. & Wang, J. C.克隆的d(pCpG)n.d(pCpG)n DNA序列通过盐、Co(III)或负超线圈从右向左螺旋结构翻转。gydF4y2Ba国家科学院学报美国gydF4y2Ba79gydF4y2Ba, 4560-4564(1982)。gydF4y2Ba
张,H. W.等。组蛋白伴侣FACT和curaxins:对基因组结构和功能的影响。gydF4y2BaJ.癌症转移治疗。gydF4y2Ba5克ydF4y2Ba, 78(2019)。gydF4y2Ba
萨芬娜,A.等。FACT是真核细胞中DNA扭转应力的传感器。gydF4y2Ba核酸测定。gydF4y2Ba45gydF4y2Ba, 1925-1945(2017)。gydF4y2Ba
Sookdeo, A., Hepp, C. M., McClure, M. A. & Boissinot, S.在基因组时代重访小鼠LINE-1的进化。gydF4y2Ba暴徒。DNAgydF4y2Ba4gydF4y2Ba, 3(2013)。gydF4y2Ba
Beknazarov, N., Jin, S. & Poptsova, M.使用组学数据预测功能Z-DNA区域的深度学习方法。gydF4y2BaSci代表。gydF4y2Ba10gydF4y2Ba, 19134(2020)。gydF4y2Ba
鲍海林,徐勇。合成含8-三氟甲基-2-脱氧鸟苷的寡核苷酸DNA观察Z-DNA结构。gydF4y2Ba咕咕叫。Protoc。gydF4y2Ba1gydF4y2Ba, e28(2021)。gydF4y2Ba
Jeronimo, C., Watanabe, S., Kaplan, C. D., Peterson, C. L. & Robert, F.组蛋白伴侣FACT和Spt6限制H2A。Z来自基因内位置。gydF4y2Ba摩尔。细胞gydF4y2Ba58gydF4y2Ba, 1113-1123(2015)。gydF4y2Ba
Denli, a.m.等。灵长类动物特异性ORF0有助于逆转录转座子介导的多样性。gydF4y2Ba细胞gydF4y2Ba163gydF4y2Ba, 583-593(2015)。gydF4y2Ba
萨默斯,K.等。curaxin CBL0137对mll重排白血病的抗白血病活性。gydF4y2BaInt。j .癌症gydF4y2Ba146gydF4y2Ba, 1902-1916(2020)。gydF4y2Ba
加斯帕里安,A. V.等。Curaxins:通过靶向FACT同时抑制NF-κB并激活p53的抗癌化合物。gydF4y2Ba科学。Transl。地中海。gydF4y2Ba3.gydF4y2Ba, 95ra74(2011)。gydF4y2Ba
卡特等人。通过抑制组蛋白伴侣FACT在成神经细胞瘤中靶向MYC信号的治疗。gydF4y2Ba科学。Transl。地中海。gydF4y2Ba7gydF4y2Ba, 312ra176(2015)。gydF4y2Ba
古,G. B.等。肿瘤中甲基化依赖的RIP3表达缺失抑制了化疗对程序性坏死的反应。gydF4y2Ba细胞Res。gydF4y2Ba25gydF4y2Ba, 707-725(2015)。gydF4y2Ba
Gabrilovich, D. I. & Nagaraj, S.骨髓源性抑制细胞作为免疫系统的调节器。gydF4y2BaNat. Rev. Immunol。gydF4y2Ba9gydF4y2Ba, 162-174(2009)。gydF4y2Ba
Weiss, s.a., Wolchok, J. D. & Sznol, M.黑色素瘤的免疫治疗:事实和希望。gydF4y2Ba中国。癌症Res。gydF4y2Ba25gydF4y2Ba, 5191-5201(2019)。gydF4y2Ba
王,J.等。在YUMMER1.7小鼠黑色素瘤模型中,紫外线诱导的体细胞突变引发功能性T细胞反应。gydF4y2Ba色素细胞黑色素瘤Res。gydF4y2Ba30.gydF4y2Ba, 428-435(2017)。gydF4y2Ba
Nirschl, C. J.等。ifn γ依赖的组织免疫稳态在肿瘤微环境中被增选。gydF4y2Ba细胞gydF4y2Ba170gydF4y2Ba, 127 - 141。e15(2017).
Benci, J. L.等。肿瘤干扰素信号调节免疫检查点封锁的多基因抵抗程序。gydF4y2Ba细胞gydF4y2Ba167gydF4y2Ba, 1540 - 1554。e12汽油(2016)。gydF4y2Ba
石井,K. J.等。TANK-binding kinase-1描述了对DNA疫苗的先天和适应性免疫反应。gydF4y2Ba自然gydF4y2Ba451gydF4y2Ba, 725-729(2008)。gydF4y2Ba
Chen, P.等。抗cd70免疫细胞因子用于开发干扰素-γ诱导的肾细胞癌rip1依赖性坏死。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba8gydF4y2Ba, e61446(2013)。gydF4y2Ba
癌症基因组图谱网络。皮肤黑色素瘤的基因组分类。gydF4y2Ba细胞gydF4y2Ba161gydF4y2Ba, 1681-1696(2015)。gydF4y2Ba
贝克特,E.等。利用基因表达估计组织浸润免疫细胞和基质细胞群体的丰度。gydF4y2Ba基因组医学杂志。gydF4y2Ba17gydF4y2Ba, 218(2016)。gydF4y2Ba
Gabitova-Cornell, L.等人。胆固醇通路抑制诱导TGF-β信号通路促进胰腺癌基础分化gydF4y2Ba癌症细胞gydF4y2Ba38gydF4y2Ba, 567 - 583。e11(2020).
Satija, R., Farrell, J. A., Gennert, D., Schier, A. F. & Regev .单细胞基因表达数据的空间重建。gydF4y2Ba生物科技Nat。》。gydF4y2Ba33gydF4y2Ba, 495-502(2015)。gydF4y2Ba
Patro, R., Duggal, G., Love, M. I., Irizarry, R. A. & Kingsford, C. Salmon提供了快速和有偏差意识的转录表达量化。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 417-419(2017)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
巴楚等人。表位标记组蛋白H3.3的多功能小鼠模型研究表观基因组动力学。gydF4y2Ba生物。化学。gydF4y2Ba294gydF4y2Ba, 1904-1914(2019)。gydF4y2Ba
Roth, S. H., Levanon, E. Y. & Eisenberg, E. ADAR腺苷-肌苷RNA编辑活性的全基因组量化。gydF4y2BaNat方法。gydF4y2Ba16gydF4y2Ba, 1131-1138(2019)。gydF4y2Ba
格鲁伯,A. R.,洛伦兹,R.,伯恩哈特,S. H.,纽博克,R. &霍法克,I. L.维也纳RNA网络套件。gydF4y2Ba核酸测定。gydF4y2Ba36gydF4y2Ba, w70-w74(2008)。gydF4y2Ba
Lo Giudice, C., Tangaro, m.a., Pesole, G. & Picardi, E.使用REDItools和REDIportal研究深层转录组数据集中的RNA编辑。gydF4y2BaProtoc Nat。gydF4y2Ba15gydF4y2Ba, 1098-1131(2020)。gydF4y2Ba
Anders, S, Pyl, P. T. & Huber, W. htseq -一个用于处理高通量测序数据的Python框架。gydF4y2Ba生物信息学gydF4y2Ba31gydF4y2Ba, 166-169(2015)。gydF4y2Ba
Kechin, A., Boyarskikh, U., Kel, A. & Filipenko, M. cutPrimers:一种用于从靶向下一代测序reads中精确切割引物的新工具。gydF4y2Baj .第一版。医学杂志。gydF4y2Ba24gydF4y2Ba, 1138-1143(2017)。gydF4y2Ba
Minimap2:核苷酸序列的成对比对。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, 3094-3100(2018)。gydF4y2Ba
李,H.等。序列对齐/映射格式和SAMtools。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 2078-2079(2009)。gydF4y2Ba
张杨,等。基于模型的ChIP-Seq (MACS)分析。gydF4y2Ba基因组医学杂志。gydF4y2Ba9gydF4y2Ba, r137(2008)。gydF4y2Ba
朱丽娟等。ChIPpeakAnno:用于注释ChIP-seq和ChIP-chip数据的Bioconductor包。gydF4y2BaBMC生物信息学gydF4y2Ba11gydF4y2Ba, 237(2010)。gydF4y2Ba
Gu, Z., Gu, L., Eils, R., Schlesner, M. & Brors, B.在R.中实现并增强了圆形可视化。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 2811-2812(2014)。gydF4y2Ba
彭茨科弗,T.等人。L1Base 2:更多逆转录转座活性LINE-1s,更多哺乳动物基因组。gydF4y2Ba核酸测定。gydF4y2Ba45gydF4y2Ba, d68-d73(2017)。gydF4y2Ba
西弗斯,F.等人。使用Clustal Omega快速,可扩展生成高质量的蛋白质多序列比对。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba7gydF4y2Ba, 539(2011)。gydF4y2Ba
Ho, P. S., Ellison, M. J., Quigley, G. J. & Rich, A.预测Z-DNA在自然序列中形成的计算机辅助热力学方法。gydF4y2BaEMBO J。gydF4y2Ba5克ydF4y2Ba, 2737-2744(1986)。gydF4y2Ba
Oki, S.等。ChIP-Atlas:一个完全集成公共ChIP-seq数据的数据挖掘套件。gydF4y2BaEMBO代表。gydF4y2Ba19gydF4y2Ba, e46255(2018)。gydF4y2Ba
新型肺I型上皮细胞模型对流感病毒感染的先天反应特征。gydF4y2Ba维罗尔将军。gydF4y2Ba95gydF4y2Ba, 350-362(2014)。gydF4y2Ba
布朗D. M. Fisher T. L. Wei C. Frelinger J. G. & Lord E. M.肿瘤可以作为体液免疫的佐剂。gydF4y2Ba免疫学gydF4y2Ba102gydF4y2Ba, 486-497(2001)。gydF4y2Ba
在坏死过程中ripk3介导的MLKL激活环磷酸化的特征。gydF4y2Ba细胞死亡不同。gydF4y2Ba23gydF4y2Ba, 76-88(2016)。gydF4y2Ba
确认gydF4y2Ba
我们感谢E. Gurova提供的CBL0137类似物;M. Bosenberg, J. Upton, A. Degterev和D.R. Green提供细胞株或抗体;M. Andrake在分子建模方面的帮助;A. Gupte和Z. Liang提供技术援助。这项工作得到了David Wiest和巴克斯县联合委员会希望种子基金会捐赠给s.b.的支持,NIH向s.b.捐赠了CA168621、CA190542、AI135025和AI144400, NIH向V.S. C.W.捐赠了GM119398,由澳大利亚国家卫生和医学研究委员会(NHMRC;APP1144049和APP1183553)。J.C.C.和P.G.T.得到了马克基金会、美国国立卫生研究院拨款U01AI150747和圣裘德儿童医院美国黎巴嫩叙利亚联合慈善机构的支持。NIH癌症中心支持基金P30CA006927向s.b.a.f.、N.B.和M.P.提供了额外的资金,由国立研究型大学高等经济学院的基础研究计划提供支持,A.H.是该计划的国际主管。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
T.Z.和C.Y.进行了大部分的细胞培养和体内实验,A.G.和r.m.w.a.f.协助进行了大部分的生物信息学分析,n.b.、L.Q、J.C.C、s.p.、P.G.T.和C.W. h.b.、S.W.和Y.X.协助进行了CD和NMR研究。C.W.提供了稳定表达ADAR1 p150及其突变体的慢病毒结构。C.W.也提供了不朽的gydF4y2Ba阿达尔月gydF4y2Ba- / -gydF4y2Bamef、主gydF4y2Ba阿达尔月gydF4y2Ba- / -gydF4y2BaIfihgydF4y2Ba- / -gydF4y2Bamef及其控制。A.A.B.提供B16-OVA细胞。A.A.B.和P.G.T.辅助荷瘤小鼠的免疫细胞分析。M.P, v.s., A.H.和S.B.设计实验,监督研究并分析数据。T.Z, C.Y, A.H.和s.b写了手稿。所有作者都参与了手稿的编辑工作。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
T.Z、C.Y.和S.B.是Fox Chase癌症中心提交的“curaxins和免疫检查点抑制剂联合治疗癌症”临时专利申请的共同发明人。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢William Haining, Jan Rehwinkel和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
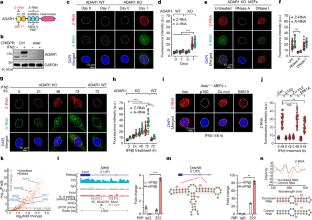
扩展数据图1 Z-RNA在gydF4y2Ba阿达尔月gydF4y2Ba消融。gydF4y2Ba
一个gydF4y2BaADAR1 WT、ADAR1 KO、ADAR1/MAVS双KO mef、抗ifnar1中和抗体(20µg/mL)处理的ADAR1 KO mef中Z-RNA和A-RNA形成的时间过程。gydF4y2BabgydF4y2BaZ-RNA和A-RNA信号荧光强度的定量gydF4y2Ba一个gydF4y2Ba.gydF4y2BacgydF4y2Ba,在基于crispr的小鼠后,WT mef中的MAVS蛋白水平gydF4y2Ba小牛gydF4y2Ba(MAVS KO)和IFNβ暴露(100 ng/mL, 24 h)。gydF4y2Ba小牛gydF4y2Ba用表达“all-in-one”sgRNA的慢病毒消融。gydF4y2BadgydF4y2Ba用IFNβ处理ADAR1 WT或ADAR1 KO的全细胞提取物6 h,用免疫印迹法检测磷酸化STAT1、总STAT1和ADAR1。gydF4y2BaegydF4y2Ba,gydF4y2BaIsg15gydF4y2Ba通过RT-qPCR检测加或不加IFNβ处理6 h的ADAR1 WT或ADAR1 KO的mRNA水平。gydF4y2BafgydF4y2Ba,原发Z-RNA和A-RNA积累的检测gydF4y2Ba阿达尔月gydF4y2Ba+/+gydF4y2BaIfih1gydF4y2Ba−−/gydF4y2Ba或以gydF4y2Ba−−/gydF4y2BaIfih1gydF4y2Ba−−/gydF4y2Ba存在或不存在IFNβ的MEFs (100 ng/mL)。gydF4y2BaggydF4y2BaZ-RNA和A-RNA信号荧光强度的定量gydF4y2BafgydF4y2Ba.gydF4y2BahgydF4y2Ba,通过基于crispr的ADAR1消融方法,在存在或不存在IFNβ (100 ng/mL)的情况下,检测永生化BMDMs中Z-RNA和A-RNA的积累。gydF4y2Ba我gydF4y2Ba,免疫印迹法检测永生化BMDMs中ADAR1 p150蛋白水平(上图)。Z-RNA和A-RNA信号荧光强度的定量gydF4y2BahgydF4y2Ba(底板)。gydF4y2BajgydF4y2Ba,在存在或不存在IFNβ的情况下(100 ng/mL),通过基于crispr的方法进行ADAR1消融后,检测气道上皮来源的永活LET1细胞中Z-RNA和A-RNA的积累。gydF4y2BakgydF4y2Ba,免疫印迹法检测LET1细胞中ADAR1 p150蛋白水平(上图)。Z-RNA和A-RNA信号荧光强度的定量gydF4y2BajgydF4y2Ba(底板)。gydF4y2BalgydF4y2BaIFNβ存在或不存在时ADAR1 WT或KO永生内皮SVEC4-10细胞Z-RNA和A-RNA积累的检测(100 ng/mL)。gydF4y2Ba米gydF4y2Ba免疫印迹法检测SVEC4-10细胞中ADAR1 p150蛋白水平(上图)。Z-RNA和A-RNA信号荧光强度的定量gydF4y2BalgydF4y2Ba(底板)。gydF4y2BangydF4y2Ba,本实验中使用的flag标记ADAR1 p150, E861A和Zα (N175A/Y179A)突变体的示意图。gydF4y2BaogydF4y2Ba,稳定重建中ADAR1结构的等效表达水平gydF4y2Ba阿达尔月gydF4y2Ba−−/gydF4y2Bamef经免疫印迹证实。gydF4y2BapgydF4y2Ba, Z-RNA在永生化中积累gydF4y2Ba阿达尔月gydF4y2Ba−−/gydF4y2Ba用空载体(Vec)或ADAR1 p150、Zα N175A/Y179A (Zα mut)或未经过IFNβ处理的编辑缺陷(E861A)突变体稳定重组mef。gydF4y2Ba问gydF4y2Ba, IFNβ (100 ng/mL)处理ADAR1 p150或E861A突变细胞后Z-RNA积累的检测。gydF4y2BargydF4y2BaZ-RNA信号荧光强度的定量gydF4y2Ba问gydF4y2Ba.gydF4y2Ba年代gydF4y2Ba,蛋白酶处理后ADAR1 p150或E861A突变细胞Z-RNA积累的检测。固定细胞染色前用蛋白酶K (0.008 U/ml)处理30 min。gydF4y2BatgydF4y2BaZ-RNA信号荧光强度的定量gydF4y2Ba年代gydF4y2Ba.数据为均数±标准差。(n = 30英寸gydF4y2BabgydF4y2Ba,gydF4y2BaggydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba,gydF4y2Ba米gydF4y2Ba,gydF4y2BargydF4y2Ba,gydF4y2BatgydF4y2Ba或者说n = 3英寸gydF4y2BaegydF4y2Ba每组)。单因素方差分析检验(gydF4y2BabgydF4y2Ba,gydF4y2BaggydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba,gydF4y2Ba米gydF4y2Ba)、带韦尔奇修正的双尾未配对t检验(gydF4y2BaegydF4y2Ba,gydF4y2BargydF4y2Ba)或双尾未配对学生t检验(gydF4y2BatgydF4y2Ba).***gydF4y2BaPgydF4y2Ba< 0.0005 (gydF4y2BaPgydF4y2Ba< 0.0001 ingydF4y2BabgydF4y2Ba,gydF4y2BaggydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba,gydF4y2Ba米gydF4y2Ba,gydF4y2BargydF4y2Ba).NS,没有意义。数据至少代表三个独立的实验。gydF4y2Ba阿达尔月gydF4y2Ba是通过基于crispr的方法消融的,如手稿中详细描述的那样。gydF4y2Ba
扩展数据图2受ADAR1抑制的z形mrna。gydF4y2Ba
一个gydF4y2BaADAR1 KO基因Z22 RIP-seq测序RNA片段的来源gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba含IFNβ的MEFs (100 ng/mL)。gydF4y2BabgydF4y2Ba, A→I来自ADAR1 WT和ADAR1的Z22下拉rna的编辑索引gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2BaIFNβ处理前后KO细胞(100 ng/mL)。gydF4y2BacgydF4y2Ba, IFNβ存在或不存在时ADAR1 WT或ADAR1 KO mef中ADAR2的表达水平(100 ng/mL, 24 h)。gydF4y2BadgydF4y2Ba, Z22中富集了ADAR1 KO的ISG mrna的重复序列分布gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Bamef。在ifn β处理的ADAR1 WT细胞Z22下拉中,定位和编辑的重复总数用红色表示(A→I编辑指数> 0.1%;每腺苷平均覆盖度> 5)。gydF4y2BaegydF4y2Ba含sin倒置mrna的3 ' utr显示Z22富集(蓝色峰)、IgG(灰色峰)、输入(橙色峰)、编辑位点(垂直红色条)、SINEs位置和qPCR引物位置(红色箭头)。qPCR定量所指示的含sine的倒置mrna (gydF4y2BaXrn1gydF4y2Ba而且gydF4y2BaKnl1gydF4y2Ba)右侧为Z22或对照IgG抗体免疫沉淀后的ADAR1 mef (100 ng/mL, 48 h)。gydF4y2BafgydF4y2Ba, qPCR分析指示gu型含mrna的简单重复序列(gydF4y2BaEif2ak2gydF4y2Ba而且gydF4y2BaIfih1gydF4y2Ba)后用来自ADAR1 KO mef的Z22抗体或对照IgG抗体免疫沉淀。潜在的二部(哑铃)z -形成RNA结构(RNA-折叠)显示在每张图的右侧,推定的z - α结合位点在粉色框中勾画。数据为均数±标准差。(n = 3英寸gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba每组)。带韦尔奇修正的双尾未配对t检验(gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba).*gydF4y2BaPgydF4y2Ba< 0.05 [gydF4y2BaPgydF4y2Ba= 0.005英寸gydF4y2BaegydF4y2Ba(gydF4y2BaXrn1gydF4y2Ba), * *gydF4y2BaPgydF4y2Ba< 0.005 [gydF4y2BaPgydF4y2Ba= 0.003英寸gydF4y2BaegydF4y2Ba(gydF4y2BaKnl1gydF4y2Ba),gydF4y2BaPgydF4y2Ba= 0.001英寸gydF4y2BafgydF4y2Ba(gydF4y2BaEif2ak2gydF4y2Ba),gydF4y2BaPgydF4y2Ba= 0.004英寸gydF4y2BafgydF4y2Ba(gydF4y2BaIfih1gydF4y2Ba)]。数据至少代表三个独立的实验。gydF4y2Ba
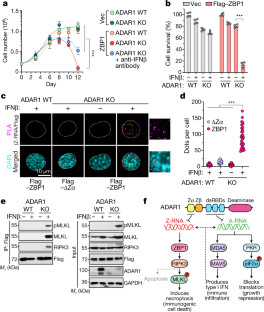
扩展数据图3 zbp1激活后的细胞死亡gydF4y2Ba阿达尔月gydF4y2Ba消融。gydF4y2Ba
一个gydF4y2Ba, IFNβ处理后ADAR1 WT或ADAR1 KO的显微照片(100 ng/mL, 48 h)。gydF4y2BabgydF4y2Ba,gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba稳定表达FLAG-ZBP1的mef,其中ADAR1存在(ADAR1 WT)或被CRISPR/Cas9消融(ADAR1 KO),用相应的细胞因子(100 ng/mL)处理。在处理后48 h测定细胞活力。(gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba),所产生的MEFs为gydF4y2BabgydF4y2Ba用IFNβ (gydF4y2BacgydF4y2Ba)或IFNγ (100 ng/mL) (gydF4y2BadgydF4y2Ba)在存在或不存在zVAD (50 mM)和RIPK3抑制剂(R3i) GSK’843(5µM)的情况下。治疗48 h后检测细胞活力。gydF4y2BaegydF4y2Ba,gydF4y2Ba阿达尔月gydF4y2Ba被基于crispr的方法消融gydF4y2BaMlklgydF4y2Ba−−/gydF4y2Ba或gydF4y2BaMlklgydF4y2Ba−−/gydF4y2BaCasp8gydF4y2Ba−−/gydF4y2Bamef。在有或没有zVAD (50 mM)的情况下,对细胞进行IFNβ (100 ng/mL)处理。治疗48 h后检测细胞活力。gydF4y2BafgydF4y2Ba在处理后24 h裂解IFNβ (100 ng/mL)处理的ADAR1 WT和ADAR1 KO mef,免疫印迹法检测RIPK1、FADD、Caspase 8 (Casp8)和ADAR1。gydF4y2BaggydF4y2Baflag标记小鼠ZBP1及其突变体示意图。gydF4y2BahgydF4y2Ba,反转录病毒重组中FLAG-ZBP1结构的等效表达水平gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba通过FLAG标记免疫印迹法确认mef。gydF4y2Ba我gydF4y2Ba,细胞活力gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba在IFNβ (100 ng/mL)处理48 h后,确定稳定表达FLAG-ZBP1或其突变体的MEFs。gydF4y2BajgydF4y2Ba,它gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba用FLAG-ZBP1或FLAG-ZBP1ΔZα突变体稳定重建的mef,通过基于crispr的方法消融ADAR1表达,用IFNβ处理(100 ng/mL, 48 h)。细胞裂解后,检测抗FLAG免疫沉淀是否存在FLAG。全细胞提取物(5%输入)平行检测FLAG。采用GAPDH作为加载对照。gydF4y2BakgydF4y2Ba, ZgydF4y2Babp1gydF4y2Ba−−/gydF4y2Ba稳定表达FLAG-ZBP1或FLAG-ZBP1ΔZα突变体的mef经IFNβ处理(100 ng/mL)后,ADAR1表达消失。使用引物RT-qPCR检测抗flag或对照IgG免疫沉淀中存在的RNAgydF4y2BaEif2ak2, Ddx58或Ifih1gydF4y2Ba.gydF4y2BalgydF4y2Ba, ZgydF4y2Babp1gydF4y2Ba−−/gydF4y2Ba用FLAG-ZBP1或FLAG-ZBP1ΔZα重组的mef,用IFNβ处理或不处理(100 ng/mL),消融ADAR1表达。从细胞裂解物中提取FLAG或对照IgG免疫沉淀,使用引物进行RT-qPCR检测3'UTRsgydF4y2BaXrn1, Knl1gydF4y2Ba或gydF4y2BaSlfn5gydF4y2Ba.gydF4y2Ba米gydF4y2Ba, Z区pMLKL(绿色)免疫荧光染色gydF4y2Babp1gydF4y2Ba−−/gydF4y2Ba稳定表达FLAG-ZBP1的MEFsgydF4y2Ba阿达尔月gydF4y2Ba(ADAR1 KO)在IFNβ治疗后(100 ng/mL)。细胞核用DAPI染色(蓝色),并用白色虚线勾勒。IFNβ暴露后,pMLKL在细胞核和细胞质中可见。gydF4y2BangydF4y2Ba,线形图描述了pMLKL阳性的动力学,条形图显示了pMLKL信号的定位。gydF4y2BaogydF4y2Ba在IFNβ (100 ng/mL)处理后48 h, ADAR1 WT或ADAR1 KO mef核膜完整性的Lamin B1(绿色)染色。gydF4y2BapgydF4y2Ba, IFNβ (100 ng/mL)处理ADAR1 WT或ADAR1 KO mef后核膜破裂动力学。数据为均数±标准差。(n = 4英寸gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BangydF4y2Ba,gydF4y2BapgydF4y2Ba或者说n = 3英寸gydF4y2BakgydF4y2Ba,gydF4y2BalgydF4y2Ba每组)。带韦尔奇修正的双尾未配对t检验(gydF4y2BabgydF4y2Ba,gydF4y2BaegydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba,gydF4y2BalgydF4y2Ba)、单因素方差分析检验(gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba)或双向方差分析(gydF4y2BapgydF4y2Ba).*gydF4y2BaPgydF4y2Ba< 0.05 [gydF4y2BaPgydF4y2Ba= 0.0178英寸gydF4y2BakgydF4y2Ba(gydF4y2BaIfih1gydF4y2Ba),gydF4y2BaPgydF4y2Ba= 0.026英寸gydF4y2BalgydF4y2Ba(gydF4y2BaSlfn5gydF4y2Ba), * *gydF4y2BaPgydF4y2Ba< 0.005 [gydF4y2BaPgydF4y2Ba= 0.0007英寸gydF4y2BabgydF4y2Ba(干扰素γ)gydF4y2BaPgydF4y2Ba= 0.001英寸gydF4y2BaegydF4y2Ba,gydF4y2BaPgydF4y2Ba= 0.0044英寸gydF4y2BakgydF4y2Ba(gydF4y2BaEif2ak2gydF4y2Ba),gydF4y2BaPgydF4y2Ba= 0.002英寸gydF4y2BakgydF4y2Ba(gydF4y2BaDdx58gydF4y2Ba),gydF4y2BaPgydF4y2Ba= 0.0018英寸gydF4y2BalgydF4y2Ba(gydF4y2BaKnl1gydF4y2Ba), * * *gydF4y2BaPgydF4y2Ba< 0.0005 [gydF4y2BaPgydF4y2Ba< 0.0001 ingydF4y2BabgydF4y2Ba(干扰素β),gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BaPgydF4y2Ba= 0.0002 ingydF4y2BalgydF4y2Ba(gydF4y2BaXrn1gydF4y2Ba)]。数据至少代表三个独立的实验。gydF4y2Ba
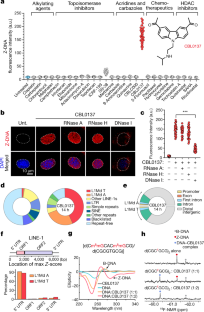
图4 CBL0137诱导Z-DNA形成。gydF4y2Ba
一个gydF4y2Ba, A-RNA, Z-RNA, Z-DNA和B-DNA的结构。gydF4y2BabgydF4y2Ba, CBL0137类似物的Z-DNA诱导活性。gydF4y2BacgydF4y2BaZ-DNA信号荧光强度的定量gydF4y2BabgydF4y2Ba.gydF4y2BadgydF4y2Ba在cbl0137处理的FLAG-ZBP1 mef中,L1Md A和L1Md T重复序列的基因组分布与Z22峰重叠。gydF4y2BaegydF4y2Ba, DeepZ分析特征的线性模型权重的分位数-分位数图。该图显示,该图的上界和下界的值不同于原假设下预期的正态分布。gydF4y2BafgydF4y2Ba,在数据集Z22和FLAG-ZBP1中评估的特征的重要性的比较图显示了在分别分析每个独立派生的数据集时DeepZ分析的可重复性。gydF4y2BaggydF4y2Ba,基于在联合Z22和FLAG-ZBP1数据集上训练的线性回归权重的特征的重要性,使用1到−1的标准化尺度进行评分。表观遗传标记、转录因子结合位点和其他预测LINE 5’UTR区域Z22和FLAG-ZBP1下拉的元素具有阳性值,并识别出与细胞中Z-DNA形成相关的特征。gydF4y2BahgydF4y2Ba,用CBL0137(5µM)处理或不处理12 h的mef染色Z-DNA(红色)和PML(绿色)。gydF4y2Ba我gydF4y2BaCBL0137未处理或处理细胞中PML病灶的定量。数据为均数±标准差。(n = 30英寸gydF4y2BacgydF4y2Ba或者n = 5英寸gydF4y2Ba我gydF4y2Ba每组)。带韦尔奇修正的双尾未配对t检验(gydF4y2Ba我gydF4y2Ba).***gydF4y2BaPgydF4y2Ba< 0.0005 (gydF4y2BaPgydF4y2Ba< 0.0001 ingydF4y2Ba我gydF4y2Ba).数据至少代表三个独立的实验。gydF4y2Ba
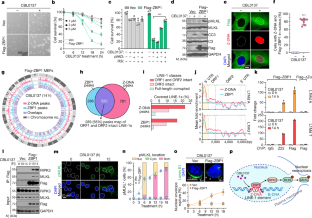
图5 CBL0137诱导zbp1依赖性细胞死亡。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BaMlklgydF4y2Ba−−/gydF4y2Ba或gydF4y2BaMlklgydF4y2Ba−−/gydF4y2BaCasp8gydF4y2Ba−−/gydF4y2Ba在存在或不存在zVAD (50 mM)的情况下,用CBL0137(5µM)处理或不处理mef,并在处理后18小时检测其活力。gydF4y2BabgydF4y2Ba初代早期传代中Z-DNA的形成(p<5)gydF4y2BaZbp1gydF4y2Ba+/+gydF4y2Ba还有垃圾控制gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2BaCBL0137(5µM)处理的mef。gydF4y2BacgydF4y2BaZ-DNA信号荧光强度的定量gydF4y2BabgydF4y2Ba.gydF4y2BadgydF4y2Ba,gydF4y2BaZbp1gydF4y2Ba+/+gydF4y2Ba而且gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba在有或没有zVAD (50 mM)和RIPK3抑制剂(R3i) GSK’843(5µM)的情况下,用CBL0137(5µM)处理mef,并在处理后18小时检测其活力。gydF4y2BaegydF4y2Ba,原发性mef中zbp1依赖性MLKL激活的免疫印迹分析。gydF4y2BafgydF4y2Ba, hIFNβ存在或不存在时,人成纤维细胞HS68中ZBP1、RIPK3和MLKL水平的免疫印迹(100 ng/mL, 6 h)。gydF4y2BaggydF4y2BaCBL0137处理HS68细胞(5µM, 12 h), Z-DNA形成。gydF4y2BahgydF4y2BaZ-DNA信号荧光强度的定量gydF4y2BaggydF4y2Ba.gydF4y2Ba我gydF4y2Ba将hIFNβ预处理(100 ng/mL, 6 h)的HS68细胞暴露于有或没有zVAD (50 mM)和RIPK3抑制剂(R3i, GSK ' 872, 5µM)的CBL0137中,并在处理后36 h检测细胞活力。gydF4y2BajgydF4y2Ba在CBL0137处理hfn β预处理(100 ng/mL, 6 h)的HS68细胞中,存在或不存在zVAD (50 mM)和RIPK3抑制剂(R3i, GSK ' 872, 5µM),在CBL0137处理30 h后,通过免疫印迹分析检测MLKL的激活情况。gydF4y2BakgydF4y2Ba, cbl0137诱导细胞死亡动力学gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Bamef与空载体(Vec)、FLAG-ZBP1或其突变体稳定重组。gydF4y2BalgydF4y2Ba, MLKL激活的免疫印迹分析gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2BaCBL0137处理后,用空载体(Vec)、FLAG-ZBP1或FLAG-ZBP1突变体重组mef。gydF4y2Ba米gydF4y2Ba、主gydF4y2BaZbp1gydF4y2Ba+/+gydF4y2Ba和littermate-matchedgydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba用CBL0137(5µM, 18 h)处理BMDMs,并染色Z-DNA(红色)和巨噬细胞标记物F4/80(绿色)。gydF4y2BangydF4y2Ba、主gydF4y2BaZbp1gydF4y2Ba+/+gydF4y2Ba而且gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba用CBL0137(5µM)处理BMDMs,并在处理后24 h检测其活力。gydF4y2BaogydF4y2Ba, CBL0137(5µM)处理14 h后富flag峰的分布。gydF4y2BapgydF4y2Ba,它gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba用CBL0137(1.5µM, 14 h)处理由FLAG- zbp1或FLAG- ΔZα突变体稳定重建的mef,并检测FLAG的抗FLAG免疫沉淀。全细胞提取物(5%输入)平行检测FLAG。采用GAPDH作为加载对照。数据为均数±标准差。(n = 4英寸gydF4y2Ba一个gydF4y2Ba,gydF4y2BadgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba,gydF4y2BangydF4y2Ba或者n = 20英寸gydF4y2BacgydF4y2Ba,gydF4y2BahgydF4y2Ba每组)。带韦尔奇修正的双尾未配对t检验(gydF4y2Ba一个gydF4y2Ba,gydF4y2BacgydF4y2Ba,gydF4y2BangydF4y2Ba)、单因素方差分析检验(gydF4y2BadgydF4y2Ba,gydF4y2Ba我gydF4y2Ba)或双向方差分析(gydF4y2BakgydF4y2Ba).***gydF4y2BaPgydF4y2Ba< 0.0005 (gydF4y2BaPgydF4y2Ba= 0.0002 ingydF4y2Ba一个gydF4y2Ba,gydF4y2BaPgydF4y2Ba< 0.0001 ingydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba,gydF4y2BangydF4y2Ba).数据至少代表三个独立的实验。gydF4y2Ba
图6 CBL0137诱导Z-DNA和免疫原性。gydF4y2Ba
一个gydF4y2Ba用CBL0137(5µM)处理HepG2、MCF7、A549、HT-29、HeLa、SK-MEL-2或A375人细胞系12 h,并染色Z-DNA。gydF4y2BabgydF4y2BaZ-DNA信号荧光强度的定量gydF4y2Ba一个gydF4y2Ba.gydF4y2BacgydF4y2Ba用CBL0137(5µM)处理缺乏坏死效应表达的细胞系(HepG2、MCF7、A549、HeLa或sk - mel2)或具有完整/重建坏死信号的细胞系(用FLAG-hZBP1重组的HT-29细胞、用hfn β (100 ng/mL)预处理6 h的HS68细胞、WT MEFs、LET1小鼠气道上皮细胞或用FLAG-mZBP1稳定重组的SVEC4-10小鼠内皮细胞),并在CBL0137处理24 h(或HS68细胞36 h)后检测细胞活力。gydF4y2BadgydF4y2Ba用空载体(Vec)或FLAG-hZBP1重组HT-29细胞,cbl0137诱导细胞死亡动力学。gydF4y2BaegydF4y2Ba用CBL0137处理稳定表达空载体或FLAG-hZBP1的人HT-29细胞的全细胞提取物,用免疫印迹法检测磷酸化的MLKL和FLAG-hZBP1。gydF4y2BafgydF4y2Ba,对小鼠皮下B16-F10黑色素瘤进行治疗,如图所示gydF4y2Ba5克gydF4y2Ba.在CBL0137(20µM,瘤内注射)和抗pd1抗体(200µg/小鼠,i.p.注射)四个周期后收集肿瘤。将这些肿瘤冷冻切片共染色用于CD8的免疫荧光检测gydF4y2Ba+gydF4y2BaCD44gydF4y2Ba+gydF4y2Ba, CD8gydF4y2Ba+gydF4y2BaGzmBgydF4y2Ba+gydF4y2Ba或CD8gydF4y2Ba+gydF4y2BaKi67gydF4y2Ba+gydF4y2BaT细胞,或CD11cgydF4y2Ba+gydF4y2BaDCs。gydF4y2BaggydF4y2Ba,小鼠患同基因皮下B16-OVA黑素瘤的治疗方案。在肿瘤体积相似(~ 100mm)的小鼠上接种肿瘤细胞后8天开始治疗gydF4y2Ba3.gydF4y2Ba).gydF4y2BahgydF4y2Ba,识别ova特异性的门控策略(SIINFEKL H-2K)gydF4y2Bab +gydF4y2Ba) CD8gydF4y2Ba+gydF4y2BaT细胞。gydF4y2Ba我gydF4y2Ba, ova特异性CD8总数gydF4y2Ba+gydF4y2Ba从肿瘤引流腹股沟淋巴结分离的T细胞(左图)和ova特异性CD8的频率gydF4y2Ba+gydF4y2BaT细胞在肿瘤引流腹股沟淋巴结,作为总CD8的一部分gydF4y2Ba+gydF4y2BaT细胞(右图)。gydF4y2BajgydF4y2Ba,双侧B16-OVA黑色素瘤小鼠治疗方案。在肿瘤体积相似(~ 100mm)的小鼠上接种肿瘤细胞后8天开始治疗gydF4y2Ba3.gydF4y2Ba).gydF4y2BakgydF4y2Ba, cbl0137治疗(同侧)和未治疗(对侧)B16-OVA肿瘤在指定治疗后的肿瘤生长曲线。gydF4y2BalgydF4y2Ba,来自CBL0137(肿瘤内,20µM)处理或未处理的WT小鼠YUMMER1.7黑素瘤切片的成纤维细胞(PDGFRα,绿色)和Z-DNA(红色)的免疫荧光染色。gydF4y2Ba米gydF4y2BaZ-DNA阳性成纤维细胞比例的定量gydF4y2BalgydF4y2Ba.gydF4y2BangydF4y2Ba, Vehicle或CBL0137注射YUMMER1.7肿瘤WT或gydF4y2BaZbp1gydF4y2Ba−−/gydF4y2Ba对小鼠进行PDGFRα(绿色)或pMLKL(红色)染色。细胞核用DAPI染色(蓝色)。gydF4y2BaogydF4y2Ba.pMLKL的定量gydF4y2Ba+gydF4y2Ba成纤维细胞在gydF4y2BangydF4y2Ba.gydF4y2BapgydF4y2Ba,静脉给药50 mg/kg 24 h后,CBL0137处理或未处理小鼠B16-F10黑素瘤切片Z-DNA(红色)和DAPI(蓝色)免疫荧光染色。gydF4y2Ba问gydF4y2BaZ-DNA阳性细胞的定量分析gydF4y2BapgydF4y2Ba.数据为均数±标准差。在gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,gydF4y2Ba米gydF4y2Ba,gydF4y2BaogydF4y2Ba,gydF4y2Ba问gydF4y2Ba或者S.E.M. ingydF4y2BafgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba(n = 20英寸gydF4y2BabgydF4y2Ba, n = 4英寸gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba, n = 6英寸gydF4y2BafgydF4y2Ba,gydF4y2Ba我gydF4y2Ba, n = 3英寸gydF4y2Ba米gydF4y2Ba,gydF4y2Ba问gydF4y2Ba或者n = 5英寸gydF4y2BaogydF4y2Ba每组)。带韦尔奇修正的双尾未配对t检验(gydF4y2BacgydF4y2Ba)、单因素方差分析检验(gydF4y2BafgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BaogydF4y2Ba)或双向方差分析(gydF4y2BadgydF4y2Ba,gydF4y2BakgydF4y2Ba).**gydF4y2BaPgydF4y2Ba< 0.005 (gydF4y2BaPgydF4y2Ba= 0.0015英寸gydF4y2Ba我gydF4y2Ba), * * *gydF4y2BaPgydF4y2Ba< 0.0005 (gydF4y2BaPgydF4y2Ba< 0.0001 ingydF4y2BadgydF4y2Ba,gydF4y2BafgydF4y2Ba,gydF4y2Ba我gydF4y2Ba,gydF4y2BakgydF4y2Ba,gydF4y2BaogydF4y2Ba).数据至少代表三个独立的实验gydF4y2Ba源数据gydF4y2Ba.gydF4y2Ba
图7用于构建Z22和FLAG-ZBP1 ChIP-Seq数据集中L1Md A和L1Md T重复序列富集谱图的算法流程图gydF4y2Ba
在映射到L1元素之后,读取的数据被归一化,并与来自UCSC基因组浏览器RepeatMasker追踪的鼠标组件mm10的一致L1序列对齐。按照补充方法计算并平滑每个位置的富集值,并与LI开放阅读框的NCBI注释对齐。gydF4y2Ba
补充信息gydF4y2Ba
补充图1gydF4y2Ba
未裁剪的免疫印迹用于准备主要数据图和扩展数据图。gydF4y2Ba
补充表1gydF4y2Ba
Z22 RIP-seq富集的z - rna。ADAR1 KO中z22富集rna的z -形成特征gydF4y2BaZbp1gydF4y2Ba- / -gydF4y2BaIFNβ处理48 h后的MEFs。还显示了ifn β处理的ADAR1 WT mef中Z22下拉序列中编辑的重复序列的位置。通过比较RIP-seq实验和匹配的对照总RNA-seq实验来测定富集程度。gydF4y2BaPgydF4y2Ba值是使用DeSeq2包中实现的单侧负二项检验(pvalue列)得出的,并且使用Benjamini-Hochberg程序来解释多次比较(padj列)。gydF4y2Ba
补充表2gydF4y2Ba
cbl0137处理细胞中通过ChIP-seq鉴定的z - dna形成序列的DeepZ特征。使用DeepZ模型评估来自ENCODE数据的874个特征,以确定它们在预测由Z22 ChIP-seq在cbl0137处理的细胞中识别的Z-DNA形成区域方面的正或负重要性,并由Flag-ZBP1 ChIP-seq验证。所分配的权重由负向正排列。特征标签使用相同的配色方案,如图扩展数据图4g所示。gydF4y2Ba
权利和权限gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
张涛,尹,C,费多洛夫,A。gydF4y2Baet al。gydF4y2BaADAR1掩盖了zbp1驱动的坏死的癌症免疫治疗前景。gydF4y2Ba自然gydF4y2Ba606gydF4y2Ba, 594-602(2022)。https://doi.org/10.1038/s41586-022-04753-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-04753-7gydF4y2Ba
这篇文章被引用gydF4y2Ba
针对细胞死亡途径的癌症治疗:坏死、焦亡、铁亡和铜腐病研究的最新进展gydF4y2Ba
血液学与肿瘤学杂志gydF4y2Ba(2022)gydF4y2Ba
z -核酸促进了免疫治疗gydF4y2Ba
自然评论癌症gydF4y2Ba(2022)gydF4y2Ba
心血管健康和疾病中的RNA修饰gydF4y2Ba
心脏病学自然评论gydF4y2Ba(2022)gydF4y2Ba
诱导Z-DNA克服免疫检查点封锁抗性gydF4y2Ba
细胞研究gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
