文摘gydF4y2Ba
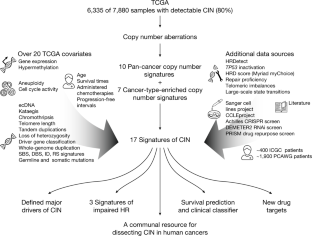
染色体不稳定(CIN)导致大规模的累积损失,收益和重组的DNAgydF4y2Ba1gydF4y2Ba。广泛的基因组的复杂性引起的CIN是癌症的一个特点gydF4y2Ba2gydF4y2Ba;然而,没有系统的框架来测量不同类型的临床表型pan-cancer CIN及其影响。我们评估的程度,多样性和CIN的起源在7880年代表33癌症肿瘤。我们目前的17个拷贝数描述特定类型的CIN的签名,与假定的目的:支持多个独立的数据源。签名预测药物反应和确定新的药物靶点。我们的框架改进同源重组受损的理解,这是一种最治疗CIN的通道类型。我们的结果说明一个基本结构潜在的人类癌症基因组的复杂性并提供资源来指导未来CIN的研究。gydF4y2Ba
这是一个预览的订阅内容,gydF4y2Ba通过访问你的机构gydF4y2Ba
相关的文章gydF4y2Ba
开放获取文章引用这篇文章。gydF4y2Ba
卵巢癌突变过程驱动站点特定的免疫逃避gydF4y2Ba
自然gydF4y2Ba开放获取gydF4y2Ba2022年12月14日gydF4y2Ba
功能分析的结构变体使用Strand-seq单个细胞gydF4y2Ba
自然生物技术gydF4y2Ba开放获取gydF4y2Ba2022年11月24日gydF4y2Ba
染色质结构在癌症gydF4y2Ba
BMC分子和细胞生物学gydF4y2Ba开放获取gydF4y2Ba2022年7月28日gydF4y2Ba
访问选项gydF4y2Ba
订阅自然+gydF4y2Ba
得到直接在线访问自然和其他55个自然杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅杂志gydF4y2Ba
得到完整的期刊访问1年gydF4y2Ba
199.00美元gydF4y2Ba
只有3.90美元的问题gydF4y2Ba
所有价格是净价格。gydF4y2Ba
增值税将被添加后在付款。gydF4y2Ba
税计算期间将完成付款。gydF4y2Ba
买条gydF4y2Ba
时间有限或全文访问ReadCube。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格是净价格。gydF4y2Ba

数据可用性gydF4y2Ba
在这项研究中使用的所有数据从公开的渠道获得,补充表1,详细描述部分的数据和代码gydF4y2Ba补充的方法gydF4y2Ba。加入一些原始数据限制访问(TCGA dbGaP号码:gydF4y2Baphs000178.v11.p8gydF4y2Ba;ICGC EGA加入号码:gydF4y2BaEGAS00001001692gydF4y2Ba)。访问可以通过应用相关的数据访问委员会(TCGA或ICGC)。作者声明,所有其他数据支持本研究的发现,包括所有数据的源数据,是公开的,也可以没有限制gydF4y2Ba补充信息gydF4y2BaGithub库。所有的数据都支持我们的拷贝数签名通航的分析通过我们的门户网站(gydF4y2Bahttps://markowetz.cruk.cam.ac.uk/cincompendium/gydF4y2Ba)。gydF4y2Ba
代码的可用性gydF4y2Ba
通过我们的中心存储库(代码公开gydF4y2Bahttps://github.com/markowetzlab/Drews2022_CIN_CompendiumgydF4y2Ba得到了CIN签名),它描述了如何以及如何重现这个发表的数据和表。存储库还包含公共可访问的数据和中间结果和生产用于这项研究。中心存储库链接到其他存储库包含专门任务的代码。gydF4y2Ba
引用gydF4y2Ba
支持,s . f . &负责人l c染色体不稳定的多方面的作用在癌症及其微环境。gydF4y2Ba细胞gydF4y2Ba174年gydF4y2Ba,1347 - 1360 (2018)。gydF4y2Ba
Hanahan d & Weinberg r . a .癌症的标志:下一代。gydF4y2Ba细胞gydF4y2Ba144年gydF4y2Ba,646 - 674 (2011)。gydF4y2Ba
Tijhuis a E。,Johnson, S. C. & McClelland, S. E. The emerging links between chromosomal instability (CIN), metastasis, inflammation and tumour immunity.摩尔。Cytogenet。gydF4y2Ba12gydF4y2Ba,17 (2019)。gydF4y2Ba
Chakravarti D。,LaBella, K. A. & DePinho, R. A. Telomeres: history, health, and hallmarks of aging.细胞gydF4y2Ba184年gydF4y2Ba,306 - 322 (2021)。gydF4y2Ba
支持,s . f . et al .染色体不稳定驱动器通过胞质DNA转移反应。gydF4y2Ba自然gydF4y2Ba553年gydF4y2Ba,467 - 472 (2018)。gydF4y2Ba
戴维斯,h . et al . HRDetect预测缺乏BRCA1和BRCA2突变特征。gydF4y2BaNat,地中海。gydF4y2Ba23gydF4y2Ba,517 - 525 (2017)。gydF4y2Ba
Cohen-Sharir, y . et al。非整倍性使癌细胞容易抑制有丝分裂检查点。gydF4y2Ba自然gydF4y2Ba590年gydF4y2Ba,486 - 491 (2021)。gydF4y2Ba
麦金太尔,g . et al .拷贝数签名在卵巢癌和突变过程。gydF4y2BaNat,麝猫。gydF4y2Ba50gydF4y2Ba,1262 - 1270 (2018)。gydF4y2Ba
斯蒂尔,c, d . et al .未分化肉瘤发展通过不同的进化历程。gydF4y2Ba癌症细胞gydF4y2Ba35gydF4y2Ba,441 - 456。e8 (2019)。gydF4y2Ba
Ben-David, &亚背景决定一切:非整倍性癌症。gydF4y2BaNat,启麝猫。gydF4y2Ba21gydF4y2Ba44 - 62 (2020)。gydF4y2Ba
斯多克,C。,Kok, Y. P., van den Tempel, N. & van Vugt, M. A. T. M. Shaping the BRCAness mutational landscape by alternative double-strand break repair, replication stress and mitotic aberrancies.核酸Res。gydF4y2Ba49gydF4y2Ba,4239 - 4257 (2021)。gydF4y2Ba
Takemon Y。gydF4y2Ba等gydF4y2Ba。Multi-omic分析揭示哺乳动物中投的角色在细胞周期调控和细胞有丝分裂富达预印本gydF4y2BabioRxivgydF4y2Bahttps://www.biorxiv.org/content/10.1101/533323v2gydF4y2Ba(2019)。gydF4y2Ba
地狱,m . P。杜达,M。,Weber, T. C., Moch, H. & Krek, W. Tumor suppressor VHL functions in the control of mitotic fidelity.癌症Res。gydF4y2Ba74年gydF4y2Ba,2422 - 2431 (2014)。gydF4y2Ba
Brownlee, p . M。,Chambers, A. L., Cloney, R., Bianchi, A. & Downs, J. A. BAF180 promotes cohesion and prevents genome instability and aneuploidy.细胞的代表。gydF4y2Ba6gydF4y2Ba,973 - 981 (2014)。gydF4y2Ba
西尔弗曼,j·S。,Skaar, J. R. & Pagano, M. SCF ubiquitin ligases in the maintenance of genome stability.学生物化学的发展趋势。科学。gydF4y2Ba37gydF4y2Ba,66 - 73 (2012)。gydF4y2Ba
Godinho s a & Pellman d中心体异常在癌症的原因和后果。gydF4y2Ba菲尔。反式。r . Soc。BgydF4y2Ba369年gydF4y2Ba,20130467 (2014)。gydF4y2Ba
Menghi f . et al,串联复印机表型是一个普遍的癌症全基因组配置由不同的基因突变。gydF4y2Ba癌症细胞gydF4y2Ba34gydF4y2Ba,197 - 210。e5 (2018)。gydF4y2Ba
Abkevich诉et al。基因杂合性丢失的模式预测卵巢癌上皮的同源重组修复缺陷。gydF4y2BaBr。j .癌症gydF4y2Ba107年gydF4y2Ba,1776 - 1782 (2012)。gydF4y2Ba
罗德,t . et al .倍性和大规模基因组不稳定性持续官腔乳房癌认同BRCA1/2失活。gydF4y2Ba癌症Res。gydF4y2Ba72年gydF4y2Ba,5454 - 5462 (2012)。gydF4y2Ba
癌症基因组图谱的研究网络。综合基因组分析卵巢癌。gydF4y2Ba自然gydF4y2Ba474年gydF4y2Ba,609 - 615 (2011)。gydF4y2Ba
Nik-Zainal, s . et al . 560年景观体细胞突变的乳腺癌的全基因组序列。gydF4y2Ba自然gydF4y2Ba534年gydF4y2Ba47-54 (2016)。gydF4y2Ba
奥格登,。,Rida, P. C. G. & Aneja, R. Prognostic value of CA20, a score based on centrosome amplification-associated genes, in breast tumors.Sci代表。gydF4y2Ba7gydF4y2Ba262 (2017)。gydF4y2Ba
广场,a &嘿,W.-D。同源重组而形成的复杂的基因重组。gydF4y2Ba细胞生物的趋势。gydF4y2Ba29日gydF4y2Ba,135 - 149 (2019)。gydF4y2Ba
Guirouilh-Barbat, J。兰伯特,S。,Bertrand, P. & Lopez, B. S. Is homologous recombination really an error-free process?前面。麝猫。gydF4y2Ba5gydF4y2Ba175 (2014)。gydF4y2Ba
Knijnenburg, t . a . et al .基因组和分子dna损伤修复的缺陷在癌症基因组图谱。gydF4y2Ba细胞的代表。gydF4y2Ba23gydF4y2Ba,239 - 254。e6 (2018)。gydF4y2Ba
Saavedra h . I。Fukasawa, K。,Conn, C. W. & Stambrook, P. J. MAPK mediates RAS-induced chromosome instability.生物。化学。gydF4y2Ba274年gydF4y2Ba,38083 - 38090 (1999)。gydF4y2Ba
Perl, a . l . et al .蛋白磷酸酶2绑定的控件进行DNA复制和调节细胞分裂周期45 (CDC45)。gydF4y2Ba生物。化学。gydF4y2Ba294年gydF4y2Ba,17043 - 17059 (2019)。gydF4y2Ba
陈,l . et al。增强R-loop骨髓增生异常综合征是一种促成一致(团结)的机制引起的高风险剪接因子突变。gydF4y2Ba摩尔。细胞gydF4y2Ba69年gydF4y2Ba,412 - 425。e6 (2018)。gydF4y2Ba
李问:et al . ERCC2解旋酶域突变赋予核苷酸切除修复缺陷和驱动顺铂敏感性muscle-invasive膀胱癌。gydF4y2Ba中国。癌症Res。gydF4y2Ba25gydF4y2Ba,977 - 988 (2019)。gydF4y2Ba
梅农诉& Povirk l . p53参与DNA双链断裂的修复:多方面的角色p53的同源重组修复(嗯)和异源端加入(NHEJ)。gydF4y2Ba亚晶胞。物化学。gydF4y2Ba85年gydF4y2Ba,321 - 336 (2014)。gydF4y2Ba
Dentro, s . c . et al .描述遗传intra-tumor跨2658年人类癌症基因组异质性。gydF4y2Ba细胞gydF4y2Ba184年gydF4y2Ba,2239 - 2254。e39 (2021)。gydF4y2Ba
杜赫斯特美国m . et al .宽容的全基因组加倍传播染色体不稳定和加速癌症基因组进化。gydF4y2Ba癌症。gydF4y2Ba4gydF4y2Ba,175 - 185 (2014)。gydF4y2Ba
Davoli t &德兰格,t .多倍体在正常发展的原因和后果和癌症。gydF4y2Ba为基础。启细胞Dev。杂志。gydF4y2Ba27gydF4y2Ba,585 - 610 (2011)。gydF4y2Ba
Berenjeno, i m . et al .致癌PIK3CA诱发中心体扩增基因组加倍和宽容。gydF4y2BaCommun Nat。gydF4y2Ba8gydF4y2Ba1773 (2017)。gydF4y2Ba
Darp, R。,Vittoria, M. A., Ganem, N. J. & Ceol, C. J. Oncogenic BRAF induces whole-genome doubling through suppression of cytokinesis. Preprint atbioRxivgydF4y2Bahttps://doi.org/10.1101/2021.04.08.439023gydF4y2Ba(2021)。gydF4y2Ba
张,问:et al . FBXW7促进nonhomologous end-joining通过K63-linked polyubiquitylation XRCC4。gydF4y2Ba摩尔。细胞gydF4y2Ba61年gydF4y2Ba,419 - 433 (2016)。gydF4y2Ba
Citri,。,Skaria, K. B. & Yarden, Y. The deaf and the dumb: the biology of ErbB-2 and ErbB-3.Exp。细胞Res。gydF4y2Ba284年gydF4y2Ba54 - 65 (2003)。gydF4y2Ba
Alexandrov l . b . et al .突变签名在人类癌症的曲目。gydF4y2Ba自然gydF4y2Ba578年gydF4y2Ba,94 - 101 (2020)。gydF4y2Ba
文卡特斯,s . et al .感应APOBEC3加剧DNA复制压力和染色体不稳定性在早期乳腺癌和肺癌进化。gydF4y2Ba癌症。gydF4y2Ba11gydF4y2Ba,2456 - 2473 (2021)。gydF4y2Ba
Crockford, a . et al .细胞周期素D抗药性genome-doubling与功能性p53肿瘤。gydF4y2Ba安。肿瘤防治杂志。gydF4y2Ba28gydF4y2Ba,149 - 156 (2017)。gydF4y2Ba
雷•乔杜里a &生理压力,a的多方面的角色PARP1在DNA修复和染色质重塑。gydF4y2BaNat。启摩尔。细胞杂志。gydF4y2Ba18gydF4y2Ba,610 - 621 (2017)。gydF4y2Ba
高尔,s . et al . CDK4/6抑制触发抗肿瘤免疫力。gydF4y2Ba自然gydF4y2Ba548年gydF4y2Ba,471 - 475 (2017)。gydF4y2Ba
Brownlee, p . M。,Meisenberg, C. & Downs, J. A. The SWI/SNF chromatin remodelling complex: Its role in maintaining genome stability and preventing tumourigenesis.DNA修复gydF4y2Ba32gydF4y2Ba,127 - 133 (2015)。gydF4y2Ba
散步流言,g . j . P。,Foltz, D. R. & Cleveland, D. W. Lethality to human cancer cells through massive chromosome loss by inhibition of the mitotic checkpoint.Proc。《科学。美国gydF4y2Ba101年gydF4y2Ba,8699 - 8704 (2004)。gydF4y2Ba
昆廷,r . j . et al .肿瘤细胞全基因组加倍赋予独特的遗传缺陷。gydF4y2Ba自然gydF4y2Ba590年gydF4y2Ba,492 - 497 (2021)。gydF4y2Ba
詹森,。,散步流言,g . j . P。L. & Medema, R. H. Elevating the frequency of chromosome mis-segregation as a strategy to kill tumor cells.Proc。《科学。美国gydF4y2Ba106年gydF4y2Ba,19108 - 19113 (2009)。gydF4y2Ba
达塔,d . et al .核仁的GTP-binding蛋白1 (NGP-1)促进G1 S相变通过激活细胞周期蛋白依赖性激酶抑制剂p21 Cip1 / Waf1。gydF4y2Ba生物。化学。gydF4y2Ba290年gydF4y2Ba,21536 - 21552 (2015)。gydF4y2Ba
马丁·l·P。,Hamilton, T. C. & Schilder, R. J. Platinum resistance: the role of DNA repair pathways.中国。癌症Res。gydF4y2Ba14gydF4y2Ba,1291 - 1295 (2008)。gydF4y2Ba
扎克,t . i . et al . Pan-cancer体细胞拷贝数改变的模式。gydF4y2BaNat,麝猫。gydF4y2Ba45gydF4y2Ba,1134 - 1140 (2013)。gydF4y2Ba
谢宁,et al。清新formalin-fixed标本的DNA拷贝数的分析与识别和排除问题地区浅全基因组测序的基因组组装。gydF4y2Ba基因组Res。gydF4y2Ba24gydF4y2Ba,2022 - 2032 (2014)。gydF4y2Ba
确认gydF4y2Ba
我们感谢m·埃尔德里奇设置在线资源和a·伯曼bug修复。R.M.D.,P.S.S., D.-L.C., J.D.B. and F.M. are funded by Cancer Research UK (core grants C14303/A17197, A22905 and A19274) and the Cambridge Cancer Centre (grant C9685/A25117). G.M. and B.H. are hosted by the Centro Nacional de Investigaciones Oncológicas (CNIO), which is supported by the Instituto de Salud Carlos III and recognized as a ‘Severo Ochoa’ Centre of Excellence (ref. CEX2019-000891-S) by the Spanish Ministry of Science and Innovation (MCIN/AEI/ 10.13039/501100011033). G.M. and B.H. were also supported by a Spanish Ministry of Science and Innovation grant PID2019-111356RA-I00 (MCIN/AEI/ 10.13039/501100011033). M.T. was supported as a postdoctoral researcher of the F.R.S.-FNRS. L.M.G. was supported by the Wellcome Trust PhD programme in Mathematical Genomics and Medicine (grant number RG92770). M.S. was supported by the Horizon 2020 (H2020) Integrated Training Network CONTRA (grant 766030-CONTRA-H2020-MSCA-ITN-2017). This work was supported by the Francis Crick Institute, which receives its core funding from Cancer Research UK (FC001202), the UK Medical Research Council (FC001202) and the Wellcome Trust (FC001202). P.V.L. is a Winton Group Leader in recognition of the Winton Charitable Foundation’s support towards the establishment of The Francis Crick Institute.
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
通用和F.M.贡献同样这项工作。R.M.D.,通用和F.M. conceived and designed the study. R.M.D., B.H., D.-L.C., M.S. and G.M. developed the methodology of the study. R.M.D., B.H., M.T., K.H., T.L., P.S.S., L.M.G., L.L., M.S. and G.M. developed the software for the study. R.M.D., M.T., K.H., T.L., P.S.S., D.-L.C. and G.M. contributed to the validation of the method and results. R.M.D., B.H., L.M.G., D.-L.C., L.L. and G.M. contributed to the formal analysis presented in this study. R.M.D., B.H., K.H., T.L., P.S.S., L.M.G. and P.V.L. provided access to data and contributed to gathering, processing and curating data. R.M.D., J.D.B., P.V.L., G.M. and F.M. wrote the original draft. R.M.D., B.H., G.M. and F.M. produced and contributed to the visualizations of the study. R.M.D., G.M. and F.M. supervised the project. All authors had access to all of the data in the study. All authors contributed to the review and the editing of the manuscript. All authors approved the manuscript before the initial submission and all other resubmissions.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
J.D.B.,通用,F.M. are co-founders, directors and share holders of Tailor Bio Ltd. Cancer Research UK, the University of Cambridge and the Spanish National Cancer Research Centre (CNIO) are in the process of applying for a patent application GB2114203.9 covering the copy number signature methodology and drug target associations that lists R.M.D., B.H., G.M. and F.M. as inventors. G.M., F.M. and J.D.B are listed on a patent on using copy number signatures to predict response to doxorubicin treatment in ovarian cancer (PCT/EP2021/065058).
同行评审gydF4y2Ba
同行审查的信息gydF4y2Ba
自然gydF4y2Ba感谢匿名评论者对他们的贡献的同行评审工作。gydF4y2Ba同行审查报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然保持中立在发表关于司法主权地图和所属机构。gydF4y2Ba
扩展数据数据和表gydF4y2Ba
扩展数据图1工作流的样本筛选和检测染色体不稳定(dCIN)。gydF4y2Ba
一个gydF4y2Ba,注意图显示流量样本的研究。gydF4y2BabgydF4y2Ba前面的卵巢,对于每一个拷贝数的特性签名:每个样本的事件数量的直方图无法分配给卵巢拷贝数TCGA卵巢群签名。红色虚线表示0.95分位数。gydF4y2BacgydF4y2Ba散点图的癌症类型比较我们的估计的可检测CIN (gydF4y2Ba补充的方法gydF4y2Ba)估计报道Mitelman数据库。gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba,箱线图比较我们的估计与非整倍性检测CIN得分和四个CNA-specific指标。盒子代表四分位范围(差)和中值作为一个粗体线。胡须扩展到最大/最小值不超过1.5 *差的铰链。离群值超出了胡须被分别标记为结束点。双边韦尔奇的t检验的结果显示在箱线图。gydF4y2Ba
扩展数据图2拷贝数的特性和签名识别的概述。gydF4y2Ba
一个gydF4y2Ba,一个示意图显示5基本拷贝数的特性,计算使用6335个样本检测CIN (dCIN)。注意,一个功能捕捉绝对拷贝数不包括在我们的方法。gydF4y2BabgydF4y2Ba,示意图显示混合模型用于全基因组特征分布分割成更小的组件通过变分贝叶斯有限高斯混合模型或混合泊松模型。生成的组件的实际数量是下面列出的每个特性分布。这些组件代表每个特性分布的基本构建块。gydF4y2BacgydF4y2Ba的概率的一个例子,CNA属于混合组件(后验概率)计算和总结。gydF4y2BadgydF4y2Ba,gydF4y2Ba(gydF4y2Ba右)结果43-dimensional为每个样本特征向量,毕竟后验概率是总结为每个组件。(左)的示意图sum-of-posterior矩阵对所有6335个样本被分裂成两个矩阵的贝叶斯实现非负矩阵factorisation (NMF),导致签名目录和一个活动目录。gydF4y2Ba
扩展数据图3签名纲要确定的示意图。gydF4y2Ba
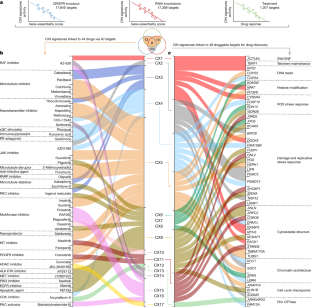
一个gydF4y2Ba,从完整的输入矩阵10 pan-cancer签名确认。gydF4y2BabgydF4y2Ba,20个癌症类型超过100个样本,128个癌症丰富签名(ct)被确定。gydF4y2BacgydF4y2Ba,所有ct被移除,余弦相似度超过0.74与任何pan-cancer签名。gydF4y2BadgydF4y2Ba,从组织的ct余弦相似性超过0.74,签名与活动最多的样本被代表签名。gydF4y2BaegydF4y2Ba,我们执行非负最小二乘法在每一对pan-cancer特定签名ct。任意组合的显示重建误差低于0.1,ct是移除。gydF4y2BafgydF4y2Ba,10套pan-cancer和7 ct被加入的17个签名。gydF4y2BaggydF4y2Ba签名活动,利用线性组合分解,计算6335年TCGA样本。gydF4y2Ba
扩展数据图4签名解释矩阵。gydF4y2Ba
显示左边的五个特性,其混合组件和组件的意思。右边的热图显示签名解释值,结合信息从sum-of-posterior矩阵,矩阵和签名签名活动定义矩阵(gydF4y2Ba补充的方法gydF4y2Ba)。只有组件与显示签名活动水平呈正相关。解释每个特性值正常化和签名。gydF4y2Ba
扩展数据图5蒙特卡洛仿真结果确定signature-specific噪声阈值。gydF4y2Ba
一个gydF4y2Ba每签名,每一个情节(1)显示了示例签名活动的四分位范围后引入的噪音拷贝数特性使用蒙特卡罗模拟。样品下令他们观察到签名活动(红线)。gydF4y2BabgydF4y2Ba,示意图显示我们如何安装所有样本的高斯分布模拟值与观测到的签名活动(0)(红色线)。水平黑线代表拟合高斯和形式的分位数0.95我们签名的基础特定的噪声阈值,值低于这条线不区分从0。gydF4y2BacgydF4y2Ba17、情节signature-specific阈值的拷贝数签名。gydF4y2Ba
扩展数据图6不同拷贝数签名稳定分析技术。gydF4y2Ba
在相同的一组478个肿瘤,我们比较了基于SNP6-array拷贝数拷贝数概要文件的概要文件和签名和签名中使用不同的拷贝数分析技术。列包含结果的不同技术和拷贝数的行包含结果比较概要文件(上),签名活动(中)和签名的定义(底部,限于pan-cancer签名)。对于每个比较我们的结果展示ASCAT是分段常数的处罚范围拟合或ASCAT。sc的圆形的二元分割。(*):设置标有一个明星是不可能获得解K = 10,而不是选择最优数量的K(低于K = 10)。gydF4y2Ba
扩展数据图7工作流评级决定签名病因学和信心。gydF4y2Ba
一个gydF4y2Ba流程图展示一个突变基因之间的联系和签名活动被用来推导一个假设为一个公认的病因学。gydF4y2BabgydF4y2Ba,流程图代表决策过程导致的分配三星级评级信心得分。gydF4y2BacgydF4y2Ba,明星CX3评级过程的例子。gydF4y2Ba
扩展数据图8总结签名和其他协变量之间的关联。gydF4y2Ba
一个gydF4y2Ba主面板显示显著的拷贝数签名和突变基因之间的关联。基因注释总结在下面的面板。盒子用红线显示显著的关联并不被认为是在确定签名目的:通过放大显著富集的基因,也居住在一个ecDNA扩增子,这可能是签名的结果而不是原因,可能导致与放大假相关签名(CX8、CX9 CX11, CX13)。gydF4y2BabgydF4y2Ba每一行显示,高度显著的签名和不同的协变量之间的关联。除非另有规定,只显示正相关性。gydF4y2Ba
扩展数据图9同源重组受损签名及其关联。gydF4y2Ba
一个gydF4y2Ba不同的患者群体,箱线图总结签名活动(行)定义为他们的司机基因突变状态。卵巢样品颜色在橙色的深绿色和乳房。盒子代表四分位范围(差)和中值作为一个粗体线。胡须扩展到最大/最小值不超过1.5 *差的铰链。离群值超出了胡须被分别标记为结束点。WT之间的意义与双边韦尔奇t测试gydF4y2BaBRCA1/2gydF4y2Ba和每个类别和纠正的多个测试通过使用Benjamini-Hochberg方法。统计上显著的比较显示右边的箱线图与明星表示意义(q < 0.05)和箭头表示两组用于统计检验。(gydF4y2BaBRCA1/2gydF4y2Ba=gydF4y2Ba乳腺癌易感基因1gydF4y2Ba和gydF4y2BaBRCA2gydF4y2Ba,WT =野生型;LOH =杂合性丢失)。gydF4y2BabgydF4y2Ba箱线图(与相同的特征gydF4y2Ba一个gydF4y2Ba)总结了签名活动TCGA 5466样品的分离低,中等和高细胞周期的分数。括号和星星(q < 0.05)显示有显著增加从低到中等到高细胞周期组测试韦尔奇的t检验和修正与Benjamini-Hochberg多个测试方法。gydF4y2BacgydF4y2Ba,火山的情节显示的结果相关性签名活动和表达的核苷酸切除修复基因(尼珥)。每个点代表一个基因,彩色圆点显示显著的相关性。gydF4y2BadgydF4y2Ba斯皮尔曼相关系数(轴)之间的相关性的签名活动和七HRD(列在顶部)的共同标准。个人系数显示受损的同源重组(《国际卫生条例》)签名和系数的分布从剩下的签名由箱线图(相同的特点gydF4y2Ba一个gydF4y2Ba)。gydF4y2Ba
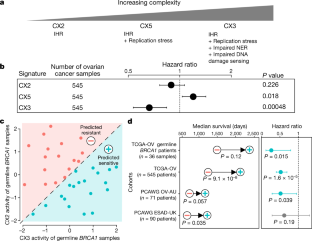
扩展数据图10个分类器的性能预测铂敏感。gydF4y2Ba
一个gydF4y2Ba,kaplan meier估计显示,TCGA卵巢癌患者的总体生存概率分成两组使用我们CX3 / CX2分类器。gydF4y2BabgydF4y2Ba,风险比率及其95%置信区间从Cox比例风险模型训练获得CX3 / CX2分类预测,TCGA卵巢癌患者的总体生存。该模型还纠正病人的年龄和癌症阶段。假定值代表瓦尔德测试的重要性。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2Ba,中位数生存和风险比率生成五癌症TCGA的军团,PCAWG和ICGC项目使用的预测从三个分类器(CX3 / CX2分类器,HRDetect和无数myChoice基于HRD分数)。平均生存考验的改善生存率较(kaplan meier生存分析)与符号代表预测耐药组和+符号预测敏感组。风险比率,他们的95%置信区间,瓦尔德测试意义的预测敏感组相比,预测耐药组获得Cox比例风险模型校正阶段和年龄的患者,除了HRDetect肿瘤阶段是省略的模型不收敛,如果包括在内。病人的数量和比例预测敏感(HRD)和耐药(没有HRD)在右边列出每个分类器。gydF4y2Ba
补充信息gydF4y2Ba
补充的方法gydF4y2Ba
这个文件包含完整的所有方法的描述,软件的链接,额外的结果和1-13补充表。gydF4y2Ba
补充数据gydF4y2Ba
这个文件包含补充1-54无花果。gydF4y2Ba
补充表gydF4y2Ba
这个压缩文件夹包含补充表14 - 64及其伴随的传说。gydF4y2Ba
权利和权限gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
德鲁兹智慧化,Hernando, B., Tarabichi, M.et al。gydF4y2Ba染色体不稳定的pan-cancer纲要。gydF4y2Ba自然gydF4y2Ba606年gydF4y2Ba,976 - 983 (2022)。https://doi.org/10.1038/s41586 - 022 - 04789 - 9gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586 - 022 - 04789 - 9gydF4y2Ba
本文引用的gydF4y2Ba
染色质结构在癌症gydF4y2Ba
BMC分子和细胞生物学gydF4y2Ba(2022)gydF4y2Ba
功能分析的结构变体使用Strand-seq单个细胞gydF4y2Ba
自然生物技术gydF4y2Ba(2022)gydF4y2Ba
在癌症基因组形式服从功能gydF4y2Ba
自然癌症gydF4y2Ba(2022)gydF4y2Ba
染色体外的DNA扩增的癌症gydF4y2Ba
自然遗传学评论gydF4y2Ba(2022)gydF4y2Ba
人类患癌症的分类器gydF4y2Ba
自然遗传学评论gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论你同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条件或准则请国旗是不合适的。gydF4y2Ba
