摘要gydF4y2Ba
细菌转座子是一种普遍存在的可移动遗传元件,它使用不同的dna结合蛋白进行水平传播。例如,gydF4y2Ba大肠杆菌gydF4y2BaTngydF4y2Ba7gydF4y2Ba家园到特定的连接站点使用TnsDgydF4y2Ba1gydF4y2Ba,而crispr相关转座子使用I型或V型Cas效应子插入引导rna指定的目标位点下游gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba.尽管这种靶向多样性,转座总是需要TnsB,一种dde家族转座酶,催化DNA切除和插入,TnsC,一种AAA+ atp酶,被认为在转座酶和靶向蛋白之间进行通信gydF4y2Ba4gydF4y2Ba.TnsC如何调节这种交流,从而调节转位保真度尚不清楚。在这里,我们使用染色质免疫沉淀与测序来监测I-F型rna引导转座体的体内形成,使我们能够在整合前解决不同的蛋白质募集事件。TniQ-Cascade复合物的DNA靶向令人惊讶地混乱——数百个基因组脱靶位点被采样,但只有其中的一个子集被授权用于TnsC和TnsB招募,这揭示了一个关键的校对检查点。为了促进对转座体组装相互作用机制的理解,我们使用低温电子显微镜确定了TnsC的结构,发现ATP结合驱动七聚环的形成,这些环将DNA穿过中心孔,从而定位下游整合的底物。总的来说,我们的研究结果强调了rna引导转位过程中连续因子结合到基因组靶位点所赋予的分子特异性,并提供了一个结构路线图来指导未来的工程工作。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
访问选项gydF4y2Ba
订阅《自然》+gydF4y2Ba
立即在线访问《自然》和其他55种《自然》杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅期刊gydF4y2Ba
获得1年的完整期刊访问权限gydF4y2Ba
199.00美元gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
增值税稍后将在结帐时添加。gydF4y2Ba
税务计算将在结账时完成。gydF4y2Ba
买条gydF4y2Ba
在ReadCube上获得时间限制或全文访问。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba


数据可用性gydF4y2Ba
下一代测序数据可在国家生物技术信息中心(NCBI)序列读取档案(BioProject加入:gydF4y2BaPRJNA759215gydF4y2Ba)和基因表达集合(gydF4y2BaGSE183114gydF4y2Ba).用于分析的已发表基因组来自NCBI (GenBank:gydF4y2BaCP001509.3gydF4y2Ba).载脂蛋白- tnsc七聚体、dna结合单环复合物和dna结合双环复合物的图谱和模型已存入电子显微镜数据库gydF4y2Baemdb - 24783gydF4y2Ba,gydF4y2Baemdb - 26476gydF4y2Ba而且gydF4y2Baemdb - 26477gydF4y2Ba,以及登入代号下的PDBgydF4y2Ba7 rzygydF4y2Ba,gydF4y2Ba7 ufigydF4y2Ba而且gydF4y2Ba7 ufmgydF4y2Ba,分别。本研究中使用的其他原子模型可在PDB上根据登录代码获得gydF4y2Ba7 n6igydF4y2Ba(TniQ-boundgydF4y2Ba商店gydF4y2BaTnsC),gydF4y2Ba7 m99gydF4y2Ba(gydF4y2Ba商店gydF4y2BaTnsC),gydF4y2Ba6 vbwgydF4y2Ba(gydF4y2BaVchgydF4y2BaTniQ-Cascade),gydF4y2Ba6 v9pgydF4y2Ba(gydF4y2BaVchgydF4y2BaTniQ)和gydF4y2Ba7 mcsgydF4y2Ba(gydF4y2Ba生态gydF4y2BaTnsC)。在当前研究中生成和分析的数据集可根据合理要求从相应作者处获得。gydF4y2Ba
代码的可用性gydF4y2Ba
用于所述ChIP-seq数据分析的自定义脚本可在GitHub (gydF4y2Bahttps://github.com/sternberglab/Hoffmann_Kim_Beh_etal_2022gydF4y2Ba).gydF4y2Ba
参考文献gydF4y2Ba
Bainton, R. J., Kubo, K. M., Feng, J. & Craig, N. L. Tn7转座:在纯化的体外系统中,靶DNA识别由多个Tn7编码蛋白介导。gydF4y2Ba细胞gydF4y2Ba72gydF4y2Ba, 931-943(1993)。gydF4y2Ba
Klompe, S. E., Vo, P. L. H., Halpin-Healy, T. S. & Sternberg, S. H.转座子编码的CRISPR-Cas系统直接rna引导DNA整合。gydF4y2Ba自然gydF4y2Ba571gydF4y2Ba, 219-225(2019)。gydF4y2Ba
Strecker, J.等。rna引导DNA插入与crispr相关转座酶。gydF4y2Ba科学gydF4y2Ba365gydF4y2Ba, 48-53(2019)。gydF4y2Ba
彼得斯,J. E. Tn7。gydF4y2BaMicrobiol。Spectr。gydF4y2Ba2gydF4y2Ba, mdna3-0010-2014(2014)。gydF4y2Ba
病毒和移动元素是进化转变的驱动因素。gydF4y2Ba费罗斯。反式。r . Soc。BgydF4y2Ba371gydF4y2Ba, 20150442(2016)。gydF4y2Ba
希克曼,A. B. &戴达,F. DNA转位机制。gydF4y2BaMicrobiol。Spectr。gydF4y2Ba3 gydF4y2Ba, mdna3-0034-2014(2015)。gydF4y2Ba
Tn7元件的靶向转座:安全位点、可移动质粒、CRISPR/Cas等。gydF4y2Ba摩尔。Microbiol。gydF4y2Ba112gydF4y2Ba, 1635-1644(2019)。gydF4y2Ba
Sarnovsky, R. J., May, E. W. & Craig, N. L. Tn7转座酶是一种异质复合物,其中DNA断裂和连接活性分布在不同的基因产物之间。gydF4y2BaEMBO J。gydF4y2Ba15gydF4y2Ba, 6348-6361(1996)。gydF4y2Ba
Choi, K. Y., Spencer, J. M. & Craig, N. L. Tn7转座调节因子TnsC与转座酶亚基TnsB和目标选择器TnsD相互作用。gydF4y2Ba国家科学院学报美国gydF4y2Ba111gydF4y2Ba, e2858-e2865(2014)。gydF4y2Ba
Peters, J. E. & Craig, N. L. Tn7利用DNA结合蛋白TnsE识别与DNA复制相关的转位靶结构。gydF4y2Ba基因的开发。gydF4y2Ba15gydF4y2Ba, 737-747(2001)。gydF4y2Ba
Stellwagen, A. E. & Craig, N. L. TnsC中的功能获得性突变,TnsC是一种atp依赖性转座蛋白,可激活细菌转座Tn7。gydF4y2Ba遗传学gydF4y2Ba145gydF4y2Ba, 573-585(1997)。gydF4y2Ba
Kuduvalli, P. N., Rao, J. E. & Craig, N. L.靶DNA结构在Tn7转座中起关键作用。gydF4y2BaEMBO J。gydF4y2Ba20.gydF4y2Ba, 924-932(2001)。gydF4y2Ba
Stellwagen, A. E. & Craig, N. L.一种atp依赖的Tn7转位调节基因功能获得突变体的分析。gydF4y2BaJ. Mol.生物学。gydF4y2Ba305gydF4y2Ba, 633-642(2001)。gydF4y2Ba
Peters, J. E., Makarova, K. S., Shmakov, S. & Koonin, E. V.通过Tn7-like转座子招募CRISPR-Cas系统。gydF4y2Ba国家科学院学报美国gydF4y2Ba114gydF4y2Ba, e7358-e7366(2017)。gydF4y2Ba
福雷,G.等人。移动基因元件中的CRISPR-Cas:反防御及其他。gydF4y2BaNat. Rev.微生物。gydF4y2Ba17gydF4y2Ba, 513-525(2019)。gydF4y2Ba
斋藤等人。crispr相关转座子归巢的双模式。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 2441-2453(2021)。gydF4y2Ba
Vo, P. L. H.等。CRISPR rna引导的高效、多重细菌基因组工程集成酶。gydF4y2Ba生物科技Nat。》。gydF4y2Ba39gydF4y2Ba, 480-489(2021)。gydF4y2Ba
Halpin-Healy, t.s., Klompe, s.e., Sternberg, s.h. & Fernández, i.s.转座子编码CRISPR-Cas系统DNA靶向的结构基础。gydF4y2Ba自然gydF4y2Ba577gydF4y2Ba, 271-274(2020)。gydF4y2Ba
Snider, J., Thibault, G. & Houry, W. A.功能多样化蛋白质的AAA+超家族。gydF4y2Ba基因组医学杂志。gydF4y2Ba9gydF4y2Ba, 216(2008)。gydF4y2Ba
Puchades, C., Sandate, C. R. & Lander, G. C.控制AAA+蛋白活性和功能多样性的分子原理。gydF4y2Ba细胞生物学。gydF4y2Ba21gydF4y2Ba, 43-58(2020)。gydF4y2Ba
霍尔德,J. W. &克雷格,N. L. Tn7转位后复合体的结构:一个复杂的核蛋白结构。gydF4y2BaJ. Mol.生物学。gydF4y2Ba401gydF4y2Ba, 167-181(2010)。gydF4y2Ba
沈,Y.等。Tn7转座子DNA靶向的结构基础。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba29gydF4y2Ba, 143-151(2022)。gydF4y2Ba
公园,J.-U。et al。rna引导的DNA转位系统中靶点选择的结构基础。gydF4y2Ba科学gydF4y2Ba373gydF4y2Ba, 768-774(2021)。gydF4y2Ba
Querques, I., Schmitz, M., Oberli, S., Chanez, C. & Jinek, M.利用V型crispr -转座子系统进行靶点选择和重构。gydF4y2Ba自然gydF4y2Ba599gydF4y2Ba, 497-502(2021)。gydF4y2Ba
Vo, P. L. H., Acree, C., Smith, M. L. & Sternberg, S. H.通过长读测序对CRISPR rna引导转位产物进行无偏见分析。gydF4y2Ba暴徒。DNAgydF4y2Ba12gydF4y2Ba, 13(2021)。gydF4y2Ba
朗宁,D. R.等。TnsC的羧基末端部分通过与TnsA的特定相互作用激活Tn7转座酶。gydF4y2BaEMBO J。gydF4y2Ba23gydF4y2Ba, 2972-2981(2004)。gydF4y2Ba
Leenay, R. T. & Beisel, C. L.破译,通信和工程CRISPR PAM。gydF4y2BaJ. Mol.生物学。gydF4y2Ba429gydF4y2Ba, 177-191(2017)。gydF4y2Ba
Kuscu, C., Arslan, S., Singh, R., Thorpe, J. & Adli, M.全基因组分析揭示了Cas9内切酶结合的脱靶位点的特征。gydF4y2Ba生物科技Nat。》。gydF4y2Ba32gydF4y2Ba, 677-683(2014)。gydF4y2Ba
吴,等。CRISPR内切酶Cas9在哺乳动物细胞中的全基因组结合。gydF4y2Ba生物科技Nat。》。gydF4y2Ba32gydF4y2Ba, 670-676(2014)。gydF4y2Ba
库珀,L. A.,斯特林格,A. M.和韦德,J. T.确定级联结合的特异性,干扰,和体内启动适应gydF4y2Ba大肠杆菌gydF4y2BaI-E型CRISPR-Cas系统。gydF4y2BamBiogydF4y2Ba9gydF4y2Ba, e02100-17(2018)。gydF4y2Ba
Thakore, p.i.等人。通过CRISPR-Cas9阻遏物对远端调控元件进行高度特异性的表观基因组编辑。gydF4y2BaNat方法。gydF4y2Ba12gydF4y2Ba, 1143-1149(2015)。gydF4y2Ba
O 'Geen, H., Henry, I. M., Bhakta, M. S., Meckler, J. F. & Segal, D. J.使用ChIP-seq和靶向序列捕获对Cas9结合特异性的全基因组分析。gydF4y2Ba核酸测定。gydF4y2Ba43gydF4y2Ba, 3389-3404(2015)。gydF4y2Ba
张杨,等。基于模型的ChIP-Seq (MACS)分析。gydF4y2Ba基因组医学杂志。gydF4y2Ba9gydF4y2Ba, r137(2008)。gydF4y2Ba
贝利,T. L.等。MEME套件:motif发现和搜索的工具。gydF4y2Ba核酸测定。gydF4y2Ba37gydF4y2Ba, w202-w208(2009)。gydF4y2Ba
Klompe, S. E.等。I-F型crispr相关转座子的进化和机制多样性。gydF4y2Ba摩尔。细胞gydF4y2Ba82gydF4y2Ba, 616-628(2022)。gydF4y2Ba
肖,杨,等。I型CRISPR-Cas系统中定向r环形成和底物切换机制的结构基础。gydF4y2Ba细胞gydF4y2Ba170gydF4y2Ba, 48-60(2017)。gydF4y2Ba
Semenova, E.等人。Cas6e核糖核酸酶不需要干扰和适应gydF4y2Ba大肠杆菌gydF4y2BaI-E型CRISPR-Cas系统。gydF4y2Ba核酸测定。gydF4y2Ba43gydF4y2Ba, 6049-6061(2015)。gydF4y2Ba
荣格等人。下一代测序芯片上CRISPR-Cas复合物的大规模并行生物物理分析。gydF4y2Ba细胞gydF4y2Ba170gydF4y2Ba, 35-47(2017)。gydF4y2Ba
陈,学术界。et al。改进的CRISPR敲除筛选的设计和分析。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, 4095-4101(2018)。gydF4y2Ba
鲁特考斯卡斯等人。通过CRISPR-Cas监测复合物级联形成的定向r环提供了有效的脱靶位点抑制。gydF4y2Ba细胞的代表。gydF4y2Ba10gydF4y2Ba, 1534-1543(2015)。gydF4y2Ba
齐,L. S.等。重新利用CRISPR作为rna引导的平台,用于基因表达的序列特异性控制。gydF4y2Ba细胞gydF4y2Ba152gydF4y2Ba, 1173-1183(2013)。gydF4y2Ba
杨,G.等人。单个生物大分子的定量质量成像。gydF4y2Ba科学gydF4y2Ba360gydF4y2Ba, 423-427(2018)。gydF4y2Ba
Zivanov, J.等人。在RELION-3中自动高分辨率冷冻- em结构测定的新工具。gydF4y2BaeLifegydF4y2Ba7gydF4y2Ba, e42166(2018)。gydF4y2Ba
Mizuno, N.等人。MuB是一种AAA+ atp酶,形成螺旋丝,以控制DNA转位的靶选择。gydF4y2Ba国家科学院学报美国gydF4y2Ba110gydF4y2Ba, e2441-e2450(2013)。gydF4y2Ba
Davey, M. J. & O 'Donnell, M.复制解旋酶装填器:破环器和环制造商。gydF4y2Ba咕咕叫。医学杂志。gydF4y2Ba13gydF4y2Ba, r594-r596(2003)。gydF4y2Ba
贾楠,谢玮,de la Cruz, M. J, Eng, E. T. & Patel, d.j . crispr - cas -转座子复合物对DNA整合初始步骤的结构功能研究。gydF4y2Ba细胞Res。gydF4y2Ba30.gydF4y2Ba, 182-184(2020)。gydF4y2Ba
阿林金,V.,斯迈什利亚耶夫,G.和巴拉巴斯,O.跳跃向前:DNA杂技驱动转位前进。gydF4y2Ba咕咕叫。当今。结构体。医学杂志。gydF4y2Ba59gydF4y2Ba, 168-177(2019)。gydF4y2Ba
沃克,D. M.弗雷德多里诺,P. L. &哈希,R. M.混合良好gydF4y2Ba大肠杆菌gydF4y2Ba基因组:通过追踪Mu转位发现广泛的接触。gydF4y2Ba细胞gydF4y2Ba180gydF4y2Ba, 703-716(2020)。gydF4y2Ba
Jackson R. N., van Erp, P. B., Sternberg, S. H. & Wiedenheft, B. crispr相关核酸酶的构象调控。gydF4y2Ba咕咕叫。当今。Microbiol。gydF4y2Ba37gydF4y2Ba, 110-119(2017)。gydF4y2Ba
Schmiedeberg, L., Skene, P., Deaton, A. & Bird, A.甲醛交联和固定的时间阈值。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba4gydF4y2Ba, e4636(2009)。gydF4y2Ba
Anzalone, A. V., Koblan, L. W., Liu d.r .利用CRISPR-Cas核酸酶、碱基编辑器、转座酶和启动编辑器进行基因组编辑。gydF4y2Ba生物科技Nat。》。gydF4y2Ba38gydF4y2Ba, 824-844(2020)。gydF4y2Ba
波诺科拉,R. P. &韦德,J. T.细菌转录控制,方法和协议。gydF4y2Ba方法分子生物学。gydF4y2Ba1276gydF4y2Ba, 327-340(2015)。gydF4y2Ba
陈淑娟,周勇,陈勇,顾杰。fastp:一种超快一体化FASTQ预处理器。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, i884-i890(2018)。gydF4y2Ba
朗米德,B. &萨尔茨伯格,S. L.快速间隙阅读对齐与领结2。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba, 357-359(2012)。gydF4y2Ba
李,H.等。序列对齐/映射格式和SAMtools。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 2078-2079(2009)。gydF4y2Ba
Ramírez, F. et al. deepTools2:用于深度测序数据分析的下一代web服务器。gydF4y2Ba核酸测定。gydF4y2Ba44gydF4y2Ba, w160-w165(2016)。gydF4y2Ba
罗宾逊,J. T.等。整合基因组学查看器。gydF4y2Ba生物科技Nat。》。gydF4y2Ba29gydF4y2Ba, 24-26(2011)。gydF4y2Ba
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 841-842(2010)。gydF4y2Ba
Russo, C. J. & Passmore, L. A.用于电子冷冻显微镜的超稳定金衬底。gydF4y2Ba科学gydF4y2Ba346gydF4y2Ba, 1377-1380(2014)。gydF4y2Ba
Suloway, C.等人。自动化分子显微镜:新的Leginon系统。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba151gydF4y2Ba, 41-60(2005)。gydF4y2Ba
Rohou, A. & Grigorieff, N. CTFFIND4:快速准确的电子显微图像离焦估计。gydF4y2Baj . Struct。医学杂志。gydF4y2Ba192gydF4y2Ba, 216-221(2015)。gydF4y2Ba
Zivanov, J., Nakane, T. & Scheres, S. H. W. reon -3.1中冷冻电磁数据集的高阶像差和各向异性放大的估计。gydF4y2BaIUCrJgydF4y2Ba7gydF4y2Ba, 253-267(2020)。gydF4y2Ba
Zivanov, J., Nakane, T. & Scheres, S. H. W.在低温em单粒子分析中光束诱导运动校正的贝叶斯方法。gydF4y2BaIUCrJgydF4y2Ba6gydF4y2Ba, 5-17(2019)。gydF4y2Ba
阿布里沙米,V.等。Scipion的局部重建加速了低温电磁数据对称错配的分析。gydF4y2Ba掠夺。Biophys。摩尔。杂志。gydF4y2Ba160gydF4y2Ba, 43-52(2021)。gydF4y2Ba
Casañal, A., Lohkamp, B. & Emsley, P.电子冷冻显微镜和晶体学数据的大分子模型构建的当前发展。gydF4y2Ba蛋白质科学。gydF4y2Ba29gydF4y2Ba, 1055-1064(2020)。gydF4y2Ba
Afonine, P. V.等。用于冷冻电镜和结晶学的PHENIX实空间细化。gydF4y2BaActa Crystallogr。DgydF4y2Ba74gydF4y2Ba, 531-544(2018)。gydF4y2Ba
穆舒多夫,G. N., Vagin, A. A. & Dodson, E. J.用最大似然方法优化大分子结构。gydF4y2BaActa Crystallogr。DgydF4y2Ba53gydF4y2Ba, 240-255(1997)。gydF4y2Ba
布朗等人。用于建立大分子模型和改进为电子冷冻显微镜重建的工具。gydF4y2BaActa Crystallogr。DgydF4y2Ba71gydF4y2Ba, 136-153(2015)。gydF4y2Ba
Prisant, M. G, Williams, C. J., Chen, V. B, Richardson, J. S. & Richardson, D. C. MolProbity验证的新工具:用于CryoEM主干网的CaBLAM,用于重新思考“水”的UnDowser,以及用于重新捕获在线3D图形的NGL Viewer。gydF4y2Ba蛋白质科学。gydF4y2Ba29gydF4y2Ba, 315-329(2020)。gydF4y2Ba
Baker, n.a., Sept, D., Joseph, S., Holst, m.j. & McCammon, J. A.纳米系统的静电学:应用于微管和核糖体。gydF4y2Ba国家科学院学报美国gydF4y2Ba98gydF4y2Ba, 10037-10041(2001)。gydF4y2Ba
确认gydF4y2Ba
我们感谢N. Jaber和S. R. Pesari对实验室的支持;D. J. Villano, X. Chen, C. Lu讨论ChIP-seq数据分析;D.陈统计咨询;C. Lu用于Covaris声纳通道;N. E. Sanjana关于crRNA文库实验的有益讨论;F. Vallese和O. B. Clarke协助iSCAMS实验和Refeyn仪器接入;L. F. Landweber用于qPCR仪器访问;以及JP苏兹伯格哥伦比亚基因组中心的工作人员为NGS提供支持。在哥伦比亚大学低温电子显微镜中心生成冷冻网格并收集筛选数据。一些冷冻电镜数据收集在西蒙斯电子显微镜中心和位于纽约结构生物学中心的国家自动化分子显微镜资源进行,由西蒙斯基金会(SF349247)和美国国立卫生研究院国家普通医学科学研究所(GM103310)资助,并得到NYSTAR和纽约州议会多数派的额外支持。 J.T.G. was supported by the International Human Frontier Science Program Organization Postdoctoral Fellowship LT001117/2021-C. This research was supported by NIH grant DP2HG011650-01, NIH grant R01EB031935-01, a Pew Biomedical Scholarship and Sloan Research Fellowship, and a start-up package from the Columbia University Irving Medical Center Dean’s Office and the Vagelos Precision Medicine Fund (to S.H.S.).
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
L.Y.B.和S.H.S.构思和设计了最初的项目,并听取了其他作者的意见。F.T.H.在L.Y.B的指导下进行了换位和ChIP-seq实验,并进行了ChIP-seq数据分析。D.R.G.和C.A.根据J.T.M.的输入,进行了额外的ChIP-seq数据分析;P.L.H.V.在L.Y.B的输入下进行crRNA文库和转录抑制实验,并帮助进行分子克隆。P.L.H.V.和C.A.进行crRNA文库数据分析。f.t.h.、m.k.、L.Y.B.和J.T.G.进行了TnsC生化实验。i.s.f., M.K.和J.W.收集了阴性染色和低温电镜数据。国际空间站分析了低温电磁数据,并确定gydF4y2BaVchgydF4y2BaTnsC结构。所有的作者都讨论了数据并撰写了手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
哥伦比亚大学(Columbia University)已经提交了与crispr -转座子系统相关的专利申请,S.H.S.和P.L.H.V.被列为发明者。S.H.S.是Dahlia Biosciences的联合创始人和科学顾问,CrisprBits和Prime Medicine的科学顾问,以及Dahlia Biosciences和CrisprBits的股权持有人。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢Hiroshi Nishimasu和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
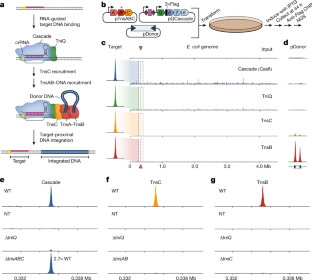
图1标记转座体的设计和ChIP数据分析。gydF4y2Ba
一个gydF4y2Ba,示意图突出显示了用于ChIP-seq实验的每个蛋白质成分中的3×Flag-tag位置。分子质量用括号表示。gydF4y2BabgydF4y2Ba,用于量化rna引导的DNA整合的体内转位实验的简化示意图gydF4y2Ba大肠杆菌gydF4y2Ba.mini -转座子(Mini-Tn) DNA底物从pDonor被动员到基因组DNA,在与crRNA互补的目标位点下游,并通过连接PCR定量整合。gydF4y2BacgydF4y2Ba,对3×Flag-tagged结构体的rna引导转位分析表明保持了接近野生型的活性。TnsB活性位点(D223N)的单点突变完全消融DNA整合。在观察到目标位点的弱富集后,TnsA-Flag不再进行进一步的检测(见图)gydF4y2BadgydF4y2Ba),因此只测试了一次。NT,非靶向crRNA对照。gydF4y2BadgydF4y2Ba,含crRNA-4的3×Flag-tagged TnsA的ChIP-seq数据,如图所示。gydF4y2Ba1 c, dgydF4y2Ba,揭示了基因组靶点的低效DNA富集(左)。然而,pDonor上两个转座子末端的显著峰值(右)表明该蛋白保留了体内功能,并可能在预整合转座体复合物中表现出与目标DNA的低效交联。读取覆盖率(RPKM)被扩展到最高峰值(gydF4y2Ba大肠杆菌gydF4y2Ba基因组图谱读取)或TnsB ChIP-seq数据(pDonor mapping读取)。gydF4y2BaegydF4y2Ba, crRNA-287实验ChIP-seq代表性数据,如图所示。gydF4y2Ba1 c, dgydF4y2Ba,揭示了多个不同因子精确招募到相应的基因组靶位点。gydF4y2BafgydF4y2Ba, ChIP-seq数据揭示了跨多个crRNA数据集的CRISPR阵列的强大自靶向性。对于标记成分的两个不同crrna,显示了映射到pQCascade的Reads,并且归一化到峰值,除了输入样本,它们归一化到相应Cas8样本的峰值。pQCascade的矢量图如图所示(上),CRISPR间隔体的位置用栗色三角形表示(下)。映射到的非唯一读取gydF4y2BalacIgydF4y2Ba未进行分析(虚线)。gydF4y2BaggydF4y2Ba在存在(浅蓝色)或不存在(ΔTniQ,深蓝色)TniQ的情况下,使用Cascade对基因组编码的mRFP进行基于CRISPR干扰(CRISPRi)的抑制。转录抑制通过相对于完全匹配(PM) crRNA的od归一化mRFP荧光来测量。元,新;1-4, crRNA错配位置1-4;25-28, crRNA在位置25-28错配。gydF4y2BahgydF4y2Ba,通过级联(crRNA-4)结合靶DNA与不含TniQ,通过荧光偏振测量。上色如图所示gydF4y2BaggydF4y2Ba.gydF4y2Ba我gydF4y2BaTnsC可能通过TnsAB的存在而稳定在整合前转座体复合体中。在实验中,3×Flag-tagged TnsC的ChIP-seq读取映射到以基因组目标位点为中心的3 kb窗口,仅在TnsA和TnsB存在或不存在时(上)存在差异,读取覆盖(RPKM)在两个图中缩放相同。请注意,图中显示了相同的数据。gydF4y2Ba1 fgydF4y2Ba.对相同的ChIP样本和非靶向对照进行定量PCR,确认省略TnsA-TnsB时靶DNA富集水平降低(下图)。相对富集计算为ΔΔgydF4y2BaCgydF4y2Ba问gydF4y2Ba在目标位点和内参基因之间。gydF4y2BajgydF4y2Ba,在缺乏TnsA和供体DNA的情况下,TnsB可以被招募到rna引导的靶位点,尽管效率会降低。在实验中,3×Flag-tagged TnsB的ChIP-seq读取映射到以基因组目标位点为中心的3 kb窗口,仅在TnsA和pDonor存在或不存在时存在差异,读取覆盖(RPKM)在所有三个图中缩放相同。数据gydF4y2BacgydF4y2Ba,gydF4y2BaggydF4y2Ba,gydF4y2BahgydF4y2Ba而且gydF4y2Ba我gydF4y2Ba表示为平均值±标准差gydF4y2BangydF4y2Ba= 3个独立的生物重复,TnsA-Flag除外(gydF4y2BacgydF4y2Ba,gydF4y2BangydF4y2Ba= 1)和ΔTnsAB (gydF4y2Ba我gydF4y2Ba,gydF4y2BangydF4y2Ba= 1)。gydF4y2Ba
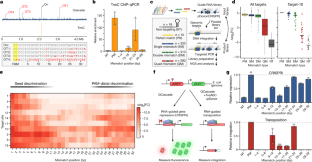
扩展数据图2来自ChIP-seq数据的脱靶DNA结合的附加分析。gydF4y2Ba
一个gydF4y2Ba,散点图显示3×Flag-tagged Cas8和crRNA-4的ChIP-seq实验的两个生物重复之间的相关性。绘制ChIP-seq富集值gydF4y2BangydF4y2Ba= 424个MACS3调用的峰值出现在两个数据集中,由重叠的峰值起始和结束坐标标识。线性回归拟合与Pearson线性相关系数(gydF4y2BargydF4y2Ba)显示;指示目标站点。gydF4y2BabgydF4y2Ba,显示Cas8和TnsC ChIP-seq实验中crRNA-287脱靶峰之间相关性的散点图。绘制ChIP-seq富集值gydF4y2BangydF4y2Ba= 60个MACS3调用的峰值出现在两个数据集中,由重叠的峰值起始和结束坐标标识。线性回归拟合与Pearson线性相关系数(gydF4y2BargydF4y2Ba)显示;对于TnsC来说,目标站点是超出规模的,因此没有被包括在内。gydF4y2BacgydF4y2Ba,中重叠峰Cas8与TnsC峰峰基因组坐标距离的直方图gydF4y2BabgydF4y2Ba.大多数山峰之间的距离都在20 bp以内。gydF4y2BadgydF4y2Ba使用热图对脱靶位点使用crRNA-291的ChIP-seq峰值进行全局可视化,并排绘制Cascade(左)、TnsC(中)和TnsB(右);数据如图所示。gydF4y2Ba2 b, cgydF4y2Ba.487个基因组位点的2 kb窗口(gydF4y2BaygydF4y2Ba轴)按照Cascade数据集称为ChIP-seq峰值富集程度递减的顺序绘制,显示RPKM比例条。TnsB的插图强调了对目标位点的强烈富集(第1行)和对其他Cascade脱靶位点的立即下降(第2-5行)。gydF4y2BaegydF4y2Ba, ChIP-seq峰值与crRNA-295的全球可视化,如图所示gydF4y2BadgydF4y2Ba.gydF4y2BafgydF4y2Ba,显示偏离目标峰重叠的维恩图称为Cascade, TnsC和TnsB。使用MACS3分别为每个ChIP-seq数据集调用峰值,并根据每个峰值的坐标分析重叠。数据显示为相同的两个crrnagydF4y2BadgydF4y2Ba而且gydF4y2BaegydF4y2Ba.gydF4y2BaggydF4y2Ba通过MEME分析,来自不同crrna实验的多个级联ChIP-seq数据集显示出共享一个共同主题的共同脱靶峰。每个数据集(从上到下)显示的是伪crrna间隔5 '端对应核苷酸上方的motif(左),以及显示在输入序列中给定位置出现motif匹配的概率的motif概率图(右)。PAM和种子区域显示在顶部,位置6(红色星号/箭头)显示的是缺乏区分。gydF4y2BangydF4y2Ba,构成母题的峰数(以及它们占总峰的百分比);gydF4y2BaEgydF4y2Ba,gydF4y2BaEgydF4y2Ba模因分析得出的模因的-value意义。gydF4y2BahgydF4y2Ba,常见脱靶母题在gydF4y2BaggydF4y2Ba可以归因于伪crrna,它们使用CRISPR阵列中末端重复序列下游的间隔状序列。该原理图显示了pQCascade编码的CRISPR阵列的结构和crRNA生物发生的假定机制。伪crrna依赖于来自第二次重复的5 ' -句柄和来自下游序列的伪间隔,这是本研究中使用的所有pQCascade载体所共有的。值得注意的是,伪crrna将缺乏重复衍生的3 ' -柄(由Cas6结合的茎环),不太可能具有单一的定义长度,并且预计将形成一个同时缺乏Cas6和TniQ的最小级联复合体。gydF4y2Ba
扩展数据图3 crRNA文库和CRISPRi数据的附加分析。gydF4y2Ba
一个gydF4y2Ba,双质粒系统用于crRNA文库的体内转位分析。我们将CRISPR阵列从pEffector重新定位到pDonor自身的迷你转座子内部(底部),相对于我们之前描述的pEffector和pDonor质粒gydF4y2Ba17gydF4y2Ba(上),从而生成pEffector(-CRISPR)和pDonor(+CRISPR)。gydF4y2BabgydF4y2Ba,中所示的两组质粒构建的rna引导转座分析gydF4y2Ba一个gydF4y2Ba,通过qPCR检测。转座子编码的crrna直接导致难以区分的整合效率。gydF4y2BacgydF4y2Ba,与相应靶位点相关的crRNA文库成员的富集分数,如图所示。gydF4y2Ba3 dgydF4y2Ba.gydF4y2BadgydF4y2Ba,位置特定富集分数绘制为日志的变化gydF4y2Ba2gydF4y2Ba(折叠变化)相对于完全匹配的crRNA,对于在所有10个目标位点上包含四倍错配的crRNA。数据沿crRNA显示为单bp分辨率的箱线图;方框表示中位数和四分位范围,胡须表示在出现异常值(点)时,从第25和第75百分位扩展1.5×四分位范围。数据揭示了pam -近端种子序列和pam -远端区域(在顶部显示)的脱靶识别。gydF4y2BaegydF4y2Ba,错配crRNA文库成员的示意图集中在pam远端区域的22-32位。成员被设计来测试非相邻不匹配对转位的影响。gydF4y2BafgydF4y2Ba,对所有10个目标位点的位置特定富集得分进行平均,并绘制出每种错配类型(SM、DM、QM)的变化对数gydF4y2Ba2gydF4y2Ba(折叠变化),与图相似。gydF4y2Ba3 egydF4y2Ba.位置22-32内聚焦错配的数据gydF4y2BaegydF4y2Ba)显示在顶部,显示在位置24和30(红色箭头)缺乏区分,在那里crRNA核苷酸被翻转。gydF4y2BaggydF4y2Ba,基因组编码的crrna定位示意图gydF4y2BamRFPgydF4y2Ba而且gydF4y2BasfGFPgydF4y2Ba基因。导子被设计成既针对模板链又针对非模板链,分别位于基因起始密码子下游约35 bp和65 bp处。gydF4y2BahgydF4y2Ba,与非靶向对照(NT)一起测量每个靶向crrna的转录抑制;针对模板链的crRNA表现出最强的抑制作用。位置1-4和25-28不匹配gydF4y2BasfGFPgydF4y2Ba-靶向crRNA显示类似的效果gydF4y2BamRFPgydF4y2Ba数据如图。gydF4y2Ba3 ggydF4y2Ba.数据gydF4y2BabgydF4y2Ba而且gydF4y2BahgydF4y2Ba表示为平均值±标准差gydF4y2BangydF4y2Ba= 3个独立的生物重复。所示的绝对样本数gydF4y2BadgydF4y2Ba是gydF4y2BangydF4y2Ba= 10,20,30,40,30,20和10,分别对应错配位置1,2,3,4 - 29,30,31和32。gydF4y2Ba
扩展数据图4 TnsC•ATP七聚体的原始电子显微照片和图像处理流程。gydF4y2Ba
一个gydF4y2Ba, TnsC•ATP七聚体在不同离焦处的代表性冷冻显微照片;相应的CTF如图所示(右上)。gydF4y2BabgydF4y2Ba代表性的无参考2D类平均,确定了四种类型的视图:顶部和底部视图,横向视图和中间视图。分类运行期间使用的圆形掩码的大小在每种类型的一级平均值中表示。gydF4y2BacgydF4y2Ba,用于TnsC•ATP七聚体的同质类粒子的识别和高分辨率细化的图像处理工作流程。7个TnsC•ATP单体中的一个表现出较弱的密度(橙色星号/箭头)。gydF4y2Ba
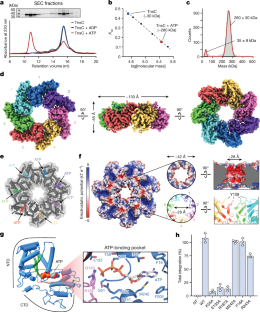
图5 TnsC的傅里叶壳层相关曲线、局部分辨率、代表性密度和二级结构图。gydF4y2Ba
一个gydF4y2Ba,傅里叶壳层相关(FSC)曲线计算两个独立细化的半图的最终子类粒子掩码前(蓝色),掩码后(黑色),和相位随机化后(红色)。曲线显示了施加C7对称条件的细化(上)和在没有施加任何对称约束的情况下对四种最佳TnsC单体进行屏蔽细化(下)。通过FSC-0.143标准,对称重建的最终分辨率为3.5 Å,没有对称强制的屏蔽细化的最终分辨率为3.6 Å。gydF4y2BabgydF4y2Ba,根据局部分辨率计算着色的未锐化地图,显示正交方向的对称地图(上)和没有对称强制的蒙面地图(下)。如图所示(右下)为C1中细化的蒙面图的欧拉角分布,根据密度分布着色。gydF4y2BacgydF4y2Ba、模型-地图FSC曲线(黑线)和模型过拟合试验,将最终模型的原子坐标随机置换0.5 Å,并将置换后的坐标相对半地图1重新精化。蓝色曲线(FSC对半地图1)与红色曲线(FSC对半地图2,未包含在细化中)的重合保证了原始模型中没有过拟合。gydF4y2BadgydF4y2Ba,来自后处理对称图的代表性最终低温电磁密度,以细棒表示。一个Mg的假定密度gydF4y2Ba2 +gydF4y2BaIon由黑色箭头表示(右)。gydF4y2BaegydF4y2Ba的二级结构图gydF4y2BaVchgydF4y2BaTnsC来源于结构数据。α-螺旋被描绘为蓝色圆柱,β-链被描绘为绿色箭头,环路被描绘为灰色痕迹。ATP结合区、面向孔区和AAA+ ATP酶家族的标志序列被指出。gydF4y2Ba
图6单体间相互作用、生化表征和TnsC七聚体形成。gydF4y2Ba
一个gydF4y2Ba、TnsC atp酶突变体纯化及七聚体组装实验。所示突变体的SDS-PAGE分析(左)和SEC色谱图(右),显示TnsC单体峰和七聚体峰。Walker A (K55A)、Walker B (E135A)和精氨酸指(R187A)基序的突变阻止稳定七聚体的形成。gydF4y2BabgydF4y2Ba相邻的TnsC单体通过两个近端环相互作用,其中一个单体的275-280残基与相邻单体的188-191残基相互作用(右侧所示)。gydF4y2BacgydF4y2Ba、TnsC界面和孔环突变体的纯化和七聚体组装实验,如图gydF4y2Ba一个gydF4y2Ba.在ATP存在的情况下,单体-单体界面的突变不能形成稳定的七聚体。凝胶源数据参见补充图1。gydF4y2BadgydF4y2Ba, CRISPR rna引导转座检测所指示的环突变体,通过qPCR检测。数据相对于WT进行归一化gydF4y2BadgydF4y2Ba表示为平均值±标准差gydF4y2BangydF4y2Ba= 3个独立的生物重复。gydF4y2Ba
图7 TnsC中柔性环的表征。gydF4y2Ba
一个gydF4y2BaNTD内的多个柔性环从七聚体TnsC盘的顶部突出(左上),这是与TniQ-Cascade复合体接合的潜在位点。顶端环E69-D75和A106-A113用红色框突出显示,假定的dna相互作用残基包括R143, S144和R146(右上)。基于与TnsAB的功能相似性,CTD上的一个长而灵活的尾巴向孔隙中心折叠,并可能与TnsAB接合gydF4y2Ba生态gydF4y2BaTnsC。A106-A113的放大视图和假定的DNA结合区域以带状表示(中右)。gydF4y2BabgydF4y2Ba, CRISPR rna引导转座分析,用于指示TnsC环突变体、CTD缺失和孔面残基(左;TniQ ZnF突变(右;用qPCR法测定。数据相对于WT进行归一化。R143和R146的突变一起取消了转位。数据以平均值±标准差表示gydF4y2BangydF4y2Ba= 3个独立的生物学重复,除R146A (gydF4y2BangydF4y2Ba= 2)及K89A、Y108A及S144A (gydF4y2BangydF4y2Ba= 1)。gydF4y2BacgydF4y2BaTnsC A106-A113环缺失突变体的纯化和七聚体组装实验。SDS-PAGE分析图(左)和SEC色谱图(右)显示TnsC单体和TnsC•ATP七聚体峰。尽管取消了转位,该环缺失突变体仍然能够形成依赖于atp的七聚体。对于TnsC(ΔA106-A113),为了清晰起见,在5 ml保留量标记处将基线设置为0。凝胶源数据参见补充图1。gydF4y2Ba
扩展数据图8 DNA结合、代表性显微图、傅里叶壳层相关曲线、局部分辨率和DNA结合TnsC的代表性密度。gydF4y2Ba
一个gydF4y2Ba,荧光极化测定TnsC对DNA的结合,用ssDNA和dsDNA底物+/ - ATP检测。数据以平均值±标准差表示gydF4y2BangydF4y2Ba= 2个实验重复。gydF4y2BabgydF4y2Ba, ssDNA和dsDNA存在或不存在时TnsC的atp酶活性;ND,未检测到。数据以平均值±标准差表示gydF4y2BangydF4y2Ba= 2个技术重复。gydF4y2BacgydF4y2Ba与ATP和dsDNA孵育后的TnsC样品的iSCAMS分析显示了三种不同的高分子量物种,与冷冻电镜观察到的dna结合的单双和三重七聚环很好地一致。gydF4y2BadgydF4y2Ba,不同离焦处dna结合TnsC样本的代表性冷冻显微照片;每个面板的右上方显示了相应的CTF。gydF4y2BaegydF4y2Ba- - - - - -gydF4y2BahgydF4y2Ba, dna结合的单七聚体复合体。gydF4y2Ba我gydF4y2Ba- - - - - -gydF4y2BalgydF4y2Ba, dna结合双七聚体复合物。gydF4y2BaegydF4y2Ba,gydF4y2Ba我gydF4y2Ba,傅里叶壳层相关(FSC)曲线(上)计算的最终独立细化半映射在掩蔽前(蓝色),掩蔽后(黑色),和相位随机化后(红色)。FSC-0.143标准的最终分辨率为3.46 Å(单七聚体)和3.9 Å(双七聚体)。下图为模型-地图FSC曲线(黑线)和模型过拟合试验,将最终模型的原子坐标随机置换0.5 Å,并对半地图1重新精化置换后的坐标。蓝色曲线(FSC对半地图1)与红色曲线(FSC对半地图2,未包含在细化中)的重合保证了原始模型中没有过拟合。gydF4y2BafgydF4y2Ba,gydF4y2BajgydF4y2Ba,根据正交方向显示的局部分辨率计算着色的未锐化地图。gydF4y2BaggydF4y2Ba,gydF4y2BakgydF4y2Ba,来自后处理图的代表性最终cryoEM密度,以细化模型表示为棒。gydF4y2BahgydF4y2Ba,gydF4y2BalgydF4y2Ba,显示最终细化地图的欧拉角分布的Mollweide图,根据密度分布着色。gydF4y2Ba
图9 dna结合的TnsC七聚体结构多样性。gydF4y2Ba
一个gydF4y2Ba,单个DNA结合的TnsC七聚体的最终冷冻电镜密度,显示未切片(左)和切片(右)侧面视图,以突出穿过中央孔的DNA。单独的子单元被标记。gydF4y2BabgydF4y2Ba中所示的视图的Ribbon表示gydF4y2Ba一个gydF4y2Ba.gydF4y2BacgydF4y2Ba, dna结合的TnsC双七聚体具有代表性的2D类平均值。gydF4y2BadgydF4y2Ba, dna结合的TnsC双七聚体的最终冷冻电镜图的两个正交视图。七个原聚体中的每一个都被标记,DNA密度以灰色显示。这两个七聚环以相同的方向结合DNA。gydF4y2BaegydF4y2Ba, dna结合的TnsC双七聚体的色带表示,如图所示gydF4y2BabgydF4y2Ba,所有原聚体中的ATP分子均显示为球体。gydF4y2BafgydF4y2Ba, dna结合的TnsC三七聚体的三个具有代表性的2D类平均。单个七聚体的动态由虚线箭头突出显示,位于结合DNA末端的TnsC七聚体相对于相邻的七聚体表现出灵活性(见补充视频)gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
扩展数据图10gydF4y2BaVchgydF4y2BaTnsC和TnsC同系物来自进化多样化的转座子。gydF4y2Ba
一个gydF4y2Ba载脂蛋白与dna结合态之间的TnsC结构高度相似。apo-TnsC和dna结合的TnsC单七聚体结构的单体结构叠加产生的全局RMSD为0.53 Å(左);与双七聚体叠加产生的全局RMSD为0.64 Å(右)。ATP分子显示为棒状,NTD和CTD被标记。gydF4y2BabgydF4y2Ba,多序列比对gydF4y2Ba生态gydF4y2BaTnsCgydF4y2Ba22gydF4y2Ba从TngydF4y2Ba7gydF4y2Ba(non-CRISPR相关),gydF4y2BaVchgydF4y2Ba来自本研究的TnsC (I-F型crispr相关),以及gydF4y2Ba商店gydF4y2Ba来自ShCAST的TnsCgydF4y2Ba3 gydF4y2Ba,gydF4y2Ba23gydF4y2Ba,gydF4y2Ba24gydF4y2Ba(V-K型crispr相关)。指出了atp酶活性的关键残基。序列恒等式如下:gydF4y2BaVchgydF4y2BaTnsC和gydF4y2Ba生态gydF4y2BaTnsC, 18.84%;gydF4y2BaVchgydF4y2BaTnsC和gydF4y2Ba商店gydF4y2BaTnsC, 22.18%;gydF4y2Ba生态gydF4y2BaTnsC和gydF4y2Ba商店gydF4y2BaTnsC, 20.83%。gydF4y2BacgydF4y2Ba,三种TnsC同源物的结构域组织比较gydF4y2Ba生态gydF4y2BaTnsC区域与TnsA, TnsB和TnsD相互作用gydF4y2Ba4gydF4y2Ba.注意N端和c端明显较短gydF4y2BaVchgydF4y2BaTnsC和gydF4y2Ba商店gydF4y2BaTnsC。gydF4y2BadgydF4y2Ba三种tniq家族蛋白同源物的结构域组织比较;注意gydF4y2Ba大肠杆菌gydF4y2BaTngydF4y2Ba7gydF4y2Ba蛋白质被称为TnsD锌指(ZnF)图案被突出。gydF4y2BaegydF4y2Ba,整体寡聚物结构(上)与APBS静电势的比较gydF4y2Ba70gydF4y2Ba(下)用于dna结合gydF4y2BaVchgydF4y2BaTnsC•ATP(左;这项研究),dna结合gydF4y2Ba生态gydF4y2BaTnsC (A225V)•AMPPNP(中心;PDB id: 7mcs)gydF4y2Ba22gydF4y2Ba和DNA-boundgydF4y2Ba商店gydF4y2BaTnsC•ATPɣ年代(右;PDB id: 7m99)gydF4y2Ba23gydF4y2Ba,孔的c端面在每个寡聚物中向上。为了清晰起见,所有的静电计算都省略了DNA。七聚体的形成gydF4y2BaVchgydF4y2BaTnsC和gydF4y2Ba生态gydF4y2BaTnsC可能有利于更有效的原聚体-原聚体堆积,这在六聚体中表现得不那么明显gydF4y2Ba商店gydF4y2BaTnsC复杂。gydF4y2BafgydF4y2Ba, dna结合的结构叠加gydF4y2BaVchgydF4y2BaTnsC•ATP(本研究)和gydF4y2Ba生态gydF4y2BaTnsC (PDB ID: 7MCS)gydF4y2Ba22gydF4y2Ba通过对NTD残基16-196 (gydF4y2BaVchgydF4y2BaTnsC)和残基70-90和128-295 (gydF4y2Ba生态gydF4y2BaTnsC), RMSD为2.42 Å。α1(标记)的构象可以创造一个更开放的原聚体结构,从而有效地包装相邻亚基,从而形成七聚体。ATP和AMPPNP以棒状表示。gydF4y2BaggydF4y2Ba, dna结合的结构叠加gydF4y2BaVchgydF4y2BaTnsC•ATP(本研究)和gydF4y2Ba商店gydF4y2BaTnsC (PDB ID: 7M99)gydF4y2Ba23gydF4y2Ba通过对NTD残基16-196 (gydF4y2BaVchgydF4y2BaTnsC)及34-198 (gydF4y2Ba商店gydF4y2BaTnsC), RMSD为1.27 Å。在gydF4y2Ba商店gydF4y2Ba在TnsC中,α1构象可能导致更封闭的原聚体结构,阻碍相邻亚基的有效包装,促进六聚体的形成。ATP和ATPɣS以棒状表示。gydF4y2BahgydF4y2Ba的结构叠加gydF4y2BaVchgydF4y2BaTniQ (PDB ID: 6V9P)gydF4y2Ba46gydF4y2Ba而且gydF4y2Ba商店gydF4y2BaTniQ单体(PDB ID: 7N6I)gydF4y2Ba23gydF4y2Ba,与全球RMSD 1.01 Å一致。ZnF基序被指示。gydF4y2Ba我gydF4y2Ba,型号gydF4y2BaVchgydF4y2BaTniQ•TnsC相互作用及其相对于dsDNA的定位,基于与TniQ结合的TnsC复合物的结构叠加gydF4y2Ba美国hofmanniigydF4y2Ba(pdb id: 7n6i;gydF4y2Ba方法gydF4y2Ba)gydF4y2Ba23gydF4y2Ba,如图所示。gydF4y2Ba5 fgydF4y2Ba.原质点3和4gydF4y2BaVchgydF4y2BaTnsC分别以黄色和橙色表示。的n端面突出的环gydF4y2BaVchgydF4y2BaTnsC,我们发现它对于rna引导的转位至关重要(扩展数据图。gydF4y2Ba7 bgydF4y2Ba),都接近假定gydF4y2BaVchgydF4y2BaTniQ接触点。E69-D75和A106-A113分别为深橙色和粉红色。gydF4y2BajgydF4y2Ba的静电渲染gydF4y2BaVchgydF4y2Ba中所示模型中的TniQ-2gydF4y2Ba我gydF4y2Ba表明离开r -环的DNA(深灰色)很好地定位于与带正电荷的表面(蓝色)相互作用(左);TniQ-1以色带表示。DNA链的再退火可能发生在富含赖氨酸和精氨酸残基的TniQ表面(右),引导DNA从Cascade进入中央孔gydF4y2BaVchgydF4y2BaTnsC七聚物。5 ' -CC-3 ' PAM核苷酸用黄色圆点表示。gydF4y2Ba
补充信息gydF4y2Ba
补充图1gydF4y2Ba
未经裁剪的SDS-PAGE图像。红色虚线框表示图中使用的裁剪区域。gydF4y2Ba
补充表1gydF4y2Ba
本研究中使用的质粒的描述和序列。gydF4y2Ba
补充表2gydF4y2Ba
本研究中使用的纯化蛋白结构的描述和序列。gydF4y2Ba
补充表3gydF4y2Ba
本研究中使用的crrna和基因组靶点。PAM序列(星号)表示紧邻目标原间隔的两个核苷酸。gydF4y2Ba
补充表4gydF4y2Ba
本研究中使用的引物和寡核苷酸的列表。gydF4y2Ba
补充表5gydF4y2Ba
ChIP-seq为本研究读取和meta信息。gydF4y2Ba
补充表6gydF4y2Ba
本研究中使用的crRNA文库间隔。gydF4y2Ba
补充视频1gydF4y2Ba
dna结合的TnsC•ATP双七聚体的最终冷冻电镜图。低温电镜图以不同的方向显示,以突出整体寡聚结构和结合的dsDNA。每个七聚体中的单体被标记和着色,如图扩展数据图9d所示。dsDNA的密度通过每个七聚体的中心孔突出,用灰色表示。这两个七聚体以相同的方向与DNA结合。gydF4y2Ba
补充视频2gydF4y2Ba
dna结合的TnsC•ATP三七聚体的2D类平均。三七聚体2D类平均序列回放突出了第三个TnsC七聚体(左上)相对于其他两个动态较少的dna结合七聚体(中间和右下)的灵活性。gydF4y2Ba
补充视频3gydF4y2Ba
DNA结合后TnsC•ATP七聚体的构象变化。TnsC•ATP单七聚体在载脂蛋白和DNA结合状态下的低温电镜模型之间的变化,从不同的方向观察,突出了与DNA结合相关的微妙构象变化。特别是,在两个亚基之间形成的裂缝表明了一种潜在的破环机制,可能使DNA加载。gydF4y2Ba
权利和权限gydF4y2Ba
根据与作者或其他权利持有人签订的出版协议,《自然》杂志或其许可方对本文拥有独家权利;作者对这篇文章接受的手稿版本的自我存档仅受此类出版协议的条款和适用法律的约束。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
霍夫曼,f.t.,金,M.,贝,L.Y.gydF4y2Baet al。gydF4y2Ba选择性TnsC招募增强rna引导转位的保真度。gydF4y2Ba自然gydF4y2Ba609gydF4y2Ba, 384-393(2022)。https://doi.org/10.1038/s41586-022-05059-4gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05059-4gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
