摘要
准确及时地检测重组谱系对于解释遗传变异、重建流行病传播、确定感兴趣的选择和变异以及准确地进行系统发育分析至关重要1,2,3.,4.在SARS-CoV-2大流行期间,基因组数据的生成超过了现有分析平台的能力,从而影响了病毒进化的实时分析5.在这里,我们使用一种新的系统基因组学方法来搜索几乎全面的SARS-CoV-2重组谱系的系统发育。在2021年5月的160万份样本树中,我们确定了589个重组事件,这表明约2.7%的测序SARS-CoV-2基因组具有可检测到的重组祖先。重组断点被推断成不成比例地发生在基因组中含有刺突蛋白的3'部分。我们的研究结果强调了及时分析重组的必要性,以确定可能增加病毒传播性或毒性的重组谱系的出现。我们预计,这种方法将能够在SARS-CoV-2大流行期间及以后全面实时跟踪病毒重组。
主要
重组是许多流行病原体(包括β -冠状病毒)新遗传变异的主要原因6包括SARS-CoV-2在内的分支。通过混合来自不同基因组的遗传物质,重组可以产生具有潜在重要表型效应的新的突变组合7.例如,重组被认为在中东呼吸综合征的近期进化史中发挥了重要作用8严重急性呼吸综合征冠状病毒(SARS-CoV)9,10,11,12.重组也有可能在未来产生具有人畜共患病潜力的病毒13.因此,准确和及时地鉴定重组是理解进化生物学和已建立和新出现的病原体在人类、农业和自然种群中的感染潜力的基础。
现在,在SARS-CoV-2人群中存在着大量的遗传多样性14已知有时会发生与不同的SARS-CoV-2变体合并感染的情况15重组有望成为大流行期间新基因变异的重要来源。自大流行早期以来,SARS-CoV-2基因组中是否存在可检测到的重组事件信号一直存在激烈的争论13.尽管如此,一些明显真正的重组世系已经确定使用特别的方法16以及通过减少对可能的重组祖先对的搜索空间来处理大量SARS-CoV-2数据集的半自动方法16,17.由于在持续的SARS-CoV-2大流行期间及时和准确监测病毒遗传变异的重要性,需要检测和表征重组单倍型的新方法,以便在新的变异基因组序列获得后尽快评估它们。这种快速转变对于推动在知情和协调的情况下对新型SARS-CoV-2变种的公共卫生应对至关重要。
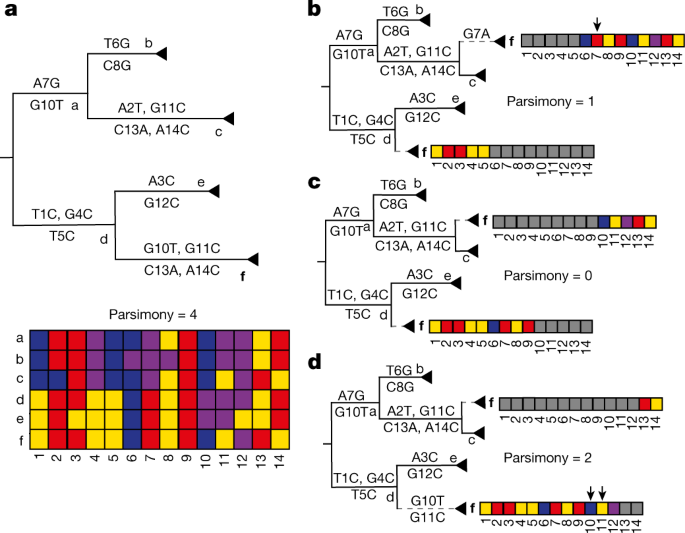
我们开发了一种检测大流行尺度系统发生中的重组的新方法,利用系统发生位置进行重组推断(涟漪,图。1)。因为重组违背了许多系统发育方法的中心假设,即单一的进化史在整个基因组中共享,从不同基因组中产生的重组谱系通常会在“长分支”上发现,这是由于适应两个亲本单倍型的不同进化史(图2)。1)。请注意,只要重组相对不常见,即使分支长度人为扩大,系统发育推断也有望保持准确18.waves通过首先识别综合SARS-CoV-2突变注释树上的长分支来利用这一信号19,20..然后,waves将潜在的重组序列详尽地分解为不同的片段,并使用最大限度的简约性将每个片段替换到一个全局系统发育中。ripple报告了两个亲本节点(下文称为供体和受体),相对于全球系统发育的原始位置,这两个亲本节点导致了最高的简约分数改善1)。因此,我们的方法利用了每个亲本谱系的系统发育信号和基因组上标记的空间相关性。我们使用基于推断的特定位点突变率的零模型来建立显著性(补充文本)2而且3.)。
一个,一个有六个内部节点(标记为A - f)的系统发育,其中节点f(粗体)是作为假定重组体进行研究的节点。根据系统发育下面的多个序列比对,节点f的初始简约得分为4,这表明了样本之间和内部节点之间的差异。请注意,内部节点在现实中可能没有相应的序列,但可以使用重建的祖先基因组进行重组测试。b- - - - - -d,两个间隔的三个部分位置(灰色单元格表示间隔之外的位置),由站点5(面板)后的断点产生b), 9(面板c)和12(面板d),以及由此得出的节俭得分。虚线表示f的部分放置所产生的新分支。箭头标记的位置增加了f的两个部分放置的总和。节点f的最佳部分放置和断点预测位于中心(c),在站点9之后有一个断点,部分位置同时作为节点c的兄弟节点和节点d的后代节点。
通过模拟的大量测试表明,涟漪是有效的,敏感的,可以自信地识别重组世系(补充文本4- - - - - -6)。正如预期的21,当重组发生在基因组边缘或基因相似序列之间时,使用waves(扩展数据图)更难检测。1而且2)。尽管如此,waves检测模拟重组的灵敏度为75.8%。在作为重组体检测的模拟样本中,waves准确地识别了90%的模拟断点(扩展数据表1及补充文本6)。此外,waves能够检测到在以前的分析中确定的所有高置信重组16(补充文本6)。利用ripple对全球约160万个SARS-CoV-2基因组进行了重组分析,结果显示,部分测序的SARS-CoV-2基因组属于可检测的重组谱系。为了减轻测序和组装错误的影响,我们排除了只有一个后代的所有节点,我们应用保守滤波器从由waves标记的重组集中去除潜在的虚假样本,并且我们使用原始序列读取数据手动确认假定重组样本子集中的突变(补充文本)7而且8,扩展数据表2和扩展数据图。3.)。在此之后,我们保留了589个独特的重组事件,这些事件总共有43104个后代样本(补充表1)。这意味着总样本SARS-CoV-2基因组中约2.7%被推断属于可检测的重组谱系。事后统计分析得出我们的统计阈值的经验错误发现率估计为11%9及扩展数据表3.)。此外,供体节点和受体节点的后代之间的地理位置和日期元数据的过度相似性支持了重组基因组的许多祖先在人类群体中共同循环的观点(补充文本)10而且11和扩展数据图。4而且5)。因为在基因相似的病毒谱系之间发生的重组事件难以检测(扩展数据图。2),我们的估计可能大大低估了重组的总体频率。因此,相对于SARS-CoV-2人群的全球重组频率,涟漪效应估计可能是保守的。
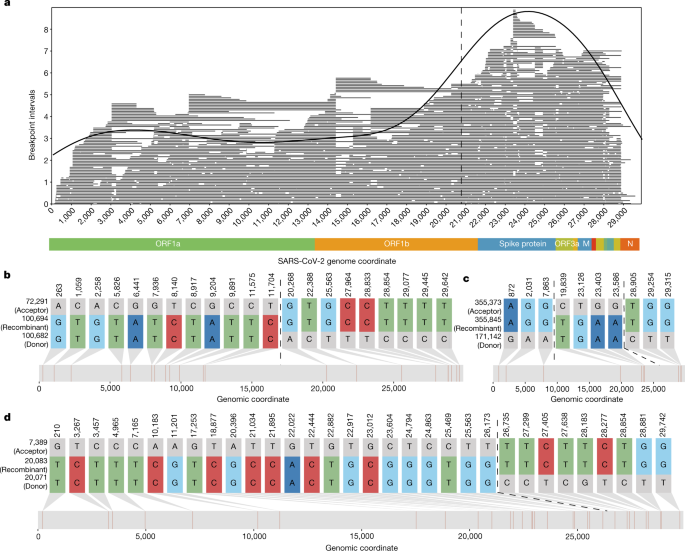
waves发现了SARS-CoV-2基因组中重组断点位置的显著不均匀分布,这与之前对β -冠状病毒的分析一致11,22.特别是,在假定的重组事件中,相对于基于随机断点位置的预期,朝向SARS-CoV-2基因组3'端的重组断点过多(P< 1 × 10−7;排列测试;补充的文本12)。值得注意的是,当我们模拟遵循均匀分布的重组断点时,这种偏差是不明显的13和扩展数据图。1)。突变点分析确定了穗蛋白区域立即5'的重组断点频率的增加(20875个碱基对;补充的文本14),当我们将自己限制在具有最大数量后代的假定节点和不同数据源之间时,这种模式是一致的,进一步表明它不是人为的(补充文本15及扩展数据表4)。假定的重组中断点的速率在变更点的3'处比5'区间高约3倍(图2)。2),这与其他人类冠状病毒基因组的相对重组率相似11.
一个,每个断点预测区间的中点分布以密度图的形式显示,底层重组预测区间以单独的灰色线表示。我们使用断点预测区间的中点,因为重组事件只能定位到预测区间,即两个具有重组信息的snp之间的区域。位置20875处的虚线划定了由变化点分析确定的重组率区域(补充文本)15)。对染色体边缘明显缺乏重组可能反映了检测偏差,这是我们上面描述的(扩展数据图。2)。b- - - - - -d,对于由waves检测到的三个示例重组三胞胎,重组信息位点(即重组节点与父节点中的任何一个匹配,但不是两个都匹配的位置)。每个序列左边的数字对应于MAT中的节点标识符。b而且d是具有单个断点的重组示例(虚线所示),c是具有两个断点的重组示例。b- - - - - -d使用SNIPIT包生成(https://github.com/aineniamh/snipit)。
几行证据表明,重组断点位置的倾斜分布不是宿主间传播动力学水平上的正向选择的结果。首先,这些重组进化支中的许多只存在了相对较短的时间,并且可能已经灭绝了。检测到的重组淋巴结的观察后代最早和最晚的日期之间的平均时间跨度仅为37天。第二,我们推断的重组事件子集发生在关注的变量之间(VOC;谱系B.1.1.7, B.1.351, B.1.617.2和P.1(参考。23)和其他谱系,VOCs对刺突蛋白突变的贡献平均略低于非VOC谱系(125个VOC/非VOC重组中有60个,P= 0.48,符号检验)。第三,重组分支的大小与其他分支的大小差别不大,这是预期的,如果重组谱系经历了强选择(P= 0.8470,排列检验)。因此,尽管重组世系宿主间传播动态的自然选择也会影响观察到的重组断点位置分布11,我们的数据表明,其他偏见影响了SARS-CoV-2基因组重组事件的分布。这可能包括影响重组断点分布的中性机械偏差。
尽管重组尚未在流行的SARS-CoV-2基因组中广泛传播,但重组已显著促进了SARS-CoV-2谱系的遗传多样性。由重组所贡献的可变位置比率(R)与由从头突变产生的基因(米),R/米,通常用来概括这两种变化源的相对影响22.利用我们的假定重组事件数据集,我们进行了估计R/米SARS-CoV-2 = 0.00264(补充文本16)。这对于冠状病毒人群来说是低的(例如,对于中东呼吸综合征,R/米估计为0.25-0.31(参考。22)),这大概反映了在大流行的最初阶段,可能的重组祖先之间的遗传多样性极低,以及我们的方法的保守性质。随着新冠病毒群体不断积累遗传多样性,并与其他病毒共同感染宿主,重组在产生功能性遗传多样性方面将发挥越来越大的作用,这一比例可能会增加24.因此,随着大流行的发展,waves将在检测新的重组谱系和量化其对病毒基因组多样性的影响方面发挥主要作用。
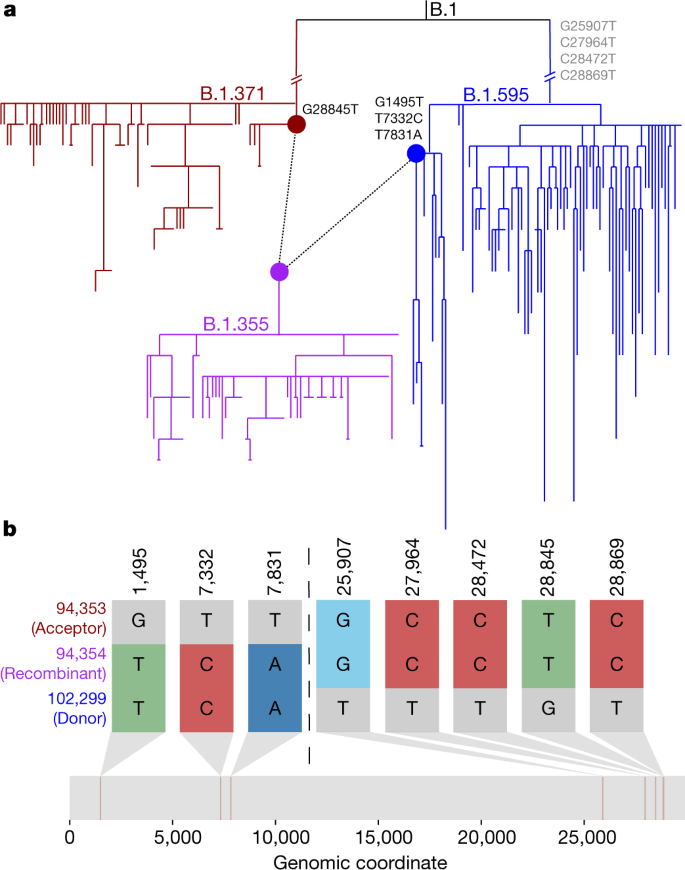
我们广泛优化的waves实现使其能够搜索整个系统进化树,并检测SARS-CoV-2谱系内部和之间的重组,而无需先验地定义一组谱系或分支定义突变。这是我们的方法相对于其他方法的一个关键优势,这些方法通过减少可能的重组事件的搜索空间来处理SARS-CoV-2数据集的规模(例如,参考文献。16,17,25)。waves在同一Pango谱系的分支中发现了223个重组事件。我们的结果还包括366个谱系间重组事件(补充表1)。此外,我们发现重组影响了穿山甲SARS-CoV-2命名系统的证据23.具体来说,我们发现B.1.355谱系的根可能是由属于B.1.595和B.1.371谱系的节点之间的重组事件引起的(图2)。3.及补充表1)。这些不同的重组事件突出了在《涟漪》中所采取的方法的多功能性和优势。
一个,亚系统发育由所有78个B.1.355样本(紫色)和最接近的78个样本分别来自谱系B.1.371和B.1.595的94,353和102,299节点组成,使用matUtils中的' k最近样本'函数20..节点94353(红色)和102299(蓝色)由虚线连接到节点94354(紫色),这是谱系B.1.355的根。含有重组信息的突变在系统发育中发生的地方被标记出来,在亲本中发生但不为重组序列所共享的突变以灰色显示。b,重组信息位点(即重组节点与任一父节点匹配但不与双亲节点都匹配的位点)按照与图相同的格式显示。两个罪犯.b是使用SNIPIT包生成的(https://github.com/aineniamh/snipit)。
在含有刺突蛋白的SARS-CoV-2基因组3'部分检测到重组率增加,突出了持续监测的实用性。刺突蛋白是病毒谱系功能新颖的主要位置,因为它们适应人类宿主内部和之间的传播。我们发现,特别是在刺突蛋白周围的重组事件过多,目前循环中的重组蛋白水平相对较高,这强调了通过实时分析病毒基因组,监测通过突变或重组产生的新病毒谱系进化的重要性。我们的工作还强调了明确考虑系统发育网络将对准确解释SARS-CoV-2序列产生的影响11.
除了SARS-CoV-2,重组是推动病毒和微生物适应的主要进化力量。它可以推动抗生素耐药性的传播7,耐药1,免疫和疫苗逃逸2.重组的鉴定是病原体进化分析管道的重要组成部分,因为重组会影响系统发育、传播和系统动力学推理的质量3..基于这些原因,近年来检测微生物重组的计算工具已经变得非常流行和重要4.SARS-CoV-2大流行推动了病原体基因组测序和数据共享的空前激增,这反过来凸显了当前软件在研究大型基因组数据集方面的一些局限性5.涟漪是为大流行规模的数据集构建的,并经过充分优化,可以在40分钟内彻底搜索有史以来推断的最大的系统发育之一的重组(补充文本)17)。我们预计waves在密集采样的基因组数据集上表现最好,这可能会成为许多全球分布的病原体的标准,但我们警告说,它还没有在其他物种上得到验证。为了方便实时分析全球不同研究小组每天产生的数万个新SARS-CoV-2序列的重组26,27,28, ripple提供了一个选项,可以在几分钟内评估任何用户提供的样本中重组血统的证据17)。因此,waves为在大量采样和快速进化的病原体群体中快速分析重组打开了大门,并为在大流行期间实时调查重组提供了工具。
方法
waves使用突变注释树(MATs)的高效空间数据结构20.,其中系统发育树的分支被注释为已经推断发生在它们身上的突变,以识别重组事件。数字1说明底层算法。waves识别假定的重组节点,包含用户指定的至少数量的突变,并通过计算从根开始的路径上分支上注释的所有突变,推断出在其相应序列上发生的突变集。然后,waves在突变位点上添加一个或两个断点,并使用与初始简约相比的部分放置来评估简约得分的提高。详情请参见补充文本1.为了确定假定的重组是否有意义,我们通过随机选择节点并添加,建立了一个零模型k从我们全局树的实际突变谱中提取的其他突变。然后,我们将这些样本放在树上,并使用waves来确定他们的节俭分数改进(补充文本)2)。对于我们全局树中的每个假定重组,我们将其简约分数改进与相同初始简约分数的零简约分数改进的分布进行比较3.)。我们首先采用2021年5月28日的公共树来开发我们的起始树19,20.,屏蔽所有有问题的网站29,以及修剪含有少于28000个非n核苷酸和含有两个或两个以上非[ACGTN-]核苷酸的样本(补充文本)5)。在此之后,我们通过运行matOptimize(补充文本)来优化该树4)两次,第一次子树修剪和再嫁接(SPR)半径为10,随后为40,并以蒙面变量调用格式(VCF)文件作为输入。使用waves的说明可在https://usher-wiki.readthedocs.io/en/latest/tutorials.html.我们在包含224个虚拟中央处理器(vcpu)的n2d-highcpu-224谷歌云平台实例上运行了waves(补充文本)18)。
为了测试waves的敏感性,我们从至少有10个后代的系统发育中随机选择两个内部节点,并在整个基因组中随机选择断点来模拟重组样本。我们分别为一个和两个断点重组生成了1000个模拟,在重组事件后,没有、一个、两个和三个额外的突变添加到序列中https://github.com/bpt26/recombination/.这些组合总共产生了2000个模拟重组谱系。然后,我们测量了作为断点位置函数的waves检测断点的能力以及重组节点到任一父节点的最小遗传距离(补充文本)6;遗传距离是根据推断出的分离病灶样本、谱系或节点的突变数量来估计的)。我们还通过确保ripple检测到Jackson等人的每一个高置信重组SARS-CoV-2聚类来评估其敏感性。16.
我们应用了几个事后过滤器来去除假定的重组节点,这些节点可能是由几个可能的错误源引起的假阳性。对于每个组成重组事件的三个内部节点(假定的重组节点、供体节点和受体节点)的每个内部节点,我们从COG-UK、GenBank、GISAID和中国生物信息学国家中心下载了每个节点最近的后代的共识基因组序列。然后,我们使用MAFFT对每个三人组的所有后代序列进行对齐30.该方法特别关注重组信息位点,也就是说,重组节点的等位基因与一个父节点匹配,而与另一个不匹配。如果重组信息突变靠近索引位点或缺失碱基,或者重组的整个碱基是20个核苷酸范围内的单簇突变(补充文本)7)。我们还通过手动检查10个样本的原始读取数据来确认序列质量,我们可以自信地将原始序列读取数据与给定的共识基因组联系起来(补充文本)8)。为了估计与我们选择的特定方法和统计阈值相关的错误发现率(FDR),我们计算了一个事后经验FDR。我们得到了我们测试的内部节点的数量,并与给定的简约分数相关联。然后,对于每个初始简约分数和简约分数的改进,我们获得了在null模型下显示简约分数改进的预期内部节点数。我们的扩展数据表3.)为给定初始和最终简约得分的期望节点数与检测到的具有相同初始和最终简约得分的重组节点数之比(补充文本)9)。
我们还使用样本元数据进行了事后分析,以确定重组节点的祖先是否具有比预期的更高的空间或时间重叠。我们将地理重叠计算为从供体节点和受体节点的后代中选择来自同一国家的样本的联合概率。对于时间重叠,我们分别记录来自供体和受体的最早到最近的样本间隔,并计算分隔这两个间隔的最小天数(重叠间隔为0)。对于每一个检测到的三元组,我们从树中选择两个随机的内部节点,它们的后代分别等于真正的供体和受体,从而为这两个类别生成一个空分布。然后,我们以同样的方式计算该随机集的地理和时间重叠(扩展数据图。4及补充文本10)。
为了确定已识别的重组断点是否显著地向基因组的3'端转移,我们进行了置换检验,比较均匀模拟断点分布的平均值与真实集中检测到的断点位置分布的平均值之间的差异(补充文本)12)。我们还使用变更点R包进行了变更点分析31并对重组预测区间中点计数进行泊松模型拟合。然后,我们计算了在已识别的变化点两侧的区间内重组断点的平均速率,以估计基因组3'部分重组率的倍增率(补充文本)13)。估计R/米,我们发现吝啬分数的下降与每个检测到的重组事件相关,作为估计R.然后我们计算米通过取这个值并从我们整个系统发育中观察到的突变总数中减去它(补充文本)16)。R/米是这些值的比值。
报告总结
有关研究设计的进一步资料,请参阅自然研究报告摘要链接到这篇文章。
数据可用性
所有资料均可在稿件或补充资料中查阅。数据集1(包含本研究中以Newick格式分析的重组系统发育)和数据集2(包含通过waves识别的重组节点的后代样本列表)可在以下地址获得https://doi.org/10.5281/zenodo.671737832.
代码的可用性
waves软件可在MIT许可下作为UShER软件包的一部分在https://github.com/yatisht/usher.我们提供了一个可复制的谷歌云平台工作流程https://github.com/yatisht/usher/tree/master/scripts/recombination.本研究中使用的特定代码和工作流的存档版本可从https://doi.org/10.5281/zenodo.6709991(ref。33)。我们使用UShER分发waves,因为它使用相同的底层数据对象,并且UShER需要推断输入MAT。waves和相关实用程序的文档可以在https://usher-wiki.readthedocs.io/en/latest/.
参考文献
Moutouh, L., Corbeil, J. & Richman, d.d .重组导致在选择性药物压力下快速出现HIV-1双耐药突变体。国家科学院学报美国93, 6106-6111(1996)。
Golubchik, T.等人。肺炎球菌基因组测序追踪通过多片段重组事件形成的疫苗逃逸变异。Nat,麝猫。44, 352-355(2012)。
史鲁普,M. H. & Hein, J.重组对传统系统发育分析的影响。遗传学156, 879-891(2000)。
Didelot, X. &威尔逊,D. J.克隆框架:有效推断重组在整个细菌基因组。公共科学图书馆第一版。医学杂志。11, e1004041(2015)。
霍德克罗夫特,e.b.等人。想更快地追踪大流行变异?解决生物信息学瓶颈。自然591, 30-33(2021)。
Forni, D., Cagliani, R. & Sironi, M.重组和阳性选择差异地塑造了β -冠状病毒亚属的多样性。病毒12, 1313(2020)。
李志刚,李志刚。重组对细菌进化的影响。Microbiol趋势。18, 315-322(2010)。
杜达斯,G. & Rambaut, A. MERS-CoV重组:对储层和适应潜力的影响。病毒的另一个星球2, vev023(2016)。
刘,s.k.p.等。严重急性呼吸综合征(SARS)冠状病毒ORF8蛋白是由大马蹄蝠的SARS相关冠状病毒通过重组获得的。j .性研究。89, 10532-10547(2015)。
霍姆斯,E. C.和兰姆波特,A.病毒进化和SARS冠状病毒的出现。费罗斯。反式。r . Soc。Lond。B杂志。科学。359, 1059-1065(2004)。
Müller, N. F., Kistler, K. E. & Bedford, T.冠状病毒的重组模式。预印在bioRxivhttps://doi.org/10.1101/2021.04.28.441806(2021)。
Bobay L.-M。,O’Donnell, A. C. & Ochman, H. Recombination events are concentrated in the spike protein region of betacoronaviruses.公共科学图书馆麝猫。16, e1009272(2020)。
李,X.等。SARS-CoV-2通过重组和强净化选择出现。科学。睡觉。6, eabb9153(2020)。
De Maio, N.等人。SARS-CoV-2同义突变的突变率和选择。基因组医学杂志。另一个星球。13, evab087(2021)。
Taghizadeh, P.等人。对伊朗SARS-CoV-2毒株的研究揭示了不同毒株的合并感染和重组对新毒株出现的潜在贡献。病毒学562, 63-73(2021)。
杰克逊等人。SARS-CoV-2大流行中谱系间重组的生成和传播。细胞184, 5179-5188(2021)。
VanInsberghe, D., Neish, A. S., Lowen, A. C.和Koelle, K.重组SARS-CoV-2基因组在大流行的第一年以低水平循环。病毒的另一个星球。7, veab059 (2021)
来自全基因组的细菌系统发育重建对重组是稳健的,但人口学推断不是。MBio5, e02158(2014)。
Turakhia, Y.等人。超快样本放置在现有的树(UShER)上,可以实现SARS-CoV-2大流行的实时系统发育。Nat,麝猫。53, 809-816(2021)。
McBroome, J.等。一个每日更新的数据库和工具,用于全面的SARS-CoV-2突变注释树。摩尔。杂志。另一个星球。38, 5819-5824(2021)。
关于不可检测的重组事件的频率。遗传学112, 923-926(1986)。
Patiño-Galindo, J. Á。,Filip, I. & Rabadan, R. Global patterns of recombination across human viruses.摩尔。杂志。另一个星球。38, 2520-2531(2021)。
兰姆波特等人。SARS-CoV-2谱系的动态命名建议以协助基因组流行病学。Microbiol Nat。5, 1403-1407(2020)。
Kim, D., Quinn, J., Pinsky, B., Shah, N. H.和Brown, I. SARS-CoV-2和其他呼吸道病原体的联合感染率。《美国医学会杂志》323, 2085-2086(2020)。
Varabyou, A., Pockrandt, C., Salzberg, S. L. & Pertea, M.用Bolotie快速检测SARS-CoV-2的分支间重组。遗传学218, iyab074(2021)。
Shu, Y.和McCauley, J. GISAID:分享所有流感数据的全球倡议——从愿景到现实。Eurosurveillance22, 30494(2017)。
塞耶斯,e.w.等人。基因库。核酸测定。49, d92-d96(2021)。
COVID-19基因组英国(COG-UK)联盟建立全国范围的新型冠状病毒基因组综合监测网络。柳叶刀的微生物1, e99-e100(2020)。
Turakhia, Y.等人。SARS-CoV-2系统发育的稳定性。公共科学图书馆麝猫。16, e1009175(2020)。
Katoh, K. & Standley, D. M. MAFFT多序列比对软件版本7:性能和可用性的改进。摩尔。杂志。另一个星球。30., 772-780(2013)。
Killick, R. & Eckley, I. changepoint:一个用于变更点分析的R包。J.统计。58, 19(2014)。
Turakhia, Y.等人。大流行尺度系统基因组学的补充揭示了SARS-CoV2重组的前景。Zenodohttps://doi.org/10.5281/zenodo.6717378(2022)。
Turakhia, Y. et al. yatisht/usher v0.5.6。Zenodohttps://doi.org/10.5281/zenodo.6709991(2022)。
确认
我们感谢作者和负责获取样本的实验室,以及通过GISAID生成和共享基因组数据的提交实验室(补充表)2)26(中国生物信息中心3.),英国COVID-19基因组公司(COG-UK)28(补充表4)和国家生物技术信息中心数据库27(补充表5),这是本研究的基础。我们感谢S. Mollenkamp对代码开发的协助。b.t., J.M.和R.C.-D。由美国国立卫生研究院资助R35GM128932。R.C.-D。由阿尔弗雷德·斯隆基金会奖学金和加州大学总统紧急COVID-19研究种子基金资助。R00RG2456。B.T.和J.M.是由国立卫生研究院资助的。T32HG008345。 B.T. was funded by the National Institutes of Health grant no. F31HG010584. N.D.M. was funded by the European Molecular Biology Laboratory. R.L. was funded by Australian Research Council grant no. DP200103151 and an Chan-Zuckerberg Initiative grant. Additional funding for this project was provided by Eric and Wendy Schmidt by recommendation of the Schmidt Futures programme.
作者信息
作者及隶属关系
贡献
R.C.-D。Y.T.开发了这种方法并撰写了手稿。R.C.-D。,Y.T., B.T. and R.L. designed experiments. Y.T., B.T., A.H. and N.D.M. conducted experiments. Y.T., B.T., A.H., J.M., N.A., K.S. and C.Y. developed code. R.C.-D. and D.H. supervised the group. Y.T., B.T., A.H., J.M., N.A., C.Y., N.D.M., D.H., R.L. and R.C.-D. edited the manuscript.
相应的作者
道德声明
相互竞争的利益
R.L.是GISAID的顾问。其余作者声明没有竞争利益。
同行评审
同行评审信息
自然感谢匿名审稿人对本工作的同行评议所作的贡献。同行评审报告是可用的。
额外的信息
出版商的注意施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
扩展的数据图形和表格
扩展数据图1推断和模拟重组断点位置的直方图。
一个)真实的模拟断点(红色)显示所有检测到的重组间隔中点(蓝色)。当蓝条的高度超过红条的高度时,这意味着相对于断点位置的真实检测率有一个多余的检测率。同样,当红色条形图的高度超过蓝色条形图的高度时,就意味着赤字。B)真实的模拟断点(红色)显示为20%最密切相关的供体-受体对(蓝色)检测到的重组间隔中点。在这两个比较中,我们通过选择最大的重组间隔,打破了相当改善的部分系统发育位置简约分数之间的联系。
扩展数据图2 waves更容易检测到导致简约分数大变化的断点。
为每个模拟样本检测到的模拟断点分布显示为一个)初始节俭分数和B)模拟样本到亲本的最小遗传距离。初始简约性(A)依赖于重组节点在树中的初始位置,指的是重组节点与其在系统发育中的直接亲本之间的突变遗传距离。样本到亲本的最小遗传距离(B)是指与重组相关的将重组节点与供体或受体分离的突变数量,不依赖于-初始系统发育。类似地,在由waves检测的模拟样本中,检测到的断点和未检测到的断点显示为C)初始节俭分数和D)与亲本的最小遗传距离。检测到的样本和断点以黑色显示,未检测到的样本和断点以红色显示。我们的条件是定位真正的断点和观察一个显著的简约分数根据我们的系统发育零模型。因此,我们排除了最小起始简约分数和遗传距离小于3的重组事件,因为这些在我们的零模型下不显著。
扩展数据图3由于序列质量问题而被过滤掉的检测到的三组例子。
一个来自经过筛选的重组节点77695、169585和77690的一致序列的部分比对,以位点28225为中心,具有跨越多个位点的大部分“N”的一致序列,这意味着重组事件的信息。当许多后代样本都有缺失数据时,就会出现这种情况。紧邻该区域的三个一致序列之间的不匹配也可能是测序质量差的结果。B来自经过筛选的重组三重奏节点173213、173209和173274的一致序列的部分比对,以位点16846为中心,在8个核苷酸窗口中有7个重组信息突变,这些突变不太可能是真正的突变事件,而是一种比对假象或复杂的indel事件。C来自经过筛选的293461、293460和211841节点的重组序列的部分比对,以29769位点为中心,在5个核苷酸窗口中有3个错配,旁边有一个大的比对间隙,不太可能是真正的突变。
图4重组祖先表现出增加的空间和时间重叠。
一个)空间及B)我们的重组三人组的时间重叠(蓝色)和零分布(灰色),顶部显示的重组祖先重叠的统计增加的曼-惠特尼秩和p值。
扩展数据图5重组体的祖先基因相似。
一个)假定的(红色)和模拟的(蓝色)重组样品放置的初始简约分数。B)推断的(红色)和模拟的(蓝色)祖先-供体对之间的遗传距离,从而产生假定的或模拟的重组。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问http://creativecommons.org/licenses/by/4.0/.
关于本文
引用本文
图拉希亚,Y.,索洛,B.,辛里奇,A.。et al。大流行尺度系统基因组学揭示SARS-CoV-2重组格局。自然609, 994-997(2022)。https://doi.org/10.1038/s41586-022-05189-9
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41586-022-05189-9