摘要gydF4y2Ba
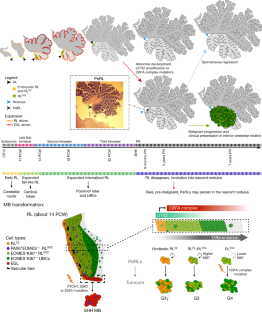
髓母细胞瘤(MB)包括一组异质性的后脑儿科胚胎性肿瘤,与后脑的早期发育有很强的联系gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.激活Sonic hedgehog信号的突变导致上菱形唇(RL)颗粒细胞谱系中的Sonic hedgehog MBgydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba.相比之下,激活WNT信号的突变导致低RL的WNT MBgydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba.然而,对更常见的4组(G4) MB知之甚少,它被认为出现在单极刷细胞谱系中gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba.在这里,我们证明了导致G4 MB的体细胞突变集中在核心结合因子α (CBFA)复合体和相互排斥的改变gydF4y2BaCBFA2T2gydF4y2Ba,gydF4y2BaCBFA2T3gydF4y2Ba,gydF4y2BaPRDM6gydF4y2Ba,gydF4y2Ba联合技术gydF4y2Ba和gydF4y2BaOTX2gydF4y2Ba.gydF4y2BaCBFA2T2gydF4y2Ba早在小脑RL室下区祖细胞中表达gydF4y2Ba智人gydF4y2Ba, G4 MB在转录上类似于这些祖细胞,但在发育时间停滞。击倒的gydF4y2BaOTX2gydF4y2Ba在模型系统中释放这种分化阻断,这允许MB细胞自发地沿着正常的发育分化轨迹进行分化。人类RL分裂的具体性质,注定会产生人类大脑中的大部分神经元,以及其高水平的易感eomegydF4y2Ba+gydF4y2BaKI67gydF4y2Ba+gydF4y2Ba单极刷状细胞祖细胞可能使我们的物种倾向于发展成g4mb。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
访问选项gydF4y2Ba
订阅《自然》+gydF4y2Ba
立即在线访问《自然》和其他55种《自然》杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅期刊gydF4y2Ba
获得1年的完整期刊访问权限gydF4y2Ba
199.00美元gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
增值税稍后将在结帐时添加。gydF4y2Ba
税务计算将在结账时完成。gydF4y2Ba
买条gydF4y2Ba
在ReadCube上获得时间限制或全文访问。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
数据可用性gydF4y2Ba
本研究中从MB肿瘤样本中生成的大量和scRNA-seq数据已在登录代码下存入欧洲基因组-表型组档案(EGA)数据库gydF4y2BaEGAS00001005826gydF4y2Ba.本研究中G3 MB细胞系HDMB03和MB3W1生成的bulk和scRNA-seq数据已根据访问代码保存在基因表达综合(GEO)数据库中gydF4y2BaGSE189238gydF4y2Ba和gydF4y2BaGSE200791gydF4y2Ba,分别。本研究中引用的已发表的MB bulk和scRNA-seq数据可在EGA数据库中查阅gydF4y2BaEGAD00001006305gydF4y2Ba,gydF4y2BaEGAD00001004435gydF4y2Ba,gydF4y2BaEGAD00001004958gydF4y2Ba,gydF4y2BaEGAS00001003170gydF4y2Ba和gydF4y2BaEGAS00001003368gydF4y2Ba.参考的GTEx正常小脑RNA-seq对照从NCBI公共库获得gydF4y2Baphs000424.v6.p1gydF4y2Ba.本研究中引用的Affymetrix SNP 6.0数据可在GEO数据库中获取gydF4y2BaGSE37385gydF4y2Ba.本研究中引用的全基因组测序数据可在EGA数据库中查阅gydF4y2BaEGAD00001003125gydF4y2Ba和gydF4y2BaEGAD00001004347gydF4y2Ba.研究期间引用的Illumina 450k甲基化数据可在GEO数据库中获取gydF4y2BaGSE85218gydF4y2Ba.研究期间引用的G3肿瘤球ChIP-seq数据可在GEO数据库中获取gydF4y2BaGSE132269gydF4y2Ba.本研究使用了多个数据库进行注释和筛选。这些包括外显子组聚合联盟/gnomAD (gydF4y2Bahttps://gnomad.broadinstitute.org/downloadsgydF4y2Ba)、NHLBI-ESP项目(gydF4y2Bahttps://esp.gs.washington.edu/drupal/gydF4y2Ba)、卡维亚基因变异资料库(gydF4y2Bahttp://db.systemsbiology.net/kaviar/: ~:文本= Kaviar % 20基因组% 20变体% 20 7 c % 20 snp数据库% 20%,和% 20频率% 20的% 20观察% 20变体。gydF4y2Ba)、单体型参考联盟(gydF4y2Bahttp://www.haplotype-reference-consortium.org/gydF4y2Ba)、大中东品种(gydF4y2Bahttp://igm.ucsd.edu/gme/gydF4y2Ba)、巴西基因组变异数据库(gydF4y2Bahttp://abraom.ib.usp.br/gydF4y2Ba)、雷达(gydF4y2Bahttp://rnaedit.com/gydF4y2Ba), GENCODE(第19节)(gydF4y2Bahttps://www.gencodegenes.org/human/release_19.htmlgydF4y2Ba), hs37d5参考基因组(gydF4y2Bahttps://ftp-trace.ncbi.nih.gov/1000genomes/ftp/technical/reference/phase2_reference_assembly_sequence/gydF4y2Ba), ERCC输入序列(gydF4y2Bahttps://www.encodeproject.org/files/ENCFF908UQN/gydF4y2Ba)和Caltech profile 3插入序列(gydF4y2Bahttps://www.encodeproject.org/references/ENCSR193ZXE/gydF4y2Ba).来自发育中的人类小脑的snRNA-seq数据是通过参考文献的通信获得的。gydF4y2Ba13gydF4y2Ba并可透过人类细胞图谱(gydF4y2Bahttps://www.covid19cellatlas.org/aldinger20gydF4y2Ba)、UCSC单元格浏览器(gydF4y2Bahttps://cbl-dev.cells.ucsc.edugydF4y2Ba)或从基因型和表型数据库(dbGaP)(登录号gydF4y2Baphs001908.v2.p1gydF4y2Ba).来自发育中的人类小脑的大量RNA-seq数据是通过参考文献的通信获得的。gydF4y2Ba12gydF4y2Ba并可通过dbGaP(登录号gydF4y2Baphs001908.v2.p1gydF4y2Ba).gydF4y2Ba源数据gydF4y2Ba提供了这篇论文。gydF4y2Ba
代码的可用性gydF4y2Ba
本研究中未使用自定义代码。只使用了开源算法,方法中详细介绍了它们的应用。关于如何使用这些算法的进一步详细信息,可根据要求从相应作者处获得。gydF4y2Ba
改变历史gydF4y2Ba
10月7日gydF4y2Ba
在这篇文章最初发布的版本中,参考文献37提供了错误的年份(2008年),在文章的HTML和PDF版本中已更正为1968年。gydF4y2Ba
11月29日gydF4y2Ba
对本文的更正已发表:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05578-0gydF4y2Ba
参考文献gydF4y2Ba
诺斯科特,p.a.等。成髓细胞瘤包括四种不同的分子变异。gydF4y2Baj .中国。肿瘤防治杂志。gydF4y2Ba29gydF4y2Ba, 1408-1414(2011)。gydF4y2Ba
卡沃利,F. M. G.等。成神经管细胞瘤亚组的肿瘤间质异质性。gydF4y2Ba癌症细胞gydF4y2Ba31gydF4y2Ba, 737 - 754。e6(2017)。gydF4y2Ba
Vladoiu, m.c.等人。儿童小脑肿瘤反映了保守的胎儿转录程序。gydF4y2Ba自然gydF4y2Ba572gydF4y2Ba, 67-73(2019)。gydF4y2Ba
Hovestadt, V.等人。用单细胞基因组学研究成神经管细胞瘤的细胞结构。gydF4y2Ba自然gydF4y2Ba572gydF4y2Ba, 74-79(2019)。gydF4y2Ba
韦氏-雷亚,R. J. &斯科特,M. P. Sonic hedgehog对小脑神经元前体增殖的控制。gydF4y2Ba神经元gydF4y2Ba22gydF4y2Ba, 103-114(1999)。gydF4y2Ba
purkinje细胞衍生的Sonic hedgehog基因调控发育中的小鼠小脑颗粒神经元前体细胞增殖。gydF4y2Ba咕咕叫。医学杂志。gydF4y2Ba9gydF4y2Ba, 445-448(1999)。gydF4y2Ba
杨,Z.-J。et al。髓母细胞瘤可由缺失修补的谱系限制祖细胞或干细胞引起。gydF4y2Ba癌症细胞gydF4y2Ba14gydF4y2Ba, 135-145(2008)。gydF4y2Ba
奥利弗,t.g.等人。髓母细胞瘤瘤前阶段颗粒细胞发育缺失和中断。gydF4y2Ba发展gydF4y2Ba132gydF4y2Ba, 2425-2439(2005)。gydF4y2Ba
吉布森等人。髓母细胞瘤的亚型有不同的发育起源。gydF4y2Ba自然gydF4y2Ba468gydF4y2Ba, 1095-1099(2010)。gydF4y2Ba
洁莎,S.等人。儿童脑瘤的根源在于发育停滞。gydF4y2BaNat,麝猫。gydF4y2Ba51gydF4y2Ba, 1702-1713(2019)。gydF4y2Ba
诺斯科特,p.a.等。成神经管细胞瘤亚型的全基因组图谱。gydF4y2Ba自然gydF4y2Ba547gydF4y2Ba, 311-317(2017)。gydF4y2Ba
Haldipur, P.等。发育中的人类小脑主要祖区时空扩展。gydF4y2Ba科学gydF4y2Ba366gydF4y2Ba, 454-460(2019)。gydF4y2Ba
阿尔丁格,K. A.等人。人类小脑发育的空间和细胞类型转录景观。gydF4y2BaNat。>。gydF4y2Ba24gydF4y2Ba, 1163-1175(2021)。gydF4y2Ba
分子亚群时代的成髓细胞瘤:综述。gydF4y2Baj . Neurosurg。Pediatr。gydF4y2Ba24gydF4y2Ba, 353-363(2019)。gydF4y2Ba
Tu, S.等。共抑制因子CBFA2T2调节多能性和种系发育。gydF4y2Ba自然gydF4y2Ba534gydF4y2Ba, 387-390(2016)。gydF4y2Ba
Nady, N.等人。ETO家族蛋白Mtgr1介导Prdm14在干细胞维持和原始生殖细胞形成中的功能。gydF4y2BaeLifegydF4y2Ba4gydF4y2Ba, e10150(2015)。gydF4y2Ba
特雷西,L. J.等。多能调节因子PRDM14需要造血调节因子CBFA2T3在小鼠中启动白血病。gydF4y2BaMol. Cancer Res。gydF4y2Ba17gydF4y2Ba, 1468-1479(2019)。gydF4y2Ba
鲍里斯,A.等人。转录辅抑制因子MTG8和MTG16调节肠干细胞从其生态位的退出以及分化为肠细胞和分泌细胞系。gydF4y2Ba胃肠病学gydF4y2Ba159gydF4y2Ba, 1328 - 1341。e3(2020)。gydF4y2Ba
Stadhouders, R.等人。通过联合共抑制因子作用控制发育启动的红系基因。gydF4y2BaCommun Nat。gydF4y2Ba6gydF4y2Ba, 8893(2015)。gydF4y2Ba
诺斯科特,p.a.等。1000个成神经管细胞瘤基因组的亚群特异性结构变异。gydF4y2Ba自然gydF4y2Ba488gydF4y2Ba, 49-56(2012)。gydF4y2Ba
Waszak, s.m.等人。超音刺猬髓母细胞瘤的生殖系伸长子突变。gydF4y2Ba自然gydF4y2Ba580gydF4y2Ba, 396-401(2020)。gydF4y2Ba
Badodi, S.等人。BMI1和CHD7在成神经管细胞瘤ERK信号通路中的收敛gydF4y2Ba细胞的代表。gydF4y2Ba21gydF4y2Ba, 2772-2784(2017)。gydF4y2Ba
Skowron, P.等。Shh成神经管细胞瘤的转录图谱。gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 1749(2021)。gydF4y2Ba
帕朗,B.等。转录辅抑制因子MTGR1调节肠道分泌谱系分配。gydF4y2Ba美国实验生物学学会联合会J。gydF4y2Ba29gydF4y2Ba, 786-795(2015)。gydF4y2Ba
布拉纳,t.c.等人。TurboID在活细胞和生物体中的高效接近标记。gydF4y2Ba生物科技Nat。》。gydF4y2Ba36gydF4y2Ba, 880-887(2018)。gydF4y2Ba
忘记吧,a等人。综合磷蛋白组分析显示,ERBB4-SRC信号异常是4组成神经管细胞瘤的标志。gydF4y2Ba癌症细胞gydF4y2Ba34gydF4y2Ba, 379 - 395。e7(2018)。gydF4y2Ba
Linggi, B. & Carpenter, G. ErbB-4 s80胞内结构域取消eto2依赖的转录抑制。gydF4y2Ba生物。化学。gydF4y2Ba281gydF4y2Ba, 25373-25380(2006)。gydF4y2Ba
诺斯科特,p.a.等。增强子劫持激活成神经管细胞瘤中的GFI1家族癌基因。gydF4y2Ba自然gydF4y2Ba511gydF4y2Ba, 428-434(2014)。gydF4y2Ba
Haldipur, P.等。Dandy-Walker畸形发病机制中菱形唇发育中断的证据。gydF4y2BaActa Neuropathol。gydF4y2Ba142gydF4y2Ba, 761-776(2021)。gydF4y2Ba
Haldipur, P.等。人小脑细胞增殖过程中Sonic hedgehog基因的表达。gydF4y2Ba阀杆。Dev细胞。gydF4y2Ba21gydF4y2Ba, 1059-1068(2012)。gydF4y2Ba
小脑的单极刷细胞产生于菱形唇,并通过发育中的白质迁移。gydF4y2Baj . >。gydF4y2Ba26gydF4y2Ba, 9184-9195(2006)。gydF4y2Ba
莱托,K.等。共识论文:小脑发育。gydF4y2Ba小脑gydF4y2Ba15gydF4y2Ba, 789-828(2016)。gydF4y2Ba
Stromecki, M.等人。新型otx2驱动干细胞程序在3组和4组成神经管细胞瘤中的特征。gydF4y2Ba摩尔。杂志。gydF4y2Ba12gydF4y2Ba, 495-513(2018)。gydF4y2Ba
Zagozewski, J.等人。OTX2-PAX3信号轴调控3组髓母细胞瘤细胞命运。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 3627(2020)。gydF4y2Ba
细胞位于第四脑室区域。gydF4y2BaAMA拱门。神经。精神病学gydF4y2Ba67gydF4y2Ba, 592(1952)。gydF4y2Ba
小脑后髓膜细胞异常聚集与成髓细胞瘤起源的关系。gydF4y2Ba拱门。神经。精神病学gydF4y2Ba52gydF4y2Ba, 163(1944)。gydF4y2Ba
罗克,L. B.,福格尔森,M. H. &里格斯,H. E.婴儿小脑异托位。gydF4y2Ba儿童神经科发展医学。gydF4y2Ba10gydF4y2Ba, 644-650(1968)。gydF4y2Ba
亚奇尼斯,A. T.,罗克,L. B.和Trojanowski, J. Q.人类小脑发育不良:发展及其与神经胶质和原始神经外胚层肿瘤的可能关系。gydF4y2Baj . Neuropathol。经验,神经。gydF4y2Ba53gydF4y2Ba, 61-71(1994)。gydF4y2Ba
杨俊,张勇。I-TASSER服务器:蛋白质结构和功能预测的新进展。gydF4y2Ba核酸测定。gydF4y2Ba43gydF4y2Ba, w174-w181(2015)。gydF4y2Ba
Mermel, C. H.等。GISTIC2.0在人类癌症中促进了对焦点体细胞拷贝数改变靶点的敏感和自信的定位。gydF4y2Ba基因组医学杂志。gydF4y2Ba12gydF4y2Ba, r41(2011)。gydF4y2Ba
Canisius, S, Martens, J. W. M. & Wessels, L. F. A.一项新的癌症体细胞改变的独立检验表明,生物学驱动互排性,但偶然性解释了大多数共发生。gydF4y2Ba基因组医学杂志。gydF4y2Ba17gydF4y2Ba, 261(2016)。gydF4y2Ba
菲纳克,G.等人。MAST:一种灵活的统计框架,用于评估单细胞RNA测序数据中的转录变化和表征异质性。gydF4y2Ba基因组医学杂志。gydF4y2Ba16gydF4y2Ba, 278(2015)。gydF4y2Ba
Ximerakis, M.等人。衰老小鼠大脑的单细胞转录组分析。gydF4y2BaNat。>。gydF4y2Ba22gydF4y2Ba, 1696-1708(2019)。gydF4y2Ba
Aibar, S.等。SCENIC:单细胞调控网络推断和聚类。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 1083-1086(2017)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
米尔德,T.等。HD-MB03是一种新型的3组成神经管细胞瘤模型,对组蛋白去乙酰化酶抑制剂治疗敏感。gydF4y2Baj . Neurooncol。gydF4y2Ba110gydF4y2Ba, 335-348(2012)。gydF4y2Ba
Dietl, S.等人。MB3W1是间变性髓母细胞瘤的原位异种移植模型,显示癌症干细胞和3组特性。gydF4y2BaBMC癌症gydF4y2Ba16gydF4y2Ba, 115(2016)。gydF4y2Ba
崔,H.等。SAINT:亲和纯化-质谱数据的概率评分。gydF4y2BaNat方法。gydF4y2Ba8gydF4y2Ba, 70-73(2011)。gydF4y2Ba
Teo, G.等人。SAINTexpress: INTeractome软件显著性分析的改进和附加特性。gydF4y2Baj .蛋白质组学gydF4y2BaOne hundred.gydF4y2Ba, 37-43(2014)。gydF4y2Ba
陈淑娟,周勇,陈勇,顾杰。fastp:一种超快一体化FASTQ预处理器。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba, i884-i890(2018)。gydF4y2Ba
Patro, R., Duggal, G., Love, M. I., Irizarry, R. A. & Kingsford, C. Salmon提供了快速和有偏差意识的转录表达量化。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 417-419(2017)。gydF4y2Ba
铃木,H.等。复发的非编码U1 snRNA突变驱动SHH成神经管细胞瘤的隐式剪接。gydF4y2Ba自然gydF4y2Ba574gydF4y2Ba, 707-711(2019)。gydF4y2Ba
Auwera, G. A.等。从FastQ数据到高置信度变量调用:基因组分析工具包最佳实践管道。gydF4y2Ba咕咕叫。Protoc。生物信息学gydF4y2Ba43gydF4y2Ba, 11.10.1-11.10.33(2013)。gydF4y2Ba
王凯,李明,王凯,李敏,韩康生。ANNOVAR:基于高通量测序数据的基因变异功能注释。gydF4y2Ba核酸测定。gydF4y2Ba38gydF4y2Ba, e164(2010)。gydF4y2Ba
劳伦斯,m.s.等人。癌症的突变异质性和寻找新的癌症相关基因。gydF4y2Ba自然gydF4y2Ba499gydF4y2Ba, 214-218(2013)。gydF4y2Ba
Mularoni, L., Sabarinathan, R., Deu-Pons, J., Gonzalez-Perez, a . & López-Bigas, N. OncodriveFML:识别癌症驱动突变的编码和非编码区域的一般框架。gydF4y2Ba基因组医学杂志。gydF4y2Ba17gydF4y2Ba, 128(2016)。gydF4y2Ba
哈斯,B. J.等。基于读映射和从头融合转录本组装方法的融合转录本检测准确性评估。gydF4y2Ba基因组医学杂志。gydF4y2Ba20.gydF4y2Ba, 213(2019)。gydF4y2Ba
Okonechnikov, K.等。灌注:从深度rna测序数据中发现融合基因和嵌合转录本。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba11gydF4y2Ba, e0167417(2016)。gydF4y2Ba
罗伯逊,G.等人。RNA-seq数据的从头组装和分析。gydF4y2BaNat方法。gydF4y2Ba7gydF4y2Ba, 909-912(2010)。gydF4y2Ba
郑国勇,等。大规模并行数字单细胞转录谱分析。gydF4y2BaCommun Nat。gydF4y2Ba8gydF4y2Ba, 14049(2017)。gydF4y2Ba
Hao, Y.等。多模态单细胞数据综合分析。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 3573 - 3587。e29(2021)。gydF4y2Ba
Hafemeister, C. & Satija, R.使用正则化负二项回归的单细胞RNA-seq数据归一化和方差稳定。gydF4y2Ba基因组医学杂志。gydF4y2Ba20.gydF4y2Ba, 296(2019)。gydF4y2Ba
帕特尔,a.p.等人。单细胞RNA-seq在原发性胶质母细胞瘤中显示瘤内异质性。gydF4y2Ba科学gydF4y2Ba344gydF4y2Ba, 1396-1401(2014)。gydF4y2Ba
Khazaei, S.等人。H3.3 G34W在骨巨细胞瘤中促进成骨样间充质祖细胞的生长并阻碍其分化。gydF4y2Ba癌症。gydF4y2Ba10gydF4y2Ba, 1968-1987(2020)。gydF4y2Ba
史崔特,K.等人。弹弓:单细胞转录组学的细胞谱系和伪时间推断。gydF4y2BaBMC基因组学gydF4y2Ba19gydF4y2Ba, 477(2018)。gydF4y2Ba
命运:R中大规模单细胞数据的扩散图。gydF4y2Ba生物信息学gydF4y2Ba32gydF4y2Ba, 1241-1243(2016)。gydF4y2Ba
阿兰,D.等人。基于参考的肺单细胞测序分析揭示了一个过渡性前纤维化巨噬细胞。gydF4y2BaImmunol Nat。gydF4y2Ba20.gydF4y2Ba, 163-172(2019)。gydF4y2Ba
Kowalczyk, m.s.等人。单细胞RNA-seq揭示了造血干细胞衰老过程中细胞周期和分化程序的变化。gydF4y2Ba基因组Res。gydF4y2Ba25gydF4y2Ba, 1860-1872(2015)。gydF4y2Ba
范纳,R. J.等。静Sox2gydF4y2Ba+gydF4y2Ba在Sonic hedgehog亚群成神经管细胞瘤中,细胞驱动分级生长和复发。gydF4y2Ba癌症细胞gydF4y2Ba26gydF4y2Ba, 33-47(2014)。gydF4y2Ba
纽曼等人。从组织表达谱中稳健地枚举细胞亚群。gydF4y2BaNat方法。gydF4y2Ba12gydF4y2Ba, 453-457(2015)。gydF4y2Ba
Van de Sande, B.等。用于单细胞基因调控网络分析的可伸缩的SCENIC工作流程。gydF4y2BaProtoc Nat。gydF4y2Ba15gydF4y2Ba, 2247-2276(2020)。gydF4y2Ba
纳吉,C.等。重度抑郁症患者前额叶皮层的单核转录组涉及少突胶质细胞、前体细胞和兴奋性神经元。gydF4y2BaNat。>。gydF4y2Ba23gydF4y2Ba, 771-781(2020)。gydF4y2Ba
古拉蒂,G. S.等。单细胞转录多样性是发育潜力的标志。gydF4y2Ba科学gydF4y2Ba367gydF4y2Ba, 405-411(2020)。gydF4y2Ba
Van den Berge, K.等人。单细胞测序数据基于轨迹的差异表达分析。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 1201(2020)。gydF4y2Ba
R.生存分析软件包3.4-0gydF4y2Bahttps://CRAN.R-project.org/package=survivalgydF4y2Ba(2022)。gydF4y2Ba
魏,Y.等。来自辅抑制因子ETO的taf4同源结构域是转录正、负调控因子的对接平台。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba14gydF4y2Ba, 653-661(2007)。gydF4y2Ba
Szklarczyk, D.等。2021年的STRING数据库:可定制的蛋白质-蛋白质网络,以及用户上传的基因/测量集的功能表征。gydF4y2Ba核酸测定。gydF4y2Ba49gydF4y2Ba, d605-d612(2021)。gydF4y2Ba
Merico, D., Isserlin, R., Stueker, O., Emili, a . & Bader, G. D.富集图:一种基于网络的基因集富集可视化和解释方法。gydF4y2Ba《公共科学图书馆•综合》gydF4y2Ba5gydF4y2Ba, e13984(2010)。gydF4y2Ba
细胞景观:生物分子相互作用网络集成模型的软件环境。gydF4y2Ba基因组Res。gydF4y2Ba13gydF4y2Ba, 2498-2504(2003)。gydF4y2Ba
顾z, Eils, R. & Schlesner, M.复杂热图揭示多维基因组数据的模式和相关性。gydF4y2Ba生物信息学gydF4y2Ba32gydF4y2Ba, 2847-2849(2016)。gydF4y2Ba
周,等。利用ProteinPaint探索儿童癌症的基因组改变。gydF4y2BaNat,麝猫。gydF4y2Ba48gydF4y2Ba4-6(2016)。gydF4y2Ba
Penas, C.等人。细胞周期退出的时间序列建模确定了Brd4依赖的小脑神经发生调节。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 3028(2019)。gydF4y2Ba
Reimand, J., Kull, M., Peterson, H., Hansen, J. & Vilo, J. g: profiler -一个基于网络的工具集,用于从大规模实验中分析基因列表的功能。gydF4y2Ba核酸测定。gydF4y2Ba35gydF4y2Ba, w193-w200(2007)。gydF4y2Ba
确认gydF4y2Ba
M.D.T.由美国国立卫生研究院(R01NS106155, R01CA159859和R01CA255369),儿童脑瘤基金会,特里福克斯研究所,加拿大卫生研究所,治愈搜索基金会,马修拉尔森基金会(IronMatt), b.r.a.i.n.child,梅根的步行,SWIFTY基金会,脑瘤慈善机构,加拿大基因组,BC基因组,魁北克基因组,安大略省研究基金,全球癌症研究,v癌症研究基金会和安大略癌症研究所通过安大略政府提供的资金。M.D.T.还得到了加拿大癌症协会研究所影响基金、英国癌症研究中心脑肿瘤奖和对抗癌症(SU2C)圣鲍德瑞克儿科梦之队转化研究基金(SU2C- aacr - dt1113)和SU2C加拿大癌症干细胞梦之队研究基金(SU2C- aacr - dt -19-15)的支持,该基金由加拿大政府通过加拿大基因组研究所和加拿大卫生研究所提供。安大略癌症研究所通过安大略政府提供的资金提供补充支持。“对抗癌症”是由美国癌症研究协会管理的娱乐产业基金会的一个项目。M.D.T.还得到了病童医院和多伦多大学儿童癌症研究加伦家庭主席的支持。K.J.M.支持NIH-R01-NS080390、R01-NS095733和R37 NS095733。T.E.W.-O。和B. Doble的研究得到了加拿大健康研究所(CIHR)和马尼托巴癌症护理基金会的支持。H.S.由日本科学促进协会(JSPS) KAKENHI (21K21001)和国家癌症中心研究与发展基金(2021-A-1)支持。 C.L.K is supported by CIHR (PJT-156086), salary awards from Fonds de Recherche du Québec-Santé (FRQS) and NSERC (RGPIN-2016-04911). P. Haldipur is supported by NIH-R21 NS117848, Brain and Behavior Young Investigator Award. We also thank the staff at Human Developmental Biology Resource London and Newcastle for their technical help and support; B. Pakuts for support with graphic design and illustrations; the Fernando Goldsztein Family for supporting this research; and L. Rorke for thoughtful discussions on cerebellar dysplasia.
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
l.d.h., P. Haldipur和O. Saulnier设计、执行和分析了本研究中的大部分实验。j.m., A.H.S.和J.G.参与了原位杂交和细胞计数分析。a.w.e对生物信息学分析和SCENIC工作流程做出了贡献。W.O.和D.S.S.对单核测序的细胞系制备有贡献。W.O.和M.C.V.对初级组织分离、单细胞制备和测序做出了贡献。V.G.和l.c.m。参与TurboID实验和OTX2-KD实验。A.L.M.参与了GCP的分离、DN-CBFA2T3结构的设计和过表达。M.S.和t.p.b。为融合CBFA2T2-TurboID结构设计做出了贡献。 M.L., R.A.S. and D. S. Scott contributed toCBFA2T2gydF4y2Ba突变验证。S.B.有助于western blot验证。H.F.对SNP6拷贝数分析做出了贡献。T.N.和S.N.对突变分析有贡献。A.B.对通路分析有贡献。J.J.D.-M。有助于scRNA-seq数据的CNV分析。K.B.对分析和模型设计做出了贡献。P.S.和E.Y.W.对RNA-seq融合调用和分析做出了贡献。O. Sirbu, s.a.k., p.b., a.v., J.J.Y.L, r.a.,陈鑫,陈晓娣和K.L.M.对实验,统计,生物信息学分析和/或解释有贡献。 B. Luu, P. Bérubé, Y.C.W., A.S.M. and X.W. contributed to RNA-seq library preparation. S.M.P., N.J., S.-K.K., F.D., O.D., F.B., J.M.-P., W.A.G., J.L., P.D., M.F.-M., A.J., P.J.F., J.M.K., K.Z., S.D.B., C.G.E., A.A.N.R., C.G., J.M.O., M.G., P. Hauser, J.J.P., Y.S.R., C.d.T., J. Mora, K.K.W.L., H.-K.N., W.S.P., I.F.P., E.L.-A., G.Y.G., T.E.V.M., T.S., R.V., R.C.T., M.K.C., J.B.R., T.K., S.J., B. Lach, A.I., V.F., P.d.A., M.Z., G.C., S.R., D. S. Stearn, E.G.V.M., P.P., G.F., M.M., C.G.C., C.C.F., M.F.R., F. Boop, J.A.C., R.E.M., E.M.T., M.A., M.L.G., F.C., P.E. and M.P. provided patient tumour material and helped design the study. K.A.A., A.S.M., F.M.G.C., J.N.R., S.J.M.J., R.A.M., M.A.M., X.H., J.R., P.H.S., R.J.W.-R., W.A.W., T.J.P., L.G., C.L.K., L.D.S., N.J., D.M. and O.A. helped analyse data and provided expert advice. F.R. and E.S. provided late-gestation human samples. X.W. and C.D. provided reagents, equipment and expert advice. D.W.E. and J.A.G. provided expert neuropathology analysis and images. T.E.W.-O. and B.D. contributed to and provided expert advice on TurboID and OTX2-KD experiments. V.R. provided MRI images and provided expert advice on their interpretation. H.S., T.E.W.-O., K.J.M. and M.D.T. jointly supervised the project. L.D.H., P.H. and M.D.T. prepared the figures and wrote the manuscript.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢匿名审稿人对本工作的同行评议所作的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
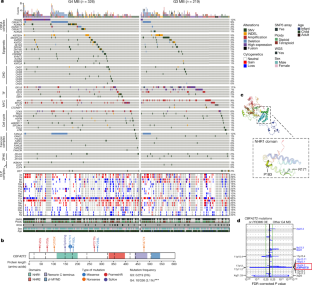
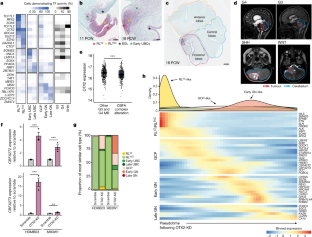
图1在G3和G4 MB中,针对相同基因的突变和拷贝数损失是互斥的。gydF4y2Ba
A b cgydF4y2Ba,总结冠心病相应基因组位点(染色体臂)基因突变和拷贝数损失的oncopprint (gydF4y2Ba一个gydF4y2Ba), fanc (gydF4y2BabgydF4y2Ba)及ELP (gydF4y2BacgydF4y2Ba)司机家庭。突变的频率低于拷贝数损失,但往往是独立发生的,这表明它们是缺失的目标。gydF4y2BadgydF4y2Ba, CHD、FANC和ELP家族靶向基因突变和拷贝数丢失事件的互斥性。gydF4y2BaegydF4y2Ba,针对CHD、FANC或ELP基因的事件重叠。大多数g4mb肿瘤通过单个缺失事件导致至少两个家族的基因单倍不足。gydF4y2Ba
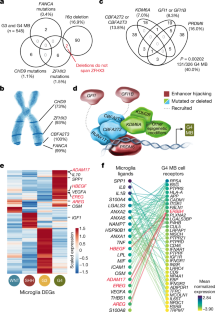
扩展数据图2 .人类基因组中G4 MB驱动基因的聚类使人类易于发展MB。gydF4y2Ba
一个gydF4y2Ba,漫画的gydF4y2Ba智人gydF4y2Ba基因组中已知的和新发现的G3和G4 MB候选驱动基因的位置显示了G3和G4 MB中已知缺失位置的基因聚类。gydF4y2BabgydF4y2Ba,全染色体臂缺失的频率与驱动基因家族的数量显著相关,具体见(gydF4y2Ba一个gydF4y2Ba)——装在手臂上。采用双侧线性回归模型评估显著性;灰色阴影区域表示95%置信区间。gydF4y2BacgydF4y2Ba,染色体臂17p、16q和8q拷贝数损失的互斥性。使用互斥性杂质测试评估显著性,在R包DISCOVER中实现gydF4y2Ba41gydF4y2Ba.gydF4y2Ba
扩展数据图3gydF4y2BaCBFA2T3gydF4y2Ba是g4mb肿瘤抑制基因。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BaPRDM6gydF4y2Ba表达gydF4y2BaCBFA2T2gydF4y2Ba突变体(红色)和gydF4y2BaCBFA2T2gydF4y2BaWT(灰色)G3和G4 MB样本表明增强子劫持介导gydF4y2BaPRDM6gydF4y2Ba表达在很大程度上限于gydF4y2BaCBFA2T2gydF4y2BaWT病例。gydF4y2BabgydF4y2Ba在G3和G4 MB病例中,沿着人类染色体16q染色体增益和损失区域的密度,表明缺失偏向于16q端粒末端,特别是已知驱动的位置gydF4y2BaCBFA2T3gydF4y2Ba.gydF4y2BacgydF4y2Ba,gydF4y2BaCBFA2T3gydF4y2Ba加和不加的样品之间的表达差异gydF4y2BaCBFA2T3gydF4y2Ba删除,按子组分割。采用Kruskal-Wallis秩和检验(FDR < 0.05), ***gydF4y2BapgydF4y2Ba< 0.0005, g3,gydF4y2BapgydF4y2Bae = 2.88gydF4y2Ba−05gydF4y2Ba;G4,gydF4y2BapgydF4y2Bae = 2.60gydF4y2Ba−09年gydF4y2Ba.G3,gydF4y2BangydF4y2Ba= 112;G4,gydF4y2BangydF4y2Ba= 206。gydF4y2BaCBFA2T3gydF4y2Ba是g4mb中一个拷贝数响应性肿瘤抑制基因。gydF4y2BadgydF4y2BaIGV分析显示2个G4 MB样本MB-0364和MB-0559为病灶缺失区。MB-0364是G3和G4 MB中16q上的最小公共删除区域(minimum common deleted region, MCDR),但在GISTIC分析中不太具有统计学意义。MB-0559是在GISTIC分析中达到统计显著性的MCDR。gydF4y2BaCBFA2T3gydF4y2Ba用一个红框标识。gydF4y2BaegydF4y2Ba,说明MCDR概念的漫画。gydF4y2BafgydF4y2Ba拷贝中性或半合子缺失的G3和G4 MB样本在chr16q24.3上MB-0364 MCDR内基因的表达差异。采用双侧Mann-Whitney U检验及FDR校正,*评估统计学意义gydF4y2BapgydF4y2Ba< 0.05, ***gydF4y2BapgydF4y2Ba< 0.0005。删除,gydF4y2BangydF4y2Ba= 86;中性的,gydF4y2BangydF4y2Ba= 232。gydF4y2BaegydF4y2Ba,拷贝中性或半合子缺失的G3和G4 MB样本中MB-0559 MCDR中chr16q24.3基因的表达差异。采用双侧Mann-Whitney U检验及FDR校正,*评估统计学意义gydF4y2BapgydF4y2Ba< 0.05, ***gydF4y2BapgydF4y2Ba< 0.0005。删除,gydF4y2BangydF4y2Ba= 86;中性的,gydF4y2BangydF4y2Ba= 232。一份完整的gydF4y2BapgydF4y2Ba中所示的基因值gydF4y2Ba(f)gydF4y2Ba和gydF4y2Ba(g)gydF4y2Ba可在补充数据表中找到gydF4y2Ba1gydF4y2Ba.gydF4y2BahgydF4y2Ba,gydF4y2BaCBFA2T2gydF4y2Ba(左)和gydF4y2BaCBFA2T3gydF4y2Ba(右)批量RNAseq在SHH、G3和G4 MB中的表达。采用Kruskal-Wallis秩和检验(FDR < 0.05), *gydF4y2BapgydF4y2Ba< 0.05, ***gydF4y2BapgydF4y2Ba< 0.0005。为gydF4y2BaCBFA2T2gydF4y2Ba: SHH-G3,gydF4y2BapgydF4y2Bae = 2.29gydF4y2Ba−47gydF4y2Ba;SHH-G4,gydF4y2BapgydF4y2Bae = 4.42gydF4y2Ba−73gydF4y2Ba;G3-G4,gydF4y2BapgydF4y2Ba= 0.035。为gydF4y2BaCBFA2T3gydF4y2Ba: SHH-G3,gydF4y2BapgydF4y2Bae = 7.10gydF4y2Ba−42gydF4y2Ba;SHH-G4,gydF4y2BapgydF4y2Bae = 1.13gydF4y2Ba−46gydF4y2Ba;G3-G4,gydF4y2BapgydF4y2Ba= 0.61。G3,gydF4y2BangydF4y2Ba= 219;G4,gydF4y2BangydF4y2Ba= 326;嘘,gydF4y2BangydF4y2Ba= 250。而gydF4y2BaCBFA2T2gydF4y2Ba和gydF4y2BaCBFA2T3gydF4y2Ba在G3和G4 MB中具有低表达,在SHH MB中观察到这两种基因的高表达和无改变。gydF4y2BaCBFA2T2gydF4y2Ba和gydF4y2BaCBFA2T3gydF4y2Ba与G3和G4 MB相比,SHH MB可能有不同的作用gydF4y2BacgydF4y2Ba,gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2Ba,gydF4y2BahgydF4y2Ba箱形图显示中位数和四分位范围,胡须显示数据范围。超出此范围的点是异常值,并单独绘制。gydF4y2Ba
CBFA多蛋白复合物包含多个已知的和新的G4 MB驱动基因。gydF4y2Ba
一个gydF4y2BaWestern blot结果显示,当TurboID结构体融合到CBFA2T2的n端,而没有融合到c端时,TurboID-CBFA2T2融合蛋白成功表达。gydF4y2BabgydF4y2Ba,新型CBFA2T2相互作用蛋白的蛋白-蛋白相互作用(PPI)网络。每个节点代表一个蛋白质,蛋白质之间的边代表已知的或新的ppi。红色边表示CBFA2T2相互作用蛋白之间的已知相互作用,绿色边表示与CBFA2T2的已知相互作用,这些作用在我们的TurboID屏幕中得到了总结。如果蛋白质之间包含已知的相互作用,则用虚线分组。gydF4y2BacgydF4y2BaCBFA2T2猎物蛋白在各生物过程中富集显著。GLI2是一个SHH致癌基因,最近已被证明维持GCP增殖和身份,涉及CBFA复合体gydF4y2Ba82gydF4y2Ba.gydF4y2BadgydF4y2Ba, TurboID富集CBFA2T2猎物蛋白的生物过程富集图(GO:BP)。每个节点代表一条显著富集的通路,边代表节点间共享的基因。节点被分组和标记为一个共同的生物学主题。使用G:Profiler评估显著性gydF4y2Ba83gydF4y2Ba罗斯福修正。gydF4y2BaegydF4y2Ba, CBFA2T2 TurboID蛋白和G3/G4 MB驱动基因的蛋白-蛋白相互作用(PPI)网络(图;gydF4y2Ba1gydF4y2Ba)使用STRINGgydF4y2Ba41gydF4y2Ba.为了简单起见,没有绘制CBFA2T2和菱形节点之间的边。连通性显著性由STRING评估,gydF4y2BapgydF4y2Ba< 0.1 egydF4y2Ba−16gydF4y2Ba.gydF4y2Ba
扩展数据图5 CBFA复合体的破坏解释了大多数G4 MB肿瘤。gydF4y2Ba
一个gydF4y2Ba,我们队列中G3和G4 MB的百分比(gydF4y2BangydF4y2Ba= 545),这是由基因改变导致的gydF4y2BaCBFA2T2gydF4y2Ba使用已知的或新的PPI(扩展数据图中显示的网络中的一个步骤)。gydF4y2Ba4 egydF4y2Ba).使用R包DISCOVER中实现的互斥性杂质测试评估的显著性gydF4y2Ba41gydF4y2Ba.gydF4y2BabgydF4y2Ba,排名表达gydF4y2BaHBEGFgydF4y2Ba(左)和gydF4y2BaEREGgydF4y2Ba(右)在g4mb内(gydF4y2BangydF4y2Ba= 326)。点由已知CBFA复合改变的存在(红色)或不存在(灰色)着色。表达最高的样本gydF4y2BaHBEGFgydF4y2Ba和gydF4y2BaEREGgydF4y2Ba通常没有CBFA复合物的改变,这表明CBFA复合物抑制的另一种机制。资料载于gydF4y2Ba一个gydF4y2Ba不进行重复试验。gydF4y2BacgydF4y2BaG3和G4 MB中中断通路的总结。改变基因按通路分组,并用改变频率标记。gydF4y2BadgydF4y2Ba(左)17 PCW人小脑的h&e染色中矢状面。(右)荧光免疫组化(IHC)显示gydF4y2BaKI67gydF4y2Ba和gydF4y2BaSOX2gydF4y2Ba人类RL区室的表达。资料载于gydF4y2BadgydF4y2Ba不进行重复试验。RLgydF4y2BaVZgydF4y2Ba和RLgydF4y2BaSVZgydF4y2Ba分别用红色和黄色星号表示。比例尺:100µm。gydF4y2Ba
扩展数据图6gydF4y2BaLMX1AgydF4y2Ba表达区分了RL的两个下游谱系gydF4y2BaSVZgydF4y2Ba.gydF4y2Ba
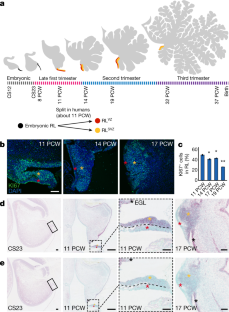
一个gydF4y2Ba,(左上)gydF4y2Ba原位gydF4y2Ba杂交(ISH)显示gydF4y2BaMKI67gydF4y2Ba表达式。In-set突出显示发育中的小脑,RL由黑盒子表示。(其他图像)发育中的人类小脑苏木精和伊红(H&E)染色正中矢状切面。其中,菱形唇由黑色方框表示。比例尺:500µm。gydF4y2BabgydF4y2Ba在17 PCW时,GFAP在发育中的人RL中表达。比例尺:100µm。的RLgydF4y2BaVZgydF4y2Ba和RLgydF4y2BaSVZgydF4y2Ba由血管丛在物理上划分,如白色星号所示。gydF4y2BacgydF4y2Ba的空间分辨RNA表达gydF4y2BaHBEGFgydF4y2Ba在发育中的人类小脑中。比例尺:50µm。gydF4y2BaHBEGFgydF4y2Ba病灶沿RL血管丛富集。gydF4y2BadgydF4y2Ba在19 PCW时,KI67在发育中的人RL中的表达。比例尺:100µm。gydF4y2BaegydF4y2Ba,出生后9个月人小脑的h&e染色中矢状面。比例尺:500µm。RL只在妊娠期出现,出生前后消失。gydF4y2Baf, ggydF4y2Ba的空间分辨RNA表达gydF4y2BaCBFA2T2gydF4y2Ba(gydF4y2BafgydF4y2Ba),gydF4y2BaCBFA2T3gydF4y2Ba(gydF4y2BaggydF4y2Ba)在发育中的人类小脑中的14pcw。比例尺:100µm。gydF4y2Bah,我gydF4y2Ba的空间分辨RNA表达gydF4y2BaCbfa2t2gydF4y2Ba(gydF4y2BahgydF4y2Ba),gydF4y2BaCbfa2t3gydF4y2Ba(gydF4y2Ba我gydF4y2Ba)在发育中的小鼠小脑的E15.5(左)和E16.5(右)。比例尺:100µm。我们在小鼠RL中没有观察到与人类RL相似的任何一种基因的表达模式,并注意到EGL中表达的富集,与人类相似。gydF4y2BajgydF4y2Ba的空间分辨RNA表达gydF4y2BaLMX1AgydF4y2Ba在人类小脑发育的11、14和17个PCW。gydF4y2BaLMX1AgydF4y2Ba在RL和RL中都高度表达gydF4y2BaVZgydF4y2Ba和RLgydF4y2BaSVZgydF4y2Ba,但gydF4y2BaLMX1AgydF4y2Ba该表达仅在从RL迁移的UBCs中保留,而在迁移到EGL的GCPs中完全不存在。资料载于gydF4y2BadgydF4y2Ba是来自三个独立实验的具有类似结果的代表性图像,其余组的数据没有在重复中进行。gydF4y2Ba
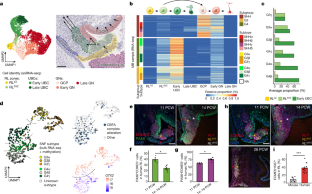
扩展数据图7用于MB与人类小脑发育之间转录映射的单细胞特征。gydF4y2Ba
一个gydF4y2Ba,显示发育中的人类小脑中RL谷氨酸能细胞类型特征标记基因表达的点图gydF4y2Ba13gydF4y2Ba.gydF4y2BabgydF4y2Ba, UMAP嵌入由Slingshot推断的伪时间着色gydF4y2Ba65gydF4y2Ba,其中,颗粒细胞谱系(左)和UBC细胞谱系(右)的伪时间方向是从深色到浅色。gydF4y2BacgydF4y2Ba,表示gydF4y2BaCBFA2T2gydF4y2Ba(左)和gydF4y2BaCBFA2T3gydF4y2Ba(右)在发育中的人类RL的每个区域通过批量RNAseqgydF4y2Ba12gydF4y2Ba.采用双侧Mann-Whitney U检验评估统计学意义,*gydF4y2BapgydF4y2Ba< 0.05;gydF4y2BaCBFA2T2gydF4y2Ba,gydF4y2BapgydF4y2Ba= 0.0078;gydF4y2BaCBFA2T3gydF4y2Ba,gydF4y2BapgydF4y2Ba= 0.0056。gydF4y2BangydF4y2Ba=每个区9个生物样本,在9至19个PCW之间获得。箱形图显示中位数和四分位范围,胡须显示数据范围。超出此范围的点是异常值,并单独绘制。gydF4y2BadgydF4y2Ba, 63296个来自G3的单细胞UMAP包埋(gydF4y2BangydF4y2Ba= 6), g4 (gydF4y2BangydF4y2Ba= 11), SHH (gydF4y2BangydF4y2Ba= 3) MB scRNAseq样本。转录相似的细胞簇被肿瘤样本着色和标记,或标注非肿瘤细胞的细胞类型。gydF4y2BaegydF4y2Ba,使用inferCNV在单细胞中检测到拷贝数变化gydF4y2Ba63gydF4y2Ba.(上)参考非肿瘤细胞没有拷贝数变异。(下)肿瘤细胞簇因样本亚组特有的拷贝数变化而富集。含有CNVs的细胞被指定为肿瘤细胞进行下游分析。gydF4y2BafgydF4y2Ba, UMAP嵌入如gydF4y2Ba(d)gydF4y2Ba通过检测拷贝数的变化来着色。gydF4y2BaggydF4y2Ba,显示SHH、G3、G4 MB特征标记基因和鉴定的非肿瘤细胞类型表达的点图。为gydF4y2Ba一个gydF4y2Ba,gydF4y2BaggydF4y2Ba,颜色表示平均表达,每个点的大小表示该簇中表达基因的细胞的百分比。gydF4y2Ba
扩展数据图8 G3和G4 MB类似于人类RL的特定成分,其分化在时间上停滞。gydF4y2Ba
一个gydF4y2Ba,单gydF4y2Ba67gydF4y2BaG3肿瘤细胞的分类(gydF4y2BangydF4y2Ba= 6), g4 (gydF4y2BangydF4y2Ba= 11), SHH (gydF4y2BangydF4y2Ba= 3) MB scRNAseq样本,通过与整个发育中的人类小脑进行比较gydF4y2Ba13gydF4y2Ba.正如预期的那样,MB细胞与谷氨酸细胞最为相似。gydF4y2BabgydF4y2Ba,肿瘤细胞的SingleR分类gydF4y2Ba(一)gydF4y2Ba通过比较谷氨酸能细胞类型。gydF4y2BacgydF4y2Ba, Kaplan-Meier图显示当前数据集中G3和G4 MB子类型的总生存期。使用对数秩检验评估显著性。审查案例,+。gydF4y2BadgydF4y2Ba,每个细胞分类的相对置信度,计算为每个子组的平均相似分gydF4y2Ba(b)gydF4y2Ba,减去每个细胞类型的其他亚组的中位数相似性得分。gydF4y2BaegydF4y2Ba, UMAP嵌入gydF4y2BangydF4y2Ba= 545 G3和G4 MB散装RNAseq样品,通过存在着色gydF4y2BaKBTBD4gydF4y2Ba突变。G4ɣCBFA复合物突变贫乏,显示高gydF4y2BaOTX2gydF4y2Ba表达,也都丰富了gydF4y2BaKBTBD4gydF4y2Ba突变。gydF4y2BafgydF4y2Ba,发育中的人类小脑snRNA-seq数据中超级增强子(SE)基因的表达。这些基因启动子已被证明能促进转录gydF4y2BaPRDM6gydF4y2Ba(gydF4y2BaSNCAIPgydF4y2Ba),gydF4y2BaGFI1BgydF4y2Ba(gydF4y2BaDDX31 / BARHL1gydF4y2Ba和gydF4y2BaPRRC2BgydF4y2Ba)在G3和G4 MB中次要于增强器劫持事件gydF4y2Ba11gydF4y2Ba,gydF4y2Ba28gydF4y2Ba.采用双侧Wilcoxon秩和检验及FDR校正,***评估显著性gydF4y2BapgydF4y2Ba< 0.0005。gydF4y2BaSNCAIPgydF4y2Ba,gydF4y2BapgydF4y2Bae = 3.39gydF4y2Ba−261gydF4y2Ba;gydF4y2BaDDX31gydF4y2Ba,gydF4y2BapgydF4y2Bae = 4.27gydF4y2Ba−71gydF4y2Ba;gydF4y2BaBARHL1gydF4y2Ba,gydF4y2BapgydF4y2Bae = 4.91gydF4y2Ba−40gydF4y2Ba.gydF4y2BangydF4y2Ba= 9208个单元格。gydF4y2BaggydF4y2Ba,表示gydF4y2BaDDX31gydF4y2Ba,gydF4y2BaBARHL1gydF4y2Ba,gydF4y2BaPRRC2BgydF4y2Ba在发育中的人类小脑的所有细胞类型中。gydF4y2BaDDX31gydF4y2Ba和gydF4y2BaBARHL1gydF4y2Ba显示特定于RL的相关表达gydF4y2BaSVZgydF4y2Ba,而gydF4y2BaPRRC2BgydF4y2Ba非特异性表达。采用双侧Wilcoxon秩和检验及FDR校正,***评估显著性gydF4y2BapgydF4y2Ba< 0.0005。gydF4y2BaDDX31gydF4y2Ba,gydF4y2BapgydF4y2Bae = 3.66gydF4y2Ba−113gydF4y2Ba;gydF4y2BaBARHL1gydF4y2Ba,gydF4y2BapgydF4y2Bae = 6.26gydF4y2Ba−191gydF4y2Ba.gydF4y2BangydF4y2Ba= 59,608个细胞。gydF4y2BahgydF4y2Ba, G3和G4 MB驱动基因的表达(见图;gydF4y2Ba1gydF4y2Ba)在发育中的人类小脑snRNA-seq数据中。gydF4y2BaI j kgydF4y2Ba, G3和G4 MB驱动基因的平均表达量gydF4y2Ba(我)gydF4y2Ba, CBFA复合基因gydF4y2Ba(j)gydF4y2Ba,以及功能驱动基因的增益gydF4y2Ba(k)gydF4y2Ba发育中的人类小脑snRNA-seq数据。采用双侧Wilcoxon秩和检验*评估显著性gydF4y2BapgydF4y2Ba< 0.05, ***gydF4y2BapgydF4y2Ba< 0.0005。gydF4y2BangydF4y2Ba= 9208个单元格。gydF4y2Ba(我)gydF4y2Ba,gydF4y2BapgydF4y2Bae = 1.5gydF4y2Ba−13gydF4y2Ba;gydF4y2Ba(j)gydF4y2Ba质量,gydF4y2BapgydF4y2Ba= 0.0085;GN早期,gydF4y2BapgydF4y2Ba= 0.047;gydF4y2Ba(k)gydF4y2BaRLgydF4y2BaSVZgydF4y2Ba,gydF4y2BapgydF4y2Bae = 1.9gydF4y2Ba−05gydF4y2Ba;质量,gydF4y2BapgydF4y2Bae = 3.2gydF4y2Ba−13gydF4y2Ba.为gydF4y2Ba我gydF4y2Ba,gydF4y2BajgydF4y2Ba,gydF4y2BakgydF4y2Ba,箱形图表示中位数和四分位范围,胡须表示数据范围。超出此范围的点是异常值,并单独绘制。gydF4y2Ba
扩展数据图9人体eomegydF4y2Ba+ vegydF4y2BaRL细胞是有丝分裂活跃的UBC祖细胞。gydF4y2Ba
一个gydF4y2Ba17岁发育中的人RL中eoms和PAX6的表达(gydF4y2Ba我gydF4y2Ba)及19 (gydF4y2Ba2gydF4y2Ba)物理化学加工。比例尺:100µm。gydF4y2BabgydF4y2Ba17岁发育中的人RL中eoms和KI67的表达(gydF4y2Ba我gydF4y2Ba)及19 (gydF4y2Ba2gydF4y2Ba)物理化学加工。比例尺gydF4y2Ba一个gydF4y2Ba.增殖加工gydF4y2Ba+ vegydF4y2BaUBC祖细胞在所有发育时间点都很常见。gydF4y2BacgydF4y2Ba, eoms +细胞在人类RL区。的RLgydF4y2BaSVZgydF4y2Ba含有明显多于RL的eoms +细胞gydF4y2BaVZgydF4y2Ba.使用非配对双尾t检验评估显著性***gydF4y2BapgydF4y2Bae = 1.048gydF4y2Ba−18gydF4y2Ba.gydF4y2BangydF4y2Ba= 3个生物重复,每N = 4个时间点;误差条,SEM。gydF4y2BadgydF4y2Ba在发育中的人RL中,eoms和KI67的表达在30 PCW晚期。比例尺gydF4y2Ba(一)gydF4y2Ba.增殖加工gydF4y2Ba+ vegydF4y2Ba在整个胎儿发育过程中都可以发现UBC祖细胞,但随着KI67表达的降低,在后期时间点出现的频率降低(图2)。gydF4y2Ba3 cgydF4y2Ba).gydF4y2BaegydF4y2Ba不同发育时间点人类RL中eoms +/KI67+细胞数量的量化。所有与11个PCW的比较使用双尾未配对t检验均不显著;14物理化学加工,gydF4y2BapgydF4y2Ba= 0.43;17个物理化学加工,gydF4y2BapgydF4y2Ba= 0.65;19物理化学加工,gydF4y2BapgydF4y2Ba= 0.33。gydF4y2BangydF4y2Ba=每个时间点3个生物重复;误差条,SEM。eoms +/KI67+ UBC祖细胞是RL的长寿和优势群体,而不是分化前的短暂状态。gydF4y2BafgydF4y2Ba,不同发育时间点小鼠RL中Eomes+/Ki67+细胞数量的量化。使用非配对双尾t检验评估显著性***gydF4y2BapgydF4y2Ba= 0.00015。gydF4y2BangydF4y2Ba=每个时间点3个生物重复;误差条,SEM。Eomes+/Ki67+ UBC祖细胞在小鼠RL中是一个罕见的群体。gydF4y2Bag hgydF4y2BaEomes和Ki67在E15.5时小鼠RL的表达gydF4y2Ba(g)gydF4y2Ba和E16.5gydF4y2Ba(h)gydF4y2Ba.RL边界用白色虚线表示。比例尺:50µm。eome + UBCs很少是Ki67+。资料载于gydF4y2BaA b g hgydF4y2Ba来自三个独立实验的代表性图像是否具有相似的结果,数据是否在gydF4y2BadgydF4y2Ba不进行重复试验。gydF4y2Ba我gydF4y2Ba, RL的致癌分化gydF4y2BaSVZgydF4y2Ba祖细胞来自正常启动的G4 MB。gydF4y2Ba
图10 G3和G4 MB肿瘤位置与gydF4y2BaOTX2gydF4y2Ba表达式,并支持原始RL单元。gydF4y2Ba
一个gydF4y2Ba,按比例缩小的gydF4y2BaOTX2gydF4y2Ba,gydF4y2BaCBFA2T2gydF4y2Ba,gydF4y2BaCBFA2T3gydF4y2Ba表达式由scRNAseq。gydF4y2BabgydF4y2Ba,表示gydF4y2BaOTX2gydF4y2Ba在发育中的人类小脑的14和17 PCW的ISH。gydF4y2BacgydF4y2Ba, G4 MB的T1增强或T2中矢状位MRI图像(gydF4y2BangydF4y2Ba= 12)肿瘤初步诊断。gydF4y2BadgydF4y2Ba, G3 MB的T1增强或T2中矢状位MRI图像(gydF4y2BangydF4y2Ba= 10)肿瘤初步诊断。G3和G4 MB肿瘤均仅存在于gydF4y2BaOTX2gydF4y2Ba+下小脑。gydF4y2BaegydF4y2Ba, SHH MB轴向T1增强、T2或FLAIR图像(gydF4y2BangydF4y2Ba= 3)初诊时。SHH肿瘤发生在小脑半球,与EGL细胞来源一致。gydF4y2BafgydF4y2Ba、WNT MB轴向T1增强、T2或FLAIR图像(gydF4y2BangydF4y2Ba= 3)初诊时。资料载于gydF4y2BabgydF4y2Ba不进行重复试验。gydF4y2Ba
扩展数据图11gydF4y2BaOTX2gydF4y2Baknockdown通过中间的上调促进G3 MB的分化gydF4y2BaCBFA2T2gydF4y2Ba和gydF4y2BaCBFA2T3gydF4y2Ba.gydF4y2Ba
一个gydF4y2Ba,gydF4y2BaOTX2gydF4y2BaChIP-seqgydF4y2Ba34gydF4y2Ba峰值富集于gydF4y2BaCBFA2T2gydF4y2Ba基因位点,但不是gydF4y2BaCBFA2T3gydF4y2Ba.gydF4y2BabgydF4y2Ba, OTX2蛋白表达在OTX2- kd后降低。样品用于批量RNA测序。β肌动蛋白用作负荷控制。gydF4y2BacgydF4y2Ba, OTX2蛋白表达在OTX2- kd后降低。样品用于单核RNA测序。β肌动蛋白用作负荷控制。gydF4y2BadgydF4y2Ba, HDMB03和MB3W1培养的OTX2-KD和置乱条件下原发肿瘤球的代表性图像。比例尺:300µm。gydF4y2Bae, fgydF4y2Ba,在HDMB03和MB3W1 G3 MB细胞系中,OTX2-KD后出现单核无偏倚聚类gydF4y2Ba(c)gydF4y2Ba.gydF4y2BaggydF4y2Ba,来自HDMB03和MB3W1的bulk RNAseq基因在OTX2-KD上的平均表达gydF4y2Ba(b)gydF4y2Ba.橙色比绿色多的细胞表明细胞具有较高的基因表达特征的G3 MB细胞系不变,反之亦然。橙色细胞可能是OTX2-KD无效的细胞。gydF4y2BahgydF4y2Ba,由CytoTRACE测定的分化评分gydF4y2Ba73gydF4y2Ba.红色为分化程度低的细胞,蓝色为分化程度高的细胞。结果支持一个模型,其中HDMB03中的第6簇和MB3W1中的第3簇代表低效的OTX2-KD细胞,这些细胞与WT肿瘤细胞保持了最大的相似性。gydF4y2Ba我gydF4y2Ba在HDMB03和MB3W1中,OTX2-KD后RBFOX3 (NeuN)蛋白表达增加,验证了OTX2-KD后GN分化。gydF4y2BajgydF4y2Ba,在正常人RL发育过程中,与颗粒神经元分化沿假时间显著相关的基因表达。(上)颗粒神经元谱系中沿着伪时间的细胞密度(扩展数据图)。gydF4y2Ba6 bgydF4y2Ba,gydF4y2Ba左gydF4y2Ba).(下)来自发育中的人类小脑snRNA-seq数据集的标记的Binned基因表达(图。gydF4y2Ba4gydF4y2Ba).当OTX2在G3 MB中被敲除时,颗粒神经元基因的逐步表达(图2)。gydF4y2Ba5我gydF4y2Ba)gydF4y2Ba惊人地反映了正常颗粒神经元分化,这表明G3和G4 MB来自失败的正常分化,而不是其他假设,如反分化或去分化。gydF4y2BakgydF4y2Ba,gydF4y2BaCBFA2T2gydF4y2Ba和gydF4y2BaCBFA2T3gydF4y2BaHDMB03(左)和MB3W1(右)的表达。gydF4y2BaCBFA2T2gydF4y2Ba在CytoTRACE评分低于0.8的细胞中,表达强烈上调gydF4y2BaCBFA2T3gydF4y2Ba遵循。研究结果表明gydF4y2BaCBFA2T2gydF4y2Ba然后gydF4y2BaCBFA2T3gydF4y2Ba在有效的OTX2-KD反应中早期上调。gydF4y2BalgydF4y2Ba,gydF4y2BaCBFA2T2gydF4y2BaqPCR检测HDMB03中CBFA2T2过表达(CBFA2T2- oe)的表达变化。gydF4y2Ba米gydF4y2BaCBFA2T2- oe后,CBFA2T2蛋白表达增加。β-肌动蛋白作为负荷控制。gydF4y2BangydF4y2Ba, CBFA2T2-OE GFP和对照RFP条件下原发肿瘤球的代表图像。比例尺:600µm。gydF4y2Bao pgydF4y2Ba、活细胞数(gydF4y2BaogydF4y2Ba)和生存能力(gydF4y2BapgydF4y2Ba)响应CBFA2T2-OE。CBFA2T2-OE显著降低了活细胞数量,但活性没有变化。数据被归一化到它们各自的控件,并显示从gydF4y2BangydF4y2Ba每N = 8或N = 5个生物重复= 3个技术重复,分别用于活细胞数量和存活率。误差条表示SEM。使用生物重复的双尾配对t检验评估显著性,**gydF4y2BapgydF4y2Ba= 0.0047。gydF4y2Ba问gydF4y2Ba,gydF4y2BaOTX2gydF4y2Ba通过CBFA复合物抑制RL祖细胞分化。资料载于gydF4y2Bad ngydF4y2Ba分别来自4个和8个独立实验的代表性图像是否具有相似的结果,数据在gydF4y2BaB c i mgydF4y2Ba不进行重复试验。凝胶源数据参见补充图。gydF4y2Ba1gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
该文件包括补充图1(未裁剪的blots)和补充表1 - 7的完整描述。gydF4y2Ba
补充表1gydF4y2Ba
16q缺失基因的显著性值。gydF4y2Ba
补充表2gydF4y2Ba
人工MB样例元数据。gydF4y2Ba
补充表3gydF4y2Ba
CBFA2T2 TurboID蛋白。gydF4y2Ba
补充表4gydF4y2Ba
区分G3和G4 MB亚型的基因。gydF4y2Ba
补充表5gydF4y2Ba
突变调用G3和G4 MB。gydF4y2Ba
补充表6gydF4y2Ba
scRNA-seq和snRNA-seq对齐指标。gydF4y2Ba
补充表7gydF4y2Ba
人谷氨酸细胞发育中细胞类型的顶级差异表达基因标记。gydF4y2Ba
权利和权限gydF4y2Ba
根据与作者或其他权利持有人签订的出版协议,自然或其许可方(例如,社会或其他合作伙伴)对本文拥有排他性权利;作者对这篇文章接受的手稿版本的自我存档仅受此类出版协议的条款和适用法律的约束。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
亨德里克斯,l.d.,哈尔迪普尔,P。gydF4y2Baet al。gydF4y2Ba人菱形唇分化失败是成神经管细胞瘤形成的基础。gydF4y2Ba自然gydF4y2Ba609gydF4y2Ba, 1021-1028(2022)。https://doi.org/10.1038/s41586-022-05215-wgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05215-wgydF4y2Ba
这篇文章被引用gydF4y2Ba
人类成神经管细胞瘤的起源gydF4y2Ba
自然gydF4y2Ba(2022)gydF4y2Ba
人脑类器官——临床神经学研究的新工具gydF4y2Ba
神经学自然评论gydF4y2Ba(2022)gydF4y2Ba
人胎儿小脑细胞图谱提示成神经管细胞瘤的起源和癌变gydF4y2Ba
自然gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
