摘要gydF4y2Ba
基因组氧化损伤是细胞代谢不可避免的结果。它通过转录激活期间的表观遗传去甲基化在基因调控元件上出现gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba.在这里,我们表明启动子通过核有丝分裂器官蛋白NuMA(也称为NUMA1)介导的过程免受氧化损伤。NuMA显示在转录起始位点附近约100 bp的基因组占用。它结合RNA聚合酶II的起始形式,暂停释放因子和单链断裂修复(SSBR)成分,如TDP1。氧化损伤后,与染色质的结合增加,NuMA促进受损染色质上TDP1的富集。NuMA的耗竭增加启动子的氧化损伤。NuMA通过限制RNA聚合酶II的多聚adp核糖化来促进转录,增加其可用性并从启动子处暂停释放。新生RNA的代谢标记可以识别依赖NuMA进行转录的基因,包括即早反应基因。numa缺陷细胞与介导与SSBR结合的突变体互补,或有丝分裂功能分离突变体,恢复SSBR缺陷。这些发现强调了DNA氧化损伤修复在基因调控元件中的重要性,并描述了一个实现这一功能的过程。gydF4y2Ba
这是订阅内容的预览,gydF4y2Ba通过你所在的机构访问gydF4y2Ba
访问选项gydF4y2Ba
订阅《自然》+gydF4y2Ba
立即在线访问《自然》和其他55种《自然》杂志gydF4y2Ba
29.99美元gydF4y2Ba
每月gydF4y2Ba
订阅期刊gydF4y2Ba
获得1年的完整期刊访问权限gydF4y2Ba
199.00美元gydF4y2Ba
每期仅需3.90美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba
增值税稍后将在结帐时添加。gydF4y2Ba
税务计算将在结账时完成。gydF4y2Ba
买条gydF4y2Ba
在ReadCube上获得时间限制或全文访问。gydF4y2Ba
32.00美元gydF4y2Ba
所有价格均为净价格。gydF4y2Ba


数据可用性gydF4y2Ba
所有凝胶和免疫印迹的原始图像可在补充图中获得gydF4y2Ba1gydF4y2Ba.质谱数据已通过PRIDE合作伙伴存储库存入ProteomeXchange联盟gydF4y2Ba75gydF4y2Ba标识符号码gydF4y2BaPXD018002gydF4y2Ba.NGS数据已保存在基因表达综合数据库下的SuperSeries登录代码gydF4y2BaGSE147015gydF4y2Ba.子系列为:rp -1 4sU-seq (gydF4y2BaGSE147012gydF4y2Ba), rp -1 OGAP-seq (gydF4y2BaGSE147013gydF4y2Ba), MRC5 NuMA ChIP-seq (gydF4y2BaGSE147014gydF4y2Ba), SH-SY5Y OGAP-seq (gydF4y2BaGSE201981gydF4y2Ba), rp -1 Pol2 ChIP-seq (gydF4y2BaGSE201982gydF4y2Ba), SH-SY5Y NuMA CUT&RUN (gydF4y2BaGSE201983gydF4y2Ba)、rp -1 NuMA及TDP1 CUT&RUN (gydF4y2BaGSE201984gydF4y2Ba).与原始数据(凝胶或印迹图像)、质谱分析和生物信息学分析相关的图包括:gydF4y2Ba1 b、f-h j-lgydF4y2Ba,gydF4y2Ba2模拟,做减法,我,kmgydF4y2Ba,gydF4y2Ba3 ggydF4y2Ba,gydF4y2Ba4 b-ggydF4y2Ba,gydF4y2Ba5 a -gydF4y2Ba而且gydF4y2Ba6 a, d, f igydF4y2Ba和扩展数据图。gydF4y2Ba1gydF4y2Ba,gydF4y2Ba3, c-jgydF4y2Ba,gydF4y2Ba4模拟gydF4y2Ba,gydF4y2Ba5中,我gydF4y2Ba,gydF4y2Ba6 b, c, h,我gydF4y2Ba,gydF4y2Ba7 a, dgydF4y2Ba,gydF4y2Ba8 bgydF4y2Ba,gydF4y2Ba9 b, c, egydF4y2Ba而且gydF4y2Ba10 hgydF4y2Ba.gydF4y2Ba
代码的可用性gydF4y2Ba
暂停比率计算器的代码可在gydF4y2Bahttps://github.com/jdparker101/Pausing_Ratio_NotebookgydF4y2Ba.gydF4y2Ba
参考文献gydF4y2Ba
组蛋白去甲基酶的分子机制和潜在功能。gydF4y2Ba细胞生物学。gydF4y2Ba13gydF4y2Ba, 297-311(2012)。gydF4y2Ba
徐,G.-L。Tet酶介导的DNA氧化去甲基化。gydF4y2Ba国家的科学。牧师。gydF4y2Ba2gydF4y2Ba, 318-328(2015)。gydF4y2Ba
马戈林,Y.克劳提尔,J.-F。,Shafirovich, V., Geacintov, N. E. & Dedon, P. C. Paradoxical hotspots for guanine oxidation by a chemical mediator of inflammation.Nat,化学。医学杂志。gydF4y2Ba2gydF4y2Ba, 365-366(2006)。gydF4y2Ba
潘,L.等。氧化鸟嘌呤碱基损伤在8-氧鸟嘌呤DNA糖基化酶-1介导的核因子κ b驱动基因表达表观遗传调控中的作用gydF4y2Ba生物。化学。gydF4y2Ba291gydF4y2Ba, 25553-25566(2016)。gydF4y2Ba
丁艳,丁艳,丁艳,丁艳。C. J.氧化性DNA损伤是通过碱基切除修复调节基因转录的表观遗传。gydF4y2Ba国家科学院学报美国gydF4y2Ba114gydF4y2Ba, 2604-2609(2017)。gydF4y2Ba
El-Khamisy, s.f.等人。脊髓小脑共济失调伴轴突神经病-1的DNA缺陷单链断裂修复。gydF4y2Ba自然gydF4y2Ba434gydF4y2Ba, 108-113(2005)。gydF4y2Ba
尼尔森,L., forstrr øm, R. J., Bjørås, M. & Alseth, I. AP内切酶独立于碱基位点的修复gydF4y2Ba粟酒裂殖酵母gydF4y2Ba.gydF4y2Ba核酸测定。gydF4y2Ba40gydF4y2Ba, 2000-2009(2012)。gydF4y2Ba
Interthal, H., Chen, H. J. & Champoux, J. J.人类Tdp1切割广泛的底物,包括磷酰胺键。gydF4y2Ba生物。化学。gydF4y2Ba280gydF4y2Ba, 36518-36528(2005)。gydF4y2Ba
Lebedeva, N. A., Rechkunova, N. I., El-Khamisy, S. F. & Lavrik, O. I.酪氨酸dna磷酸二酯酶1启动无嘌呤/无嘧啶位点的修复。gydF4y2BaBiochimiegydF4y2Ba94gydF4y2Ba, 1749-1753(2012)。gydF4y2Ba
怀特豪斯,c.j.等。XRCC1刺激受损DNA末端的人多核苷酸激酶活性,加速DNA单链断裂修复。gydF4y2Ba细胞gydF4y2Ba104gydF4y2Ba, 107-117(2001)。gydF4y2Ba
El-Khamisy, S. F., Masutani, M., Suzuki, H. & Caldecott, K. W.在氧化性DNA损伤部位XRCC1核病灶的组装或稳定性需要PARP-1。gydF4y2Ba核酸测定。gydF4y2Ba31gydF4y2Ba, 5526-5533(2003)。gydF4y2Ba
Das, B. B.等。PARP1-TDP1偶联修复拓扑异构酶i诱导的DNA损伤。gydF4y2Ba核酸测定。gydF4y2Ba42gydF4y2Ba, 4435-4449(2014)。gydF4y2Ba
Awwad, s.w., Abu-Zhayia, e.r., Guttmann-Raviv, N. & Ayoub, N. NELF-E被招募到DNA双链断裂位点促进转录抑制和修复。gydF4y2BaEMBO代表。gydF4y2Ba18gydF4y2Ba, 745-764(2017)。gydF4y2Ba
Hoch, n.c.等人。XRCC1突变与PARP1过度激活和小脑性共济失调有关。gydF4y2Ba自然gydF4y2Ba541gydF4y2Ba, 87-91(2017)。gydF4y2Ba
Takashima, H.等。TDP1的突变,编码拓扑异构酶i依赖的DNA损伤修复酶,在脊髓小脑共济失调与轴突神经病。gydF4y2BaNat,麝猫。gydF4y2Ba32gydF4y2Ba, 267-272(2002)。gydF4y2Ba
Giampetruzzi等人。肌动蛋白聚合的调节影响多种形式的肌萎缩性侧索硬化症的核质转运。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 3827(2019)。gydF4y2Ba
梅纳德等人。Lamin A/C促进DNA碱基切除修复。gydF4y2Ba核酸测定。gydF4y2Ba47gydF4y2Ba, 11709-11728(2019)。gydF4y2Ba
Lydersen, B. K. & Pettijohn, D. E.与有丝分裂器官极性区相关的人类特异性核蛋白:在人/仓鼠杂交细胞中的分布。gydF4y2Ba细胞gydF4y2Ba22gydF4y2Ba, 489-499(1980)。gydF4y2Ba
Hueschen, C. L., Kenny, S. J., Xu K. & Dumont, S. NuMA在有丝分裂时将动力蛋白活性招募到微管负端。gydF4y2BaeLifegydF4y2Ba6gydF4y2Ba, e29328(2017)。gydF4y2Ba
Merdes, A., Ramyar, K., Vechio, J. D.和Cleveland, D. W. NuMA和细胞质动力蛋白复合物对有丝分裂纺锤体组装至关重要。gydF4y2Ba细胞gydF4y2Ba87gydF4y2Ba, 447-458(1996)。gydF4y2Ba
Merdes, A. & Cleveland, D. W. NuMA在间期核中的作用。gydF4y2Ba细胞科学。gydF4y2Ba111gydF4y2Ba, 71-79(1998)。gydF4y2Ba
Vidi,中国。et al。NuMA通过调节DNA断裂处ISWI atp酶SNF2h的积累促进同源重组修复。gydF4y2Ba核酸测定。gydF4y2Ba42gydF4y2Ba, 6365-6379(2014)。gydF4y2Ba
Salvador-Moreno, N.等人。核结构蛋白NuMA是53BP1在DNA双链断裂修复中的负调控因子。gydF4y2Ba核酸测定。gydF4y2Ba47gydF4y2Ba, 2703-2715(2019)。gydF4y2Ba
塞拉-马奎斯,A.等人。有丝分裂蛋白NuMA在核的形成和力学中起着纺锤体独立的作用。gydF4y2BaJ.细胞生物学。gydF4y2Ba219gydF4y2Ba, e202004202(2020)。gydF4y2Ba
Bahrami, S. & Drabløs, F.基因在即早反应过程中的调控。gydF4y2Ba放置杂志。Regul。gydF4y2Ba62gydF4y2Ba, 37-49(2016)。gydF4y2Ba
图莱,J. W.等。即早反应基因和延迟反应基因具有不同的功能和基因组结构。gydF4y2Ba生物。化学。gydF4y2Ba282gydF4y2Ba, 23981-23995(2007)。gydF4y2Ba
萨姆森,l。d。抑制炎症的目标。gydF4y2Ba科学gydF4y2Ba362gydF4y2Ba, 748-749(2018)。gydF4y2Ba
维斯尼斯,T.等。OGG1小分子抑制剂抑制促炎基因表达和炎症反应。gydF4y2Ba科学gydF4y2Ba362gydF4y2Ba, 834-839(2018)。gydF4y2Ba
Dellino, g.i.等人。在特定位点释放暂停的RNA聚合酶II有利于DNA双链断裂的形成并促进癌症易位。gydF4y2BaNat,麝猫。gydF4y2Ba47gydF4y2Ba, 1-21(2019)。gydF4y2Ba
Boehler, C.等人。Poly(adp -核糖)聚合酶3 (PARP3),细胞对DNA损伤和有丝分裂进程的反应中的新人。gydF4y2Ba国家科学院学报美国gydF4y2Ba108gydF4y2Ba, 2783-2788(2011)。gydF4y2Ba
张,P. Coughlin, M. & Mitchison, T. J.聚(adp -核糖)的tankyase -1聚合是纺锤体结构和功能所必需的。gydF4y2Ba细胞生物学。gydF4y2Ba7gydF4y2Ba, 1133-1139(2005)。gydF4y2Ba
Haren, L. & Merdes, a . NuMA与微管蛋白的直接结合是由尾域中的一种新型序列基序介导的,该序列基序捆绑并稳定微管。gydF4y2Ba细胞科学。gydF4y2Ba115gydF4y2Ba, 1815-1824(2002)。gydF4y2Ba
Hendriks, i.a., Larsen, s.c. & Nielsen, m.l.使用质谱为基础的蛋白质组学全面分析adp核糖基化位点的先进策略。gydF4y2Ba细胞蛋白质组学gydF4y2Ba18gydF4y2Ba, 1010-1026(2019)。gydF4y2Ba
Palazzo, L.等。丝氨酸是DNA损伤后adp核糖基化的主要残基。gydF4y2BaeLifegydF4y2Ba7gydF4y2Ba, e34334(2018)。gydF4y2Ba
Páhi, Z. G., Borsos, B. N., Pantazi, V., Ujfaludi, Z. & Pankotai, T.转录过程中的PARylation:对微调机制和调控的洞察。gydF4y2Ba癌症gydF4y2Ba12gydF4y2Ba, 183(2020)。gydF4y2Ba
Ting, X.等。USP11作为一种组蛋白去泛素酶,在DNA修复过程中参与染色质重组。gydF4y2Ba核酸测定。gydF4y2Ba47gydF4y2Ba, 9721-9740(2019)。gydF4y2Ba
库兹涅佐夫,n.a.等人。酪氨酸-DNA磷酸二酯酶对DNA结合和碱基位点水解的预稳态动力学。gydF4y2Baj . Biomol。结构体。直流发电机。gydF4y2Ba35gydF4y2Ba, 2314-2327(2017)。gydF4y2Ba
Poetsch, a.r., Boulton, S. J. & Luscombe, n.m.氧化性DNA损伤和修复的基因组景观揭示了突变的区域选择性保护。gydF4y2Ba基因组医学杂志。gydF4y2Ba19gydF4y2Ba, 215(2018)。gydF4y2Ba
Wu, J., McKeague, M. & Sturla, S. J.核苷酸分辨率的氧化DNA损伤全基因组图谱点击-代码- seq。gydF4y2Baj。化学。Soc。gydF4y2Ba140gydF4y2Ba, 9783-9787(2018)。gydF4y2Ba
毛,P.等。酵母中烷基化损伤、修复和突变的全基因组图谱揭示了突变异质性的机制。gydF4y2Ba基因组Res。gydF4y2Ba27gydF4y2Ba, 1674-1684(2017)。gydF4y2Ba
Reid, d.a.等人。合并核苷类似物映射基因组修复位点在有丝分裂后的人类神经元。gydF4y2Ba科学gydF4y2Ba372gydF4y2Ba, 91-94(2021)。gydF4y2Ba
吴,W.等。神经元增强子是DNA单链断裂修复的热点。gydF4y2Ba自然gydF4y2Ba593gydF4y2Ba, 440-444(2021)。gydF4y2Ba
Radulescu, A. E. & Cleveland, D. W. NuMA 30年后:矩阵重访。gydF4y2Ba细胞生物学趋势。gydF4y2Ba20.gydF4y2Ba, 214-222(2010)。gydF4y2Ba
Ohata, H.等人。NuMA是选择性诱导p53靶基因所必需的。gydF4y2Ba摩尔。细胞。医学杂志。gydF4y2Ba33gydF4y2Ba, 2447-2457(2013)。gydF4y2Ba
Jayaraman, S.等人。核有丝分裂器蛋白NuMA控制rDNA转录,并以p53独立的方式介导核仁应激反应。gydF4y2Ba核酸测定。gydF4y2Ba45gydF4y2Ba, 11725-11742(2017)。gydF4y2Ba
Chang, W., Dynek, J. N. & Smith, S. NuMA是有丝分裂中坦基酶1多聚(adp -核糖化)的主要受体。gydF4y2Ba物化学。J。gydF4y2Ba391gydF4y2Ba, 177-184(2005)。gydF4y2Ba
阿尔特梅尔等人。本质无序蛋白质的液体分离由聚(adp -核糖)播种。gydF4y2BaCommun Nat。gydF4y2Ba6gydF4y2Ba, 8088(2015)。gydF4y2Ba
奈尔,S. J.等。配体激活增强子的相分离允许合作染色体增强子组装。gydF4y2BaNat。结构。摩尔。杂志。gydF4y2Ba26gydF4y2Ba, 193-203(2019)。gydF4y2Ba
Endo, A., Moyori, A., Kobayashi, A. & Wong, R. W.核有丝分裂器蛋白NuMA在癌细胞中调节p53介导的转录。gydF4y2Ba细胞死亡。gydF4y2Ba4gydF4y2Ba, e713-e713(2013)。gydF4y2Ba
Perera, D.等人。差异DNA修复是癌症基因组中活性启动子突变热点的基础。gydF4y2Ba自然gydF4y2Ba532gydF4y2Ba, 259-263(2016)。gydF4y2Ba
Janssens, D. & Henikoff, S. CUT&RUN:针对低细胞数量的高效原位全基因组分析。gydF4y2Baprotocols.iogydF4y2Bahttps://doi.org/10.17504/protocols.io.zcpf2vngydF4y2Ba(2019)。gydF4y2Ba
Liu, N. Library prep for CUT&RUN with NEBNext UltragydF4y2BaTMgydF4y2BaII DNA文库准备试剂盒Illumina (E7645) V.1。gydF4y2Baprotocols.iogydF4y2Bahttps://doi.org/10.17504/protocols.io.wvgfe3wgydF4y2Ba(2019)。gydF4y2Ba
Cox, J. & Mann, M. MaxQuant能够实现高肽识别率,个性化p.p.b.范围的质量精度和蛋白质组范围的蛋白质定量。gydF4y2Ba生物科技Nat。》。gydF4y2Ba26gydF4y2Ba, 1367-1372(2008)。gydF4y2Ba
Tyanova, S.等。用于(蛋白质)组学数据综合分析的Perseus计算平台。gydF4y2BaNat方法。gydF4y2Ba13gydF4y2Ba, 731-740(2016)。gydF4y2Ba
Li, D. et al. pFind:一种通过串联质谱自动识别肽和蛋白质的新型数据库搜索软件系统。gydF4y2Ba生物信息学gydF4y2Ba21gydF4y2Ba, 3049-3050(2005)。gydF4y2Ba
Wang, L. et al. pFind 2.0:通过串联质谱鉴定肽和蛋白质的软件包。gydF4y2Ba快速Commun。质量范围。gydF4y2Ba21gydF4y2Ba, 2985-2991(2007)。gydF4y2Ba
Dobin, A.等人。STAR:超快通用RNA-seq对准器。gydF4y2Ba生物信息学gydF4y2Ba29gydF4y2Ba, 15-21(2013)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
爱,i.m.,安德斯,S. & Huber, W.计数数据的差分分析- DESeq2包。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
Patro, R., Duggal, G., Love, M. I., Irizarry, R. A. & Kingsford, C. Salmon提供了快速和有偏差意识的转录表达量化。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 417-419(2017)。gydF4y2Ba
Soneson, C., Love, m.i.和Robinson, m.d. RNA-seq的差异分析:转录水平的估计提高了基因水平的推断。gydF4y2BaF1000Res。gydF4y2Ba4gydF4y2Ba1521(2016)。gydF4y2Ba
Durinck, S., Spellman, P. T., Birney, E. & Huber, W.将基因组数据集与R/Bioconductor包biomaRt集成的映射标识符。gydF4y2BaProtoc Nat。gydF4y2Ba4gydF4y2Ba, 1184-1191(2009)。gydF4y2Ba
Giannakakis, A.等。氧化应激下人类基因组编码部分和非编码部分的表达模式对比。gydF4y2BaSci代表。gydF4y2Ba5gydF4y2Ba, 9737(2015)。gydF4y2Ba
Dellino, g.i.等人。在特定位点释放暂停的RNA聚合酶II有利于DNA双链断裂的形成并促进癌症易位。gydF4y2BaNat,麝猫。gydF4y2Ba51gydF4y2Ba, 1011-1023(2019)。gydF4y2Ba
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 841-842(2010)。gydF4y2Ba
Sun, Z. & Chadwick, B. P. SETDB1的缺失部分通过IL1RAPL1基因增强子的再激活来解压缩失活的X染色体。gydF4y2BaEpigenet。染色质gydF4y2Ba11gydF4y2Ba, 45(2018)。gydF4y2Ba
亨利克先生,K.-O。et al。整合基因组尺度分析确定不利成神经细胞瘤转录调控的表观遗传机制。gydF4y2Ba癌症Res。gydF4y2Ba76gydF4y2Ba, 5523-5537(2016)。gydF4y2Ba
李,H. & Durbin R.快速和准确的长读对齐与Burrows-Wheeler变换。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba, 589-595(2010)。gydF4y2Ba
Sims, D.等人。计算基因组学分析工具包。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 1290-1291(2014)。gydF4y2Ba
李,H.等。序列对齐/映射格式和SAMtools。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba, 2078-2079(2009)。gydF4y2Ba
加斯帕,j.m.改进了MACS2的峰值调用。预印在gydF4y2BaBiorxivgydF4y2Bahttps://doi.org/10.1101/496521gydF4y2Ba(2018)。gydF4y2Ba
泽比诺,D. R.等。运用2018年。gydF4y2Ba核酸测定。gydF4y2Ba46gydF4y2Ba, d754-d761(2017)。gydF4y2Ba
Liao, Y., Smyth, G. K. & Shi, W. featuremets:一种高效的通用程序,用于分配序列读取基因组特征。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 923-930(2014)。gydF4y2Ba
哈德利,W。gydF4y2Baggplot2:数据分析的优雅图形gydF4y2Ba(斯普林格出版社,2016年版)。gydF4y2Ba
Perez-Riverol, Y.等。2019年PRIDE数据库及相关工具和资源:改善对量化数据的支持。gydF4y2Ba核酸测定。gydF4y2Ba47gydF4y2Ba, d442-d450(2019)。gydF4y2Ba
确认gydF4y2Ba
我们感谢L. Ferraiuolo对iPS细胞衍生神经元的建议,感谢S. Dumont提供包含稳定集成spCas9 (Tet-On启动子)和NuMA sgRNA的hTERT (RPE-1)细胞,感谢Sheffield Wolfson光学显微镜设备用于成像实验,感谢Sheffield biOMICS设备用于蛋白质质谱实验。这项工作由惠康信托基金研究员奖(103844)和李斯特预防医学研究所奖学金(137661)资助给s.f.e.k。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
贡献gydF4y2Ba
S.R.进行了蛋白质相互作用和体外转录实验。a.a.a进行了parylylation实验。S.R.和A.A.A.进行了成像、DNA链断裂修复、全基因组占用和氧化损伤实验。J.P.在I.S. k.l.、N.M.P.和C.L.的建议和支持下进行了所有的生物信息学分析,进行了彗星实验、体外结合和泛素化。A.E.A.-M。进行并分析了蛋白质质谱实验。C.D.S.S.生成iPS细胞来源的神经元。M.J.协助彗星实验并进行细胞周期分析。S.F.E.-K。,S.R. and A.A.A wrote the manuscript. All authors edited the manuscript. S.F.E.-K. conceived the study, and led and managed the project.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者声明没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢Rami Aqeilan、Zhao-Qi Wang和其他匿名审稿人对本工作的同行评议所作的贡献。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
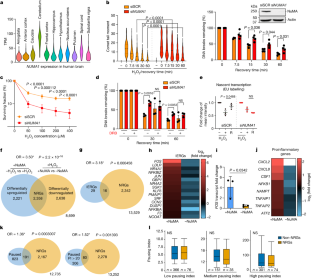
图1 SSBR基因在不同脑区表达情况。gydF4y2Ba
的表达gydF4y2BaTDP1gydF4y2Ba,gydF4y2BaXRCC1gydF4y2Ba而且gydF4y2BaPARP1gydF4y2Ba在GTEx v8数据集中的不同大脑区域。gydF4y2Ba
扩展数据图2 DRB治疗可消除SSBR中numa依赖性缺陷。gydF4y2Ba
转染siSCR和sinuma的MRC5细胞分别用DMSO或DRB预处理2 h后再进行hgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba治疗后恢复30、60分钟。代表性的小提琴图显示了来自150个核的彗星尾矩的传播,n = 3个生物重复(双面,未配对t检验)。gydF4y2Ba
扩展数据图3 NuMA促进氧化损伤后的转录gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)对rp -1细胞裂解物进行4sU-Seq免疫印迹。, n = 2个生物重复。(gydF4y2BabgydF4y2Ba) 4sU-Seq的原理图。(gydF4y2BacgydF4y2Ba) 4sU-Seq显示-loggydF4y2Ba10gydF4y2Ba调整p值与Log的关系gydF4y2Ba2gydF4y2BaWT (+HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)与(−H .gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)细胞。(DEseq2,双侧Wald检验,Benjamini-Hochberg程序调整p值≤0.05)。日志gydF4y2Ba2gydF4y2Ba折量随WT (+H)的变化gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)细胞。(gydF4y2BadgydF4y2Ba暂停基因与上调基因重叠(gydF4y2Ba左gydF4y2Ba)及调低(gydF4y2Ba正确的gydF4y2Ba)在WT (+HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)与(−H .gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)细胞。(gydF4y2BaegydF4y2Ba) 4sU-Seq显示-loggydF4y2Ba10gydF4y2Ba根据Log调整p值gydF4y2Ba2gydF4y2BaKD与WT (+HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)细胞。(DEseq2,双侧Wald检验,Benjamini-Hochberg调整p值≤0.05)。日志gydF4y2Ba2gydF4y2BaKD细胞的折叠变化。(gydF4y2BafgydF4y2Ba暂停基因与上调基因重叠(gydF4y2Ba左gydF4y2Ba)及调低(gydF4y2Ba正确的gydF4y2Ba)在KD与WT (+HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)细胞。(gydF4y2BaggydF4y2Ba)含有脆弱启动子(FPG)的基因与NRGs重叠。背景集是差异表达基因的总集。(gydF4y2BahgydF4y2Ba) Log中平均转录水平的分布gydF4y2Ba2gydF4y2BaNRGs的百万分转录本(TPM)和基因在WT(+HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba),并在重复间取平均值。只有WT(+HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)重复被纳入分析。在箱形图中,中心线为中值,上下铰链表示上下四分位数,上下须表示最大和最小值分别不超过上下铰链的1.5倍四分位数范围;双面Wilcoxon秩和检验。(gydF4y2Ba我gydF4y2BaLog的散点图gydF4y2Ba2gydF4y2BaWT与KD(+HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba)细胞相对于基因长度(KB)的对数gydF4y2Ba10gydF4y2Ba规模。相关系数(R)由斯皮尔曼秩和显著性使用渐近t近似(双边)。(gydF4y2BajgydF4y2Ba)gydF4y2Ba左gydF4y2Ba;所示SSBR基因新生RNA转录本的折叠变化。gydF4y2Ba正确的gydF4y2Ba;SSBR和DSBR基因4sU-Seq谱的一个代表性轨迹的IGV快照。gydF4y2Ba
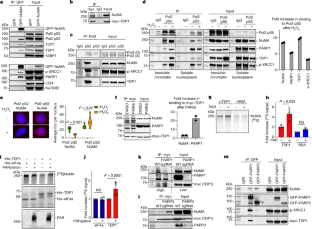
扩展数据图4 GFP下拉中质谱鉴定的聚集蛋白。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)转染GFP-empty或GFP-NuMA的HEK-293细胞用HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba细胞裂解物进行GFP-trap免疫沉淀,珠上胰蛋白酶消化,随后进行质谱分析。火山图显示NuMA相互作用蛋白的定量富集,通过使用Student 's t检验对数据集进行统计分析确定。实线表示经过过滤后,误发现率为0.05,组内人为方差S0 = 1,相互作用蛋白显著富集。Pol2和SSBR机械的组件被突出显示为红色方块。(gydF4y2BabgydF4y2Ba) GFP下拉图中质谱鉴定的聚集蛋白热图。根据样本条件,LFQ蛋白强度的层次聚类显示出两个优势样本聚类,GFP和GFP- numa - l (LTR)。(gydF4y2BacgydF4y2BaHEK293细胞分别用DMSO或PARG (PDD00017273)、PARP3 (ME0328)或PARP1 (Olaparib)抑制剂处理。Western免疫印迹法显示聚adp核糖基化(PAR)水平,n = 3个生物重复。(gydF4y2BadgydF4y2Ba) GFP下拉图中质谱鉴定的聚集蛋白热图。根据样本条件,LFQ蛋白强度的层次聚类显示出GFP、GFP- numa - s (STR)和GFP- numa - l (LTR)三个优势样本聚类。右边是NuMA-S (STR)和NuMA-L (LTR)强度分布的放大热图。gydF4y2Ba
图5 NuMA-L促进SSBR和转录恢复功能。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba人类NuMA异构体的示意图。(gydF4y2BabgydF4y2Ba) GFP-免疫沉淀来自GFP- numa - l和numa - s转染的HEK-293细胞,HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba并进行质谱分析。NuMA-L的注释串联质谱(gydF4y2Ba前gydF4y2Ba)及NuMA-S (gydF4y2Ba底gydF4y2Ba)在免疫沉淀物中鉴定出特异性肽。前体质量偏差LTAQVEQLEVFQR肽段为2.07 ppm, LTAQVEELSK肽段为0.66 ppm。(gydF4y2BacgydF4y2Ba从(b)中定量富集NuMA-L和NuMA-S之间的相互作用蛋白。使用Student t检验进行统计分析,确定numa相互作用蛋白。实线表示筛选FDR为0.05和人工组内方差(S0 = 1)后富集的相互作用蛋白。注释的蓝色和红色点表示NuMA-S (STR), NuMA-L (LTR)和其他感兴趣的蛋白质。未标注的淡红点表示与GFP对照相比,在每个NuMA下拉中显著富集的蛋白质。绿点是NuMA-S和NuMA-L下拉中都富集的蛋白质,红点是只在NuMA-L下拉中富集的蛋白质。(gydF4y2BadgydF4y2Ba)转染GFP-empty、GFP- numa - l和GFP- numa - s的HEK-293细胞中GFP-免疫沉淀的免疫印迹gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba, n = 3个生物重复。(gydF4y2BaegydF4y2Ba)转染的MRC5细胞免疫印迹。, n = 5个生物重复。(gydF4y2BafgydF4y2Ba) MRC5细胞如(e)所示转染,HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba并在媒体中恢复。来自250个原子核的彗尾矩分布的代表性图(双面,未配对,t检验)。(gydF4y2BaggydF4y2Ba)条形表示剩余DNA链断裂的百分比。误差条(±s.e.m,), n = 5个生物重复(双面,未配对t检验)。(gydF4y2BahgydF4y2Ba)如(e)所示转染的MRC5细胞的克隆性存活,以半对数尺度表示,n = 3个生物重复(双面,未配对t检验)。(gydF4y2Ba我gydF4y2Ba)转染的MRC5细胞免疫印迹。(gydF4y2BajgydF4y2Ba) MRC5细胞按(gydF4y2Ba我gydF4y2Ba)和新出现的带有EU标签的成绩单。散点图显示平均荧光的折叠变化。误差柱(±s.e.m.), n = 3个生物重复(双面,未配对t检验)。gydF4y2Ba
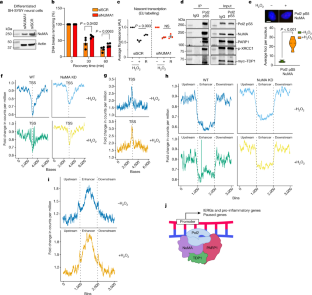
扩展数据图6 NuMA c端尾或有丝分裂功能分离突变体促进SSBR。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba由NuMA-FL生成的截断突变体的示意图,其c端尾部(aa 1700-2115)被指定为球状结构域(GD),微管结合结构域(aa 1866-1936)被指定为MD (gydF4y2BabgydF4y2BaHEK293细胞与全长或不同截断的NuMA共转染,如(gydF4y2Ba一个gydF4y2Ba),以及myc-TDP1。采用免疫印迹法分析gfp免疫沉淀,n = 3个生物重复。(gydF4y2BacgydF4y2Ba)对转染siSCR或siNuMA的MRC5细胞进行免疫印迹,然后与靶向耐药gfp标记的NuMA-FL、NuMA-GD或EV互补。(gydF4y2BadgydF4y2Ba)代表性的小提琴图显示了从3个生物重复中转染的150个MRC5细胞的彗星尾矩分布,如图(gydF4y2BacgydF4y2Ba),通过gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba治疗和恢复在指定时间点(双侧,未配对t检验)。(gydF4y2BaegydF4y2Ba)柱状图表示去掉H后剩余的断裂百分比gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba误差柱(±s.e.m), n = 3个生物重复,(双侧,未配对t检验)。(gydF4y2BafgydF4y2Ba) HEK293细胞转染NuMA-FL或NuMA-MD后,在收获前进行诺可达唑处理。代表图像显示NuMA(绿色),α-微管蛋白(红色),细胞核(DAPI/蓝色),n = 6个生物重复,比例尺,10 μm。(gydF4y2BaggydF4y2Ba)在有丝分裂极处,NuMA细胞的百分比被量化。误差条(±s.e.m), n = 100个细胞,来自6个生物重复(双面,未配对t检验)。(gydF4y2BahgydF4y2Ba)对转染GFP-empty、NuMA-FL或NuMA-MD的HEK-293细胞的gfp免疫沉淀进行免疫印迹,同时对myc-TDP1进行处理gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba, n = 3个生物重复。(gydF4y2Ba我gydF4y2Ba)对转染了siSCR或siNuMA的MRC5细胞的裂解物进行免疫印迹,并补充靶向抗gfp标记的NuMA-FL, NuMA-MD或EV。(gydF4y2BajgydF4y2Ba) MRC5细胞如(i)所示转染,HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba并在指定时间点在介质中恢复。代表性小提琴图显示来自3个生物重复的150个细胞的彗星尾矩分布(双面,未配对t检验)。(gydF4y2BakgydF4y2Ba)条形表示去除H后剩余的股断裂百分比gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba在完全介质中恢复。误差柱(±s.e.m.), n = 3个生物重复(双面,未配对t检验)。gydF4y2Ba
图7 NuMA促进TDP1在受损染色质上的富集。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)gydF4y2Ba最高;gydF4y2Ba采用抗NuMA抗体免疫印迹法分析NuMA的敲除效率。gydF4y2Ba底;gydF4y2Ba在光漂白实验的细胞中测量GFP-TDP1在时间0(秒)时的表达水平。误差条(±s.e.m.), n = 3个生物重复。(gydF4y2BabgydF4y2BaMRC5细胞被镀在玻璃底皿上,并与GFP-TDP1和siSCR或siNuMA共转染。细胞用DMSO或奥拉帕尼预培养。用紫外A激光(405 nm)局部照射表达类似总GFP信号的细胞,并在指定的时间点量化GFP - tdp1在损伤部位的积累。比例尺bar-5µm。(gydF4y2BacgydF4y2Ba)数据绘制为微辐照轨迹中平均百分比荧光(任意单位)±s.e.mgydF4y2Ba∼gydF4y2Ba测量30个细胞,n = 3个生物重复(双侧,未配对t检验)。(gydF4y2BadgydF4y2Ba用免疫印迹法分别或联合转染siSCR、siNuMA和siTDP1的MRC5细胞的裂解物进行分析,n = 3个生物重复。(gydF4y2BaegydF4y2Ba)转染MRC5细胞,如(gydF4y2BadgydF4y2Ba),用HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba并在指定的时间点恢复。代表性小提琴图显示150个细胞的彗星尾矩分布,n = 3个生物重复。(gydF4y2BafgydF4y2Ba)柱状图表示去掉H后剩余的断裂百分比gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba在完全介质中恢复。误差柱(±s.e.m.), n = 3个生物重复(双面,未配对t检验)。(gydF4y2BaggydF4y2Ba)转染MRC5细胞的存活情况如下(gydF4y2BadgydF4y2Ba)与HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba.结果以半对数尺度表示,代表三个生物重复的平均值±s.e.m。Ns -不显著(双侧未配对t检验)。gydF4y2Ba
扩展数据图8 NuMA与PAF1的交互gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba用GFP或GFP- numa - l质粒转染HEK-293细胞,用HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba用质谱分析细胞裂解物。火山图显示NuMA相互作用蛋白的定量富集,通过使用Student 's t检验对数据集进行统计分析确定。实线表示经过过滤后,误发现率为0.05,组内人为方差S0 = 1,相互作用蛋白显著富集。PAF1复合体的组成部分突出显示为蓝色方块。(gydF4y2BabgydF4y2BaHEK-293细胞用HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba并将其分为不溶性染色质结合部分和可溶性核质部分。采用Pol2 pS5抗体免疫沉淀内源性RNA Pol2。采用抗Pol2 pS5、NuMA和PAF1抗体免疫印迹法分析免疫沉淀复合物,n = 3个生物重复。gydF4y2Ba
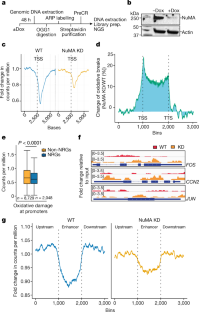
图9 NuMA缺失增加8-oxoG水平,降低启动子上TDP1的占用,而氧化应激增加启动子上NuMA和TDP1的可用性。gydF4y2Ba
(a) rp -1细胞未处理,HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba或凯擘gydF4y2Ba3.gydF4y2Ba.提取基因组DNA,用高效液相色谱- qqq质谱法定量8-oxoG / 1,000,000脱氧鸟苷(dG)水平。误差柱(±s.e.m), n = 3个生物重复,(双面,未配对t检验)(b和c) RPE-1细胞中(b) NuMA和(c) TDP1 CUT&RUN相对于IgG的TSS中每百万次计数变化的meta剖面。(d)用Flag-TDP1转染rp -1细胞,在启动子处进行ChIP-qPCRgydF4y2Ba”丛书gydF4y2Ba,gydF4y2BaCCN2gydF4y2Ba而且gydF4y2BaSRFgydF4y2Ba.误差柱(±s.e.m.), n = 3个生物重复(双面,未配对t检验)。(e)在未受干扰的条件下,RPE-1细胞中NuMA CUT&RUN相对于IgG的增强子区每百万次计数变化的meta分析。增强子两侧的区域为2500 bp。gydF4y2Ba
图10缺乏numa的神经元细胞表现出SSBR和转录恢复缺陷。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba在含有维甲酸(RA)的低血清培养基中,SH-SY5Y细胞分化为神经元形态(类似于长突起的原代神经元)的Brightfield图像。比例尺,100µm。(gydF4y2BabgydF4y2Ba)显示RA分化后神经突生长量化的Violin图,使用Fiji (ImageJ)软件和NeuronJ插件进行量化,n = 16/条件(双面,未配对t检验)。(gydF4y2BacgydF4y2Ba)未分化和分化SH-SY5Y细胞的MAP2免疫荧光图像,n = 2个生物重复,比例尺,10µm。(gydF4y2BadgydF4y2Ba)代表性的小提琴图显示,在从H恢复后或恢复期间,来自4个生物重复的200个分化SH-SY5Y细胞的彗星尾时刻的分布gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba(双侧未配对t检验)。(gydF4y2BaegydF4y2Ba)显示人类ipsc衍生运动神经元的Brightfield图像,n = 3个生物重复,比例尺,100µm。(gydF4y2BafgydF4y2Ba人类shSCR或shnuma转导的ipsc衍生运动神经元的代表性免疫荧光图像显示NuMA的相对水平(绿色),n = 2个生物重复,标度条,10µm。(gydF4y2BaggydF4y2Ba)小提琴图显示NuMA荧光强度n = 20个细胞/条件(双面,未配对t检验)。(gydF4y2BahgydF4y2Ba)对shSCR和shnuma转导神经元的裂解物进行免疫印迹,n = 2个生物重复。(gydF4y2Ba我gydF4y2Ba)柱状图表示去掉H后剩余的断裂百分比gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba在完全介质中的回收率,由碱性彗星试验定量。误差条表示范围,n = 2个生物重复。(gydF4y2BajgydF4y2Ba)代表性的小提琴图,显示从H恢复后立即或期间从100个细胞传播的彗星尾矩gydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba(双尾未配对t检验)(gydF4y2BakgydF4y2Ba)用HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba新生的成绩单也被欧盟贴上了标签。散点图显示平均荧光的折叠变化。误差条(±s.e.m.), n = 4个独立的封面卡瓦(双面,未配对t检验)。(gydF4y2BalgydF4y2Ba)在模拟处理或HgydF4y2Ba2gydF4y2BaOgydF4y2Ba2gydF4y2Ba治疗。对于每个样本,都显示了一个有代表性的图像,它是红色和蓝色通道的合并。小提琴图显示Pol2 pS5-NuMA复合体/核的平均信号强度,n = 125个细胞,来自三个独立孔,(双面,未配对t检验),比例尺,10µm。gydF4y2Ba
扩展数据图11描述NuMA在氧化应激反应中的功能。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba) GTEx v8数据集PARP1/NuMA在脑组织中的表达分布。(gydF4y2BabgydF4y2Ba)描述NuMA在氧化应激反应中的作用的示意图。NuMA在启动子处富集,并与RNA聚合酶2 (Pol2)和SSBR蛋白TDP1和PARP1相互作用。由于该复合物的富集,细胞能够有效地修复启动子的氧化损伤。NuMA作为PAR汇,确保发生在核质中的Pol2 PARylation的生理水平,从而增加Pol2启动子的可用性,并增强其暂停释放。这导致NuMA调控基因(NRGs)的转录增加(gydF4y2BacgydF4y2BaNuMA水平的降低会增加启动子的氧化损伤。此外,NuMA的缺失增加了核质中的Pol2 PARylation,从而降低了Pol2在启动子上的可用性,导致nrg的转录减少。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
该文件包含用于8-oxoG质谱的siRNA和gRNA序列、抗体、引物、质粒、寡核苷酸以及过渡和保留时间的补充表1-6。它还包含未裁剪的图像凝胶和西方印迹,与红框显示裁剪带。gydF4y2Ba
补充数据1gydF4y2Ba
该文件包含火山图中GFP vs NuMA-L所表示的蛋白质的完整列表。gydF4y2Ba
补充数据2gydF4y2Ba
该文件包含火山图NuMA-S vs NuMA-L中表示的蛋白质的完整列表。gydF4y2Ba
权利和权限gydF4y2Ba
根据与作者或其他权利持有人签订的出版协议,《自然》杂志或其许可方对本文拥有独家权利;作者对这篇文章接受的手稿版本的自我存档仅受此类出版协议的条款和适用法律的约束。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
Ray, S., Abugable, a.a., Parker, J。gydF4y2Baet al。gydF4y2Ba基因调控元件氧化损伤修复机制研究。gydF4y2Ba自然gydF4y2Ba609gydF4y2Ba, 1038-1047(2022)。https://doi.org/10.1038/s41586-022-05217-8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05217-8gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba
