文摘gydF4y2Ba
严重急性呼吸系统综合症冠状病毒2 (SARS-CoV-2)出现在2019年底,冠状病毒的破坏性的全球大流行疾病引起的2019 (COVID-19),部分原因是它能够有效地抑制宿主细胞的反应gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba。在极少数情况下,病毒蛋白抑制抗病毒反应通过模仿人类蛋白质组蛋白的关键区域gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8 gydF4y2Ba,尤其是那些包含所需的转录后修饰转录监管gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba。最近的研究表明,SARS-CoV-2明显破坏宿主细胞表观遗传调控gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba。然而,SARS-CoV-2如何控制宿主细胞表观基因组以及是否使用组蛋白模仿仍不清楚。在这里,我们表明,SARS-CoV-2蛋白质编码gydF4y2BaORF8gydF4y2Ba(ORF8)函数作为一个组蛋白模拟方舟图案的组蛋白H3破坏宿主细胞表观遗传调控。ORF8与染色质,扰乱监管关键组蛋白的转录后修饰,促进染色质压实。删除的gydF4y2BaORF8gydF4y2Ba基因或组蛋白模拟网站变弱SARS-CoV-2破坏宿主细胞染色质的能力,影响感染和转录反应减弱病毒基因组拷贝数。这些发现表明一个新的函数ORF8和SARS-CoV-2破坏宿主细胞表观遗传调控机制。此外,这项工作提供了一种建立在分子理论基础上的发现SARS-CoV-2缺乏gydF4y2BaORF8gydF4y2Ba与减少COVID-19的严重性。gydF4y2Ba
主要gydF4y2Ba
严重急性呼吸系统综合症冠状病毒2 (SARS-CoV-2)已被证明是一个高度致命的病毒导致毁灭性的和全球大流行。虽然最近的研究结果表明,SARS-CoV-2感染会影响表观遗传调控gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba14gydF4y2Ba并且抑制了天生的抗病毒宿主细胞反应gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba,目前尚不清楚如何发生这种情况。在极少数情况下,干扰其他高毒性病毒宿主细胞通过模仿宿主细胞蛋白质的表观遗传调控gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,特别是组蛋白gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8 gydF4y2Ba。组蛋白DNA包装成复杂的结构和功能,在这一过程中,基因组进行访问控制。组蛋白蛋白质是由广泛的转录后修饰改性(天车)动态控制基因表达的调节gydF4y2Ba9gydF4y2Ba,gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba。组蛋白模仿允许病毒破坏宿主细胞调节基因的表达和应对的能力有效地感染。但是,没有确认的病例组蛋白模仿曾被报道在冠状病毒。尽管SARS-CoV-2可能使用许多机制干扰宿主细胞的功能,我们检查它是否使用染色质组蛋白拟态扰乱监管和转录对感染的反应。gydF4y2Ba
ORF8包含一组蛋白H3模仿gydF4y2Ba
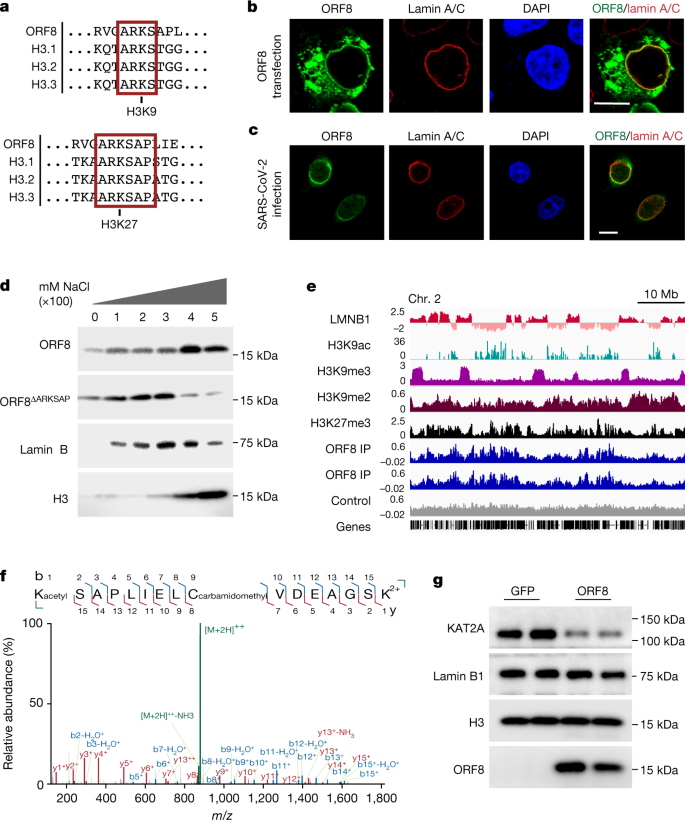
确定是否使用组蛋白拟态SARS-CoV-2,我们首先进行了科学的比较,SARS-CoV-2病毒蛋白质gydF4y2Ba18gydF4y2Ba与人类所有组蛋白蛋白质(扩展数据图。gydF4y2Ba1 a, bgydF4y2Ba)。大多数SARS-CoV-2蛋白质非常类似于在冠状病毒毒株导致前面的主要冠爆发与蛋白质编码的例外gydF4y2BaORF3bgydF4y2Ba和gydF4y2BaORF8gydF4y2Ba最发散,ORF8 SARS-CoV-2(参考文献gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba)。值得注意的是,我们发现一个编码的蛋白质氨基酸50 - 55之间相同的匹配gydF4y2BaORF8gydF4y2Ba和关键区域组蛋白H3氨基端尾(图。gydF4y2Ba1gydF4y2Ba)。此外,ORF8对齐到一个长序列的氨基酸(6残留物)比任何先前描述和验证组蛋白拟态gydF4y2Ba4 gydF4y2Ba,gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba21gydF4y2Ba或一个假定的组蛋白模仿SARS-CoV-2包膜蛋白gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba(扩展数据图。gydF4y2Ba1 c, dgydF4y2Ba)。ORF8的晶体结构的基础上,这些残留物位于一个无序区域表面的蛋白质ORF8单体gydF4y2Ba24gydF4y2Ba。最咄咄逼人地,主题包含了“方舟”序列,发现在两个不同的地点在组蛋白H3尾巴(图。gydF4y2Ba1gydF4y2Ba),并建立了H3内最重要的监管的地区之一。H3方舟网站都有多个多功能天车的修改,包括mono - di - trimethylation和乙酰化在H3赖氨酸9 (H3K9me和H3K9ac)和H3赖氨酸27 (H3K27me和H3K27ac)。这种氨基酸拉伸缺席前冠gydF4y2BaORF8gydF4y2Ba编码的蛋白质生成之前和之后都删除gydF4y2BaORF8agydF4y2Ba和gydF4y2BaORF8bgydF4y2Ba25gydF4y2Ba但是存在于蝙蝠SARS-CoV-2和变异的关注(扩展数据图。gydF4y2Ba1 e, fgydF4y2Ba)。在感染ORF8高度表达gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba,gydF4y2BaORF8gydF4y2Ba转录表达水平高于组蛋白H3和ORF8蛋白质表达水平超过20%以上的蛋白质最丰富的组蛋白H3 24 h内的感染gydF4y2Ba28gydF4y2Ba(扩展数据图。gydF4y2Ba1 g hgydF4y2Ba)。最后,蛋白质组学鉴定SARS-CoV-2蛋白质绑定合作伙伴表明ORF8结合DNA甲基转移酶1 (DNMT1)gydF4y2Ba22gydF4y2Ba,gydF4y2Ba29日gydF4y2Ba。gydF4y2Ba
一个gydF4y2Ba,ORF8包含一个方舟图案50氨基酸相匹配的组蛋白H3尾巴。gydF4y2BabgydF4y2Ba核纤层蛋白A / C染色HEK293T细胞转染表达Strep-ORF8。gydF4y2BacgydF4y2Ba、ORF8和核纤层蛋白A / C染色SARS-CoV-2-infected A549gydF4y2BaACE2gydF4y2Ba细胞在莫伊= 1,48 h后感染。gydF4y2BadgydF4y2Ba连续盐提取HEK293T细胞表达ORF8或ORF8gydF4y2BaΔARKSAPgydF4y2Ba。gydF4y2BaegydF4y2Ba,基因跟踪ORF8 ChIP-seq规范化输入控件。gydF4y2BafgydF4y2Ba的,有针对性的质谱分析trypsin-digested ORF8表明ORF8在赖氨酸乙酰化52。完整的肽或前体为879.9508gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba2 +收费孤立和分散。串联质谱光谱显示unfragmented前体(绿色)匹配产品的离子的质量误差内10 ppm。片段强度是相对于整个最高的离子强度gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba的范围内。每个片段显示颜色,字母和数字的序列片段中包含较大的肽(上)。y(红色)和b(蓝色)表明C -和N-terminus-matched碎片,碎片。gydF4y2BaggydF4y2Ba,ORF8表达式结果KAT2A水平下降。规模的酒吧、10μm。凝胶源数据,请参阅补充图。gydF4y2Ba1 b lgydF4y2Ba。gydF4y2Ba
确定ORF8功能组蛋白模拟,我们开始通过检查它的胞内定位。尽管ORF8没有一个定义良好的核本地化序列,它是15 kDa的大小,因此足够小,扩散到细胞核。我们转染HEK293T细胞构造编码Strep-tagged ORF8和可视化ORF8 Strep-Tactin-conjugated荧光探针。外貌虽然ORF8本地化是变量,ORF8通常是位于细胞质和细胞核的边缘在使用免疫荧光(图gydF4y2Ba1 bgydF4y2Ba),如之前报道gydF4y2Ba30.gydF4y2Ba使用时,在细胞质和细胞核细胞分离(扩展数据图。gydF4y2Ba2gydF4y2Ba)。鉴于观察到的表达模式,我们下一个被问及ORF8 colocalizes核纤层蛋白的蛋白质。我们发现ORF8与核纤层蛋白B1和核纤层蛋白A / C细胞中转染表达ORF8(无花果。gydF4y2Ba1 bgydF4y2Ba和扩展数据图。gydF4y2Ba2 b, cgydF4y2Ba)。接下来,我们感染一个A549肺epithelial-derived ACE2受体(A549细胞株表达gydF4y2BaACE2gydF4y2Ba)与SARS-CoV-2,染色细胞与一个特定于ORF8抗血清(扩展数据图。gydF4y2Ba2 d, egydF4y2Ba),证实感染细胞(图相似的表达模式。gydF4y2Ba1 cgydF4y2Ba)。值得注意的是,虽然其他功能提出了ORF8(参考文献gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31日gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba),一个潜在的角色ORF8宿主细胞的细胞核和专门调节染色质没有被探索。gydF4y2Ba
我们下一个测试是否ORF8与染色质通过增加盐浓度检查染色质绑定。我们发现ORF8分离染色质分数的盐浓度类似核纤层蛋白和组蛋白蛋白质分离(无花果。gydF4y2Ba1 dgydF4y2Ba)。相比之下,ORF8删除ARKSAP主题(ORF8gydF4y2BaΔARKSAPgydF4y2Ba)在低盐浓度和分离出现染色质分数较低水平相比ORF8与这个主题(无花果。gydF4y2Ba1 dgydF4y2Ba和扩展数据图。gydF4y2Ba2 f, ggydF4y2Ba),表明假定的组蛋白模拟网站影响的强度与染色质ORF8协会。我们下一个执行染色质免疫沉淀反应与测序(ChIP-seq) ORF8是否与基因组DNA, ORF8 associates。尽管ORF8没有明确定义的山峰,ORF8免疫沉淀反应显示浓缩在输入(图。gydF4y2Ba1 egydF4y2Ba)和ORF8浓缩在特定基因组区域,尤其是那些与H3K27me3(扩展数据图。gydF4y2Ba2 h-kgydF4y2Ba)。gydF4y2Ba
ORF8的定位的基础上核的外围及其与染色质协会(观察使用生化和测序的方法),我们进一步测试是否与lamin-complex蛋白质ORF8 associates。我们发现ORF8 co-immunoprecipitated与核纤层蛋白B1,组蛋白H3和HP1α相关核纤层蛋白的蛋白质和蛋白质组蛋白(扩展数据图。gydF4y2Ba3gydF4y2Ba)。互惠co-immunoprecipitation核纤层蛋白B1和组蛋白H3证实ORF8绑定(扩展数据图。gydF4y2Ba3 bgydF4y2Ba)。接下来,我们测试是否也ORF8 co-immunoprecipitates histone-modifying酶这一目标在组蛋白H3方舟的主题。我们发现ORF8与组蛋白乙酰转移酶KAT2A(也称为GCN5),目标H3K9(无花果。gydF4y2Ba1 fgydF4y2Ba)。尽管ORF8和ORF8gydF4y2BaΔARKSAPgydF4y2Ba免疫沉淀反应与先前建立的细胞质绑定合作伙伴,hla a2 (ref。gydF4y2Ba30.gydF4y2Ba),我们没有检测ORF8gydF4y2BaΔARKSAPgydF4y2Ba与染色质蛋白,这表明ARKSAP主题加强ORF8协会与染色质蛋白质(扩展数据图。gydF4y2Ba3 c, dgydF4y2Ba)。进一步,ORF8没有绑定到BRD4优先结合乙酰化组蛋白H4(扩展数据图。gydF4y2Ba3 egydF4y2Ba)。最后,我们使用质谱识别额外的约束力的合作伙伴之外发现通过候选人的方法集中在染色质修饰符(补充表gydF4y2Ba1克ydF4y2Ba)。全细胞溶解产物主要是耗尽染色质蛋白质被用在一个互补的方法主要是胞质蛋白被发现。然而,转录因子SP2被发现和证实了绑定到ORF8 co-immunoprecipitation(扩展数据图。gydF4y2Ba3 fgydF4y2Ba)。gydF4y2Ba
的基础上与KAT2A ORF8 associates的观察,我们使用有针对性的质谱分析,确定该ORF8类似于组蛋白组蛋白模拟网站修改。使用自底向上方法,ORF8从细胞纯化,减少、烷基化和消化。与液相色谱分离是紧随其后的是平行反应监测质谱(LC-PRM-MS)针对可能未经改装的形式的ORF8常见组蛋白,包括丝氨酸磷酸化和赖氨酸monomethylation dimethylation, trimethylation和乙酰化作用。这些目标,修改的赖氨酸乙酰化和识别。乙酰化肽含有大量转移+ 42 Da,证明离子几乎完全覆盖所有可能的产品。高分辨率质谱分化trimethylated肽的前体和匹配所有产品离子的质量误差内10 ppm(无花果。gydF4y2Ba1 fgydF4y2Ba和扩展数据图。gydF4y2Ba3 ggydF4y2Ba)。这表明ORF8提出方舟内是赖氨酸乙酰化组蛋白模拟网站,类似于组蛋白H3。值得注意的是,存在内的乙酰化赖氨酸ARKSAP主题可能是不兼容ORF8二聚作用,包括在这个残渣形成氢键相互作用gydF4y2Ba24gydF4y2Ba,从而表明ORF8可以作为一个单体细胞内存在。最后,鉴于ORF8促进另一个绑定合作伙伴的溶酶体降解gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,我们检查是否ORF8同样影响chromatin-associated蛋白质。ORF8表达导致明显减少大量的KAT2A(无花果。gydF4y2Ba1克gydF4y2Ba),而核水平叶片蛋白质和lamina-associated异染色质持平或略有增加(扩展数据图。gydF4y2Ba3 h lgydF4y2Ba)。这些发现表明,不仅ORF8如乙酰转移酶与蛋白质,但它也可能是修改它们类似于组蛋白H3和诱发他们的退化。总的来说,这些发现表明,ORF8将作为组蛋白模拟的基础上,其与染色质和chromatin-modifying酶和其消耗的组蛋白乙酰转移酶KAT2A的能力。gydF4y2Ba
ORF8扰乱了染色质调节gydF4y2Ba
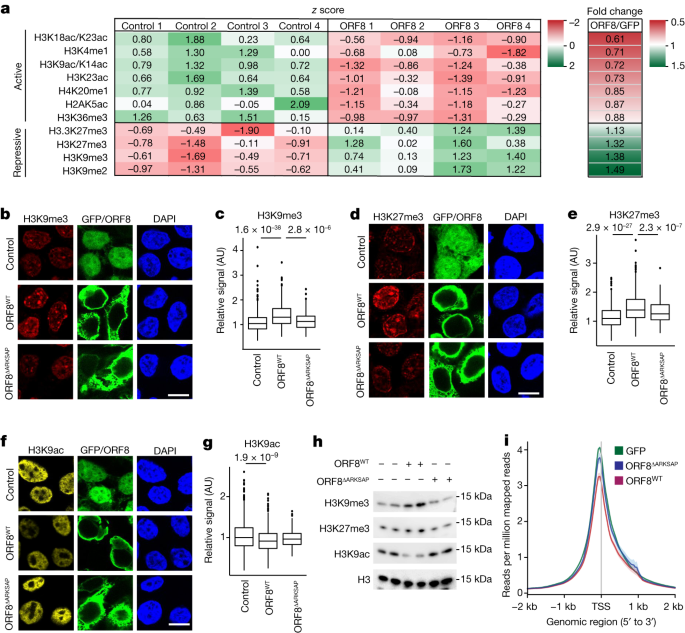
我们下一个检查ORF8是否会影响组蛋白表达天车使用一个公正的质谱分析方法。HEK293T细胞转染与控制质粒编码绿色荧光蛋白或质粒编码ORF8喉炎的标签。转染细胞,GFP荧光或Strep-Tactin-conjugated识别荧光探针,孤立使用fluorescence-activated细胞排序(流式细胞仪)。组蛋白通过酸提取、纯化和自底向上的无偏质谱进行量化检测组蛋白天车。值得注意的是,组蛋白修饰和转录镇压增加相关,而大量的组蛋白修饰与基因表达活跃在细胞表达ORF8耗尽(无花果。gydF4y2Ba2gydF4y2Ba)。特别是,H3方舟内修改图案非常破坏。例如,包含甲基化的多肽H3K9 H3K27,与转录镇压,显示,应对ORF8强劲增加丰富的表情。相反,包含H3K9ac和H3K14ac肽,这两个有一个的链接活跃的基因表达,表现为应对ORF8丰度降低的表情。这些数据支持作用ORF8作为假定的组蛋白模拟,证明它能够干扰组蛋白天车监管在组蛋白在许多关键的站点。gydF4y2Ba
一个gydF4y2Ba组蛋白的质谱分析铝电解控制(GFP-expressing)或ORF8-expressing HEK293T细胞通过流式细胞仪分离。的gydF4y2BazgydF4y2Ba分数和褶皱变化所示修改显著改变以应对ORF8表达式,发现超过1%的总肽丰富和完善的功能(补充表所示的全部结果gydF4y2Ba2gydF4y2Ba)。gydF4y2BabgydF4y2Ba- - - - - -gydF4y2BaggydF4y2Ba,免疫荧光分析HEK293T细胞转染GFP表达或Strep-ORF8表明ORF8表达增加H3K9me3 (gydF4y2BabgydF4y2Ba,gydF4y2BacgydF4y2Ba)和H3K27me3 (gydF4y2BadgydF4y2Ba,gydF4y2BaegydF4y2Ba),同时减少H3K9ac (gydF4y2BafgydF4y2Ba,gydF4y2BaggydF4y2Ba)。相反,ORF8删除的组蛋白模拟网站ARKSAP (ORF8gydF4y2BaΔARKSAPgydF4y2Ba)不影响这些组蛋白天车。gydF4y2BangydF4y2Ba= 614 (GFP), 497 (ORF8)和170 (ORF8gydF4y2BaΔARKSAPgydF4y2BaH3K9me3)细胞;616年、550年和154年为H3K27me3细胞;到666年,568年和170年为H3K9ac编译从三个独立的转染细胞。单向方差分析与事后两面gydF4y2BatgydF4y2Ba测试和Bonferroni调整。gydF4y2BahgydF4y2Ba组蛋白,免疫印迹分析隔绝FACS-sorted转染细胞。gydF4y2Ba我gydF4y2BaATAC-seq HEK293T细胞表达绿色荧光蛋白,ORF8或ORF8gydF4y2BaΔARKSAPgydF4y2Ba通过流式细胞仪分离。读取每百万映射读取周边转录起始站点(TSS)的表达基因都是平均。gydF4y2BangydF4y2Ba= 2独立复制。原始的屁股补充图所示。gydF4y2Ba1克ydF4y2Ba。规模的酒吧、10μm。流式细胞仪控制策略和细胞数量孤立补充图所示。gydF4y2Ba2gydF4y2Ba。凝胶源数据,请参阅补充图。gydF4y2Ba1 egydF4y2Ba。盒子情节集中在中位数与边界第25和第75百分位,定义的最小和最大平均±1.5×四分位范围和胡须扩展范围的最低和最高价值。gydF4y2Ba
确认质谱分析发现,我们用免疫荧光成像来检测甲基化和乙酰化H3K9 H3K27。我们发现细胞表达ORF8展出增加H3K9me3 H3K27me3和减少H3K9ac染色与转染质粒(图与控制。gydF4y2Ba2 b-ggydF4y2Ba)。ORF8表达没有明显破坏H3K27ac,全球乙酰化作用,H3S10磷酸化,H3K9me2或核纤层蛋白B(扩展数据图。gydF4y2Ba4 a、bgydF4y2Ba)。尽管ORF8gydF4y2BaΔARKSAPgydF4y2Ba在相似的水平表示ORF8(扩展数据图。gydF4y2Ba4摄氏度gydF4y2Ba),它不会增加H3K9me3 H3K27me3和非重要中间的影响H3K9ac(无花果。gydF4y2Ba2 b-ggydF4y2Ba)。接下来,我们检查获得的突变gydF4y2BaORF8gydF4y2Ba通常存在于编码S84L替换(ORF8 SARS-CoV-2菌株gydF4y2BaS84LgydF4y2Ba)。这个网站不太可能影响蛋白质的稳定性gydF4y2Ba31日gydF4y2Ba,gydF4y2Ba39gydF4y2Ba和谎言在组蛋白模拟区域,因此替代不会影响ORF8调节组蛋白天车的能力。表达ORF8gydF4y2BaS84LgydF4y2Ba也增加了H3K9me3 H3K27me3水平,同时减少H3K9ac(扩展数据图。gydF4y2Ba4 d-fgydF4y2Ba),这表明,正如预测的那样,这种常见的变异并不改变ORF8的组蛋白模拟功能。同样,另一个非结构化的six-residue删除地区ORF8与氨基酸组成相似,但不同的序列(AGSKSP)作为组蛋白模拟网站并不影响ORF8破坏组蛋白调控的能力(扩展数据图。gydF4y2Ba4 ggydF4y2Ba)。gydF4y2Ba
我们下一个试图证实这些发现使用独立的方法。确保平等的表达水平ORF8 ORF8gydF4y2BaΔARKSAPgydF4y2Ba我们孤立的转染细胞,流式细胞仪(扩展数据图。gydF4y2Ba5gydF4y2Ba)。然后我们孤立的组蛋白通过酸萃取和证实ORF8增加H3K9me3 H3K27me3和已故H3K9ac ARKSAP-dependent地通过免疫印迹分析(无花果。gydF4y2Ba2 hgydF4y2Ba)。同样,CUT&Tag H3K9ac证明ORF8测序,但不是ORF8gydF4y2BaΔARKSAPgydF4y2Ba已故H3K9ac(扩展数据图。gydF4y2Ba5 b, cgydF4y2Ba)。最后,分析transposase-accessible染色质的高通量测序(ATAC-seq)证明ORF8,但不是ORF8gydF4y2BaΔARKSAPgydF4y2Ba,减少了染色质可访问性(扩展数据图。gydF4y2Ba5 dgydF4y2Ba和无花果。gydF4y2Ba2我gydF4y2Ba)。H3K9ac和染色质的变化可访问性主要是全球但特别明显的基因高表达(扩展数据图的中间。gydF4y2Ba5种情况gydF4y2Ba)。gydF4y2Ba
确定这些染色质中断影响基因表达,我们使用RNA序列(RNA-seq)定义差异表达基因转染细胞(扩展数据图。gydF4y2Ba6 a - cgydF4y2Ba)。而ORF8和ORF8gydF4y2BaΔARKSAPgydF4y2Ba共享的差异表达基因的一个子集,组蛋白的存在导致更少的动态模拟图案基因表达变化。不同的基因群体也ORF8和ORF8之间的差异表达gydF4y2BaΔARKSAPgydF4y2Ba,相对于ORF8 ORF8降低基因表达gydF4y2BaΔARKSAPgydF4y2Ba,特别是高表达基因(扩展数据图。gydF4y2Ba6 d - igydF4y2Ba和补充表gydF4y2Ba3 gydF4y2Ba)。基因表达下调的反应相对于ORF8 ORF8表达式gydF4y2BaΔARKSAPgydF4y2Ba也有更高的基础水平H3K9ac和可访问性大于基因调节(扩展数据图。gydF4y2Ba6 j, kgydF4y2Ba),这表明他们可能更敏感H3K9ac损耗。在一起,这些结果支持模型ORF8有多个功能正如前面提出的gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31日gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba40gydF4y2Ba和激活基因表达的途径,尤其是在缺乏ARKSAP图案。然而,存在ARKSAP主题抑制了宿主细胞转录反应和减少可访问性和H3K9ac高表达的基因。在一起,这些数据定义一个角色ORF8破坏宿主细胞组蛋白的天车通过一个新的组蛋白的拟态的方舟在组蛋白H3图案。gydF4y2Ba
SARS-CoV-2扰乱了染色质调节gydF4y2Ba
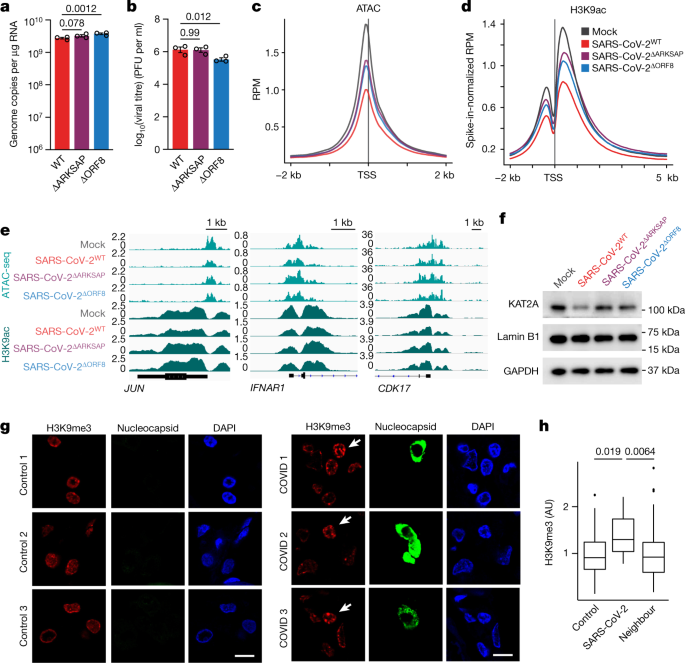
有显示ORF8足以破坏染色质调节,我们接下来检查ORF8对组蛋白的影响铝电解监管在病毒感染的上下文中。我们生成一个重组突变SARS-CoV-2病毒删除gydF4y2BaORF8gydF4y2Ba(SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba)利用cDNA反向遗传学系统gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba。我们感染A549gydF4y2BaACE2gydF4y2Ba细胞与SARS-CoV-2或SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba和病毒基因组的水平相比,存在和缺乏ORF8传染性病毒生产。因为他们ACE2受体的过度表达,这些细胞很容易和快速感染SARS-CoV-2,从而提供了一个理想的系统,比较细胞反应突变形式的病毒没有感染的混杂因子不同的利率。没有基因组拷贝数的差异或病毒滴定度检测在24小时,观察,只有细微的差别在48 h(扩展数据图。gydF4y2Ba7 a、bgydF4y2Ba和无花果。gydF4y2Ba3 a, bgydF4y2Ba),允许直接比较这两种病毒感染后在这些早期的时间点。我们因此感染A549gydF4y2Ba王牌gydF4y2Ba细胞与SARS-CoV-2或SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba和使用ChIP-seq激增正常化(ChIP-RX)允许全球变化的检测组蛋白天车。我们发现SARS-CoV-2感染导致H3K9me3强劲增长和H3K27me3相比mock-infected细胞(扩展数据图。gydF4y2Ba7汉英gydF4y2Ba),镜像ORF8表达的影响。然而,删除gydF4y2BaORF8gydF4y2Ba大大减弱这种效果,表明SARS-CoV-2在压抑的组蛋白修饰的效果在一定程度上是由于ORF8表达式。同样,ATAC-seq证明感染野生型SARS-CoV-2导致大量SARS-CoV-2染色质缩合而感染gydF4y2BaΔORF8gydF4y2Ba导致一个中间表型。最后,ChIP-RX表明SARS-CoV-2感染导致H3K9ac下降,这效果又减毒细胞感染SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba(无花果。gydF4y2Ba3 d, egydF4y2Ba)。这些数据表明,ORF8有助于SARS-CoV-2感染的影响在宿主细胞染色质可访问性和组蛋白修饰。gydF4y2Ba
一个gydF4y2Ba,gydF4y2BabgydF4y2Ba与定量逆转录PCR(存在)SARS-CoV-2基因的表达分析gydF4y2BaRDRPgydF4y2Ba(gydF4y2Ba一个gydF4y2Ba)和空斑实验分析病毒滴定度(gydF4y2BabgydF4y2Ba在A549gydF4y2Ba王牌gydF4y2Ba与野生型细胞在感染后48 h SARS-CoV-2 (SARS-CoV-2gydF4y2BaWTgydF4y2Ba),SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba或SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba在莫伊= 1。双向方差分析与Dunnett多重比较的测试(额外的时间点补充表所示gydF4y2Ba4 gydF4y2Ba)。代表两个独立的感染。PFU,点状单位。gydF4y2BacgydF4y2Ba,gydF4y2BadgydF4y2BaATAC-seq (gydF4y2BacgydF4y2Ba)和H3K9ac ChIP-RX (gydF4y2BadgydF4y2Ba)的A549gydF4y2Ba王牌gydF4y2Ba细胞与SARS-CoV-2gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba,SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba或模拟感染48 h后感染。莫伊= 1。gydF4y2BangydF4y2BaATAC-seq除了= 3gydF4y2BangydF4y2Ba为SARS-CoV-2 = 2gydF4y2BaΔARKSAPgydF4y2Ba。gydF4y2BangydF4y2BaChIP-RX除了= 3gydF4y2BangydF4y2Ba为SARS-CoV-2 = 2gydF4y2BaWTgydF4y2Ba。RPM,读取每百万。gydF4y2BaegydF4y2Ba,ChIP-seq和ATAC-seq基因信号通路相关基因的病毒的反应。gydF4y2BafgydF4y2Ba免疫印迹分析KAT2A A549gydF4y2Ba王牌gydF4y2Ba与野生型和突变SARS-CoV-2病毒感染后细胞。gydF4y2BaggydF4y2Ba,事后从患者肺组织COVID-19彩色H3K9me3和核衣壳蛋白识别SARS-CoV-2-infected细胞。箭头表示被感染的细胞。gydF4y2BahgydF4y2Ba、量化的H3K9me3相比,感染细胞与邻近的细胞和组织的控制。gydF4y2BangydF4y2Ba= 12感染细胞,131感染邻近细胞从三个COVID-19患者和60细胞从三个人的控制。单向方差分析与事后两面gydF4y2BatgydF4y2Ba测试和Bonferroni调整。规模的酒吧、10μm。凝胶源数据,请参阅补充图。gydF4y2Ba1阿gydF4y2Ba。盒子情节集中在中位数在第25和第75百分位数与范围,定义的最小和最大平均±1.5×四分位范围和胡须扩展范围的最低和最高价值。gydF4y2Ba
因为很可能ORF8有多个对细胞功能的影响,最近puplications的基础上gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31日gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba,gydF4y2Ba40gydF4y2Ba和我们机械的数据,我们也试图确定这些影响特别由于组蛋白模拟主题。要做到这一点,我们生成的一种突变SARS-CoV-2删除只有ARKSAP主题(SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba)。在A549gydF4y2BaACE2gydF4y2Ba细胞,SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba复制类似于野生型病毒(无花果。gydF4y2Ba3 a, bgydF4y2Ba),但大大减轻感染的影响染色质可亲地H3K9ac,匹配的影响gydF4y2BaORF8gydF4y2Ba删除(图。gydF4y2Ba3汉英gydF4y2Ba)。鉴于SARS-CoV-2 H3K9ac和健壮的影响ORF8耗尽KAT2A(图的能力。gydF4y2Ba1克gydF4y2Ba),我们还研究了感染KAT2A水平的影响。野生型SARS-CoV-2感染KAT2A表达减少,而SARS-CoV-2感染gydF4y2BaΔORF8gydF4y2Ba或SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba没有(图。gydF4y2Ba3 fgydF4y2Ba)。这些数据表明,ORF8,特别是在ORF8 ARKSAP图案,有助于SARS-CoV-2在宿主细胞表观基因组的影响。gydF4y2Ba
以确保宿主细胞中观察到的差异之后SARS-CoV-2调控染色质和SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba感染并不是由于利率的任何细微的差异之间的感染病毒,我们试图进一步证实了这些发现使用一个方法独立于被感染细胞的数量。我们用免疫细胞化学染色组蛋白修饰的兴趣,使用染色双链RNA(极)来识别和专门检查感染的细胞。在感染后24小时,细胞感染SARS-CoV-2增加了H3K9me3 H3K27me3和减少H3K9ac相比mock-infected细胞或邻近细胞无毒性(扩展数据图。gydF4y2Ba8 fgydF4y2Ba)。作为ChIP-seq数据观察,这种效应在很大程度上失去了与删除gydF4y2BaORF8gydF4y2Ba。gydF4y2Ba
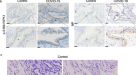
是否类似的效果也发生在患者人群的上下文中,我们获得死后肺组织样本三冠状病毒疾病患者2019 (COVID-19)和匹配控制。我们染色的组织为SARS-CoV-2 H3K9me3以及核衣壳蛋白识别感染细胞。我们发现,在所有患者样本,感染细胞显示增加H3K9me3染色与邻近的细胞在相同的组织,以及与控制组织(无花果。gydF4y2Ba3 g hgydF4y2Ba和扩展数据图。gydF4y2Ba8 ggydF4y2Ba)。当样品的可用性限制了结论,可以从这个试验,这一发现表明组蛋白天车也打乱了严重COVID-19疾病患者。总之,我们发现SARS-CoV-2感染组蛋白的影响铝电解和染色质压实要求ORF8表达式和镜像ARKSAP-dependent ORF8的影响。gydF4y2Ba
SARS-CoV-2对转录的影响gydF4y2Ba
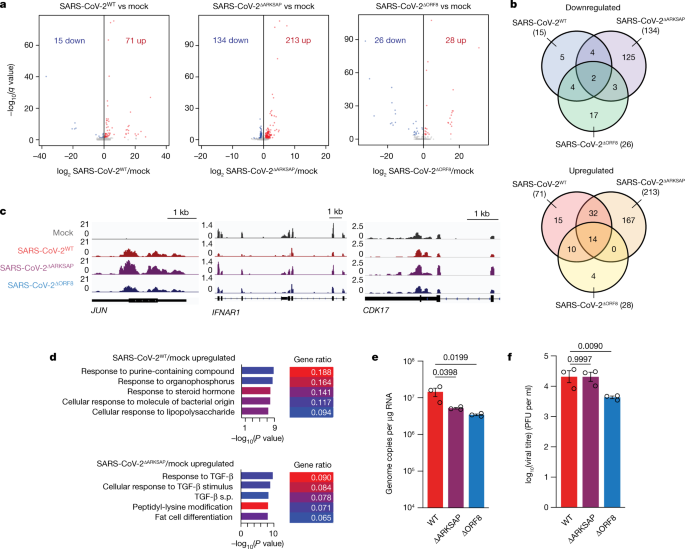
接下来,我们研究如何改变组蛋白天车通过ChIP-seq发现与使用RNA-seq基因表达。所有病毒包含类似数量的读取,唯一的区别在SARS-CoV-2转录表达gydF4y2BaORF8gydF4y2Ba在SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba(扩展数据图。gydF4y2Ba9模拟gydF4y2Ba)。然而,在野生型病毒,gydF4y2BaORF8gydF4y2Ba成绩单是高度表达和更丰富的组蛋白H3-encoding成绩单(扩展数据图。gydF4y2Ba9 egydF4y2Ba)。有趣的是,在感染早期,三个病毒测试每个中断了一组不同的基因,这表明组蛋白的模仿主题改变转录响应感染(无花果。gydF4y2Ba4得了gydF4y2Ba)。病毒感染后48 h,所有三个由绝大多数映射读取和导致强大的基因表达的变化与mock-infected细胞(扩展数据图。gydF4y2Ba9 c、f, ggydF4y2Ba)。基因的官能团大部分由三种病毒感染也不同,说明不同的宿主细胞反应在早期的时间点(无花果。gydF4y2Ba4 dgydF4y2Ba和扩展数据图。gydF4y2Ba10gydF4y2Ba)。这是考虑到野生型SARS-CoV-2和SARS-CoV-2显著gydF4y2BaΔARKSAPgydF4y2Ba有几乎相同的数字拷贝和复制在A549利率吗gydF4y2BaACE2gydF4y2Ba细胞(图。gydF4y2Ba3 a, bgydF4y2Ba不同的转录反应),因此不太可能是由于不同数量的细胞感染或感染细胞内的病毒载量。有趣的是,SARS-CoV-2的直接比较gydF4y2BaΔORF8gydF4y2Ba和SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba还显示不同的基因表达变化和官能团浓缩(扩展数据图。gydF4y2Ba10 b, cgydF4y2Ba),再次表明,ORF8可能有多个功能之外的ARKSAP域。此外,基因表达的变化应对感染与H3K9ac(扩展数据图的变化。gydF4y2Ba10 d-fgydF4y2Ba)。值得注意的是,这些数据进一步支持最新发现表明SARS-CoV-2导致早期转录反应有限gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba43gydF4y2Ba并证明ORF8 ARKSAP域与基因表达的变化。gydF4y2Ba
一个gydF4y2Ba差异基因表达分析RNA-seq A549gydF4y2BaACE2gydF4y2Ba细胞感染SARS-CoV-2后24小时gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba或SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba与模拟相比,感染。莫伊= 1。显著差异表达基因所示蓝色(下调)和红色(调节)。gydF4y2BangydF4y2Ba= 3。意义基于DESeq2分析和多重比较修正。gydF4y2BabgydF4y2Ba,差异表达基因的重叠感染SARS-CoV-2gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba或SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba。gydF4y2BacgydF4y2Ba,基因信号通路相关基因的病毒的反应。gydF4y2BadgydF4y2Ba顶级基因本体论(去)条款供SARS-CoV-2基因调节gydF4y2BaWTgydF4y2Ba和SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba感染。基于Benjamini-Hochberg-adjusted clusterProfiler分析的意义gydF4y2BaPgydF4y2Ba值。gydF4y2BaegydF4y2Ba,gydF4y2BafgydF4y2Ba,存在分析SARS-CoV-2基因的表达gydF4y2BaRDRPgydF4y2Ba(gydF4y2BaegydF4y2Ba)和空斑实验分析病毒滴定度(gydF4y2BafgydF4y2Ba)在iAT2肺细胞在感染后48 h SARS-CoV-2gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba或SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba在莫伊= 1。gydF4y2BangydF4y2Ba= 3复制。单向(gydF4y2BaegydF4y2Ba)或双向(gydF4y2BafgydF4y2Ba)与Dunnett多重比较的方差分析测试(额外的时间点gydF4y2BafgydF4y2Ba和所有复制补充表所示gydF4y2Ba4 gydF4y2Ba)。酒吧情节表明意味着±s.e.m。gydF4y2Ba
考虑到强劲的影响gydF4y2BaORF8gydF4y2Ba删除在宿主细胞染色质调节转录对感染的反应,我们试图测试是否ORF8介导的复制SARS-CoV-2使用生理相关的细胞类型。人类诱导多能干细胞肺肺泡II型(iAT2)肺细胞gydF4y2Ba44gydF4y2Ba感染了SARS-CoV-2, SARS-CoV-2gydF4y2BaΔORF8gydF4y2Ba或SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba(感染复数(MOI) = 1)。值得注意的是,我们观察到,同时降低了基因组拷贝数变异病毒感染后48 h在大多数复制(无花果。gydF4y2Ba4 egydF4y2Ba和补充表gydF4y2Ba4 gydF4y2Ba),这表明ORF8,特别是ARKSAP领域,影响SARS-CoV-2宿主细胞基因组复制。然而,病毒通过斑块化验证明SARS-CoV-2滴定度测量gydF4y2BaΔORF8gydF4y2Ba生成传染性微粒比野生型SARS-CoV-2而SARS-CoV-2少gydF4y2BaΔARKSAPgydF4y2Ba出现类似于野生型病毒,甚至在某些情况下表现出更多菌斑的形成(图。gydF4y2Ba4 fgydF4y2Ba和补充表gydF4y2Ba4 gydF4y2Ba)。拟合与以前的工作表明ORF8影响内质网(ER)应力路径gydF4y2Ba32gydF4y2Ba,这可能表明ORF8 ARKSAP-independent函数,可以促进病毒粒子的形成。总的来说,这项工作提出了一种特定SARS-CoV-2蛋白质和表观遗传之间的联系中断发生感染和提供了一个机械的解释越来越多的证据gydF4y2Ba12gydF4y2Ba,gydF4y2Ba13gydF4y2Ba,gydF4y2Ba45gydF4y2Ba表观基因中断导致COVID-19的严重程度。gydF4y2Ba
讨论gydF4y2Ba
这里描述的工作确定了一个新的组蛋白在感染SARS-CoV-2模仿和定义了一个机制,通过这个机制SARS-CoV-2行为破坏宿主细胞染色质的监管。我们发现SARS-CoV-2蛋白质编码gydF4y2BaORF8gydF4y2Ba基因包含一个方舟主题,ORF8扰乱了组蛋白表达天车的监管。ORF8与chromatin-associated相关蛋白,组蛋白和核板本身就是在组蛋白模拟主题类似于组蛋白乙酰化。ORF8表达式扰乱了多个重要的组蛋白天车和促进染色质压实,而ORF8缺乏组蛋白模拟主题不。此外,SARS-CoV-2感染人类细胞系和死后患者肺组织引起类似的全球中断染色质通过组蛋白模拟表演部分。此外,删除的gydF4y2BaORF8gydF4y2Ba基因或序列编码的组蛋白模拟影响宿主细胞转录反应SARS-CoV-2感染。最后,失去ORF8减少SARS-CoV-2复制人类诱导性多功能干细胞iAT2肺细胞而损失的组蛋白模拟主题特别影响病毒基因组拷贝数。gydF4y2Ba
值得注意的是,在染色质ORF8中断的作用在感染早期不符合其他提出角色ORF8在其他细胞车厢或感染的后期gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31日gydF4y2Ba,gydF4y2Ba32gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba46gydF4y2Ba并不排除其他转录机制提出中断响应SARS-CoV-2 (ref。gydF4y2Ba23gydF4y2Ba)。事实上,我们对模型中,数据点ORF8具有多重功能,包括作为组蛋白模拟主题。删除附件蛋白质的影响从SARS-CoV-2转基因小鼠模型显得复杂,与ORF8损失导致复制和病毒载量下降但有有限的对生存的影响gydF4y2Ba47gydF4y2Ba。然而,COVID-19患者的数据被用来检查一个罕见的382 -核苷酸缺失在新加坡SARS-CoV-2孤立的变化,导致一小部分的损失gydF4y2BaORF7BgydF4y2Ba和大多数的gydF4y2BaORF8gydF4y2Ba基因。这项工作发现,这种SARS-CoV-2变异与温和的感染患者COVID-19和一种改进的干扰素反应gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba。指向我们的发现在人类iAT2肺细胞的损失ORF8作为这些差异的可能原因,并提供一个表观遗传机制的作用在促进SARS-CoV-2 ORF8患者人群中的毒性。最后,本文描述的工作有着至关重要的意义,可以让人们了解新兴病毒株携带缺失和突变gydF4y2BaORF8gydF4y2Ba基因gydF4y2Ba50gydF4y2Ba并在病人COVID-19发病机理。gydF4y2Ba
方法gydF4y2Ba
A549gydF4y2Ba王牌gydF4y2Ba细胞gydF4y2Ba
ACE2-expressing A549细胞生成的如前所述gydF4y2Ba3 gydF4y2Ba。A549gydF4y2BaACE2gydF4y2Ba细胞生长在rpmi - 1640和10%的边后卫和1% penicillin-streptomycin是支原体的免费维护。细胞被感染的莫伊1和固定或细胞溶解在24或48小时后感染。gydF4y2Ba
HEK293T细胞gydF4y2Ba
HEK293T细胞从美国获得类型文化集合(写明ATCC),在DMEM培养(4.5 g LgydF4y2Ba1gydF4y2Ba葡萄糖,gydF4y2BalgydF4y2Ba谷氨酰胺和丙酮酸钠)补充10%的边后卫(Sigma-Aldrich f2442 - 500毫升)和1% penicillin-streptomycin (Gibco, 15140122)和维护自由的支原体。磷酸钙转染是用来介绍质粒DNA编码绿色荧光蛋白,ORF8和突变ORF8 HEK293T细胞。免疫细胞化学实验,细胞被镀聚gydF4y2BadgydF4y2Ba赖氨酸)涂盖玻片。细胞被洗后24 h转染培养基和固定或颗粒状和flash冷冻转染后48 h。细胞被固定用4%多聚甲醛(PFA) PBS 8分钟。颗粒细胞,细胞分离培养板的使用TrypLE表达(Gibco, 12605010)分离试剂、旋转5分钟在180gydF4y2BaggydF4y2Ba和flash在液态氮冷冻。gydF4y2Ba
iAT2细胞gydF4y2Ba
代人为诱导肺泡上皮类型II-like (iAT2)细胞进行了描述gydF4y2Ba44gydF4y2Ba。保持一个稳定的和纯文化iAT2细胞系,SFTPCgydF4y2BatdTomato +gydF4y2Ba细胞分类和连续通道每14 d。细胞生长在使用90%瀑样格式基底膜基质的细胞密度每µl 400细胞。细胞被美联储使用CK + DCI培养基+摇滚第一48 h后分裂抑制剂,然后改为5 d K + DCI媒介CK + DCI媒介7 d紧随其后。每14 d, alveolosphere瀑样通道,瀑样被释放从基底膜基质使用2毫克毫升gydF4y2Ba1gydF4y2BaDispase 1 h在37°C和单个细胞然后生成使用0.05%胰蛋白酶为15分钟37°C。使用锥虫蓝细胞数量和可行性进行评估,最后细胞通过新的人工基底膜滴留给聚合为30分钟37°C公司5%gydF4y2Ba2gydF4y2Ba孵化器,之后凝固基底膜基质细胞被喂以根据板的格式。gydF4y2Ba
生成的二维(2 d)肺泡细胞为病毒感染,当alveolosphere瀑样通道,细胞被镀上预镀1:30人工基底膜板每厘米125000个细胞的细胞密度gydF4y2Ba2gydF4y2Ba使用CK + DCI培养基+第一48 h,岩石抑制剂和中被改为CK + DCI的媒介。镀七十二小时后细胞,细胞被感染SARS-CoV-2病毒使用的莫伊1 48 h。gydF4y2Ba
细胞系验证和测试gydF4y2Ba
细胞系验证如前所述gydF4y2Ba3 gydF4y2Ba。HEK293T和州立E6细胞从写明ATCC获得这个项目的开始。所有被证实是负的细胞株用于支原体和每年两次测试。gydF4y2Ba
ORF8构造gydF4y2Ba
从Addgene ORF8表达质粒了,pLVX-EF1alpha-SARS-CoV-2-orf8-2xStrep-IRES-Puro (Addgene质粒141390)。gydF4y2BaORF8gydF4y2Ba删除构造产生在ORF8骨干利用Pfu涡轮HotStart DNA聚合酶(安捷伦600322 - 51),和引物使用的DNA引物设计特色创建在线PrimerX工具。结构被Sanger测序验证。gydF4y2Ba
SARS-CoV-2感染gydF4y2Ba
病毒的一代gydF4y2Ba
SARS-CoV-2 (USA-WA1/2020应变)是来自贝和传播州立E6细胞。RNA基因组测序,发现是相同的基因库gydF4y2BaMN985325.1gydF4y2Ba。突变病毒生成使用cDNA反向遗传学系统如前所述gydF4y2Ba42gydF4y2Ba。gydF4y2Ba
感染gydF4y2Ba
细胞感染了野生型和突变体的莫伊SARS-CoV-2 1每细胞(A549微升gydF4y2BaACE2gydF4y2Ba每个细胞)或5 PFU (iAT2)如前所述gydF4y2Ba3 gydF4y2Ba。病毒加入细胞1 h在37°C和被删除,取而代之的是媒体。细胞细胞溶解在感染后48 h和RNA是孤立的。所有感染和病毒操作进行了生物安全三级实验室(BSL3)使用适当的防护设备和协议。gydF4y2Ba
病毒生长动力学和斑块化验gydF4y2Ba
生长动力学进行了分析和斑块化验如前所述gydF4y2Ba3 gydF4y2Ba。总之,在指定的时间点,200µl上层清液从细胞收集并存储在−80°C的传染性病毒的滴定。样本稀释血清DMEM和维罗E6细胞吸附到37°C 1 h之前增加了液体覆盖(与2%的边后卫,DMEM 1×丙酮酸钠和0.1%琼脂糖)。3 d后,覆盖被删除和细胞被固定为4% PFA和结晶紫染色斑块的可视化和计数。所有斑块化验进行生物一式三份和技术复制。gydF4y2Ba
病毒基因组中存在量化gydF4y2Ba
病毒基因组RNA收集、存在和量化进行了如前所述gydF4y2Ba3 gydF4y2Ba。总之,在指定的时间点,感染细胞细胞溶解使用RLT +缓冲,提取基因组DNA和RNA删除使用试剂盒RNeasy迷你包(试剂盒,74134)。互补脱氧核糖核酸是使用高容量cDNA逆转录酶生成工具(应用生物系统公司,4368814)。互补脱氧核糖核酸是放大使用特定存在引物针对病毒gydF4y2BaNSP12gydF4y2Ba(向前,5′-GGTAACTGGTATGATTTCG-3′;相反,5′-CTGGTCAAGGTTAATATAGG-3′),智商SYBR绿色Supermix (Bio-Rad, 1708880)和QuantStudio 3 PCR系统(热费希尔)。量化SARS-CoV-2基因组拷贝执行使用标准曲线生成的连续稀释一个已知浓度的pcDNA6B-nCoV NSP12-FLAG构造编码gydF4y2BaRDRPgydF4y2Ba基因(g·斯塔克的礼物,克利夫兰诊所)与XhoI消化后。基因组拷贝数测定QuantStudio 3中使用标准曲线分析软件,并复制数字/微克的RNA计算使用cDNA反应量和输入RNA cDNA反应。gydF4y2Ba
细胞分离gydF4y2Ba
颗粒状细胞被短暂地在冰上融化。缓冲区1(15毫米Tris-HCl (pH值7.5),60毫米氯化钾,15毫米氯化钠,MgCl 5毫米gydF4y2Ba2gydF4y2Ba1毫米CaClgydF4y2Ba2gydF4y2Ba与1毫米PMSF和0.25蔗糖,1毫米德勤和一个完整的蛋白酶抑制剂鸡尾酒平板电脑添加立即使用前)添加到颗粒在大约5倍体积的颗粒,轻轻用移液器吸取上下分离颗粒。样本在冰上孵化为5分钟,其次是增加同等体积的缓冲与0.4% NP-40样品1。样本然后倒置的混合5分钟在4°C。样品将在200年gydF4y2BaggydF4y2Ba在prechilled离心机离心10分钟颗粒细胞核。上层清液(细胞质分数)被转移到一个新管。丸resuspended轻轻地在0.5毫升缓冲区1洗核然后颗粒状的上层的丢弃。核颗粒溶解缓冲区(150毫米氯化钠,50 mM Tris-HCl (pH值8.0),1% NP-40 MgCl和5毫米gydF4y2Ba2gydF4y2BaPMSF 1毫米,1毫米德勤在250 Uµl Benzonase酶gydF4y2Ba1gydF4y2Ba添加使用)前不久被添加到缓冲1的颗粒体积的一半。样品在室温下被孵化thermoshaker直到颗粒完全溶解。Benzonase酶的数量翻了一倍的样品与未溶解的材料20分钟后离开。样本然后在13000 r.p.m.离心20分钟在4°C。上层清液(核分数)收集。样品的浓度是由BCA化验,样本在西方加载缓冲煮10分钟前通过免疫印迹分析。gydF4y2Ba
染色质顺序盐萃取gydF4y2Ba
盐提取进行了描述gydF4y2Ba51gydF4y2Ba。简而言之,一个2×里帕的解决方案是让(100毫米三羟甲基氨基甲烷(pH值8.0),液钠脱氧胆酸盐NP-40 2%和0.5%)和与不同浓度的5 M氯化钠溶液混合生成里帕包含0,100,200,300、400和500毫米氯化钠。颗粒状细胞在缓冲resuspended与蛋白酶抑制剂(0.3蔗糖,60毫米氯化钾,60毫米三羟甲基氨基甲烷(pH值8.0)液,2毫米NP-40 EDTA和0.5%)和旋转在4°C 10分钟。细胞核被离心6000颗粒状gydF4y2BaggydF4y2Ba5分钟在4°C。上层清液被删除并保存,200µl里帕0毫米氯化钠和蛋白酶抑制剂的添加到样本。样品被移液混合和孵化15倍冰离心法在6500前3分钟gydF4y2BaggydF4y2Ba3分钟在4°C。上层清液是保存和里帕的步骤重复氯化钠浓度。样本然后煮熟,用免疫印迹分析。gydF4y2Ba
ATAC-seqgydF4y2Ba
HEK293T细胞被染色和分类隔离转染细胞使用相同的方法如下所述。排序在寒冷的裂解细胞resuspended缓冲区(10µl每10000个细胞;10毫米Tris-Cl (pH值7.5),MgCl氯化钠10毫米,3毫米gydF4y2Ba2gydF4y2Ba,0.1% NP-40(卷/卷),0.1%(卷/期)Tween-20和0.01%(卷/期)洋地黄皂苷)和洗了洗缓冲区(10毫米Tris-Cl (pH值7.5),10毫米氯化钠,MgCl 3毫米gydF4y2Ba2gydF4y2Ba和0.1%(卷/期)Tween-20)。与Tagment换位进行DNA TDE1 (Illumina公司,15027865)。换位反应是清洁与AMPure XP珠子(贝克曼,A63880)和库生成通过PCR NEBNext高保真2×PCR反应混合液(内,M0541)。库大小确认生物分析仪在测序550年NextSeq平台上(40-bp读取长度,搭配)。gydF4y2Ba
感染A549gydF4y2Ba王牌gydF4y2Ba细胞被固定在ATAC-seq集合。协议执行如上除了0.05% Igepal ca - 630添加到裂解缓冲。此外,在转座酶反应后,反向交联的解决方案是添加(50 mM Tris-Cl最终浓度,1毫米EDTA, 1% SDS, 0.2 ng氯化钠和5毫升gydF4y2Ba1gydF4y2Ba蛋白酶K) 200μl。的混合是在65°C的环境震动1000 r.p.m.热块中过夜,然后纯化如上所述。gydF4y2Ba
对于ATAC-seq分析,比对进行Bowtie2(魅惑gydF4y2Ba52gydF4y2Ba使用hg38基因组的管道gydF4y2Bahttps://github.com/shenlab-sinai/chip-seq_preprocessgydF4y2Ba。读取映射使用门店阴谋。ATAC-seq HEK293T细胞,基因具有高、中级,低,没有定义的表达式从HEK293T细胞RNA-seq DESeq2规范化basemean值数据和下2 basemean non-expressing基因和其余的基因被分成三组低,中间高表达。对A549gydF4y2Ba王牌gydF4y2Ba细胞ATAC-seq,三个生物复制每2 - 3技术进行复制。一千万从每个读取技术复制子集(SAMtools v1.9,种子1)和合并,然后每个条件都合并在生物复制。对于平均轮廓图,每个条件downsampled到4000万读和策划对所有基因被DESeq2 A549表示超过gydF4y2Ba王牌gydF4y2BaRNA-seq数据。gydF4y2Ba
ChIP-seqgydF4y2Ba
对于ORF8 ChIP-seq,转染后2 d,细胞被固定为5分钟1% PFA在PBS和反应是然后用2.5米甘氨酸淬火。细胞被洗两次,收集在PBS蛋白酶和磷酸酶抑制剂,然后颗粒状1200 r.p.m. 5分钟。细胞被旋转在裂解缓冲1(50毫米HEPES-KOH (pH值7.5),140毫米氯化钠,1毫米EDTA、10%甘油、0.5% NP-40和0.25% Triton x - 100) 10分钟在4°C和1350gydF4y2BaggydF4y2Ba5分钟在4°C分离细胞核。浮在表面的丢弃和细胞裂解缓冲2 resuspended(10毫米Tris-HCl pH值(8),200毫米氯化钠,EDTA 1毫米和0.5毫米EGTA)溶解核。样品在室温下旋转10分钟,在1350年再次旋转gydF4y2BaggydF4y2Ba5分钟在4°C。浮在表面的丢弃,颗粒在裂解缓冲resuspended 3(10毫米Tris-HCl pH值(8),100毫米氯化钠,EDTA 1毫米,0.5毫米EGTA, 0.1% EDTA和0.5%gydF4y2BaNgydF4y2Ba-lauroylsarcosine)。溶菌产物用在Covaris超声发生器40分钟(200次/破裂)。特里同x - 100添加到最终浓度为1%,和溶菌产物被旋转20000gydF4y2BaggydF4y2Ba10分钟在4°C。Strep-Tactin磁珠(MagStrep类型3 XT珠子;IBA,一夜之间,2-4090-002)被添加到溶解产物与旋转在4°C。珠子被用低盐缓冲(Triton x - 100年0.1% SDS, 1%, 2毫米EDTA, 20毫米三(8)pH值和150毫米氯化钠),高盐缓冲(Triton x - 100年0.1% SDS, 1%, 2毫米EDTA, 20毫米三(8)pH值和500毫米氯化钠),氯化锂洗缓冲区(150毫米氯化锂,NP-40 1%, 1%钠脱氧胆酸盐,1毫米EDTA和10毫米三(8)pH值),然后用50 mM TE氯化钠。染色质是筛选了珠子的30分钟在室温下用颤抖的55年µl BXT洗脱缓冲(IBA, 2-1042-025)紧随其后的150年µl洗脱缓冲(50毫米Tris-HCl (pH值8.0),10毫米EDTA和1% SDS) 30分钟在65°C。样本从珠子和交联被进一步扭转孵化染色质一夜之间在65°C。RNA与核糖核酸酶消化1 h在37°C,和蛋白质与蛋白酶K消化为30分钟55°C。DNA纯化PCR Zymo净化设备。Illumina公司TruSeq芯片净化设备被用来准备样品测序Illumina公司NextSeq 500乐器(42-bp读取长度,搭配)。gydF4y2Ba
为ORF8 ChIP-seq分析,进行了比对和Bowtie2(2.1.0的)gydF4y2Ba52gydF4y2Ba使用hg38基因组ChIP-seq管道(gydF4y2Bahttps://github.com/shenlab-sinai/chip-seq_preprocessgydF4y2Ba)。ORF8读取映射使用门店阴谋。组蛋白的化学修饰与ChIP-seq数据集、编码和4 d nucleome数据被用于H3K9ac(实验ENCSR000ASV),核纤层蛋白(4 dn组实验4 dnes24xa7u8), H3K9me3(实验ENCSR000FCJ和ENCSR179BUC) H3K9me2(实验ENCSR55LYM)和H3K27me3 ENCSR000AKD(实验)。定义ORF8-enriched地区,HiddenDomains用于每个两个ORF8 ChIP-seq实验规范化输入。输出文件与bedtools合并(v2.18.1)选择子集相交丰富地区发现的复制。DiffBind(3.4.11)是用于检查H3K27me3浓缩ORF8-enriched区域内。Deeptools (3.3.0) plotEnrichment工具被用来计算百分比读取的组蛋白修饰编码ChIP-seq ORF8-enriched区域内的数据集。ngs.plot。r(2.63)是用于生成块ORF8浓缩在感兴趣的基因组区域。gydF4y2Ba
组蛋白天车ChIP-seq,十百万细胞resuspended 1毫升的裂解缓冲1(50毫米HEPES-KOH (pH值7.5),140毫米氯化钠,1毫米EDTA、10%甘油、0.5% NP-40和0.25% Triton x - 100)在4°C和旋转10分钟,其次是离心分离和去除浮层。细胞被resuspended 1毫升的裂解缓冲2(10毫米Tris-HCl (pH值8.0),200毫米氯化钠,EDTA 1毫米和0.5毫米EGTA)和旋转10分钟在4°C,紧随其后的是上层清液的离心和删除。细胞被resuspended 1毫升的裂解缓冲3(10毫米Tris-HCl (pH值8.0),100毫米氯化钠,EDTA 1毫米,0.5毫米EGTA,脱氧胆酸盐钠0.1%和0.5%gydF4y2BaNgydF4y2Ba再次-lauroylsarcosine)和旋转10分钟在4°C。细胞被用与Covaris S220超声发生器35分钟(入射功率峰值,140;占空因数,5%;循环每破裂,200)。其次是增加110µl Triton x在最大速度(20000 - 100年和离心gydF4y2BaggydF4y2Ba在4°C) 15分钟溶解产物。根据DNA染色质溶解产物浓度是平衡的内容(衡量一个量子位荧光计)。这之后,等同于对染色质的5%gydF4y2BaCamponotus floridanusgydF4y2Ba蛹被添加到所有样本根据染色质的浓度,和50µl溶解产物的保存作为输入剪切控制。然后,250µl平衡的溶解产物添加洗,antibody-conjugated蛋白质A / G Dynabeads(2µg抗体结合到15µl蛋白质A / G Dynabeads resuspended 50µl /免疫沉淀反应),和免疫沉淀反应在一夜之间旋转在4°C 300µl的最后一卷。第二天,免疫沉淀反应在里帕洗五次洗缓冲区(50毫米HEPES-KOH (pH值7.5),500毫米氯化锂,1毫米EDTA,钠脱氧胆酸盐NP-40 1%和0.7%),一旦在TE (pH值8.0)。洗过后,两洗脱至75年µl洗脱缓冲(50毫米Tris-HCl (pH值8.0),10毫米EDTA和1% SDS)在65°C 45分钟用颤抖的(1100 r.p.m)。DNA纯化了苯酚:氯仿:异戊醇(25:24:1)提取乙醇沉淀紧随其后。颗粒状DNA在25µl resuspended TE。库测序是准备使用NEBNext超二世DNA库准备包Illumina公司(内,E7645)所描述的制造商,但使用所有试剂和原料数量的一半。PCR扩增的PCR循环的最佳数量决定使用qPCR副反应以10%的adaptor-ligated, size-selected DNA。七到十周期的PCR用于组蛋白天车库和5周期被用于输入控件。样本测序NextSeq 500仪器(42-bp读取长度,搭配)。gydF4y2Ba
分析组蛋白天车ChIP-seq数据,读取使用bcl2fastq2去复用(Illumina公司)选项”——mask-short-adapter-reads 20 minimum-trimmed-read-length 20——no-lane-splitting barcode-mismatches 0 '。使用TRIMMOMATIC读取被剪掉gydF4y2Ba53gydF4y2Ba选项的ILLUMINACLIP:(适配器。f一个]:2:30:10 LEADING:5 TRAILING:5 SLIDINGWINDOW:4:15 MINLEN:15’ and aligned to a hybrid hg38 + C。gydF4y2BafloridanusgydF4y2Ba(v7.5 RefSeq)基因组组装使用bowtie2 (v2.2.6)gydF4y2Ba52gydF4y2Ba选择“——sensitive-local”。比对映射质量低于5(使用SAMtools)和重复读取被移除。山峰被称为使用MACS2 (v2.1.1.20160309)gydF4y2Ba54gydF4y2Ba的选项——call-summits——nomodel B。微分芯片使用DiffBind山峰被称为gydF4y2Ba55gydF4y2Ba选项的bFullLibrarySize = FALSE, bSubControl = TRUE, bTagwise = FALSE”为dba.analyze ()。对于DiffBind测试,与使用阻塞DESeq2算法,芯片复制的阻碍因素,而测试用于模拟和感染样本之间的差异。芯片信号跟踪、个人复制跟踪生产的RPM和褶皱浓缩在输入控件,合并和平均。gydF4y2Ba
占潜在的全球不同的组蛋白天车丰富,否则错过了更标准的分位数normalization-type方法,高质量的删除处理人类和阅读数量生产gydF4y2BaC。gydF4y2BafloridanusgydF4y2Ba映射读取,读取的比例映射到每个组蛋白的外源基因天车。输入控件也以这种方式对待潜在的差异占初始激增之间除了样本。每个组蛋白天车的比例激增读取被适当的输入控件的值归一化。因为spike-ins应该目标染色质的浓度成反比,SARS-CoV-2 /模拟值之比为每个组蛋白天车×复制生产,和SARS-CoV-2样品产生的信号值除以这个比例。这导致每个碱基对信号值调整全球不同的程度在一个给定的样本类型之间的组蛋白天车的水平。gydF4y2Ba
所有抗体都描述的补充表gydF4y2Ba6gydF4y2Ba。gydF4y2Ba
RNA-seqgydF4y2Ba
使用试剂盒RNA RNA提取净化设备。样本准备使用Illumina公司测序TruSeq净化装备和测序的Illumina公司NextSeq 500仪器(75 - bp读取长度、单读)。库大小确认生物分析仪在测序550年NextSeq平台上(单头,75周期)。gydF4y2Ba
RNA-seq分析SARS-CoV-2感染实验,参考基因组比对是由连接人类(GRCh38大会)和SARS-CoV-2 (WA-CDC-WA1/2020组装;gydF4y2BaMN985325.1gydF4y2Ba)的基因组。RNA-seq分析HEK293T细胞实验中,使用GRCh38大会。RNA-seq,读取使用明星(v2.6.1a)使用默认参数一致,只有独特的映射读取保留了下游分析。使用IGVtools TDF文件生成的。读取被数对人类基因(GENCODE v35)和SARS-CoV-2基因(WA-CDC-WA1/2020组装;gydF4y2BaMN985325.1gydF4y2Ba)使用featureCounts (v1.6.2)。低、基因被过滤掉,因此只有基因计数每百万(CPM)值大于1至少在三个样本使用。数据规范化和差异基因表达分析进行使用DESeq2 R包(v1.26.0)。我们定义基因重要使用错误发现率(罗斯福)截止0.05和1.5×褶皱变化。去富集分析差异表达基因实施clusterProfiler R包(v3.14.3),使用人类基因组注释org.Hs.eg记录。db R包(v3.10.0)和Benjamini-Hochberg-adjustedgydF4y2BaPgydF4y2Ba截止值为0.05。gydF4y2Ba
免疫沉淀反应gydF4y2Ba
Anti-Strep标记亲和纯化、全细胞溶解产物和细胞质hla a2 co-immunoprecipitationgydF4y2Ba
蛋白质和约束力的合作伙伴与亲和纯化喉炎标记净化。对于ORF8天车分析和质谱分析绑定合作伙伴分析、全细胞溶解产物准备如下所述。简要解冻冷冻细胞颗粒和悬浮在裂解缓冲(免疫沉淀反应(IP)缓冲区(50毫米Tris-HCl (pH值7.5)在4°C, 150毫米氯化钠,1毫米EDTA和10毫米丁酸钠)补充0.5%诺乃清洁剂P 40替代(NP-40;丙烯酰胺分析)和完整的迷你EDTA-free蛋白酶和PhosSTOP磷酸酶抑制剂鸡尾酒(罗氏))。样本孵化在管旋转30分钟在4°C。碎片被离心13000颗粒状gydF4y2BaggydF4y2Ba15分钟在4°C。溶菌产物被孵化与Strep-Tactin磁珠(40µl;MagStrep类型3 XT珠子;IBA, 2-4090-002)与旋转在4°C 2 h。珠子被洗了三次1毫升洗缓冲缓冲补充0.05% NP-40 (IP),然后用1毫升IP缓冲区。Strep-tagged ORF8复合物被筛选了珠子的BXT缓冲区(IBA, 2-1042-025)与1100 r.p.m颤抖。30分钟。gydF4y2Ba
为染色质绑定合作伙伴Anti-Strep标记亲和纯化gydF4y2Ba
细胞裂解缓冲1中旋转(50毫米HEPES-KOH (pH值7.5),140毫米氯化钠,丁酸钠10毫米,1毫米EDTA、10%甘油、0.5% NP-40和0.25% Triton x - 100)补充0.5%诺乃清洁剂P 40替代(NP-40;丙烯酰胺分析)和完整的迷你EDTA-free蛋白酶和PhosSTOP磷酸酶抑制剂鸡尾酒(罗氏)10分钟4°C和1350gydF4y2BaggydF4y2Ba5分钟在4°C分离细胞核。浮在表面的丢弃和细胞裂解缓冲2 resuspended(10毫米Tris-HCl pH值(8),200毫米氯化钠,丁酸钠10毫米,1毫米EDTA和0.5毫米EGTA)溶解核。细胞被旋转10分钟在室温和在1350年再次旋转gydF4y2BaggydF4y2Ba5分钟在4°C。上层的丢弃,染色质颗粒在裂解缓冲resuspended 3(10毫米Tris-HCl pH值(8),100毫米氯化钠,丁酸钠10毫米,EDTA 1毫米,0.5毫米EGTA, 0.1% EDTA和0.5%gydF4y2BaNgydF4y2Ba-lauroylsarcosine)。溶菌产物用使用小费超声发生器和三个5 s在50%电量与寒冷的冰之间爆发。声波降解法后,溶解产物被带到特里同x - 100浓度1%破坏叶片蛋白质的相互作用。碎片被离心16000颗粒状gydF4y2BaggydF4y2Ba在4°C,和上层的孵化与Strep-Tactin磁珠(40µl;MagStrep类型3 XT珠子;Iba, 2-4090-002)与旋转在4°C 2 h。珠子被洗了三次1毫升洗缓冲缓冲补充0.05% NP-40 (IP),然后用1毫升IP缓冲区。Strep-tagged ORF8复合物被筛选了珠子的BXT缓冲区(IBA, 2-1042-025)与1100 r.p.m颤抖。30分钟。分析相对ORF8细胞质与染色质结构水平分数通过免疫印迹,样本取自裂解缓冲1和裂解缓冲3,分别。gydF4y2Ba
反向免疫沉淀反应gydF4y2Ba
染色质颗粒溶解产物得到如上所述的染色质免疫沉淀反应的蛋白质。溶菌产物是结合antibody-conjugated蛋白质Dynabeads(15µg抗体结合100µl Dynabeads)和旋转一夜之间在4°C。第二天,珠被洗了三次1毫升清洗缓冲缓冲补充0.05% NP-40 (IP),然后用1毫升IP缓冲区。染色质蛋白复合物被筛选了珠子的洗脱缓冲(50毫米Tris-HCl (pH值8.0),10毫米EDTA和1% SDS) 30分钟用颤抖的在65°C。gydF4y2Ba
所有抗体都描述的补充表gydF4y2Ba6gydF4y2Ba。gydF4y2Ba
免疫细胞化学gydF4y2Ba
HEK293T细胞A549的荧光免疫细胞化学gydF4y2BaACE2gydF4y2Ba细胞gydF4y2Ba
细胞被固定在4% PFA 10分钟,洗PBS。固定细胞permeabilized在PBS使用0.5% Triton x - 100 20分钟。细胞被封锁在屏蔽解决方案(PBS 3% BSA, 2%血清和0.1% Triton x - 100)至少1 h和沾指定主要抗体在一夜之间在4°C。第二天,电池盖玻片和PBS和孵化洗二次抗体在室温下1 h。检测Strep-tagged ORF8, Strep-Tactin dy - 488 (IBA, 2-1562-050;1:50 0)添加到二次抗体的解决方案。核与DAPI染色(PBS 1:1,000) 10分钟洗在PBS。盖玻片被安装到显微镜幻灯片使用延长黄金不变色试剂(热费希尔)。gydF4y2Ba
荧光免疫细胞化学分析的核纤层蛋白B1、核纤层蛋白A / C和H3K9me2gydF4y2Ba
HEK293T细胞被固定为2% PFA(电子显微镜科学,15710)在室温下8分钟,洗了三次DPBS (Gibco, 14190 - 136)。细胞permeabilized 0.25% Triton x - 100(热费希尔,28314)10分钟,洗了三次DPBS每个5分钟洗和阻止1% BSA(σ,A4503) PBST (DPBS Tween-20 0.05%, pH值7.4(热费舍尔,28320))60分钟。细胞被孵化主要抗体稀释在阻断缓冲区1 h,洗了三次对PBST 5分钟每个洗和孵化与二次抗体稀释阻挡缓冲60分钟。细胞被洗两次PBST和与PBS为每个洗5分钟,然后被安装在一个幻灯片使用Duolink原位安装介质与DAPI(σ,DUO82040-5ML)。所有程序都是在室温下进行。gydF4y2Ba
Immunohistological染色的病人的肺部组织gydF4y2Ba
Formalin-fixed,石蜡包埋幻灯片取自佩恩的病理临床服务中心。幻灯片deparaffinized和水化如下:孵化与二甲苯(两次)10分钟,10分钟以100%乙醇(两次),5分钟以95%乙醇、70%乙醇5分钟,5分钟,50%乙醇,然后运行蒸馏水。幻灯片被加工使用燥热引起抗原决定基检索(在这里)。幻灯片是在炎热的柠檬酸钠缓冲孵化(10毫米Tween-20柠檬酸钠和0.05%,pH值6.0),放置在一个压力锅和加热水浴用高压设置了25分钟。幻灯片是在室温下冷却和在TBS洗两次。膜在TBS permeabilized 0.4% Triton x - 100 20分钟。幻灯片被孵化在屏蔽解决方案(TBS山羊血清,10% 1% BSA和0.025% Triton x - 100) 2 h。幻灯片在鼠标孵化主要抗体溶液含有anti-SARS-CoV-2核衣壳和兔子anti-H3K9me3抗体溶液在一夜之间在4°C。第二天,幻灯片在二级抗体TBS清洗和孵化的解决方案。核与DAPI染色(5µg毫升gydF4y2Ba1gydF4y2Ba在TBS) 10分钟洗和TBS紧随其后。盖玻片是安装延长黄金不变色试剂(热费希尔)。所有抗体都描述的补充表gydF4y2Ba6gydF4y2Ba。gydF4y2Ba
图像采集gydF4y2Ba
荧光免疫细胞化学ORF8和组蛋白天车gydF4y2Ba
细胞成像在6000年一个正直的徕卡DM, TCS SP8激光扫描共焦显微镜与405 nm、488 nm、552 nm和638 nm激光。显微镜使用两个海德拉巴探测器和三个PMT探测器。使用的目的是一个×63 HC PL APO CS2石油客观的NA 1.40。F型浸液(莱卡)是用于石油的目标。图片是175.91×171.91µmgydF4y2Ba2gydF4y2Ba1024×1024像素,每个像素16位。天车量化,HEK293T细胞和人类肺组织在一个z平面成像和A549细胞成像gydF4y2BazgydF4y2Ba通过细胞核堆栈。gydF4y2Ba
荧光免疫细胞化学分析的核纤层蛋白B1、核纤层蛋白A / C和H3K9me2gydF4y2Ba
共聚焦免疫荧光图像都是获得使用徕卡SP8激光扫描共焦系统×63/1.40-NA HC PL APO CS2客观和海德拉巴探测器在标准模式下100%的涨幅。核纤层蛋白A / C和核纤层蛋白B1的对比模拟和ORF8-positive细胞之间的信号强度,单排共焦图像。所有图片都获得相同的显微镜设置(放大、激光功率、增益等)。分析H3K9me2-marked染色质的组织的核板,中间的三维(3 d)图像gydF4y2BazgydF4y2Ba作为飞机的核gydF4y2BazgydF4y2Ba栈使用0.1 -μm间隔1μm /核。共焦三维图像与惠更斯deconvoluted专业软件使用显微镜参数,标准的背景估计PSF和自动设置。gydF4y2Ba
图像分析gydF4y2Ba
图像进行分析使用ImageJ软件(版本2.0.0-rc-69/1.52p,构建269 a0ad53f)。单gydF4y2Ba- zgydF4y2Ba平面的图像HEK293T细胞和人类的肺组织和总结gydF4y2BazgydF4y2Ba通过A549核被用于多功能天车量化栈。聚焦的图像感兴趣区域(roi)核半自动生成使用DAPI通道定义,分析粒子的功能与手动修正。HEK293T组蛋白天车在转染细胞和non-transfected邻近细胞使用量化的平均灰度值。信号Strep-tagged ORF8构造(喉炎的症状- tactin - 488)和GFP是用来定义转染细胞,和HEK293T组蛋白转染细胞被相对化铝水平的组蛋白天车水平non-transfected邻近细胞。组蛋白天车量化在A549细胞和人类肺组织使用集成密度值。dsRNA, SARS-CoV-2核衣壳信号用于定义感染A549细胞和人类肺细胞,分别。的总荧光强度核纤层蛋白A / C和核纤层蛋白B1信号从模拟的整个核测量和ORF8-positive细胞。分析外围异染色质组织进行比较的一小部分H3K9me2-marked染色质核板/外围的模拟和ORF8-positive细胞。一小部分H3K9me2信号在核板/外围测量使用核纤层蛋白B信号掩码或DAPI信号来创建一个0.6的面具-μm-thick核外围区。gydF4y2Ba
蛋白质对齐gydF4y2Ba
识别潜在的组蛋白拟态,SARS-CoV-2蛋白质序列对齐的人类蛋白质组蛋白序列(H2A、H2B H3.1, H3.2, H4, H2A。X, H2A。Z, macroH2A和H3.3)使用多个序列比较Log-Expectation(肌肉),默认设置。SARS-CoV-2蛋白质序列从蛋白质序列获得出版第一武汉隔离gydF4y2Ba56gydF4y2Ba。gydF4y2Ba
流式细胞仪gydF4y2Ba
HEK293T细胞颗粒轻轻resuspended 1毫升流式细胞仪缓冲区(CagydF4y2Ba2 +gydF4y2Ba/毫克gydF4y2Ba2 +gydF4y2Ba无PBS BSA)和颗粒状在500年为2%gydF4y2BaggydF4y2Ba5分钟在4°C;上层清液被移除。细胞转染与ORF8构造和non-transfected控制细胞在1毫升流式细胞仪轻轻resuspended缓冲区的1:50 0稀释Strep-Tactin dy - 488和旋转在4°C 1 h,免受光。细胞被洗两次1毫升流式细胞仪缓冲区,resuspended 1毫升流式细胞仪缓冲区和过滤35-µm网在流式细胞仪管。BD涌入细胞分类器被用来分析细胞。Strep-Tactin dy - 488和GFP是兴奋和488 nm激光信号收集530/40-nm探测器。不包括紧身衣和细胞碎片,细胞被封闭在Strep-Tactin dy - 488信号,在阈值设置使用non-transfected控制细胞等,< 1%的控制细胞被认为是阳性Strep-Tactin dy - 488。Strep-Tactin dy - 488阳性细胞在流式细胞仪收集缓冲和颗粒状为后续实验。流式细胞仪控制策略和细胞数量孤立补充图所示。gydF4y2Ba2gydF4y2Ba。gydF4y2Ba
组蛋白提取gydF4y2Ba
转染细胞被孤立的流式细胞仪如上所述。排序细胞颗粒状,resuspended 1毫升冷HgydF4y2Ba2gydF4y2Ba所以gydF4y2Ba4 gydF4y2Ba一夜之间,旋转在4°C。隔夜孵化后,细胞以最大速度和颗粒状浮在表面的被转移到一个新的管。三氯乙酸加入25%体积,这些细胞被留在冰在一夜之间在4°C。细胞被再次以最大速度,颗粒状和上层的丢弃。Prechilled丙酮被用来轻轻洗球两次。第二次洗后,管了空气干燥颗粒resuspended之前在水里。样本然后分解由声波降解法交替10分钟和30分钟50°C的摇晃直到丸完全溶解。gydF4y2Ba
质谱分析gydF4y2Ba
组蛋白天车通过定量质谱分析gydF4y2Ba
净化验证了组蛋白sds - page紧随其后Coomassie染色证明足够的浓缩。BCA分析(热费希尔)进行蛋白质估计使用制造商的指示,和20µg组蛋白用于化学衍生化和消化如前所述gydF4y2Ba57gydF4y2Ba。在短暂的,修改的赖氨酸derivatized两次1:3比例的乙腈proprionic酸酐。组蛋白在1:20然后用胰蛋白酶消化酵素蛋白质比一夜之间在37°C。与新成立的N末端消化组蛋白derivatized完成先前的两倍。最后,组蛋白与真空浓缩器干。干样本重组在0.1%三氟乙酸(组织)和脱盐C18微旋转列(哈佛装置)。列准备200μl 100%乙腈和平衡200μl加载缓冲组织为0.1%。肽是装上的列,加载缓冲清洗和筛选了200μl甲酸乙腈70% 0.1%。所有步骤加载、洗涤和洗脱台式离心(300gydF4y2BaggydF4y2Ba2分钟)。筛选了肽被干真空离心浓缩器。gydF4y2Ba
干组蛋白肽在0.1%甲酸还原。合成库93年沉重的标签和derivatized肽通常包含测量组蛋白天车gydF4y2Ba58gydF4y2Ba上升到内生样本的最终浓度大约100 ngµl吗gydF4y2Ba1gydF4y2Ba内源性肽和100年fmolµlgydF4y2Ba1gydF4y2Ba对于每个重标记合成分析物。为每一个分析,1µl样本注入到列data-independent Q-Exactive高频仪器分析(热科学)连接到一个最终3000 nano-UPLC系统和Nanospray Flex离子源(热科学)。使用水溶液0.1%甲酸作为缓冲和有机溶液的80%乙腈和0.1%甲酸作为缓冲B,肽在63分钟的梯度分离400 nl分钟gydF4y2Ba1gydF4y2Ba从4%开始缓冲缓冲B / B和增加32% 58分钟然后缓冲B / 5分钟增加到98%。列然后洗98%缓冲B / 5分钟和平衡缓冲3% B Data-independent收购进行以下设置。一个完整的女士gydF4y2Ba1克ydF4y2Ba扫描从300年到950年gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba60000年收购了一项决议,自动增益控制(AGC) 3×10的目标gydF4y2Ba6gydF4y2Ba的最大注射时间55 ms。然后,一系列的25 MSgydF4y2Ba2gydF4y2Ba与顺序扫描了在相同的质量范围隔离的窗户24gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba碰撞能量的28岁,30000年的一项决议,1×10的AGC的目标gydF4y2Ba6gydF4y2Ba的最大注射时间55 ms。数据分析和手动检查使用合成库作为参考进行与天际线(MacCoss实验室)。比率是使用R工作室和生成统计分析在Excel在较早的组蛋白进行分析。gydF4y2Ba
胰蛋白酶和胰凝乳蛋白酶消化ORF8 ORF8修改的识别gydF4y2Ba
凝胶乐队包含ORF8使退色了50 mM碳酸氢铵50%乙腈。乐队当时减少10毫米德勤在50 mM碳酸氢铵为30分钟55°C。接下来,乐队在50毫米和100毫米碘乙酰胺烷基化碳酸氢铵在黑暗中在室温下30分钟。蛋白质与胰凝乳蛋白酶和胰蛋白酶消化孵化一个约1:20酶蛋白质比一夜之间在37°C。消化后,上层的收集。从凝胶中提取更多的多肽,150μl增加了50%的乙腈和1%的组织和样本孵化不断摇晃了30分钟。乙腈的上层清液收集和100μl添加孵化常摇动10分钟紧随其后。最后的上层清液收集。所有三个浮在表面的结合和干。然后干样本脱盐如上所述。gydF4y2Ba
ORF8与控制免疫沉淀反应的识别约束力的合作伙伴gydF4y2Ba
ORF8免疫沉淀反应elutants减少和烷基化如上所述。蛋白质被消化和脱盐迷你S-Trap (Protifi)后,制造商的指示。总之,25μl elutant加上25μl 10% SDS烷基化后最后一个SDS浓度的5%。样本然后用磷酸和酸化沉淀通过添加90%的甲醇100毫米triethylammonium碳酸氢盐(TEAB) 6:1(卷/期)的比例。蛋白质被添加到陷阱台式离心(4000gydF4y2BaggydF4y2Ba1分钟),洗在1:10酶和胰蛋白酶消化蛋白质比一夜之间在37°C。消化后,抗菌肽筛选了100毫米的陷阱与40个μl TEAB, 40μl 0.2%的甲酸和40μl 50%乙腈0.2%甲酸。结合elutant卷被干。gydF4y2Ba
胰凝乳蛋白酶质/ MS和LC-PRM-MS分析gydF4y2Ba
干缩氨酸与甲酸0.1%,重组和2µg每个样本的注入。在Q-Exactive Chymotrypsin-digested ORF8样本分析(热科学)耦合到一个简单的缴送工作1000 UHPLC系统和Nanospray Flex离子源(热科学)。LC仪器配有75µm×20厘米列装在房子使用Reprosil-Pur C18 AQ(2.4µm;Maisch博士)。使用相同的列和缓冲条件如前所述,肽分离的85 - 400 nl分钟最小梯度gydF4y2Ba1gydF4y2Ba从缓冲缓冲B / B和增加32% 3% 79分钟,然后增加到50%缓冲B / 5分钟最后缓冲B / 1分钟增加到90%。列然后洗缓冲90% B / 5分钟和平衡缓冲3% B .视数据采集与动态排除40多岁了。一个完整的女士gydF4y2Ba1克ydF4y2Ba扫描从350年到1200年gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba分辨率为70000,收购1×10的AGC的目标吗gydF4y2Ba6gydF4y2Ba和最大注入50毫秒的时间。然后,一系列的女士gydF4y2Ba2gydF4y2Ba扫描获得了前15名前体的电荷状态2 - 7日,28岁,一个隔离窗的碰撞能量2.0gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba。每一个女士gydF4y2Ba2gydF4y2Ba扫描分辨率17500收购,AGC 2×10的目标gydF4y2Ba5gydF4y2Ba和最大注入50毫秒的时间。进行数据库搜索使用人类SwissProt序列和ORF8序列与蛋白质组发现者2.3或2.4(热科学)使用以下搜索条件:carboxyamidomethylation半胱氨酸残基作为一个固定的修改;氧化在蛋氨酸和赖氨酸乙酰化变量的修改;两个最大允许错过分裂;前体女士10 ppm的宽容;0.02 - da MS / MS。计划外的平行反应监测方法gydF4y2Ba59gydF4y2Ba开发识别或验证45可能的修改和未修改的肽ORF8的目标。肽分离LC梯度相同的条件。一个完整的女士gydF4y2Ba1克ydF4y2Ba扫描从300年到900年gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba分辨率为70000,收购1×10的AGC的目标吗gydF4y2Ba6gydF4y2Ba和最大注入50毫秒的时间。然后,一系列的女士gydF4y2Ba2gydF4y2Ba扫描循环计数的收购23前兆,28岁,一个隔离窗的碰撞能量1.2gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba。每一个女士gydF4y2Ba2gydF4y2Ba扫描分辨率17500收购,AGC 1×10的目标gydF4y2Ba6gydF4y2Ba和最大注入100毫秒的时间。数据分析和人工检查进行轮廓gydF4y2Ba60gydF4y2Ba(MacCoss实验室)和”gydF4y2Ba61年gydF4y2Ba。gydF4y2Ba
胰蛋白酶ORF8质/女士和LC-PRM / MS分析和IP质/ MS分析gydF4y2Ba
干缩氨酸与甲酸0.1%,重组和2µg每个样本的注入。视数据采集运行分析Q-Exactive高频或HF-X(热科学)连接到一个最终3000纳米UPLC系统和Nanospray Flex离子源(热科学)。使用相同的列和缓冲条件如上所述,肽在112分钟的梯度分离400 nl分钟gydF4y2Ba1gydF4y2Ba缓冲B从5%,提高到35%缓冲B / 104分钟然后缓冲B / 8分钟增加到60%。在95%的缓冲B列然后洗5分钟和平衡缓冲5% B .视数据采集与动态执行排除45 s。一个完整的女士gydF4y2Ba1克ydF4y2Ba扫描从380年到1200年gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba收购分辨率为120000,3×10的AGC的目标吗gydF4y2Ba6gydF4y2Ba的最大注射时间32女士。然后,一系列的女士gydF4y2Ba2gydF4y2Ba扫描获得了前20名前体的电荷状态2 - 5,28岁,一个隔离窗的碰撞能量1.2gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba。每一个女士gydF4y2Ba2gydF4y2Ba扫描分辨率30000收购,AGC 1×10的目标gydF4y2Ba6gydF4y2Ba女士的最大注射时间32(高频)或55 ms (HFX)。进行数据库搜索使用人类SwissProt序列和ORF8序列与蛋白质组发现者2.3或2.4(热科学)以下搜索条件:carboxyamidomethylation半胱氨酸残基作为一个固定的修改;氧化在蛋氨酸和赖氨酸乙酰化变量的修改;两个最大允许错过分裂;前体女士gydF4y2Ba1克ydF4y2Ba10 ppm的宽容;0.02 - da女士gydF4y2Ba2gydF4y2Ba宽容。计划外的平行反应监测方法gydF4y2Ba59gydF4y2Ba是识别开发的16个可能的修改和未修改的肽ORF8的目标。肽分离LC梯度相同的条件。一个完整的女士gydF4y2Ba1克ydF4y2Ba扫描从350年到950年gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba收购分辨率为120000,3×10的AGC的目标吗gydF4y2Ba6gydF4y2Ba和最大注入100毫秒的时间。然后,一系列的女士gydF4y2Ba2gydF4y2Ba扫描获得16个循环计数的前兆,28岁,一个隔离窗的碰撞能量1.2gydF4y2Ba米gydF4y2Ba/gydF4y2BazgydF4y2Ba。每一个女士gydF4y2Ba2gydF4y2Ba扫描分辨率30000收购,AGC 1×10的目标gydF4y2Ba6gydF4y2Ba和最大注入100毫秒的时间。数据分析和人工检查进行轮廓gydF4y2Ba60gydF4y2Ba(MacCoss实验室)和”gydF4y2Ba61年gydF4y2Ba。gydF4y2Ba
统计和再现性gydF4y2Ba
Box-and-whisker情节显示中值为中线,盒子上、下四分位数限制,胡须1.5×四分位范围和异常值点。方差分析测试执行和情节生成与r . Bonferroni调整申请多个比较。斐济是用于图像分析。成像和分析与实验者进行盲目的实验条件。在某些情况下,例如病人组织成像,分析需要有针对性的选择、成像和分析受感染的细胞与未感染的细胞。这需要实验人员需要注意的细胞感染状态而成像。然而,在这些情况下,感兴趣的测量(如组蛋白改性染色)并不认为在选择字段来避免选择的偏差。gydF4y2Ba
图片代表多个复制如下:gydF4y2Ba
图gydF4y2Ba1 bgydF4y2Ba:> 5个独立的实验。gydF4y2Ba
图gydF4y2Ba1 cgydF4y2Ba:两个独立的实验。gydF4y2Ba
图gydF4y2Ba1 dgydF4y2Ba:三个独立的实验。gydF4y2Ba
图gydF4y2Ba1克gydF4y2Ba:五个独立样本两个独立运行的流式细胞仪分选。gydF4y2Ba
图gydF4y2Ba2 b, d, fgydF4y2Ba图中描述的:准确的细胞数量和复制。gydF4y2Ba2 c、d、ggydF4y2Ba。gydF4y2Ba
图gydF4y2Ba2 hgydF4y2Ba:两个显示四个独立样本的流式细胞仪。gydF4y2Ba
图gydF4y2Ba3 fgydF4y2Ba:每三个独立样本条件从一个感染。gydF4y2Ba
图gydF4y2Ba3 ggydF4y2Ba图中描述的:准确的细胞数量和复制。gydF4y2Ba3 hgydF4y2Ba。gydF4y2Ba
扩展的数据图。gydF4y2Ba2gydF4y2Ba:三个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba2 b, cgydF4y2Ba:> 5个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba二维gydF4y2Ba:两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba2 egydF4y2Ba:两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba3 bgydF4y2Ba:两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba4gydF4y2Ba:核纤层蛋白和组蛋白H3,三个独立的实验;HP1αKAT2A,两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba4 bgydF4y2Ba:两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba4摄氏度gydF4y2Ba:两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba4 dgydF4y2Ba:一个独立的实验,重复先前公布的数据。gydF4y2Ba
扩展的数据图。gydF4y2Ba4 egydF4y2Ba:两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba4 fgydF4y2Ba:两个独立的实验。gydF4y2Ba
扩展的数据图。gydF4y2Ba5gydF4y2Ba:准确的细胞数量和复制扩展数据图中描述。gydF4y2Ba5 bgydF4y2Ba。gydF4y2Ba
扩展的数据图。gydF4y2Ba5度gydF4y2Ba:准确的细胞数量和复制扩展数据图中描述。gydF4y2Ba5 dgydF4y2Ba。gydF4y2Ba
扩展的数据图。gydF4y2Ba5 egydF4y2Ba:扩展数据图的图像一样。gydF4y2Ba5度gydF4y2Ba。gydF4y2Ba
扩展的数据图。gydF4y2Ba6 bgydF4y2Ba:三个显示五个独立样本的流式细胞仪分选。gydF4y2Ba
扩展的数据图。gydF4y2Ba10gydF4y2Ba:准确的细胞数量和复制中所描述的扩展D ata无花果。gydF4y2Ba10 bgydF4y2Ba。gydF4y2Ba
扩展的数据图。gydF4y2Ba10 cgydF4y2Ba:准确的细胞数量和复制扩展数据图中描述。gydF4y2Ba10 dgydF4y2Ba。gydF4y2Ba
扩展的数据图。gydF4y2Ba10 egydF4y2Ba:准确的细胞数量和复制扩展数据图中描述。gydF4y2Ba10 fgydF4y2Ba。gydF4y2Ba
报告总结gydF4y2Ba
进一步研究信息设计是可用的gydF4y2Ba自然研究报告摘要gydF4y2Ba与这篇文章有关。gydF4y2Ba
数据可用性gydF4y2Ba
所有全基因组测序数据加入号码是可用的gydF4y2BaGSE186628gydF4y2Ba。质谱的蛋白质组学数据被存入ProteomeXchange财团通过骄傲合作伙伴库gydF4y2Ba62年gydF4y2Ba数据集的标识符gydF4y2BaPXD034379gydF4y2Ba。公开的数据用于分析包括编码数据ENCSR000ASV、ENCSR000FCJ ENCSR179BUC ENCSR55LYM ENCSR000AKD和4 d 4 dnes24xa7u8 nucleome数据集。gydF4y2Ba
改变历史gydF4y2Ba
2023年2月06gydF4y2Ba
这篇论文已经发表的校正:gydF4y2Bahttps://doi.org/10.1038/s41586 - 023 - 05764 - 8gydF4y2Ba
2023年1月09gydF4y2Ba
这篇论文已经发表的校正:gydF4y2Bahttps://doi.org/10.1038/s41586 - 022 - 05631 - ygydF4y2Ba
引用gydF4y2Ba
Hadjadj, j . et al . I型干扰素活动和受损严重COVID-19患者的炎症反应。gydF4y2Ba科学gydF4y2Ba369年gydF4y2Ba,718 - 724 (2020)。gydF4y2Ba
Blanco-Melo, d . et al .主机不平衡响应SARS-CoV-2 COVID-19驱动发展。gydF4y2Ba细胞gydF4y2Ba181年gydF4y2Ba,1036 - 1045 (2020)。gydF4y2Ba
李,y . et al . SARS-CoV-2诱发双链rna介导呼吸epithelial-derived细胞和心肌细胞的先天免疫反应。gydF4y2BaProc。《科学。美国gydF4y2Ba118年gydF4y2Bae2022643118 (2021)。gydF4y2Ba
Marazzi, et al .抑制流感的抗病毒反应组蛋白模拟。gydF4y2Ba自然gydF4y2Ba483年gydF4y2Ba,428 - 433 (2012)。gydF4y2Ba
Schaefer U。,Ho, J. S. Y., Prinjha, R. K. & Tarakhovsky, A. The ‘histone mimicry’ by pathogens.冷泉哈布。计算机协会。定量,杂志。gydF4y2Ba78年gydF4y2Ba,81 - 90 (2013)。gydF4y2Ba
Avgousti特区等。核心病毒蛋白结合主机核小体隔离免疫危险信号。gydF4y2Ba自然gydF4y2Ba535年gydF4y2Ba,173 - 177 (2016)。gydF4y2Ba
Avgousti特区et al .腺病毒蛋白质7核心会使宿主基因组DNA损伤反应。gydF4y2Baj .性研究。gydF4y2Ba91年gydF4y2Bae01089-17 (2017)。gydF4y2Ba
Tarakhovsky, a & Prinjha r·k·利用障碍:病毒如何使用组蛋白模仿他们的优势。gydF4y2Baj . Exp。地中海。gydF4y2Ba215年gydF4y2Ba,1777 - 1787 (2018)。gydF4y2Ba
Strahl b d & alli, c, d .共价组蛋白修饰的语言。gydF4y2Ba自然gydF4y2Ba403年gydF4y2Ba41 - 45,(2000)。gydF4y2Ba
Jenuwein t & a, c, d .翻译组的代码。gydF4y2Ba科学gydF4y2Ba293年gydF4y2Ba,1074 - 1080 (2001)。gydF4y2Ba
伯杰,s . l .染色质的复杂语言在转录调控。gydF4y2Ba自然gydF4y2Ba447年gydF4y2Ba,407 - 412 (2007)。gydF4y2Ba
Ho j . s . y . et al . TOP1抑制疗法预防SARS-CoV-2-induced致命的炎症。gydF4y2Ba细胞gydF4y2Ba184年gydF4y2Ba,2618 - 2632 (2021)。gydF4y2Ba
李,s . et al。病毒诱导衰老是一个司机,在COVID-19治疗目标。gydF4y2Ba自然gydF4y2Ba599年gydF4y2Ba,283 - 289 (2021)。gydF4y2Ba
Zazhytska, m . et al . Non-cell-autonomous破坏核COVID-19-induced嗅觉缺失的架构作为一个潜在的原因。gydF4y2Ba细胞gydF4y2Ba185年gydF4y2Ba,1052 - 1064 (2022)。gydF4y2Ba
Elde: c & Malik h . s .病原体的进化难题拟态。gydF4y2Ba启Microbiol Nat。gydF4y2Ba7gydF4y2Ba,787 - 797 (2009)。gydF4y2Ba
戴维:E。,Travé, G. & Gibson, T. J. How viruses hijack cell regulation.学生物化学的发展趋势。科学。gydF4y2Ba36gydF4y2Ba,159 - 169 (2011)。gydF4y2Ba
Chaurushiya, m . s . et al .病毒性E3泛素ligase-mediated退化细胞E3:病毒的模仿细胞磷酸化RNF8 FHA领域目标。gydF4y2Ba摩尔。细胞gydF4y2Ba46gydF4y2Ba,79 - 90 (2012)。gydF4y2Ba
陈,j . F.-W。et al . 2019年的小说《人类病原冠状病毒的基因组特征与非典型性肺炎患者在访问武汉。gydF4y2Ba紧急情况。微生物感染。gydF4y2Ba9gydF4y2Ba,221 - 236 (2020)。gydF4y2Ba
陆,r . et al . 2019年的小说《冠状病毒基因组描述和流行病学:影响病毒的起源和受体结合。gydF4y2Ba《柳叶刀》gydF4y2Ba395年gydF4y2Ba,565 - 574 (2020)。gydF4y2Ba
袁,K.-S。,你们,z。,Fung, S.-Y., Chan, C.-P. & Jin, D.-Y. SARS-CoV-2 and COVID-19: the most important research questions.细胞Biosci。gydF4y2Ba10gydF4y2Ba40 (2020)。gydF4y2Ba
位于,s . c . et al .组蛋白的甲基化模拟G9a的组蛋白甲基转移酶调节蛋白质复杂的装配。gydF4y2Ba摩尔。细胞gydF4y2Ba27gydF4y2Ba,596 - 608 (2007)。gydF4y2Ba
戈登,d . e . et al . SARS-CoV-2蛋白质交互地图显示药物再利用的目标。gydF4y2Ba自然gydF4y2Ba583年gydF4y2Ba,459 - 468 (2020)。gydF4y2Ba
凡,k . r . et al .绑定SARS-CoV-2信封E蛋白对人类BRD4感染至关重要。gydF4y2Ba结构gydF4y2Ba30.gydF4y2Ba,1224 - 1232 (2022)。gydF4y2Ba
花,t . g . et al。结构SARS-CoV-2 ORF8,一个快速发展的免疫逃避的蛋白质。gydF4y2BaProc。《科学。美国gydF4y2Ba118年gydF4y2Bae2021785118 (2021)。gydF4y2Ba
Muth, d . et al。衰减的29个核苷酸删除复制的发热期间获得人际传播的早期阶段。gydF4y2Ba科学。代表。gydF4y2Ba8 gydF4y2Ba15177 (2018)。gydF4y2Ba
Hachim, a . et al . ORF8 ORF3b抗体是准确的早期和晚期SARS-CoV-2感染的血清学标记。gydF4y2BaImmunol Nat。gydF4y2Ba21gydF4y2Ba,1293 - 1301 (2020)。gydF4y2Ba
Wang x et al .准确诊断的COVID-19小说免疫原性分泌SARS-CoV-2 orf8蛋白质。gydF4y2BamBiogydF4y2Ba11gydF4y2Bae02431-20 (2020)。gydF4y2Ba
Bojkova, d . et al .蛋白质组学SARS-CoV-2-infected宿主细胞显示治疗目标。gydF4y2Ba自然gydF4y2Ba583年gydF4y2Ba,469 - 472 (2020)。gydF4y2Ba
Bouhaddou, m . et al .全球磷酸化SARS-CoV-2感染的景观。gydF4y2Ba细胞gydF4y2Ba182年gydF4y2Ba,685 - 712 (2020)。gydF4y2Ba
张,y . et al . SARS-CoV-2 ORF8蛋白的调节免疫逃避通过显示MHC-Ι。gydF4y2BaProc。《科学。美国gydF4y2Ba118年gydF4y2Bae2024202118 (2021)。gydF4y2Ba
拉希德,f . et al . SARS-CoV-2突变gydF4y2BaORF8gydF4y2Ba改变了结合网络与干扰素调节因子3逃避宿主的免疫系统。gydF4y2Ba前面。Microbiol。gydF4y2Ba12gydF4y2Ba703145 (2021)。gydF4y2Ba
拉希德,F。,Dzakah, E. E., Wang, H. & Tang, S. The ORF8 protein of SARS-CoV-2 induced endoplasmic reticulum stress and mediated immune evasion by antagonizing production of interferon β.病毒Res。gydF4y2Ba296年gydF4y2Ba198350 (2021)。gydF4y2Ba
耿,h . et al . SARS-CoV-2 ORF8形式细胞内聚集,抑制IFNγ-induced抗病毒基因表达在人类肺上皮细胞。gydF4y2Ba前面。Immunol。gydF4y2Ba12gydF4y2Ba679482 (2021)。gydF4y2Ba
林,x et al . ORF8有助于细胞激素风暴期间SARS-CoV-2感染通过激活IL-17途径。gydF4y2BaiSciencegydF4y2Ba24gydF4y2Ba102293 (2021)。gydF4y2Ba
吴x et al .病毒拟态的interleukin-17A SARS-CoV-2 ORF8。gydF4y2BamBiogydF4y2Ba13gydF4y2Bae0040222 (2022)。gydF4y2Ba
陈,j . et al .严重急性呼吸系统综合症冠状病毒2 ORF8蛋白能抑制I型干扰素生产针对HSP90B1信号。gydF4y2Ba前面。细胞。感染。Microbiol。gydF4y2Ba12gydF4y2Ba899546 (2022)。gydF4y2Ba
李,J.-Y。et al, ORF6 SARS-CoV-2 ORF8和核衣壳蛋白的I型干扰素抑制信号通路。gydF4y2Ba病毒Res。gydF4y2Ba286年gydF4y2Ba198074 (2020)。gydF4y2Ba
松岗,k . et al . SARS-CoV-2配件蛋白质ORF8糖蛋白为分泌细胞外。gydF4y2Ba生物。化学。gydF4y2Ba298年gydF4y2Ba101724 (2022)。gydF4y2Ba
Ohki, S。,Imamura, T., Higashimura, Y., Matsumoto, K. & Mori, M. Similarities and differences in the conformational stability and reversibility of ORF8, an accessory protein of SARS-CoV-2, and its L84S variant.物化学。Biophys。Commun >,gydF4y2Ba563年gydF4y2Ba,92 - 97 (2021)。gydF4y2Ba
Zinzula l .迷失在删除:神秘ORF8 SARS-CoV-2的蛋白质。gydF4y2Ba物化学。Biophys。Commun >,gydF4y2Bahttps://doi.org/10.1016/j.bbrc.2020.10.045gydF4y2Ba(2020)。gydF4y2Ba
谢,x et al . SARS-CoV-2的传染性cDNA克隆。gydF4y2Ba细胞宿主细菌gydF4y2Ba27gydF4y2Ba,841 - 848 (2020)。gydF4y2Ba
谢,x等。工程SARS-CoV-2使用反向遗传系统。gydF4y2BaProtoc Nat。gydF4y2Ba16gydF4y2Ba,1761 - 1784 (2021)。gydF4y2Ba
李,y . et al . SARS-CoV-2诱发双链rna介导呼吸epithelial-derived细胞和心肌细胞的先天免疫反应。gydF4y2BaProc。《科学。美国gydF4y2Ba118年gydF4y2Bae2022643118 (2021)。gydF4y2Ba
雅各,a . et al .推导人类自我更新的肺肺泡上皮II型细胞的多能干细胞。gydF4y2BaProtoc Nat。gydF4y2Ba14gydF4y2Ba,3303 - 3332 (2019)。gydF4y2Ba
王,r . et al . SARS-CoV-2重组宿主染色质结构。预印在gydF4y2BabioRxivgydF4y2Bahttps://doi.org/10.1101/2021.07.20.453146gydF4y2Ba(2021)。gydF4y2Ba
Valcarcel,。,Bensussen, A., Álvarez-Buylla, E. R. & Díaz, J. Structural analysis of SARS-CoV-2 ORF8 protein: pathogenic and therapeutic implications.前面。麝猫。gydF4y2Ba12gydF4y2Ba693227 (2021)。gydF4y2Ba
席尔瓦,j . a . et al .贡献K18 SARS-CoV-2配件蛋白质病毒致病性的人类gydF4y2BaACE2gydF4y2Ba转基因小鼠。gydF4y2Baj .性研究。gydF4y2Ba95年gydF4y2Bae0040221 (2021)。gydF4y2Ba
年轻,b . e . et al。的影响主要在SARS-CoV-2基因组中删除感染和炎症反应的严重程度:一项观察性队列研究。gydF4y2Ba《柳叶刀》gydF4y2Ba396年gydF4y2Ba,603 - 611 (2020)。gydF4y2Ba
方,S.-W。et al。强劲的特异性适应性免疫COVID-19患者SARS-CoV-2Δ382变种感染。gydF4y2Baj .中国。Immunol。gydF4y2Bahttps://doi.org/10.1007/s10875 - 021 - 01142 - zgydF4y2Ba(2021)。gydF4y2Ba
DeRonde, S。,Deuling, H., Parker, J. & Chen, J. Identification of a novel SARS-CoV-2 variant with a truncated protein inORF8gydF4y2Ba新一代测序的基因。gydF4y2Ba科学。代表。gydF4y2Ba12gydF4y2Ba4631 (2022)。gydF4y2Ba
波特,e . G。,Connelly, K. E. & Dykhuizen, E. C. Sequential salt extractions for the analysis of bulk chromatin binding properties of chromatin modifying complexes.j .粘度实验。gydF4y2Bahttps://doi.org/10.3791/55369gydF4y2Ba(2017)。gydF4y2Ba
Langmead, b &扎尔茨贝格,s . l .快gapped-read符合领结2。gydF4y2BaNat方法。gydF4y2Ba9gydF4y2Ba,357 - 359 (2012)。gydF4y2Ba
博尔格、a . M。,Lohse, M. & Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data.生物信息学gydF4y2Ba30.gydF4y2Ba,2114 - 2120 (2014)。gydF4y2Ba
张y等。基于模型分析ChIP-Seq (mac)。gydF4y2Ba基因组医学杂志。gydF4y2Ba9gydF4y2BaR137 (2008)。gydF4y2Ba
斯塔克,r &布朗,g . DiffBindgydF4y2Bahttps://doi.org/10.18129/B9.BIOC.DIFFBINDgydF4y2Ba(2017)。gydF4y2Ba
吴,f . et al。一种新的冠状病毒与人类呼吸道疾病在中国。gydF4y2Ba自然gydF4y2Ba579年gydF4y2Ba,265 - 269 (2020)。gydF4y2Ba
Sidoli, S。,Bhanu, N. V., Karch, K. R., Wang, X. & Garcia, B. A. Complete workflow for analysis of histone post-translational modifications using bottom-up mass spectrometry: from histone extraction to data analysis.j .粘度实验。gydF4y2Bahttps://doi.org/10.3791/54112gydF4y2Ba(2016)。gydF4y2Ba
林,s . et al . Stable-isotope-labeled组蛋白肽库组蛋白翻译后修饰和变体量化质谱分析。gydF4y2Ba摩尔。细胞。蛋白质组学gydF4y2Ba13gydF4y2Ba,2450 - 2466 (2014)。gydF4y2Ba
彼得森,a . C。,Russell, J. D., Bailey, D. J., Westphall, M. S. & Coon, J. J. Parallel reaction monitoring for high resolution and high mass accuracy quantitative, targeted proteomics.摩尔。细胞。蛋白质组学gydF4y2Ba11gydF4y2Ba,1475 - 1488 (2012)。gydF4y2Ba
MacLean b . et al .天际线:开源文档编辑器创建和分析目标蛋白质组学实验。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba,966 - 968 (2010)。gydF4y2Ba
Brademan, d R。,莱利:M。,Kwiecien, N. W. & Coon, J. J. Interactive Peptide Spectral Annotator: a versatile web-based tool for proteomic applications.摩尔。细胞。蛋白质组学gydF4y2Ba18gydF4y2BaS193-S201 (2019)。gydF4y2Ba
Perez-Riverol y . et al ., 2022年骄傲数据库资源:中心质量spectrometry-based蛋白质组学证据。gydF4y2Ba核酸Res。gydF4y2Ba50gydF4y2BaD543-D552 (2022)。gydF4y2Ba
确认gydF4y2Ba
我们感谢并承认m . Weitzman反馈和建议,a的显微镜支持,c . Comar病毒感染,r . Jain和p . Shaw协议,m·费尔德曼和k . Montone提供患者样本,和美国狼和p .科博监督和支持。J.K.,S.T., E.K. and research were supported by the Penn Center for Coronavirus and Other Emerging Pathogens and the Dean’s Innovation Fund from the Perelman School of Medicine at the University of Pennsylvania. E.K. was supported by the Sloan Research Fellowship, the Klingenstein-Simons Fellowship, the NARSAD Young Investigator Award and NIH grants R00MH111836 and 1DP2MH129985. Viral work was supported by NIH grant RO1AI140442 supplement for SARS-CoV-2 and funds from the Penn Center for Coronavirus Research and Other Emerging Pathogens. D.M.R. was supported in part by T32AI055400. A.P., A.A.K. and J.A.E. were supported by NIH grant R35HL140018. R.T. and W.Y. were supported in part by institutional funds from the University of Pennsylvania Perelman School of Medicine to the iPSC Core. iPSC cell line generation and sharing were supported by NIH grants NO175N92020C00005 and U01TR001810. Mass spectrometry was supported by AI118891. P.-Y.S. was supported by NIH grants HHSN272201600013C, U01AI151801 and U19AI171413 and by awards from the Sealy Smith Foundation, the Kleberg Foundation, the John S. Dunn Foundation, the Amon G. Carter Foundation, the Gillson Longenbaugh Foundation and the Summerfield Robert Foundation.
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
J.K.设计、执行和分析结果对于大多数实验。钢桁架生成细胞,DNA样本和构造和执行分析。D.M.R.和李y执行SARS-CoV-2病毒感染和病毒感染的分析。kg。Z.Z.执行和ChIP-seq实验的结果进行了分析。伙夫ATAC-seq和生成的样本组蛋白天车执行分析。J.C.执行和分析质谱实验的结果指导B.A.G. X.Z.,X.X. P.-Y.S.生成删除病毒。y局域网提供生物信息学分析。F.L.C.-D。R.T. iAT2细胞生成和指导从W.Y.和E.M. D.N.K. K.D.A.干细胞系提供。美联社, A.A.K. and J.A.E. performed ORF8 and nuclear lamina imaging and analysis. S.L.B. provided input and expertise and led ChIP–seq studies. S.R.W. provided input and expertise and led viral work. E.K. led the project and wrote the manuscript.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
P.-Y.S.已收到资金支持的实验室从辉瑞公司赞助的研究协议,基诺,默克公司,葛兰素史克,IGM生物科学和Atea制药。P.-Y.S.是科学顾问委员会的一员AbImmune FlaviTech创始人。S.R.W.的科学顾问委员会Ocugen Immunome。gydF4y2Ba
同行评审gydF4y2Ba
同行审查的信息gydF4y2Ba
自然gydF4y2Ba感谢匿名评论者对他们的贡献的同行评审工作。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然保持中立在发表关于司法主权地图和所属机构。gydF4y2Ba
扩展数据数据和表gydF4y2Ba
扩展数据图1 Orf8是一个假定的组蛋白模拟。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)比对识别执行公认的组蛋白模拟网站SARS-CoV-2基因组内。(gydF4y2BabgydF4y2Ba)的确切数量顺序重叠SARS-CoV-2之间发现蛋白质和氨基酸组蛋白蛋白质。(gydF4y2BacgydF4y2Ba)Orf8和H3与其他提出的重叠病例组蛋白模仿。精确的氨基酸重叠显示在深蓝色和浅蓝色所示结构相似的氨基酸重叠。NS1来自甲型H3N2。蛋白E SARS-CoV-2先前提出的模拟。G9a模仿H3是人类蛋白质。(gydF4y2BadgydF4y2Ba)之前提出的组蛋白H3模仿SARS-CoV-2蛋白e (gydF4y2BaegydF4y2Ba)重叠H3的冠状蛋白质。(gydF4y2BafgydF4y2Ba)方舟图案出现在蝙蝠冠Orf8和变异的担忧,但没有找到与冠Orb8a / b或冠状前体Orf8突变之前生成两个不同的蛋白质。(gydF4y2BaggydF4y2Ba- - - - - -gydF4y2BahgydF4y2Ba)Orf8成绩单(g)和蛋白质(h)表达SARS-CoV-2 Caco-2感染细胞数据集出版。莫伊= 1。情节表明意味着±SEM。gydF4y2Ba
扩展数据图2 Orf8本地化。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)。亚细胞分离与Strep-Orf8 HEK293T细胞转染表明Orf8存在于细胞质和细胞核。(gydF4y2BabgydF4y2Ba与核纤层蛋白B) HEK293T细胞表达Orf8 co-stained streptactin检测Orf8。(gydF4y2BacgydF4y2Ba)的旋转z-stacks(右和底部面板每个染色)显示colocalization Orf8和核纤层蛋白b (gydF4y2BadgydF4y2Ba)Orf8专门抗血清检测Strep-tagged Orf8通过免疫印迹与Strep-Orf8 HEK293T细胞转染。(gydF4y2BaegydF4y2Ba)Orf8专门污渍感染A549抗血清gydF4y2Ba王牌gydF4y2Ba细胞后48 h SARS-CoV-2感染没有染色观察模拟感染。(gydF4y2BafgydF4y2Ba)删除构造用于测试Orf8 ARKSAP主题的影响。(gydF4y2BaggydF4y2Ba)细胞分数生成用于后续chromatin-immunoprecipitation Orf8 ChIP-sequencing显示比率越低gydF4y2BaΔARKSAPgydF4y2Ba出现在染色质分数然后Orf8。(gydF4y2BahgydF4y2Ba)基因跟踪Orf8 ChIP-sequencing规范化输入控件。控制显示负控制免疫球蛋白ChIP-sequencing。(gydF4y2Ba我gydF4y2Ba基因组地区)Orf8丰富H3K27me3高和相对较低的输入控件H3K9me2浓缩H3K9me3地区高。(gydF4y2BajgydF4y2Ba)的比例从编码读取Orf8-enriched区域内数据集。N = 4样本ENCSR000FCJ ENCSR179BUC H3K9me3, N = 2样本ENCSR55LYM H3K9me2,和N = 4 H3K27me3 ENCSR000AKD样本。(gydF4y2BakgydF4y2Ba)Diffbind分析H3K27me3 vs Orf8丰富地区的输入控件。比例尺= 5μm在a - b,在d。10μm凝胶源数据,见补充图。gydF4y2Ba1,f, kgydF4y2Ba。酒吧情节表明意味着±SEM。gydF4y2Ba
扩展数据图3 Orf8蛋白质相互作用和修改。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba与核纤层蛋白)Orf8 co-immunoprecipitates复杂联系蛋白质包括LaminB HP1α,H3, KAT2A。”——“显示细胞不表达负面控制Orf8 IPs并行执行。(gydF4y2BabgydF4y2Ba)互惠co-immunoprecipitation核纤层蛋白和H3证实Orf8绑定。(gydF4y2BacgydF4y2Ba)Orf8gydF4y2BaΔARKSAPgydF4y2Ba与LaminB co-immunoprecipitation HP1α,H3, KAT2A没有检测到。(gydF4y2BadgydF4y2Ba)Orf8 Orf8gydF4y2BaΔARKSAPgydF4y2Baco-immunoprecipitates MHC1复杂蛋白质hla a2。(gydF4y2BaegydF4y2Ba)Orf8 co-immunoprecipitation Brd4未被检测到。(gydF4y2BafgydF4y2Ba)确认转录因子SP2的质谱分析。Orf8产生免疫沉淀反应Streptactin珠子和溶菌产物被探测SP2和Streptactin。蛋白质纯化的质谱分析孤立使用完整的细胞溶解产物条件而不是通过染色质浓缩的分数。这种方法(方法)中描述了主要是胞质约束力的合作伙伴和数量有限的chromatin-associated蛋白质。(gydF4y2BaggydF4y2Ba)有针对性的质谱分析trypsin-digested Orf8包含拟议中的组蛋白的肽在Orf8模仿。Orf8乙酰化在检测到K52 3 +肽通过有针对性的质谱。MS / MS谱匹配产品的离子(b离子蓝色,y离子红、c离子黄色)在10 ppm质量错误。(gydF4y2BahgydF4y2Ba)代表共焦图像HEK293T细胞转染orf8表达式构造(绿色)和彩色核纤层蛋白A / C(青色)和核纤层蛋白B1(红色)。DAPI复染色显示为蓝色。5μm比例尺。(gydF4y2Ba我gydF4y2Ba)棒图显示分布的核纤层蛋白A / C或核纤层蛋白B1每个细胞在模拟信号强度和Orf8-expressing细胞。n = 129和86个细胞样本。统计分析是使用Mann-Whitney执行测试。(gydF4y2BajgydF4y2Ba)代表共焦图像HEK293T细胞转染Orf8表达式构造(绿色)和彩色H3K9me2(红色)和核纤层蛋白B(青色)。DAPI复染色显示为蓝色。5μm比例尺。(gydF4y2BakgydF4y2Ba)条形图显示一部分H3K9me2-marked染色质在核板/外围控制和Orf8-expressing细胞。n≥32细胞/样品。(gydF4y2BalgydF4y2Ba)代表共焦图像所示j HEK293T细胞转染Orf8表达式构造(绿色)和染色与Orf8核纤层蛋白B1(红色)和核纤层蛋白B1浓缩细胞沿虚线所示绘制图像。5μm比例尺。使用韦尔奇双向t测试执行统计分析。箱形图显示值,第25和第75百分位数。须展示图基的置信区间。凝胶源数据,请参阅补充图。gydF4y2Ba1 c, d, g-jgydF4y2Ba。gydF4y2Ba
扩展数据图4 Orf8影响组蛋白天车。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)Orf8 H3K27ac没有显著影响。N = 616 (GFP), 550 (Orf8)从3独立的转染。(gydF4y2BabgydF4y2BaH3磷酸化丝氨酸10 (H3S10ph), H3K9 dimethylation (H3K9me2),核纤层蛋白B和全球乙酰化作用(组蛋白或non-histone蛋白质)的GFP或Orf8表达细胞通过流式细胞仪分离。(gydF4y2BacgydF4y2Ba)Orf8构造测试显示类似的HEK293T细胞中的表达水平。N = 137 (Orf8), 87 (Orf8gydF4y2BaΔARKSAPgydF4y2Ba),120 (S84L)从4个独立的转染。(gydF4y2BadgydF4y2Ba- - - - - -gydF4y2BafgydF4y2Ba)Orf8 SARS-CoV-2基因组变异常见,Orf8-S84L,显示了相同的影响组蛋白天车H3K9me3 (d), H3K27me3 (e)和H3K9ac (f)。N = 332 (GFP), 237 (S84L) H3K9me3细胞;186年、166年为H3K27me3细胞;332年,从2独立H3K9ac转染237细胞。(gydF4y2BaggydF4y2Ba6)Orf8氨基酸删除外的对H3K9me3 ARKSAP图案显示了相同的影响。N = 216 (GFP), 120 (Orf8)、88 (AGSKSP)细胞2独立的转染。(g) 1路的方差分析与因果双向的t检验和Bonferroni调整。(b、d、e、f)双向的学习任务。酒吧= 10μm规模。凝胶源数据,请参阅补充图。gydF4y2Ba1 n - mgydF4y2Ba。gydF4y2Ba
扩展数据图5 Orf8影响染色质组蛋白天车和可访问性。gydF4y2Ba
(gydF4y2Ba一个gydF4y2BaWT Orf8和Orf8)表达式gydF4y2BaΔARKSAPgydF4y2BaFAC排序细胞用于免疫印迹分析。(gydF4y2BabgydF4y2BaWT Orf8和Orf8)表达式gydF4y2BaΔARKSAPgydF4y2BaFAC排序细胞用于H3K9ac CUT&TAG。(gydF4y2BacgydF4y2Ba)H3K9ac CUT&TAG测序平均轮廓表达基因。(gydF4y2BadgydF4y2BaWT Orf8和Orf8)表达式gydF4y2BaΔARKSAPgydF4y2Ba从FAC细胞用于ATAC-seq排序。(gydF4y2BaegydF4y2Ba)H3K9ac CUT&TAG平均配置高,中,低,non-expressing基因。(gydF4y2BafgydF4y2Ba)ATAC-seq平均配置高,中,低,non-expressing基因。(gydF4y2BaggydF4y2Ba)H3K9ac CUT&TAG和ATAC-seq基因跟踪相关的基因病毒反应HEK 293 t细胞表达质粒,Orf8或Orf8gydF4y2BaΔARKSAPgydF4y2Ba。(gydF4y2BahgydF4y2Ba有限的)基因跟踪例子Orf8和Orf8之间变化gydF4y2BaΔARKSAPgydF4y2Ba在H3K9ac CUT&TAG ATAC-seq。N = 2生物独立样本a, b, d。酒吧情节表明平均值。流式细胞仪控制策略和细胞数量孤立无花果所示。SgydF4y2Ba2gydF4y2Ba。gydF4y2Ba
扩展数据图6 Orf8扰乱基因表达。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)的表达Orf8gydF4y2BaWTgydF4y2Ba和Orf8gydF4y2BaΔARKSAPgydF4y2Ba从FAC细胞用于RNA-seq排序。(gydF4y2BabgydF4y2Ba)火山的微分Orf8的基因表达分析gydF4y2BaWTgydF4y2Ba表达细胞GFP表达相比。(gydF4y2BacgydF4y2Ba)火山的微分Orf8的基因表达分析gydF4y2BaΔARKSAPgydF4y2Ba表达细胞GFP表达相比。红色代表显著差异表达的基因。(gydF4y2BadgydF4y2Ba)重叠的基因或调节Orf8 Orf8gydF4y2BaΔARKSAPgydF4y2Ba相对于GFP表达细胞。(gydF4y2BaegydF4y2Ba)基因本体分析的基因表达下调的Orf8 Orf8相比gydF4y2BaΔARKSAPgydF4y2Ba。(gydF4y2BafgydF4y2Ba- - - - - -gydF4y2BaggydF4y2Ba)火山的微分Orf8的基因表达分析gydF4y2BaWTgydF4y2Ba表达细胞Orf8相比gydF4y2BaΔARKSAPgydF4y2Ba表达细胞画的p值(f)和平均表达式(g)。gydF4y2BahgydF4y2Ba)跟踪Orf8基因诱导的基因gydF4y2BaΔARKSAPgydF4y2Ba但Orf8抑制反应。(gydF4y2Ba我gydF4y2Ba)基因的基因不受Orf8表达式。(gydF4y2BajgydF4y2Ba)H3K9ac CUT&Tag读取在Orf8诗句Orf8下来调节度gydF4y2BaΔARKSAPgydF4y2Ba。(gydF4y2BakgydF4y2Ba)ATAC-seq读取在Orf8诗句Orf8下来调节度gydF4y2BaΔARKSAPgydF4y2Ba。度:差异表达基因。火山的情节显示差异表达基因与多重比较修正DESeq2分析。基因本体论意义基于clusterProfiler分析Benjamini-Hochberg假定值调整。RNA-seq, N = 2 GFP和Orf8gydF4y2BaΔARKSAPgydF4y2Ba并为Orf8 N = 3gydF4y2BaWTgydF4y2Ba。流式细胞仪控制策略和细胞数量孤立补充图所示。gydF4y2Ba2gydF4y2Ba。gydF4y2Ba
扩展数据图7 Orf8介导SARS-CoV-2对压抑的组蛋白铝的影响。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)中存在的分析SARS-CoV-2基因的表达在A549 RdRp病毒效价分析gydF4y2Ba王牌gydF4y2Ba肺细胞在24和48 h后感染SARS-CoV-2gydF4y2BaWTgydF4y2Ba或SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba在莫伊= 1。(gydF4y2BabgydF4y2Ba)在A549空斑实验分析病毒效价gydF4y2Ba王牌gydF4y2Ba肺细胞在24和48 h后感染SARS-CoV-2gydF4y2BaWTgydF4y2Ba或SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba在莫伊= 1。N = 3并行复制从感染了。(gydF4y2BacgydF4y2Ba- - - - - -gydF4y2BadgydF4y2Ba)ChIP-RX H3K9me3 (c)和H3K27me3 A549的(d)gydF4y2Ba王牌gydF4y2Ba细胞与SARS-CoV-2gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba或模拟感染莫伊= 1,48 h后感染。N = 3。山峰所示面板的c - d表明sic峰从合并ChIP-RX数据区域。灰色的线表示边界和峰值within-peak距离两端固定的高峰。(gydF4y2BaegydF4y2Ba)ChIP-seq基因跟踪信号通路相关基因的病毒的反应。P值表明双向方差分析与Šidak多个比较修正。酒吧情节表明意味着±SEM。RPM,读取每百万映射读取。gydF4y2Ba
扩展数据图8 Orf8介导SARS-CoV-2影响组蛋白天车。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba,gydF4y2BacgydF4y2Ba,gydF4y2BaegydF4y2Ba)H3K9me3 (a), H3K27me3 (c)或H3K9ac (e) A549的染色gydF4y2Ba王牌gydF4y2Ba细胞SARS-CoV-2后24小时gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba或模拟感染莫伊= 1。(gydF4y2BabgydF4y2Ba,gydF4y2BadgydF4y2Ba,gydF4y2BafgydF4y2Ba)量化H3K9me3 (b), H3K27me3 (d)或H3K9ac (f)。H3K9me3 N = 39 (SARS-CoV-2) 35 (SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba),H3K27me3 N = 48 (SARS-CoV-2), 37 (SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba),H3K9ac N = 94 (SARS-CoV-2), 120 (SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba)细胞规范化邻居细胞无毒性1 - 2独立感染。虚线表示相对信号模拟感染状况。(gydF4y2BaggydF4y2Ba)量化H3K9me3在感染细胞和邻近的细胞来自同一组织切片分别为每个病人样本显示,相对于控制样本。示例1,N = 3感染和48未感染,样品2 N = 125感染和未感染,感染样本3,N = 5 - 55未感染的细胞。(b, d, f) 1路的方差分析与因果双向的t检验和Bonferroni调整。酒吧= 10μm规模。盒子情节集中在中间,范围在25gydF4y2BathgydF4y2Ba和75年gydF4y2BathgydF4y2Ba百分位,最小值和最大值定义为±1.5 x四分位范围,中值与胡须在范围扩展到最低/最高价值。gydF4y2Ba
扩展数据图9 RNA-sequencing读度比较和48小时。gydF4y2Ba
(gydF4y2Ba一个gydF4y2Ba)读取对齐到人类和SARS-CoV-2基因组在感染后24小时。(gydF4y2BabgydF4y2Ba水平细胞感染SARS-CoV-2 SARS-CoV-2成绩单gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba,或者SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba24小时后感染。(gydF4y2BacgydF4y2Ba)读取对齐到人类和SARS-CoV-2基因组48 h后感染。(gydF4y2BadgydF4y2Ba水平细胞感染SARS-CoV-2 SARS-CoV-2成绩单gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba,或者SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba感染后48 h。(gydF4y2BaegydF4y2Ba)规范化读取SARS-CoV-2 Orf8和人类记录的组蛋白H3基因在细胞感染SARS-CoV-2gydF4y2BaWTgydF4y2Ba或SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba。(gydF4y2BafgydF4y2Ba)重叠SARS-CoV-2反应的差异表达基因gydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba,或者SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba相比模拟感染莫伊= 1,48 h后感染。(gydF4y2BaggydF4y2BaRNA-seq A549的)差异基因表达分析gydF4y2Ba王牌gydF4y2Ba细胞SARS-CoV-2后48 hgydF4y2BaWTgydF4y2Ba,SARS-CoV-2gydF4y2BaΔOrf8gydF4y2Ba,或者SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba相比,模拟感染莫伊= 1。显著差异表达基因(相对于模拟感染)所示蓝色(下降)和红色(了)。N = 3。火山的情节显示差异表达基因与多重比较修正DESeq2分析。酒吧情节表明意味着±SEM。gydF4y2Ba
扩展数据图10在A549 ChIP-sequencing和RNA-sequencing数据进行比较gydF4y2Ba王牌gydF4y2Ba细胞。gydF4y2Ba
(gydF4y2Ba一个gydF4y2BaSARS-CoV-2)基因本体分析的基因调节gydF4y2BaΔOrf8gydF4y2Ba感染。(gydF4y2BabgydF4y2Ba)SARS-CoV-2之间基因表达的变化gydF4y2BaΔOrf8gydF4y2Ba和SARS-CoV-2gydF4y2BaΔARKSAPgydF4y2Ba。(gydF4y2BacgydF4y2BaSARS-CoV-2)基因本体分析的基因调节gydF4y2BaΔARKSAPgydF4y2BaSARS-CoV-2相比gydF4y2BaΔOrf8gydF4y2Ba。(gydF4y2BadgydF4y2Ba)意味着H3K9me3峰值差异基因,改变,或调节对感染的反应。(gydF4y2BaegydF4y2Ba)意味着H3K27me3峰值差异基因,改变,或调节对感染的反应。(gydF4y2BafgydF4y2Ba)意味着H3K9ac峰值差异的基因差异表达对感染的反应。高峰值取决于diffbind最重要的峰值相交TSS的基因。N = 3 ChIP-sequencing每个修改除了N = 2 H3K9ac野生型SARS-CoV-2gydF4y2BaWTgydF4y2Ba。P值表明克鲁斯卡尔-沃利斯检验结果,后跟一个因果Dunn的测试和Bonferroni调整。n表示不显著的组间差异表达的基因。盒子情节集中在中间,范围在25gydF4y2BathgydF4y2Ba和75年gydF4y2BathgydF4y2Ba百分位,最小值和最大值定义为±1.5 x四分位范围,中值与胡须在范围扩展到最低/最高价值。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
这个文件包含补充图1(原始免疫印迹图像),补充图2(流式细胞仪控制策略)和传说为补充表1 - 6(见单独的Excel文件表格)。gydF4y2Ba
补充表1gydF4y2Ba
ORF8约束力的合作伙伴。gydF4y2Ba
补充表2gydF4y2Ba
基因本体论分析ORF8后基因表达变化的表达式。gydF4y2Ba
补充表3gydF4y2Ba
组蛋白与ORF8天车变化表达式。gydF4y2Ba
补充表4gydF4y2Ba
所有感染数据。gydF4y2Ba
补充表5gydF4y2Ba
基因本体论分析A549 SARS-CoV-2感染后基因表达的变化gydF4y2Ba王牌gydF4y2Ba细胞。gydF4y2Ba
补充表6gydF4y2Ba
抗体在手稿中使用。gydF4y2Ba
源数据gydF4y2Ba
权利和权限gydF4y2Ba
Springer性质或其许可方(例如一个社会或其他合作伙伴)拥有独占权下本文与作者出版协议(s)或其他情况下(年代);作者self-archiving接受这篇文章的手稿版本是完全由这样的出版协议的条款和适用法律。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
凯,J。,Thudium, S., Renner, D.M.et al。gydF4y2Ba通过组蛋白拟态SARS-CoV-2破坏主机表观遗传调控。gydF4y2Ba自然gydF4y2Ba610年gydF4y2Ba,381 - 388 (2022)。https://doi.org/10.1038/s41586 - 022 - 05282 - zgydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586 - 022 - 05282 - zgydF4y2Ba
本文引用的gydF4y2Ba
SARS-CoV-2重组宿主染色质结构gydF4y2Ba
微生物学性质gydF4y2Ba(2023)gydF4y2Ba
表观遗传观点COVID-19感染及相关细胞因子风暴:更新的审核gydF4y2Ba
感染gydF4y2Ba(2023)gydF4y2Ba
SARS-CoV-2模仿宿主蛋白质旁路防御gydF4y2Ba
自然gydF4y2Ba(2022)gydF4y2Ba
组蛋白被SARS-CoV-2模仿gydF4y2Ba
自然评论微生物学gydF4y2Ba(2022)gydF4y2Ba
评论gydF4y2Ba
通过提交评论你同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条件或准则请国旗是不合适的。gydF4y2Ba