文摘gydF4y2Ba
DNA从胞质细胞器转移到细胞核内共生场比赛- - -多数nuclear-mitochondrial遗留下来的片段(NUMTs)被认为是古代,前人类物种形成gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba。这里我们分析全基因组序列与癌症以及展示66083人12509人的线粒体DNA转移到细胞核,导致一个复杂NUMT景观。超过99%的人至少有1637种不同的NUMTs之一,与1的8个人拥有一件NUMT存在在不到0.1%的人口。超过90%的现存NUMTs我们评估插入人类从猿分化后的核基因组。一旦嵌入,序列不再在线粒体内的进化约束下,和NUMT-specific突变有不同的线粒体DNA突变的签名。新创NUMTs观察生殖系每10次gydF4y2Ba4gydF4y2Ba在每10出生和一次gydF4y2Ba3 gydF4y2Ba癌症。NUMTs优先参与非编码线粒体DNA,与核转录和复制链接到其起源、插入涉及多个机制包括双链断裂修复与公关相关域锌指蛋白9 (PRDM9)绑定。肿瘤特异的频率NUMTs癌症之间的不同,可能包括一个因果插入黏液样脂肪肉瘤。我们发现选择的证据的基础上对NUMTs大小和基因组的位置,形成一个高度异构和动态人类NUMT景观。gydF4y2Ba
主要gydF4y2Ba
从胞质细胞器基因的转移到细胞核内共生理论支撑的线粒体的起源gydF4y2Ba3 gydF4y2Ba。高阶生物有逐渐较小的线粒体基因组,反映出线粒体基因易位到核基因组在进化的时间,促进协调由胞质转化机械organellar蛋白质的合成gydF4y2Ba2gydF4y2Ba。这个过程使得non-expressed线粒体DNA片段(mtDNA)在非编码空间,与许多NUMTs跨物种共享,反映出他们古老的起源gydF4y2Ba4gydF4y2Ba。最近,全基因组测序(WGS)已经确认件NUMTs在人类身上gydF4y2Ba5克ydF4y2Ba,这意味着mtDNA-nuclear转移是一个持续的过程,但生殖系NUMT形成仍是未知的。小说mtDNA-nuclear入侵具有重要意义,因为他们可能会破坏蛋白质编码基因,导致疾病gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba,并创建混合种群mtDNA文物相似gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba(pseudo-heteroplasmy)。无意中解读NUMT序列作为mtDNA变体可能混淆线粒体疾病的诊断gydF4y2Ba12gydF4y2Ba和提高关于mtDNA可能的父亲继承的问题gydF4y2Ba13gydF4y2Ba。gydF4y2Ba
大型WGS项目提供了一个机会在更大的深度描述人类NUMTs比其他物种。这里我们描述人类的景观NUMTs在66083人,其中包括8201 mother-father-child三人赛和12509年tumour-normal内组织对100000人基因工程在英格兰。这为mtDNA变异的解释提供了一个资源跨不同人口和我们理解核基因组进化。结果在一个可搜索的在线数据库gydF4y2Bahttps://wwei.shinyapps.io/numts/gydF4y2Ba。gydF4y2Ba
人类生殖系NUMTs的阿特拉斯gydF4y2Ba
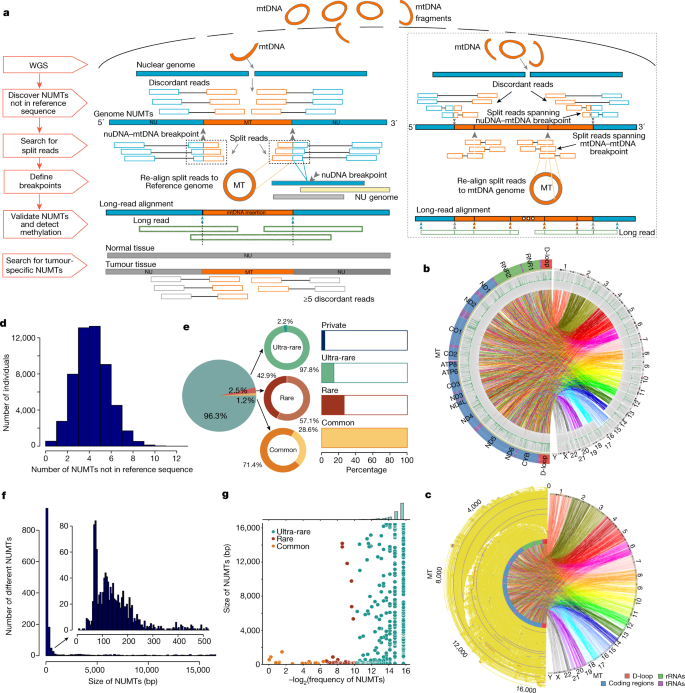
我们最初68348基因组67875个参与者的基因组学研究英格兰罕见的疾病项目gydF4y2Ba14gydF4y2Ba。毕竟质量控制(QC)步骤(方法),我们对25436名男性和28138名女性从0到99岁(扩展数据图。gydF4y2Ba1 a, bgydF4y2Ba),包括8201三人小组的报告亲缘与基因组预测(方法)是相一致的。使用验证短内容NUMT检测管道gydF4y2Ba5克ydF4y2Ba,gydF4y2Ba15gydF4y2Ba(无花果。gydF4y2Ba1gydF4y2Ba),我们发现335891 NUMTs参考序列中不存在基于至少两对不和谐的阅读中发现53535人(> 99.9%),其中包括3829种不同NUMTs(扩展数据图。gydF4y2Ba1 d, egydF4y2Ba)。增加紧缩NUMT检测至少5不和谐的阅读对精制254195 NUMTs收益率(53507年1637种不同NUMTs(99.87%)个人),参考序列(图中不存在。gydF4y2Ba1 b, cgydF4y2Ba和补充表gydF4y2Ba1克ydF4y2Ba)。这higher-stringency数据集的基础研究结果,我们引用NUMTs常见(频率(gydF4y2BaFgydF4y2Ba(≤0.1%)≥1%),罕见gydF4y2BaFgydF4y2Ba< 1%)、件(gydF4y2BaFgydF4y2Ba< 0.1%)或私人(只有一个家庭中发现)。读测序验证我们NUMT调用管道在99%的情况下(182年184 NUMTs 39个人;无花果。gydF4y2Ba1gydF4y2Ba)(方法)。gydF4y2Ba
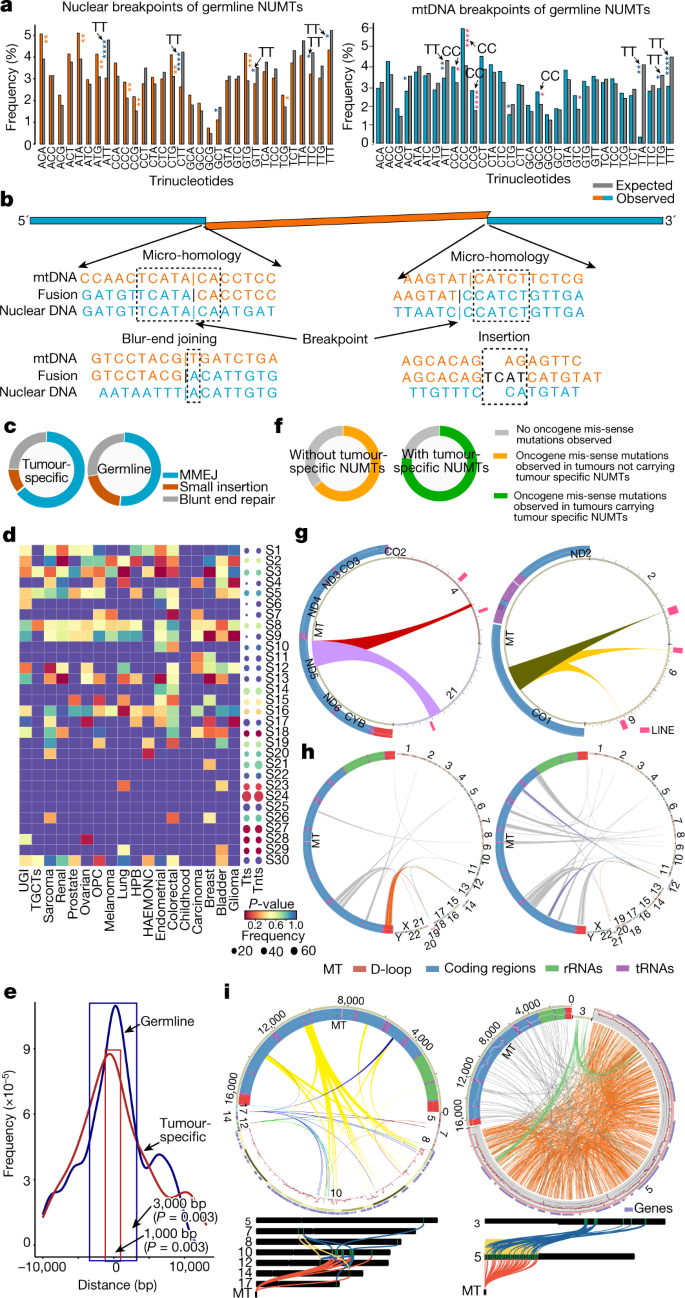
一个gydF4y2Ba不存在,生物信息学管道检测NUMTs参考序列,包括连接NUMTs(盒装)。简短的写着:mtDNA是橙色,核DNA (nuDNA)蓝色所示。长读绿色所示。太,线粒体基因组;ν基因组,核基因组。gydF4y2BabgydF4y2Ba在53574年发现了1637种不同NUMTs个人。从外面:(1)核染色体(右)和mtDNA基因(左);(2)件和稀有NUMTs频率;(3)共同NUMTs的频率;(4)链接连接mtDNA和核断点。gydF4y2BacgydF4y2BamtDNA碎片的1637种不同NUMTs从53574个人。离开,大小和位置NUMTs mtDNA。链接连接mtDNA和核碎片插入站点。gydF4y2BadgydF4y2Ba,平均每个个人的NUMTs数量参考序列中不存在,被检测到至少5个不和谐的读取。gydF4y2BaegydF4y2Ba(左)的比例由人口频率(NUMTs常见,gydF4y2BaFgydF4y2Ba≥1%;罕见,≤0.1%gydF4y2BaFgydF4y2Ba< 1%;件,gydF4y2BaFgydF4y2Ba< 0.1%)。中间,甜甜圈图显示的比例(深颜色)和新(浅色)NUMTs识别。正确的,条形图显示个人携带常用的频率,罕见,件和私人NUMTs。99.87%的人携带至少一个共同NUMT (gydF4y2BaFgydF4y2Ba> 1%),26.2%的人携带至少一个NUMTgydF4y2BaFgydF4y2Ba< 1%,14.2%的个人随身携带至少一个NUMTgydF4y2BaFgydF4y2Ba< 0.1%和3.6%的人携带至少一个私人NUMT。gydF4y2BafgydF4y2Ba生殖系NUMTs大小分布。NUMTs小于500 bp的插图所示。gydF4y2BaggydF4y2Ba,NUMT频率和大小之间的相关性。gydF4y2Ba
个人平均4.7 NUMTs(其中。= 1.6),没有参考序列(图中。gydF4y2Ba1 dgydF4y2Ba)。男性和女性之间没有差别(gydF4y2BaPgydF4y2Ba值= 0.834,Wilcoxon rank-sum测试;扩展的数据图。gydF4y2Ba1 fgydF4y2Ba随着年龄的增长)或(gydF4y2BaPgydF4y2Ba值= 0.95,皮尔森的相关性;扩展的数据图。gydF4y2Ba1克gydF4y2Ba)。共有1615个不同的NUMTs(98.7%)在26.2%的人没有出现在参考序列和罕见或件(gydF4y2BaFgydF4y2Ba< 1%),1567个不同NUMTs(96.1%)在14.2%的人是件(gydF4y2BaFgydF4y2Ba< 0.1%)和1039例(63.7%)NUMTs看到3.6%的个人私人(NUMTs中发现只有一个家庭)(图gydF4y2Ba1 egydF4y2Ba和扩展数据图。gydF4y2Ba1 dgydF4y2Ba)。正如预期的那样,绝大多数(71.4%)的常见NUMTs (gydF4y2BaFgydF4y2Ba≥1%)之前报告的gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba(补充表gydF4y2Ba2gydF4y2Ba)。因此,结合稀有件NUMTs公共NUMT数据,我们发现1564 NUMTs,据我们所知,没有报道之前(图。gydF4y2Ba1 egydF4y2Ba)(方法)。定义mtDNA断点两端(无花果。gydF4y2Ba1gydF4y2Ba),NUMTs的大小不等,从24日英国石油公司对整个线粒体基因组(平均156个基点,意味着1597个基点和年代。d 3651个基点)。大多数NUMTs短插入(63.2%的NUMTs少于200 bp和77.8%的人不到500个基点大小)(图gydF4y2Ba1 fgydF4y2Ba),逆NUMT大小和频率(人口之间的关系gydF4y2BaPgydF4y2Ba= 0.021,gydF4y2BaRgydF4y2Ba2gydF4y2Ba=−0.058,皮尔森相关测试;无花果。gydF4y2Ba1克gydF4y2Ba),与正在进行的选择对大型NUMTs一致。符合这一点,我们观察到的主要频率和分布的差异NUMTs不同民族之间,与非洲和东亚人最明显的与NUMT频率和染色体的位置(图。gydF4y2Ba2gydF4y2Ba和扩展数据图。gydF4y2Ba2gydF4y2Ba)。gydF4y2Ba
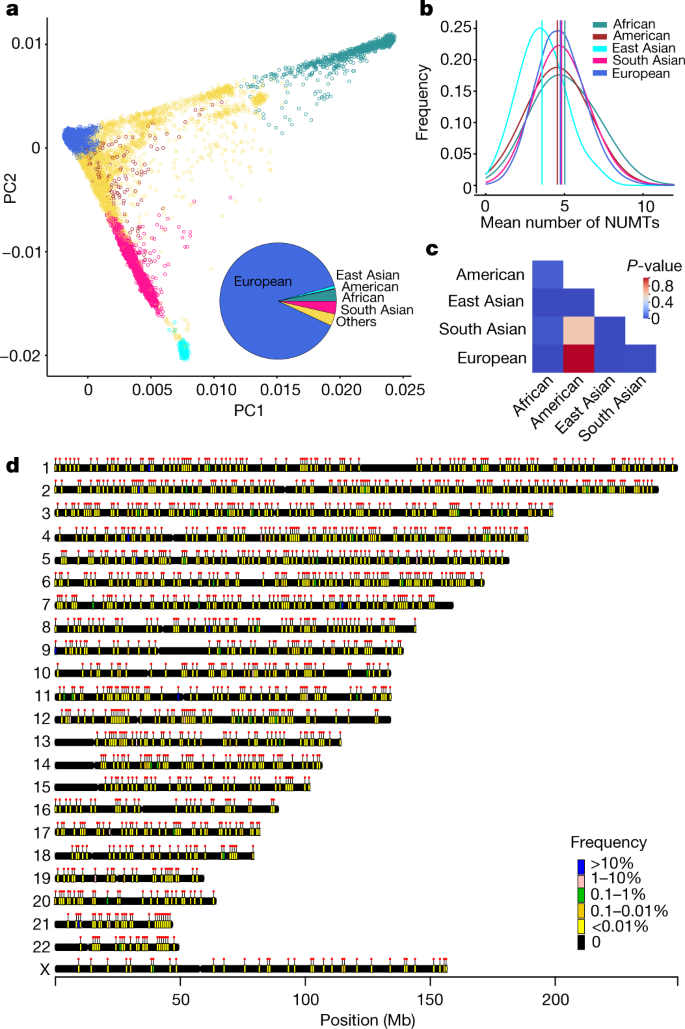
一个gydF4y2Ba,核基因型常见的单核苷酸多态性(SNPs)投射到两大主要组件(PC1和PC2)。个人彩色根据分配的核基因组的祖先。饼图显示每组整体的比例:东亚(青色)、南亚(粉红色),非洲(绿色),美国(红色)、欧洲(蓝色)和未赋值的(黄色)。gydF4y2BabgydF4y2Ba的平均数量NUMTs在人群中发现不同的祖先。垂直线显示NUMTs从每个人口的平均数量。gydF4y2BacgydF4y2Ba、热地图gydF4y2BaPgydF4y2Ba值成对比较的平均数量NUMTs发现群体间不同的血统(双面Wilcoxon rank-sum测试)。gydF4y2BadgydF4y2Ba,染色体NUMT插入的位置检测到在这项研究中,彩色NUMTs的频率。点显示NUMTs的位置。为每个祖先染色体的位置不同NUMT插入检测扩展数据图所示。gydF4y2Ba2gydF4y2Ba。gydF4y2Ba
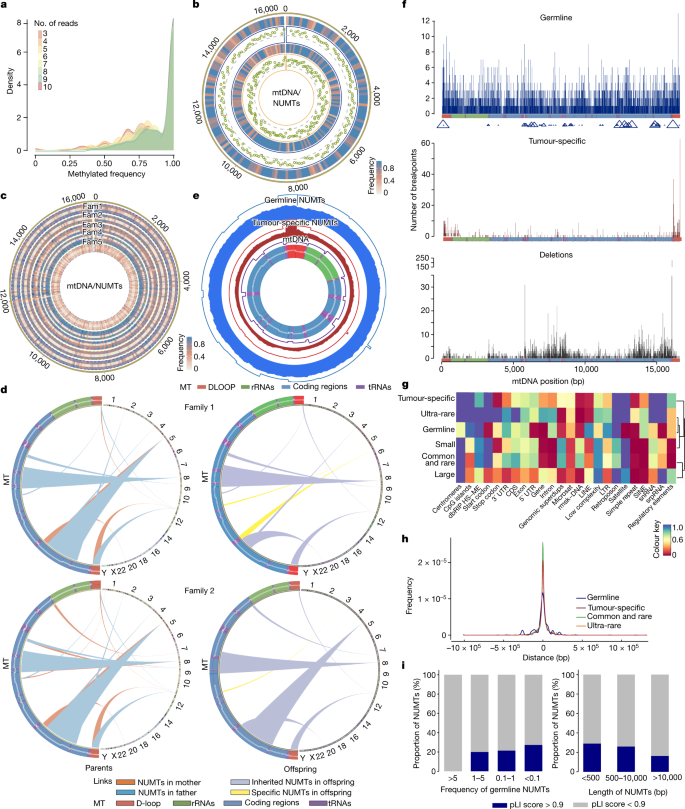
一些NUMTs表现出复杂的结构,确定通过检测分割读取映射只mtDNA紧随其后的是严格的QC过滤(图。gydF4y2Ba1gydF4y2Ba和扩展数据图。gydF4y2Ba3模拟gydF4y2Ba)。分析5885年mtDNA-mtDNA分裂读3197年发现三人小组表明,544年于560年继承了父亲和来自母亲。一百年和11个人共享相同的罕见mtDNA-mtDNA分裂读取在58件NUMTs (gydF4y2BaFgydF4y2Ba< 0.1%),作为连接NUMTs可能见过gydF4y2Ba5克ydF4y2Ba(扩展数据图。gydF4y2Ba3 a, bgydF4y2Ba)。牛津纳米孔测序读进行五个家庭(图。gydF4y2Ba1gydF4y2Ba和扩展数据图。gydF4y2Ba3 cgydF4y2Ba),验证连接NUMT结构。牛津纳米孔测序也使我们能够确定甲基化状态gydF4y2Ba20.gydF4y2Ba(图39个人的NUMTs检测。gydF4y2Ba3gydF4y2Ba)(方法)。检查NUMTs显示增加CpG甲基化相对于真实mtDNA读取,非甲基化gydF4y2Ba21gydF4y2Ba,包括paternally-transmitted连接mega-NUMTs,共享相同的甲基化模式在两代人(无花果。gydF4y2Ba3 b, cgydF4y2Ba和扩展数据图。gydF4y2Ba3 egydF4y2Ba),压制他们的核基因组中的表达gydF4y2Ba22gydF4y2Ba。连接NUMTs mtDNA可以模仿父亲的传播,产生了混合单体型类似mtDNA heteroplasmygydF4y2Ba5克ydF4y2Ba。这里我们展示他们增加检测混合等位基因的可能性类似heteroplasmy(与个人相比没有携带连接NUMTs;gydF4y2BaPgydF4y2Ba< 6.02×10gydF4y2Ba−8gydF4y2Ba等位基因分数(AF) > 2%,gydF4y2BaPgydF4y2Ba< 3.09×10gydF4y2Ba−15gydF4y2Ba对于房颤> 1%;Wilcoxon rank-sum测试)(扩展数据图。gydF4y2Ba3 fgydF4y2Ba)。gydF4y2Ba
一个gydF4y2Ba、甲基化频率的NUMTs 39人。颜色对应的长阅读数量不受测序深度的影响。gydF4y2BabgydF4y2Ba甲基化状态的连接从一双father-proband NUMT。从外面:(1)甲基化频率连接NUMT的父亲;(2)之间的甲基化频率比NUMT和non-methylated mtDNA序列的父亲;(3)甲基化频率连接NUMT渊源者;(4)之间的甲基化频率比NUMT和non-methylated mtDNA序列渊源者。绿点甲基化展示网站。此分析仅包括读取明确核的起源。颜色对应于甲基化频率。gydF4y2BacgydF4y2Ba五个家庭,甲基化概要(fam1-fam5)连接NUMTs(补充表gydF4y2Ba7gydF4y2Ba)。从外面:父亲,母亲,兄弟姐妹(当可用)和渊源者。个人窝藏连接NUMTs已经没有连接NUMTs甲基化水平高于个人。颜色对应于甲基化频率。gydF4y2BadgydF4y2Ba,三个新创NUMTs两三人小组。gydF4y2BaegydF4y2Ba的频率,从生殖系和肿瘤特异NUMTs mtDNA插入。从外面:(1)频率从生殖系NUMTs断点;(2)频率从生殖系NUMTs mtDNA碎片;(3)从肿瘤特异NUMTs频率的断点;(4)频率从肿瘤特异NUMTs mtDNA碎片;(5)频率mtDNA序列所期望的机会;(6)mtDNA地区。gydF4y2BafgydF4y2Ba的断点、分布与生殖系NUMTs线粒体基因,肿瘤特异NUMTs和线粒体删除(窗口大小= 100个基点)。三角形的大小表明NUMTs在每个窗口的频率。gydF4y2BaggydF4y2Ba,gydF4y2BaPgydF4y2Ba值富集分析基因组不同区域(补充无花果。gydF4y2Ba1克ydF4y2Ba- - - - - -gydF4y2Ba3 gydF4y2Ba和方法)。Microsat,微卫星;rmsk-DNA重复DNA;核内小RNA、小核RNA;srpRNA、信号识别颗粒RNA;superdups superduplications。gydF4y2BahgydF4y2Ba,从TSS NUMT位置的距离。gydF4y2Ba我gydF4y2Ba,NUMTs在基因的比例高和低pLI分数按NUMT频率分组(左)和按NUMT分组大小(右)。gydF4y2Ba
分析NUMT隔离在8201年完成mother-father-child三人小组发现,三个来自两个家庭的私人NUMTs未见的父母,表明新创生殖系NUMT 2.44×10的突变率gydF4y2Ba−4gydF4y2Ba每一代(95%置信区间为2.95×10gydF4y2Ba−5gydF4y2Ba8.81×10gydF4y2Ba−4gydF4y2Ba)(图。gydF4y2Ba3 dgydF4y2Ba和扩展数据图。gydF4y2Ba4gydF4y2Ba)。在每种情况下,更始NUMT序列不一致与其他网站在孩子的核基因组的组装,使它不太可能NUMTs起源于核内DNA。没有其他NUMTs发现每个孩子和他们的父母一样NUMT新创NUMT插入序列,即使映射敏感性增加下降的需求至少五不和谐的读两个不和谐的读。新创NUMTs也没有出现在参考基因组或发表NUMTs列表(补充表gydF4y2Ba2gydF4y2Ba)。新创NUMT频率很可能被低估了,因为短NUMTs源头的确定的困难,虽然我们不能绝对排除的可能性明显新创NUMTs起源于核基因组的其他部分,而不是一个新的mtDNA插入事件。gydF4y2Ba
NUMT插入的特点gydF4y2Ba
接下来,我们研究了mtDNA NUMTs和核DNA上下文,它被发现在所有核染色体(无花果。gydF4y2Ba二维gydF4y2Ba),涉及整个mtDNA(无花果。gydF4y2Ba1 b, cgydF4y2Ba)。3184年mtDNA断点被浓缩在非编码对应位移循环(D-loop) (gydF4y2BaPgydF4y2Ba= 0.001)——特别是在三个变异度高的部分地区(HV1,就gydF4y2BaPgydF4y2Ba= 0.002;HV2,gydF4y2BaPgydF4y2Ba= 0.001;HV3,gydF4y2BaPgydF4y2Ba= 0.006)——重链(OHR,gydF4y2BaPgydF4y2Ba= 0.002)和轻链(OLRgydF4y2BaPgydF4y2Ba= 0.016)的起源,较少涉及gydF4y2BaMT-ATP6gydF4y2Ba(gydF4y2BaPgydF4y2Ba= 0.001),gydF4y2BaMT-ND2gydF4y2Ba(gydF4y2BaPgydF4y2Ba= 0.015)和gydF4y2BaMT-ND3gydF4y2Ba(gydF4y2BaPgydF4y2Ba= 0.034)(图gydF4y2Ba3 egydF4y2Ba和扩展数据图。gydF4y2Ba5 a、bgydF4y2Ba)。这是由mtDNA片段的分布(gydF4y2BaPgydF4y2Ba优势比= 1.14 = 0.03,95%置信区间1.01 - -1.28,确切概率法)(无花果。gydF4y2Ba3 egydF4y2Ba)。之间存在弱相关的生殖系NUMT mtDNA断点和已知的位置在mtDNA删除断点,表现出边际意义(gydF4y2BaPgydF4y2Ba= 0.047,gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 0.24,皮尔逊相关性(图)。gydF4y2Ba3 fgydF4y2Ba和扩展数据图。gydF4y2Ba5度gydF4y2Ba)。总的来说,我们观察到每个染色体的长度之间有很强的正相关关系,NUMTs发现后对每个染色体的数量占其他基因组功能(gydF4y2BaPgydF4y2Ba= 1.42×10gydF4y2Ba−6gydF4y2Ba线性回归测试)。然而,染色体3、6和21有更多的比剩下的常染色体NUMTs / Mb(3号染色体gydF4y2BaPgydF4y2Ba= 0.03;6号染色体gydF4y2BaPgydF4y2Ba= 0.005;21号染色体,gydF4y2BaPgydF4y2Ba= 0.03,双尾排列测试)和X染色体数量减少的NUMTs / Mb (gydF4y2BaPgydF4y2Ba= 0.001)。二百二十八NUMTs X染色体上观察,预期的大约两个更多的女性比男性(151 28138,和75年的25426男性;Fisher精确检验gydF4y2BaPgydF4y2Ba= 1.713×10gydF4y2Ba−5gydF4y2Ba优势比= 1.824,95%置信区间1.374 - -2.441)。Y染色体不是分析由于复杂的重复结构限制自信对齐。gydF4y2Ba
先前的报道当地的序列特征与NUMT插入有关gydF4y2Ba23gydF4y2Ba促使邻近的独特的综合分析NUMTs着丝粒,基因组重复,简单的重复,dbRIP HS-ME(逆转录转座子插入多态性,人类移动元素),监管元素,CpG岛,卫星和反转位子活动(包括长点缀元素(行)和短点缀元素(正弦))。常见和罕见NUMTs (gydF4y2BaFgydF4y2Ba≥0.1%)更有可能接近或发生在基因组复制(gydF4y2BaPgydF4y2Ba= 0.030),件NUMTs浓缩在监管元素(gydF4y2BaPgydF4y2Ba= 0.011),sin (gydF4y2BaPgydF4y2Ba= 0.003),简单的重复(gydF4y2BaPgydF4y2Ba= 0.006)和内含子gydF4y2BaPgydF4y2Ba= 0.003(图。gydF4y2Ba3 ggydF4y2Ba和补充无花果。gydF4y2Ba1克ydF4y2Ba- - - - - -gydF4y2Ba3 gydF4y2Ba)。没有在500个基点常见NUMTs地区转录起始点的侧翼(TSS),符合选择对NUMTs扰乱基因功能(无花果。gydF4y2Ba3 hgydF4y2Ba和扩展数据图。gydF4y2Ba5 dgydF4y2Ba)。一致,基因宽容的分数gydF4y2Ba24gydF4y2Ba(pLI)呈负相关的频率NUMTs人群中(图gydF4y2Ba3我gydF4y2Ba)。gydF4y2Ba
阿特拉斯的肿瘤特异NUMTsgydF4y2Ba
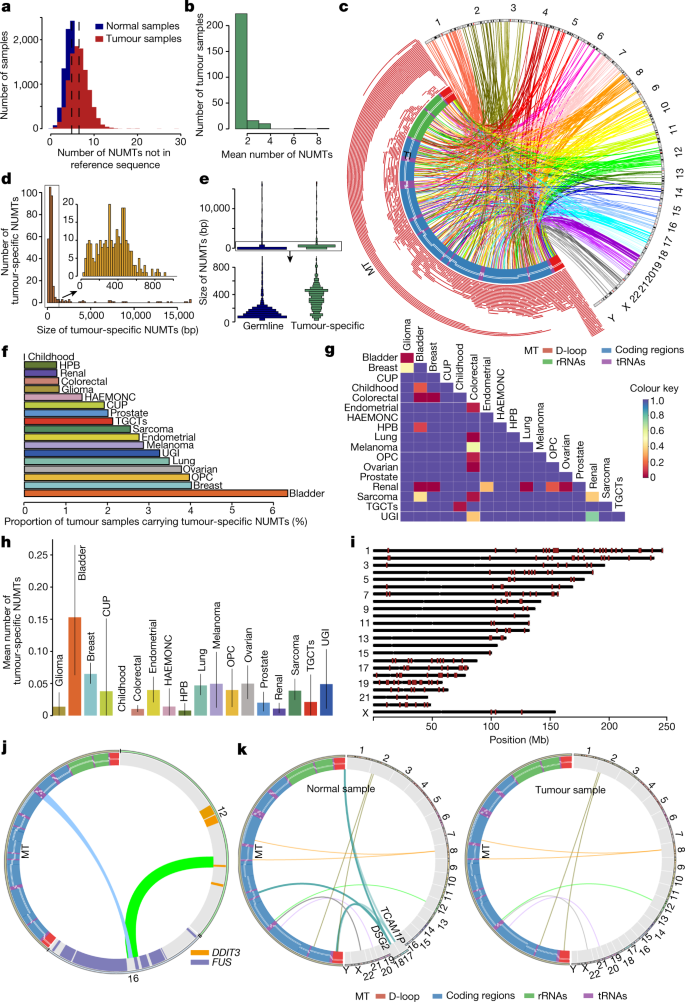
接下来,我们研究了26488名癌症WGS英格兰从基因组学项目。质量控制措施(方法)后,我们分析了12509配对WGS代表生殖系肿瘤和健康组织21癌症类型(扩展数据图。gydF4y2Ba6模拟gydF4y2Ba和补充表gydF4y2Ba3 gydF4y2Ba)。总的来说,肿瘤有较高意味着许多NUMTs(6.5±2.2(意味着南达科他州±。))不存在的参考序列比相应的正常组织(4.8±1.6;gydF4y2BaPgydF4y2Ba< 2.2×10gydF4y2Ba−16gydF4y2Ba,Wilcoxon rank-sum测试)(图gydF4y2Ba4gydF4y2Ba和补充图。gydF4y2Ba4gydF4y2Ba)。这种差异可能反映了肿瘤本身,而不是正常组织在每种情况下,因为NUMTs的平均数不同正常组织类型之间没有差别(平均检测唾液细胞NUMT是4.7,5在皮肤成纤维细胞和血液样本的4.9;唾液和血液,gydF4y2BaPgydF4y2Ba= 0.24,估计=−0.1;成纤维细胞与血液gydF4y2BaPgydF4y2Ba= 0.67,=−0.1估计,线性回归测试)(扩展数据图。gydF4y2Ba6 egydF4y2Ba)。癌症的频率生殖系NUMTs并不不同于生殖系NUMTs的频率以罕见的疾病项目参与者(gydF4y2BaPgydF4y2Ba= 0.924,线性回归测试占测序深度)(扩展数据图。gydF4y2Ba6 fgydF4y2Ba)。没有性别差异在NUMT分布(补充图。gydF4y2Ba5克ydF4y2Ba)。对于大多数肿瘤,没有个人的年龄之间的相关性在诊断和NUMTs(扩展数据图的数量。gydF4y2Ba6 bgydF4y2Ba和补充图。gydF4y2Ba6gydF4y2Ba)。然而,NUMTs更低的平均数量在血液学的从老年人恶性肿瘤,可能反映出他们的起源克隆造血作用gydF4y2Ba25gydF4y2Ba(gydF4y2BaPgydF4y2Ba= 3.29×10gydF4y2Ba−3gydF4y2Ba线性回归,估计=−0.007)。gydF4y2Ba
一个gydF4y2Ba,平均每个正常的NUMTs发现数和肿瘤样本中不存在参考序列。gydF4y2BabgydF4y2Ba,平均数量的肿瘤特异NUMTs中发现肿瘤。gydF4y2BacgydF4y2Ba在12509年发现肿瘤特异NUMTs normal-tumour对。离开,NUMT mtDNA大小和位置。mtDNA和核基因组之间的链接连接断点。gydF4y2BadgydF4y2Ba、大小分布的肿瘤特异NUMTs(红色)和肿瘤特异NUMTs小于1000个基点(橙色)。gydF4y2BaegydF4y2Ba,大小分布的生殖系肿瘤特异NUMTs(上)和生殖系肿瘤特异NUMTs小于1000个基点(底部)。gydF4y2BafgydF4y2Ba不同类型的肿瘤的比例,至少一个肿瘤特异NUMT。gydF4y2BaggydF4y2Ba,gydF4y2BaPgydF4y2Ba值成对比较肿瘤特异的平均数NUMTs不同肿瘤类型。gydF4y2BahgydF4y2Ba,平均每个肿瘤的肿瘤特异NUMTs类型。数据均值±s.e.m。神经胶质瘤,gydF4y2BangydF4y2Ba= 359;膀胱,gydF4y2BangydF4y2Ba= 268;乳腺癌、gydF4y2BangydF4y2Ba= 2038;杯,gydF4y2BangydF4y2Ba= 52个;童年,gydF4y2BangydF4y2Ba= 170;结直肠,gydF4y2BangydF4y2Ba= 1934;子宫内膜,gydF4y2BangydF4y2Ba= 579;HAEMONC,gydF4y2BangydF4y2Ba= 72;HPB,gydF4y2BangydF4y2Ba= 258;肺癌、gydF4y2BangydF4y2Ba= 1061;黑色素瘤,gydF4y2BangydF4y2Ba= 244;OPC,gydF4y2BangydF4y2Ba= 151;卵巢,gydF4y2BangydF4y2Ba= 423;前列腺,gydF4y2BangydF4y2Ba= 298;肾,gydF4y2BangydF4y2Ba= 1022;肉瘤,gydF4y2BangydF4y2Ba= 979;TGCTs,gydF4y2BangydF4y2Ba= 47;UGI,gydF4y2BangydF4y2Ba= 184。gydF4y2Ba我gydF4y2Ba、肿瘤特异NUMTs染色体位置显示为红酒吧。gydF4y2BajgydF4y2Ba,NUMTs参与gydF4y2BaFUS-DDIT3gydF4y2Ba嵌合融合。NUMTs蓝色链接和显示gydF4y2BaFUS-DDIT3gydF4y2Ba融合显示为绿色链接。染色体数目和线粒体基因组。gydF4y2BakgydF4y2Ba,在乳腺肿瘤样本中失去NUMTs的例子。链接代表NUMTs中发现肿瘤或正常(左)(右)样本。染色体数目和线粒体基因组。杯,未知的主癌;子宫内膜,子宫内膜癌;神经胶质瘤,成人神经胶质瘤;HAEMONC haemato-oncology;HPB hepato-pancreato-biliary癌症;黑色素瘤,恶性黑色素瘤;OPC、口腔口咽癌; TGCTs, testicular germ cell tumours; UGI, upper gastrointestinal cancer.
接下来,我们致力于肿瘤特异的一个子群NUMTs没有出现在其他非癌症基因组,提供高的信心,这些NUMTs出现在体细胞组织导致癌症,或在癌症本身。三百七十九这些新创NUMTs 251年肿瘤(2.3%)从10713年tumour-normal双,3.56×10的速度gydF4y2Ba−2gydF4y2Ba每癌症基因组(95%置信区间为3.38×10gydF4y2Ba−2gydF4y2Ba3.74×10gydF4y2Ba−2gydF4y2Ba)(图。gydF4y2Ba4 b, cgydF4y2Ba和补充表gydF4y2Ba4gydF4y2Ba;方法),这是高于生殖系率(gydF4y2BaPgydF4y2Ba= 2.08×10gydF4y2Ba−59gydF4y2Ba,确切概率法)和与先前的报道一致gydF4y2Ba15gydF4y2Ba,gydF4y2Ba26gydF4y2Ba。八十二年肿瘤进行多个新创NUMT,超过预期的机会(gydF4y2BaPgydF4y2Ba< 2.2×10gydF4y2Ba−16gydF4y2Ba,确切概率法)(图。gydF4y2Ba4 bgydF4y2Ba)。肿瘤特异NUMTs的平均数是0.035(其中。= 0.29),平均长度为396个基点(第一四分位数250个基点,第三四分位数524个基点,意味着= 1197个基点),这是高于生殖系NUMTs的数量(值= 156个基点,第一四分位数97个基点,第三四分位数382个基点)(gydF4y2BaPgydF4y2Ba< 2.2×10gydF4y2Ba−16gydF4y2Ba,Wilcoxon rank-sum测试)(图gydF4y2Ba4 d, egydF4y2Ba)。这些发现与癌症驾驶NUMT形成一致。肿瘤的比例与新创NUMT取决于肿瘤类型、肾和结直肠肿瘤有四倍NUMTs少于乳腺癌(gydF4y2BaPgydF4y2Ba= 1.93×10gydF4y2Ba−6gydF4y2Ba,确切概率法)和7.5倍少于膀胱癌(gydF4y2BaPgydF4y2Ba= 3.42×10gydF4y2Ba−4gydF4y2Ba,确切概率法)(图。gydF4y2Ba4 fgydF4y2Ba),它有更多的NUMTs比其他肿瘤类型(图。gydF4y2Ba4 g hgydF4y2Ba),如图所示gydF4y2Ba26gydF4y2Ba。肿瘤特异的平均数NUMTs并不与年龄(补充图。gydF4y2Ba6 bgydF4y2Ba),这意味着他们出现在致癌作用,而不是在一生中体细胞在癌症的形成。裁判做了对比。gydF4y2Ba26gydF4y2Ba补充表所示gydF4y2Ba5克ydF4y2Ba。gydF4y2Ba
新创NUMTs签名的癌症gydF4y2Ba
mtDNA段形成新创肿瘤NUMTs不同于生殖系(图。gydF4y2Ba3 egydF4y2Ba):他们不太可能涉及gydF4y2BaMT-CO3gydF4y2Ba(gydF4y2BaPgydF4y2Ba= 7.7×10gydF4y2Ba−3gydF4y2Ba),gydF4y2BaMT-ND4gydF4y2Ba(gydF4y2BaPgydF4y2Ba= 3.1×10gydF4y2Ba−3gydF4y2Ba),gydF4y2BaMT-ND4LgydF4y2Ba(gydF4y2BaPgydF4y2Ba= 3.4×10gydF4y2Ba−3gydF4y2Ba),gydF4y2BaMT-ND5gydF4y2Ba(gydF4y2BaPgydF4y2Ba= 5.3×10gydF4y2Ba−3gydF4y2Ba),但超过2.5倍更有可能涉及D-loop (gydF4y2BaPgydF4y2Ba= 3.36×10gydF4y2Ba−36gydF4y2Ba),主要是因为一个大约四倍代表的断点termination-associated序列2 (TAS2) (gydF4y2BaPgydF4y2Ba= 1.03×10gydF4y2Ba−7gydF4y2Ba,确切概率法)(扩展数据图。gydF4y2Ba5 a、bgydF4y2Ba),也反映在mtDNA片段(D-loop,gydF4y2BaPgydF4y2Ba= 5.51×10gydF4y2Ba−30gydF4y2Ba优势比= 2.00,95%置信区间1.77 - -2.25,确切概率法)(无花果。gydF4y2Ba3 egydF4y2Ba和gydF4y2Ba4摄氏度gydF4y2Ba)。这可以解释观察到的相关性新创NUMT断点和mtDNA删除断点(gydF4y2BaPgydF4y2Ba= 0.004,gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 0.44,皮尔逊相关性(图)。gydF4y2Ba3 fgydF4y2Ba和扩展数据图。gydF4y2Ba5度gydF4y2Ba),也倾向于集群在D-loop 3′末端gydF4y2Ba27gydF4y2Ba。肿瘤特异NUMTs更常见的19号染色体上(gydF4y2BaPgydF4y2Ba= 9.08×10gydF4y2Ba−6gydF4y2Ba和不太常见的6号染色体上gydF4y2BaPgydF4y2Ba= 1.53×10gydF4y2Ba−3gydF4y2Ba)(图。gydF4y2Ba4我gydF4y2Ba)和更有可能包含重复元素(gydF4y2BaPgydF4y2Ba= 4.24×10gydF4y2Ba−16gydF4y2Ba),特别是卫星重复(gydF4y2BaPgydF4y2Ba= 0.023)和微卫星重复(gydF4y2BaPgydF4y2Ba=比生殖系NUMTs(图0.007)。gydF4y2Ba3 ggydF4y2Ba和补充图。gydF4y2Ba1克ydF4y2Ba)。最后,一个大比例的肿瘤特异NUMTs被发现在500个基点,2000个基点和5000个基点的TSS比生殖系NUMTs(无花果。gydF4y2Ba3 hgydF4y2Ba和扩展数据图。gydF4y2Ba5 dgydF4y2Ba)。结合在一起,这些发现表明,当地的序列特征,基因组不稳定和更少的机会选择删除特定NUMTs由于放松的进化约束解释了为什么NUMT景观不同于生殖系。gydF4y2Ba
NUMT插入的不良后果gydF4y2Ba
九百四十六人(58%)生殖系NUMTs观察基因区域,绝大多数(85.8%,gydF4y2BangydF4y2Ba= 812)丰富的内含子与外显子(gydF4y2BaPgydF4y2Ba= 0.01,排列测试)(无花果。gydF4y2Ba3 ggydF4y2Ba和补充无花果。gydF4y2Ba1克ydF4y2Ba- - - - - -gydF4y2Ba3 gydF4y2Ba)。不常见或罕见NUMTs (gydF4y2BaFgydF4y2Ba> 0.1%)被发现在编码DNA序列(CDS) (gydF4y2BaPgydF4y2Ba= 0.039排列测试),并没有将导致罕见的疾病(gydF4y2Ba方法gydF4y2Ba和gydF4y2Ba补充信息gydF4y2Ba“结果”),符合NUMTs受到进化的约束。二百二十年肿瘤特异NUMTs被发现在基因区域,包括cd、13 3影响停止密码子,4影响密码子开始,16日在3′,5′非翻译区(utr)。怀有八个肿瘤肿瘤特异NUMTs插入基因在宇宙癌症基因普查名单上gydF4y2Ba28gydF4y2Ba(两个gydF4y2BaFHIT基因gydF4y2Ba,这是一个脆弱的基因组gydF4y2Ba29日gydF4y2Ba,分别gydF4y2BaCTNNA2gydF4y2Ba,gydF4y2BaDDIT3gydF4y2Ba,gydF4y2BaWIF1gydF4y2Ba,gydF4y2BaBCL11BgydF4y2Ba,gydF4y2BaKDM5AgydF4y2Ba和gydF4y2BaAKT2gydF4y2Ba)(补充表gydF4y2Ba4gydF4y2Ba)。一个肿瘤NUMT插入的基因内区gydF4y2BaFANCIgydF4y2Ba参与DNA修复。复杂的重组NUMT插入染色体易位的现场也看到在三(图8个肿瘤样本。gydF4y2Ba4 jgydF4y2Ba和扩展数据图。gydF4y2Ba7一个gydF4y2Ba)。一个带有黏液样脂肪肉瘤肿瘤gydF4y2Ba付家gydF4y2Ba- - - - - -gydF4y2BaDDIT3gydF4y2Ba嵌合融合复杂重排引起的癌蛋白涉及NUMT插入(图。gydF4y2Ba4 jgydF4y2Ba和扩展数据图。gydF4y2Ba7一个gydF4y2Ba)。FUS-DDIT3融合中存在90%的黏液样脂肪肉瘤gydF4y2Ba30.gydF4y2Ba在致癌作用,暗示NUMT个人在我们的研究中。三个私人NUMTs non-tumour组织没有发现匹配的乳房肿瘤,可能影响预后的损失gydF4y2BaDSG2gydF4y2Ba31日gydF4y2Ba和gydF4y2BaTCAM1PgydF4y2Ba32gydF4y2Ba(无花果。gydF4y2Ba4 kgydF4y2Ba和扩展数据图。gydF4y2Ba7 bgydF4y2Ba)。两个正常组织从患有血癌的NUMTs极高的数字没有出现在肿瘤组织(扩展数据图。gydF4y2Ba7 cgydF4y2Ba),可能反映出克隆增殖。gydF4y2Ba
NUMT插入和修改gydF4y2Ba
NUMT断点更有可能涉及nCC / CCn三核苷酸mtDNA基因组和不太可能涉及nTT / TTn核基因组和mtDNA(无花果。gydF4y2Ba5gydF4y2Ba,扩展数据图。gydF4y2Ba8gydF4y2Ba和补充表gydF4y2Ba6gydF4y2Ba)。分析扩展到2 bp, 3 bp和4 bp mtDNA断点显示poly-C大片以外的8日12和18倍比预期更多的机会(gydF4y2BaPgydF4y2Ba= 7.57×10gydF4y2Ba−10gydF4y2Ba,gydF4y2BaPgydF4y2Ba= 2.13×10gydF4y2Ba−5gydF4y2Ba和gydF4y2BaPgydF4y2Ba= 6.3×10gydF4y2Ba−5gydF4y2Ba),暗示microhomology NUMT插入通过重组事件。我们还观察到重叠序列microhomology (≥1 bp)在51.9%的NUMT断点(gydF4y2BaPgydF4y2Ba= 2.05×10gydF4y2Ba−45gydF4y2Ba,确切概率法),符合microhomology-mediated结束加入(MMEJ)在一些NUMT形成;钝端修复NUMT断点的27.6%和20.5%的NUMT short-nucleotide插入断点,暗示异源端加入gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba(无花果。gydF4y2Ba5 b, cgydF4y2Ba)。更大比例的肿瘤特异NUMTs(64.1%)比生殖系重叠序列microhomology NUMTs (gydF4y2BaPgydF4y2Ba= 5.22×10gydF4y2Ba−10gydF4y2Ba,确切概率法)(图。gydF4y2Ba5度gydF4y2Ba)。gydF4y2Ba
一个gydF4y2Ba在核基因组中,三核苷酸频率NUMT断点(左)和mtDNA(右)(细节扩展数据图。gydF4y2Ba8gydF4y2Ba)。箭头指向nCC / CCn或nTT / TTn三核苷酸NUMTs大大丰富。*gydF4y2BaPgydF4y2Ba< 0.05,* *gydF4y2BaPgydF4y2Ba< 0.01,* * *gydF4y2BaPgydF4y2Ba< 0.001,* * * *gydF4y2BaPgydF4y2Ba< 0.0001。gydF4y2BabgydF4y2Ba期间,Microhomology-mediated加入NUMTs的形成。gydF4y2BacgydF4y2Ba,microhomology序列的比例、小核和mtDNA序列之间插入和钝端加入NUMT断点。gydF4y2BadgydF4y2Ba癌症,癌症签名浓缩为每个类型(heat map)和所有癌症类型(点)。圆点大小正比于样品的数量与每个签名在肿瘤特异NUMTs (Tts)和non-tumour-specific NUMTs (tnt)。gydF4y2BaegydF4y2Ba,NUMTs和PRDM9-binding站点之间的距离在生殖系肿瘤特异NUMTs。gydF4y2BafgydF4y2Ba,NUMTs肿瘤有无错义突变在人类DNA修复基因。gydF4y2BaggydF4y2Ba,两个例子相同的mtDNA片段检测到两个地点在核基因组中,展示的证据NUMT插入到一个位置,然后移动到另一个。gydF4y2BahgydF4y2Ba(左)一个mtDNA片段插入到染色体14至19岁之间的易位染色体14和19。NUMTs在染色体发现了14到19岁,这表明NUMTs插入易位发生前核基因组,然后搬到另一个位置易位。吧,一个mtDNA片段插入到12号染色体易位染色体12到21。NUMTs被认为12号染色体上,但不是在21号染色体,这表明NUMTs插入易位发生后的核基因组。gydF4y2Ba我gydF4y2Ba,两个样品的例子mito-chromothripsis观察在这个研究。圆环图显示的位置NUMTs核和mtDNA基因组,在核基因组结构变异。核基因组测序深度红线所示。地图显示染色体结构变异参与多个核基因组的染色体。从整合基因组学读对齐查看器扩展数据图所示。gydF4y2Ba9 c, dgydF4y2Ba。gydF4y2Ba
我们还观察到浓缩的癌症三核苷酸突变签名gydF4y2Ba35gydF4y2BaS2 (gydF4y2BaPgydF4y2Ba= 6.93×10gydF4y2Ba−7gydF4y2Ba),S3 (gydF4y2BaPgydF4y2Ba= 4.68×10gydF4y2Ba−13gydF4y2Ba)和向(gydF4y2BaPgydF4y2Ba= 1.72×10gydF4y2Ba−18gydF4y2Ba)在癌症肿瘤特异NUMTs(无花果。gydF4y2Ba5 dgydF4y2Ba)。NUMT插入像转座子跳之前与S2和向有关gydF4y2Ba35gydF4y2Ba和S3与失败的双链断裂(双边带)通过同源重组修复,NUMTs有一个角色gydF4y2Ba35gydF4y2Ba。签名2和13也丰富APOBEC-mediated点突变,这也能导致双边带gydF4y2Ba36gydF4y2Ba。因此似乎是常见的体细胞突变在癌症和NUMT背后的分子机制的形成。gydF4y2Ba
符合这一点,生殖系NUMTs更有可能被发现在3 kb PRDM9-binding网站(gydF4y2BaPgydF4y2Ba= 0.003,排列测试)和肿瘤特异NUMTs更有可能被发现在1 kb的PRDM9-binding网站(gydF4y2BaPgydF4y2Ba= 0.003,排列测试)(无花果。gydF4y2Ba5 egydF4y2Ba和扩展数据图。gydF4y2Ba8 cgydF4y2Ba)。PRDM9涉及双边带修复并确定交叉热点在减数分裂gydF4y2Ba37gydF4y2Ba,所以协同定位符合NUMTs双边带修复作用gydF4y2Ba33gydF4y2Ba,gydF4y2Ba34gydF4y2Ba。因此,不同的分子机制参与NUMT形成,所有这些都与核基因组不稳定。符合这一点,肿瘤样本在DNA修复错义突变致癌基因gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba更有可能比其余港口肿瘤特异NUMTs肿瘤(77.7%比63.1%,确切概率法gydF4y2BaPgydF4y2Ba= 5.05×10gydF4y2Ba−6gydF4y2Ba,95%置信区间1.44 - -2.68,比值比= 1.95)(图gydF4y2Ba5 fgydF4y2Ba和补充图。gydF4y2Ba7gydF4y2Ba)。gydF4y2Ba
有人建议,NUMTs移动在最初插入事件gydF4y2Ba40gydF4y2Ba这条其他传染性元素。我们发现几个例子支持这个假设(无花果。gydF4y2Ba5克gydF4y2Ba)。我们还发现NUMTs与大型删除,插入,拷贝数损益,尤其是复杂的结构性变异(扩展数据断点的无花果。gydF4y2Ba9 a、bgydF4y2Ba)。一些癌症包含广泛NUMT重组,插入到核基因组的证据之前电子易位(无花果。gydF4y2Ba5 hgydF4y2Ba)。我们还发现在这两个例子的多个片段mtDNA是跨多个染色体(图嵌入整个基因组。gydF4y2Ba5我gydF4y2Ba和扩展数据图。gydF4y2Ba9 c, dgydF4y2Ba)类似的极端chromothripsis重组gydF4y2Ba41gydF4y2Ba(mito-chromothripsis)。gydF4y2Ba
分子进化的NUMT序列gydF4y2Ba
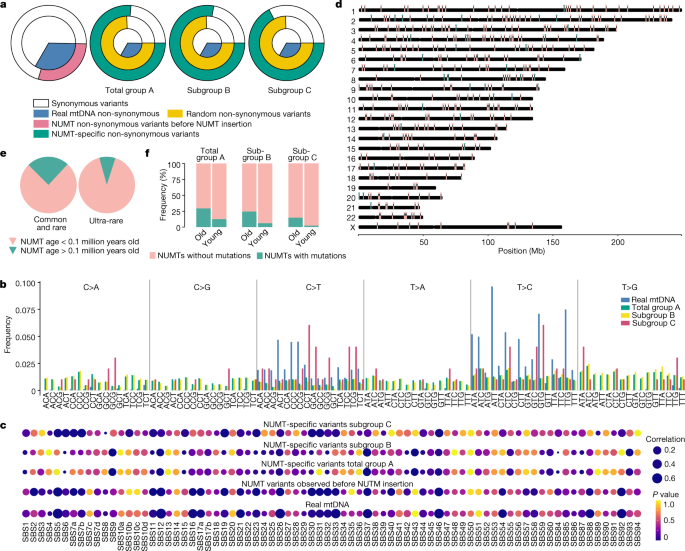
理解的分子进化mtDNA序列插入到核基因组后,我们确定完整的931个不同的核苷酸序列NUMTs包含144805个基点,当地完成组装NUMTs从短内容排序是可能的(gydF4y2Ba方法gydF4y2Ba)。这个分析的结果发表在无花果。gydF4y2Ba6gydF4y2Ba和gydF4y2Ba补充信息gydF4y2Ba“结果”。最后,我们估计429岁NUMT插入(gydF4y2Ba方法gydF4y2Ba)。绝大多数(90%以上)小于010万岁和41(9.5%)超过0.1 milion岁,与一系列375万年(无花果。gydF4y2Ba6 dgydF4y2Ba和补充表gydF4y2Ba1克ydF4y2Ba)。正如预期的那样,老NUMTs人群中(图中较为常见。gydF4y2Ba6 egydF4y2Ba),尤其是在非洲基因组(扩展数据图。gydF4y2Ba8 dgydF4y2Ba),更有可能携带比年轻NUMTs NUMT-specific突变(总A组:gydF4y2BaPgydF4y2Ba= 7.2×10gydF4y2Ba−3gydF4y2Ba优势比= 2.92,95%置信区间1.27 - -6.39;B组:gydF4y2BaPgydF4y2Ba= 3.9×10gydF4y2Ba−4gydF4y2Ba优势比= 2.92,95%置信区间1.27 - -6.39;子群C:gydF4y2BaPgydF4y2Ba= 9.0×10gydF4y2Ba−4gydF4y2Ba优势比= 8.06,95%置信区间2.18 - -28.27,确切概率法)(无花果。gydF4y2Ba6 fgydF4y2Ba)。在一起这些发现表明持续NUMT插入在人类进化和演化。gydF4y2Ba
讨论gydF4y2Ba
NUMTs之前被认为是古代的残余mtDNA易位事件通常是相关的物种之间共享gydF4y2Ba42gydF4y2Ba。在这里,我们表明,NUMT形成是一个持续的过程,与新创生殖系事件发生大约每10次gydF4y2Ba4gydF4y2Ba在每10出生和体细胞插入发生一次gydF4y2Ba3 gydF4y2Ba癌症。这将导致高NUMT多样性在人口中,有14.2%的人携带一件NUMT发现在小于1 1000人。批发mtDNA片段转移到细胞核基因组将不可避免地增加人类基因组的大小gydF4y2Ba3 gydF4y2Ba。然而,逆相关性NUMT大小和人群中发生的频率指向一个选择性的过程平衡NUMT插入、维持基因组大小和删除NUMTs影响基因的表达。协同定位的NUMTs PRDM9-binding网站会促进他们清除生殖系因为PRDM9决定在减数分裂重组热点的网站gydF4y2Ba37gydF4y2Ba。这样,NUMTs可以作为“临时修复”类似膏药,修复双边带直到他们在减数分裂过程中被除去。NUMTs的高负担和分布在癌症可能反映了一种高度基因组不稳定状态没有选择在短的时间内。gydF4y2Ba
尽管NUMTs可以涉及整个mtDNA分子,非编码D-loop NUMT断点更常见,包括重型和轻型链复制的起源。这就提出了一个可能性,mtDNA删除参与NUMT形成。然而,更令人信服的解释涉及mtDNA转录和复制有关,它起源于D-loopgydF4y2Ba43gydF4y2Ba。最近的描述线粒体形成疝和BSX-BAK大孔隙提供一种途径gydF4y2Ba44gydF4y2Ba后,可能涉及RNA中间体泄漏到细胞质mtDNA双边带gydF4y2Ba45gydF4y2Ba。这也可以发生在单个细胞,导致躯体镶嵌性。gydF4y2Ba
细胞器基因的易位到细胞核中有关键作用建立真核细胞线粒体和之间的共生关系。在这里,我们表明,DNA转移的机制保持活跃和修改生殖系大约每4000年出生。因此可以想见,一个endosymboisis始于大约14.5亿年前还没有完成。gydF4y2Ba
方法gydF4y2Ba
研究样本gydF4y2Ba
我们研究了68348从遗传基因组DNA在英格兰罕见疾病基因组学项目和26488年从基因组学英国癌症癌症基因组项目。基于基因组DNA提取和加工英格兰样品处理指南(gydF4y2Bahttps://legacy.genomicsengland.co.uk/about -基因组学- england/the - 100000 -基因组project/information - - gmc staff/sample guidance/——处理gydF4y2Ba)。DNA样本接收FluidX管(布鲁克斯)和增加到实验室管理信息系统(LIMS)英国Biocentre。自动化图书馆准备后,图书馆是量化使用自动定量PCR,集群和测序。图书馆是准备使用Illumina公司TruSeq DNA PCR-Free高通量样品制备设备或Illumina公司TruSeq纳米高通量样品制备设备gydF4y2Ba46gydF4y2Ba。gydF4y2Ba
伦理批准gydF4y2Ba
伦理批准提供了英格兰剑桥的东部南部国家研究伦理委员会参考13号/ EE / 0325下,参与者提供书面知情同意批准的研究。所有的参与者在100000年罕见的疾病的基因工程为通过在国民医疗服务制度(NHS) 13个中心涵盖所有在英国NHS病人。gydF4y2Ba
质量控制检查罕见疾病的基因组gydF4y2Ba
所有的样品都通过了最初的QC检查基于测序质量和覆盖从基因组学测序提供者(Illumina公司)和英格兰内部质量控制检查(gydF4y2Bahttps://research-help.genomicsengland.co.uk/display/GERE/Sample +质量控制gydF4y2Ba)。我们只包括样品一致gydF4y2Ba智人gydF4y2BaNCBI GRCh38组装与诱饵(gydF4y2BaNgydF4y2Ba= 58335)。所有样本测序产生至少85 Gb的数据序列测序的至少30质量。比对了至少95%的基因组15 x或以上已有读取(映射质量> 10)后丢弃重复。此外,所有包括样品通过了一套基本的质量控制指标:(1)样本污染(VerifyBamID freemixgydF4y2Ba47gydF4y2Ba)< 0.03,(2)单核苷酸变异的比率(SNV) Heterozygous-to-Homozygous (Het-to-Hom)调用< 3,(3)SNVs总数3.2米- 4.7米之间,(4)数组索引> 90%,(5)中间片段大小> 250个基点,(6)超过嵌合读< 5%,(7)映射器读取比例> 60%,和(8)在辍学的比例< 10%。57961个基因组通过WGS qc。我们进一步排除下面的样品与线粒体基因组的平均深度500 x后重塑了线粒体读取(参见下面的细节)。罕见疾病的基因组研究中,我们包括53574人,25436名男性和28138名女性,年龄从0到99年(扩展数据图。gydF4y2Ba1 a, bgydF4y2Ba)。南达科他州WGS的平均深度是42 x (x = 7.7)和mtDNA的平均深度是1990 x(其中= 866 x)(扩展数据图。gydF4y2Ba1 cgydF4y2Ba)。gydF4y2Ba
家庭质量控制检查gydF4y2Ba
家庭的相关分析,WGS家庭选择质量检查处理对于罕见疾病的基因组,报告性染色体异常和报道与基因性总结检查(计算从家庭亲缘,孟德尔不一致,和性染色体检查)。性别决定,X和Y染色体的覆盖率数据与样本的平均覆盖率常染色体使用叮铃声v1.90gydF4y2Ba48gydF4y2Ba(gydF4y2Bawww.cog-genomics.org/plink/1.9/gydF4y2Ba)。生成的输出与参与者相比性提供了在样本收集。亲缘检查是基于验证孟德尔的三/家人之间的矛盾。个人VCF文件合并成一个家庭VCF BCFTools (v1.3.1)gydF4y2Ba49gydF4y2Ba并与叮铃声孟德尔矛盾再次检查。的关系也被计算所有成对基因identity-by-descent值关系在家庭使用叮铃声和报告关系(与预期值gydF4y2Bahttps://research-help.genomicsengland.co.uk/gydF4y2Ba)。我们进一步处理一个独立的亲缘检查使用我们以前公布的方法gydF4y2Ba50gydF4y2Ba。总之,32665年常染色体snp的列表选择估计亲缘。通过过滤合并VCF和1000基因组参考集gydF4y2Ba51gydF4y2Ba与选定的单核苷酸多态性,pc-relate函数从创世纪包应用于获得成对亲缘gydF4y2Ba52gydF4y2Ba。前20主成分用于人口结构,重量和参考集用于增加遗传多样性占了主成分分析。最后,我们包括8201个家庭之间的羁绊是一致的两个独立的预测方法和临床记录。gydF4y2Ba
质量控制检查癌症的基因组gydF4y2Ba
我们最初从基因组学英国癌症研究了26488名癌症基因组项目。样本准备使用一个Illumina公司TruSeq DNA纳米,TruSeq DNA PCR-Free或FFPE图书馆准备装备,然后测序HiSeq X生成150个基点paired-end读取。生殖系样本测序产生至少85 Gb的序列测序的至少30质量。对肿瘤样本需要至少212.5 Gb。比对的生殖系样本覆盖至少95%的基因组15 x或以上已有读取(映射质量> 10)后丢弃重复(gydF4y2Bahttps://research-help.genomicsengland.co.uk/gydF4y2Ba)。gydF4y2Ba
样本交叉污染的检查,生殖系与VerifyBamID样品处理gydF4y2Ba47gydF4y2Ba算法,通过状态分配不到3%的样品污染。肿瘤样本处理ConPair算法gydF4y2Ba53gydF4y2Ba与通过状态指示所述污染低于1%gydF4y2Bahttps://research-help.genomicsengland.co.uk/display/GERE/10。+ +阅读+和+文档?预览癌症% 2520分析% = / 38047056/45023724 / 2520技术% 2520信息% 2520的文件% 2520 v1-11 % 2520 - 10. - furtherreadinganddocumentation technicaldocumentation main.pdf # idgydF4y2Ba。gydF4y2Ba
上述质量控制措施后,12509个tumour-normal组织对12509年肿瘤样本和11913年与正常组织(生殖系)样本11909人。样本准备用5种不同的方法(FF, FFPF CD128排序细胞,EDTA和送气音)和三种不同的图书馆类型(PCR, PCR-FFPE和PCR-free)。我们执行额外的质量控制通过比较NUMTs被检测到的平均数量的样品由不同的方法和库类型。我们观察到的平均数量NUMTs不同群体之间显著不同(补充图。gydF4y2Ba8gydF4y2Ba)。为了避免可能的偏见造成的样品制备和库类型,我们只包括10713年tumour-normal样本对准备使用FF和图书馆类型PCR-free从9648个人在21癌症类型(扩展数据图。gydF4y2Ba6gydF4y2Ba)。肿瘤样本的平均深度WGS 117 x(其中10.1 x)和生殖系的平均WGS深度是43 x(其中。9.3 x)(补充图。gydF4y2Ba8 bgydF4y2Ba)。肿瘤样本的平均mtDNA深度27119 x(其中13642 x)和生殖系的平均mtDNA深度是3549 x(其中2452 x)(补充图。gydF4y2Ba8 cgydF4y2Ba)。gydF4y2Ba
从核基因组测序数据推测祖先gydF4y2Ba
广泛使用种族遗传祖先估计从1000人基因工程三期(1 kgp3)gydF4y2Ba51gydF4y2Ba真理,通过生成电脑1 kgp3样品和所有参与者情绪投射到这些。我们包括五大super-populations:非洲(误判率),混在美国(AMR)、东亚(EAS)、南亚(SAS)和欧洲(欧元)。简要步骤如下:(1)所有样本选自1 kgp3无关的,(2)188382年我们选择高质量的单核苷酸多态性在我们的数据集,(3)我们进一步过滤加器> 0.05 1 kgp3(以及我们的数据),(4)我们计算第一个使用GCTA 20个主要组件gydF4y2Ba54gydF4y2Ba,(5)我们预计个人数据到1 kgp3主成分载荷,(6)我们训练随机森林模型来预测血统的基础上(我)前8 1 kgp3主成分,(2)设置Ntrees = 400,(3)训练和预测1 kgp3 AMR,误判率、东亚峰会、欧元和SAS super-populations。可以找到的全部细节gydF4y2Bahttps://research-help.genomicsengland.co.uk/display/GERE/Ancestry +推理gydF4y2Ba。遗传祖先也预测和检查使用我们以前公布的方法gydF4y2Ba50gydF4y2Ba。5个人没有分配给任何super-populations被贴上“其他”。我们预测1280年的误判率,170 AMR 342东亚峰会,5758 SAS 42202欧元和3363其他在这项研究中(图。gydF4y2Ba2gydF4y2Ba)。在癌症生殖系基因组,我们包括312误判率,17 AMR, 71东亚峰会,338 SAS 8348欧元和314其他(扩展数据图。gydF4y2Ba6 c, dgydF4y2Ba)。gydF4y2Ba
我们执行统一的歧管近似和投影(UMAP)gydF4y2Ba55gydF4y2Ba基于独特的NUMTs每个人口在罕见疾病的基因组。UMAP分析用UMAP包默认参数R和可视化使用M3C包gydF4y2Ba56gydF4y2Ba在R。gydF4y2Ba
提取线粒体DNA序列和检测变体gydF4y2Ba
的子集序列读取对齐的线粒体基因组提取从每个使用Samtools WGS BAM文件gydF4y2Ba57gydF4y2Ba。我们跑MToolBox (v1.0)gydF4y2Ba58gydF4y2Ba由此产生的较小的BAM文件生成重塑mtDNA BAM文件。重塑BAM文件被用来调用变体。我们也使用第二个调用者VarScan2变体gydF4y2Ba59gydF4y2Ba调用mtDNA变异从重塑BAM文件(————min-var-freq 0.001, strand-filter 1——min-reads2 1——min-avg-qual 30)。mpileup文件用于VarScan2 Samtools生成的选项- d 30 0 - q - q 30。等位基因分数从VarScan2提取。我们只保留了单核苷酸多态性(snp)超过2读取每个微小等位基因链。变异减少低区域内(66 - 71,300 - 316,513 - 525,3106 - 3107,12418 - 12425和16182 - 16194年)被排除在外。gydF4y2Ba
使用HaploGrep2线粒体DNA haplogroup执行任务gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61年gydF4y2Ba。gydF4y2Ba
检测NUMTs和断点没有出现在参考序列gydF4y2Ba
检测NUMTs,我们使用之前发表和验证方法gydF4y2Ba5克ydF4y2Ba,gydF4y2Ba15gydF4y2Ba。从对齐WGS BAM文件使用samblaster我们提取的不和谐的阅读对gydF4y2Ba62年gydF4y2Ba和包括阅读对一端对齐,核基因组和另一端对齐mtDNA参考序列。读取映射质量等于零是丢弃。不和谐的读取被聚集在一起基于共享同一方向和他们是否在500个基点的距离。我们发现支持的集群至少两对不和谐的读取,并过滤掉不到5支持的集群对不和谐的读入主要分析。在1000个基点的距离NUMTs核DNA和mtDNA NUMT分组是相同的。我们生成的两套NUMTs基于NUMTs支持至少两对不和谐的读取和至少5对不和谐的读取(补充表gydF4y2Ba1克ydF4y2Ba)。我们观察到的弱相关的平均数量NUMTs和WGS深度(gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 0.134,gydF4y2BaPgydF4y2Ba< 2.2×10gydF4y2Ba−16gydF4y2Ba)和线粒体基因组深度(gydF4y2BaRgydF4y2Ba2gydF4y2Ba= 0.092,gydF4y2BaPgydF4y2Ba< 2.2×10gydF4y2Ba−16gydF4y2Ba)(补充无花果。gydF4y2Ba9 a、bgydF4y2Ba)表明,尽管一些NUMTs可能错过由于低深度,他们不太可能对我们产生影响的结论。没有发现差异的数量检测读取NUMTs的频率,建议检测NUMTs没有偏见的测序质量(补充图。gydF4y2Ba9 cgydF4y2Ba)。gydF4y2Ba
确定假定的断点跨越核DNA和mtDNA-derived序列(nuclear-mtDNA断点),我们寻找分裂读取1000个基点的距离内的不和谐的读取然后用咩咩的重塑gydF4y2Ba63年gydF4y2Ba。我们进一步分析了重塑读取,读取映射到核DNA的一端,另一端相同的阅读映射到mtDNA-derived序列。我们定义断点的至少三个读同一个NUMT分裂。每个NUMT应该有一个核断点和两个线粒体断点,除了NUMTs与其他核基因组结构发生变化。200个基点的断点侧翼地区核基因组注释使用gencode v29gydF4y2Ba64年gydF4y2Ba,gnomAD公益诉讼的分数gydF4y2Ba65年gydF4y2Ba和数据集的列表从UCSC的下载gydF4y2Ba66年gydF4y2Ba出版物(参见下面的细节)。当NUMTs参与多种基因,我们保持最高的基因公益诉讼的分数。线粒体基因组上的断点是使用MitoMap注释gydF4y2Ba67年gydF4y2Ba。gydF4y2Ba
探测连接NUMTsgydF4y2Ba
检测的连接NUMTs,首先我们寻找mtDNA-derived序列上的断点跨越两个地点(mtDNA-mtDNA断点)。我们提取分割读取,只有mtDNA序列对齐。这些分裂读取进一步重塑使用咩咩的叫声。我们分析了读取相同的两端读mtDNA序列映射到两个地点。然后过滤断点如下:(1)每个断点至少有3分裂读取中观察到至少一个个体,每个断点(2)至少有2分读中观察到同一个人,(3)我们排除了分裂读取映射到附近的开始和结束mtDNA基因组(D-loop区域的开始和结束),(4)我们排除了两个连接不到50个基点(他们可能mtDNA删除)。注意我们的方法有其局限性我们无法单独mtDNA-mtDNA断点NUMTs内真的mtDNA如果断点位于D-loop区域的开始和结束。因此,我们分析可能错过了连接NUMTs mtDNA-mtDNA断点D-loop区域的开始和结束。然而,我们的目的是检测自信连接NUMTs和显示连接NUMTs存在于人类。应用严格的过滤(上图)后,我们发现8686年从8450年的151个不同的mtDNA-mtDNA断点断点个人(扩展数据图。gydF4y2Ba3 dgydF4y2Ba)。279的8686个断点(140不同的断点)从148人件(频率< 0.1%)。一个断点(12867 - 14977)是特别常见的频率(38.4%),这也是常见的一个独立的数据集在我们先前的研究gydF4y2Ba5克ydF4y2Ba。确认从核基因组mtDNA-mtDNA断点,我们进行了两个独立的分析:(1)我们比较了mtDNA-mtDNA断点中观察到后代和他们的两个父母。如果mtDNA-mtDNA断点出现在后代和他们的父亲,而不是他们的母亲,我们定义father-transmitted mtDNA-mtDNA断点。如果mtDNA-mtDNA断点出现在孩子和他们的母亲,而不是他们的父亲,我们定义mother-transmitted mtDNA-mtDNA断点。注意我们没有能够识别传播模式如果mtDNA-mtDNA断点出现在所有三名家庭成员使用短内容测序技术。(2)罕见和件mtDNA-mtDNA断点(gydF4y2BaFgydF4y2Ba< 1%),我们检查个人携带相同的是否也进行同样的NUMT mtDNA-mtDNA断点。gydF4y2Ba
比较已知NUMTsgydF4y2Ba
已知NUMTs从UCSC的下载和以前的出版物gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba。BedtoolsgydF4y2Ba49gydF4y2Ba被用来寻找已知的NUMTs在我们的数据集。使用一个保守的方法,我们定义了NUMTs众所周知提供已知NUMTs在1000 bp NUMT侧翼(上游500 bp +下游500 bp)检测到核基因组在这项研究中,无论mtDNA插入片段的序列。gydF4y2Ba
富集分析gydF4y2Ba
浓缩的核和mtDNA基因组分析,我们研究了1637种不同的信心NUMTs至少5不和谐的读取使用2-tailed排列测试。基因组重复,简单的重复,dbRIP_HS-MEgydF4y2Ba90年gydF4y2Ba、监管元素,CpG岛、卫星、反转位子活动(包括线路和正弦)和TSS从UCSC的下载gydF4y2Ba66年gydF4y2Ba(gydF4y2Bahttps://genome.ucsc.edu/gydF4y2Ba)。使用这些信息来计算每个数据集的频率在200个基点NUMT侧翼(上游100 bp +下游100个基点)。经验gydF4y2BaPgydF4y2Ba值的计算是通过重采样随机位置匹配观察NUMTs 1000套。对于每个核基因组染色体上的浓缩,我们排除了Y染色体由于复杂的重复结构的Y染色体序列限制自信对齐。gydF4y2Ba
调查不同的染色体和NUMTs之间的关系,我们在R(应用线性回归gydF4y2Bahttp://CRAN.R-project.org/gydF4y2Ba)gydF4y2Ba68年gydF4y2Ba。gydF4y2Ba
Nnumt哪里NUMTs数量在每一个染色体,发现Lchr是染色体的长度,Pcentro, Pcpg,多段线,Pltr, Pretroposon, Psine, Pmicrosat, Prmsk, Prepeats, Pdups和怀孕的日志吗gydF4y2Ba2gydF4y2Ba转换比例的着丝粒,CpG岛、线、公升,逆转录子,正弦,微卫星重复,简单的重复,每个染色体基因组复制和监管元素。gydF4y2Ba
比较NUMTs和线粒体DNA缺失gydF4y2Ba
研究之间的关系NUMT插入和线粒体删除,我们将NUMT断点的频率与线粒体DNA的频率删除断点。1312 mtDNA删除列表从mitoBreak下载数据库gydF4y2Ba69年gydF4y2Ba。我们计算断点的频率在不同mtDNA regions-D-loop, 13个编码基因,相比2 rna结合22图示,断点的分布与生殖系肿瘤特异NUMTs使用线性回归。gydF4y2Ba
寻找新创NUMTs罕见的疾病三人小组和肿瘤特异NUMTs癌症基因组gydF4y2Ba
我们使用最保守的方法来定义的新创NUMTs father-mother-offspring三人小组。我们只包括与至少五双NUMTs不和谐的读取后代并没有不和谐的阅读中发现的父母。gydF4y2Ba
我们申请同样的方法来定义肿瘤特异NUMTs癌症基因组。肿瘤特异NUMTs被定义为至少5对不和谐的读取肿瘤样本并没有不和谐的读取与正常样本。失去NUMTs癌症基因组被定义为至少5对不和谐的读取正常样本和不超过一个的不和谐的读取与肿瘤样本。gydF4y2Ba
估计的速度新创NUMTs三人小组和肿瘤特异NUMTs癌症基因组gydF4y2Ba
新创NUMT插入率在三人小组和癌症基因组估计如下:gydF4y2Ba
在哪里gydF4y2BaρgydF4y2Ba(生殖系)是新创NUMT插入的速度在三人小组,gydF4y2BaρgydF4y2Ba(肿瘤)的肿瘤特异NUMT插入肿瘤样本,NumtTtrio是新创NUMT事件的数量在三人小组,NumtTumour是肿瘤特异的数量NUMTs, Ntrio总三人小组的数量,Ngenome总normal-tumour双的数量。gydF4y2Ba
分析肿瘤特异NUMTs和癌症的关联类型gydF4y2Ba
理解之间的关系供体年龄、性别和NUMTs的平均数量,我们应用线性回归到每个数据集使用R (gydF4y2Bahttp://CRAN.R-project.org/gydF4y2Ba)。gydF4y2Ba
模型1 <−lm (gydF4y2BaNgydF4y2Ba∼gydF4y2Ba年龄+性别+ DPmt)gydF4y2Ba
模型2 <−lm (NsomagydF4y2Ba∼gydF4y2Ba年龄+性别+ DPmt)gydF4y2Ba
在哪里gydF4y2BaNgydF4y2Ba和Nsoma平均数字NUMTs和肿瘤特异NUMTs年龄供体年龄、性别是捐赠性和DPmt平均线粒体DNA测序深度。gydF4y2Ba
检测癌症SNVs indels和结构变异gydF4y2Ba
读一致性对人类基因组参考GRCh38-Decoy +执行EBV与以撒(iSAAC-03.16.02.19版)gydF4y2Ba70年gydF4y2BaSNVs和短insertions-deletions (indels)变体叫一起肿瘤−正常执行减法使用Strelka(2.4.7版)gydF4y2Ba71年gydF4y2Ba。Strelka过滤掉以下生殖系变异电话:(1)所有调用与样本深度三倍染色体的意思是,(2)基因型与近端indel调用冲突,(3)轨迹读证据显示不平衡逐步模式,(4)基因型变异的电话来电者不与染色体倍性一致,(5)的比例basecalls过滤掉网站> 0.4,(6)轨迹质量分数< 14为杂合的或纯合子的SNP,(7)轨迹质量分数< 6为杂合的,纯合子或het-alt indels,(8)轨迹质量分数< 30其它小变体类型或质量分数不计算。Strelka过滤掉以下体细胞变异电话:(1)所有调用与正常样本深度三倍染色体的意思是,(2)所有调用该网站在正常样本不是一个纯合子的参考,(3)体细胞SNV与经验的电话安装VQSR得分< 2.75(调整质量分数表达phr比例概率的躯体被称为假阳性观察),(4)体细胞indels一部分basecalls过滤掉在一个窗口延长50基地的两侧indel设位置> 0.3,(5)体细胞indels与质量分数< 30(体细胞变异的联合概率,homo ref正常基因型),(6)所有调用重叠线重复区域。gydF4y2Ba
结构变异(sv)和长indel(> 50个基点)调用执行了蝠鲼(0.28.0版)gydF4y2Ba72年gydF4y2Ba结合配对和SV split-read证据发现和得分。拷贝数变异(CNVs)被称为画布(1.3.1版本)gydF4y2Ba73年gydF4y2Ba员工保险和次要的等位基因频率分配拷贝数。这些工具过滤掉以下变体电话:(1)Manta-called sv与正常样本深度附近的一个或两个变体假期结束三倍染色体的意思是,(2)Manta-called sv与体质量分数< 30岁(3)Manta-called体细胞缺失和重复长度> 10 kb, (4) Manta-called体细胞小变体(< 1 kb)分数的读取与周围MAPQ0 break-end > 0.4, (5) Canvas-called体细胞基因拷贝数异变与长度< 10 kb, (6) Canvas-called体细胞基因拷贝数异变与质量分数< 10。生物信息学可以找到管道的全部细节gydF4y2Bahttps://research-help.genomicsengland.co.uk/pages/viewpage.action?pageId=38046624gydF4y2Ba。gydF4y2Ba
寻找NUMT插入机制的证据gydF4y2Ba
PRDM9gydF4y2Ba
PRDM9决定减数分裂重组热点的位置在减数分裂DNA双边带形成。调查NUMT插入的机制,我们比较了NUMTs组170198发表PRDM9-binding山峰跨基因组gydF4y2Ba74年gydF4y2Ba。我们清点的数量NUMTs重叠PRDM9-binding高峰和排列进行分析(见“浓缩分析”的细节)。接下来,我们计算每个NUMT的断点之间的距离(从生殖系和肿瘤特异NUMTs)最近的PRDM9-binding网站。gydF4y2Ba
人类DNA修复基因gydF4y2Ba
一组已知的人类DNA修复基因从人类DNA修复基因下载网站(gydF4y2Bahttps://www.mdanderson.org/documents/Labs/Wood-Laboratory/human-dna-repair-genes.htmlgydF4y2Ba)gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba。我们提取的DNA修复基因的体细胞错义突变所有癌症样本,并对比样品携带突变基因和肿瘤特异NUMTs之间的关系。gydF4y2Ba
体细胞突变签名gydF4y2Ba
体细胞突变签名多个突变过程的结果,人类的身体是一生。每个不同的流程生成一个独一无二的结合被称为突变的突变类型签名(gydF4y2Bahttps://cancer.sanger.ac.uk/signatures/signatures_v2/gydF4y2Ba)。突变的签名是计算使用R包nnls (gydF4y2Bahttps://CRAN.R-project.org/package=nnlsgydF4y2Ba)。签名是如何计算出来的细节描述Alexandrov et al ., 2013gydF4y2Ba75年gydF4y2Ba和在线文档gydF4y2Bahttps://research-help.genomicsengland.co.uk/pages/viewpage.action?pageId=38046624gydF4y2Ba。gydF4y2Ba
评估临床意义gydF4y2Ba
罕见疾病的参与者没有已知的基因诊断gydF4y2Ba
基因组学英格兰PanelApp (gydF4y2Bahttps://panelapp.genomicsengland.co.uk/gydF4y2Ba)gydF4y2Ba76年gydF4y2Ba基因和基因组的实体列表被用来提供一个潜在的疾病基因列表(gydF4y2BaNgydF4y2Ba= 5883)。NUMTs被确定的频率< 1%,断点200 bp的侧翼区域内这些基因之一。结果做了注释与gencode v29,包括基因、基因内区,外显子,cd,起始密码子,终止密码子,5 ' UTR和3′UTR区域gydF4y2Ba64年gydF4y2Ba。NUMTs标注为下降的一个外显子进行了详细的分析。对于每一个基因,我们考虑证据的强度与疾病相关的基因,遗传模式的障碍,据报道类型的致病性变异和疾病的机制(例如,haploinsufficiency,增加功能或重复扩张),使用来自人类的信息(gydF4y2Bahttps://omim.org/gydF4y2Ba)gydF4y2Ba77年gydF4y2Ba并通过搜索PubMed (gydF4y2Bahttps://pubmed.ncbi.nlm.nih.gov/gydF4y2Ba)。建立疾病基因,我们认为每个渊源者可用的临床信息,包括它们的人类表型本体术语gydF4y2Ba91年gydF4y2Ba在入学时,家族史和年龄。我们假定罕见NUMT只出现在一个等位基因,除非是出现在父母双方或有记录的血缘关系(父母的数据不可用)。隐性障碍基因包含NUMT,我们研究是否出现在父母一方或双方(如果可用),是否存在血缘关系的家族病史,在序列数据是否有第二个罕见的变异。NUMT插入的位置是探索UCSC基因组浏览器gydF4y2Ba66年gydF4y2Ba。gydF4y2Ba
参与者罕见疾病的基因诊断gydF4y2Ba
参与者与基因组医学的发现证实了基因诊断中心退出问卷(gydF4y2Bahttps://research-help.genomicsengland.co.uk/pages/viewpage.action?pageId=38046767gydF4y2Ba)。基因致病变种的坐标比较基因组坐标的使用bedtools NUMTsgydF4y2Ba49gydF4y2Ba。gydF4y2Ba
罕见疾病NUMTs参与者与线粒体DNA维护障碍gydF4y2Ba
与线粒体DNA维护障碍的参与者gydF4y2Ba78年gydF4y2Ba从基因组医学鉴定中心退出问卷和我们之前分析的参与者与疑似线粒体疾病gydF4y2Ba79年gydF4y2Ba。我们还发现影响家庭成员基因组测序的数据可用。122 NUMTs被发现从20个人。只有4 NUMTs不同NUMTs(2)外显子的两个家庭。我们比较的平均数量在这些参与者NUMTs罕见疾病的其他参与者。gydF4y2Ba
癌症基因组gydF4y2Ba
确定NUMT插入是司机突变在癌症的发展,与200个碱基对NUMTs侧翼地区被确定定位感兴趣的基因。我们感兴趣的基因被定义为那些在宇宙(体细胞突变在癌症的目录)癌症基因普查名单(一级和二级)包括已知含有突变会涉及癌症的基因gydF4y2Ba28gydF4y2Ba。我们也使用已知的人类DNA修复基因的列表gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba。NUMT插入的位置相对于这些基因列表中探索UCSC基因组浏览器。gydF4y2Ba
验证NUMTs使用读测序gydF4y2Ba
在短内容测序验证NUMT检测,我们进行了全基因组测序在牛津纳米孔PromethION 39个人罕见疾病的基因组。DNA测序最大化收益,4μg生殖系从100年kgp参与者是分散到15 - 30 Kb Covaris g-tubes (4000 rpm, 1分钟,1 - 3传递,直到所需的长度达到了),然后耗尽的低分子量DNA与短(< 10 Kb)读取器套件(Circulomics ss - 100 - 101 - 01)所描述的制造商。在检查DNA安捷伦毫微微脉冲系统的大小分布,与牛津纳米孔测序库生成SQK-LSK109装备,从1.2µg高分子weight-enriched DNA。样本量化与一个量子位荧光计(表达载体,Q33226)和500 ng加载到PromethION R.9.4.1流动细胞遵循制造商的指示。在实验吞吐量快速增长是有限的不可用毛孔,图书馆是持久化,核酸酶后冲洗~ 20小时后初始运行。与Guppy-3.2.6/3.2.8 Base-calling进行高精度模式。可以发现协议的全部细节gydF4y2Bahttps://research-help.genomicsengland.co.uk/display/GERE/Genomic +数据+ +永久吗? = / 38046759/38047942 / v1_protocol_ONT_LSK109.pdf预览gydF4y2Ba。测序读GRCh38使用minimap2对齐gydF4y2Ba80年gydF4y2Ba2.17版。质量控制统计和土地使用Nanoplot生成gydF4y2Ba81年gydF4y2Ba1.26.0版本。生物信息学可以找到管道的全部细节gydF4y2Bahttps://research-help.genomicsengland.co.uk/display/GERE/Genomic +数据+ +永久吗? = / 38046759/38047944 /预览PromethION % 20 sv调用% 20管道% 20 grch38.docx % 20gydF4y2Ba。然后提取长读对齐使用短内容相同的地区检测到NUMT测序来自同一个人。提取的长阅读使用咩咩的叫声被重塑。观察到NUMTs也对整合基因组学手动检查浏览器(进口)gydF4y2Ba82年gydF4y2Ba。182 184 NUMTs(29的31不同NUMTs)检测到使用短内容排序也看到读测序数据。同一个人失踪的两个NUMTs读测序可能由于低读序列对齐的读取次数。gydF4y2Ba
NUMTs使用读测序检测甲基化状态gydF4y2Ba
Whole-genome-wide甲基化检测进行了使用call-methylation函数从Nanopolish v0.13.3gydF4y2Ba83年gydF4y2Ba在39人。甲基化检测的输出包括CG二核苷酸的位置参考基因组,读的ID用于打电话,。和对数似然比我们提取了长的读取映射到mtDNA基因组,并进一步分组分成两组:(1)长读也映射到核基因组,(2)长只读取映射到mtDNA基因组。接下来,我们使用calculate_methylation_frequency甲基化频率计算每个站点。py脚本包的每一个阅读小组。甲基化调用检测到第一组从NUMTs和调用从真实mtDNA检测到第二组。我们使用真mtDNA的甲基化剖面作为参考,和NUMTs甲基化估计日志gydF4y2Ba2gydF4y2Ba甲基化频率比NUMTs和真实之间的每个站点mtDNA来自同一个人。注意,如果个人进行连接NUMTs,调用检测到第二组从真实mtDNA和连接NUMTs混合。我们不能单独的漫长的读取映射到中间的连接NUMTs读取也只有映射到mtDNA基因组和真正mtDNA基因组。gydF4y2Ba
在这个分析中,我们专注于连接NUMTs和大型NUMTs长读NUMTs自信地对齐。我们只包括与至少3读取调用映射到NUMTs和至少10读取映射到真正mtDNA序列。我们也使用4读5读取6读、7:8读9读和10读否决检测NUMTs甲基化。我们观察到相同的甲基化分布频率不同的碎屑(无花果。gydF4y2Ba3gydF4y2Ba),说明read-thresholds并不影响我们的结果。gydF4y2Ba
检测在NUMT插入突变gydF4y2Ba
我们执行一个新创的所有335891 NUMTs发现在这项研究中。过程的步骤是:(1)我们从每个NUMT集群不和谐的读取检测到同一个人。(2)NUMT的共识序列重叠群使用CAP3生成gydF4y2Ba84年gydF4y2Ba。(3)叠连群然后对齐对线粒体基因组参考gydF4y2Ba85年gydF4y2Ba使用粗嘎的声音gydF4y2Ba63年gydF4y2Ba和ClustalωgydF4y2Ba86年gydF4y2Ba。(4)Clustalω的一致序列被用来检测NUMT之间的核苷酸的变化序列,并使用BioPython线粒体基因组参考序列gydF4y2Ba87年gydF4y2Ba。确保自信的电话,我们应用额外的过滤如下:(1)我们只包括NUMTs短于1000个基点;(2)我们排除了NUMT断点的变体在5英国石油(bp);(3)我们移除对齐参考等位基因的变异不同于mtDNA参考基因组在同一位置;(4)我们只包括单一核变化;(5)我们排除了个人携带更多的变异比总人口(>的意思是南达科他州变异数+ 3×)。gydF4y2Ba
定义NUMT-specific变异,我们应用额外的过滤:(1)我们排除了变异超过50%个人携带相同的常见或罕见NUMTs和75%个人携带同一件NUMTs。这种严格的过滤策略旨在提供最大的信心,任何NUMT-specific变体是极有可能发生NUMT序列插入核基因组后,影响的敏感性分析。(2)我们排除了变异仅发现1个人测序错误的可能性降到最低;(3)获取最自信NUMT-specific突变,我们只包括变异检测到至少两个人来自同一个家庭。在主文本,我们报道3组NUMT-specific变体。后A组,应用步骤(1);子群B,在步骤(2);(3)和子群C,后一步。gydF4y2Ba
估计NUMTs的年龄gydF4y2Ba
NUMTs的年龄估计使用前面描述的方法gydF4y2Ba19gydF4y2Ba。我们从人类线粒体序列对齐,从每个NUMT黑猩猩和共识序列重叠群使用Clustalω。黑猩猩的祖先线粒体序列是从运用下载(Pan_tro_3.0)。对齐序列被用来生成使用BioPython核苷酸的变化。我们计算的网站数量的比例匹配人类等位基因总数的网站在人类和祖先的线粒体序列在每个NUMT地区有所不同。比率是用来获得一个为每个NUMT大致年龄,相对于估计human-chimpanzee分歧时间为600万年。确保自信的结果,我们应用过滤如下:(1)我们只包括NUMTs长度为50到1000个基点;(2)我们排除了NUMTs没有不同的等位基因在人类和黑猩猩之间;(3)的年龄估计超过50%的人携带相同的NUMT和至少2个人。应用这种过滤后,我们排除了所有的私人NUMTs只出现在一个个体。 (4) We excluded concatenated NUMTs.
统计分析和策划gydF4y2Ba
所有统计分析在这项研究中提出了文本和使用R执行gydF4y2Ba68年gydF4y2Ba(gydF4y2Bahttp://CRAN.R-project.org/gydF4y2Ba)和Python (gydF4y2Bahttp://www.python.orggydF4y2Ba)。数据生成使用R和Matplotlib (gydF4y2Bahttps://matplotlib.orggydF4y2Ba在Python中)。圆环图是使用圆环(gydF4y2Bahttp://circos.ca/gydF4y2Ba)gydF4y2Ba88年gydF4y2Ba。使用chromoMap染色体地图了gydF4y2Ba89年gydF4y2Ba。gydF4y2Ba
一个web接口来存款NUMTs发现在这项研究中开发使用闪亮的v1.7.1 (gydF4y2Bahttps://CRAN.R-project.org/package=shinygydF4y2Ba)(gydF4y2Bahttps://cran.r-project.org/web/packages/shiny/index.htmlgydF4y2Ba)gydF4y2Ba92年gydF4y2Ba。gydF4y2Ba
Web资源gydF4y2Ba
NUMTs公开在本研究发现通过一个web界面gydF4y2Bahttps://wwei.shinyapps.io/numts/gydF4y2Ba。gydF4y2Ba
报告总结gydF4y2Ba
进一步研究信息设计是可用的gydF4y2Ba自然研究报告摘要gydF4y2Ba与这篇文章有关。gydF4y2Ba
数据可用性gydF4y2Ba
WGS参与者参加100000人基因工程的数据可以通过访问过程概述了后基因组学英国有限公司:gydF4y2Bahttps://www.genomicsengland.co.uk/about-gecip/joining-research-community/gydF4y2Ba。总之,申请人注册机构可以申请加入一个英格兰的基因组学临床解释关系,然后注册一个项目使访问英格兰基因组学研究环境2 h后完成在线培训。gydF4y2Ba智人gydF4y2Ba可以发现在NCBI GRCh38组装gydF4y2Bahttps://www.ncbi.nlm.nih.gov/assembly/gydF4y2Ba。Gencode v29可以找到gydF4y2Bahttps://www.gencodegenes.org/human/release_29.htmlgydF4y2Ba。可以发现在人类基因组注释文件gydF4y2Bahttps://hgdownload.soe.ucsc.edu/goldenPath/hg38/database/gydF4y2Ba。黑猩猩的祖先线粒体序列可以找到gydF4y2Bahttps://www.ensembl.org/Pan_troglodytes/Info/IndexgydF4y2Ba。gydF4y2Ba
代码的可用性gydF4y2Ba
可以在代码中使用的研究gydF4y2Bahttps://github.com/WeiWei060512/NUMTs-detection.gitgydF4y2Ba和gydF4y2Bahttps://doi.org/10.5281/zenodo.6966017gydF4y2Ba。gydF4y2Ba
引用gydF4y2Ba
罗杰,a·J。,Munoz-Gomez, S. A. & Kamikawa, R. The origin and diversification of mitochondria.咕咕叫。医学杂志。gydF4y2Ba27gydF4y2BaR1177-R1192 (2017)。gydF4y2Ba
灰色,m . W。,Burger, G. & Lang, B. F. Mitochondrial evolution.科学gydF4y2Ba283年gydF4y2Ba,1476 - 1481 (1999)。gydF4y2Ba
Hazkani-Covo E。,Zeller, R. M. & Martin, W. Molecular poltergeists: mitochondrial DNA copies (numts) in sequenced nuclear genomes.公共科学图书馆麝猫。gydF4y2Ba6gydF4y2Bae1000834 (2010)。gydF4y2Ba
洛佩兹,j . V。Yuhki, N。,Masuda, R., Modi, W. & O'Brien, S. J. Numt, a recent transfer and tandem amplification of mitochondrial DNA to the nuclear genome of the domestic cat.j·摩尔。另一个星球。gydF4y2Ba39gydF4y2Ba,174 - 190 (1994)。gydF4y2Ba
魏,w . et al . Nuclear-mitochondrial DNA片段像父亲一般地在人类遗传的线粒体DNA。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba1740 (2020)。gydF4y2Ba
Willett-Brozick, j·E。Savul, s。,R我chey, L. E. & Baysal, B. E. Germ line insertion of mtDNA at the breakpoint junction of a reciprocal constitutional translocation.嗡嗡声。麝猫。gydF4y2Ba109年gydF4y2Ba,216 - 223 (2001)。gydF4y2Ba
Borensztajn, k . et al .表征两个小说剪切位点突变在人类第七因子基因造成严重的等离子第七因子缺乏和出血素质。gydF4y2BaBr。j . Haematol。gydF4y2Ba117年gydF4y2Ba,168 - 171 (2002)。gydF4y2Ba
特纳,c . et al .人类遗传疾病引起的新创mitochondrial-nuclear DNA转移。gydF4y2Ba嗡嗡声。麝猫。gydF4y2Ba112年gydF4y2Ba,303 - 309 (2003)。gydF4y2Ba
艾哈迈德,z . m . et al . Nonsyndromic隐性耳聋DFNB18和亚瑟综合征类型IC是USHIC的等位基因突变。gydF4y2Ba嗡嗡声。麝猫。gydF4y2Ba110年gydF4y2Ba,527 - 531 (2002)。gydF4y2Ba
Pagnamenta, a . T。魏,W。,R一个hman, S. & Chinnery, P. F. Biparental inheritance of mitochondrial DNA revisited.Nat,启麝猫。gydF4y2Ba22gydF4y2Ba,477 - 478 (2021)。gydF4y2Ba
罗,s . et al .双亲遗传的线粒体DNA。gydF4y2BaProc。《科学。美国gydF4y2Ba115年gydF4y2Ba,13039 - 13044 (2018)。gydF4y2Ba
莫德,h . et al . NUMT混杂偏见线粒体heteroplasmy调用的引用等位基因。gydF4y2Ba前面。细胞。Dev,杂志。gydF4y2Ba7gydF4y2Ba201 (2019)。gydF4y2Ba
萨拉斯。,Schonherr, S., Bandelt, H. J., Gomez-Carballa, A. & Weissensteiner, H. Extraordinary claims require extraordinary evidence in asserted mtDNA biparental inheritance.法医科学。Int,麝猫。gydF4y2Ba47gydF4y2Ba102274 (2020)。gydF4y2Ba
斯梅德利,d . et al . 100000基因组飞行员健康Care-preliminary罕见的疾病诊断报告。gydF4y2Ba心血管病。j .地中海。gydF4y2Ba385年gydF4y2Ba,1868 - 1880 (2021)。gydF4y2Ba
居,y s . et al。频繁的体细胞的线粒体DNA转移到人类癌症细胞的核基因组。gydF4y2Ba基因组Res。gydF4y2Ba25gydF4y2Ba,814 - 824 (2015)。gydF4y2Ba
西蒙,D。,花茎甘蓝,f M。朗,M。,Gasparre, G. & Attimonelli, M. The reference human nuclear mitochondrial sequences compilation validated and implemented on the UCSC genome browser.BMC基因组学gydF4y2Ba12gydF4y2Ba517 (2011)。gydF4y2Ba
花茎甘蓝,f M。,西蒙,D。&Attimonelli, M. Primates and mouse NumtS in the UCSC Genome Browser.BMC Bioinf。gydF4y2Ba13gydF4y2BaS15 (2012)。gydF4y2Ba
李米。,Schroeder, R., Ko, A. & Stoneking, M. Fidelity of capture-enrichment for mtDNA genome sequencing: influence of NUMTs.核酸Res。gydF4y2Ba40gydF4y2Bae137 (2012)。gydF4y2Ba
Dayama G。,Emery, S. B., Kidd, J. M. & Mills, R. E. The genomic landscape of polymorphic human nuclear mitochondrial insertions.核酸Res。gydF4y2Ba42gydF4y2Ba,12640 - 12649 (2014)。gydF4y2Ba
Gigante, et al。使用读测序检测印DNA甲基化。gydF4y2Ba核酸Res。gydF4y2Ba47gydF4y2Bae46 (2019)。gydF4y2Ba
Bicci,我。,Calabrese, C., Golder, Z. J., Gomez-Duran, A. & Chinnery, P. F. Single-molecule mitochondrial DNA sequencing shows no evidence of CpG methylation in human cells and tissues.核酸Res。gydF4y2Ba49gydF4y2Ba,12757 - 12768 (2021)。gydF4y2Ba
黄,c . Y。,Grunheit, N., Ahmadinejad, N., Timmis, J. N. & Martin, W. Mutational decay and age of chloroplast and mitochondrial genomes transferred recently to angiosperm nuclear chromosomes.植物杂志。gydF4y2Ba138年gydF4y2Ba,1723 - 1733 (2005)。gydF4y2Ba
居,y s . et al .起源和功能体线粒体DNA突变在人类癌症的后果。gydF4y2BaeLifegydF4y2Ba3 gydF4y2Bae02935 (2014)。gydF4y2Ba
列克,m . et al . 60706年分析蛋白质编码基因变异人类。gydF4y2Ba自然gydF4y2Ba536年gydF4y2Ba,285 - 291 (2016)。gydF4y2Ba
热那亚、g . et al .克隆造血和血癌风险推断从血液的DNA序列。gydF4y2Ba心血管病。j .地中海。gydF4y2Ba371年gydF4y2Ba,2477 - 2487 (2014)。gydF4y2Ba
元,y . et al。全面的线粒体基因组的分子特性在人类癌症。gydF4y2BaNat,麝猫。gydF4y2Ba52gydF4y2Ba,342 - 352 (2020)。gydF4y2Ba
塞缪尔,d . C。,Schon, E. A. & Chinnery, P. F. Two direct repeats cause most human mtDNA deletions.趋势麝猫。gydF4y2Ba20.gydF4y2Ba,393 - 398 (2004)。gydF4y2Ba
Sondka, z . et al。宇宙癌症基因普查:描述在所有人类癌症基因功能障碍。gydF4y2BaNat。启癌症gydF4y2Ba18gydF4y2Ba,696 - 705 (2018)。gydF4y2Ba
松山,a . et al .脆弱的网站直接同源FHIT基因/ FRA3B和FHIT基因/ Fra14A2:进化保守但高度recombinogenic。gydF4y2BaProc。《科学。美国gydF4y2BaOne hundred.gydF4y2Ba,14988 - 14993 (2003)。gydF4y2Ba
Goransson m . et al,黏液样脂肪肉瘤FUS-DDIT3融合癌蛋白放松与NFKBIZ NF-κB目标基因的交互。gydF4y2Ba致癌基因gydF4y2Ba28gydF4y2Ba,270 - 278 (2009)。gydF4y2Ba
秦,s . et al . DSG2表达式与不良预后相关,促进早期宫颈癌。gydF4y2Ba癌细胞Int。gydF4y2Ba20.gydF4y2Ba206 (2020)。gydF4y2Ba
饶,x et al . MicroRNA-221/222乳腺癌fulvestrant阻力通过调节多个信号通路。gydF4y2Ba致癌基因gydF4y2Ba30.gydF4y2Ba,1082 - 1097 (2011)。gydF4y2Ba
布兰查德,j·l·&施密特,g . w .酵母和人类线粒体DNA迁移事件:由一个共同的end-joining机制集成和替代观点核苷酸替换模式。gydF4y2Ba摩尔。杂志。另一个星球。gydF4y2Ba13gydF4y2Ba893 (1996)。gydF4y2Ba
里凯蒂、M。,Tekaia, F. & Dujon, B. Continued colonization of the human genome by mitochondrial DNA.公共科学图书馆杂志。gydF4y2Ba2gydF4y2BaE273 (2004)。gydF4y2Ba
Alexandrov l . b . et al .突变签名在人类癌症的曲目。gydF4y2Ba自然gydF4y2Ba578年gydF4y2Ba,94 - 101 (2020)。gydF4y2Ba
Seplyarskiy v . b . et al . APOBEC-induced突变在人类癌症是强烈丰富的滞后在复制DNA链。gydF4y2Ba基因组Res。gydF4y2Ba26gydF4y2Ba,174 - 182 (2016)。gydF4y2Ba
迈尔斯,s . et al。在灵长类动物对热点主题涉及PRDM9基因在减数分裂重组。gydF4y2Ba科学gydF4y2Ba327年gydF4y2Ba,876 - 879 (2010)。gydF4y2Ba
木材、r D。,Mitchell, M. & Lindahl, T. Human DNA repair genes, 2005.Mutat。Res。gydF4y2Ba577年gydF4y2Ba,275 - 283 (2005)。gydF4y2Ba
木材、r D。,Mitchell, M., Sgouros, J. & Lindahl, T. Human DNA repair genes.科学gydF4y2Ba291年gydF4y2Ba,1284 - 1289 (2001)。gydF4y2Ba
Hazkani-Covo E。,Sorek, R. & Graur, D. Evolutionary dynamics of large numts in the human genome: rarity of independent insertions and abundance of post-insertion duplications.j·摩尔。另一个星球。gydF4y2Ba56gydF4y2Ba,169 - 174 (2003)。gydF4y2Ba
李,y . et al .宪法和体细胞的21号染色体重排急性淋巴细胞白血病。gydF4y2Ba自然gydF4y2Ba508年gydF4y2Ba,98 - 102 (2014)。gydF4y2Ba
Bensasson D。,Feldman, M. W. & Petrov, D. A. Rates of DNA duplication and mitochondrial DNA insertion in the human genome.j·摩尔。另一个星球。gydF4y2Ba57gydF4y2Ba,343 - 354 (2003)。gydF4y2Ba
Gustafsson, c . M。,F一个lkenberg, M. & Larsson, N. G. Maintenance and expression of mammalian mitochondrial DNA.为基础。学生物化学启。gydF4y2Ba85年gydF4y2Ba,133 - 160 (2016)。gydF4y2Ba
麦克阿瑟,k . et al . BAK /伯灵顿大孔隙促进线粒体形成疝和mtDNA射流在细胞凋亡。gydF4y2Ba科学gydF4y2Ba359年gydF4y2Baeaao6047 (2018)。gydF4y2Ba
Tigano, M。,Vargas, D. C., Tremblay-Belzile, S., Fu, Y. & Sfeir, A. Nuclear sensing of breaks in mitochondrial DNA enhances immune surveillance.自然gydF4y2Ba591年gydF4y2Ba,477 - 481 (2021)。gydF4y2Ba
·考尔菲德,m . et al .国家基因组研究和医疗知识库。figsharegydF4y2Bahttps://doi.org/10.6084/m9.figshare.4530893.v5gydF4y2Ba(2019)。gydF4y2Ba
小君,g . et al .检测和估计的污染人类DNA样本测序和基于数组的基因型数据。gydF4y2Ba点。j .的嗡嗡声。麝猫。gydF4y2Ba91年gydF4y2Ba,839 - 848 (2012)。gydF4y2Ba
Chang, c . c . et al .第二代叮铃声:上升到更大的挑战和更丰富的数据集。gydF4y2BaGigasciencegydF4y2Ba4gydF4y2Ba7 (2015)。gydF4y2Ba
昆兰,a。r . &大厅,i m . BEDTools:一套灵活的工具来比较基因组的特性。gydF4y2Ba生物信息学gydF4y2Ba26gydF4y2Ba,841 - 842 (2010)。gydF4y2Ba
魏,w . et al .生殖系选择形状人类线粒体DNA多样性。gydF4y2Ba科学gydF4y2Ba364年gydF4y2Baeaau6520 (2019)。gydF4y2Ba
1000人基因组计划财团。一个全球参考人类遗传变异。gydF4y2Ba自然gydF4y2Ba526年gydF4y2Ba,68 - 74 (2015)。gydF4y2Ba
Conomos, m . P。,Re我ner, A. P., Weir, B. S. & Thornton, T. A. Model-free estimation of recent genetic relatedness.点。j .的嗡嗡声。麝猫。gydF4y2Ba98年gydF4y2Ba,127 - 148 (2016)。gydF4y2Ba
伯格曼,大肠。陈,b . J。Arora, K。,Vacic, V. & Zody, M. C. Conpair: concordance and contamination estimator for matched tumor-normal pairs.生物信息学gydF4y2Ba32gydF4y2Ba,3196 - 3198 (2016)。gydF4y2Ba
杨,J。,Lee, S. H., Goddard, M. E. & Visscher, P. M. GCTA: a tool for genome-wide complex trait analysis.点。j .的嗡嗡声。麝猫。gydF4y2Ba88年gydF4y2Ba,76 - 82 (2011)。gydF4y2Ba
麦克因尼斯,L。,Healy, J. & Melville, J. UMAP: uniform manifold approximation and projection for dimension reduction. Preprint athttps://arxiv.org/abs/1802.03426gydF4y2Ba(2020)。gydF4y2Ba
约翰·c·r . et al . M3C:蒙特卡罗reference-based共识集群。gydF4y2Ba科学。代表。gydF4y2Ba10gydF4y2Ba1816 (2020)。gydF4y2Ba
李,h . et al .序列比对/地图格式和SAMtools。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba,2078 - 2079 (2009)。gydF4y2Ba
花茎甘蓝,c . et al . MToolBox:高度自动化管道heteroplasmy注释和优先级人类线粒体变异在高通量测序分析。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba,3115 - 3117 (2014)。gydF4y2Ba
Koboldt特区et al . VarScan 2:体细胞突变在癌症和拷贝数改变发现外显子组测序。gydF4y2Ba基因组Res。gydF4y2Ba22gydF4y2Ba,568 - 576 (2012)。gydF4y2Ba
范烤箱,m &凯塞,m .更新全球人类线粒体DNA变异的综合系统树。gydF4y2Ba嗡嗡声。Mutat。gydF4y2Ba30.gydF4y2BaE386-E394 (2009)。gydF4y2Ba
Weissensteiner, h . et al . HaploGrep 2:线粒体haplogroup分类在高通量测序的时代。gydF4y2Ba核酸Res。gydF4y2Ba44gydF4y2BaW58-W63 (2016)。gydF4y2Ba
浮士德,g . g . &大厅,i m . SAMBLASTER:快速阅读提取重复标记和结构变体。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba,2503 - 2505 (2014)。gydF4y2Ba
肯特,w . j . BLAT-the爆炸之类的校准工具。gydF4y2Ba基因组Res。gydF4y2Ba12gydF4y2Ba,656 - 664 (2002)。gydF4y2Ba
法兰克,a . et al . GENCODE参考人类和小鼠基因组的注释。gydF4y2Ba核酸Res。gydF4y2Ba47gydF4y2BaD766-D773 (2019)。gydF4y2Ba
Karczewski, k . j . et al . 141456年从变异突变约束谱量化人类。gydF4y2Ba自然gydF4y2Ba581年gydF4y2Ba,434 - 443 (2020)。gydF4y2Ba
肯特,w . j . et al。人类在UCSC基因组浏览器。gydF4y2Ba基因组Res。gydF4y2Ba12gydF4y2Ba,996 - 1006 (2002)。gydF4y2Ba
Kogelnik, a . M。洛特,m . T。,Brown, M. D., Navathe, S. B. & Wallace, D. C. MITOMAP: a human mitochondrial genome database.核酸Res。gydF4y2Ba24gydF4y2Ba,177 - 179 (1996)。gydF4y2Ba
R核心团队。的语言和环境统计计算(R统计计算的基础,2019)。gydF4y2Ba
花缎,J。,Carneiro, J., Amorim, A. & Pereira, F. MitoBreak: the mitochondrial DNA breakpoints database.核酸Res。gydF4y2Ba42gydF4y2BaD1261-D1268 (2014)。gydF4y2Ba
Raczy et al。艾萨克:超高速全基因组辅助分析Illumina公司测序平台。gydF4y2Ba生物信息学gydF4y2Ba29日gydF4y2Ba,2041 - 2043 (2013)。gydF4y2Ba
金姆,s . et al . Strelka2:快速、准确调用生殖系和体细胞变异。gydF4y2BaNat方法。gydF4y2Ba15gydF4y2Ba,591 - 594 (2018)。gydF4y2Ba
陈,x et al .外套:快速检测生殖系和癌症的结构变异和indels测序应用程序。gydF4y2Ba生物信息学gydF4y2Ba32gydF4y2Ba,1220 - 1222 (2016)。gydF4y2Ba
辊、E。,Ivakhno, S., Lee, S., Royce, T. & Tanner, S. Canvas: versatile and scalable detection of copy number variants.生物信息学gydF4y2Ba32gydF4y2Ba,2375 - 2377 (2016)。gydF4y2Ba
Altemose: et al。人类的地图PRDM9绑定提供了证据小说PRDM9和其他锌指蛋白在减数分裂的行为。gydF4y2BaeLifegydF4y2Ba6gydF4y2Bae28383 (2017)。gydF4y2Ba
Alexandrov, l . B。、Nik-Zainal年代。楔形,d . C。,Campbell, P. J. & Stratton, M. R. Deciphering signatures of mutational processes operative in human cancer.细胞的代表。gydF4y2Ba3 gydF4y2Ba,246 - 259 (2013)。gydF4y2Ba
Martin a . r . et al . PanelApp大佬专家知识建立共识诊断基因面板。gydF4y2BaNat,麝猫。gydF4y2Ba51gydF4y2Ba,1560 - 1565 (2019)。gydF4y2Ba
Hamosh,。,Scott, A. F., Amberger, J., Valle, D. & McKusick, V. A. Online Mendelian Inheritance in Man (OMIM).哼MutatgydF4y2Ba15gydF4y2Ba57 - 61 (2000)。gydF4y2Ba
亚当,m p等人gydF4y2BaGeneReviewsgydF4y2Ba(eds El-Hattab a . w . et al。)线粒体DNA维修缺陷概述。(华盛顿大学,1993)。gydF4y2Ba
舍恩k . r . et al .使用全基因组测序确定疑似线粒体疾病的遗传基础:队列研究。gydF4y2Ba英国人。地中海。J。gydF4y2Ba375年gydF4y2Bae066288 (2021)。gydF4y2Ba
李,h . Minimap2:核苷酸序列两两对齐。gydF4y2Ba生物信息学gydF4y2Ba34gydF4y2Ba,3094 - 3100 (2018)。gydF4y2Ba
De Coster W。D 'Hert, S。,Schultz, D. T., Cruts, M. & Van Broeckhoven, C. NanoPack: visualizing and processing long-read sequencing data.生物信息学gydF4y2Ba34gydF4y2Ba,2666 - 2669 (2018)。gydF4y2Ba
罗宾逊,j . t . et al .综合基因组浏览器。gydF4y2Ba生物科技Nat。》。gydF4y2Ba29日gydF4y2Ba26 (2011)。gydF4y2Ba
鲁曼:J。,Quick, J. & Simpson, J. T. A complete bacterial genome assembled de novo using only nanopore sequencing data.Nat方法。gydF4y2Ba12gydF4y2Ba,733 - 735 (2015)。gydF4y2Ba
黄,x &马丹,A . CAP3: DNA序列组装程序。gydF4y2Ba基因组Res。gydF4y2Ba9gydF4y2Ba,868 - 877 (1999)。gydF4y2Ba
安德鲁斯,r . m . et al .再分析和修正的剑桥参考人类线粒体DNA序列。gydF4y2BaNat,麝猫。gydF4y2Ba23gydF4y2Ba147 (1999)。gydF4y2Ba
西弗斯,et al。快,可伸缩的一代的高质量蛋白质多序列比对使用Clustalω。gydF4y2Ba摩尔。系统。医学杂志。gydF4y2Ba7gydF4y2Ba539 (2011)。gydF4y2Ba
旋塞,p . j . et al . Biopython:免费提供Python计算分子生物学和生物信息学的工具。gydF4y2Ba生物信息学gydF4y2Ba25gydF4y2Ba,1422 - 1423 (2009)。gydF4y2Ba
Krzywinski m . et al .圆环:比较基因组学的审美信息。gydF4y2Ba基因组Res。gydF4y2Ba19gydF4y2Ba,1639 - 1645 (2009)。gydF4y2Ba
Anand, l . &罗德里格斯洛佩兹,c . m . ChromoMap: R包multi-omics的交互式可视化的数据和注释的染色体。gydF4y2BaBMC生物信息学gydF4y2Ba23gydF4y2Ba33岁(2022年)。gydF4y2Ba
王等人dbRIP:人类逆转录转座子插入多态性的一个高度集成的数据库。gydF4y2Ba嗡嗡声。Mutat。gydF4y2Ba27gydF4y2Ba,323 - 329 (2006)。gydF4y2Ba
科勒,s . et al . 2021年人类表型本体。gydF4y2Ba核酸Res。gydF4y2Ba49gydF4y2BaD1207-D1217 (2020)。gydF4y2Ba
温斯顿常等人的:Web应用程序框架为R R包版本1.7.1上。gydF4y2Bahttps://CRAN.R-project.org/package=shinygydF4y2Ba(2021)。gydF4y2Ba
确认gydF4y2Ba
本研究利用数据生成的基因组学英国100000人基因工程。资金的主要来源生物和英格兰基因组学是由英国国家卫生研究所(NIHR,gydF4y2Bahttp://www.nihr.ac.ukgydF4y2Ba)。这项工作也是通过资金来自英国医学研究理事会(MRC)来创建临床基因组学公司设在英国。P.F.C.是威康信托基金会首席研究员(212219 / Z / 18 / Z),和一个英国NIHR高级调查员,他收到来自英国医学研究理事会的支持线粒体生物学单位(MC_UU_00015/9),英国医学研究理事会(MRC)国际基因组医学中心神经肌肉疾病(先生/ S005021/1),利华休姆信托赞助(rpg - 2018 - 408),一个MRC研究格兰特(先生/ S035699/1),一个阿尔茨海默氏病协会项目格兰特(pg - 18 b - 022)和国家卫生研究所(NIHR)基于生物医学研究中心的剑桥大学医院NHS信托基金会和剑桥大学。由MRC K.R.S.支持战略奖建立一个国际基因组医学中心神经肌肉疾病(ICGNMD) / S005021/1先生。M.J.C. NIHR高级调查员,这项工作形式组合的一部分NIHR巴兹医院生物医学研究中心的研究。基因组学英格兰和100000人基因组计划由国家卫生研究所,威康信托基金会,医学研究委员会,英国癌症研究中心、卫生部和社会关怀和英国国民健康保险制度。我们感谢所有的参与者和医疗团队使得这一研究成为可能。作者的观点是(s)和不一定NHS, NIHR或卫生部和社会关怀。gydF4y2Ba
作者信息gydF4y2Ba
作者和联系gydF4y2Ba
贡献gydF4y2Ba
P.F.C.和一部构思的整体研究。一部关于生物信息学和统计分析完成,其他来自K.R.S.和m . Tischkowitz癌症基因分析,K.R.S.罕见疾病基因分析,和通用电气公司,同,M. Tanguy and A.G. for the long-read sequencing. M.J.C. had a key role in advising and overseeing access to the Genomics England dataset. P.F.C. and W.W. wrote the first draft of the manuscript, which all authors critically edited. P.F.C. supervised the study and sought the funding.
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称没有利益冲突。gydF4y2Ba
同行评审gydF4y2Ba
同行审查的信息gydF4y2Ba
自然gydF4y2Ba由于年轻的Seok榉,布雷特·考夫曼和其他,匿名的,审稿人(s)为他们的贡献的同行评审工作。同行评审报告。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然保持中立在发表关于司法主权地图和所属机构。gydF4y2Ba
扩展数据数据和表gydF4y2Ba
扩展数据图1 53574人全基因组测序的基因组学英格兰罕见的疾病项目和检测NUMT插入。gydF4y2Ba
一个gydF4y2Ba。个人的年龄的柱状图。gydF4y2BabgydF4y2Ba。饼图罕见疾病的个人的性别决定基因。gydF4y2BacgydF4y2Ba。Letter-value块测序深度的全基因组测序(左)和线粒体基因组测序从罕见的疾病基因组(右)。中间的线表示中位数(50百分位)。每个连续的水平向外包含剩余的数据的一半。前两个部分从中心行包含50%的数据。接下来的两个小节包含25%的数据。这个过程一直持续到在离群值级别。离群值绘制为钻石。gydF4y2BadgydF4y2Ba。概述的频率NUMTs检测到至少2条不和谐的读取。常见=人口频率(F) > = 1%;罕见= F < 1%,但> = 0.1%;件F < 0.1%的人口。gydF4y2BaegydF4y2Ba。直方图的平均每个个体所不具备的NUMTs数量的参考序列和被检测到至少2条不和谐的读取。gydF4y2BafgydF4y2Ba。Letter-value块NUMTs检测到的平均数量至少5对不和谐的读取从每个个体,男性和女性分别显示。中间的线表示中位数(50百分位)。每个连续的水平向外包含剩余的数据的一半。前两个部分从中心行包含50%的数据。接下来的两个小节包含25%的数据。这个过程一直持续到在离群值级别。离群值绘制为钻石。gydF4y2BaggydF4y2Ba。相关的个人年龄和NUMTs检测到的平均数量。回归线红色所示。gydF4y2Ba
扩展数据图2 NUMTs检测不同人群。gydF4y2Ba
一个gydF4y2Ba。染色体NUMTs地图中发现非洲、美国、东亚、南亚和欧洲的基因组。染色体的位置不同NUMT插入彩色NUMTs的频率(F)。点显示NUMTs的位置。gydF4y2BabgydF4y2Ba。统一的歧管近似和投影(UMAP)的生殖系NUMTs人口和4个亚类。gydF4y2BacgydF4y2Ba。染色体的位置NUMTs明显更大的/更少的检测不同人群。gydF4y2Ba
扩展数据图3连接NUMTs和读测序验证。gydF4y2Ba
一个gydF4y2Ba。圆环图显示4个人从2家庭共享5 mtDNA-mtDNA断点只出现在4个人,同时也分享一件NUMT插入也只在4个人。gydF4y2BabgydF4y2Ba。圆环图显示8个人共享1 mtDNA-mtDNA断点只出现在这8个人,也分享了NUMT插入也只在8个人。蓝色箭头指向共享NUMTs。红色箭头指向共享mtDNA-mtDNA断点。gydF4y2BacgydF4y2Ba。模型显示连接NUMTs和我们的战略的形成检测使用读测序和短内容排序。mtDNA和核基因组序列是橙色和蓝色所示。读取映射到mtDNA和核基因组序列都显示在灰色,映射到只有mtDNA序列在橙色和映射到只有蓝色的核基因组序列。gydF4y2BadgydF4y2Ba。圆环的情节mtDNA-mtDNA断点罕见疾病基因组中发现。mtDNA-mtDNA断点被分裂只读取映射mtDNA检测。复杂的连接NUMTs包含多个mtDNA碎片。检测支持公认的连接NUMTs mtDNA-mtDNA断点。常见和罕见mtDNA-mtDNA断点(频率> = 0.1%)所示红色链接。件mtDNA-mtDNA断点(频率< 0.1%)蓝色链接所示。gydF4y2BaegydF4y2Ba。圆环图显示了罕见的甲基化频率NUMT(插入mt.12314 - 9526 bp,频率= 0.26%)中发现4来自同一家庭成员(父亲、母亲、兄弟和渊源者)。圈从外到内显示如下:(1)甲基化频率分裂long-reads NUMTs发现的父亲,母亲,兄弟和渊源者,(2)之间的甲基化频率比NUMTs和“true”mtDNA序列在所有4个家庭成员。绿点在NUMTs甲基化的网站。颜色键对应于甲基化频率。gydF4y2BafgydF4y2Ba。Letter-value块的平均数量观察mtDNA变体(左-变异频率> 1%,右-变异频率> 2%),个人携带的连接NUMTs没有公认的连接NUMTs显示,分别。变体中观察到的个体携带假定的连接NUMTs mtDNA序列和NUMTs混合变异。中间的线表示中位数(50百分位)。每个连续的水平向外包含剩余的数据的一半。前两个部分从中心行包含50%的数据。接下来的两个小节包含25%的数据。这个过程一直持续到在离群值级别。离群值绘制为钻石。gydF4y2Ba
扩展数据图4 IGV对齐gydF4y2Ba新创gydF4y2BaNUMTs罕见疾病的基因组。gydF4y2Ba
整合基因组学查看器(进口)截图显示,读取对应三个保持一致gydF4y2Ba新创gydF4y2BaNUMTs中观察到两个家庭。水鸭条指示一致读它映射到配偶映射到mtDNA核DNA。在家庭1,后代携带两个NUMTs在相同的基因,但未见的父母。在家庭2,后代携带NUMT未见的父母中的任何一个。gydF4y2Ba
扩展数据图5的频率NUMT断点mtDNA基因组和转录起始点的核NUMT位置的距离(TSS)。gydF4y2Ba
一个gydF4y2Ba。归一化频率在每个mtDNA地区NUMT断点。黑色线条预计频率。上面的蓝色区域图显示的频率从生殖系NUMTs断点。底红色区域图显示了肿瘤特异NUMTs断点的频率。线粒体区域显示在底部的不同颜色的每一个情节。红框突出的地区频率明显大于预期的偶然。蓝盒子强调了地区的频率明显低于预期的机会。gydF4y2BabgydF4y2Ba。规范化的NUMTs数量在每个Dloop地区。星星代表NUMTs明显富集在每个地区(排列测试)。圆圈标记P值来自生殖系的比较和肿瘤特异NUMTs(双边确切概率法)。gydF4y2BacgydF4y2Ba。删除断点和频率的相关性NUMT断点在每个从生殖系肿瘤特异NUMTs mtDNA区域。gydF4y2BadgydF4y2Ba。直方图距离的NUMTs转录起始点的位置(TSS)。生殖系,生殖系常见和罕见,件和肿瘤特异NUMTs,分别。gydF4y2Ba
扩展数据图6 12509年全基因组测序normal-tumour从基因组学对英国癌症项目和检测NUMT插入。gydF4y2Ba
一个gydF4y2Ba。饼图的样本量的比例每个癌症类型包括在这项研究中。gydF4y2BabgydF4y2Ba。肿瘤供体年龄从所有癌症类型的柱状图(右下角),每个癌症类型。gydF4y2BacgydF4y2Ba。投影核基因型常见的单核苷酸多态性在两大主成分(PC1和PC2)计算与1000年从癌症基因组、基因组数据集与个人彩色分配核血统。gydF4y2BadgydF4y2Ba。样本大小的比例从每个人口癌症基因组。gydF4y2BaegydF4y2Ba。NUMTs发现数量不同的组织类型匹配的正常组织样本癌症的参与者。中间的线表示中位数(50百分位)。每个连续的水平向外包含剩余的数据的一半。前两个部分从中心行包含50%的数据。接下来的两个小节包含25%的数据。这个过程一直持续到在离群值级别。离群值绘制为钻石。gydF4y2BafgydF4y2Ba。数量NUMTs罕见疾病的血液样本中发现和匹配的正常组织样本取自癌症的参与者。中间的线表示中位数(50百分位)。每个连续的水平向外包含剩余的数据的一半。前两个部分从中心行包含50%的数据。接下来的两个小节包含25%的数据。这个过程一直持续到在离群值级别。离群值绘制为钻石。gydF4y2Ba
扩展数据图7 IGV对齐NUMTs的例子。gydF4y2Ba
一个gydF4y2Ba。IGV对齐肿瘤特异的例子NUMTs加上其他易位核基因组的变化。水鸭条指示一致读它映射到配偶映射到mtDNA核DNA。其他non-grey色彩条指示对齐读取映射到一个核染色体配偶映射到不同的核染色体。例如,癌症样本1有一个NUMT(蒂尔酒吧)5号染色体上,另一个5号染色体和染色体易位变化13(橙色酒吧)在同一地区(左)。同样也出现在染色体易位变异13(右)。对齐读取映射到13号染色体,配偶映射到5号染色体(钢蓝色酒吧)。gydF4y2BabgydF4y2Ba。失去NUMTs IGV对齐的肿瘤的一个例子。IGV截图显示对齐读取对应NUMTs丢失在一个乳房肿瘤样本。水鸭条指示一致读它映射到配偶映射到mtDNA核DNA。NUMTs只出现在匹配的正常样本而不是肿瘤样本,平均测序深度的肿瘤样本(128 x)超过三次比匹配的正常样本(40 x)。gydF4y2BacgydF4y2Ba。Cirocs情节展示了一个例子,失去了NUMT血液学的肿瘤样本。代表所有的链接NUMTs检测正常样本或肿瘤样本。肿瘤样本失去了许多NUMTs整个基因组测序深度平均的肿瘤样本(116 x)是两倍多比匹配的正常样本(40 x)。gydF4y2Ba
扩展数据图8 NUMT核断点,关系PRDM9结合位点,NUMT年龄。gydF4y2Ba
一个gydF4y2Ba。频率的三核苷酸在生殖系NUMTs断点。核基因组的断点显示在顶部和底部mtDNA基因组,常见的,件NUMTs和期望频率中显示不同的颜色。三核苷酸的断点侧翼更可能发生在nCC / CCn mtDNA基因组和不太可能在nTT / TT核和mtDNA基因组,尤其是件NUMTs。有同样的趋势未见肿瘤特异NUMTs (gydF4y2BabgydF4y2Ba),表示信号是由生物学、但不是测序文物。gydF4y2BabgydF4y2Ba。频率的三核苷酸肿瘤特异NUMTs断点在核基因组(上)和mtDNA基因组(底部),肿瘤特异NUMTs和期望频率中显示不同的颜色。# P值< 0.1,< 0.05,< 0.01 * *,< 0.001 * * *,< 0.0001 * * * *(双边确切概率法)(补充表gydF4y2Ba6gydF4y2Ba)。gydF4y2BacgydF4y2Ba。PRDM9结合位点之间的距离分布和肿瘤特异NUMTs在每个肿瘤类型。gydF4y2BadgydF4y2Ba。岁NUMTs估计在这个研究。Y轴显示的频率NUMTs在非洲和非洲以外的人群。NUMTs的频率不同非洲和非洲以外的,特别是对老NUMTs更常见的在非洲的人口。gydF4y2Ba
扩展数据图9 IGV比对NUMTs和核染色体结构变异。gydF4y2Ba
一个gydF4y2Ba。mtDNA片段插入两条边的一个例子CNV的重复。gydF4y2BabgydF4y2Ba。mtDNA片段插入两条边的一个例子的删除。水鸭条指示一致读它映射到配偶映射到mtDNA核DNA,并强调在蒂尔。gydF4y2BacgydF4y2Ba。gydF4y2BadgydF4y2Ba。两个例子的癌症基因组携带mito-chromothripsis观察在这个研究。gydF4y2BacgydF4y2Ba。核基因组的测序深度显示在顶部面板。例子的读一致性NUMTs IGV底部所示。阅读是由一对彩色的取向和可以找到配偶的染色体。gydF4y2BadgydF4y2Ba。核基因组的测序深度显示在顶部面板。蒂尔点NUMT插入的位置。例子的读一致性NUMTs IGV底部所示。阅读是由一对彩色的取向和可以找到配偶的染色体。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
这个文件包含补充的结果,参考补充结果和补充无花果。1 - 9。gydF4y2Ba
补充表gydF4y2Ba
这个文件包含补充表1 - 7。gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到Creative Commons许可,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
魏,W。,Schon, K.R., Elgar, G.et al。gydF4y2Ba在66083年人类基因组Nuclear-embedded线粒体DNA序列。gydF4y2Ba自然gydF4y2Ba611年gydF4y2Ba,105 - 114 (2022)。https://doi.org/10.1038/s41586 - 022 - 05288 - 7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586 - 022 - 05288 - 7gydF4y2Ba
评论gydF4y2Ba
通过提交评论你同意遵守我们的gydF4y2Ba条款gydF4y2Ba和gydF4y2Ba社区指导原则gydF4y2Ba。如果你发现一些滥用或不符合我们的条件或准则请国旗是不合适的。gydF4y2Ba