摘要gydF4y2Ba
急性病毒感染可以在恢复后对免疫系统产生持久的功能影响,但它们如何影响稳态免疫状态和对未来扰动的反应仍然知之甚少gydF4y2Ba1克ydF4y2Ba,gydF4y2Ba2 gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba,gydF4y2Ba4 gydF4y2Ba.在这里,我们使用系统免疫学方法,包括纵向多模态单细胞分析(表面蛋白、转录组和V(D)J序列),比较评估33名从轻度、非住院的COVID-19康复后的健康个体(诊断后平均151天)和40名从未患过COVID-19的年龄和性别匹配的对照个体的基线免疫状态和对流感疫苗的反应。在COVID-19后的基线和独立于时间的情况下,康复者的T细胞激活特征升高,先天性免疫基因(包括单核细胞中的toll样受体)表达降低。与从COVID-19中恢复的健康男性个体和从COVID-19中恢复的女性个体相比,从COVID-19中恢复的男性个体在接种疫苗后具有更高的先天性流感特异性浆母细胞和抗体反应,部分原因是男性康复者在接种疫苗后早期具有更高的IL-15反应的单核细胞,同时“虚拟记忆”类CD8的预防接种频率升高gydF4y2Ba+gydF4y2Ba在IL-15刺激后,T细胞会产生更多的IFNγ。此外,在接种疫苗后的第1天至第28天,恢复的单核细胞中被抑制的先天免疫基因的表达增加,因此向健康对照个体的疫苗接种前基线移动。相比之下,对照组的这些基因在第1天下降,并在第28天恢复到基线水平。我们的研究揭示了先前轻度COVID-19的性别二形态效应,并表明人类的病毒感染可以建立新的免疫设定点,以抗原不可知的方式影响未来的免疫反应。gydF4y2Ba
主要gydF4y2Ba
已经描述了慢性和已解决的病毒感染的长期免疫效应的例子;例如,从自然急性麻疹感染中恢复后,在数月至数年内,体液免疫显著降低,对非麻疹感染的易感性增加gydF4y2Ba1克ydF4y2Ba.如芽孢杆菌Calmette-Guérin(卡介苗)和麻疹等活疫苗可以对先天免疫细胞(如单核细胞)及其长寿祖细胞产生“训练”作用,这可能是卡介苗在降低婴儿全因死亡率方面的病原体非特异性作用的基础gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba.COVID-19可导致感染后数月持续的临床后遗症,无论是住院患者还是轻症患者gydF4y2Ba7gydF4y2Ba.虽然急性后COVID-19综合征(又称长冠状病毒)的临床表现范围正在扩大,但我们对SARS-CoV-2感染后恢复后的分子和细胞免疫变化缺乏了解。更好地了解轻度COVID-19的功能性免疫印迹可能具有特别重要的公共卫生意义,因为这一人群构成了大多数COVID-19康复者。更广泛地说,病毒感染是否以及如何改变了稳态基线免疫状态,以及任何这种改变是否会影响对未来挑战(如感染或疫苗接种,共享或不同的抗原)的反应,这些基本问题仍然知之甚少。gydF4y2Ba
在这里,我们利用了2020年初秋的独特机会和流行病学环境,当时COVID-19轻度患者已临床康复,但尚未再次感染SARS-CoV-2或接受COVID-19疫苗接种(直到2020年底才开始接种);此外,在此期间,其他呼吸道感染的流行率极低gydF4y2Ba8gydF4y2Ba.我们招募并比较评估了来自同一地理区域的健康个体,他们(1)从未住院的轻度COVID-19病例中康复,(2)年龄和性别匹配的从未患过COVID-19的对照组。除了评估covid -19后的免疫状态外,我们还使用流感疫苗在血清学、转录、蛋白质组学和细胞水平上评估这两个人群的免疫反应。这些分析揭示了人类在两次明确的免疫接触后免疫系统发生变化的基本原则:轻度COVID-19作为一种自然传染性扰动,流感疫苗接种作为一种非sars - cov -2抗原的受控和定时干预。gydF4y2Ba
既往有SARS-CoV-2感染症状者(gydF4y2BangydF4y2Ba= 31;经鼻腔PCR检测确诊)或无症状感染(gydF4y2BangydF4y2Ba= 2;经抗体试验诊断;gydF4y2Ba方法gydF4y2Ba),以及年龄和性别匹配的健康对照组(gydF4y2BangydF4y2Ba= 40),无COVID-19病史(抗体测试阴性),于2020年秋季从社区招募,并进行纵向随访(图2)。gydF4y2Ba1gydF4y2Ba而且gydF4y2Ba方法gydF4y2Ba).COVID-19诊断后恢复的平均时间为151天(扩展数据图)。gydF4y2Ba1gydF4y2Ba及扩展数据表gydF4y2Ba1克ydF4y2Ba),这些患者在急性疾病期间有临床轻度疾病,不需要住院治疗(自我报告的平均患病时间为16.5天),并且没有重大的医疗合并症,包括入组时的感染、肥胖(体重指数> 30)或自身免疫性疾病(图2)。gydF4y2Ba1 bgydF4y2Ba).所有参与者都没有参加COVID-19疫苗试验,在本研究中,他们在接种季节性流感疫苗之前也没有最近接种过任何类型的疫苗。在研究登记时,少数个体继续有轻微的自我报告的疾病后遗症(3名男性和8名女性),最常见的是味觉和/或嗅觉的丧失(扩展数据表)gydF4y2Ba1克ydF4y2Ba).女性参与者更有可能有后遗症(Fisher的精确测试,gydF4y2BaPgydF4y2Ba= 0.09gydF4y2BaPgydF4y2Ba年龄<65岁的= 0.03),与其他大型研究报告的比率相似gydF4y2Ba9gydF4y2Ba.gydF4y2Ba
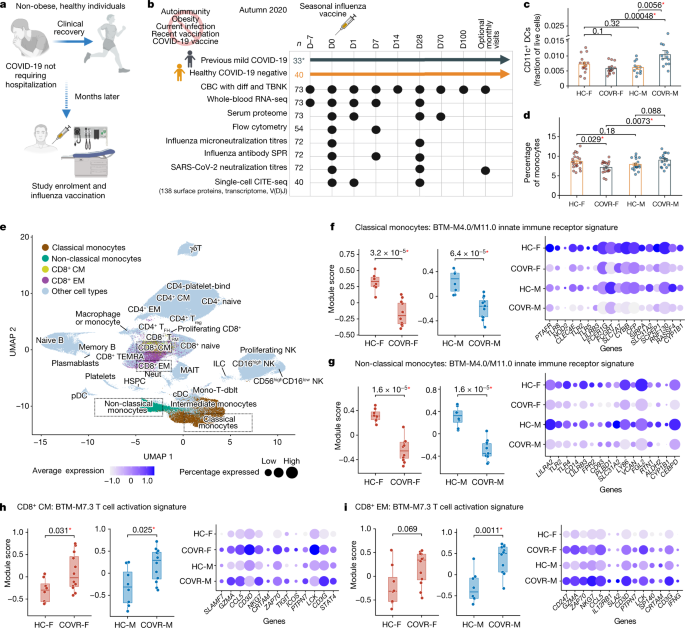
一个gydF4y2Ba,研究概念和设计示意图。gydF4y2BabgydF4y2Ba,研究中产生的数据。从COVID-19中恢复的参与者和健康对照组均在疫苗接种前7天(D - 7)入组,并在与流感疫苗接种日相关的指定时间点进行采样。为每种数据类型分析的参与者数量被指出。CBC有差异,全血计数有差异;SPR,表面等离子体共振;TBNK, T和B淋巴细胞和自然杀伤细胞表型。用星号(*)表示包括2名无症状个体(基于抗体)。gydF4y2BacgydF4y2Ba, CD11c比例比较gydF4y2Ba+gydF4y2Badc(作为流式细胞术中活细胞的百分比)在COVR-F (gydF4y2BangydF4y2Ba= 15), hc-f (gydF4y2BangydF4y2Ba= 16), covr-m (gydF4y2BangydF4y2Ba= 12)及HC-M (gydF4y2BangydF4y2Ba= 11)组在D0。误差条表示每组的s.e.m.。gydF4y2BadgydF4y2Ba,类似于gydF4y2BacgydF4y2Ba,但对于单核细胞(来自CBC;gydF4y2BaygydF4y2Ba轴)之间的COVR-F (gydF4y2BangydF4y2Ba= 17), covr-m (gydF4y2BangydF4y2Ba= 16), hc-f (gydF4y2BangydF4y2Ba= 21)及HC-M (gydF4y2BangydF4y2Ba= 19)组(D−7和D0的平均值)。gydF4y2BaegydF4y2Ba,对CITE-seq单细胞数据进行均匀流形近似和投影(UMAP)分析,显示基于细胞表面蛋白标记物表达的细胞聚类(来自所有时间点的632,100个单细胞与CITE-seq数据:第0,1,28天)。染色和盒状细胞团进一步检查gydF4y2BafgydF4y2Ba- - - - - -gydF4y2Ba我gydF4y2Ba.CD4-platelet-bind: CD4gydF4y2Ba+gydF4y2Ba血小板标记的T细胞;CM,中央存储器;ILC,先天淋巴样细胞;Mono-T-dblt,单核细胞和T细胞双合体;TgydF4y2Ba跳频gydF4y2Ba, T滤泡辅助细胞;TgydF4y2Ba注册gydF4y2Ba,调节性T细胞;TgydF4y2BaRMgydF4y2Ba,组织常驻记忆T细胞;造血干细胞和祖细胞;中性粒细胞,中性粒细胞;pDC,浆细胞样树突状细胞;cDC,传统树突状细胞;MAIT:粘膜相关不变T细胞。gydF4y2BafgydF4y2Ba,先天免疫受体(IIR)特征评分比较(gydF4y2Ba方法gydF4y2Ba)与HC-F (gydF4y2BangydF4y2Ba= 8)和COVR-F (gydF4y2BangydF4y2Ba= 12)(左方框)及HC-M (gydF4y2BangydF4y2Ba= 8)和COVR-M (gydF4y2BangydF4y2Ba= 12)(右框)组使用CITE-seq经典单核细胞假体表达数据在D0(左)。每个点代表一个参与者。右,所选基因的平均基因表达量,包括基因本体论(GO)模式识别受体活性和免疫受体活性基因集。gydF4y2BaggydF4y2Ba,类似于gydF4y2BafgydF4y2Ba,但在D0处显示非经典单核细胞群。gydF4y2BahgydF4y2Ba,类似于gydF4y2BafgydF4y2Ba,但显示CD8的T细胞激活(BTM-M7.3)模块评分gydF4y2Ba+gydF4y2BaCM T细胞在D0。从基因集富集分析(GSEA)中选取的男性和女性共有的前沿基因的平均基因表达量如下所示(gydF4y2Ba方法gydF4y2Ba).gydF4y2Ba我gydF4y2Ba,类似于gydF4y2BahgydF4y2Ba,但显示CD8gydF4y2Ba+gydF4y2Ba在D0处的EM T细胞数量。所有的箱形图都显示了中位数(中心线),第一和第三分位数(盒限),以及每个方向(上下须)从盒限开始的最大1.5 ×四分位数范围(IQR)。除特别说明外,采用双尾Wilcoxon秩和检验来确定组间差异的统计学意义。重要的(gydF4y2BaPgydF4y2Ba< 0.05)的差异用红色星号(*)突出显示。的图表gydF4y2Ba一个gydF4y2Ba而且gydF4y2BabgydF4y2Ba是使用BioRender创建的。gydF4y2Ba
轻度COVID-19基线恢复gydF4y2Ba
纵向多组分析使用全血转录组(WBT)分析,138个表面蛋白的单细胞分析,转录组和V(D)J序列分析,使用转录组的细胞索引和测序表位(CITE-seq)gydF4y2Ba10gydF4y2Ba,血清蛋白谱,抗体特征,外周血免疫细胞频率与全血细胞计数(CBC)的血液学参数,以及临床和研究流式细胞术覆盖主要免疫细胞谱系和亚群(图2)。gydF4y2Ba1 bgydF4y2Ba和补充图。gydF4y2Ba1克ydF4y2Ba).我们首先评估了恢复者与年龄和性别匹配的健康对照个体之间的基线疫苗接种前差异。据报道,COVID-19有性别依赖性免疫反应gydF4y2Ba11gydF4y2Ba,我们的分析明确地搜索了性别依赖的特征。即使在症状消退后,感染后的免疫解决也可能随着时间的推移而展开,并且确实有参数显示了我们队列中持续变化的证据-定义为自COVID-19诊断以来与时间相关的参数(TSD;gydF4y2Ba方法gydF4y2Ba及补充表gydF4y2Ba1克ydF4y2Ba),如预期的那样,包括sars - cov -2中和抗体滴度gydF4y2Ba12gydF4y2Ba(扩展数据图。gydF4y2Ba1 bgydF4y2Ba).然而,我们主要感兴趣的是发现持续的、不依赖于TSD的COVID-19后免疫印迹,因此我们专注于与先前轻度COVID-19相关但与TSD无关的暂时稳定免疫状态。因此,我们评估了(1)从COVID-19康复的女性参与者(COVR-F)与健康对照女性参与者(HC-F)之间的差异;(2)从COVID-19康复的男性参与者(COVR-M)与健康对照男性参与者(HC-M);(3)考虑健康对照个体的男女差异后的COVR-M与COVR-F(以下为性别差异;补充表gydF4y2Ba2 gydF4y2Ba).与HC-M和/或COVR-F组相比,COVR-M组中单核细胞和常规/髓系树突状细胞(cdc)等髓系细胞的频率趋于更高(图2)。gydF4y2Ba1 c, dgydF4y2Ba和扩展数据图。gydF4y2Ba1 c, dgydF4y2Ba),这与COVID-19中髓细胞破坏的报告一致,特别是在严重急性疾病中gydF4y2Ba13gydF4y2Ba.在这里,即使在轻度疾病恢复几个月后,也能检测到男性特异性单核细胞频率的升高。gydF4y2Ba
WBT数据还揭示了与先前轻度COVID-19相关的性别依赖特征(扩展数据图)。gydF4y2Ba1 egydF4y2Ba;例如,来自血液转录模块(BTM)收集的与单核细胞相关的M11.0和M4.0,包括氧化磷酸化等代谢特征(补充表)gydF4y2Ba3 gydF4y2Ba).WBT差异可由细胞组成和细胞内转录变化共同驱动。事实上,先天免疫、代谢和T细胞相关的特征至少部分是由COVR-M组中循环单核细胞频率增加和相应的T细胞频率降低所驱动的(图2)。gydF4y2Ba1 dgydF4y2Ba和扩展数据图。gydF4y2Ba1 fgydF4y2Ba),因为当考虑到单核细胞频率时,这些转录富集信号在统计学上变得无关紧要(数据未显示)。gydF4y2Ba
为了评估独立于细胞频率的转录改变,我们使用CITE-seq检测了上述WBT特征背后的细胞类型特异性贡献。我们对单个细胞进行聚类,并使用表面蛋白表达谱对结果的聚类进行注释。gydF4y2Ba1 egydF4y2Ba而且gydF4y2Ba方法gydF4y2Ba).细胞类型特异性转录分析表明,从COVID-19康复的参与者与健康对照个体之间存在性别依赖和独立差异(补充表)gydF4y2Ba4 gydF4y2Ba).在上述WBT分析中富集的基因集中(扩展数据图。gydF4y2Ba1 egydF4y2Ba),但现在没有了细胞频率混淆,在从COVID-19康复的参与者中,BTM M11.0/4.0基因集在经典和非经典单核细胞中相对于健康对照个体表现出降低的表达,而在CD8中T细胞激活签名(BTM M7.3)的基因则相反gydF4y2Ba+gydF4y2Ba中央记忆和效应记忆(EM) T细胞(图。gydF4y2Ba外:我1gydF4y2Ba扩展数据图gydF4y2Ba1克gydF4y2Ba及补充表gydF4y2Ba5gydF4y2Ba).CD8中的T细胞激活特征gydF4y2Ba+gydF4y2BaEMs在COVR-M组尤其明显。gydF4y2Ba1我gydF4y2Ba).驱动单核细胞抑制富集的基因(即前沿基因(LEGs))包括许多表面受体,如编码模式识别受体(gydF4y2BaTLR2gydF4y2Ba,gydF4y2BaTLR4gydF4y2Ba而且gydF4y2BaTLR8gydF4y2Ba)、肽聚糖识别受体(gydF4y2BaNOD2gydF4y2Ba)、高亲和IgE FC受体(gydF4y2BaFCER1GgydF4y2Ba)和c型凝集素受体(gydF4y2BaCLEC4EgydF4y2Ba)(图。gydF4y2Ba1 f, ggydF4y2Ba).单核细胞中的先天免疫受体(IIR)特征以及T细胞激活特征主要与男性和女性个体的TSD无关(扩展数据图)。gydF4y2Ba1 hgydF4y2Ba).gydF4y2Ba
T细胞激活特征可能在急性COVID-19期间出现并在急性COVID-19后持续存在(参考文献)。gydF4y2Ba14gydF4y2Ba),但IIR的签名就不那么清楚了。因此,我们研究了这一特征是否与急性COVID-19中出现的基因表达变化有关。使用先前发表的CITE-seq数据集,该数据集是我们从意大利患有严重COVID-19住院的老年男性群体中生成的gydF4y2Ba15gydF4y2Ba,我们注意到,在经典单核细胞中,急性COVID-19患者的IIR LEGs平均表达明显低于健康对照个体,且与疾病严重程度呈负相关(扩展数据图)。gydF4y2Ba1我gydF4y2Ba).因此,这种抑制的IIR特征可能源于感染的急性反应,并稳定地持续存在。先前的研究报道了急性COVID-19中几种(可能重叠的)单核细胞改变类型,包括抗原提呈低、NF-κB/炎症抑制或骨髓来源抑制细胞样表型的单核细胞gydF4y2Ba13gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba.然而,与我们队列中健康对照组相比,在流感疫苗接种前基线时,从COVID-19康复的参与者的单核细胞中,这些单核细胞表型均无显著差异(补充图2)。gydF4y2Ba2 gydF4y2Ba),这表明我们的抑郁单核细胞基因特征包括模式识别和IIR基因,与早期急性疾病中发现的特征不同。总之,我们的研究结果表明,即使是轻微的、未住院的SARS-CoV-2感染,也可能在临床康复几个月后建立新的、暂时稳定的、性别依赖的免疫印迹。gydF4y2Ba
为了评估其他自然呼吸道病毒感染是否会留下类似的未解决的性别特异性免疫状态,我们使用了已发表的WBT数据集,评估了两个不同季节确诊为社区甲型流感(主要是H1N1大流行)感染的独立队列患者gydF4y2Ba18gydF4y2Ba(2009-2010和2010-2011;扩展数据图。gydF4y2Ba2gydF4y2Ba).通过比较每个季节前后(即感染前和恢复后)的WBT谱,我们发现这两个独立队列之间的感染后变化仅在男性个体中具有一致性(女性个体的变化在这两个队列/季节之间不一致;扩展数据图。gydF4y2Ba2 bgydF4y2Ba及补充表gydF4y2Ba6gydF4y2Ba).在我们的队列中,与COVR-F组相比,男性个体恢复后表达增加的基因也富集了COVR-M组中更高表达的基因(在考虑了健康参与者中存在的预期性别差异后;扩展数据图。gydF4y2Ba2摄氏度gydF4y2Ba).此外,男性从流感感染中恢复后表达较低的基因富集了上述抑制IIR的特征,包括gydF4y2BaTLR5gydF4y2Ba而且gydF4y2BaVCANgydF4y2Ba(无花果。gydF4y2Ba1 f, ggydF4y2Ba及补充表gydF4y2Ba6gydF4y2Ba).这些观察结果提供了独立的支持,即暴露于呼吸道病毒病原体可导致可在血液中检测到的持续免疫印迹,即使在患有轻度疾病的健康个体中也是如此。然而,不同的病毒感染,例如具有不同取向和炎症表现的病毒感染,也可能留下具有不同基因和过程的病原体依赖印迹;例如,流感后和轻度COVID-19后之间的重叠信号只是我们检测到的COVID-19后性别变化的一小部分。gydF4y2Ba
对比流感疫苗接种反应gydF4y2Ba
接下来,我们研究了以前的COVID-19是否会影响个体对非sars - cov -2免疫挑战的反应。研究参与者接种了季节性流感四价疫苗,并进行了长达100天的纵向随访,包括第1天(D1)、D7和D28天,以评估血清学、分子和细胞水平上的疫苗反应(图2)。gydF4y2Ba1 a, bgydF4y2Ba而且gydF4y2Ba2gydF4y2Ba).选择这种疫苗的部分原因是其对公共卫生的重要性——在我们的研究开始时,2020-2021年流感季节即将到来,目前尚不清楚以前的COVID-19感染是否会影响流感疫苗的反应。此外,健康成人对季节性流感疫苗接种的反应已经有很好的特征,包括疫苗接种后D1上的早期先天/炎症和干扰素(IFN)反应,以及D7附近强烈但短暂的浆母细胞峰值,最终产生流感特异性抗体gydF4y2Ba19gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba.因此,流感疫苗接种为探测先前轻度SARS-CoV-2感染的功能影响提供了极好的扰动。gydF4y2Ba
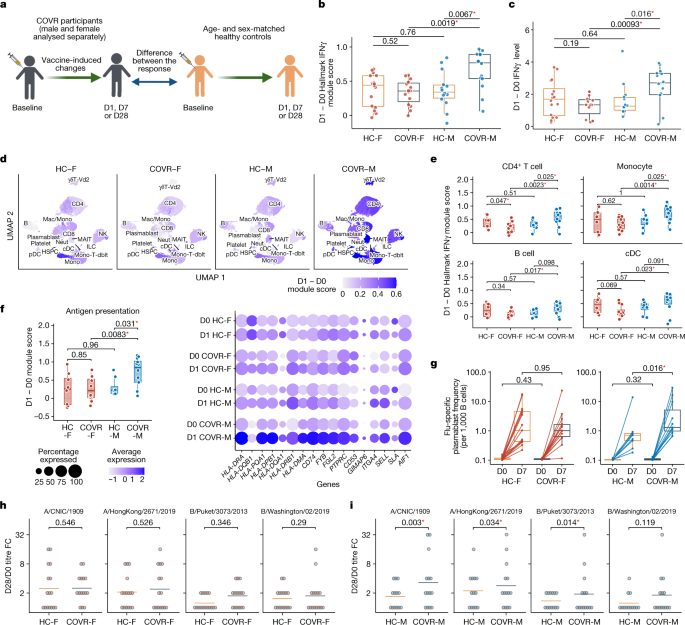
一个gydF4y2Ba从COVID-19康复的参与者和健康对照参与者之间,在接种疫苗后的时间点(D1、D7和D28),疫苗诱导的变化的性别特异性比较示意图。分析适用于65岁以下的参与者(因为年龄较大的受试者接种了更高剂量的疫苗;gydF4y2Ba方法gydF4y2Ba).gydF4y2BabgydF4y2Ba, COVR-F的D1全血IFNγ应答转录评分(D1−D0,使用Hallmark IFNγ应答基因集的基因计算)(gydF4y2BangydF4y2Ba= 15), covr-m (gydF4y2BangydF4y2Ba= 14), hc-f (gydF4y2BangydF4y2Ba= 16)及HC-M (gydF4y2BangydF4y2Ba= 14)组。gydF4y2BacgydF4y2Ba,参与者血清IFNγ蛋白水平的D1反应(D1−D0)显示在gydF4y2BabgydF4y2Ba.gydF4y2BadgydF4y2Ba,基于表面蛋白表达的UMAP分析(如图。gydF4y2Ba1 egydF4y2Ba),细胞根据D1 IFNγ反应转录评分(D1 - D0;看到gydF4y2BabgydF4y2BaHC-F的每个细胞亚群(gydF4y2BangydF4y2Ba= 8), covr-f (gydF4y2BangydF4y2Ba= 12)及HC-M (gydF4y2BangydF4y2Ba= 8)和COVR-M (gydF4y2BangydF4y2Ba= 12)组。颜色越深表示所示细胞子集D1和D0之间的差异越大。Mac,巨噬细胞;Mono,单核细胞。gydF4y2BaegydF4y2Ba,类似于gydF4y2BabgydF4y2Ba,但对于HC-F中指定的细胞子集(使用细胞子集的CITE-seq伪体mRNA表达数据计算)(gydF4y2BangydF4y2Ba= 8), covr-f (gydF4y2BangydF4y2Ba= 12), hc-m (gydF4y2BangydF4y2Ba= 8)和COVR-M (gydF4y2BangydF4y2Ba= 12)组。gydF4y2BafgydF4y2Ba,相同受试者在经典单核细胞中抗原呈递相关基因的D1转录应答评分(D1 - D0)gydF4y2BaegydF4y2Ba(左)(gydF4y2Ba方法gydF4y2Ba).右,来自抗原呈递基因的单个LEGs的平均表达(gydF4y2Ba方法gydF4y2Ba)在经典单核细胞中。gydF4y2BaggydF4y2Ba,流感特异性浆母细胞(PB;所有的哈gydF4y2Ba+gydF4y2BaCD27gydF4y2Ba+gydF4y2BaCD38gydF4y2Ba+gydF4y2BaCD20gydF4y2Ba低gydF4y2BaCD21gydF4y2Ba低gydF4y2Ba细胞;gydF4y2Ba方法gydF4y2Ba和补充图。gydF4y2Ba3 gydF4y2Ba)在D0和D7的频率,分别为COVR-F (gydF4y2BangydF4y2Ba= 14), hc-f (gydF4y2BangydF4y2Ba= 15), covr-m (gydF4y2BangydF4y2Ba= 11)及HC-M (gydF4y2BangydF4y2Ba9)组。这些线连接来自同一参与者的D0和D7的数据点。gydF4y2BahgydF4y2BaCOVR-F组和HC-F组季节性流感疫苗四种毒株(列)D28/D0微量中和滴度折叠变化(FC)的分析。每个点代表一个人。橙色和灰色线分别表示HC-F组和COVR-F组的平均折叠变化。未经调整的gydF4y2BaPgydF4y2Ba数值来源于广义线性模型,包括年龄、种族、流感疫苗接种史和基线流感滴度(gydF4y2Ba方法gydF4y2Ba).gydF4y2Ba我gydF4y2Ba,类似于gydF4y2BahgydF4y2Ba,但COVR-M和HC-M组除外。所有的箱形图都显示了中位数(中心线),第一和第三分位数(盒限),以及每个方向(上下须)盒限的最大1.5 × IQR。未经调整的gydF4y2BaPgydF4y2Ba数值会显示出来。除非另有说明,组间差异的统计学意义是用双尾Wilcoxon秩和检验确定的。重要的(gydF4y2BaPgydF4y2Ba< 0.05)的差异用红色星号(*)突出显示。图中gydF4y2Ba一个gydF4y2Ba是使用BioRender创建的。gydF4y2Ba
WBT、外周免疫细胞频率、CITE-seq、流感特异性B细胞和抗体滴度分析(评估D1、D7和D28相对于D0的反应)共同指出了对疫苗的协调的、性别特异性的先天和适应性反应差异,与健康组和COVR-F组相比,COVR-M组通常表现出更强的反应(图2)。gydF4y2Ba2我gydF4y2Ba扩展数据图gydF4y2Ba3 a、c、d、ggydF4y2Ba及补充表格gydF4y2Ba7gydF4y2Ba而且gydF4y2Ba8gydF4y2Ba).这些包括更强的先天/炎症反应,特别是ifn相关的转录反应(图。gydF4y2Ba2 bgydF4y2Ba和扩展数据图。gydF4y2Ba3gydF4y2Ba),在COVR-M组中,D1使血清中循环IFNγ蛋白水平相应增加更多(图。gydF4y2Ba2摄氏度gydF4y2Ba).正如单细胞CITE-seq分析所揭示的那样,IFNγ的这种系统性增加影响了表达IFNγ信号成分的多种细胞类型——与其他组相比,COVR-M组中大多数外周免疫细胞在D1上具有更高的IFN响应特征(基于D1与D0的比较;无花果。gydF4y2Ba二维gydF4y2Ba;无花果。gydF4y2Ba2 egydF4y2Ba表明CD4gydF4y2Ba+gydF4y2BaT细胞,B细胞,单核细胞和cdc为例)。从COVID-19康复的参与者和健康对照个体之间,基线和疫苗接种前ifn相关的转录活性基本上没有区别(扩展数据图)。gydF4y2Ba3 bgydF4y2Ba).此外,在COVR-M组的经典单核细胞中,对抗原呈递基因(包括MHC I类和II类基因)观察到更强的反应(图2)。gydF4y2Ba2 fgydF4y2Ba).因此,COVR-M组的个体在流感疫苗接种后通过D1在先天和适应性免疫细胞中产生更强的循环IFNγ和相应的转录反应。gydF4y2Ba
根据以往对健康成人流感疫苗接种的研究,由于已知佐剂引起的先天免疫反应增强可增强适应性反应gydF4y2Ba21gydF4y2Ba,我们假设COVR-M组较强的早期炎症反应将导致更强的体液反应。事实上,我们发现在COVR-M组中D7 B细胞相关和浆细胞相关的转录特征增加(扩展数据图)。gydF4y2Ba3 a, cgydF4y2Ba).此外,与HC-M组相比,COVR-M组在D7时流感特异性浆母细胞有更大的增加(图7)。gydF4y2Ba2 ggydF4y2Ba和补充图。gydF4y2Ba3 gydF4y2Ba).与之前对健康成年人的观察结果一致gydF4y2Ba22gydF4y2Ba以及COVR-M组中较强的早期IFN反应有助于诱导更强的B细胞反应的假设,我们检测到这两个参数之间的正相关,包括流感特异性浆母细胞增加的程度(扩展数据图)。gydF4y2Ba3 dgydF4y2Ba).与HC-M组相比,COVR-M组在D28相对于基线的所有疫苗株中也有更高的流感特异性抗体反应(图2)。gydF4y2Ba2 h,我gydF4y2Ba,gydF4y2Ba方法gydF4y2Ba扩展数据图gydF4y2Ba3比gydF4y2Ba及补充表gydF4y2Ba8gydF4y2Ba).虽然流感感染和疫苗接种史会影响流感疫苗的反应gydF4y2Ba23gydF4y2Ba,它们本身不太可能解释上述发现,因为covid -19康复组和健康对照组具有相似的基线抗体滴度(扩展数据图)。gydF4y2Ba3 e, fgydF4y2Ba),年龄/性别匹配,并且来自2020-2021赛季流感感染/传播非常低的同一地理区域gydF4y2Ba8gydF4y2Ba.此外,用于评估滴度反应差异的统计模型将疫苗接种前的流感滴度作为协变量(gydF4y2Ba方法gydF4y2Ba).COVID-19后时间依赖性免疫消退的程度可能不是一个因素,因为TSD和D28滴度反应在男女中都不相关(数据未显示)。总之,这些观察结果表明,以前轻度感染SARS-CoV-2可导致急性疾病几个月后对非SARS-CoV-2抗原免疫的先天和适应性反应发生性别依赖的协调变化。gydF4y2Ba
将基线与先天反应联系起来gydF4y2Ba
在确定先前轻度COVID-19与流感疫苗接种前新的基线免疫状态相关后(图2)。gydF4y2Ba1克ydF4y2Ba和扩展数据图。gydF4y2Ba1克ydF4y2Ba)和接种疫苗后covr - m组特异性反应(图。gydF4y2Ba2 gydF4y2Ba和扩展数据图。gydF4y2Ba3 gydF4y2Ba),我们接下来试图将两者联系起来,并检查哪些基线变量和细胞回路可能导致COVR-M组中ifn相关反应的增强,从而导致他们更强大的体液反应(图2)。gydF4y2Ba3gydF4y2Ba).使用流式细胞仪(补充图。gydF4y2Ba1克ydF4y2Ba)和quote -seq数据,我们首先使用多元线性模型来确定基线/预接种免疫细胞,其频率预测D1 IFN相关反应(血清IFNγ蛋白水平和IFN转录特征评分的D1与D0)。CD8的一个子集gydF4y2Ba+gydF4y2Ba具有EM表型的T细胞(CD45RAgydF4y2Ba−gydF4y2BaCCR7gydF4y2Ba−gydF4y2BaCD28gydF4y2Ba+gydF4y2BaCD27gydF4y2Ba−gydF4y2Ba;早期效应剂样)是COVR-M组的首选候选者,因此可能是接种疫苗后IFNγ的细胞来源(扩展数据图。gydF4y2Ba4 a、bgydF4y2Ba和补充图。gydF4y2Ba4 gydF4y2Ba);在健康对照个体中未观察到相同的关系(补充图。gydF4y2Ba5 a、bgydF4y2Ba).gydF4y2Ba
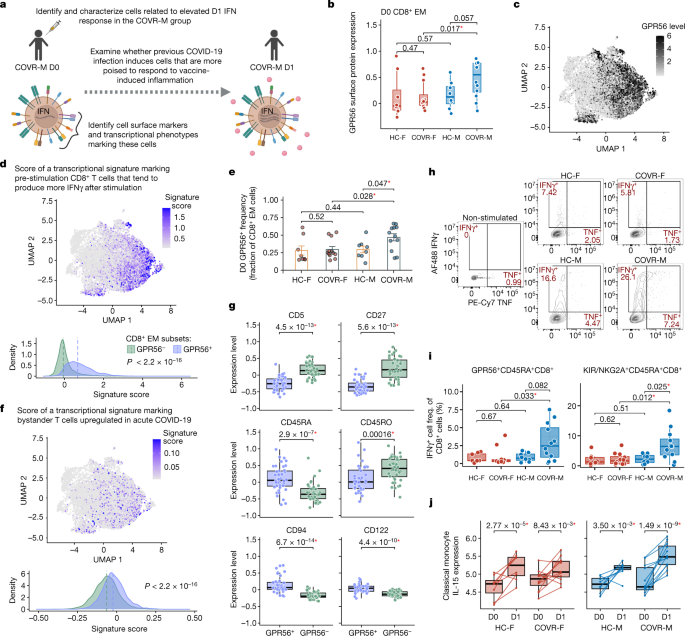
一个gydF4y2Ba,评估COVR-M组早期IFNγ反应升高的方法示意图。gydF4y2BabgydF4y2Ba, CD8中GPR56表面表达的样本均数比较gydF4y2Ba+gydF4y2Ba在D0处的EM T细胞用于COVR-F (gydF4y2BangydF4y2Ba= 12), hc-f (gydF4y2BangydF4y2Ba= 8), covr-m (gydF4y2BangydF4y2Ba= 12)及HC-M (gydF4y2BangydF4y2Ba= 8)组。gydF4y2BacgydF4y2Ba, UMAP分析D0表面GPR56蛋白在CD8上的表达gydF4y2Ba+gydF4y2BaEM细胞来自所有40名参与者,并带有CITE-seq数据。UMAP是利用CD8中排名前60位的可变表面蛋白获得的gydF4y2Ba+gydF4y2Ba电磁细胞(gydF4y2Ba方法gydF4y2Ba).gydF4y2BadgydF4y2Ba, UMAP分析,如gydF4y2BacgydF4y2Ba,但显示了使用CD29相关基因计算的D0基因表达签名评分gydF4y2Ba高gydF4y2BaCD8gydF4y2Ba+gydF4y2BaT细胞早前在一项独立研究中被发现gydF4y2Ba25gydF4y2Ba(gydF4y2Ba方法gydF4y2Ba)(上)。密度图显示上述签名分数在GPR56中的分布gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba和GPR56gydF4y2Ba−gydF4y2BaCD8gydF4y2Ba+gydF4y2BaEM细胞(下)。虚线表示分布的中位数。在单细胞水平上确定了两个细胞亚群之间的特征-得分差异的统计学意义。gydF4y2BaegydF4y2Ba, GRP56比例比较gydF4y2Ba+gydF4y2Ba细胞(CD8的部分)gydF4y2Ba+gydF4y2BaEM细胞在CITE-seq数据)之间的差异gydF4y2BabgydF4y2Ba在D0。误差条表示每组的s.e.m.。gydF4y2BafgydF4y2Ba,类似于gydF4y2BadgydF4y2Ba,但显示了在基线(D0)的旁观者T细胞签名评分(签名基因来源于参考文献。gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba;gydF4y2Ba方法gydF4y2Ba).gydF4y2BaggydF4y2Ba,比较GPR56指示记忆细胞表面蛋白标记物的平均表达gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba与GPR56gydF4y2Ba−gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba相同参与者在D0处的EM细胞gydF4y2BabgydF4y2Ba.每个点代表一个参与者。gydF4y2BahgydF4y2Ba, IFNγ的代表性流式细胞仪等高线图gydF4y2Ba+gydF4y2Ba和肿瘤坏死因子gydF4y2Ba+gydF4y2BaGPR56内的门gydF4y2Ba+gydF4y2BaCD45RAgydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaT细胞在IL-15刺激后体外表达。每个门显示的数字表示亲本细胞(即GPR56)的百分比gydF4y2Ba+gydF4y2BaCD45RAgydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaT细胞)。gydF4y2Ba我gydF4y2Ba, IFNγ的频率gydF4y2Ba+gydF4y2BaGPR56gydF4y2Ba+gydF4y2BaCD45RAgydF4y2Ba+gydF4y2BaVM-like CD8gydF4y2Ba+gydF4y2BaT细胞(左;作为CD8的分数gydF4y2Ba+gydF4y2BaT细胞)和IFNγgydF4y2Ba+gydF4y2Ba吉珥/ NKG2AgydF4y2Ba+gydF4y2BaCD45RAgydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaT细胞(右;作为CD8的分数gydF4y2Ba+gydF4y2BaT细胞)在相同的参与者中gydF4y2BabgydF4y2Ba在体外IL-15刺激后。gydF4y2BajgydF4y2Ba, D0和D1伪体的比较gydF4y2BaIL15gydF4y2BamRNA表达(gydF4y2BaygydF4y2Ba轴)在经典单核细胞中为相同的参与者gydF4y2BabgydF4y2Ba.使用考虑年龄、种族和流感疫苗接种史的线性模型确定显著性(gydF4y2Ba方法gydF4y2Ba).所有的箱形图都显示了中位数(中心线),第一和第三分位数(盒限),以及每个方向(上下须)盒限的最大1.5 × IQR。除非另有说明,组间差异的统计学意义均采用双尾Wilcoxon秩和检验。重要的(gydF4y2BaPgydF4y2Ba< 0.05)的差异用红色星号(*)突出显示。图中gydF4y2Ba一个gydF4y2Ba是使用BioRender创建的。gydF4y2Ba
接下来我们关注所有的CD8gydF4y2Ba+gydF4y2BaT细胞来自EM表型(CD8gydF4y2Ba+gydF4y2BaEM细胞)在CITE-seq数据中基于表面蛋白标记和mRNA表达(gydF4y2Ba方法gydF4y2Ba;排名靠前的簇蛋白标记见补充表gydF4y2Ba10gydF4y2Ba).我们在这些CD8中寻找细胞平均表面标记物表达的差异gydF4y2Ba+gydF4y2Ba四个参与者组的EM聚类,发现GPR56是COVR-M组中相对于HC-M和COVR-F组中表达增加的最高差异表达标记(图2)。gydF4y2Ba3 b, cgydF4y2Ba及补充表gydF4y2Ba10gydF4y2Ba).这很有趣,因为CD4gydF4y2Ba+gydF4y2BaEM和TEMRA(重表达CD45RA的终分化EM细胞)T细胞表面GPR56表达基线(刺激前)已被报道在PMA和离子霉素(PMAI)刺激后产生更多的IFNγ。gydF4y2Ba24gydF4y2Ba.与此一致的是GPR56gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba我们数据中的EM细胞具有丰富的转录特征(来源于一项独立研究)gydF4y2Ba25gydF4y2Ba)标记CD8gydF4y2Ba+gydF4y2BaEM细胞在PMAI刺激后能够分泌更高水平的IFNγ(图。gydF4y2Ba3 dgydF4y2Ba).因此,GPR56gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaEM细胞可能是流感疫苗接种后COVR-M组IFNγ产量升高的一个来源。事实上,疫苗接种前,相对于HC-M和COVR-F组,这些细胞在COVR-M组中出现的频率有所升高。gydF4y2Ba3 egydF4y2Ba),但与TSD不相关,因此在时间上是稳定的(通过Spearman的相关性评估:gydF4y2BaPgydF4y2Ba= 0.18 (COVR-F)和gydF4y2BaPgydF4y2Ba= 0.51 (covr-m))。此外,gydF4y2BaIFNGgydF4y2BaCOVR-M组接种流感疫苗后,D1上这些细胞的转录物显著增加(扩展数据图)。gydF4y2Ba4 c, dgydF4y2Ba).这些数据表明,以前的COVID-19增加了GPR56的频率gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba男性个体中的EM细胞和这些细胞在流感疫苗接种后早期准备制造更多的IFNγ,这共同促成了COVR-M组中更高的IFNγ产量;与这一假设一致,在GPR56中没有观察到这种情况gydF4y2Ba−gydF4y2Ba单元(扩展数据图。gydF4y2Ba4 dgydF4y2Ba和补充图。gydF4y2Ba5度gydF4y2Ba).gydF4y2Ba
据报道,轻度、非住院的COVID-19可诱导CD8的旁观者激活(非sars - cov -2特异性)gydF4y2Ba+gydF4y2BaT细胞gydF4y2Ba26gydF4y2Ba.值得一提的是GPR56gydF4y2Ba+gydF4y2Ba细胞也富集了与旁观者T细胞激活相关的转录签名gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba(无花果。gydF4y2Ba3 fgydF4y2Ba).此外,GPR56gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaEM细胞频率与T细胞激活特征评分正相关,如上图所示,在COVR-M组中,T细胞激活特征评分基线时升高。gydF4y2Ba1我gydF4y2Ba和扩展数据图。gydF4y2Ba4 egydF4y2Ba).这表明,在感染的急性期,其中一些细胞可能以旁观者的方式扩张。这促使我们考虑这些GPR56gydF4y2Ba+gydF4y2Ba细胞类似于旁观者激活的虚拟内存(VM) CD8gydF4y2Ba+gydF4y2BaT细胞,其特征是它们能够在没有T细胞受体(TCR)刺激的情况下,单独被炎症细胞因子(例如IL-12, IL-18和IL-15)快速激活,产生IFNγgydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba.VM CD8gydF4y2Ba+gydF4y2BaT细胞通过细胞因子刺激而扩增,包括病毒感染诱导的IL-15(已知IL-15浓度在急性COVID-19患者中升高,并与疾病严重程度相关gydF4y2Ba30.gydF4y2Ba),以表达CD45RA的分化EM表型为特征gydF4y2Ba28gydF4y2Ba.我们评估了几种报道的这些细胞的表面标记物gydF4y2Ba28gydF4y2Ba在GPR56gydF4y2Ba+gydF4y2Ba与GPR56gydF4y2Ba−gydF4y2Ba发现GPR56gydF4y2Ba+gydF4y2Ba细胞表型确实与VM细胞相似(图。gydF4y2Ba3 ggydF4y2Ba).例如GPR56gydF4y2Ba+gydF4y2Ba与GPR56相比,细胞具有更高的CD122,但较低的CD5表面表达gydF4y2Ba−gydF4y2Ba同行;CD5表面表达与先前IL-15(或潜在的其他炎症细胞因子)相遇的程度有关gydF4y2Ba28gydF4y2Ba,gydF4y2Ba31gydF4y2Ba.值得注意的是,基于CD45RA和CD45RO的表面水平,GPR56gydF4y2Ba+gydF4y2Ba细胞表型位于GPR56之间gydF4y2Ba−gydF4y2Ba和TEMRA单元(扩展数据图。gydF4y2Ba4 fgydF4y2Ba).gydF4y2Ba
为了进一步验证我们的假设,我们进行了体外刺激实验,以评估GPR56gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaT细胞可以在疫苗接种或感染诱导的几种细胞因子的作用下产生IFNγ(补充图)。gydF4y2Ba6gydF4y2Ba).IL-15刺激显示GPR56gydF4y2Ba+gydF4y2BaCD45RAgydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba与COVR-F组相比,来自COVR-M组的T细胞产生了更多的IFNγ(图2)。gydF4y2Ba3 h,我gydF4y2Ba).CD8gydF4y2Ba+gydF4y2Ba使用表面标记CD45RA鉴定vm样T细胞gydF4y2Ba+gydF4y2Ba,吉珥gydF4y2Ba+gydF4y2Ba和/或NKG2AgydF4y2Ba+gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2BaCOVR-M组在这些细胞中产生了更高水平的IFNγ(图2)。gydF4y2Ba3我gydF4y2Ba).IL-12、IL-15和IL-18的刺激也显示出类似的趋势(补充图)。gydF4y2Ba6 bgydF4y2Ba).单独使用IL-18或IL-12和IL-18一起刺激也显示出类似的趋势,但这些条件诱导的IFNγ强度低于IL-15刺激(数据未显示)。接下来,我们使用CITE-seq数据评估了流感疫苗接种后IL-15的细胞来源,发现来自COVR-M组的经典单核细胞显示出最显著的增加gydF4y2BaIL15gydF4y2Ba流感疫苗接种后第1天mRNA水平(图;gydF4y2Ba3 jgydF4y2Ba).总之,这表明疫苗接种后COVR-M组IFNγ反应的增加可能归因于对炎症刺激本质上更敏感的细胞基线(疫苗接种前)频率的增加,包括产生IL-15和CD8升高的经典单核细胞gydF4y2Ba+gydF4y2Ba仅对细胞因子刺激产生更强IFNγ反应的vm样T细胞。gydF4y2Ba
由于VM T细胞可以快速激活产生细胞因子,无需克隆,抗原特异性扩增gydF4y2Ba28gydF4y2Ba,我们评估了GPR56的克隆性gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba利用CITE-seq的V(D)J/TCR数据分析流感疫苗接种后不同时间点的EM细胞。两个GPR56的克隆性gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaEM和TEMRA细胞在D0(疫苗接种前)、D1和D28(流感疫苗接种后)保持稳定(扩展数据图)。gydF4y2Ba4 g hgydF4y2Ba).GPR56的频率gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba个体内不同时间点共享的EM克隆也相似(扩展数据图。gydF4y2Ba4我gydF4y2Ba).总之,这些数据反驳了GPR56被高度激活的观点gydF4y2Ba+gydF4y2Ba在COVR-M组中,流感疫苗接种后早期细胞的增加仅仅是由于tcr依赖的T细胞激活和克隆扩增。如前所述gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba和以上的体外刺激数据,一个更合理的解释是这些CD8gydF4y2Ba+gydF4y2Ba流感疫苗诱导的炎症细胞因子以抗原不依赖的方式激活vm样细胞产生IFNγ。尽管它们与VM细胞相似,但一些GPR56gydF4y2Ba+gydF4y2Ba细胞可以通过传统的非旁观者途径从原始细胞发育而来(例如,一些细胞可以在急性COVID-19期间发育,并且对SARS-CoV-2具有特异性),尽管这些细胞中没有一个CDR3序列与被认为对SARS-CoV-2具有特异性的公共克隆细胞相匹配(数据未显示)。真正的抗原特异性记忆CD8gydF4y2Ba+gydF4y2Ba在小鼠中,通过TCR刺激从原始细胞发育而来的T细胞也被证明能单独对炎症细胞因子产生IFNγgydF4y2Ba34gydF4y2Ba,gydF4y2Ba35gydF4y2Ba.gydF4y2Ba
我们的数据还揭示了其他类型的细胞可能有助于在接种COVR-M组疫苗后在D1上观察到IFNγ产量的增加(补充图。gydF4y2Ba7得了gydF4y2Ba).gydF4y2BaIFNGgydF4y2BaCD16 D1位点COVR-M组的转录本明显高于HC-M组和COVR-F组gydF4y2Ba低gydF4y2Ba自然杀伤细胞(NK)gydF4y2Ba7 cgydF4y2Ba及补充表gydF4y2Ba4 gydF4y2Ba).此外,CD16的基线频率gydF4y2Ba低gydF4y2BaNK细胞与两者D1的增加程度相关gydF4y2BaIFNGgydF4y2Ba表达和血清蛋白水平(补充图;gydF4y2Ba7 bgydF4y2Ba).然而,在COVR-M组中,IL-15体外刺激后总NK细胞中的IFNγ反应并没有显著提高(扩展数据图)。gydF4y2Ba4 jgydF4y2Ba),可能是因为CD16gydF4y2Ba低gydF4y2BaNK细胞是NK细胞总数的一小部分。相比之下,体外IL-15刺激显示,与COVR-F和HC-M组相比,COVR-M组MAIT细胞中的IFNγ反应更高(扩展数据图)。gydF4y2Ba4 jgydF4y2Ba),但gydF4y2BaIFNGgydF4y2Ba根据CITE-seq数据,流感疫苗接种后COVR-M组D1上的mRNA表达无统计学意义(补充图gydF4y2Ba7 cgydF4y2Ba).CD8gydF4y2Ba+gydF4y2Ba带有TEMRA的T细胞(CD45RAgydF4y2Ba+gydF4y2BaCD45ROgydF4y2Ba−gydF4y2BaCCR7gydF4y2Ba−gydF4y2Ba)的表型可能也有作用,因为在体外IL-15刺激后,COVR-M组的IFNγ反应高于COVR-F和HC-M组(扩展数据图)。gydF4y2Ba4 jgydF4y2Ba),与CITE-seq数据一致(补充图。gydF4y2Ba7 cgydF4y2Ba).gydF4y2Ba
综上所述,我们证明了CD8的种群gydF4y2Ba+gydF4y2Ba异种疫苗接种后,GPR56表达标记的EM T细胞和具有抗原不可知促炎潜能的vm样标记。重要的是,这些细胞,可能还有CD16gydF4y2Ba低gydF4y2BaNK, MAIT和CD8gydF4y2Ba+gydF4y2BaTEMRA细胞(尽管我们的CITE-seq数据支持较少)出现在其他临床健康的个体中,并且在从轻度SARS-CoV-2感染中恢复数月的男性个体中尤其升高,更容易做出反应,这为与先前轻度COVID-19相关的性别特异性、功能相关的免疫设定点提供了额外的证据。gydF4y2Ba
接种疫苗改变单核细胞印迹gydF4y2Ba
考虑到疫苗诱发的训练效果gydF4y2Ba6gydF4y2Ba,gydF4y2Ba36gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,我们接下来研究了流感疫苗是否可以改变我们之前检测到的一些covid -19后转录印迹(图2)。gydF4y2Ba4gydF4y2Ba).由于上述报告的IIR特征明显降低,我们将重点放在单核细胞上(在从COVID-19康复的参与者与健康对照参与者中;无花果。gydF4y2Ba1 f, ggydF4y2Ba),因为疫苗可能会在这些细胞中诱导持久的变化gydF4y2Ba6gydF4y2Ba,gydF4y2Ba36gydF4y2Ba.使用健康对照基线(D0)作为健康参考,我们使用CITE-seq数据评估从COVID-19康复的参与者在接种疫苗前后的签名基因(如上所述)的平均表达,分别用于经典(图0)。gydF4y2Ba1 fgydF4y2Ba)和非经典单核细胞(图;gydF4y2Ba1克gydF4y2Ba)在男性和女性个体中(扩展数据图。gydF4y2Ba5 a、bgydF4y2Ba).如上所述,与接种疫苗前D0时的健康对照组相比,从COVID-19康复的参与者中,这些基因的平均表达水平较低。然而,在COVR-F组和COVR-M组中,它们的平均表达量在D1时向健康对照个体增加,并持续到D28,尽管在COVR-F组中这种效应似乎更强(扩展数据图。gydF4y2Ba5 a、bgydF4y2Ba).gydF4y2Ba
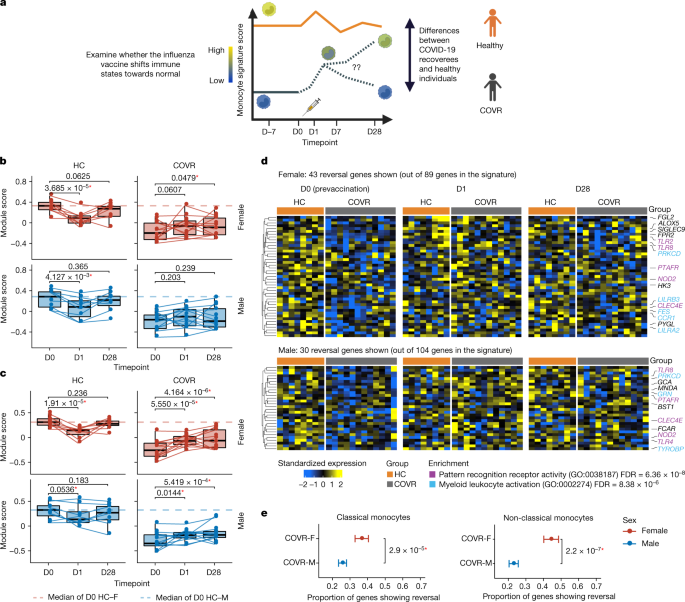
一个gydF4y2Ba,研究问题的示意图。gydF4y2BabgydF4y2Ba, IIR签名的模块得分(图;gydF4y2Ba1 fgydF4y2Ba)在HC-F (gydF4y2BangydF4y2Ba= 8), hc-m (gydF4y2BangydF4y2Ba= 8), covr-f (gydF4y2BangydF4y2Ba= 12)和COVR-M (gydF4y2BangydF4y2Ba= 12)组在D0, D1和D28使用CITE-seq假体基因表达数据在经典单核细胞。虚线表示的是同性别健康对照个体D0得分的中位数。这些线连接来自同一参与者在不同时间点的数据点。使用考虑年龄、种族和流感疫苗接种史的混合效应模型确定差异的统计学意义(gydF4y2Ba方法gydF4y2Ba).未经调整的gydF4y2BaPgydF4y2Ba数值会显示出来。gydF4y2BacgydF4y2Ba,类似于gydF4y2BabgydF4y2Ba,但对于非经典单核细胞(图;gydF4y2Ba1克gydF4y2Ba).gydF4y2BadgydF4y2Ba,经典单核细胞中逆转基因表达的热图(行标准化;参见扩展数据图。gydF4y2Ba5度gydF4y2Ba对于非经典单核细胞)。逆转基因被定义为基线IIR特征中的基因(图2)。gydF4y2Ba1 fgydF4y2Ba),其中在接种疫苗后D1和D28时从COVID-19中恢复的参与者的表达向健康对照组的基线(疫苗接种前)表达移动。COVR-F组(上)和COVR-M组(下)分别显示;健康对照个体也被纳入比较。行是基因,列是个体样本(按参与者/时间点分组),时间点和参与者组标签显示在顶部,包括与中相同的参与者gydF4y2BabgydF4y2Ba在每个时间点。显示了属于功能感兴趣的基因集的基因名称。False-discovery-rate-corrected浓缩gydF4y2BaPgydF4y2Ba数值会显示出来。gydF4y2BaegydF4y2Ba, IIR特征基因比例比较(图;gydF4y2Ba1 f, ggydF4y2Ba),在经典和非经典单核细胞中,COVR-F组与COVR-M组显示部分逆转。平均置信区间和95%置信区间(由柱状表示)来自自举过程(gydF4y2Ba方法gydF4y2Ba).使用双尾Wilcoxon检验来确定自举样本之间的显著性。所有的箱形图都显示了中位数(中心线),第一和第三分位数(盒限),以及每个方向(上下须)盒限的最大1.5 × IQR。重要的(gydF4y2BaPgydF4y2Ba< 0.05)的差异用红色星号(*)突出显示。图中gydF4y2Ba一个gydF4y2Ba是使用BioRender创建的。gydF4y2Ba
量化这些性别和细胞类型依赖的基因集的平均表达(模块得分)(图。gydF4y2Ba1 f, ggydF4y2Ba)随着时间的推移,在个体参与者中证实了向健康对照个体转移的类似而显著的趋势(图。gydF4y2Ba4 b, cgydF4y2Ba).该分析进一步表明,基因表达的这种变化在非经典单核细胞中比在经典单核细胞中更为明显(图2)。gydF4y2Ba4 b, cgydF4y2Ba).值得注意的是,这些基因在健康对照个体中的行为是不同的——基因模块得分在D1上呈下降趋势,并在健康对照个体中于28天恢复到疫苗接种前的水平(图2)。gydF4y2Ba4 b, cgydF4y2Ba).尽管这种差异的潜在机制尚不清楚,但健康对照个体中的单核细胞可能通过以负前馈机制下调某些免疫受体基因和相关信号基因来避免过度反应,从而对疫苗诱导的炎症做出反应。而从COVID-19中恢复的参与者体内的“抑郁”单核细胞则通过增加这些基因的表达来做出反应,从而向正常(健康基线)水平移动。gydF4y2Ba
接下来,我们确定了这些基因集中向健康对照基线移动的单个基因(gydF4y2Ba方法gydF4y2Ba).在经典和非经典单核细胞中,与男性参与者相比,女性的还原基因比例显著更高(图2)。gydF4y2Ba4 d, egydF4y2Ba和扩展数据图。gydF4y2Ba5度gydF4y2Ba),虽然有几个tlr(例如,gydF4y2BaTLR2gydF4y2Ba而且gydF4y2BaTLR4gydF4y2Ba),gydF4y2BaNOD2gydF4y2Ba在两性的一个或两个单核细胞亚群中均显著。这些变化可能不是由于感染后持续的免疫消退,因为这些基因的基线(D0)表达与TSD无关(扩展数据图)。gydF4y2Ba1 hgydF4y2Ba),且在接种后D1急剧增加,并持续到D28。值得注意的是,与这种抑制的IIR特征相反(图。gydF4y2Ba1 f, ggydF4y2Ba和扩展数据图。gydF4y2Ba1我gydF4y2Ba),其他已知在急性covid -19期间表达较低的单核细胞相关转录标记,如与抗原提呈、炎症和NF-κB激活以及髓系抑制细胞相关的基因gydF4y2Ba13gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba-在D0/基线时,从COVID-19康复的参与者与健康对照组之间相似;尽管COVR-M组在D1上的非经典单核细胞中抗原递呈转录反应升高,但在D28上仍保持轻度升高,但疫苗接种也没有始终引起这些签名到D28的更持久的变化(扩展数据图)。gydF4y2Ba5 d, egydF4y2Ba).gydF4y2Ba
总之,CITE-seq分析显示,对流感疫苗的早期(D1)反应使从COVID-19康复的参与者单核细胞中一组先前(即接种疫苗前)抑制的IIR基因在接种疫苗后至少升高到D28。尽管这些变化的功能相关性仍有待确定,但这些结果表明,流感疫苗接种的早期炎症反应有助于将单核细胞的covid -19后免疫状态转变为健康个体的免疫状态,特别是女性恢复者。gydF4y2Ba
讨论gydF4y2Ba
尽管有报道称COVID-19住院患者出现急性和长期免疫紊乱gydF4y2Ba13gydF4y2Ba,gydF4y2Ba40gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba,gydF4y2Ba43gydF4y2Ba对于急性发病后几个月有轻度、未住院的SARS-CoV-2感染、无肥胖、自身免疫或免疫缺陷等混杂合并症的健康康复患者,我们所知较少。在这里,我们揭示了先前未住院的COVID-19临床健康康复患者除了具有SARS-CoV-2特异性免疫外,还具有性别特异性免疫印迹,其中一些仅在接种与SARS-CoV-2不同的抗原后才变得明显。我们的发现与SARS-CoV-2和其他免疫挑战急性反应的性别二态性质一致gydF4y2Ba11gydF4y2Ba.健康女性个体对感染和疫苗的炎症反应往往会增强gydF4y2Ba44gydF4y2Ba;因此,令人惊讶的是,在这里发现质的相反,发现COVR-M在基线时具有更稳定的免疫状态,并且对流感疫苗有更强的先天和适应性反应。尽管有报道称长冠患者的免疫状态持续变化(持续数月)gydF4y2Ba41gydF4y2Ba,我们研究中的大多数人报告没有或轻微的covid -19后后遗症。未来的研究可以评估一些性别特异性印迹,包括疫苗接种反应的差异,是否与长COVID有关gydF4y2Ba7gydF4y2Ba.gydF4y2Ba
我们的研究结果表明,在COVR-M组中,稳定的基线免疫状态有助于在流感疫苗接种后的第1天、第7天和第28天分别建立更强大的IFN、浆母细胞和抗体反应。早期IFN反应可能是由于单核细胞具有较高的表达gydF4y2BaIL15gydF4y2Ba接种疫苗后早期的转录反应加上接种前vm样CD8频率的升高gydF4y2Ba+gydF4y2Ba在IL-15刺激后,T细胞会产生更多的IFNγ。我们描述的单核细胞印迹包括平稳gydF4y2BaIL15gydF4y2Ba男性的mRNA产量恢复,两性中转录抑制的先天受体基因签名与训练过的先天免疫的概念一致gydF4y2Ba6gydF4y2Ba.值得注意的是,尽管后一种特征可以在急性COVID-19重症患者中检测到,但它不同于以往急性COVID-19研究中发现的抗原提呈抑制或骨髓抑制细胞样状态。gydF4y2Ba13gydF4y2Ba,gydF4y2Ba15gydF4y2Ba,gydF4y2Ba16gydF4y2Ba,gydF4y2Ba17gydF4y2Ba,gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba).由于训练有素的先天免疫可以通过多种机制介导,包括细胞内的染色质和代谢变化,未来的研究可以探索单核细胞中的这些潜在机制,包括性别/性别、急性疾病严重程度和年龄对患有一系列covid -19后临床后遗症的参与者的影响。鉴于循环单核细胞的半衰期相对较短(可短于28天)gydF4y2Ba45gydF4y2Ba,我们检测到的部分逆转可能归因于骨髓髓样祖细胞,因为在SARS-CoV-2感染后,造血干细胞和祖细胞显示出染色质可及性变化gydF4y2Ba46gydF4y2Ba.gydF4y2Ba
在自然病毒感染后,旁观者T细胞活化已被报道gydF4y2Ba47gydF4y2Ba,包括SARS-CoV-2(参考。gydF4y2Ba26gydF4y2Ba).最近,旁观者激活CD8gydF4y2Ba+gydF4y2BaEM T细胞已被确定在控制早期感染中发挥重要作用,包括先前没有抗原暴露或TCR接触的VM细胞gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba.由于这些细胞可以在单独的细胞因子刺激后出现,因此在急性疾病期间,男性相对于女性个体对SARS-CoV-2的细胞因子反应更强或更持久可能导致GPR56频率升高gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaCOVR-M组的vm样细胞。这一假设与报告一致,即与女性个体相比,因COVID-19住院的男性个体往往经历更大的先天免疫激活(通过循环细胞因子测量)gydF4y2Ba48gydF4y2Ba,gydF4y2Ba49gydF4y2Ba.gydF4y2Ba
我们观察到的一些免疫印迹可能在不同类型的病毒感染中共享,但根据我们与自然流感感染的比较,有些可能是SARS-CoV-2所特有的。我们的研究结果指出,任何感染或免疫挑战都可能改变免疫状态,从而建立新的基线设定值,这些设定值不仅由单细胞谱系的状态编码,而且由VM T细胞和单核细胞等相互作用的细胞类型网络编码。此外,虽然预测未来反应的基线免疫状态在个体之间往往是不同的,并且在几个月的时间尺度内是稳定的gydF4y2Ba50gydF4y2Ba,gydF4y2Ba51gydF4y2Ba我们的研究结果表明,这种基线免疫状态可能是由过去的感染建立的,并且在下一次扰动之前是稳定的。因此,个体的基线免疫状态是由大量以前的暴露所决定的,它可能以抗原特异性和抗原不可知的方式影响未来的反应gydF4y2Ba2 gydF4y2Ba,gydF4y2Ba3 gydF4y2Ba.除了揭示关于两次明确的自然免疫相遇(轻度COVID-19和人类流感疫苗接种)后发生的情况的基本原则外,我们的观察还为研究更复杂的情况提供了基础,例如在更长的时间范围内发生额外的炎症相遇。我们的研究提出了一个概念,即即使是轻微的病毒感染也可以建立新的免疫设定值,以抗原不可知的方式影响未来的免疫反应,并说明了如何将异体疫苗接种作为揭示这种功能印迹的工具。gydF4y2Ba
方法gydF4y2Ba
患者群体和样本收集gydF4y2Ba
在2020年8月至12月期间,从当地(马里兰州、弗吉尼亚州和哥伦比亚特区)招募了至少18岁的参与者,并参加了美国国立卫生研究院(NIH)的19-I-0126方案(季节性流感疫苗免疫反应的系统分析)。这项研究得到了NIH机构审查委员会的批准(ClinicalTrials.gov:gydF4y2BaNCT04025580gydF4y2Ba),并遵守所有有关的道德守则。获得所有参与者的知情同意。在获得知情同意后,进行基线病史和体格检查。参与者被要求描述过去SARS-CoV-2感染的任何当前持续症状。排除标准包括肥胖(BMI≥30);自身免疫、自身炎症或免疫缺陷疾病病史或疑似病史;过去30天内(减毒活疫苗)或14天内(非减毒活疫苗)接种过任何疫苗;任何实验性疫苗的历史;过去一年有寄生虫、阿米巴、真菌或分枝杆菌感染史;或者是当前感染。 The COVID-19 vaccine was not available at the time of the study, and no study participants participated in any COVID-19 vaccine trials. All study visits occurred at the NIH Clinical Center (CC) in Bethesda, MD, USA. Blood samples were collected by phlebotomy staff at the NIH CC. The samples were collected between September 2020 and April 2021. No sample size calculations were done prior to enrolment, in part because there were no reliable effect size estimates related to the impact of prior COVID-19 infection on vaccine responses. The number of subjects in the study was the number that were able to be recruited during the recruitment period.No blinding or randomization was performed.
从三组参与者中收集样本:(1)既往有症状性SARS-CoV-2感染史(定义为在方案筛查时鼻腔PCR检测阳性和美国食品和药物管理局(FDA)紧急使用授权(EUA) SARS-CoV-2抗体检测阳性);2)有无症状SARS-CoV-2感染史(定义为在方案检查时使用FDA EUA SARS-CoV-2抗体检测呈阳性,但无covid - cov -2样症状史;这组患者没有确诊COVID-19感染或诊断的时间,他们被排除在所有TSD分析之外);(3)无SARS-CoV-2感染史(在方案筛查时,FDA EUA SARS-CoV-2抗体检测为阴性)。gydF4y2Ba
在与季节性流感疫苗接种相关的以下每个时间点(第0天)收集外周血单个核细胞(PBMCs)、血清、全血RNA (Tempus血液RNA管,赛默飞世尔科学公司)、全血计数(CBC)和淋巴细胞表型:−7天、0天、1天、7天、14天、28天、70天和100天。分别于第0天、第28天和第100天采集粪便样本。为参与者提供了Cardinal Health粪便收集包(Cardinal Health)和带冰袋的泡沫聚苯乙烯储存容器,以便在家中收集粪便样本,并亲自返回NIH。在第100天之后,参与者可以选择继续每月提供pbmc、血清、全血RNA、差异和淋巴细胞表型的CBC血液样本,直到2021年8月。gydF4y2Ba
在研究登记后的每个时间点,使用NIH托管的REDCap (v.8.5.27)电子数据捕获工具收集和管理数据gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba.REDCap(研究电子数据采集)是一个安全的基于web的软件平台,旨在支持研究数据采集,提供:(1)验证数据采集的直观界面;(二)跟踪数据操纵和导出程序的审计跟踪;(3)自动导出程序,无缝地将数据下载到通用统计包;(4)与外部数据源进行数据集成和互操作的程序。通过两项独立的irb批准的调查,使用REDCap电子问卷收集参与者的信息。在研究第1天和第7天进行了一项评估疫苗相关不良事件或症状的调查,并在第0天开始的每次随访中进行了一项评估任何健康变化或新药的单独调查。调查通过电子邮件发送给参与者,研究团队将回答从REDCap系统转移到NIH临床研究信息管理系统(CRIMSON)系统。gydF4y2Ba
接种流感疫苗gydF4y2Ba
年龄在18岁至64岁之间的参与者接种了氟克斯四价季节性流感疫苗(2020-2021年;Seqirus)。65岁及以上的参与者接受高剂量Fluzone四价季节性流感疫苗(2020-2021年;赛诺菲巴斯德)。gydF4y2Ba
流感微中和滴度gydF4y2Ba
基于美国疾病控制和预防中心(CDC)大流行性流感参考实验室的方法,使用低致病性疫苗病毒和MDCK细胞,采用微中和法测定了接种前和接种后血清的病毒中和滴度。X‐179A病毒是一种5:3重组疫苗gydF4y2Ba哈gydF4y2Ba,gydF4y2BaNAgydF4y2Ba而且gydF4y2BaPB1gydF4y2Ba来自A/California/07/2009 (H1N1pdm09)的基因和来自A/PR/8/34的其他5个基因由高生长病毒nyymc X‐157捐献。此外,还检测了季节性疫苗株H1N1 A/Brisbane/59/07、H3N2 A/乌拉圭/716/07和B/Brisbane/60/2001的免疫血清中和滴度。所有分析的内部对照都是由FDA生物制剂评估和研究中心针对相应菌株生成的羊血清。所有个体血清均连续稀释(从1:10开始稀释两倍),并在96孔板(1:1混合)中对每种菌株的100中位组织培养感染剂量进行重复检测。滴度代表完全抑制病毒复制的最高稀释度。gydF4y2Ba
SARS-CoV-2假病毒生产及中和试验gydF4y2Ba
人sars - cov - 2s糖蛋白编码密码子优化cDNA (GenBank:gydF4y2BaNC_045512gydF4y2Ba)克隆到真核细胞表达载体pcDNA 3.1中,表达在BamHI和XhoI位点之间。使用Lipofectamine 3000将Lenti‐X 293T细胞与psPAX2(gag/pol)、pTrip-luc慢病毒载体和pcDNA 3.1 SARS-CoV-2-spike-deltaC19共转染产生假病毒粒子。转染后48小时收集上清液,通过0.45µm膜过滤,并用293T-ACE2细胞(表达ACE2蛋白的HEK293T细胞)滴定。通过BEI Resources, NIAID, NIH获得以下试剂:表达人血管紧张素转换酶2的人胚胎肾细胞(HEK293T), HEK293T- hace2细胞系,NR-52511。gydF4y2Ba
为了进行中和试验,将50 μ l SARS-CoV-2 S假病毒粒子与等体积的不同稀释度的血清在室温下预孵育1小时,然后将病毒-抗体混合物添加到96孔板中的293T-ACE2细胞中。培养3 h后,将接种物替换为新鲜培养基。24小时后,细胞裂解,荧光素酶活性测定如前所述gydF4y2Ba56gydF4y2Ba,gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba.对照组包括只有细胞的对照组、没有任何抗体的病毒对照组和阳性对照血清。Lenti‐X 293T细胞来自Takara Bio (Cat。632180号)。293T-ACE2细胞来源于ATCC。对293T-ACE2细胞进行ACE2表达检测,并通过FACS分析进行验证。这两种细胞系都没有通过核型分析或其他基因组技术进行鉴定。两种细胞系检测均为阴性gydF4y2Ba支原体gydF4y2Ba.gydF4y2Ba
基于spr的人血清抗体结合动力学gydF4y2Ba
如前所述,使用ProteOn表面等离子体共振(BioRad)系统在25°C下监测血清的稳态平衡结合gydF4y2Ba59gydF4y2Ba,gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba.纯化的重组SARS-CoV-2或其他蛋白被捕获到Ni-NTA传感器芯片(BioRad, 176-5031),在测试流通道中具有200个共振单元(RU)。芯片上的蛋白质密度被优化,如测量独立于抗体同型的单价相互作用。以50µl min的流速将新制备的样品连续稀释(10倍、30倍和90倍)于BSA- pbst缓冲液中(PBS pH 7.4缓冲液,含有Tween-20和BSA)gydF4y2Ba−1gydF4y2Ba(120秒接触时间)进行关联,间隔600秒进行分离。来自蛋白质表面的响应被校正为来自模拟表面的响应和来自仅缓冲的注射的响应。使用BioRad ProteOn管理软件计算总抗体结合(v.3.1)。所有的SPR实验都进行了两次,并且进行分析的研究人员对样品的身份不知情。在这些优化的SPR条件下,每个样品在重复SPR运行中的变化<5%。图中显示的最大共振单位(max RU)数据是10倍稀释血清样品的RU信号。gydF4y2Ba
PBMC隔离gydF4y2Ba
PBMC样本是从Vacutainer EDTA管(通用实验室供应商)中使用sepate -50管(STEMCELL Technologies)收集的血液中分离出来的,并对制造商的方案进行了以下修改:血液样本用室温PBS 1:1稀释,并通过移液混合。稀释后的血液分层在SepMate试管中15 ml Cytiva Ficoll PAQUE-Plus (Cytiva Life Sciences)层上。SepMate试管在1200度离心gydF4y2BaggydF4y2Ba在室温下制动设置为5,10分钟。离心后,在不干扰PBMC层的情况下,尽可能去除顶层等离子体层。如果有任何细胞粘在试管壁上,就用移液管将它们从试管壁上轻轻刮下来,这样它们就可以和其他细胞一起重新悬浮。细胞从SepMate试管中倒入50毫升锥形试管中。将含有细胞的试管用冷洗缓冲液(PBS加2% FBS)填充至50 ml,并通过倒置混合。试管在300度离心gydF4y2BaggydF4y2Ba在室温下制动设置为5,10分钟。离心后,在不干扰细胞颗粒的情况下,去除上清液。用冷洗缓冲液重新悬浮颗粒后,使用Guava Muse细胞分析仪(Luminex)对细胞进行计数。试管再次在300度下离心gydF4y2BaggydF4y2Ba在室温下制动设置为5,10分钟。在不干扰细胞颗粒的情况下,去除上清液。gydF4y2Ba
在细胞计数的基础上,每个样品的每瓶都冷冻了600 - 1000万个pbmc。由于在最后一次离心前对细胞进行计数,假设细胞损失为50%,并在细胞计数计算中予以考虑。细胞颗粒重悬gydF4y2BangydF4y2Ba× 600µl(其中gydF4y2BangydF4y2Ba为待冷冻的PBMC瓶数)冷冻介质(RPMI + 10% FBS)通过温和移液。冷冻介质后,gydF4y2BangydF4y2Ba滴加× 600 μ l DMSO冷冻液(含15% DMSO的FBS),同时轻轻摇动试管。换句话说,对于每一瓶待冷冻的PBMC,加入600µl的冷冻介质和600µl的DMSO冷冻液,使每瓶的总体积达到1.2 ml。在将1.2 ml细胞溶液转移到每个1.8 ml冷冻瓶(一般实验室供应商)之前,通过移液轻柔地混合溶液。细胞瓶被放置到CoolCell容器(Thomas Scientific)中,容器被放置到−80°C的冰箱中。至少4小时后,PBMC小瓶转移到液氮中。gydF4y2Ba
RNA隔离gydF4y2Ba
血液被直接抽到Tempus血液RNA管(赛默飞世尔科学公司)根据制造商的协议。在每个研究时间点收集两个Tempus管。从每个Tempus管中提取的血液样本被倒入两个4.5 ml低温瓶(一般实验室供应商)。这些冷冻瓶直接储存在−80°C。gydF4y2Ba
RNA样本在分离前经过仔细的批处理,每批12-22个样本进行分离,以减少年龄、性别和患者群体的混杂因素。gydF4y2Ba
使用QIAsymphony RNA Kit (Qiagen)和QIAsymphony SP仪器(Qiagen)从Tempus管中的血液中分离RNA。血液样本在冰上解冻,然后将每个样本转移到一个50毫升的锥形管中。加入1× PBS使样品总体积达到12 ml。管子全速旋转30秒,然后在3500下离心gydF4y2BaggydF4y2Ba在4°C下放置1小时。离心后,将试管中的上清液滗出,并将试管倒置放在干净的纸巾上2分钟,使残留液体排出。为了重新悬浮颗粒,将800 μ l RLT+缓冲液添加到每个管的底部,并旋转几秒钟。所有800 μ l的每个样品转移到2 ml螺旋盖管(Sarstedt)。这些管被放置在#3b适配器(Qiagen)中,并加载到QIAsymphony系统上。gydF4y2Ba
在QIAsymphony系统上,选择了RNA CT 800协议进行RNA分离。仪器按照制造商的协议设置,RNA样品的洗脱量设置为100µl。洗脱RNA样品的最终体积为65 μ l至95 μ l。gydF4y2Ba
RNA产量根据产量使用Qubit RNA BR试剂盒或Qubit RNA HS试剂盒(赛默飞世尔科学公司)测定。RNA RIN数使用RNA screenentape (Agilent Technologies)测量。平均RIN为8.3,平均产率为81.3 ngµlgydF4y2Ba−1gydF4y2BaRNA样本。gydF4y2Ba
RNA-seqgydF4y2Ba
RNA-seq文库根据制造商的协议,使用Universal Plus mRNA-Seq与NuQuant, Human Globin AnyDeplete (Tecan Genomics)手动制备。对于每个样本,500ng总RNA用于poly(A)选择分离mRNA。将捕获的mRNA清洗、片段化,并用随机和寡核苷酸(dT)引物混合引物进行引物。cDNA合成后,用独特的双指数适配器对修复和结扎末端。使用AnyDeplete模块去除rRNA、mtRNA和球蛋白中多余的大量转录本。其余文库经14次PCR扩增,用AMPure XP试剂(Beckman Coulter)纯化。gydF4y2Ba
文库浓度使用定量it PicoGreen dsDNA检测试剂盒(Thermo Fisher Scientific)在BioTek Synergy H1平板阅读器(BioTek Instruments)上使用2 μl样品进行测定。在4200 TapeStation系统(安捷伦科技)上使用D1000 ScreenTape(安捷伦科技)确定库大小分布。从每个板中随机选取32个样本,测量文库大小分布。为了确定片段大小,电泳图上的区域设置为200 bp到700 bp。其余库使用片段大小的平均值来计算物质的量浓度。gydF4y2Ba
为了创建一个平衡的测序池,使用QIAgility液体处理机器人(Qiagen)将一个培养皿中的所有文库稀释到相同的摩尔浓度,并将等量的归一化样品混合。从板1-4的每个板中总共汇集了96个样本,从板5中汇集了35个样本。为了准确地定量池文库,使用KAPA文库定量试剂盒(Roche)进行定量PCR。gydF4y2Ba
所有文库都在美国国家癌症研究所癌症研究测序中心的NovaSeq 6000仪器(Illumina)上进行测序。使用NovaSeq 6000 S4 Reagent Kit(200个循环)和NovaSeq XP 4-Lane Kit (Illumina)对1-4板的文库进行测序,测序参数为100 bp对端reads。使用NovaSeq 6000 SP Reagent Kit(300循环;Illumina),以150 bp配对末端reads作为测序参数。gydF4y2Ba
此外,经过质量控制,11个样品在NextSeq 500仪器上以100 bp配对末端reads为测序参数,使用NovaSeq 6000 S4 Reagent Kit(200个周期)作为第6板进行重测序。在每个板上放置技术重复,以控制板的变异性。gydF4y2Ba
CITE-seqgydF4y2Ba
单单元CITE-seq处理gydF4y2Ba
冷冻的PBMC样品用RPMI培养基(含10% FBS和10 mg ml)解冻、回收和洗涤gydF4y2Ba−1gydF4y2BaDNase I (STEMCELL),然后像前面描述的那样进行处理gydF4y2Ba15gydF4y2Ba用于CITE-seq染色。简而言之,来自不同捐赠者的样本被汇集起来,来自同一捐赠者的不同时间点被分别汇集起来,这样每个池只包含来自一个捐赠者的一个时间点。PBMC池Fc阻断(Human TruStain FcX, BioLegend),用Totalseq-C人类“标签”抗体(BioLegend)染色,用CITE-seq染色缓冲液(2% BSA在PBS中)洗涤。然后结合标签PBMC池,细胞用含有137个表面蛋白的TotalSeq-C人冻干板(BioLegend)鸡尾酒染色(包括7个同型对照;补充表gydF4y2Ba11gydF4y2Ba)和sars - cov - 2s1蛋白探针。然后将细胞清洗,在PBS中重悬并计数,然后立即进行单细胞分割步骤。gydF4y2Ba
单细胞CITE-seq文库构建与测序gydF4y2Ba
如前所述,使用10x 5 '铬单细胞免疫分析Next GEM v2化学试剂盒(10x Genomics)将PBMC样品分割成乳状液中的单细胞凝胶珠(GEM)与逆转录混合物混合gydF4y2Ba15gydF4y2Ba.反转录步骤在Veriti Thermal Cycler (Thermo Fisher Scientific)中进行。单细胞基因表达、细胞表面蛋白、T细胞受体(TCR)和B细胞受体(BCR)文库根据10x Genomics用户指南制备(gydF4y2Bahttps://www.10xgenomics.com/resources/user-guides/gydF4y2Ba).所有文库都使用Bioanalyzer(安捷伦)进行质量控制,并使用Qubit荧光定量(赛默飞世尔科学公司)进行量化。10x Genomics 5 '单细胞基因表达、细胞表面蛋白标签、TCR和BCR文库在Illumina NovaSeq平台(Illumina)上进行合并和测序,使用以下测序参数:read1-100 cycle, i7-10-, i5-10, read2-100。gydF4y2Ba
血清分离和蛋白质特性gydF4y2Ba
直接在血清分离管中收集血清,在室温下至少凝固30分钟。在采集血液2小时内,将管子离心1800gydF4y2BaggydF4y2Ba室温10分钟。使用移液管去除顶部(血清)层,并在−80°C下存储在单个小瓶中。使用Olink Target 96免疫肿瘤学和Olink Target 96炎症组(Olink Proteomics, Uppsla,瑞典)分析血清蛋白,每个组包含92种蛋白质,并使用基于接近延伸试验的方法。数据以归一化蛋白表达(NPX)单位报告。gydF4y2Ba
CBCs和淋巴细胞表型gydF4y2Ba
对于参与者,标准全血细胞计数与差异(CBCs)在NIH实验室医学中心进行。淋巴细胞(T细胞、B细胞、NK细胞)流式细胞术定量采用BD FACSCanto II流式细胞仪(BD Biosciences)。gydF4y2Ba
体外刺激PBMCgydF4y2Ba
将pmcs解冻并在含有10%胎牛血清、2 mM谷氨酰胺、0.055 mM β -巯基乙醇、1%青霉素-链霉素、1 mM丙酮酸钠、10 mM HEPES和1%非必需氨基酸的PRMI1640中培养,并在以下条件下刺激:(1)IL-15 (10 ng mlgydF4y2Ba−1gydF4y2Ba), IL-12 (20 ng mlgydF4y2Ba−1gydF4y2Ba), IL-18 (20 ng mlgydF4y2Ba−1gydF4y2Ba) 48小时;(2) IL-15 (50 ng mlgydF4y2Ba−1gydF4y2Ba) 48小时;(3) IL-18 (50 ng mlgydF4y2Ba−1gydF4y2Ba) 48小时;(4) IL-12 (20 ng mlgydF4y2Ba−1gydF4y2Ba), IL-18 (20 ng mlgydF4y2Ba−1gydF4y2Ba) 48小时;(5) anti-CD3 (1 μg mlgydF4y2Ba−1gydF4y2Ba), anti-CD28 (1 μg mlgydF4y2Ba−1gydF4y2Ba) 24小时;(6)非刺激对照组。在收集前4小时添加蛋白转运抑制剂(BD Biosciences, 554724)和Brefeldin A (BFA, Invitrogen, 00-4506-51)。以下细胞因子从BioLegend购买:IL-15 (570304), IL-12(573004)和IL-18(592104)。gydF4y2Ba
流式细胞术gydF4y2Ba
B细胞表型面板,包括流感HA探针gydF4y2Ba
解冻的pmcs在含50 U ml的RPMI培养基中洗涤gydF4y2Ba−1gydF4y2Ba再用PBS冲洗。细胞用活/死固定蓝染料(Life Technologies)孵育,用于排除死亡细胞的分析。细胞在4°C黑暗环境下孵卵30分钟,孵卵物为B型流感的荧光色素偶联HAs (B/Washington/02/2019和B/Phuket/3073/2013结合在同一荧光色素上)、甲型流感H1 (A/Hawaii/70/2019)和H3 (A/Hongkong/2671/2019)以及抗IgM、IgA、CD21、CD85J、FCRL5、CD20、IgG、CD38、CD14、CD56、CD3、CD27、CD71、CD19和IgD的荧光色素偶联抗体。研究小组(Sarah Andrews, National Institute of Allergy and Infectious Diseases, NIH, Vaccine Research Center, Vaccine Research Center)中的染料和抗体的详细信息汇总在补充表中gydF4y2Ba12gydF4y2Ba.抗体孵育30分钟后,细胞用FACS缓冲液(0.1% BSA/PBS (pH 7.4))洗涤两次,并固定在1%多聚甲醛中。Cytek Aurora光谱学细胞仪(Cytek Biosciences;SpectroFlo (v.2.2.0))。数据分析使用FlowJo (v.10;BD生物科学)。gydF4y2Ba
一般免疫表型组gydF4y2Ba
解冻的pmcs在含50 U ml的RPMI培养基中洗涤gydF4y2Ba−1gydF4y2Ba再用PBS冲洗。细胞用活/死固定蓝染料(Life Technologies)孵育,用于排除死亡细胞的分析。细胞在FACS染色缓冲液(1× PBS、0.5%胎牛血清、0.5%正常小鼠血清和0.02% NaN)中洗涤gydF4y2Ba3 gydF4y2Ba),用人Fc阻断试剂(BD Bioscience, 564220)在室温下孵育5分钟。用针对CCR7, CCR6, CXCR5, CXCR3和TCRgd的荧光偶联抗体在室温下黑暗中染色10分钟。然后用荧光素偶联CD45RA、CD16、CD11c、CD56、CD8、CD123、CD161、IgD、CD3、CD20、IgM、IgG、CD28、PD-1、CD141、CD57、CD45、CD25、CD4、CD24、CD95、CD27、CD1c、CD127、HLA-DR、CD38、ICOS、CD21、CD19和CD14的抗体在室温黑暗中染色30分钟。细胞用FACS染色缓冲液(1× PBS、0.5%胎牛血清、0.5%正常小鼠血清和0.02% NaN)洗涤2次gydF4y2Ba3 gydF4y2Ba)并固定在1%多聚甲醛中。补充表gydF4y2Ba13gydF4y2Ba显示表型面板中使用的抗体的克隆和信息。使用Cytek Aurora波谱细胞仪(Cytek Biosciences;SpectroFlo (v.2.2.0))。使用FlowJo分析主要人群的频率(v.10;BD Biosciences)基于先前描述的手动门控策略gydF4y2Ba62gydF4y2Ba,gydF4y2Ba63gydF4y2Ba,gydF4y2Ba64gydF4y2Ba.gydF4y2Ba
体外刺激T细胞面板gydF4y2Ba
收集体外模拟pmcs并在PBS中洗涤。细胞用僵尸UV固定活性染料(BioLegend)在黑暗中(室温下)孵育20分钟。然后将细胞清洗并与Human TruStain FcX (BioLegend)孵育10分钟,随后与抗ccr7抗体孵育10分钟。加入针对CD8、CD4、HLA-DR、CD69、CD45RA、CD11c、CD5、CD3、TCRVa7.2、CD45RO、CD56、CD122、CD158e/k (KIR3DL1/DL2)、KIR2D、NKG2A、CD14、CD29和GPR56的荧光偶联抗体鸡尾酒,并在黑暗中染色30分钟。使用固定/渗透试剂盒(BD Biosciences)清洗和固定细胞。固定后染色细胞内蛋白IFNγ、TNF、Ki-67。使用BD FACSymphony流式细胞仪(BD Biosciences)收集样本,并使用FlowJo进行分析(v.10)。检测组中使用的抗体列表见补充表gydF4y2Ba14gydF4y2Ba.gydF4y2Ba
数据处理与转换gydF4y2Ba
批量RNA-seq数据处理gydF4y2Ba
用Trimmomatic (v.0.38.0)对5号板的测序读数进行适配器和质量校正至100 bpgydF4y2Ba65gydF4y2Ba匹配其他板的读取长度(读取小于100 bp的结果将被丢弃)。然后使用STAR (v.2.6.0b)校准器将Reads对准人类基因组hg38。使用ui -tools根据唯一的分子标识符(v.0.5.3)去除PCR扩增的重复读段。使用featurets进行基因表达量化gydF4y2Ba66gydF4y2BaSubread包中的函数(v.1.6.2)。少于500万个指定reads的样本被重新测序和替换。读取是标准化和日志转换使用limma轰击gydF4y2Ba67gydF4y2Ba.低表达基因,定义为有少于5个样本的>0.5 / million reads,被删除。来自同一健康对照参与者的预疫苗接种(第7天和第0天)样本被认为是重复,并使用RUVSeq的RUVs函数来估计潜在的技术因素gydF4y2Ba68gydF4y2BaR包(v.1.18)。包括四个潜在变量,以获得用于可视化和特别注意的标准化基因表达值。基于健康对照个体和跨技术重复的疫苗接种前样本参与者变异的可变基因被过滤掉,总共有10017个剩余基因用于下游分析。gydF4y2Ba
CITE-seq数据处理gydF4y2Ba
单细胞样本解复用和预处理gydF4y2Ba
单细胞测序数据被解复用,转换为FASTQ格式,映射到人类hg19参考基因组,并使用CellRanger (10x Genomics)管道进行计数。样本级解复用是基于前面描述的两个级别执行的gydF4y2Ba15gydF4y2Ba:(1)标签抗体染色,区分不同时间点的样本与同一参与者;(2)从全血RNA-seq数据中调用单核苷酸多态性(SNPs)来识别不同的参与者。具体来说,CellRanger (v.6.0.1)被用于生成计数矩阵和软件包demuxlet (v.2,来自popscale软件套件)gydF4y2Ba69gydF4y2Ba用于将单细胞基因表达数据与每个供体匹配,并识别空液滴和双胞体。gydF4y2Ba
单单元数据聚类和单元注释gydF4y2Ba
使用运行在R v.4.1.1中的Seurat (v.4.0.3)进一步处理单细胞数据。我们移除了检测到的基因少于200个和多于5000个的细胞;超过60%的reads映射到单个基因;线粒体读数大于15%;细胞表面蛋白标签大于20000;标签抗体数量超过2万。使用DSB方法对蛋白质数据进行归一化和去噪(v.0.3.0)gydF4y2Ba70gydF4y2Ba.dsb归一化函数中使用了以下参数:假计数= TRUE,假计数。use = 10, denoise_counts = TRUE, use.isotype.control = TRUE。dsb归一化蛋白质数据用于生成顶部变量特征(gydF4y2BangydF4y2Ba= 100)和主部件(PCs)。共享最近邻图后面跟着gydF4y2BakgydF4y2Ba-最近邻集群是在Seurat (v.4.0.3)的前15台pc上分别使用FindNeighbors和FindClusters函数构建的。细胞簇的质量控制基于它们最近的邻居和细胞表面蛋白。然后在Seurat中使用加权最近邻(WNN)分析在每个主要细胞群中进一步聚类gydF4y2Ba71gydF4y2Ba(v.4.1.0)通过整合细胞表面蛋白和基因表达方式。使用细胞表面蛋白和可变特征RNA的前10个pc进行WNN FindMultiModalNeighbors。使用表面蛋白和基因表达对WNN聚类进行手工注释和质量控制。gydF4y2Ba
CD8gydF4y2Ba+gydF4y2BaCITE-seq集群的EM单元注释gydF4y2Ba
所有CD8gydF4y2Ba+gydF4y2Ba采用上述小波神经网络对细胞进行聚类。CD8gydF4y2Ba+gydF4y2Ba根据所报道的表面标记对聚类进行了注释gydF4y2Ba72gydF4y2Ba连同基因表达谱。CD8的RNA表达gydF4y2Ba+gydF4y2Ba使用Seurat标签传输方法将cell映射到外部数据集gydF4y2Ba71gydF4y2Ba,gydF4y2Ba73gydF4y2Ba(v.4.1.0)。集群注释为CD8gydF4y2Ba+gydF4y2BaEM是表面CD45ROgydF4y2Ba高gydF4y2Ba, CD45RAgydF4y2Ba低gydF4y2Ba, CD95gydF4y2Ba+gydF4y2Ba、CD62LgydF4y2Ba低gydF4y2Ba而且gydF4y2BaCCR7gydF4y2Ba信使核糖核酸gydF4y2Ba−gydF4y2Ba大多数细胞(约90%)映射到CD8gydF4y2Ba+gydF4y2Ba外部数据集中的EM表型细胞gydF4y2Ba71gydF4y2Ba,gydF4y2Ba73gydF4y2Ba.gydF4y2Ba
单单元TCR数据处理gydF4y2Ba
使用CellRanger (V .6.0.1)组装V(D)J contigs。V(D)J赋值和克隆类型来自经过过滤的contig_annotations.csv文件的CellRanger输出,用于每个10x车道。使用scRepertoire R包(v.1.4.0)对所有通路的数据进行组合,并对每个单细胞的配对TCRα和TCRβ链进行组合。gydF4y2Ba74gydF4y2Ba并与单单元CITE-seq Seurat对象元数据集成。细胞标记为CD8gydF4y2Ba+gydF4y2Ba对检测到α和β链的T细胞进行筛选和分析。CD8gydF4y2Ba+gydF4y2Ba子集和GPR56gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba使用Circlize R包通过Circos图显示EM细胞的克隆性(v.0.4.14)gydF4y2Ba75gydF4y2Ba.为了可视化的目的,每个子集的细胞都进行了相同数量的下采样(子集之间的比较见扩展数据图。gydF4y2Ba4 ggydF4y2Ba)或每个时间点(时间点之间的比较见扩展数据图。gydF4y2Ba4 h,我gydF4y2Ba).当细胞具有相同的CDR3 (α和β链)时,它们被认为是相同的克隆。在每个样本或每个参与者中,相同的克隆在时间点上用线连接起来。gydF4y2Ba
OLINK血清蛋白质组学gydF4y2Ba
缺失值是使用gydF4y2BakgydF4y2Ba-最近的邻居接近gydF4y2BakgydF4y2Ba= 10使用impute R包gydF4y2Ba76gydF4y2Ba(v.1.60.0)。对于每个样本,探针针对相同的蛋白质取平均值。gydF4y2Ba
Cytek流式细胞仪gydF4y2Ba
通过将细胞计数转换为指定的活细胞或淋巴细胞的分数来生成细胞频率。频率数据为对数gydF4y2Ba2 gydF4y2Ba-转换为线性建模。对于任何样本中计数为零的总体,在整个样本中添加一个等于最小非零值的一半的偏移量。gydF4y2Ba
CBCs和淋巴细胞表型gydF4y2Ba
绝对计数和相对计数均为对数gydF4y2Ba2 gydF4y2Ba-转换为线性建模。缺失值由gydF4y2BakgydF4y2Ba-最近的邻居接近。对于任何样本中值为零的参数,在整个样本中添加一个等于最小非零值的一半的偏移量。gydF4y2Ba
统计分析gydF4y2Ba
基线差异表达分析gydF4y2Ba
利用梦想gydF4y2Ba77gydF4y2Ba在variancePartition R包(v.1.16.1)中,应用混合效应模型来确定从COVID-19康复的参与者和健康对照组参与者之间的分析物差异水平(即全血基因表达、血清蛋白、细胞频率、流感滴度和SPR以及血清学参数),按性别特异性方式如下:~ 0 +组:性别+年龄+种族+ batch.effects +(1|参与者。id)。gydF4y2Ba
根据试验类型,将批效应相关的协变量添加到特定模型中。对于大量RNA-seq,这些包括四个潜在的技术因素(参见“大量RNA-seq数据处理”部分)和来自CBC面板的时间点匹配的%中性粒细胞参数。对于Cytek和Olink平台,取样批次/板被作为协变量。除第0天外,第7天的可用样本(在RNA-seq和CBC面板中)也被作为基线重复纳入建模。gydF4y2Ba
性别特异性组差异由对比计算。女性-健康。女性和covid。男性-健康。男性。通过结合这两个对比来确定COVID-19与健康对照的总体差异,即(covid.Female−health . female)/2 + (covid.Male−health . male)/2。与SARS-CoV-2感染相关的性别差异来自于对比(covid.male - covid.female) -(健康。男性-健康。女性),以解释男性和女性之间的正常差异。gydF4y2BaPgydF4y2Ba使用Benjamini-Hochberg方法在每种测定类型和对比组合中进行多次测试调整值gydF4y2Ba78gydF4y2Ba.gydF4y2Ba
与TSD的关系gydF4y2Ba
为了评估基线检测到的任何差异是否已经稳定或可能仍在解决,使用线性模型来测试从COVID-19恢复的参与者中相关参数与COVID-19诊断时间(TSD)的相关性:~ 0 +性别+性别:量表(TSD) +年龄+种族+(1|参与者。id)。gydF4y2Ba
两名无已知TSD的无症状参与者被排除在模型之外。分别评估女性和男性个体的相关性,并通过联合对比(女性:TSD +男性:TSD)/2进行联合评估。因变量在模型中转换为等级,以减少潜在异常值的影响。gydF4y2Ba
使用保守方法,如果基因有未调整的tsd相关gydF4y2BaPgydF4y2Ba< 0.05,按规定排除后续分析。为了确定是否有任何基线差异基因集与TSD相关,LEG模块来自来自不同对比的同一基因集的所有LEG的并集(参见“Bulk RNA-seq基因集模块评分”部分)。如果关联模型中检测的三种对比均不显著(使用未调整的样本),则认为该基因集是稳定的gydF4y2BaPgydF4y2Ba值阈值为0.05)。gydF4y2Ba
接种后差异表达分析gydF4y2Ba
与基线差异表达分析中使用的工作流程类似,创建混合效应模型来评估接种后每个可用时间点的变化和组间差异。65岁及以上的参与者被排除在外,因为他们接种了不同类型的疫苗。除了基线协变量外,该模型还考虑了参与者近10年内的流感疫苗接种史,如下所示:~ 0 +访问次数:分组:性别+年龄+种族+ flu.vax.count.10yr + batch.effects +(1|参与者。id)。gydF4y2Ba
使用该模型检查了三种类型的比较:gydF4y2Ba
特定时间点组差异:与基线模型中的对比相似,但适用于接种疫苗后的个别时间点(第1天至第100天)。gydF4y2Ba
疫苗诱导的组差异变化:类似于上述特定时间点的对比,但另外减去相应的基线对比,以评估相对于基线的变化。例如,用(D1.covid.female−D1.health .female)−(基线.covid.female−基线.health .female)来评估感染COVID-19的女性个体与健康对照个体在D1上疫苗诱导变化的差异。gydF4y2Ba
逆转COVID-19与健康对照的差异:不使用相同相应时间点的健康对照参与者作为参考,将从COVID-19中恢复的参与者的疫苗接种后样本与基线健康对照个体进行比较,对比[时间点].covid.female -基线.healthy。女性和[时间点].covid.男性−基线。健康。男性。这些对比可以告知从COVID-19康复的参与者中观察到的任何疫苗接种前差异是否在接种后恢复到健康基线水平。逆转被定义为在接种后的D1和D28与基线绝对效应量相比具有更小的绝对效应量(使用来自梦函数的z.std值)。gydF4y2Ba
PgydF4y2Ba使用Benjamini-Hochberg方法对每个时间点、检测类型和对比组合进行多次检测。gydF4y2Ba
差异表达基因的基因集富集gydF4y2Ba
使用clusterProfiler R包(v3.17.0)中实现的预排序GSEA算法识别富集的基因集gydF4y2Ba79gydF4y2Ba.使用符号−log对基因进行排序gydF4y2Ba10gydF4y2Ba改变了gydF4y2BaPgydF4y2Ba来自差分表达式模型的值。利用MSigDB的Hallmark收集的基因集列表评估富集程度gydF4y2Ba80gydF4y2Ba,血液转录组模块gydF4y2Ba81gydF4y2Ba以及细胞类型基因特征gydF4y2Ba53gydF4y2Ba.只考虑10到300个基因的基因集。gydF4y2BaPgydF4y2Ba使用Benjamini-Hochberg方法对每个对比的基因集列表进行值调整,并使用fdr调整基因集gydF4y2BaPgydF4y2Ba< 0.05为显著。基线富集基因集通过交叉从差异表达模型中提取的显著基因集来获得,这些差异表达模型使用的样本独立于-7天、0天和两者结合。基线与TSD相关的基因(见“与TSD相关”部分;补充表gydF4y2Ba1克ydF4y2Ba)被排除在疫苗接种后富集分析之外,以帮助将疫苗接种的效果与SARS-CoV-2感染的自然时间消退区分开。gydF4y2Ba
伪体微分表达式与GSEAgydF4y2Ba
从给定样本中提取的单个细胞,通过对给定基因的所有读数求和,根据它们的细胞类型进行计算汇总。从每种细胞类型的分析中去除由少数细胞组成的假体积文库,因此可能无法正确使用体积差异表达方法进行建模,以去除包含少于4个细胞且池化后文库大小小于35,000个的样本。使用edgeR的filterByExpr函数分别去除每种细胞类型的低表达基因(v.3.26.8)gydF4y2Ba82gydF4y2Bamin.count = 2。使用calcNormFactors函数提供的用于文库大小归一化的标度因子计算每个基因的log -transform counts per million (CPM)。差异表达分析使用“接种后差异表达分析”部分中描述的相同模型进行,没有单独运行基线模型,因为整个CITE-seq队列的年龄在65岁以下。在该模型中,批处理效应包括批量分配和每个样品的条形码/单元数。gydF4y2Ba
类似地,对每种细胞类型进行GSEA的方式与对大量RNA-seq数据的描述相同(参见“差异表达基因的基因集富集”部分),其中特别关注大量RNA-seq分析确定的基线富集基因集。摩纳哥基因集从单细胞分析中被排除,因为细胞簇已被注释,不需要进一步的细胞类型分解。gydF4y2Ba
散装RNA-seq基因集模块评分gydF4y2Ba
基因集模块得分由RUVseq (v.1.18)归一化基因表达值(见“大量RNA-seq数据处理”部分)使用GSVA R包(v.1.30.0)中的基因集变异分析(GSVA)方法生成。gydF4y2Ba83gydF4y2Ba.使用GSEA识别的LEGs来计算代表富集通路活性的相关样本的LEG模块评分,以提高信噪比。第7天和0天之间的平均分数用于计算相对于基线的疫苗接种后的变化。gydF4y2Ba
伪体基因集模块得分计算gydF4y2Ba
计算特定细胞类型的每个假体样本的模块分数(基因集签名分数),代表富集通路活性。假体基因计数使用limma包(v.3.42.2)中的removeBatchEffect函数进行校正,以去除实验批次和细胞数量效应,然后用voom归一化gydF4y2Ba84gydF4y2Ba.然后使用基因集变异分析(GSVA)方法从GSVA R包(v.1.42.0)中生成分数。gydF4y2Ba83gydF4y2Ba.具体来说,对于单核细胞特征,通过GSEA从(1)D0中鉴定BTM模块M4.0和M11.0的leg。COVR-F与D0。HC-F和(2)D0。COVR-M vs . D0。HC-M模型。女性和男性样本的得分计算采用LEGs的并集。gydF4y2Ba
对于图中所示的BTM-M7.3 T细胞激活特征和急性COVID-19数据中的其他特征,分别使用所示对照组的LEGs来计算女性和男性个体的评分。gydF4y2Ba
对于单核细胞抗原呈递签名,使用富集抗原呈递(I)的BTM-M71和富集抗原呈递(II)基因集的M95.0的LEGs生成模块评分:COVR-M组与HC-M组之间D1−D0的变化(图2)。gydF4y2Ba2 fgydF4y2Ba).gydF4y2Ba
对于Hallmark IFNγ反应模块评分,基因集中的所有基因都用于计算每种细胞类型的模块评分,以便比较细胞类型之间的差异。gydF4y2Ba
单细胞模块得分计算和可视化gydF4y2Ba
为了使用单个数据来可视化特定基因特征在不同研究对象组之间的差异,我们使用所示基因集中的基因来计算每个单个细胞的相应模块得分。模块分数使用Seurat (v.4.1.0)中的AddModuleScore函数计算,然后在UMAP图中可视化。对于D1与D0, Hallmark IFNγ反应模块评分差异(D1 - D0)显示在UMAP投影中(图2)。gydF4y2Ba二维gydF4y2Ba)、D1细胞。HC-F D1。COVR-F D1。HC-M and D1.COVR-M groups were downsampled to the same number of cells. The UMAP embeddings of cells coloured with the average difference (D1–D0) of each high-resolution cell subsets are shown (each of the major cell clusters shown can contain one or more high-resolution cell subsets).
体外急性COVID-19单细胞CITE-seq数据的单细胞模块评分计算与检验gydF4y2Ba
单细胞数据来自ref. Brescia队列。gydF4y2Ba15gydF4y2Ba从基因表达综合(GEO)中下载。提取单个单核细胞数据,并从Brescia队列中收集单细胞数据,如“假体差异表达和GSEA”部分所述。所有样本的BTM模块M4.0和M11.0的基因集模块分数是在“基因集模块分数计算”部分使用男性和女性的并集LEGs生成的。使用DEseq2 R包(v.1.34.0)的variancestablilizingtransform函数对假体基因计数进行归一化。gydF4y2Ba85gydF4y2Ba.然后使用GSVA方法从GSVA R包(v.1.42.0)中生成分数gydF4y2Ba83gydF4y2Ba.考虑到每个参与者有多个样本,患者组之间的差异(健康对照组,轻重度和重度,对应于HC,参考文献中的DSM-low和DSM-high)。gydF4y2Ba15gydF4y2Ba)使用Limma (v.3.50.1)线性模型进行检验,其中使用duplicateCorrelation将来自同一供体的样本视为重复样本。gydF4y2BaPgydF4y2Ba的值gydF4y2BatgydF4y2Ba所示对比线性模型的统计数字。gydF4y2Ba
热图中基因表达的可视化gydF4y2Ba
使用ComplexHeatmap R包(v.2.10.0)生成显示伪批量数据的热图gydF4y2Ba86gydF4y2Ba.通过池化细胞计算给定细胞类型的每个样本的log[CPM]归一化表达,如“伪体差异表达和GSEA”部分所述。热图显示gydF4y2BazgydF4y2Ba-每个样本中每个基因归一化表达的得分。gydF4y2Ba
数据可视化gydF4y2Ba
除非特别注明,否则使用ggplot2 (v.3.3.5)和ggpubr (v.0.4.0)创建图进行统计计算。gydF4y2Ba
端点协会gydF4y2Ba
为了评估相关参数,包括基因集模块评分和细胞频率,与IFN或抗体滴度折叠变化终点的相关性,应用以下模型:终点~组:性别+量表(参数):组:性别+年龄+种族+流感.vax.count.10yr。gydF4y2Ba
终点值转换为等级,以减少潜在异常值的影响。对同一参与者的重复数据进行平均。gydF4y2Ba
血清学gydF4y2Ba
检测限为1:20以下的流感抗体滴度设为1:10。每个菌株的最大滴度是通过将D0和D28的所有样品的滴度水平分别归一化,然后对每个样品取最大标准化滴度来计算的。gydF4y2Ba
基线滴度差异分析gydF4y2Ba
对于四种菌株,应用线性模型,按性别确定从COVID-19恢复的参与者和健康对照组参与者之间的基线滴度差异,方法如下:第0天滴度~组:性别+年龄+种族。gydF4y2Ba
滴度值为对数gydF4y2Ba10gydF4y2Ba在模型中转换,并根据对比计算性别特异性组差异。女性-健康。女性和covid。男性-健康。男性。65岁及以上的参与者被排除在分析之外。gydF4y2Ba
D28滴度差异分析gydF4y2Ba
对于疫苗接种后滴度反应,流感疫苗接种史和基线滴度被作为协变量,部分解释了之前的暴露,类似于美国食品和药物管理局用于流感疫苗评估的方法(第27页)gydF4y2Bahttps://www.fda.gov/media/135687/downloadgydF4y2Ba).评估D28滴度和D28/D0 FC作为终点,以确定四种菌株中从COVID-19恢复的参与者与健康对照组参与者之间的分组差异:终点~组:性别+年龄+种族+流感.vax.count.10年+第0天滴度。gydF4y2Ba
对于D28 FC,采用负二项对数链路模型。MASS R包中的nb函数(v.7.3-53)。采用线性模型拟合D28滴度。菌株特异性滴度值为对数gydF4y2Ba10gydF4y2Ba-在模型中转换。使用基线分析中相同的参与者和对比来评估组间差异。gydF4y2Ba
采用与HA1和HA2的滴度数据相同的方法分析了使用SPR测量的流感抗体的亲和度,不同之处是对折叠变化应用了线性模型。gydF4y2Ba
自然流感感染队列的一致性gydF4y2Ba
一项前瞻性队列研究,参与者在两个季节的自然流感感染之前和至少21天后进行研究gydF4y2Ba18gydF4y2Ba分别在男性和女性个体中评估感染的残留效应。基因表达数据从GEO (gydF4y2BaGSE68310gydF4y2Ba).只感染甲型流感病毒的参加者(gydF4y2BangydF4y2Ba= 51名女性及gydF4y2BangydF4y2Ba= 35名男性)被鉴定并纳入本分析。去除低表达的探针,其余数据转换为基于基因的表达。由于数据已经标准化,因此没有执行额外的处理步骤。gydF4y2Ba
对于每个季节,使用variancePartition R包(v.1.16.1)中的dream函数分别对来自同一个体的基线(感染前)和春季(感染后长期)样本进行差异表达分析。考虑流感疫苗接种史和疾病严重程度(基于发烧等级:无、低和高)的混合效应模型构建如下:~ 0 +时间点:性别+年龄+接种次数。流感.疫苗+发烧。等级+(1|参与者。id)。gydF4y2Ba
差异表达基因通过对比Spring.F−Baseline进行鉴定。F和Spring.M−基线。M分别代表女性和男性个体。通过对比(Spring.M−Baseline.M)−(Spring.F−Baseline.F)来评估性别差异。根据基因间效应大小的相关性(梦产生的z.std值),评估两个季节之间差异表达结果的一致性。gydF4y2Ba
进行富集分析,以确定来自这一独立队列的流感感染前和流感感染后的参与者以及从COVID-19中恢复的参与者与接种疫苗前的健康对照组相比,相同的一组基因是否存在差异。为了更好地匹配两项研究中参与者的年龄范围,对COVID-19队列中65岁以下的参与者再次进行了基线差异基因分析(见“基线差异表达分析”部分)。考虑到男性参与者在两个流感季节之间表现出更强的一致性(扩展数据图)。gydF4y2Ba2 bgydF4y2Ba),用符号−log对COVID-19差异表达基因进行排序gydF4y2Ba10gydF4y2Ba改变了gydF4y2BaPgydF4y2Ba与差异表达的(gydF4y2BaPgydF4y2Ba在流感感染队列男性参与者的基因中< 0.05)。gydF4y2Ba
弹性净多元预测模型gydF4y2Ba
使用eNetXplorer R包(v.1.1.3)构建弹性网络模型gydF4y2Ba87gydF4y2Ba以CITE-seq和流式细胞仪在D0时的细胞频率为预测指标,预测流感疫苗接种后第1天(D1)的INFγ反应。共纳入33例(COVR-F = 11, HC-F = 8, COVR-M = 9, HC-M = 5)均有CITE-seq和流式细胞仪数据。在20次五次交叉验证的基础上,正则化参数网格(gydF4y2BaαgydF4y2Ba而且gydF4y2BaλgydF4y2Ba)进行测试,以确定具有最佳性能的模型和具有一致预测能力的细胞子集。模型的性能是根据预测响应和观测响应之间的均方误差进行评估的。细胞群的重要性由模型选择它的频率决定(即具有非零系数)。gydF4y2BaPgydF4y2Ba通过与用排列响应构造的空模型进行比较,得到模型性能和特征重要性的值。gydF4y2Ba
TCR分集度量计算gydF4y2Ba
香农熵(gydF4y2BaHgydF4y2Ba’指数)作为衡量TCR多样性的指标gydF4y2Ba88gydF4y2Ba,gydF4y2Ba89gydF4y2Ba.每个CD8样本gydF4y2Ba+gydF4y2Ba从计算中过滤出小于50个单元格的子集。所有样本都被减少到50个细胞,因为多样性指标可能受到样本细胞数量的影响。该过程重复1000次,对50个细胞进行随机下采样,并使用Shannon指数的中位数作为给定样本的多样性估计。不同CD8之间多样性度量的差异gydF4y2Ba+gydF4y2Ba子集或时间点采用双尾Wilcoxon检验。gydF4y2Ba
逆转基因和自举推断COVR-F组和COVR-M组之间单核细胞抑制信号逆转差异的意义gydF4y2Ba
逆转基因被定义为covid -19康复患者相对于D0健康患者控制绝对效应量的基因(z.std值来自梦境;见“接种后差异表达分析”部分)在D1和D28位点均小于D0位点。gydF4y2Ba
使用自引导法来确定COVR-F组和COVR-M组从单核细胞抑制特征(BTM M4.0和M11.0)向健康对照基线移动的基线leg比例差异的显著性。在每一轮自举中,每个参与者组的成员被随机抽样并进行替换,他们的样本按照“接种后差异表达分析”部分所述进行分析。分别计算每轮接种后COVR-F组和COVR-M组经典单核细胞和非经典单核细胞中LEGs逆转的比例gydF4y2BaPgydF4y2Ba图中所示值。gydF4y2Ba4 egydF4y2Ba是在20轮手术的基础上确定的。gydF4y2Ba
报告总结gydF4y2Ba
有关研究设计的进一步资料,请参阅gydF4y2Ba自然组合报告摘要gydF4y2Ba链接到这篇文章。gydF4y2Ba
数据可用性gydF4y2Ba
全血体积RNA-seq和单细胞CITE-seq的原始和处理数据可从NCBI GEO获得,登录号如下gydF4y2BaGSE194378gydF4y2Ba而且gydF4y2BaGSE206265gydF4y2Ba,分别。其他数据集,包括临床,蛋白质组学,流式细胞仪和流感抗体测量,可在Zenodo (gydF4y2Bahttps://doi.org/10.5281/zenodo.5935845gydF4y2Ba).我们使用的流感感染数据集是直接从GEO (gydF4y2BaGSE68310gydF4y2Ba).gydF4y2Ba
代码的可用性gydF4y2Ba
分析代码,扩展患者和样本元数据可在GitHub (gydF4y2Bahttps://github.com/niaid/covid-flugydF4y2Ba).gydF4y2Ba
参考文献gydF4y2Ba
米娜,m.j.等人。麻疹病毒感染会减少现有的抗体,这些抗体可以保护人们免受其他病原体的侵害。gydF4y2Ba科学gydF4y2Ba366gydF4y2Ba, 599-606(2019)。gydF4y2Ba
托马尔卡,J. A.,苏塔尔,m.s.,戴蒙德,m.s. &塞卡利,r.p.先天抗病毒免疫:先前的暴露如何指导未来的反应。gydF4y2BaImmunol趋势。gydF4y2Ba43gydF4y2Ba, 696-705(2022)。gydF4y2Ba
曾,J. S.等。提高疫苗诱导免疫:基线能预测结果吗?gydF4y2BaImmunol趋势。gydF4y2Ba41gydF4y2Ba, 457-465(2020)。gydF4y2Ba
古德里奇,H. S.等人。利用疫苗接种的有益异源效应。gydF4y2BaNat. Rev. Immunol。gydF4y2Ba16gydF4y2Ba, 392-400(2016)。gydF4y2Ba
Aaby, P., Netea, M. G. & Benn, C. S.活疫苗对COVID-19和其他不相关感染的有益非特异性作用。gydF4y2Ba柳叶刀感染。说。gydF4y2Ba23gydF4y2Ba, e34-e42(2022)。gydF4y2Ba
Netea, M. G.等。定义训练免疫及其在健康和疾病中的作用。gydF4y2BaNat. Rev. Immunol。gydF4y2Ba20.gydF4y2Ba, 375-388(2020)。gydF4y2Ba
Nalbandian, A.等人。COVID-19急性后综合征。gydF4y2BaNat,地中海。gydF4y2Ba27gydF4y2Ba, 601-615(2021)。gydF4y2Ba
奥尔森,S. J.等。COVID-19大流行期间流感和其他呼吸道病毒活动的变化-美国,2020-2021年。gydF4y2BaMMWR Morb。凡人。工作代表。gydF4y2Ba70gydF4y2Ba, 1013-1019(2021)。gydF4y2Ba
苏德瑞,C. H.等。长冠肺炎的属性和预测因素。gydF4y2BaNat,地中海。gydF4y2Ba27gydF4y2Ba, 626-631(2021)。gydF4y2Ba
斯托克修斯等人。单细胞中同时测定表位和转录组。gydF4y2BaNat方法。gydF4y2Ba14gydF4y2Ba, 865-868(2017)。gydF4y2Ba
Ursin, R. L. & Klein, S. L.呼吸道病毒发病机制和治疗的性别差异。gydF4y2Ba为基础。启性研究。gydF4y2Ba8gydF4y2Ba, 393-414(2021)。gydF4y2Ba
惠特利,a.k.等人。轻-中度COVID-19患者对SARS-CoV-2免疫反应的演变gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 1162(2021)。gydF4y2Ba
舒尔茨,J. L.和Aschenbrenner, a.c. COVID-19和人类先天免疫系统。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 1671-1692(2021)。gydF4y2Ba
塞特,A. &克罗蒂,S.对SARS-CoV-2和COVID-19的适应性免疫。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 861-880(2021)。gydF4y2Ba
刘,C.等。时间分辨系统免疫学揭示了与致命COVID-19相关的晚期节点。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 1836-1857(2021)。gydF4y2Ba
Paludan, S. R. & Mogensen, t.h. COVID-19发病机制中的先天免疫途径。gydF4y2Ba科学。Immunol。gydF4y2Ba7gydF4y2Ba, eabm5505(2022)。gydF4y2Ba
雷耶斯等人。细菌性败血症或严重COVID-19患者的血浆诱导体外造血祖细胞产生抑制性骨髓细胞。gydF4y2Ba科学。Transl。地中海。gydF4y2Ba13gydF4y2Ba, eabe9599(2021)。gydF4y2Ba
翟,Y.等。宿主对流感和其他急性呼吸道病毒感染的转录反应——一项前瞻性队列研究gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba11gydF4y2Ba, e1004869(2015)。gydF4y2Ba
系统疫苗学:用疫苗探索人类不同的免疫系统。gydF4y2Ba国家科学院学报美国gydF4y2Ba111gydF4y2Ba, 12300-12306(2014)。gydF4y2Ba
利用种群变异、疫苗接种和系统生物学来研究人类免疫学。gydF4y2BaImmunol趋势。gydF4y2Ba36gydF4y2Ba, 479-493(2015)。gydF4y2Ba
Pulendran, B., S. **am, P. & O 'Hagan, D. T.疫苗佐剂科学中的新兴概念。gydF4y2BaNat. Rev.药物发现gydF4y2Ba20.gydF4y2Ba, 454-475(2021)。gydF4y2Ba
布卡萨斯,K. L.等。基因表达的早期模式与人类对流感疫苗的体液免疫反应相关。gydF4y2Baj .感染。说。gydF4y2Ba203gydF4y2Ba, 921-929(2011)。gydF4y2Ba
奥拉德尔等人。流感病毒感染史决定了抗体对流感疫苗的反应。gydF4y2BaNat,地中海。gydF4y2Ba28gydF4y2Ba, 363-372(2022)。gydF4y2Ba
Truong K.-L。et al。杀手样受体和GPR56的渐进性表达决定了CD4细胞因子的产生gydF4y2Ba+gydF4y2Ba记忆T细胞。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 2263(2019)。gydF4y2Ba
尼科莱,B. P.等。CD29识别产生IFN-γ的人CD8gydF4y2Ba+gydF4y2Ba细胞毒性增强的T细胞。gydF4y2Ba国家科学院学报美国gydF4y2Ba117gydF4y2Ba, 6686-6696(2020)。gydF4y2Ba
Bergamaschi, L.等人。纵向分析显示,延迟的旁观者CD8gydF4y2Ba+gydF4y2BaT细胞活化和早期免疫病理将COVID-19重型和轻型疾病区分开来。gydF4y2Ba免疫力gydF4y2Ba54gydF4y2Ba, 1257-1275(2021)。gydF4y2Ba
刘斯,s.c.等。人类CD4gydF4y2Ba+gydF4y2Ba记忆T细胞是旁观者激活和凋亡的优先目标。gydF4y2Baj . Immunol。gydF4y2Ba182gydF4y2Ba, 1962-1971(2009)。gydF4y2Ba
怀特,J. T.克罗,E. W. & Kedl, R. M.抗原无经验记忆CD8gydF4y2Ba+gydF4y2BaT细胞:它们来自哪里,为什么我们需要它们。gydF4y2BaNat. Rev. Immunol。gydF4y2Ba17gydF4y2Ba, 391-400(2017)。gydF4y2Ba
莫里斯,N. J.,泰伯,a . K. & Prlic, M.丑小鸭变成天鹅:旁观者激活记忆CD8 T细胞感知的变化。gydF4y2Baj . Immunol。gydF4y2Ba206gydF4y2Ba, 455-462 2021)。gydF4y2Ba
Abers, m.s.等人。基于免疫的生物标志物特征与COVID-19患者的死亡率相关。gydF4y2Ba江森自控的洞察力gydF4y2Ba6gydF4y2Ba, 144455(2021)。gydF4y2Ba
Herndler-Brandstetter, D.等人。人记忆T细胞中CD5水平的胸腺后调控与对白细胞介素-15的反应强度呈负相关。gydF4y2Ba嗡嗡声。Immunol。gydF4y2Ba72gydF4y2Ba, 627-631(2011)。gydF4y2Ba
Jacomet, F.等人。表达eomesdermin的先天性样CD8的证据gydF4y2Ba+gydF4y2Ba吉珥/ NKG2AgydF4y2Ba+gydF4y2Ba成人和脐带血样本中的T细胞。gydF4y2Ba欧元。j . Immunol。gydF4y2Ba45gydF4y2Ba, 1926-1933(2015)。gydF4y2Ba
金,黄永发。et al。虚拟内存CD8gydF4y2Ba+gydF4y2BaT细胞抑制hiv -1感染患者的病毒库,抗逆转录病毒治疗通过解除抑制基尔介导的抑制。gydF4y2Ba细胞。摩尔。Immunol。gydF4y2Ba17gydF4y2Ba, 1257-1265(2020)。gydF4y2Ba
Yang J., Zhu H., Murphy, T. L., Ouyang, W. & Murphy, K. M. il -18刺激的GADD45β在细胞因子诱导的IFN-γ生成中需要,而不是tcr诱导的。gydF4y2BaImmunol Nat。gydF4y2Ba2 gydF4y2Ba, 157-164(2001)。gydF4y2Ba
Kastenmüller, W., Torabi-Parizi, P., Subramanian, N, Lämmermann, T. & Germain, R. N.淋巴结中空间组织的多细胞先天免疫反应限制了系统性病原体传播。gydF4y2Ba细胞gydF4y2Ba150gydF4y2Ba, 1235-1248(2012)。gydF4y2Ba
威默斯,F.等人。流感疫苗免疫的单细胞表观基因组和转录景观。gydF4y2Ba细胞gydF4y2Ba184gydF4y2Ba, 3915-3935(2021)。gydF4y2Ba
Debisarun, p.a.等人。通过流感疫苗诱导训练免疫-对COVID-19的影响。gydF4y2Ba公共科学图书馆Pathog。gydF4y2Ba17gydF4y2Ba, e1009928(2021)。gydF4y2Ba
**am, p.s.等。人类对轻度和重度COVID-19感染的免疫系统生物学评估。gydF4y2Ba科学gydF4y2Ba369gydF4y2Ba, 1210-1220(2020)。gydF4y2Ba
Schulte-Schrepping, J.等人。重症COVID-19的特征是骨髓细胞间隔失调。gydF4y2Ba细胞gydF4y2Ba182gydF4y2Ba, 1419-1440(2020)。gydF4y2Ba
Pérez-Gómez, A.等。在SARS-CoV-2感染后,树突状细胞缺陷持续7个月。gydF4y2Ba细胞。摩尔。Immunol。gydF4y2Ba18gydF4y2Ba, 2128-2139(2021)。gydF4y2Ba
Phetsouphanh, C.等人。在最初轻度至中度SARS-CoV-2感染后,免疫功能障碍持续8个月。gydF4y2BaImmunol Nat。gydF4y2Ba23gydF4y2Ba, 210-216(2022)。1 - 7。gydF4y2Ba
utroo - rico, A.等人。循环单核细胞的改变可以预测COVID-19的严重程度,包括恢复六个月后仍可检测到的染色质变化。gydF4y2Ba共同参与gydF4y2Ba9gydF4y2Ba, 1253(2021)。gydF4y2Ba
你,m等人。外周免疫细胞的单细胞表观基因组图谱揭示了COVID-19恢复期个体训练免疫的建立。gydF4y2Ba细胞生物学。gydF4y2Ba23gydF4y2Ba, 620-630(2021)。gydF4y2Ba
克莱因,S. L.和弗拉纳根,K. L.免疫反应的性别差异。gydF4y2BaNat. Rev. Immunol。gydF4y2Ba16gydF4y2Ba, 626-638(2016)。gydF4y2Ba
帕特尔,a.a.等人。稳态和全身炎症状态下人类单核细胞亚群的命运和寿命。gydF4y2Ba实验,医学。gydF4y2Ba214gydF4y2Ba, 1913-1923(2017)。gydF4y2Ba
畅,J.-G。et al。先天免疫细胞及其祖细胞中COVID-19的表观遗传记忆。预印在gydF4y2BabioRxivgydF4y2Bahttps://doi.org/10.1101/2022.02.09.479588gydF4y2Ba(2022)。gydF4y2Ba
李志强,李志强,李志强,等。病毒和I型干扰素诱导T细胞增殖的研究。gydF4y2Ba科学gydF4y2Ba272gydF4y2Ba, 1947-1950(1996)。gydF4y2Ba
高桥,T.等。免疫反应的性别差异是COVID-19疾病结果的基础。gydF4y2Ba自然gydF4y2Ba588gydF4y2Ba, 315-320(2020)。gydF4y2Ba
史高丽,e.p.等。检测、入院、临床表现以及COVID-19严重后果的驱动因素方面的性别差异。gydF4y2Ba打开论坛感染。说。gydF4y2Ba8gydF4y2Ba, ofab448(2021)。gydF4y2Ba
曾,J. S.等。人类免疫变异的全球分析揭示了疫苗接种后反应的基线预测因子。gydF4y2Ba细胞gydF4y2Ba157gydF4y2Ba, 499-513(2014)。gydF4y2Ba
Kotliarov, Y.等。广泛的免疫激活奠定了健康个体疫苗反应性和狼疮患者疾病活动性的共同设定点特征。gydF4y2BaNat,地中海。gydF4y2Ba26gydF4y2Ba, 618-629(2020)。gydF4y2Ba
Nakaya, H. I.等。人类季节性流感疫苗接种的系统生物学。gydF4y2BaImmunol Nat。gydF4y2Ba12gydF4y2Ba, 786-795(2011)。gydF4y2Ba
摩纳哥,G.等。由mRNA丰度归一化的RNA-seq签名允许人类免疫细胞类型的绝对反褶缩。gydF4y2Ba细胞的代表。gydF4y2Ba26gydF4y2Ba, 1627-1640(2019)。gydF4y2Ba
哈里斯,p.a.等人。研究电子数据捕获(REDCap) -提供转化研究信息学支持的元数据驱动方法和工作流过程。gydF4y2Baj .生物医学。通知。gydF4y2Ba42gydF4y2Ba, 377-381(2009)。gydF4y2Ba
哈里斯,p.a.等人。REDCap联盟:构建国际软件平台合作伙伴社区。gydF4y2Baj .生物医学。通知。gydF4y2Ba95gydF4y2Ba, 103208(2019)。gydF4y2Ba
Ravichandran, S.等。SARS-CoV-2刺突蛋白免疫原诱导兔抗体标记。gydF4y2Ba科学。Transl。地中海。gydF4y2Ba12gydF4y2Ba, eabc3539(2020)。gydF4y2Ba
Ravichandran, S.等。“轻度”和“重度”COVID-19患者的纵向抗体库揭示了与疾病严重程度和缓解相关的免疫标志物。gydF4y2Ba科学。睡觉。gydF4y2Ba7gydF4y2Ba, eabf2467(2021)。gydF4y2Ba
唐,J.等。抗体亲和成熟度和血浆IgA与COVID-19住院患者的临床结局相关gydF4y2BaCommun Nat。gydF4y2Ba12gydF4y2Ba, 1221(2021)。gydF4y2Ba
Khurana, S.等。MF59佐剂增强大流行性流感疫苗抗体介导免疫应答的多样性和亲和性gydF4y2Ba科学。Transl。地中海。gydF4y2Ba3 gydF4y2Ba, 85ra48(2011)。gydF4y2Ba
Khurana, S.等。vsv -埃博拉疫苗接种后的人抗体库确定了新的靶点和病毒中和IgM抗体。gydF4y2BaNat,地中海。gydF4y2Ba22gydF4y2Ba, 1439-1447(2016)。gydF4y2Ba
Khurana, S.等。重复接种会降低人体内不同流感疫苗平台的抗体亲和成熟度。gydF4y2BaCommun Nat。gydF4y2Ba10gydF4y2Ba, 3338(2019)。gydF4y2Ba
De Biasi, S.等。COVID-19肺炎患者的T细胞活化、衰老、衰竭和向TH17倾斜。gydF4y2BaCommun Nat。gydF4y2Ba11gydF4y2Ba, 3434(2020)。gydF4y2Ba
海特,A.等。接种疫苗可以在人的血液中建立生发中心T细胞的克隆亲属。gydF4y2Ba实验,医学。gydF4y2Ba214gydF4y2Ba, 2139-2152(2017)。gydF4y2Ba
Park, L. M., Lannigan, J. & Jaimes, M. C. OMIP-069:四十色全光谱流式细胞仪面板用于人外周血主要细胞亚群的深层免疫分型。gydF4y2Ba血细胞计数一次gydF4y2Ba97gydF4y2Ba, 1044-1051(2020)。gydF4y2Ba
Bolger, a.m., Lohse, M. & Usadel, B. Trimmomatic:用于Illumina序列数据的灵活修剪器。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 2114-2120(2014)。gydF4y2Ba
Liao, Y., Smyth, G. K. & Shi, W. featuremets:一种高效的通用程序,用于分配序列读取基因组特征。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 923-930(2014)。gydF4y2Ba
Law, c.w ., Chen, Y., Shi, W. & Smyth, G. K. voom:精确权重解锁线性模型分析工具用于rna序列读取计数。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, r29(2014)。gydF4y2Ba
Risso, D., Ngai, J., Speed, T. P. & Dudoit, S.使用控制基因或样本的因子分析进行RNA-seq数据的标准化。gydF4y2Ba生物科技Nat。》。gydF4y2Ba32gydF4y2Ba, 896-902(2014)。gydF4y2Ba
Kang, h.m.等。利用自然遗传变异的多液滴单细胞rna测序。gydF4y2Ba生物科技Nat。》。gydF4y2Ba36gydF4y2Ba, 89-94(2018)。gydF4y2Ba
Mulè, M. P., Martins, A. J. & Tsang, J. S.从基于液滴的单细胞分析中标准化和去噪蛋白表达数据。gydF4y2BaCommun Nat。gydF4y2Ba13gydF4y2Ba, 2099(2022)。gydF4y2Ba
斯图尔特,T.等人。单细胞数据的全面集成。gydF4y2Ba细胞gydF4y2Ba177gydF4y2Ba, 1888-1902(2019)。gydF4y2Ba
Martin, m.d.和Badovinac, v.p.定义记忆CD8 T细胞。gydF4y2Ba前面。Immunol。gydF4y2Ba9gydF4y2Ba, 2692(2018)。gydF4y2Ba
郑国勇,等。大规模并行数字单细胞转录谱分析。gydF4y2BaCommun Nat。gydF4y2Ba8gydF4y2Ba, 14049(2017)。gydF4y2Ba
Borcherding, N., Bormann, N. L. & Kraus, G. scRepertoire:一个基于r的单细胞免疫受体分析工具包。gydF4y2BaF1000Res。gydF4y2Ba9gydF4y2Ba, 47(2020)。gydF4y2Ba
Gu, Z., Gu, L., Eils, R., Schlesner, M. & Brors, B.在R.中实现并增强了圆形可视化。gydF4y2Ba生物信息学gydF4y2Ba30.gydF4y2Ba, 2811-2812(2014)。gydF4y2Ba
朱晓明,朱晓明,王晓明。微阵列数据的归一化方法。R包版本1.72.1(2022年)。gydF4y2Ba
霍夫曼,g.e.和鲁索斯,P.梦想:重复测量设计的强大差分表达式分析。gydF4y2Ba生物信息学gydF4y2Ba37gydF4y2Ba, 192-201(2021)。gydF4y2Ba
Benjamini, Y. & Hochberg, Y.控制错误发现率:多重测试的一种实用而强大的方法。gydF4y2BaJ. R.统计社BgydF4y2Ba57gydF4y2Ba, 289-300(1995)。gydF4y2Ba
Wu, T.等人。clusterProfiler 4.0:解释组学数据的通用丰富工具。gydF4y2Ba创新gydF4y2Ba2 gydF4y2Ba, 100141(2021)。gydF4y2Ba
Liberzon等人。分子特征库(MSigDB) 3.0。gydF4y2Ba生物信息学gydF4y2Ba27gydF4y2Ba, 1739-1740(2011)。gydF4y2Ba
李,S.等。抗体反应的分子特征来源于对五种人类疫苗的系统生物学研究。gydF4y2BaImmunol Nat。gydF4y2Ba15gydF4y2Ba, 195-204(2014)。gydF4y2Ba
麦卡西,陈永勇,史密思。多因素rna序列实验中生物变异的差异表达分析。gydF4y2Ba核酸测定。gydF4y2Ba40gydF4y2Ba, 4288-4297(2012)。gydF4y2Ba
Hänzelmann, S., Castelo, R. & Guinney, J. GSVA:微阵列和RNA-seq数据的基因集变异分析。gydF4y2BaBMC生物信息学gydF4y2Ba14gydF4y2Ba, 7(2013)。gydF4y2Ba
Ritchie, m.e.等人,limma为rna测序和微阵列研究的差异表达分析提供了动力。gydF4y2Ba核酸测定。gydF4y2Ba43gydF4y2Ba, e47(2015)。gydF4y2Ba
Love, m.i, Huber, W. & Anders, S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。gydF4y2Ba基因组医学杂志。gydF4y2Ba15gydF4y2Ba, 550(2014)。gydF4y2Ba
顾z, Eils, R. & Schlesner, M.复杂热图揭示多维基因组数据的模式和相关性。gydF4y2Ba生物信息学gydF4y2Ba32gydF4y2Ba, 2847-2849(2016)。gydF4y2Ba
Candia, J. & Tsang, J. S. eNetXplorer:用于广义线性模型的弹性网族定量探索的R包。gydF4y2BaBMC生物信息学gydF4y2Ba20.gydF4y2Ba, 189(2019)。gydF4y2Ba
罗萨蒂,E.等人。t细胞受体库分析方法综述。gydF4y2Ba生物科技BMC》。gydF4y2Ba17gydF4y2Ba, 61(2017)。gydF4y2Ba
莫里斯,e.k.等人。选择和使用多样性指数:来自德国生物多样性探索的生态应用见解。gydF4y2Ba生态。另一个星球。gydF4y2Ba4 gydF4y2Ba, 3514-3524(2014)。gydF4y2Ba
确认gydF4y2Ba
我们感谢研究参与者,他们帮助我们扩大了对COVID-19和一般病毒感染的科学理解;K. Sadtler协助开发Cytek 36色流式细胞仪面板;Q.徐先生协助设计细胞因子刺激实验;F.张用于计算辅助;M. Mulè提供软件(dsb);NCI CCR测序设施的工作人员提供测序支持;NIAID高性能计算团队提供计算支持;以及R. Germain阅读手稿。无花果。gydF4y2Ba1 a, bgydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3gydF4y2Ba而且gydF4y2Ba4gydF4y2Ba和扩展数据图。gydF4y2Ba2gydF4y2Ba是使用BioRender创建的。这项研究得到了NIAID的校内研究项目和支持NIH人类免疫学中心的NIH研究所的校内项目的支持。j。h。的部分资金来自国家癌症研究所,国家卫生研究院的联邦基金,合同编号为。75 n91019d00024。本出版物的内容不一定反映美国卫生与公众服务部的观点或政策,也不代表美国政府对商品名称、商业产品或组织的认可。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
财团gydF4y2Ba
贡献gydF4y2Ba
概念化和J.S.T.方法:W.W.L,中一段和J.S.T.软件和形式分析:W.W.L.中一段调查:R.S, W.W.L,部件,K.L.H。注意,赵硕,,G.G。L.R.K, G.K。,李鸿源,j.t和A.J.M.资源:R.S, W.W.L,部件,K.L.V,地勤人员,司仪,S.F.A。注意,L.E.F, S.L。l ., P.B。,,交流,J.H, A.K.M, V.M, C.P。个终身制,点,硕士,r.a., A.B.M, E.M.B, H.G。”栏目和J.S.T.数据管理:R.S, W.W.L,部件,K.L.V, M.C。注意:和L.E.F.原创作品草案:写作评审和编辑:R.S., W.W.L., C.L.和J.S.T.可视化:R.S., W.W.L.和C.L.监督:R.S., h.g., S.K.和J.S.T.项目管理:R.S.和J.S.T.资金获取:R.S.和J.S.T.人的研究和样本获取:R.S., k.l.v., g.s., m.c., n.b., l.e.f., s.l., l.s., p.b., d.c., a.c., j.h., a.k.m., v.m., c.p., D.S.和A.M.全血RNA-seq分析:G.K.和N.B.流感微中和滴度,流感抗体SPR和SARS-CoV-2中和滴度:j.m., g.g., L.R.K.和J.T.引用-seq数据生成:c.l., A.J.M.和J.C., G.K.和K.L.V. PBMC细胞因子刺激实验:流式细胞仪、CITE-seq、血液RNA-seq、循环蛋白和抗体数据的综合分析:w.w.l和c.l, J.S.T.概念性贡献gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
相互竞争的利益gydF4y2Ba
E.M.B.报告在提交的工作之外,来自Terumo BCT、Abbott Laboratories、Tegus和UptoDate的个人费用和非经济支持;是美国FDA血液产品咨询委员会的成员。本手稿中所表达的任何观点或意见都是作者的观点,基于他自己的科学知识和专业判断;它们不一定代表血液制品咨询委员会的观点或FDA的正式立场,也不约束或强制咨询委员会或FDA接受所表达的观点。其他作者宣称没有利益竞争。gydF4y2Ba
同行评审gydF4y2Ba
同行评审信息gydF4y2Ba
自然gydF4y2Ba感谢Andreas Wack和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2Ba同行评审报告gydF4y2Ba是可用的。gydF4y2Ba
额外的信息gydF4y2Ba
出版商的注意gydF4y2Ba施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
扩展的数据图形和表格gydF4y2Ba
扩展数据图1 covid -19康复参与者与匹配对照组之间的基线差异。gydF4y2Ba
一个gydF4y2Ba,显示诊断以来时间分布的箱形图(TSD;COVR-F (n = 16)和COVR-M (n = 15)中的x轴)。2名无症状COVID-19感染,因此未知TSD的参与者不包括在内。采用双尾Wilcoxon检验确定组间差异的显著性。gydF4y2BabgydF4y2Ba,显示TSD (x轴)与SARS-CoV-2中和滴度(WA1株;y轴)为流感疫苗接种前D0的COVR个体。斯皮尔曼等级相关和未调整gydF4y2BaPgydF4y2Ba数值会显示出来。无症状COVID-19感染的参与者不包括在TSD分析中。阴影区域表示95%置信区间。gydF4y2BacgydF4y2Ba,类似于(gydF4y2BabgydF4y2Ba),但对于D0时由全血细胞计数(y轴)测量的外周血单核细胞百分比。虚线表示HC-F和HC-M的中位水平。gydF4y2BadgydF4y2Ba,类似于(gydF4y2BabgydF4y2Ba),但CD11c+树突状细胞(dc;作为活细胞的一部分;y轴),用流式细胞术测定D0时pmcs的含量。gydF4y2BaegydF4y2Ba, COVR组和HC组稳定基线(预疫苗接种)差异的血液转录组分析。富集图显示不同比较所选基因集的归一化富集评分(NES) (GSEA FDR < 0.05;gydF4y2Ba方法gydF4y2Ba;参见补充表gydF4y2Ba3 gydF4y2BaFDR < 0.05的所有显著基因集)。分别绘制COVR-F与HC女性(HC- f)、COVR-M与HC男性(HC- m)的NES,或两组比较之间的差异(考虑健康性别差异的COVR-M与COVR-F)。阳性(阴性)NES表示基因集得分在第一组高于(低)在比较中列出的第二组。在COVR个体中,只有基线时与TSD不相关的基因集被认为是稳定的。gydF4y2BafgydF4y2Ba,与图相似。gydF4y2Ba1 dgydF4y2Ba,但CD3+细胞(T细胞)的百分比。gydF4y2BaggydF4y2Ba,类似于(gydF4y2BaegydF4y2Ba),但对于单核细胞和T细胞激活基因组的一个子集具有显著的富集(gydF4y2BaPgydF4y2Ba< 0.05)使用指定细胞类型的D0 CITE-seq伪体表达(gydF4y2Ba方法gydF4y2Ba;参见补充表gydF4y2Ba5gydF4y2Ba完整的结果)。gydF4y2BahgydF4y2Ba,显示TSD和前沿基因(LEG)模块得分之间关系的散点图[左两个方框:T细胞激活基因集(BTM-M7.3);右边两个方框:来自BTM M4.0和M11.0基因集LEGs的并集;gydF4y2Ba方法gydF4y2Ba在D0的COVR-F (n = 12)(上一行)和COVR-M (n = 12)(下一行)中使用所示细胞类型的CITE-seq伪体数据。每个点代表一个COVR个体。虚线表示所示比较中性别匹配的HC组在D0处的中位数分数。斯皮尔曼秩相关和gydF4y2BaPgydF4y2Ba数值会显示出来。阴影区域表示95%置信区间。gydF4y2Ba我gydF4y2Ba,(左)比较图中使用的LEGs的经典单核细胞假体模块评分的箱线图。gydF4y2Ba1 fgydF4y2Ba(女性(F)和男性(M)基因集的结合)在急性COVID-19 CITE-seq数据集中gydF4y2Ba等gydF4y2Ba.gydF4y2Ba15gydF4y2Ba.M (n = 50)和F (n = 9)个体均包含在所有三组中(HC n = 13,轻重度n = 21,重度n = 25)。每个点代表一个样本。未经调整的gydF4y2BaPgydF4y2Ba所示的两组比较值。gydF4y2BaPgydF4y2Ba值是使用被调节的生成的gydF4y2BatgydF4y2Ba来自线性模型的统计数据,其中来自同一捐赠者的样本被视为重复样本(gydF4y2Ba方法gydF4y2Ba).(右)图中基因表达的气泡图。gydF4y2Ba1 fgydF4y2Ba右面板为Liu的经典单核细胞CITE-seq数据gydF4y2Ba等gydF4y2Ba.在左边显示的三组病人中。所有图元素均如图所示。gydF4y2Ba1克ydF4y2Ba.gydF4y2Ba
图2自然流感感染后持续的感染后基因表达变化。gydF4y2Ba
一个gydF4y2Ba,翟发表了用于评估两个不同季节自然流感感染前(感染前基线)和几个月后血液基因表达变化的方法示意图gydF4y2Ba等gydF4y2Ba.gydF4y2Ba18gydF4y2Ba在这项研究中,这些基因变化可能与先前COVID-19导致的性别特异性差异有关。gydF4y2BabgydF4y2Ba,散点密度图显示基因表达变化之间的相关性(补充表gydF4y2Ba6gydF4y2Ba) 2009年(x轴)和2010年(y轴)女性自然甲型流感感染(秋季)之前(春季)和之后(春季)(F;左),男性(M;中),以及M与F的对比(右)。所示为斯皮尔曼的秩相关和未调整gydF4y2BaPgydF4y2Ba值。gydF4y2BacgydF4y2Ba, 2009-2010年和2010-2011年秋季(感染前)和春季(感染后)M基因增加的基因集富集图。基因按照有符号的-log排序gydF4y2Ba10gydF4y2Ba-转换(未经调整)的P值在covid -19恢复(COVR)-M vs COVR- f对比的基线,仅使用65岁以下的参与者。标记表示这些基因在流感基因集中的位置。图中gydF4y2Ba一个gydF4y2Ba是使用BioRender创建的。gydF4y2Ba
图3 covid -19康复个体和匹配对照组对流感疫苗接种的分子、细胞和体液反应的性别特异性差异。gydF4y2Ba
一个gydF4y2Ba,类似于扩展数据图。gydF4y2Ba1 egydF4y2Ba但这里显示全血中富集的基因集,比较女性COVID康复(COVR)与健康对照(HC)参与者早期(D1和D7)流感疫苗接种反应(F;对照1),男性(M;对比2)和性别差异(对比2和1;即考虑到健康性别差异的COVR-M与COVR-F;看到gydF4y2Ba方法gydF4y2Ba).绘制的是各组中显示与基线(D-7和D0)相比有显著变化的基因集[例如D1中的COVR-F和HC-F],以及两组在指示时间点上的显著差异(FDR < 0.05;参见补充表gydF4y2Ba5gydF4y2Ba).gydF4y2BabgydF4y2Ba,与图相似。gydF4y2Ba2 egydF4y2Ba,但显示了来自CITE-seq伪体表达数据的指示细胞类型的D0 Hallmark IFNγ响应模块评分。Cd4 = Cd4gydF4y2Ba+gydF4y2BaT细胞;cDC =常规/髓系树突状细胞;B = B细胞。gydF4y2BacgydF4y2Ba,箱形图显示中谷基因的D7全血特征评分gydF4y2Ba等gydF4y2Ba.gydF4y2Ba52gydF4y2Ba其D7/D0折叠变化与D28流感血凝抑制滴度呈正相关。仅包括65岁以下的参与者[COVR-F (n = 15), COVR-M (n = 14), HC-F (n = 16), HC-M (n = 14)]。差异显著性采用双尾Wilcoxon秩和检验。gydF4y2BadgydF4y2Ba,显示全血D1 - D0 Hallmark干扰素γ反应基因集模块评分(x轴)与全血D7 - D0浆母细胞(PB)基因集模块评分相关性的散点图(左y轴;摩纳哥gydF4y2Ba等gydF4y2Ba.gydF4y2Ba53gydF4y2Ba)和流感特异性PB的D7 - D0差异(所有HAgydF4y2Ba+gydF4y2BaCD27gydF4y2Ba+gydF4y2BaCD38gydF4y2Ba+gydF4y2BaCD20gydF4y2Ba低gydF4y2BaCD21gydF4y2Ba低gydF4y2Ba)流式细胞仪频率(右y轴;作为CD19的一部分gydF4y2Ba+gydF4y2BaB细胞)。仅包括年龄< 65岁的研究参与者。斯皮尔曼等级相关和未调整gydF4y2BaPgydF4y2Ba数值会显示出来。gydF4y2BaegydF4y2Ba,显示65岁以下女性(COVR-F和HC-F)季节性流感疫苗中四种毒株的D0(预接种)微中和滴度的箱形图(列)。未经调整的gydF4y2BaPgydF4y2Ba数值来自考虑年龄和种族的线性模型(gydF4y2Ba方法gydF4y2Ba).gydF4y2BafgydF4y2Ba,类似于(gydF4y2BaegydF4y2Ba),但适用于65岁以下男性(COVR-M和HC-M)。gydF4y2BaggydF4y2Ba女性(左)和男性(右)分别为接种后D28时(疫苗中四种毒株之间)的最大标准化流感疫苗滴度。未经调整的gydF4y2BaPgydF4y2Ba数值来自线性回归模型,考虑年龄、种族、流感疫苗接种史和基线流感滴度(gydF4y2Ba方法gydF4y2Ba).未经调整的gydF4y2BaPgydF4y2Ba数值会显示出来。所有图元素均如图所示。gydF4y2Ba2 gydF4y2Ba.gydF4y2Ba
扩展数据图4 GPR56gydF4y2Ba+gydF4y2Ba像CD8这样的虚拟内存gydF4y2Ba+gydF4y2Ba在covid -19康复的男性中,T细胞有助于增加第1天的IFNγ反应。gydF4y2Ba
一个gydF4y2Ba,表示第0天(D0)日志之间的相关性的散点图gydF4y2Ba2 gydF4y2Ba早期效应子样CD8的频率gydF4y2Ba+gydF4y2Ba用流式细胞术测定T细胞(作为活淋巴细胞的部分;见《补充表》人口50gydF4y2Ba9gydF4y2Ba和补充图。gydF4y2Ba1克ydF4y2Ba;x轴)和OLINK平台测量的covid -19康复(COVR)女性血清干扰素γ (IFNγ)蛋白水平的变化(D1 - D0) (y轴)(COVR- f;top, n = 14)和COVR雄性(COVR- m;底;n = 11)。斯皮尔曼秩相关和gydF4y2BaPgydF4y2Ba数值会显示出来。gydF4y2BabgydF4y2Ba,类似于gydF4y2Ba一个gydF4y2Ba但显示了D0日志之间的相关性gydF4y2Ba2 gydF4y2Ba早期效应子样CD8的频率gydF4y2Ba+gydF4y2Ba用流式细胞术测定T细胞(活淋巴细胞的百分比;x轴)和Hallmark IFNγ反应基因集全血特征评分的变化(D1 - D0) (y轴)。gydF4y2BacgydF4y2Ba,比较D0和D1伪体IFNγ基因的箱线图(gydF4y2BaIFNGgydF4y2Ba)表达(y轴)gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba新兴市场人口HC-F (n = 8), COVR-F (n = 12), HC-M (n = 8)和COVR-M (n = 12)。显著性由考虑年龄、种族和流感疫苗接种史的线性模型确定(gydF4y2Ba方法gydF4y2Ba).gydF4y2BadgydF4y2Ba,条形图显示gydF4y2BatgydF4y2Ba疫苗诱导的变化(D1 - D0)的统计gydF4y2BaIFNGgydF4y2Ba表达式使用CITE-seq伪批量数据(x轴)在GPR56gydF4y2Ba+gydF4y2Ba和GRP56gydF4y2Ba−gydF4y2BaCD8gydF4y2Ba+gydF4y2BaHC-F EM (n = 8), COVR-F (n = 12), HC-M (n = 8)和COVR-M (n = 12)。*gydF4y2BaPgydF4y2Ba< 0.05,具体数值见gydF4y2Ba(c)gydF4y2Ba.gydF4y2BaegydF4y2Ba,显示GPR56之间相关性的散点图gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaEM细胞频率(作为总CD8的分数gydF4y2Ba+gydF4y2BaEM在CITE-seq数据中;x轴)和BTM-M7.3的CD8 T细胞激活特征评分gydF4y2Ba+gydF4y2BaEM细胞计算使用CITE-seq假体基因表达数据(y轴)。斯皮尔曼相关gydF4y2BaPgydF4y2Ba数值会显示出来。阴影部分表示95%置信区间。gydF4y2BafgydF4y2Ba,与无花果有关。gydF4y2Ba3 hgydF4y2Ba但只有CD8显示CD45RA和CD45ROgydF4y2Ba+gydF4y2BaTEMRA细胞包括作为额外的比较物。gydF4y2BaggydF4y2Ba,(左)不同CD8下T细胞受体(TCR)克隆性的Circos图gydF4y2Ba+gydF4y2BaT细胞亚群在D0。外圈的节段代表不同的CD8gydF4y2Ba+gydF4y2BaT细胞群。内圈部分代表COVR和HC参与者的男性(M)和女性(F)。灰线连接每个个体内共享相同CDR3序列的克隆。为了可视化,降低了细胞子集的采样(gydF4y2Ba方法gydF4y2Ba).(右)显示每种CD8在D0处的香农熵指数(y轴)的箱形图gydF4y2Ba+gydF4y2Ba人群。采用双尾Wilcoxon检验确定差异显著性。Shannon熵指数用于评估每个样本的TCR基因库多样性。指数越高表明多样性越高(即Circos图中显示的共享无性系越少)。EM,效应器存储器;CM,中央存储器;TEMRA, EM细胞重表达CD45RA。gydF4y2BahgydF4y2Ba,(左)GPR56的TCR克隆Circos图gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2Ba不同时间点的EM细胞。外圈的部分代表研究中的不同天数(D0, D1, D28)。内圈的部分代表COVR和HC参与者的男性(M)和女性(F)。灰线连接每个样本中共享相同CDR3序列的克隆。时间点为可视化目的而降低采样(gydF4y2Ba方法gydF4y2Ba).(右)显示TCR克隆性在每个指定时间点(D0, D1, D28;x轴)为GPR56gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaEM T细胞(左)和GPR56gydF4y2Ba+gydF4y2BaCD8gydF4y2Ba+gydF4y2BaTEMRA(右)。采用双尾Wilcoxon检验确定差异显著性。gydF4y2Ba我gydF4y2Ba,(左)类似于(gydF4y2BahgydF4y2Ba),但显示了不同时间点之间的共享克隆(外圆部分)。内圈中的段表示每个个体的唯一克隆。克隆和连接共享克隆的线是彩色的。细胞数量少于30个的样本被过滤掉以进行可视化。(右)Circos图(左)显示每个参与者在D0、D1和D28处每个克隆的频率(y轴)折线图。gydF4y2BaPgydF4y2Ba用配对Wilcoxon检验的-值比较了D0、D1和D28间克隆频率的差异。gydF4y2BajgydF4y2Ba,与无花果有关。gydF4y2Ba3我gydF4y2Ba还显示了IFNγ的频率gydF4y2Ba+gydF4y2BaNK,干扰素γgydF4y2Ba+gydF4y2BaCD45RAgydF4y2Ba+gydF4y2BaCD45ROgydF4y2Ba+gydF4y2BaTEMRA CD8gydF4y2Ba+gydF4y2BaT细胞和IFNγgydF4y2Ba+gydF4y2BaIL-15体外刺激MAIT细胞。所有图元素均如图所示。gydF4y2Ba3 gydF4y2Ba.未经调整的gydF4y2BaPgydF4y2Ba数值会显示出来。gydF4y2Ba
图5 covid -19康复患者接种流感疫苗后免疫状态的变化。gydF4y2Ba
一个gydF4y2Ba,先天免疫受体(IIR)特征基因水平差异的分布(见图)。gydF4y2Ba1 fgydF4y2Ba),分别在女性(F)和男性(M)的经典单核细胞中[在每个基因水平上以z-score表示,捕捉指定时间点(从上到下:D0、D1和D28)的COVID康复(COVR)与D0时健康对照(HC)之间的平均差异;看到gydF4y2Ba方法gydF4y2Ba].红色虚线表示分布的中位数。底部的黑色标记代表单个基因,彩色圆点突出感兴趣的特定基因。用成对双尾Wilcoxon秩和检验确定与D0的差异的显著性。gydF4y2BabgydF4y2Ba,类似于(gydF4y2Ba一个gydF4y2Ba),但对于非经典单核细胞(见图。gydF4y2Ba1克gydF4y2Ba).gydF4y2BacgydF4y2Ba,与图相似。gydF4y2Ba4 dgydF4y2Ba但对于非经典单核细胞(见图。gydF4y2Ba1克gydF4y2Ba对于非经典单核细胞中的先天受体特征)。gydF4y2BadgydF4y2Ba,与图相似。gydF4y2Ba4 bgydF4y2Ba但仅对COVR-F(红色)和COVR-M(蓝色)进行检测,并在每个图的顶部显示基因集。显示基因集的经典单核细胞LEG模块评分(y轴)的箱形图,来自补充图。gydF4y2Ba2 gydF4y2Ba抗原呈递相关基因集,标志性炎症反应,标志性TNF-α信号通路NF-κB,以及来自Reyes的MS-1信号gydF4y2Ba等gydF4y2Ba.gydF4y2Ba17gydF4y2Ba.在刘的急性COVID-19患者中,发现前三个基因组中的leg被抑制gydF4y2Ba等gydF4y2Ba.gydF4y2Ba15gydF4y2Ba.gydF4y2BaegydF4y2Ba,类似于(gydF4y2BadgydF4y2Ba),但对非经典单核细胞。所有图元素均如图所示。gydF4y2Ba4 gydF4y2Ba.未经调整的gydF4y2BaPgydF4y2Ba数值会显示出来。gydF4y2Ba
补充信息gydF4y2Ba
补充信息gydF4y2Ba
补充讨论,补充参考文献和补充图1-7。gydF4y2Ba
补充表1gydF4y2Ba
基线参数TSD关联结果(所有参与者)。gydF4y2Ba
补充表2gydF4y2Ba
基线参数DE结果(所有参与者)。gydF4y2Ba
补充表3gydF4y2Ba
基线全血GSEA结果(所有参与者)。gydF4y2Ba
补充表4gydF4y2Ba
重要的(gydF4y2BaPgydF4y2Ba< 0.05)单细胞时间点DE结果。gydF4y2Ba
补充表5gydF4y2Ba
时间点全血和单细胞GSEA结果。gydF4y2Ba
补充表6gydF4y2Ba
翟等自然流感感染队列DE结果。gydF4y2Ba
补充表7gydF4y2Ba
流感疫苗接种后时间点DE结果。gydF4y2Ba
补充表8gydF4y2Ba
流感疫苗抗体滴度和SPR D0和D28模型结果。gydF4y2Ba
补充表9gydF4y2Ba
36色Cytek面板的门控人群。gydF4y2Ba
补充表10gydF4y2Ba
COVR-M CD8gydF4y2Ba+gydF4y2BaEM标记来自CITE-seq。gydF4y2Ba
补充表11gydF4y2Ba
用于CITE-seq的蛋白质表面标记面板,BioLegend Total-seq-C人类通用鸡尾酒,V1.0。gydF4y2Ba
补充表12gydF4y2Ba
抗体和流感探针用于B细胞流动面板。gydF4y2Ba
补充表13gydF4y2Ba
抗体和活性染料用于36色Cytek流式细胞仪面板。gydF4y2Ba
补充表14gydF4y2Ba
T细胞刺激试验中使用的抗体和活性染料。gydF4y2Ba
权利和权限gydF4y2Ba
根据与作者或其他权利持有人签订的出版协议,自然或其许可方(例如,社会或其他合作伙伴)对本文拥有排他性权利;作者对这篇文章接受的手稿版本的自我存档仅受此类出版协议的条款和适用法律的约束。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
斯派克,R.,刘伟伟,刘,C.。gydF4y2Baet al。gydF4y2Ba流感疫苗接种显示既往轻度COVID-19的性别二态印记。gydF4y2Ba自然gydF4y2Ba614gydF4y2Ba, 752-761(2023)。https://doi.org/10.1038/s41586-022-05670-5gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
发行日期gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1038/s41586-022-05670-5gydF4y2Ba
这篇文章被引用gydF4y2Ba
疫苗接种后血清细胞因子水平与突破性流感感染相关gydF4y2Ba
科学报告gydF4y2Ba(2023)gydF4y2Ba
评论gydF4y2Ba
通过提交评论,您同意遵守我们的gydF4y2Ba条款gydF4y2Ba而且gydF4y2Ba社区指导原则gydF4y2Ba.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2Ba