摘要gydF4y2B一个
在目前富含肉类的饮食中增加当地生产的植物蛋白的比例,可以大大减少温室气体排放和生物多样性的丧失gydF4y2B一个1gydF4y2B一个.然而,植物蛋白质生产受到缺乏与大豆同等农艺价值的冷季豆科植物的阻碍gydF4y2B一个2 gydF4y2B一个.蚕豆(gydF4y2B一个蚕豆根尖gydF4y2B一个L.)产量潜力高,非常适合在温带地区种植,但基因组资源稀缺。在这里,我们报告了蚕豆基因组的高质量染色体尺度组装,并表明它已经通过反转录转座子和卫星重复序列的扩增和消除率之间的不平衡扩展到巨大的13 Gb大小。基因和重组事件均匀地分散在染色体上,考虑到基因组大小,基因空间非常紧凑,尽管有大量的拷贝数变化由串联复制驱动。为了展示基因组序列的实际应用,我们开发了一种靶向基因分型试验,并使用高分辨率全基因组关联分析来解剖种子大小和种门颜色的遗传基础。这些资源构成了一个基于基因组学的蚕豆育种平台,使育种家和遗传学家能够加速改善地中海、亚热带和北温带农业生态区的可持续蛋白质生产。gydF4y2B一个
主要gydF4y2B一个
蚕豆(gydF4y2B一个蚕豆根尖gydF4y2B一个L。2gydF4y2B一个ngydF4y2B一个在近东被驯化了一万多年gydF4y2B一个英国石油公司gydF4y2B一个3.gydF4y2B一个,gydF4y2B一个4 gydF4y2B一个它广泛的适应性、轮作修复作物的价值和高营养密度使它成为除南极洲以外所有大陆都种植的全球作物gydF4y2B一个5gydF4y2B一个.尽管它在全球具有重要意义,但还没有发现现存的野生祖先。尽管如此,新石器时代烧焦的野生蚕豆种子的发现表明,狩猎采集者在驯化之前就使用了这个物种,并可能在黎凡特进行了驯化gydF4y2B一个3.gydF4y2B一个.几个近亲物种的存在(gydF4y2B一个野豌豆属narbonensisgydF4y2B一个,gydF4y2B一个野豌豆属巴勒斯坦gydF4y2B一个而且gydF4y2B一个野豌豆属kalakhensisgydF4y2B一个)在同一地区gydF4y2B一个6gydF4y2B一个给了我们找到野生祖先的希望。蚕豆在种子大小上表现出如此极端的变化,以至于一些分类学家将其定义为原始的小种子“paucijuga”形式gydF4y2B一个7gydF4y2B一个或者小种子的“小”形式gydF4y2B一个8gydF4y2B一个作为中大型“蚕豆”类型的独立亚种。然而,这些形式之间没有繁殖障碍,这意味着“大”、“小”、“马科”和“少丘加”形式现在被认为是几千年来人类对生长习惯和种子大小持续选择的植物类型gydF4y2B一个9gydF4y2B一个.蚕豆在21世纪仍然具有重要意义,因为人类正在努力用植物性替代品取代肉类或牛奶蛋白,以降低农业温室气体排放gydF4y2B一个10gydF4y2B一个.它是所有谷物豆类中产量最高的gydF4y2B一个11gydF4y2B一个与其他冷季豆类(如豌豆、扁豆和鹰嘴豆)相比,它具有良好的蛋白质含量(约29%),使其成为满足未来蛋白质需求的合适候选人。蚕豆具有较高的生物固氮率gydF4y2B一个12gydF4y2B一个花蜜丰富,有利于传粉的花朵持续时间长gydF4y2B一个13gydF4y2B一个提供重要的生态系统服务,这意味着蚕豆种植越来越被视为可持续集约化战略的关键。相反,它的部分异体交配系统和估计的13gb基因组大小,加上低种子繁殖率,使它成为育种家具有挑战性的目标gydF4y2B一个14gydF4y2B一个.蚕豆基因组学和前期育种研究已取得重大进展。第一个蚕豆转录组的挖掘和基于单核苷酸多态性(SNP)的遗传图谱的开发,显示出与模式豆科植物的强共线性,为鉴定WD40转录因子奠定了基础gydF4y2B一个零Tannin1gydF4y2B一个轨迹gydF4y2B一个15gydF4y2B一个,而结合高分辨率作图,转录组学和代谢组学方法导致克隆gydF4y2B一个VC1gydF4y2B一个基因控制了抗营养物质vicine和jine的种子含量,为在人类食物链中更安全地开发这种作物铺平了道路gydF4y2B一个16gydF4y2B一个.然而,参考基因组序列的缺乏使这些研究变得非常复杂,迫切需要改进蚕豆基因组资源来加速作物改良。gydF4y2B一个
巨型蚕豆基因组序列gydF4y2B一个
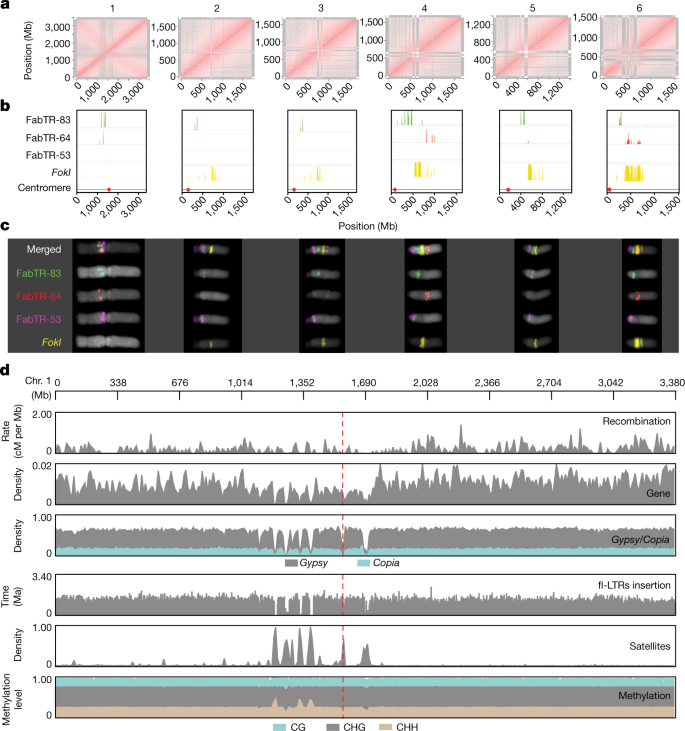
13 gb蚕豆基因组(2gydF4y2B一个ngydF4y2B一个= 2gydF4y2B一个xgydF4y2B一个= 12)是最大的二倍体大田作物之一(扩展数据图。gydF4y2B一个1 a, bgydF4y2B一个),其主要重复家族成员较长gydF4y2B一个17gydF4y2B一个,gydF4y2B一个18gydF4y2B一个(高达25kb),而不是类似大小的多倍体谷物基因组gydF4y2B一个19gydF4y2B一个.它的六条染色体中最大的一条相当于一个完整的人类基因组。虽然有助于细胞遗传学gydF4y2B一个20.gydF4y2B一个在长而准确的解读出现之前,这些特性使得基因组组装非常具有挑战性。我们选择Hedin/2自交系作为参考基因型,因为它具有较高的自育性和生产力,结合早熟春季习性和优异的纯合子程度。我们用PacBio HiFi长reads对其基因组进行了20倍覆盖测序,组装了11.9 Gb的序列,其中超过一半的序列由长度超过2.7 Mb的contigs表示(扩展数据表gydF4y2B一个1gydF4y2B一个).遗传图谱提供的连锁信息(补充表gydF4y2B一个1gydF4y2B一个)和染色体构象捕获测序(Hi-C)数据将11.2 Gb(94%)放入染色体假分子中(图。gydF4y2B一个1gydF4y2B一个和补充图。gydF4y2B一个1gydF4y2B一个).着丝粒组蛋白H3的染色质免疫沉淀测序确定了着丝粒在Hedin/2组合中的位置,臂比与核型一致(补充图)。gydF4y2B一个1 bgydF4y2B一个).单稳心染色体1是唯一一条采用Rabl构型的染色体,从Hi-C相互作用图中该染色体上主对角线和反对角线的存在可以明显看出(图2)。gydF4y2B一个1gydF4y2B一个).这支持了这样一种观点,即染色体臂需要具有大约相等的大小才能在间期空间上并置。Hi-C接触矩阵的一些区域由于缺乏映射的短reads而为空。gydF4y2B一个1gydF4y2B一个).这些白色区域与巨大(高达752 Mb)卫星阵列的位置一致,并与这些重复序列的细胞学图对齐。gydF4y2B一个1 b, cgydF4y2B一个).用merquy进行装配评估gydF4y2B一个21gydF4y2B一个显示基因组的完整性为96.3%,一致质量值为60.5,表明我们的Hedin/2组装具有很高的准确性(扩展数据表gydF4y2B一个1gydF4y2B一个和扩展数据图。gydF4y2B一个1 cgydF4y2B一个).遗传图和物理图之间良好的共线一致性进一步验证了contigs与染色体的精确分配(补充图)。gydF4y2B一个1gydF4y2B一个).此外,长终端重复(LTR)装配指数得分为10.5,支持我们的装配的连续性。我们还收集了德国品种“蒂芙尼”的HiFi数据(十倍覆盖率),并将这些数据组装成一组N50为1.6 Mb的contigs,覆盖11.4 Gb(扩展数据表)gydF4y2B一个1gydF4y2B一个).与Hedin/2类似,merquury评估支持Tiffany组件的高质量(扩展数据表gydF4y2B一个1gydF4y2B一个和扩展数据图。gydF4y2B一个1 dgydF4y2B一个).这种水平的完整性和邻近性足以将contigs排列成Hedin/2参考文献(扩展数据表)指导的假分子gydF4y2B一个1gydF4y2B一个和扩展数据图。gydF4y2B一个2 gydF4y2B一个).在未来,Hedin/2组装有望成为蚕豆泛基因组的核心。gydF4y2B一个
一个gydF4y2B一个,组装染色体的染色体内接触矩阵。红色强度表示每条染色体上1mb窗口之间的归一化Omni-C Hi-C链接。1号染色体的反对角线模式代表Rabl配置。gydF4y2B一个bgydF4y2B一个卫星重复序列的主要家族分布(绿色为FabTR-83,红色为FabTR-64,品红为FabTR-53,品红为FabTR-53)gydF4y2B一个FokIgydF4y2B一个在黄色)。gydF4y2B一个cgydF4y2B一个多色荧光原位杂交显示中期染色体上卫星重复序列主要家族的分布。gydF4y2B一个dgydF4y2B一个,基因组组分的分布,包括重组(cM / Mb),基因密度,LTR逆转录转座子gydF4y2B一个吉普赛gydF4y2B一个而且gydF4y2B一个CopiagydF4y2B一个1号染色体上有全长ltr -逆转录转座子(fl-LTR)插入、卫星重复和DNA甲基化(CH, CHG和CHH上下文)。红色虚线表示着丝粒的位置。gydF4y2B一个
基因组大小扩张的驱动力gydF4y2B一个
Hedin/2的基因组序列用9个不同组织的RNA测序数据进行了注释(补充表gydF4y2B一个2 gydF4y2B一个),结果共得到34221个蛋白质编码基因(补充表gydF4y2B一个3.gydF4y2B一个).在蒂芙尼组装中也预测了类似数量的基因模型(34,043)。预测的Hedin/2基因模型根据BUSCO度量捕获了胚胎植物中保存的96%的单拷贝同源基因(补充表gydF4y2B一个4 gydF4y2B一个).基因密度沿染色体是均匀的(除了卫星DNA阵列的位置),没有近端-远端梯度通常观察到的草染色体gydF4y2B一个22gydF4y2B一个.减数分裂重组显示了类似的分布,平均27个基因每厘摩根(图。gydF4y2B一个1 dgydF4y2B一个和扩展数据图。gydF4y2B一个3.gydF4y2B一个).因此,尽管蚕豆的基因组很大,但它可能比谷物更容易进行基因定位,谷物中多达三分之一的基因被锁定在非重组的中心周围区域gydF4y2B一个22gydF4y2B一个.基因顺序与其他豆科植物高度共线和共线(图2)。gydF4y2B一个2gydF4y2B一个).为了进一步验证基因注释,我们对齐了262gydF4y2B一个Medicago truncatulagydF4y2B一个与根瘤菌或丛枝菌根真菌共生相关的基因,并发现了它们的假定同源基因。此外,通过RNA测序,我们验证了这些基因的很大一部分对接种有反应,正如预期的那样gydF4y2B一个23gydF4y2B一个,gydF4y2B一个24gydF4y2B一个,gydF4y2B一个25gydF4y2B一个(补充表gydF4y2B一个5gydF4y2B一个).gydF4y2B一个
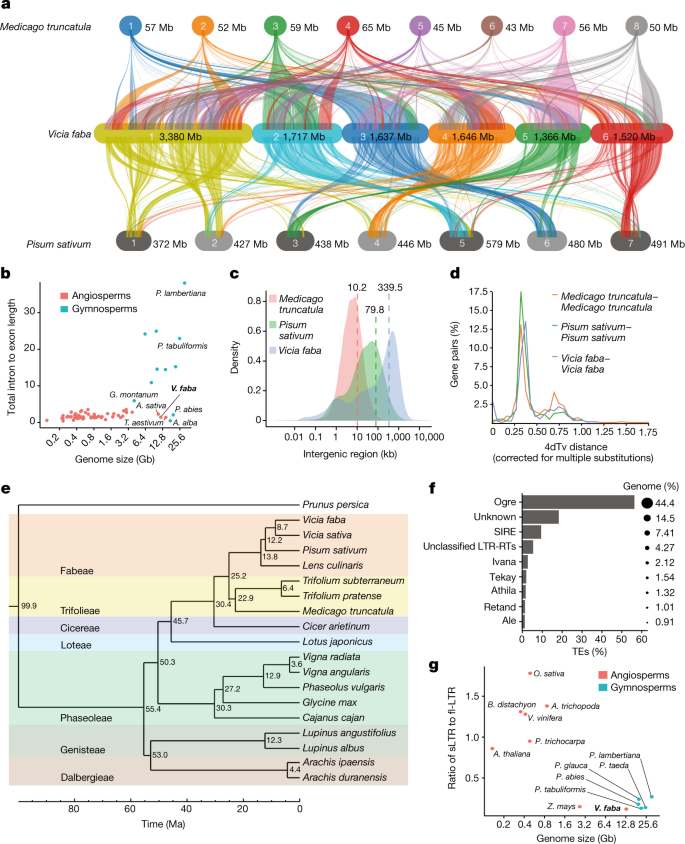
一个gydF4y2B一个,蚕豆(中)与gydF4y2B一个MedicagogydF4y2B一个(上)和豌豆(下)。gydF4y2B一个bgydF4y2B一个,被子植物和裸子植物中内含子与外显子的长度。gydF4y2B一个答:阿尔巴gydF4y2B一个,gydF4y2B一个冷杉属阿尔巴gydF4y2B一个;gydF4y2B一个答:漂白亚麻纤维卷gydF4y2B一个,gydF4y2B一个燕麦属漂白亚麻纤维卷gydF4y2B一个;gydF4y2B一个g . montanumgydF4y2B一个,gydF4y2B一个水杨梅属植物gydF4y2B一个montanumgydF4y2B一个;gydF4y2B一个p .冷杉属gydF4y2B一个,gydF4y2B一个挪威云杉gydF4y2B一个;gydF4y2B一个p . lambertianagydF4y2B一个,gydF4y2B一个松果体lambertianagydF4y2B一个;gydF4y2B一个p . tabuliformisgydF4y2B一个,gydF4y2B一个松果体tabuliformisgydF4y2B一个;gydF4y2B一个t . aestivumgydF4y2B一个,gydF4y2B一个小麦gydF4y2B一个.gydF4y2B一个cgydF4y2B一个,豆科植物基因间区长度。gydF4y2B一个dgydF4y2B一个,副同源基因对四重退化位点(4dTv)的转位率分布。gydF4y2B一个egydF4y2B一个,蚕豆与凤蝶科其他豆科植物的系统发育关系。分支上的数字表示估计的发散时间(Ma)。gydF4y2B一个fgydF4y2B一个蚕豆逆转录转座子的组成按科概述。gydF4y2B一个ggydF4y2B一个裸子植物和被子植物sLTR与fl-LTR的比值与基因组大小的关系。其他物种的比率从参考文献中检索。gydF4y2B一个32gydF4y2B一个.gydF4y2B一个答:trichopodagydF4y2B一个,gydF4y2B一个Amborella trichopodagydF4y2B一个;gydF4y2B一个b . distachyongydF4y2B一个,gydF4y2B一个Brachypodium distachyongydF4y2B一个;gydF4y2B一个o .漂白亚麻纤维卷gydF4y2B一个,gydF4y2B一个栽培稻gydF4y2B一个;gydF4y2B一个p .冷杉属gydF4y2B一个,gydF4y2B一个挪威云杉gydF4y2B一个;gydF4y2B一个p . glaucagydF4y2B一个,gydF4y2B一个云杉glaucagydF4y2B一个;gydF4y2B一个p . taedagydF4y2B一个,gydF4y2B一个松果体taedagydF4y2B一个;gydF4y2B一个p . trichocarpagydF4y2B一个,gydF4y2B一个杨树trichocarpagydF4y2B一个;gydF4y2B一个诉酿酒用葡萄gydF4y2B一个,gydF4y2B一个葡萄gydF4y2B一个;gydF4y2B一个z梅斯gydF4y2B一个,gydF4y2B一个玉米gydF4y2B一个.gydF4y2B一个
与裸子植物相比,它们有着同样巨大的基因组gydF4y2B一个26gydF4y2B一个,gydF4y2B一个27gydF4y2B一个蚕豆基因中的内含子并不比基因组较小的被子植物中的内含子大(图2)。gydF4y2B一个2 bgydF4y2B一个),但基因间隙更大(图;gydF4y2B一个2摄氏度gydF4y2B一个).此外,蚕豆的多拷贝基因家族数量与相关的二倍体物种相似(补充表gydF4y2B一个6gydF4y2B一个和补充图。gydF4y2B一个2 gydF4y2B一个),而大豆则被认为是部分二倍体的四倍体gydF4y2B一个28gydF4y2B一个.同样,同源和同源基因对之间的核苷酸替代率表明,蚕豆谱系中最后一次全基因组复制(WGD)事件发生在5500万年前(Ma),远远早于从其他papilionideae中分裂出来gydF4y2B一个29gydF4y2B一个(无花果。gydF4y2B一个2 egydF4y2B一个和补充图。gydF4y2B一个3.gydF4y2B一个),这个分类单元还包括豌豆和扁豆(gydF4y2B一个镜头culinarisgydF4y2B一个),与蚕豆的差异分别在12.2 Ma和13.8 Ma左右。虽然我们没有在蚕豆中发现最近WGD的证据,但与豌豆和扁豆相比,更多的基因被串联复制(补充图)。gydF4y2B一个4gydF4y2B一个).这些重复发生在最后一次WGD之后,且发生时间晚于序列重复gydF4y2B一个拟南芥gydF4y2B一个而且gydF4y2B一个m . truncatulagydF4y2B一个(补充图。gydF4y2B一个4 bgydF4y2B一个),这两个物种的基因组也富含此类事件,并与最近的转座因子(TE)扩张相吻合。Hedin/2和Tiffany共1108个串联复制基因共聚簇,部分共聚簇拷贝数不同。值得注意的是,与农学相关的豆科血红蛋白家族已经扩大(补充表gydF4y2B一个7gydF4y2B一个).尽管如此,没有谱系特异性的WGD或广泛的基因家族扩展意味着重复元件的增殖在很大程度上解释了为什么蚕豆基因组比它的近亲普通野豌豆基因组大7倍以上(gydF4y2B一个野豌豆属漂白亚麻纤维卷gydF4y2B一个)gydF4y2B一个30.gydF4y2B一个.gydF4y2B一个
大约79%的Hedin/2组装被注释为转座子衍生的(补充表gydF4y2B一个8gydF4y2B一个).到目前为止,最大的一组是LTR逆转录转座子(RLX),占基因组序列的63.7%。其他组的TEs仅代表基因组的一小部分(补充表gydF4y2B一个8gydF4y2B一个).在RLX中,那些gydF4y2B一个吉普赛gydF4y2B一个(RLG)超级家族数量超过gydF4y2B一个CopiagydF4y2B一个(RLC)元素比大于2:1(图;gydF4y2B一个1 dgydF4y2B一个和扩展数据图。gydF4y2B一个3.gydF4y2B一个).的gydF4y2B一个食人魔gydF4y2B一个的家庭gydF4y2B一个吉普赛gydF4y2B一个单是元素就占了基因组的近一半(44%),这证实了它在豆科基因组大小的主要决定因素的地位gydF4y2B一个18gydF4y2B一个(无花果。gydF4y2B一个2 fgydF4y2B一个).单个元素的大长度(最高可达35kbgydF4y2B一个食人魔gydF4y2B一个32kb的gydF4y2B一个陛下gydF4y2B一个(分别为最长和第二长元素),加上它们的丰度,部分解释了蚕豆基因组的大尺寸(补充图。gydF4y2B一个5gydF4y2B一个).此外,大量不同的卫星重复家族的单体序列和基因组丰度不同gydF4y2B一个31gydF4y2B一个占总装配长度的9.4%,拥有最丰富的卫星家族gydF4y2B一个FokIgydF4y2B一个占4% (0.475 Gb)。gydF4y2B一个FokIgydF4y2B一个,与其他几个高度扩增的卫星一起,在蚕豆染色体上形成显著的异色带(图2)。gydF4y2B一个1 cgydF4y2B一个).TE密度沿所有6条染色体显著不变,反映基因密度和重组率,与卫星阵列的密度相反(图2)。gydF4y2B一个1 dgydF4y2B一个和扩展数据图。gydF4y2B一个3.gydF4y2B一个).gydF4y2B一个
逆转录转座子作为全长拷贝的持久性可以告诉我们基因组大小通过逆转录转座子扩大和通过重组消除缩小之间的平衡。将单链链(sLTRs)建模为单个元素的单链链(lts)之间重组的产物,并假设正则gydF4y2B一个食人魔gydF4y2B一个395,657个sLTRs由4161 bp的lts和11,655 bp的内部结构域组成,从基因组中损失了6.26 Gb的DNA(占当前组装大小的55.6%)。如果不同个体的ltr之间进行重组,这种损失将更大gydF4y2B一个食人魔gydF4y2B一个元件以及DNA双链断裂修复介导的内部截断被考虑。然而,与基因组较小的植物物种不同,蚕豆中相对于全长ltr的数量,sLTRs通常要少得多,类似于大型裸子植物基因组(图2)。gydF4y2B一个2 ggydF4y2B一个),表明RLX的清除比扩散慢gydF4y2B一个32gydF4y2B一个.的gydF4y2B一个诉漂白亚麻纤维卷gydF4y2B一个1.65 Gb的基因组早前报道占22.5%gydF4y2B一个食人魔gydF4y2B一个元素,并且每个全长有1.6个sltrgydF4y2B一个食人魔gydF4y2B一个,比发现的要多得多gydF4y2B一个诉较好gydF4y2B一个18gydF4y2B一个.gydF4y2B一个
高效的全基因组甲基化gydF4y2B一个
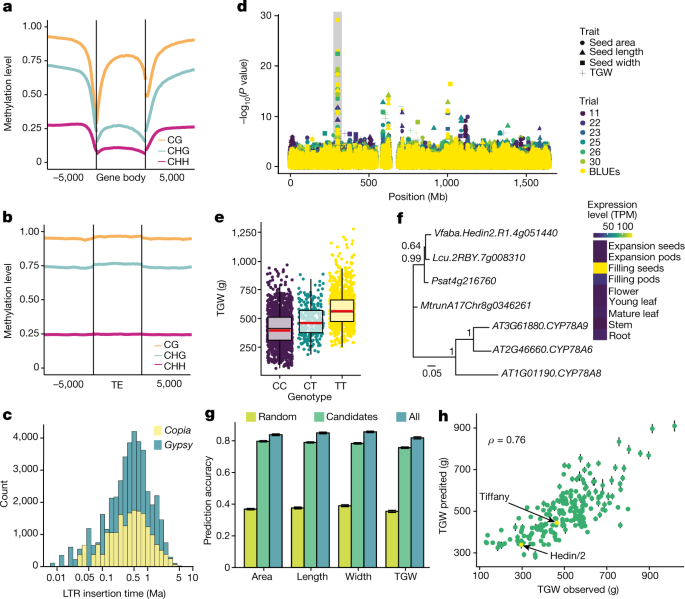
除了相对较慢的RLX消除速度外,也有可能是低水平的甲基化通过低效率的沉默加速了TE的增殖。我们发现蚕豆基因组中的大多数胞嘧啶被甲基化:CG中95.8%,CHG中88.2%,CHH中14%(图2)。gydF4y2B一个1 dgydF4y2B一个和扩展数据图。gydF4y2B一个3.gydF4y2B一个),使其成为甲基化程度最高的植物基因组之一gydF4y2B一个32gydF4y2B一个.基因体甲基化遵循典型模式(图;gydF4y2B一个3gydF4y2B一个)在其他植物中也有gydF4y2B一个33gydF4y2B一个CG甲基化在内部外显子和内含子中富集(补充图。gydF4y2B一个6gydF4y2B一个),与第一外显子的低甲基化相反,可能与转录抑制有关gydF4y2B一个34gydF4y2B一个.基因体甲基化水平高的基因在幼叶组织中表达较高(补充图。gydF4y2B一个6 bgydF4y2B一个),亦较长(平均3.3 kb)。RLX的主要超族元素,gydF4y2B一个吉普赛gydF4y2B一个而且gydF4y2B一个CopiagydF4y2B一个,分别占据了48%和11%的基因组。它们的甲基化程度也很高,在它们体内的甲基化程度比它们侧翼的甲基化程度更高。gydF4y2B一个3 bgydF4y2B一个).最近的转座子爆发发生在不到1 Ma的时间内,但许多结构完整的元素都在300万到500万年之间(图5)。gydF4y2B一个3 cgydF4y2B一个).在所有三个序列环境中,年轻插入和年老插入都无一例外地发生甲基化(扩展数据图)。gydF4y2B一个4gydF4y2B一个).与其他植物类群相比gydF4y2B一个35gydF4y2B一个, RLX插入次数与甲基化水平不耦合。CHH甲基化升高的显著岛屿也与丰富的卫星重复FabTR-83相吻合(扩展数据图)。gydF4y2B一个4 bgydF4y2B一个),占基因组的1.1%。一般来说,蚕豆的甲基化机制似乎功能齐全,有效地甲基化了所有类别的重复元素,这表明甲基化缺陷不太可能在基因组扩增中起作用。这得到了对rna定向DNA甲基化相关基因的研究的支持gydF4y2B一个36gydF4y2B一个,其基因拷贝数在蚕豆中与之相似gydF4y2B一个诉漂白亚麻纤维卷gydF4y2B一个,豌豆和扁豆(补充表gydF4y2B一个9gydF4y2B一个).gydF4y2B一个
一个gydF4y2B一个蛋白质编码基因的DNA甲基化水平的全球分布,包括转录起始位点上游和转录结束位点下游的5kb区域。gydF4y2B一个bgydF4y2B一个, TEs及其5-kb侧翼区域的DNA甲基化模式。gydF4y2B一个cgydF4y2B一个,分布gydF4y2B一个CopiagydF4y2B一个(RLC)和gydF4y2B一个吉普赛gydF4y2B一个(RLG)基于插入时间的逆转录转座子。gydF4y2B一个dgydF4y2B一个结合4号染色体种子面积、种子宽、种子长和千粒重(TGW)的GWAS曼哈顿图分析。BLUE,最佳线性无偏估计器。gydF4y2B一个egydF4y2B一个,其中SNP标记的TGW效应图gydF4y2B一个Vfaba.Hedin2.R1.4g051440gydF4y2B一个在4号染色体的299,823,118号位置(部分用灰色条标出)gydF4y2B一个dgydF4y2B一个).gydF4y2B一个ngydF4y2B一个= 2499个数据点分布在6个试验中。在盒状图中,水平的黑色中心线表示中位数,红线表示平均值,盒子的范围从第一到第三四分位数,垂直的黑线延伸到1.5×四分位数范围内的最小或最大点。gydF4y2B一个fgydF4y2B一个,系统发育树显示之间的关系gydF4y2B一个Vfaba.Hedin2.R1.4g051440gydF4y2B一个它的同类豌豆,扁豆,gydF4y2B一个MedicagogydF4y2B一个而且gydF4y2B一个拟南芥gydF4y2B一个Hedin/2 in transcripts per million (TPM)在9种不同组织中的表达水平。分支长度以每个位点的替换数来衡量。分支旁边的数字表示支持引导。gydF4y2B一个ggydF4y2B一个,利用15个随机选择的SNPs(随机)、15个种子大小相关标记(候选)或所有标记(全部)对种子性状进行基因组最佳线性无偏预测精度。gydF4y2B一个hgydF4y2B一个,利用15个种子大小相关的snp与平均观测值进行TGW的平均基因组最佳线性无偏预测。给出了Pearson相关系数。误差条表示五倍基因组最佳线性无偏预测交叉验证方案与十次重复运行计算的预测值的标准偏差(gydF4y2B一个ggydF4y2B一个,gydF4y2B一个hgydF4y2B一个;看到gydF4y2B一个方法gydF4y2B一个).gydF4y2B一个
QTL和变异数据的整合gydF4y2B一个
蚕豆基因组序列为遗传作图、基因表达谱和比较基因组学提供了统一的参考框架。为了帮助蚕豆育种家和遗传学家采用新的基础设施,我们绘制了两种常用的基因分型平台Illumina Infinium 1536 SNP和Illumina Oligo Pool Array测定的标记。此外,我们将不同双亲杂交的遗传图谱投射到基因组组装上,并获得了共识遗传图谱。这为抗病性和物候学提供了数量性状位点(qtl)的物理坐标。标记图和QTL区间可以交互式浏览gydF4y2B一个https://pulses.plantinformatics.iogydF4y2B一个(补充图。gydF4y2B一个7gydF4y2B一个及补充说明gydF4y2B一个1gydF4y2B一个).gydF4y2B一个
基因组序列也为基于序列的基因分型铺平了道路。我们挖掘Hedin/2组装寡核苷酸探针用于单引物富集技术(SPET)。gydF4y2B一个37gydF4y2B一个这是一种具有高通量和低样本成本的减少代表性基因分型方法。从一个用于性状定位的多样性集合中提取197个栽培种质,使用90,000探针SPET分析,每个预测基因至少有一个探针(补充表gydF4y2B一个10gydF4y2B一个).序列读取被映射到Hedin/2组装,并被称为沿基因组均匀分布的1081031个分离变异(SNPs)。通过分析在基因区发现的SNPs和短插入和缺失(indels)的功能影响,我们在197份材料中的至少一份中鉴定出1042个SNPs和65个引入过早终止密码子的indels。过早终止密码子中断了933个基因的转录,其中包括39个抗性基因类似物(扩展数据图)。gydF4y2B一个5gydF4y2B一个及补充表gydF4y2B一个11gydF4y2B一个).我们提供了携带过早终止密码子的完整基因图谱,以促进功能研究(补充表)gydF4y2B一个12gydF4y2B一个).gydF4y2B一个
种子大小的遗传学gydF4y2B一个
尽管存在巨大的变异和重要的农艺重要性,但蚕豆种子大小的遗传学仍然不清楚,只有少数大种子重量的QTL区域被检测到gydF4y2B一个38gydF4y2B一个.我们在3年的时间里收集了两个地点197份种质的种子大小数据,并将这些数据与SPET标记数据结合起来,进行了高分辨率的全基因组关联研究(GWAS)。这确定了15个标记-性状关联,这些关联在试验和GWAS方法中是稳定的。gydF4y2B一个3 dgydF4y2B一个扩展数据图gydF4y2B一个6gydF4y2B一个,补充图。gydF4y2B一个8gydF4y2B一个- - - - - -gydF4y2B一个11gydF4y2B一个及补充表格gydF4y2B一个13gydF4y2B一个而且gydF4y2B一个14gydF4y2B一个).最显著的信号位于第4染色体gydF4y2B一个Vfaba.Hedin2.R1.4g051440gydF4y2B一个基因(图。gydF4y2B一个3 d, egydF4y2B一个和补充图。gydF4y2B一个12gydF4y2B一个),在蚕豆种子中高度表达,位于先前确定的种子重量QTL区域gydF4y2B一个38gydF4y2B一个(补充注gydF4y2B一个1gydF4y2B一个),与gydF4y2B一个拟南芥gydF4y2B一个CYP78AgydF4y2B一个已知的调节种子大小的基因gydF4y2B一个39gydF4y2B一个(无花果。gydF4y2B一个3 fgydF4y2B一个).gydF4y2B一个Vfaba.Hedin2.R1.4g051440gydF4y2B一个因此可能导致蚕豆种子大小的变化,但并不能解释这一复杂性状的大部分变化(图2)。gydF4y2B一个3 egydF4y2B一个).相比之下,使用所有15个高置信度标记时,我们能够以几乎与使用全套基因组标记时一样高的准确度预测种子大小(图2)。gydF4y2B一个3 g hgydF4y2B一个),表明我们已经确定了很大一部分控制蚕豆种子大小的关键位点,以及相关的候选基因。为了研究种子大小是否一直是种群分化的驱动因素,我们通过基于模型的祖先估计进行了种群结构分析,并通过主成分分析将多样性面板分为4组,与它们的地理起源相对应(扩展数据图)。gydF4y2B一个7gydF4y2B一个).所有居群的种子增大等位基因比例相似,所有居群中均存在所有的种子增大等位基因,但居群4中很少有全部携带的种子增大等位基因gydF4y2B一个Vfaba.Hedin2.R1.4g051440gydF4y2B一个(扩展数据图。gydF4y2B一个8gydF4y2B一个及补充表gydF4y2B一个13gydF4y2B一个).这种等位基因在种群间的分布表明,不同地理区域的育种家在历史上有广泛的种质共享。gydF4y2B一个
门部候选颜色基因定位gydF4y2B一个
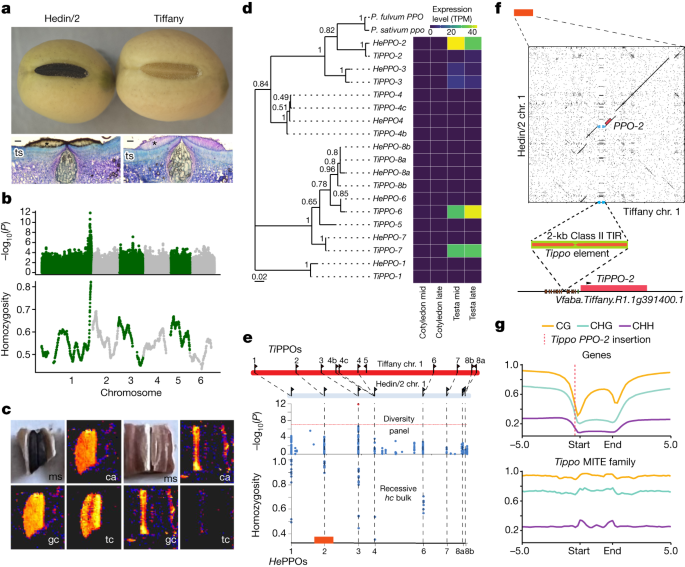
Hedin/2和Tiffany这两种测序基因型不仅在种子大小上有差异,而且在种门颜色上也有差异(图2)。gydF4y2B一个3 hgydF4y2B一个而且gydF4y2B一个4gydF4y2B一个).这是一个重要的孟德尔质量特征,人类消费者更喜欢浅色的希拉gydF4y2B一个40gydF4y2B一个.与种子大小类似,目前还没有发现候选基因。为了揭示该性状的分子基础,我们对种门颜色进行了GWAS,并确定了一个与先前绘制的两者一致的显著峰gydF4y2B一个肝门颜色(HC)gydF4y2B一个轨迹gydF4y2B一个40gydF4y2B一个和白门和黑门蚕豆品种杂交的隐性白门分离体的峰值纯合度(图2)。gydF4y2B一个4 bgydF4y2B一个).我们在多酚氧化酶中发现了高度相关的GWAS标记(gydF4y2B一个PPOgydF4y2B一个)基因以8个完整且高度保守的基因为一簇gydF4y2B一个PPOgydF4y2B一个Hedin/2组合中的基因。在豌豆中,PPO变异控制着种门颜色。在共音上gydF4y2B一个πgydF4y2B一个轨迹,一种框架转换的,非功能形式的单一gydF4y2B一个PPOgydF4y2B一个在所有现代豌豆品种中,都有固定的浅色种脐gydF4y2B一个41gydF4y2B一个.色素沉着的模式(图。gydF4y2B一个4gydF4y2B一个)以及蚕豆色素和非色素部位表面绿原酸、没食子儿茶素、咖啡酸四聚物二聚体和三聚体等低聚酚类化合物的含量(图。gydF4y2B一个4摄氏度gydF4y2B一个和扩展数据图。gydF4y2B一个9gydF4y2B一个)与在豌豆中观察到的非常相似gydF4y2B一个40gydF4y2B一个.结合遗传数据,这表明不同的PPO活性是豌豆和蚕豆门部颜色变化的原因,但尚不清楚哪一种PPO(或PPOs)可能是造成蚕豆门部颜色变化的原因。gydF4y2B一个
一个gydF4y2B一个上图为黑色门(左)和白色门(右)的横切面(ts)的光学显微镜图像,显示黑色门(Hedin/2)和白色门(Tiffany)的完整种子。星号表示肝门的反栅栏细胞,其中PPO活性由棕色色素沉着表示。比例尺,20µm。gydF4y2B一个bgydF4y2B一个在NORFAB多样性面板(上)中,带有门部颜色的GWAS被标记为二元性状,并在84组分隐性伪f中被标记为苍白门部亲本等位基因的纯合度gydF4y2B一个2 gydF4y2B一个苍白的肺门体积(下)。gydF4y2B一个cgydF4y2B一个, Hedin/2(左)和Tiffany(右)肝门标本激光解吸-电离质谱成像(ms)的光学图像,以及绿原酸(ca)、表没食子儿茶素(gc)和四聚咖啡酸(tc)的激光解吸-电离质谱成像信号分布,显示这些化合物在苍白肝门基因型的肝门区域没有信号。gydF4y2B一个dgydF4y2B一个系统发育树显示了致病豌豆基因与Hedin/2和Tiffany串联排列中发现的8和11个PPO拷贝之间的关系gydF4y2B一个HCgydF4y2B一个分别轨迹。gydF4y2B一个他gydF4y2B一个而且gydF4y2B一个“透明国际”gydF4y2B一个前缀表示Hedin/2-和tiffany特定版本的gydF4y2B一个PPOgydF4y2B一个paralogues。分支长度以每个位点的替换数来衡量。分支旁边的数字表示支持引导。gydF4y2B一个egydF4y2B一个,从上到下:1号染色体PPO簇的比例示意图,显示Tiffany和Hedin/2中PPOs的顺序和方向,虚线连接共位PPO拷贝;NORFAB多样性面板中肝门颜色相关性的特写和苍白肝门的纯合子性(gydF4y2B一个hcgydF4y2B一个)散装也显示出来。红色块表示在面板f中的点图中扩展的区域。gydF4y2B一个fgydF4y2B一个, 20 kb的上下游点图gydF4y2B一个HePPO-2gydF4y2B一个(3291947464)和gydF4y2B一个TiPPO-2gydF4y2B一个(3,263,562,398),显示一个约2 kb的螨区,名为'gydF4y2B一个TippogydF4y2B一个的转录因子结合位点(棕色椭圆形)中,靠近RNA聚合酶ii结合位点(TATA框为绿色椭圆形)和转录起始位点(箭头)gydF4y2B一个PPO-2gydF4y2B一个.终末倒转重复。gydF4y2B一个ggydF4y2B一个,基因的全基因组甲基化状态(上)与gydF4y2B一个TippogydF4y2B一个螨家族(下)。gydF4y2B一个
为了澄清,我们比较了Hedin/2(深色)和Tiffany(浅色)两种完全测序基因型的PPO簇的系统发育和结构。gydF4y2B一个VfPPO-2gydF4y2B一个与豌豆基因具有最高水平的同一性gydF4y2B一个Psat1g2063360gydF4y2B一个(无花果。gydF4y2B一个4 dgydF4y2B一个),而最强相关的GWAS标记在gydF4y2B一个VfPPO-3gydF4y2B一个,而苍白的肝门体纯合子峰位于之间gydF4y2B一个VfPPO-2gydF4y2B一个而且gydF4y2B一个VfPPO-3gydF4y2B一个,表明因果多态性存在于聚类的近端(图2)。gydF4y2B一个4 egydF4y2B一个).结构上,除了同位PPO基因间距离差异较大外,主要是由gydF4y2B一个食人魔gydF4y2B一个插入时,Hedin/ 2-Tiffany比较最显著的特征是三倍的gydF4y2B一个VfPPO-4gydF4y2B一个和蒂芙尼的缺失gydF4y2B一个VfPPO-5gydF4y2B一个在Hedin/2(图;gydF4y2B一个4 egydF4y2B一个),促使我们研究这些结构变化是否与PPO基因表达的变化有关。我们首先确定了PPO基因簇的转录几乎只局限于母睾丸组织(包括门部),而不是两种基因型的子叶(图。gydF4y2B一个4 dgydF4y2B一个、补充表格gydF4y2B一个15gydF4y2B一个而且gydF4y2B一个16gydF4y2B一个和补充图。gydF4y2B一个13gydF4y2B一个).在Hedin/2 testa中,gydF4y2B一个VfPPO-2gydF4y2B一个,在较小的程度上gydF4y2B一个VfPPO-3gydF4y2B一个,几乎占全部PPO表达。相比之下,Tiffany testa的PPO表达以gydF4y2B一个VfPPO-6gydF4y2B一个而且gydF4y2B一个VfPPO-7gydF4y2B一个(无花果。gydF4y2B一个4 dgydF4y2B一个).PPO聚类区域的详细注释和对比重复分析(补充图。gydF4y2B一个14gydF4y2B一个)突出显示了一个大约2 kb的AT-rich MITE插入gydF4y2B一个TiPPO-2gydF4y2B一个启动子区(图;gydF4y2B一个4 fgydF4y2B一个),它会中断预测的序列gydF4y2B一个VfPPO-2gydF4y2B一个启动子,属于一类与高水平甲基化相关的螨(图。gydF4y2B一个4 ggydF4y2B一个).综上所述,我们的研究结果表明gydF4y2B一个VfPPO-2gydF4y2B一个控制蚕豆门部颜色变异。除了提示蚕豆苍白门的致病机制外,我们的分析表明,增加的拷贝数并不一定与性状表达相关,并强调了来自多个基因型的全基因组序列的效用。gydF4y2B一个
讨论gydF4y2B一个
蚕豆是最早被驯化的作物之一。这是新石器时代作物的一部分,早期农民离开肥沃的新月地带时带走了这些作物gydF4y2B一个42gydF4y2B一个.蚕豆毒性的担忧在古典时代就有了gydF4y2B一个43gydF4y2B一个.在21世纪,营养质量仍然是育种的一个中心目标:新的蚕豆品种应该是低生物碱糖苷vicine和convine以及单宁。此外,必须更好地平衡必需氨基酸以适应人类的饮食需求,而种子植酸盐和蛋白酶抑制剂应减少以提高营养物质的生物利用度,同时注意不改变种子大小或损害害虫抗性,同时提高产量稳定性。蚕豆育种者现在可以在基因组资源和见解的支持下面对这些复杂的挑战。普遍存在和频繁的重组将允许新性状快速渗透到优秀材料中,并允许利用SPET基因分型提供的高SNP密度的强大和广泛适用的映射方法。在具有串联复制基因的基因组区域,精确定位致病变异仍然很困难,但我们对门颜色的研究表明,这些挑战可以通过使用高质量的长读集结合转录组学来克服。重复序列及其甲基化影响基因组进化,但也会影响插入基因调控区域的重复序列元件的基因表达变异。我们丰富的全基因组重复注释现在阐明了这些影响,为基于基因组学的育种平台增加了一个重要的组成部分。蚕豆似乎是一个孤立的物种,不与其他属杂交gydF4y2Ba野豌豆属gydF4y2B一个38gydF4y2B一个,有效地禁止在蚕豆育种中使用野生亲缘种。然而,稳定gydF4y2B一个农杆菌属gydF4y2B一个介导的蚕豆胚轴转化已被报道gydF4y2B一个44gydF4y2B一个.与目标基因鉴定一起,这为基因编辑开辟了可能性。通过编目和尽可能多地利用驯化蚕豆的分离变异来进一步扩大平台尤其重要,因为我们不知道它的野生祖先。在本文提供的资源和方法的支持下,突变体、基因库和优良品种的群体规模重测序,以及主要种质类群代表的泛基因组组装现在可以进行。gydF4y2B一个
方法gydF4y2B一个
基因组组装与验证gydF4y2B一个
PacBio HiFi读取使用hifiasm v0.11-r302(参考。gydF4y2B一个45gydF4y2B一个),使用默认参数。使用minimap2 v2.20将dovetail omnic数据与结果contigs对齐。gydF4y2B一个46gydF4y2B一个)来精确地排序和定位contigs。类似地,先前已经报道了来自共识遗传图谱的遗传标记gydF4y2B一个47gydF4y2B一个,将在NV644 × NV153重组自交系(F6)中定位的25K SNP阵列标记用minimap2与初步扩增的contigs进行比对,将contigs分配到染色体上。随后,利用TRITEX管道进行假分子构建gydF4y2B一个48gydF4y2B一个.在omnic和NV644 × nv153衍生遗传图谱的辅助支持下,对每条染色体上contigs的最终顺序和方向进行人工检测和校正。使用Kraken2 v2.1.1对组装的contigs进行分类。gydF4y2B一个31gydF4y2B一个)与数据库包括植物,昆虫和细菌的序列,并与BlobTools v1.1(参考。gydF4y2B一个49gydF4y2B一个).通过merquury v1.3(参考文献)评估基因组完整性和一致性准确性。gydF4y2B一个50gydF4y2B一个).用merquy、findse v1.94(参考文献)等工具评估纯合度、杂合度和重复水平。gydF4y2B一个21gydF4y2B一个)和GenomeScope v1.0(参考。gydF4y2B一个51gydF4y2B一个).如先前报道的那样,通过染色质免疫沉淀,然后用CENH3(着丝粒特异性组蛋白H3变体)抗体测序(ChIP-seq),在每条染色体中鉴定出着丝粒区域gydF4y2B一个52gydF4y2B一个.简而言之,来自ChIP-seq的原始读取被cutadapt v.1.15(参考。gydF4y2B一个53gydF4y2B一个),并使用minimap2映射到初步的假分子。使用SAMtools v1.15.1将对齐转换为BAM格式。gydF4y2B一个54gydF4y2B一个),并按Novosort v3.06.05 (gydF4y2B一个http://www.novocraft.comgydF4y2B一个).然后以100kb窗口为单位计算读取深度。最后,根据着丝粒位置(由短臂到长臂)确定每条染色体的顺序,并与蚕豆核型图相匹配。gydF4y2B一个
用流式细胞术估计基因组大小gydF4y2B一个
如前所述,用流式细胞术估计核基因组大小gydF4y2B一个55gydF4y2B一个.简而言之,完整的叶组织gydF4y2B一个诉较好gydF4y2B一个Hedin/2和gydF4y2B一个Secale cerealegydF4y2B一个简历。丹科夫斯克(2C = 16.19 pg DNA)gydF4y2B一个56gydF4y2B一个,作为内参标准品,切碎后放入含有500 μl Otto I溶液(0.1 M柠檬酸和0.5% v/v吐温20;奥托,1990)。粗悬浮液经50 μm尼龙网过滤。然后将细胞核制成球团(300gydF4y2B一个ggydF4y2B一个2分钟),再悬浮于300 μ l奥托I溶液中。在冰上孵育15分钟后,600µl的Otto II溶液中加入50µg mlgydF4y2B一个−1gydF4y2B一个RNase和50µg mlgydF4y2B一个−1gydF4y2B一个加入碘化丙啶。样品分析使用CyFlow Space流式细胞仪(Sysmex Partec GmbH),配备532 nm绿色激光。调整仪器的增益,使在使用512通道尺度时,代表参考标准物G1核的峰值大约位于相对丙啶荧光强度直方图的100通道上。将低水平阈值设置为通道20,以消除直方图中荧光强度最低的颗粒;所有剩余的荧光事件被记录,没有进一步的门控使用。对12株Hedin/2植物进行采样,每个样品分析三次,每次在不同的一天。使用FloMax软件(Sysmex Partec GmbH)分析每个样品至少有5000个核,2C DNA含量(单位为pg)由G1峰位置的平均值计算,公式为:2C核DNA含量=(样品G1峰平均值)×(标准2C DNA含量)/(标准G1峰平均值)。然后计算每个物种的平均核DNA含量(2C),并使用转换因子1 pg DNA = 0.978 Gb(参考文献)将DNA含量(pg)转换为碱基对的数量(bp)。gydF4y2Ba57gydF4y2B一个).gydF4y2B一个
基因组大小估计和质量评估gydF4y2B一个
的分布gydF4y2B一个kgydF4y2B一个- m (gydF4y2B一个KgydF4y2B一个= 101)频率从PacBio HiFi读取使用水母v2.2.10(参考。gydF4y2B一个58gydF4y2B一个).使用findGSE v1.94(参考文献),使用输出直方图来估计基因组大小和杂合度。gydF4y2B一个52gydF4y2B一个).通过两种独立的方法评估程序集的完整性:(1)通过minimap2 v2.20对HiFi读取进行自对齐,然后使用Sniffles v1.0.11调用单个变体(SV)。gydF4y2B一个59gydF4y2B一个);(2) BUSCO v3.0.2bgydF4y2B一个60gydF4y2B一个利用胚胎植物数据库进行分析。gydF4y2B一个
酶甲基化测序gydF4y2B一个
使用Qiagen dnasy Plant 96试剂盒按照制造商的说明提取用于甲基组测序的DNA,并在1%琼脂糖凝胶上检查完整性,并使用Thermo Fisher quantum - it PicoGreen dsDNA Assay进行定量。在Hedin/2基因组DNA中,200 ng与0.001 ng cpg甲基化的pUC19对照DNA和0.02 ng未甲基化的噬菌体Lambda对照DNA结合,然后使用EB缓冲液使体积达到50µl。输入DNA在S220聚焦超声仪(Covaris)上被剪切至350-400 bp,使用以下协议:占空因数= 10;峰值入射功率= 175;每脉冲周期= 200;时间= 2乘以30秒。剪切的DNA被用来准备一个大的插入NEBnext酶甲基-seq文库按照制造商的说明(gydF4y2B一个https://www.neb.com/-/media/nebus/files/manuals/manuale7120.pdfgydF4y2B一个).采用不同的测序指标构建了4个文库。进行5个PCR循环,包括指标和扩增文库。在Novaseq6000系统(Illumina)的SP-flow cell上进行500个循环(2 × 250 bp对端reads)测序,并以等浓度聚合。gydF4y2B一个
蒂芙尼基因组组装gydF4y2B一个
的分布gydF4y2B一个kgydF4y2B一个即(gydF4y2B一个KgydF4y2B一个= 51)从PacBio HiFi读取KAT v2.4.2(参考文献。gydF4y2B一个61gydF4y2B一个).输出直方图使用findse v1.94估计基因组大小和杂合度。使用hifiasm v0.15.5-r350执行组装。通过将HiFi read对齐回contigs并使用Sniffles v2.0.7调用结构变体,评估了组装的完整性。虽然没有明显的杂合子峰gydF4y2B一个kgydF4y2B一个-mer图中,我们观察到Tiffany中BUSCO重复基因的比例高于Hedin/2,并且findGSE对基因组大小略有高估。此外,我们还注意到一些短的contigs,其阅读覆盖率约为预期的一半,这表明在其他大部分纯合的基因组中存在杂合性区域。因此,我们使用purge_haplotigs v1.1.2执行haplotig清除。gydF4y2B一个62gydF4y2B一个) (purge_haplotigs cov -l 3 -m 7 -h 25)。使用merquury v1.3进一步评估了净化组件的质量。使用RagTag v2.0.1构建染色体水平支架(参考文献)。gydF4y2B一个63gydF4y2B一个)使用haplotig-purged组件。为了确认脚手架的成功,使用GSAlign v1.0.22对Hedin/2和Tiffany染色体进行了比对。gydF4y2B一个64gydF4y2B一个).我们比较了Tiffany注释的两种方法,以选择最适合进行比较分析的方法:(1)基因组的个体注释;和(2)“转移和间隙填充”方法(补充图。gydF4y2B一个15gydF4y2B一个).我们观察到,当使用相同的管道对基因组进行单独注释时,一部分共程基因具有不同的外显子结构。当Hedin/2注释转移到Tiffany上时,这些差异大大减少,这表明它们可能并不能反映真正的生物学差异。即使在使用相同的管道时,注释中的人为差异也可能混淆比较分析,这在以前已经报道过gydF4y2B一个65gydF4y2B一个,gydF4y2B一个66gydF4y2B一个.因此,我们使用了转移和间隙填充方法,其中Hedin/2注释使用Liftoff v1.6.1转移到Tiffany。gydF4y2B一个67gydF4y2B一个).为了防止嵌合基因模型的形成,例如由sv引起的嵌合基因模型,将具有帧内终止密码子的转移模型移除,并由Tiffany基因取代。蒂芙尼特有的基因模型也被添加到注释中。总的来说,我们观察到转移和间隙填充方法在两个材料中都产生了更多的同位基因和更多具有相同编码序列(CDS)长度的基因。gydF4y2B一个
重复DNA注释gydF4y2B一个
使用RepeatModeler v2.0.1(参考文献)对Hedin/2假分子进行从头重复发现。gydF4y2B一个68gydF4y2B一个),样品量为1,000,000 bp,并使用ltr_retrierver v2.9.0(参考。gydF4y2B一个69gydF4y2B一个)和LTRharvestgydF4y2B一个70gydF4y2B一个.De novo元素使用cd-hit-est v4.8.1(参考。gydF4y2B一个71gydF4y2B一个);元素分类是通过与RepBase 20181926版本的比较来辅助的,来自GyDB 2.0的核心重复域(参考文献)。gydF4y2B一个72gydF4y2B一个)和REXdb Viridiplantae v3.0(参考。gydF4y2B一个73gydF4y2B一个).对于逆转录转座子,sltr和全长元素由ltr_retrierver和LTRharvest指定。重复屏蔽使用RepeatMasker v4.2.1完成(gydF4y2B一个http://www.repeatmasker.orggydF4y2B一个)使用从头重复库。基于编码保守蛋白结构域的转座元件序列与REXdb v3.0数据库的相似性,使用DANTE v0.1.1 (gydF4y2B一个https://github.com/kavonrtep/dantegydF4y2B一个).卫星重复序列使用相似度搜索对我们以前研究中描述的卫星DNA家族定制数据库进行注释gydF4y2B一个18gydF4y2B一个,gydF4y2B一个31gydF4y2B一个,gydF4y2B一个74gydF4y2B一个,gydF4y2B一个75gydF4y2B一个.染色体中卫星重复序列的分布gydF4y2B一个诉较好gydF4y2B一个采用荧光原位杂交(FISH)检测。如前所述,进行染色体制备、探针标记和FISHgydF4y2B一个31gydF4y2B一个杂交和洗涤温度调整,以考虑探针AT/CG含量,以允许10-20%的不匹配。染色体用4 ',6-二氨基氨基-2-苯基吲哚(DAPI)反染色,安装在Vectashield培养基(Vector Laboratories)中,并使用蔡司AxioImager进行检测。Z2显微镜与Axiocam 506单相机。图像采集和处理使用ZEN 3.2软件(卡尔蔡司)。gydF4y2B一个
基因模型注释gydF4y2B一个
重复序列使用RepeatMasker v4.1.1 (gydF4y2B一个http://www.repeatmasker.orggydF4y2B一个)使用由RepeatModeler v2.0.1生成的自定义重复库(使用Hedin/2程序集)。基因注释使用BRAKER v2.1.6(参考文献。gydF4y2B一个76gydF4y2B一个(etpmode, min_contig 10000)。RNA测序文库(补充表gydF4y2B一个2 gydF4y2B一个)使用STAR 2.7.8a进行对齐gydF4y2B一个77gydF4y2B一个,gydF4y2B一个78gydF4y2B一个.蛋白质数据库Viridiplantae OrthoDB v10.1(参考;gydF4y2B一个79gydF4y2B一个) (gydF4y2B一个https://onlinelibrary.wiley.com/doi/10.1111/tpj.14546mergedgydF4y2B一个),并附上先前发表的译本gydF4y2B一个诉较好gydF4y2B一个转录组的组装gydF4y2B一个16gydF4y2B一个,连同使用GMAP v2020-10-14绘制蚕豆转录组组装图生成的比对(参考文献)一起被用作BRAKER的输入。gydF4y2B一个80gydF4y2B一个).此外,gydF4y2B一个m . truncatulagydF4y2B一个基因(' Mt4.0v2_Genes ')和gydF4y2B一个p .一gydF4y2B一个基因(' pissa. camor .gnm1.ann1.7 szr ')使用GMAP v2020-10-14进行比对。生成的比对被用于优化BRAKER基因模型。为了解释任何被BRAKER预测遗漏但存在于Hedin/2转录组组装中的基因模型,使用bedtools v2.30.0对来自GMAP蚕豆转录组比对的基因模型和BRAKER进行比较,仅保留与BRAKER基因模型没有交集的GMAP基因。对于这些基因,进一步过滤以消除任何短(少于50个氨基酸)翻译蛋白,帧内终止密码子或低(少于200个reads)表达特征。gydF4y2B一个81gydF4y2B一个).gydF4y2B一个
通过对准一个Iso-Seq数据集,评估Hedin/2和Tiffany注释的完整性gydF4y2B一个82gydF4y2B一个并组装了蚕豆品种Hiverna、Dozah和Farah的转录组。转录组使用GMAP v2020-10-14进行映射,这些映射与注释之间的比较使用床工具进行gydF4y2B一个83gydF4y2B一个.如果转录本不是一个假定的转座因子,则被抛光去除但与映射的转录本相交的基因模型被挽救。gydF4y2B一个RgydF4y2B一个使用rgauury v1.0(参考文献)在未抛光和抛光的注释上检测基因。gydF4y2B一个84gydF4y2B一个).gydF4y2B一个RgydF4y2B一个在未抛光的注释中存在的基因也被拯救了,但在抛光的注释中没有。使用CPC2 v2.0(参考文献)计算每个转录本的编码势。gydF4y2B一个85gydF4y2B一个).低编码潜能的mrna被重新分类为长非编码rna。去除至少50%与转座元件结构域重叠的基因。最后,相位校正后任何含有帧内停止密码子的蛋白质也被移除。使用BUSCOv5.2.2和embryophyta_odb10和fabales_odb10数据库评估最终基因集的完整性。gydF4y2B一个
共生基因的发现gydF4y2B一个
每种条件进行3个生物重复的总RNA测序。制备了18个文库,采用GeneWiz对Illumina HiSeq mRNA测序(2 × 100 bp RNA测序),平均每个文库约产生2 × 7000万reads。使用CLC Genomics Workbench 11 (CLC Bio Workbench, Qiagen)除去适配器序列。只有至少30 nt的插入被保留用于进一步分析。根据制造商的建议,使用CLC Genomics Workbench 11将Reads映射到Hedin/2基因组。每个转录本的映射读数被归一化为总数,并用于计算基因表达。完好的和破损的都算为一对。在不同条件下,各转录本的总计数用比例检验统计量进行比较gydF4y2Ba86gydF4y2B一个在CLC基因组工作台套件中实现。这种β二项检验比较了一组样本与另一组样本的计数比例。根据样品的大小(总数),给予不同的重量。权重是通过假设一组中比例的β分布来获得的,并通过矩量法估计这些比例,以及二项分布的比例。结果是加权的gydF4y2B一个tgydF4y2B一个-type测试统计量。然后,我们计算了多重假设检验的错误发现率修正gydF4y2B一个87gydF4y2B一个.分析中只考虑在所有比较条件下至少有10个reads的基因。gydF4y2B一个
同源基因家族鉴定gydF4y2B一个
19种豆科植物的基因(补充表gydF4y2B一个6gydF4y2B一个)聚类,以确定正交关系。利用BLASTP v2.2.26(参考文献)对这些物种的蛋白质序列进行了比对。gydF4y2B一个88gydF4y2B一个) (- value 1 × 10gydF4y2B一个−5gydF4y2B一个).然后用OrthoMCL v2.0.9对基因家族进行聚类。gydF4y2B一个89gydF4y2B一个).gydF4y2B一个
系统发育分析和分化时间估计gydF4y2B一个
从19种豆科植物中鉴定的单拷贝基因(补充表gydF4y2B一个6gydF4y2B一个)采用OrthoMCL v2.0.9进行系统发育分析。通过PhyML v3.0(参考文献)提取四重简并同义位点(4d位点)构建进化树。gydF4y2B一个90gydF4y2B一个)和TreeBest v1.9.2 (gydF4y2B一个https://github.com/Ensembl/treebestgydF4y2B一个).使用PAML v4.5包中的MCMCTREE v4.4估算分子钟和发散时间gydF4y2B一个91gydF4y2B一个利用系统发育树和已知物种的分化时间(来自已发表的文献或使用timeree (gydF4y2B一个http://www.timetree.org/gydF4y2B一个))。gydF4y2B一个
全基因组复制gydF4y2B一个
的全基因组复制gydF4y2B一个诉较好gydF4y2B一个,gydF4y2B一个m . truncatulagydF4y2B一个而且gydF4y2B一个p .一gydF4y2B一个利用每个基因组内的共线性进行估计。首先,使用MCScanX v2.0(参考。gydF4y2B一个92gydF4y2B一个).然后,利用同向区域的基因对进行4dtv(四重简并转换)计算。反转率由HKY校正gydF4y2B一个93gydF4y2B一个模型。同义替换(Ks)和非同义替换(Ka)由KaKs_Calculator v1.2估计。gydF4y2B一个94gydF4y2B一个).gydF4y2B一个
串联复制基因发现gydF4y2B一个
使用CRBHits v0.0.4包也发现了串联复制基因gydF4y2B一个95gydF4y2B一个tandemdups函数。为了确认结果,还使用DupGen_finder v25Apr2019对基因进行了分类。gydF4y2B一个96gydF4y2B一个),gydF4y2B一个答:芥gydF4y2B一个作为外组的gydF4y2B一个诉漂白亚麻纤维卷gydF4y2B一个被排除在TD分析之外,因为怀疑其结构注释的碎片化,这可能导致注释为串联重复(TDs)的基因数量膨胀(补充表gydF4y2B一个17gydF4y2B一个).重复的年龄估计使用gydF4y2B一个TgydF4y2B一个= k / 2gydF4y2B一个rgydF4y2B一个,gydF4y2B一个r =gydF4y2B一个1.5 × 10gydF4y2B一个−8gydF4y2B一个.通过CRBHits方法“Li”计算Ks。使用CRBHits v0.0.4包函数rbh2dagchainer (type = ' idx ', gap_length = 1, max_dist_allowed = 20)分析Hedin/2基因和Tiffany基因的Synteny,该函数内部使用DAGchainer算法gydF4y2B一个97gydF4y2B一个.通过使用Hedin/2和Tiffany之间发现的Syntenic串联复制基因对连接个体基因组中的TDG簇,发现了Syntenic串联复制基因(TDG)簇。为了尽量减少未放置基因对拷贝数变异分析的影响,因为未放置基因会导致虚假的拷贝数变异调用,我们使用Orthofinder v2.5.4验证了基于syntene的结果。gydF4y2B一个98gydF4y2B一个)分析。基于synteny和Orthofinder结果(Orthofinder仅考虑匹配染色体和未放置的contigs上的基因),仅保留相同基因型中具有相同或更高拷贝数的聚类进行进一步分析。使用prot-scriber v0.1.0 (gydF4y2B一个https://github.com/usadellab/prot-scribergydF4y2B一个).gydF4y2B一个
SPET文库的制备和测序gydF4y2B一个
使用Qubit 2.0荧光计(Invitrogen)进行量化的基因组DNA用于文库制备,应用Allegro靶向基因分型方案(NuGEN Technologies),该方案依赖于一组探针。DNA溶液,20ngµlgydF4y2B一个−1gydF4y2B一个按照制造商的说明作为输入。所有文库均使用Qubit 2.0荧光计进行量化,文库大小使用Bioanalyzer(安琪伦科技)的高灵敏度DNA检测或Caliper LabChip GX (Caliper生命科学)的高灵敏度DNA检测进行验证。使用CFX96触摸实时PCR检测系统(Bio-Rad Laboratories)对文库进行定量PCR。样品在IGA Technology Services (IGATech)进行测序。在Illumina NovaSeq 6000 (Illumina)上以2 × 150 PE配置进行DNA测序,每次接入平均产生773万对测序读对。gydF4y2B一个
表型和现场试验gydF4y2B一个
使用MARViN种子分析仪(MARViTECH)对2019年在Sejet Plant Breeding(55.82°N, 9.94°E)(试验23)、2020年(试验26)和2021年(试验30)以及2018年在Nordic Seed, Dyngby(55.96°N, 10.25°E)(试验11)、2019年(试验22)和2020年(试验25)收获的种子进行了种子性状量化。通过目视检查来评定肝门颜色。gydF4y2B一个
SNP呼叫和GWASgydF4y2B一个
SPET原始reads使用cutadapt v1.15修剪,并使用minimap2 v2.20对准Hedin/2基因组。使用Novosort v3.06.05 (gydF4y2B一个http://www.novocraft.comgydF4y2B一个), BCFtools v.1.8用于调用snp和短索引。VCF文件中缺失的数据是使用Beagle v5进行计算的。使用admix v1.3.0进行种群结构分析。gydF4y2B一个99gydF4y2B一个),gydF4y2B一个KgydF4y2B一个取值范围从2到10。每一个都有五倍的交叉验证误差gydF4y2B一个KgydF4y2B一个用来选择最好的gydF4y2B一个KgydF4y2B一个.使用Plink v1.90b6.9进行主成分分析,使用LDBlockshow v1.40识别连杆不平衡(LD)块。GWAS使用GEMMA v0.98.5(参考文献)。gydF4y2B一个One hundred.gydF4y2B一个),眨眼gydF4y2B一个101gydF4y2B一个, FarmCPUgydF4y2B一个102gydF4y2B一个和EMMAX+EMMA200(参考。gydF4y2B一个103gydF4y2B一个)使用估算SNP矩阵。BLINK和FarmCPU使用GAPIT3 v3.1 R包运行,包含三个主要组件。只有通过至少两种方法发现的snp才被认为是候选的,并且进一步要求信号在不止一次试验中被发现。用于GWAS的值是每个试验中每个基因型的平均值和最佳线性无偏估计量(BLUE)。使用R中的lme4包计算BLUEs,首先使用模型:gydF4y2B一个
在哪里gydF4y2B一个ygydF4y2B一个ijkgydF4y2B一个分数是多少gydF4y2B一个我gydF4y2B一个在环境gydF4y2B一个jgydF4y2B一个在块gydF4y2B一个kgydF4y2B一个,gydF4y2B一个µgydF4y2B一个是性状的总体均值,gydF4y2B一个GgydF4y2B一个我gydF4y2B一个继承的效力是什么gydF4y2B一个我gydF4y2B一个,gydF4y2B一个EgydF4y2B一个jgydF4y2B一个是环境的影响吗gydF4y2B一个jgydF4y2B一个,gydF4y2B一个GgydF4y2B一个我gydF4y2B一个×gydF4y2B一个EgydF4y2B一个jgydF4y2B一个加入之间的交互作用是什么gydF4y2B一个我gydF4y2B一个和环境gydF4y2B一个jgydF4y2B一个,gydF4y2B一个BgydF4y2B一个jkgydF4y2B一个是挡的效果吗gydF4y2B一个kgydF4y2B一个在环境gydF4y2B一个jgydF4y2B一个,gydF4y2B一个εgydF4y2B一个ijkgydF4y2B一个是残差。除均值外,其他效应均为随机效应。每个随机效应的意义除外gydF4y2B一个GgydF4y2B一个然后在r中使用lmertest包一次测试一个gydF4y2B一个PgydF4y2B一个大于0.05的值纳入最终模型。最终的模型gydF4y2B一个GgydF4y2B一个而且gydF4y2B一个µgydF4y2B一个固定效应,其他效应都是随机的。然后从gydF4y2B一个GgydF4y2B一个.gydF4y2B一个
采用基因组最佳线性无偏预测(gBLUP)方法进行五倍交叉验证,研究了种子大小相关性状的预测精度。拟合的矩阵表示模型如下所示gydF4y2B一个ygydF4y2B一个=gydF4y2B一个1gydF4y2B一个μ+gydF4y2B一个祖茂堂gydF4y2B一个+gydF4y2B一个egydF4y2B一个,在那里gydF4y2B一个ygydF4y2B一个是观察到的表型记录(BLUEs)的载体,gydF4y2B一个μgydF4y2B一个是截距,gydF4y2B一个1gydF4y2B一个是1的向量,gydF4y2B一个ZgydF4y2B一个是一个连接记录和资料的设计矩阵,gydF4y2B一个ugydF4y2B一个是否假设了种质的(基因组)育种价值的载体gydF4y2B一个ugydF4y2B一个∼gydF4y2B一个N (gydF4y2B一个0gydF4y2B一个,gydF4y2B一个GgydF4y2B一个\({\σ}_ {g} ^ {2} \)gydF4y2B一个),gydF4y2B一个\({\σ}_ {g} ^ {2} \)gydF4y2B一个加性遗传方差和gydF4y2B一个GgydF4y2B一个为基因组关系矩阵(GRM)。GRM构造为gydF4y2B一个GgydF4y2B一个=gydF4y2B一个ZZgydF4y2B一个”gydF4y2B一个/ 2gydF4y2B一个pgydF4y2B一个我gydF4y2B一个(1−gydF4y2B一个pgydF4y2B一个我gydF4y2B一个)gydF4y2B一个104gydF4y2B一个,在那里gydF4y2B一个ZgydF4y2B一个SNP矩阵是否以等位基因频率和为中心gydF4y2B一个pgydF4y2B一个我gydF4y2B一个标记的等位基因频率是多少gydF4y2B一个我gydF4y2B一个.最后,gydF4y2B一个egydF4y2B一个假设一个随机残差向量gydF4y2B一个egydF4y2B一个∼gydF4y2B一个N (gydF4y2B一个0gydF4y2B一个,gydF4y2B一个我gydF4y2B一个\({\σ}_ {e} ^ {2} \)gydF4y2B一个),gydF4y2B一个我gydF4y2B一个是单位矩阵和吗gydF4y2B一个\({\σ}_ {e} ^ {2} \)gydF4y2B一个是剩余方差。通过改变用于GMR计算的可用标记,研究了三种预测情景:(1)仅候选全基因组关联信号,(2)重复100次的相同大小的随机样本,以及(3)所有可用的SNP标记。交叉验证重复10次,并报告平均值±标准差。“混合。解决'函数从rrBLUP v4.6.1(引用。gydF4y2B一个105gydF4y2B一个)所有计算均采用R包。gydF4y2B一个
早熟终止密码子及抗性基因类似物鉴定gydF4y2B一个
筛选SNPs和indes,仅保留双等位变异。SNPEff v4.3注释为“stop_gain”的变体(参考。gydF4y2B一个106gydF4y2B一个),只保留至少一个纯合参考基因型和一个纯合替代基因型的多态性变异。使用rgauury v1.0管道鉴定抗性基因类似物gydF4y2B一个84gydF4y2B一个.使用regionerR v1.18.1的permTest函数计算抗性基因类似物中过早终止密码子的富集。gydF4y2B一个107gydF4y2B一个)和1000种排列(随机。funct我on=resampleRegions, evaluate.function=numOverlaps). All genes were provided as a universe for resampleRegions function.
种子大小候选基因的鉴定gydF4y2B一个
与种子大小相关的最显著和最稳定的SNPs的位置与Hedin/2蛋白编码基因的位置进行了比较。使用Orthofinder v2.5.4 (-M msa -S diamond -A mafft -T fasttree)检测基因重叠变异的同源物。使用Clustal Omega v1.2.4对所选蛋白质序列进行多序列比对。使用最大似然方法和基于JTT矩阵的模型推断了进化史,该模型在MEGA X v10.2.6中实现。gydF4y2B一个108gydF4y2B一个),自举重复100次。公开可用的表达式数据gydF4y2B一个16gydF4y2B一个用Kallisto v0.44.0(参考文献)定量Hedin/2 9个不同组织的基因表达。gydF4y2B一个109gydF4y2B一个).使用LDBlockShow v1.40(参考文献)研究候选基因周围基因组区间的LD模式。gydF4y2B一个110gydF4y2B一个).gydF4y2B一个
肝门颜色和组织学gydF4y2B一个
为了检查种门形态,从成熟的干种子中分离出含有种皮的Hedin/2(深色种门)和Tiffany(浅色种门),用2%蔗糖溶液在真空下饱和1 h,并将其嵌入冷冻凝胶介质(冷冻凝胶Leica)中。样品在低温机(Leica CM1950)中切割成15µm横切面,用甲苯胺蓝O (0.01%, w/v在水中;Sigma Aldrich)gydF4y2B一个41gydF4y2B一个,gydF4y2B一个111gydF4y2B一个.观察和摄影是在奥林巴斯BX 51显微镜(奥林巴斯)在明亮的视野中完成的,并用Apogee U4000数码相机(Apogee成像系统)记录数字。为了用激光解吸-电离成像质谱(LDI-MS)研究种门表层的代谢物含量,用机械裂解种子,用微剪刀(MicroSupport)将种门及其周围的一小部分组织与其余种皮分离,用双面胶带固定在MALDI板上,外表面朝上,并按前面描述的那样进行分析gydF4y2B一个41gydF4y2B一个,gydF4y2B一个112gydF4y2B一个.LDI-MSI实验使用Synapt G2-S (Waters)的高分辨率串联质谱仪(HRTMS)完成。使用的真空MALDI离子源配备了350 nm 1 khz Nd:YAG固体激光器。质谱仪参数设置如下:提取电压为10 V,碰撞能量为4 eV的陷阱碰撞能量(TrapCE)和2 eV的转移碰撞能量(TransferCE)。采用25 eV的TrapCE和10 eV的低质量(LM)分辨率进行MS/MS实验。用红磷(1 mg.mL)进行仪器校准gydF4y2B一个−1gydF4y2B一个,悬浮在丙酮中)。大量成像数据采集由HDImaging 1.5软件(Waters)驱动。激光束尺寸为60 μm。在激光能量为300 arb的正电离和负电离模式下采集光谱。激光重复频率设置为1000hz。质量范围50 - 1200 Da。精细映射gydF4y2B一个HCgydF4y2B一个自交系Disco(♀白色)与Hedin/2(♂深色)作杂交。FgydF4y2B一个4 gydF4y2B一个种子来自337 FgydF4y2B一个3.gydF4y2B一个21 F的后代gydF4y2B一个2 gydF4y2B一个个体通过侧翼标记分析显示为杂合跨gydF4y2B一个HCgydF4y2B一个对肝门颜色进行间隔评分,结果显示,肺门深色为253,浅色为84 (gydF4y2B一个χgydF4y2B一个2 gydF4y2B一个= 0.00098,gydF4y2B一个PgydF4y2B一个= 0.9749,拟合与预期3:1的比例)。由84个隐性伪f基因中每一个DNA的等摩尔数量组成的DNA池gydF4y2B一个2 gydF4y2B一个创建个体,并与亲本系的DNA样本一起进行SPET重测序。为了研究PPO基因家族在豆荚填充中后期的表达,Hedin/2和Tiffany的单株在温室中生长,直到较低节点上最成熟的豆荚接近成熟,最上面的节点仍在花中,形成种子发育的梯度。所有的豆荚被收获并解剖成豆荚壁、种皮、子叶、珠柄和胚轴样本。gydF4y2B一个16gydF4y2B一个);记录每个组织的新鲜重量。因为给定节点上的所有豆荚都不是同步受精的,也不一定以相同的速度发育,这是基于我们之前对蚕豆种子发育的研究gydF4y2B一个113gydF4y2B一个,根据子叶重量与总种子重量的比值,我们将单个豆荚分为灌浆中期和后期(补充图。gydF4y2B一个17gydF4y2B一个).gydF4y2B一个
PPO位点比较序列分析gydF4y2B一个
为了鉴定Hedin/2和Tiffany蛋白质组中的PPO同源物,从豌豆中提取了蛋白质序列gydF4y2B一个PPO1 / PlgydF4y2B一个基因(gydF4y2B一个Psat1g206360gydF4y2B一个)被用作BLAST v2.12.0查询。采用Clustal Omega v1.2.4对PPO蛋白序列进行多序列比对。利用最大似然法和基于JTT矩阵的模型推断了进化历史,并在MEGA X中实现了100个自举重复。利用minimap2 v2.24-r1122提取完整的PPO区域(从第一个PPO基因开始到最后一个PPO基因结束,两侧10000 bp侧翼序列),并对其进行比对。然后,从下游和上游20,000 bp的转录开始gydF4y2B一个PPO-2gydF4y2B一个序列之间的相似性使用FlexiDot v1.06 (ref。gydF4y2B一个114gydF4y2B一个).gydF4y2B一个
基因表达分析gydF4y2B一个
根据制造商的说明,使用Sigma Spectrum Kit (STRN250)从100mg快速冷冻的解剖组织(睾丸和子叶)中提取RNA,除了DNA消化后在室温下孵育。尽管从子叶中提取RNA完全按照制造商的规范进行,但在由CTAB、PVP、2 M Tris pH 8、0.5 M EDTA pH 8、4 M NaCl、亚精胺和β-巯基乙醇组成的提取缓冲液中破坏了睾丸组织,然后用8 M氯化锂沉淀(而不是试剂盒的裂解步骤)。在使用标准方法制备定向mRNA测序文库之前,用Qubit RNA IQ法定量总RNA,并将其归一化。每个文库产生410万到560万Illumina PE150短读(3×重复,2×组织和2×基因型)。利用Kallisto v0.44.0伪对齐RNA测序,对Hedin/2和Tiffany基因的表达进行定量分析。利用tximport将转录水平丰度转化为基因水平丰度。gydF4y2B一个
报告总结gydF4y2B一个
有关研究设计的进一步资料,请参阅gydF4y2B一个自然组合报告摘要gydF4y2B一个链接到这篇文章。gydF4y2B一个
数据可用性gydF4y2B一个
原始数据可在欧洲核苷酸档案研究ID下获得gydF4y2B一个PRJEB52541gydF4y2B一个.Hedin/2和Tiffany的基因组组装和注释可在以下网址下载gydF4y2B一个www.fabagenome.dkgydF4y2B一个并可通过交互式基因组浏览器(gydF4y2B一个http://w3lamc.umbr.cas.cz/lamc/resources.htmlgydF4y2B一个).gydF4y2B一个
参考文献gydF4y2B一个
Hyland, J. J., Henchion, M., McCarthy, M. & McCarthy, S. N.肉类在战略中实现可持续饮食降低温室气体排放的作用:综述。gydF4y2B一个肉类科学。gydF4y2B一个132gydF4y2B一个, 189-195(2017)。gydF4y2B一个
Zander, P.等人。欧洲农业的谷物豆类衰退和潜在复苏:综述。gydF4y2B一个阿格龙。维持。Dev。gydF4y2B一个36gydF4y2B一个, 26(2016)。gydF4y2B一个
Tanno K.-i。种植的起源gydF4y2B一个中投arietinumgydF4y2B一个l .,gydF4y2B一个蚕豆根尖gydF4y2B一个L.:叙利亚西北部Tell el-Kerkh的早期发现,公元前10世纪后期。gydF4y2B一个素食主义者。嘘。Archaeobot。gydF4y2B一个15gydF4y2B一个, 197-204(2006)。gydF4y2B一个
Caracuta, V.等人的14000年前的种子表明,失去的蚕豆祖先起源于黎凡特。gydF4y2B一个科学。代表。gydF4y2B一个6gydF4y2B一个, 37399(2016)。gydF4y2B一个
瓦萨米,A. O., O 'Sullivan, D. M. & Tosi, P.蚕豆种子贮存蛋白(gydF4y2B一个蚕豆根尖gydF4y2B一个L):遗传改良的现状和前景。gydF4y2B一个j·阿格利司。食品化学。gydF4y2B一个66gydF4y2B一个, 12617-12626(2018)。gydF4y2B一个
哈塔布,A., Maxted, N. & Bisby, F. A.来自叙利亚的豆豆的近亲:一个新种gydF4y2B一个野豌豆属gydF4y2B一个还有关于gydF4y2B一个诉hyaeniscyamusgydF4y2B一个(豆科)。gydF4y2B一个丘公牛。gydF4y2B一个43gydF4y2B一个, 535-540(1988)。gydF4y2B一个
Muratova, V。gydF4y2B一个遗传与植物育种应用植物学通报gydF4y2B一个补充gydF4y2B一个50gydF4y2B一个, 1-298(1931)。gydF4y2B一个
Hanelt, P., Schäfer, H. & schultz - motel, J. Die Stellung vongydF4y2B一个蚕豆根尖gydF4y2B一个L. in der GattunggydF4y2B一个野豌豆属gydF4y2B一个文化艺术之旅。gydF4y2B一个KulturpflanzegydF4y2B一个20.gydF4y2B一个, 263-275(1972)。gydF4y2B一个
库贝罗,j。i。苏索,m。j。原始和现代形式gydF4y2B一个蚕豆根尖gydF4y2B一个.gydF4y2B一个KulturpflanzegydF4y2B一个29gydF4y2B一个, 137-145(1981)。gydF4y2B一个
Vranken, L., Avermaete, T., Petalios, D. & Mathijs, E.遏制全球肉类消费:第二次营养转型的新证据。gydF4y2B一个环绕。科学。政策gydF4y2B一个39gydF4y2B一个, 95-106(2014)。gydF4y2B一个
Cernay, C., Pelzer, E. & Makowski, D.一个用于评估谷物豆类生产的全球实验数据集。gydF4y2B一个科学。数据gydF4y2B一个3.gydF4y2B一个, 160084(2016)。gydF4y2B一个
Herridge, d.f., Peoples, M. B. & Boddey, R. M.农业系统中生物固氮的全球投入。gydF4y2B一个植物的土壤gydF4y2B一个311gydF4y2B一个, 1-18(2008)。gydF4y2B一个
贝勒斯,派崔克,J. G. &格洛弗,B. J.田间豆提供的能量奖励分析(gydF4y2B一个蚕豆根尖gydF4y2B一个)花:花蜜、花粉和作用力。gydF4y2B一个生态。另一个星球。gydF4y2B一个8gydF4y2B一个, 3161-3171(2018)。gydF4y2B一个
Adhikari, K. N.等。加速蚕豆遗传增益的常规和分子育种工具(gydF4y2B一个蚕豆根尖gydF4y2B一个l .)。gydF4y2B一个前面。植物科学。gydF4y2B一个12gydF4y2B一个, 744259(2021)。gydF4y2B一个
韦伯,A.等人。基于snp的蚕豆共位性状定位一致性遗传图谱(gydF4y2B一个蚕豆根尖gydF4y2B一个l .)。gydF4y2B一个生物科技植物》。J。gydF4y2B一个14gydF4y2B一个, 177-185(2016)。gydF4y2B一个
Björnsdotter, E.等。VC1催化蚕豆中vicine生物合成的一个关键步骤。gydF4y2B一个Nat。植物gydF4y2B一个7gydF4y2B一个, 923-931(2021)。gydF4y2B一个
Macas, J. & Neumann, P. Ogre元素——一组独特的植物Ty3/吉普赛样逆转录转座子。gydF4y2B一个基因gydF4y2B一个390gydF4y2B一个, 108-116(2007)。gydF4y2B一个
马卡斯,J.等。通过对23种植物基因组重复DNA的深入分析,揭示了豆科豆科植物基因组大小变异的来源。gydF4y2B一个《公共科学图书馆•综合》gydF4y2B一个10gydF4y2B一个, e0143424(2015)。gydF4y2B一个
张伟,Jääskeläinen, M.,李s.p。BARE逆转录转座子通过不同的RNA池进行翻译和复制。gydF4y2B一个《公共科学图书馆•综合》gydF4y2B一个8gydF4y2B一个, e72270(2013)。gydF4y2B一个
Fuchs, J., Strehl, S., Brandes, A., Schweizer, D. & Schubert, I.分子细胞遗传学的特征gydF4y2B一个蚕豆根尖gydF4y2B一个基因组异染色质分化,复制模式和序列定位。gydF4y2B一个染色体Res。gydF4y2B一个6gydF4y2B一个, 219-230(1998)。gydF4y2B一个
rihie, A., Walenz, B. P., Koren, S. & Phillippy, A. M. merquury:基因组组装的无参考质量、完整性和分期评估。gydF4y2B一个基因组医学杂志。gydF4y2B一个21gydF4y2B一个, 245(2020)。gydF4y2B一个
马舍尔等人。一个染色体构象捕获大麦基因组的有序序列。gydF4y2B一个自然gydF4y2B一个544gydF4y2B一个, 427-433(2017)。gydF4y2B一个
德布鲁因,F. J. ingydF4y2B一个模范豆类gydF4y2B一个截叶苜蓿(de Bruijn, F. J.)第8章(威利,2019)。gydF4y2B一个
Courty, p.e., Smith, P., Koegel, S., Redecker, D. & Wipf, D.有益植物根-微生物相互作用中无机氮的吸收和运输。gydF4y2B一个暴击。植物科学gydF4y2B一个34gydF4y2B一个, 4-16(2015)。gydF4y2B一个
Wipf, D., Krajinski, F., van Tuinen, D., Recorbet, G. & Courty, p.e。丛枝菌根市场交易:从丛枝到普通菌根网络。gydF4y2B一个新植醇。gydF4y2B一个223gydF4y2B一个, 1127-1142(2019)。gydF4y2B一个
De La Torre, A. R.等人。针叶树千兆基因组的洞察。gydF4y2B一个植物杂志。gydF4y2B一个166gydF4y2B一个, 1724-1732(2014)。gydF4y2B一个
牛,S.等。中国松基因组和甲基组揭示了针叶树进化的关键特征。gydF4y2B一个细胞gydF4y2B一个185gydF4y2B一个, 204 - 217。e14(2022)。gydF4y2B一个
Schmutz, J.等。古多倍体大豆基因组序列。gydF4y2B一个自然gydF4y2B一个463gydF4y2B一个, 178-183(2010)。gydF4y2B一个
坎农,S. B.等人。结瘤和非结瘤豆科植物早期辐射中的多重多倍体事件。gydF4y2B一个摩尔。杂志。另一个星球。gydF4y2B一个32gydF4y2B一个, 193-210(2014)。gydF4y2B一个
奚海华,阮文伟,刘志强,刘志强,李志强,李志强。黄豆属植物的染色体水平组装(gydF4y2B一个野豌豆属漂白亚麻纤维卷gydF4y2B一个)参考基因组。gydF4y2B一个ggydF4y2B一个https://doi.org/10.46471/gigabyte.38gydF4y2B一个(2022)。gydF4y2B一个
Ávila Robledillo, L.等。卫星DNAgydF4y2B一个蚕豆根尖gydF4y2B一个其特点是在序列组成、与着丝粒的关联和复制时间方面具有显著的多样性。gydF4y2B一个科学。代表。gydF4y2B一个8gydF4y2B一个, 5838(2018)。gydF4y2B一个
Cossu, R. M.等。LTR逆转录转座子在大型植物基因组中表现出低水平的不平等重组和高比率的元素内基因转换。gydF4y2B一个基因组医学杂志。另一个星球。gydF4y2B一个9gydF4y2B一个, 3449-3462(2017)。gydF4y2B一个
李志强,李志强,李志强,等。植物基因体DNA甲基化研究。gydF4y2B一个咕咕叫。当今。植物医学杂志。gydF4y2B一个36gydF4y2B一个, 103-110(2017)。gydF4y2B一个
Brenet, F.等人。第一外显子的DNA甲基化与转录沉默密切相关。gydF4y2B一个《公共科学图书馆•综合》gydF4y2B一个6gydF4y2B一个, e14524(2011)。gydF4y2B一个
王,L.等。DNA甲基化组分析提供了证据,证明茶叶基因组的扩张与TE爆发有关。gydF4y2B一个生物科技植物》。J。gydF4y2B一个17gydF4y2B一个, 826-835(2019)。gydF4y2B一个
Erdmann, R. M. & Picard, C. L. rna定向DNA甲基化。gydF4y2B一个公共科学图书馆麝猫。gydF4y2B一个16gydF4y2B一个, e1009034(2020)。gydF4y2B一个
Barchi, L.等人。单引物富集技术在番茄和茄子种质资源高通量基因分型中的应用。gydF4y2B一个前面。植物科学。gydF4y2B一个10gydF4y2B一个, 1005(2019)。gydF4y2B一个
Khazaei, H., O 'Sullivan, D. M., Sillanpää, M. J. & Stoddard, F. L.蚕豆气孔性状QTL的拟合性分析(gydF4y2B一个蚕豆根尖gydF4y2B一个l .)。gydF4y2B一个定理。达成。麝猫。gydF4y2B一个127gydF4y2B一个, 2371-2385(2014)。gydF4y2B一个
Adamski, n.m., Anastasiou, E., Eriksson, S., O 'Neill, C. M. & Lenhard, M.对种子大小的本地母本控制gydF4y2B一个KLUH / CYP78A5gydF4y2B一个依赖生长信号。gydF4y2B一个国家科学院学报美国gydF4y2B一个106gydF4y2B一个, 2015 - 20120(2009)。gydF4y2B一个
Khazaei, H.等人。蚕豆中大豆碱浓度的SNP标记(gydF4y2B一个蚕豆根尖gydF4y2B一个l .)。gydF4y2B一个摩尔。育种gydF4y2B一个35gydF4y2B一个, 38(2015)。gydF4y2B一个
Balarynová, J.等。多酚氧化酶功能的丧失与豌豆门色素沉着有关,在豌豆驯化过程中已被选择。gydF4y2B一个新植醇。gydF4y2B一个235gydF4y2B一个, 1807-1821(2022)。gydF4y2B一个
戈斐,A.,列弗-亚顿,S. &阿博,S.。gydF4y2B一个破土:新石器时代黎凡特的植物驯化:“核心区单一事件”模型gydF4y2B一个(Emery和Claire Yass在考古学上的出版物,特拉维夫大学考古研究所,2021年)。gydF4y2B一个
斯卡伯勒,J. Beans,毕达哥拉斯,禁忌,和古代营养学。gydF4y2B一个经典。世界gydF4y2B一个75gydF4y2B一个, 355-358(1982)。gydF4y2B一个
Hanafy, M., Pickardt ., Kiesecker, H. & Jacobsen, H.- j。gydF4y2B一个农杆菌属gydF4y2B一个-介导的蚕豆转化(gydF4y2B一个蚕豆根尖gydF4y2B一个l)利用胚轴。gydF4y2B一个EuphyticagydF4y2B一个142gydF4y2B一个, 227-236(2005)。gydF4y2B一个
程浩,李国涛,冯晓霞,张海华,李海华。基于相位装配图的单倍型解析从头装配。gydF4y2B一个Nat方法。gydF4y2B一个18gydF4y2B一个, 170-175(2021)。gydF4y2B一个
Minimap2:核苷酸序列的成对比对。gydF4y2B一个生物信息学gydF4y2B一个34gydF4y2B一个, 3094-3100(2018)。gydF4y2B一个
Carrillo-Perdomo, E.等。蚕豆新遗传资源的开发(gydF4y2B一个蚕豆根尖gydF4y2B一个L.)通过发现基于基因的SNP标记和构建高密度共识图谱进行育种。gydF4y2B一个科学。代表。gydF4y2B一个10gydF4y2B一个, 6790(2020)。gydF4y2B一个
Monat, C.等。TRITEX:染色体尺度序列组装麦类基因组与开源工具。gydF4y2B一个基因组医学杂志。gydF4y2B一个20.gydF4y2B一个, 284(2019)。gydF4y2B一个
Wood, D. E. & Salzberg, S. L. Kraken:使用精确比对的超快速宏基因组序列分类。gydF4y2B一个基因组医学杂志。gydF4y2B一个15gydF4y2B一个, r46(2014)。gydF4y2B一个
Laetsch, D. & Blaxter, M. BlobTools:基因组组装的询问[版本1;同行评议:2人通过,但有保留]。gydF4y2B一个F1000Res。gydF4y2B一个6gydF4y2B一个, 1287(2017)。gydF4y2B一个
孙浩,丁俊,Piednoël, M. & Schneeberger, K. findGSE:估计人类基因组大小变异gydF4y2B一个拟南芥gydF4y2B一个使用k-mer频率。gydF4y2B一个生物信息学gydF4y2B一个34gydF4y2B一个, 550-557(2017)。gydF4y2B一个
Vurture, G. W.等。GenomeScope:短读快速无参考基因组分析。gydF4y2B一个生物信息学gydF4y2B一个33gydF4y2B一个, 2202-2204(2017)。gydF4y2B一个
Martin, M. Cutadapt从高通量测序读取中移除适配器序列。gydF4y2B一个EMBnet J。gydF4y2B一个17gydF4y2B一个, 10-12(2011)。gydF4y2B一个
李,H.等。序列对齐/映射格式和SAMtools。gydF4y2B一个生物信息学gydF4y2B一个25gydF4y2B一个, 2078-2079(2009)。gydF4y2B一个
杜瑞泽,李志强,李志强,等。利用流式细胞仪估计植物核DNA含量。gydF4y2B一个Protoc Nat。gydF4y2B一个2 gydF4y2B一个, 2233-2244(2007)。gydF4y2B一个
dolezel, J., Sgorbati, S. & Lucretti, S.三种DNA荧光染料用于流式细胞术估计植物核DNA含量的比较。gydF4y2B一个杂志。工厂。gydF4y2B一个85gydF4y2B一个, 625-631(1992)。gydF4y2B一个
鳟鱼和人类的核DNA含量和基因组大小。gydF4y2B一个血细胞计数一次gydF4y2B一个51gydF4y2B一个, 127-128(2003)。gydF4y2B一个
Marçais, G. & Kingsford, C.一种快速,无锁的k-mers发生的有效并行计数方法。gydF4y2B一个生物信息学gydF4y2B一个27gydF4y2B一个, 764-770(2011)。gydF4y2B一个
Sedlazeck, F. J.等。利用单分子测序精确检测复杂结构变异。gydF4y2B一个Nat方法。gydF4y2B一个15gydF4y2B一个, 461-468(2018)。gydF4y2B一个
Simão, F. A., Waterhouse, R. M., Ioannidis, P., Kriventseva, E. V. & Zdobnov, E. M. BUSCO:评估基因组组装和单拷贝orthologs注释完整性。gydF4y2B一个生物信息学gydF4y2B一个31gydF4y2B一个, 3210-3212(2015)。gydF4y2B一个
Mapleson, D., Garcia Accinelli, G., Kettleborough, G., Wright, J. & Clavijo, B. J. KAT:质量控制NGS数据集和基因组组装的k-mer分析工具包。gydF4y2B一个生物信息学gydF4y2B一个33gydF4y2B一个, 574-576(2017)。gydF4y2B一个
罗奇,M. J.,施密特,S. A. & Borneman, A. R.清除单倍体:第三代二倍体基因组组装的等位基因contig重分配。gydF4y2B一个BMC生物信息学gydF4y2B一个19gydF4y2B一个, 460(2018)。gydF4y2B一个
Alonge, M.等人。使用RagTag的自动化组装脚手架提升了一个新的番茄系统,用于高通量基因组编辑。gydF4y2B一个基因组医学杂志。gydF4y2B一个23gydF4y2B一个, 258(2022)。gydF4y2B一个
林,H.-N。&徐,w - l。GSAlign:一种高效的种内基因组序列比对工具。gydF4y2B一个BMC基因组学gydF4y2B一个21gydF4y2B一个, 182(2020)。gydF4y2B一个
König, S., Romoth, L. W., Gerischer, L. & Stanke, M.在多个基因组中同时发现基因。gydF4y2B一个生物信息学gydF4y2B一个32gydF4y2B一个, 3388-3395(2016)。gydF4y2B一个
拜耳,p.e.等。装配与比较两者密切相关gydF4y2B一个芸苔属植物显著gydF4y2B一个基因组。gydF4y2B一个生物科技植物》。J。gydF4y2B一个15gydF4y2B一个, 1602-1610 (2017)gydF4y2B一个
Shumate, A. & Salzberg, S. L. Liftoff:基因注释的精确定位。gydF4y2B一个生物信息学gydF4y2B一个37gydF4y2B一个, 1639-1643(2021)。gydF4y2B一个
弗林,J. M.等人。RepeatModeler2用于自动发现转座因子家族的基因组。gydF4y2B一个国家科学院学报美国gydF4y2B一个117gydF4y2B一个, 9451-9457(2020)。gydF4y2B一个
Ou, S. & Jiang, N. LTR_retriever:用于鉴定长末端重复逆转录转座子的高度准确和敏感的程序。gydF4y2B一个植物杂志。gydF4y2B一个176gydF4y2B一个, 1410-1422(2017)。gydF4y2B一个
Ellinghaus, D., Kurtz, S. & Willhoeft, U. LTRharvest,一种高效灵活的LTR逆转录转座子从头检测软件。gydF4y2B一个BMC生物信息学gydF4y2B一个9gydF4y2B一个, 18(2008)。gydF4y2B一个
黄勇,牛斌,高勇,付林,李伟。CD-HIT套件:一个用于聚类和比较生物序列的网络服务器。gydF4y2B一个生物信息学gydF4y2B一个26gydF4y2B一个, 680-682(2010)。gydF4y2B一个
Llorens, C.等人。移动遗传元素的吉普赛数据库(GyDB): 2.0版。gydF4y2B一个核酸测定。gydF4y2B一个39gydF4y2B一个, d70-d74(2010)。gydF4y2B一个
Neumann, P. Novák, P. Hoštáková, N. & Macas, J.植物ltr -反转录转座子的系统研究阐明了其多蛋白结构域的系统发育关系,并为元素分类提供了参考。gydF4y2B一个暴徒。DNAgydF4y2B一个10gydF4y2B一个, 1(2019)。gydF4y2B一个
Ávila Robledillo, L.等。豆科Fabeae族中心粒卫星的异常序列多样性和杂乱性。gydF4y2B一个摩尔。杂志。另一个星球。gydF4y2B一个37gydF4y2B一个, 2341-2356(2020)。gydF4y2B一个
Vondrak, T.等。超长纳米孔中重复序列阵列的特征揭示了卫星DNA的频繁起源,这些重复序列来源于逆转录转座子。gydF4y2B一个植物J。gydF4y2B一个101gydF4y2B一个, 484-500(2020)。gydF4y2B一个
brzhenna, T., Hoff, K. J., Lomsadze, A., Stanke, M. & Borodovsky, M. BRAKER2:在蛋白质数据库支持下,使用genmark - ep +和AUGUSTUS自动注释真核基因组。gydF4y2B一个NAR染色体组。Bioinform。gydF4y2B一个3.gydF4y2B一个, lqaa108(2021)。gydF4y2B一个
Dobin, A.等人。STAR:超快通用RNA-seq对准器。gydF4y2B一个生物信息学gydF4y2B一个29gydF4y2B一个, 15-21(2013)。gydF4y2B一个
Dobin, A. & Gingeras, T. R.用STAR测序RNA‐seq。gydF4y2B一个咕咕叫。Protoc。生物信息学gydF4y2B一个51gydF4y2B一个, 11.14.1-11.14.19(2015)。gydF4y2B一个
Kriventseva, e.v.等。OrthoDB v10:对动物、植物、真菌、原生生物、细菌和病毒基因组的多样性进行采样,用于orthorths的进化和功能注释。gydF4y2B一个核酸测定。gydF4y2B一个47gydF4y2B一个, d807-d811(2019)。gydF4y2B一个
Wu, T. D. & Watanabe, C. K. GMAP: mRNA和EST序列的基因组定位和比对程序。gydF4y2B一个生物信息学gydF4y2B一个21gydF4y2B一个, 1859-1875(2005)。gydF4y2B一个
Liao, Y., Smyth, G. K. & Shi, W. featuremets:一种高效的通用程序,用于分配序列读取基因组特征。gydF4y2B一个生物信息学gydF4y2B一个30.gydF4y2B一个, 923-930(2014)。gydF4y2B一个
吕俊毅等。解开蚕豆的复杂性(gydF4y2B一个蚕豆根尖gydF4y2B一个L.)利用长读异构体测序技术揭示冷应激反应基因。gydF4y2B一个科学。代表。gydF4y2B一个11gydF4y2B一个, 21094(2021)。gydF4y2B一个
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2B一个生物信息学gydF4y2B一个26gydF4y2B一个, 841-842(2010)。gydF4y2B一个
Li, P.等。rgauury:用于植物抗性基因类似物(RGAs)全基因组预测的管道。gydF4y2B一个BMC基因组学gydF4y2B一个17gydF4y2B一个, 852(2016)。gydF4y2B一个
Kang Y.-J。et al。CPC2:基于序列内在特征的快速准确编码势计算器。gydF4y2B一个核酸测定。gydF4y2B一个45gydF4y2B一个, w12-w16(2017)。gydF4y2B一个
巴格里,K. A.,邓,L.,莫里斯,J. S. &阿尔达兹,C. M. SAGE的差异表达:解释正常的库间变异。gydF4y2B一个生物信息学gydF4y2B一个19gydF4y2B一个, 1477-1483(2003)。gydF4y2B一个
Benjamini, Y. & Hochberg, Y.控制错误发现率:多重测试的一种实用而强大的方法。gydF4y2B一个J. R.统计社gydF4y2B一个57gydF4y2B一个, 289-300(1995)。gydF4y2B一个
Altschul, s.f.等人。gapping BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。gydF4y2B一个核酸测定。gydF4y2B一个25gydF4y2B一个, 3389-3402(1997)。gydF4y2B一个
李,L., Stoeckert, C. J. & Roos, D. S. OrthoMCL:真核生物基因组的正交基团鉴定。gydF4y2B一个基因组Res。gydF4y2B一个13gydF4y2B一个, 2178-2189(2003)。gydF4y2B一个
金登,S.,德尔苏斯,F.,杜法亚德,J.-F.。& Gascuel, O.用PhyML估计最大似然系统发生。gydF4y2B一个方法分子生物学。gydF4y2B一个537gydF4y2B一个, 113-137(2009)。gydF4y2B一个
Yang Z. PAML:最大似然分析系统发育的程序包。gydF4y2B一个第一版。达成。Biosci。gydF4y2B一个13gydF4y2B一个, 555-556(1997)。gydF4y2B一个
王毅,等。MCScanX:一个用于检测和进化分析基因同步性和共线性的工具包。gydF4y2B一个核酸测定。gydF4y2B一个40gydF4y2B一个, e49(2012)。gydF4y2B一个
长谷川,M.,岸野,H. &矢野,t . a .。用线粒体DNA分子钟测定人猿分裂的年代。gydF4y2B一个J. Mol. EvolgydF4y2B一个22gydF4y2B一个, 160-174(1985)。gydF4y2B一个
张哲,等。KaKs_Calculator:通过模型选择和模型平均计算Ka和Ks。gydF4y2B一个蛋白质组学生物信息学gydF4y2B一个4 gydF4y2B一个, 259-263(2006)。gydF4y2B一个
Ullrich, K. K. CRBHits:从条件互惠最佳命中到密码子对齐和R中的Ka/Ks。gydF4y2B一个J.开源软件。gydF4y2B一个5gydF4y2B一个, 2424(2020)。gydF4y2B一个
乔,X.等。植物重复多倍体-二倍体循环中的基因复制和进化。gydF4y2B一个基因组医学杂志。gydF4y2B一个20.gydF4y2B一个, 38(2019)。gydF4y2B一个
哈斯,B. J.,德尔彻,a . L.,沃特曼,J. R. &萨尔茨伯格,S. L. DAGchainer:用于挖掘片段基因组复制和同向性的工具。gydF4y2B一个生物信息学gydF4y2B一个20.gydF4y2B一个, 3643-3646(2004)。gydF4y2B一个
Emms, D. M. & Kelly, S. OrthoFinder:比较基因组学的系统发育矫形学推断。gydF4y2B一个基因组医学杂志。gydF4y2B一个20.gydF4y2B一个, 238(2019)。gydF4y2B一个
Alexander, D. H. & Lange, K.对个人祖先估计的admix算法的增强。gydF4y2B一个BMC生物信息学gydF4y2B一个12gydF4y2B一个, 246(2011)。gydF4y2B一个
周,X. & Stephens, M.关联研究的全基因组高效混合模型分析。gydF4y2B一个Nat,麝猫。gydF4y2B一个44gydF4y2B一个, 821-824(2012)。gydF4y2B一个
黄,明,刘,X,周,Y, Summers, r.m.,张哲。BLINK:针对个体和数百万标记的下一层次的全基因组关联研究的一揽子方案。gydF4y2B一个GigaSciencegydF4y2B一个8gydF4y2B一个, 2(2018)。gydF4y2B一个
刘旭,黄明,范B, Buckler, E. S. &张卓。迭代使用固定和随机效应模型进行强大而有效的全基因组关联研究。gydF4y2B一个公共科学图书馆麝猫。gydF4y2B一个12gydF4y2B一个, e1005767(2016)。gydF4y2B一个
Kang, h.m.等。方差分量模型解释全基因组关联研究中的样本结构。gydF4y2B一个Nat,麝猫。gydF4y2B一个42gydF4y2B一个, 348-354(2010)。gydF4y2B一个
计算基因组预测的有效方法。gydF4y2B一个乳制品科学gydF4y2B一个91gydF4y2B一个, 4414-4423(2008)。gydF4y2B一个
Endelman, J. B. Ridge回归和其他R包rrBLUP基因组选择的核心。gydF4y2B一个植物基因组gydF4y2B一个4 gydF4y2B一个, 250-255(2011)。gydF4y2B一个
Cingolani, P.等人。注释和预测单核苷酸多态性的影响的程序,SnpEff:在基因组的SNPsgydF4y2B一个黑腹果蝇gydF4y2B一个应变w1118;iso-2;iso-3。gydF4y2B一个飞(奥斯丁)gydF4y2B一个6gydF4y2B一个, 80-92(2012)。gydF4y2B一个
Gel, B. et al. regioneR:一个R/Bioconductor包,用于基于排列测试的基因组区域关联分析。gydF4y2B一个生物信息学gydF4y2B一个32gydF4y2B一个, 289-291(2015)。gydF4y2B一个
Kumar, S., Stecher, G., Li, M., Knyaz, C. & Tamura, K. MEGA X:跨计算平台的分子进化遗传学分析。gydF4y2B一个摩尔。杂志。另一个星球。gydF4y2B一个35gydF4y2B一个, 1547-1549(2018)。gydF4y2B一个
Bray, N. L, Pimentel, H., Melsted, P. & Pachter, L.近最优概率RNA-seq量化。gydF4y2B一个生物科技Nat。》。gydF4y2B一个34gydF4y2B一个, 525-527(2016)。gydF4y2B一个
盾,s。et al。LDBlockShow:一个基于可变调用格式文件的链接不平衡和单倍型块可视化的快速方便的工具。gydF4y2B一个简短。Bioinform。gydF4y2B一个22gydF4y2B一个, bbaa227(2020)。gydF4y2B一个
Zablatzká, L., Balarynová, J., Klčová, B., Kopecký, P. & Smýkal, P.野生植物种皮发育的解剖与组织化学gydF4y2B一个Pisum一gydF4y2B一个无性系种群。gydF4y2B一个elatiusgydF4y2B一个穿上灰西服时(m)。Asch。et Graebn。和家养豌豆(gydF4y2B一个Pisum一gydF4y2B一个无性系种群。gydF4y2B一个一种gydF4y2B一个l .)。gydF4y2B一个Int。理学。gydF4y2B一个22gydF4y2B一个, 4602(2021)。gydF4y2B一个
Krejčí, P.等。电子驱动的微操作与激光解吸电离质谱相结合-分析种皮层和揭示种子休眠之谜的独特工具。gydF4y2B一个TalantagydF4y2B一个242gydF4y2B一个, 123303(2022)。gydF4y2B一个
Warsame, a.o., Michael, N., O 'Sullivan, d.m. & Tosi, P.蚕豆种子发育与蛋白质积累模式(gydF4y2B一个蚕豆根尖gydF4y2B一个l .)。gydF4y2B一个j·阿格利司。食品化学。gydF4y2B一个70gydF4y2B一个, 9295-9304(2022)。gydF4y2B一个
Seibt, K. M. Schmidt, T. & Heitkam, T. FlexiDot:用于可视化序列分析的高度可定制、可识别歧义的点状图。gydF4y2B一个生物信息学gydF4y2B一个34gydF4y2B一个, 3575-3577(2018)。gydF4y2B一个
确认gydF4y2B一个
本研究由丹麦创新基金(' NORFAB ', 5158-00004B)向J.S.;face - jpi ERA-NET SusCrop Profaba到S.U.A, D.M.O, F.L.S.和A.H.S.;德国莱布尼茨协会框架下的莱布尼茨青年研究小组(J118/2021/REPLACE);德国联邦教育与研究部(de.NBI, 031A536B)授予H.G.;诺和诺德基金(#NNF20OC0065157);Jane and Aatos Erkko基金会(PanFaba)对美国卫生服务协会的资助;亚历山大·冯·洪堡基金会在索菲亚·科瓦列夫斯卡娅奖的框架下授予A.A.G.;生物技术和生物科学研究理事会授予D.M.O. BB/P023509/1;德国研究基金会(DFG)项目编号497667402给A.A.G.和B.U.;捷克科学基金会项目编号GACR 20-24252S给J.M.; the European Research Council under the European Union’s Horizon 2020 research and innovation programme (grant agreement no. 834221) to J.S.; the BMBF-funded de.NBI Cloud within the German Network for Bioinformatics Infrastructure (de.NBI) (031A532B, 031A533A, 031A533B, 031A534A, 031A535A, 031A537A, 031A537B, 031A537C, 031A537D and 031A538A); and Grains Research Development Corporation as well as Agriculture Victoria for PacBio sequencing, parts of the public dataset curation for pulses.plantinformatics.io (DAV 1905-003RTX) and RNA sequencing data (BioProject ID PRJNA395480). We acknowledge CSC-IT Center for Science, Finland and ELIXIR CZ Research Infrastructure (Czech Ministry of Education, Youth and Sports grant no. LM2018131) for generous computational resources; and A. Fiebig for help with data submission.
作者信息gydF4y2B一个
作者及隶属关系gydF4y2B一个
贡献gydF4y2B一个
M.J.进行了Hedin/2基因组组装,甲基化数据分析,门部颜色GWAS,并撰写了第一份手稿草稿。A.A.G.进行了Tiffany基因组组装,分析了种皮和胚胎表达数据,并编制了过早终止密码子图谱。a.a.g., J.K.和bi分析了同步性,串联基因重复和内含子大小的变化。j.k., A.A.G.和L.I.F.贡献了基因注释和数据提交。S.U.A, D.M.O, L.I.F, D.A.和P.S.设计了SPET试验。d.a和a.o.w行肝门颜色体积分离分析(BSA)。P.B.负责种皮,MS. E.B.和l.l.j负责SPET数据分析和种子大小的提交和预测。p.e.c.和R.B.生成并分析了根瘤菌和菌根共生RNA测序数据。K.C.生成了种皮和胚胎RNA测序文库。J.D.和J.C.用流式细胞仪估计了基因组的大小。 H.G., A.H.S., J.M., P.Neumann, P.Novák, J.T. and J.K. annotated and analysed repetitive elements. A.Hallab, P.T., B.U. and L.U.H. performed gene functional annotation. A.Himmelbach, S.P. and N.S. generated methylome sequencing data. S.K. and G.K.-G. contributed to PacBio sequencing, provided RNA sequencing data used for variant discovery, and integrated and visualized QTL and variation data online. A.K. and J.M. generated and analysed ChIP–seq data. L.K. and P.S. performed seed coat histochemistry. P.K. contributed seed coat analytical chemistry. T.W.M. carried out phenotype data validation and seed size GWAS. M.M., D.M.O., A.H.S., S.U.A., I.S., G.A., M.N. and H.K. edited the manuscript with input from all authors. J.O., L.K.N. and A.S. carried out SPET sample preparation and hilum colour phenotyping. P.Novák developed the faba bean genome browser. K.C. and T.R.-S.-H. prepared seed coat and embryo RNA sequencing libraries. L.A.R. contributed cytogenetic analysis. A.H.J.W. and R.J.S. generated HiFi data. H.Z. provided gene family, evolution, diversity and phylogenetic analysis. F.L.S. provided plant material. S.U.A., J.S., N.T. and A.M.T. provided resources. D.M.O. and D.A. genotyped pure Hedin/2 stock and generated the genetic map. S.U.A., A.H.S., D.M.O. and M.J. designed the study. S.U.A. coordinated the project.
相应的作者gydF4y2B一个
道德声明gydF4y2B一个
相互竞争的利益gydF4y2B一个
作者声明没有利益竞争。gydF4y2B一个
同行评审gydF4y2B一个
同行评审信息gydF4y2B一个
自然gydF4y2B一个感谢Aureliano Bombarely, Eric von Wettberg和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2B一个同行评审报告gydF4y2B一个是可用的。gydF4y2B一个
额外的信息gydF4y2B一个
出版商的注意gydF4y2B一个施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2B一个
扩展的数据图形和表格gydF4y2B一个
图1基于K-mer和流式细胞术的基因组大小估计。gydF4y2B一个
一个gydF4y2B一个,核基因组大小的估计gydF4y2B一个诉较好gydF4y2B一个Hedin/2流式细胞仪检测。经碘化丙染色的大鼠细胞核经流式细胞术分析,得到相对细胞核DNA含量的直方图gydF4y2B一个诉较好gydF4y2B一个赫定/ 2,gydF4y2B一个Secale cerealegydF4y2B一个简历。Dankovske (2C = 16.19 pg),作为内部参考标准。低水平阈值设置为相对荧光强度512通道尺度直方图上的通道20;记录所有剩余的荧光事件。2C核DNA含量gydF4y2B一个诉较好gydF4y2B一个Hedin/2估计为26.36 pg(±0.26 s.d)。gydF4y2B一个bgydF4y2B一个,基于k-mer的基因组大小估计,使用Hedin/2原始HiFi测序数据。gydF4y2B一个cgydF4y2B一个, merquury制作的Hedin/2基因组组装k-mer谱图。K-mer多重性是指一个K-mer在测序读取中被发现的次数。颜色与k-mer在集合中出现的次数相对应。gydF4y2B一个dgydF4y2B一个, merquury为Tiffany基因组组装制作的k-mer谱图。gydF4y2B一个
图2 Hedin/2和Tiffany染色体排列。gydF4y2B一个
点状图显示Hedin/2和Tiffany染色体之间的对齐。gydF4y2B一个
图3 Hedin/2染色体2 - 6的基因组特征。gydF4y2B一个
基因组成分分布包括重组(cM/Mb),基因密度,逆转录转座子gydF4y2B一个吉普赛gydF4y2B一个而且gydF4y2B一个CopiagydF4y2B一个超家族,全长(FL) LTR插入,卫星重复和DNA甲基化(CH, CHG和CHH上下文)在染色体2至6上。gydF4y2B一个
图4蚕豆的甲基化格局。gydF4y2B一个
一个gydF4y2B一个,不同TE插入年龄的CG(上)、CHG(中)和CHH(下)甲基化情况。gydF4y2B一个bgydF4y2B一个, CHH甲基化在FabTR-83卫星重复序列上达到峰值。gydF4y2B一个
扩展数据图5导致提前终止密码子的变体。gydF4y2B一个
从SPET数据中识别出导致过早终止密码子(ptc)的变体(snp和短indel)。gydF4y2B一个一个gydF4y2B一个,等位基因计数分布,包括缺失的基因型呼叫。gydF4y2B一个bgydF4y2B一个,排除缺失基因型呼叫的等位基因频率分布。gydF4y2B一个cgydF4y2B一个Hedin/2基因组注释中鉴定的抗性基因类似物(RGAs)总结。gydF4y2B一个dgydF4y2B一个, rga中导致PTC变异的等位基因频率分布。gydF4y2B一个egydF4y2B一个, ptc在rga中过度表达的排列试验。gydF4y2B一个
扩展数据图6种子大小GWAS的曼哈顿区。gydF4y2B一个
种子性状以形状表示。试验用颜色表示。蓝色-最佳线性无偏估计器。候选snp以虚线表示。gydF4y2B一个
图7遗传多样性与群体结构。gydF4y2B一个
一个gydF4y2B一个,按admix分组的种群结构。gydF4y2B一个bgydF4y2B一个主成分分析(PCA)对197个种质按亚群体分组的结果进行了分析。gydF4y2B一个cgydF4y2B一个, PCA对197个产地根据地理来源着色。一个非洲聚类延伸至南美洲,一个欧洲聚类横跨北美,一个亚洲聚类和其他聚类与观测到的admix结果一致。根据隶属度系数(>=0.6)分析,113份(57.36%)可归属于特定类群,84份被鉴定为4个类群中2个或2个以上类群杂交的杂合体。gydF4y2B一个dgydF4y2B一个,邻居连接(NJ)树显示了197个连接之间的关系。gydF4y2B一个
扩展数据图8种群间种子增大等位基因。gydF4y2B一个
一个gydF4y2B一个15个显著相关SNPs(列)的种子增大等位基因在各品系和亚群体(行)中的分布。gydF4y2B一个bgydF4y2B一个,各亚群体中种子增大等位基因的比例。gydF4y2B一个
扩展数据图9蚕豆叶门中的酚类化合物。gydF4y2B一个
激光解吸电离质谱成像(LDI-MSI)显示与蚕豆暗门颜色相关的酚类化合物的聚合。绿原酸(1和2)和没食子儿茶素(3和4)二聚体和三聚体在着色(A, Hedin/2)和非着色(B, Tiffany)胞门表面的分布绿原酸单体、二聚体和三聚体的放大光谱(5;信号在m/z 353.0811;705.1667和1057.2427)和没食子儿茶素(6;在m/z 305.0646, 609.1223, 913.1827)从色素(A, Hedin/2)和非色素(B, Tiffany)希拉中收集(显示特定信号的光谱在A和B基因型的相同强度下放大,例如强度1.30x10gydF4y2B一个5gydF4y2B一个为5A、5B等光谱设置),从不含种皮碎片门槽区和边缘的致密表面采集光谱。绿原酸和没食子儿茶素二聚体和三聚体的特征片段(即绿原酸二聚体和三聚体分别在m/z 179.0341和353.0811处裂解出咖啡酸和绿原酸阴离子,没食子儿茶素二聚体和三聚体分别在m/z 179.0375和125.0232处裂解出反- diels Alder产物)进一步证实了绿原酸和没食子儿茶素二聚体和三聚体的同质性。gydF4y2B一个
补充信息gydF4y2B一个
补充信息gydF4y2B一个
此文件包含补充图1-17,补充表3,4,6,13和补充说明(椒盐饼干说明)。该文件支持组装和结构注释质量控制,基因家族和基因组重复分析,转座元件和DNA甲基化分析,GWAS和基因表达谱质量控制,进一步支持种门颜色和种子大小候选位点的信息。补充说明包括使用Pretzel访问集成蚕豆数据集的说明。gydF4y2B一个
补充表gydF4y2B一个
该文件包含补充表1、2、5、7-12和14-17。该文件支持Hedin/2和Tiffany种子的基因组组装和注释、根和结节的表达谱、拷贝数变异和转座因子分析、SPET数据和GWAS分析以及表达谱。gydF4y2B一个
权利和权限gydF4y2B一个
开放获取gydF4y2B一个本文遵循知识共享署名4.0国际许可协议,允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可协议中,除非在材料的信用额度中另有说明。如果材料未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。如欲查看本牌照的副本,请浏览gydF4y2B一个http://creativecommons.org/licenses/by/4.0/gydF4y2B一个.gydF4y2B一个
关于本文gydF4y2B一个
引用本文gydF4y2B一个
Jayakodi, M., Golicz, a.a., Kreplak, J。gydF4y2B一个et al。gydF4y2B一个巨大的二倍体蚕豆基因组揭示了全球蛋白质作物的变异。gydF4y2B一个自然gydF4y2B一个(2023)。https://doi.org/10.1038/s41586-023-05791-5gydF4y2B一个
收到了gydF4y2B一个:gydF4y2B一个
接受gydF4y2B一个:gydF4y2B一个
发表gydF4y2B一个:gydF4y2B一个
DOIgydF4y2B一个:gydF4y2B一个https://doi.org/10.1038/s41586-023-05791-5gydF4y2B一个
评论gydF4y2B一个
通过提交评论,您同意遵守我们的gydF4y2B一个条款gydF4y2B一个而且gydF4y2B一个社区指导原则gydF4y2B一个.如果您发现一些滥用或不符合我们的条款或指导方针,请标记为不适当。gydF4y2B一个