文摘
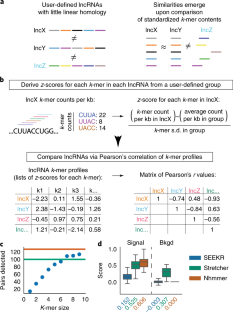
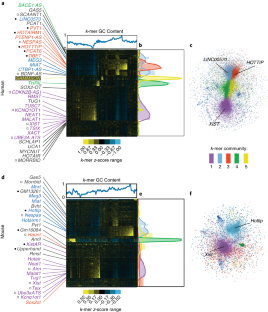
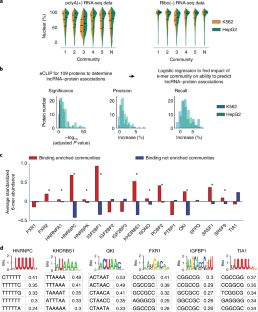
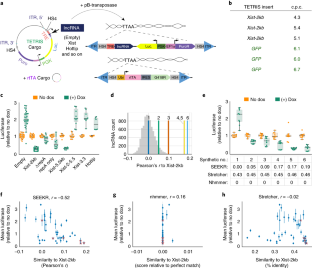
大多数长非编码rna的功能(lncRNAs)是未知的。与蛋白质相比,具有相似功能的lncRNAs往往缺乏线性序列同源性;因此,识别功能在一个lncRNA很少告诉别人的识别功能。我们开发了一个序列比较法来解构线性序列关系lncRNAs和评估基于大量的短序列相似性称为k-mers。我们发现lncRNAs相关函数通常有类似k-mer资料尽管缺乏线性同源,这k-mer概要文件与蛋白结合lncRNAs和亚细胞定位。使用新颖的分析量化Xist-like监管潜力,我们直接证明进化无关lncRNAs可以编码类似的功能通过不同的空间安排相关序列的图案。K-mer-based分类是一种强大的方法来检测复发lncRNAs序列和功能之间的关系。
这是一个预览的订阅内容,通过访问你的机构
相关的文章
开放获取文章引用这篇文章。
多效性的健身效果的lncRNA Uhg4在黑腹果蝇
BMC基因组学开放获取2022年11月30日
表观遗传肿瘤异质性与纳米孔测序单细胞分析的时代
临床实验胚胎学开放获取2022年8月27日
AptaNet作为aptamer-protein互动的深度学习方法预测
科学报告开放获取2021年3月16日
访问选项
订阅自然+
得到直接在线访问自然和其他55个自然杂志
29.99美元
每月
订阅杂志
得到完整的期刊访问1年
79.00美元
只有6.58美元的问题
所有价格是净价格。
增值税将被添加后在付款。
税计算期间将完成付款。
买条
时间有限或全文访问ReadCube。
32.00美元
所有价格是净价格。

数据可用性
期间生成的数据集和/或分析在当前研究中可用的文章及其补充信息文件。
引用
艾耶,m . k . et al .长非编码rna的景观在人类转录组。Nat麝猫。。47,199 - 208 (2015)。
聊聊,s &科勒,j . RNA在意想不到的地方:长非编码RNA功能不同的细胞环境。Nat。启摩尔。细胞杂志。14,699 - 712 (2013)。
Holoch d &莫札,d . rna介导基因表达的表观遗传调控。Nat,启麝猫。16,71 - 84 (2015)。
刘,X。,Hao, L., Li, D., Zhu, L. & Hu, S. Long non-coding RNAs and their biological roles in plants.基因组蛋白质组生物信息学13,137 - 147 (2015)。
Rinn, j·l·& Chang h . y .长非编码rna基因调控。为基础。学生物化学启。81年,145 - 166 (2012)。
Gutschner, t & Diederichs美国癌症的特点:长非编码RNA的观点。RNA杂志。9,703 - 719 (2012)。
李,j . t . & Bartolomei m . s . X-inactivation印记,长非编码rna在健康和疾病。细胞152年,1308 - 1323 (2013)。
吴,x &锋利,p .答:(不同的转录新基因起源的驱动力?细胞155年,990 - 996 (2013)。
切赫,t·r·&施泰茨j . a .非编码RNA revolution-trashing旧规则建立新的。细胞157年,77 - 94 (2014)。
Hezroni h . et al。长非编码RNA进化原理来自17个物种的转录组的直接比较。细胞的代表。11,1110 - 1122 (2015)。
Cabili, m . n等。人类大型综合注释基因间的非编码rna显示全局属性和具体的子类。Dev的基因。25,1915 - 1927 (2011)。
Derrien, t . et al。人类长非编码rna的GENCODE v7目录:分析他们的基因结构、进化,和表达式。基因组Res。22,1775 - 1789 (2012)。
贝特曼,a . et al . UniProt:蛋白质信息的中心。核酸Res。43D204-D212 (2015)。
伯曼,H。,Henrick, K. & Nakamura, H. Announcing the worldwide Protein Data Bank.Nat。结构。医学杂志。10980 (2003)。
Ulitsky, i & Bartel d . p . lincRNAs:基因组学、进化和机制。细胞154年26-46 (2013)。
库特,et al。快速的周转率长非编码rna和基因表达的进化。公共科学图书馆麝猫。8e1002841 (2012)。
Necsulea, a . et al . lncRNA体验和表达模式的进化在四足动物。自然505年,635 - 640 (2014)。
艾迪,s . r .守恒的RNA二级结构的计算分析转录组和基因组。为基础。启Biophys。43,433 - 456 (2014)。
奎因,j。j。快速进化的营业额是守恒的lncRNA-genome交互。Dev的基因。30.,191 - 207 (2016)。
艾迪,s . r .同源性搜索结构rna:从原理到实际使用的证据。核糖核酸21,605 - 607 (2015)。
t·j·惠勒和艾迪,s . r . nhmmer: DNA同源性搜索与概要文件摘要。生物信息学29日,2487 - 2489 (2013)。
大米,P。,Longden, I. & Bleasby, A. EMBOSS: the European Molecular Biology Open Software Suite.趋势麝猫。16,276 - 277 (2000)。
雷,d . et al。rna结合主题的纲要解码基因调控。自然499年,172 - 177 (2013)。
Stefl, R。,Skrisovska, L. & Allain, F. H. RNA sequence- and shape-dependent recognition by proteins in the ribonucleoprotein particle.EMBO代表。633-38 (2005)。
埃德加,r c & Batzoglou多重序列比对。咕咕叫。当今。Struc。医学杂志。16,368 - 373 (2006)。
Altschul, s F。、《W。,Miller, W., Myers, E. W. & Lipman, D. J. Basic local alignment search tool.j·摩尔,杂志。215年,403 - 410 (1990)。
Pervouchine, d, d . et al。增强从多个鼠标组织转录组地图显示进化基因表达的约束。Commun Nat。65903 (2015)。
查德威克,b . p . Xi染色质组织的变化和相关性H3K27me3染色质区域的转录序列通过微阵列分析。Chromosoma116年,147 - 157 (2007)。
Engreitz, j . m . et al . RNA-RNA交互使特定目标的非编码rna新生Pre-mRNAs和染色质网站。细胞159年,188 - 199 (2014)。
麦,w . et al . Mitotically稳定polycomb协会组与不活跃的x染色体蛋白质速度和enx1滋养层干细胞。咕咕叫。医学杂志。12,1016 - 1020 (2002)。
西方,j . a . et al。长非编码rna NEAT1 MALAT1结合活性染色质网站。摩尔。细胞55,791 - 802 (2014)。
克莱姆森,c . M。,McNeil, J. A., Willard, H. F. & Lawrence, J. B. XIST RNA paints the inactive X chromosome at interphase: evidence for a novel RNA involved in nuclear/chromosome structure.j .细胞。医学杂志。132年,259 - 275 (1996)。
花茎甘蓝j . m . et al .因地制宜沉默的监管元素X失活的机制。细胞151年,951 - 963 (2012)。
他们批判,诉D。,Guillaume, J. L., Lambiotte, R. & Lefebvre, E. Fast unfolding of communities in large networks.j . Stat。机械。理论E。https://doi.org/10.1088/1742-5468/2008/10/P10008(2008)。
邓纳姆,等。一个集成的百科全书在人类基因组中DNA的元素。自然489年57 - 74 (2012)。
Carlevaro-Fita, J。Rahim,。Guigo, R。,Vardy, L. A. & Johnson, R. Cytoplasmic long noncoding RNAs are frequently bound to and degraded at ribosomes in human cells.核糖核酸22,867 - 882 (2016)。
Van Nostrand e . l . et al .健壮transcriptome-wide发现rna结合蛋白结合位点与增强剪辑(eCLIP)。Nat方法。13,508 - 514 (2016)。
霍金斯,d . m .过度拟合的问题。j .化学。正无穷。第一版。科学。441 - 12 (2004)。
Spitale, r . c . et al .结构性痕迹体内解码RNA监管机制。自然519年,486 - 490 (2015)。
兰伯特:et al。RNA Bind-n-Seq:定量评估的顺序和结构结合特异性RNA结合蛋白。摩尔。细胞54,887 - 900 (2014)。
Smola, m . j . et al .形状显示transcript-wide交互,复杂的结构域,蛋白质相互作用在Xist lncRNA活细胞。Proc。《科学。美国113年,10322 - 10327 (2016)。
迪马特奥,m . et al . PiggyBac工具箱。摩尔。生物方法。859年,241 - 254 (2012)。
叮,s . et al。有效的换位piggyBac (PB)转座子在哺乳动物细胞和老鼠。细胞122年,473 - 483 (2005)。
Dowen, j . m . et al .基因控制细胞的身份出现在绝缘社区在哺乳动物染色体。细胞159年,374 - 387 (2014)。
Wutz,。,Rasmussen, T. P. & Jaenisch, R. Chromosomal silencing and localization are mediated by different domains of Xist RNA.Nat,麝猫。30.,167 - 174 (2002)。
刘,F。,Somarowthu, S. & Pyle, A. M. Visualizing the secondary and tertiary architectural domains of lncRNA RepA.Nat,化学。医学杂志。13,282 - 289 (2017)。
Tyner, c . et al . UCSC基因组浏览器数据库:2017更新。核酸Res。45D626-D634 (2017)。
R核心团队。接待员:统计计算的语言和环境(R统计计算的基础,2017年)。
沙尔丹哈,a . j . Java Treeview-Extensible微阵列数据的可视化。生物信息学20.,3246 - 3248 (2004)。
堰,w . H。,Emmons, S., Gibson, R., Taylor, D. & Mucha, P. J. Post-processing partitions to identify domains of modularity optimization.算法1093 (2017)。
多布林,a . et al .明星:超速普遍RNA-seq对准器。生物信息学29日,15至21 (2013)。
廖,Y。,Smyth, G. K. & Shi, W. featureCounts: an efficient general purpose program for assigning sequence reads to genomic features.生物信息学30.,923 - 930 (2014)。
f . et al . Scikit-learn Pedregosa:机器学习在Python中。j·马赫。学习。Res。12,2825 - 2830 (2011)。
贝利,t . l . et al . MEME套件:主题发现和搜索工具。核酸Res。37W202-W208 (2009)。
Machanick p &贝利,t . l . MEME-ChIP:主题分析DNA的大型数据集。生物信息学27,1696 - 1697 (2011)。
Darty, K。,Denise, A. & Ponty, Y. VARNA: interactive drawing and editing of the RNA secondary structure.生物信息学25日(1974 - 1975 (2009)。
釜山,s &周k m RNA结构模型的可视化综合基因组内查看器。核糖核酸23,1012 - 1018 (2017)。
确认
我们感谢UNC的同事讨论,j . Cheng帮助俄罗斯方块克隆。这项工作是由美国国立卫生研究院(NIH)拨款UL1TR002489 GM121806, GM105785,罗勒奥康纳奖。5100683从3月角基金会和资金Eshelman创新研究所Lineberger综合癌症中心和北卡罗来纳大学药理学系(J.M.C.),詹姆斯·s·麦克唐纳基金会21世纪科学Initiative-Complex系统格兰特没有学者奖。220020315 (P.J.M.), NIH米拉奖R35 GM122532 (K.M.W.)。J.M.K.是一个NSF研究生研究员(格兰特dge - 1650116)和支持部分由国家卫生研究院培训格兰特在生物信息学和计算生物学(T32 GM067553)。D.M.L.支持部分由国家卫生研究院培训格兰特在遗传学和分子生物学(T32 GM007092)。时是一个NSF研究生研究员(格兰特dge - 1144081)和支持部分由国家卫生研究院培训格兰特在分子和细胞生物物理学(批准T32 GM08570)。
作者信息
作者和联系
贡献
J.M.K.,P.J.M., and J.M.C. conceived the study. J.M.K., D.S., and J.M.C. performed the computational analysis. S.O.K., K.I., D.M.L., M.D.S., J.S.W., A.R.B., K.M.W., and J.M.C. designed and performed the TETRIS assays. D.W.C., C.R.H., S.W., Q.C., and J.M.K. built the website. J.M.K. and J.M.C. wrote the paper.
相应的作者
道德声明
相互竞争的利益
作者宣称没有利益冲突。
额外的信息
出版商的注意:施普林格自然保持中立在发表关于司法主权地图和所属机构。
补充信息
补充文本和数字
补充图1 - 11和补充表2 - 6,9、10、13 - 17和21
补充表1
策划在人类和小鼠基因工程lncRNAs列表
补充表7
人类lncRNA社区作业和描述
补充表8
鼠标lncRNA社区作业和描述
补充表11
人类社会k-mer概要文件
补充表12
鼠标社区k-mer概要文件
补充表18
k-mer丰富核和胞质lncRNAs
补充表19
蛋白质对数似结果比较空的预测能力和逻辑回归模型
补充表20
蛋白质逻辑回归(LR)精度和召回的结果
补充表22
TETRIS-lncRNA片段信息
补充表23
俄罗斯方块测定寡核苷酸引物
辅助软件
图书馆小k-mer计数频率的核苷酸序列
权利和权限
关于这篇文章
引用这篇文章
柯克,,Kim, S.O., Inoue, K.et al。长非编码rna的功能分类k- m内容。Nat麝猫50,1474 - 1482 (2018)。https://doi.org/10.1038/s41588 - 018 - 0207 - 8
收到了:
接受:
发表:
发行日期:
DOI:https://doi.org/10.1038/s41588 - 018 - 0207 - 8
本文引用的
表观遗传肿瘤异质性与纳米孔测序单细胞分析的时代
临床实验胚胎学(2022)
多效性的健身效果的lncRNA Uhg4在黑腹果蝇
BMC基因组学(2022)
PCirc:随机与森林有关的植物circRNA识别软件
BMC生物信息学(2021)
发现深深地守恒的图案组合在快速发展的非编码序列
基因组生物学(2021)
AptaNet作为aptamer-protein互动的深度学习方法预测
科学报告(2021)
