摘要gydF4y2B一个
完整准确的参考基因组和注释为功能基因组学和作物育种提供了基础资源。本文报道了一个豌豆品种ZW6的重组和注释,其contig N50长度为8.98 Mb,与现有的豌豆品种相比,其contig长度增加了243倍,在复杂重复区序列的连续性和质量均有明显提高。118株栽培豌豆和野生豌豆的基因组多样性证明了这一点gydF4y2B一个Pisum abyssinicumgydF4y2B一个一个单独的物种不同于gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p .一gydF4y2B一个在gydF4y2B一个PisumgydF4y2B一个.定量性状位点分析发现了两个已知的与茎长相关的孟德尔氏基因(gydF4y2B一个勒/勒gydF4y2B一个)和种子形状(gydF4y2B一个R / RgydF4y2B一个)以及孟德尔研究的荚果形态的一些候选基因。构建了116份豌豆材料的泛基因组,结果表明gydF4y2B一个p . abyssinicumgydF4y2B一个而且gydF4y2B一个p . fulvumgydF4y2B一个功能富集明显,表明其作为豌豆育种资源的潜在价值。gydF4y2B一个
主要gydF4y2B一个
识别和理解驯化过程中表型变异的遗传基础是现代遗传学和进化生物学的主要焦点之一gydF4y2B一个1gydF4y2B一个,gydF4y2B一个2gydF4y2B一个.在过去的几十年里,下一代测序(NGS)技术极大地促进了作物基因组学研究,从而更好地理解基因组结构和复杂性gydF4y2B一个3.gydF4y2B一个,gydF4y2B一个4gydF4y2B一个.高质量的参考基因组和完整的注释为群体基因组学和分子遗传学研究提供了重要的工具,以了解作物的驯化和加速遗传改良gydF4y2B一个5gydF4y2B一个,gydF4y2B一个6gydF4y2B一个.大量基于单核苷酸多态性(SNPs)和小插入/缺失(indel)多态性的作物群体基因组学研究和全基因组关联分析为了解作物驯化和重要性状的基因挖掘奠定了重要基础gydF4y2B一个7gydF4y2B一个,gydF4y2B一个8gydF4y2B一个,gydF4y2B一个9gydF4y2B一个,gydF4y2B一个10gydF4y2B一个,gydF4y2B一个11gydF4y2B一个.许多研究已经确定了涉及定义基因组结构、基因功能和表达水平的结构变异(SVs),并描述了它们在植物进化、表型多样性和作物改良中的重要作用gydF4y2B一个12gydF4y2B一个,gydF4y2B一个13gydF4y2B一个,gydF4y2B一个14gydF4y2B一个,gydF4y2B一个15gydF4y2B一个,gydF4y2B一个16gydF4y2B一个.然而,SV的长度、类型、分布和群体频率及其对表型的贡献尚未得到充分的描述gydF4y2B一个15gydF4y2B一个,gydF4y2B一个17gydF4y2B一个,gydF4y2B一个18gydF4y2B一个.gydF4y2B一个
越来越多的研究证明,单一的参考基因组不足以代表一个物种,特别是由于与作物长期驯化相关的遗传结构的多样化和改变,而由不同个体构建的泛基因组作为捕获物种内部多样性的工具越来越受欢迎gydF4y2B一个9gydF4y2B一个,gydF4y2B一个14gydF4y2B一个,gydF4y2B一个16gydF4y2B一个,gydF4y2B一个19gydF4y2B一个,gydF4y2B一个20.gydF4y2B一个,gydF4y2B一个21gydF4y2B一个,gydF4y2B一个22gydF4y2B一个.近年来对植物泛基因组的研究成功地揭示了功能重要基因存在/缺失变异(PAVs),核心基因/同源基因簇的比例在33% ~ 92%之间gydF4y2B一个21gydF4y2B一个.大规模sv的发现及其与基因组进化、基因表达和农艺性状的关系也有报道。这些研究为了解作物驯化、探索基因功能和利用育种资源作出了贡献gydF4y2B一个18gydF4y2B一个,gydF4y2B一个21gydF4y2B一个,gydF4y2B一个22gydF4y2B一个.gydF4y2B一个
豌豆(gydF4y2B一个Pisum一gydF4y2B一个L., 2n = 2x = 14),一年生冷季豆科植物,属豆科、凤蝶科和gydF4y2B一个PisumgydF4y2B一个基因组大小约为4.45 GbgydF4y2B一个23gydF4y2B一个,gydF4y2B一个24gydF4y2B一个.豌豆作为新鲜蔬菜和干粮,在食品和饲料工业中是一种多功能作物gydF4y2B一个24gydF4y2B一个,gydF4y2B一个25gydF4y2B一个.豌豆的收获面积在豆科植物中排名第四,仅次于大豆、菜豆和鹰嘴豆(gydF4y2B一个http://www.fao.org/faostat/gydF4y2B一个).作为蛋白质,淀粉,纤维和矿物质的来源gydF4y2B一个26gydF4y2B一个,gydF4y2B一个27gydF4y2B一个其生物固氮能力强,具有明显的生态可持续性优势gydF4y2B一个28gydF4y2B一个在美国,豌豆一直受到人们的关注,尤其是在孟德尔通过豌豆育种实验揭示了遗传规律之后gydF4y2B一个29gydF4y2B一个,gydF4y2B一个30.gydF4y2B一个.据推测,豌豆大约在1万年前由近东和中东的新石器时代的农民驯化,被认为是最早驯化的豆类作物之一gydF4y2B一个31gydF4y2B一个,gydF4y2B一个32gydF4y2B一个,gydF4y2B一个33gydF4y2B一个.然而,尽管它在推进植物遗传学方面发挥了关键作用,但它的驯化过程仍然是一个谜,栽培豌豆和野生豌豆的遗传多样性也是一个谜gydF4y2B一个PisumgydF4y2B一个还没有完全发现。gydF4y2B一个
最近,基于NGS技术构建的豌豆参考基因组的可用性为豆科植物的基因组进化提供了新的视角gydF4y2B一个34gydF4y2B一个.然而,为了更好地理解豌豆的表型变异和基因组进化,需要改进基因组组装和基因组注释gydF4y2B一个6gydF4y2B一个,gydF4y2B一个35gydF4y2B一个,gydF4y2B一个36gydF4y2B一个.基于PacBio单分子实时(SMRT)测序技术,结合10x Genomics测序、Bionano光学作图和染色体构象捕获(Hi-C)测序以及Illumina NGS技术,构建了豌豆品种ZW6的全新基因组组合。该组合为豌豆的参考基因组和注释提供了明显的改进。我们进一步鉴定了全基因组变异(SNPs、indels和SVs),并基于全基因组重测序数据展示了118种栽培豌豆和野生豌豆基因型的群体遗传结构。通过基因组选择和QTL分析,发现了一批与驯化和育种改良性状相关的候选基因,其中包括几个孟德尔基因候选基因。我们还报告了基于这118个附加基因和序列的豌豆泛基因组,这些附加基因和序列在参考基因组中不存在。高质量的参考基因组和泛基因组为豌豆基因组的进化和驯化提供了新的视角,为豌豆遗传育种研究提供了宝贵的基因组资源gydF4y2Ba22gydF4y2B一个,gydF4y2B一个37gydF4y2B一个.gydF4y2B一个
结果gydF4y2B一个
基因组组装体PeaZW6的构建与评价gydF4y2B一个
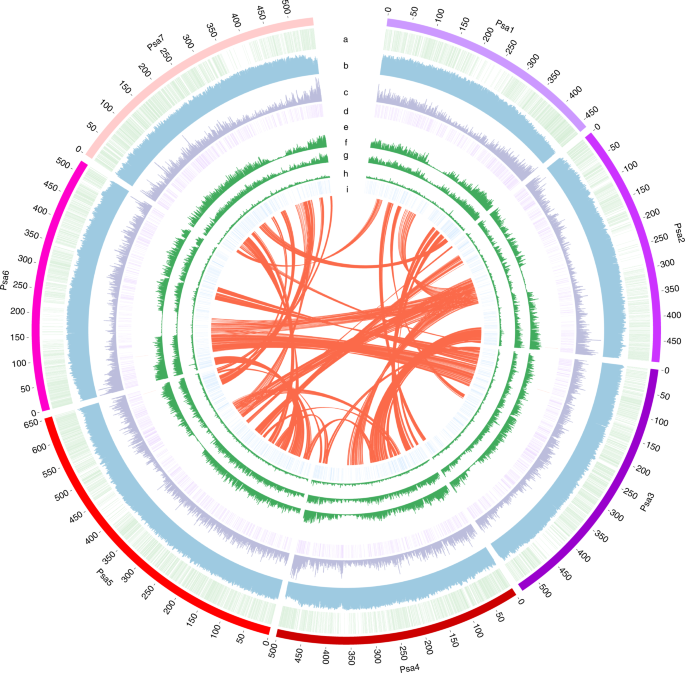
ZW6是一种广泛种植的豌豆品种。gydF4y2B一个1gydF4y2B一个).流式细胞术估计ZW6的基因组大小为4.28 Gb。gydF4y2B一个2gydF4y2B一个及补充表gydF4y2B一个1gydF4y2B一个)和4.26 Gb K-mer分析(补充图。gydF4y2B一个3.gydF4y2B一个).这些估计值小于先前报道的基因组大小(4.45 Gb)。gydF4y2B一个23gydF4y2B一个,gydF4y2B一个24gydF4y2B一个.K-mer分析也显示ZW6具有非常低的杂合度(0.08%)和较高的重复序列比例(83%)(补充图)。gydF4y2B一个3.gydF4y2B一个).使用PacBio SMRT测序、10x Genomics脚手架、Bionano光学作图、Hi-C脚手架和Illumina NGS技术的组合(补充图)。gydF4y2B一个4gydF4y2B一个及补充表gydF4y2B一个2gydF4y2B一个),构建了一个高质量、高连续性的ZW6染色体水平参考组装体(PeaZW6)。gydF4y2B一个1gydF4y2B一个和表gydF4y2B一个1gydF4y2B一个).基于379.34 Gb PacBio reads (~85.2×基因组覆盖率)的初始组装总大小为3796.7 Mb, contig N50大小为8.98 Mb。经过打磨,迭代搭建,人工策展(补充图。gydF4y2B一个5gydF4y2B一个),最后的组装被锚定到7个染色体水平的假分子中,其中有两个细胞器基因组和1572个未放置的contigs(图2)。gydF4y2B一个1gydF4y2B一个和表gydF4y2B一个1gydF4y2B一个).锚定contigs的总大小为3719.6 Mb,占PeaZW6的97.96%,而锚定contigs仅占Caméor (PeaCaméor)之前基于ngs的组装体的82.51%。gydF4y2B一个34gydF4y2B一个.未知序列的累计长度为10.3 Mb,远小于PeaCaméor的760.8 MbgydF4y2B一个34gydF4y2B一个.将修正后的reads映射到PeaZW6后,发现99.41%和99.16%的assembly分别被至少20个PacBio reads和20个NGS reads覆盖,这证实了PeaZW6的高质量gydF4y2B一个3.gydF4y2B一个,补充图。gydF4y2B一个6gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).gydF4y2B一个
改良PeaZW6的BUSCO完整性(99.38%,基因组模式)也高于PeaCaméor(96.78%,基因组模式)(补充表)gydF4y2B一个4gydF4y2B一个).大多数组织中RNA测序(RNA-seq) reads的映射率大于99%(补充表)gydF4y2B一个5gydF4y2B一个).此外,merquury分析显示PeaZW6的共识质量值(QV)(44.5)几乎是PeaCaméor(24.3)的两倍(补充表)gydF4y2B一个6gydF4y2B一个),证实PeaZW6的质量更高,准确度更高。具体而言,PeaZW6含有98.5%的独特遗传标记,表明染色体水平组装与先前报道的遗传图谱之间存在高度共线性gydF4y2B一个38gydF4y2B一个(补充图。gydF4y2B一个7gydF4y2B一个).豌豆和紫花苜蓿基因组间的同步区(gydF4y2B一个Medicago truncatulagydF4y2B一个)的检测结果表明,PeaZW6/Medicago同线区域内的同源基因数量明显且一致地大于PeaCaméor/Medicago在不同参数下的同源基因数量(补充表gydF4y2B一个7gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个),验证了PeaZW6的长程连续性。gydF4y2B一个
PeaZW6基因组注释gydF4y2B一个
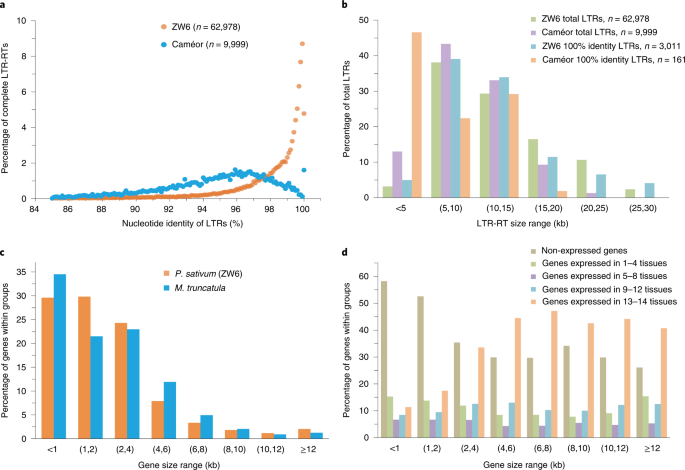
PeaZW6的重复元素总长度为3249.5 Mb,大于PeaCaméor的2662.5 Mb。Gypsy是转座因子的主导类型,占PeaZW6的54.34%(补充数据gydF4y2B一个1gydF4y2B一个).长端重复(LTR)组装指数(LAI)分析表明,与PeaCaméor (LAI = 2.09)相比,PeaZW6的LTR-逆转录转座子(LTR- rt)完整性有显著改善(LAI = 13.31)(补充表gydF4y2B一个8gydF4y2B一个).PeaZW6具有比PeaCaméor更多的全长ltr - r,并且活动和较长的ltr比例更高(图2)。gydF4y2B一个2 a, bgydF4y2B一个).这些结果可以解释为什么基于pacbio的PeaZW6 (10.3 Mb)和基于ngs的PeaCaméor (760.8 Mb)之间的间隙大小存在明显差异。LTR-RT完整性的提高表明,最近活跃的长重复序列的组装受益于基于PacBio长读的组装。gydF4y2B一个
在PeaZW6中共鉴定出47,526个编码基因gydF4y2B一个9gydF4y2B一个而且gydF4y2B一个10gydF4y2B一个).基因平均长度为2563.7 bp,编码序列平均长度为1122.3 bpgydF4y2B一个9gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).在PeaZW6中,3kb侧翼编码序列中存在缺口的基因数量从PeaCaméor的20%下降到1%(补充图)。gydF4y2B一个8gydF4y2B一个),每个基因的转录数从1.29增加到1.42(补充图;gydF4y2B一个8 bgydF4y2B一个),表明调控区序列的完整性和选择性剪接的注释得到了改善。PeaZW6基因的蛋白模式BUSCO完整性(97.77%)也高于PeaCaméor(93.99%)(补充表)gydF4y2B一个4gydF4y2B一个).PeaZW6蛋白编码基因的长度分布与紫花苜蓿相当,其基因组大小约为豌豆的八分之一(图2)。gydF4y2B一个2摄氏度gydF4y2B一个).此外,长度超过2 kb的基因具有相似的表达宽度模式(图2)。gydF4y2B一个二维gydF4y2B一个).这些结果表明,豌豆基因组中重复序列的高含量可能对基因结构或蛋白编码基因的表达谱影响不大。gydF4y2B一个
基因多态性gydF4y2B一个
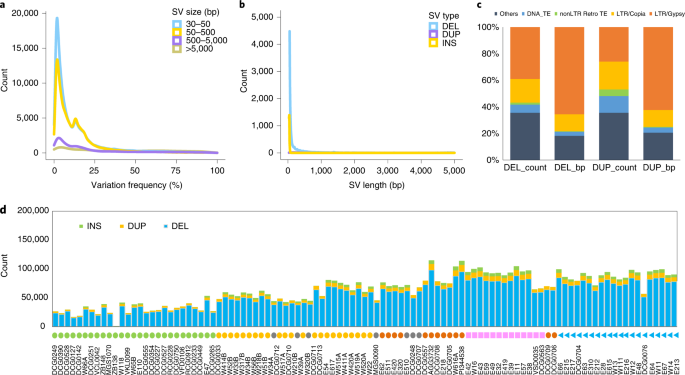
目的:研究栽培豌豆和野生豌豆的基因多态性gydF4y2B一个PisumgydF4y2B一个共鉴定出26,250,039个高质量SNPs和1,443,829个小索引gydF4y2B一个PisumgydF4y2B一个(补充数据gydF4y2B一个2gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个),其中64.1%的SNPs和53.0%的索引位于基因间区,只有2.4%的SNPs和1.1%的索引位于外显子(补充表gydF4y2B一个11gydF4y2B一个).从118个大于30个bp的SVs中筛选出了376,309个gydF4y2B一个PisumgydF4y2B一个主要由删除组成(94.5%)(补充表gydF4y2B一个12gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).对SVs的分析表明,大多数SVs较小,且存在相对较低的变异频率(图2)。gydF4y2B一个3 a, bgydF4y2B一个).此外,还发现85.5%的缺失和77.4%的重复来自重复序列,其中以LTR/Copia和LTR/Gypsy为主(图2)。gydF4y2B一个3 cgydF4y2B一个).每次加入的sv数量在916至114,900之间,平均为63,987。相比于栽培的gydF4y2B一个p .一gydF4y2B一个,加入gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p . abyssinicumgydF4y2B一个对PeaZW6参考基因组有更多的sv(图;gydF4y2B一个3 dgydF4y2B一个).gydF4y2B一个
群体遗传结构gydF4y2B一个
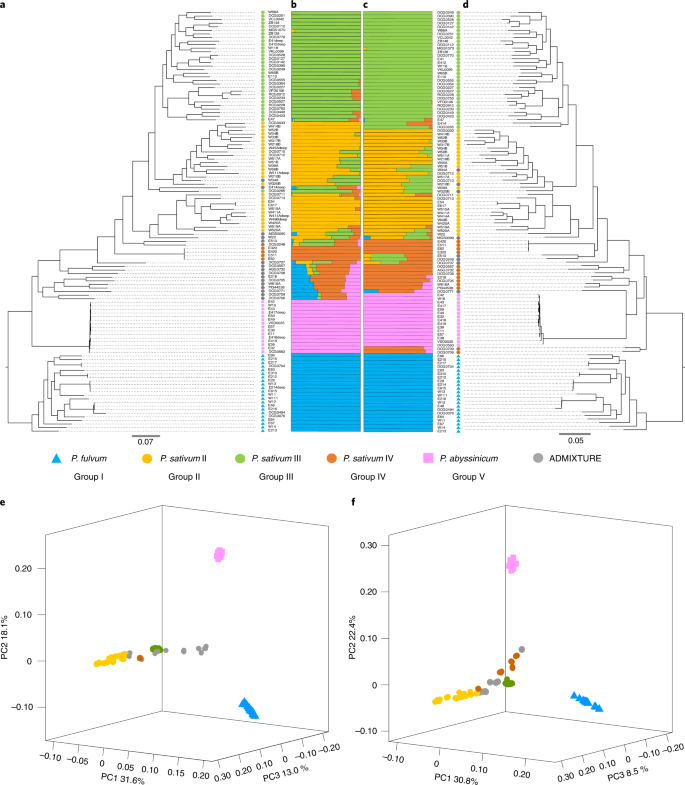
目的:阐明栽培豌豆与野生豌豆的系统发育关系及群体遗传结构gydF4y2B一个PisumgydF4y2B一个, admix应用于SNP和SV数据集,结果高度一致(图2)。gydF4y2B一个4 b, cgydF4y2B一个和补充图。gydF4y2B一个9gydF4y2B一个).有三个不同物种的结构gydF4y2B一个PisumgydF4y2B一个,gydF4y2B一个p . fulvumgydF4y2B一个,gydF4y2B一个p .一gydF4y2B一个而且gydF4y2B一个p . abyssinicumgydF4y2B一个获得一致支持。其中鉴定出了三个遗传群gydF4y2B一个p .一gydF4y2B一个,其中gydF4y2B一个p .一gydF4y2B一个IV (PSIV)代表分化较早的组(图。gydF4y2B一个4 b, cgydF4y2B一个).gydF4y2B一个p .一gydF4y2B一个II (PSII)和gydF4y2B一个p .一gydF4y2B一个III (PSIII)主要对应代表不同地理区域(即亚洲和欧洲)栽培豌豆的两个遗传群,这可能与豌豆驯化后的传播途径有关(图2)。gydF4y2B一个4 b, cgydF4y2B一个).利用SNP和SV数据集构建系统发育树(图。gydF4y2B一个4 dgydF4y2B一个)在主要分支上表现出相似的系统发育关系,与admix结果的主要遗传类群有良好的对应关系。此外,gydF4y2B一个p . fulvumgydF4y2B一个,gydF4y2B一个p . abyssinicumgydF4y2B一个和培育gydF4y2B一个p .一gydF4y2B一个的gydF4y2B一个PisumgydF4y2B一个形成了三个独立的演化支(图;gydF4y2B一个4 dgydF4y2B一个),这也得到了SNP和SV数据集主成分分析的支持(图。gydF4y2B一个4 e, fgydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).gydF4y2B一个
一个gydF4y2B一个,基于snp的系统发育树。gydF4y2B一个bgydF4y2B一个,基于snp的admix分析gydF4y2B一个KgydF4y2B一个= 5。gydF4y2B一个cgydF4y2B一个,基于sv的admix分析gydF4y2B一个KgydF4y2B一个= 5。gydF4y2B一个dgydF4y2B一个,基于sv的系统发育树。gydF4y2B一个egydF4y2B一个,基于snp的主成分(PC)分析。gydF4y2B一个fgydF4y2B一个,基于sv的主成分分析。颜色和形状表示植物的遗传群和分类种gydF4y2B一个PisumgydF4y2B一个分别是每一次加入。gydF4y2B一个
PisumgydF4y2B一个多样性与连锁失衡gydF4y2B一个
基于admix的结果,首先计算了内部各种的遗传多样性gydF4y2B一个PisumgydF4y2B一个每一个基因组gydF4y2B一个p .一gydF4y2B一个单核苷酸多态性。在这三个物种中,gydF4y2B一个p .一gydF4y2B一个具有最高的核苷酸多样性(π = 9.40 × 10gydF4y2B一个−4gydF4y2B一个)接着是gydF4y2B一个p . fulvumgydF4y2B一个π = 7.22 × 10gydF4y2B一个−4gydF4y2B一个),gydF4y2B一个p . abyssinicumgydF4y2B一个π = 2.44 × 10gydF4y2B一个−4gydF4y2B一个)(补充图gydF4y2B一个10gydF4y2B一个).的三个遗传群gydF4y2B一个p .一gydF4y2B一个,gydF4y2B一个p .一gydF4y2B一个II保留了最大的核苷酸多样性(π = 9.13 × 10gydF4y2B一个−4gydF4y2B一个);核苷酸的多样性gydF4y2B一个p .一gydF4y2B一个π = 6.32 × 10gydF4y2B一个−4gydF4y2B一个)(补充图gydF4y2B一个10 bgydF4y2B一个).gydF4y2B一个
此外,群体遗传分化(gydF4y2B一个FgydF4y2B一个圣gydF4y2B一个)在具有SNPs的物种和遗传群中进行了估计。种间分化明显大于种内分化gydF4y2B一个PisumgydF4y2B一个(补充图。gydF4y2B一个10gydF4y2B一个).在三个物种之间,遗传分化gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p . abyssinicumgydF4y2B一个最高的(gydF4y2B一个FgydF4y2B一个圣gydF4y2B一个= 0.563),其次是betweengydF4y2B一个p . abyssinicumgydF4y2B一个而且gydF4y2B一个p .一gydF4y2B一个(gydF4y2B一个FgydF4y2B一个圣gydF4y2B一个= 0.522),之间gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p .一gydF4y2B一个(gydF4y2B一个FgydF4y2B一个圣gydF4y2B一个= 0.440)(补充图。gydF4y2B一个10gydF4y2B一个).在三个基因组中,gydF4y2B一个p .一gydF4y2B一个二世和gydF4y2B一个p .一gydF4y2B一个III的遗传分化最低(gydF4y2B一个FgydF4y2B一个圣gydF4y2B一个= 0.175)(补充图。gydF4y2B一个10 bgydF4y2B一个),这与系统发育分析相一致(图;gydF4y2B一个4 dgydF4y2B一个).gydF4y2B一个
连杆不平衡(LD) (gydF4y2B一个RgydF4y2B一个2gydF4y2B一个)用SNPs计算,但在不同物种之间有所不同gydF4y2B一个PisumgydF4y2B一个和不同的基因群gydF4y2B一个p .一gydF4y2B一个(补充图。gydF4y2B一个11gydF4y2B一个).在6kb处,LD降至最大值的一半gydF4y2B一个p . fulvumgydF4y2B一个,而LD范围为gydF4y2B一个p .一gydF4y2B一个约25 kb,与野生大豆相近(gydF4y2B一个甘氨酸大豆gydF4y2B一个, 27kb)gydF4y2B一个7gydF4y2B一个野生玉米(gydF4y2B一个玉米gydF4y2B一个ssp。gydF4y2B一个parviglumisgydF4y2B一个, 22 kb)gydF4y2B一个39gydF4y2B一个.在gydF4y2B一个p .一gydF4y2B一个二世和gydF4y2B一个p .一gydF4y2B一个III, LD衰减距离增大,分别为80 kb和35 kb。gydF4y2B一个
豌豆驯化过程中的选择信号gydF4y2B一个
为了确定在豌豆驯化过程中被选择的假定的选择性基因组区域,跨种群复合似然比检验(XP-CLR)gydF4y2B一个40gydF4y2B一个进行不同的比较gydF4y2B一个p . fulvumgydF4y2B一个与gydF4y2B一个p .一gydF4y2B一个而且gydF4y2B一个p . fulvumgydF4y2B一个与gydF4y2B一个p . abyssinicumgydF4y2B一个.之间的gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p .一gydF4y2B一个共鉴定了514次扫描,共7279个基因,覆盖15.54% (~578 Mb)的组装基因组(补充数据gydF4y2B一个3.gydF4y2B一个和补充图。gydF4y2B一个12 a, c, egydF4y2B一个).之间的gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p . abyssinicumgydF4y2B一个,共检测到609个扫描,包含10,132个基因,占组装基因组的19.34% (~719 Mb)gydF4y2B一个4gydF4y2B一个,补充图。gydF4y2B一个12 b, d, fgydF4y2B一个),候选选择区域含有多个与荚果开裂和种子休眠相关基因同源的基因gydF4y2B一个g·马克斯gydF4y2B一个而且gydF4y2B一个m . truncatulagydF4y2B一个(补充数据gydF4y2B一个5gydF4y2B一个).对假定选择区域内的基因进行分析表明,发现有1494个基因是共同的gydF4y2B一个p .一gydF4y2B一个而且gydF4y2B一个p . abyssinicumgydF4y2B一个而这两种基因分别为5785个和8638个。基因本体论(GO)分析8,638个候选基因的独特选择gydF4y2B一个p . abyssinicumgydF4y2B一个揭示了参与对非生物和生物刺激反应的基因的富集(补充表gydF4y2B一个13gydF4y2B一个).gydF4y2B一个
孟德尔基因位点的QTL分析与重新发现gydF4y2B一个
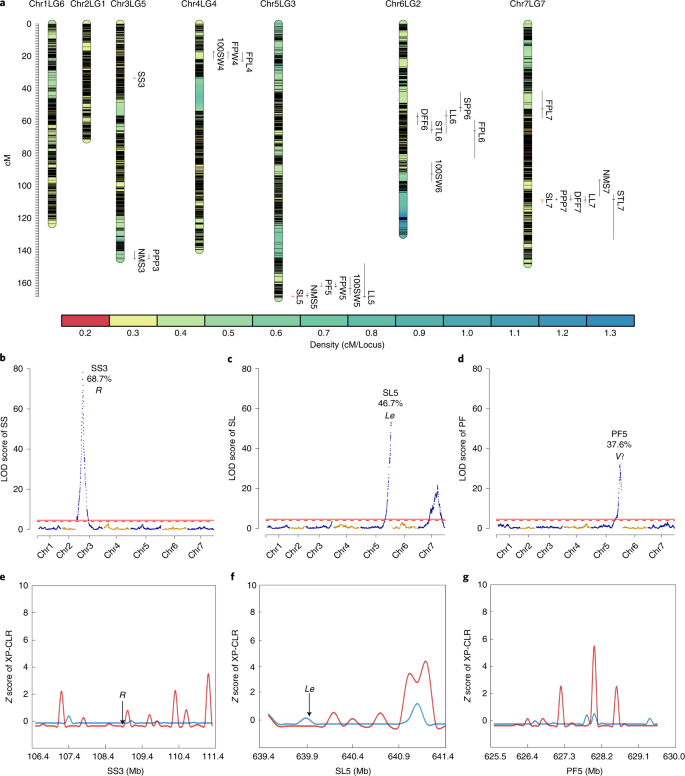
为探索豌豆重要农艺性状的遗传基础,对300 F的12个农艺性状进行了QTL分析gydF4y2B一个2gydF4y2B一个人群(WJ×ZW6)使用基因测序分型(补充数据gydF4y2B一个6gydF4y2B一个而且gydF4y2B一个7gydF4y2B一个,补充图。gydF4y2B一个13gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).将124900个高质量SNP标记聚为2950个bin标记,构建了7个跨度924.1 cM的高密度(0.31 cM)遗传连锁图谱(Supplementary Table)gydF4y2B一个14gydF4y2B一个和补充图。gydF4y2B一个14gydF4y2B一个).共发现25个qtl与12个农艺性状相关,对数比值(LOD)值在4.2 ~ 78.1之间,最大表型解释变异(PVE)高达68.7%(图5)。gydF4y2B一个5gydF4y2B一个及补充资料gydF4y2B一个8gydF4y2B一个).在Mendel分析的25个QTL中,与3个性状相关的SS3、SL5和PF5具有较高的LOD(78.1、53.1和31.9)和PVE(68.7%、46.7%和37.6%),在基因组中出现了明显的QTL峰值(4.87 Mb、1.85 Mb和4.43 Mb)(图5)。gydF4y2B一个5罪犯gydF4y2B一个及补充资料gydF4y2B一个8gydF4y2B一个).对SS3、SL5和PF5的同源性比对和功能注释的结果发现了两个先前已知的孟德尔性状的遗传位点,gydF4y2B一个RgydF4y2B一个41gydF4y2B一个而且gydF4y2B一个勒gydF4y2B一个42gydF4y2B一个(补充数据gydF4y2B一个9gydF4y2B一个而且gydF4y2B一个10gydF4y2B一个),以及一个可能的候选基因(gydF4y2B一个Psat05G0794700gydF4y2B一个)与豆荚形态相关(补充数据gydF4y2B一个11gydF4y2B一个而且gydF4y2B一个12gydF4y2B一个).然而,这些基因中没有一个属于假定的选择区域,这意味着它们可能与豌豆的驯化没有密切的联系(图2)。gydF4y2B一个5比gydF4y2B一个).gydF4y2B一个
一个gydF4y2B一个共鉴定出25个与12个农艺性状相关的qtl,其中红色条表示4个与种子形状(SS)、茎长(SL)和荚果形态(PF) 3个孟德尔性状相关的qtl。gydF4y2B一个bgydF4y2B一个- - - - - -gydF4y2B一个dgydF4y2B一个、SS3中LOD评分、PVE及候选基因的分布(gydF4y2B一个bgydF4y2B一个), sl5 (gydF4y2B一个cgydF4y2B一个)及PF5 (gydF4y2B一个dgydF4y2B一个),其中红色实线和折线分别代表0.01和0.05的阈值。gydF4y2B一个egydF4y2B一个- - - - - -gydF4y2B一个ggydF4y2B一个, SS3中的候选选择信号(gydF4y2B一个egydF4y2B一个), sl5 (gydF4y2B一个fgydF4y2B一个)及PF5 (gydF4y2B一个ggydF4y2B一个)基于物种间的XP-CLR分析结果gydF4y2B一个PisumgydF4y2B一个,其中红线表示gydF4y2B一个p . fulvumgydF4y2B一个与gydF4y2B一个p .一gydF4y2B一个与gydF4y2B一个αgydF4y2B一个0.05gydF4y2B一个= 2.18,蓝线表示gydF4y2B一个p . fulvumgydF4y2B一个与gydF4y2B一个p . abyssinicumgydF4y2B一个与gydF4y2B一个αgydF4y2B一个0.05gydF4y2B一个= 0.39。gydF4y2B一个
118株栽培和野生豌豆的泛基因组分析gydF4y2B一个
为了更深入的了解gydF4y2B一个PisumgydF4y2B一个对118份栽培和野生豌豆种质的个体从头组装进行了泛基因组分析(补充数据gydF4y2B一个13gydF4y2B一个).通过比对PeaZW6参考基因,我们发现在一个遗传群体中,新序列和基因的比例是相似的,但随着该群体与ZW6遗传距离的增加而增加(补充数据)gydF4y2B一个14gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).同时,在合并新序列以去除PeaZW6之外的冗余后,我们还发现来自所有接入的新序列的百分比高于任何遗传组(补充数据gydF4y2B一个15gydF4y2B一个),表明植物的大部分多样性gydF4y2B一个PisumgydF4y2B一个主要是在不同类群之间以基因组序列唯一性的形式存在。gydF4y2B一个
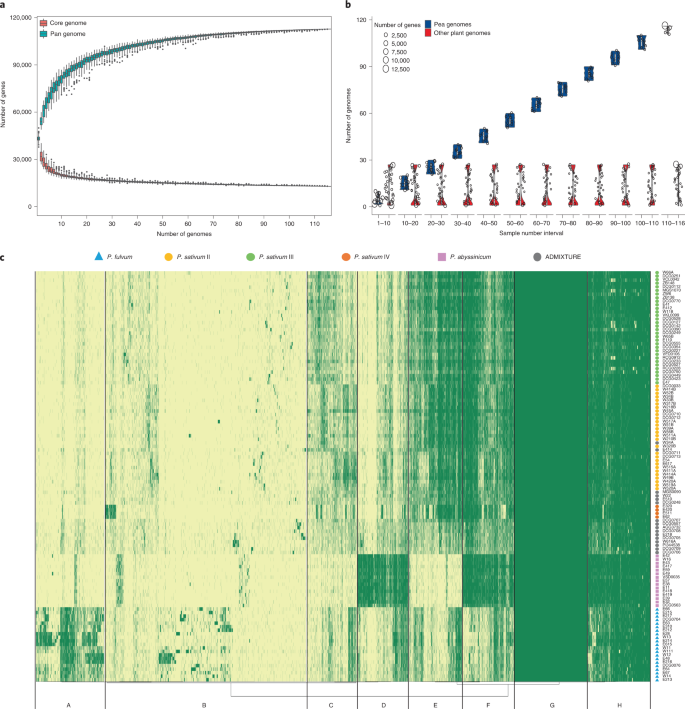
为了进一步研究与性状或功能相关的新序列,本文对植物的PAV模式进行了分析gydF4y2B一个PisumgydF4y2B一个进行泛基因分析(gydF4y2B一个补充笔记gydF4y2B一个).随着新基因组的加入,核心基因数量减少,泛基因数量增加,逐渐趋于饱和(图2)。gydF4y2B一个6gydF4y2B一个).经过质量控制,来自PeaZW6和115个合格基因组的基因,基于跨基因组同源物的系统发育,聚类为112,776个泛基因代表系统发育等级正构群(HOGs)(图。gydF4y2B一个6gydF4y2B一个及补充资料gydF4y2B一个16gydF4y2B一个).在gydF4y2B一个PisumgydF4y2B一个,核心基因、软核基因、壳基因和云基因数量分别为15470、6170、41028和50108,分别占聚类前基因总数的35.19%、15.54%、44.28%和4.99%(补充数据)gydF4y2B一个16gydF4y2B一个).各组核心基因所占比例均高于对照组gydF4y2B一个PisumgydF4y2B一个总体(补充数据)gydF4y2B一个16gydF4y2B一个),与新序列百分比一致。值得注意的是,群体的核心百分比可能对应于其计算的遗传多样性(补充图。gydF4y2B一个10gydF4y2B一个),这表明遗传多样性也可能对核心基因的比例有贡献。与此同时,在其他27种植物基因组中,核心基因也趋于保守。gydF4y2B一个6 bgydF4y2B一个及补充资料gydF4y2B一个17gydF4y2B一个),说明它们的基本功能。此外,PAVs的邻域连接树也显示出116的明显分离gydF4y2B一个PisumgydF4y2B一个这与基于SNPs和sv的结果高度一致(补充图;gydF4y2B一个15gydF4y2B一个),表明重要的遗传变异有助于驯化gydF4y2B一个PisumgydF4y2B一个也被埋在pav里。gydF4y2B一个
为了检测泛基因组中的基因偏好和功能富集,PAV模式进一步将hog聚类为8个聚类,命名为A至H(图2)。gydF4y2B一个6摄氏度gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).该模式显示gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p . abyssinicumgydF4y2B一个种质资源具有丰富的独特基因,具有潜在的育种价值。许多gydF4y2B一个p .一gydF4y2B一个这可能反映了其育种历史中基因渗透的潜在事件。gydF4y2B一个
最后,PAV聚类、遗传群和遗传群中独特泛基因的GO功能富集在保守基因(核心和软核基因)和可变基因(壳和云基因)之间呈现分化的功能富集。保守基因在碳水化合物和脂类代谢过程等基本功能中富集。变异基因在应激和刺激反应等辅助功能上富集。值得注意的是,独特的泛基因gydF4y2B一个p . abyssinicumgydF4y2B一个刺激和化学反应丰富,而泛基因gydF4y2B一个p . fulvumgydF4y2B一个在发育、生长、繁殖、细胞骨架和向性中富集(补充图。gydF4y2B一个16gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).这进一步证实了的潜在价值gydF4y2B一个p . abyssinicumgydF4y2B一个而且gydF4y2B一个p . fulvumgydF4y2B一个作为今后提高豌豆品种抗性和产量的育种材料。gydF4y2B一个
讨论gydF4y2B一个
豌豆是重要的豆科作物之一,具有较高的营养价值和生物固氮能力gydF4y2B一个43gydF4y2B一个,gydF4y2B一个44gydF4y2B一个自孟德尔遗传定律被发现以来,它也一直是遗传研究的模式植物物种gydF4y2B一个45gydF4y2B一个.高质量的参考基因组和注释为作物遗传性状的鉴定提供了基础资源。遗憾的是,该作物长期缺乏高质量的参考基因组和遗传转化系统,从而失去了主导地位,成为现代基因组时代的孤儿作物gydF4y2B一个46gydF4y2B一个,gydF4y2B一个47gydF4y2B一个,gydF4y2B一个48gydF4y2B一个.在本研究中,通过生成基于完整PacBio SMRT长读测序的新型组装,该基因组的contig长度增加了243倍,显示出在之前的参考基因组中作为间隙的复杂重复区域和转座元件的连续性和质量的显著改善gydF4y2B一个34gydF4y2B一个(表gydF4y2B一个1gydF4y2B一个,补充图。gydF4y2B一个7gydF4y2B一个而且gydF4y2B一个8gydF4y2B一个,补充表gydF4y2B一个8gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).新的参考基因组拓宽了我们对巨大豌豆基因组背后的遗传学知识,并将促进未来的育种研究,这可能有助于养活世界。gydF4y2B一个
尽管很多研究都集中在分类上gydF4y2B一个PisumgydF4y2B一个在美国,这个长期存在的问题仍然没有解决,关于豌豆驯化的许多困惑仍然存在gydF4y2B一个49gydF4y2B一个,gydF4y2B一个50gydF4y2B一个,gydF4y2B一个51gydF4y2B一个,gydF4y2B一个52gydF4y2B一个,gydF4y2B一个53gydF4y2B一个.争论的焦点之一是gydF4y2B一个p . abyssinicumgydF4y2B一个,即是将其视为一个独立的物种还是内部的亚种gydF4y2B一个p .一gydF4y2B一个54gydF4y2B一个.鉴于其独特的形态特征、繁殖隔离程度和特定的分布区域gydF4y2B一个51gydF4y2B一个,gydF4y2B一个54gydF4y2B一个,gydF4y2B一个55gydF4y2B一个,以及利用泛基因组中确定的基因组SNPs、SVs和PAV进行系统发育分析的结果,我们强烈支持gydF4y2B一个p . abyssinicumgydF4y2B一个毫无疑问是一个独立的物种gydF4y2B一个p . fulvumgydF4y2B一个而且gydF4y2B一个p .一gydF4y2B一个在gydF4y2B一个PisumgydF4y2B一个(无花果。gydF4y2B一个4gydF4y2B一个而且gydF4y2B一个6gydF4y2B一个,补充图。gydF4y2B一个9gydF4y2B一个而且gydF4y2B一个15gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).此外,对传统意义上对gydF4y2B一个p .一gydF4y2B一个无性系种群。gydF4y2B一个elatiusgydF4y2B一个可能是现代豌豆的祖先gydF4y2B一个56gydF4y2B一个.在中观察到高的浸润率gydF4y2B一个p .一gydF4y2B一个无性系种群。gydF4y2B一个elatiusgydF4y2B一个(无花果。gydF4y2B一个4gydF4y2B一个),暗示它可能是栽培豌豆与野生豌豆杂交的产物。这种杂交起源也得到了最近一项野生杂交分析的支持gydF4y2B一个p .一gydF4y2B一个包括北方gydF4y2B一个阿根廷蚂蚁gydF4y2B一个南部gydF4y2B一个阿根廷蚂蚁gydF4y2B一个而且gydF4y2B一个p .一gydF4y2B一个无性系种群。gydF4y2B一个elatiusgydF4y2B一个57gydF4y2B一个.gydF4y2B一个
豆荚开裂和种子休眠是豆科植物驯化过程中的两个关键性状gydF4y2B一个58gydF4y2B一个.分子遗传学研究已经确定了控制这两个性状的几个基因,并证明了豆科植物物种间的平行选择gydF4y2B一个59gydF4y2B一个,gydF4y2B一个60gydF4y2B一个.一个被认为与豌豆荚果开裂有关的基因是gydF4y2B一个Dpo1gydF4y2B一个61gydF4y2B一个,gydF4y2B一个62gydF4y2B一个,肽聚糖结合结构域蛋白(PGBD)的同源物gydF4y2B一个m . truncatulagydF4y2B一个(gydF4y2B一个Medtr2g079050gydF4y2B一个)gydF4y2B一个58gydF4y2B一个.基于同源比对,gydF4y2B一个Dop1gydF4y2B一个注释为gydF4y2B一个Psat05G0678800gydF4y2B一个在PeaZW6基因组中,并定位于假定的选择区域gydF4y2B一个p . abyssinicumgydF4y2B一个但不是在gydF4y2B一个p .一gydF4y2B一个(补充数据gydF4y2B一个5gydF4y2B一个),表明它可能在两个物种中经历了独立的驯化,正如之前的研究所提到的那样gydF4y2B一个55gydF4y2B一个.gydF4y2B一个GmHs1-1gydF4y2B一个而且gydF4y2B一个GmGgydF4y2B一个对大豆种子休眠有控制作用gydF4y2B一个63gydF4y2B一个,gydF4y2B一个64gydF4y2B一个.两个同源基因gydF4y2B一个Psat02G0081200gydF4y2B一个而且gydF4y2B一个Psat02G0507900gydF4y2B一个对应于gydF4y2B一个GmHs1-1gydF4y2B一个而且gydF4y2B一个GmGgydF4y2B一个,分别在PeaZW6中被鉴定出来,并且都存在于推定的选择区域gydF4y2B一个p . abyssinicumgydF4y2B一个(补充数据gydF4y2B一个5gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).gydF4y2B一个
孟德尔通过研究豌豆的七个特征,开创了基因研究的先河gydF4y2B一个29gydF4y2B一个,gydF4y2B一个30.gydF4y2B一个.在过去的几十年里,控制种子形状的孟德尔基因位点(gydF4y2B一个RgydF4y2B一个/gydF4y2B一个rgydF4y2B一个)gydF4y2B一个41gydF4y2B一个,茎长(gydF4y2B一个勒gydF4y2B一个/gydF4y2B一个勒gydF4y2B一个)gydF4y2B一个42gydF4y2B一个子叶颜色(gydF4y2B一个我gydF4y2B一个/gydF4y2B一个我gydF4y2B一个)gydF4y2B一个65gydF4y2B一个,以及种皮和花的颜色(gydF4y2B一个一个gydF4y2B一个/gydF4y2B一个一个gydF4y2B一个)gydF4y2B一个66gydF4y2B一个,已进行功能分析,而其他三个孟德尔性状的基因身份,包括荚果颜色(gydF4y2B一个全科医生gydF4y2B一个/gydF4y2B一个全科医生gydF4y2B一个),豆荚形态(gydF4y2B一个VgydF4y2B一个/gydF4y2B一个vgydF4y2B一个)和花位(gydF4y2B一个足总gydF4y2B一个/gydF4y2B一个足总gydF4y2B一个)尚未探索gydF4y2B一个29gydF4y2B一个,gydF4y2B一个30.gydF4y2B一个.利用参考基因组PeaZW6,对4个克隆的Mendel’s基因进行了精确定位。gydF4y2B一个1gydF4y2B一个及补充资料gydF4y2B一个12gydF4y2B一个).有趣的是,三个基因显示出与之前研究中发现的相同的突变等位基因gydF4y2B一个rgydF4y2B一个ZW6基因(gydF4y2B一个Psat03G0136800gydF4y2B一个),在第22外显子插入9 bp,而不是插入0.8 kbgydF4y2B一个41gydF4y2B一个导致种子发生核状转变,而不是种子起皱(补充数据)gydF4y2B一个12gydF4y2B一个).与此同时,QTL分析使两个孟德尔基因得以重新发现,gydF4y2B一个rgydF4y2B一个而且gydF4y2B一个勒gydF4y2B一个,以及候选人gydF4y2B一个vgydF4y2B一个3个主要qtl(图;gydF4y2B一个5gydF4y2B一个、补充数据gydF4y2B一个8gydF4y2B一个- - - - - -gydF4y2B一个12gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).gydF4y2B一个
一些研究强调了泛基因组的必要性,以充分了解一个物种的基因组复杂性gydF4y2B一个18gydF4y2B一个,gydF4y2B一个20.gydF4y2B一个,gydF4y2B一个21gydF4y2B一个,gydF4y2B一个67gydF4y2B一个.单个基因组可能包含塑造独特特征的独特基因,而许多基因组之间共享的核心基因可能解释是什么塑造了一个物种gydF4y2B一个16gydF4y2B一个,gydF4y2B一个19gydF4y2B一个,gydF4y2B一个22gydF4y2B一个,gydF4y2B一个67gydF4y2B一个.由于NGS的技术限制,118个接入的初始组装是碎片化和不完整的。为了克服这个问题,我们引入了一种策略,将两种不同的基于算法的程序集与引用引导的脚手架结合起来,以改进各个程序集。通过高质量的PeaZW6参考,从头组装的完整性有了明显的提高(补充数据gydF4y2B一个13gydF4y2B一个).在我们的泛基因组分析中,我们还结合使用了从头和基于map-to-pan的策略来发现PAV。这种方法使我们能够尽可能多地使用NGS重测序数据来了解豌豆的泛基因组(补充数据gydF4y2B一个18gydF4y2B一个).核心、软核壳和云基因的百分比和功能富集与前人研究一致或相当gydF4y2B一个16gydF4y2B一个,gydF4y2B一个21gydF4y2B一个,gydF4y2B一个22gydF4y2B一个,gydF4y2B一个37gydF4y2B一个,证实了我们改进策略的可行性。总体而言,泛基因组分析揭示了豌豆基因组保守和发散部分的位置,增强了我们对豌豆基因组多样性和潜在价值的认识。然而,基于ngs的数据,泛基因组分析是相当有限的。例如,基于图的基因组的泛基因组长度比合并和增强的基因组要小得多(补充数据gydF4y2B一个15gydF4y2B一个),表明许多SVs在图中没有被识别。这种限制有望通过更多基于长读取的单独程序集得到改进。gydF4y2B一个
综上所述,本文提供的高质量参考基因组和泛基因组为豌豆基因组进化和驯化提供了新的视角,为豌豆遗传育种研究提供了宝贵的基因组资源gydF4y2B一个22gydF4y2B一个,gydF4y2B一个37gydF4y2B一个.这项研究将填补以前的基本模型和现代基因组学之间的空白,以促进豌豆的研究和作物改良。gydF4y2B一个
方法gydF4y2B一个
取样和基因组测序gydF4y2B一个
中国国家基因库豌豆品种中万6号(ZW6) G0005527经三代单籽遗传纯化。采用ZW6幼叶进行基因组DNA提取。使用Illumina NovaSeq 6000或Illumina HiSeq X Ten测序平台(Illumina)共生成1031.25 Gb NGS数据。同时使用PacBio Sequel平台(Pacific Biosciences) 379.34 Gb SMRT测序数据进行组装分析。gydF4y2B一个
基因组大小估计gydF4y2B一个
通过流式细胞术估计ZW6的基因组大小gydF4y2B一个68gydF4y2B一个.样品置于500 μl核萃取缓冲液中,用锋利的刀片切碎,60 s后用50 μm过滤器过滤。每个样品收集5000个细胞,加入2000 μl RNase染色缓冲液,黑暗处理30分钟。采用CyFlow Space Flow Cytometer (Sysmex Partec)和FloMax (v2.3)软件对细胞核悬液进行分析。gydF4y2B一个2gydF4y2B一个).K-mer方法使用JellyFish (v2.3.0)gydF4y2B一个69gydF4y2B一个(gydF4y2B一个KgydF4y2B一个= 21)使用~800 Gb Illumina测序数据(~187×)获得不同K-mers的频率分布。根据分布,GCE (gydF4y2B一个ftp://ftp.genomics.org.cn/pub/gcegydF4y2B一个)用于估计基因组大小、杂合子比例和重复序列百分比(补充图;gydF4y2B一个3.gydF4y2B一个).gydF4y2B一个
10x基因组文库构建和测序gydF4y2B一个
在10x Genomics测序中,根据Genome Reagent Kit Protocol (10x Genomics)提取高分子量基因组DNA,进行索引和条形码。然后制备文库,用HiSeq 2500 (Illumina)进行测序。gydF4y2B一个
Bionano测序gydF4y2B一个
根据Bionano Prep植物组织DNA分离规程,从幼苗叶片中提取高分子量DNA。然后模拟酶消化,选择内切酶DLE1进行消化。标记和染色过程根据Bionano Prep直接标记和染色(DLS)协议进行。使用Bionano Saphyr芯片(Bionano Genomics)进行测序。gydF4y2B一个
Hi-C实验和测序gydF4y2B一个
新鲜叶片用甲醛固定,过滤去核。提取的染色质用gydF4y2B一个后gydF4y2B一个III限制性内切酶(New England Biolab),构建4个Hi-C文库(gydF4y2B一个补充笔记gydF4y2B一个)gydF4y2B一个70gydF4y2B一个.经过质量控制后,Hi-C文库在Illumina HiSeq X Ten测序仪上测序。gydF4y2B一个
RNA-seq和公共数据收集gydF4y2B一个
2014年,在中国农业科学院北京作物科学研究所昌平实验站自然条件下的温室中,种植了10颗ZW6种子。在开花和结荚期收获根、叶、卷须、茎、花、花芽、绿色荚果和未成熟种子等8种组织,并立即置于液氮中,在−80°C保存。使用基于trizol的RNA提取试剂盒(Novogene)提取每个组织样本的总RNA。随后使用Kapa转录组试剂盒进行mRNA提取和mRNA-seq文库,并使用Illumina HiSeq 2000平台进行测序。8个RNA-seq文库共生成32.1 Gb的配对端读,并保存在NCBI BioProject中gydF4y2B一个PRJNA730094gydF4y2B一个.公共RNA-seq数据gydF4y2B一个PRJNA267198gydF4y2B一个,gydF4y2B一个PRJNA517587gydF4y2B一个,gydF4y2B一个PRJNA277074gydF4y2B一个而且gydF4y2B一个PRJNA328997gydF4y2B一个也用于转录组分析。gydF4y2B一个
基因组组装gydF4y2B一个
PacBio读取是使用Canu (v1.8)重新组装的gydF4y2B一个71gydF4y2B一个.使用Pilon (v1.23)对组装的contigs进行校正gydF4y2B一个72gydF4y2B一个.使用PurgeHaplotigs清除潜在的复制或单倍体contigs (v1.1.1)gydF4y2B一个73gydF4y2B一个.使用ARCS (v1.0.4),用10x基因组数据进一步构建纯化的contigs。gydF4y2B一个74gydF4y2B一个和LINKS (v1.8.6)gydF4y2B一个75gydF4y2B一个.然后使用带有DLE1标记光学图的Bionano Solve包(v3.4_06042019a)对10倍支架进行校正并将其提升至超级支架。然后使用Juicer (v1.5.6)将超级支架固定到染色体水平支架上。gydF4y2B一个76gydF4y2B一个和3d-dna管道(v180922)gydF4y2B一个77gydF4y2B一个使用JuiceBox Assembly Tools (JBAT) (v1.11.08)进行手动优化gydF4y2B一个78gydF4y2B一个.使用ALLMAPS (v1.0)评估Hi-C支架并将其固定在染色体上gydF4y2B一个79gydF4y2B一个用之前研究的基因标记gydF4y2B一个38gydF4y2B一个.使用BLAST (v2.5.0 +)从组装的contigs中人工恢复叶绿体基因组。gydF4y2B一个80gydF4y2B一个而且gydF4y2B一个NC_014057.1gydF4y2B一个参考RefSeq。使用BLAT手动恢复线粒体基因组(v34)gydF4y2B一个81gydF4y2B一个用NCBI中所有可用的线粒体基因作为种子,在组装的contigs中寻找候选。其他基本序列操作和统计使用SeqKit (v0.15.0)完成gydF4y2B一个82gydF4y2B一个.PeaZW6组件下载、浏览器和基本分析工具可在豌豆基因组数据库(gydF4y2B一个https://www.peagdb.com/gydF4y2B一个).看到gydF4y2B一个补充笔记gydF4y2B一个有关详细信息。gydF4y2B一个
基因组组装评估gydF4y2B一个
使用基准通用单拷贝Orthologs (BUSCO) (v5.0.0)评估ZW6和Caméor v1a组装的基因完整性。gydF4y2B一个83gydF4y2B一个.用merquury (v1.3)评价两组基因组的K-mer完整性和杂合度gydF4y2B一个84gydF4y2B一个.为了映射摘要和统计,使用BWA-MEM (v0.7.15)映射原始NGS读取gydF4y2B一个85gydF4y2B一个使用Minimap2 (v2.1)映射修正后的PacBio读数。gydF4y2B一个86gydF4y2B一个.使用LTR组装指数(LTR Assembly Index, LAI)评估重复基因组区域的质量。gydF4y2B一个87gydF4y2B一个(1) LTRharvest在GenomeTools (v1.6.0)gydF4y2B一个88gydF4y2B一个和LTR_FINDER (v1.0.7)gydF4y2B一个89gydF4y2B一个用于从头预测两个豌豆装配序列中的候选LTR-RTs(全长ltr逆转录转座子),以及(2)ltr_retrieval (v2.9.0)gydF4y2B一个90gydF4y2B一个然后用于组合和重构所有候选,以得到最终的全长ltr - rt。LAI的计算公式为:LAI = (LTR-RT完整长度/ LTR-RT总长度)× 100。看到gydF4y2B一个补充笔记gydF4y2B一个有关详细信息。gydF4y2B一个
基因组注释gydF4y2B一个
RepeatModeler和RepeatMasker (v4.1.1;gydF4y2B一个http://repeatmasker.org/gydF4y2B一个)通过识别PeaZW6组件中的重复族来构建zw6特定的重复库,并屏蔽PeaZW6组件中的重复序列。全长LTR-RT由LTR_FINDER_parallel (v1.0.7)识别gydF4y2B一个89gydF4y2B一个,gydF4y2B一个91gydF4y2B一个.gydF4y2B一个
蛋白质编码基因的注释使用从头算,同源和基于转录组的预测相结合。使用HISAT2 (v2.1.0)绘制了共71个RNA-seq文库,其中8个来自本研究,63个来自公共数据库。gydF4y2B一个92gydF4y2B一个,并且使用StringTie (v1.3.4)构建转录本gydF4y2B一个93gydF4y2B一个.构建的转录本使用TACO (v0.7.3)进行组合gydF4y2B一个94gydF4y2B一个.使用TransDecoder (v5.5.0)提取转录本上的开放阅读帧(orf)gydF4y2B一个95gydF4y2B一个.使用来自TransDecoder的完整orf作为BRAKER2管道从头算预测的训练集(v2.1.5)gydF4y2B一个96gydF4y2B一个.对于基于同源性的预测,使用genomic ethreader (v1.7.1)从近缘物种和已发表的豆类基因组中收集的蛋白质序列进行绘制。gydF4y2B一个97gydF4y2B一个.注释管道和工具箱Funannotate (v1.7.4) (gydF4y2B一个https://funannotate.readthedocs.io/en/latest/index.htmlgydF4y2B一个)gydF4y2B一个98gydF4y2B一个用于将不同的证据组合为初步注释集。采用多级策展工作流来减少潜在的错误预测。初步注释基因上的蛋白质结构域由HMMER识别(v3.3.1)gydF4y2B一个99gydF4y2B一个针对PFAM数据库(v31)gydF4y2B一个One hundred.gydF4y2B一个去除带有逆转录转座子结构域的基因。去除从头算证据提示的无表达或同源的单外显子基因。基于同源的搜索由BLASTP (v2.5.0 +)执行gydF4y2B一个80gydF4y2B一个针对UniProtKB / SwissProtgydF4y2B一个101gydF4y2B一个, NR和KEGGgydF4y2B一个102gydF4y2B一个数据库,和蛋白质从密切相关的物种和发表的豆类基因组,以删除基因没有同源性。最后,在Cufflinks (v0.11.6)中使用GFFRead去除帧移和部分基因gydF4y2B一个103gydF4y2B一个.使用InterProScan (v5.0)执行功能注释gydF4y2B一个104gydF4y2B一个和eggNOG-mapper (v2.1.6)gydF4y2B一个105gydF4y2B一个根据同源性确定它们的潜在功能。此外,还使用BLASTP (v2.5.0 +)搜索NR和KEGG数据库,进行标注率和其他交叉检查。统计学中使用的基因长度定义为起始密码子和终止密码子之间的染色体距离。对于叶绿体和线粒体,使用遗传密码11进行从头预测和ORF提取。看到gydF4y2B一个补充笔记gydF4y2B一个有关详细信息。gydF4y2B一个
基因表达分析gydF4y2B一个
原始RNA-seq读取使用Trimmomatic(v0.39)进行质量控制gydF4y2B一个106gydF4y2B一个FastQC (v 0.11.9)gydF4y2B一个107gydF4y2B一个.在HISAT2 (v2.1.0)基因注释模型的引导下,修剪后的reads被映射到最终染色体水平的PeaZW6组装。gydF4y2B一个92gydF4y2B一个.用StringTie (v1.3.4)检测各基因的表达水平gydF4y2B一个93gydF4y2B一个.gydF4y2B一个
比较基因组分析gydF4y2B一个
为了尽量减少同源基因对Medicago/PeaZW6和Medicago/PeaCaméor同位基因块检测的影响,MCScanXgydF4y2B一个108gydF4y2B一个用于识别PeaZW6/Medicago和PeaCaméor/Medicago的共联区,使用对MedtrA17_4.0gydF4y2B一个109gydF4y2B一个.简单地说,一个基因组中的所有蛋白质都是在另一个基因组的蛋白质数据库中进行BLASTP搜索的,反之亦然。E值阈值为1 × 10gydF4y2B一个−10gydF4y2B一个.如果两个蛋白是彼此最好的BLASTP命中,就可以确定同源性。由于MCScanX中的两个参数(' s '和' m ')对于检测到的同形块数量和同形块内同源基因的数量很重要,我们运行MCScanX,使用不同的' s '和' m '组合,分别计算同形块的数量和所含基因的数量。OrthoFinder (v2.5.4)gydF4y2B一个110gydF4y2B一个用于基因家族构建,选择最长的蛋白代表具有多个转录本的位点。gydF4y2B一个
snp, indes和sv的重测序和鉴定gydF4y2B一个
76份资料的5粒种子,代表不同的分类群gydF4y2B一个PisumgydF4y2B一个50gydF4y2B一个于2020年在中国农业科学院北京作物科学研究所自然条件温室栽培。每次获取一株植物的新鲜叶片,提取基因组DNA,并使用Illumina NovaSeq 6000测序平台(Illumina)重新测序。共生成6.2 T 150 bp配对端Illumina reads,平均覆盖率14.98× /次(补充数据)gydF4y2B一个2gydF4y2B一个).此外,还发表了42份的重测序数据gydF4y2B一个PisumgydF4y2B一个在以前的研究中使用的被包括在变异呼叫和群体遗传分析中gydF4y2B一个34gydF4y2B一个.gydF4y2B一个
使用Trimmomatic删除了适配器和低质量的原始读取序列gydF4y2B一个106gydF4y2B一个,利用BWA-MEM (v0.7.15)将干净的reads定位到ZW6的参考基因组。gydF4y2B一个85gydF4y2B一个.SNP调用使用Genome Analysis Toolkit 4 (GATK4,gydF4y2B一个https://gatk.broadinstitute.orggydF4y2B一个),使用默认参数。原始snp和indel首先用GATK推荐的变体过滤过滤,然后使用VCFtools过滤(v0.1.15)gydF4y2B一个111gydF4y2B一个(gydF4y2B一个补充笔记gydF4y2B一个).使用snpEff 4.3t注释变量gydF4y2B一个112gydF4y2B一个基于PeaZW6基因组注释。gydF4y2B一个
sv是用Delly (v0.8.3)识别的gydF4y2B一个113gydF4y2B一个使用重测序数据的BAM格式映射结果。首先,在每个个体上从头开始运行SV调用,然后将结果合并到一个VCF文件中作为指导参考。其次,在合并后的VCF文件的指导下重新运行SV调用。接下来,保留过滤中带有PASS标签的sv进行进一步分析。最后将所有品种的sv与BCFtools (v1.8)进行组合gydF4y2B一个114gydF4y2B一个使用VCFtools进行过滤(v0.1.15)gydF4y2B一个111gydF4y2B一个(gydF4y2B一个补充笔记gydF4y2B一个).gydF4y2B一个
PisumgydF4y2B一个群体遗传分析gydF4y2B一个
最后生成一个包含118个样本的SNP数据集,用于系统发育分析和其他群体遗传分析。系统发育树使用FastTree (v2.1.10)构建gydF4y2B一个115gydF4y2B一个使用GTR模型,并用FigTree (v1.4.3)进行可视化(gydF4y2B一个http://tree.bio.ed.ac.uk/software/figtree/gydF4y2B一个).采用ADMIXTRUE (v1.3.0)对群体遗传结构进行了研究gydF4y2B一个116gydF4y2B一个和簇号gydF4y2B一个KgydF4y2B一个取值范围为1 ~ 10。的gydF4y2B一个KgydF4y2B一个假设CV误差最小的值为最佳聚类gydF4y2B一个问gydF4y2B一个在进一步的遗传多样性、遗传分化和选择分析中,将每个个体的初级遗传成分的值排除在60%以下。使用默认设置的PLINK (v1.90b4.6)执行主成分分析gydF4y2B一个117gydF4y2B一个.保留前三个特征向量,使用R (v3.6.0) (gydF4y2B一个https://www.r-project.org/gydF4y2B一个).对SNP数据集的相同群体遗传分析也使用SVs进行,包括缺失、插入和重复,而易位和倒置由于Illumina测序技术短读的潜在不确定性而被排除在外。gydF4y2B一个
核苷酸多样性(π)和gydF4y2B一个FgydF4y2B一个圣gydF4y2B一个使用VCFtools (v0.1.15),根据admix的最佳聚类结果计算各组gydF4y2B一个111gydF4y2B一个窗口为1,000 kb,步长为100 kb。gydF4y2B一个
LD用PopLDdecay估计gydF4y2B一个118gydF4y2B一个管道与默认参数不同的物种gydF4y2B一个PisumgydF4y2B一个还有子组gydF4y2B一个p .一gydF4y2B一个基于SNP数据集的群体遗传结构结果。gydF4y2B一个
基因组扫描选择性信号gydF4y2B一个
我们使用更新的Python版本的跨种群复合似然比方法(XP-CLR)进行了基因组扫描。gydF4y2B一个40gydF4y2B一个发布于gydF4y2B一个https://github.com/hardingnj/xpclrgydF4y2B一个.在物种分化过程中,跨越基因组的选择信号gydF4y2B一个PisumgydF4y2B一个被分为两组进行评估:gydF4y2B一个p . fulvumgydF4y2B一个与gydF4y2B一个p . abyssinicumgydF4y2B一个而且gydF4y2B一个p . fulvumgydF4y2B一个与gydF4y2B一个p .一gydF4y2B一个.基因组扫描是用1,000 kb的滑动窗口完成的,整个基因组的步长为100 kb。在每个窗口中测定的snp的最大数量固定为600。对XP-CLR值进行归一化处理,将最高值前5%以上的区域视为选择性区域。此外,多样性减少前50%的选择区域(根据人工种群与野生种群的π比计算)被认为是候选选择区域。最后,使用bedtools将相邻的选择性区域合并为选择性扫描(v2.30.0)gydF4y2Ba119gydF4y2B一个.用R包CMplot (gydF4y2B一个https://github.com/YinLiLin/CMplotgydF4y2B一个).gydF4y2B一个
遗传连锁图谱构建及QTL定位gydF4y2B一个
双亲群体由300个F组成gydF4y2B一个2gydF4y2B一个这些个体来自WJ(雌性)和ZW6(雄性)的杂交,并于2017年在中国北京自然条件下的温室中生长。研究了18个农艺性状,包括15个数量性状和3个质量性状gydF4y2B一个6gydF4y2B一个而且gydF4y2B一个补充笔记gydF4y2B一个).采用SPSS 16.0软件对各性状进行相关分析。gydF4y2B一个
来自FgydF4y2B一个2gydF4y2B一个由Novogene (Novogene生物信息研究所,北京,中国)通过基因测序分型进行基因分型。使用BWA-MEM (v0.7.15)将共805.58 Gb 150-bp对端Illumina清洁reads定位到PeaZW6参考基因组。gydF4y2B一个85gydF4y2B一个.SNP呼叫使用GATK 4 (gydF4y2B一个https://gatk.broadinstitute.orggydF4y2B一个),使用默认参数。原始snp首先用GATK推荐的变体过滤,然后用VCFtools过滤(v0.1.15)gydF4y2B一个111gydF4y2B一个.在Tassel中使用Perl脚本run_piline .pl将最终VCF文件转换为abh格式的映射数据文件(v 5.2.40)gydF4y2B一个120gydF4y2B一个筛选合适的标记,利用R/qtl构建遗传连锁图谱gydF4y2B一个121gydF4y2B一个.SNPbinnergydF4y2B一个122gydF4y2B一个用于计算断点和构建基因型箱(gydF4y2B一个补充笔记gydF4y2B一个).利用R/qtl的Kosambi图谱功能,利用bin标记构建遗传连锁图谱gydF4y2B一个121gydF4y2B一个.QTL分析采用区间映射法进行R/ QTL分析gydF4y2B一个121gydF4y2B一个.显著性阈值(gydF4y2B一个αgydF4y2B一个= 0.05和gydF4y2B一个αgydF4y2B一个= 0.01),通过1000个排列来估计gydF4y2B一个123gydF4y2B一个对于每一个特质。采用单个QTL模型和多个QTL模型对LOD值高于阈值的QTL进行识别,并确定每个性状的最佳拟合QTL模型。遗传图谱和QTL分析结果采用R包LinkageMapView进行可视化gydF4y2B一个124gydF4y2B一个和CMplot (gydF4y2B一个https://github.com/YinLiLin/CMplotgydF4y2B一个).gydF4y2B一个
已鉴定孟德尔基因的图谱gydF4y2B一个
从以前的参考文献中搜索了四个确定的孟德尔基因gydF4y2B一个29gydF4y2B一个,gydF4y2B一个30.gydF4y2B一个.圆形种子蛋白ID为CAA56319.1(参考文献)。gydF4y2B一个41gydF4y2B一个).高性状的蛋白ID为AAC49792.1(参考文献)。gydF4y2B一个42gydF4y2B一个).彩色种皮和非彩色花的蛋白id分别为ADO13282.1和ADO13283.1gydF4y2B一个66gydF4y2B一个.黄色子叶和绿色子叶的蛋白id分别为BAF76351.1和BAF76352.1gydF4y2B一个65gydF4y2B一个.BLASTP工具与高信心(1egydF4y2B一个−6gydF4y2B一个)用于在参考基因PeaZW6中定位4个已鉴定的孟德尔氏基因。gydF4y2B一个
PisumgydF4y2B一个泛基因组组装、注释和PAV分析gydF4y2B一个
每个接入都是使用基于dbg的MEGAHIT (v1.2.9)重新测序数据重新组装的gydF4y2B一个125gydF4y2B一个和基于olc的MaSuRCA (v3.4.0)gydF4y2B一个126gydF4y2B一个独立。这两个程序集使用CD-HIT (v4.8.1)进行合并gydF4y2B一个127gydF4y2B一个并使用RagTag锚定到PeaZW6引用(v2.0.1)gydF4y2B一个128gydF4y2B一个类似于全景管道gydF4y2B一个129gydF4y2B一个.使用BUSCO(补充数据)对118个组件的质量进行评估gydF4y2B一个13gydF4y2B一个),以排除缺乏(gydF4y2B一个CgydF4y2B一个< 90%)的BUSCO完整性。gydF4y2B一个
使用MUMmer (v4.0)将来自每个组件的contig对齐到PeaZW6参考gydF4y2B一个130gydF4y2B一个.将contigs对齐段(同源性≥90%,长度≥100 bp)修剪掉。保留的序列被认为是PeaZW6基因组的附加序列(补充数据gydF4y2B一个14gydF4y2B一个).为了消除装配间冗余,使用了一种vg和类似模型的“增强”策略。从PeaZW6参考开始,我们迭代对齐每个基因组,并将额外的序列添加到先前的增强参考中,作为下一轮的新参考。同时,基于图的泛基因组也使用minigraph从所有组件生成(v0.13)gydF4y2B一个131gydF4y2B一个使用参数-l 500 -d 500,统计数据由gfatools(v0.5)报告gydF4y2B一个131gydF4y2B一个.这一工作流程也重复了所有遗传组(补充数据gydF4y2B一个15gydF4y2B一个).gydF4y2B一个
在使用RepeatMasker软屏蔽重复序列后,BRAKER2管道gydF4y2B一个96gydF4y2B一个采用PeaZW6模型,以PeaZW6、PeaCaméor和SwissProt数据库中的蛋白序列为线索,预测每个基因组上的基因。使用CD-HIT (v4.8.1)对预测的蛋白质序列进行聚类gydF4y2B一个127gydF4y2B一个去除重复的基因。去除重复元件重叠(长度≥50%)的基因。此外,使用hmm (v3.3.1)将基因与PFAM数据库进行比对。gydF4y2B一个99gydF4y2B一个UniRef90数据库使用BLASTP (v2.5.0 +)过滤出靶序列长度覆盖<50%的片段基因。最后,利用BLASTP (v2.5.0 +)将保留的基因与PeaZW6基因进行比对,以确定是否为附加基因(补充数据)gydF4y2B一个14gydF4y2B一个).gydF4y2B一个
使用OrthoFinder (v2.5.4)对所有来源的蛋白质进行聚类gydF4y2B一个110gydF4y2B一个(−gydF4y2B一个ygydF4y2B一个能够将副同源基因分裂为不同的hog),并将其划分为系统发育hog作为泛基因的代表。我们进一步使用“map-to-pan”策略来恢复由于测序偏差或部分基因预测而在每次加入时错误遗漏的hog。以所有品系的完整基因序列为参考,利用Minimap2 (v2.1)对所有116个品系的原始基因序列进行测序。gydF4y2B一个86gydF4y2B一个并使用samtools限制NM≤1gydF4y2B一个132gydF4y2B一个.长度≥99%和覆盖深度≥3×的基因被认为存在于一个附加项中,其对应的hog被标记为存在于PAV表中。gydF4y2B一个
在确定了来自猪的116个基因组的PAV模式和map-to-pan(补充数据gydF4y2B一个18gydF4y2B一个),最终PAV模式以病房为单位聚类。hclust包中的D方法,并由R (v3.6.0)中的pheatmap包进行了说明。根据Roary的定义,根据共享的基因组百分比,将hog分为核心基因(≥99%的基因组)、软核基因(≥90%和<99%)、壳基因(≥15%和<90%)和云基因(<15%)gydF4y2B一个133gydF4y2B一个,适用于所有品系及遗传组别。每个组的唯一核心基因和唯一泛基因是通过去除至少两个组之间共享的基因来确定的。gydF4y2B一个
为了研究泛基因的功能,用cutree in R将聚成簇的泛基因切割成标记为A ~ H的8组(图。gydF4y2B一个6摄氏度gydF4y2B一个).由于hclust包中只有65,535列,因此使用randomForest包构建分类器,将112,776个hog重新分配到8个预构建组中,100次运行的曲线下平均面积达到0.98 (Supplementary Fig。gydF4y2B一个17gydF4y2B一个).使用EggNOG-mapper (v2.1.6)评估所有组的假定功能富集gydF4y2B一个105gydF4y2B一个基于EggNOG数据库(v5.0)gydF4y2B一个134gydF4y2B一个.GO富集分析采用AgriGO (v2.0)进行。gydF4y2B一个135gydF4y2B一个和TBtoolsgydF4y2B一个136gydF4y2B一个并使用R (v3.6.0)中的pheatmap包进行了说明。gydF4y2B一个
看到gydF4y2B一个补充笔记gydF4y2B一个有关详细信息。gydF4y2B一个
统计分析gydF4y2B一个
在氧化石墨烯富集分析中,采用单边Fisher精确检验gydF4y2B一个PgydF4y2B一个使用Benjamini-Hochberg方法调整值gydF4y2B一个137gydF4y2B一个.gydF4y2B一个
报告总结gydF4y2B一个
有关研究设计的进一步资料,请参阅gydF4y2B一个自然研究报告摘要gydF4y2B一个链接到这篇文章。gydF4y2B一个
数据可用性gydF4y2B一个
来自全基因组霰弹枪项目的数据gydF4y2B一个Pisum一gydF4y2B一个品种中万6号(PeaZW6)已存入DDBJ/ENA/GenBank,编号JAMSHJ000000000。所有原始测序数据和118个泛基因组组合已在生物计划下存放在NCBIgydF4y2B一个PRJNA730094gydF4y2B一个.PeaZW6组件(gydF4y2B一个https://doi.org/10.5281/zenodo.6622409gydF4y2B一个),以及118个泛基因组组合(gydF4y2B一个https://doi.org/10.5281/zenodo.6622578gydF4y2B一个)也可作为Zenodo数据集使用。PeaZW6组件以及基因组浏览器和基本分析工具也可在豌豆基因组数据库(gydF4y2B一个https://www.peagdb.com/gydF4y2B一个).gydF4y2B一个
代码的可用性gydF4y2B一个
用于PeaZW6基因组和泛基因组计划的自定义脚本已存放在ZenodogydF4y2B一个138gydF4y2B一个(gydF4y2B一个https://doi.org/10.5281/zenodo.6614849gydF4y2B一个).gydF4y2B一个
参考文献gydF4y2B一个
梅耶,R. S. & Purugganan, m.d.作物物种的进化:驯化和多样化的遗传学。gydF4y2B一个Nat. Rev. Genet。gydF4y2B一个14gydF4y2B一个, 840-852(2013)。gydF4y2B一个
李文杰,李文杰。植物适应与多样化研究。gydF4y2B一个前面。工厂。科学。gydF4y2B一个4gydF4y2B一个, 290(2013)。gydF4y2B一个
Bevan, m.w.等人。作物改良的基因组创新。gydF4y2B一个自然gydF4y2B一个543gydF4y2B一个, 346-354(2017)。gydF4y2B一个
袁勇,拜耳,彭培,巴特利,爱德华,D.基因组技术的改进:在作物基因组学中的应用。gydF4y2B一个生物科技趋势》。gydF4y2B一个35gydF4y2B一个, 547-558(2017)。gydF4y2B一个
爱德华兹,D,巴特利,J. &斯诺登,R. J.利用新一代测序技术获取复杂的作物基因组。gydF4y2B一个定理。达成。麝猫。gydF4y2B一个126gydF4y2B一个, 1-11(2013)。gydF4y2B一个
焦,Y.等。单分子技术改良玉米参考基因组。gydF4y2B一个自然gydF4y2B一个546gydF4y2B一个, 524-527(2017)。gydF4y2B一个
周哲,等。对302份野生和栽培大豆种质进行重测序,鉴定出与大豆驯化改良相关的基因。gydF4y2B一个生物科技Nat。》。gydF4y2B一个33gydF4y2B一个, 408-414(2015)。gydF4y2B一个
Varshney, R. K.等。292份鸽豌豆种质的全基因组重测序确定了与驯化和农艺性状相关的基因组区域。gydF4y2B一个Nat,麝猫。gydF4y2B一个49gydF4y2B一个, 1082-1088(2017)。gydF4y2B一个
王,w.s.,等。亚洲栽培水稻3,010个不同种质的基因组变异。gydF4y2B一个自然gydF4y2B一个557gydF4y2B一个, 43-49(2018)。gydF4y2B一个
魏,T.等。445的全基因组重测序gydF4y2B一个摘要以gydF4y2B一个资料揭示了栽培莴苣的驯化历史。gydF4y2B一个Nat,麝猫。gydF4y2B一个53gydF4y2B一个, 752-760(2021)。gydF4y2B一个
吴,J.等。对683种普通大豆基因型进行重测序,确定了南北斜向的产量组成性状相关性。gydF4y2B一个Nat,麝猫。gydF4y2B一个52gydF4y2B一个, 118-125(2020)。gydF4y2B一个
Feuk, L., Marshall, C. R., Wintle, R. F. & Scherer, S. W.结构变异:改变染色体的景观和疾病研究设计。gydF4y2B一个嗡嗡声。摩尔,麝猫。gydF4y2B一个15gydF4y2B一个, r57-r66(2006)。gydF4y2B一个
王毅,等。拷贝数变化gydF4y2B一个GL7gydF4y2B一个遗传位点对水稻籽粒大小多样性有贡献。gydF4y2B一个Nat,麝猫。gydF4y2B一个47gydF4y2B一个, 944-948(2015)。gydF4y2B一个
Alonge, M.等人。广泛结构变异对番茄基因表达和作物改良的重大影响。gydF4y2B一个细胞gydF4y2B一个182gydF4y2B一个, 145-161(2020)。gydF4y2B一个
Kou, Y.等。亚洲水稻结构变异的进化基因组学(gydF4y2B一个栽培稻gydF4y2B一个)驯化。gydF4y2B一个摩尔。杂志。另一个星球。gydF4y2B一个37gydF4y2B一个, 3507-3524(2020)。gydF4y2B一个
刘,Y.等。野生和栽培大豆的泛基因组。gydF4y2B一个细胞gydF4y2B一个182gydF4y2B一个, 162-176(2020)。gydF4y2B一个
周,Y.等。葡萄驯化中结构变异的群体遗传学研究。gydF4y2B一个Nat。植物gydF4y2B一个5gydF4y2B一个, 965-979(2019)。gydF4y2B一个
汗,A. W.等人。通过整合一个物种的野生侧来加速作物改良的超级泛基因组。gydF4y2B一个植物科学gydF4y2B一个25gydF4y2B一个, 148-158(2020)。gydF4y2B一个
泰特林,H.等人。多株病原菌的基因组分析gydF4y2B一个链球菌agalactiaegydF4y2B一个:微生物“泛基因组”的含义。gydF4y2B一个国家科学院学报美国gydF4y2B一个102gydF4y2B一个, 13950-13955(2005)。gydF4y2B一个
高利兹,A. A.巴特利,J. &爱德华兹,D.植物泛基因组学。gydF4y2B一个生物科技植物》。J。gydF4y2B一个14gydF4y2B一个, 1099-1105(2016)。gydF4y2B一个
Golicz, a.a., Bayer, p.e., Bhalla, p.l., Batley, J. & Edwards, D.泛基因组学成熟:从细菌到植物和动物应用。gydF4y2B一个植物科学gydF4y2B一个36gydF4y2B一个, 132-145(2020)。gydF4y2B一个
高,L.等。番茄泛基因组揭示了新的基因和一种罕见的调节水果味道的等位基因。gydF4y2B一个Nat,麝猫。gydF4y2B一个51gydF4y2B一个, 1044-1051(2019)。gydF4y2B一个
Dolezel, J. & Greilhuber, J.核基因组大小:我们正在接近吗?gydF4y2B一个血细胞计数一次gydF4y2B一个77gydF4y2B一个, 635-642(2010)。gydF4y2B一个
Smýkal, P.等。豌豆(gydF4y2B一个Pisum一gydF4y2B一个L.)在基因组时代。gydF4y2B一个农学gydF4y2B一个2gydF4y2B一个, 74-115(2012)。gydF4y2B一个
Tayeh, N.等人。豌豆育种计划中的基因组工具:现状与展望。gydF4y2B一个前面。植物科学。gydF4y2B一个6gydF4y2B一个, 1037(2015)。gydF4y2B一个
豆科植物的碳水化合物部分:在人体营养中的用途和健康潜力。gydF4y2B一个Br。j .减轻。gydF4y2B一个88gydF4y2B一个, s293-s306(2002)。gydF4y2B一个
达尔,W. J.,福斯特,L. M. &泰勒,R. T.豌豆健康益处综述(gydF4y2B一个Pisum一gydF4y2B一个l .)。gydF4y2B一个Br。j .减轻。gydF4y2B一个108gydF4y2B一个, s3-s10(2012)。gydF4y2B一个
MacWilliam, S. Wismer, M. & Kulshreshtha, S.加拿大西部脉冲系统的生命周期和经济评估:在作物轮作中包含脉冲。gydF4y2B一个Agr。系统。gydF4y2B一个123gydF4y2B一个, 43-53(2014)。gydF4y2B一个
埃利斯,T. H.,霍费尔,J. M.,蒂默曼-沃恩,G. M.,科因,C. J.和海伦斯,R. P.孟德尔,150年。gydF4y2B一个植物科学gydF4y2B一个16gydF4y2B一个, 590-596(2011)。gydF4y2B一个
里德,J. B.和罗斯,J. J.孟德尔的基因:朝着完整的分子特征。gydF4y2B一个遗传学gydF4y2B一个189gydF4y2B一个, 3-10(2011)。gydF4y2B一个
Zohary, D. & Hopf, M.旧大陆豆类的驯化:当近东农业开始时,豆类是小麦和大麦的伙伴。gydF4y2B一个科学gydF4y2B一个182gydF4y2B一个, 887-894(1973)。gydF4y2B一个
Smýkal, P.等。植物的系统发育、系统地理学和遗传多样性gydF4y2B一个PisumgydF4y2B一个属。gydF4y2B一个植物麝猫。Resour。gydF4y2B一个9gydF4y2B一个, 4-18 (2010)gydF4y2B一个
Smýkal, P.等。用于科学和育种的豆类作物系统发育和遗传多样性。gydF4y2B一个暴击。植物科学gydF4y2B一个34gydF4y2B一个, 43-104(2015)。gydF4y2B一个
Kreplak, J.等人。豌豆的参考基因组为豆科植物的基因组进化提供了见解。gydF4y2B一个Nat,麝猫。gydF4y2B一个51gydF4y2B一个, 1411-1422(2019)。gydF4y2B一个
罗伯茨,R. J.,卡内罗,M. O. &沙茨,M. C. SMRT测序的优势。gydF4y2B一个基因组医学杂志。gydF4y2B一个14gydF4y2B一个, 405(2013)。gydF4y2B一个
柴森,m.j.p.等。利用单分子测序解决人类基因组的复杂性。gydF4y2B一个自然gydF4y2B一个517gydF4y2B一个, 608-611(2015)。gydF4y2B一个
孙,x等。阶段性的二倍体基因组组装和泛基因组为了解苹果驯化的遗传历史提供了依据。gydF4y2B一个Nat,麝猫。gydF4y2B一个52gydF4y2B一个, 1423-1432(2020)。gydF4y2B一个
Tayeh, N.等人。开发了两大豌豆基因组学资源:GenoPea 13.2K SNP阵列和高密度、高分辨率共识遗传图谱。gydF4y2B一个植物J。gydF4y2B一个84gydF4y2B一个, 1257-1273(2015)。gydF4y2B一个
赫福德,m.b.等人。玉米驯化改良的比较群体基因组学研究。gydF4y2B一个Nat,麝猫。gydF4y2B一个44gydF4y2B一个, 808-811(2012)。gydF4y2B一个
陈,H.,帕特森,N.和赖希,D.种群分化作为选择性扫描的测试。gydF4y2B一个基因组Res。gydF4y2B一个20.gydF4y2B一个, 393-402(2010)。gydF4y2B一个
巴塔查里亚,M. K.,史密斯,a . M.,埃利斯,T. H.,赫德利,C.和马丁,C.孟德尔所描述的豌豆褶皱种子特性是由编码淀粉分支酶的基因中一个类似转座子的插入引起的。gydF4y2B一个细胞gydF4y2B一个60gydF4y2B一个, 115-122(1990)。gydF4y2B一个
马丁,D. N., Proebsting, W. M. & heden, P. Mendel的矮化基因:来自gydF4y2B一个勒gydF4y2B一个表达蛋白的等位基因及其功能。gydF4y2B一个国家科学院学报美国gydF4y2B一个94gydF4y2B一个, 8907-8911(1997)。gydF4y2B一个
鲍尔斯,S. E.和塔瓦拉贾,D.检查农业的脉搏:田间豌豆(gydF4y2B一个Pisum一gydF4y2B一个L.)、可持续性和磷利用效率。gydF4y2B一个前面。植物科学。gydF4y2B一个10gydF4y2B一个, 1489(2019)。gydF4y2B一个
科因,C. J.等。豌豆、扁豆和鹰嘴豆野生亲缘改良的潜力和局限性。gydF4y2B一个豆类科学。gydF4y2B一个2gydF4y2B一个, e36(2020)。gydF4y2B一个
Smýkal, P.等。从孟德尔对豌豆的发现到今天的植物遗传学和育种。gydF4y2B一个定理。达成。麝猫。gydF4y2B一个129gydF4y2B一个, 2267-2280(2016)。gydF4y2B一个
叶春英,范丽丽。基因组时代的孤儿作物及其野生亲缘。gydF4y2B一个摩尔。植物gydF4y2B一个14gydF4y2B一个, 27-39(2021)。gydF4y2B一个
莫雷尔,P. L.,巴克勒,E. S. &罗斯-伊巴拉,J.作物基因组学:进展和应用。gydF4y2B一个Nat. Rev. Genet。gydF4y2B一个13gydF4y2B一个, 85-96(2012)。gydF4y2B一个
潘迪,a.k.等人。组学资源和组学支持的方法,以实现高生产力和提高豌豆质量(gydF4y2B一个Pisum一gydF4y2B一个l .)。gydF4y2B一个定理。达成。麝猫。gydF4y2B一个134gydF4y2B一个, 755-776(2021)。gydF4y2B一个
Zong, x.x等。分析一个多元的全球gydF4y2B一个PisumgydF4y2B一个sp集合和与中国本地的比较gydF4y2B一个p .一gydF4y2B一个用微卫星标记收集。gydF4y2B一个定理。达成。麝猫。gydF4y2B一个118gydF4y2B一个, 193-204(2009)。gydF4y2B一个
刘,R.等。栽培与野生豌豆群体遗传结构及分类(gydF4y2B一个PisumgydF4y2B一个sp.)基于形态性状和SSR标记。gydF4y2B一个j .系统。另一个星球。gydF4y2B一个60gydF4y2B一个, 85-100(2022)。gydF4y2B一个
马思德;安布罗斯gydF4y2B一个地中海豆科植物遗传资源gydF4y2B一个(eds Maxted, N. & Bennet, S. j) 181-190(施普林格,2001)。gydF4y2B一个
Kosterin, O. E. & Bogdanova, V. S.野生和栽培形式的关系gydF4y2B一个PisumgydF4y2B一个L.从质体、线粒体和核基因组三个标记的分析推断。gydF4y2B一个麝猫。Resour。另一个星球。gydF4y2B一个55gydF4y2B一个, 735-755(2008)。gydF4y2B一个
博格达诺娃,V. S.等。该属的隐晦的分化gydF4y2B一个PisumgydF4y2B一个L.(豌豆),质体基因组系统发育分析显示。gydF4y2B一个摩尔。Phylogenet。另一个星球。gydF4y2B一个129gydF4y2B一个, 280-290(2018)。gydF4y2B一个
阿比西尼亚豌豆(gydF4y2B一个Lathyrus schaeferigydF4y2B一个Kosterin职业gydF4y2B一个Pisum abyssinicumgydF4y2B一个a . Br.):一个有问题的分类单元。gydF4y2B一个学报杂志。有血缘关系的。gydF4y2B一个3.gydF4y2B一个, 97-110(2017)。gydF4y2B一个
豌豆的驯化(gydF4y2B一个Pisum一gydF4y2B一个l):以阿比西尼亚豌豆为例。gydF4y2B一个前面。植物科学。gydF4y2B一个9gydF4y2B一个, 515(2018)。gydF4y2B一个
Ben-Ze 'ev, N. & Zohary, D.属的种间关系gydF4y2B一个PisumgydF4y2B一个lgydF4y2B一个Isr。j .机器人。gydF4y2B一个22gydF4y2B一个, 73-91(1973)。gydF4y2B一个
赫尔维格,T., Abbo, S. & Ophir, R.系统发育和不同的选择特征表明两个基因独立的豌豆驯化事件(gydF4y2B一个PisumgydF4y2B一个l .)。gydF4y2B一个植物J。gydF4y2B一个110gydF4y2B一个, 419-439(2022)。gydF4y2B一个
Hradilová, I.等。豌豆荚裂和种子休眠两个关键驯化性状的转录组学、代谢组学和解剖学综合比较分析(gydF4y2B一个PisumgydF4y2B一个sp)。gydF4y2B一个前面。工厂。科学。gydF4y2B一个8gydF4y2B一个, 542(2017)。gydF4y2B一个
帕克,t.a., Lo, S. & Gepts, P.籽粒豆科植物的荚果破碎:新兴的遗传和环境相关模式。gydF4y2B一个植物细胞gydF4y2B一个33gydF4y2B一个, 179-199(2021)。gydF4y2B一个
张,M.等。近十年来大豆功能基因组学研究进展。gydF4y2B一个生物科技植物》。J。gydF4y2B一个20.gydF4y2B一个, 256-282(2022)。gydF4y2B一个
S.基因突变。gydF4y2B一个Pisum。阿格利司。长的矮。麝猫。gydF4y2B一个30.gydF4y2B一个, 1-293(1972)。gydF4y2B一个
韦登,N. F.,布劳内,S. & Przyborowski, J. A.豌豆荚果开裂的遗传分析(gydF4y2B一个Pisum一gydF4y2B一个l .)。gydF4y2B一个细胞。摩尔。杂志。列托人。gydF4y2B一个7gydF4y2B一个, 657-663(2002)。gydF4y2B一个
孙,L.等。gydF4y2B一个GmHs1-1gydF4y2B一个编码一种钙调神经磷酸酶样蛋白,控制大豆的硬籽性。gydF4y2B一个Nat,麝猫。gydF4y2B一个47gydF4y2B一个, 939-943(2015)。gydF4y2B一个
王,M.等。多科作物驯化过程中休眠基因的平行选择。gydF4y2B一个Nat,麝猫。gydF4y2B一个50gydF4y2B一个, 1435-1441(2018)。gydF4y2B一个
Sato, Y., Morita, R., Nishimura, M., Yamaguchi, H. & Kusaba, M. Mendel的绿色子叶基因编码叶绿素降解途径的正调控因子。gydF4y2B一个国家科学院学报美国gydF4y2B一个104gydF4y2B一个, 14169-14174(2007)。gydF4y2B一个
海伦斯,R. P.等。孟德尔白花性状鉴定。gydF4y2B一个《公共科学图书馆•综合》gydF4y2B一个5gydF4y2B一个, e13230(2010)。gydF4y2B一个
Varshney, R. K.等。基于3366个基因组测序的鹰嘴豆遗传变异图。gydF4y2B一个自然gydF4y2B一个599gydF4y2B一个, 622-627(2021)。gydF4y2B一个
Hare, E. E. & Johnston, J. S.利用流式细胞术测定碘化丙啶染色细胞核的基因组大小。gydF4y2B一个方法分子生物学。gydF4y2B一个772gydF4y2B一个, 3-12(2011)。gydF4y2B一个
Marçais, G. & Kingsford, C.一种快速,无锁的k-mers发生的有效并行计数方法。gydF4y2B一个生物信息学gydF4y2B一个27gydF4y2B一个, 764-770(2011)。gydF4y2B一个
王,M.等。棉花多倍体化后三维基因组结构的进化动态。gydF4y2B一个Nat。植物gydF4y2B一个4gydF4y2B一个, 90-97(2018)。gydF4y2B一个
柯伦,S.等人。Canu:通过自适应k-mer加权和重复分离可伸缩和精确的长读取组装。gydF4y2B一个基因组Res。gydF4y2B一个27gydF4y2B一个, 722-736(2017)。gydF4y2B一个
沃克,B. J.等。Pilon:综合微生物变异检测和基因组组装改进的集成工具。gydF4y2B一个《公共科学图书馆•综合》gydF4y2B一个9gydF4y2B一个, e112963(2014)。gydF4y2B一个
罗奇,M. J.,施密特,S. A. & Borneman, A. R.清除单倍体:第三代二倍体基因组组装的等位基因contig重分配。gydF4y2B一个BMC生物信息学gydF4y2B一个19gydF4y2B一个, 460(2018)。gydF4y2B一个
Yeo, S., Coombe, L., Warren, R. L., Chu, J. & Birol, I. ARCS:链接读取的脚手架基因组草图。gydF4y2B一个生物信息学gydF4y2B一个34gydF4y2B一个, 725-731(2018)。gydF4y2B一个
沃伦,R. L.等。链接:具有长读取的可伸缩的、无对齐的基因组草图脚手架。gydF4y2B一个GigaSciencegydF4y2B一个4gydF4y2B一个, 35(2015)。gydF4y2B一个
杜兰德,n.c.等人。Juicer提供了一键式系统,用于分析循环分辨率Hi-C实验。gydF4y2B一个细胞系统。gydF4y2B一个3.gydF4y2B一个, 95-98(2016)。gydF4y2B一个
杜琴科,O.等。从头组装的gydF4y2B一个埃及伊蚊gydF4y2B一个使用Hi-C的基因组产生染色体长度支架。gydF4y2B一个科学gydF4y2B一个356gydF4y2B一个, 92-95(2017)。gydF4y2B一个
罗宾逊,J. T.等。Juicebox.js为Hi-C数据提供了一个基于云的可视化系统。gydF4y2B一个细胞系统。gydF4y2B一个6gydF4y2B一个, 256-258(2018)。gydF4y2B一个
Tang, H.等。ALLMAPS:基于多个映射的健壮的脚手架排序。gydF4y2B一个基因组医学杂志。gydF4y2B一个16gydF4y2B一个, 3(2015)。gydF4y2B一个
阿特舒,S. F.,吉什,W.,米勒,W.,迈尔斯,E. W. &李普曼,D. J.基本的局部对齐搜索工具。gydF4y2B一个J. Mol.生物学。gydF4y2B一个215gydF4y2B一个, 403-410(1990)。gydF4y2B一个
Kent, W. J. blat -类似blast的对齐工具。gydF4y2B一个基因组Res。gydF4y2B一个12gydF4y2B一个, 656-664(2002)。gydF4y2B一个
沈玮,乐胜,李永和胡峰。SeqKit:一个跨平台的FASTA/Q文件操作工具包。gydF4y2B一个《公共科学图书馆•综合》gydF4y2B一个11gydF4y2B一个, e0163962(2016)。gydF4y2B一个
沃特豪斯,r.m.等。BUSCO从质量评估到基因预测和系统基因组学的应用。gydF4y2B一个摩尔。杂志。另一个星球。gydF4y2B一个35gydF4y2B一个, 543-548(2018)。gydF4y2B一个
rihie, A., Walenz, B. P., Koren, S. & Phillippy, A. M. merquury:基因组组装的无参考质量、完整性和分期评估。gydF4y2B一个基因组医学杂志。gydF4y2B一个21gydF4y2B一个, 245(2020)。gydF4y2B一个
李,H. & Durbin R.快速和准确的短读对齐与Burrows-Wheeler变换。gydF4y2B一个生物信息学gydF4y2B一个25gydF4y2B一个, 1754-1760(2009)。gydF4y2B一个
Minimap2:核苷酸序列的成对比对。gydF4y2B一个生物信息学gydF4y2B一个34gydF4y2B一个, 3094-3100(2018)。gydF4y2B一个
欧胜,陈娟,蒋宁。利用LTR组装指数(LAI)评估基因组组装质量。gydF4y2B一个核酸测定。gydF4y2B一个46gydF4y2B一个, e126(2018)。gydF4y2B一个
Ellinghaus, D., Kurtz, S. & Willhoeft, U. LTRharvest,一个高效和灵活的软件gydF4y2B一个新创gydF4y2B一个检测LTR逆转录转座子。gydF4y2B一个BMC生物信息学gydF4y2B一个9gydF4y2B一个, 18(2008)。gydF4y2B一个
徐卓,王宏。LTR_FINDER:预测全长LTR逆转录转座子的有效工具。gydF4y2B一个核酸测定。gydF4y2B一个35gydF4y2B一个, w265-w268(2007)。gydF4y2B一个
Ou, S. & Jiang, N. LTR_retriever:用于鉴定长末端重复逆转录转座子的高度准确和敏感的程序。gydF4y2B一个植物杂志。gydF4y2B一个176gydF4y2B一个, 1410-1422(2018)。gydF4y2B一个
Ou, S. & Jiang, N. LTR_FINDER_parallel: LTR_FINDER并行化,实现长端重复逆转录转座子的快速识别。gydF4y2B一个暴徒。DNAgydF4y2B一个10gydF4y2B一个, 48(2019)。gydF4y2B一个
Kim, D., Langmead, B. & Salzberg, S. L. HISAT:低内存需求的快速拼接对齐器。gydF4y2B一个Nat方法。gydF4y2B一个12gydF4y2B一个, 357-360(2015)。gydF4y2B一个
Pertea, M., Kim, D., Pertea, g.m., Leek, J. T. & Salzberg, S. L. RNA-seq实验的转录水平表达分析与HISAT, StringTie和Ballgown。gydF4y2B一个Protoc Nat。gydF4y2B一个11gydF4y2B一个, 1650-1667(2016)。gydF4y2B一个
Niknafs, Y. S, Pandian, B., Iyer, H. K., Chinnaiyan, A. M. & Lyer, M. K. TACO从RNA-seq中产生强大的多样本转录组组装。gydF4y2B一个Nat方法。gydF4y2B一个14gydF4y2B一个, 68-70(2017)。gydF4y2B一个
哈斯,B. J.等。改善gydF4y2B一个拟南芥gydF4y2B一个基因组注释使用最大转录本比对汇编。gydF4y2B一个核酸测定。gydF4y2B一个31gydF4y2B一个, 5654-5666(2003)。gydF4y2B一个
Hoff, K. J., Lomsadze, A., Borodovsky, M. & Stanke, M.全基因组注释与BRAKER。gydF4y2B一个方法分子生物学。gydF4y2B一个1962gydF4y2B一个, 65-95(2019)。gydF4y2B一个
格莱姆,G.,布伦德尔,V.,斯巴克斯,M. E.和库尔茨,S.工程软件工具的基因结构预测在高等生物。gydF4y2B一个正,Softw。抛光工艺。gydF4y2B一个47gydF4y2B一个, 965-978(2005)。gydF4y2B一个
Palmer, J. Funannotate:基因组注释管道(2016);gydF4y2B一个https://funannotate.readthedocs.io/en/latest/index.htmlgydF4y2B一个
Mistry, J., Finn, R. D., Eddy, S. R., Bateman, A. & Punta, M.同源搜索中的挑战:HMMER3和卷曲线圈区域的收敛演化。gydF4y2B一个核酸测定。gydF4y2B一个41gydF4y2B一个, e121(2013)。gydF4y2B一个
El-Gebali, S.等。2019年Pfam蛋白家族数据库。gydF4y2B一个核酸测定。gydF4y2B一个47gydF4y2B一个, d427-d432(2019)。gydF4y2B一个
T. U. UniProt:全球蛋白质知识中心。gydF4y2B一个核酸测定。gydF4y2B一个47gydF4y2B一个, d506-d515(2019)。gydF4y2B一个
绪方,H.等。KEGG:京都基因和基因组百科全书。gydF4y2B一个核酸测定。gydF4y2B一个27gydF4y2B一个, 29-34(1999)。gydF4y2B一个
Ghosh, S. & Chan, C. K.使用TopHat和袖扣分析RNA-seq数据。gydF4y2B一个方法分子生物学。gydF4y2B一个1374gydF4y2B一个, 339-361(2016)。gydF4y2B一个
琼斯等人。InterProScan 5:基因组级蛋白质功能分类。gydF4y2B一个生物信息学gydF4y2B一个30.gydF4y2B一个, 1236-1240(2014)。gydF4y2B一个
& Cantalapiedra, C.P.等人。eggNOG-mapper v2:宏基因组尺度上的功能注释、orthology赋值和域预测。gydF4y2B一个摩尔。杂志。另一个星球。gydF4y2B一个38gydF4y2B一个, 5825-5829(2021)。gydF4y2B一个
Bolger, a.m., Lohse, M. & Usadel, B. Trimmomatic:用于Illumina序列数据的灵活修剪器。gydF4y2B一个生物信息学gydF4y2B一个30.gydF4y2B一个, 2114-2120(2014)。gydF4y2B一个
Andrews, S. FastQC:高通量序列数据的质量控制工具(2010);gydF4y2B一个https://www.bioinformatics.babraham.ac.uk/projects/fastqc/gydF4y2B一个
王毅,等。MCScanX:一个用于检测和进化分析基因同步性和共线性的工具包。gydF4y2B一个核酸测定。gydF4y2B一个40gydF4y2B一个, e49(2012)。gydF4y2B一个
Tang, H.等。模型豆科植物Medicago truncatula的改良基因组释放(Mt4.0版本)。gydF4y2B一个BMC基因组学gydF4y2B一个15gydF4y2B一个, 312(2014)。gydF4y2B一个
Emms, D. M. & Kelly, S. OrthoFinder:比较基因组学的系统发育矫形学推断。gydF4y2B一个基因组医学杂志。gydF4y2B一个20.gydF4y2B一个, 238(2019)。gydF4y2B一个
Danecek, P.等人。变量调用格式和VCFtools。gydF4y2B一个生物信息学gydF4y2B一个27gydF4y2B一个, 2156-2158(2011)。gydF4y2B一个
Cingolani, P.等人。注释和预测单核苷酸多态性的影响的程序,SnpEff: snpin基因组gydF4y2B一个黑腹果蝇gydF4y2B一个应变gydF4y2B一个wgydF4y2B一个1118gydF4y2B一个;gydF4y2B一个isogydF4y2B一个2;gydF4y2B一个isogydF4y2B一个3所示。gydF4y2B一个飞gydF4y2B一个6gydF4y2B一个, 80-92(2012)。gydF4y2B一个
劳施,T.等人。DELLY:通过综合配对端和分裂读分析发现结构变异。gydF4y2B一个生物信息学gydF4y2B一个28gydF4y2B一个, 333-339(2012)。gydF4y2B一个
Danecek, P. & McCarthy, s.a. BCFtools/csq:单倍型感知变异后果。gydF4y2B一个生物信息学gydF4y2B一个33gydF4y2B一个, 2037-2039(2017)。gydF4y2B一个
Price, M. N. Dehal, P. S. & Arkin, a . P. FastTree:用剖面代替距离矩阵计算大型最小进化树。gydF4y2B一个摩尔。杂志。另一个星球。gydF4y2B一个26gydF4y2B一个, 1641-1650(2009)。gydF4y2B一个
Alexander, D. H., Novembre, J. & Lange, K.基于模型的不相关个体祖先估计。gydF4y2B一个基因组Res。gydF4y2B一个19gydF4y2B一个, 1655-1664(2009)。gydF4y2B一个
Purcell, S.等人。PLINK:全基因组关联和基于人群的连锁分析工具集。gydF4y2B一个点。j .的嗡嗡声。麝猫。gydF4y2B一个81gydF4y2B一个, 559-575(2007)。gydF4y2B一个
张春,董松松,徐建勇,何文明,杨铁林。基于可变调用格式文件的链不平衡衰减分析方法。gydF4y2B一个生物信息学gydF4y2B一个35gydF4y2B一个, 1776 - 1788(2019)。gydF4y2B一个
Quinlan, a.r. & Hall, i.m. BEDTools:一套用于比较基因组特征的灵活工具。gydF4y2B一个生物信息学gydF4y2B一个26gydF4y2B一个, 841-842(2010)。gydF4y2B一个
Bradbury, P. J.等。TASSEL:用于不同样本中复杂性状关联映射的软件。gydF4y2B一个生物信息学gydF4y2B一个23gydF4y2B一个, 2633-2635(2007)。gydF4y2B一个
Broman, kw, Wu, H., Sen, S. & Churchill, G. A. R/qtl:实验杂交的qtl定位。gydF4y2B一个生物信息学gydF4y2B一个19gydF4y2B一个, 889-890(2003)。gydF4y2B一个
刚达,我,等等。基于测序的番茄映射群体bin map构建,便于高分辨率定量性状位点检测。gydF4y2B一个植物基因组gydF4y2B一个12gydF4y2B一个, 180010(2019)。gydF4y2B一个
邱吉尔,g.a.和多吉,r.w.定量特征映射的经验阈值。gydF4y2B一个遗传学gydF4y2B一个138gydF4y2B一个, 963-971(1994)。gydF4y2B一个
Ouellette, L. A., Reid, R. W., Blanchard, S. G. & Brouwer, C. R. linkagemapview -渲染高分辨率链接和QTL地图。gydF4y2B一个生物信息学gydF4y2B一个34gydF4y2B一个, 306-307(2018)。gydF4y2B一个
Li, D., Liu, c.m, Luo, R., Sadakane, K. & Lam, t.w. MEGAHIT:通过简洁的de Bruijn图进行大型和复杂宏基因组组装的超快速单节点解决方案。gydF4y2B一个生物信息学gydF4y2B一个31gydF4y2B一个, 1674-1676(2015)。gydF4y2B一个
齐敏,A. V.等。MaSuRCA基因组汇编器。gydF4y2B一个生物信息学gydF4y2B一个29gydF4y2B一个, 2669-2677(2013)。gydF4y2B一个
傅丽玲,牛斌,朱智,吴松,李伟。CD-HIT:用于下一代测序数据聚类的加速技术。gydF4y2B一个生物信息学gydF4y2B一个28gydF4y2B一个, 3150-3152(2012)。gydF4y2B一个
Alonge, M.等人。RaGOO:快速和准确的参考指导草图基因组搭建。gydF4y2B一个基因组医学杂志。gydF4y2B一个20.gydF4y2B一个, 224(2019)。gydF4y2B一个
Glick, L. & Mayrose, I.全景:构建真核泛基因组的软件包。gydF4y2B一个摩尔。生态。Resour。gydF4y2B一个21gydF4y2B一个, 1393-1403(2021)。gydF4y2B一个
Marçais, G.等。MUMmer4:一个快速和通用的基因组比对系统。gydF4y2B一个公共科学图书馆第一版。医学杂志。gydF4y2B一个14gydF4y2B一个, e1005944(2018)。gydF4y2B一个
李海华,冯晓霞,朱晨。基于迷你图的参考泛基因组图设计与构建。gydF4y2B一个基因组医学杂志。gydF4y2B一个21gydF4y2B一个, 265(2020)。gydF4y2B一个
李,H.等。序列对齐/映射格式和SAMtools。gydF4y2B一个生物信息学gydF4y2B一个25gydF4y2B一个, 2078-2079(2009)。gydF4y2B一个
佩奇,A. J.等人。Roary:快速大规模原核生物泛基因组分析。gydF4y2B一个生物信息学gydF4y2B一个31gydF4y2B一个, 3691-3693(2015)。gydF4y2B一个
Huerta-Cepas, J. et al. eggNOG 5.0:基于5090种生物和2502种病毒的分层、功能和系统发育注释的orthology资源。gydF4y2B一个核酸测定。gydF4y2B一个47gydF4y2B一个, d309-d314(2019)。gydF4y2B一个
田,T.等人agriGO v2.0:农业社区GO分析工具包,2017年更新。gydF4y2B一个核酸测定。gydF4y2B一个45gydF4y2B一个, w122-w129(2017)。gydF4y2B一个
陈,C.等。TBtools:为大生物数据的交互式分析而开发的集成工具包。gydF4y2B一个摩尔。植物gydF4y2B一个13gydF4y2B一个, 1194-1202(2020)。gydF4y2B一个
Benjamini, Y. & Hochberg, Y.控制错误发现率:一个实用而强大的方法来多个假设检验。gydF4y2B一个J. R.统计社BgydF4y2B一个57gydF4y2B一个, 289-300(1995)。gydF4y2B一个
高淑华。自定义脚本在豌豆ZW6基因组和泛基因组计划中的应用。gydF4y2B一个ZenodogydF4y2B一个https://doi.org/10.5281/zenodo.6614849gydF4y2B一个(2022)。gydF4y2B一个
确认gydF4y2B一个
感谢L. Li, B. Redden和J. Hu在样品采集和材料准备方面的支持。我们非常感谢北京生物标记技术有限公司和诺和基因生物信息研究所(中国北京)在PacBio测序、NGS、10x基因组测序和Hi-C测序方面的技术支持。我们感谢北京Grandomics Biosciences对Bionano测序的技术支持。国家重点研发计划项目(2018YFD1000701/2018YFD1000700 - T.Y.)、中国科学院青年创新促进会项目(2017140 - y.f.l)、山东省农业品种改良计划项目(2019LZGC017 - H.F.D.)、农业部中国农业研究系统项目(car -08 - X.X.Z)、国家自然科学基金项目(31801428 - R.L.)资助。山东省农业科学院科技创新项目课题团队(CXGC2018E15 to H.F.D.)、作物种质资源保护项目(2130135 to X.X.Z)、山东省农业科学院科技创新项目产业团队(CXGC2016A02 to H.F.D.)、山东省现代农业产业技术体系粗粮创新团队(SDAIT-15-01 to H.F.D.)中国农科院农业科技创新计划(aspp ~ X.X.Z)、山东省农业科学院青年科研基金(2016YQN19 ~ D.W.)、澳大利亚默多克大学食品期货研究所。gydF4y2Ba
作者信息gydF4y2B一个
作者及隶属关系gydF4y2B一个
贡献gydF4y2B一个
X.X.Z, s.h.g., H.F.D.和R.K.V.策划了该项目并设计了研究。T.Y, R.L, Y.F.L, S.N.H, S.H.G.和D.W.进行了数据分析并起草了手稿。Y.H.H Y.J.L。,X.J.W李从先和N.N.L.收集样本。Y.N.H G.L。,Y.S.J N.N.L M.W.L.和X.Y.进行实验。C.Y.W, M.K.P, s.g., Q.L.X.和R.S.协调基因组和转录组数据准备。X.X.Z, R.K.V, S.H.G.和H.F.D.修改了手稿。所有作者都阅读并批准了手稿。gydF4y2Ba
相应的作者gydF4y2B一个
道德声明gydF4y2B一个
相互竞争的利益gydF4y2B一个
作者声明没有利益竞争。gydF4y2B一个
同行评审gydF4y2B一个
同行评审信息gydF4y2B一个
自然遗传学gydF4y2B一个感谢Aureliano Bombarely, Michael Alonge和其他匿名审稿人对这项工作的同行评审所做的贡献。gydF4y2B一个
额外的信息gydF4y2B一个
出版商的注意gydF4y2B一个施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2B一个
补充信息gydF4y2B一个
权利和权限gydF4y2B一个
开放获取gydF4y2B一个本文遵循知识共享署名4.0国际许可协议(Creative Commons Attribution 4.0 International License),允许以任何媒介或格式使用、分享、改编、分发和复制,只要您对原作者和来源给予适当的署名,提供知识共享许可协议的链接,并注明是否有更改。本文中的图像或其他第三方材料包含在文章的创作共用许可中,除非在材料的信用额度中另有说明。如果内容未包含在文章的创作共用许可协议中,并且您的预期使用不被法定法规所允许或超出了允许的使用范围,您将需要直接获得版权所有者的许可。要查看此许可证的副本,请访问gydF4y2B一个http://creativecommons.org/licenses/by/4.0/gydF4y2B一个.gydF4y2B一个
关于本文gydF4y2B一个
引用本文gydF4y2B一个
杨涛,刘荣,罗勇。gydF4y2B一个et al。gydF4y2B一个改进的豌豆参考基因组和泛基因组突出了基因组特征和进化特征。gydF4y2B一个Nat麝猫gydF4y2B一个54gydF4y2B一个, 1553-1563(2022)。https://doi.org/10.1038/s41588-022-01172-2gydF4y2B一个
收到了gydF4y2B一个:gydF4y2B一个
接受gydF4y2B一个:gydF4y2B一个
发表gydF4y2B一个:gydF4y2B一个
发行日期gydF4y2B一个:gydF4y2B一个
DOIgydF4y2B一个:gydF4y2B一个https://doi.org/10.1038/s41588-022-01172-2gydF4y2B一个